摘要
背景
春小麦是哈萨克斯坦种植的最大农作物,2016年年播种面积达1200万公顷。每年优质粮食产量约1500万吨。尽管存在环境压力因素,但据预测,使用新技术可能会使产量从目前的每公顷1.5吨提高到3吨。提高小麦产量的途径之一是在植物育种项目中应用新的面向基因组的方法。全基因组关联研究(GWAS)正在成为利用高通量基因分型技术和植物采集表型评估来理解复杂性状遗传的有力工具。本研究通过对来自哈萨克斯坦、俄罗斯、欧洲和CIMMYT的194份春小麦材料的表型和基因分型数据进行分析,利用GWAS技术对农艺性状的标记-性状关联(MTA)进行鉴定。
结果
哈萨克斯坦中央和南部地区北部,中央和南部地区的田间试验使用194春小麦加入,揭示了引导日期,植物高度,生物量,每株峰值数的强烈相关性,以及每穗粒数。与本地品种的表现相比,欧洲和CIMMYT的欧洲和中部地区的育种潜力很高。使用每个植物的平均产量的GGE双粒子方法表明,清楚地分离到其三种环境中的三种育种起源中,以与其评估的三种环境。使用3245多态性SNP(单核苷酸多态性)标记进一步研究三组释放遗传变异。主要坐标分析的应用明确将194名中的一种加入194种进入育种来分为三分支。来自九个现场试验的数据上的GWA允许识别114 MTA,用于12种不同的农艺性状。
结论
国外种质在哈萨克斯坦北部的田间评价结果表明,该种质在哈萨克斯坦主要小麦产区的产量表现较差。但研究发现,EU和CIMMYT种质在提高中南部地区产量方面具有较高的育种潜力。使用主坐标分析清楚地将面板分为三个不同的组根据他们的育种来源。基于TASSEL 5.0包装的GWAS可鉴定12个农艺性状的114个mta。这项研究确定了一个关键基因网络,以提高哈萨克斯坦小麦种植区的产量。
背景
六倍体面包小麦(Triticum Aestivum.L.)是哈萨克斯坦出口的主要商品,每年种植超过1200万公顷。哈萨克斯坦的小麦种植史表明,大多数小麦品种都与俄罗斯育种者合作开发,并使用俄罗斯小麦遗传资源[1].即使在苏联分手之后,这一趋势仍然存在于该地方,因为两国在基于CIMMYT伞下的双边项目和国际活动的基础上分享了他们的专业知识和遗传资源[2,3.].研究还发现哈萨克-俄罗斯小麦种质在遗传上与美国小麦相近[4].据推测,在19世纪末俄罗斯门诺派成功将土耳其红小麦引入美国之后,俄罗斯大量进口了这种作物[5可能是哈萨克人和美国人遗传关系密切的主要原因[4].
哈萨克斯坦80%以上的小麦收获地区是春季种植的,在该国的部分地区,包括哈萨克斯坦北部和东北部,都在高纬度地区种植。其他重要的生长区域沿着天山山脉南部和东南部的部分延伸,在那里冬春两种类型都能成功生长。这些地区的气候条件变化很大,如土壤类型、生长季节的温度、降水水平和光周期长度等[6].因此,研究不同生态位的产量表现对我国小麦种植区当前和未来的育种活动具有重要意义。据估计,该国农艺的改进和新育种方法的使用可能会导致新品种的开发,从而使产量提高到每公顷3吨[7].过去,植物育种家成功地依靠传统的工具和方法。如今,新的基因组工具和资源的可用性为分析与产量提高相关的复杂性状的遗传机制带来了新的机会[8].
随着高通量基因分型成本的降低,全基因组关联研究(GWAS)正成为检测小麦农艺性状QTL (quantitative trait loci)的一种强有力的方法,最终目标是基于标记辅助选择的应用,加快本地育种活动[9,10].GWAS在小麦中的成功主要基于Affymetrix的高密度SNP基因分型平台的发展[11,12]和illumina [9,13],这是现在的高通量基因分型小麦多样性面板数据提供了丰富的资源。在GWAS,显著关联的检测主要依赖于遗传标记覆盖,个体的研究和连锁的多态性的数目,和致病之间的连锁不平衡(LD)[14,15].目前,GWAS已成功应用于六倍体小麦的产量构成QTL鉴定[16,17,18,非生物抗压力[19,20.,21]疾病抵抗[22,23],和谷物质量[24,25].一项对文献的调查表明,GWAS被积极应用于世界许多不同地区的小麦研究,包括北美[26],中美洲[25],欧洲[18,24],非洲[19),澳大利亚(22],以及亚洲[20.].
尽管GWAS已经被证明是捕获重要标记-性状关联(MTA)的非常有效的方法,来自世界不同地区的研究结果表明,生长环境对产量有很强的影响,其中产量QTL具有显著的基因型x环境互作揭示(GEI)。例如,在欧洲进行的三项不同的GWAS研究中,获得了与产量表现QTL鉴定相关的结果[24)、印度(27],以及墨西哥[16]表明不同的响应,QTL在基因组的不同部分的产量组件。这一趋势也证实了在研究时,同一种质亚洲[不同地区进行测试20.].这一结果与Quarrie等人(2005)在使用双亲本制图群体的研究中报告的结果一致[28]并且可以通过对关键生长阶段的环境因子的敏感性来解释,该阶段决定每只耳朵的潜在颗粒数[29].因此,区域项目的成功可能在很大程度上取决于使用基因型适应性种质的独立的、地方的GWAS试验。这项工作的主要目标是GWAS利用来自哈萨克斯坦、俄罗斯、欧洲和CIMMYT(墨西哥)的春小麦材料,在哈萨克斯坦三个不同环境的田间试验中鉴定MTA。该研究是利用GWAS技术鉴定春小麦重要QTL和加强春小麦育种项目的首次尝试。
方法
春小麦小组包括来自哈萨克斯坦和俄罗斯联邦的96个商业和预期品种,来自欧洲的38个品种和60个CIMCOG (CIMMYT墨西哥核心种质)系(CIMMYT,墨西哥)(补充文件)1).目前,来自哈萨克斯坦和俄罗斯的61个品种已通过哈萨克斯坦共和国国家种子试验委员会(2015年)注册,并在哈萨克斯坦正式种植。该小组还包括在哈萨克斯坦和俄罗斯开发的29个潜在品种(附加文件)1).欧洲品种系列主要包括源自英国的追加。Cimcog线是为研究光合作用和生物量改进的机会而开发的特殊人口[30.].田间试验在哈萨克斯坦的三个不同纬度地区进行(补充文件)2),特别是在Karabalyk配种站(北哈萨克斯坦),卡拉干达育种研究所(哈萨克斯坦中部),并在哈水稻研究所(南哈萨克斯坦)。收集在随机实验种植在每个站点各三次重复在2013-2015季节。行之间的距离为15cm和行内的植物之间的距离为5厘米。在北和南区中的实验1分块进行的,而在南部地区的种质种植在每个重复3行。总体上,对于在九个环境收获194份六倍体小麦种质的12个农艺性状的平均值的数据进行进一步的统计分析。12个性状包括如下:天引导(BD),天标题时间(HT),天至成熟(MT),热时间标题(TT-H),热时间到期时(TT-M),植物高度(PH),花序梗长度(PL),沃尖峰(NFS)的数目,每穗(NKS),千粒重(TKW),每株植物的生物量干重(BPP)和每株植物(YPP)收率粒数。
DNA样品中提取并使用商业试剂盒(Qiagene,CA,USA)个别品种的单粒种子进行纯化。每个样品的DNA浓度调节至50ng /μl。使用小麦90ķIllumina的I选择SNP阵列如[描述种质进行基因分型4].
数据的统计分析,包括多因素Anova,Pearson的相关性和t检验使用软件包GraphPad Prism 5.0计算计算[31].使用GenStat软件包(第17版,VSN国际公司,赫特福德郡,英国)使用GGE Biplot方法。两种方法的对称比例选择和所有三个站点的可用现场数据都用于估计。
利用TASSEL 5.0软件包对194份供试材料生育期和产量参数的QTL进行了GWAS分析[32].为此,SNP数据集对缺失数据使用10%的截止值进行过滤,GWAS只考虑小等位基因频率≥0.10的标记。结构及结构收割机[29]程序用于确定确定的聚类的delta K值(ΔK)和q矩阵。
结果
哈萨克斯坦三个地区春小麦采集的田间表现
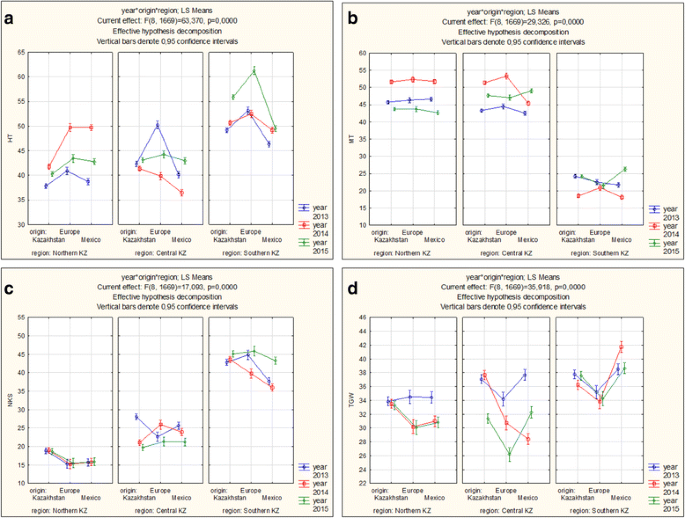
关于194年春小麦地区的现场性能数据(欧盟),CIMMYT(CIMCOG线)以及来自哈萨克斯坦和俄罗斯联邦的线条(附加档案1)在2013-2015年的哈萨克斯坦北部、中部和南部地区的实验站进行了分析。从3个育种来源收集的植物生长阶段的长度和产量构成的方式在区域间有显著差异(图2)。1).与哈萨克斯坦南部相比,北部和中部地区的三个地区的样品的开花时间都更早(图2)。1),成熟时间长度完全相同(图。1 b).产量组分,包括每个尖峰(NKS)的粒子数量在南部繁殖站中总是更高的(图。1 c).三个组的平均YPP(2013-2015)在三个地区之间没有相关性,除了南部地区的CIMCOG品种,该品种与中部地区的本地和欧盟品种显著相关(附加文件)3.).在哈萨克斯坦北部,这是该国最重要的小麦生长地区,在研究中,产量最低。Pearson的相关测试表明,哈萨克斯坦北部的ypp领先的导致因素是BD,pH,BPP,NFS和NKS(附加文件4).在哈萨克斯坦北部、中部和南部共测量了10个不同的植物生长阶段,包括TT-H和TT-M日期。Pearson相关指数表明,在这三个地区,TT-H和TT-M对产量构成因素,包括每穗粒数(NKS)和千粒重(TKW)均有极显著影响(附加文件)4).
本研究中TT-H和TT-M这两个发育阶段在不同育种来源间存在差异(表中O效应)1和图1),并在九个环境(O X R X Y)和生长场所(O X R)中显示出与繁殖起源的显着相互作用。
主要影响的收率的幅度(年,地区和产地),以及它们之间的相互作用进行了排名区>产地>年份> Y X R> Y X○×R>○×R,其效果至少为Y×○,由F-值所指示的(表1).唯一在区域效应上较大的唯一情况是从pH接收的结果,其中原点效果的F值比区域效应高1.7倍。
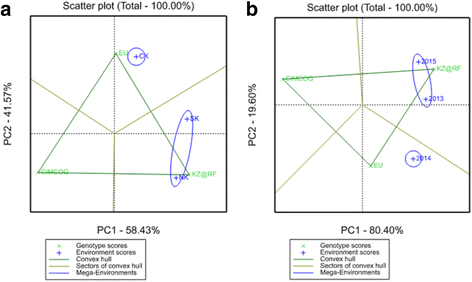
GGE双图分析基于9种环境下的产量表现,根据地区和年份的影响将不同的育种来源分组(图1)。2).在区域效应分析中,双曲线图表明,哈萨克斯坦南部和北部更适合哈萨克斯坦和俄罗斯联邦的入盟,而哈萨克斯坦中部更适合欧洲的入盟(图2)。2).在年效应分析中,来自哈萨克斯坦和俄罗斯联邦的小麦品种组与2013年和2015年的环境相匹配,而2014年对来自欧洲的小麦品种更有利(图2)。2 b).虽然在两次扫描中,三组不同育种来源的材料被很好地分离,但环境效应的分离是不同的。在区域效应下,两个主坐标都能有效区分两个超级环境,而在年效应下,两个超级环境的分离主要基于第二个主坐标(PC2)。
基于SNP数据的不同育种起源的春小麦遗传变异
来自哈萨克斯坦,俄罗斯和西欧的194年春小麦加入均使用含有81,587个SNP的90 K SNP Iselect阵列进行基因分型。基因分型允许生成66,223可被批量的SNP和Blastn搜索允许识别IWGSC的染色体调查测序项目中的55,165个SNP命中[33允许标记物在染色体上的位置。CIMCOG细胞系的基因分型数据可在[34],并通过使用35ķAxiom®阵列[生成12] [32].从这些数据中,194次春小麦加入,包括60条CIMCOG线之间的遗传变异性,是根据3245个多态性SNP标记进行了研究。三组加入,由此最小数据集是用于CIMCOG组(3852个SNP标记)的对准之后多态型标志的数目减少。在SNP图谱所有小麦染色体的总长度为3109.9 cM的。平均SNP密度为0.96 SNP / cM和在B基因组在基因组d从0.68到不等2.31。Nei氏无偏多样性指数的三组在哈萨克斯坦和俄罗斯种质的混合组从CIMCOG线0247变化到0339种质的范围内(表2).基因组A、B和D的多样性指数分别为0.286、0.284和0.269。D基因组的LD最大(r20.1)嵌段(26.8cm),然后是基因组(17.5cm)和B基因组(14.0cm)基因组(附加文件5).
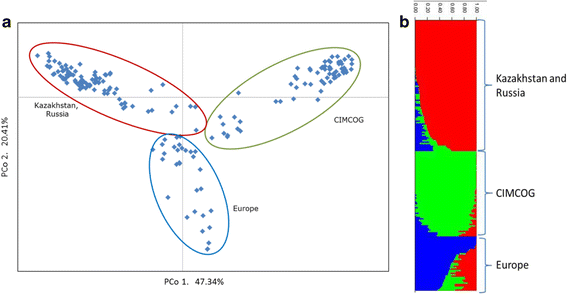
主坐标分析显示,根据其育种来源,将供试材料分为三个不同的亚群(图。3.).第一个坐标(47.34%)清晰地将哈萨克斯坦和俄罗斯样本从CIMCOG线中分离出来,第二个坐标(20.41%)将欧洲样本从其他两组中分离出来(图2)。3).这一结果与结构分析的结果一致,其中三种疏水物的含量根据其繁殖源分离(图。3 b).
基于GWAS的生育期和产量构成的SNP标记鉴定
在本研究中,194份春小麦材料和3245个多态SNP标记组成了基因分型的HapMap文件。对缺失数据和小等位基因频率≥0.10的标记,以10%的截断阈值进行筛选,得到多态SNP数据集。运行Structure Harvester后,通过对delta K值的分析(ΔK),对识别出的三个聚类选择q -矩阵进行进一步分析。对哈萨克斯坦北部、中部和南部的9个研究环境分别进行基因分型分析。
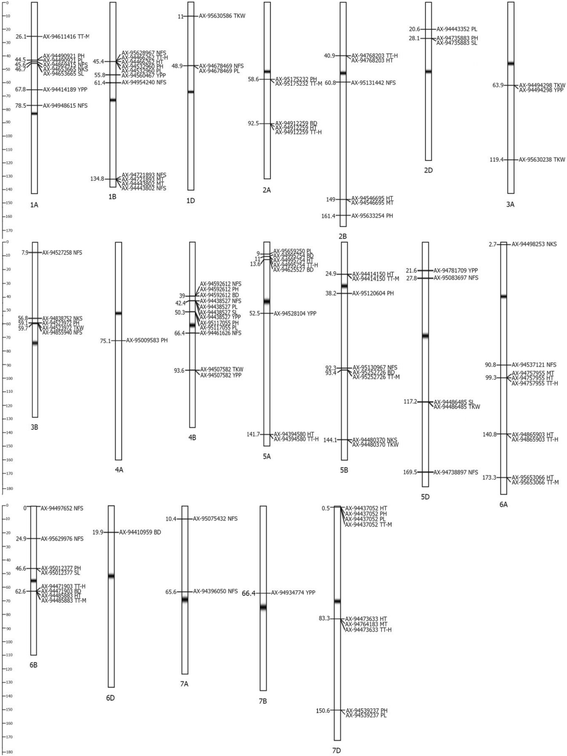
在小麦基因组的19条染色体上共鉴定出114个mta(图。4。,附加文件6)在染色体4B(12MTA)上检测到最大数量的相关标记物。南部,中心和南部地区识别的MTA的曼哈顿和QQ(Quintile - 五分之一)绘制的额外文件7,8,9,分别。与生育期有关的性状鉴定出46个mta,形态性状和产量构成性状鉴定出68个mta,其中6个标记对两组性状均有显著性6).十二个QTL在区域内的两个环境位点同时确定。只有两个PL的MTA映射到1B和7D染色体(AX-94532960和AX-94539237)是为地区的环保网站显著。其中的MTA的农艺性状,21个协会还确定了NFS,四为NKS,以及七项TGW(附加文件6).
讨论
为促进mta的发现,对三组不同育种来源的小麦材料进行了研究。在遗传方面,遗传资源被清楚地分为三个亚组,在分析中,第一亚组(俄罗斯/哈萨克斯坦)是最多样化的,第三亚组(CIMCOG)是最不多样化的组。利用structure软件对种群结构进行分析,结果表明,将种群划分为三个亚群是一致的。
哈萨克斯坦北部3年的产量表现最低。南方地区的产量表现一直较高。然而,该地区的耕地面积有限,与哈萨克斯坦北部的小麦种植区相比微不足道。结果表明,EU收集的品种可以成功地用于中部地区获得更好的产量,而选定的CIMCOG品种可以有效地用于所有三个地区的TKW的改进。基于产量数据的GGE双标图有助于确认三个育种来源内的群体具有不同的遗传背景,并且本地种质很好地适应于北方和南方地区。
在分析中掺入CIMCOG线导致GWA可用的多态SNP标记的减少,因为这些线的基因分型数据仅限于分析与Illumina SNP阵列之间共用的那些。因此,在GWA中仅使用3245个对准的多晶型SNP,其中1340个SNP定位在B基因组中的1448中,在D基因组中457。A和B基因组的多样性指数相对较高,D基因组中较低,这与先前的观察结果很好[10,18].GWAS在3年的时间里分别进行了9次现场试验,确定了114次mta(附加文件)6).在区域内和区域之间的两个环境中,只有12个MTA是显著的(附加文件6),表明这些协会的稳定性受到环境因素的强烈影响。
将本研究中检测到的mta与其他出版物中报道的mta进行比较,发现有许多相似之处。例如,Rht-D1已知基因位于4b染色体上的31.5cm [35],接近AX-94592612 (39.0 cM),本研究结果与株高、孕穗期和可育穗数有关。其他相似的例子是mta在花序梗长(20.6 cM)、株高和穗长(28.1 cM)的位置3.)的位置Rht8染色体2D (23.0 cM)上的基因[36].本研究中6A染色体(2.7 cM)上NKS的MTA定位与Guo等人的研究相似[24].4B染色体上PH的mta (AX-94592612和AX-95117055)与Rht-B1使用在哈萨克斯坦东南部进行测试的双重父母映射群体鉴定的植物高度的基因[28].在Quarrie等人的同一研究中。[28]、NKS (1A染色体)、TKW (1D、3A、4B染色体)和NFS (1A、4B、5D、7A染色体)的QTL在本研究发现的NKS、TKW和NFS的mta的相似位置(表3.).
尽管在与其他研究中鉴定的MTA的遗传态势中存在相似之处,但在不同地理区域进行的GWA中缺少该研究的许多MTA。本研究揭示了与NKS相关的四种MTA,它是小麦的主要产量组分之一[28].文献调查表明,在3B (56.9 cM)和5B (144.1 cM)染色体上发现的位点在世界其他地区的GWAS研究中未发现[17,25,28也没有Quarrie等人[28].因此,我们假设这些mta以及表中所示的其他关联3.是新的mta。
最近,有一些讨论涉及到多样性面板中遗传变异的大小和水平对GWAS项目成功的重要性[14,15,37].有人指出小于384份的实验[14]及大型楼宇[15]可能导致识别假阳性关联。Turner等人(2016)的研究表明,较小的面板可能允许检测在较大的面板中无法检测到的假阴性关联[37].本研究使用相对较小的面板(n = 194) are largely in agreement with the study by Turner et al. [37].
结论
该研究证实了GWAS对标记重要农艺性状的分子标记的有效性。利用哈萨克斯坦、俄罗斯、欧盟和CIMMYT春小麦样品测定了12个生理农艺性状的114个mta,在哈萨克斯坦3个地区的田间条件下进行了研究。株高mta位点与相应的遗传位点相似rht-b1,rht-d1,和Rht8基因的小麦。此外,从田间试验发现,EU和CIMMYT种质在提高我国中南部地区产量方面具有很高的育种潜力。使用主坐标分析清楚地将面板分为三个不同的组根据他们的育种来源。这项研究确定了一个关键基因网络,将进一步验证在哈萨克斯坦小麦种植区提高产量。
参考文献
- 1.
Martynov SP, Dobrovotvorskaya TV, Morgounov AI, Urazaliev RA, abattarova AS。哈萨克斯坦1929-2004年春面包小麦品种多样性的家谱分析。作物学报。2005;53:261-72。
- 2.
关键词:小麦,铁和锌粒密度,小麦,中铁和锌粒密度Euphytica。2007;155:193 - 203。
- 3.
Gomez-Becerra高频,Abugalieva, Morgounov Abdullaev K, Bekenova L, Yessimbekova M, Sereda G, Shpigun年代,Tsygankov V, Zelenskiy Y,佩纳RH, Cakmak表型的相关性,GЈE交互和广义遗传分析的谷物和面粉质量特征在高纬度春天面包小麦从哈萨克斯坦和西伯利亚。Euphytica。2010;171:23-38。
- 4.
基于小麦90k单核苷酸多态性阵列的哈萨克斯坦小麦品种系统发育分析植物遗传资源。2015;doi:10.1017 / S1479262115000325.
- 5.
Olmstead A,Rhode P.红色女王和硬度:美国小麦的生产力增长,1800-1940。j econ stay。2002; 62(4):929-66。
- 6。
哈萨克斯坦不同地区春大麦产量基因型x环境互作模式研究遗传学报。2013;49(2):224-34。
- 7。
Bruinsma J.“资源前景至2050年:通过2050年的土地,水和作物收益率增加了多少?”在粮农组织专家会议上“如何在2050年养活世界,粮农组织,罗马,意大利,意大利,2009年6月。第1-1-32号。
- 8。
Pérez-de-Castro Am,Vilanova S,CañizaresJ,等。基因组工具在植物育种中的应用。目前的基因组学。2012; 13(3):179-95。
- 9.
Cericola楼Jahoor A,OrabiĴ,安德森JR,Janss LL,詹森J.优化训练群体的大小和使用的关联的研究结果和系谱信息的基因组基因分型预测策略。学习先进的小麦育种线的情况。Plos一个。2017; 12(1):e0169606。
- 10.
Lopes MS, Dreisigacker S, Peña RJ, Sukumaran S, Reynolds MP。小麦关联定位计划(WAMI)面板用于春小麦复杂性状分析的遗传特征。Theor Appl Genet. 2015; 128:453-64。
- 11.
崔F,张n,风扇x,张w,zhao ch,阳lj,潘雷克,陈m,韩俊,赵xq,ji j,童捷,张hx,jiajz,赵gy,李杰。利用小麦660K SNP阵列衍生的高密度遗传图谱,用于高分辨率映射的主要QTL,用于内核数量。SCI批准。2017; 7(1):3788。
- 12.
Allen AM, Winfield MO, Burridge AJ, Downie RC, Benbow HR, Barker GL, Wilkinson PA, Coghill J, Waterfall C, Davassi A, Scopes G, Pirani A, Webster T, Brew F, Bloor C, Griffiths S, Bentley AR, Alda M, Jack P, Phillips AL, Edwards KJ。适用于全球六倍体面包小麦高通量SNP基因分型的小麦育种家阵列的特性(Triticum Aestivum.).生物技术学报2017;15(3):390-401。
- 13.
王,王D,福勒斯特K,艾伦,曹国伟年代,黄,Maccaferri M, Salvi年代,米尔纳SG, Cattivelli L, Mastrangelo,星期几,巴克G, Stephen S Wieseke R, Plieske J,国际小麦基因组测序协作组,Lillemo M,马瑟D, appel R, Dolferus R, Brown-Guedira G,各地,Akhunova AR, Feuillet C,泥火山J, Morgante M, Pozniak C,利用高密度90000单核苷酸多态性阵列研究多倍体小麦基因组多样性。中国生物技术学报(英文版);
- 14.
张志刚,张志刚,张志刚,张志刚。关联定位:从基因分型到实验设计的关键考量。植物细胞。2009;21(8):2194 - 202。
- 15.
Zegeye H, Rasheed A, Makdis F, Badebo A, Ogbonnaya FC。合成六倍体小麦幼苗和成株抗条锈病的全基因组关联作图。《公共科学图书馆•综合》。2014;9 (8):e105593。
- 16.
Sukumaran S,Dreisigacker S,洛佩斯男,查韦斯P,雷诺MP。粮食产量和相关性状在温带生长的精英春小麦人口全基因组关联分析灌溉环境。TheorAppl遗传学。2015; 128:353-63。
- 17。
马米迪,马文杰,熊敏。全基因组关联研究揭示了与春小麦多斑病抗性相关的新的数量性状位点。《公共科学图书馆•综合》。2014;9 (9):e108179。
- 18。
Zanke C, Ling J, Plieske J, Kollers S, Ebmeyer E, Korzun V, Argillier O, Stiewe G, Hinze M, Beier S, Ganal MW, Röder MS.欧洲冬小麦抽穗期主效QTL的遗传结构。植物科学学报,2014;(5):217。doi:10.3389 / fpls.2014.00217.
- 19。
门格斯图DK,基达内YG,Catellani男,Frascaroli E,Fadda C,PE ME,在黑人硬粒小麦地方品种Dell'Acqua的M.高密度分子表征和关联作图揭示了小麦育种高多样性和潜力。植物生物技术J. 2016; 14:1800至12年。
- 20.
OYIGA BC,Sharma RC,Baum M,Ogbonnaya Fc,LéonJ,Ballvora A.在小麦盐胁迫耐受性耐盐耐受性的定量特质位置检测到等位基因变化和差异表达。植物,细胞和环境。2017; DOI:10.1111 / pce.12898.
- 21.
陈志强,王志强,王志强,等。高温胁迫下不同基因型小麦穗乙烯的表型和全基因组关联分析。新植醇。2017;214:271 - 83。
- 22.
Jighly A, Alagu M, Makdis F, Singh M, Singh S, embiri LC, OgbonnayaFC。合成六倍体小麦抗多种真菌病原体的基因组区域。摩尔繁殖。2016;36:127。
- 23.
Naruoka Y,·Garland-Campbell Ka,卡特啊。用于条纹锈病(PUCCINIA Striformis F. sp.tritici)的基因组 - 宽协会映射在美国太平洋西北冬小麦(Triticum Aestivum L.)。Theorappl Genet.2015; 128:1083-1101。
- 24.
郭志,陈丹,Alqudah AM, MS. Rӧder3, Ganal MW, Schnurbusch T.小麦小花育性54个性状的多位点关联分析。新植醇。2017;214:257 - 70。
- 25.
Arora S,Singh N,Kaur S,Bains NS,Uauy C,Poland J,Chhuneja P. Genome-Interive野外谷物建筑的基因组协会研究Aegilops Tauschii..前面。植物科学。2017;doi:10.3389 / fpls.2017.00886.
- 26.
林敏,张东,刘旭国,于军,Fritz AK,白光。美国冬小麦采前发芽抗性与籽粒颜色的全基因组关联分析。BMC基因组学。2016;17:794。
- 27.
小麦部分重要农艺性状的全基因组单位点、单性状、多位点和多性状关联作图(t . aestivumL.)Plos一个。2016. DOI:10.1371 / journal.pone.0159343
- 28.
Quarrie SA, Steed A, Calestani C, Semikhodskii A, Lebreton C, Chinoy C, Steele N, Pljevljakusic D, Waterman E, Weyen J, Schondelmaier J, Habash DZ, Farmer P, Saker L, Clarkson DT, Abugalieva A, Yessimbekova M, Turuspekov Y, Abugalieva S, Tuberosa R, Sanguineti MC, Hollington PA, Aragues R, Royo A,D.六倍体小麦的高密度遗传图谱(Triticum Aestivum.中国春季× SQ1杂交组合及其在一系列环境下比较籽粒产量qtl的应用。Theor Appl Genet. 2005; 110:865-80。
- 29。
Reynolds MP,Trethowan R,Crossa J,Vargas M,Sayre Kd。小麦间环型与基因型相关的生理因素。现场作物RES。2002; 75:139-60。
- 30.
Gonzales-Navarro O,Griffiths S,Molero G,Reynolds M,Slafer G.精英小麦系列中发育模式的变化以及与产量,产量组分和尖峰生育的关系。现场作物RES。2016; 196:294-304。
- 31.
格拉夫派得软件公司http://www.graphpad.com/faq/viewfaq.cfm?faq=1362.2017年7月7日生效。
- 32.
张志,kon DE, Casstevens TM, Ramdoss Y, Buckler ES。TASSEL:用于不同样本中复杂性状关联制图的软件。生物信息学。2007;23:2633-5。
- 33.
国际小麦基因组测序联盟(IWGSC)。六倍体面包小麦(Triticum Aestivum.)基因组。科学。2014;345(6194):1251788。
- 34.
威尔金森PA,温菲尔德MO,巴克GLA,艾伦AM,布里奇A,科格希尔JA,布里奇A,爱德华兹KJ。谷物DB 2.0:用于植物育种家和科学家的综合资源。BMC生物信息学。2012; 13:219。
- 35.
面包小麦的高密度微卫星共识图(Triticum Aestivum.l .)。Theor Appl Genet. 2004; 109:1105-14。
- 36.
矮化基因的遗传分析(Rht8在小麦。第一部分分子定位Rht8在面包小麦2D染色体短臂上(Triticum Aestivum.l .)。Theor Appl Genet. 1998; 96:1104-9。
- 37.
Turner MK, Kolmer JA, Pumphrey MO, Bulli P, Chao S, Anderson JA。春小麦核心种质抗叶锈病基因座的关联作图。Theor Appl Genet. 2016;doi10.1007 / s00122 - 016 - 2815 - y.
确认
这项工作得到了欧盟第7框架项目ADAPTAWHEAT项目和哈萨克斯坦共和国教育和科学部1784/GF4项目的资助。
资金
本文的发表得到了哈萨克斯坦共和国教育和科学部1784/GF4项目的资助。
可用性数据和材料
支持这篇文章的结论的数据集包括在项目之内。
关于这个补充剂
本文已作为BMC植物生物学2017年第17卷增编1:PlantGen 2017节选文章。该补充的全部内容可在网上找到//www.cinefiend.com/articles/supplements/volume-17-supplement-1.
作者信息
从属关系
贡献
YT和SA进行了实验设计。LT,VC和GS在哈萨克斯坦的三个不同地区进行了现场试验,MG和SG提供了SNP基因分型数据,YT,AB和KY进行了GWAS分析。YT,SA和SG参加了准备稿件。所有作者审查了稿件的草案。所有作者均阅读并批准最终手稿。
相应的作者
道德声明
伦理批准和同意参与
本研究不包含任何需要伦理同意或批准的研究。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
额外的文件1:
研究分析了哈萨克斯坦、俄罗斯、欧洲和墨西哥的加入名单。(XLS 43 kb)
附加文件2:
哈萨克斯坦三个繁殖地的位置和气候数据。(DOC 44 kb)
额外的文件3:
各试验点3年平均YPP与种质起源的相关性。(XLS 27 kb)
附加文件4:
2013-2015年哈萨克斯坦北部地区性状间的Pearson相关分析(XLS 70 kb)
附加文件5:
LD衰减线(阈值r2基于3245个SNP标记的A、B、D基因组和全基因组。答:一个基因组;B B基因组;c D基因组;d .整个基因组。(医生294 kb)
附加文件6:
MTA的列表中标识出基于使用TASSEL 5.0 3245个SNP标记。(XLS 61 KB)
附加文件7:
曼哈顿和QQ图基于使用Tassel 5.0包分析来自北哈萨克斯坦的场绘图数据的分析。(XLS 6271 KB)
额外的文件8:
利用TASSEL 5.0软件包,在分析哈萨克斯坦中心田间小区数据的基础上,分析了曼哈顿和QQ小区。(XLS 11565 kb)
附加文件9:
利用TASSEL 5.0软件包对哈萨克斯坦南部的田间小区数据进行分析,得到曼哈顿和QQ小区。(XLS 10628 kb)
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
图鲁斯佩科夫,拜布拉托娃,叶尔梅克巴耶夫,K。等等。春小麦各生育期及产量构成因素的GWAS (Triticum Aestivum.L.)在哈萨克斯坦的三个地区收获。BMC植物杂志17,190(2017)。https://doi.org/10.1186/s12870-017-1131-2
关键词
- 基因组协会研究(GWAS)
- Marker-trait协会(放在)
- 原创繁殖
- 高繁殖潜力
- 春小麦加入