摘要
背景
半纤维素是一组复杂的非纤维素多糖,约占植物细胞壁的三分之一,可用作膳食纤维、食品添加剂和生物燃料的原料。参与半纤维素合成的基因尚未在小粒谷物中得到广泛研究。
结果
为了分离出基因序列纤维素synthase-like(Csl)基因家族,共鉴定出108个基因(以下简称TaCsl).每个基因由2到3个同等位基因代表,它们被命名为TaCslXY_ZA,TaCslXY_ZB,或TaCslXY_ZD式中,X表示Csl亚家族,Y是基因数目,Z是它所在的小麦染色体。这些基因中有四分之一被预测有2到3个剪接变体,导致总共137个假定的翻译产物。大约45%TaCsl基因位于2号和3号染色体上。C亚科和D亚科的序列分布在双科植物和禾本科植物之间,而A亚科的序列则集中在每一类植物中。双科特有的B亚科和G亚科分别与草特有的H亚科和J亚科接近,表明它们有共同的起源。在不同组织中的表达分析表明,大部分基因是普遍表达的,有些基因是组织特异性的。超过一半的基因在第0阶段含有内含子,三分之一在第2阶段,少数在第1阶段。
结论
小麦的详细特征Csl基因增强了对其结构、功能和进化特征的理解。这些信息将有助于设计半纤维素合成的遗传操作实验,以开发用于生物燃料生产的改良品种和提高对各种胁迫的耐受性。
背景
植物细胞壁由三种主要的多糖组分组成:纤维素、半纤维素和果胶,另外两种组分为木质素和蛋白质。草壁主要含有三种多糖组分中的两种,果胶是相当次要的成分。半纤维素是植物细胞壁基质多糖,具有不同的线性或分支结构[1,2]。这些主要包括草中的1 - 4-β-葡聚糖、1,3;1,4-β-葡聚糖、半乳聚糖和葡甘露聚糖[3.]。此外,葡萄糖醛酸阿拉伯木聚糖是草壁的主要成分。由于在其聚合物骨架上存在异相取代基或其他键,半纤维素是非结晶的,与纤维素相比容易水解。这些多糖可以通过氢键与纤维素微原纤维相互作用[4]。
植物骨架中的半纤维素多糖是由纤维素类合酶(Csl)酶,它们是一个更大的基因超家族的成员,被称为糖基转移酶2(GT2) [5]。其他几个GTs,也就是说,xyloglucanα1,6-xylosyltransferases(GT34),xyloglucan focusyltransferases(GT37),xyloglucan半乳糖基转移酶(GT47)参与木葡聚糖的生物合成[6]。编码Csl酶的基因与dna序列相似纤维素合酶A(中国极限运动协会)在整个植物界形成纤维素的基因家族[7]。数目不定的Csl据报道,在不同的植物物种中有30至50个基因,可分为9个亚科(里昂证券- - - - - -CslH和CslJ) [8,9]。谷物普遍缺乏CslB和CslG家庭。在剩下的家庭中,里昂证券,CslC,CslD在所有陆生植物中均有保存,而该论坛,CslH仅限于草类[10,11]。一个鲜为人知的亚家族,CslJ,在禾本科和菊科植物中都有报道,这与之前只在禾本科中出现的说法形成了对比[12,13]。同样,亚科CslB和CslG以前的报道是针对dicots的[14]。然而,最近的一份报告证实了CslB单子叶的亚科[12]。有几个Csl据报道,亚家族参与不同细胞壁多糖的生物合成。例如,subfamily里昂证券形成半乳甘露聚糖和葡甘露聚糖的β-1,4-甘露聚糖骨架[15,16]。同样的,该论坛和CslH亚家族在草中产生1-3;1-4 -β-葡聚糖[17,18),而CslC基因与木葡聚糖和其他一些多糖的1-4 -β-葡聚糖主链的形成有关[19]。
小麦是世界上耕地面积最大的主要谷类作物,在粮食产量上仅次于玉米,养活了世界约40%的人口[20.]。它具有大的基因组大小(~ 17gb),其中~ 80-90%是重复的[21]。即使在获得完整的基因组序列之后[22],Csl面包小麦的基因仍未被识别和鉴定。一般来说,大多数基因的同源拷贝位于属于每个亚基因组(A, B和D)的三条染色体上,这表明Csl基因预计大约是水稻等二倍体物种的三倍。我们利用公开资源检索小麦基因组序列。利用Pfam域模型进行了大规模的数据挖掘Csl基因家族成员,在这项研究中有报道。
方法
数据源和序列检索
小麦基因组数据从Ensembl Plants FTP服务器(ftp://ftp.ensemblgenomes.org/pub/current/plants/fasta/triticum_aestivum/),由国际小麦基因组测序联盟(IWGSC)生成,并使用UNIX管道转换为本地BLAST数据库。BLAST分析(BLASTN和BLASTP)使用NCBI (National Center for Biotechnology Information)的独立命令行版本进行。BLAST 2.2.28+ (ftp://ftp.ncbi.nih.gov/blast/executables/LATEST/),于2013年3月19日发布。从Pfam域模型生成查询文件;PF00535 (GT2)域和PF03552 (Cellulose_synt)下载自2016年6月30日发布[23]。剪接变异序列也从Ensembl Plants浏览器(http://plants.ensembl.org/Triticum_aestivum/Info/Index).根据Kim等人(2007)的描述对剪接变异体进行了分析[24]。之前所知Csl序列从拟南芥,栽培稻,玉米从细胞壁导航数据库下载[25]。对于Brachypodium,从phytomine (https://phytozome.jgi.doe.gov).上述CSL蛋白的氨基酸序列在附加文件中给出1:图S1
Blast搜索小麦同源物
利用所有包含PF00535和PF03552两个Pfam域模型的查询文件,在本地面包小麦的blast数据库中进行BLASTn搜索。所有e值为>1.0的爆炸命中被移除。使用截止E值<1.0,所有先前已知中国极限运动协会基因被检索。将所有低于截止值的序列编译后,使用CD-hit程序获得非冗余序列。采用较高的截止E值确定了Pfam结构域PF00535和PF03552的所有基因。这些基因与先前已知的来自拟南芥、短茅、玉米和水稻的CSL蛋白一起通过系统发育分析进一步筛选,其中反映了一些从进一步分析中删除的非靶向基因[26]。系统发育分析也被用于分类不同的Cslsub-families。中国极限运动协会基因被区分于Csl具有CXC基序的基因,该基序是CesA的诊断性基因,但在Csl蛋白中不存在[7,27]。守恒域的存在Cellulose_synt / GT2在NCBI的CDD(保守域数据库)上进行了批量爆炸搜索。三个基因组的同源基因分别被命名TaCslXY_ZA,TaCslXY_ZB,或TaCslXY_ZD,在那里X表示Csl亚科,Y基因数目和Z小麦染色体的位置。所有新鉴定的小麦的序列比对Csl基因在附加文件中给出2:图S2。
蛋白质结构和基序/结构域鉴定
蛋白质序列从Ensembl Plants FTP服务器(ftp://ftp.ensemblgenomes.org/pub/current/plants/fasta/triticum_aestivum/),由国际小麦基因组测序联盟(IWGSC)开发[22]。使用Clustal Omega (http://www.ebi.ac.uk/Tools/msa/clustalo/) [28]。结果比对分析了保守基序(D, D, DXD, QXXRW)的存在GT2总科。使用序列操作套件突出显示已对齐序列的保守模式:颜色对齐保存(http://www.bioinformatics.org/sms2/color_align_cons.html) [29]。利用CCD数据库(http://www.ncbi.nlm.nih.gov/Structure/cdd/cdd.shtml) [22,30.,31]。小麦Csl基因的命名基于它们的序列一致性、覆盖范围、保守结构域的存在以及与先前鉴定的水稻相似的基序Csl基因。在一个亚家族中基因数量超过水稻,额外的基因被赋予了新的名称。因为…的相似CslD基因与中国极限运动协会基因及其在纤维素合成中的可能作用,我们特别关注TaCslD亚科。基因结构与内含子演化TaCslD使用基因结构显示服务器2.0 (http://gsds.cbi.pku.edu.cn/),使用基因组和cDNA序列。
的进化关系Csl基因
利用MAAFT (v1.3.6)对拟南芥、玉米、水稻和小麦的215个CSL蛋白进行了比对[32]。没有延伸到保守核心区域的序列被删除。超过40%的序列包含间隙的位置也被删除。使用Seqboot (v3.696)推断这些序列的系统发育和1000次bootstrap复制[33和FastTree (v2.1.10)在桂林集群上实现[34]。
的系统发育CslD在拟南芥、短茅、玉米、水稻和小麦中也分别发现了该亚科。在系统发育分析中,使用MUSCLE对CSL蛋白的氨基酸序列进行比对,并使用Neighbor-Joining方法推断其进化史[35]。这棵树是按比例绘制的,分支长度相当于用来推断系统发育树的进化距离。进化距离用泊松校正计算,并以每个位点的氨基酸取代数给出。站点之间的变化率用gamma分布(形状参数= 1)建模,所有包含间隙和缺失数据的位置都被删除。对MEGA6进行了进化分析[36]。
RNA-seq表达分析
利用从面包小麦(var. Chinese Spring)中获得的公开RNA-seq数据来研究新鉴定小麦的表达Csl基因。这些数据收集自小麦在苗期、营养发育阶段和生殖发育阶段的五种不同组织(穗、叶、茎、根和籽粒)。37]。每一个的相对表达式TaCsl使用expVIP(每1000万次读取)支持的小麦表达浏览器,以热图形式呈现每个基因的相对转录本丰度(每1000万次读取)。http://www.wheat-expression.com).
结果
的鉴定和分类Csl面包小麦的基因家族成员
数据库使用保守的pfam基序PF00535和PF03552搜索面包小麦,这两个基序是特异性的GT2结果鉴定出108个纤维素合酶样(TaCsl)基因(表1).A、B和D基因组中每个基因的2到3个同源拷贝是常见的。鉴定出的基因是根据水稻的命名法命名的,这与小麦有共同的合成。为了避免命名法的复杂性,每个基因名称都使用了与染色体数目和特定小麦基因组标识符(a、B或D)相对应的后缀[7]。例如,亚家族的第一个基因里昂证券;CslA1位于基因组A、B和D的1号染色体长臂上的染色体被命名为TaCslA1_1AL,TaCslA1_1BL,TaCslA1_1DL,分别。
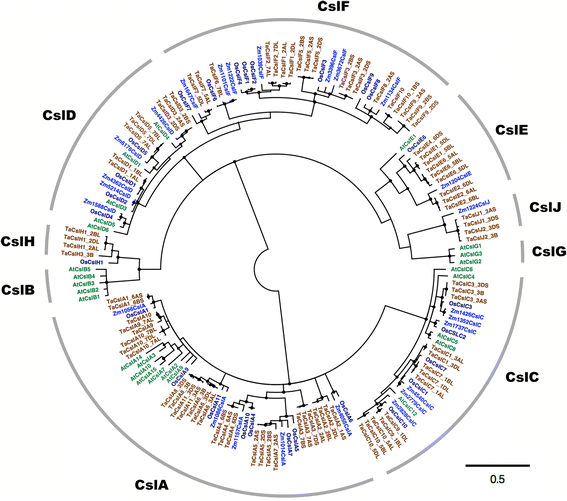
从拟南芥、玉米、水稻和小麦中获得的215个Csl蛋白的无根邻接(NJ)树如图所示。1。TaCsl蛋白分为7个亚家族:TaCslA(32个蛋白))、TaCslC(13个蛋白)、tacslld(12个蛋白)、TaCslE(10个蛋白)、TaCslF(29个蛋白)、TaCslH(8个蛋白)、TaCslJ(4个蛋白)(图2)。2).TaCslA和TaCslC亚科在分类分布和系统发育上具有密切的亲缘关系。正如预期的那样,这些亚科在整个植物物种中都是保守的。虽然tacslld存在于所有植物物种中,而TaCslF只存在于禾草中,但它们彼此接近表明它们有共同的起源[12]。在菊科植物和禾本科植物共有的序列中,CslA亚科在这两类植物之间的差异最大。在拟南芥和禾本科植物中,CslC和CslD亚科的序列是穿插分布的,而拟南芥的所有CslA亚科序列都聚集在一起,与禾本科植物的CslA序列分开。CslB和CslH亚科的接近性表明它们在禾本科和菊科分离之前就有共同的起源。同样,CslG和CslJ显然有一个共同的起源。
的剪接变体Csl基因
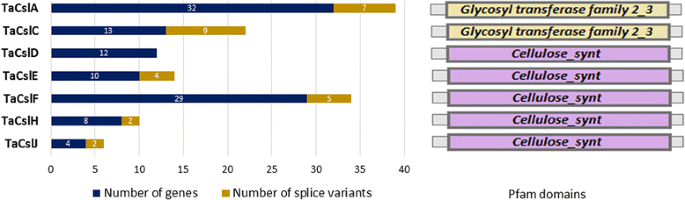
根据Ensembl数据库的预测,108个基因中有22个基因由于存在备选剪接位点而编码两种或两种以上的蛋白质,这将导致137种可能的Csl蛋白产物(表1)2).在所有亚家族中都预测了剪接变异TaCsl除了基因TaCslD(表2).在亚族中TaCslA, 6个基因交替剪接形成13个假定的蛋白质,而在亚家族中TaCslC, 5个基因被交替剪接,得到14个推测的蛋白。类似地,对于亚科TaCslE和TaCslF,选择性剪接分别产生7个和10个剪接变体。1号和2号基因的选择性剪接分别产生了3个和4个推测的蛋白质CslH和CslJ亚科(无花果。2).超过一半(51%)的剪接变异源于外显子跳变,约24%来自5 '和3 '剪接位点的选择,其余约24%来自内含子保留(表1)2).
保守基序和域
所有预测的TaCSL蛋白都含有pfam糖基转移酶家族2_3(GT)域(PF13641)或cellulose_synt域(PF03552),被认为是GT2总科(12,26]。亚科TaCslA和TaCslC包含GT 2 _3,CslD、CslE、CslF、CslH和CslJ包含了cellulose_synt域(无花果。2).所有的TaCsl移植产物含有D、DXD、D和QXXRW基序,但有8个截短的基因显然由于缺失序列而缺少这些基序(TaCslA7_2DS,TaCslD4_1BS,TaCslD4_5BS,TaCslF2_7BL,TaCslF6_7AL,TaCslF6_7DL,TaCslH3_3AS,TaCslH2_3B).大米CesA10,11和CslH3也只包含DXD但缺少D和QXXRW基序[38]。保守基序DXD和QXXRW的可变氨基酸在不同亚科中存在差异Csl例如,基因TaCslA(demand media, QQH / FRW);TaCslC(demand media, QQHRW);TaCslD论证,QVLRW);TaCslE论证,QHKRW);TaCslF(直流/ GD,气/重要/ VRW);TaCslH论证QF / YKRW);TaCslJ论证,QNKRW)。这些图案在文本文件S_2a-f中的对齐文件中突出显示。
植物的系统发育分析CslD亚科
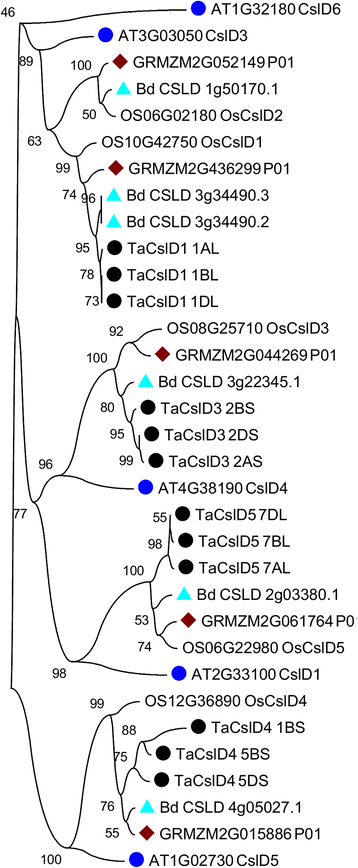
的进化史CslD利用Neighbor-Joining方法从拟南芥、短叶菊、水稻、玉米和小麦中推断出亚家族,MEGA6 [36],将不同物种的直系同源物分组为不同的枝(图2)。3.).大米Csl基因被用作参考,因为它们的完整命名法有很好的文献记载。所有的基因分为三个分支。第一个分支包含CslD2和CslD1来自水稻的基因及其来自其余物种的直系同源物。小麦的三个同源基因与OsCslD1;这一分支下的小麦基因被命名TaCslD1_1AL,TaCslD1_1BL,TaCslD1_1DL。第二个分支包含两个与水稻基因同源的亚群CslD3和CslD5来自不同的物种。第一个亚群中的基因被命名TaCslD3_2AS,TaCslD3_2BS,TaCslD3_2DS,还有第二组的人TaCslD5_7AL,TaCslD5_7BL,TaCslD5_7DL。最后的进化支是由水稻的同源物组成的CslD4小麦基因TaCslD4_5BS,TaCslD4_1BS和TaCslD4_5DS。这里我们只发现了两个同源物TaCslD4,但一种来自bs基因组的基因(TaCslD4_1BS(由小麦组成的)TaCslD4基因(bootstrap = 1000),指向其原始a基因组的易位(表2)1).该基因的序列同源性为85%TaCslD4_5BS在氨基酸水平上。OsCslD与小麦同源基因序列同源性为73 ~ 86%。
基因结构与内含子演化TaCslD亚科
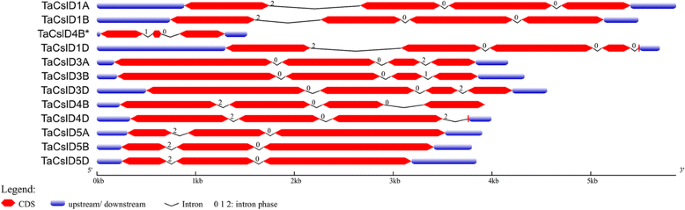
12个TaCslD从面包小麦中鉴定出的基因大小在1519 ~ 5864 bp之间。的TaCslD4_1BS吉恩是最矮的TaCslD1_1AL是最长的。所有基因的同源拷贝在核苷酸水平上具有87%至94%的序列一致性。不同基因之间大小的差异主要是由于内含子的数量和长度,但也因为数据库中缺乏完整的序列(图2)。4).所有基因的内含子数量从2个到4个不等。两个homeologs:TaCslD1_1AL和TaCslD1_1BL每个内含子包含三个内含子,而第三个同源物(TaCslD1_1DL)有四个。的基因TaCslD3,TaCslD4它们的同源物各含有三个内含子,除了TaCslD4_1BS只有两个内含子。TaCslD5它的同源物也各有两个内含子。对于内含子的阶段,来自于TaCslD亚族表现出不同的分布模式。的内含子1,2,3TaCslD1_1AL,TaCslD1_1BL和TaCslD1_1DL在2 0和0相而第4个内含子TaCslD1_1DL处于0相。的内含子1和2TaCslD3_2AS,TaCslD3_2BS和TaCslD3_2DS两者都处于0相。这些基因的第三个内含子分别处于2期、1期和2期。的基因TaCslD4_5BS,TaCslD4_5DS,TaCslD5_7AL,TaCslD5_7BL和TaCslD5_7DL内含子1和2分别在阶段2和0,第三个内含子是TaCslD4_5BS和TaCslD4_5DS分别处于0期和2期。TaCslD4_1BS在阶段1和阶段0有内含子1和2。所有基因中内含子在0期所占比例最大(60%),其次是2期(34%),第1期较少(6%)。
的表达分析TaCsl来自面包小麦的基因
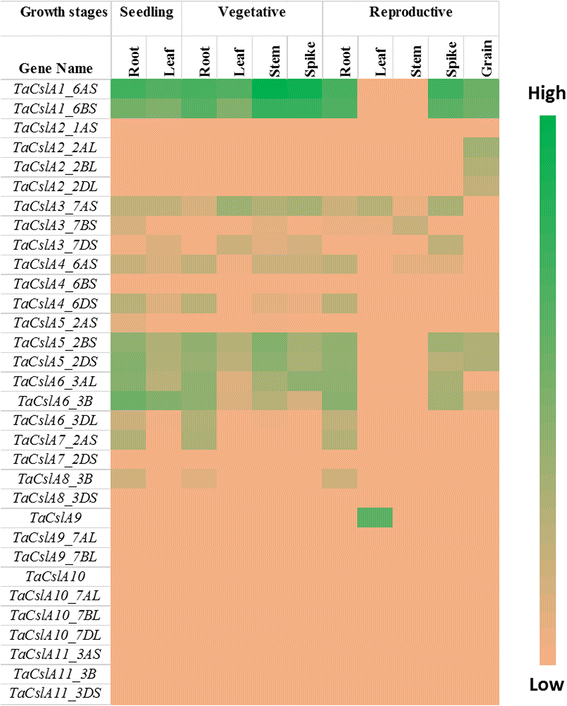
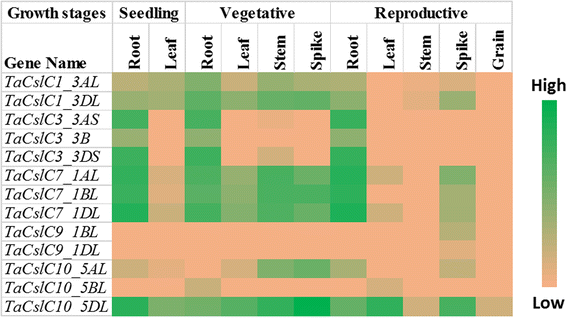
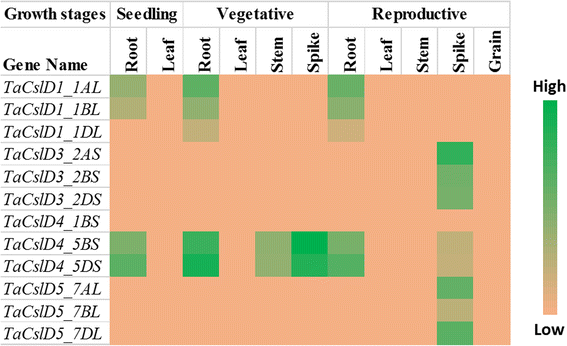
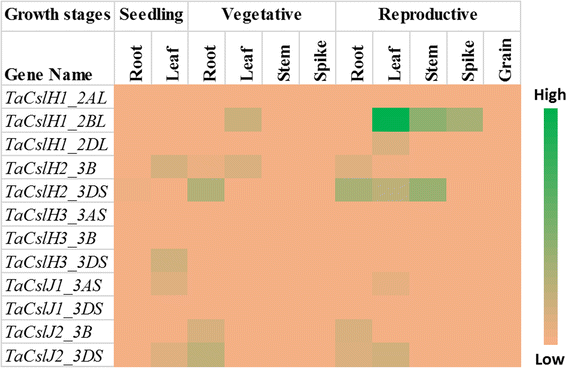
公开可用的RNA-Seq数据集用于分析TaCsl小麦三个发育阶段和不同组织的基因,包括根、茎、叶、穗和粒。其中32例的表达数据可用TaCslA基因。两个基因(TaCslA1_6AS和TaCslA1_6BS)在除生殖茎和叶外的所有组织中均有表达。四个基因(TaCslA5_2BS,TaCslA5_2DS,TaCslA6_3B,TaCslA6_3AL)表达适度。TaCslA9基因从繁殖阶段开始在叶片组织中高表达,而其余基因的转录丰度较低(图2)。5).TaCslC亚家族基因,WTHT除外TaCslC3, TaCslC9还有两个同源物TaCslC10,在根和穗组织中高度表达。两个基因,TaCslC1和TaCslC7其同源物在苗期和营养期各组织中均有中高表达。一个基因(TaCslC10_5DL)在除生殖茎和籽粒外的所有组织中均有中高表达(图2)。6).大部分基因的表达TaCslD亚家族在穗和根组织中从中等到高不等,但在所有其他组织中都很低(图2)。7).10个中的3个TaCslE亚家族基因(TaCslE2_6AL,TaCslE2_6BL和TaCslE3)在所有组织中由中到高水平表达。的remaining genes were expressed at a very low level in all the tissues (Fig.8).在大鼠中观察到混合的表达模式TaCslF亚科。三个基因(TaCslF6_7AL,TaCslF6_7BL和TaCslF6_7DL)在除叶片外的所有组织中均高表达。两个基因(TaCslF4_2BS和TaCslF4_2DS)在茎组织中高度表达,但在其他组织中仅低或中等水平表达。所有其他基因在一个或多个组织中低或中等水平表达(图2)。9).在TaCslH亚家族,八个基因之一,TaCslH1_2BL,在叶、茎和穗组织中由中到高表达。其余基因在所有组织中从低水平到中等水平表达(图2)。10).亚家族四分之三的成员TaCslJ在叶和根组织中由低到中等水平表达,而其中一个基因(TaCslJ1_3DS)在所有研究组织中表达不良(图2)。10).
讨论
草细胞壁含有20-40%的非纤维素多糖。这些多糖的比例和组成在不同的植物种类中各不相同[39]。在第一份报告证明β-葡聚糖合成酶在aCsl-编码蛋白发表[15的几个成员Csl据报道,基因家族参与了半纤维素多糖骨架的形成[16,18,19,26,38,40,41]。作为身份的信息Csl小麦缺乏基因,我们进行这项研究来填补这一空白。
我们检索到108TaCsl使用两个保守结构域PF00535和PF03552从小麦中提取的基因,这两个保守结构域先前被证明存在于所有小麦的衍生蛋白中Csl基因(12]。这些基因包括来自面包小麦A、B和D基因组的同源基因。同源基因的相似模式被发现开花地点(英国《金融时报》),配对同源1(Ph1),ADP-glucose焦磷酸化酶(AGPase六倍体小麦的基因家族。大约有四分之一的人被确定Csl基因被预测为可选择性剪接,可能有助于编码酶的多样性。最近的一项研究表明,选择性剪接在植物中很常见,约占水稻叶片和穗组织中转录位点的20%山羊草属tauschii。在萌发的大麦胚胎中,14-20%的内含子基因被选择性剪接[42]。这种现象显然是为了提高生物体的适应性,但到目前为止还没有报道Csl来自其他物种的基因[43]。
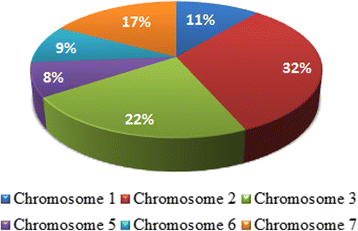
的TaCsl基因分布在除4号染色体外的所有小麦染色体上(图2)。11).类似的趋势Csl在大麦中观察到基因分布[9,44,45]。超过一半的人TaCsl基因仅位于两条染色体上:2(32%)和3(22%)。这表明了Csl这些染色体上的基因,尽管这种现象的原因尚不清楚。不过,似乎独联体复制Csl基因比反式小麦的重复。九个中的五个该论坛大麦基因位于染色体2H上[40]。事实上,大麦该论坛早在几乎没有基因组序列可用之前,基因就通过与水稻的同源性确定了其在混合链接葡聚糖(MLG)形成中的作用[40]先前发表的对大麦中MLG的已知QTL对应的水稻合成区域的详细分析,最初导致了作用的突破该论坛在这种多糖的形成过程中[40])。一组类似的该论坛在短柄草和高粱的1号染色体和2号染色体的保守共合区也分别检测到该基因[9]。
观察发现只有一半的基因来自亚家族里昂证券在被研究的组织中以不同的水平表达,这表明表面上沉默的基因可能在压力条件下提供了备份。或者,它们可能只在专门的细胞或细胞部分中短暂表达,表达水平太低,无法通过研究表达的方法检测到。的关系的第一个生化证据里昂证券甘露聚糖合成酶活性基因来源于瓜尔豆的表达里昂证券大豆体细胞胚cDNA [15]。随后对昆虫细胞的研究证实了里昂证券葡甘露聚糖合成酶的家族成员[16,46]。逆向遗传和生化方法拟南芥和石斛兰officinale还准有关联的吗里昂证券葡甘露聚糖生物合成基因[41,47]。最近对小麦的一项研究表明,一种来自小麦的基因参与了这一过程里昂证券分蘖发育、细胞壁组成和茎强的亚科。这项研究进一步提出了……的可能作用里昂证券植物碳分配中的基因转录水平[48]。
对于亚科来说TaCslC和TaCslD在营养期和生殖期,大部分基因在根和穗组织中表达量相对较高。异源表达毕赤酵母属揭示了CslC-编码的酶产生β-1,4-葡聚糖,这是木葡聚糖的主干[19]。的CslD亚科在所有陆生植物中均有保存,与植物的亲缘关系最为密切中国极限运动协会在氨基酸水平上具有40-50%序列相似性的基因家族[49]。类似于中国极限运动协会,CslD亚科在迄今为止所研究的所有植物基因组中普遍存在,不像其他的分类群特异性Csl亚科(50]。以前的报告还显示,该组织的某些成员也参与其中CslD尖端生长的亚科,如根毛发育和花粉管伸长[51,52],植物正常生长[50,53]和分生组织形态学[53,54]。最近,它们在抵抗生物压力中的作用已被描述[55]。除了这个讨论之外,我们的计算机表达分析表明某些因素的参与TaCslD穗发育中的基因。这个观点得到了一个观察结果的支持,一个突变体,细长叶1 (sle1)该基因在水稻中编码CSLD4蛋白,可减少穗部小穗的数量和宽度[56]。
两组Csl的基因,该论坛和CslH,在草类中独立进化[57]。第三种人CslJ,最初被认为是草类特有的,最近在一些菊科植物中被发现[11,13]。虽然TaCslF6除生殖期的叶片组织外,该基因在所有组织中均有较高的表达TaCslF在粒组织中高度表达的亚科。几项研究已经证明了CslF6和CslH合成MLG [18,44,58,59]。这些家族中只有一个基因成员CslF6,在籽粒中表达,表明它与MLG的形成有关。MLG是一种理想的膳食纤维多糖,但不适合酿酒业,因为它会使啤酒产生雾霾。应该有可能为基因的表达选择自然变体CslF6根据谷物的目标市场,选择增加或减少MLG含量的基因。
在面包小麦的3个不同基因组的同源拷贝中发现了不同的表达模式,这与之前的研究结果一致,即3个基因组对基因表达的贡献不相等。有趣的是,同源的TaCslD基因在内含子相进化方面也存在差异,这表明同源基因拷贝在结构和功能上存在差异(图2)。4).大多数内含子存在于0期,这与先前的研究结果一致,表明内含子偏向于0期[7,60,61]。本研究报告的所有基因均未观察到每个基因的三个同源性。这可能是由于序列信息不完整或在小麦异源多倍体化过程中基因被消除所致。
结论
我们已经确定了108个TaCsl面包小麦的基因分类及7个亚科(CslA、CslC、CslD、CslE、CslF、CslH、和CslJ).在大多数情况下,鉴定出两个或三个同源等位基因Csl基因。尽管在除4号染色体外的所有小麦染色体上都有Csl基因特别集中在2号和3号染色体上,这表明在独联体阶段。29个里只有一个该论坛的基因,CslF6,在籽粒中表达,表明其在混合连接葡聚糖形成中的作用。既不CslJ也不CslH是在谷物中表达的。本报告的信息将有助于设计改变小麦壁成分的实验,以改善籽粒品质、秸秆强度或用于生物燃料的秸秆成分。
缩写
- 中国极限运动协会:
-
纤维素合酶
- Csl:
-
纤维素synthase-like
- GT:
-
糖基转移酶
- MLG:
-
Mixed-linked葡聚糖
参考文献
- 1.
李建军,李建军,李建军,等。细胞壁碳水化合物及其在生物燃料中的应用。植物学报,2008;34(4):559 - 568。
- 2.
Sandhu APS, Randhawa GS, Dhugga KS。植物细胞壁基质多糖的生物合成。植物学报,2009;2(5):840-50。
- 3.
李建军,刘建军,李建军,等。生物质化学成分对生物燃料的影响。生物科学。2014;64(3):192 - 201。
- 4.
刘磊,刘丽娟,张建军,张建军,张建军。半纤维素生物合成技术研究进展。足底。2013;238(4):627 - 42。
- 5.
Richmond TA, Somerville CR.纤维素合酶超家族。植物生理学报,2000;24(2):495 - 498。
- 6.
李建军,李建军,李建军,李建军。高粱细胞壁相关基因的克隆与表达分析一种粮食、饲料和生物燃料作物。植物科学,2016;1287。
- 7.
李建军,李建军,李建军,等。小麦纤维素合成酶基因的克隆与功能分析。科学通报,2016;11(1):e0147046。
- 8.
Hazen SP, Scott-Craig JS, Walton JD。水稻纤维素合酶样基因。植物生理学报,2002;32(2):356 - 356。
- 9.
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军。植物生理学报,2015;36(3):968 - 983。
- 10.
Burton RA, Collins HM, Kibble NA, Smith JA, Shirley NJ, Jobling SA, Henderson M, Singh RR, Pettolino F, Wilson SM等。转基因大麦中特异性HvCslF纤维素合酶样基因的过度表达增加了细胞壁(1,3;1,4)- β -d-葡聚糖的水平,并改变了它们的精细结构。植物学报,2011;9(2):117 - 135。
- 11.
刘建军,刘建军,刘建军,刘建军,刘建军。植物细胞壁生物合成:鉴定关键基因的遗传、生化和功能基因组学方法。植物学报,2006;4(2):145 - 145。
- 12.
尹颖,马琼,曹华,Rupani M.植物和藻类基因组和转录组的研究揭示了纤维素合成酶超家族的进化和功能。生物医学工程学报,2014;15(1):1。
- 13.
Vogel JP, Garvin DF, Mockler TC, Schmutz J, Rokhsar D, Bevan MW, Barry K, Lucas S, harmono - smith M, Lail K.模式草短茅(Brachypodium Distachyon)基因组测序与分析。大自然。2010;463(7282):763 - 8。
- 14.
Dhugga KS。植物细胞壁非纤维素多糖的生物合成。植物化学。2012;74:8-19。
- 15.
Dhugga KS, Barreiro R, Whitten B, Stecca K, Hazebroek J, Randhawa GS, Dolan M, Kinney AJ, Tomes D, Nichols S.瓜尔籽ß-甘露醇合成酶是纤维素合成酶超基因家族的成员。科学。2004;303(5656):363 - 6。
- 16.
Liepman AH, Wilkerson CG, Keegstra K.纤维素合成酶样(Csl)基因在昆虫细胞中的表达揭示了Csl家族成员编码甘露聚糖合成酶。科学通报,2005;32(6):1221 - 1221。
- 17.
Burton RA, Wilson SM, Hrmova M, Harvey AJ, Shirley NJ, Medhurst A, Stone BA, Newbigin EJ, Bacic A, Fincher GB。纤维素合酶样CslF基因介导细胞壁的合成(1,3;1、4)——ß-D-glucans。科学。2006;311(5769):1940 - 2。
- 18.
Doblin MS, Pettolino FA, Wilson SM, Campbell R, Burton RA, Fincher GB, Newbigin E, Bacic A.大麦纤维素合酶样CSLH基因介导(1,3;1,4)- β - d -葡聚糖合成。科学通报,2009;35(4):591 - 591。
- 19.
Cocuron JC, Lerouxel O, Drakakaki G, Alonso AP, Liepman AH, Keegstra K, Raikhel N, Wilkerson CG。来自纤维素类合成酶C家族的基因编码-1,4葡聚糖合成酶。中国科学d辑,2007;32(5):555 - 555。
- 20.
古普塔PK, Mir RR, Mohan A, Kumar J.小麦基因组学的现状与展望。植物基因组学学报。2008;33(2):444 - 444。
- 21.
Mayer KF, Rogers J, Doležel J, Pozniak C, Eversole K, Feuillet C, Gill B, Friebe B, Lukaszewski AJ, Sourdille P.基于染色体的六倍体面包小麦(Triticum Aestivum)基因组草图序列。科学。2014;345(6194):1251788。
- 22.
财团IWGS。基于染色体的六倍体面包小麦(Triticum Aestivum)基因组草图序列。科学。2014;345(6194):1251788。
- 23.
Finn RD, Coggill P, Eberhardt RY, Eddy SR, Mistry J, Mitchell AL, Potter SC, Punta M, Qureshi M, Sangrador-Vegas a . Pfam蛋白家族数据库:面向可持续发展的未来。中国生物医学工程学报,2016;44(1):379 - 379。
- 24.
李建军,李建军,李建军,等。真核生物选择性剪接的研究进展。核酸学报,2007;35(1):125-31。
- 25.
周建军,陈建军,陈建军,陈建军。细胞壁导航数据库的研究进展。一种基于系统的方法,生物体不受限制地挖掘参与细胞壁代谢的蛋白质家族。植物生理学报,2004;36(2):393 - 398。
- 26.
尹勇,黄娟,徐勇。植物和藻类纤维素合成酶超家族的全序列分析。植物学报,2009;9(1):99。
- 27.
高等植物纤维素合酶。中国生物医学工程学报,2000;1(4):563 - 567。
- 28.
Sievers F, Wilm A, Dineen D, Gibson TJ, Karplus K, Li W, Lopez R, McWilliam H, Remmert M, Söding J.利用Clustal omega快速、可扩展地生成高质量的蛋白质多序列比对。中国生物医学工程学报,2011;7(1):539。
- 29.
序列操作套件:用于分析和格式化蛋白质和DNA序列的JavaScript程序。生物学技术。2000年,28(6):1102 - 4。
- 30.
Marchler-Bauer A, Derbyshire MK, Gonzales NR, Lu S, Chitsaz F, Geer LY, Geer RC, He J, Gwadz M, Hurwitz DI。CDD: NCBI的保守域数据库。核酸学报,2014:gku1221。
- 31.
根特异性壁相关激酶基因HvWAK1调控大麦和其他谷物的根生长,并具有高度分化性。函数集成基因组学,2013;13(2):167-77。
- 32.
kaoh K, Misawa K, Ki K, Miyata T.一种基于快速傅里叶变换的快速多序列比对方法。核酸学报,2002;30(14):3059-66。
- 33.
Felsenstein J.系统发育推断包(版本3.2)。支序分类学。1996;5:164-6。
- 34.
Price MN, Dehal PS, Arkin AP. FastTree 2 -用于大排列的近似最大似然树。科学通报,2010;5(3):e9490。
- 35.
李建平,李建平。基于邻域连接的系统发育树重建方法。中华生物医学杂志,1987;4(4):406 - 406。
- 36.
田村K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学6.0版本。中华生物医学杂志,2013;30(12):2725-9。
- 37.
李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军,李建军。科学。2014;345(6194):1249721。
- 38.
王丽,郭凯,李艳,涂艳,胡慧,王斌,崔霞,彭丽。水稻CESA/CSL超家族基因表达谱及整合分析。植物学报,2010;10
- 39.
王晓明,王晓明,王晓明,等。木醋杆菌纤维素合成酶基因acsAII的鉴定。[J] .中华微生物学杂志。1995;17(6):576 - 583。
- 40.
Burton RA, Wilson SM, Hrmova M, Harvey AJ, Shirley NJ, Medhurst A, Stone BA, Newbigin EJ, Bacic A, Fincher GB。纤维素合酶样CslF基因介导细胞壁(1,3;1,4)- β - d -葡聚糖的合成。科学。2006;311(5769):1940 - 2。
- 41.
Goubet F, Barton CJ, Mortimer JC, Yu X, Zhang Z, Miles GP, Richens J, Liepman AH, Seffen K, Dupree P.拟南芥细胞壁葡甘聚糖的CSLA糖基转移酶合成及其对胚胎发生的影响。植物学报,2009;36(3):527 - 538。
- 42.
张强,张翔,王松,谭超,周刚,李晨。大麦种子萌发过程中选择性剪接的参与。科学通报,2016;11(3):e0152824。
- 43.
周勇,周超,叶磊,董军,徐宏,蔡磊,张磊,魏磊。植物已知交替剪接基因数据库及分析。基因组学,2003;82(6):584 - 95。
- 44.
Schreiber M, Wright F, MacKenzie K, Hedley PE, Schwerdt JG, Little A, Burton RA, Fincher GB, Marshall D, Waugh R.大麦基因组序列测序发现了另外三个CslF成员(1,3;1,4)-β-葡聚糖合成酶基因家族。科学通报,2014;9(3):e90888。
- 45.
Burton RA, Jobling SA, Harvey AJ, Shirley NJ, Mather DE, Bacic A, Fincher GB。大麦纤维素合酶样HvCslF基因家族的遗传和转录谱分析。植物生理学报,2008;36(4):521 - 523。
- 46.
李磊,孙永华,蒋伟林。毛杨纤维素合酶基因超家族及木质部特异性纤维素合酶样基因的生化功能。植物生理学报,2006;42(3):1233 - 1245。
- 47.
何超,吴康,张健,刘翔,曾森,于忠,张翔,da Silva JAT,邓荣,谭杰。铁皮石斛多糖的细胞化学定位及DoCSLA6在甘露聚糖合成中的作用。植物科学,2017;8:173。
- 48.
Hyles J, Vautrin S, Pettolino F, MacMillan C, Stachurski Z, Breen J, Berges H, Wicker T, Spielmeyer W.小麦纤维素合成酶样基因重复长度变异与分蘖数和干细胞壁组成的关系。实验学报,2017;68(7):1519-29。
- 49.
Doblin MS, De Melis L, Newbigin E, Bacic A, Read SM。烟叶花粉管表达来自不同β-葡聚糖合成酶家族的两个基因。植物生理学报,2001;25(4):559 - 561。
- 50.
Hunter CT, Kirienko DH, Sylvester AW, Peter GF, McCarty DR, Koch KE。纤维素合酶样D1是玉米正常细胞分裂、扩张和叶片发育的重要组成部分。植物生理学报,2012,31(2):444 - 444。
- 51.
Kim CM, Park SH, Je BI, Park SH, Park SJ,朴海龙,Eun MY, Dolan L, Han CD.水稻根毛形态发生所需的纤维素合酶样D1基因OsCSLD1。植物生理学报,2007;43(3):1220-30。
- 52.
Yuo T, Shiotani K, Shitsukawa N, Miyao A, Hirochika H, Ichii M, Taketa S.水稻根系无毛2 (rth2)突变体描述了纤维素合酶样基因OsCSLD1的功能缺失等位基因。品种科学,2011;61(3):225-33。
- 53.
李敏,熊刚,李锐,崔健,唐东,张波,保利明,程铮,周勇。水稻纤维素合成酶样D4对正常细胞壁生物合成和植物生长至关重要。植物学报,2009;60(6):1055 - 1069。
- 54.
刘建军,刘建军,刘建军,刘建军,刘建军,刘建军。ATCSLD5的破坏导致拟南芥生长减慢,木聚糖和高半乳糖酸合成酶活性降低,木聚糖发生改变。植物学报,2007;32(5):791 - 792。
- 55.
杜奇可夫,Lueck S, Hensel G, Kumlehn J, Rajaraman J, Johrde A, Doblin MS, Beahan CT, Kopischke M, Fuchs R.大麦(Hordeum Vulgare)纤维素合酶样D2基因(HvCslD2)对白粉病真菌的渗透抗性。新植物学报,2016;22(2):421 - 33。
- 56.
Yoshikawa T, Eiguchi M, Hibara K-I, Ito J-I, Nagato Y.水稻细长叶1基因编码纤维素合酶样D4,并在M期细胞中特异性表达,调控细胞增殖。实验学报,2013;64(7):2049-61。
- 57.
Burton RA, Collins HM, Kibble NA, Smith JA, Shirley NJ, Jobling SA, Henderson M, Singh RR, Pettolino F, Wilson SM。转基因大麦中特异性HVCSLF纤维素合酶样基因的过表达增加了细胞壁水平(1,3;1,4)-β- d -葡聚糖并改变其精细结构。植物学报,2011;9(2):117 - 135。
- 58.
武田S,田友T,田野香T, Tsumuraya Y, Inagaki Y, Haruyama N, Larroque O, Jobling SA。大麦无葡聚糖突变体的功能表征表明,CslF6在(1,3;1,4)- β - d -葡聚糖生物合成中具有独特的作用。实验学报,2012;63(1):381-92。
- 59.
Nemeth C, Freeman J, Jones HD, Sparks C, Pellny TK, Wilkinson MD, Dunwell J, Andersson AAM, Aman P, Guillon F,等。CSLF6基因的下调导致小麦胚乳中(1,3;1,4)- β - d -葡聚糖的减少。植物生理学报,2010;32(3):1109 - 1109。
- 60.
林奇M.内含子进化作为种群遗传过程。科学通报,2002;19(9):618 - 623。
- 61.
董波。纤维素合酶基因内含子的起源及其进化意义。系统进化学报,2017;55(2):142-8。
致谢
这项工作得到了CGIAR联盟研究计划WHEAT (KSD)、加拿大创新基金会(CFI)、Économie部、quacimbe科学与创新部(MESI)、quacimbe自然与技术研究基金会(FRQ-NT) (RB)和加拿大自然科学与工程研究理事会通过发现计划(JS)的支持。
计算是在麦吉尔大学的超级计算机“吉利民”上进行的,该计算机由加拿大计算机公司和加拿大计算机公司管理。这台超级计算机的运行是由加拿大创新基金会(CFI)、Économie部、quacimbe科学与创新部(MESI)和quacimbe自然与技术研究基金会(FRQ-NT)资助的。
资金
加拿大自然科学与工程研究委员会。
数据和材料的可用性
是的,所有的数据都已经包含在附录里了。
作者信息
从属关系
贡献
SK提取序列,进行分析,撰写论文;KSD与JS一起构思项目,分析数据,撰写部分论文,编辑稿件;RB进行系统发育分析,构建系统发育树;JS构思并监督该项目,并帮助撰写论文。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
N/A
发表同意书
N/A
相互竞争的利益
作者宣称他们没有竞争利益。
出版商的注意
b施普林格《自然》杂志对已出版的地图和机构的管辖权要求保持中立。
附加文件
附加文件1:图S1。
用于系统发育分析的CSL蛋白FASTA序列。(PDF 453kb)
附加文件2:图S2。
Csl亚家族基因列表,蛋白质大小(氨基酸数量),不同亚家族的多个蛋白质序列比对。CSL蛋白的保守基序(D, D, DXD, QXXRW)诊断用红色框突出显示每个亚家族。(PDF 465kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
Kaur, S., Dhugga, k ., Beech, R.。et al。全基因组分析类纤维素合成酶(Csl)面包小麦的基因家族(小麦l .)。BMC Plant Biol17日,193(2017)。https://doi.org/10.1186/s12870-017-1142-z
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-017-1142-z
关键字
- 阿糖基木聚糖
- 生物能源
- 生物燃料
- 细胞壁
- 纤维素
- 中国极限运动协会
- Csl
- Glucuronoarabinoxylan
- Mixed-linked葡聚糖
- 小麦