抽象的
背景
SET是一个保守的蛋白结构域,具有甲基转移酶活性。在植物谱系(Archaeplastida)中已有一些基因组和转录组数据,但SET结构域蛋白在大多数植物谱系中的地位尚未得到全面分析。
结果
本文介绍了从16个植物家族(古原生质体)成员中计算鉴定的506个SET结构域蛋白的系统发育和结构域结构。以水稻和拟南芥的SET域蛋白为参考。该分析揭示了古原生质体中SET结构域蛋白的保守性和独特性。植物谱系的SET结构域蛋白可分为E(z)、Ash、Trx、Su(var)和Orphan 5类。SET蛋白孤儿类主要在早期古原生质体中含有独特的结构域。与之前的研究相反,本研究首次在绿藻中而不是苔藓中出现了SRA等几个结构域在SET结构域蛋白上。
结论
本研究是一个框架,用于在植物谱系中进行实验表征设定结构域蛋白质。
背景
表观遗传细胞记忆在有丝分裂和/减数分裂期间是可遗传的,但不在遗传物质中编码[1,2].组蛋白翻译后修饰、DNA甲基化、非编码rna和染色质重塑是表观遗传的主要组成部分[3.].许多组蛋白修饰,如乙酰化,甲基化和磷酸化主要发生在组蛋白的N-末端尾部[2,4,5,6].这些组蛋白修饰与转录抑制或激活、DNA修复和DNA重组相关[7].在各种组蛋白修饰中,赖氨酸甲基化在植物、真菌和动物模型中进行了严格的研究[8,9].赖氨酸残基可以是单体,二和三甲基化的[8].赖氨酸甲基化是由130-150个氨基酸组成的蛋白质结构域催化的。SET这个名字来源于果蝇的蛋白质,苏(var)3 - 9[10],Zeste的增强剂,e(z) [11),而Trithorax(硫氧还蛋白) [12有这个域名。唯一的例外是由不含设定结构域的Dot1甲基转移酶沉积的H3K79甲基化标记[13].
基于对果蝇同源物的设定结构域序列相似性,植物设定域蛋白分为4个主要阶级[14], Zeste增强子,E(z)同源物;无,小的,或同源盘(灰)同源物和相关蛋白质;三胸(Trx)同源物及相关蛋白;和杂色抑制基因、Su(var)同源物及相关蛋白[14,15,16].Su(var)和e(z)组蛋白主要参与转录抑制,而TRX和ASH组主要涉及转录降压[16,17].稍后将单独的类别添加以包括具有中断设定结构域的蛋白质,低保守的设定结构域和具有TPR和Rubisco结构域的蛋白质[18,19,20.].这种类别的蛋白质并不具备其生物化学活性。
在这里,我们已经采取的计算方法来分析16种不同的类群原始色素体生物的SET结构域序列。506种蛋白质被识别被分成五大类。我们的工作将作为植物系的SET蛋白质的功能和生化特性的指导原则。
方法
在原始色素体生物SET结构域蛋白的鉴定
要在archaeplastida中揭开诱导蛋白,保守的设定结构域氨基酸序列来自拟南芥(在),奥雅萨苜蓿(操作系统),挪威云杉(PA),卷柏meollendorffii(Sm),Physcomitrella金属盘(PP),micromonas pusila.(微控制器),micromonas rcc299(先生)欧洲核心糖陶里(Ot),Ostreococcus lucimerinus(ol),小球藻vulgaris.(CV),衣藻reinhardtii(CR)和Volvox carteri(Vc)通过BLAST搜索,使用SET域(PF00856)作为PFAM数据库的查询http://pfam.xfam.org/family/PF00856) [21.].还从UniProt检索设定的结构域蛋白序列(http://www.uniprot.org/)和Phytozome v12.1 (https:///phytozome.jgi.doe.gov/pz/portal.html.)在PFAM中不可用。为了更好地了解植物谱系中含有域含有域的蛋白质的进化历史和功能,我们已经使用了组装的转录组数据,Nitella君子兰(Nm) (https://www.ncbi.nlm.nih.gov/bioproject/PRJNA158153158153), Klebsormidium flaccidum(kf)(https://www.ncbi.nlm.nih.gov/bioproject/ prjna 51159)。Cyanophora.悖论(http://cyanophora.rutgers.edu/cyanophora/)和预测的蛋白质组数据地钱(MP)(https://www.ncbi.nlm.nih.gov/bioproject/PRJNA218052)和裸子植物,挪威云杉(PA)(http://marchantia.info/tom/blast/blast.html汤姆/爆炸/ blast.html)。在硅蛋白组数据集中,如果两个或多个重叠的蛋白质序列产生于相同的遗传位点,我们选择最长的序列。从NCBI网站获得的转录本序列使用转码器(https://transdecoder.github.io/)给出了最长的开放阅读框架。本地BLASTP(ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/latest/.nih.gov /爆炸/可执行文件/爆炸+ /最新/)用这些预测的蛋白质对来自的130-150长度的SET域序列进行拟南芥来过滤包含序列的SET域。然后,从在线搜索得到的所有SET域序列中去除多余的SET域氨基酸序列。的拟南芥和奥雅萨苜蓿由于SET域蛋白序列是开花植物的代表,具有相对完整的注释蛋白信息,所以使用它作为查询。
域架构
特定结构域及其组织的存在或缺失可能提示不同蛋白质组的功能差异。此外,一般认为具有相同结构域的蛋白质可能具有密切的进化关系和功能关联。使用NCBI Batch CD搜索工具搜索PFAM和local Blast预测蛋白的特征SET域及其相关域(http://www.ncbi.nlm.nih.gov/structure/bwrpsb/bwrpsb.cgi/)和NCBI-CD Hit (https://ww w.ncbi.nlm. .)nih.gov /结构/ cdd / wrpsb.cgi).
系统发育分析
用于系统发生树结构中,251组SET结构域的序列与E(z)的,灰,Trx的和苏(VAR)组中检索包括在内。含有来自孤儿,SETD,和TPR蛋白SET结构域被排除由于与E(z)的,灰,Trx的和苏(VAR)低的序列相似性。SET结构域序列的多序列比对所使用的ClustalW比程序执行的。进化分析在MEGA7.0程序进行[22.].进化历史,通过使用基于与1000次重复用于内部分支可靠性的引导测试的JTT矩阵基于模型的最大似然方法和使用的所有站点的选择,因为间隙/缺失的数据处理推断。最高数似然(-39,059.190)树所示。初始树(S)为启发式搜索通过施加邻接法和BioNJ算法以成对距离的矩阵估计用JTT模型,然后选择具有优异的对数似然值拓扑自动获得的。
设置域序列的多个对齐
对参考植物谱系中未注释的保守SET域序列进行多序列比对拟南芥和水稻SET结构域序列(http://multalin.toulouse.inra.fr/multalin/),使用默认参数。SET域序列与拟南芥和米饭。SET结构域序列被归类到SET结构域的相应基团。分类是基于相邻域组织还解释。
SET结构域蛋白的命名和分类
SET结构域蛋白根据与一般的大写字母,使用常见的特异性蛋白名称后面加上种名在小写标注的植物和动物的SET结构域的组蛋白甲基转移酶的命名法来命名。SET结构域蛋白是根据最接近的名字命名拟南芥和奥雅萨苜蓿同族体。如果两种或两种以上的蛋白质与一种相同拟南芥和奥雅萨苜蓿含有蛋白质的SET域,然后使用相同的名称,后面跟着字母“a”、“b”等。含有SET结构域的蛋白质的类和子类采用以下命名法:例如,蛋白质类型,II- 2c属于II类、2类和C类的细分。
结果
原质体中SET结构域蛋白的鉴定与分类
为了了解从植物谱系的植物域和可能的功能关系中的集合域蛋白质,我们从16个archaeplastida对从PFAM和NCBI转录机组件中检索的集结构域序列进行了同源性搜索。鉴定了总共506个候选结构结构域(图。1额外的文件1:表S1和附加文件2:表S2)。该集合域同源性搜索确定了每种植物物种中含有蛋白质的25-50个域的范围。在本研究中鉴定的蛋白质的长度范围为200至3500氨基酸。含有结构域的蛋白质的数量显示出具有粗略的演变的非线性增量(图。1).
SET结构域蛋白可分为5类。类I, E (z),二类灰,第三类硫氧还蛋白和四级苏(var)和类v类II, III和IV每个也称为孤儿一个子类,集域与守恒的显著低同源组域或没有签名各自类的域。Class V包括Orphan、SETD和TPR, SET域与其他4组序列相似性低。SETD蛋白家族具有典型的Rubisco结构域。TPR具有四三肽结构域,而Orphan家族具有中断的SET结构域或具有少量相关结构域的SET结构域(图)。1).这里呈现的分类是具有来自其他植物物种的已经注释的组蛋白甲基转移酶的均匀性,其修饰几种。在本研究中考虑的少数基因组是草案阶段,因此我们分析中的一些蛋白质可能缺失或可能在预测的蛋白质结构中具有误差。
含有单细胞胶质阴性物种中蛋白质的设定结构域的数量,Cyanophora paradoxa,(16)是约3倍相比蕨类植物物种降低,卷柏meollendorffii(51)。然而,少量的SET结构域蛋白在Cyanophora paradoxa也可能是由于目前可用蛋白质组中缺失的蛋白质。e(Z)蛋白质蛋白没有检测到,肺泡糖和叶绿藻SP。Volvox carteri虽然它们存在于另一种绿藻中,衣藻reinhardtii.灰分和Trx组蛋白早在绿藻中就已被发现,灰分和Trx组蛋白的数量随物种复合作用的增加呈非线性增加。在4个主要亚科中,灰分蛋白在植物种类中保持了几乎稳定的数量。然而,一个值得注意的和令人惊讶的特点是,灰组蛋白质的缺乏micromonas pusila.(图。1).TRX蛋白显示龟豆,Marchantiophyta,Bryophyta,Bryophyta的数量突然加倍,与叶绿素,肺霉菌相比。苏(var)蛋白质不存在于青光膜和叶绿藻物种中,volvox carteri,乌欧鸵鸟科SP。和micromonas rcc.299再有就是在中,地钱门,苔藓植物,蕨类植物和数量逐渐增加。三十四肽重复(TPR)的蛋白的组中的绿藻,首先识别,衣藻reinhardtii在蕨类植物中含量最高。SETD蛋白在绿藻中被发现,但在不同种类的植物谱系中数量没有成比例的增加。在绿藻门和轮藻门中,SETD的缺失一直存在争议。不太可能,孤儿蛋白自绿藻门出现后就被鉴定出来。孤儿蛋白在micromonas rcc299,而地钱具有最低数量的孤儿家族蛋白。序列比较来自其他原体类群成员的含有SET域的蛋白和参考种的蛋白,拟南芥和奥雅萨苜蓿.胚胎植物中编码的SET域蛋白数量和多样性的增加可能反映了多细胞程序的多样性和复杂性的增加。这些结构域的特性将有助于理解SET结构域蛋白的功能。
系统发育分析
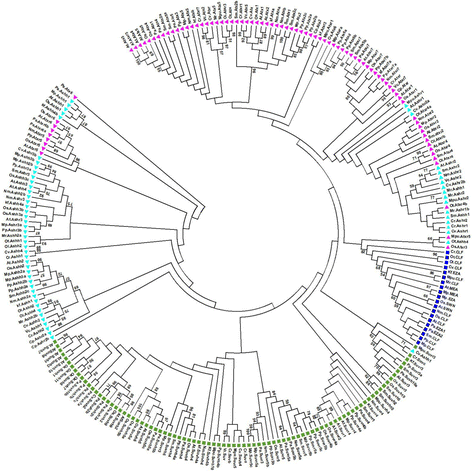
250个SET域蛋白分别属于E(z)、Ash、Trx和Su(var),用于系统发育聚类。由于序列相似度低,孤儿、SETD和TPR中剩余的SET域候选蛋白被排除在系统发育分析之外。251个SET域蛋白序列的多序列比对在附加文件中提供3.:图S1。基于组特异性结构基序的存在和缺失以及序列比对,将这251条SET结构域蛋白序列分为E(z)、Ash、Trx和Su(var)。值得注意的一点是,许多Ash和Trx相关蛋白从它们各自的分支分离出来(图。2).
原质体中set结构域蛋白的系统发育树。利用ClustalW对来自16个物种的251个蛋白的SET结构域序列进行了比对。系统发育分析使用MEGA 7.0进行。原质体的SET结构域蛋白被分为四个不同的组:E(z)、Ash、Trx和Su(var) 3-9蛋白。随附图例所述的各分支的分支节点颜色明显。其中深蓝色矩形表示E (z)组,青蓝三角形表示Ash组,粉色三角形表示Trx组,绿色矩形表示Suv组。利用基于JTT矩阵模型的最大似然法推导了该模型的演化历史。显示了具有最高log likelihood(−39,445.1504)的树。启发式搜索的初始树是通过将Neighbor-Join和BioNJ算法应用于使用JTT模型估计的成对距离矩阵,然后选择具有较优对数似然值的拓扑来自动获得的
趣味i类增强器,E(z)
E(z)是Polycomb抑制复合物2的催化成分,在植物谱系的少数成员中具有特征,催化H3K27的二甲基化和三甲基化[23.].E(Z)类蛋白质被命名为我研究中的I类蛋白。基于组合结构结构域的组合存在其他结构域,注意到七种e(z)蛋白的变化。与其他课程不同,这类蛋白质似乎具有严格且较少多样化的域组合。(图。3.).预设结构域在单晶,稻米和蕨类植物中存在,卷柏.甲钒结合结构域存在于Physcomitrella金属盘和地钱.富含半胱氨酸/丝氨酸的核蛋白质,(CSR)和男性特异性致死(MSL)结构域是鸵鸟科特定.Cyanophora.和Volvox.缺乏这种蛋白质。有趣的是,植物谱系中的大多数成员只有一个E(z)同源物,而Marchantia和拟南芥包含三个和云杉包含两个同源染色体。
e(z)家庭的域架构。示意图显示了e(z)蛋白的域组织。对于E(Z)家族,显示了通过定位域和相关域的定位不同的七个主要代表性子组。共享特定域架构的物种的相应名称被放置在右侧。发散域通过图中的不同颜色指示。SANT:SWI3,ADA2,N-COR和TFIIIB;TCR:Tesmin / TSO1;VN:钒结合蛋白;Aldh-SF:醛脱氢酶超家族;CSR:富含半胱氨酸/丝氨酸的核蛋白质; MSL: Male-specific lethal. Domains are not drawn to scale. Scale bars indicate 100 amino acids. (At-拟南芥;操作系统- - - - - -奥雅萨苜蓿;Pa -挪威云杉;Sm -卷柏meollendorffii;页-Physcomitrella金属盘;国会议员-Marchantia多形;纳米-Nitella杆菌;Kf -Klebsormidium flaccidium;先生。-Micromonas RCC299;微控制器-Micromonas pusila;不-StreoCoccus tauri;ol-ostreococcus lumiferans;简历-Chleslella Venlarla;Cr-chlamydomonas Reinhardtii;风投-Volvox carteri;Cp-cyanophora悖论)
II类-缺失的,小的,或同源的盘状(灰分)和相关蛋白质
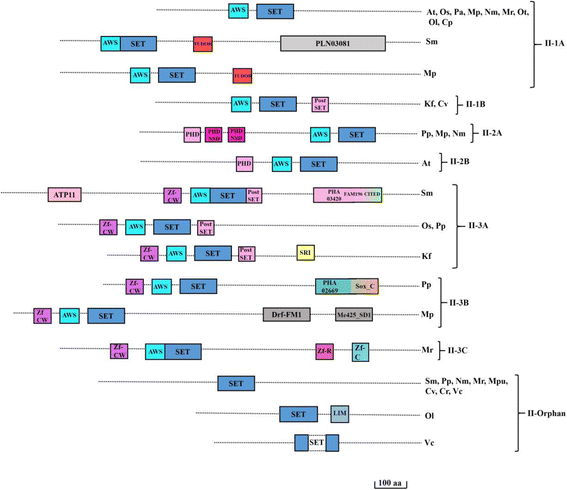
ASH类蛋白质具有不同的域组合。基于域组合,这里提出了一种新的灰蛋白的分类。灰分蛋白分为4级,II-1级和II-3和II-Orphan(表1).第II-1类和第II-2类分为2个子类,而第II-3类又分为3个子类。除孤类外,所有蛋白均具有AWS (Associated with SET domain),表明其在Ash类蛋白的功能中起着不可或缺的作用(图)。4).Class II-1A是AWS域,其次是SET域,而Class II-1B是PostSET域。绿藻门成员,小球藻vulgaris.示出了在灰家庭后集域的初始存在(附加文件4Class II-2有Plant Homeo Domain (PHD),其次是AWS和SET Domain。非常有趣的是,Physcomitrella金属盘,地钱,Nitella君子兰在串联的3个PHD结构域。多数类II-3蛋白具有锌指。您正在访问的是轮藻门的物种引进,micromonas rcc299(附加文件4S2:图).这类蛋白质可能使组蛋白中不同位置的多个氨基酸甲基化。
Ash家族的域架构。Ash家族蛋白的示意图。三个主要的具有代表性的子组由于SET域的定位和相关域域的类型不同而与孤儿组一起显示。这些组又进一步细分。该蛋白质的右侧表示具有特定蛋白质结构的物种的缩写形式。相关的域由不同颜色的带有名称的框表示。AWS:与SET相关;博士:植物homeodomain;PHD_NSD:在核受体结合的SET结构域中发现的植物同源结构域;Zf-MYND:骨髓神经,耳聋; Zf_C: Zinc binding motif composed of cysteine motif; TPR: Tetratricopeptide Repeat; Zf-CW: Zinc finger domain with conserved cysteine and tryptophan residues; TUDOR: Royal family protein; PLN03081: pentatricopeptide (PPR) repeat containing protein; PKC: Protein kinase catalytic domain; TNG2: T-cell leukemia neighbouring genes; ATP_11: Adenosine tri phosphate 11; PHA_03420: E4 protein; PHA02669: Hypothetical protein; CITED: CBP/p300-interacting transactivator with ED-rich tail, FAM196: Family of unknown function; SRI: Set2 Rbp1 interacting; Zf_R: Zn-finger in Ran binding protein and others; SoxC: Sry-related HMG box; Drf_FM1: Diaphanous related formin homology region1; Me425_SD1: Mediator complex 25 iynapsin 1; LIM: Lin11, Isl-1 & Mec-3. Domains are not drawn to scale. Scale bars indicate 100 amino acids. (At-拟南芥;操作系统- - - - - -奥雅萨苜蓿;Pa -挪威云杉;Sm -卷柏meollendorffii;页-Physcomitrella金属盘;国会议员-Marchantia多形;纳米-Nitella杆菌;Kf -Klebsormidium flaccidium;先生。-Micromonas RCC299;微控制器-Micromonas pusila;不-StreoCoccus tauri;ol-ostreococcus lumiferans;简历-Chleslella Venlarla;Cr-chlamydomonas Reinhardtii;风投-Volvox carteri;Cp-cyanophora悖论)
iii类-三胸同源物及相关蛋白(Trx)
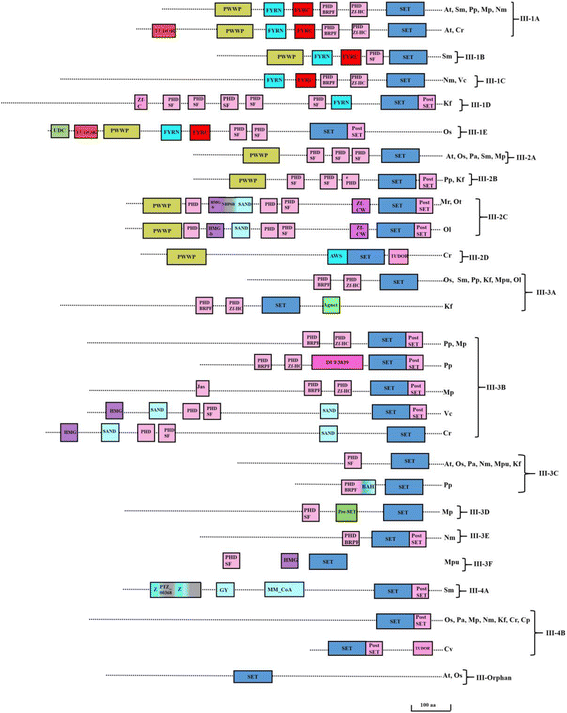
根据早先的分类,Trx蛋白被指定为III类,并细分为4个亚类,III-1类至III-4类[24.].每个亚类进一步分为亚类- iii - 1a至E、III-2A至D、III-3A至F、III-4A、B和孤儿(表1)2).除了SET结构域外,III类蛋白还具有若干结构域,如脯氨酸-色氨酸-色氨酸-脯氨酸残基结构域(PWWP)、苯丙氨酸-酪氨酸残基结构域(FYR)、PHD和PostSET结构域(图)。5).PWWP,PHD,早期检测到叶绿素物种中的FYR结构域,Volvox carteri(附加文件5:图S3)。Trx中的III-1类唯一具有FYR和PWWP域。FYR结构域由一个FYR n端部分和一个FYR c端部分组成,它们通常彼此靠近。一些物种如衣藻reinhardtii,拟南芥和奥雅萨苜蓿在这类蛋白质中具有皇室结构域(TUDOR)。在III-1类的子类中,PHD域的数量有所变化。III-2组通常包含一个PWWP域、两个到三个PHD域和一个不含FYR域的PostSET域(图2)。5).III-2A类缺少PostSET域。III-2C类的一个独特特征是锌指(ZnF), Sp100, air -1, NucP41/75, deaf1 (SAND)和高迁移率基团盒(HMGb)结构域以及典型的PWWP, PHD, SET和PostSET结构域的存在。另一个显著的特征是在III- 2d的一个蛋白类型中同时存在AWS和PWWP域(分别为II类和III类的签名域)。III-3类缺乏PWWP域,以PHD和SET域的存在为特征。子类III-3B和III-3E有一个额外的PostSET域。Class III-3D具有单一的预设域,而Class III-3F具有HMG域。III-3类除典型结构域外,还具有aptala 2 (AP2)、Agnet(皇室结构域)、溴邻同源(BAH)和恶性脑肿瘤(MBT)结构域。除SET和PostSET外,蓝藻没有其他结构域。III-4A类包含一个额外的甘氨酸-酪氨酸-苯丙氨酸残基(GYF), ZnF_C2H2和HMG结构域。 These domains are reported for RNA binding or single-stranded DNA binding in eukaryotic proteins [25.].III-4B类通常没有额外的域,除了SET和PostSET域没有额外的域。
Trx家族的域架构。Trx蛋白的代表性结构域。Trx蛋白被分为四组,每组再细分。右边的图是物种共享域排列的简写形式。PHD1:植物homeodomain1;植物同源结构域超家族;扩展的植物同源结构域;FYRN_C: n端和c端富苯丙氨酸/酪氨酸结构域;锌关节是一个锌结合基序组成的半胱氨酸残基序发现大部分从逆转录病毒gag蛋白;HMG-b:高迁移率组盒; Bro-Bromodomain; AP2: Apetala2; TUDOR: Royal family protein; NHP6B: Chromatin-associated proteins containing the HMG domain; Agnet: Royal family domain; DUF_3839: Domain of unknown function; Jas: Jasmonate motif; BAH: Bromo-adjacent homology; MBT: Malignant brain tumor; PTZ_00368: hypothetical protein; GYF: GYF domain: contains conserved Gly-Tyr-Phe; MM_CoA: Methylmalonyl CO enzyme A; SAND: Sp100, AIRE-1, NucP41/75, DEAF-1 domain. Domains are not drawn to scale. Scale bars indicate 100 amino acids. (At-拟南芥;操作系统- - - - - -奥雅萨苜蓿;Pa -挪威云杉;Sm -卷柏meollendorffii;页-Physcomitrella金属盘;国会议员-Marchantia多形;纳米-Nitella杆菌;Kf -Klebsormidium flaccidium;先生。-Micromonas RCC299;微控制器-Micromonas pusila;不-StreoCoccus tauri;ol-ostreococcus lumiferans;简历-Chleslella Venlarla;Cr-chlamydomonas Reinhardtii;风投-Volvox carteri;Cp-cyanophora悖论)
iv类杂色抑制性基因同源和亲缘Su(var) 3-9
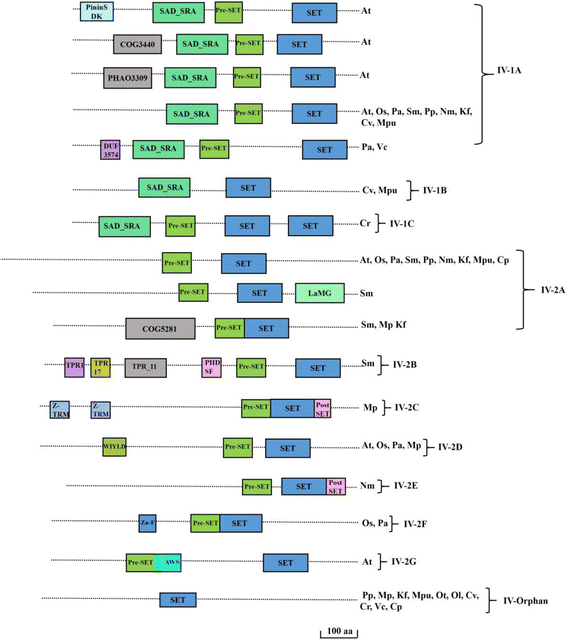
Su(var)由一大群蛋白组成,其特征是存在预设、SET和PostSET结构域。这个类别在这里被指定为第四类(图。6).该类分为两种 - IV-1和2. IV-1被细分为IV-1A至C. IV-2被细分为IV-2a至G.大多数IV-1的成员具有特征安排设置和戒指指状物关联(SAD_SRA)域,后跟一个预设的n终端到集合域。只有异常是IV-1B类,在SAD_SRA和SET域之间不存在预设域。SAD_SRA结构域与甲基化胞嘧啶结合[26.].类IV-1B蛋白存在于小球藻vulgaris.和micromonas pusila..IV-C类有两个SET域,分别位于c端SAD_SRA和PreSET域。这类蛋白质存在于衣藻reinhardtii.IV-2类的所有成员在SET域之前都有一个预设域,不包含SAD_SRA域。类4 - 2c在其n端包含一个或多个ZnF_C2H2结构域,仅在地钱而类IV-2D包含一个泛素结合在其N-末端带有预设和SET沿WIYLD域(WIYLD),在发现拟南芥,奥雅萨苜蓿,挪威云杉,地钱(桌子3.).这个领域组织可能是最近才出现的Marchantia polymorpha,属于胚胎植物(附加文件6:图S4)。值得注意的是,不像我们看到的,SRA_SET已经存在于叶绿素中,这与之前关于它首次出现在苔藓植物中的报道相反[27.].我们的分析表明PostSET域只存在于地钱和Nitella Mirabilis,分别归入IV-2C类和IV-2E类。PostSET域可能在随后的发展过程中被一些成员丢失了(附加文件6:图S4)。
Su(var)3-9家族的域组织。Su(var)3-9个家庭蛋白质分为两组,并进一步亚组,共17种不同的域组合。分享特定域安排的物种在右侧表示。如图所示,不同的蛋白质结构域不同。SRA:YDG集和无戒指的关联;TPR:四氢肽重复;Z-TRM:TRNA 2'-O-甲基转移酶TRM13;wiyld:泛素绑定wiyld域;duf3574:未知功能的域名;COG5281:噬菌体相关的次尾蛋白; LaMG: Laminin G domain; UBA: Ubiquitin associated domain. Domains are not drawn to scale. Scale bars indicate 100 amino acids. (At-拟南芥;操作系统- - - - - -奥雅萨苜蓿;Pa -挪威云杉;Sm -卷柏meollendorffii;页-Physcomitrella金属盘;国会议员-Marchantia多形;纳米-Nitella杆菌;Kf -Klebsormidium flaccidium;先生。-Micromonas RCC299;微控制器-Micromonas pusila;不-StreoCoccus tauri;ol-ostreococcus lumiferans;简历-Chleslella Venlarla;Cr-chlamydomonas Reinhardtii;风投-Volvox carteri;Cp-cyanophora悖论)
v类孤儿,SETD和TPR
V类V类蛋白质与保守的设定结构域蛋白共享低水平的序列同一性,因此分别分组。V类包括孤儿蛋白,SetD和TPR。在v类中不存在在其他组中发现的不同相关域名(附加文件7:图S5和附加文件8:图S6)。很少有成员也表现出中断设定域。孤儿组中许多独特的域,如染色体凝结调节剂(RCC1);BHLH-MYC(基本螺旋循环螺旋与MYC转录因子结合);HLH(螺旋循环螺旋);发现Basp1(脑酸可溶蛋白1),核糖体蛋白L1(RP1A)和DUF4239(未知功能的结构域),其在其他设定结构域蛋白中是不常见的[28.,29.,30.].SetD蛋白质含有Rubisco赖氨酸丝氨酸甲基转移酶(LSMT)底物结合结构域,其允许蛋白质结合组蛋白H3和H4的N-末端尾部[31.].与典型SET蛋白相比,SETD的SET结构域也具有高度分化的序列,并可能甲基化非组蛋白靶标[32.,33.].(附加文件7:图S5和附加文件8:图S6)。然而,设定区域内的特定甲基化特征保留[31.,32.].TPR组蛋白质对规范设定结构域序列缺乏显着的同源性。该蛋白质家族具有最小34个氨基酸的四肽的独特域。
讨论
在本研究中,我们从已发表的16个植物始祖质体成员的基因组和转录组中计算出506个SET结构域蛋白。我们对SET结构域进行了系统发育分析,并根据其结构域组织对蛋白质进行了分类。发表了水稻和水稻的SET域系统发育拟南芥用作模板[14,15,19].该分类可以反映功能相关性,并且是选择候选者以实验阐明设定结构域蛋白的功能的基础。未来,看看这里呈现的设定领域蛋白中各个领域的损失和增益是否与增长模式,栖息地和植物血统的进化史上的改变有关。在TRX组中引入灰组蛋白蛋白的蛋白质,Zn手指C2H2和Su(var)的次曲线促进令人兴奋地推测这些变化对底栖的生活方式。
与拟南芥一样,水稻和其他开花植物的植物谱系似乎比酵母和动物有相对较多的SET域蛋白[15,33.].有趣的是,单独的类蛋白质的号码指示谱系特异性或这些蛋白质的多余的功能不同的植物群体显着变化。
植物集域蛋白有较少的结构域。在植物谱系中可能,在较大数量的蛋白质中保持域。Tesmin / TSO1(TCR)结构域与E(Z)类蛋白质有关。Glaucophyte在设定结构域蛋白中没有E(Z)蛋白质也不是TCR。
E(Z)蛋白的演化中的其他有趣特征是SWI3,ADA2,N-COR和TFIIIB(SANT)结构域的存在,仅在水稻中表明单子叶特异性功能。醛脱氢酶超家族(Aldh-SF);半胱氨酸/丝氨酸核蛋白(CSR);男性特异性致死(MSI)是与e(Z)成员的设定结构域相关的生物化学上无表特征域。
灰类蛋白质具有不同的结构域组合,因此我们对它们进行了新的分类。除孤儿外,AWS域与Ash类蛋白的SET域N端连接非常紧密。一个有趣的特征是,在II-2B类蛋白上有一个PHD结构域,而在II-2A类蛋白上有三个PHD结构域。TUDOR结构域据报道在植物谱系中不存在,但我们证实了它在植物Ash类蛋白中存在卷柏meollendorffii和地钱[19].该域也存在于TRX类蛋白质中。
在一般的Trx类蛋白质中具有较大数量的结构域。这是朝向动物域蛋白的趋势。主要是PWWP和PHD结构域与TRX类蛋白质相关联。铎域已经出现在叶绿素中的结构域蛋白中。
Su(var) 3-9 SET蛋白的系统发育分类是基于SRA结构域的存在或不存在。我们的分析表明,IV类的WIYLD域和锌指域起源于marchantiophyta (地钱).SRA Domain早已作为血糖和Su(var)的秋季域3-9次追溯到甲型细胞,然后在随后的演变期间丢失了一些成员。(图。6额外的文件6:图S43.).我们推测,与其他陆生植物的分化之后人数较多苏(VAR)的开花植物品种3-9组蛋白质是由于近期重复和它们在植物发育的作用有关开花。这是,KRYPTONITE花发育这一类的成员之一的作用是一致的[34.].
在生命形式的多样性及其与表观遗传规则进化的联系的背景下,值得注意的是,一个物种在多细胞、栖息地和形式方面的复杂性被认为与表观遗传规则无关。这一分析表明,SET域蛋白的多样性可能不是与物种的不同生物学相关的唯一决定因素。另一种可能是同源蛋白在不同的物种中扮演着不同的角色。由此可见,在演化过程中,在SET域的基础上又获得了一个额外的域来实现补充功能。因此,我们认为,目前对Archaeplastida SET结构域蛋白的鉴定和分类,将加速这些蛋白的生化表征。
结论
通过序列同源性和系统发育分析方法,鉴定和分析了来自16个古原生质体成员的506个SET结构域蛋白。我们将这些SET结构域蛋白分为5类:E(z), Ash, Trx, Su(var)和Orphan。我们的工作为植物SET结构域蛋白的实验表征提供了框架。
参考
- 1.
Russo Ve,Martienssen Ra,Riggs广告。基因调控的表观遗传机制。纽约,美国:冷泉港实验室出版社;1996年。
- 2.
通过在植物中有丝分裂和减数分裂Saze H.后生内存传输。研讨会在细胞和发育生物学。2008; 19(6):527-36。
- 3.
哥德堡AD,艾利斯CD,伯恩斯坦e。表观遗传学:一个景观的形成。细胞。2007;128(4):635 - 8。
- 4.
伯杰sl。转录过程中染色质调控的复杂语言。自然。2007; 447(7143):407-12。
- 5。
斯特拉尔BD,Allis CD。共价组蛋白修饰的语言。自然。2000; 403(6765):41-5。
- 6.
帕特尔DJ,表观遗传修饰的王Z.读数。Annu启生物化学。2013; 82:81-118。
- 7.
Ho L,Crabtree Gr。染色质在开发期间重塑。自然。2010; 463(7280):474-84。
- 8.
刘超,陆芳,崔旭,曹晓霞。高等植物组蛋白甲基化的研究进展。植物学报2010;61:395-420。
- 9.
真菌中SET结构域蛋白的组蛋白甲基化。微生物学报。2017;8(71):413-39。
- 10.
由果蝇位置效应斑变抑制基因Su (var) 3-9编码的蛋白结合了同源性基因复合物的拮抗调节域。EMBO j . 1994; 13(16): 3822。
- 11.
Jones R,Gelbart W. Zeste的果蝇Polycomb-Group基因增强剂含有与Trithorax序列相似的区域。Mol细胞Biol。1993年; 13(10):6357-66。
- 12.
Stassen MJ,Bailey D,Nelson S,Chinwalla V,Harte PJ。果蝇曲率蛋白含有新的核受体类型DNA结合结构域的新型变体,以及在其他染色体蛋白中发现的古老保守基质。MECH DEV。1995年; 52(2):209-23。
- 13。
冯Q,王H,NG HH,Erdjument-Bromage H,Tempst P,struHl K,张Y.H3-赖氨酸79的甲基化由没有设定结构域的新的HMTase家族介导。Curr Biol。2002; 12(12):1052-8。
- 14。
Baumbusch LO,Thorstensen T,克劳斯V,费A,瑙曼K,Assalkhou R,舒尔茨I,路透G,阿伦RB。拟南芥基因组包含至少29倍编码,可以分配到四个进化上保守的类SET结构域的蛋白的活性的基因。核酸RES。2001; 29(21):4319-33。
- 15.
施普林格NM, Napoli CA, Selinger DA, Pandey R, Cone KC, Chandler VL, Kaeppler HF, Kaeppler SM。玉米和拟南芥SET结构域蛋白的比较分析表明,在单子叶和双子叶分化之前存在多个重复。植物杂志。2003;132(2):907 - 25所示。
- 16.
张晓东,张晓东,程晓东。蛋白质结构域蛋白超家族:蛋白质赖氨酸甲基转移酶。基因组医学杂志。2005;6(8):227。
- 17.
Jenuwein T,Allis CD。翻译组型代码。科学。2001; 293(5532):1074-80。
- 18.
英Z,穆里根RM,詹尼N,Houtz RL。Rubisco小和大SubunitN甲基转移双和单功能是甲基化Rubisco酶的小和大亚基甲基转移酶。J Biol Chem。1999; 274(51):36750-6。
- 19.
复杂的进化历史和不同的结构域组织表明不同的调控相互作用。新植醇。2012;195(1):248 - 63。
- 20.
吴DW,王T,Chandrasekharan MB,阿拉马约R,Kertbundit S,霍尔TC。植物含有SET结构域的蛋白:结构,功能和调节。生物化学生物生物物理学学报。2007; 1769(5):316-29。
- 21.
Finn RD, Coggill P, Eberhardt RY, Eddy SR, Mistry J, Mitchell AL, Potter SC, Punta M, Qureshi M, Sangrador-Vegas a . Pfam蛋白家族数据库:迈向更可持续的未来。核酸Res. 2016;44(D1): D279-85。
- 22.
MEGA7:用于更大数据集的分子进化遗传学分析7.0版本。生物化学学报。2016;33(7):1870-4。
- 23.
曹锐,王磊,王海龙,夏磊,王海龙。组蛋白H3 -赖氨酸27甲基化在多冠基因沉默中的作用。科学。2002;298(5595):1039 - 43。
- 24.
Zhu X,Chen C,Wang B.完全测序陆地植物中TRX设定基因的系统发育及演化。基因组。2012; 55(4):269-80。
- 25。
GYF结构域是一种新型结构褶皱,通过富含脯氨酸的序列参与淋巴样信号传导。中国生物医学工程学报。1999;6(7):656-60。
- 26。
Arita K,Ariyoshi M,Tochio H,Nakamura Y,Shirakawa M.通过碱性翻转机构通过SRA蛋白UHRF1识别Hemi-甲基化DNA。自然。2008; 455(7214):818-21。
- 27。
朱晓霞,马洪,陈志强。陆生植物Su (var) 3-9 SET基因的系统发育与进化:结构和功能的快速多样性。BMC Evol Biol. 2011;11(1):63。
- 28.
Garbarini n,Delpire E.与Myc(PAM)相关的蛋白质的RCC1结构域与并调节KCC2。细胞生物学生物化学。2008; 22(1-4):031-44。
- 29.
Huq E, Quail PH. PIF4是一种与光敏色素相互作用的bHLH因子,在拟南芥中起着光敏色素B信号转导的负调控作用。EMBO j . 2002; 21(10): 2441 - 50。
- 30.
韩志强,王志强,王志强。CAZy以外的植物糖基转移酶:对DUF家族的展望。植物学报2012;3
- 31.
Trievel RC, Flynn EM, Houtz RL, Hurley JH。SET结构域酶Rubisco LSMT多重赖氨酸甲基化的机制。中国生物医学工程学报。2003;10(7):545-52。
- 32.
Trevel RC,Beach BM,Dirk LM,Houtz RL,Hurley JH:设定结构域甲基转移酶的结构和催化机制。电池2002,111(1):91-103。
- 33.
Yadav CB,Muthamilarasan M,AnandIni SS,Prasad M.在Foxtail Millet中的集结构域基因系列综合分析识别SISET14在非生物胁迫耐受性的推定作用。SCI REP。2016; 6
- 34.
王志强,王志强,王志强。KRYPTONITE组蛋白H3甲基转移酶对CpNpG DNA甲基化的控制大自然。2002;416(6880):556 - 60。
致谢
作者希望感谢Chhavi Dawar和Divya Tej Sowpati从Cellular和Molecular Biology(CSIR),海德拉巴,印度,为帮助计算工作。
资金
ML非常感谢生物技术(DBT),GOVT。印度和CSIR-CCMB进行财政援助。SS承认DBT印度提供博士后研究奖学金。
数据和材料的可用性
支持本文结论的数据集包含在其附加文件中。
作者信息
从属关系
贡献
SS收集数据,进行生物信息学分析,并起草手稿。ML构思并指导了这项研究,起草并修改了手稿。两位作者都阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
附加文件1:表S1。
本研究中考虑的物种列表,具有含有蛋白质的相应组域。(PDF 10 KB)
附加文件2:表S2。
本研究考虑的物种中含有SET结构域的蛋白质及其序列id的列表。(PDF 65 kb)
附加文件3:图S1。
来自e(z),灰,灰,Trx和su(var)的251设定结构域蛋白序列的多序列对准。(PDF 74 KB)
附加文件4:图S2。
在隐花植物谱系中介绍Ash SET蛋白的结构域。黑色箭头表示在特别提到的古原生质体物种中引入了指定的域。(PDF 212 kb)
附加文件5:图S3。
在植物谱系中引入TRX设定蛋白中的结构域。黑色箭头表示在特别提到的古原生质体物种中引入了指定的域。(PDF 221 KB)
附加文件6:图S4。
在植物谱系中引入SU(var)蛋白质中的结构域。黑色箭头表示在特别提到的古原生质体物种中引入了指定的域。(PDF 85 KB)
附加文件7:图S5。
孤儿蛋白结构域的示意图。(PDF 550 kb)
附加文件8:图S6。
A) SETD类和B) TPR类的领域架构。(PDF 507 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
陈志刚,陈志刚,陈志刚,等。古原生质体中SET结构域蛋白的系统发育关系及结构域结构。BMC植物BIOL.17,238(2017)。https://doi.org/10.1186/s12870-017-1177-1
收到了:
接受:
发表:
关键字
- Archaeplastida
- 组蛋白修饰
- 表观遗传学
- 设置域
- Polycomb
- 系统发育分析
- 进化