摘要
背景
蕨类植物是维管植物中未被开发的大型类群(约11000种)。目前可获得的基因组数据包括6个物种的低覆盖核基因组序列和部分线粒体基因组序列和一些质体基因组。
结果
我们描述了三种植物的质体基因组粗茎鳞毛,是最大的蕨类属之一,利用叶绿体DNA富集样本的测序,并与蕨类植物中物种最丰富的Polypodiales的可用质体进行比较分析。我们也测序了质体Adianthum hispidulum(Pteridaceae).出乎意料的是,我们在红外区发现了高变异性,包括重复rrn16在d . blanfordii,完全丧失trnI高斯在d . filix-mas的外显子缺失导致的假基因化d . blanfordii。对以往报道的水龙足科植物质体的分析表明Woodwardia unigemmata而且Lepisorus clathratus在红外区有不寻常的插入。这些插入区域的序列与Polypodiales以外蕨类植物的几个LSC片段和间隔具有高度的相似性tRNA-CGA而且tRNA-TTT线粒体基因组的基因Asplenium病灶.我们认为,这反映了古老的DNA从线粒体转移到质体基因组发生在一个共同的蕨类祖先。我们测定了水龙足科植物IRs中基因含量的显著保守性和基因间间隔的相对进化速率。4个基因间区进化较快(trnA——或者f42,rrn16-rps12, rps7-psbA而且ycf2-trnN).
结论
Polypodiales plasomes的IRs是动态的,由基因丢失、复制和推测的线粒体横向转移等事件驱动。
背景
Сhloroplast陆地植物的基因组(质体)在大小、基因含量和顺序上一般是保守的。叶绿体和线粒体的进化起源可以追溯到古代的内共生细菌,这种细菌大大减少了基因组,只包含祖先基因的一小部分[1,2].
植物质体粒具有四部分结构,由大的单拷贝区(LSC)和小的单拷贝区(SSC)组成,由两部分倒置重复序列(IR)分割[2,3.].高等植物的质体体长度通常在150000 bp左右,由大约120-130个基因组成,其中约75个基因编码光系统I和光系统II的蛋白质,以及与光合作用有关的其他蛋白质[参见,例如,[4]],而其他基因则编码核糖体RNA和蛋白质并转移RNA。陆地植物的质体的基因顺序和含量偏差最大的是在非光合作用物种中发现的,其中发现了极度减少的质体-高达11 Kbp [5].然而,在光合植物中也观察到特定基因的丢失,以及改变基因顺序的易位[6,7,8].
IRs通常被认为是质体中最稳定的部分。事实上,与单个复制区域相比,IR序列的替换率是最低的[3.];没有IRs的质体体(如豆科的IRLC分支)表现出较高的替代率[9反之亦然,从SC转移到IR的基因减缓了它们的替代率[10].IRs的大小一般在15至30 kbp之间,包含一组核心基因,由4个rRNA基因(4.5秒,5 s,16 s而且23 s rRNA)、tRNA基因(trnAUG,trnI-GAU,trnN-GUU,trnR-ACG而且trnV-GAC).许多陆生植物的IRs也含有许多其他基因,这是系系特异性扩张和收缩的结果[3.].很少有进化谱系表现出IR的大规模扩张(超过几个kbp并包含许多基因)[3.,11].特别是,几个重叠的倒置影响了蕨类植物中IR的大小、基因含量和顺序,这一分支包括大多数蕨类物种[8,10].
另一个影响质体结构的进化事件是水平基因转移(HGT)。核、线粒体和质体之间的HGT发生率很高,通过基因的重新定位和重组,对植物基因组进化有重要贡献,从而促进了遗传多样性。DNA片段从线粒体或质体转移到细胞核是最常见的报道[12,13,14,15,16].线粒体基因组也经常被质体衍生序列入侵;来自核基因组的DNA也出现在许多开花植物的谱系中[17,18,19,20.,21,22,23,24和蕨类植物[25].线粒体dna片段转位到质体基因组是非常罕见的。目前已知的病例只有两例,均来自开花植物。在胡萝卜胡萝卜(订单Apiales(蜂科)质体具有约1.5 Kb的区域,具有高度的相似性葡萄(订单至关重要的)发现线粒体序列[26].这个区域不包含任何典型的质体基因。描述的胡萝卜属丝裂基因组与其他质体的筛选伞形科提示它是从该属的一个共同祖先的线粒体基因组插入到质体上的胡萝卜属[27].另一个例子是水平基因转移是一个2.4 kb的线粒体DNA片段进入rps2-rpoC2植物质体的基因间间隔Asclepias syriaca(夹竹桃科) [28].因此,与受质体和核序列插入影响的线粒体基因组不同,开花植物的质体很少从其他细胞间隔的DNA转移中获益[22,29,30.].
现存的蕨类植物是一种无种子维管植物,目前已知有45科。280属),其中一半以上属于水螅目,符合[31].大部分的水足纲物种可分为两个姐妹枝-正足类I和正足类II,其余的为科Pteridaceae,姬蕨科,Saccolomataceae,Lindsaeaceae(基底进化枝)32].沃尔夫等人。8]首次研究了几个分布广泛的代表性蕨类类群的质体结构,但还没有人研究过水螅足科。朱等人。[3.]分析了陆生植物IR边界的演化速率和变化,包括7种蕨类植物,但没有考虑水螅足类植物。拉曼等人[33)的特点Cyrtomium falcatum质体基因组与同属种有一定差异c . devexiscapulaetRNA基因含量和起始密码子。在24个蕨类植物样本中,其中5个为水龙蹄目(其余为现存的10个目),该地区的部分(rpoB-psbZ)进行了分析,发现遥远物种(属于不同目)的基因组发生了相当大的变化[34].通过比较几个分类学范围内的蕨类植物质体组(包括番茄植物科、裸叶蕨科、Equisetopsida、Marattiopsida和Polypodiopsida),发现一些谱系经历了多种红外变化,包括扩展和反转,而另一些谱系则表现出停滞[35].近年来,许多蕨类植物质体的新序列(包括Polypodiales种)被公布。然而,相应的研究只报道了这些序列的系统发育分析结果,没有详细分析其基因含量和结构[36].
这表明蕨类植物质体结构的多样性研究还不够充分。在此前提下,我们从水螅属植物中鉴定了另外四个质体序列,其中三个来自水螅属植物粗茎鳞毛和一个来自Adianthum,并对所有蕨类植物质体进行了比较分析。
结果
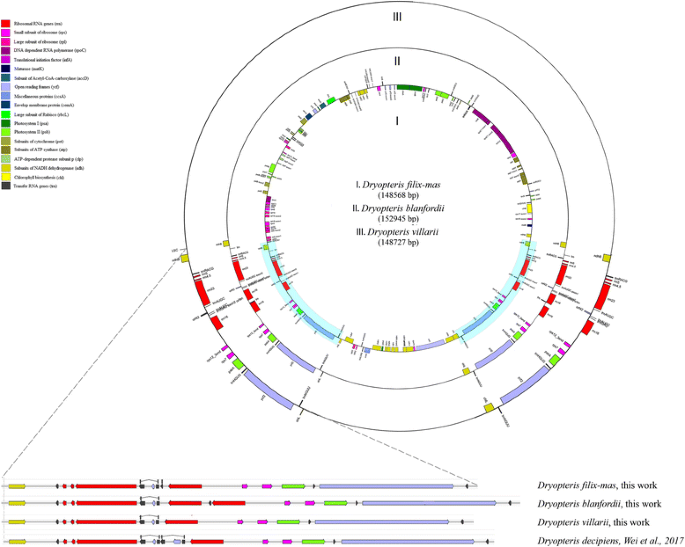
我们对三种eupolypod I的新质体序列进行了测序和组装:粗茎鳞毛filix-mas,粗茎鳞毛blanfordii而且粗茎鳞毛villarii.质体具有典型的四边形结构,大小相似(148,568,分别为152,945和148,727 bp)。共对130个基因进行了doga注释d . filix-mas其中蛋白质编码基因91个(其中5个在IRs中重复),tRNA基因25个(其中5个在IRs中重复),rRNA基因4-5个(全部在IRs中重复)。质体系的粗茎鳞毛物种,包括D. blanfordii, D. villarii, D. filix-mas和之前报道d . decipiensLSC和SSC基因含量相同,但IRs不同(图1)。1).
我们还对一个质体进行了测序Adianthum hispidulum,属蕨科,与正多足纲植物相对的基群.完整的质体序列答:hispidulum长度为151327 bp,由LSC (83,188 bp)、SSC (21,459 bp)和IRs (23,340 bp)组成。叶绿体中的基因含量答:hispidulum与同属种略有不同答:capillus-veneris(发表在37),在答:hispidulum trnT-UGU基因(定位于IR)完全缺失答:capillus-veneris它由一个假基因表示(图5)。2).
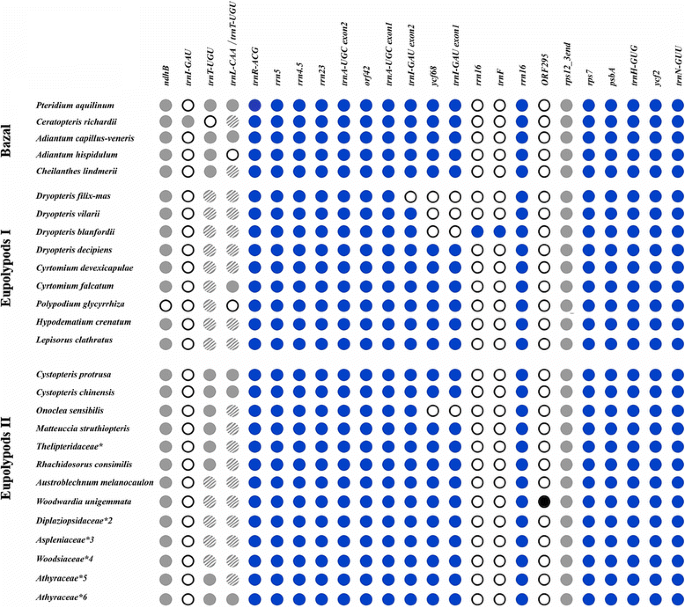
蕨类水杨属植物的IR基因含量。填充的蓝色圆圈和打开的蓝色圆圈分别表示在相应物种中存在和不存在的基因。浅灰色填充的圆圈表示被DOGMA标记为假基因的序列,阴影浅灰色的圆圈表示没有被DOGMA发现的假基因,仅通过使用BLAST进行手动检查。* - - - - - -Macrothelypteris torresiana,Thelypteris aurita,Stegnogramma sagittifolia,Cyclosorus降眉间肌,Ampelopteris prolifera;* 2 -Homalosorus pycnocarpos,Diplaziopsis是,Diplaziopsis cavaleria;* 3 -Asplenium prolongatum,Asplenium pekinense* 4 -Woodsia macrochlaena,Woodsia polystichoides;* 5 -Conopteris opaca(syn。Athyrium opacum)和甲壳raceae的所有Deparia种,*6 -Anisocampium sheareri(syn。Athyrium sheareri)及其他Athyrium种Athyraceae
红外序列比较数据集包括公共序列数据库中所有的polypodiaceae类的质体组,我们的新数据包括45个序列:31个属于eupolypod I, 9个属于eupolypod II,其余5个属于Pteridaceae或Dennstaedtiaceae。我们对已发表的Polypodiales plasomes进行了重新注释。所有物种的IR/SSC边界都位于ndhF基因和IR/LSC -内ndhB基因。类似的IR边界以前被定义为许多单生植物类群(Psilotales, ophiolossales, Equisetales, Marattiales和Polypodiopsida) [35].通常,Polypodiales的叶绿体基因组携带高度相似的序列(~ 246 bp)ycf68ORF在IR区域,尽管他们之前没有被作者注释。的ycf68是否有功能基因位于trnI-GAU内含子,存在于许多陆生植物叶绿体基因组中,但由于其功能尚不清楚,常常未被注释[38,39].另一个假定的基因,ORF42,在trnI-GAU内含子的所有物种包括在分析中。ORF42之前在?的内含子区域发现的trnA-UGC例如,一些开花植物的质体基因,Veratrum patulumo .卫矛。(Melanthiaceae) [40),天竺葵属植物×hortorum柳青(香叶科)[11].具有高度相似性的序列ORF42是在线粒体基因组中发现的菜豆;可能是由于质体到线粒体的横向基因转移[19].
Polypodiales的红外结构如图所示。2和额外的文件1.基因的数量通常从14到16不等。Polypodiales的质体质体在IR范围内主要积累了0 ~ ~ 3kbp和~7 ~ ~11,5 Kbp两个区域的基因数量变异性。这些区域也表现出较低的序列相似性。另外两个区域(~3 ~ ~ 7kbp和~11,5 Kbp ~ IR结束)在基因数量和序列上非常保守。
0 ~ ~ 3kbp区域含有tRNA(伪)基因(图1)。2).虽然它被注释为功能双外显子trnT-UGU在许多物种(如33,37)的tRNA基因中,tRNAscan-SE对其结构的预测并不支持其功能。在其他物种中,它被注释为假基因(trnT-UGU或trnL-CAA)或完全丢失。然而,人工检查显示假基因在所有被分析的物种中都存在。2)除了Adianthum hispidulum在那里它完全消失了。
在~7 ~ ~11,5 Kbp可变区有3个基因出现部分或全部缺失或复制(trnI-GAU,ycf68, rrn16)(图。2)(额外的文件1).一些种类的水螅类已经部分或完全消失了trnI-GAU而且ycf68的内含子trnI-GAU),即:o . sensibilis四种中的三种粗茎鳞毛物种(D. blanfordii, D. villarii, D. filix-mas,科)。在d . filix-mas trnI-GAU而且ycf68完全失去了。d . blanfordii部分失去了trnI-GAU基因(内含子和一个外显子被删除),但有复制的大部分rrn16基因。d . villarii失去内含子和一个外显子trnI-GAU但是没有rrn16重复。由于删除/重复的结果,IR的大小粗茎鳞毛物种变化:IR的d . filix-mas而且d . villarii是否比1450-1570 bp短d . blanfordii相反,IRs长647 bp (Cyrtomium以物种为参考)。令人惊讶的是d . decipiens报告(36,在该区域没有删除.o . sensibilis(蛇麻科)质体有一个缺失ycf68-trnI-GAU地区。Onoclea而且粗茎鳞毛物种属于不同的进化支:真多足类II和真多足类iycf68-trnI-GAU区域是独立的事件。
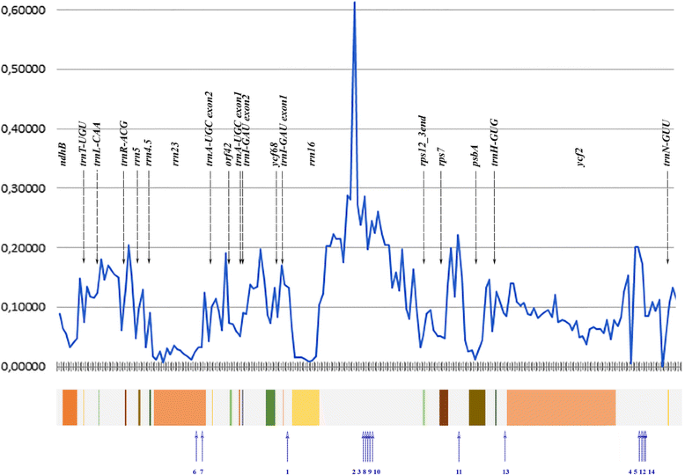
由于缺失和重复,Polypodiales的IR大小从22 Kbp不等(Ceratopteris richardii[KM052729])至26,9 Kbp (Cystopteris protrusa,[KP136830]。特别是,我们在上述高度可变的基因间间隔(intergene 14)中发现了大部分的大型插入(370 bp以上)rrn16而且rps12基因间(intergene 16)rps7而且psbA)和基因间19(在ycf2而且trnN-GUU),见图。3..插入的长度、定位和与已知的高等植物序列的相似性见表1.
水龙足科植物IRs的核苷酸多样性及其插入位置。横轴为所有水螅属植物的IRs内坐标。纵轴表示p-distance的滑动窗口分析(窗口= 200 b.p.),即由Masafumi Nozawa编写的perl脚本计算的序列之间每个位点的核苷酸差异的比例。下面的彩色框标记了基因的编码区域(在平均Polypodiales的IR中)。蓝色编号的箭头表示不同蕨类植物的红外序列中插入的位置(另见表)2)
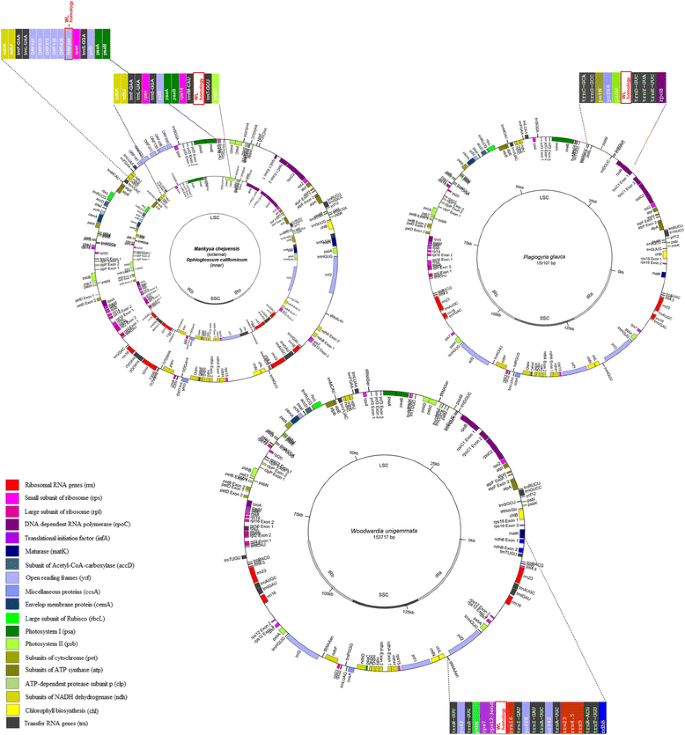
发现了一个不寻常的1663 bp插入rrn16而且rps12基因的Woodwardia unigemmata(坐标93404…95067)。w . unigemmata蕨类植物是家族的吗Blechnaceae(eupolypod II),其质体组由Lu等人测序,2015 [41],之前没有在这个区域注释过基因。大部分的w . unigemmata插入与的插入具有高度的序列相似性Lepisorus clathratus(水龙科,真波科I)位于同一区域的IR,之间rrn16而且rps12基因。约1160 bpw . unigemmata而且l . clathratus插入之间表现出65-78%的相似性。在其他水脚科植物的红外光谱中,除在水脚科植物中有少量(约150bp)的序列外,未发现类似的序列Matteuccia struthiopteris(Onocleaceae, eupolypod II),有62-66%的相似性l . clathratus而且w . unigemmata插入(进一步称为WL-sequences)。
令人惊讶的是,类似wl的序列竟然出现在距离很远的蕨类植物分类学类群的LSC区域,在Polypodiales之外2).首先,WL序列中有小部分与LSC质体片段相似Plagiogyria物种(Plagiogyria是独型科草蕨科的一个属,例如参见«中国植物志»[42])。更准确地说,WL序列的772 bp部分与该区域具有67%的同一性p . glauca质体系(坐标29128…29895, KP136831)和p .粳稻质体(部分序列,座标4503-5273,HQ658099)。在两个物种中都存在与WL同源的序列trnD-GUC-psbM基因间的间隔。其他蕨类植物在LSC中也含有wl样序列Ophioglossum californicum(KC117178) [34),Mankyua chejuensis(KP205433)。这两个物种都属于蛇舌科(基础蕨类);它们远离Polypodiales(核心细囊蕨类植物)和Cyatheales。蛇舌蕨中与WL序列相似的片段长度约为255 ~ 275 bp,分布于LSC的不同区域。在o . californicum(KC117178)发现于基因间间隔trnT-GGU而且trnfM-CAU的基因,m . chejuensis(KP205433, JF343520)它也位于LSC区域,但在其他基因间间隔(之间)trnL-UAA而且rps4).它被注释为ORF295(无花果。4).综上所述,这表明在蕨类质体的进化过程中存在着从LSC到IR(或反之)的DNA片段易位。
有趣的是,这部分WL序列与约5,9 Kb的基因间间隔203-bp片段具有较高的一致性(67%)tRNA-CGA而且tRNA-TTT线粒体基因组的基因Asplenium病灶(部分序列,坐标3188…2986,AM6006412).Asplenium是一个属,属于Polypodiales - eupolypod II的同一分支Woodwardia而是在另一个科,无脾科。我们对犹他州立大学知识库中可用的蕨类线粒体contigs进行了wl序列相似性搜索[45;http://digitalcommons.usu.edu/fern_genome/].在Plagiogyria formosana- 244bp(恒等式72%,contig 439,坐标5336-5585)和277 bp(恒等式72%,contig 93,坐标1-277)。另一种蕨类植物(d . conjugata,Pteridium aquilinum,c . richardii,多桅草).
我们估计了Polypodiales IR的基因和基因间间隔的相对进化速率。对于蛋白质编码基因,我们使用分支位点模型分析dN/dS,对于非编码区域使用两种模型:同质替代参数和非同质替代参数(详见材料和方法)。我们发现,对于四个非编码(基因间)区域,该模型对不同分支具有不同的替代参数,与基于不同谱系中相同替代参数假设的模型相比,具有显著的高似然(LRT)(见表)3.).所有这些区域都是间隔:间隔trnA-UGC而且或f42 (intergene8)之间rrn16而且rps12(intergene14),之间的rps7而且psbA(intergene16)和最后一个之间ycf2而且trnN-GUU(intergene19)。
讨论
与以往对蕨类植物红外区稳定性的观察相比,我们发现蕨类植物的红外序列和基因含量具有较高的变异性。有两个高变区,一个位于IR的开始,0 - 3kb,另一个是7 - 11kb。这些区域是独立发生在不同演化支中的类似进化变化的主体。大多数物种的第一个区域含有tRNA假基因。真多足类I和真多足类II的成员都表现出独立的缺失trnI-GAU-ycf68地区(即。粗茎鳞毛而且Onoclea).Polypodiales(连同Salviniales和Cyatheales)属于一个被称为核心细囊类的分支[43].它们的质体与真囊蕨类植物的质体有很大的不同(裸叶蕨类,蛇舌蕨类,马齿苋类,马齿苋类)[8,33].然而,需要注意的是,由于tRNA基因注释的不确定性,蕨类植物质体的比较分析受到了干扰。这尤其涉及到内含子trnT-UGU,这是在IR(之间ndhB而且trnR-ACG)的几种蕨类质体[37,43,44,被认为是核心钩端囊状血管的特殊特征。但intron-containingtrnT-UGU在其他蕨类植物谱系中没有发现[45,46或在蕨类植物以外的任何其他植物中;只有intronlesstrnT-UGU存在.这是不寻常的,因为与蛋白质编码基因相比,质体tRNA基因具有高度保守的外显子-内含子结构。Gao及其同事认为,在蕨类植物的进化过程中,tRNA基因可能会反复独立丢失,并可能丢失trnT-UGU是那些事件之一[45].我们的分析包括人工重新注释和使用tRNA预测程序tRNAscan-SE检查,但不支持含有内含子的ir定位功能trnT-UGU在任何有报告的水螅属植物中。相反,我们发现trnT-UGU在几乎所有的水脚属植物的IR中发现了假基因。最有可能的是,这个假基因很难被识别,因此自动注释的结果可能被解释为基因丢失。值得注意的是,高和他的同事[45比较了假定含有内含子和不含内含子的序列trnT-UGU可以看出前者具有异常的发散性,远远高于功能性tRNA基因的预期。我们的结论是含有内含子的ir定位trnT-UGU是人工制品引起的自动注释的缺点。此外,该伪基因的两个部分(“外显子”)可以被自动化注释程序识别为不同的tRNAs -,如dogatrnT-UGU而且trnL-CAA.
在第二个高变量区域,7 - 11kb,我们发现一个不寻常的插入(wl序列)在两个不相关的Polypodiales -Woodwartia unigemmata而且Lepisorus clathrathus.在同一区域发现了与wl序列高度相似的小插入Mattheucia sthruttiopteris.此外,与wl序列高度相似的插入也出现在蛇舌科植物(与水脚科植物相距较远)的质体组中,但位置不同——位于LSC区域。
该序列与线粒体基因组区域具有较高的相似性Asplenium病灶.这有两种可能的解释:要么是整合在质体组中的线粒体起源序列,要么是整合到线粒体基因组中并从大多数蕨类植物的质体基因组中丢失的质体起源序列w . unigemmata而且l . clathrathus.由于没有蕨类植物完整的线粒体基因组序列,到目前为止我们还无法得出这两种假设中哪一种是正确的结论。
无论如何,这是线粒体和质体之间横向基因组片段转移的结果。横向基因组片段转移是在原核和真核生物中常见的一种现象。在植物中,一个细胞间隔内的三个基因组(线粒体、叶绿体和细胞核)的存在导致了不同类型的细胞内基因组片段交换:细胞器和细胞核之间,线粒体和叶绿体之间,双向的[47].遗传物质从细胞器转移到细胞核似乎是原核生物祖先基因组减少的一个持续的进化过程[48,49].许多报告断言,与叶绿体和核基因组相比,植物线粒体基因组异常容易发生外来序列的渗入[47,50].关于蕨类植物线粒体的资料很少。没有完整的线粒体基因组组合,只有部分。[检测到多个与质体DNA序列高度相似的区域51,但它们与6种蕨类植物的总基因组组的质体序列没有关联Dipteris conjugata(Gleicheniales),Plagiogyria formosana(Cyatheales),Pteridium aquilinum(姬蕨科),Ceratopteris richardii(Pteridaceae),Polypodium甘草(eupolypods)和Cystopteris protrusa(eupolypods)。作者推测,质体样序列存在于细胞核或线粒体基因组中[42].假设这是事实,这意味着在蕨类植物的进化中,细胞器基因组片段的水平转移并不罕见。
结论
本文研究了蕨类植物Polypodiales蕨类植物质体IRs的结构和进化稳定性。IRs的两个区域是高度可变的:(i)之间的序列ndhB而且trnR-ACG基因(~ 3kbp)和(ii)包含rrn16基因和侧翼邻近区(~4,5 Kbp)。这种闪烁的trnI-CAU,trnT-UGU,ndhB而且rps12, trnI-GAU, ycf68, rn16在不同的物种中观察到与这些区域相关的基因。三个的质体粗茎鳞毛物种表现出trnI-GAU消除的动态过程rrn16重复。
二水螅目种-w . unigemmata而且l . clathratus-在红外区域有不寻常的序列。它与LSC垫片相似trnL-rps4瓶尔小草目和pbsM-trnD的部分线粒体基因组Asplenium(真蕨科)。我们认为这些特征是蕨类植物进化过程中质体内重排以及叶绿体和线粒体基因组之间转移的结果。
方法
植物材料
两者的成熟叶粗茎鳞毛filix-mas (l)Schott,粗茎鳞毛blanfordii(C. Hope) C. Christensen和粗茎鳞毛villarii (Bellardi)Woyn。Schinz和Thell的标本来自莫斯科国立大学植物园的室外部分。
粗茎鳞毛filix-mas(l)Schott是俄罗斯森林中常见的蕨类物种,因此该标本的采集地点仅被大致描述为“在莫斯科附近”。
粗茎鳞毛blanfordii(C.希望)C.克里斯滕森长大了云杉或冷杉属中国(甘肃、四川、西藏、云南)、阿富汗、印度、克什米尔、尼泊尔和巴基斯坦海拔2900-3500米的森林[52,53,54].这种植物的母体是2003年在印度采集的。孢子在莫斯科国立大学植物园温室的人工条件下萌发。然后将发育好的孢子体移植到植物园的室外部分。
粗茎鳞毛villarii (Bellardi)Woyn。来自Schinz & Thell.-亚高山种,生长于丘陵、石灰岩悬崖的露头,包括中欧和南欧的高山[55].孢子由Zürich植物园种子部提供(采集于瑞士联邦自然栖息地),于2013-2017年在莫斯科国立大学植物园的小温室中萌发和生长。
铁线蕨hispidulum西南。泛热带,古热带种,分布于非洲东部经印度南部、泰国和锡兰到太平洋岛屿、波利尼西亚、新西兰和大洋洲[56,57,58,59].的成年叶铁线蕨hispidulum采自莫斯科国立大学植物园温室。标本保存于莫斯科国立大学生物系植物标本室。
叶绿体基因组测序,从头组装和注释
利用Illumina MiSeq高通量测序平台对叶绿体DNA (cpDNA)进行测序。作为样品准备,成年活植物从莫斯科国立大学植物园收集。使用cpDNA提取协议从2,6 g(鲜重)叶中提取cpDNA [60,61进行小的修改:用蒸馏水清洗后,在+4℃(Tris-HCl (pH 8,0) 50 mM, EDTA 7 mM, 1% PVP-40, NaCl 1,25 M,抗坏血酸0,25 M,甲亚硫酸钠10 mM,硼砂0.0124 M)将叶片在35 ml分离缓冲液中均匀化,并用软抹布过滤匀浆。均匀液在200 g下4℃离心15分钟(丢弃细胞壁碎片),在1000 g下+4℃离心20分钟(丢弃沉淀物),最后在2000 g下+4℃离心20分钟。在后一种情况下,沉淀被重新悬浮在3ml的洗涤缓冲液中(Tris-HCl (pH 8,0) 50mm, EDTA 25mm),并小心地装入15 ml的蔗糖梯度管中,其中包含7ml 52%的蔗糖洗涤缓冲液,并覆盖4ml 52%的蔗糖洗涤缓冲液。将有样品和蔗糖梯度的试管在4℃下3500 g离心60分钟。收集52% - 30%蔗糖(约1ml)的界面,以12000 g离心。重悬于900 μl洗涤缓冲液中,加入100 μl 10% CTAB裂解1 h, 55°C。然后使用[描述的协议]进行DNA纯化步骤。62].
使用TruSeq协议(NEBNext®DNA文库准备主混合集,用于Illumina, E6040, NEB试剂)制备基因组文库。我们制作PE序列(2 × 300 bp.),每个库的两倍数约为1.2-1.97 m。63],使用RefSeq数据库和Bowtie2中的13个完整和5个部分蕨类叶绿体基因组序列对测序reads进行筛选[64].然后使用Velvet Assembler为过滤的和未过滤的读取集生成两个contig集[65]及MIRA4 [66].如果组装好的片段和支架显示出与已发表的蕨类叶绿体基因组相似,则选择它们进行下一个组装。最终的从头组装是通过几个迭代步骤完成的。草稿顺序由PE读取映射手动更正。
我们获得了由大单拷贝区(LSC)、小单拷贝区(SSC)和两个反向重复区(IR)组成的完整循环叶绿体基因组的reads。最后,将初始读取的映射执行到程序集,以检查潜在的程序集工件。组装的叶绿体基因组中的蛋白质编码基因注释用DOGMA进行注释[67].使用Bowtie2、VarScan (v.2.3.7)和SAMtools/BCFtools软件包进行读取和变量调用的映射[64,68,69].
叶绿体基因组分析
本研究纳入序列的Genbank或ENA登录号列于表中2.
将本研究测序的物种与先前报道的物种进行叶绿体全基因组分析。从基因库中下载了9个质体。已分析的物种的完整列表可在表中找到1.首先,用Kalign (www.ebi.ac.uk /工具/ msa / kalign).系统发育分析采用Mega 6.0最大似然(ML)方法进行[70].用mVista web-tool (http://genome.lbl.gov/vista/mvista/submit.shtml).
对于IRs的每个区域(分别为基因和基因间区域)以及所有编码序列的连接物的进化速率分析,使用MUSCLE [71].使用串联对齐构建了ML树。使用phangorn包中的modelTest函数选择BIC评分最低的替代模型[72].利用核苷酸取代矩阵、gamma、不变位点比例和gamma分布参数对树拓扑结构进行优化。对于非蛋白编码区域,树枝长度由两个模型计算(均质替代参数- nhomo = 1,非均质- nhomo = 4),使用baseml [73],然后采用LRT对模型进行比较。对于蛋白质编码区域,使用PAML包中的codeml估计每个基因的dN和dS [73],通过分支和M0模型估计各谱系的dN/dS比值。baseml和codeml分析都使用phagorn连接树与最近邻交换。使用猿包中的baseml/codeml树计算距离矩阵[74].然后计算每个区域(编码和非编码)的相对进化速率,使用ERaBLE [75].p-distance的滑动窗口分析(窗口= 200 b.p.),即序列之间每个位点的核苷酸差异比例,由Masafumi Nozawa编写的perl脚本计算[76].
参考文献
- 1.
灰色兆瓦。细胞器基因组的起源和进化。中华医学会论文集1993;3(6):884-90。
- 2.
Olejniczak SA, Lojewska E, Kowalczyk T, Sakowicz T.叶绿体:质体测序的研究现状和实际应用。足底。2016;244(3):517 - 27所示。
- 3.
朱安,郭伟,古普塔S,范伟,Mower JP。质体倒置重复的进化动力学:扩张、收缩和损失对替代率的影响。新植醇。2016;209(4):1747 - 56。
- 4.
张文华,林春春,于敏,常文杰。叶绿体基因组:多样性、进化和在基因工程中的应用。基因组医学杂志。2016;17(1):134。
- 5.
在内寄生虫属(apoodanthaceae)的两个物种的质体组中,每个都只保留了5或6个可能的功能基因。中国生物学报2015;8(1):189-201。
- 6.
Haberle RC, Fourcade HM, Boore JL, Jansen RK。青色Trachelium caeruleum叶绿体基因组的大量重排与重复序列和tRNA基因有关。中华分子生物学杂志2008;66(4):350-61。
- 7.
Guisinger MM, Kuehl JV, Boore JL, Jansen RK。香叶科被子植物质体基因组的极端重组:重排、重复和密码子的使用。中华分子生物学杂志2011;28(1):583-600。
- 8.
沃尔夫PG,罗珀JM,达菲AM。蕨类植物叶绿体基因组结构的演化。基因组。2010;53(9):731 - 8。
- 9.
佩里AS,沃尔夫KH。豆科植物叶绿体DNA中的核苷酸取代率取决于反向重复的存在。中华分子生物学杂志2002;55(5):501-8。
- 10.
李富伟,郭丽丽,Pryer KM, Rothfels CJ。转位到质体倒置重复的基因取代率降低,GC含量升高。基因组生物学学报2016;8(8):2452-8。
- 11.
Chumley TW, Palmer JD, Mower JP, Fourcade HM, Calie PJ, Boore JL, Jansen RK。天竺葵(pelargonium x hortorum)叶绿体全基因组序列:陆地植物最大、高度重排的叶绿体基因组的组织和进化。分子生物学与生物学报2006;23(11):2175-90。
- 12.
Gantt JS, Baldauf SL, Calie PJ, Weeden NF, Palmer JD。rpl22向细胞核的转移大大早于它从叶绿体中丢失,并涉及到内含子的获得。EMBO j . 1991; 10(10): 3073 - 8。
- 13.
赖斯DW,帕尔默JD。质体中一个异常的水平基因转移:由一个遥远的细菌同源基因替换,并有证据表明,裸生植物和隐生植物质体是姐妹。BMC医学杂志。2006;31。
- 14.
DNA的交换与获取:植物中的水平基因转移。植物学报,2010;15(1):11-22。
- 15.
将有限转移窗口假说推广到细胞器间DNA迁移。基因组生物学学报2011;3:743-8。
- 16.
李文华,李文华。非绿色菱形科植物质体基因组还原过程中的细胞内基因转移。新植醇。2016;210(2):680 - 93。
- 17.
Bergthorsson U, Adams KL, Thomason B, Palmer JD。开花植物线粒体基因的广泛水平转移。自然。2003;424(6945):197 - 201。
- 18.
Bergthorsson U, Richardson AO, Young GJ, Goertzen LR, Palmer JD。从不同陆生植物供体的线粒体基因大量水平转移到基底被子植物amborela。中国生物医学工程学报2004;29(5):559 - 561。
- 19.
Woloszynska M, Bocer T, Mackiewicz P, Janska H.一个叶绿体DNA片段可能从非eudicots水平转移到Phaseolus线粒体基因组。植物生物学杂志2004;56(5):811-20。
- 20.
公共科学图书馆麝猫。2013;9 (2):e1003265。
- 21.
Hepburn NJ, Schmidt DW, Mower JP。三叶木兰线粒体cox2基因中两个内含子的丢失暗示着基因水平转移和基因转换是内含子丢失的一种新的机制。中华分子生物学杂志2012;29(10):3111-20。
- 22.
Rice DW, Alverson AJ, Richardson AO, Young GJ, Sanchez-Puerta MV, Munzinger J, Barry K, Boore JL, Zhang Y, dePamphilis CW,等。在被子植物amborrela中通过线粒体融合的整个基因组的水平转移。科学。2013;342(6165):1468 - 73。
- 23.
Park S, Grewe F, Zhu A, Ruhlman TA, Sabir J, Mower JP, Jansen RK。通过多个水平和细胞内基因转移的天竺葵线粒体基因组的动态进化。新植醇。2015;208(2):570 - 83。
- 24.
王东,吴艳伟,史ac,吴春春,王茵,周思明。叶绿体基因组DNA向线粒体基因组转移至少发生在300 MYA。分子生物学与生物学报2007;24(9):2040-8。
- 25.
戴维斯CC,安德森WR,沃达克KJ。从寄生开花植物到蕨类植物的基因转移。中国生物医学工程学报,2005;32(5):447 - 447。
- 26.
葡萄的线粒体DNA和横向基因转移猖獗问题。分子生物学与生物学报2009;26(1):99-110。
- 27.
Iorizzo M, Grzebelus D, Senalik D, Szklarczyk M, Spooner D, Simon P.反对交通:线粒体DNA转移到质体基因组的第一个证据。中国生物医学工程学报2012;2(6):261-6。
- 28.
Straub SC, Cronn RC, Edwards C, Fishbein M, Liston A.从线粒体到质体基因组的DNA水平转移及其在乳筋草(夹罗布科)中的后续进化。基因组生物学学报2013;5(10):1872-85。
- 29.
库林琴科M, Konstantinov Y, Dietrich A.植物线粒体通过渗透过渡孔复合体主动导入DNA。EMBO j . 2003; 22(6): 1245 - 54。
- 30.
陆生植物线粒体DNA:系统发育的特征。咕咕叫麝猫。2004;46(3):123 - 39。
- 31.
石松和蕨类植物现存科属的线性序列。Phytotaxa。2011;19:7-54。
- 32.
罗斯菲尔斯CJ,马sundue MA,李国,Larsson A, Kato M, Schuettpelz E, Pryer KM。全足蕨类植物II科分类的修正(水龙足科:水龙足科)。分类单元。2012;61(3):515 - 33所示。
- 33.
张志强,张志强,张志强,等。基于叶绿体基因组测序的竹蕨属植物Cyrtomium Falcatum的系统发育关系。基因。2016;7(12):115。doi:10.3390 / genes7120115.http://www.mdpi.com/2073-4425/7/12/115.
- 34.
高亮,周勇,王志伟,苏玉军,王涛。蕨类植物质体rpoB-psbZ区基因组的进化:显著的结构重排和高度可变的基因间间隔。植物生物学杂志。2011;11:64。
- 35.
Grewe F, Guo W, Gubbels EA, Hansen AK, Mower JP。从ophianthrossum calnicum, Psilotum Nudum和Equisetum Hyemale的完整质体基因组揭示了一个古老的陆地植物基因组结构,并确定了Equisetales在单生植物中的位置。BMC Evol . 2013;13:8。
- 36.
魏锐,闫永华,Harris AJ,康建生,沈华,项清鹏。质体系统基因组学解决了具有快速辐射和速率异质性的蕨类植物之间的深层关系。基因组生物学进展2017;9(6):1646-57。
- 37.
Wolf PG, Rowe CA, Sinclair RB, Hasebe M.细囊蕨叶绿体基因组的完整核苷酸序列,铁线蕨capillus-venerisL. DNA Res. 2003; 10:59-65。
- 38.
Picramnia、Alvaradoa和Leitneria的系统基因组分析支持独立的Picramniales。系统进化学报2017;55(3):171-6。
- 39.
Roper JM, Hansen SK, Wolf PG, Karol KG, Mandoli DF, Everett KDE, Kuehl J, Boore JL江蚕(Angiopteris Evecta, G. Forst.)全质体基因组序列Hoffm。观音座莲科。王晓明。2007;32(2):49 - 52。
- 40.
做HD, Kim JS, Kim JH。从百合属植物Veratrum Patulum O. Loes全叶绿体基因组序列推断百合属4个科的比较基因组学。(Melanthiaceae)。基因。2013;530(2):229 - 35。
- 41.
陆建明,张楠,杜晓霞,文杰,李德智。叶绿体系统基因组学研究蕨类植物的关键关系。系统疾病学报,2015;53(5):448-57。
- 42.
Lycophytes &蕨类植物。见:郑义W, Raven PH, Deyuan H,编。中国植物志2-3卷。北京和圣路易斯:科学出版社和密苏里植物园出版社。
- 43.
Wolf PG, Der JP, Duffy AM, Davidson JB, Grusz AL, Pryer KM。蕨类植物叶绿体基因和基因组的进化。植物分子生物学杂志,2011;76(3-5):251-61。
- 44.
高亮,易鑫,杨烨,苏玉杰,王涛。桫椤叶绿体全基因组序列:蕨类植物叶绿体基因组进化变化的洞察。生物学报。2009;9:30 30。
- 45.
高丽,王波,王志伟,周勇,苏玉杰,王婷。日本叶蕨和马氏囊蕨的质体序列揭示了从基部蕨类植物到核心细囊类植物的基因组组织转化。基因组生物学学报2013;5(7):1403-7。
- 46.
金海涛,郑明明,金金杰。早期分化细囊蕨类植物的叶绿体基因组进化。37摩尔细胞。2014;(5):372 - 82。
- 47.
高超,任鑫,Mason AS,刘宏,肖明,李娟,付东。植物横向基因转移研究。功能整合基因组学,2014;14(1):23-9。
- 48.
黄春春,陈晓燕,陈晓燕。内共生基因转移:细胞器基因组锻造真核染色体。科学通报2004;5(2):123-35。
- 49.
苏茜,黄杰,高加腾JP。横向基因转移:构建生命之网。中国生物医学工程学报2015;16(8):472-82。
- 50.
阿奇博尔德JM,理查兹TA。基因转移:任何东西进入植物线粒体。BMC医学杂志。2010;8:147。
- 51.
Wolf PG、Sessa EB、Marchant DB、Li FW、Rothfels CJ、Sigel EM、Gitzendanner MA、Visger CJ、Banks JA、Soltis DE、Soltis PS、Pryer KM、Der JP。蕨类植物基因组空间的探索。中国生物学报2015;7(9):2533-44。
- 52.
Mir SA, Mishra AK, Reshi ZA, Sharma议员。标题印度克什米尔河谷鳞翅蕨科四新记录种。Biodiversitas。2014;15(1):6尺11寸。
- 53.
Mir SA, Mishra AK, Pala SA, Reshi ZA, Sharma议员。蕨类植物和蕨类植物的盟友区Shopian,克什米尔谷,印度。Biodiversitas。2015;16(1):27-43。
- 54.
印度次大陆鳞蕨属(蕨类目:鳞蕨科)专著,大英博物馆公报(自然历史)。伦敦:大英博物馆(自然历史);1989.386 - 9页。
- 55.
Olsen S:长尾鳞毛蕨。收录于:花园蕨类百科全书。中国:木材出版社;2007.
- 56.
帕里斯BS。新西兰铁线菊和毛铁线菊(铁线菊科:丝状科)。林志军学报1980;18:503-6。
- 57.
祁力马扎罗山南坡蕨类植物生态学研究。植物生态。2002;159:211-39。
- 58.
陆建明,文杰,Lutz S,王一平,李德泽。基于5种质体标记的中国铁线属植物系统发育关系。植物生态学报,2012;29(2):369 - 369。
- 59.
泰国蕨类植物区系新记录种绒蕨(Adiantum Hispidulum,蕨科)的注释。科学技术,2013;35(5):513-6。
- 60.
石超,胡楠,黄辉,高健,赵玉军,高丽珍。一种改进的用于全质体基因组测序的叶绿体DNA提取程序。《公共科学图书馆•综合》。2012;7 (2):e31468。
- 61.
维埃拉LD, Faoro H, Fraga HPD, Rogalski M, de Souza EM, Pedrosa FD, Nodari RO, Guerra MP。针叶树完整叶绿体和cpDNA分离方法的改进。《公共科学图书馆•综合》。2014;9 (1):e84792。
- 62.
Krinitsina AA, Sizova TV, Zaika MA, Speranskaya AS, Sukhorukov AP.一种快速、经济的从档案植物标本中提取DNA的方法。生物化学(Mosc)。2015, 80(11): 1478 - 84。
- 63.
Bolger AM, Lohse M, Usadel B. Trimmomatic:一种用于Illumina序列数据的灵活微调器。生物信息学。2014;30(15):2114 - 20。
- 64.
Langmead B, Salzberg SL.快速间隙读取对准领结2。Nat方法。2012;9 (4):357 - u354。
- 65.
Zerbino DR, Birney E. Velvet:使用de Bruijn图的从头短读组装算法。基因组研究》2008;18(5):821 - 9。
- 66.
Chevreux B, Pfisterer T, Drescher B, Driesel AJ, Muller WEG, Wetter T, Suhai s使用miraEST组装器进行可靠和自动化的mRNA转录本组装和测序est中的SNP检测。基因组研究》2004;14(6):1147 - 59。
- 67.
Wyman SK, Jansen RK, Boore JL。利用DOGMA自动注释细胞器基因组。生物信息学,2004;20(17):3252 - 5。
- 68.
Koboldt DC, Zhang QY, Larson DE, Shen D, McLellan MD, Lin L, Miller CA, Mardis ER, Ding L, Wilson RK。VarScan 2:通过外显子组测序发现癌症中的体细胞突变和拷贝数改变。基因组研究》2012;22(3):568 - 76。
- 69.
李H, Handsaker B, Wysoker A, Fennell T,阮j, Homer N, Marth G, Abecasis G, Durbin R, Proc GPD。序列对齐/映射格式和SAMtools。生物信息学。2009;25(16):2078 - 9。
- 70.
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析版本6.0。中华分子生物学杂志2013;30(12):2725-9。
- 71.
埃德加钢筋混凝土。MUSCLE:多序列对齐,高精度,高吞吐量。核酸学报,2004;32(5):1792-7。
- 72.
Schliep KP。生物信息学杂志,2011;27(4):592-3。
- 73.
杨振帕林4:最大似然系统发育分析。分子生物学与生物学报2007;24(8):1586-91。
- 74.
王晓燕,王晓燕,王晓燕。R语言的系统发育和进化分析。生物信息学,2004;20(2):289 - 90。
- 75.
Binet M, Gascuel O, Scornavacca C, Douzery EJ, Pardi F.快速准确的系统发育树分支长度估计。BMC生物信息学。2016;17:23。
- 76.
王晓燕,王晓燕。果蝇microRNA基因的起源与进化。基因组生物学学报2010;2:180-9。
确认
我们非常感谢Christopher R. Fraser-Jenkins博士纠正了我们对粗茎鳞毛blanfordii的标本和植物园,提供孢子d . villarii.
资金
这项工作得到了俄罗斯基础研究基金会的资助。14-04-01852(测序工作除外)。测序工作得到了俄罗斯科学基金资助。14-50-00029。出版费用由通讯作者提供。
数据和材料的可用性
全叶绿体基因组序列已保存在欧洲核苷酸档案(ENA)。分析中使用的其他数据包含在本文和附加文件中。
关于这个补充
本文已发布的一部分BMC植物生物学2017年增刊第17卷第2期:Belyaev会议2017:植物生物学文章选集。该补刊的全部内容可在网上查阅//www.cinefiend.com/articles/supplements/volume-17-supplement-2.
作者信息
从属关系
贡献
SK为本次研究选择并提供了植物材料;AK分离叶绿体,提取cpDNA,进行系统发育分析;AS和ML准备文库并进行测序工作;AS、ML和AK对基因组数据进行比较分析;KK和MB进行生物信息学分析,组装叶绿体基因组;MB和AS制备的基因组注释;EK -进行进化速率分析;AS和ML构思了这项研究并撰写了手稿。所有作者阅读并批准了最终稿件。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明他们没有竞争利益。
出版商的注意
施普林格自然对出版的地图和机构附属的管辖权要求保持中立。
额外的文件
额外的文件1:
注释的基因列表d . filix-mas而且d . blanfordii用DOGMA分析叶绿体基因组。(XLSX 46 kb)
权利和权限
开放获取本文根据创作共用署名4.0国际许可协议发布(http://creativecommons.org/licenses/by/4.0/),该协议允许在任何媒体上不受限制地使用、分发和复制,前提是您适当地注明原作者和来源,提供创作共用许可的链接,并说明是否有更改。创作共用公共领域奉献放弃书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条提供的资料。
关于这篇文章
引用这篇文章
洛切娃,医学博士,克里斯汀娜,a.a.,贝莱尼金,理学硕士et al。多足蕨(Polypodiales)质体组倒置重复序列的比较分析揭示了两个高变量区域。BMC植物杂志17日,255(2017)。https://doi.org/10.1186/s12870-017-1195-z
关键字
- Asplenium病灶
- Hispidula
- Pteridaceae
- 质体基因组
- 相对发展速度