摘要
背景
KASP (KBioscience Competitive Allele Specific PCR)和Amplifluor (Amplification with fluorescence) SNP标记是基于相同的等位基因特异性PCR平台的两项重要技术。
方法
使用自己设计的通用探针(UPs)和基因特异性引物(GSPs)进行扩增样SNP和KASP分析。
结果
与KASP相比,类放大器系统的优势包括成本显著降低,在“自行设计”双荧光标记UPs和常规GSPs的调整和开发方面具有更大的灵活性。提出的结果包括UPs和GSPs中“尾”长度的优化,方案调整,以及不同qPCR仪器中各种荧光团的使用。KASP主混配器在原系统和类放大器系统中的兼容性已在本文的结果中得到证明,证明了它们的相似原理。除了出现频率较高的等位基因外,还显示了罕见等位基因的SNP评分结果。
结论
amplifluo -like系统产生的SNP基因分型结果具有与KASP相当的敏感性和准确性,但成本明显更低,而且对于使用自行设计的GSPs的UPs具有更大的灵活性。
背景
基于SNPS(单核苷酸多态性)的低通量技术是植物基因分型领域的蓬勃发展。具有若干类型的SNP分析,具有非常不同的原理和应用程序(在[1,2,3.,4,5,6,7])。基于荧光(Förster)共振能量转移(FRET)方法的“等位基因特异性PCR”(AS-PCR)实现了一项革命性的新技术[8].AS-PCR方法的主要优点是将两种组分结合使用进行扩增:(2)非标记基因特异性引物(GSPs), 3 '端设计与SNP匹配,触发两个荧光标记探针中的任何一个进行扩增。在早期的报告中,UPs被定义为“通用能量转移标记引物”[9,10.,11.].然而,为了避免“探针”和“引物”之间的进一步混淆,我们将只使用术语“通用探针”作为上面提到的那些同义词。
KASP和Amplifluor SNP分析
两个成熟的技术是基于一个共享的AS-PCR平台:(1)KASP或卡斯帕(Kompetitive等位基因特异性PCR或KBiosience竞争等位基因特异性PCR),和(2)Amplifluor(扩增与荧光)的SNP标记。
KASP标记由LGC Genomics (www.lgcgroup.com.),他们通过自己专有的高通量机器人系统制造和应用这项技术,从树叶样本开始,直到完成基因分型结果。KASP的基本原理与amplfluor类似,但其基本化学成分和探针/引物分析从未被披露,这代表了公司的“商业秘密”。在进行KASP标记物分析时,研究人员有三种选择。大型大学和研究中心可以全部或部分购买用于KASP分析的仪器模块,包括LGC Genomics的试剂分析;这显然是一笔巨大的投资。第二种选择是将用于KASP分析的DNA样本直接提交给LGC Genomics办公室,这要便宜得多,成本大约是其他方法(如TaqMan方法)的10倍[12.].研究人员的最后一个选择是订购KASP Master-mix,并使用自行设计的GSPs进行自己的基因分型。这种方法更受欢迎,因为它为研究人员在开发各种GSPs提供了更多的灵活性。然而,这一选择的局限性在于,LGC Genomics生产的KASP Master-mix的供应是垄断的,相对昂贵,特别是对于小型实验。通常情况下,无论研究人员是购买了Robotic lines和KASP Master-mix,还是仅仅作为客户提交了DNA样本,并获得了自动的KASP基因分型结果,KASP标记的结果都是高度准确和有效的[13.].
Amplifluor SNP基因分型系统由Millipore开发,该公司最近与Merck合并(http://www.merckmillipore.com),并可作为试剂分析或服务。与KASP相反,amplfluor系统中所有组件的化学、设计和序列都在在线小册子和发表的论文中公开(例如,[9,10.,14.,15.])。使用该平台,研究人员可以选择在Millipore订购试剂/服务,或者在简单的指令指导下设计并订购他们自己的类amplfluor系统,与KASP标记物相比,成本降低了10 - 20倍。非常低的成本是最重要的,但不是“自制”扩增类SNP标记的唯一优势。研究人员有更大的灵活性,可以设计、测试、重新设计(如果需要的话),并分析尽可能多的SNP引物,使用一次性订购的两个用不同染料标记的UPs库存。事实上,在新的实验中,研究人员只需要以常规寡聚物为代价订购新的GSPs。在这种情况下,扩增样标记的目标、准确性和重现性完全被置于研究人员的“头脑中”和“手中”。
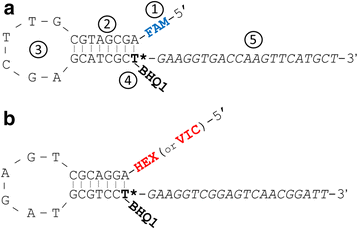
amplfluor万能探头结构原理
UPs的基本设计较早时已公布[9,10.].每个UP包含:(1)5 '端一个特定的荧光团;(2)发夹的茎和(3)圈;(4)带有猝灭剂的修饰胸腺嘧啶(T);(5)与荧光团对应的3 '端特定的'尾'(图5)。1).两种荧光团用于标记UPS,以在每个PCR中产生两种特异性UPS的混合物,其中扩增根据SNP和GSPS差异(图。1A和b).
荧光团
荧光团FAM常用在所有基于fret的技术中[9].第二个荧光团的选择是可变的;历史上是SR [10.,11.但后来被JOE [3.,14.,15.].HEX仍然是一个流行的第二荧光团的选择[16.],目前用于所有KASP标记[17.].Abi-Thermofer制造的QPCR仪器中的通道专为荧光团VIC设计[18.].适用于SNP分析的所有荧光团(FAM, SR, JOE, HEX和VIC)的详细信息在附加文件中提供1.
发夹
大小和发夹环的长度和干UPS已被报告为Amplifluor变化很大,并自行设计Amplifluor样SNP [9].然而,后来对其进行了优化,并公布了不同类型SNP分析的最合适参数[10.,15.,与图中所示相同。1.尽管如此,研究人员仍有可能对UPs的发夹结构进行进一步的修改,甚至是全新的设计,以进一步证明这些修改的有效性。
猝灭剂
最初,早期Amplifluor SNP标记开发仅使用DABSYL猝灭器[9].随着在474处最大吸光度,当与FAM配对DABCYL是优秀的,但它由于其较低的吸光度发射光谱是较不适合于其它的荧光团(附加文件1).然而,DABSYL在FAM和SR SNP分析中的成功应用[10.,11., FAM和JOE [14.,15.,19.,20.]、FAM和HEX [16.的报道。
黑洞淬灭剂,BHQ1, BHQ2和BHQ3,被开发并用于植物病理学的TaqMan PCR [21.,22.]和医学研究中[23.].在所有荧光团的光谱中,只有BHQ1的吸光度最大,FAM, JOE, VIC和HEX(附加文件1).BHQ1已成功用于我们以前对面包小麦的放大器SNP标记的研究[24.].
3特定尾部
在3 '端,每两个UPs都有一个特定的21 bp长尾。在带有猝灭剂的修饰T后,尾部的前六个核苷酸是相同的。3 '端的最后15个核苷酸对每条尾巴来说都是独特的。最初,FAM和DABSYL猝灭器标记的UPs使用较短的尾部[9].在以后的纸张中,指示尾部1和2,并用于扩增SNP分析[10.,11.,14.,15.这两条尾巴与图中所示的是相同的。1.
非标记基因特异性引物(GSPs)的设计
必须为每个特定的感兴趣基因(GOI)设计一套三种非标签GSPs,遵循早些时候发表的原则[10.,11.,14.,15.].两个正向引物设计为具有分别匹配SNP核苷酸的确定的两个变体3的端部。因此,这两个正向引物通过仅在其3'末端的单核苷酸差异。单个共用反向引物被设计成产生具有50至120个碱基之间的最佳大小的扩增子。与SNP特异性每个正向引物3'端也必须在其5'端尾延伸。这些扩展是相同的(但不互补)尾1和两个相应的UPS 2,在表1.该组需要三个GSP的是常规的非标记的寡核苷酸,成本一样购买了常规PCR引物一般,所有的分子生物学实验室使用。
Amplifluor SNP和KASP标记的PCR方法
有许多不同的已发表的协议适用于Amplifluor SNP和KASP标记,使用各种PCR热循环器。Amplifluor SNP标记包含35-40相同周期[10.,11.,19.,20.]或两组20-25个周期[14.,15.].与KASP Master-mix使用的协议包含10个循环,梯度温度降低,并进一步25-35个循环[25.,26.,27.,28.,29.,30.,31].的所有协议细节存在于其他文件2.
Kasp与“自制”主机的兼容性
如上所述,使用KASP标记的研究人员可以购买并使用整个机器人生产线对各种作物进行SNP基因分型,例如在澳大利亚阿德莱德大学。另一些人则将DNA样本送交LGC办事处,并公布所收到的SNP基因分型结果,如大麦[32Peanut [33].
然而,许多研究人员也订购了KASP Master-mix,并在自己的实验室使用常规热循环仪或qPCR仪器进行SNP基因分型,例如在小麦中[26.,28.,34,35,大豆27.,29.,36]和玉米[37].有趣的是,在这些论文中提到GSPs与KASP Master-mix一起使用时,其尾部1和尾部2完全相同,如Table所示1.因此,为KASP和amplflur -like分析而开发的两种不同类型的GSPs具有相同的尾部。目前还没有关于KASP中完整的UPs结构的信息,但如图所示,两个尾巴似乎与Amplifluor SNP标记中使用的相同。1.因此,我们可以推测,商业生产的KASP主混合物和自行设计的放大器样SNP标记兼容,可互换使用。
本文的目的是:(1)评估试剂、仪器、主混合、PCR、荧光信号检测和SNP调用的修改和优化方案的效果;(2)对“自制”扩增类标记用于植物SNP分析的基本方案进行评价,并与已发表文章中免费提供的方法进行比较;(3)比较自制Amplifluor-like标记和KASP标记针对同一SNP的基因分型结果。
方法
植物材料和DNA提取
在哈萨克斯坦的Akmola地区进行了大田试验,种植了多种品种的面包小麦和大麦。如我们之前的论文所述,我们收集了树叶并提取了DNA [24.],然后采用苯酚-氯仿萃取法[38,用PCR检测DNA质量。
通用探头(UPS)和基因特异性引物设计(GSP的)
在我们的实验中,我们使用了带有四种特定尾部的UPs(见表)1A).前两个尾巴(1和2)与之前发表的相同[10.,11.,14.,15.].最后两条尾(3和4)较短,设计时相应的尾1和尾2缺失核苷酸2 - 14(表)1A).四种UPs的时序如表所示1B.,其中前两个通用探针,UP-1和位置UP-2,是相同的那些图1中。1.最后两个通用探针,UP-3和UP-4,如所描述的缩短了尾巴。设计两组的GSP(F1,F2,R和F3,F4,R),并用于每个GOI的分别与对应的UP-1 / UP-2和UP3 / UP-4,。一个这样的GSP为大麦GOI,重叠群ABC08184的一个例子,在表被呈现1C在附加文件中3..所有标记FAM, VIC或HEX的UPs均由俄罗斯莫斯科DNA合成公司(http://www.oligos.ru),所有基因特异性引物均由俄罗斯新西伯利亚Biosan公司(http://www.biosan-nsk.ru.).
放大器样SNP和KASP分析
哈萨克斯坦阿斯塔纳农业技术大学使用Quant Studio-7 Real-Time PCR仪器、qPCR (ThermoFisher Scientific, Paisley, UK)和flustar Omega微标仪(BMG LabTech, Ortenberg, Germany)。Real-Time qPCR系统,CFX96模型(BioRad, Gladesville, NSW, Australia)在澳大利亚贝德福德公园的弗林德斯大学使用。
正如我们前面论文中描述的PCR条件相同[24.[PCR鸡尾酒组成改变。用96或384孔微量培养板用在每个孔中加入10μl或5微升总反应体积分别使用。将PCR混合物含有2×主混合物与终浓度以下试剂:1×PCR缓冲液,1.8毫摩尔MgCl2,每一种dNTP为0.2mM,0.25μM每种荧光标记探针,0.15μM每个正向引物,反向引物为0.78μM和0.5单位的TaqDNA聚合酶(GenLab,阿斯塔纳,哈萨克斯坦)或0.1单位Maxima热启动Taq聚合酶(美国ThermoFisher)。PCR量的一半为基因组DNA,调整为10 ng/μl。PCR程序优化见附加文件2.基因分型的SNP呼叫是自动确定的,如前面描述的24.].每个实验重复两次,技术重复证实了SNP呼叫的可信度。
在澳大利亚阿德莱德大学植物基因组学中心的LGC Genomics高通量基因分型系中,根据制造商的协议进行了KASP分析。KASP Master-mix和消耗品由LGC Genomics (Middlesex, UK)提供。
结果
具有不同尾部的Amplifluor通用探针的优化
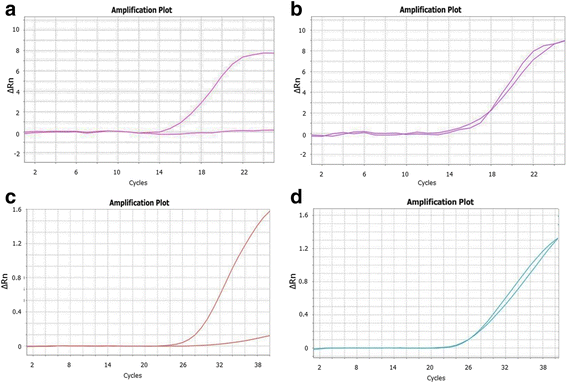
使用尾长为21 bp的UPs和尾长为21 bp的UPs进行扩增,以及使用尾长为9 bp的UPs和尾长为3和4进行扩增,使用qPCR仪器得到了相似的结果(图)。2).使用尾1和尾2的UPs荧光标记产品的扩增开始得更早,在第14个周期,在homo和杂合等位基因中(图)。2和b).相对放大,ΔRn,一个等位基因扩增达到8个单位,等位基因间分化非常强(图。2).两个杂合等位基因扩增速度快,均为ΔR的9个单位n在PCR终点。
FAM-和vic标记的不同尾部UPs的放大曲线。扩增样SNP标记被开发并与纯合子的“标准”尾1和2一起使用(一个)和杂合(b)基因型。使用两个尾较短的UPs(尾3和尾4)对同一纯合子(c)和杂合(d)基因型。数据取自[24.用于设计用于一个SNP从哈萨克斯坦不同面包小麦品种,与Amplifluor状标记KATU48基因分型的植物TaDREB5转录因子。y轴为FAM或VIC荧光信号的相对放大单位ΔRn
在尾数为3和4的UPs中,同源和杂合等位基因的扩增开始较晚,在第24个周期开始(图)。2C和D.).纯合子等位基因的区分是基于ΔR中较小的差异n,相对扩增仅达到3个单位,但经过30-35个PCR循环后仍然非常清晰(图)。2摄氏度).
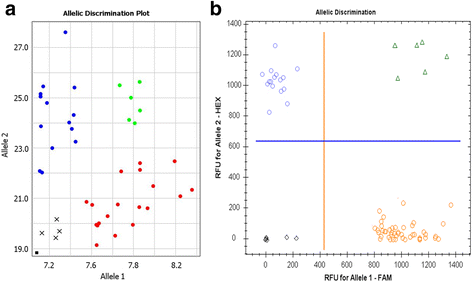
不同QPCR仪器和荧光团的兼容性等位基因歧视
没有差异在Amplifluor状SNP基因分型的用标记有FAM和VIC UPS相同的实验中发现,通过使用赛默和BioRad公司(图制造qPCR仪器。3.).这些仪器有专为FAM和VIC设计的通道(图。3)和FAM和HEX(图。3 b分别)吸收。在我们的实验中,FAM和hex标记的UPs在相同的qPCR系统中也记录了类似的结果(数据未显示)。
FAM荧光团和VIC荧光团标记相同UPs的扩增类SNP标记应用比较Quant Studio-7 Real-Time PCR仪器(ThermoFisher Scientific, Paisley, UK),专为FAM和VIC评分(一个)和Real-Time qPCR系统,CFX96型(BioRad, Gladesville, NSW, Australia),专为FAM和HEX识别而设计(b),使用了。对等位基因自动调用以不同颜色显示,分别使用辅助软件,每台仪器。X轴和Y轴显示相对放大单元,ΔRn值,对于FAM和VIC荧光信号(一个);FAM和HEX的相对荧光单位RFU (b);分别用qPCR ThermoFisher和BioRad仪器测定
扩增SNP标记的PCR方案
三种类型的协议中使用各种PCR基因扩增仪我们的实验中进行了测试(附加文件2).前两个协议被设计并用于SNP Amplifluor方法。方案1是一个非常通用的三步循环PCR,延长时间相对较长(40 s), 35-45个循环。在方案2中,在前20个周期和后22个周期中,分别使用较高/较低的退火温度和较短/较长的退火/延长时间的两个PCR周期。作为KASP技术的制造商,LGC Genomics已发布协议3,用于类似生物化学的KASP标记。
我们用自己设计的ampliflur -like SNP标记进行实验,并对面包小麦、大麦和鹰嘴豆的各种GOI进行分析,结果显示这三种PCR方法的使用没有显著差异(数据未显示)。我们自己设计的协议4(附加文件2)最类似于协议2 [14.,15.不同之处在于,在每个双周期的第一部分和第二部分中,包含了“双周期”,分别将较高和较低的退火温度与较短和较长的退火/延伸时间结合起来。数字2结果显示,PCR扩增使用20和40个周期的不同Amplifluor-like SNP探针,但后一种情况下,30-35个双周期就足以明确等位基因分型。在本节中,我们可以得出结论,无论PCR方案的差异如何,UPs在Amplifluor-like SNP标记中的应用都是稳定的,因此研究人员可以从已公布的PCR方案中选择或设计自己的PCR方案来满足自己的具体需要。
调整扩增类SNP标记的PCR方案以解决杂合子的假阳性
效率和放大的水平发挥SNP基因分型中最重要的角色。尽管精确的等位基因特异性,Amplifluor状SNP判定可能会失败,并显示“假阳性”的结果,通常作为杂合子“ab这些基因型的缺失或存在的数量非常少。图中给出了一个将基因型错误识别为杂合子的例子。4A从哈萨克斯坦小麦种质资源收集与Amplifluor般的SNP标记W58在设计TFTanfyc-A7.图2中的完美对角线。4A在每个基因型中FAM和VIC荧光标记均显示相同的扩增信号。这种情况也发生在其他发达的ampliflur -like SNP标记。然而,这不能真实反映情况,因为在任何给定的印度国土邦中,种质收集将大部分包含很少或没有杂合子。
通过降低浓度,解决了导致错误基因型鉴定的冲突Taq- 聚合酶使用。在图呈现的情况。4, Maxima热态启动Taq-聚合酶,由ThermoFisher制造。它的放大能力Taq采用0.5个酶单位/ 10 μl的总PCR,最终扩增出两种荧光标记的片段。数字4B.显示了我们的结果,使用相同的面包小麦收集基因型和相同的SNP引物W58,只是Maxima Hot-Start少了5倍Taq-聚合酶(每10 μl总PCR反应0.1酶单位)。在研究的小麦材料中观察到明显的等位基因区分,杂合子完全缺失,只有少数不确定的基因型(图。4B.).
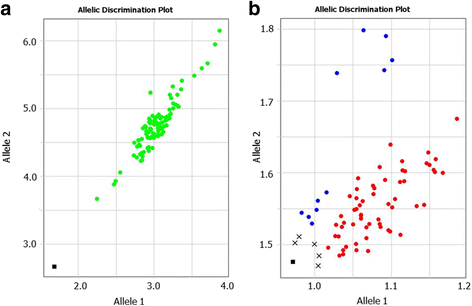
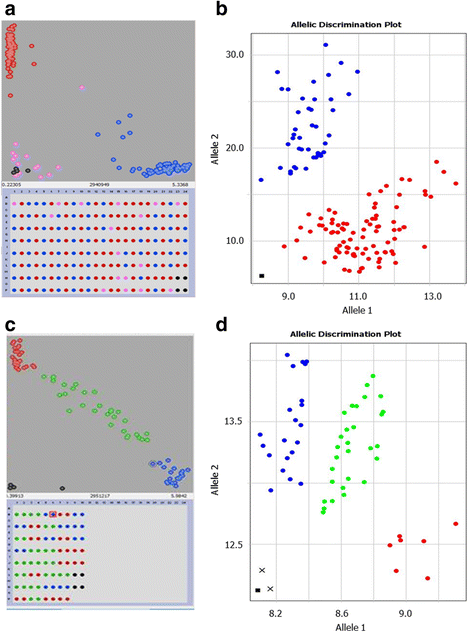
KASP和类放大系统在相同主混合下的比较
同样KASP主混合物在原始KASP都在使用的自主研发Amplifluor类系统。数字5展示了在大麦中研究的两种GOI的例子,其中使用相同的KASP Master-mix与不同的GSPs和工具进行扩增和等位基因识别。配对面板a/b和c/d在图中。5显示两个系统的非常相似的输出,唯一的差异是屏幕的设计和等位基因歧视的颜色变化,与仪器的技术设置相关。
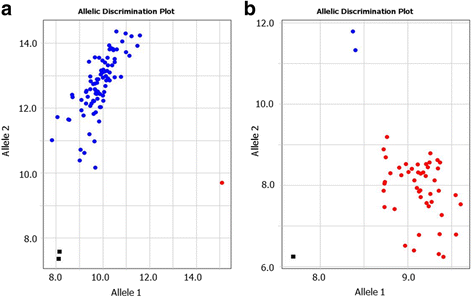
罕见SNP在罕见等位基因基因分型中的应用
哈萨克斯坦小麦和大麦中罕见等位基因的基因分型得分的例子见图。6.这些结果是使用我们自己设计的GSPs类放大器系统产生的。
讨论
KASP和Amplifluor-like标记在SNP基因分型方面具有相似的原理和应用。这一点得到了可用信息的支持,显示GSPs中的尾1和尾2在两种方法中是相同的。此外,之前发表的信息和本文提供的数据证实,KASP Master-mix可以与原始的KASP技术和自主开发的Amplifluor-like系统一起用于SNP基因分型。在KASP系统中UPs的结构仍未公开,但在LGC Genomics的网站上提到了带有荧光团(FAM和HEX)的“两个通用FRET磁带”[17.].也没有关于UPs中猝灭器类型的可用信息,这对于KASP基因分型来说仍是未知的,但我们可以推测,在KASP和Amplifluor-like系统中,UPs的一般结构非常相似,如果不是完全相同的话。
UPs的完美优化和相关的组织良好的业务和营销使KASP标记在植物生物学家和更广泛的研究人员中非常受欢迎。大量且不断增长的引用KASP分析的出版物表明人们对这一技术的兴趣持续高涨(见表)2).然而,即使在研究人员的实验室订购和使用KASP Master-mix, KASP标记物的成本仍然相对较高。相比之下,ampliflur -like自开发的SNP标记共享相同的分子平台,但比KASP便宜得多。发表的文章数量的减少表明,科学家可能不知道Amplifluor-like SNP系统,因此,提交样本用于KASP分析或订购KASP Master-mix,而不是开发和使用他们自己的Amplifluor-like标记(见表)2).
我们的结果表明,根据公布的序列或自己的设计订购UPs并不复杂。如图所示的两个UPs。1和表1在探针中间有一个淬灭器,在5 '端有不同的染料。因此,UPs是相对昂贵的试剂。但是这两款UPs在所有的实验中都是常见的,所以只需要批量订购一次即可。然后,它们可以在相当长的时间内用于所有进一步的AS-PCR实验,没有限制,这大大降低了成本。与UPs类似的KASP Master-mix价格更高。GSPs,表中有序列示例1和额外的文件3.,可以简单地下令定期寡核苷酸。
snp可以通过研究人员自己的实验来识别,也可以从已发表的论文或公开可用的数据库中获取。根据研究人员的目标,可能需要在印度政府内部或侧翼区域内进行SNP。强烈建议对推测为SNPs的基因片段进行测序,以验证其存在,然后将显示SNPs的测序基因型作为扩增类或KASP方法进行SNP分析的参考。然而,由于时间或资金的限制,这并不总是可能的,特别是在小型实验室。在这种情况下,可以根据已发表的结果或SNP数据库对假定的SNP进行初步分析。UPs的优势在于,它提供了额外的机会,可以根据研究人员的意愿订购和筛选尽可能多的假定snp,而不需要测序,花费也更少。然而,技术上对使用的工具进行等位基因区分的要求必须在以后的基因型测序中得到验证,以显示最清晰的SNP评分结果。经过SNP验证后,这些参考基因型可用于进一步的基因分型。
在无花果。3.我们在两种不同的仪器中展示了相同的自我开发的放大器SNP标记的应用。实时QPCR系统分别由Thermofisher和BioRad制造。然而,任何类似的qPCR循环仪可用于分析Amplifluor状SNP标记,如果这种仪器具有至少两个通道的吸光度在两个不同的谱分析与所选择的荧光团[兼容39,40].
同时,目前大多数研究人员,特别是在使用KASP Master-mix时,根本不需要使用任何实时qPCR系统。可以使用常规PCR,与任何普通PCR循环器,包括旧型号。PCR产物可以使用分光光度计进行分析,分光光度计吸收的波长与所使用的荧光团相对应(附加文件)1),然后可以手动转换为SNP等位基因识别。
然而,更富有成效和适当使用酶标仪高吞吐量所需的方法时。在这种情况下,96孔和384孔板是最适合的格式,由光学透明胶带密封并直接PCR完成后用酶标仪筛选。大范围的制造酶标仪给人的机会,研究人员根据厂家价格,可用性和个人喜好来选择最合适的产品。维克多1420珀-Elmer公司WALLAC,USA:在发表论文,SNP Amplifluor和KASP标记使用以下酶标仪大多得分11.], flustar Optima或Omega, BMG LabTec,德国[20.,25.和瑞士泰坎萨菲尔[28.,36].
在我们的实验中,使用不同的PCR循环器测试了三种类型的协议(附加文件)2).前两个协议是为SNP Amplifluor方法设计并使用的。方案1是一个非常通用的三步循环PCR,延长时间相对较长(40 s), 35-45个循环。在方案2中,在前20个周期和后22个周期中,分别采用较高/较低的退火温度和较短/较长的退火/延长时间的两个PCR周期。这在开始时产生了更特异的PCR产物扩增,但在方案的第二部分更增强的扩增和延伸。协议3发表并用于类似生物化学的KASP标记,由KASP技术的制造商LGC Genomics规定。该协议包含初始10个周期,持续时间相对较长(60秒),具有温度梯度。在方案3的每个周期中,这种长时间退火和温度下降的组合允许在初始PCR步骤中具有最大可变性的扩增。以下周期有60秒的退火,没有专门的扩展步骤。在[26.,28.],“扩增子小于120 bp,扩增步骤是不必要的”这一点是正确的,但它与之前需要很长的退火步骤(60 s)相冲突。在一般的PCR设置中,10 s就足够扩增子的退火了。然而,上面提到的注释完全匹配协议1和协议2中退火+扩展的总时间,与协议3中完全相同(60 s)。我们认为,之所以需要这么长时间的退火+延伸,是因为SNP ampliflur -like和KASP标记(包括UPs和GSPs)的性质相似。在我们的实验中,最优的PCR条件是协议4(附加文件)所述的条件2),尾为1和2的UPs为20个“双倍循环”,尾为3和4的UPs为30-40个循环(图。2).
必须仔细调整PCR协议,特别注重使用哪种品牌的效率和放大水平Taq- 聚合酶是优选的。主要目的是从只有单一荧光标记实现扩增曲线温柔的开始,只有从第二荧光标记非常小的放大。高浓度的使用Taq- 聚合酶具有较强的放大能力产生掩蔽通过虚假放大真实结果的风险更大,因为两个GSP的通过仅在3'-末端的单核苷酸差异。因此,PCR协议的调整来结合在较低浓度Taq- 聚合酶和更少的PCR循环需要选择在荧光信号放大的最佳启动。与已知序列的参考基因型可以在初始PCR协议的调整非常有益的。
SNP可以在研究基因型中具有差异分布。当在研究的附加过程中发生大量等位基因时,SNP的最清晰的基因分型结果发生。然而,这种情况并非总是如此,主要是与研究中的基因库的性质有关。例如,分享来自适应局部环境的父母的同一血统或分离群体的高级育种线,没有异国情调的种质,通常具有有限的遗传多态性,特别是在一些GOI中。因此,显示大部分SNP等位基因的病例不经常发生。相比之下,稀有SNP等位基因,其中只有一个或两个基因型从研究的种质资源收集的其余部分不同,或从分离群体其他后代,发生更频繁。在我们的研究中,最多60%的小麦和30%的大麦种质中起源于哈萨克斯坦的特征可以稀有SNP等位基因。罕见的SNP等位基因的高概率可以使其在交叉计划中进一步考虑和应用尤其重要,以产生完美的映射群,预测GOI的隔离。稀有等位基因鉴定的扩增荧光系统的应用显示我们的实验中的完美结果(图。6).
结论
我们的研究已经表明,Amplifluor样系统,用于SNP基因分型提供了与灵敏度和精确度的结果等于那些KASP的在显著成本便宜,并具有与自行设计的GSP UPS更大的灵活性。为了筛选的目的,当研究人员需要的测试和检查数百或数千推定的SNP的“自制” Amplifluor般的SNP系统更加适合开发简单,价格便宜,准确的SNP标记。这种说法是植物遗传学和作物育种,其中来自后代或品系个人成千上万必须同时分析,或在很短的时间表现最好的植物分子标记辅助选择尤为重要。此外,“自制” Amplifluor样系统SNP基因分型也是最适合医学,兽医学和微生物学的研究。在Amplifluor样SNP系统的这一研究和应用的呈现结果可以开辟新的途径科学家开发并为自己的其他改进适应SNP标记的现有系统。
缩写
- Amplifluor:
-
与荧光放大
- GSP:
-
基因特异性引物
- KASP:
-
Kompetitive等位基因特异性PCR或KBIONIES竞争等位基因特异性PCR
- SNP:
-
单核苷酸多态性
- 向上:
-
通用探针
参考文献
- 1。
李志刚,李志刚。SNP基因分型技术在生物医学分类中的应用。生物工程2007;9:289-320。
- 2。
SNP基因分型:促成一场革命的六项技术。Nat方法。2008;5(5):447 - 53年。
- 3.
Gašparič MB, Tengs T, La Paz JL, Holst-Jensen A, Pla M, Esteve T, Žel J, Gruden k。生物化学。2010;396(6):2023-9。
- 4.
Peatman E. SNP基因分型平台。在:刘Z,主编。新一代测序技术在水产养殖全基因组选择。牛津:Blackwell出版;2011. p。123-32。
- 5.
通过下一代测序发现SNP及其应用。《植物基因组学》2012;
- 6。
Kumpatla SP, Buyyarapu R, Abdurakhmonov IY, Mammadov JA。21世纪基因组学辅助植物育种:技术进步与进步。印:Abdurakhmonov I,编辑。植物育种。InTech里耶卡:的哲理;2012.p . 131 - 84。
- 7。
汤姆森乔丹。高通量SNP基因分型加速作物改良。植物育种技术。2014;2(3):195-212。
- 8。
L, lider KW, Vavra SH, Kwiatkowski RW。未扩增基因组DNA的大规模SNP评分。药物基因组学。2000;1:219-29。
- 9。
纳扎仁科IA,纳加尔SK,Hohman RJ。基于能量转移用于扩增和检测DNA的封闭管形式。核酸RES。1997; 25(12):2516-21。
- 10.
Myakishev MV,Khripin Y,胡S,哈默DH。高通量SNP基因分型的等位基因特异性PCR与通用能量转移标记的引物。Genome Res。2001; 11:163-9。
- 11.
Bengra C, Mifflin TE, Khripin Y, Manunta P, Williams SM, Jose PA, Felder RA。基于通用能量转移引物的均相PCR方法对原发性高血压单核苷酸多态性进行基因分型。化学2002;48(12):2131 - 40。
- 12.
2 .单核苷酸多态性基因分型:生物化学、基因工程、成本和通量。药物基因组学j . 2003; 3:77 - 96。
- 13。
Babiker EM,TC戈登,晁S,劳斯MN,Wanyera R,等人。电阻的遗传作图到Ug99真菌种族组的小麦锈菌f . sp。Tritici.在一个春小麦地方品种CItr 4311。中国科学(d辑:地球科学)2016;
- 14.
人类基因组DNA单核苷酸多态性基因分型方法的改进:扩增等位基因特异性聚合酶链反应与连接检测反应- taqman。学生物化学肛门。2004;330:288 - 97。
- 15.
Khripin Y.高通量基因分型与能量转移标记的引物。在:Didenko VV,编辑。方法在分子生物学:荧光能量转移的核酸探针:设计和协议,卷。335.托托瓦:Humana公司新闻公司;2006.215-40。
- 16。
Kreuzer KA, Bohn A, Lupberger J, Solassol J, le Coutre P, Schmidt CA.使用4-(4’-二甲氨基苯基偶氮)苯甲酸作为暗猝灭染料,实时荧光逆转录- pcr同时绝对定量的目标和控制模板。中国化学2001;47(3):486 - 90。
- 17。
LGC集团有限公司:http://www.lgcgroup.com/products/kasp-genotyping-chemistry.2017年访问。
- 18。
蒋可拉,李志强,李志强,等。利用三种高通量SNP基因分型方法,GOOD, Amplifluor和TaqMan,对二倍体和多倍体植物进行分析。Theor applet . 2006; 112:1115-24。
- 19。
以Mon810事件特异性分析为模型的实时PCR检测化学和循环模式的比较。中国农业科学。2007;55(11):4312-8。
- 20.
汉密尔顿A,梁R,使用FLUOSTAR OPTIMA酶标仪Amplifluor的SNP基因分型系统的Whittl B.应用。BMG LABTECH。2010; 187。
- 21.
Schena L, Hughes KJD, cook DEL。检测和定量Phytophthora Ramorum.,P. kernoviae,p . citricola和P. quercina在症状叶中进行多重实时PCR检测。植物学报。2006;7(5):365-79。
- 22.
Jawhari男,Abrahamian P,萨特AA,Sobh H,Tawidian P,阿布 - Jawdah Y.特异性PCR和实时PCR测定法的检测和定量“CandidatusPhytoplasma phoenicium”。分子细胞探针。2015;29(1):63-70。
- 23.
Fenati RA, Connolly AR, Ellis AV.有和没有溴化乙锭插入剂的单核苷酸多态性鉴别。化学学报。2017;954:121-8。
- 24。
引用本文Shavrukov Y, zhu alin A, serkbay D, Botayeva M, Otemisova A, abattarova A, et al.。通过Amplifluor SNP标记鉴定出dreb2型基因的表达水平与哈萨克斯坦北部面包小麦品种的性能和脱水耐受性相关。植物学报2016;7:1736。
- 25。
Saxena RK, Penmetsa RV, Upadhyaya HD, Kumar A, Carrasquilla-Garcia N, Schlueter JA,等。用于鸽子豆遗传作图和豆科比较作图的低成本单核苷酸多态性标记分析的大规模开发。DNA研究》2012;19(6):449 - 61。
- 26.
结合新一代测序数据的SNP发现和大量分离分析(BSA),在多倍体小麦中精细定位基因。BMC Plant Biol. 2012;12:14。
- 27.
袁Ĵ,文Z,顾C,高吞吐量的王D.介绍和成本有效的大豆SNP基因分型平台。植物基因组遗传学生物技术。2014; 2(1):90-4。
- 28。
Ramirez-Gonzalez RH, Segovia V, Bird N, Fenwick P, Holdgate S, Berry S,等。RNA-Seq批量分离分析使高分辨率遗传标记的鉴定在六倍体小麦育种。中国生物技术学报(英文版);2015;
- 29。
史志强,刘胜,李志强,李志强。大豆包囊线虫抗性的SNP位点鉴定与高产筛选。BMC基因组学。2015;16:314。
- 30.
Graves H, Rayburn AL, Gonzalez-Hernandez JL, Nah G, Kim DS, Lee DK。利用KASP法验证草原网茅DNA多态性(摘要pectinata链路)在美国前植物科学群。2016; 6:1271。
- 31.
Pasquariello M, Ham J, Burt C, Jahier J, Paillard S, Uauy C, Nicholson P.眼斑抗性基因PCH1和PCH 2小麦不是homoeoloci。Theor Appl Genet. 2017;13.0(1):91–107.
- 32.
谷物作物大麦DUS表型评估的诊断分子标记的评价(Hordeum Vulgare.ssp。vulgarel)。Al Appl Genet。2012; 125:1735至1749年。
- 33.
凯拉P,乌帕德亚雅HD,潘迪MK,Roorkiwal男,Sriswathi男,Janila P,等人。单核苷酸多态性为基础的参考集中花生的遗传多样性(花生通过开发和应用具有成本效益的竞争性等位基因特异性聚合酶链反应基因分型分析。植物基因组。2013;6 (3):doi:10.3835 / plantgenome2013.06.0019.
- 34.
Jayatilake DV,塔克EJ,布吕格曼Ĵ,刘易斯Ĵ,加西亚男,Dreisigacker S,等人。的遗传作图CRE8抗谷物囊肿线虫的轨迹(异皮线虫属avenae沃尔)小麦。摩尔品种。2015; 35(2):66。
- 35.
程P,Gedling CR,帕蒂尔G,VUONG TD,香农JG,多兰斯AE,阮HT。的轨迹的用于定量抗性遗传作图和单倍型分析Fusarium Graminearum.大豆品种PI 567516C。Theor Appl Genet. 2017;10.1007 / s00122 - 017 - 2866 - 8.
- 36。
Pham AT, Harris DK, Buck J, Hoskins A, Serrano J, Abdel-Haleem H,等。精细定位和表征控制抗性的候选基因Cercospora SojinaK.原两个大豆种质。Plos一个。2015; 10(5):e0126753。
- 37。
苏安,宋伟,邢建军,张锐,李超,等。利用大体积分离RNA-Seq鉴定与玉米s型细胞质雄性不育性不稳定性潜在相关的基因《公共科学图书馆•综合》。2016;11 (9):e0163489。
- 38.
小麦及其亲缘种β-淀粉酶序列定位。Theor Appl Genet 1988; 75:286-90。
- 39.
Löfström C, Josefsen MH, Hansen T, Søndergaard MSR, Hoorfar J.基于荧光的实时定量聚合酶链反应(qPCR)技术高通量筛选病原体。在:Bhunia AK, Kim MS, Taitt CR,编辑。高通量食品安全评估筛选。阿姆斯特丹:爱思唯尔;2015.219 - 48页。
- 40。
堪萨斯州立大学。http://www.ksre.k-state.edu/igenomics/equipment.Accessed 14 Aug 2015。
致谢
我们要感谢S.Seifullin哈萨克农业技术大学,阿斯塔纳(哈萨克斯坦)和弗林德斯大学,SA(澳大利亚)的工作人员和学生支持这项研究。我们表示特别感谢丽贝卡·福克斯和肯查尔莫斯在植物基因组中心,阿德莱德大学(澳大利亚)KASP分析进行。我们也感谢卡莉·施拉姆在手稿的批评意见。
资金
研究和出版费用由哈萨克斯坦教育和科学部研究计划n0106 /PTSF资助。
数据和材料的可用性
本研究期间生成或分析的所有数据都包含在本发布的文章及其补充信息文件中。
关于这个补充
本文已作为一部分发布BMC植物生物学2017年第17卷增刊2:2017年Belyaev会议植物生物学文章选集。该补充的全部内容可在网上找到//www.cinefiend.com/articles/supplements/volume-17-supplement-2.
作者信息
从属关系
贡献
SJ和AK协调研究和组织实验,LZ和GK进行SNP基因分型,DS, AsZ MB提取和调整DNA样本,对在该领域进行实验和植物取样,AiZ与PCR协议修改,KS协调qPCR研究和修订相应的部分,PL监督了这个项目,并修改了手稿的最终版本。YS用UPs和GSPs进行实验,并撰写了第一版手稿。所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用
同意出版
不适用
相互竞争的利益
作者宣称,这项研究是在没有任何商业或财务关系的情况下进行的,这些关系可以被解释为潜在的利益冲突。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Jatayev, S., Kurishbayev, A., Zotova, L。et al。扩增型SNP标记在植物基因分型中的优势BMC植物杂志17,254(2017)。https://doi.org/10.1186/s12870-017-1197-x
关键字
- Amplifluor
- 基因特异性引物
- KASP
- 单核苷酸多态性
- SNP标记
- 通用探针