抽象的
背景
MicroRNA作为细胞信号传导,植物生长和发育的分子调节剂,并调节各种初级和次生植物代谢过程。在本研究中,进行小RNA的深度测序,以鉴定来自药学上重要植物的已知和新的miRNA,吊兰borivilianum.
结果
从幼叶小RNA库中共鉴定出442个已知mirna和5个新mirna。茎环RT-PCR实验验证证实了其在硅胶上的鉴定。基于根和叶的转录组数据C.博伊西亚姆,奥里扎萨蒂瓦,和拟南芥使用psRNAtarget和mirRanda进行靶基因预测。BLAST2GO帮助定位预测靶点,KEGG (Kyoto Encyclopedia for Genes and Genomes)通路分析得出miR9662、miR894、miR172和miR166可能参与调控皂苷生物合成通路。通过RT-qPCR分析进一步验证了miRNA与其目的基因的相关性。
结论
本研究提供了首次详细阐述的miRNA池c . borivilianum,有助于了解miRNA依赖对皂苷生物合成的调控,并设计进一步的代谢工程实验来提高其在植物中的含量。
背景
吊兰borivilianumSantapau&Fernandes是Liliaceae家族的单圈常年草药。Genus.吊兰由大约215个物种组成,其中C.博尔西氏菌,C. arundinateum Baker,C.Tuberosum(Roxb。)Baker,C. Laxum R.Br.,C.Attenuatum Baker和C. Breviscapum Dalz.产于印度[1那2].c . borivilianum为四倍体种(2n = 4× = 28),基本染色体数为7 [3.那4.].该植物提取物具有免疫调节作用[5.],抗糖尿病[6.], pendiculatory [7.和雄激素活动[8.]主要是由于植物中含有大量的皂苷[5.].由于壮阳性质的特性c . borivilianum,这种植物也被称为“草药伟哥”。
化学上,皂苷被归类为三萜类化合物和甾体糖苷。甾体皂苷是27个C-原子分子,而三萜类皂苷是30℃分子。皂甙由非极性糖酮(三萜或类固醇)和一种或多种烯醇(单糖)部分组成[9.].由于亲水和疏水部分存在,Saponins是优异的乳化剂和发泡剂。这些性质可能有助于降低人类的血清胆固醇水平。皂苷作为抗氧化剂,有助于减少自由基以防止氧化应激。Saponins味道甜蜜苦味,并拥有额外的性质,如发泡,药理学,药用,溶血,抗微生物,杀虫,软体动物活动,并在化妆品行业,饮料和糖果中找到一些地方[10].皂苷在各种植物物种中以高量存在c . borivilianum那光甘草、人参、苦参、巴拉圭冬青等等。 [11].
在C.博伊西亚姆,已报道了多种甾体皂苷,如糠烷型甾体皂苷:鲍里维酸苷a (C56.H94.O.27.),硼化石苷-b(c57.H96.O.27.),硼柳甙-c(c57.H96.O.28.)和硼丙苷-D(c56.H92.O.27.) [12].四种螺柱烷类甾体皂苷,如硼酰亚甲苷e (C73.H120O.39.na),硼ilianosides-f(c73.H118O.39.na),硼梨糖苷-g(c51.H82.O.24.), Borivilianosides-H (C50.H80O.24.) [13].另一种皂苷,叶绿素-i(3b,5a,22r,25r)-26-(β-d-吡喃葡萄糖氧基)-22-羟基 - futostan-12-一三甘油苷(1-4)荧光葡萄酒孤立 [14].最近,发现了1'-乙酰氧基吡啶醇(ACA)c . borivilianum根(15].因此,该植物中存在多种皂苷。皂苷通过甲基醇酸途径(MVA)在细胞质和非甲戊酯途径(MEP)中的生物合成。以前,转录组的根和叶子研究c . borivilianum揭示了与皂苷、类黄酮和生物碱生物合成途径有关的基因[16].皂苷生物合成途径相关酶编码基因的差异表达研究证实,该途径的早期和晚期基因分别在叶和根中被激活[17那18那19].
由于这些植物成分具有重要的药用价值,目前的研究重点是提高其皂苷的含量c . borivilianum.有报道称,当幼苗接种菌根真菌时,皂苷含量增加[20.].然而,尚未确定调节皂苷含量的产生的分子因素。由于miRNA的发现,这些是公知的在转录和转录后水平调节基因表达。近年来,发现MIRNA是调节许多植物中次生代谢产量的主要生物因素[21.].在各种spp。那据报道,miR5021通过调控酶的基因表达来检测精油的生物合成,如参与2- c -甲基-d -赤藓糖醇4-磷酸/1-脱氧-d -木糖5-磷酸(DOXP)途径的香叶醇二磷酸合酶[22.].发现MIR5021和MIR5293调节MVA途径的第一次酶活性田七[23.].基于上述研究,我们假设必须有一些miRNA调节次级代谢途径c . borivilianum.我们的目的是分析c . borivilianum并预测所鉴定的mirna的靶点。进一步研究了mirna靶点在生物化学途径中的作用,重点研究了皂苷生物合成途径。为此,我们制备了小RNA库,并利用Illumina平台进行测序。预测已知和新的mirna及其基因靶标。RT-qPCR分析mirna与计算分析预测的靶蛋白之间的相关性。
方法
植物材料
c . borivilianum用白光(通量200 μmolm)照射植物生长室,在27℃昼夜温度条件下,在调节的环境下,从老植株的营养芽中培养出1∶1.5的土钙混合植株-2S.-1印度昌迪加尔的旁遮普大学[纬度:30°44 ' 14 N;经度76°47的14 e;海拔350米]。取2月龄植株的幼叶,分离富含小RNA的总RNA。
RNA分离和小RNA测序
总RNA包括小RNA的提取是结合Ghawana等人2011 [24.]协议与miRNeasy试剂盒(Qiagen,德国)。嫩叶组织细粉(100mg)用溶液- i和溶液- ii均质。用200 μl氯仿处理,分离有机相。分离上水相,加入1.5体积的100%乙醇。将溶液上到miRNeasy柱上洗涤,DNase酶切。经柱上DNase酶切和洗涤后,用核酸酶游离水洗脱富集小RNA的总RNA。利用Nanodrop分光光度计和Agilent 2100生物分析仪分别检测RNA的数量和质量。小RNA测序使用Illumina HiSeq 2000平台进行,该平台位于印度喀拉拉邦Kakanad信息公园路智慧城市科奇AgriGenome实验室私人有限公司(http://www.aggenome.com/).
小RNA测序原始数据提交至NCBI (national Centre for Biotechnology Information) SRA (Sequence Read Archive)数据库。利用Illumina平台生成50 bp长的小RNA片段。根据GC含量、平均基础质量和phred评分删除低质量读。然后处理原始读取,使用Cutadapt工具(v-1.3)消除5 '和3 '适配器序列[25.].然后使用Bowtie2程序(version 2.1.0)对GtRNAdb、Rfam、piRNABank、siRNAdb、NCBI Genbank、deepBase等数据库进行比对,分别剔除tRNA、rRNA、piRNA、siRNA、snRNA和snoRNA等非编码rna [26.].其余的reads用于预测已知和新的miRNAs。
鉴定已知和新的miRNA
过滤后的小RNA reads (17-35 bp)用于已知miRNA的鉴定。这些reads与miRBase-21中viridiplantae数据集的成熟mirna和前体序列进行了比对[27.]使用Bowtie程序(版本0.12.9)只允许2个不匹配与成熟miRNA [28.].
使用miRDeep2 (version 2.0.0.7)进一步进行新的miRNAs预测,在已知miRNAs预测后剩余的未对齐reads [29.].读取与叶片和根转录组序列对齐c . borivilianum。使用来自MFOLD WebServer的RNA折叠形式(2.3个能量)应用检查这些读取的发夹环形成[30.].在茎环臂部发现了miRNA和miRNA*序列。最终,这些reads被认为是新型mirna的先驱。具有以下特征的序列可以被认为是新型miRNAs的潜在前体:(一)序列形成茎环结构成熟的microrna的发夹结构(b)序列在一个手臂的发夹结构(c)小于6之间的不匹配允许microrna的microrna的* (d)二级结构和最小自由能折叠低于或等于-18千卡每摩尔(e)盟% 30 - 70%的范围内(31.].
用茎环RT-PCR验证miRNA
选择5个已知的miRNA和所有5个新型miRNA用于验证。根据Kramer,2011年,MiRNA特异性茎 - 环RT引物进行设计,2011年[32.](附加文件1).对每个miRNA制备cDNA (iScript™Select cDNA Synthesis Kit, Biorad),使用miRNA特异性正向引物和通用反向引物对每个miRNA进行PCR扩增。PCR产物洗脱,连接至pGEM®-T easy载体,并转化为活性体大肠杆菌DH5α细胞。分离重组质粒,分离和分析。
鉴定miRNA的目标预测
在硅靶基因预测中已知和新的miRNAsc . borivilianum使用靶基因预测软件,psrnatarget进行了完成[33.和米兰达[34.].鉴定的miRNA针对根和叶片转录组文库靶向c . borivilianum那栽培稻(rice, transcript, tiger (The Institute for Genomic Research) genome cDNA OSA1 Release 5 (OSA1R5), version 5)拟南芥(转录物,除去MiRNA基因,TAIR(拟南芥信息资源)版本10,2010_12_14发布的第10版)使用具有默认参数的PSRNATargetv2(2017释放)[最高期望:3;互补性得分的长度(HSPSIZE):19;目标可访问性 - 最大能量为目标网站(UPE):25;目标位点周围的侧翼长度用于靶辅助性分析:17 bp(上游)/ 13 bp(下游);导致翻译抑制的中央不匹配范围:10-11 nt]。psrnatarget可能针对每个miRNA序列和特定mRNA分子的多个靶位点分配多个靶,因为它考虑了目标的互补性和现场可访问性。一旦获得潜在的靶mRNA序列,将靶转录物经受BLASTX(基本局部对准搜索工具)从NCBI搜索NR(非冗余)数据库以预测潜在目标的函数。为了进一步了解MiRNA在皂苷生物合成途径中的作用,已知的已知和新的miRNA专门针对使用Miranda参与参与MVA和MEP途径的酶的转录物。使用Miranda使用Miranda进行了使用默认参数的最小自由能量(MFE)的详细统计研究[间隙开放罚分:-0.9;间隙延伸:-0.4; Score threshold: 50.0; Energy default: −0.20 Kcal/mol].
最后,使用Blast2GO 4.1软件在生物过程(BP)、分子功能(MF)和细胞成分(CC)水平检索所有识别的靶标的基因本体(GO)注释[35.].最后,鉴定的靶标在基因和基因组(KEGG)途径分析中受到京都百科全书的生化途径。
基于RT-qPCR的mirna表达分析及其预测靶标
为了确定mirna与靶点的关系,研究了植物的叶片组织c . borivilianum收集了两个阶段。一个,在年轻阶段(2个月大)和另一个在休眠阶段(7个月大)。选择总计11个保守的miRNA,其计算地预测,以调节编码皂苷生物合成中涉及的酶的基因。如前所述,设计了保守的miRNA的茎环RT-QPCR的引物[32.]使用底漆3.0输入软件设计靶基因的引物(附加文件1).
总RNA以及小RNA分离[24.根据前面描述的方案进行CDNA制备。使用SSOFAST™Evagreen®SuperMix制备MiRNA的RT-QPCR的反应鸡尾酒及其靶标,使用Bio-Rad CFX96™实时PCR系统进行实验。β-肌动蛋白用作内部对照。将1μl每种cDNA样品一起用于分析,以及10μl的Syber Green,7μl核酸酶的游离水,每种miRNA特异性前底漆和茎环互补的通用反向引物,在95℃下热开始的循环条件下对于2分钟,在95℃下的45个循环的变性15秒,在55℃下退火30秒并在72℃下延伸30秒。所有实验均以三倍一次进行。最后计算miRNA及其目标的折叠变化表达ΔδCT.使用方法[36.].2 ^( -ΔδCT.值更改为(log .)2)生成折叠变化表达式。
结果
原始数据分析
利用Illumina Hiseq 2000平台共生成1 × 50 bp的原始reads 79419,700条。去除低质量reads、短片段和适配器序列后,共得到22,155,316条干净reads(见表)1).清洁读取与各种数据库的对准鉴定了SIRNA(0.51%),PIRNA(3.10%),SNRNA(0.02%),Snorna(0.04%),TRNA(0.52%)和rRNA等其他非编码RNA的序列(11.30%)(11.30%)。然后过滤剩余的18,723,614(84.51%)读取,其中18,057,758次读取的长度17-35bp用于鉴定已知的miRNA。
已知的microrna的识别
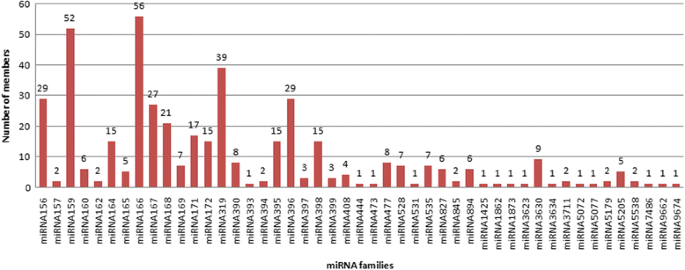
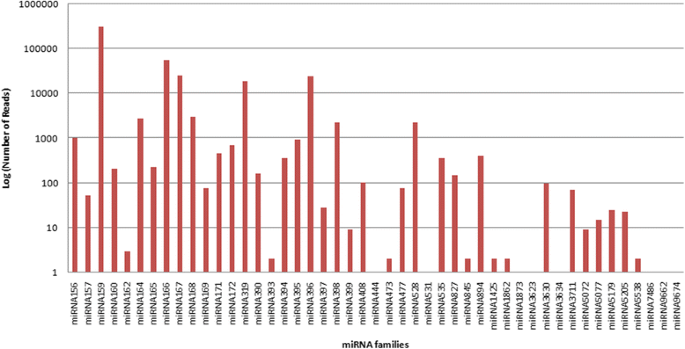
共鉴定出47个miRNA家族的442个已知miRNAc . borivilianum(附加文件2).通过添加数字后缀来分化不同成熟miRNA的命名法,通过添加一个数字后缀进行分化[37.那38.].通过这种方法,从一个共同的前体切片的miRNA物种可以被区分。在我们的研究中,我们发现每个miRNA都有多个同源物,认为miRNA有多个同源物但序列相似,属于同一家族。miR166家族有56个成员,其次是miR159(52个成员)家族,而miR393、miR444、miR473、miR531、miR1425、miR1862、miR1873、miR3623、miR3634、miR5072、miR5077、miR7486、miR9662、miR9674等14个miRNA家族只有1个成员。相同miRNA家族的成员并不反映核苷酸(nt)序列的相似程度,而是功能的对等,即具有共同的功能。每个家庭的成员数见图。1.观察到每个miRNA的总读计数,从而对miRNA的表观表达水平提供了一个思路c . borivilianum年轻的叶组织。其中,在生物信息学分析的基础上,观察到miR159家族的最大表达量为315,441个reads,其次是miR166和miR167家族,分别为56,445和25,592个reads,有17个miRNA家族的表达量小于10个reads。不同mirna的广泛表达预示着不同的mirna在植物的生长发育中发挥着不同的作用。图中提到了每个miRNA家族丰度的图形表示。2.在C.博伊西亚姆,miRNA长度在18-24 nt范围内,包含21 nt的miRNA最多(31.45%),其次是长度为20 nt的miRNA(附加文件)3.).
新的miRNA鉴定
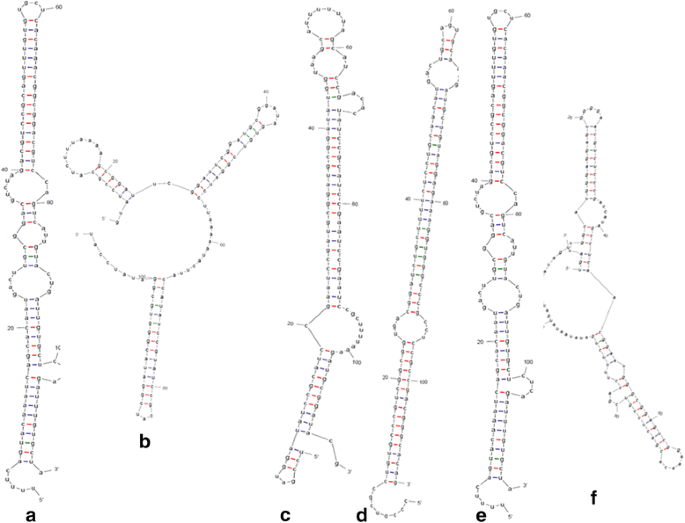
由于无法获得全基因组序列C.博伊西亚姆,根和叶片转录组数据被用作新的MiRNA预测的参考。总共审查了5个新的miRNA(表2), 2个与叶片转录组相关,3个与根转录组相关。有趣的是,一种miRNA,即cbo-miR1,在叶和根中有不同的前体,表达水平相同。这表明cbo-miR1在根和叶中都有功能,但有离散的起源。新发现的miRNA范围在21到24 nt之间,其中miRNA cbo-miR3最为丰富。植物特异性mirna的丰度变化范围很广,从cbo-miR2的28个reads到cbo-miR3的23252个reads。与以往许多研究相反,对新mirna的序列分析表明它们都不是以5'U开头的。miRNA前体形成茎环的能力可以证实其生物合成成熟miRNA的能力。因此,预测的新mirna前体被用于利用Mfold web服务器的RNA折叠形式(能量版本2.3)形成二级结构。这为每个折叠结构提供了dG(自由能)。图中描述了所有5种新型mirna的二级结构。3..发现CBO-miR3(Dg = -63.60 kcal / mol在37°C)是基于自由能和CON-miR5(Dg = -35.70千卡/摩尔在37℃)的最稳定的新型miRNA,最小稳定性c . borivilianum.茎 - 环RT-PCR进行,以确认在硅片鉴定已知的和新的miRNA。完成了miRNA的克隆,重组质粒的测序验证了计算识别的miRNA(附加文件4.).在随后的miRNA克隆和测序的硅分析中证实,新的miRNA确实是miRNA家族的一部分C.博伊兰人.
目标预测与基因本体
更好地理解识别的miRNA的功能c . borivilianum,对已知和新的mirna进行靶标预测。对nr (Non冗余)数据库进行BLASTX搜索,对潜在靶标进行标注。这些靶标被注释为转录因子,如GAMYB (Gibberellin和aba -regulated MYB)转录因子、生长素反应因子(Auxin Response factor, ARF)家族蛋白、生长调节因子、D-tyrosyl-tRNA(Tyr)脱酰酶、Squamosa启动子结合样(Squamosa promoter-binding-like, SPL)蛋白、UDP-glucuronate脱羧酶、富亮氨酸重复受体样丝氨酸/苏氨酸蛋白激酶,磷酸烯醇丙酮酸磷酸酶样,蛋白HUA ENHANCER 2-LIKE 1。大量的靶点被确定,表明mirna在调控植物关键功能中的作用,如非编码RNA生物发生、抗病、信号转导和胁迫反应(附加文件)5.).利用的转录组数据对miRNAs进行了平行靶预测o.苜蓿和A. Thaliana..为此,还发现了已知的miRNA的目标o.苜蓿转录组,A. Thaliana.转录组。目标的描述o.苜蓿和A. Thaliana.是否在附加文件中说明6.和7.分别。次生代谢物主要是影响黄酮类化合物广泛药用特性的主要因素c . borivilianum。因此,研究针对皂苷生物合成途径中mrna的mirna具有重要意义。利用miRanda对已知和新的mirna进行靶预测后,对转录本进行BLASTX分析,以找到功能蛋白。从根和叶的转录组中,发现了参与皂苷生物合成的靶点。角鲨烯环氧化酶、角鲨烯合酶、角鲨烯单加氧酶、甲戊酸二磷酸脱羧酶、甲戊酸激酶、磷酸evalonate激酶、叶绿体1-脱氧-d-木酮糖-5-磷酸合酶、香叶酰二磷酸合酶、二磷酸evalonate脱羧酶、环artenol合酶、细胞色素p450-90b1-like、4-羟基-3-甲基-2-烯基二磷酸还原酶、氧化角鲨烯环化酶、法尼基焦磷酸合酶、udp -糖基转移酶家族蛋白、糠醇糖苷26- o - β -葡萄糖苷酶、异戊烯基二磷酸异构酶2、羟甲基戊二醇辅酶a合酶、4-羟基-3-甲基-2-烯-1-基二磷酸合酶。
此外,还发现新的miRNA来调节重要的生物过程。来自叶片和根转录组的4个新型miRNA的总计7个靶标c . borivilianum被发现。Cbo-miR3调控编码转酮醇酶、细胞色素P450家族蛋白和环氧化物水解酶a的基因表达。新型miRNAs cbo-miR4和cbo-miR5分别靶向细胞色素P450家族蛋白、谷胱甘肽s -转移酶1和cbo-miR1靶向β -葡萄糖苷酶12样亚型的转录本编码。这表明,mirna与其靶标直接参与皂苷生物合成在附加文件中进行了描述8..
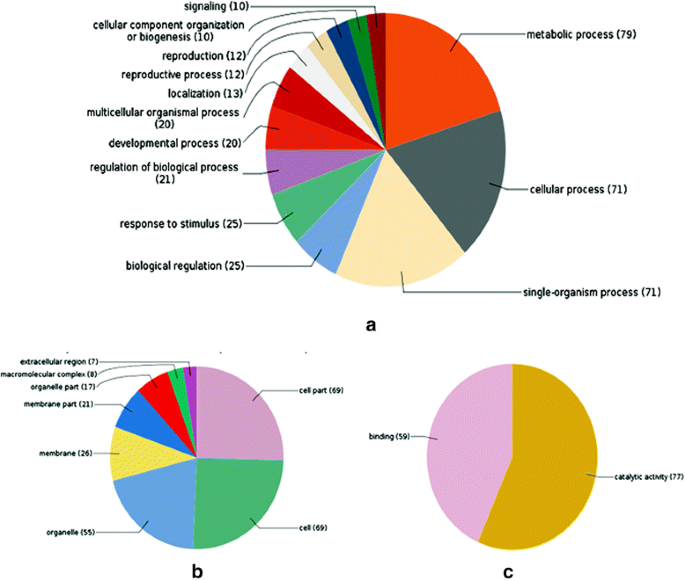
使用Blast2GO 4.1软件进行基因功能注释。在这里,我们根据它们在细胞过程、代谢过程和生物学作用中的作用对这些靶标进行了分类。对psRNATarget和miRanda预测的135个靶标进行BLASTX搜索、mapping和注释,使用GO分析进行分析和分类(图3)。4.).
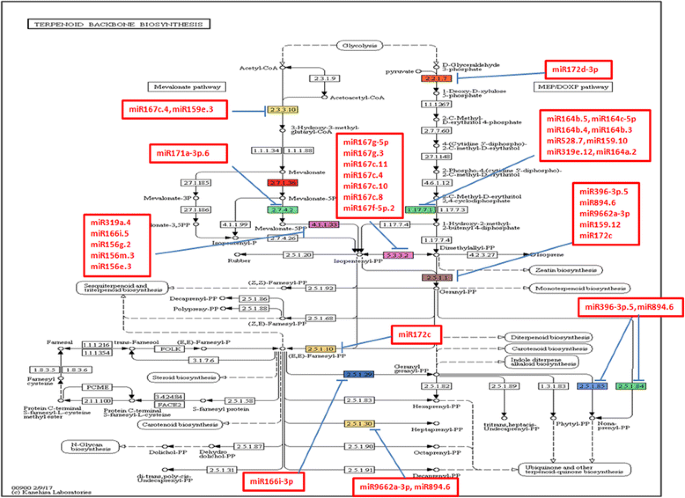
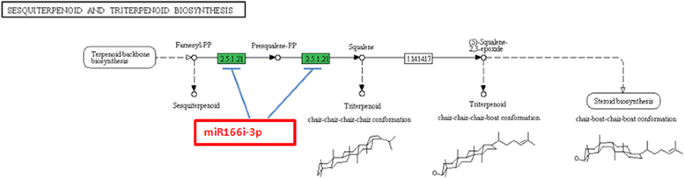
KEGG通路分析共发现135个靶点c . borivilianum转录组参与了43个代谢网络,包括淀粉和蔗糖代谢、半胱氨酸和蛋氨酸代谢、半乳糖代谢、花生四烯酸代谢、萜类主干生物合成、类固醇生物合成、丁酸代谢、倍半萜和三萜生物合成、戊糖磷酸途径。共29个mirna (miR172d-3p, miR164b。5、miR164c-5p miR164b。4, miR164b。3.那miR528.7, miR159.10, miR319e.12, miR164a.2, miR396-3p.5, miR894.6, miR9662a-3p, miR159.12, miR172c, miR167g-5p, miR167g.3, miR167c.11, miR167c.4, miR167c.10, miR167c.8, miR167f-5p.2, miR159e.3, miR171a-3p.6, miR319a.4, miR166i.5, miR156g.2, miR156m.3, miR156e.3, miR166i-3p) were found to regulate a single pathway i.e. terpenoid backbone biosynthesis (Fig.5.).然而,发现单一miRNA(miR166i-3p)调节筛氏萜素和三萜类生物合成(图。6.).预测的KEGG通路提供了有关mirna靶点功能的信息。
萜类化合物骨架生物合成表明microrna的位置和他们的目标基因,EC: 2.5.1.85-all-trans-nonaprenyl-diphosphate合成酶,EC: 2.5.1.84-all-trans-nonaprenyl-diphosphate合成酶,EC: 2.5.1.1-farnesyl二磷酸合酶和geranylgeranyl二磷酸合酶,EC: 2.3.3.10-hydroxymethylglutaryl-CoA合成酶,EC:5.3.3.2-isopentenyl-diphosphate Delta-isomerase EC: 2.5.1.10-farnesyl二磷酸合酶和geranylgeranyl二磷酸合酶,EC: 2.2.1.7-1-deoxy-D-xylulose-5-phosphate合成酶,EC: 2.7.4.2-phosphomevalonate激酶,EC: 4.1.1.33-diphosphomevalonate脱羧酶,EC: 2.7.1.36-mevalonate激酶,EC:2.5.1.29-香叶酰香叶酰二磷酸合酶,EC: 2.5.1.30-庚二磷酸基合酶,EC: 1.17.7.1-(E)-4-羟基-3-甲基-丁-2-烯基二磷酸合酶
miRNA及其目标的相对表达分析
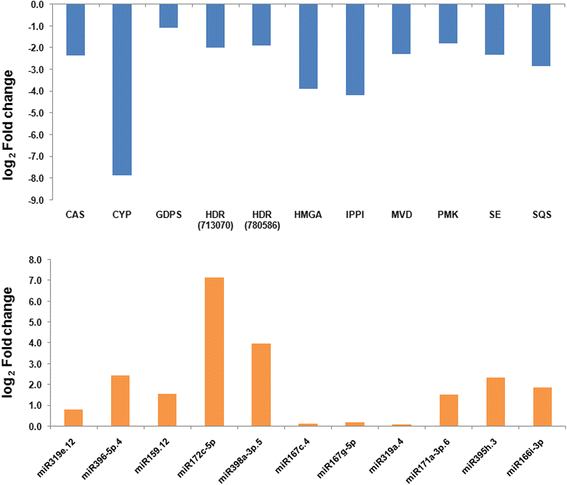
为了研究miRNA和相应的靶基因的关联,通过RT-QPCR验证了保守miRNA的11个选定靶基因的表达模式。表中提到了基因列表以及其靶向miRNA3..幼株叶组织的相对表达研究及成熟植物达到休眠阶段。幼株被认为是参考计算折叠变化表达。观察到皂苷生物合成中涉及的基因在休眠期间的表达低于活性生长期。同时,观察到miRNA表达模式与目标相反。表达细胞色素p450 90 b1-like(CYP.)下调了7.9 log倍,而相应的miRNA (miR396e-5p.4)上调了1.5 log倍。在相同的模式下,MVA途径的关键酶角鲨烯合成酶(Squalene synthase, SQS)基因编码的对数倍下调2.8倍,靶向miRNA (miR166i-3p)的相对上调1.9倍。图中很好地解释了所有靶标及其靶向mirna表达折叠变化的反向模式。7..mirna的这一链接及其靶点提示mirna在调控植物皂苷合成基因中的作用。mirna表达及其靶点的相对变化并不完全相同,这意味着每个靶点都可以被多个mirna调控[39.].
讨论
mirna是一种小型、非编码的单链调控元件,近二十年来一直受到人们的深入研究。最近,已经鉴定的植物mirna被发现有助于植物在逆境条件下的适应。这些发现表明,在干旱、盐度、极端温度、营养剥夺和重金属等非生物胁迫条件下,mirna表达的改变调节了植物的生长和发育。因此,mirna可以进一步用于基因调控,使作物更具抗逆性。在过去的几年里,科学研究一直致力于理解涉及mirna的次级代谢物的转录后调控。mirna直接改变植物化学物质的有效作用已经有报道,了解这一机制将有助于进一步提高植物次生代谢产物的浓度。2011年首次发表这类报告,揭示了miR156通过靶向调控花青素的数量SPL基因(40].此后,许多研究报告了miRNA在许多二级成分中的调节和生物合成中的作用,例如黄酮类,萜类化合物和生物碱[41.].Zhang等人2012年的里程碑式研究表明,食品中的miRNA可以安全地进入哺乳动物的肠道并进入血液.这表明,植物miRNAs可以用于调节靶基因的表达,甚至用于治疗许多人类疾病。因此,mirna被报道作为生物工程工具来改变植物和动物中的基因表达[42.].到目前为止,已经探索了许多植物。在水稻中,发现miR168结合人/小鼠低密度脂蛋白受体接合蛋白1(LDLRAP1)mRNA并降低肝脏中的蛋白质水平,从而降低了小鼠等离子体的LDL [43.].在姜黄,发现ath-miR167d同源物与eif2ak2.(酪氨酸蛋白激酶)和ZFYVE16.(Zinc Finger FYVE-Type)基因负责阻断内质网蛋白加工通路和调节内体通路的膜运输。这对治疗动脉硬化和高血糖等疾病很有帮助[44.].在Gmelina arborea那6个推测的mirna被发现在预防癌症、血源性疾病和其他尿路感染等疾病中发挥重要作用[45.].此外还有豆科、菊科、十字花科、山茶科、菊苣科、夹竹桃科、芸香科、茜草科、姜科、玄参科、没药科、马鞭草科、亚麻科、大戟科、茄科、五加科、松柏科、杨柳科、唇形科、锦葵科、Ericaceae、Vitaceae、和禾本科植物已被研究以探索它们的miRNA库[46.].这是对百合科单子叶药用植物miRNA的首次研究。广泛的治疗和药用特性c . borivilianum使miRNA学习做出了选择。c . borivilianum近十年来一直专注于二级代谢产物生物合成途径的研究。到目前为止,mirna及其在c . borivilianum是不知道。了解miRNA有助于了解该植物的其他分子和代谢研究中的环孔。在本研究中,我们应用深度测序结合生物信息学分析来识别和表征mirna及其靶标c . borivilianum.目前世界上还没有关于百合科植物sRNA的信息。因此,本研究可为进一步探索和应用生物活性物质的复杂代谢机制奠定基础c . borivilianum.通过深度测序产生的大量数据有助于识别数量更少和较小的mirna,因为分析更多的reads增加了恢复罕见转录本的几率[47.].
共有442个属于47个家庭和5个新的miRNA的MiRNAc . borivilianum叶srna图书馆。它表示,存在不同和高度复杂的小RNA人口c . borivilianum.在其他单码植物中已经观察到几乎类似的趋势。在山羊草属tauschii那Brachypodium distachyon那o.苜蓿那高粱,小麦,和Zea Mays.,已报道成熟mirna的数量分别为173、525、713、241、119和321 [27.].尽管miRNA基因序列可能不同,但属于同一家族的miRNA的种子区域可能是相同的[48.].关于miRNA注释的另一个困难是基因组中重复出现的同源miRNA位点,产生相同或几乎相同的成熟miRNA。基于这一原理,这些mirna可以被归为相同的家族[49.].其他物种miR156、miR157、miR159、miR160、miR162、miR164、miR165、miR166、miR167、miR168、miR169、miR171、miR172、miR319、miR390、miR393、miR394、miR395、miR396、miR397、miR398、miR399、miR408和miR444等高度保守的mirna [50.)也被观察到c . borivilianum在丰度高。新mirna或物种特异性mirna被认为在植物中发挥特定功能,而保守mirna被认为参与信号转导、叶和花的发育等广义功能[51.].保守mirna具有在测序基因组中由多个位点代表的独特特性;它们中的大多数是通过基因组复制事件产生的,这在一定程度上表明了它们的古老[52.].
不同物种的microrna保存模式不同。大多数情况下,mirna的保守性与其碱基含量和裂解位点等序列特征有关.植物miRNA显示出保护和Au / gc含量之间的负相关性/正相关。在5'结束时,保守的miRNA通常以基础U开始,而较少保守的miRNA在哺乳动物中的开始位置具有非U碱基。但在昆虫和植物的情况下,这是不是真的[48.].根据数据分析,可以预测miR159和miR166在叶片中表达量最大的时期是叶片的活跃生长时期。据报道,在其他植物中也有大量相同的miRNAsPanax Ginseng,甜叶菊雷博尼亚,和A. Thaliana.[53.那54.那55.].miR159是植物界最古老的mirna之一[52.].miR159家族成员通过调控MYB转录因子水平调控ABA胁迫反应和植物种子萌发A. Thaliana.[56.那57.].在番茄中,miR159通过靶向SGN-U567133调控叶片和花的发育[58.].由于样品是在植物生长活跃期采集的,miR159的高丰度表明其对叶和根的发育有积极的调节作用。
单子叶植物特有的mirna如miR437, miR444, miR396在单子叶植物物种如水稻,玉米,高粱和甘蔗中被报道[50.].在我们的研究中,我们发现只有miR396和miR444存在,而没有miR437。
完全参考基因组应该是SRNA-SEQ的主要限制因素。不幸的是,基因组c . borivilianum尚未公布,但转录的根和叶组织的研究c . borivilianum已经进行过。来自先前研究的转录组数据用作新的miRNA预测的参考。但是更多的mirnas来自c . borivilianum可以在未来得到完整的基因组信息时进行注释。此外,我们的研究发现了5种推测的新miRNAsc . borivilianum以该植物的现有序列数据为参考。MFE (Minimum Fold Energy)、序列长度和碱基组成是预测植物mirna的重要特征。因此,预测的新mirna通过形成其前体的二级结构得到了证实。所有新型miRNA前体均具有茎环发夹结构,且这种折叠式发夹结构的自由能较低,根据Mfold软件的预测。小说microrna在c . borivilianum发现源自前体,AU百分比为40 - 53%。miRNA的种子区可以结合靶mRNA的3 ' -UTR、5 ' -UTR和ORF区域,但在所有这些3 ' -UTR靶标中比其他两个靶标的频率要高得多[59.].
在本研究中,随着叶子和根的转录组数据c . borivilianum那o.苜蓿和A. Thaliana.转录组也被用来寻找最大的miRNAs靶标。靶抑制被发现是更频繁的裂解。这一结果支持了Argonaute2介导的切割可能是许多已知植物miRNAs基因抑制的主要方式[60.].此外,我们发现A. Thaliana.miR167靶向ARF6和ARF8, miR160和in靶向ARF10、ARF16和ARF17o.苜蓿ARF16 miR160;miR167靶向ARF6、ARF8.在c . borivilianumARF18和ARF17被miR160靶向;和miR167的ARF12。miR167下调番茄ARF6和ARF8,影响番茄花的发育和雌性不育[61.].这提示了miRNAs在功能上的相似性C. Borivilianum,O. Sativa,和A. Thaliana..
本研究通路特异性靶基因预测显示,miRNA可靶向全反式非戊二烯二磷酸合成酶、法尼基二磷酸合成酶、香叶基香叶基二磷酸合成酶、羟甲基戊二烯-辅酶a合成酶、异戊二烯基二磷酸delta-异构酶、1-脱氧-d -木酮糖-5-磷酸合成酶、磷酸evalonate激酶,二磷酸evalonate脱羧酶,甲戊酸激酶,庚二磷酸合成酶,和(E)-4-羟基-3-甲基-2-烯基二磷酸合成酶。
叶片转录组中已知mirna的靶标c . borivilianum被发现为蛋白质损伤,ARF,转录因子GamyB,生长调节因子,D-酪氨酸酶,SPL蛋白,含有NAC域蛋白,HD-ZIP蛋白,UDP-葡萄糖醛酸的蛋白质,HD-ZIP蛋白,UDP-葡萄糖醛酸酯脱羧酶蛋白,推定ABC1蛋白,乙烯 - 不敏感2同种型,UDP-N-乙酰葡糖胺二磷酸化酶,富含纯度的重复受体样丝氨酸/苏氨酸 - 蛋白激酶,色氨酸合酶,DEA(D / H) - 盒RNA螺旋酶家庭蛋白,和磷酸丙烯酸磷酸酶样。在靶分析期间,观察到以前据报道的一些miRNA调节初级代谢途径正在调节初级和二次代谢途径c . borivilianum.例如,miR172d-3p。2target ABC1 protein and Chloroplast 1-deoxy-d-xylulose-5-phosphate synthase; miR172c target APETALA2-like protein and Farnesyl pyrophosphate synthase; miR172c-5p Ethylene-Insensitive 2 isoform X2 and 4-Hydroxy-3-methylbut-2-enyl diphosphate reductase; miR156g.2, miR156e.3 and miR156m.3 target SPL12 and Farnesyl pyrophosphate synthase. Detailed list of such miRNAs are mentioned in Additional file9..
利用Blast2GO进一步分析目标预测结果。研究发现,信号传递、对刺激的反应、发育过程、生殖过程等生物过程都是由mirna调控的。这提示了miRNA的广泛调控c . borivilianum.确定的两个最重要的途径是萜类主干生物合成和倍半萜和三萜生物合成。这提示了mirna在次级代谢中的特殊作用。预测的靶基因及其产物的功能,可能为研究人体必需的生物学过程和代谢提供有价值的线索c . borivilianum.
采用RT-qPCR方法计算mirna及其靶mrna的表达水平。在植物两个生长期的叶片中测定其表达。所有11个mirna均在休眠期间表达增加。miR172c-5p和miR398a-3p的表达变化最大。5,靶向4-羟基-3-甲基-2-烯基二磷酸还原酶。这说明多种mirna可以共同调控同一基因。mirna在休眠过程中各自的上下调节及其作用靶点,表明mirna在调控次生代谢产物积累中发挥了重要作用。它确保了mirna直接参与负调控。目前的研究遵循了其他一些研究人员之前讨论过的相同的情景[41.].
结论
利用生物信息学工具共鉴定出442个已知mirna和5个新mirna。发现植物特异性miRNA和已知miRNA与MVA/MEP通路相关。这有助于建立miRNA和次级代谢之间的相关性。mirna靶向参与次级代谢产物生物合成途径的基因,可以进一步细化mirna对基因的调控,从而调节其表达。我们的研究还发现一些mRNA编码转录因子作为mirna的靶点,提示mirna在植物生长发育中的作用。GO和KEGG分析为进一步研究提供了基础。RT-qPCR结果提示,休眠期间次级代谢产物的合成减慢,有助于理解mirna在次级代谢中的作用。这将有助于开发的数据库c . borivilianum有助于设计调控次生代谢产物含量的RNAi实验。
缩写
- 0.C:
-
摄氏度
- A. Thaliana.:
-
拟南芥
- ACA:
-
1'-乙酰氧基吡啶醇乙酸盐
- 东盟地区论坛:
-
养阴呼吸因子
- 爆炸:
-
基本的局部比对搜索工具
- 英国石油公司:
-
碱基对
- 英国石油公司:
-
生物过程
- c . borivilianum:
-
吊兰borivilianum
- 答:
-
蜂窝组件
- DG:
-
自由能
- doxp:
-
1-脱氧-D-木糖糖5-磷酸盐
- EIF2AK2:
-
酪氨酸蛋白激酶
- 去:
-
基因本体论
- Hiseq:
-
高通量测序
- HSP:
-
互补性得分的长度
- KEGG:
-
Kyoto基因和基因组的百科全书
- ldlrap1:
-
低密度脂蛋白受体适配器蛋白1
- 议员:
-
非麦克风
- MF:
-
分子功能
- mf:
-
最低折能量
- mg:
-
毫克
- MVA:
-
甲戊酸酸
- NT:
-
核苷酸
- o.苜蓿:
-
栽培稻
- SPL:
-
Squamosa启动子结合蛋白质
- TAIR:
-
拟南芥信息资源
- UPE:
-
目标可及性-最大能量解对目标位置
- ZFYVE16:
-
锌指FYVE-Type
- μl:
-
微升
参考文献
- 1.
胡克JD。《英属印度植物志》,第6卷,伦敦:L. Reeve & Co;1894.
- 2。
标题沙塞特岛吊兰属一新种。J Bombay Nat Hist Soc. 1955; 52:896-900。
- 3。
提高植物药物生产的基因组和倍性操作。植物遗传资源。2005;3(2):170-7。
- 4。
王志强,王志强。印度一种重要药用植物吊兰的特性、遗传改良及栽培。植物遗传资源。2005;3(2):264-72。
- 5。
Thakur M,Bhargava S,Dixit V.叶绿素的免疫调节活动。F.基于EVID的补充替代MED。2007; 4(4):419-23。
- 6。
叶兰水提物对链脲佐菌素诱导的高血糖大鼠抗糖尿病活性的初步研究。中国生物医学工程学报2009;2(1):5-53。
- 7。
王志强,王志强,王志强,等。几种瓦吉卡兰中药对雄性小鼠悬垂活性和体外精子数的影响。性Disabil。2007;25(4):203 - 7。
- 8。
Thakur M,Dixit V.叶绿素疾病对雄性大鼠雄激素和性行为的影响。印度药物 - 孟买。2006; 43(4):300。
- 9。
植物皂苷的色谱测定。中国科学(d辑:地球科学)2002;
- 10.
Rohit S,Nidhi S,Gulab Ts,Bhagwan SS,Pallavi J.来自叶绿素的皂苷提取的常规方法。et fernand。Global J Res Med植物土着Med。2014; 3(2):33。
- 11.
关键词:植物,皂苷,定量,定性分析吊兰borivilianum.2015;6(1): 826-35。
- 12.
Acharya D, Mitaine-Offer AC, Kaushik N, Miyamoto T, Paululat T, Lacaille-Dubois MA。从吊兰根中提取的糠甾体类皂苷。Helvetica Chimica Acta 2008;91(12): 2262-9。
- 13。
Acharya D,Mitaine-Report A-C,Kaushik N,Miyamoto T,Paululat T,Mirjolet JFO,Duchamp O,Lacaille-Dubois MA。细胞毒素螺旋烷型皂苷来自叶绿素的根源。J Nat Prod。2009; 72(1):177-81。
- 14。
吊兰中植物成分的分离与鉴定。制药研究》2010;2(6):343。
- 15.
Chua B,Abdullah Z,Pin Ky,Abdullah LC,Choong Tsy,尤斯英国。1'-乙酰氧基吡啶(ACA)的分离,结构阐明,鉴定和定量分析来自叶绿素肺炎(SAFED Musli)的根源。j eng sci technol。2017; 12(1):198-213。
- 16。
新转录组测序揭示了植物吊兰的重要分子网络和代谢途径。PLoS ONE。2013; 8 (12): e83336。
- 17。
Kumar S,Kalra S,Kumar S,Kaur J,Singh K.从叶绿素博洛安的叶片和根组织的差异表达转录物:一种具有高药用价值的植物。基因。2012; 511(1):79-87。
- 18。
叶绿藻角鲨烯合成酶基因的克隆与表达分析。弗尔南多。)。生物科技摩尔》。2013;54(3):944 - 53。
- 19。
Kumar S,Kalra S,Singh B,Kumar A,Kaur J,Singh K.RNA-SEQ介导的叶绿素疾病疾病的根转录组分析,用于鉴定皂苷生物合成中涉及的基因的基因。Funct综合基因组学。2016; 16(1):37-55。
- 20.
Dave S,Tarafdar JC。安全穆斯林(叶绿素玻利维族)菌株菌根真菌的刺激合成皂苷。int res jageric sci土壤sci。2011; 1:137-41。
- 21.
Bulgakov vp,Avramenko电视。植物中次生新陈代谢调节的新机会:专注于微大罗纳斯。Biotechnol Lett。2015; 37(9):1719-27。
- 22.
3 .陈志刚,陈志刚,陈志刚,陈志刚。Mentha spp次生代谢途径中mirna的鉴定及其靶点。计算机生物化学。2016;64:154-62。
- 23.
魏河,邱德,威尔逊IW,赵立,鲁斯,苗族,冯斯,白升,吴Q,涂Panax诺辛根的新型和保守的小罗风的识别。BMC基因组学。2015; 16(1):835。
- 24.
Ghawana S,Paul A,Kumar H,Kumar A,Singh H,Bhardwaj PK,Rani A,Singh Rs,Raizada J,Singh K.富含次生代谢物的植物组织RNA隔离系统。BMC RES笔记。2011; 4(1):85。
- 25。
Martin M. Cutadapt从高通量测序读取中移除适配器序列。EMBnet j . 2011; 17(1): 10 - 2。
- 26。
Langmead B,Salzberg SL。与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9(4):357-9。
- 27。
Griffiths-Jones S,Saini H,Van Dongen S,Engright A. Mirbase:MicroRNA基因组学的工具。核酸RES。2007; 36:154-58。
- 28。
Langmead B,Trapnell C,Pop M,Salzberg SL。超快和记忆高DNA序列对人类基因组的对准。基因组Biol。2009; 10(3):R25。
- 29。
Friedländer MR, Chen W, Adamidi C, Maaskola J, Einspanier R, Knespel S, Rajewsky N.利用miRDeep从深度测序数据中发现microrna。生物科技Nat》,2008,26(4):407 - 15所示。
- 30.
用于核酸折叠和杂交预测的zucker M. Mfold web服务器。核酸学报2003;31(13):3406-15。
- 31.
张波林,潘小平,王清良,George PC, Anderson TA。利用EST分析鉴定植物新microrna。细胞杂志2005;15(5):336 - 60。
- 32.
克莱默曼氏金融。茎环RT-qPCR用于miRNAs。2011;第15章,第15.10单元。
- 33.
戴X,赵px。psrnatarget:植物小型RNA目标分析服务器。核酸RES。2011; 39(SUPPLY_2):W155-9。
- 34.
John E. Gaul Microrna在果蝇中的目标。基因组Biol。2003; 5(1):R1。
- 35.
Blast2GO:植物基因组学功能分析的综合套件。植物基因组学杂志。2008;2008
- 36.
Livak KJ,Schmittgen TD。实时定量PCR的相对基因表达数据分析及2ΔΔCT方法。方法。2001; 25(4):402-8。
- 37。
孟Y, Shao C, Wang H, Chen M. Are all the mirbase registered microrna true?基于结构和表达的植物复核。RNA杂志。2012;9(3):249 - 53。
- 38。
MicroRNA命名法和修订命名处方的必要性。功能基因组学。2015;15(1):65-71。
- 39。
microrna与胃癌靶基因的多对多关系。8。2013;(5):e62589。
- 40.
苟建勇,王建伟。mir156靶向的SPL转录因子对拟南芥花青素合成的负调控植物细胞。2011;23(4):1512 - 22所示。
- 41.
Gupta Op,Karkute SG,Banerjee S,Meena NL,Dahuja A.当代对植物中次级代谢物生物合成的基于MiRNA的调控。前植物SCI。2017; 8:374。
- 42.
张丽丽,侯东,陈晓丽,朱丽,张颖,李军,Bian Z,梁晓霞,蔡晓霞。外源植物MIR168a对哺乳动物LDLRAP1的调控作用:microRNA的跨界调控。细胞研究》2012;22(1):107 - 26所示。
- 43.
王克,张某,马铃木B,脱脚P,Brighman A,Hu Z,Hood Le,Galas DJ。循环微小RNA,潜在的药物诱导肝损伤的生物标志物。PROC NATL ACAD SCI。2009; 106(11):4402-7。
- 44.
Rameshwari R,Singhal D,Narang R,Maheshwari A,Prasad T.在Curcuma Longa中MiRNA的硅预测中,他们在人类代谢组学中的作用。int j adv biotecres。2013; 4:253-9。
- 45.
Dubey A,Kalra SS,Trivedi N.MiRNA在Gmelina arborea的计算预测及其在人类代谢组中的作用。Am J Biosci Bioeng。2013; 1:62-74。
- 46.
miRNA转移对植物治疗活性的影响。生物医学Pharmacother。2015;74:228-32。
- 47.
Jones-Rhoades兆瓦。植物microrna的保守与分化。植物学报。2012;80(1):3-16。
- 48.
Shi B,Gao W,Wang J.微小Ranna保守的序列指纹。Plos一个。2012; 7(10):E48256。
- 49。
Meyers Bc,Axtell MJ,Bartel B,Bartel DP,Baulcombe D,Bowman JL,Cao X,Carchton JC,Chen X,Green PJ。植物微稻草注释标准。植物细胞。2008; 20(12):3186-90。
- 50.
Sunkar R,Jagadeeswaran G.在大量多种植物物种中的Silico识别保守MicroRNA。BMC植物BIOL。2008; 8(1):37。
- 51.
张斌,潘X, Cobb GP, Anderson TA。植物microRNA:一种影响很大的小调控分子。Dev杂志。2006;289(1):3-16。
- 52.
Axtell MJ,Bowman JL。植物微小RNA的演变及其目标。趋势植物SCI。2008; 13(7):343-9。
- 53.
关键词:人参,microrna,小RNA转录组,高通量测序《公共科学图书馆•综合》。2012;7 (9):e44385。
- 54.
Mandhan V, Kaur J, Singh K. smRNAome profiling鉴定甜菊糖(Stevia Rebaudiana Bertoni)中保守的和新的microrna。植物学报。2012;12(1):197。
- 55.
Rajagopalan R,Vaucheret H,Trejo J,Bartel DP。在拟南芥中的微小Radorna多样化和进化的流体套。基因开发。2006; 20(24):3407-25。
- 56.
Reyes JL,Chua NH。ABA诱导MIR159控制拟南芥种子萌发期间两种MYB因子的转录水平。工厂J. 2007; 49(4):592-606。
- 57.
Achard P,Herr A,Baulcombe DC,Harberd NP。胃植物监管微稻草的花卉发育调节。发展。2004; 131(14):3357-65。
- 58.
Buxdorf K,Hendelman A,Stav R,Lapidot M,Ori N,Arazi T.鉴定和表征与番茄中的MyB无关的新型miR159靶标无关。Planta。2010; 232(5):1009-22。
- 59.
Bartel DP。MicroRNAs:目标识别和调控功能。细胞。2009;136(2):215 - 33所示。
- 60.
ARGONAUTE蛋白的结构、功能和调控。植物细胞。2010;22(12):3879 - 89。
- 61.
Liu N, Wu S, Van Houten J, Wang Y, Fei Z, Fei Z ., Clarke TH, Reed JW, Van Der Knaap E. effect of AUXIN RESPONSE factor 6 and 8 by microRNA 167下调番茄花发育缺陷和雌性不育。[J] .机器人。2014;65(9):2507-20 .]
致谢
作者感谢印度CSIR为开展研究工作提供资金。Monika Kajal感谢印度大学拨款委员会提供的奖学金。我们非常感谢Ravindresh Chhabra博士在RT-qPCR数据分析方面的帮助。
资金
作者感谢印度CSIR为开展研究工作提供资金。
数据和材料的可用性
与本研究相关的SRA数据已以生物工程ID PRJNA415558 (SRP121214)提交给NCBI。
作者信息
从属关系
贡献
KS;构思构思,设计实验,分析结果,最终确定MS. MK;完成了所有的实验,分析了数据,编辑了结果,写了ms。所有的作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用
同意出版物
不适用
利益争夺
提交人声明他们没有竞争利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
额外的文件1:
miRNA茎环RT-PCR引物序列及miRNA序列。(多克斯16 kb)
额外的文件2:
已知的miRNA鉴定在幼叶小RNA群体中c . borivilianum.(XLSX 31 kb)
额外的文件3:
中每个miRNA长度的频率百分比c . borivilianum。(TIFF 23 KB)
额外的文件4:
茎环RT-PCR的测序结果。(DOCX 11 KB)
额外的文件5:
从叶和根的转录组中鉴定了已知mirna的靶点c . borivilianum使用psrnatarget。(docx 77 kb)
额外的文件6:
已知的MIRNA的鉴定目标栽培稻转录组使用psrnatarget。(XLSX 88 KB)
额外的文件7:
已知的MIRNA的鉴定目标拟南芥转录组使用psrnatarget。(XLSX 81 KB)
额外的文件8:
使用Miranda的皂苷生物合成途径特异性靶标来自根和叶片转录组。(docx 23 kb)
额外的文件9:
参与调节初级和次级代谢的mirna列表。(多克斯15 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Kajal, M., Singh, K. Small RNA profiling for identification of microrna involved in regulation of saponins biosynthesis in吊兰borivilianum.BMC植物BIOL.17,265(2017)。https://doi.org/10.1186/s12870-017-1214-0.
收到了:
公认:
发表:
关键字
- 微
- 吊兰borivilianum
- illumina测序
- 皂苷
- 甲羟戊酸途径