抽象的
背景
长末端重复(LTR)反转座子在植物基因组中非常丰富,它们的增殖复制方式需要转录活性。这些序列存在于植物基因组中,作为主要元素超科中的不同亚系(即,吉普赛和copia).虽然这些元素的转录活性越来越被认为是植物转录组的一个常规属性,但目前尚不清楚这些元素的不同亚系在物种内部和跨物种的转录活性的程度。在本报告中,我们利用下一代测序方法检测了三种二倍体野生向日葵中不同LTR逆转录转座子亚系的基因组拷贝数丰度及其相应的转录活性水平。向日葵agrestis),H. Carnosus.和H. Porteri。
结果
所调查的二倍体向日葵品种的基因组大小差异为2.75倍,2C值为22.93h . agrestis),12.31 for.H. Carnosus.8.33,H. Porteri..同样多样化吉普赛和copiaLTR反转录转座子的亚系被跨物种鉴定,但与吉普赛序列总是比copia和全球合作吉普赛序列丰富与核基因组大小正相关。检测到转录活动copia和吉普赛序列,检测到的活性水平明显更高copia与吉普赛.有趣的是,在被鉴定为转录活性的11个元素中,5个在所有3个物种中均可检测到表达,3个在两个物种中均可检测到表达。
结论
对三种向日葵的LTR逆转录转座子基因组丰度和转录活性的联合分析,为这组向日葵的基因组大小进化和转座子动态提供了新的见解。尽管不同物种的核基因组大小存在相当大的差异,但仍观察到相对保守的LTR逆转录转座子转录活性模式,其序列高度重叠copia和吉普赛序列被观察到跨物种的转录活性。较高的比例copia与吉普赛元素被发现是转录活性的,这些序列也在更高的水平表达。
背景
长末端重复(LTR)反转座子是在多种生命形式的核基因组中发现的第1类转座子[1].这些元素在植物基因组中复制尤其成功[2,3.在那里,它们通常构成大部分的核DNA。吉普赛和copia元素代表在植物中发现的主要自治超科,但也认识到其他非自治形式(如TRIMs, LARDs) [4].
LTR回收转换的“复制和粘贴”转置模式需要转录激活,并用作用于翻译和逆转录的模板的结果RNA分子[5].翻译产物(如gag、RT、INT)在多步自主生命周期中起作用,而逆转录产物则作为插入基因组新位置的物理子拷贝。这种以转录为基础的复制转座模式使LTR逆转录转座子能够实现植物基因组中异常高的拷贝数。
长期以来一直有人认为,在正常条件下,由于宿主基因组的表观遗传抑制,LTR逆转录转座子转录不活跃。自然选择应该倾向于抑制元素,因为不受抑制的元素活性可能产生负的诱变作用。然而,越来越多的人认识到,LTR反转录转座子的转录活性比以前认为的更为普遍,实际上可能是植物转录组的一个常规属性[6,7,8,9,10.,11.,12.].
虽然高度重复,但LTR逆转录转座子存在于植物基因组中,作为更大的分类群(即超科)中不同亚系所代表的不同序列群体吉普赛和copia).基因组拷贝数不同亚系中元素的丰度可能是高度可变的,来自少数亚系的元素通常构成总基因组丰度的不成比例的大比例[13.,14.,15.,16.,17.].转录活性的LTR反转座子在多大程度上代表了植物基因组中存在的全部元素多样性尚不清楚,也不清楚特定亚系的表达水平与基因组拷贝数丰度之间的关系。在目前的报告中,我们使用NGS测序来研究这些关系在三个二倍体野生体向日葵通过比较向日葵多个亚系的基因组丰度和转录活性水平吉普赛和copiaLTR反转位子活动。我们特别关注基因组丰度和转录活性的多样性面板的40个全长吉普赛12个全长copia以前所示的元素表示向日葵中的LTR回收转换多样性的大部分[16.,17.,18.,19.].普通向日葵中这些元素的插入年龄估计向日葵表明大多数是最近活跃的,平均插入年龄为70万年h . annuus基因组[19.].
向日葵物种一直是一系列调查的焦点,检查LTR反转座子及其对基因组进化的贡献(在Giordani等人的综述[20.]),这些序列的转录活性已在该属内的多个物种中被报道[7,12.,17.,21.,22.,23.].目前研究中正在调查的物种包括h . agrestis),估计二倍体基因组规模最大的向日葵品种[17.,24.,以及其他的物种H. Carnosus.和H. Porteri..这三种向日葵品种在美国东南部的地理分布范围相对有限,其种群为h . agrestis)和H. Carnosus.主要限于佛罗里达州[25.,26.]和人口H. Porteri.局限于阿拉巴马、乔治亚和南卡罗莱纳东部皮埃蒙特地区南部的花岗岩露头[25.,26.,27].尽管范围和地理发生了类似,但这些物种占据了不同的位置向日葵系统发生,有H. Carnosus.属于东南多年生地区,h . agrestis)一年生的一年生植物,包含所有多年生植物的单系群向日葵,用H. Porteri,也是一年生植物,位于所有的基部向日葵[26.].这些物种的系统发育放置使LTR转回转化基因组的丰度和转录活性模式在向日葵属中广泛地解释。
方法
植物材料和生长条件
本研究中使用的植物种类的种子是从美国农业部(USDA)国家植物种质制度(表1).种子在培养皿中潮湿的滤纸上发芽,然后转移到10厘米的塑料罐中,罐中装有米洛混合350:万能砂的混合物(2:1)。植物在堪萨斯州立大学的温室设施中生长,光照周期为16小时:8小时,并有补充照明。在同一日期,从每个物种的三个生物复制中,从成熟植株中收获成熟的、完全展开的叶子,在液氮中快速冷冻,并在进行以下处理之前在−70°C下保存。
基因组大小估计
核基因组大小由流式细胞术测定,使用的是番石榴PCA-96微毛细管系统(Guava Technologies, Hayward, CA.)。样品制备包括用刀片共切约30毫克的新鲜叶子组织作为样品和一个内部标准(Secale麦片简历。Dankovské)在Bino等人改良的核隔离缓冲液中[28由15 mM HEPES、1 mM EDTA、80 mM KCl、20 mM NaCl、300 mM蔗糖、0.5 mM精胺、0.25 mM PVP-40、15 mM β-巯基乙醇和0.2% Triton-X组成。样品经30 μm尼龙网过滤,离心收集细胞核,用碘化丙啶溶液(BioSure)染色。对3个不同居群的核基因组大小进行了估算h . agrestis)和H. Porteri.对于两种不同的人群H. Carnosus.(桌子1).每个种群进行3次生物重复,共24个样本。
DNA和RNA提取,文库构建和测序
使用DNeasy植物迷你试剂盒(Qiagen,valencia,Ca)和Trizol试剂(Invitrogen,Carlsbad,CA)从叶组织中提取DNA和RNA,以后的制造商说明。纯化总RNA以避免使用DNase I和RNEANY MINI KIT(QIAGEN,VALENCIA,CA)的任何基因组DNA污染。每种样品的一个微图谱和RNA的总DNA和RNA在Illumina Hiseq2500平台上用于文库制备和测序,为两个数据集产生2×100bp配对读数。在Illumina Truseq DNA PCR-Free文库制备试剂盒和Illumina Truseq RNA样品制备试剂盒V2(Illumina Inc.,San Diego,Ca,Ca,美国)进行图书馆制剂。平均刀片尺寸分别为DNA和RNA文库分别为470bp和260bp。图书馆建设和测序是在堪萨斯大学基因组测序核心设施,劳伦斯,KS,USA(http://gsc.drupal.ku.edu/).
使用CLC基因组学工作台V8.5.1(QIAGEN,VALENCIA,CA)修剪来自DNA和RNA-SEQ的原始读数,以分别去除低质量序列(PHRED分数<30)和序列<80 bp和<40 bp。根据程序重复交换机进行更严格过滤DNA-SEQ数据[29要求输入长度≥80 bp的序列进行分析。通过质量筛选,鉴定叶绿体和线粒体来源的序列,并对其叶绿体全基因组(GenBank登录号DQ383815)和线粒体全基因组(KF815390)进行mapping reads向日葵.
基因组重复的部分
使用由Novak等人开发的基于图的聚类方法测定每种物种的基因组重复部分。[29],并在RepeatExplorer中实现[30.]在Galaxy Server上(http://www.repeatexplorer.org/).简单地说,从每个物种的每个生物复制中随机抽取大约3m个单端DNA-seq reads,并基于序列相似性(≥90%)和序列重叠(≥55%)的全域比较进行聚类。如果包含≥0.01%的起始采样序列(例如,3 M序列,最小聚类大小= 300个序列),则对个体聚类进行识别,并按基因组重复部分计数。这些参数值表示RepeatExplorer的默认设置。
LTR-RT识别和全局表达
从DNA-SEQ读数获得的序列簇被确定为吉普赛和copia基于相似性搜索的LTR反转座子,在RepeatExplorer中实现,针对一个由40个全长组成的定制数据库进行聚类输出吉普赛12个全长copiaLTR反转座子来源于h . annuus基因组[18.,19.].这些序列代表了在基因组中发现的多样化和可变的丰富元素。h . annuus[19.],几个额外的向日葵物种(17.],以及在外面的菊科的其他物种向日葵[16.].这些序列可作为Tetreault和Ungerer中的补充文件S1 [17.].我们遵循了[17.]进行LTR-RT鉴定和表达分析。
通过使用CLC基因组学工作台V8.5.1(QIAGEN,VALENCIA,CA)绘制滤波后的RNA-SEQ读取滤波的RNA-SEQ读取滤波器的RNA-SEQ估计转录活动≥80%。每个映射的配对端读数被计算为一个映射片段,每个映射映射读取。每个upblineage的映射片段的映射片段被焦点内全长元素的平均长度标准化吉普赛和copia通过筛选的reads总数(每千碱基每百万序列的片段数,FPKM)来量化表达水平。转录活性元件定义为FPKM值≥1。转录活性分析中只使用了唯一的mapping reads,尽管当使用多重mapping reads进行分析时,结果在定性上是相似的。
基因组重复部分与表达水平的相关性
皮尔森的r计算以评估内部超级素数的基因组重复级分和表达水平之间的相关性吉普赛和copia超级美食。为了考虑不同upblineoges的进化历史,用Phylip V3.695的对比进行系统发育独立对比(图片)[31,32].的极大似然树吉普赛和copia元素使用Mega V6.0构建[33基于逆转录酶结构域的氨基酸序列。通过ProtTest v2.0估计最佳的蛋白序列进化模型[34].
结果
核基因组大小,测序数据和基因组重复分数
在种内,核基因组大小估计的差异为22.29-23.31h . agrestis),12.07-12.55H. Carnosus.和8.16-8.56H. Porteri..基于取样,同一物种种群间2C值的差异不显著(h . agrestis),F= 1.8792,P= 0.23;H. Carnosus.,t = 0.7403,P= 0.50;h . poteri,F= 3.1982,P= 0.11)。核基因组大小在不同物种间的差异为2.75倍,平均估计为22.93倍h . agrestis)12.31H. Carnosus.8.33,H. Porteri.(桌子1).
Illumina Hi-Seq平台对被调查物种的DNA-和RNA-seq分别生成了34.0 M到63.5 M和35.8 M到56.9 M的原始reads(表1)1和额外的文件1:表S1)。修剪和质量过滤后,最终数据集中的读数的数量分别为24.6米至48.0米和32.6米至51.7米。从原始尺寸的101个基础的读取长度降低到99.6个碱基的平均长度用于RNA-SEQ的DNA-SEQ和93.8基础(附加文件1:表S1)。
在RepeatExplorer中实现的基于图的每个物种~ 3m DNA-seq读取聚类[30.]产生的基因组重复分数估计为83.33±0.02%h . agrestis)74.48±0.04%为H. Carnosus.,74.78±0.20%H. Porteri.(桌子1).的重复分数估计h . agrestis)(82.12±0.15%)[17.和新生成的估算H. Carnosus.和H. Porteri.是在上限,但在范围内,报告其他二倍体向日葵品种(68.17% - 75.26%)[17.].
LTR-RT鉴定和丰度
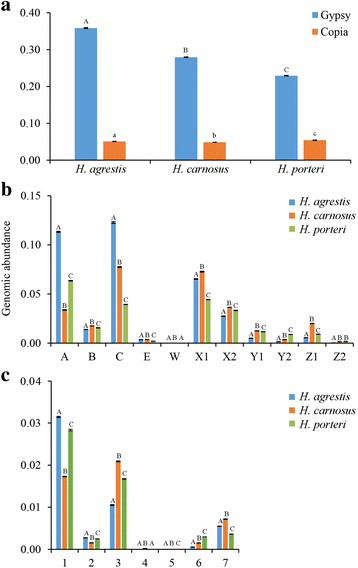
识别和量化属于不同注释面板的DNA-seq读取吉普赛和copia之前在普通向日葵中发现的LTR反转座子(h . annuus)的基因组,基于图的聚类分析的聚类输出数据的序列相似性搜索对40个小组进行了分析吉普赛和12copia全长LTR-RT序列最初在Buti等人报道[18.和Staton等[19.].22.92%±0.07 (H. Porteri.)及35.85%±0.12 (h . agrestis)),鉴定为吉普赛, 4.86%±0.01 (H. Carnosus.)及5.41%±0.04 (H. Porteri.)被确认为copia(无花果。1),吉普赛序列的丰富程度是copia这些物种基因组中的元素。而两种read类型的物种之间的丰度存在显著差异(吉普赛,F = 5.246,P < 10−9;copia,F= 118,P < 10−4,无花果。1),只有吉普赛序列丰度与核基因组大小呈正相关(r= 0.998,P = 0.040 for吉普赛;r=−0.494,P= 0.671copia额外的文件1:图S1)。
在本研究中使用的LTR-RT参考面板中,序列多样性相当大,有40个全长序列吉普赛12个全长copia基于逆转录酶氨基酸结构域的系统发育分析(附加文件,包含多个良好的良好的余介质的序列(附加文件1:图S2)。因此,我们检查了这些不同的基因组丰度吉普赛和copia在这种多样性背景下的分支。3种向日葵不同亚系的DNAseq reads的密度存在较大差异,密度为吉普赛读数范围从3.97 × 10−6为0.12h . agrestis)从8.56 × 10−6为0.08H. Carnosus.从3.56 × 10−6为0.06H. Porteri.和密度copia读数范围从5.67 × 10−7为0.03h . agrestis)从2.78 × 10−6为0.02H. Carnosus.从1.16×10−5为0.03H. Porteri.(无花果。1 b和c).在个体的分支内吉普赛和copia,在所有情况下,物种间的DNA-seq read丰度均存在显著差异,最大的种间差异表现为在h . agrestis)两个分支吉普赛(属A和属C;无花果。1 b).
表达特点
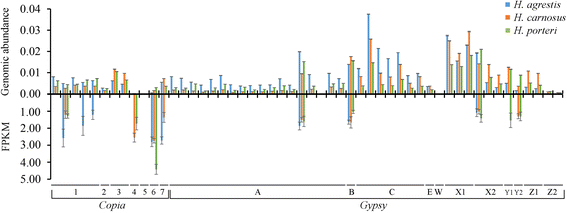
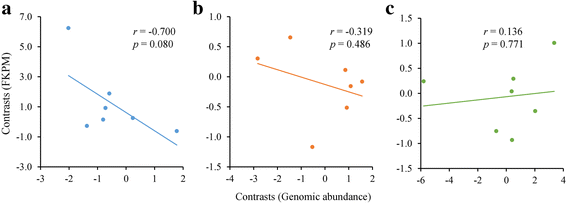
的转录活动copia和吉普赛通过将RNASEQ数据映射到全长LTR回收输送器的参考面板来确定序列。表达被检测到总共11个单独的元素,由6组成copia和5吉普赛序列(图。2).成比例的增加copia(50%)和吉普赛(12.5%)发现要表达的元素给出了用于检测的1个FPKM阈值,并在物种中汇集,表达水平copia元素显著高于对照组吉普赛(t= 2.77;P= 0.011)。有趣的是,同样的元素经常在多个物种中被检测到表达,11个元素中有5个在所有3个物种中表达,11个元素中有3个在2个物种中表达,剩下的3个在一个物种中表达。对于被确定为转录活性的元素,FPKM和基因组丰度的回归分析产生了不显著的结果(图。3得了),尽管对h . agrestis)和h . carnosus。基因组丰度估计最高的元素(例如,在吉普赛sublineages c和x1)缺乏转录活动和单一copia元素,在所有三个中检测到其最高表达向日葵物种(即copiaSublegeage 6)在最低基因组丰度估计中展出(图。2).
40个个体的基因组丰度和表达水平(FPKM)吉普赛和12个人copia三种向日葵的元素。FPKM≥1作为检测表达的截止值。误差棒代表基于三个生物重复的±1 SE。数字和字母的名称指的是附加文件中描述的不同分支1:图S2
表达水平(FPKM)和基因组丰度之间的系统发育独立对比吉普赛和copia表现出FPKM≥1的元素(参见图12).每个数据点代表个人对比度,Pearson的r和P的PIC分析计算的值h . agrestis)(一个),H. Carnosus.(b) 和H. Porteri.(c)
讨论
我们对植物基因组中LTR逆转录转座子多样性和丰度的理解近年来随着基因组技术的进步和模型和非模型物种序列数据生成速度的迅速提高而大幅提高[13.,14.,15.,16.,17.,35].迄今为止审查的大多数植物物种吉普赛和copia超家族作为不同的亚系存在,其中少数亚系通常代表不成比例的大比例的LTR逆转录转座子,因此基因组成分([4,但参见[35])。由无偏的RNAseq研究支持的越来越多的证据也表明,LTR逆转录转座子的表达比以前认为的更普遍,尽管长期以来认为这些序列转录不活跃。然而,植物基因组中不同元素在可检测水平上的表达程度,以及个体元素的基因组丰度与转录活性的可能性或水平之间的确切关系仍是未知的。在本研究中,我们结合DNAseq和RNAseq数据集,研究了三种二倍体野生向日葵品种中不同LTR反转录转座子的基因组丰度和转录活性水平。
对DNAseq数据集的分析表明有相当高的基因组丰度吉普赛与copia这一结果与之前对其他向日葵的分析一致[16.,17.,18.,19.,36]和其他植物分类群[37,38,39]但虽然升高了偏见吉普赛植物基因组中的丰度不是普遍存在的[40,41,42,43].本文所研究的三种向日葵在属的分布上大体相似吉普赛和copia在基因组中是最丰富的还是最不丰富的,尽管在大多数亚系中,物种之间的reads密度存在显著差异。读出密度最高的是在h . agrestis)用2表示的序列吉普赛SublineAges包含约23%的物种所阅读的约23%。这些观察结果与具有最大估计核基因组大小的物种一致(表1),基因组重复率最高(表2),并指出具体吉普赛这些分支很可能促进了基因组的扩张。
两者的转录活性吉普赛和copiaLTR逆转录转座子以前在栽培和野生向日葵中都有记录,使用靶向[12.,23.,44,45和无偏见的[7,21.,22.]方法。多次观察到低但可检测的转录活性吉普赛和copia元素在当前研究中所占比例较高copia这些元素在转录上是活跃的copia在更高层次表达的元素。这些观察结果与基因组中更丰富的元素(例如,吉普赛)更有可能成为宿主基因组沉默的目标,而那些丰度较低的更有可能逃脱这种目标。在其他植物物种中也观察到LTR逆转录转座子的表达变异[46,47,48].例如,在玉米中,发现被鉴定为低拷贝的逆压力被表达,而被确定为丰富的逆阻表不是[49].然而,这种结果并不是普遍观察到的,因为在不同元素的逆转录转座子拷贝数和转录水平之间也观察到正相关(尽管不显著)[48].
本研究的一个意外发现是发现涉及转录活性的特定元素的高度重叠模式,其中包含在所有三种物种中转录活性的11种,共11个中有2种。考虑到这些物种的不同位置,这是一个有趣的结果向日葵1.7-8.2 mya的文学系统和估计年龄[50].这些结果表明,相同或高度相似的元素保留了转录活性和/或避免跨属宿主特异性靶向的能力。目前尚不清楚这些LTR逆转录转座子的转录活性是否会导致相关的插入活性,因为向日葵中TE抑制的转录后机制尚不清楚。转录后抑制可能是由表现某些最高表达水平的个体元素也表现出某些最低基因组丰度的模式所暗示的(例如,copia4和copia6).然而,这种模式应谨慎解释,因为所有元素的表达水平一般都很低。
本研究中观察到的LTR反转录转座子的转录活性模式不太可能来自于RNA样本的DNA污染。当检测高度重复序列的转录活性时,污染问题是一个明确的关注,如果污染是一个问题,我们的结果与预期相反。首先,污染将导致元素丰度水平和转录活性水平之间的正相关。表现出最高丰度水平的单个元素通常被发现是转录非活性的。其次,对于被发现具有转录活性的元素,FPKM与丰度之间没有观察到正相关关系,事实上,在研究的三个物种中,有两个存在明显的负相关趋势。
表观遗传沉默机制已知在宿主基因组中起作用,以控制逆转录转座子增殖,并防止元件转座子活性潜在的负致突变作用[51].这些机制可以通过转录前(如rna导向的DNA甲基化;[51])和转录后机制(如RNA干扰;[52,53])。对这些沉默机制的研究主要集中在模式植物系统上,如拟南芥蒂利亚纳,米饭和玉米([51];和引用)。向日葵基因组中甲基化和小RNA靶向的比较分析可能有助于更好地了解宿主与te相互作用的动态。
结论
虽然目前研究的三种向日葵的基因组大小存在显著差异,但LTR逆转录转座子转录活性的模式在不同的物种间相对保守,具有高度重叠的组copia和吉普赛被发现具有转录活性的元素。对于被分类为转录活性的元素,在三个物种中有两个的表达水平(FPKM)和基因组丰度之间观察到负的但不显著的趋势。未检测到具有最高丰度的单个元素的转录活性。需要更多的实验来阐明转录控制和抑制的机制。
缩写
- FPKM:
-
每千碱基次数的碎片
- INT(警官):
-
整合酶
- 猪油:
-
大型回复扫描衍生物
- LTR:
-
长末端重复
- ngs:
-
新一代测序
- 图片:
-
系统独立的对比
- RT:
-
逆转录酶
- 修剪:
-
末端重复反转座子的缩影
参考文献
- 1。
Eickbush TH, Malik HS。反转录转座子的起源与进化。在:Craig NL, Craigie R, Gellert M, Lambowitz AM,编辑。移动的DNA二世。华盛顿:ASM出版社;2002.1111 - 44页。
- 2。
王文德。植物基因组大小变化机制的新见解。J机器人。2010;2010:382732。
- 3.
库马尔,贝内特岑JL。植物反转位子活动。Annu Rev Genet, 1999; 33:479-532。
- 4.
Sabot F, Schulman, AH。植物的寄生和逆转录转座子生命周期:基因组的搭便车指南。遗传。2006;97(6):381 - 8。
- 5.
舒尔曼啊。反转录转座子在植物中的复制。病毒学报。2013;3(6):604-14。
- 6.
Grandbastien马。LTR逆转录转座子,植物调控和胁迫反应的便利搭便车者。Acta botanica yunnanica(云南植物研究),2015;
- 7.
Kawakami T,Darby BJ,Ungerer MC。多年生向日葵的转录组资源Helianthus maximiliani从生态上不同的种群中获得。生态学报。2014;14(4):812-9。
- 8。
Neumann P, Pozarkova D, Macas J.高度丰富的豌豆LTR逆转录转座子ogre构成转录和部分剪接。植物学报。2003;53(3):399-410。
- 9。
陈建平,陈建平,陈建平。RNA干扰对水稻着丝粒反转录转座子转录的影响。遗传学。2007;176(2):749 - 61。
- 10。
一个生态重要树种的转录组测序:组装、注释和标记发现。BMC基因组学。2010;11:180。
- 11.
vicent CM, Jaaskelainen MJ, Kalendar R, Schulman AH。活性反转录转座子是草基因组的共同特征。植物杂志。2001;125(3):1283 - 92。
- 12.
关键词:向日葵,Copia,吉普赛人,反转录转座子向日葵l .)。BMC Plant Biol. 2009;9:150。
- 13.
Baucom RS, Estill JC, Chaparro C, Upshaw N, Jogi A, Deragon JM, Westerman RP, SanMiguel PJ, Bennetzen JL。B73玉米基因组中反转录因子的异常多样性、非随机分布和快速进化。公共科学图书馆麝猫。2009;5 (11):e1000732。
- 14.
El Baidouri M, Panaud O.植物界的比较基因组古生物学揭示了te驱动的基因组进化动力学。基因组生物学进展。2013;5(5):954-65。
- 15.
Hawkins JS, Kim H, Nason JD, Wing RA, Wendel JF。转座元件的差异谱系特异性扩增是导致棉基因组大小变化的原因。基因组研究》2006;16(10):1252 - 61。
- 16.
Staton SE, Burke JM。菊科的进化转变与转座元素丰度的显著变化一致。BMC基因组学。2015;16:623。
- 17.
8个二倍体向日葵种的长末端重复逆转录转座子的含量。G3(贝塞斯达)。2016; 6(8): 2299 - 308。
- 18.
Buti M,Giordani T,Cattonaro F,Cossu RM,Pistelli L,Vukich M,Morgante M,Cavallini A,Natali L.在向日葵基因组的演变中的演化中的时间动态,其三种大型基因组区域的测序和注释揭示。Al Appl Genet。2011; 123(5):779-91。
- 19。
Staton SE, Bakken BH, Blackman BK, Chapman MA, Kane NC, Tang S, Ungerer MC, Knapp SJ, Rieseberg LH, Burke JM。向日葵(向日葵基因组反映了转座因子的偏置积累的近期历史。植物j . 2012; 72(1): 142 - 53年。
- 20。
向日葵基因组的重复组成。植物学报。2014;1:45-54。
- 21。
Gill n,Buti M,Kane N,Bellec A,Helmstetter N,Berges H,Rieseberg LH。基于序列的结构组织分析与栽培向日葵的组成(向日葵l .)基因组。生物学。2014;3(2):295 - 319。
- 22。
雷诺S, Rowe HC, Ungerer MC, Rieseberg LH。同倍体杂交物种形成的基因组学:杂交向日葵长末端重复反转录转座子的多样性和转录活性。(1)中国科学(d辑:地球科学)2014;
- 23。
UNGERER MC,Kawakami T. LTR Roctionsposons早期发电和古代向日葵杂交种的转录动态。基因组Biol Evol。2013; 5(2):329-37。
- 24。
SIMS LE,价格HJ。核DNA含量变化向日葵(菊科)。我是J机器人。1985; 72(8):1213-9。
- 25.
Heiser CB, Smith DM, Clevenger SB, Martin WC。北美向日葵(向日葵).《Mem Torrey Bot Club》1969;22:1-218。
- 26.
Stephens JD, Rogers WL, Mason CM, Donovan LA, Malmberg RL。二倍体树种树的估计向日葵(菊科)利用目标富集。植物学报。2015;102(6):910-20。
- 27.
Gevaert SD,Mandel Jr,Burke Jm,Donovan La。搬运工向日葵的高遗传多样性和低人口结构(Helianthus porteri.).J在这里。2013;104(3):407 - 15所示。
- 28.
Bino RJ,Lanteri S,Verhoeven Ha,Kraak HL。流式细胞术测定种子组织中核复制阶段的测定。安乐伦敦。1993; 72(2):181-7。
- 29.
基于图的新一代测序数据中重复序列的聚类和表征。BMC生物信息学。2010;11:378。
- 30.
RepeatExplorer:一个基于星系的网络服务器,用于从下一代序列读取真核重复元素的全基因组特性。生物信息学。2013;29(6):792 - 3。
- 31。
系统发育学与比较方法。Nat。1985;125(1):1 - 15。
- 32。
Felsenstein J. Phylip - 系统发生推理包(版本3.2)。克莱斯。1989; 5:164-6。
- 33。
Tamura K,SteCher G,Peterson D,Filipski A,Kumar S. Mega6:分子进化遗传学分析6.0版。mol Biol Evol。2013; 30(12):2725-9。
- 34。
蛋白质进化的最佳拟合模型的选择。生物信息学。2005;21(9):2104 - 5。
- 35。
Kelly LJ, Renny-Byfield S, Pellicer J, Macas J, Novak P, Neumann P, Lysak MA, Day PD, Berger M, Fay MF,等。巨型基因组的分析贝母(百合科)表明缺乏DNA去除特征是基因组大小的极端扩大。新植醇。2015;208:596 - 607。
- 36。
Natali L, Cossu RM, Barghini E, Giordani T, Buti M, Mascagni F, Morgante M, Gill N, Kane NC, Rieseberg L,等。向日葵基因组的重复组成部分通过不同的程序来组装下一代测序reads。BMC基因组学。2013;14:686。
- 37.
国际水稻基因组测序P.基于图谱的水稻基因组序列。自然。2005;436(7052):793 - 800。
- 38.
Ming R,Hou S,Feng Y,Yu Q,Dionne-Laporte A,Saw JH,Senin P,Wang W,Ly Bv,Lewis Kl等。转基因热带果树番木瓜的基因组草案(番木瓜Linnaeus)。自然。2008; 452(7190):991-6。
- 39.
Paterson AH, Bowers JE, Bruggmann R, Dubchak I, Grimwood J, Gundlach H, Haberer G, Hellsten U, Mitros T, Poliakov A, et al.;高粱双色基因组与禾本科植物的多样性。大自然。2009;457(7229):551 - 6。
- 40.
Argout X, Salse J, Aury JM, Guiltinan MJ, Droc G, Gouzy J, Allegre M, Chaparro C, Legavre T, Maximova SN,等。可可树的基因组。43 Nat麝猫。2011;(2):101 - 8。
- 41.
亚麻中转座因子的鉴定、表征和分布Linum Usitatissimum.l .)基因组。BMC基因组学。2012; 13:644。
- 42.
黄SW,Li Rq,张Zh,Li L,Gu XF,粉丝W,Lucas WJ,王XW,Xie,Ni PX等。黄瓜的基因组,Cucumis巨大成功李志刚。2009;41(12):1275-81。
- 43。
Jaillon O, Aury JM, Noel B, Policriti A, Clepet C, Casagrande A, Choisne N, Aubourg S, Vitulo N, Jubin C, et al.;葡萄基因组序列表明主要被子植物门的祖先六倍体化。大自然。2007;449(7161):463 - 7。
- 44。
Buti M, Giordani T, Vukich M, Gentzbittel L, Pistelli L, Cattonaro F, Morgante M, Cavallini A, Natali L. HACRE1,最近插入的向日葵copia-like逆转录转座子(向日葵l .)。基因组。2009;52:904-11。
- 45。
Kawakami T, Dhakal P, Katterhenry AN, Heatherington CA, Ungerer MC.尽管LTR逆转录转座子具有广泛的转录活性,但转座子增殖和基因组扩展在当代向日葵杂种群体中非常罕见。基因组生物学进展。2011;3:156-67。
- 46。
Domingues DS, Cruz GMQ, Metcalfe CJ, Nogueira FTS, Vicentini R, Alves CD, Van Sluys MA。植物ltr反转录转座子在精细的家族水平上的分析揭示了个体的分子模式。BMC基因组学。2012;13:137。
- 47。
Ming R, VanBuren R, Wai CM, Tang HB, Schatz MC, Bowers JE, Lyons E, Wang ML, Chen J, Biggers E, et al。菠萝基因组与CAM光合作用的进化。Nat麝猫。2015;47(12):1435 - 42。
- 48。
富有厘米。玉米转移元素的转录活性。BMC基因组学。2010; 11:601。
- 49.
玉米基因组中重复元件的丰度、分布和转录活性。基因组研究》2001;11(10):1660 - 76。
- 50.
先令EE。系统发育分析向日葵(菊科)基于叶绿体DNA限制位点数据。中国科学(d辑:地球科学)1997;94(6-7):925-33。
- 51.
Bousios A,Gaut BS。可转产元素及其植物主持人的表观遗传冲突的机械和进化问题。CurrOp植物BIOL。2016; 30:123-33。
- 52.
Slotkin Rk,Martienssen R.转换元素和基因组的表观遗传调节。NAT Rev Genet。2007; 8(4):272-85。
- 53.
植物转座因子的表观遗传调控。植物学报2009;60:43-66。
确认
我们感谢Hannah Tetreault和Anastasia Weston进行技术援助。
资金
这项研究得到了堪萨斯州立大学的支持。没有贡献。17-217-J来自堪萨斯农业实验站。本文的出版部分由堪萨斯州立大学开放获取出版基金资助。
数据和材料的可用性
原始序列读取可从NCBI Short Read Archive (SRA)项目#PRJNA418245获得。
作者信息
隶属关系
贡献
FQ和MCU是本研究的构思和设计。FQ和MCU进行了实验,FQ进行了所有的分析。FQ和MCU撰写手稿。两位作者均已阅读并批准本手稿。
通讯作者
道德声明
伦理批准和同意参与
本研究所用植物的种子来自美国农业部(USDA)国家植物种质系统。本研究对植物进行的实验研究符合机构、国家和国际准则。
同意出版
不适用
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
额外的文件
附加文件1:图S1。
阅读密度和核基因组大小之间的皮尔逊相关吉普赛和copia元素。图S2。(A)不同亚系间的系统发育关系吉普赛和(B)copia根据逆转录酶结构域的氨基酸序列(吉普赛173同上;copia, 250 a.)使用最大可能性。>50%支持的内部分支的引导评分。表S1。DNA-SEQ和RNA-SEQ数据的概述统计。(PDF 488 KB)
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
邱,F.,Ungerer,M.C.不同的基因组丰富和转录活动吉普赛和copia三种野生向日葵的长末端重复反转录转座子。BMC植物杂志18,6(2018)。https://doi.org/10.1186/s12870-017-1223-z
已收到:
接受:
发表:
关键字
- 反转位子活动
- 吉普赛
- Copia
- 基因组丰度
- 转录活动
- 向日葵