摘要
背景
现在迫切需要增加全球农作物产量。识别和结合控制不同生物过程的特定基因具有提高作物产量的潜力。转录组学是深入了解构成这些性状的复杂基因调控网络的有力工具,但依赖于高质量参考序列和精确基因模型的可用性。此前,我们在小麦5A染色体上鉴定了一个粒重QTL (5A QTL),该QTL在籽粒发育早期通过果皮细胞扩张来增加粒长。在本研究中,我们对近等基因系(NILs)进行了rna测序,分离出了5A QTL,并使用最新的基因模型识别了可能影响小麦果皮细胞大小和粒重的差异调控基因和通路。
结果
我们在花后4天和8天取样,发现在这一时期,两种NILs中与代谢、生物合成、蛋白质水解和防御反应相关的基因都被上调。我们鉴定了112个在任何时间点5A NILs之间差异表达的转录本(DE),其中包括8个可能的因果5A基因及其下游靶点的候选转录本。112份DE转录本具有非编码RNA、转座子相关、细胞周期控制、泛素相关、热休克、转录和组蛋白相关等功能注释。许多被确认的基因属于以前与其他物种的种子/谷物发育相关的家族。值得注意的是,我们在与泛素介导的蛋白质降解相关的途径的几乎所有步骤中都发现了DE转录本。在DE转录本子集的启动子中,我们发现了与C2H2、MYB/SANT、YABBY、AT-HOOK和Trihelix转录因子家族相关的结合位点富集。
结论
在这项研究中,我们鉴定了具有多种预测生物学功能的DE转录本,反映了控制早期谷物发育途径的复杂性。这些基因中很少有与其他物种的粒级基因直接同源的,而且没有一个在小麦中被鉴定过。这些候选基因的进一步功能特征以及它们如何相互作用,将为谷物粒度的控制提供新的见解。
背景
作物产量必须增加,以满足全球人口的需求,据估计到2050年全球人口将超过90亿[1].事实上,目前有九分之一的人生活在粮食不安全状态下[2].在农业扩张机会有限的情况下,提高现有土地的产量可以显著减少面临饥饿风险的人数[3.].据估计,到2050年,作物产量至少需要增加50% [4.那5.]然而,目前的产量增长率不足以实现这一目标[6.].因此,我们判断增加作物产量的方法是至关重要的。
作物最终产量受许多遗传和环境因素的相互作用影响。这种复杂性阻碍了它的研究,也意味着控制这一特性的机制还没有被很好地理解。然而,粒重是最终产量的一个重要组成部分,它比产量本身更容易遗传,也更容易被理解[7.]。粒重主要由粒径决定,粒径本身由细胞增殖和扩展过程的协调控制。对作物和模式物种的研究表明,这些过程受广泛的基因和分子机制调控(综述见[8.那9.])。水稻转录因子(TF)证明了转录水平的对照OsSPL16通过细胞增殖影响晶粒尺寸[10,而WRKY域TF,TTG2,影响拟南芥种子表皮细胞的扩张[11].还鉴定了与蛋白质转换有关的重要途径,例如E3泛素 - 连接酶,GW2,通过对细胞划分的控制,对水稻的粒重和宽度负调节[12].GW2其他物种的同源物,包括拟南芥和小麦,也作为种子/颗粒大小的负调节者,这表明这些机制可能在物种之间是保守的[13那14].影响颗粒大小的其他途径/机制包括微管动力学[15那16, g蛋白信号[17那18]和植物激素生物合成及信号传递[19那20.那21].
小麦是一种全球重要的作物,约占人类消耗热量的20% [22].然而,与水稻和拟南芥相比,我们对小麦颗粒大小控制机制的了解仍然有限。比较基因组学方法提供了一些见解[13那23],并鉴定了许多与粒级和形状成分(粒面积、长度和宽度)相关的数量性状位点(QTL) [24那25那26那27那28那29].然而,这些QTL尚未被克隆,其机制也知之甚少。此前,我们在小麦染色体5A上鉴定了一个与粒重增加相关的QTL。使用公元前4.在近代源线(NIL)近,我们确定QTL在谷物开发的早期阶段起作用,以通过增加果罗的细胞扩张增加粒度[29].这项研究和其他研究表明,小麦粒/胚珠发育的早期阶段对决定最终粒的大小/形状很重要[13那30.那31].
转录组织是一种强大的工具,可以进入基因的复杂基因监管网络的洞察,该网络利用特定的特征和生物过程。几项研究使用了转录组织方法来查看小麦籽粒发育期间表达的基因[32那33那34那35那36那37那38].然而,这些研究主要集中在谷物发展的后期阶段,通常关注胚乳中的淀粉积累。此外,使用微阵列进行这些研究中的许多研究[33那36那37,它们代表转录组的一部分,不能区分同源基因副本。最近的研究使用了rna测序(RNA-seq) [34那35],这是一个开放式平台,提供优异的特定分辨率。然而,RNA-SEQ的准确性取决于高质量参考序列和准确基因模型的可用性。直到最近,六倍体小麦基因组的大(〜17 GB)和高度重复性意味着基因组资源受到限制和不完整。然而,这在过去几年中,这种全部基因组序列和注释的释放情况发生了急剧改变[39那40那41.那42.].迄今为止,RNA-seq谷物发育研究使用了表达序列标签(EST)[35那38]或染色体测量序列(CSS)[34作为引用。然而事后看来,这些注释对于最新的基因模型来说是不完整的[39那41.].这些新资源为小麦转录组学的更详细和准确的研究提供了新的机会。
转录组学研究的一个潜在缺陷是,跨品种、组织或时间点的比较可能导致大量转录本的差异表达。虽然这有助于我们理解生物学机制,但很难为下游分析确定特定基因的优先级。使用更精确定义的遗传物质、组织和发育时间点的比较转录组方法可以通过定义更小的差异调节转录本集来帮助实现这一点。例如,比较野生型和RNAi敲减系的旗叶转录组谷物蛋白质含量1(GPC)基因被用来鉴定下游靶点GPCTFs [43.].同样,比较了染色体臂4AL上一个主要的籽粒休眠QTL的NILs转录组,并鉴定了该QTL的特定候选基因[44.].据我们所知,还没有在已知粒级差异的小麦等基因系上进行过这样的实验。
在本研究中,我们对NILs进行RNA-seq分离,在染色体臂5AL上获得一个主要粒重QTL。此前,我们发现QTL在籽粒发育早期起作用,携带5A阳性等位基因的NILs (5A+ NILs)显著增加千粒重(TGW;(7%),粒长(4%)和果皮细胞长(10%),而携带负5A等位基因的NILs (5A- NILs) [29].利用重组自交系,根据IWGSC RefSeq v1.0,将粒长效应精确定位到5A染色体长臂上的75mb区域。本研究的目的是通过RNA-seq识别在5A-和5A+ NILs之间有差异调节的基因,识别潜在影响粒长和果皮细胞大小的生物学途径。
结果
5a附近的RNA排序
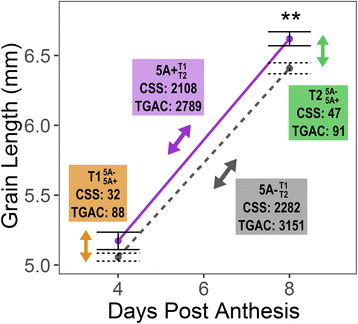
我们对来自两个5A NILs的全粒进行了RNA-seq,对比了基于2014年生长季节的粒长[29].我们选择NILs在粒长上首次出现显著差异的时间点(花后8天(dpa));T2)和之前的时间点(4 dpa;T1)来捕捉这一时期基因表达的差异(图。1).我们假设,虽然在T1时粒长表型没有显著差异,但表型差异已经开始出现,影响这一差异的基因表达变化可能已经发生。我们在所有12个样本(2个时间点,2个NILs, 3个生物重复)中获得了超过362 M reads,单个样本的reads范围从15.0 M到53.6 M,每个样本平均30.2 M reads(标准误差±3.5 M reads)(见表)1).
跨时间的差异表达基因。RNA-SEQ在2014年生长的4种不同条件下的全晶RNA样品上进行[29]:花后4 d 5A-(短粒)和5A+(长粒)NILs (dpa;T1)和8 dpa (T2)。这些被选择为第一个显著差异(P. < 0.01, asterisks) in grain length was observed between 5A- (grey, dashed line, short grains) and 5A+ (purple, solid line, long grains) and the preceding time point. Differentially expressed (DE) transcripts were identified for four comparisons (q-value < 0.05). Coloured boxes indicate the numbers of DE transcripts identified for each comparison using alignments to either the IWGSC Chinese Spring Survey Sequence (CSS) or the TGACv1 (TGAC) Chinese Spring reference transcriptomes. Two ‘across time’ comparisons:\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)(灰色盒子;比较5a-nil的t1和t2样品)和\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)(紫色框;比较T1和T2样品的5A+ NIL),以及两个‘between NIL’比较:\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)(橙盒;比较T1时5A-和5A+ NILs)和\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \)(绿色的盒子;比较T2时5A-和5A+ NILs)
中国春季参考转录组比较
我们将读取从中国春天参考加入中的两种不同的转录组序列,IWGSC染色体测量序列(CSS)[40]及TGACv1 (TGAC) [41.)参考。在样本中,平均69.8±0.3%的读对齐到CSS参考,而84.4±0.2%的读对齐到TGAC参考。
我们将一个转录本定义为:在四种条件(2个NILs × 2个时间点)中至少有一种条件下,其平均丰度为> 0.5 transcripts per million (tpm)。这导致在CSS和TGAC转录组中分别表达了64,020和101,652个转录本。我们使用sleuth定义差异表达(DE)转录本(q值< 0.05)[45.然后进行了四次两两比较:两次‘跨时间’比较和两次‘无时间’比较。“跨时间”分析包括T1和T2样本之间的5A- NIL(以下符号为\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \);无花果。1,灰色)和5a + nil样本的相应比较(以下\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \);无花果。1, 紫色的)。在“跨时间”分析中,转录物被认为是对T1 I.E的上调或下调的。在时间的表达中增加或减少。“尼尔之间”分析中的“在T1的5A-和5A +”之间的比较包括(以下\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\);无花果。1,橙色),以及T2处5A-和5A+ NILs的比较(下文)\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \);无花果。1、绿色)。在所有情况下,与CSS转录组相比,TGAC中鉴定出了更多的DE转录本,在四项比较中,两项参考文献都观察到了类似的趋势(图)。1).
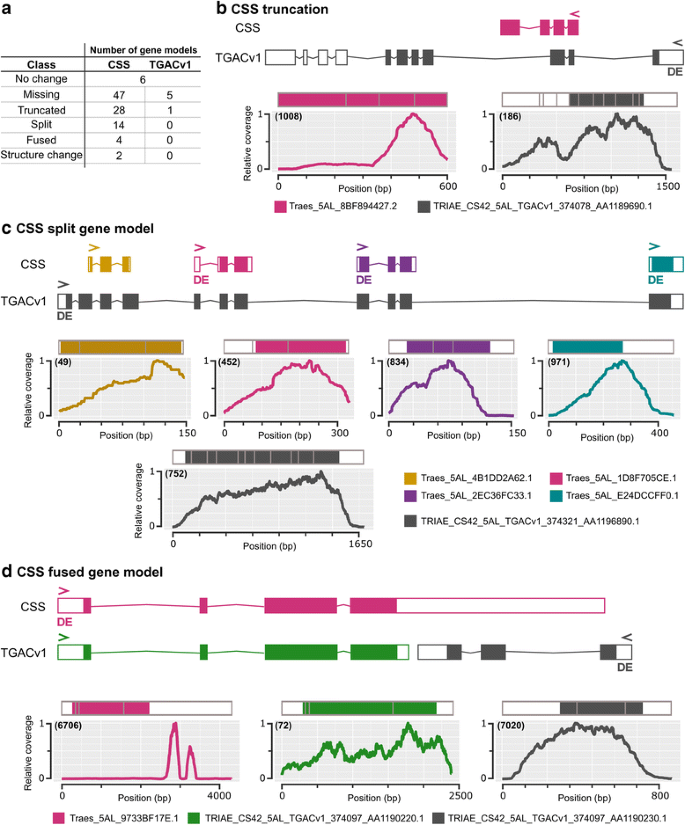
我们选择与最少的成绩单(\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\);32和88 DE抄本的CSS和TGAC,分别)进行更深入的比对和参考文献分析。对于每个比对的所有DE转录本,我们使用其他参考序列中的等效转录本/基因模型进行鉴定运用植物释放35个,并比较基因模型(附加文件1).对于64个TGAC DE转录本,我们没有发现一个等效的CSS DE转录本,要么是因为没有相应的CSS基因模型(47个转录本),要么是NILs之间的表达变化对CSS转录本不显著。同样,11个CSS DE转录本没有等效的TGAC基因模型DE,其中5个是由于没有相应的TGAC基因模型注释。结合这两组基因,我们鉴定了42组等效基因模型,其中26组在两种比对中都有差异表达。比较这42组数据,并考虑每个数据集中的融合和分裂基因模型,两个数据集中共有97个基因模型(50个CSS + 47个TGAC)(图。2A,附加文件1).其中,只有6个在CSS和TGAC引用之间是相同的。所有其他不一致的基因模型都属于这一类,包括任一参考文献中的截断、在一个参考序列中分裂/融合的基因模型,以及在总体结构上存在巨大差异的基因模型。
CSS与TGACv1基因模型比较。一种CSS和TGAC参考序列中基因模型之间鉴定的差异以及落入类别的基因模型的数量。面板(B.),(C) 和 (D.)显示具体的差异示例。在每个面板中,未亮相基因模型的表示显示为彩色盒子,未翻译的区域作为白色盒子,以及内含物作为细线。图表显示了扫描曲折上的相对读取覆盖,其中结构在每个图中直接表示。括号中的数字显示每个基因模型的最大绝对读取深度。> <在基因结构中,表明转录方向和'de'表明基因模型在差异上表达\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)(q值< 0.05)。对于每个小组,成绩单名称显示在彩色图例中
对于所有不同的基因模型,我们使用转录组阅读图谱和物种间比较来确定哪个基因模型似乎最可信。无花果。2B.显示了最常见的差异示例,其中CSS参考(粉红色)中的基因模型相对于TGAC参考(灰色)被截断。我们的转录组数据支持DE TGAC基因模型,因为我们观察到整个基因模型的读取覆盖率,而CSS基因模型的覆盖率在TGAC模型中预测内含子的位置下降。另一个常见的差异是一个参考文献中的单基因模型在另一个参考图。2C展示单个DE TGAC基因模型包含四种单独的CSS基因模型的实例。在这种情况下,所有五种基因模型在整个基因体上覆盖,但是单个TGAC基因模型与来自其他物种的蛋白质更类似于其他物种的蛋白质,表明这种单一基因模型最有可能是正确的。最后的例子(图。2D)显示两种TGAC基因模型,其融合成单个CSS基因模型。CSS基因模型的覆盖范围不一致,大多数读数集中在3'未转换区域(UTR)中。两种TGAC基因模型在整个基因模型上具有更一致的覆盖率,并且均由蛋白质对准与其他物种的支撑。有趣的是,只有较短的TGAC基因模型是de(图。2D这表明CSS基因模型的差异表达是由标记到假定的3 ' UTR的reads驱动的,而不是转录本的编码区域(图2)。2D粉色)。一起在一个更高比例的读取映射到TGAC基因模型和许多更多的检查TGAC基因模型支持的种间比较,比CSS基因表达数据模型,我们决定继续使用比对分析TGAC基因模型。
早期谷物发育期间的许多成绩单都在尼尔之间共享
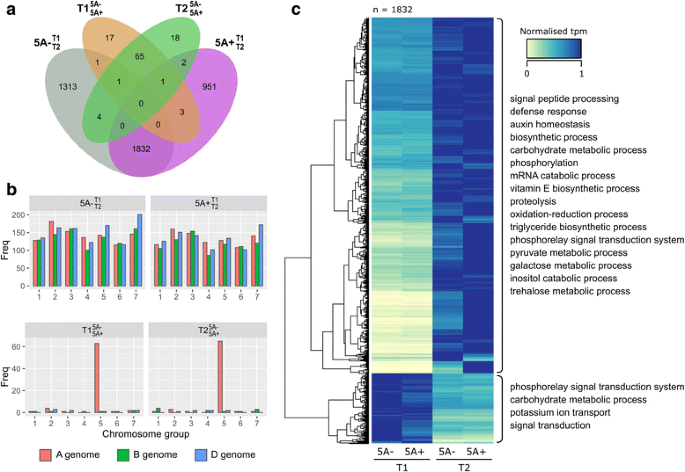
我们在早期谷物发育中鉴定了3151和2789个DE转录本\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)和\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)分别(无花果。1, 无花果。3A).DE转录本均匀分布在21条染色体上,没有显示出对任何染色体组或亚基因组的总体偏倚(图)。3B.)。约60%(1832)的DE转录本由\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)和\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)(无花果。3A)84%(1532)的共享转录本随时间上调(图。3C)我们在上调转录本中鉴定了41个显著富集的基因本体(GO)术语(附加文件)2).GO术语的十六个与生物过程有关,可以在三个父母GO条款下进行分组:代谢过程(GO:0008152),防御响应(GO:0006952)和生物调节(GO:0065007)(图。3B.).在代谢过程中,我们发现了与碳水化合物(GO:0005975)和丙酮酸代谢(GO:0006090)、维生素E (GO:0010189)和甘油三酯生物合成(GO:0019432)、mRNA分解代谢(GO:0006402)、蛋白质水解(GO:0006508)和磷酸化(GO:0016310)相关的术语。下调的转录本(300)富集了7个GO项,其中4个与生物过程有关:钾离子转运(GO:0006813)、信号转导(GO:0007165)、磷酸化信号转导(GO:0000160)和碳水化合物代谢(GO:0005975)。在上调和下调的转录本(如碳水化合物代谢)中富集的氧化石墨烯术语之间的重叠表明,这些过程的不同方面在谷物发育的早期阶段受到不同的调节。
差异表达的成绩单概述。一种差异表达(DE)转录本的维恩图(q < 0.05)在4个两两比较中鉴定:\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)(橘子),\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \)(绿色),\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)(灰色)和\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)(紫色)。B.位于每种染色体上的DE转录物的数量,用于所有比较。这\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)和\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)DE转录本(上图)均匀分布在所有21条染色体上\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)和\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \)DE转录物(底部图)浓缩在染色体5A上。C中常见DE转录本的标准化tpm(每百万份转录本)热图\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)和\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)(N= 1832)。分级聚类将这些转录本分成上调的转录本(N= 1532)和下调(N= 300)跨越时间。热图右侧显示各组显著富集的氧化石墨烯(仅生物功能)
我们还鉴定了许多转录本,这些转录本仅在两个基因型中的一个基因型的早期籽粒发育中存在差异(即,这两个基因型中的任何一个都是唯一的)\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)或\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)比较)。然而,在相反的基因型比较中,许多转录本的边界不显著,q值的分布向显著性倾斜(附加文件)3.).此外,对GO术语的唯一DE转录本进行了丰富,类似于共享转录本(附加文件)2).然而,一些氧化石墨烯术语只富集在独特的DE转录本中,例如细胞壁组织或生物合成(GO:0071554)和对非生物刺激的反应(GO:0009628)。综上所述,虽然基因型之间存在一定的差异,但在发育早期,两种5A NILs的籽粒发生了大致相似的生物学过程。
NILs之间的DE转录本集中在染色体5A上
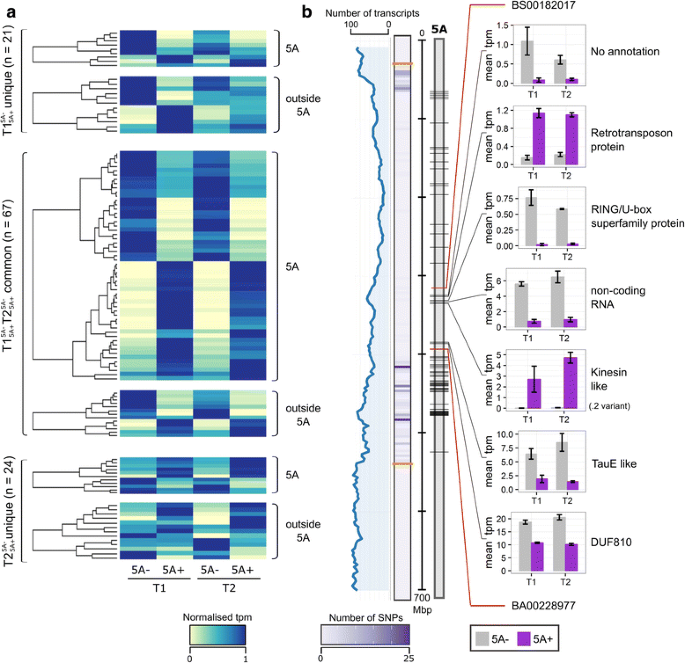
我们在NILs之间找到了88到91份DE转录本\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)和\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \),比确定的要少很多\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)或\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)这是预期的,因为NIL在基因上非常相似,因此T1和T2时间点之间发育阶段的差异导致基因表达的更大变化。对于DE转录子的子集,我们使用qRT PCR确认表达变化(参见方法详情)。在179个DE转录本中,67个是常见的\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)和\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \)而45个基因型之间的DE转录本是唯一的,仅在一个时间点被识别(导致112个DE转录本在任何时间点;无花果。3A).这些群体中没有大幅度富集。在67个常见的DE转录物中,54(80%)位于染色体5a上,同时在染色体5a上位于小于50%的T1和T2的独特基团中,同时位于染色体5a上(图。4A).相同数量的DE转录本在两种基因型中都有更高的表达,在独特组和普通组之间没有观察到明显的模式。
5A NILs在T1和T2的差异表达转录本。一种NILs之间DE(差异表达)转录本的标准化tpm(每百万份转录本)热图(\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)和\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \)比较)。转录本首先根据它们在两个时间点是否有差异表达进行分组(\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)和\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \)普通)或只在T1或T2 (\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)独特的,\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \)分别是独特的),然后它们是否位于染色体5a上。B.dna转录本在5A染色体上的定位(灰色矩形上的黑线)。线形图(蓝色)显示了位于3个Mbp bins的转录本数量的滚动平均数,同时还有热图显示了3个类似大小bins中5A-和5A+ NILs之间的90k iSelect SNPs的数量。SNP热图上的橙色线定义了491 Mbp渗入,这在NILs之间是不同的。染色体上的红色标记为5A粒长QTL (BS00182017和BA00228977)精细定位区域的侧翼标记位置。柱状图显示了位于精细映射区域的DE转录本(5A- NILs为灰色,5A+ NILs为紫色)T1和T2处tpm值的平均值。只显示了一个驱动蛋白样基因的转录变体(2)。误差棒是三个生物重复的标准误差
我们特别观察了位于染色体5A上的74个DE转录本的位置,发现所有转录本都位于NILs的491 Mbp渗入区域内(图)。4B.).在5A NILs之间SNP密度增加的区域中,发现了更多的DE转录本。此前,我们在5AL上精细定位了粒长效应的75 Mbp区间(BS00182017 (317 Mbp)和BA00228977 (392 Mbp;[29]),八个DE转录本位于这段时间内。其中3个转录本在5A+ NILs中表达较高(5A +)高转录本),其中两个是同一基因的转录变体(一种激肽样蛋白;图中只有2种变体。4B.).另一个5A +高转录本被注释为假定的逆转录转座子蛋白。其中5个转录本中5A- NIL表达较高(5A -高其余4个被注释为非编码RNA、RING/U-box蛋白、taulike蛋白和DUF810家族蛋白。
染色体5A外的DE转录本富集于特定的转录因子结合位点
由于染色体5a上的所有转录物位于491MBp狭窄的区域内,差异表达可能是NILS之间的序列变异的直接后果。在推动者区域。然而,位于染色体5a外部的38转录物具有相同的核苷酸序列,因为它们通过下降相同(BC4.NILs与90k iSelect SNP标记数据确认[29])。这些38 DE转录本不太可能代表假阳性(见方法),因此我们假设这些转录本是DE基因的下游靶点,如转录因子(TFs),位于5A基因渗入内。
为了评估这一点,我们鉴定了38个DE转录本启动子区域中存在的转录因子结合位点(TFB)。我们鉴定了与这组转录本中存在的91个不同TF家族相关的TFB(附加文件4.),其中五个相对于所有表达的转录本富集(表2;罗斯福调整P.< 0.05)。TFBS家族为C2H2、Myb/SANT、AT-Hook、YABBY和MADF/Trihelix。
为了确定上游调节器的潜在候选基因,我们确定了位于染色体5A上的导入区内的所有TF[46.].我们共鉴定了200个注释的TF,属于35个TF家族。其中,4个家族(跨越29个基因)与丰富的TFBS家族重叠。29个转录因子中有4个位于染色体5A精细定位的粒长区域内,包括C2H2、MYB和myb_相关转录因子5.).
DE转录本功能注释
根据染色体位置分析了NIL之间的DE转录物,我们根据其功能注释查看了112个转录物(附加文件6.)我们确定了多个类别,包括与泛素介导的蛋白质降解、细胞周期、代谢、转运、转座子和非编码RNA相关的转录本(表1)3.).少数类只位于5A上或5A外,或在5A-或5A+ NIL中有较高的表达。
DE转录本最多的类别是非编码RNA (ncRNA, 15个转录本),尽管相对于所有表达的转录本而言,这一类别并不丰富。所有的ncRNA转录本被分类为长非编码rna (> 200 bp, [47.])我们发现,4个NCRNA与编码转录物重叠(两者在反义方向上)和一个NCRNA是推定的miRNA前体(Ta-miR132-3p; [48.])。在TGAC文献中,我们确定了13个转录本作为Ta-miR132-3p的假定靶点,但这些靶点转录本在我们的数据集中均无差异表达。第二大转录本类别是转座子相关转录本(14个;FDR-adjustedP.= 0.008),而第三大类是与泛素和蛋白酶体相关的DE转录本(12个;P.= 0.008)。被注释为同源盒的DE转录本也被富集(4个转录本;FDR-adjustedP.= 0.001)。有趣的是,我们在38个5A DE外转录本中发现了27个同源结构域TFBS,尽管这并没有显著富集(fdr调整)P.= 0.166,附加文件4.).
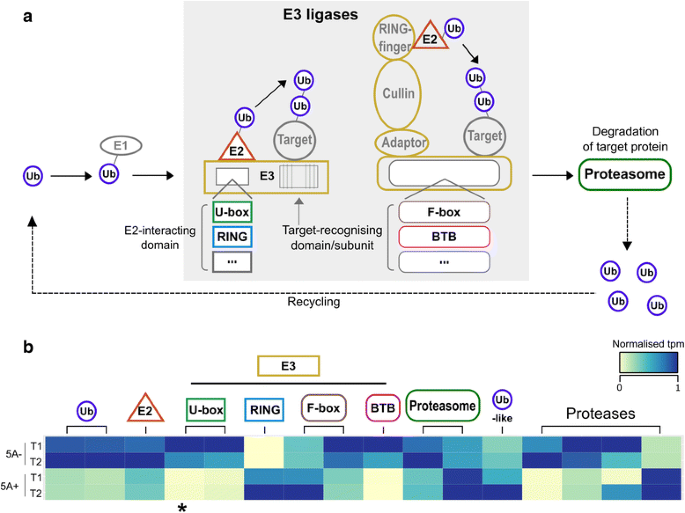
与遍霉素相关的转录物特别感兴趣,因为泛素介导的蛋白质转运先前与小麦种子/晶粒尺寸的控制有关[13]和其他物种,包括水稻和拟南芥[12那14那49.].这一途径通过酶的级联作用而起作用(见图)。5A将泛素(ub)的多个副本添加到底物蛋白中,然后将底物蛋白作为蛋白酶体降解的目标。我们鉴定了该途径几乎所有步骤(不包括E1)的转录本的差异表达:两个泛素蛋白和一个泛素样蛋白,一个E2接合酶,六个潜在的E3连接酶成分和两个推定的蛋白酶体成分(图)。5.).除此之外,我们还鉴定了四个注释为蛋白酶的DE转录本(图)。5.),它们是已知的受此途径调控的底物[50.那51.那52.通过调节细胞增殖来影响器官大小。差异表达的泛素途径的大部分组分在5A- NIL中表达更高(11/16,包括蛋白酶)(图。5B.).
5A NILs中泛素通路的差异调控。一种与泛素介导的蛋白转换相关的功能注释的差异表达(DE)转录本相对于整个基因组富集(一种).该途径通过三种酶的级联作用,将泛素(Ub)的多个拷贝添加到底物蛋白上:E1 (Ub激活酶)、E2 (Ub结合酶)和E3 (Ub连接酶)。标记的底物被26S蛋白酶体降解,Ub蛋白被循环利用。E3连接酶是三种酶中最多样化的,既存在单亚基蛋白,也存在多亚基复合物。这些类的子集显示在(一种),根据DE成绩单的注释进行选择。单个亚基E3连接酶有一个e2相互作用域(例如U-box, RING等(…))和一个底物识别域。多亚基配合物也有e2相互作用配合物和底物识别亚基(如F-box、BTB等(…))。在器官大小控制的背景下,一些蛋白酶已被确定为该途径的下游靶点(如DA1, UBP15 [50.那51.])。B.泛素、蛋白酶体和蛋白酶相关DE转录本的标准化tpm热图。*指示位于5A粒长QTL精细定位区域的转录本(图。4B.;环/U盒超家族蛋白)
讨论
在本研究中,我们对果皮细胞大小、粒长和最终粒重已知差异的5A NILs的粒进行了RNA-seq。我们之前确定NILs之间的第一个表型差异出现在早期谷物发育过程中[29].我们假设在这些早期阶段期间NIL之间的基因表达的差异使我们能够鉴定影响终结细胞尺寸和转录水平的晶粒尺寸的特定基因和途径。
高质量参考序列的重要性
我们最初将RNA-SEQ数据映射到两个不同的参考转录om:CSS和TGAC。我们发现TGAC在对齐和在基因模型本身的读数的术语中术语术语表现优于CSS转录组。这很可能是由于CSS上的TGAC参考的序列旁观性方面的显着改善(分别分别为<10 kB,分别为<10 kB),允许更准确地预测基因模型。我们的研究突出了这种改进的实际重要性,因为我们在大多数情况下使用TGAC参考检测到64种变量转录物,因为在CSS参考(46转录物)中没有相应的基因模型。我们还确定了CSS参考中的基因模型不正确导致误导性结果的情况。例如,在CSS融合基因模型案例研究中(图。2D)来自CSS参考的单个DE转录本有大量的读映射到3 ' UTR。这个基因是拟南芥的同源基因NPY1,在生长素调控的器官发生中发挥作用[53.,因此可能与控制晶粒尺寸有关。然而,在TGAC的引用中,除了NPY1orthologue,一个替代的基因模型被注释在3 ' UTR。这个替代基因模型在NPY1直系同源基因表达水平很低,没有差异表达。
支架尺寸、邻近性和基因注释的改进为小麦研究提供了新的机遇。在这里,我们使用新的物理序列为112个DE转录本中的107个分配位置,使我们能够确定哪些DE转录本位于QTL精细定位区间内。同样,对启动子序列的分析为这一特定的生物过程提供了新的假说,也将有助于理解基因组间启动子差异如何影响不同同源性的相对转录本丰度。这说明了正确注释的基因模型和改进的基因组装配在获得更准确的底层生物学观点方面的重要性。
差异表达分析提供了对早期谷物发育中发生的生物过程的洞察
我们在4 dpa和8 dpa取样,以包含发育阶段,在这一阶段,5A NILs的粒长首次出现显著差异。在这个阶段,颗粒大小的增加主要是由果皮细胞的扩张驱动的[54.那55.,这与我们之前的发现一致,即果皮细胞大小的增加导致了最终粒长的差异。这些时间点也相对较早,与其他关注后期籽粒灌浆过程的籽粒相关RNA-seq研究相比[34那35那36].“跨越时间”的比较(\ (5 \ mathrm{一}{-}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \)和\ (5 \ mathrm{一}{+}_ {\ mathrm T} {2} ^ {\ mathrm T} {1} \))在每个NIL中鉴定出> 2700 DE转录本,并且在差异调控的生物过程中存在大量重叠。我们发现大多数DE转录本随着时间的推移而上调,其中许多与代谢和生物合成有关,这与谷物经历一段快速生长时期以及胚乳细胞化开始这一发育阶段相一致[32].随着时间的推移,与蛋白质水解和mRNA分解代谢相关的转录本也上调,这与谷物发育这一阶段特定蛋白酶和其他水解酶的增加一致[56.].这可能是细胞程序性死亡的迹象,这种死亡发生在珠心和果皮的发育中粒达12dpa [54.]我们还发现了与防御反应和氧化还原过程相关的转录物的上调,这与先前的报告一致,即在谷物发育的早期中期,与防御病原体和氧化应激相关的蛋白质积累[57.].转录研究总是警告说,基因表达的变化可能不会转化为蛋白质水平的变化[58.].然而,对谷物发育相似阶段的蛋白质组学分析已经确定了相似本体的差异调节[57.那59.表明这些转录变化反映了谷物中蛋白质的整体状态。
比较转录组学作为鉴定QTL候选基因的方法
与高分辨率遗传映射相比,在高度同学材料上进行RNA-SEQ对高分辨率遗传造影的相对缓解和低成本使其成为鉴定QTL下面的候选基因的吸引方法,并且更好地理解相关性状。对于具有大型基因组的物种,例如小麦的物种尤其如此,其全基因组测序的成本保持高。然而,转录组学方法的潜在缺点是在比较不同品种或发育阶段时经常观察到的大规模基因表达变化。通过定义在线之间的非常特异性的染色体间隔来解决高度同学线来解决这一点。我们的研究中的5A尼尔斯通过允许直接比较谷物发育期间两次滴定在基因表达上的效果(\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)和\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \)).这导致基因型之间的定义112种转录物。多数的\ (\ mathrm {T} {1} _ {5 \ mathrm{一}+}^ {5 \ mathrm{一}-}\)和\(\ mathrm {t} {2} _ {5 \ mathrm {a} +} ^ {5 \ mathrm {a} - } \)DE转录本均位于5A染色体上,且均位于5A基因渐渗区内。考虑到NILs的序列变异局限于染色体5A区域,这是预料之中的。
位于染色体5A精细定位区间内的DE转录本为进一步鉴定提供了很好的候选基因。基于功能注释,激肽样基因和RING/U-box超家族蛋白是特别有力的候选基因。以往的研究表明,激肽样蛋白可以通过参与微管动力学来调节颗粒长度和细胞膨胀[15那16那60.].RING/U-box蛋白是一种推测的E3连接酶,这是一类与颗粒大小控制有关的酶(稍后将详细讨论;[12那13])。
然而,当仅仅基于转录本的差异表达推测一个(或多个)因果基因的身份时,应谨慎行事。在5A NILs的情况下,很难预测精细定位区间内的DE转录本是否真的与5A QTL的影响有关,还是仅仅是亲本品种之间序列变异的结果,即“关联内疚”。一个相关的例子是最近使用转录组学来定义一个潜在的种子休眠QTL的候选基因(PM19) [44.].随后的研究表明,一个不同的基因在近距离的物理接近(TaMKK3) [61.]是观测到的自然变化的原因[62.].转录组学数据的错误解释是由于DE之间的完全连锁不平衡PM19基因和因果关系TaMKK3原始研究中使用的种质中的基因。
另外考虑是QTL底层的因果基因可能不会差异表达。QTL的表型效应可能是等位基因变异的结果,其改变了与表达水平无关的潜在基因的功能。实际上,可以使用RNA-SEQ数据识别编码区域序列多态性[63.].这些多态性可以转化为遗传标记来进一步精细定位基因,尽管我们在研究中没有采用这种策略。
可以认为,在利用RNA-seq等方法进行进一步研究之前,资源应该更好地集中在QTL的精细定位和克隆上。事实上,通过TILLING或基因编辑对因果基因的直接操作可以精确地询问基因功能及其与兴趣特征的关系(在[64.]). 然而,除了鉴定致病基因本身之外,我们认为使用高度等基因材料的比较转录组学对于理解该性状的潜在生物学是一种有价值的方法。由于RNA-seq(本研究中的两条测序通道)的成本相对较低,这一点变得越来越普遍。RNA-seq是目前可用于多种物种的改良基因组组合[65.,以及改进后的软件(例如[66.那67.),以促进数据的下游分析。将转录组学研究与之前的精细定位和表型特征相结合,可以选择功能特征的候选者,这可以与QTL的进一步精细定位一起进行。在本研究中,发现与粒级组分差异相关的基因将是小麦粒级控制的有趣的新候选基因,无论它们是否是5A QTL的因果基因。的情况下PM19再提供一个相关的例子,如直接操纵PM19在转基因株系中产生了一种谷物休眠表型,尽管事实上它不是所讨论的谷物休眠QTL的因果基因[44.那62.].
最终,在5A QTL的情况下,将需要进一步细微映射轨迹以识别底层基因。然而,我们将考虑通过提供额外的SNP和洞察QTL特征的生物机制来实现转录组织的使用互补方法。
染色体5A外的DE转录本是5A QTL下游的候选靶点
我们认为染色体5A外的DE转录本作为位于5A渐渗基因下游靶点的候选基因,因为差异表达不可能通过序列变异产生。这包括位于所有三个基因组上的基因,这意味着在A、B和D基因组之间存在转录水平的交叉。我们发现,在这些基因的启动子中,与TF家族相关的TF结合位点富集,这些位点以前都被证明在器官发育的控制中发挥不同的作用[68.那69.]例如YABBY基因,一个植物特有的TFs家族,在模式形成和器官极性的建立中起着关键作用[70]及水果大小[71].另一个例子是C2H2 tf,小瘤和锯齿状的,这参与了确定拟南芥中的心皮[72].AT-Hook转录因子在玉米和水稻花器官发育中的作用[73那74]调节拟南芥缺点中的细胞伸长[75].这些转录因子家族很少在小麦中被鉴定,尽管这些相互作用需要实验验证,但它们可能是操纵粒级的潜在靶点。
DE转录本具有控制种子/器官大小的功能
对水稻和拟南芥等物种的研究表明,种子大小是由一个复杂的基因网络和不同的机制调控的,最终通过细胞增殖和扩张的协调。8.那9.])。5A+ NILs的果皮细胞明显较长,提示潜在基因影响细胞扩展[29].在物理上改变细胞壁的基因已经被证明可以直接控制细胞的扩张(在[76])我们鉴定了细胞壁合成和重塑中具有潜在作用的三种转录物。我们还鉴定了与细胞周期相关的许多转录物和细胞增殖的控制。在种子发育过程中,观察到除典型的有丝分裂循环之外的多种细胞周期类型。一种这样的替代循环类型是结束的,其特征在于,在没有细胞分裂的情况下复制染色体,其与细胞扩大相关联(在[77])。其中两个DE转录本是在器官发育中具有特定作用的拟南芥基因中最接近的小麦同源基因:a平相互作用因子(GIF),苏斯(SEU).在拟南芥中,GIF基因与生长调节因子(平)TFs和作为转录共激活因子通过细胞增殖调节器官大小[78].相反,SEU作为一种转录辅助抑制因子,并与重要的发育调节器相互作用,以控制许多过程,包括花器官的发育[79].
种子的发育需要多种组织之间的协调过程,即种皮、胚乳和胚。这些组织的发育和生长是内在相连的,已经有人提出,母体种皮/果皮施加的机械约束对种子/粒的大小施加了一个上限[29那30.那80].表观遗传调控似乎在这些组织的相互交流和协调中发挥着重要作用[81].34个非编码转录本、转座子和组蛋白相关转录本在NILs之间的差异表达可能表明与控制果皮细胞大小有关的表观遗传状态的差异。对这些非编码rna进行特征鉴定的额外工作将有助于确定它们在谷物发育中的作用。
泛素介导的种子/颗粒大小控制在许多物种中已被记录(在[82),包括小麦[13那83].与泛素途径相关的DE转录本在5A NILs中显著富集,尽管只有一个位于精细定位的区间内。该通路通过酶级联的顺序作用,将泛素蛋白的多个拷贝标记到底物蛋白上:E1 (Ub激活)、E2 (Ub结合酶)和E3 (Ub连接酶)。然后将泛素化的底物蛋白对准26S蛋白酶体进行降解[84].GW2,是一种环状E3连接酶,负调控细胞分裂,被鉴定为水稻粒宽和粒重QTL的原因基因[12].拟南芥orthologue,DA2,通过相同的机制调节拟南芥种子大小[14].另一个e3连接酶,EOD1 / BB还负面调节拟南芥的种子大小[49.]通常,E3连接酶决定底物蛋白的特异性[84),DA2和EOD1.可能有不同的底物靶点,但它们会聚并都以泛素激活的蛋白酶为靶点DA1.DA1还负调控细胞增殖,并与两者协同作用DA2和EOD1.,尽管尚不清楚这两种E3连接酶是通过独立的遗传途径作用,还是作为相同机制的一部分[14那50.那85].UBP15(一种泛素特异性蛋白酶)是该途径的下游靶点,反过来通过促进细胞增殖作为种子大小的正调控因子[51.].其他与泛素相关的器官/颗粒大小调节因子已被确定,包括26S蛋白酶体的成分、具有去泛素化活性的酶和已在体外显示与泛素结合的蛋白质[52.那86那87]与该途径相关的DE转录本不是这些先前特征化基因的直接同源物。因此,这些假定的新成分的功能特征可以为泛素介导的谷物粒径控制提供新的见解。
结论
在这项研究中,我们对5A QTL的因果基因的产生了候选者,并且还确定了控制晶粒尺寸的潜在下游途径。这些候选者的子集正在使用耕作突变体在功能上进行测试[88和其他方法,为谷物粒度的控制提供了新的见解。最终确定调控颗粒大小的单个成分和途径,并了解它们如何相互作用,将使我们能够更准确地控制小麦的最终产量。
方法
植物材料
公元前54.本研究中使用的NILs以前有过描述[29].简单地说,NILs是由英国品种‘Charger’和‘Badger’之间的一个双单倍体群体产生的,以Charger作为轮回亲本。在5A染色体上,NILs相差约491 Mbp。我们用一个基因型分别为5A- (Charger等位基因,短粒)和5A+ NIL (Badger等位基因,长粒)。在2014年生长季,采用完全随机区组设计,共5个重复,在1.1 × 6 m的地块(试验单元)种植植株,并在穗完全出苗时标记穗状花序[29].使用开花时间最相似的三个区块进行取样。根据2014年中概述的发育时间过程,在花后4天(时间点1:T1)和8天(时间点2:T2)对植物进行取样[29].对于每个基因型,我们在实验单元内从不同植物的三个独立穗上取样三个粒。因此,每个生物复制由每个基因型汇集9粒谷物组成。籽粒从三个穗的中间部分的外层小花(F1和F2位置)取样。在田间从穗上取下谷粒,立即用液氮冷冻保存在-80°C。在每个时间点,对每个NIL进行3次生物重复(取自野外3个区)。
RNA提取和测序
对于每个生物复制,将九粒颗粒汇集在一起并在液氮下研磨。使用Ambion植物RNA分离辅助剂(Thermo Fisher Scientific)在RE缓冲液(0.1 M Tris pH 8.0,5 mM EDTA pH 8.0,0.1 M NaCl,0.5%SDS,1%β-巯基乙醇)中提取RNA。用1:1酸性苯酚(pH 4.3)提取上清液:氯仿。通过添加异丙醇和3 M醋酸钠(pH 5.2),在-80°C下沉淀RNA。在70%乙醇中洗涤RNA颗粒两次,并在无RNA酶的水中重新悬浮。使用RNeasy Plant Mini kit(Qiagen)对RNA进行DNA酶处理和纯化根据制造商的说明。RNA质控、文库构建和测序由诺里奇厄勒姆研究所(前身为基因组分析中心)执行。文库构建使用TruSeq RNA协议v2(Illumina 15026495 Rev.F)在Perkinlemer Sciclone上执行。文库汇集(2个6个文库)并在HiSeq 2500(Illumina)的两条通道上,使用100 bp配对末端读取和V3化学在高输出模式下进行测序。使用fastQC对读取进行初始质量评估[89].
读取对齐和差异表达分析
读取与来自同一小麦加入,中国春季的两种参考序列对齐,染色体测量序列(CSS; [40]下载了运用TGACv1参考序列[41.].CSS参考包含124,201个高可信度的转录本,其中基于Ensembl plants release 29选择了一个单一的转录本(导致102,503个总转录本)。然而,TGACv1转录组包括来自高可信度和低可信度基因模型的转录变体(分别为164、623和109,116),相当于总转录本的273,239个。我们使用kallisto-0.42.3进行了读取比对和表达量化[66.]使用默认参数,30个引导(−b 30)和–pseudobam选项。Kallisto先前已被证明适合以同源特异性方式对小麦转录组数据进行比对[90].
差异表达分析使用sleuth-0.28.0进行[45.],使用默认参数。假发现率(FDR)调整的转录本P.-value (q value) < 0.05视为差异表达。在所有四种条件下,平均丰度< 0.5 tpm的转录本被认为不表达,因此被排除在进一步分析之外。我们还随机抽取样本,以确定由差异表达分析产生的假阳性的预期数量。我们对基因型和时间点的相对样本进行分组,发现在这种情况下平均有1.83个转录本差异表达(标准误差= 0.32,N= 130)。
对每个条件,计算所有三个生物重复的tpm的平均值。所有的热图显示平均表达值为标准化tpm,在0到1的范围内,1是转录本的最高表达值。使用bedtools-2.24.0 genome cov获得基因模型的Read覆盖率[91,然后组合起来得到每个位置的总覆盖值。一个基因模型的覆盖率以0到1的比例绘制成相对覆盖率,其中1相当于该基因模型的最高覆盖率。
我们还使用定量反转录PCR (qRT-PCR)测量了来自“NIL之间”比较的5个DE转录本子集的表达。qRT-PCR获得的表达值与RNA-seq获得的表达值高度一致(附加文件)7.).对于qRT-PCR,使用与RNA-seq相同的12个RNA样本(即每个时间点每NIL 3个生物重复),并使用M-MLV逆转录酶(Life Technologies)制作cDNA。使用基因组特异性正向和反向引物测定每个转录本的转录水平(附加文件)8.).利用从所有12个样品汇集的cDNA测定引物效率(所有> 88%)。使用LightCycler 480 SYBR Green I Master Mix和LightCycler 480仪器(罗氏应用科学公司,英国)进行qRT-PCR反应,条件如下:在95°C下5分钟;45个循环,95°C下10秒,60°C下15秒,72°C下30秒。通过解离曲线分析(60 ~ 95℃)确定引物的特异性。所有反应都经过三个技术重复。计算相对于肌动蛋白的相对表达水平[92]和线性化的值确定使用(1 +效率)−ΔΔCt方法(93].
去词浓缩
我们使用了R包GOseq v1.26 [94]检测氧化石墨烯在特定组DE转录本中的富集情况。我们考虑了过度代表的GO术语与本杰明·霍克伯格罗斯福调整P.-值< 0.05则显著富集。
功能注释
TGACv1注释获得转录本的功能性注释[41.].此外,对于编码转录本,我们对非冗余NCBI蛋白数据库和保守结构域数据库进行BLASTP,在每个情况下基于e值的最高命中都被保留。如果三个注释都一致,则报告TGAC注释。如果三个注释产生了不同的结果,则报告所有注释。从拟南芥和水稻等其他物种中获得同源物运用植物释放36。112份DE转录本中有8份与其他物种没有注释或蛋白质序列相似。我们根据剩余的104份DE转录本的预测功能对其进行了手工分类。大小为1的文本被归类为“其他”。对于非编码转录本,我们使用BLASTN识别潜在的miRNA前体,使用一组保守的小麦特异性miRNA序列,这些序列来自Sun et al., 2014 [48.].我们使用BLAST的-task blastn-short选项对短序列进行处理,只考虑没有错配的miRNA序列的全长命中作为潜在前体。我们使用了psRNAtarget工具(http://plantgrn.noble.org/psrnatarget/)确定miRNA的靶点。
转录因子结合位点的鉴定
我们在cDNA开始站点的上游提取了1000bp的序列,以搜索转录因子结合位点(TFB)。在分析中不使用参考序列中<1000bp上游的转录物。我们从MEME套件中使用了FIMO工具(V 4.11.4; [95])的位置权重矩阵(PWM),该矩阵由plantPAN 2.0 (http://plantpan2.itps.ncku.edu.tw/; [96])。我们用FIMOP.<1E-4(默认)的值阈值,将最大存储的分数增加到1,000,000,以考虑数据集的大小,并根据Peng等人推荐使用1E-8的-Motif-Pseudo。[97]以配合pwm使用。我们使用MEME的fasta-get-markov命令对所有提取的启动子序列生成背景模型。
浓缩试验
为了测试相对于所有表达的转录物的不同类别的转录物,我们使用R-3.2进行了Fisher的确切测试。对于功能性注释类别,浓缩测试仅在可以根据TGAC参考中的注释使用GO术语和关键词来提取的类别执行。在富集试验中使用可以用该方法提取的DE转录物。例如,我们鉴定了与泛素相关的12名转录物。通过TGAC注释和手动注释的组合获得这些转录物的注释。但是,可以使用GO术语和来自整个参考注释的关键词来提取这些成绩单中的七个。因此,只使用七种转录物用于富集试验。
参考文献
- 1.
联合国。世界人口前景:2015年修订。https://esa.un.org/unpd/wpp/publications/files/key_findings_wpp_2015.pdf.2017年2月27日。
- 2.
粮农组织,IFAD,WFP。2015年世界粮食不安全状况。召开2015年国际饥饿目标:占据不均匀进展。罗马,粮农组织2015年。
- 3.
Rosegrant MW, Tokgoz S, Bhandary P, Msangi S.展望未来:食物的前景。2012年全球粮食政策报告。华盛顿特区:国际粮食政策研究所;2013.p . 88 - 101。
- 4.
王志强,王志强。全球粮食需求与农业可持续集约化。中国科学院院刊。2011;108(50):20260-4。https://doi.org/10.1073/pnas.1116437108.
- 5。
粮农组织。2050年全球农业。罗马:粮农组织;2009.
- 6。
Ray DK, Mueller ND, West PC, Foley JA。到2050年,产量趋势不足以使全球作物产量翻番。《公共科学图书馆•综合》。2013;8 (6):e66428-e。https://doi.org/10.1371/journal.pone.0066428.
- 7.
陈志强,陈志强,陈志强,等。面包小麦籽粒产量的遗传分析。一、QTL分析。应用计算机学报。2007;115(8):1029-41。https://doi.org/10.1007/S00122-007-0629-7..
- 8.
李娜,李勇。植物种子大小的母体控制。中国生物医学工程学报。2015;66(4):1087-97。https://doi.org/10.1093/jxb/eru549..
- 9.
黄锐,姜玲,郑军,王涛,王华,黄勇,等。米粒形状的遗传基础:如此多的基因,如此鲜为人知。植物科学进展。2013;18(4):218-26。https://doi.org/10.1016/j.tplants.2012.11.001.
- 10.
王胜,吴科,袁强,刘旭,刘震,林旭,等。控制晶粒尺寸、形状和质量OsSPL16《水稻自然遗传学》2012;44(8):950-4。https://doi.org/10.1038/ng.2327.
- 11.
Garcia D,Gerald JNF,Berger F. Endosperm Growth对细胞伸长率和对胚胎生长的引发控制的母体控制,以确定拟南芥中的种子大小。植物细胞。2005; 17(1):52-60。https://doi.org/10.1105/tpc.104.027136.
- 12.
宋晓军,黄伟,史敏,朱明志,林宏旭。水稻粒宽和粒重的QTL编码一个未知的环型E3泛素连接酶。Nat麝猫。2007;(5):623 - 30。https://doi.org/10.1038/ng2014.
- 13.
Simmonds J, Scott P, Brinton J, Mestre TC, Bush M, Blanco A,等。中剪接受体位点突变TaGW2-A1在四倍体和六倍体小麦中,通过更宽和更长的谷粒增加千粒重。[j] .计算机应用。2016;129(6):1099-112。https://doi.org/10.1007/s00122-016-2686-2.
- 14。
夏T,李N,杜明尼J,李J,卡门斯基A,贝万MW,等。泛素受体DA1与E3泛素连接酶相互作用DA2调节拟南芥中的种子和器官大小。植物细胞。2013; 25(9):3347-59。https://doi.org/10.1105/tpc.113.115063.
- 15.
Kitagawa K,Kurinami S,Oki K,Abe Y,Ando T,Kono I,等。一种调节水稻种子长度的新型驱动蛋白13。植物细胞生理学。2010;51(8):1315-29。https://doi.org/10.1093/pcp/pcq092.
- 16。
Fujikura U,Elsaesser L,Breuninger H,Sánchez-RodríguezC,Ivakov A,Laux T等人。Atkinesin-13a调节细胞壁合成和细胞扩展拟南芥通过THESEUS1途径。公共科学图书馆麝猫。2014;10 (9):e1004627。https://doi.org/10.1371/journal.pgen.1004627.
- 17。
范超,邢勇,毛洪,陆涛,韩波,徐超,等。GS3是水稻粒长和粒重的主要QTL,粒宽和粒厚的次要QTL,编码一个可能的跨膜蛋白。应用计算机学报。2006;112(6):1164-71。https://doi.org/10.1007/s00122-006-0218-1.
- 18.
黄X,钱Q,刘Z,Sun H,He S,Luo D等人。DEP1基因座的自然变化增强了水稻中的谷物产量。NAT Genet。2009; 41(4):494-7。https://doi.org/10.1038/ng.352.
- 19.
Riefler M,Novak O,Strnad M,SchmüllingT.拟南芥Cytokinin受体突变体揭示了芽生长,叶片衰老,种子大小,萌发,根部发育和细胞蛋白代谢的功能。植物细胞。2006; 18(1):40-54。
- 20
Schruff Mc,Spielman M,Tiwari S,Adams S,Fenby N,Scott RJ。Auxin响应因子2拟南芥链接养肝信号传导,细胞分裂和种子和其他器官的大小。发展。2006; 133(2):251-61。https://doi.org/10.1242/dev.02194.
- 21
姜文波,黄海英,胡永文,朱绍文,王志勇,林文华。油菜素内酯调节拟南芥种子的大小和形状。植物杂志。2013;162(4):1965 - 77。
- 22
粮农组织。在线统计数据库:食品平衡。遗传邮件。2017年。http://www.fao.org/faostat/en/.通过2017年2月27日。
- 23.
Ma M,Zhao H,Li Z,Hu S,Song W,Liu X. Tacyp78a5调节小麦的种子大小(小麦).J Exp Bot. 2016;67(5): 1397-410。https://doi.org/10.1093/jxb/erv542.
- 24.
Simmonds J, Scott P, Leverington-Waite M, Turner AS, Brinton J, Korzun V,等。六倍体小麦6A染色体稳产千粒重QTL的鉴定与独立验证(小麦L。)。BMC植物生物学。2014; 14(1):191。https://doi.org/10.1186/s12870-014-0191-9..
- 25.
Breseghello F, Sorrells ME。小麦两个六倍体定位群体籽粒大小和形状的QTL分析。中国农业科学(英文版);2010 - 03https://doi.org/10.1016/j.fcr.2006.11.008.
- 26.
Gegas VC, Nazari A, Griffiths S, Simmonds J, Fish L, Orford S,等。小麦粒度和形状变异的遗传框架。植物细胞。2010;22(4):1046 - 56。https://do.org/10.1105/TPC.110.074153.
- 27.
Farré A, Sayers L, Leverington-Waite M, Goram R, Orford S, Wingen L,等。应用近等基因系文库了解面包小麦籽粒产量和适应性性状QTL的背景依赖表达。BMC Plant Biol. 2016;16https://doi.org/10.1186/s12870-016-0849-6.
- 28.
等。关键词:人工智能,人工智能,人工智能利用高密度SNP连锁图谱分析精英x非适应杂交小麦籽粒形状和大小的遗传因素植物Genome-Us。2016; 9 (1)https://doi.org/10.3835/plantgenome2015.09.0081.
- 29.
Brinton J、Simmonds J、Minter F、Leverington Waite M、Snape J、Uauy C.果皮细胞长度增加是六倍体小麦粒重的主要数量性状位点的基础。新植物醇。2017;215(3):1026–38.https://doi.org/10.1111/nph.14624.
- 30.
Hasan AK, Herrera J, Lizana C, Calderini DF。心皮重量、粒长和稳定的籽粒含水量是小麦粒重测定的生理驱动因素。中国农业科学(d辑:地球科学)2011;https://doi.org/10.1016/j.fcr.2011.05.019.
- 31.
郭志,陈德,陈德华。小麦花发育过程中花药和子房大小的方差、遗传力和相关分析。J Exp Bot. 2015;https://doi.org/10.1093/jxb/erv117.
- 32.
张志强,张志强,张志强,等。小麦籽粒发育的综合研究。至)。谷物科学。2012;56(1):21-30。https://doi.org/10.1016/j.jcs.2011.11.007.
- 33.
Wan Y, Poole RL, hutly AK, Toscano-Underwood C, Feeney K, Welham S, et al.;六倍体小麦籽粒发育的转录组分析。BMC基因组学,2008;9https://doi.org/10.1186/1471-2164-9-121.
- 34.
6 .陈志强,陈志强,陈志强,陈志强。六倍体面包小麦转录组基因组间的相互作用。科学。2014;345(6194):1250091。
- 35.
Pellny TK,Lovegrove A,Freeman J,Tosi P,Love CG,Knox JP等.发育中小麦淀粉胚乳的细胞壁:成分和RNA-Seq转录组的比较.植物生理学.2012;158(2):612-27。https://doi.org/10.1104/pp.111.189191.
- 36.
于宇,朱东,马超,曹辉,王勇,徐勇,等。转录组分析揭示了参与小麦籽粒发育的关键差异表达基因。作物学报。2016;4(2):92-106。https://doi.org/10.1016/j.cj.2016.01.006.
- 37.
Laudencia-Chingcuanco DL, Stamova BS, You FM, Lazo GR, Beckles DM, Anderson OD。利用cDNA微阵列对小麦颖果发育的转录分析。植物学报。2007;63(5):651-68。https://doi.org/10.1007/S11103-006-9114-Y..
- 38.
刘伟,吴智,张勇,郭丁,徐勇,陈伟,等.基于RNA序列的小麦籽粒转录组分析.农业科学与工程前沿.2014;1(3):214-22。https://doi.org/10.15302/j-fase-2014024.
- 39。
IWGSC RefSeq v1.0。https://wheat-urgi.versailles.inra.fr/Seq-Repository/Assemblies.
- 40.
IWGSC。六倍体面包小麦(小麦)基因组。科学。2014;345(6194):1251788。https://doi.org/10.1126/science.1251788.
- 41.
Clavijo BJ,Venturini L,Schudoma C,Garcia Accinelli G,Kaithakottil G,Wright J等人。Allohexaploid小麦基因组的改进组装和注释鉴定了农艺基因的完整系列,并为染色体易位提供基因组证据。Genome Res。2017; 27:885-96。https://doi.org/10.1101/gr.217117.116.
- 42
Zimin AV,Puiu D,Hall R,Kingan S,Clavijo BJ,Salzberg SL.第一个接近完整的六倍体面包小麦基因组组装。小麦bioRxiv。2017;https://doi.org/10.1101/159111.
- 43
Cantu D,Pearce SP,Distelfeld A,Christiansen MW,Uauy C,Akhunov E等。高表达基因下调的效应谷物蛋白质含量(GPC)基因在单果衰老过程中的作用。BMC基因组学。2011;12(1):492。https://doi.org/10.1186/1471-2164-12-492.
- 44
作者单位:国家自然科学基金青年基金,国家自然科学基金青年基金。小麦近等基因系鉴定的转录组分析PM19-A1和A2作为主要休眠QTL的候选。基因组生物学。2015;16(1):93。https://doi.org/10.1186/s13059-015-0665-6.
- 45
基于不确定度的RNA-seq差异分析。Nat方法。2017;14(7):687 - 90。https://doi.org/10.1038/nmeth.4324.
- 46
小麦NAC转录因子家族的全基因组序列及表达分析。G3: | |基因组遗传基因。2017; 7(9): 3019 - 29。https://doi.org/10.1534/g3.117.043679..
- 47.
Guttman M, Rinn JL。大型非编码rna的模块化调控原理。大自然。2012;482(7385):339 - 46所示。
- 48.
Sun F,Guo G,Du J,Guo W,Peng H,Ni Z等。麦克纳斯的全基因组和他们在小麦的目标(小麦L。).acta botanica yunnanica(云南植物研究). 2014;14(1):142。https://doi.org/10.1186/1471-2229-14-142.
- 49.
Disch S,Anastasiou E,Sharma VK,Laux T,Fletcher JC,Lenhard M. e3泛素连接酶大哥控制拟南芥器官大小与剂量有关。咕咕叫医学杂志。2006;16(3):272 - 9。https://doi.org/10.1016/j.cub.2005.12.026.
- 50.
董辉,卢芳华,王志强,等。泛素化激活肽酶,促进其激活E3连接酶和多种生长调节蛋白的切割和不稳定,以限制拟南芥细胞增殖。基因Dev。2017;31(2):197 - 208。https://doi.org/10.1101/gad.292235.116.
- 51.
Du L,Li N,Chen L,Xu Y,Li Y,Zhang Y,等。泛素受体DA1通过调节泛素特异性蛋白酶UBP15/SOD2的稳定性来调节种子和器官的大小拟南芥.植物细胞,2014,26(2):665 - 77。https://doi.org/10.1105/TPC.114.122663.
- 52.
黄凯,王东,段鹏,张斌,徐锐,李宁,等。宽而厚粒1,它编码一种具有去泛素活性的类似于otubain的蛋白酶,影响着大米的颗粒大小和形状。植物j . 2017; 91(5): 849 - 60。https://doi.org/10.1111/tpj.13613.
- 53.
关键词:植物生长素,器官发生,NPY1, bbt - nph3样蛋白拟南芥.中国科学(d辑:地球科学)2007;104(47):18825-9。https://doi.org/10.1073/pnas.0708506104.
- 54.
大麦母体种子组织的发育受细胞扩张和解体的调控,并与胚乳生长协调。中国生物医学工程学报。2011;62(3):1217-27。https://doi.org/10.1093/jxb/erq348.
- 55.
Drea S, Leader DJ, Arnold BC, Shaw P, Dolan L, Doonan JH。小麦颖果发育过程中基因表达的系统空间分析。植物细胞。2005;17(8):2172 - 85。https://doi.org/10.1105/tpc.105.034058.
- 56.
Dominguez F, Cejudo FJ。小麦籽粒发育过程中出现的内蛋白酶的特性。植物杂志。1996;112(3):1211 - 7。https://doi.org/10.1104/pp.112.3.1211.
- 57.
王志强,王志强,王志强,等。无标记蛋白质组分析揭示了年轻大麦粒的发育依赖模式。蛋白质组学杂志。2016;143:106-21。https://doi.org/10.1016/j.jprot.2016.04.007.
- 58.
皮雷JC,科南特GC。稳健但脆弱:表达噪音,蛋白质错误折叠,基因在基因组进化中的剂量。Annu Rev Genet. 2016; 50:113-31。https://doi.org/10.1146/annurev-genet-120215-035400.
- 59.
杨米,高X,董俊,甘地,蔡H,冯Wettstein dh等。通过蛋白质组学分析鉴定的麦粒蛋白表达的模式。前植物SCI。2017; 8:962。https://doi.org/10.3389/fpls.2017.00962.
- 60。
李军,蒋军,钱强,徐勇,张超,肖军,等。水稻BC12/GDD1编码一种与GA生物合成基因启动子结合的激肽样蛋白,其突变导致细胞伸长受损的侏儒症。植物细胞。2011;23(2):628 - 40。https://doi.org/10.1105/tpc.110.081901.
- 61。
等。小麦4A染色体种子休眠原因基因编码MAP激酶激酶。咕咕叫医学杂志。2016;26(6):782 - 7。https://doi.org/10.1016/j.cub.2016.01.063.
- 62。
Shorinola o,Balcárkováb,hyles j,jfg t,hayden mj,Holušovak,等。Haplotype分析预收获前发芽阻力位于Phs-A1揭示了因果关系TaMKK3-A在全球种质中。前植物SCI。2017; 8:1555。https://doi.org/10.3389/fpls.2017.01555.
- 63.
Ramirez-Gonzalez RH, Segovia V, Bird N, Fenwick P, Holdgate S, Berry S,等。RNA-Seq批量分离分析使高分辨率遗传标记的鉴定在六倍体小麦育种。acta botanica yunnanica(云南植物研究);2015;13(5):613-24。https://doi.org/10.1111/pbi.12281.
- 64.
Uauy C,Wulff BBH,Dubcovsky J.将传统诱变与新的高通量测序和基因组编辑相结合,以揭示多倍体小麦中隐藏的变异。遗传学年度评论。2017;51(1)https://doi.org/10.1146/annurev-genet-120116-024533.
- 65.
周建平,黄建军,黄建军,黄建军。基因组技术在作物改良中的应用。大自然。2017;543(7645):346 - 54。https://doi.org/10.1038/nature22011.
- 66
Bray NL,Pimentel H,熔化P,Pachter L.近乎最佳概率RNA-SEQ定量。NAT BIOTECH。2016; 34(5):525-7。https://doi.org/10.1038/nbt.3519.
- 67
Patro R,Duggal G,Love Mi,Irizarry Ra,Kingsford C. Salmon提供了快速和偏见的转录表达式的量化。NAT Meth。2017; 14(4):417-9。https://doi.org/10.1038/nmeth.4197.
- 68
转录因子的三螺旋家族-光、应激和发育。植物科学进展。2012;17(3):163-71。https://doi.org/10.1016/j.tplants.2011.12.002.
- 69
Barg R,Sobolev I,Eilon T,Gur A,Chmelnitsky I,Shabtai S等人。番茄早期果实特异性基因Lefsm1定义了一种新型的植物特异性Sant / MyB结构域蛋白。Planta。2005; 221(2):197-211。https://doi.org/10.1007/S00425-004-1433-0..
- 70.
等。关键词:人工神经网络,神经网络,神经网络区分拟南芥由Yabby活动组合的叶子射击。植物细胞。2010; 22(7):2113-30。https://doi.org/10.1105/tpc.110.075853.
- 71.
Cong B,Barrero LS,Tanksley SD。雅比特的转录因子的监管变化导致番茄驯化期间极端果实大小的演变。NAT Genet。2008; 40(6):800-4。https://doi.org/10.1038/ng.144.
- 72.
dineny JR, Weigel D, Yanofsky MF。瘤状和锯齿状定义雄蕊和心皮形状拟南芥.发展。2006; 133(9):1645-55。https://doi.org/10.1242/dev.02335.
- 73.
金勇,罗强,童华,王安,程志,唐军,等。水稻胚层形成和花器官数量控制需要一个AT-hook基因。Dev杂志。2011;359(2):277 - 88。https://doi.org/10.1016/j.ydbio.2011.08.023.
- 74.
Gallavotti A,Malcomber S,Gapes C,Stanfield S,Whipple C,Kellogg E等人。贫瘠的茎快速1是形成玉米耳朵所需的钩子蛋白。植物细胞。2011; 23(5):1756-71。https://doi.org/10.1105/tpc.111.084590.
- 75.
SOB3/AHL29和ESC/AHL27是拟南芥下胚轴生长的负调控因子。植物j . 2008; 54(1): 1 - 14。https://doi.org/10.1111/j.1365 - 313 x.2007.03393.x。
- 76.
Cosgrove DJ.植物细胞壁的生长.美国国家自然科学院摩尔细胞生物学版.2005;6(11):850-61。https://doi.org/10.1038/nrm1746.
- 77.
Dante Ra,Larkins Ba,Sabelli Pa。细胞周期控制和种子发展。前植物SCI。2014; 5:493。https://doi.org/10.3389/fpls.2014.00493.
- 78.
李BH, Ko J-H, Lee S, Lee Y, Pak J-H, Kim JH。拟南芥grf - interaction FACTOR基因家族在决定器官大小和多种发育特性方面发挥重叠功能。植物杂志。2009;151(2):655 - 68。https://doi.org/10.1104/pp.109.141838.
- 79.
Bao F,Azhakanandam S,Franks RG。Seuss和Seuss样转录适配器调节花卉和胚胎发育拟南芥.植物理性。2010; 152(2):821-36。https://doi.org/10.1104/pp.109.146183.
- 80。
Adamski NM, Anastasiou E, Eriksson S, O ' ' ' CM, Lenhard M. Local mother control of seed size by KLUH/CYP78A5-dependent growth signaling。中国科学(d辑:地球科学)2009;106(47):2015 - 20。https://doi.org/10.1073/pnas.0907024106..
- 81。
植物生长素对拟南芥和玉米种子发育的影响。植物科学学报2014;5:412。https://doi.org/10.3389/fpls.2014.00412.
- 82。
Li N, Li Y. ubiquitin介导的植物种子大小调控。植物科学学报。2014;5:332。https://doi.org/10.3389/fpls.2014.00332.
- 83。
杨志强,白志强,王鹏,杨磊,等。小麦粒重相关基因TaGW2的SNP鉴定和等位基因特异性PCR标记的建立。应用计算机学报。2012;125(5):1057-68。https://doi.org/10.1007/s00122-012-1895-6.
- 84。
Hershko A, cichanover A,泛素系统。《生物化学学报》1998;67:425-79。https://doi.org/10.1146/annurev.biochem.67.1.425.
- 85。
Vanhaeren H,Nam Y-J,De Mille L,Chae E,Storme V,Weigel D等。永远年轻:泛素受体DA1和E3连接酶BIG BROTHER在控制叶片生长发育中的作用。植物生理学。2016;01410https://doi.org/10.1104/pp.16.01410.
- 86。
王胜,李勇,李玉林,王胜。26S蛋白酶体功能的丧失导致拟南芥芽器官的细胞大小增加和细胞数量减少。植物杂志。2009;150(1):178 - 89。https://doi.org/10.1104/pp.109.135970.
- 87.
翁军,顾胜,万旭,高华,郭涛,苏宁,等。水稻粒宽和粒重相关QTL GW5的分离及初步鉴定。细胞研究》2008;18(12):1199 - 209。
- 88.
等。发现多倍体小麦的隐性变异。中国科学院院刊。2017;114(6):E913-E21https://doi.org/10.1073/pnas.1619268114.
- 89.
FastQC:用于高通量序列数据的质量控制工具。2010.http://www.bioinformatics.babraham.ac.uk/projects/fastqc.2015年9月9日访问。
- 90。
Borrill P,Ramirez Gonzalez R,Uauy C.expVIP:一个可定制的RNA序列数据分析和可视化平台。植物生理学。2016;170(4):2172-86。
- 91.
昆兰AR, Hall IM。BEDTools:用于比较基因组特征的一套灵活的工具。生物信息学,2010,26(6):841 - 2。https://doi.org/10.1093/bioinformatics/btq033.
- 92.
Uauy C,Distelfeld A,Fahima T,Blechl A,Dubcovsky J.调节衰老的NAC基因改善了小麦中的晶粒蛋白,锌和铁含量。科学。2006; 314(5803):1298-301。
- 93.
Schmittgen TD, Livak KJ。采用对比CT方法分析实时荧光定量PCR数据。Nat Protoc。2008;3:1101。https://doi.org/10.1038/nprot.2008.73.
- 94.
Young MD,Wakefield MJ,Smyth GK,Oshlack A.RNA序列的基因本体分析:选择偏差的解释.基因组生物学.2010;11(2):R14。https://doi.org/10.1186/gb-2010-11-2-r14.
- 95.
格兰特CE,Bailey TL,Noble WS。FIMO:扫描给定主题的发生。生物信息学。2011; 27(7):1017-8。https://doi.org/10.1093/bioinformatics/btr064.
- 96.
周志宁,郑华强,吴宁勇,钱志华,黄宏东,李天勇,等。PlantPAN 2.0:重建植物转录调控网络的植物启动子分析导航仪的更新。44(数据库版):D1154-D60。https://doi.org/10.1093/nar/gkv1035.
- 97.
彭凤飞,胡震,杨荣昌。模式和温带谷类植物光周期和春化反应基因启动子区转录因子结合位点的生物信息学预测BMC基因组学。2016;17:573。https://doi.org/10.1186/s12864-016-2916-7.
确认
我们感谢David Swarbreck和Gemy Kaithakottil(emperham学院)在TGACv1基因模型的Silico映射和IWGSC中进行预发布访问Refseq V.1.0。
资金
这项工作由英国生物技术和生物科学研究理事会(BBSRC)拨款BB/P013511/1和BB/P016855/1资助。JB得到了英国农业和园艺发展局(AHDB)和约翰·英尼斯基金会的支持。
数据和材料的可用性
支持本文结果的数据集包含在本文及其附加文件中。原始序列读取保存在生物工程PRJNA396738下的NCBI序列读取档案中。
作者信息
隶属关系
贡献
JB设计研究,进行RNA提取,分析数据,进行统计分析,撰写手稿;JS协调了田间试验,开发了本研究使用的种质;CU设计了研究并撰写了手稿。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
作者声明他们没有相互竞争的利益
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Brinton, J., Simmonds, J. & Uauy, C.在对比六倍体小麦果皮细胞大小和粒重的等基因系中有差异表达的泛素相关基因。BMC植物BIOL.18,22(2018)。https://doi.org/10.1186/s12870-018-1241-5
收到了:
公认:
发表:
关键词
- 小麦
- RNA-SEQ.
- 泛素
- 粒重
- 果皮
- 转录组学
- 晶粒尺寸
- 近代源线