摘要
背景
大多数植物的叶绿体基因组(质体)是高度保守的结构,基因内容,基因和顺序。寄生的植物,包括那些完全光合作用,往往含有原质重排。这些最显着的包括导致较小的质体大小的基因缺失。基因缺失基因组结构重排的性质已经在几个寄生植物了研究,但他们的时间和贡献,这些寄生虫的适应还需要进一步调查,特别是在欠研究半寄生虫。
结果
采用从头测序、组装和注释的方法,研究了五种来自Orobanchaceae的光合寄生虫的叶绿体基因组。其中4个发生了主要的结构重排,包括基因复制和基因丢失,这使得类群发生了分化。兼性寄生虫Aureolaria virginica与非寄生近亲的基因组含量最相似,Lindenbergia philippensis,具有类似的基因组大小和组织,没有基因含量的差异。相比之下,兼产寄生虫Buchnera美国和三个寄生寄生虫斯特所有这些塑料的放大,主要是由倒置的膨胀引起的,该倒置重复(IRS)是标准的塑料特征。其中一些IR增加由多种调查的物种共享,但其他人对特定谱系是独一无二的。基因缺失和伪析化也既有共享和谱系特异性,特别频繁和独立丧失ndh与电子回收有关的基因。
结论
完全组装并比较了五种新的塑性基因组。结果表明,寄生植物中含有塑性不稳定性,甚至那些保留需要像光合作用这样的需要进行必要的塑性功能的那些。基因损失在分类群中缓慢而且不相同,这表明不同的谱系对其一些塑性基因含量不同的用途或需求,包括参与光合作用一些方面的基因。最近的重复区域扩展,一些独特的终端物种分支,在分歧之后被观察到Buchnera /斯特这表明这一罕见的事件在这一谱系中具有特殊的价值。
背景
除了在光合作用中的重要作用外,叶绿体也是一些色素、脂类、氨基酸和硫化合物合成的特殊场所[1,2].叶绿体包含它自己的基因组,即质体。维管植物的质体被认为有一个单一的内吞起源,超过10亿年前,从一个蓝藻样的有机体[3.,4,5].随着时间的推移,睾丸的大多数基因已经丢失或已迁移到核和/或线粒体基因组,因此现代植物塑料含有约10%的祖先肠胃基因基因[6].
在大多数植物中,质体处于强大的选择压力下,因此在基因含量和顺序、核苷酸替换率、结构和大小方面趋于高度保守[7].大多数质体的大小在120到160 kb之间[1].最初,质体被认为是单环分子。虽然有百分之一的质体具有这种结构,但它们也存在于二聚体和高阶串联体中,有圆形的,也有线性的[2].质体具有由两个几乎相同的反向重复序列(IR)分隔的大单拷贝区和小单拷贝区组成的四元结构一个和红外B)地区[8].这两个反向重复序列之间的重组在稳定质体方面起着重要作用[9].在被子植物中,LSCs通常为80 - 90kb, SSCs为16 - 27kb,反向重复序列为20 - 30kb [2].原质体系含有编码参与光合作用以及其它生化途径的蛋白质的基因在叶绿体中进行。它也编码30点的tRNA和其它几个结构的RNA [10,11,12].
与大多数植物不同,寄生植物通常含有高度分化的质体[13,14,15,16,17,18,19,20.,21,22,23,24,25,26,27,28].即获得来自寄主植物所有的营养物质非光合植物被称为holoparasites,而hemiparasites是获得主机和从光合作用其余的一些营养素的光合植物。Hemiparasites可以进一步细分为需要它们的生命周期或兼性寄生虫,其能够完成其整个生命周期中没有宿主植物中的至少一部分期间宿主植物,但如果一个主机可用可寄生寄生菌。寄生植物plastomes可以具有较低的选择性压力由于对光合作用的依赖。毫不奇怪,holoparasites要失去所有的光合作用的基因,或者是整个叶绿体基因组[潜力24而半寄生虫则需要维持大部分或全部的光合基因。缺乏或较少的选择保留光合作用和其他重要的叶绿体功能,可以允许较高的核苷酸取代率和重排在质体。这些变化可能包括通过假基因化为功能基因丢失或通过基因缺失造成的物理损失,从而导致更小的质体尺寸[14,16,17,19,20.,21,22,23,26].在某些情况下,其中一个反转重复的整个副本已经丢失[19,20.,29].由于重复在质体结构中起稳定作用,这将导致质体进一步的不稳定和重排[30.].
Orobanchaceae是最大的寄生植物科。它们只有一个非寄生属,Lindenbergia[31],此外还有全寄生虫和兼性和专性半寄生虫[32].在这项研究中,我们研究了两种兼hemiparasites的,Aureolaria virginica和Buchnera美国,和属于属的三种迫使血管血清斯特:S. hermonthica,S. forbessii., 和美国粗.自养生物的质体Lindenbergia philippensis已经进行完全测序,并通过Wicke等注释。2013年,所以它提供了宝贵的外类群比较我们的干扰物。尽管我们所有研究的物种的依靠光合作用,我们发现基因丢失和其他形式的plastomes重排的许多案件。
方法
样本采集、DNA提取、DNA测序
从叶组织的DNA制备斯特物种与之前描述的相同[33].B. Americana.和答:virginica叶组织取自佐治亚大学植物生物学教学收藏中的活标本。用改进的十六烷基三甲基溴化铵法从这些样品中分离出高分子量dna [34根据制造商的说明,使用NEBNext®Ultra™II DNA文库准备试剂盒进行Illumina文库制备。Illumina MiSeq PE250试验是在乔治亚基因组研究所进行的。
塑料组装
从总细胞DNA序列中选择质体同源序列Striga Hermonthica然后用De Novo集装管道组装,如前所述[35],创建10个重叠群。为了填补在该组件的十个间隙,设计引物对每个重叠群的端部和PCR设置为对于每个引物对。每个PCR反应由5-20纳克模板DNA,10μLQ5反应缓冲液,10μL的甜菜碱,2.5微升DMSO,1个微升10毫摩尔dNTP,2.5μL正向引物,2.5微升反向引物,0.5微升Q5高保真DNA聚合酶和20微升水。递降PCR,是对一个MJ研究PTC-200 Peltier热循环仪每个反应进行。所用的程序为98℃30秒;10个循环在98℃20秒由变性,在68℃退火 - 1°每个循环15秒,伸长率下以72℃1分钟;25个循环在98℃15秒,58由变性℃进行15秒,伸长率在72℃下1分钟;并在72℃的最终延伸步骤为10分钟。PCR产物送Macrogen美国中心(Rockville,MD)的Sanger测序。 Sequences were manually assembled to the original contig ends in order to fill gaps.
聚集在一起S. hermonthica质体与Lindenbergia philippensis使用淡紫色2.3.1的质体[36].所有路口的预测重排S. hermonthica通过PCR扩增和Sanger测序(美国Macrogen公司)验证其装配正确。
通过鉴定对我们的S.Hermonthica组件的读数同源的读数,从原始的总DNA读取中提取了我们的每个靶种类的血浆读取。用毒性8.1.6的De Novo组件用于诊断37].因此,每种物种都独立地进行每个DE Novo组件。
每个组件的重复区域由塑料的区域识别,与塑料的其余部分相比,覆盖的覆盖率约为两倍。如果是S. hermonthica,我们对所有重复序列与LSC和SSC之间的连接点进行了PCR。这些PCR产物的测序证实了这些连接的组装。
质体注释和比对
来自注释的基因l . philippenis从NCBI下载了叶绿体基因组数据库(Chloroplast Genome DB)中所有植物的质体基因[38].我们对每个装配好的质体对所有基因进行BLAST试验。所有在质体中发现的基因都被geneous定位到质体中。Reads也分别被映射到每个基因上,以确认序列组装是正确的,并且在我们的组装中没有缺失Reads映射到它们的基因。所有五种寄生物种的个体基因都是一致的l . philippensis使用Clustal W [39来识别突变、插入和缺失。含有一个或多个移码突变或过早终止密码子的基因被认为是潜在的伪基因。对大多数可能产生伪基因的5 '终止密码子或移码突变的原始读取进行了目视检查,以评估读取调用的准确性。这些位点的阅读深度从34X到548X不等,其中18个病例与突变呼叫100%一致,其他病例显示> 95%一致(数据未显示)。
用Mauve 2.3.1对质体进行比对。系统发育树是由所有五个物种共享的完整基因序列数据生成的。这些基因被连接成一个单一序列,使用Mega 7.02中的Clustal W比对[40].l . philippensis作为外群体被纳入。贝叶斯系统发育分析在MrBayes 3.2.1上进行[41采用广义时间可逆+ Γ + I进化模型。我们最初运行的是2.0 × 107每10000代采样一次的树。第一个5.0 × 106树木不包括在最终分析中。剩下的树木被用来产生共识树。使用相同的数据来使用Mega产生系统发育树[40],并且该生成的几乎相同的结果,因此只MrBayes树被呈现。
结果
塑料组装
叶片总DNA的Illumina序列分析为5种植物的全基因组组装提供了充足的质体-同源序列数据。通过PCR和PCR产物Sanger测序,确认了所有的初始连续序列,并填补了空白。因此,获得了完整的组装,并且通过比较这些物种的基因组所预测的重排也通过PCR和Sanger测序得到了证实。
我们的装配表明塑料答:virginica是153,547 bp,大小接近155,103 bp的质体L. Philippensis。LSC,SSC和IR的长度(84,317bp,17,168 bp和26,031 bp)答:virginica都是在尺寸上和l . philippensis(分别为85,584 bp、17,885 bp和25,812 bp)。来自其他四种物种(S. hermonthica, S. aspera,S.forbessii和B. americana)在总质体大小和各区域大小上均存在较大差异.
重复扩张
他们三个斯特物种和B. Americana.均表现出较大的质体尺寸,范围为166,596 bpB. Americana.至190,233 bpS. forbessii..较大的塑料尺寸与较大的红外长度相关,范围从43,864 bpB. Americana.至63,240 bpS. forbessii..相反,四种物种的LSC和SSC区域大小均有所减小。SSC长度为3377 bpBuchnera到11,191 bpS. forbessii., LSC长度为51,628 bpS. hermonthica至75,491 bp美国。所有塑料组件尺寸都显示在表中1,并与之前研究过的一些寄生植物进行比较。
在B. Americana., LSC和SSC区域均发生了重复扩张。大约8 kb的LSC序列侧翼的重复l . philippensis现在包含在重复区域中B. Americana..这个区域包含基因trnH,PSBA.,matK,trnK,rps16,trnQ, 和PSBK..扩张也发生在SSC的两侧区域。在SSC的一端,NDHF.迷失在B. Americana.虽然相邻的6 kB区域现在是重复区域的一部分.ndhE,PSAC.,n,ccsA,trnL, 和rpl32.都在这个区域内。在SSC的另一端,一个大约5 kb的区域包含rpl32.和ycf1已经转入重复中了吗B. Americana..
他们三个斯特物种的IR扩张模式基本一致,大部分扩张发生在LSC区域。两个lsc侧翼区域以及一个内部区域现在都是这三个区域重复的一部分斯特物种。一个大约12.5 kb的区域在IR的侧面l . philippensis是IR的一部分吗Striga。这个区域包含trnH、psbA、matK、trnK、rps16、trnQ、psbK、psbI、trnS、trnG、trnR、和atpA.在LSC的另一端,IR区域扩展到约18.8kb的相邻序列。该区域包含15个基因:CLPP,PSBB,PSBT,PSBN,PSBH,RPOA,RPS11,RPL36,INFA,RPS8,RPL14,RPL16,RPS3,RPL22, 和rps19.内部区域是5.5 kb,包含ycf4 cemA petA psbJ psbL psbF, 和Psbe..在L. Philippensis,在这个区域和一侧的IR之间有61 kb,在另一侧的IR之间有18 kb。在这三个区域中,与重复区域相邻的SSC区域大约有4.5 kb,现在是重复区域的一部分斯特物种。这个区域包含ycf1基因。重复扩展的所有细节见表1.
整体基因的内容
在这5个物种中,没有任何tRNA或rRNA基因丢失。所有的质体编码30种不同的tRNAs和4种不同的rnas。与外群体相比Lindenbergia,Aureolaria没有丢失任何蛋白质编码基因,总共包含79个单拷贝基因和6个包含在重复区域内的基因,因此存在于两个拷贝中。Buchnera总共有77个单拷贝基因,加上17个在重复区域,其中9个基因可能是假基因。S. forbessii.含有79个单拷贝基因,加上重复内的35种,包括14个潜在的假生素。S. hermonthica有76个单拷贝蛋白编码基因,35个重复序列和10个潜在的伪基因。美国粗含有77个单拷贝基因,在重复区中加35,具有12个潜在的伪原(表2)。
基因损失
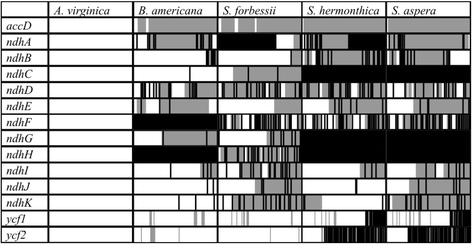
在l . philippensis和答:virginica,ndha.是2.2 kB基因,含有两个外显子,539bp和553bp,由1.1 kb内含子分开。S. hermonthica缺失的第二个外显子ndha., 尽管S. forbessii.就是缺失了第一个外显子B. Americana.和美国粗有两个外显子,但在第一个外显子中有预测的移码突变和过早终止密码子。所有ndh基因的结构与功能基因在答:virginica.B. Americana.和三个斯特物种都有一个或多个停止密码子或移码ndhb,从氨基酸446英寸开始B. Americana.和448年S. forbessii..S. hermonthica和美国粗它们在氨基酸24处都有一个移码,这表明它们的共同祖先中有一个共同的事件,随后有多个移码和终止密码子。NDHC.存在于B. Americana.但含有氨基酸82的终止密码子。S. forbessii.包含NDHC.从氨基酸23开始移码,然后有多个终止密码子。NDHC.两者都缺少S. hermonthica和美国粗.所有四种物种都包含多个架构和终止密码子n从36in的氨基酸开始B. Americana., 25S. forbessii.4在S. hermonthica和40美国粗.S. forbessii.包含一个终止密码子ndhE氨基酸101。B. Americana.在氨基酸11处具有止芯密码子。S. hermonthica在氨基酸40处有一个终止密码子。B. americana, S. hermonthica,和美国粗都含有5 '移码突变,在氨基酸34B. Americana.和氨基酸32斯特物种,但都是独立事件。这三个物种在移码开始后都有额外的停止密码子。NDHF.中没有B. Americana.并含有氨基酸20的框架S. forbessii.后面有多个终止密码子。S. hermonthica和美国粗它们都在第三个氨基酸处包含一个终止密码子,然后是附加的终止密码子和移码。ndhg.包含一个移码和多个终止密码子从氨基酸63开始B. Americana.和氨基酸3S. forbessii..ndhg.中没有S. hermonthica和美国粗.ndhh.中没有B. Americana.,S. hermonthica, 和美国粗.ndhh.存在于S. forbessii.但含有从氨基酸11开始的架构和多个止挡密码子。NDHI.包含一个终止密码子B. Americana.在氨基酸90和在S. forbessii.在氨基酸17,然后是框架和额外的止挡密码子。S. hermonthica含有从氨基酸47开始的移码美国粗包含从氨基酸42开始的移码,都有多个终止密码子。ndhj.包含从140英寸的氨基酸开始的终止密码子B. Americana..在s . forbessii ndhJ在氨基酸71上包含一个移码,后面有多个终止密码子。ndhj.在S. hermonthica含有起始于氨基酸102的终止密码子。ndhj.包含帧移美国粗从氨基酸44开始,然后是终止密码子。NDHK.含有氨基酸120的终止密码子B. Americana.和氨基酸14S. forbessii..两个物种都包含额外的终止密码子和S. forbessii.包含一个框架。NDHK.同时S. hermonthica和美国粗在氨基酸4上包含一个移码,然后是终止密码子。相关的移码和终止密码子如图所示。1.表格3.显示两个或两个以上物种在相同氨基酸上共有的突变,导致移码或终止密码子。
阻止潜在伪基因中的密码子和框架移位。在五个物种中至少有一个缺失或预测为无功能的基因。纯白色条表示该基因存在并在该物种中具有功能,而带有灰色阴影或黑色线条的条则可能是假基因。该基因的蛋白质编码部分的结构显示在每个盒子内,5 '端在最左边,3 '端在最右边。每个盒子的大小是蛋白质编码区域的100%,所以它们不会根据每个基因编码区域的不同大小进行校正。灰色阴影表示部分基因有一个移动的框架,而黑色线表示停止密码子。纯黑色的条状物是基因或基因片段在那个物种中完全缺失。例如,图中显示5 '端ndha.中缺少S. forbessii.3 '末端也不见了S. Hermonthica,但是在5 '末端有多个终止密码子和帧移S. hermonthica应该保证它是一个无功能基因吗
accD存在于所有五种物种中。在答:virginica,它看起来与差异相比很少l . philippensis.在其他四个物种中,有许多预测的突变,包括多次移帧。然而,这个基因的3 '端比5 '端保守得多.这是可能的accD具有由基因的3'末端编码的保守蛋白质域。ycf1和ycf2似乎在所有物种中都有多次移码突变,除了ycf2在答:virginica.
质体比对和系统发育分析
6个质体均与Mauve 2.3.1对齐,如图所示。2.答:virginica基本上与l . philippensis,除了SSC处于反向方向之外。所有其他塑料含有多种重排。在这内斯特属,S. hermonthica和美国粗它们之间没有重新排列,而S. forbessii.在重复区域有一个单一的重排。在更高的分辨率下,如图。2 b显示了just的淡紫色对齐示例S. hermonthica和A. Virginica。
基因丢失事件发生的时间
在许多情况下,一个预测会使基因失活的序列变化的特定核苷酸位置被多个物种共享。因此,这些事件最有可能发生在同一物种的共同祖先身上。由于这些缺失、移码和/或终止密码子的创造被预测会使基因失活,因此,将每个基因的谱系和失活的相对时间绘制到比较物种的系统发育树上是合适的。系统发育树l . philippensis作为外群体,如图所示。3.,预测共享基因灭活作为特定分支的基因名称。
讨论
质体的保存和改变
质体在几乎所有的维管植物中发挥着共同和重要的功能,因此是系统发育分析的非常有力的工具。非光合作用的寄生植物失去了对参与光合作用的许多质体基因的强选择能力,但有望保留参与其他重要叶绿体作用的基因的编码能力。在以前对非光合作用植物的研究中[13,14,18,19,20.,21,大多数或所有光合基因被认为是假基因化或丢失。这个过程似乎是由随机突变引起的,因此被认为是一个渐进的过程,只会在寄生/非光合作用生活方式建立后的某个遥远的时间完成。我们的观察,类似于Wicke等人的观察[20.]的研究表明,即使在完全进行光合作用的寄生虫中,某些叶绿体基因也会丢失,这表明除了产生光合产物以外,叶绿体的作用现在可能由宿主提供。因此,从质体中大量的基因丢失并不仅仅与全寄生的生活方式有关。如果半寄生是向全寄生进化的常规步骤,那么可能是质体基因丢失过程既是全寄生的原因,也是结果。
在被子植物中,寄生至少进化了12到13次[42].Orobanchaceae的寄生被认为已经进化过一次,估计发生在32-64 mya [43,44,45].有趣的是,这些不同的质体最终的基因内容有所不同,尽管失去了明显的功能,但许多序列仍然存在。这表明基因丢失过程非常缓慢,特别是与核DNA丢失相比[46,并提出了一种可能性,即这些不同物种的不同适应性导致了不同的质体基因丢失结果。例如,大多数ndh基因在形成该属的早期谱系中已经丢失斯特,但ndhE,ndhh.和ndhj.只在末端分支中丢失,这表明这些基因可能具有过渡性作用,即使当另一个ndh基因不再具有功能性。相似地,Pedicularis cheilanthifolia显然只有一个质体基因因假基因化而丢失,NDHF.,已经在许多谱系[已失去了独立的基因28].所有这些结果都证实了在半寄生虫中ndh途径是不必要的或甚至可能选择的。进一步分析塑料基因组变化的性质应提供额外的见解,遗传(以及因此生理)功能丢失,在哪些谱系中以及进化率和时间。
质体基因组大小
大多数植物的塑料尺寸为120至160 kB。l . philippensis在这个范围内。的答:virginica塑料略小于l . philippensis,但仍然落在典型的范围内。LSC,SSC和IR的答:virginica都是在尺寸上相似的l . philippensis在被子植物的一般大小范围内。B. Americana.和三个斯特然而,所有物种在整个质体长度和LSCs、SSCs和IRs长度上都表现出有趣的大小差异。它们的质体都比l . philippensis以及在植物中常见的范围以上.这是出乎意料的,因为迄今为止研究的大多数寄生物种的质体都较小[14,16,17,19,20.,21,22,23].尺寸的增加可以解释为反向重复的长度的增加。不过,这五个物种的倒重复大小都有所增加Aureolaria靠近Lindenbergia(26,031 bp, 25,812 bp),属于被子植物反向重复大小的典型范围。其他四种有相当大的重复,超过20到30 kb大小,通常在被子植物中看到,从43,864 bpBuchnera至63,240 bpS. forbessii..倒置重复大小的几百BP的少量变化在土地厂中是常见的[47,48,49],但这里看到的更大变化很少[47,48,49,50,51,52].最大的已知重复属于天竺葵属植物hotorum,75,741 bp [53].寄生质体中可见轻微的IR膨胀Schwalbea美国和Cistanche phelypaea.然而,这些扩张的大小小得多,包括2个基因和3个基因[19,与我们在图中看到的展开进行了比较斯特和B. Americana..
我们观察到的重复大小的增加是由于扩展到单个拷贝区域。所有五种寄生性物种的SSC和LSC区域均小于l . philippensis.的斯特扩展与中扩展有很大的不同美国。在B. Americana.在美国,大多数重复扩张都进入了南中国海地区。在这三个斯特物种,在SSC区域有一定的重复扩张,但大部分扩张是在LSC区域。他们之间的扩展模式有两个相似之处斯特物种和Buchnera。第一个是靠近IR的LSC区域的8kb(从TRN.H到PSB.这个区域包含在所有四个物种的重复序列中,尽管斯特IRS进入该地区进一步的4.5千克地区。在物种分歧之前发生初始8 kB膨胀,然后在额外的4.5 kB扩展之后斯特血统。第二个相似之处是扩展到SSC,其中包含ycf1基因。的B. Americana.IR包含额外的0.5 kb包含rpl这表明这是世系分化后的一次额外扩张。除了这两个相似之处,其余重复扩展都是独一无二的B. Americana.或者斯特并且很可能发生在两种世系分化之后。与其他被研究的植物相比,这些独立的重复扩展要么是对这种现象的选择,要么是这些谱系中的一种机制异常,使得这种结果非常可能发生。在功能上,这些复制区域的增加似乎没有创造任何新的基因。因为这些重组重复的基因转换过程[3.],预计任何新的基因功能都会通过这种IR扩展演变。
美国粗和S. hermonthica没有显示它们之间的重排或重复展开之间的差异。两者的质体在结构上有一个主要的区别S. forbessii..在重复区域内,2.5 kb的区域包含CEMA和佩塔以反方向位于该区域的不同部分。一种可能是重复膨胀独立地发生在S. forbessii.血统与共享S. hermonthica / aspera两个谱系分歧后的谱系。另一种可能性是,在这三种物种的共同祖先发散之前发生重复膨胀,然后在发散后进行谱系特异性重排。
缺失的基因
NAD(P) h -脱氢酶复合物参与了围绕光系统I回收电子的多种途径之一[54,55,60].NAD(P)H复杂功能在压力条件下,对于高度可变光强度条件下的光合作用可能是必不可少的[56,57].质体编码了11个亚单位:ndhA B C D E F G H I J和K[58].这些基因在陆生植物中已经丢失过几次,在寄生植物中也经常丢失[13,16,17,19,20.,23,59,60,61,62,63].
Ycf1是第一个鉴定出的质体编码蛋白,它的存在对绿色植物的生存至关重要。Ycf蛋白质是在绿色植物中负责叶绿体蛋白运输的转位子的重要组成部分[64].但是,没有ycf在Poales中检测到1个同源物,除了蒲菜[65].在我们研究的五个物种中,ycf1被观察到高度发散,具有预测的移码突变。的基因ycf2仍有未知的功能[66]并具有功能结构答:virginica,但包含多个帧移突变B. Americana.和三个斯特物种。这两个ycf1,ycf2在包括非寄生虫在内的大多数陆生植物中具有很高的替代率,并可能在某些谱系中成为假基因[67,68].两个基因的5 '端往往相对保守,而基因的其余部分则更分散。我们观察到一个相似的模式,两个基因最保守的区域都在5 '端。双方完全损失ycf1,ycf2发生在一些单子叶植物中[69].
ACCD.在脂肪酸合成过程中,乙酰辅酶a羧化酶介导乙酰辅酶a向丙二酰辅酶a的转化。的accD基因在Angiosperms中已经丢失了几次[70],其功能被核复制物取代[71].3 '区域acc我们研究的所有5个物种的D基因都比5 '端保守得多。更高的散度或截断的5 '端accD基因也已在几种物种中观察到[19,20.].我们没有任何在这个手稿调查物种的核基因组序列,但我们预测,核accD同源物现在在B. Americana.和斯特谱系。我们还预测,3'结束accD基因可能具有单独的、质体特异性的功能。
质体基因组比对
答:virginica与?相比没有重大基因重排l . philippensis除外倒置SSC区域。的SSC区域经常翻转在plastomes,具有相反取向SSC通常存在于50:50的比例在植物细胞中[1].对比答:virginica和B. Americana.都很有趣,因为它们都是兼性半寄生虫。由于它们都能在没有寄主的情况下生存,因此严重依赖光合作用,因此可以预计,与自养植物质体相比,它们的重组数量相对较少。这就是A. Virginica。然而,B. Americana.与多重重排相比呢答:virginica和l . philippensis.他们三个斯特物种也有几次重排答:virginica和l . philippensis,以及与之相比B. Americana..之间没有重排S. hermonthica和美国粗这两个和之间以及单个重排S. forbessii..这些模式都属于一个适当的系统发育顺序,可以预测它们的时间和起源谱系。
该系统发育树与现有的系统发育分析相同意,就像关系l . philippensis,答:virginica,B. Americana.,斯特属(72,以及三者之间的关系斯特物种之间的相互关系S. hermonthica和美国粗比彼此更密切相关S. forbessii.[33].
质体不稳定性与寄生植物功能
虽然在本研究中调查的所有五种植物都是专性光合生物,但发现四种植物的质体不稳定性广泛存在。这些结果与以前对其他寄生植物的观察结果一致[13,14,15,16,17,18,19,20.,21],而且在非寄生虫中也较少见[29,45,53,65,73,74,75,76,77,78,79].因此,塑性不稳定性是基因组变异的来源,其提供用于自然选择的原料。在任何绿色植物中,这种选择都预计会导致基因含量的总体守恒,任何叶绿体功能都可以直接提供的任何叶绿体功能。对于寄生植物,宿主植物是主要的环境贡献者,可能用于光合酸酯,并且许多由局部化到叶绿体的途径产生的许多小分子。的ndh通路在许多世系中丢失,这表明它在光合作用中的作用通常是可有可无的。鉴于这一通路在如此多的世系中迅速和几乎完全丧失,这表明ndh可以实际上选择在一些植物谱系中。未来的研究将这种途径归还它自然失去的物种将提供调查该谱系和条件的绝佳机会ndh基因序列可以使植物受益,也可以使植物衰弱。
在这种相同的静脉中,可以研究本研究中观察到的所有基因变化,以便在植物健身中作用。这些基因在这些分类群中不同的基因的表达水平,例如由于其纳入或缺乏美国国税局,可以提出在许多植物谱系中产生和保留这种重排的作用。塑料结构的自然变化应该在更多的奥古氏菌物种上进行测试,无论是如何获得更好的谱系和这种重排的速率的思考,还要提供用于比较这些重排的功能结果的原料。随着现在在许多植物物种中可用的转变叶绿体的能力[80,81,82,83,自然质体变异及其在机体功能中的作用的研究可以直接与工程转基因中测试这些基因效应的实验进行比较。
结论
本文对五种半寄生植物的叶绿体基因组进行了测序和组装。我们比较了组装的可用的质体组装Lindenbergia philippensis,一种Orobanchaceae的自养生物。Aureolaria virginica几乎和l . philippensis在质体结构、基因含量和基因顺序方面。B. americana, S. forbessii, S. hermonthica, 和美国粗这些都显示了大量的重排和生理和功能基因的丢失。最有趣的结果是这四种寄生虫的质体尺寸增大,因为大多数寄生虫的质体尺寸减小。这种增长是由于重复区域扩展到单个复制区域,尽管这对质体有什么影响尚不清楚。B.美国,S.她的月, 和美国粗都有缺失ndh基因,而这三个物种S. forbessii.有多个ndh基因是潜在的非功能由于预测移码突变和终止密码子。没有其他的基因从任何物种的缺失,但的accD,ycf1,和ycf2都有很高的偏离度,在B. americana, S. forbessii, S. hermonthica,和美国粗以及停止密码子ycf1和ycf2在S. hermonthica和美国粗.
参考文献
- 1.
帕默JD。叶绿体基因组的比较组织。《Genet Rev》1985;19:325-54。
- 2.
王志强,王志强,王志强,等。陆地植物质体染色体的进化:基因含量、基因顺序、基因功能。植物学报。2011;76:273-97。
- 3.
帕默JD。分子进化:所有质体的单一诞生?大自然。2000;405:32-3。
- 4.
McFadden GI, van Dooren GG.进化论:红藻基因组确认所有质体的共同起源。咕咕叫医学杂志。2004;R514:516。
- 5.
等。中国生物科学(英文版);2010;
- 6.
Martin W, Rujan T, rich E, Hansen A, Cornelsen S, Lins T, Leister D, Stoebe B, Hasegawa M, Penny D.进化分析拟南芥、蓝藻和叶绿体基因组揭示了质体系统发育和细胞核中数以千计的蓝藻基因。美国国家科学研究院2002;99:12246-51。
- 7.
拉布斯顿洛杉矶,詹森卢克。植物的叶绿体基因组。在:编辑亨利·雷吉。植物多样性与进化:高等植物的基因型和表型变异。沃林福德,英国:Cabi出版;2005. p。45-68。
- 8.
Kolodner R, Tewari KK。高等植物叶绿体DNA的反向重复。美国国立科学院。1979; 76:41-5。
- 9.
MaréchalA,父母J,Véronneau-Lafortune F,Joyeux A,Lang BF,Brisson N.Wirly蛋白维持体液基因组稳定性拟南芥.国家科学院院刊美A. 2009; 106:14693-8。
- 10.
帕默JD。质体染色体:结构与进化。在:Bogorad L, Vasil IK,编辑。植物的细胞培养和体细胞遗传学,第7A卷。质体的分子生物学。圣地亚哥:学术出版社;1991.5-53页。
- 11.
叶绿体基因组。植物学报1992;19:149-68。
- 12.
质体基因组的结构、功能和遗传。在:Bock R,编辑。质体的细胞与分子生物学。柏林海德堡,63:施普林格。29页。
- 13.
dePamphilis CW,帕尔默JD。从寄生开花植物的质体基因组光合chlororespiratory基因的损失。自然。1990; 348:337-9。
- 14.
Wolfe Kh,Morden CW,Palmer JD。非分水性寄生植物最小血浆基因组的功能和演变。Proc Natl Acad Sci U S A. 1992; 89:10648-52。
- 15.
Nickrent DL, Ouyang R, Joel D, dePamphilis CW。非小行星全寄生开花植物有质体基因组吗?植物学报1997;34:717-29。
- 16.
2种寄生性开花植物质体基因组的完整DNA序列五角reflexa和Cuscuta Gronovii..BMC Plant Biol. 2007;7:45。
- 17.
叶绿体基因组的完整序列表明,寄生植物属的光合基因具有很强的保留选择性五角.BMC植物BIOL。2007; 7:57。
- 18.
krausek.从叶绿体到“神秘”血浆:寄生植物中的塑性基因组的演变。Curr Genet。2008; 54:111-21。
- 19.
Wicke S,MüllerKF,Depamphilis CW,Quandt D,Wideet NJ,Zhang Y,Renner SS,Schneeweiss Gm。武装系列光合作用和非水性寄生植物的功能性和物理基因组的机制。植物细胞。2013; 25:3711-25。
- 20。
王志强,王志强,王志强。植物非光合作用过程中进化速率变化的机制模型。中国科学(d辑:地球科学)2016;
- 21。
samigullin th,logacheva md,penin aa,vallejo cm。近期Holoparasites的完全塑性基因组Lathraea squamaria揭示了Orobanchaceae中最早期的质体减少阶段。《公共科学图书馆•综合》。2016;https://doi.org/10.1371/journal.pone.0150718.
- 22.
Delavault PM,Russo NM,Lusson Na,Thalouarn P.组织减少的塑性基因组Lathraea clandestina一个achlorophyllous寄生植物。physiol植物。1996; 96:674-82。
- 23.
作者简介:卫克特(Wickett NJ, Zhang Y, Hansen SK), Roper JM, Kuehl JV, Plock SA, Wolf PG, dePamphilis CW, Boore JL, Goffinet B.Aneura mirabilis..mol Biol Evol。2008; 25:393-401。
- 24.
Molina J,Hazzouri Km,Nickrent D,Geisler M,Meyer Rs,Pentony MM,Flowers JM,Pelser P,Barcelona J,Inovejas SA等。寄生开花植物中叶绿体基因组的可能丧失rafflesia lagascae.(rafflesiaceae)。mol Biol Evol。2014; 31:793-803。
- 25.
Naumann J, Der JP, Wafula EK, Jones SS, Wagner ST, Honaas LA, Ralph PE, Bolin JF, Maass E, Neinhuis C,等。非光合寄生植物高度分化质体基因组的检测和特征Hydnora visseri(菌花科)。基因组生物学进展。2016;8:345-63。
- 26.
内寄生属两种植物的质体皮原士(Apodanthaceae)每次保留只有五个或六种可能的功能基因。基因组Biol Evol。2015; 8:189-201。
- 27.
王志强,王志强,王志强,等。全寄生性植物的叶绿体基因组结构及其生物学意义。植物学报,2003,23 (4):494 - 498簇花草属hypocistis(三尿膜)。Ann Bot。2016; 118:885-96。
- 28。
张锐,王杰,韩科,任涛,曾胜,Biffin E,刘志强Pedicularis cheilanthifolia,中国的高山植物。保守遗传资源。2017; DOI:https://doi.org/10.1007/S12686-017-0740-2.
- 29。
老唐尼,帕尔默。叶绿体DNA反向重复的限制性位点定位:星科的分子系统发育。《安莫Bot Gard》1992;79:266-83。
- 30.
伍尔夫KH。豆科植物叶绿体DNA中的核苷酸替换率依赖于反向重复的存在。中华医学杂志。2002;55:501-8。
- 31。
olmstead rg,depamphilis cw,wolfe广告,年轻的nd,elisons wj,Reeves pa。骨科的崩解。我是J机器人。2001; 88:348-61。
- 32.
王志强,王志强,王志强,等。羊齿科全寄生植物的系统发育和起源。Am J Bot. 2013; 100:971-83。
- 33.
step MC, Gowda BS, Huang K, Timko MP, Bennetzen JL。利用样本序列分析对属寄生杂草的基因组特征进行分析。植物基因组。2012;5:30-41。
- 34.
道尔JJ,道尔JL。小数量新鲜叶组织的快速DNA分离程序。Phytochem公牛。1987;19:11-5。
- 35.
Vaughn JN, Chaluvadi SR, Tushar RL, Bennetzen JL。5种生姜的整个质体序列有助于标记的发展和确定条形码方法的界限。《公共科学图书馆•综合》。2014;https://doi.org/10.1371/journal.pone.0108581.
- 36.
Darling ACE, Mau B, Blattner FR, Perna NT.淡紫色:保守基因组序列的多重比对与重排。麝猫杂志2004;14:394 - 1403。
- 37.
Kearse M, Moir R, Wilson A, Stones-Havas S,张M, Sturrock S, Buxton S, Cooper A, Markowitz S, Duran C, Thierer T, Ashton B, Mentjies P, Drummond A. Geneious basic:用于序列数据组织和分析的集成的、可扩展的桌面软件平台。生物信息学。2012;28:1647-9。
- 38.
崔莉,李志强,李志强,李志强,李志强。叶绿体基因组数据库。核酸学报2006;34:D692-6。
- 39.
Larkin MA, Blackshields G, Brown NP, Chenna R, mcgetigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R, Thompson JD, Gibson TJ, Higgins DG。clusteral W和clusteral X 2.0版本。生物信息学。2007;23:2947-8。
- 40.
分子进化遗传学分析7.0版。Mol Biol Evol. 2016; 33:1870-4。
- 41.
Ronquist F, Huelsenback JP。MRBAYES 3:混合模型下的贝叶斯系统发育推断。生物信息学。2003;19:1572-4。
- 42.
Westwood Jh,Yoder Ji,Timko MP,Depamphilis CW。植物中寄生派的演变。趋势植物SCI。2010; 15:227-35。
- 43。
Bremer K,Fijs Em,Bremer B.小行星开花植物的分子系统发育约会显示出早期的白垩纪多样化。系统中的生物学。2004; 53:496-505。
- 44。
刘林,柯士泰。系统发育和生物地理学列当科.叶形线Geobot。2005;40:115-34。
- 45。
Naumann J,Salomo K,Der JP,Wafula ek,Bolin JF,Maass E,Frenzke L,Samain Ms,Neinhuis C,Depamphilis CW,Wanke S.单拷贝核基因在派对中占据普通氢牛皮,并揭示了多个白垩纪寄生的Anviosperm谱系。Plos一个。2013;https://doi.org/10.1371/journal.pone.0079204.
- 46.
KH,李万辉,夏普总理。在植物线粒体、叶绿体和核dna中,核苷酸替代率差异很大。美国国家科学研究院1987;84:9054-8。
- 47.
Goulding SE, Wolfe KH, Olmstead RG, modern CW。叶绿体的涨落倒转重复。摩尔Gen Genet 1996; 252:195-206。
- 48.
王荣军,程春林,常聪,吴春林,苏明明,张淑敏。单子叶植物叶绿体基因组中反向重复-大单拷贝连接的动态和进化。BMC Evol Biol. 2008;8:36。
- 49.
唐尼SR,詹森RK。蜂目全质体基因组的比较分析:反向重复序列的扩张和收缩,DNA的线粒体到质体转移,以及高度分化的非编码区域的鉴定。系统机器人。2015;40:336-51。
- 50.
Knox EB, Palmer JD。的叶绿体基因组排列半边莲thuliniana(Lobeliaceae):扩张的倒置重复在一个祖先园林.acta botanica sinica(云南植物研究),1999;
- 51。
Dugas DV, Hernandez D, Koenen EJM, Schwarz E, Straub S, Hughes CE, Jansen RK, Nageswara-Rao M, Staats M, Trujillo JT, Hajrah NH, Alharbi NS, AL - malki AL, Sabir JSM, Bailey CD. Mimosoid legome plasome evolution: IR expansion, tandem repeat expansion, and accelerated rate inCLPP..SCI REP。2015; 5:16958。
- 52。
朱艾,郭伟,古普塔,风扇W,割草机jp。塑体倒置的进化动力学重复:膨胀,收缩和替代率损失的影响。新植物。2016; 209:1747-56。
- 53。
Chumley TW, Palmer JD, Mower JP, Fourcade HM, Calie PJ, Boore JL, Jansen RK。完整的叶绿体基因组序列天竺葵xhortorum:陆生植物叶绿体基因组中最大、重排程度最高的组织和进化。生物化学学报2006;23:2175-90。
- 54。
氯呼吸与循环电子传递的平衡。中国生物化学杂志2000;275:942-8。
- 55。
尼克松PJ。Chlororespiration。中国生物科学(英文版);2000;541 - 541。
- 56.
MartínM,Funk HT,Serrot pH,Poltnigg P,Sabater B.通过地点针对突变的囊体NDH复合磷酸化的功能表征NDHF.基因。BBA-Bioenergetics。2009年; 1787年:920-8。
- 57.
Martín M, Sabater B. Plastidndh植物演化中的基因。植物理性生物化学。2010; 48:636-45。
- 58.
弗里德里希T,SteinmüllerK,魏斯H.菌和线粒体和叶绿体同源的质子泵呼吸系统复杂的我。FEBS快报。1995; 367:107-11。
- 59.
McCoy SR, Kuehl JV, Boore JL, Raubeson LA。质体的完整基因组序列千岁兰君子兰:一种异常紧密的质体,分化速度加快。BMC Evol Biol. 2008;8:130。
- 60.
吴超,赖勇,林晨,王勇,周胜,等。分子系统学进展。2009;52:115-24
- 61.
张传聪,林慧聪,林叶,周泰,陈洪辉,陈文华,郑春华,林超英,刘思敏,张传聪,周思敏。的叶绿体基因组蝴蝶兰属阿佛洛狄忒兰科植物与禾本科植物进化速度的比较分析及其系统发育意义。生物学报2006;23:279-91。
- 62.
吴芳,陈敏,廖丹丹,徐超,李勇,林志刚金蝶兰属植物Oncidiinae鉴定与育种的分子标记评价。BMC Plant Biol. 2010;10:68。
- 63.
植物基因组降解的研究进展[J] .浙江农业学报,2017,29(4):559 - 564。生物科学进展。2014;31:3095-112。
- 64.
De Vries J,Sousa FL,Bölterb,soll j,gould sb。YCF1:绿色TIC?植物细胞。2015; 27:1827-33。
- 65.
Guisinger MM, Chumley TW, Kuehl JV, Boore JL, Jansen RK。typha (typhaceae, poales)质体基因组序列对了解类人猿基因组进化的意义。中国科学(d辑:地球科学)2010;
- 66.
Logacheva MD, Schelkunov MI, Shtratnikova VY, Matveeva MV, Penin AA。非光合作用杜鹃花科及其光合亲缘植物质体基因组的比较分析。科学报告。2016;https://doi.org/10.1038/srep30042.
- 67。
Oliver M, Murdock A, Mishler BD, Kuehl J, Boore J, Mandoli D, Everett K, Wolf PG, Duffy A, Karol KG。苔藓叶绿体基因组序列Tortula Ruralis.:基因含量、多态性和相对于其他绿色植物叶绿体基因组的结构安排。BMC基因组学。2010;11:143。
- 68。
狼PG,DERĴ,达菲A,戴维森Ĵ,Grusz A,窥探者KM。叶绿体基因和基因组中的蕨类植物的进化。植物mol biol。2010;https://doi.org/10.1007/s11103-010-9706-4.
- 69。
唐妮斯SR,Katz-Downie DS,Wolfe Kh,Calie PJ,Palmer JD。最大叶绿体基因的结构和演变(ORF2280):内在塑性和高血管培养过程中的多基因损失。Curr Genet。1994年; 25:367-3781。
- 70.
Jansen RK, Cai Z, Raubeson LA, Daniell H, de Pamphilis CW, Leebens-Mack JH, Müller KF, Guisinger-Bellian M, Haberle RC, Hansen AK, Chumley TW, Lee SB, Peery R, JR MN, Kuehl JV, Boore JL。通过对64个质体基因组中的81个基因的分析,揭示了被子植物的亲缘关系,并确定了基因组规模的进化模式。美国国家科学研究院2007;104:19369-74。
- 71.
Nakkaew A, Chotigeat W, Eksomtramage T, Phongdara A.质体编码亚基β羧转移酶基因的克隆和表达(accD)和来自油棕的乙酰-CoA羧化酶的核编码亚基亚单位羧化酶(Elaeis guineensisjacq。)。植物SCI。2008; 175:497-504。
- 72.
王志刚,王志刚。植物的生长发育及其对植物生长的影响。植物学报。2006;
- 73.
帕默JD。叶绿体DNA进化和叶绿体DNA变异的生物系统学应用。Nat。1987;130:S6-S29。
- 74.
Tsudzuki J, Nakashima K, Tsudzuki T, Hiratsuka J, Shibata M, Wakasugi T, Sugiura M.黑松叶绿体DNA保留了一个残差的反向重复缺失rRNA基因:核苷酸序列TRNQ,TRNK,PSBA,TRNI和trnH以及缺少rps16.孟根。1992;232:206-14
- 75。
碧藓科Apioideae亚科叶绿体反向重复的扩张和收缩。系统机器人。2000:648 - 67。
- 76。
Daniell H, Lee S, Grevich J, Saski C, Quesada-Vargas T, Guda C, Tomkins J, Jansen RK。完整的叶绿体基因组序列Solanum bulbocastanum,solanum lycopersicum与其他茄科基因组进行比较分析。Al Appl Genet。2006; 112:1503-18。
- 77。
Wolfe Pg,roper JM,Duffy Am。蕨类植物叶绿体基因组结构的演变。基因组。2010; 53:731-8。
- 78。
Grewe F,Viehoever P,Weisshaar B,Knoop V.在肝脏的线粒体基因组中,Intrace群I内含子和TRNA Hyperiting水韭engelmannii.核酸RES。2009; 37:5093-104。
- 79。
郭伟,葛伟,柯博-克拉克A,范伟,段铮,Adams RP, Schwarzbach AE, Mower JP。质体基因组中的主要和亚化学计量异构体共存Juniperus植物并在愚蠢的演变期间已经多次转移。基因组Biol Evol。2014; 6:580-90。
- 80.
植物质体的稳定转化。美国国家科学学院1990;87:8526-30。
- 81.
Sikdar SR,Serino G,Chaudhuri S,Maliga P. Plastid转型拟南芥蒂利亚纳.Plant Cell rep 1998; 18:20-4。
- 82.
Sidorov Va,Kasten D,Pang Sz,Hajdukiewicz PTJ,Staub JM,Nehra NS。马铃薯的稳定叶绿体转化:使用绿色荧光蛋白作为塑性标记物。工厂J. 1999; 19:209-16。
- 83.
Wani Sh,Haider N,Kumar H,Singh NB。植物塑料工程。Curr基因组学。2010; 11:500-12。
确认
我们感谢J. Palmer教授(印第安纳州大学)和S. Wicke教授(Muenster大学),为他们的乐于助人的评论。
资金
这项研究得到了USDA / Nifa Grant奖的支持#2012-67013-19473和与格鲁吉亚大学遗传学系的吉尔斯教授相关的捐赠。
数据和材料的可用性
当前研究中生成和/或分析的数据集可在GenBank存储库中找到,https://www.ncbi.nlm.nih.gov/genbank/.登录号为MF780870、MF780871、MF780872、MF780873和MF780874。
作者信息
隶属关系
贡献
DCF在塑植组装,注释,分析和手稿准备和修订上。SRC进行了实验,辅助塑料组装,注释和稿件修订。JNV辅助塑料组装。CGC辅助塑料测序。JLB构思了该项目,并帮助实验,数据分析,稿件准备和修订。所有作者均致力于稿件的修订。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
弗莱利,华盛顿特区,查卢瓦迪,s.r.,沃恩,J.N.et al。羚羊族五次血管基岩体积的基因丧失和基因组重排。BMC植物杂志18,30(2018)。https://doi.org/10.1186/s12870-018-1249-x.
收到了:
接受:
发表:
关键字
- 叶绿体
- 染色体重排
- 基因缺失
- 寄生虫
- 质体系
- 斯特