抽象的
背景
DiazotrophicBradyrhizobiumSPP。众所周知,它们能够触发各种豆科物种的结节形成。在结节中,Bradyrhizobium利用植物源碳水化合物交换固定的氮。结瘤和固氮特性所必需的基因聚集在一个基因组区域,这被称为“共生岛”。最近,小说non-diazotrophicBradyrhizobiumSPP。已被发现在土壤中非常丰富,这表明这些物种也可以拥有“自由生活”的生命历史。但是,无论是非真正营养的Bradyrhizobiumsp .能否与植物共生尚不清楚。
结果
在这项研究中,我们展示了这一点Bradyrhizobium属非豆科植物常见的根内生菌,包括拟南芥蒂利亚纳(拟南芥) - 生长在生态环境中。从一个拟南芥根,四个Bradyrhizobium分离菌株MOS001 ~ MOS004。比较基因组分析表明,这些菌株在遗传和功能上具有高度多样性,但不存在结瘤和固氮基因簇。用MOS菌株和固氮共生菌株进行比较定殖实验,结果均表明BradyrhizobiumSPP。可以殖民征集拟南芥的根内生物隔室。
结论
本研究提供了虚拟营养和非真正营养的证据Bradyrhizobium在许多植物种的根内生菌室定殖,包括模式种拟南芥。这说明植物根是植物的主要生态位BradyrhizobiumSPP。,这可能是该属的染色和氮固定性状的演变的祖先。
背景
植物可以与土壤细菌发展关系,从根际的松散联系到植物组织中细胞间和细胞内的亲密寄主。豆科植物和一组固氮的α-和β-变形菌(统称根瘤菌)之间的关系是一个著名的和被充分研究的亲密相互作用的例子[1].豆类可以在其根系上形成特殊的器官 - 称为结节 - 促进根茎转化为铵的根茎转化为铵,随后可以通过工厂使用2].这种固氮共生的细菌基因是结瘤(点头和nol)和氮固定(nif和使固定)基因。这些基因在基因组或共生质粒上形成一个或多个簇。这点头/nol和nif/使固定在根瘤菌属的不同范围内鉴定出的基因具有高度同源性,因此这些共生基因是通过水平基因转移传播的观点已被广泛接受[3.].这表明祖先根瘤菌的生活方式与根瘤共生无关。
共生性根瘤菌最深刻的种植物之一是属Bradyrhizobium(属于慢生根瘤菌科)。定义为“豆科植物生长缓慢、不产酸的根瘤细菌”[4.],该属现包括37种[5.].这些物种的类型菌株都有结瘤,并有广泛的豆科植物(包括大豆;大豆)和非豆科植物Parasponiasp . as host [6.那7.].的基因组Bradyrhizobium分枝相对较大(7-10 Mb),结节(点头/ nol)和氮固定(nif /修复)基因簇位于基因组内的一个共生岛上[8.].
最近的研究表明Bradyrhizobium在没有豆科植物的土壤中,spp也非常丰富。首先,证据来自于对橡树土壤和根际细菌群落的非培养研究。这些社区有大量的人口Bradyrhizobiumspp。9.那10那11].丰富的额外证据Bradyrhizobium在北美针叶林土壤中发现没有豆科植物的一种植物[12].通过数量群体基因组学和全基因组测序,鉴定出BradyrhizobiumSPP。缺乏点头/ nol和nif /修复基因簇,因此不能使豆科植物结瘤或固定大气中的氮。相反,这些菌株具有与复杂碳代谢和芳香族化合物降解相关的多个基因簇。同样,两个Bradyrhizobium从裸露的休耕地和草地的土壤中分离出来的种属菌株不具有共生岛[13].总之,这些研究表明Bradyrhizobium豆科植物不仅可以与豆科植物共生,还可以有一个“自由生活”的生活史。然而,这种非氮固结作用是否存在尚不清楚Bradyrhizobium spp。可以和植物有亲密的关系
如结瘤和固氮BradyrhizobiumSPP。可以用豆科植物主持人建立一个细胞内的生活方式,我们假设这一点Bradyrhizobium缺乏共生岛的物种可以与植物紧密共生。在本研究中,我们调查了是否如此BradyrhizobiumSPP。在自然生态系统中发生非豆类的根部或内部。我们在荷兰的生态测试领域采用了一个名为Mossel的荷兰的生态测试领域,我们以前研究过九种物种的根微生物组,其中拟南芥蒂利亚纳(拟南芥)(Schneijderberg等,准备)。
结果
Bradyrhizobiumsp .定殖植物根
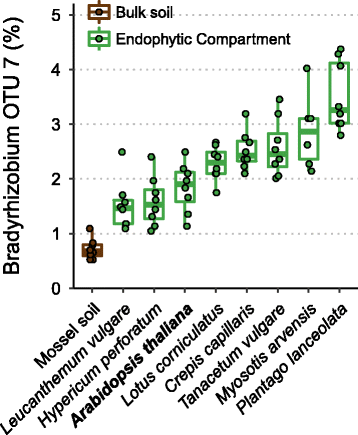
以确定是否Bradyrhizobium物种可以殖民,非豆科植物根源,我们分析了一个单独但同时实验的16S rdna扩增子数据集(Schneijderberg等人。准备)。该数据集包括位于荷兰的威尔圭地区(称为豆类)的野外实验,包括代表六个分类命令的九种植物物种(表1).其中8种非豆科植物,其中拟南芥(accession Mossel;火星科学实验室)。2016年夏季,在莫塞尔(Mossel)的一个试验田上生长8周后,通过16S rDNA扩增子测序,确定了这些物种的根内生菌室的细菌群落(Schneijderberg等人在准备中)。分析揭示了隶属于该属的四个操作分类单元(OTUs)Bradyrhizobium其中一个OTU(#7)在土壤(0.5%)和植物根内生物室中的高度丰富(图。1).这种OTU不仅在豆类的根内内科舱中浓郁Lotus corniculatus,也是所有八个非豆类的根样本,范围为1.5%Leucanthemum vulgare超过3%Plantago生长状况(无花果。1).广泛的丰富Bradyrhizobium结果表明,在莫塞尔地区,该属植物的根定殖是一种属现象。
16S RDNA扩增子测序显示出高度丰富Bradyrhizobium在所有测试的植物物种中。植物物种(列在表格中)1),在荷兰维鲁韦莫塞尔地区试验田种植7周。的相对丰度BradyrhizobiumOTU 7在根内生菌室。每个点代表一个复制(N= 8)
以确定是否Bradyrhizobium我们分析了公开的16S rDNA扩增数据集。这表明,BradyrhizobiumOTUs存在于多种植物的内生菌室中,包括拟南芥、水稻(栽培稻)和玉米(Zea Mays.)(表2).这有力地支持了BradyrhizobiumSpp.是植物根系中常见的内生菌。然而,是否这些菌株组成了Bradyrhizobium在那些研究中,OTUs拥有共生岛,仍然难以捉摸。
拟南芥植物内生Bradyrhizobium sp。MOS菌株缺乏氮固定性状
基于16S rDNA V4区域的OTU可以代表多个菌株甚至物种。为了确定OTU 7是否有多个菌株,以及这些菌株是否具有结瘤固氮基因簇,我们提出了一种分离和鉴定OTU 7的策略Bradyrhizobium从拟南芥mosel实验地的单根中分离到102株具有拟南芥生长特性的菌株Bradyrhizobium.使用Box-PCR的DNA指纹识别这些菌株在12组中分类(附加文件1).接下来,我们对每个组的一个代表性菌株的16S rDNA位点进行了测序。结果显示有四种菌株(占分离株的85%)与之匹配Bradyrhizobium在RDP数据库中[14)(附加文件1).这4株菌株的16S rDNA V4区与OTU 7的一致序列同源性达到99%以上,表明这4株菌株代表该OTU。我们给这些菌株命名Bradyrhizobiumsp。MOS001Bradyrhizobiumsp. MOS004,以它们的起源地区为准。第五种菌株被命名为MOS005,分类为Bradyrhizobium基于16S rdNA基因,也被测序。但是,由于全基因组序列无法确保这是一个Bradyrhizobiumsp.,我们选择从分析中排除它(附加文件1).
来确定这些Bradyrhizobium基于Illumina HiSeq平台上测序的150对碱基对端文库,构建了拟合的基因组序列。这导致了大小从7.6 (MOS003)到9.1 Mb (MOS001)的草案组装,代表6879 (MOS003)到8622 (MOS001)注释基因模型(附加文件)2).这样的基因组大小以及相应的GC含量(64-66%)与之前的测序结果相当BradyrhizobiumSPP。基因组[12那13那15那16].来验证基因组装配草图的完整性Bradyrhizobiumsp. MOS菌株进行Busco分析[17].这表明,在mo004中有97.6%的基因全长存在,在根瘤菌科的其他MOS菌株中有99.1%的基因全长存在,表明接近完整的基因组覆盖(附加文件2).
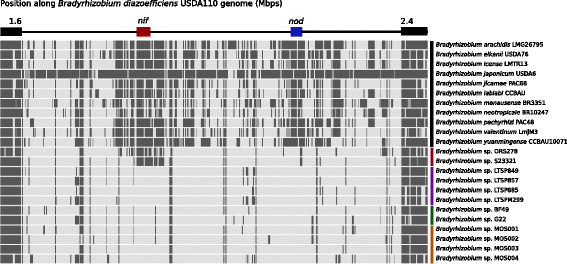
以确定是否Bradyrhizobiumsp。MOS菌株具有染色和/或氮固定基因,我们使用了氮气固定的Symbiont倒数最佳爆炸次算法Bradyrhizobium diazoeffiens.菌株USDA110作为参考[16].这揭示了这四个Bradyrhizobiumsp. MOS菌株在上述两种菌株中均无明显的特异性点头/nol基因簇或nif /修复基因簇(无花果。2).因此我们得出结论Bradyrhizobiumsp. MOS菌株不能结瘤和固氮。
倒数最佳爆击分析表明Bradyrhizobiumsp。MOS菌株缺乏共生岛。关于共生参考菌株的示意图B. diazoeffiens.USDA110,在黑色保守区,在红色中nif /修复基因簇和蓝色的点头/ nol基因簇。在右边,菌株名称(颜色编码如下。黑色:图中的型株。3.;红色:只有株的菌株nif /修复基因簇;紫色:非共生LTSP菌株[12];绿色:非共生菌株G22和BF49 [13]:橙色,MOS菌株。深灰色条表示参考基因组的击中,覆盖率≥50%,相似度≥70%。这Bradyrhizobiumsp。MOS菌株没有受到打击点头/ nol或nif /修复基因群,表明共生岛的缺失。如预期的那样,LTSP菌株和G22 / BF49菌株的共生基因均未出现命中
拟南芥植物内生Bradyrhizobiumsp。菌株高度多样化
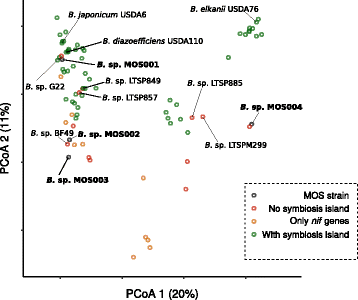
根据遗传指纹图谱,这4个分离株是不同的。为了深入了解这四种分离物所代表的分类学多样性Bradyrhizobiumsp。MOS菌株,测定了系统发育关系。由于我们获得了全基因组序列,我们基于31 Amphora基因的核苷酸序列的系统发育分析[18,包括一组高度保守的标记基因。我们将这些基因与所有基因进行了比对Bradyrhizobium已发表基因组的种,包括在森林地区确定的非共生菌株(指定LTSP [12])及草原(指定为G22及BF49 [13])。随后,重建了系统发育关系(图。3.).系统发生树的拓扑结构与之前报道的基于单或多序列比对的系统发生一致[12那19那20.].这Bradyrhizobiumsp. MOS菌株在属内代表四个不同的谱系(图。3.).Bradyrhizobiumsp. MOS001和MOS002属于以型菌株为代表的一个分支的不同谱系b .日本血吸虫USDA6和集群与来自草原上分离的两个非共生菌株(即Bradyrhizobiumsp. G22和BF49)。Bradyrhizobiumsp. MOS003属于一个大的分支,其中包括类型菌株b . arachidisLMG26795和b . yuanmingenseCCBau10071,但是Bradyrhizobiumsp. MOS004与Bradyrhizobiumsp。DFCI-1,内部B. elkanii.USDA76思工。
由于四种菌株起源于唯一的拟南芥根,因此我们质疑分离株是否在功能上相似,尽管它们遗传分歧。我们使用了先前发布的自定义r管道[21]使用kegg orthology(ko)数据库预测每个隔离中的所有开放阅读帧的功能组[22].在确定KO基团的存在和不存在之后并使用二进制距离测量产生一对不相似矩阵之后,我们沿着前两个主坐标绘制每个功能轮廓。这导致PCOA,其中物种之间的距离类似于菌株的系统发育关系(图。4.).沿着前两个主坐标Bradyrhizobiumsp。MOS001,MOS002和MOS003应变簇相对接近B. diazoeffiens.USDA110和b .日本血吸虫USDA6,而MOS004则相对接近B. elkanii.进化枝。这表明MOS分离株不仅具有遗传多样性,而且具有功能多样性。
MOS分离物重新播放拟南芥根
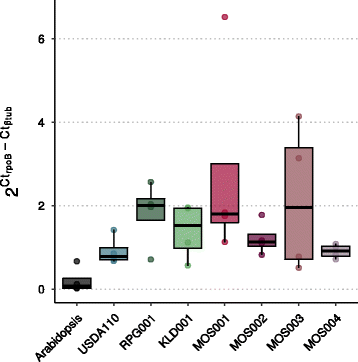
我们旨在确定是否Bradyrhizobium在实验系统中,sp. MOS001, MOS002, MOS003和/或MOS004具有定殖植物根系内生菌室的能力。此外,我们质疑这种定殖能力在多大程度上与重氮营养体的定殖能力相似BradyrhizobiumSPP。为了调查这一点,我们在体外种植拟南芥幼苗(加入MSL)移植到接种的高压灭菌河砂中,在其中设立增长测定Bradyrhizobium应变为密度与密度相当Bradyrhizobium在天然豆类土壤中,约为2.5×105.每克土壤的细胞。2周后,分析植物。四个都不是Bradyrhizobium三种固氮共生菌株对拟南芥的鲜重均无影响3.).为了量化不同菌株的根殖民,我们在高度保守的细菌上使用QPCRRPOB.基因并归一化,这与拟南芥的微管蛋白结合共聚因子C [23].未接种对照植株的根中细菌DNA检测接近于零,而根内生菌室样品中所有菌株的DNA检测量约为每个植株基因组拷贝2个细菌基因组拷贝(图1)。5.).比较这四个Bradyrhizobiumsp. MOS菌株与3种固氮共生体的定殖能力无显著差异。这说明了共生和非共生Bradyrhizobium在我们的生长系统中,菌株同样能很好地定植拟南芥的根。5.).
qPCR-based量化显示Bradyrhizobium拟南芥根的定植。y轴是每个植物基因组拷贝的细菌基因组拷贝数的近似值。这是通过将rpoB(靶向细菌DNA)和Btub(靶向拟南芥B微管蛋白)的Ct值的差异乘以2来计算的。在添加了细菌的蒸压河沙中,将2株植物的根进行池积,每处理共4个重复。拟南芥:模拟处理的植物。USDA110:Bradyrhizobium diazoeffiens.USDA110。RPG001:Bradyrhizobiumsp.隔绝Chamaecrista模仿结节。KLD001:Bradyrhizobiumsp.隔绝Parasponia andersonii结节。这些菌株具有共生岛
讨论
在这里,我们证明了多种非豆科植物(包括拟南芥)的内生菌室是由不同范围的非固氮定殖Bradyrhizobium物种。早期的研究表明Bradyrhizobium非豆科植物根际土壤中物种丰富。例如,在最近的一项研究中Bradyrhizobium,但也根瘤菌和Bulkholderia在由31种植物组成的热带时序的根际核心微生物群中发现OTUs [24].这与越来越多的数据相一致,这些数据表明根茎菌与植物的相互作用是进化保守的,非共生相互作用可能是广泛存在的[12那13那24].这些研究中发现的根瘤菌是否具有共生基因还有待检验。事实上Bradyrhizobiumsp。MOS菌株,我们在拟南芥的根生成室和其他非豆类物种中鉴定,没有共生岛,表明相互作用与已知的染色和固定基因无关。此外,我们得出结论,内心舱的定植Bradyrhizobiumspp是一种共同的特征BradyrhizobiumSPP。大多数可能具有广泛的主机范围。
这四个Bradyrhizobiumsp. MOS菌株从单个拟南芥根中分离得到。令人惊讶的是,我们发现它们的基因高度多样化。利用31个保守双耳基因,对MOS菌株和所有公开的慢生根瘤菌分离株进行系统发育分析。根据“平均核苷酸识别”软件所作的分析[25,我们认为MOS001很可能代表ab .日本血吸虫与USDA6型菌株具有95%的同源性。而MOS002、MOS003和MOS004与已知型株序列相似性小于95%,因此这些菌株可能代表新种。这些分离株具有高度多样化的功能特征,这一事实表明,共生岛的缺失与特定的非共生生活方式无关,但共生岛的缺失是普遍存在的,这些非重氮营养物种具有多样化的生活策略。
在拟南芥上的再接种实验在我们测试的条件下没有显示出任何菌株对植物生长的影响。然而,很有可能在一个复杂的微生物群中,植物和微生物都从紧密的联系中受益。我们证明了孤立的Bradyrhizobiumsp. MOS菌株定殖根的效率可与固氮共生菌相媲美。与此相一致,我们认为内生植物的生活方式可能是Bradyrhizobium此前已有研究表明,结瘤基因和固氮基因已通过水平基因转移在豆科植物的不同属内分布,从而与豆科植物建立结瘤共生关系[3.那26].因此,似乎有可能Bradyrhizobium具有内心生命风格的菌株获得了点头/ nol和nif /修复基因通过水平基因转移而获得了特殊的固氮共生生活方式。
结论
这项研究表明Bradyrhizobium在许多植物种的根内生菌室定殖,包括模式种拟南芥。来自拟南芥单个根的4个分离株具有较高的遗传和功能变异,但均缺乏结瘤和固氮基因。这些non-diazotrophicBradyrhizobiumsp. MOS菌株,以及重氮营养菌株,重新定殖拟南芥根内生菌室。综上所述,本研究表明,植物根系是植物的主要生态位BradyrhizobiumSPP。,这可能是该属的染色和氮固定性状的演变的祖先。
方法
土壤收集与田间实验
2016年5月,在荷兰“Hoge Veluwe”的莫塞尔地区(坐标:N52°03 ' 35.5″E5°45 ' 06.4″),从100米半径内的四个不同地点收集了土壤。如有植被存在,则去除表层5 ~ 10 cm土壤。土壤均质,清除所有碎屑。在使用前,土壤被保存在4°C的冷室内。所有植物用4倍稀释家用漂白剂消毒10分钟,用无菌MQ水洗涤7次,用湿滤纸转移到板上,4°C放置48小时,然后移到21°C黑暗培养箱中。用70%乙醇对拟南芥种子进行短时间漂洗l . corniculatus并用H治疗2分钟2所以4.在接触家用漂白水之前。的种子T. Officinale.那T.Vulgare.那l . vulgare和薄荷直接在21℃培养箱中置于没有冷处理的培养箱中。豆类土壤被置于带3×3厘米的罐和浇水的托盘中。为了去除内源性种子种群,将托盘放入温室中2天。在除草后,将板上的无菌幼苗用豆类土壤移植到托盘上,并在7天后将包括土壤的植物种植到豆类领域。
植物收获和微生物群落的DNA分离
经过6周的田间,在植物底部围绕植物3-4厘米,植物被挖掘出来。因此,孔宽6-8厘米,深约10厘米。在将植物(包括CLOC)运送到实验室之后,我们施加了如前所述的收获方案[27].四株单独的植物被汇集到一个样本中。分别使用Mo Bio PowerSoil试剂盒(Qiagen)和Fast DNA Spin kit for soil (MP Biomedicals)从土壤和内生菌室样品中分离DNA。通过纳米滴和凝胶电泳检测DNA的质量和数量。约400 ng送往北京基因组研究所进行16S rDNA测序。
16S RDNA扩增子测序和数据处理
使用引物515f和806r [28],V4区在Hiseq2500测序平台(Illumina)上的BGI测序。使用先前报告的自定义实施处理来自BGI的原始数据[29的[30.在Schneijderberg等人(准备中)中进行了详细的修改。简而言之,使用ChimeraSlayer对嵌合体的读取进行了质量过滤和过滤。使用97%的身份阈值,确定新的OTUs,并使用RDP分类器与GreenGenes数据库进行分类分配。去除与线粒体和叶绿体序列相关的OTUs,以及至少5个样本中没有25个reads的OTUs。获得…的相对丰度Bradyrhizobium,从匹配的OTUs读取Bradyrhizobium将每个样本加起来,然后除以过滤稀有类群后该样本的读总数。
拟南芥生长试验
将无菌拟南芥MSL种子转移至1/2 MS-平板,并在21℃下用16H / 8H光周期孵育。7天后,根是加利福尼亚州。3厘米的长度,并移植到如下制备的盆中:细菌培养物循环,用10mM MgSO洗涤4.加入15毫升¼Hoagland 's培养基中[31].将这种培养基添加到4 × 4厘米装满无菌河沙的花盆中。土壤湿度为保水能力的70%,细菌细胞密度为4 × 105.每克无菌沙粒的细胞数。无菌对照仅用¼霍格兰氏剂处理。在生长期间,5个单独的花盆称重以估计水分损失,如果必要的话用无菌水灌溉。两周后,植物被小心地从沙子中挖出来,嫩枝与根分开。立即测定地上部生物量。根经历了前面描述的收获协议[27],置于−80℃保存至提取DNA:用金属珠在Tissue Lyzer中研磨根,加入500 μL CTAB缓冲液。样品在65℃下孵育30分钟。加入氯仿500 μL,以14000 g纺丝5 min。然后,将上相转移到含有400 μL异丙醇的新的Eppendorf管中。样品在−80℃孵育1 h。孵育后,在14000 g中纺丝10 min,用500 μL 70%乙醇洗涤DNA颗粒。再离心一步,去除乙醇,室温干燥15 min。最后,将颗粒重悬于50 μL MilliQ水中,并在−2℃下保存,直到进一步使用。
定量聚合酶链反应
瞄准的底漆RPOB.基因BradyrhizobiumSPP。(RPOB_FW1; 5'-CGCTGAAGAACCTCGACGAAGCC-3'和RPOB_RV1; 5'-CGGCGTGATCTTGCCGACG-3')设计了佐锯8.1。Arabidopsis B-小管蛋白基因引物(前进:5'-AgaaAccggaaacgagc-3'和反向5'-AcaAgacacttccGcttgg-3')用于促进植物DNA [23].在QPCR之前测试两个引物套的效率。通过遵循制造商的协议(Power Sybr Green,Life Technologies),在每次治疗中进行四种植物DNA样本进行QPCR。要测定细菌与植物DNA,我们使用的公式:
这是细菌基因组拷贝数与植物基因组拷贝数的近似值。
菌株分离培养
超声处理和清洁的拟南芥根组织用研钵和杵在1ml磷酸盐缓冲液中研磨(每升:6.33 g NaH)2阿宝4.·H2O, 10.96 g Na2HPO.4.·2H.2200 μL Silwet L-77)。将100 μL复镀在1/10 TSA和YEM介质板上。剩余溶液500 μL保存在40%甘油中备用。另外,还分别镀100×和1000×倍稀释液。选取3天和7天后出现的菌落(附加文件)2),以96孔格式储存于YEM介质中。
用于菌株测序的基因组DNA分离
将菌株接种于10 ml YEM液体培养基中。生长5天后,细胞按4000 g离心,弃去培养基。根据制造商的说明,使用Qiagen血液和组织试剂盒进行DNA分离。通过纳米滴和凝胶电泳检测DNA的数量和纯度。由于总数量不足以满足测序的最低要求,MOS003又进行了一轮DNA分离。
对菌株进行测序
使用Illumina Hiseq 2500 PE 150平台在BGI进行测序,具有350bp插入库尺寸。配对的Illumina阅读是用FASTQC评估的质量[32],并相应地用Trimmomatic v. 0.35进行修剪[33,对所有样本进行PHRED33阈值评分大于20的筛选。原始读取由SPAdes v. 3.9.0汇编[34]根据不同的k-mer大小,最好的装配判断为包含最小数量的contigs。从基因组中移除Contigs <1000bp(仅存在mos002中)。用QUAST v评估装配统计信息.4.5 [35].Contig的GC含量揭示了MOS001和MOS004中的污染。用maxbin v除去污染。2.2.4 [36].用ProDigal v预测豆晶组件的ORF和蛋白质组。2.6.1 [37].程序集使用Prokka v1.11工具进行注释[38].BLAST数据库由隶属于该基因的RefSeq蛋白组成Bradyrhizobium属的BLAST+包[39].通过指定属旗,Prokka首先对该数据库中的基因进行了注释。
同源共生的岛
所有公开的草案和属于的完整组合Bradyrhizobium从2017年1月从NCBI撤回了属。用抛光互易击中算法鉴定共生基因[39].Symbiotic岛上的基因B. diazoeffiens.USDA110装配用作正轨搜索的参考。与参考基因相比,存在或不存在作为推定的正代,其具有至少70%同一性和50%的覆盖率。
系统发育多样性分析
发现31种Amphora基因在所有组件中存在。Inphora基因的核苷酸序列与分类ωV。1.2.4 [40]和非保守片段用Gblocks去除[41].使用Geneious 8.1中的邻居连接方法,通过1000个bootstrap生成系统发育树。使用交互式生命树(iTol)平台对树进行可视化[42].
功能多样性分析
通过使用Bai等人公开发布的HMM模型,在ORF中测定Kegg直疏原素(KO)基团的存在或不存在。[21].使用Hmmer v.3.1b2 [43hmmsearch工具,其阈值设置为小于10 × 10的e值−5大于70%的覆盖范围。对于符合此标准的多次命中,仅保留了最小电子值的命中。数据从每个KO组的绝对计数转换为缺席或存在。使用vegdist.从R中的素食包中的命令使用二进制距离测量来创建距离矩阵。PCOA由此进行cmdscale.命令,使用Bray-Curtis作为不同的方法。
参考文献
- 1.
Masson-Boivin C,Giraud E,Perret X,Batut J.与豆类建立氮素固定的共生:大麻食谱?趋势微生物。2009; 17:458-66。
- 2.
udvardi m,poole ps。豆类根瘤菌中的运输和新陈代谢。Annu Rev植物Biol。2013; 64:781-805。
- 3.
Remigi P,朱家,年轻JPW,Masson-Boivin C. symbiosis中的共生:进化氮素固定豆科豆科植物。趋势微生物。2016; 24:63-75。
- 4.
约旦DC。注意:将japonicum buchanan 1980转移到Bradyrhizobium gen。11月,一种缓慢生长的生长,根瘤菌来自豆科植物的细菌。int J SYST Bacteriol。1982; 32:136-9。
- 5.
在命名法中有地位的原核生物名称的清单。核酸Res. 2014;42: D613-6。
- 6.
Trinick MJ,Hadobas Pa。Parasponia-Bradyrhizobium共生的生物学。植物土壤。1988; 110:177-85。
- 7.
王志强,王志强。植物根瘤菌与植物共生关系的研究。Microbiol启1995;59:124-42。
- 8。
α -proteobacteria的翻译结果:微生物学报2004;2:933-45。
- 9。
Sachs JL, Ehinger MO, Simms EL。野生慢生根瘤菌的欺骗和共生损失的起源。《生态学杂志》2010;23:1075-89。
- 10.
Hartmann M,Howes Cg,Vaninsberghe D,Yu H,Bachar D,Christen R等。木材收割对北极草林土壤微生物群落的显着和持续影响。ISME J. 2012; 6:2199-218。
- 11.
urz S, Buée M, Murat C, Frey-Klett P, Martin F.焦磷酸测序揭示了橡树根际和周围土壤的细菌多样性。环境微生物代表2010;2:281-8。
- 12.
Vaninsberghe D,Maas Kr,Cardenas E,Strachan Cr,Hallam Sj,Mohn WW。非共生的Bradyrhizobium Ecotypes主导北美森林土壤。ISME J. 2015; 9:2435-41。
- 13.
新欧洲自由生活的非重氮营养慢生根瘤菌分离物从对比土壤缺乏结瘤和固氮基因-基因组比较。Sci众议员2016;6:25858。
- 14.
王强,Garrity GM, Tiedje JM, Cole JR. Naïve基于贝叶斯分类器的细菌rRNA序列快速分类。应用环境微生物。2007;73:5261-7。
- 15.
Kaneko T, Nakamura Y, Sato S, Minamisawa K, Uchiumi T, Sasamoto S,等。固氮共生菌日本慢生根瘤菌USDA110的全基因组序列。DNA研究》2002;9:189 - 97。
- 16.
Sugawara M, Tsukui T, Kaneko T, Ohtsubo Y, Sato S, Nagata Y, et al.;重氮慢生根瘤菌USDA 122的全基因组序列,一种固氮大豆共生体。基因组Announc。2017;5:e01743-16。
- 17.
Simão FA, Waterhouse RM, Ioannidis P, Kriventseva EV, Zdobnov EM. BUSCO:用单拷贝同源性评估基因组组装和注释完整性。生物信息学。2015;31:3210-2。
- 18.
吴敏,艾森JA。一种简单、快速、准确的系统基因组推断方法。基因组医学杂志。2008;9:R151。
- 19。
徐志强,李志强,李志强,等。慢生根瘤菌的多位点序列分析。微生物学报。2009;32:101-10。
- 20.
张XX,Guo Hj,Wang R,Sui Xh,Zhang Ym,Wang et等。伴有共生岛内外的基因多层序列分析揭示了大豆的遗传分歧。申请环境微生物。2014; 80:3181-90。
- 21.
王志强,王志强,王志强,等。拟南芥叶片和根菌群的功能重叠。大自然。2015;528:364-9。
- 22.
Ogata H, Goto S, Sato K, Fujibuchi W, Bono H, Kanehisa M. KEGG:京都基因和基因组百科全书。核酸Res. 1999; 27:29-34。
- 23.
李洪波,李洪波,李洪波,等。植物特异性组蛋白去乙酰化酶HDT½调控GIBBERELLIN 2- oxidase 2的表达以控制拟南芥根分生组织细胞数量。植物细胞。2017; 29: tpc。2017:00366。
- 24.
Yeoh Yk,Dennis Pg,Paungfoo-Lonhienne C,Weber L,Brackin R,Ragan Ma,等。沿热带土壤计量术中植物植物中核心根微生物体的进化储存。NAT Communce。2017; 8:215。
- 25.
尹胜,何淑敏,林俊,权S,田俊。一种计算平均核苷酸识别的大规模算法评估。范列文虎克,中华微生物学杂志。2017;110:1281-6。
- 26.
Sachs JL,Skophammer Rg,Regus Ju。细菌共生中的进化转变。PROC NATL ACAD SCI。2011; 108(补充2):10800-7。
- 27.
Lundberg DS, Lebeis SL, Paredes SH, Yourstone S, Gehring J, Malfatti S,等。确定拟南芥核心根微生物组。大自然。2012;488:86 - 90。
- 28.
Parada AE, Needham DM, Fuhrman JA。每个基础都很重要:用模拟群落、时间序列和全球现场样本评估海洋微生物群系的小亚基rRNA引物。环境Microbiol。2016;18:1403-14。
- 29.
Pérez-Jaramillo JE, Carrión VJ, Bosse M, Ferrão LFV, De Hollander M, Garcia AAF,等。野生和驯化菜豆根际微生物组组成与基因型和根系表型性状的联系ISME j . 2017; 11:2244-57。
- 30.
Caporaso JG,Kuczynski J,Stombaugh J,Bittinger K,Bushman FD,Costello EK等。Qiime允许分析高吞吐量社区测序数据。NAT方法。2010; 7:335-6。
- 31。
Hoagland Dr,Arnon Di。没有土壤种植植物的水培养方法。加利福尼亚州大九欧元科技。1950; 347:1-32。
- 32.
FastQC:用于高通量序列数据的质量控制工具。https://www.bioinformatics.babraham.ac.uk/projects/fastqc/.
- 33.
Bolger Am,Lohse M,Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014; 30:2114-20。
- 34.
王志强,王志强,王志强,等。一个新的基因组组装算法及其在单细胞测序中的应用。J computer Biol. 2012; 19:455-77。
- 35.
Gurevich A,Saveliev V,Vyahhi N,Tesler G. Quast:基因组组件的质量评估工具。生物信息学。2013; 29:1072-5。
- 36.
吴耀伟,唐耀华,丁奇SG,西蒙斯学士,歌手SW。MaxBin:一种自动分类方法,使用期望最大化算法从宏基因组中恢复个体基因组。微生物。2014;2:26。
- 37.
凯悦D,Chen G-L,Locascio PF,Land ML,Larimer FW,Hauser LJ。浪费:原核基因识别和翻译起始位点鉴定。BMC生物信息学。2010; 11:119。
- 38.
Seamann T.Prokka:快速原核基因组注释。生物信息学。2014; 30:2068-9。
- 39.
公鸡PJA,谢尔顿·杰姆,朱宁B,约翰逊·杰,索兰佐N. NCBI Blast +融入了Galaxy。傻瓜。2015; 4:39。
- 40。
李伟,李志伟,李伟,等。使用Clustal omega快速、可扩展的高质量蛋白质多序列比对生成。生物学杂志。2011;7:539。
- 41。
关键词:系统发育分析,序列分析,保守序列生物化学学报2000;17:540-552。
- 42。
交互式生命树(iTOL) v3:用于显示和注释系统发育和其他树的在线工具。核酸Res. 2016;44: W242-5。
- 43。
涡流SR。配置文件隐藏马尔可夫模型。生物信息学。1998年; 14:755-63。
- 44.
等。拟南芥根寄生菌群的结构和组装线索。大自然。2012;488:91-5。
- 45.
Lebeis SL,Paredes Sh,Lundberg DS,Breakfield N,Gehring J,McDonald M等人。水杨酸通过特异性细菌征草调节根微生物组的定子。科学。2015; 349:860-4。
- 46.
王志强,王志强,王志强,等。水稻根相关微生物群的结构、变异和组装。中国科学院院刊。2015;112:e111 - 20。
- 47.
牛斌,郑兴国,郑兴国。玉米根系细菌群落结构的简化和代表性研究。中国科学院院刊。2017;114:E2450-9。
致谢
我们要感谢课程分子工具箱#60和#63的学生在Wageningen大学执行试点实验,Marnix Medema博士为他的宝贵评论和Zosapol Harnvanichvech和Zhichun Yan在编制时为他们的帮助手稿。
资金
本研究由ERC的3100000843号基金资助。
数据和材料的可用性
这Bradyrhizobiumsp. MOS草案基因组可在NCBI上获得,登录号为SAMN07975002 (MOS001)和SAMN08687197 - SAMN08687199 (MOS002 - MOS004)。16S扩增数据是准备中的手稿的一部分。该手稿的草稿版本和相应的16S放大器数据可根据要求提供。
作者信息
从属关系
贡献
MS、SP、XC和CF分离并鉴定菌株。MS和LS组装基因组并进行硅分析。MS, XC和CF进行了其他实验。MS, RG和TB撰写了手稿。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
陈志强,陈志强。et al。一种遗传和功能上不同的非重氮营养生物Bradyrhizobiumsp .定殖于根内生菌室拟南芥蒂利亚纳.BMC植物杂志18,61(2018)。https://doi.org/10.1186/s12870-018-1272-Y.
收到了:
公认:
发表:
关键字
- Bradyrhizobium
- 拟南芥
- 根殖民
- 内科舱