摘要
背景
人参(Panax Ginseng.梅耶)是一种多年生草本植物,在传统的东方医学中已使用了数千年。人参皂苷是人参中最主要的生物活性成分,对人体健康具有重要的药理作用。人们已经认识到该植物对人类的重要性,一个完整的组学资源对促进该植物的基因组研究、分子育种和药理学研究是必不可少的。
描述
第一个基因组序列草图p .人参最近举报了“Chunpoong”。这里,使用基因组草稿,转录组和功能注释数据集p .人参,我们构建了人参基因组数据库http://ginsengdb.snu.ac.kr/,第一个开放式接入平台,提供全面的基因组资源p .人参.该数据库的当前版本提供了最新的迄今为止的基因组序列(大约3000 MBP的支架序列)以及59,352基因的结构和功能注释以及基因的基因的数字表达,基于来自不同组织的转录组数据,生长阶段和治疗。此外,还提供了用于可视化的工具和来自各种分析的基因组数据。数据库中的所有数据都在手动策划并集成在用户友好的查询页面中。
结论
该数据库为一系列相关的研究领域提供了有价值的资源p .人参和其他属于蜂目的物种,以及一般的植物研究团体。人参基因组数据库可访问http://ginsengdb.snu.ac.kr/.
背景
人参(Panax Ginseng.是一种多年生草本植物人参五加科五加属植物,在东亚和北美被广泛用作传统药物。人参的主要生物活性成分是人参皂苷(统称为三萜类皂苷),通过类异戊二烯途径生物合成[1].人参对人体有多种疗效,包括治疗癌症、糖尿病、心血管、压力等。2,3.,4,5,6].p .人参已知是四倍体(2n = 4×= 48),估计基因组大小约为3.6 gbp [7,8].它庞大的、高度重复的基因组经历了全基因组复制,阻碍了全基因组测序的进展p .人参[7].此外,人参栽培田的世代时间长(4年)和维护困难也限制了人参的遗传研究p .人参.然而,随着新的测序技术的出现,表达序列标签(ESTs)和RNA-Seq数据已经从不同的组织和生长阶段产生p .人参[9,10,11,12],并以此为基础对参与人参皂苷生物合成途径的一些基因进行了表征[10,11].近日,完整的叶绿体基因组序列p .人参对品种及近缘种进行了鉴定[13,14].此外,还确定了人参品种和物种的鉴定,还鉴定了叶绿体基因组多样性[13,14,15,16,17].
在该项目的一端,来自NCBI DB-EST(截至2017年1月)的共有17,773个EST)和不定根的数据库[9]是人参的公开供应品。但这些数据对人参功能基因组学、比较基因组学和分子育种的研究尚不充分。尽管人参是一种重要的药用作物,具有很高的药理价值,但却没有公开的人参综合数据库。鉴于人参对人类健康的众多影响,一个基因组和转录组数据库对人参研究社区和其他Apiales目的近亲属至关重要。此外,一个完整的人参遗传、基因组和代谢资源数据库也有望成为翻译基因组学的宝贵资源。最近,我们生成了大量的基因组和转录组数据p .人参品种“Chunpoong”[18].
在这项研究中,我们建立了集成了基因组序列草图,转录谱和人参的注解数据集的动态数据库。这人参基因组数据库现在是公开可用的(http://ginsengdb.snu.ac.kr/)供全球科学界使用,以探索广阔的可能性。
这种用户友好的数据库将作为挖掘基因序列和来自各种组织,发育阶段,和治疗样品的他们的数字表达数据集线器。我们的数据库接口将促进使用InterPro中,KEGG,BLAST和基因本体论(GO)数据库,基因家族和相关功能注释的方便检索。为了加快人参代谢组学,我们已经做了一个独立的部分,其分类的各种代谢途径,包括人参皂甙生物合成途径相关的基因。此外,我们还包括强大的工具,如BLAST和基因组浏览器(JBrowse)19[学人参基因组特征的调查和可视化。该数据库将定期使用新的基因组序列和有关注释的信息进行更新,并将提供参考基因组信息进行研究p .人参以及相关物种。
建筑和内容
全基因组测序、组装和基因模型
的基因组序列数据p .人参从利用Illumina HiSeq平台精英品种“Chunpoong”产生。A total of 746 Gb paired-end and 365 Gb mate-paired raw data were produced and assembled, yielding the draft genome sequence of about ~ 3.0 Gb in size. The repeat sequences were identified and masked using RepeatModeler [20.]和RepeatMasker [21].利用证据模型(EVM)进行自动基因预测[22用从头算预测(BRAKER 1 [23)、蛋白质证据、ESTs和RNA-Seq证据[24].除去转座子序列后,预测了总共59,352个诱导的蛋白质编码基因。这些基因在功能上使用Interpro注释[25], Blast2Go [26], KEGG [27[BLASTP搜索已知的蛋白质数据库。
转录组数据
从利用Illumina HiSeq和PacBio平台(各种组织和人参的非生物胁迫处理的样品所产生的转录组数据http://ginsengdb.snu.ac.kr/transcriptome.php.).大约120 Gb的原始RNA- seq reads经过四步预处理以获得高质量的RNA reads。最初,使用BWA对可用细菌基因组进行读图,去除细菌污染读[28].预处理后,使用FastUniq过滤掉重复的读[29].第三步是使用SortMeRNA去除核糖体RNA (rRNA)读取[30.].最后,使用NGS QC Toolkit删除低质量的读取[31.].高质量的RNA-Seq reads被Trinity用于从头组装[32.[hisat&stringtie [33.],然后用于在基因组序列草图基因预测。此外,高品质PacBio序列用于细化预测基因模型。
基因家族和代谢途径
根据蛋白结构域(Pfam)和InterPro结构域对基因进行分组。通过KAAS服务器预测代谢途径[27的基因注释参考信息拟南芥蒂利亚纳,素类,大豆,vitis Vinifera和茄属植物lycopersicum.可以访问此信息http://ginsengdb.snu.ac.kr/gene_family.php和http://ginsengdb.snu.ac.kr/metabolic_pathway.php.
基因组级代谢网络
基于基因注释,重建了一个分区化的基因组代谢网络,提供了人参中所有代谢物,酶,反应和途径的全球概述。该网络总共占4946个基因,映射到2194次酶催化和蛋白质介导的传输反应,涉及六个细胞内隔室的2003个独特的代谢物。可以访问人参基因组代谢网络的全球概述http://ginsengdb.snu.ac.kr/network/index.html.这个网络也可以作为一个系统生物学标记语言(SBML)文件下载。
转录因素
转录因子(TFS)是开发和刺激反应的关键调节因素。基于PLNTFDB的标准来确定TFS [34.]使用iTAK (http://bioinfo.bti.cornell.edu/cgi-bin/itak/index.cgi.).共鉴定出4439个TF和转录调节基因,分为94个TF家族(http://ginsengdb.snu.ac.kr/tf_class.php).
人参皂苷生物合成途径中的基因
人参皂苷是通过甲羟戊酸(MVA)和2-C-甲基d赤藓糖醇-4-磷酸(MEP)途径[生物合成10,35.].基于KEGG以及BLASTP注释,鉴定了参与人参皂苷生物合成的基因数。在该途径的最终步骤中,还基于Interpropatepatterns(PS00375)并爆炸同源搜索,还识别了在该途径的最后步骤中负责生产各种种植皂苷的UDP糖基转移酶(UGT)基因。可以访问推定的途径和相关基因http://ginsengdb.snu.ac.kr/pathway.php.
数字基因表达谱
利用所有RNA-Seq数据确定数字基因表达谱。使用RSEM计算每个样本中所有基因的FPKM值[36.].此外,使用M值(TMM)的修剪均值来归一化表达数据,以解决测序深度的差异。可以访问数字表达式配置文件http://ginsengdb.snu.ac.kr/gene_exp.php.
效用和讨论
数据库实现
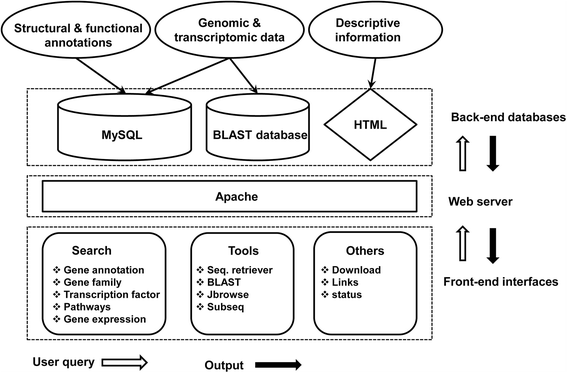
在Linux(CentOS 6.6)操作系统中建立了人参基因组数据库,具有Apache HTTP服务器。PHP,HTML,JavaScript和Python脚本用于构建用户友好的界面和设计网页。为了可视化基因组,我们包括jbrowse 1.11.6,它是基于JavaScript的基因组浏览器,允许对基因组注释进行视觉分析[19].我们还包括一个BLAST服务器,以使用GINSENG的不同数据集进行同源性搜索。此外,我们开发了一种基于Python的工具来检索或下载特定的支架和基因序列。人参基因组数据库架构的概述如图4所示。1.
查询搜索
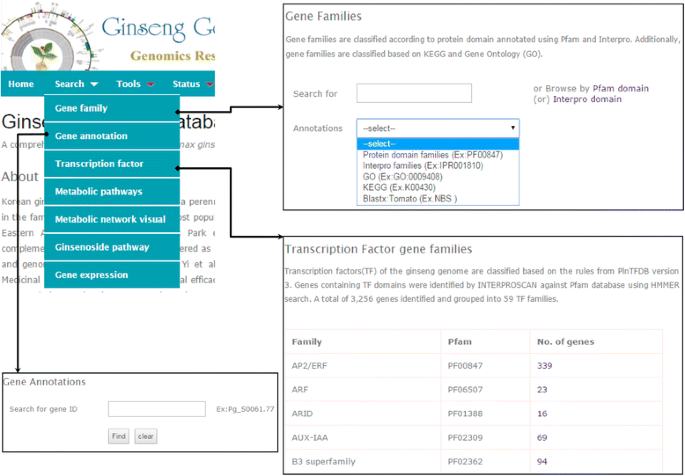
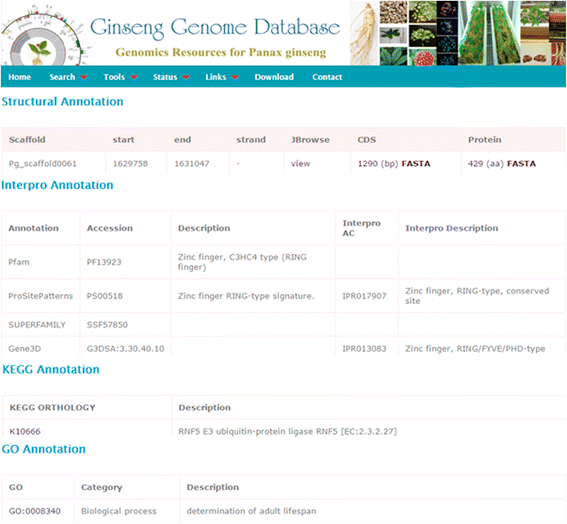
人参基因组数据库提供了两个主要的面板,即“搜索”面板和“工具”面板,这两个面板都以易于使用的方式组成了所有的信息。在“搜索”面板下,可以通过基因ID、InterPro结构域、Pfam结构域、GO和KEGG orthology (KO)标识符(ID)和关键字搜索基因或基因家族(图)。2).此外,用户可以使用“InterPro”和“Pfam”域浏览分类的基因家族。“基因家族”选项提供了一个子菜单,用于检索与用户定义的功能域或关键字相关的基因组。用户可以从输出页面下载特定基因家族的所有编码序列(CDSs)或用户选择的FASTA格式CDSs。“基因注释”部分提供了详细的注释,包括用户查询基因的结构和功能注释(图。3.).在“输出”页面中,用户可以找到指定基因,CD和蛋白的支架被注释,然后可以通过jbrowse可视化这些基因。此外,还可以浏览基于包括PFAM,ProsePatterns和Superfamily,Go,KegG和Blast的Interprojinnotation的功能描述。
注释TF家族的列表包括在'转录因素的部分(图。2).用户可以探索与特定TF家族相关的TF基因,下载相应的CDSs。在“代谢路径”部分,用户只需输入路径名称或点击“浏览路径”,即可检索特定路径中涉及的基因。我们的数据库还提供链接,用户可以从KEGG数据库中查询相应基因和完整通路的“酶委托(EC)编号”。人参皂苷生物合成的已知途径和相应的基因列在“人参皂苷途径”部分。在“基因表达”部分,用户可以输入一个特定的基因标识符,并可以选择比较人参植物组织或非生物胁迫之间的表达。这将以不同颜色的条形图形式返回表达式数据。
序列寻回犬
我们结合了使用Python脚本一个“序列取回工具”。这可以通过在所输入的查询框中输入单个或批量基因(CDS中和肽)或支架的ID来使用。我们的定制与“观点”和“下载”的输出选项。用户可以通过选择“查看”选项查看网页上FASTA格式的单个或多个序列。在许多序列的情况下,用户可以选择“下载”选项来下载FASTA格式的序列。
爆炸
该数据库还提供了同源搜索工具BLAST,该工具使用ncbi-wwwblast包(v2.2.26)嵌入到数据库中,为用户提供图形界面。建立了BLAST-able数据库,包括全基因组草图序列、编码序列(CDSs)和蛋白序列,用于BLAST搜索。此外,还提供了人参全基因组研究所需的各种组织和非生物胁迫处理的转录组数据,以及此前发表的RNA-Seq组装和est序列,用于BLAST搜索。用户可以直接在“查询文本框”中粘贴查询序列,通过选择适当的搜索程序(BLASTN, BLASTP, BLASTX, TBLASTN或TBLASTX)执行BLAST搜索,其中BLASTP和TBLASTN仅根据氨基酸序列进行查询。选项来过滤低复杂度和设置e值在“其他选项”部分下可用。结果格式也可以使用“结果选项”下的选项自定义。
jbrowse.
在“工具”小组下,包含'jbrowse'以显示人参的基因组特征。所有组装的支架和预测基因都用于构建基因组浏览器。“jbrowse”的主页包含几个不同子部分下的曲目。用户可以选择“脚手架”(只有30个脚手架可以在下拉菜单中看到)或在搜索框中使用或没有位置的位置键入脚手架的名称。用户可以可视化各种基因组特征,如“基因模型”,“从头开始“为基因注释管道”、“组装好的转录组结构”和“重复序列”生成的基因模型。此外,RNA-Seq的比对读取直接由二进制比对/MAP (BAM)和使用GMAP的PacBio contig比对生成的基因组序列[37.]也纳入活跃起来的基因的结构标注。此外,非编码基因的蛋白质序列包括微RNA(miRNA)和长的非编码RNA(lncRNA)可以与他们的基因特征一起可以看出。除了Panax Ginseng.,我们已经整合了其他基因引导的转录组组装人参物种,即三七和p . quinquefolius这将有助于比较基因结构或发现缺失的基因任何其他人参物种。
下载
所有组装的基因组和转录组序列都可用http://ginsengdb.snu.ac.kr/data.php.我们的数据库提供HTTP链接,以FASTA格式下载基因组序列草案(v1)和假定CDSs和蛋白质序列(v1.1)。基因和重复结构注释可用通用文件格式(GFF3)。数据文件列表包括为全基因组研究生成的从头和参考导向转录组组装,以及之前发表的由我们的研究生成的转录组序列,也可以以FASTA格式访问。此外,筛选后用于基因组分析的RNA-Seq数据和人参基因组规模代谢网络也可以作为SBML文件下载。
结论
人参基因组数据库,人参的原始,全包数据库,建立在基因组序列的最新信息和准确的注释中。它用作开放式接入界面,以从基因组到基因水平检索基因组信息并可视化基因组的所有不同组分。人参基因组数据库将形成一个有价值的资源,增强各种研究领域,如功能/比较基因组学,代谢组科,分子育种和人参的进化分析。
缩写
- 英国石油公司:
-
基对
- CD:
-
编码序列
- 电子商务:
-
酶佣金
- 美国东部时间:
-
表达序列标签
- 维生素:
-
证据建模
- FPKM:
-
每百万片碎片每千碱基碎片
- 人造石铺地面:
-
一般特性的格式
- kb:
-
千濑对
- lncRNA:
-
长不用RNA
- 门店:
-
下一代测序
- TF:
-
转录因子
- TMM:
-
M值的修剪平均值
参考文献
- 1.
王j,高风,张俊,佐,张lm,黄lq。人参皂苷生物合成途径研究进展Panax Ginseng.c·a·迈耶。植物生理学报。2012;34(2):397-403。
- 2.
Saito H,Yoshida Y,Takagi K.效果人参根对力竭运动小鼠。日本Ĵ药理学。1974; 24(1):119-27。
- 3.
彭东,王浩,曲春,谢磊,Wicks SM,谢军。人参皂苷的化学、代谢和药代动力学。下巴医学。2012;2。
- 4.
吴佳,袁春生。人参药理:多成分、多作用。58生物化学杂志。1999;(11):1685 - 93。
- 5。
汤伟,杨勇,周玲,姜波,金华,陈明。人参皂苷Rb1通过胰岛素样信号通路促进脂肪细胞对葡萄糖的吸收。J性。2008;198(3):561 - 9。
- 6。
Radad K,Gille G,Liu L,Rausch Wd。人参在医学中的用途重点在于神经变性障碍。J pharmacol sci。2006; 100(3):175-86。
- 7。
Choi HI, Waminal NE, Park HM, Kim NH, Choi BS, Park M, Choi D, Lim YP, Kwon SJ, Park BS,等。主要重复成分占人参的三分之一(Panax Ginseng.C.A.Meyer)基因组和同种异体中的证据。工厂J. 2014; 77(6):906-16。
- 8。
Waminal NE, Park HM, Ryu KB, Kim JH, Yang TJ, Kim HH。核型分析Panax Ginseng.C.a.meyer,1843(Araliaceae)基于RDNA基因座和DAPI带分布。Comp cytogenet。2012; 15:425-41。
- 9.
Jayakodi M, Lee SC, Park HS, Jang W, Lee YS, Choi BS, Nah GJ, Kim DS, Natesan S, Sun C,等。转录组分析和比较分析Panax Ginseng.不定根根。j ginseng res。2014; 38(4):278-88。
- 10.
Jayakodi M,Lee Sc,Lee Ys,Park HS,Kim NH,Jang W,Lee Ho,Joh Hj,Yang TJ。综合分析Panax Ginseng.根转录om。BMC植物BIOL。2015; 15(1):138。
- 11.
Lee Y,Park Hs,Lee Dk,Jayakodi M,Kim NH,Koo Hj,Lee Sc,Kim Yj,Kwon SW,杨TJ。五个人参品种的整合转录组和代谢组分揭示了人参皂苷生物合成的动态。前植物SCI。2017; 8:1048。
- 12.
关键词:人参皂苷,生物合成基因,微rna,序列重复,转录组分析Panax Ginseng.c·a·迈耶。BMC基因组学。2013;14:245。
- 13。
Kim K, Lee SC, Lee J, Lee HO, johj, Kim NH, Park HS, Yang TJ。人参叶绿体基因组和45S nrdna遗传多样性的综合研究。《公共科学图书馆•综合》。2015;10:e0117159。
- 14。
Kim K,Lee Sc,Lee J,Yu Y,Yang K,Choi Bs,Koh Hj,Waminal Ne,Choi Hi,Kim NH。完全叶绿体和核糖体序列30种载体阐明菊族AA基因组物种的演变。SCI REP。2015; 5:15655。
- 15.
Kim K,Nguyen VB,Dong Jz,Wang Y,Park Jy,Lee Sc,杨TJ。从完全叶绿体基因组中推断的Araliaceae家族的演变和10个NRDNA人参有关的物种。Sci众议员2017;7:4917。
- 16。
阮vb, Park HS, Lee SC, Lee J, Park JY, Yang TJ。身份验证标记主要有五种人参通过叶绿体全基因组序列的比较分析发展的物种。中国农业科学。2017;65(30):6298-306。
- 17。
DNA条形码、微阵列和下一代测序:药用植物遗传多样性评估和鉴定的最新工具。生物技术进展。2016;36(2):191-203。
- 18。
Kim NH, Jayakodi M, Lee SC, Choi BS, Jang W, Lee J, Kim HH, Waminal NE, Lakshmanan M, Binh NV,等。需要遮荫的药草的基因组和进化Panax Ginseng..生物技术学报2018;https://doi.org/10.1111/pbi.12926.
- 19.
Skinner ME, Uzilov AV, Stein LD, Mungall CJ, Holmes IH。JBrowse:下一代基因组浏览器。基因组研究》2009;19(9):1630 - 8。
- 20.
Smit A, Hubley R. RepeatModeler Open-1.0。在:重复遮罩网站;2010.
- 21.
SMIT AF,Hubley R,Green P:RepectMasker Open-3.0。1996年。
- 22.
Haas BJ, Salzberg SL, Zhu W, Pertea M, Allen JE, Orvis J, White O, Buell CR, Wortman JR. .使用evencemodeler和程序组装拼接对齐的真核生物基因结构自动注释。基因组医学杂志。2008;9 (1):R7。
- 23.
Hoff KJ, Lange S, Lomsadze A, Borodovsky M, Stanke M. BRAKER1: genmark - et和AUGUSTUS的非监督rna - seq基因组注释。生物信息学。2015;32:767-9。
- 24.
Haas Bj,Delcher Al,Mount Sm,Wortman Jr,Smith JR RK,Hannick Li,Maiti R,Ronning Cm,Rusch DB,Town CD。利用最大转录对准组件改善拟南芥基因组注释。核酸RES。2003; 31(19):5654-66。
- 25。
猎人S,Apweiler R,Attwood TK,Bairoch A,Bateman A,Binns D,Bork P,Das U,Daugherty L,Duquenne L. Interpro:综合蛋白签名数据库。核酸RES。2009; 37:D211-5。
- 26。
Conesa A,Gotz S,Garcia-Gomez JM,Terol J,Talon M,Robles M.Baster2Go:功能基因组学研究中的注释,可视化和分析的普遍工具。生物信息学。2005; 21(18):3674-6。
- 27。
守谷Y,伊藤男,奥田S,吉泽AC,Kanehisa M. KAAS:一种自动基因组注释和通路重建服务器。核酸RES。2007; 35:W182-5。
- 28。
李H,Durbin R.快速准确的短读对齐与挖洞轮转变换。生物信息学。2009; 25(14):1754-60。
- 29。
徐辉,罗X,钱Ĵ,彭X,宋Ĵ,钱G,陈军,陈S. FastUniq:快速从头重复去除短配对工具读取。Plos一个。2012; 7:e52249。
- 30.
Kopylova E,的NoéL,Touzet H. SortMeRNA:在metatranscriptomic数据核糖体RNA的快速和准确的筛选。生物信息学。2012; 28(24):3211-7。
- 31。
Patel RK,Jain M.NGS QC Toolkit:用于下一代测序数据的质量控制的工具包。Plos一个。2012; 7(2):E30619。
- 32.
Grabherr Mg,Haas Bj,Yassour M,Levin JZ,Thompson Da,Amit I,Adiconis X,Fan L,Raychowdhury R,Zeng Q等。来自RNA-SEQ数据的全长转录组件没有参考基因组。NAT BIOTECHNOL。2011; 29(7):644-52。
- 33.
Pertea M, Pertea GM, Antonescu CM, Chang T-C, Mendell JT, Salzberg SL. StringTie能够从RNA-seq读取改进的转录组重建。生物科技Nat》。2015;33(3):290 - 5。
- 34.
Jin J,张H,Kong L,Gao G,Luo J.Planttfdb 3.0:一种门户网站,用于植物转录因子的功能和进化研究。核酸RES。2013; 42:D1182-7。
- 35.
赵胜,王玲,刘玲,梁艳,孙颖,吴建军。甲戊酸和非甲戊酸5途径参与人参皂苷的生物合成。植物学报2014;33(3):393-400。
- 36.
李B,杜威CN。RSEM:从RNA测序数据精确转录定量具有或不具有参照基因组。BMC生物信息学。2011; 12:323。
- 37。
吴丹丹,渡边CK。GMAP: mRNA和EST序列的基因组定位和比对程序。生物信息学。2005;21(9):1859 - 75。
资金
本研究得到了下一代生物绿色21计划(PJ01311901, PJ01334605)、韩国农村发展管理局和新加坡国立大学合成生物学计划(DPRT/943/09/14)的资助。
可用性数据和材料
人参基因组数据库中的数据集可为非营利性和学术组织的研究目的免费访问http://ginsengdb.snu.ac.kr.此外,所有序列数据存放到国家农业生物技术信息中心(NG-0858-000001〜NG-0858-009845)(http://nabic.rda.go.kr.).数据库针对Internet Explorer、Mozilla Firefox、谷歌Chrome和Safari进行了优化。
作者信息
从属关系
贡献
JM和TJY开发了该方法并进行了研究。JM,BSC,SCL,NK,WJ和ML参与了数据库和生物信息学工具开发。JM,DYL,JYP,SVM和TJY起草了稿件,由所有作者修订。所有作者均阅读并批准最终手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Jayakodi M, Choi, BS。李,SC。等等。人参基因组数据库:对基因组学的开放式接入平台Panax Ginseng..BMC植物BIOL.18,62(2018)。https://doi.org/10.1186/s12870-018-1282-9
收到了:
接受:
发表:
关键词
- Panax Ginseng.
- 基因组数据库
- 人参注释
- 人参基因组浏览器