摘要
背景
对孤儿作物的研究往往因缺乏基因组资源而受阻。随着负担得起的测序技术的出现,对整个基因组或大基因组物种的基因组的代表性部分进行基因分型对任何作物都变得可行。然而,大多数基因测序分型(GBS)方法都是为了获得大量低序列深度的标记,这就排除了它们在杂合个体中的应用。此外,生物信息学管道通常缺乏处理成对末端reads或应用于多倍体物种的灵活性。
结果
UGbS-Flex将公开可用的软件与内部的python和perl脚本相结合,可以有效地从测序基因分型读取snp,而不考虑物种的倍性水平、育种系统和参考基因组的可用性。UGbS-Flex管道值得注意的特性是能够使用成对端读取作为输入,这是一种有效的方法,可以通过增强的输出跨样本进行群集读取,以及最大化SNP调用。我们演示了管道的使用,以识别数千个高可信度snp与高代表性的跨F样本3.派生F2种群中异体四倍体指谷。使用经过时间考验的测绘程序MAPMAKER构建了健壮的高密度遗传图谱,我们将其升级为在Windows命令提示符环境中以半自动化的方式高效运行。我们利用手指小米的一个二倍体祖先的比较GBS,将连锁基团分配到亚基因组,并证明了染色体重排的存在。
结论
这篇论文结合了GBS协议的修改、一种新的灵活的GBS分析管道UGbS-Flex、最大限度地识别SNP的建议、更新的遗传图谱软件和第一张指谷子的高密度图谱。UGbS-Flex管道中使用的模块和用于遗传定位的模块被应用于指谷子,一种没有参考基因组的异体四倍体自交物种,作为案例研究。UGbS-Flex模块可以独立运行,很容易转移到具有其他育种系统或倍性水平的物种。
背景
有效的基因分型方法,无论是用于定位还是用于群体遗传研究,都必须简单可靠,并提供覆盖整个基因组的数千个或更多多态位点的等位基因组成[1].尽管测序技术最近取得了进展,但对大基因组物种来说,全基因组测序仍然不具有成本效益,特别是在需要对数百个个体实现多倍覆盖的情况下。基于限制性内切片段选择性测序的几种简化表达方法已被开发出来,以同时进行高通量标记发现和基因分型[2,3.,4].我们将这些方法统称为“基因测序分型”(GBS)。为了保持低测序成本,通常需要在标记数量和测序深度之间进行权衡。因此,测序基因分型数据集往往存在大量缺失数据,且每个位点的序列覆盖率很低[2,5,6].imputation可用于推断缺失的基因型[7].然而,在分析异交种和双亲本回交的多样性面板时,低序列深度是很有问题的1(异交种)和F2在每个位点上有足够的序列深度是明确区分纯合和杂合等位基因的先决条件。因此,我们对Elshire等人开发的实验性GBS协议进行了一些修改。[2]和波兰等。[8],并测试了它们在减少GBS片段池和为高置信度无imputation单核苷酸多态性(SNP)鉴定提供更均匀的汇总样本读覆盖方面的效果。
如果全基因组序列数据可用,GBS读数可以对齐,那么GBS读数的分析是相当直接的。基于参考的GBS分析管道包括tsel -GBS [9,10], Fast-GBS [11]和堆栈[12].tasser - uneak等管道[5]、堆栈[12]和GBS-SNP-CROP [13可以生成一个新创GBS参考来自实验数据,旨在分析没有参考基因组的物种的GBS数据。大多数管道都是针对来自二倍体生物体的单端测序数据。
手指小米,Eleusine coracana(l)Gaertn。无性系种群。coracana,是一种近交同种异体四倍体(AABB)谷物,属于Chloridoideae亚科,单倍体基因组大小为1.7 Gb [14].尽管它是东非和印度南部部分地区重要的粮食安全作物,但它一直被国际研究界忽视。指谷的野生祖先,大肠coracana无性系种群。非洲象,起源于杂交之间大肠籼(AA基因组)和一个未知的b基因组物种。异源多倍体化事件发生的时间尚不清楚。迄今为止,仅生成了由332个位点组成的单一连锁图谱,主要由限制性片段长度多态性(限制性片段长度多态性,RFLP)标记和单链构象多态性表达序列标记检测[15,16].该映射是在F2由野生品系MD-20和栽培品系Okhale-1杂交而来的种群。基于a -基因组中RFLP片段大小的相似性大肠籼而且大肠coracana,连锁基团被分配到亚基因组[15].该连锁图谱还用于建立指谷和水稻基因组之间的总体比较关系[16].目前需要高密度的小米SNP图谱来辅助性状分析和计划中的基因组测序工作。
即使只有不到10,000个标记,构建一个精确的连锁图也是极具挑战性的,特别是当处理不太完美的数据集时,例如,在F .3.重组F2人群。传统软件如MAPMAKER [17,18]及JoinMap [19,20.]使用基于最大似然的三点和多点分析,可以提供高度精确的标记排序,但对于大型数据集来说是高度内存和时间密集型的。为了处理大量标记,MSTmap [21]及Lep-Map [22基于旅行推销员原则而开发出来的。地图生成速度快,但标记排序对基因分型错误更敏感。为了能够利用MAPMAKER订购大型标记集的高精度,我们修改了原始的MAPMAKER包,使其在Windows命令提示符环境中有效运行,并开发了内部python脚本来自动化MAPMAKER映射过程的几个步骤。应用该图谱管道生成了由数千个高质量SNP标记组成的新的指谷子高密度遗传图谱。
因此,我们的论文提供了一种改进的GBS协议,一种新的管道(UGbS-Flex),用于分析配对端GBS数据,适用于具有不同倍性水平、育种系统和多态性水平的物种,而不考虑参考基因组序列的可用性。我们还为gbs后的数据分析提供了一个全面的解决方案,以及一个高密度的手指谷子遗传图谱,提供了关于异体四倍体手指谷子基因组组织的新信息。
方法
植物材料和DNA提取
F2映射种群是由之间的交叉生成的大肠coracana无性系种群。非洲象加入MD-20和大肠coracana无性系种群。coracana接入奥克海尔-1 [15].一百三十四2:3每株F3.这个家庭和父母是在佐治亚大学的温室里,昼夜温度在26-30°C。KNE 796,其全基因组测序正在进行中,以及大肠籼在肯尼亚野外采集的Ei-0、Ei-2和Ei-5在同样的条件下生长。所有种质都是按照国家和国际关于用于研究目的的种子进出口协定获得的。DNA提取采用改良的CTAB方法,改良的CTAB方法来自Doyle和Doyle [23]从每棵F3.家苗从八周大。在Nanodrop (Thermo Scientific)上测量DNA浓度,并将样品稀释至50 ng/μl。DNA质量在0.8%琼脂糖凝胶上进行评估。
GBS样品制备
200纳克的高分子量DNA被任意一种混合的混合物消化太平洋标准时间我/Msp我,太平洋标准时间我/濒死经历我或太平洋标准时间我/Msp我+猿吻。消化量为30 μl,温度为4u太平洋标准时间I-HF和8uMsp我在NEB CutSmart缓冲器太平洋标准时间我/Msp我消化,4个你太平洋标准时间I, 8 uMsp我和你猿KI在NEB-buffer 3.1用于太平洋标准时间我/Msp我加猿KI消化,和4 U太平洋标准时间I-HF和4u濒死经历我在NEB CutSmart缓冲器太平洋标准时间我/濒死经历我来进行。样品在37°C下孵育2小时。然后在75°C下对含有该酶的反应混合物再孵育2小时猿吻。样品没有猿KI在75°C下孵育20分钟以灭活限制性内切酶。
20微升限溶物与1 μl条形码混合太平洋标准时间I适配器(原装:0.1 μl),常见1.5 μlMsp我或濒死经历I型y型接头(原液:10 μM)、4 μl 10X T4-DNA连接酶缓冲液和200 U T4 DNA连接酶(NEB),总体积为40 μl。常见的y型适配器和条形码适配器由Poland等人描述。[8].在22°C下结缔组织2小时。结缔组织完成后,用0.7体积根据Rohland和Reich制备的Sera-Mag SpeedBeads (GE Healthcare Life Sciences)孵育样品,去除小于300 bp的片段。24],室温下5 min。磁架分离上清液,用200 μl新鲜制备的70%乙醇洗涤3次。用40 μl 10 mM Tris从风干珠中洗脱DNA。HCl (pH 8.0)。
将得到的3微升洗脱液加入16 μl H的鸡尾酒中2O、5 μl 5X Taq主混合液(NEB)、0.5 μl针对条形码适配器的正向引物(原液:10 μM)和0.5 μl与通用适配器同源的反向引物(原液:10 μl)。分别对每个样品进行PCR扩增,初始变性在95°C下30秒,变性在95°C下30秒,16个变性循环,引物在62°C下退火20秒,片段延伸在68°C下15秒,然后最后片段延伸步骤在68°C下5分钟。在1.5%琼脂糖凝胶上检测8微升PCR产物。每个GBS文库的DNA浓度使用Qubit™dsDNA HS检测试剂盒在Qubit 2.0上测量。只有浓度为> 5.0 ng/μl的GBS库被测序。每个GBS库中有30纳克。收集的样本数量取决于所使用的测序平台;我们的目标是每个样本获得200万个阅读量。用0.7体积的AMPure Beads或Sera-Mag SpeedBeads从DNA池中去除引物、dNTP和小DNA片段。合并的GBS文库(100 ng)在Illumina NextSeq平台上测序,对端reads为150 bp。家长和115楼2:3样品测序作为同一测序操作的一部分。另外3个和26个F2:3样本测序作为两次单独的NextSeq运行的一部分。从独立生成的库中对10个样本进行了重复测序,以确保库和运行之间的一致性。
GBS分析管道具有可选性新创引用的生成
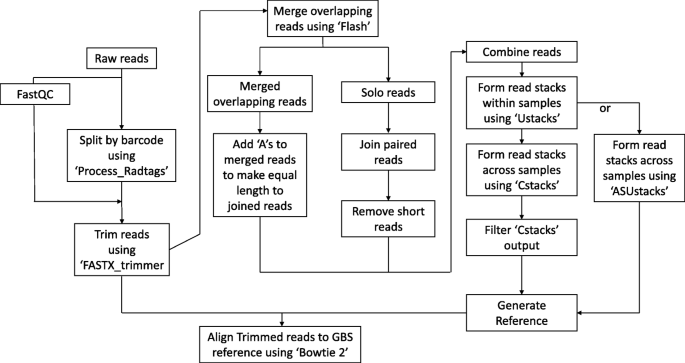
下面描述了完整的UGbS-Flex管道。在UGbS-Flex管道中使用的所有内部perl和python脚本及其使用信息都在“程序和脚本”中提供http://research.franklin.uga.edu/devoslab/.关于如何应用UGbS-Flex管道的详细信息在附加文件中给出1:数据S1和附加文件2:图S1。
读取的预处理
使用' FastQC ' v. 0.11.4检查读取质量[25].读取被条形码分割,使用' Stacks '程序中的' Process_Radtags '模块[12选项-r(救援条形码和rad标签)。前向读取如果同时携带条形码和太平洋标准时间我限制网站。由于在一些Illumina NextSeq测序中,反读的前几个碱基质量较低,因此没有对第二酶的酶切位点进行选择(Msp我或濒死经历I)反向读。' FASTX_trimmer '从' FASTX Toolkit '包(http://hannonlab.cshl.edu/fastx_toolkit/)用于删除(1)限制位点,(2)每次读取的最后5个bp(通常)更有可能包含错误(或者,对于低质量运行,读取的3 '端FastQC质量评分低于20的所有碱基)以及(3)正向读取的3 '端额外的0(对于10 bp条形码)到5个碱基(对于5 bp条形码),以使所有读取的长度相同。相同的读取长度是' Stacks '程序的先决条件[12用于生成a新创参考从GBS读取。
新创生成一个GBS引用
为了在生成GBS引用期间方便处理成对的末端读取,使用' Flash '合并重叠的正向和反向读取[26].从非重叠的读取文件中,我们通过运行python脚本' EL.1.2.py '删除了任何比预期(修剪)大小短的读取文件。如果反向读取被移除,相应的正向读取也会被移除反之亦然.' EL.1.2.py '脚本然后反向补充其余读对中的非重叠反向读取,并人为地将它们连接到相应的正向读取的3 '端。在正向读和反向补反读的交界处未加n。因为通过' ustacks ' [12]要求相同长度的读取,' EL.1.2.py '也在3 '端用' As '扩展合并的重叠读取,使它们与连接的非重叠读取长度相同。a延伸的重叠片段通常对所有样品都是共同的,因此polyA束不会产生多态性。每个样本中的读取都使用' Stacks '程序中的' ustacks '模块(选项:-m 2 - m1 -N 1)进行聚类[12].堆栈中的' cstacks '模块(选项:- b1 -n 1)用于通过聚类从' ustacks '获得的两个父类和117 F的读堆栈来生成一组有代表性的标签2后代。只是F的一个子集2样本被包括在“cstacks”分析中,因为运行具有大量样本的“cstacks”需要很高的内存。我们还测试并验证了一种替代方法,称为“跨样本ustacks”(“ASustacks”)来取代“cstacks”。使用内部python脚本,“ustacks”在每个样本中生成的共识序列被提取出来,给出一个包含样本标识符的唯一名称,并通过向每个共识序列添加由Es组成的序列质量行,将其放置在一个人工fastq文件中。' ustacks '模块然后使用与' cstacks '中应用的参数应用于此文件。形成堆栈所需的最小读取数(−m)设置为1。图中显示了使用UGbS-Flex管道生成GBS引用所涉及的步骤的概述。1.
筛选GBS代表性标记以生成引用
对通过“cstacks”或“ASustacks”在样本中识别的代表性标签进行了两个过滤步骤(图2)。1)。首先,在' cstacks '输出中,使用内部perl脚本' FCT.pl '删除了出现在低于用户定义的样本百分比(本研究中为50%和70%)的代表性标签。这个过滤步骤已经集成到“ASustacks”模块中。其次,我们删除了与另一个代表性标签的相似程度等于或高于用户定义的百分比(本研究为98%)的代表性标签。为了实现这一点,对共识标签进行了全面的分析(e值阈值:10e−5)。对于同源性≥98%的标签,使用python脚本' Ref_98.py '只保留一个标签。剩下的代表性标签被用作GBS参考。
SNP/indel调用和过滤
预处理读使用Bowtie 2 v. 2.2.0与默认参数的GBS引用对齐[27].如果全基因组参考序列可用,新创可以省略GBS参考基因的生成,并将修剪过的reads对准参考基因组。对于SNP/indel呼叫,我们在GATK套件中测试了“统一基因型(GATK v. 3.4.0)”(参数-dcov 1000,−glm both)和“单倍型Caller (GATK v. 3.4.0)”(参数——genotyping_mode DISCOVERY -stand_emit_conf 10 -stand_call_conf 30 -minPruning 1 -emitRefConfidence GVCF)模块[28].为了简单起见,我们使用术语“SNPs”来涵盖snp和Indels。在GATK中过滤原始snp,仅保留等位基因频率在10 - 90%之间的双等位基因snp。我们还删除了相邻的snp(脚本' SNPs_ISL.pl '和' Rm_adj_SNPs.pl '),因为其中一些snp是由正向读取和反向读取交界处的错位引起的人工产物。然后使用GATK .vcf文件中提供的等位基因深度(AD)信息对每个位点上snp的等位基因状态进行评分(脚本' snp_genotype .py ')。总AD < 8的位点被记为缺失数据点(−)。带有广告的基因位点裁判(erence等位基因)/广告alt (ernate等位基因)比例≥10的分为A(亲本1等位基因纯合)、AD裁判/广告alt≤0.10为B, 10> AD裁判/广告alt> 4作为D(模棱两可的A或杂合(H))和0.25 > AD裁判/广告alt> 0.1为C(模棱两可的B或H),其他比例的位点记为H2由两个近交系亲本产生的群体,如指小米的情况,父母中不同等位基因的所有非纯合的snp都被去除(脚本' snp_selectbypparent .py ')。在超过30%的样品中缺失或A/B比值< 10%或> 90%的snp也被去除。这是在Excel中手动完成的。同样,缺失数据超过30%的样本被移除。
UGbS-flex和GBS-SNP-CROP管道的比较
比较UGbS-Flex和GBS-SNP-CROP的性能[13Melo等人使用的48个四倍体猕猴桃基因型的原始序列数据。[13]已从NCBI (SRR2296676)下载。在fastQC报告的指导下,我们将正向和反向读数削减到121 bp。用于“ustacks”的参数为- m2 - m2 - n4,用于“ASustacks”的参数为- m1 - m2 - n4。我们选择了与Melo及其同事相同的缺失数据阈值(25%)和SNP评分的测序深度[13].h阈值设置为4。
基因映射
通过合并位于相同GBS标签中的snp来减少数据集
位于相同GBS参考标签内且处于连锁不平衡状态的SNP位点信息被整合,以进一步提高映射评分的稳健性(函数包含在' snp_genotype .py '中)。如果一个代表性标签内的基因座得分为' a ', ' D '和' - '的组合,则合并得分为' a '(表1)。同样,“B”、“C”和“-”的组合被合并为“B”,“H”、“C”、“D”和“-”的组合被合并为H。如果一个GBS代表性标签中的所有位点都被评分为“-”、“C”或“D”,这些分数被保留。冲突的分数(A和B, A和C, A和H, B和H,以及B和D)被标记为“F”,并被视为地图生成中的缺失数据。
通过删除共隔离标记来减少数据集
为了生成一组用于基因图谱构建的高质量非冗余标记,每个SNP标记都被给予一个“C”或“D”(惩罚= 1)或一个缺失的数据点(惩罚= 2)的惩罚分数。使用内部python脚本“snp_cosegg .py”,使用贪婪算法在所有对所有分析中比较所有SNP标记的映射分数。筛选出多位点基因型相同的SNP标记,并选取每组中惩罚分数最小的标记进行作图。
基因图谱构建
为了构建基因图谱,我们删除了超过20%的后代中所有缺失数据的snp。利用MSTmap将SNP标记集拆分为连锁组[21].使用MST map建立初始映射顺序,并使用MAPMAKER检查双重组事件(改编自[18];改编版本可从http://research.franklin.uga.edu/devoslab/).去除双重组事件超过定义数量(本研究为4个)的标记。重复MSTmap映射和检查双重组事件的过程,直到每个标记与其侧翼标记之间的双重组事件数≤4。相应的MSTmap地图被用作使用MAPMAKER生成地图的起点(改编自[18];改编版本可从http://research.franklin.uga.edu/devoslab/)。
由于固有的程序限制,使用的MAPMAKER版本被限制为订购~ 100个标记,具有超过100个标记的MSTmap映射被分成60到100个标记的更小的子组。亚组由40个标记重叠。使用“order”和“try”命令为每个子组构建遗传图谱。基于重叠段的共同标记顺序合并子组映射。每个链接组再次被分割为< 100个标记的子组,并且使用' ripple '命令进一步调整标记顺序。' Try '和' ripple '分别使用' Try .py '和' ripple.py '脚本以半自动化的方式完成。最终标记顺序在子组间合并。遗传图谱距离(在Kosambi中)使用MAPMAKER中“错误检测开启”的“map”命令获得。地图订单是人工审查的,如有必要,还会进一步调整。将具有相同多位点基因型的标记添加到图谱中,与它们的代表性标记进行共分离。 Finally, we placed markers with ambiguous orders (not separated by clear recombination events) in bins.
鉴定a和B基因组连锁群
阅读三篇大肠籼采用Bowtie 2 v. 2.2.9和默认参数,将AA基因组与GBS参考标签进行比对[27].存在感(至少出现在三种类型中的两种大肠籼资料分析)和缺席(三个方面都缺席)大肠籼的GBS标记大肠籼用Excel散点图沿着遗传图谱的长度绘制基因组。与大肠籼标签沿着它们的整个长度被分配到A基因组和那些基本上没有的基因组大肠籼标签被分配到b基因组。
结果
不同酶组合产生多态标记的效率
我们测试了两种双酶组合(太平洋标准时间我/Msp我和太平洋标准时间我/濒死经历I)和一个三酶组合(太平洋标准时间我/Msp我+猿KI)对三个指状谷子资源进行了研究,因为它们能有效地生成大量重叠的片段池,当测序时,在所有三个资源中至少8×深度都产生了snp。所有样品在Illumina NextSeq平台上测序(配对端150 bp)。9个样品/酶组合(3个接入,3个酶组合)中的每一个获得的读取数在附加文件中给出3.:表S1。为了估计读取深度对数据的影响新创在生成GBS参考时,我们用我们新开发的UGbS-Flex管道分析了每个接入/酶组合的20万(M)、0.5 M、1 M、2 M和3 M配对端reads的子集(图2)。1)。较小的读数是较大读集的子集。
对于酶的组合太平洋标准时间我/Msp我和太平洋标准时间我/Msp我加猿KI,正如预期的那样,测试的所有三种访问所共有的GBS标记的数量随着总读取数的增加而增加,在2m读取左右达到一个平台(附加文件3.:表S1“通过酶组合”)。对于酶的组合太平洋标准时间我/濒死经历然而,我,普通GBS标签的总读取次数从0.2 M增加到1 M,但当总读取次数从1 M增加到3 M时,又减少了。12我们开发了脚本' across-sample ustacks ' (' ASustacks ')。“ASustacks”从每个样本的“ustacks”输出中生成一个人工的fastq文件,这些文件被用作“ustacks”的输入。“ASustacks”方法产生的参考标签数量与“cstacks”相似,除了酶组合中的读取数≥1 M太平洋标准时间我/濒死经历I.我们现在看到,随着所有酶组合的读取数增加,常见GBS标记的预期增加。超过97%的被识别为“cstacks”的GBS参考标签也在使用“ASustacks”生成的参考中找到。有趣的是,由“cstacks”和“ASustacks”识别的GBS参考标签的读取深度明显低于由“ASustacks”唯一识别的GBS参考标签的读取深度(附加文件4:表S2)。这表明高读取深度阻碍了“cstacks”的性能,可能是因为更高的读取深度导致了更高的snp绝对存在,这是由PCR或等位基因读取中的测序错误引起的。“cstacks”模块可能已经消除了这些可能由重复DNA组成的集群。我们对“ASustacks”在3 M reads子集中识别的所有手指小米参考标签进行了blast分析太平洋标准时间我/濒死经历我在重复掩盖的米饭上消化样品(栽培稻)基因组。30%的“cstacks”和“ASustacks”共有的标签被识别出爆击(e值阈值为e-5), 37%的“ASustacks”唯一识别的标签被识别出爆击。这表明被' cstacks '消除的标签没有被重复充实。为了进一步验证“ASustacks”方法,我们比较了由“cstacks”和“ASustacks”生成的GBS数据中的读簇,这些数据来自一组96种不同的狐尾百合系的DNAEremurus用太平洋标准时间我/MspI.狐尾百合基因组约7.9 Gb (1C) (I. Leitch, pers。通讯)。基于“cstacks”和“ASustacks”的至少50%的行中共有的标签数量分别为376个和3552个,98%的“cstacks”集群出现在“ASustacks”输出中。除了产生更多的参考标签之外,“ASustacks”方法在计算上比“cstacks”方法要少得多。
为了估计不同的酶对减少测序片段池的影响,我们比较了在所有三种被测试的资料中存在的多态性GBS标签的数量,以及在三种酶组合中识别的snp的数量(表2)2;额外的文件3.:表S1 ' By Read Number ')。SNP评分的最小读取深度为8×。第三种酶的使用大大减少了GBS参考标签的数量,因此识别出的SNP的数量,但与我们的预期相反,只略微增加了SNP的读取深度(表2)。百分比猿位于太平洋标准时间我/Msp我读到的数据是太平洋标准时间我/Msp我+猿KI读取集,并可与GBS中找到的参考标签(附加文件5:表S3)。然而,在三重文摘中,猿与读取相比,包含ki的读取在GBS参考中严重不足(附加文件5:表S3)。这表明猿三重摘要中包含ki的读不能聚类。它们很可能起源于适配器-结扎步骤,通过随机结扎猿KI片段来自不同的基因组区域。通过酶联,在最小读深为8×的条件下,三个样本中多态性snp的数量最高太平洋标准时间我/MspI用于读数≥1m和带太平洋标准时间我/濒死经历I当每个样本测序的片段数≤50万个时(附加文件3.:表S1 ' By Read Number ')。
在MD-20 x Okhale-1映射种群中生成GBS参考
146个样本共获得278,880,767对末端reads (SRA研究)SRP136342)。使用适用于SNP巩固的规则合并重复样本的基因型得分。大约2%的snp在重复的样本之间不一致,并作为缺失数据输入。每个样本的平均阅读数为1,910,142,中位数为1,317,595。一个读数少于60万的样本被从分析中移除。在进行“cstacks”分析后,至少50%的样本(以下称为Ref50)中出现的代表性GBS标记的数量为34,960个(表3.)。对于至少70%的样本(Ref70)中存在的标记,这一数字下降到16,725。在去除同源性≥98%的代表性标签后,在Ref50参考文献(Ref50_98)中保留了28,579个标签,在Ref70参考文献(Ref70_98)中保留了15,397个标签。
SNP打电话
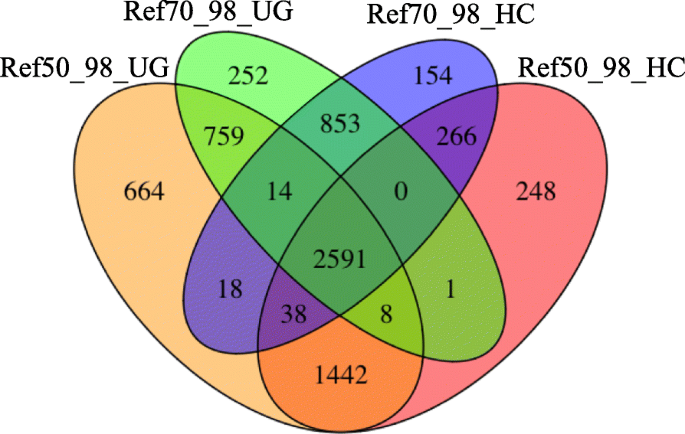
修剪后的读对生成的Ref50、Ref50_98、Ref70和Ref70_98 GBS引用进行对齐,并将这些对齐用于SNP调用。采用GATK的统一基因型和单倍型调用器,并对其输出进行比较。表中给出了每个SNP调用者/对齐组合携带SNP的参考GBS标签的数量3..图中显示了不同引用和不同SNP调用者共有的含SNP的GBS标签的数量。2和附加文件6:图S2在构建遗传图谱时,将使用统一基因型和单倍型调用器识别的SNPs与参考文献Ref50_98和Ref70_98进行合并,共得到17245个SNPs,分布在7307个标签上。将位于同一标签上的SNP合并为每个标签上的一个一致SNP,将SNP(标签)的数量减少到7125。总共有182个标签被移除,因为它们在20%或更多的后代中携带了与基因型得分冲突的snp。位于同一GBS标记上的snp之间的大量冲突得分可能是由于A和B基因组读对同一GBS参考标记造成的。
UGbS-flex和GBS-SNP-CROP管道的比较
使用与Melo及其同事报告的相同数据集,并尽可能使用相同的阈值[13], UGbS-Flex在过滤前共获得86,810个snp,而Melo及其同事在GBS-SNP-CROP中报告的结果为56,598个7:表S4)。过滤后,UGbS-Flex管道保留的snp总数为50139,而GBS-SNP-CROP保留的snp总数为213187:表S4)。GBS-SNP-CROP管道报告的SNP号是指在生成引用时使用了读数最多的单个接入。当所有48份材料都被使用时,GBS-SNP-CROP筛选得到的SNPs总数为14,712。我们使用所有48个访问用UGbS-Flex生成引用。
基因映射
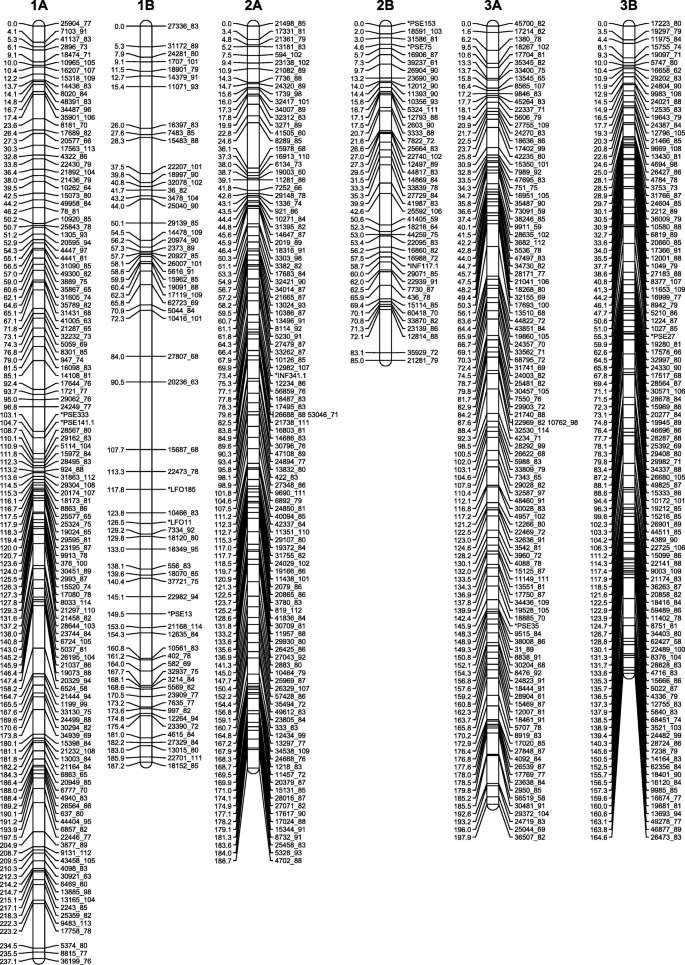
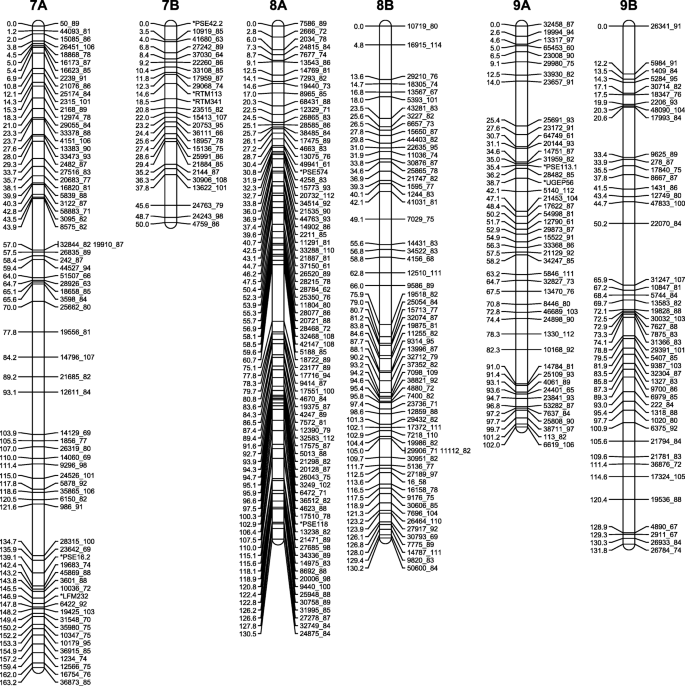
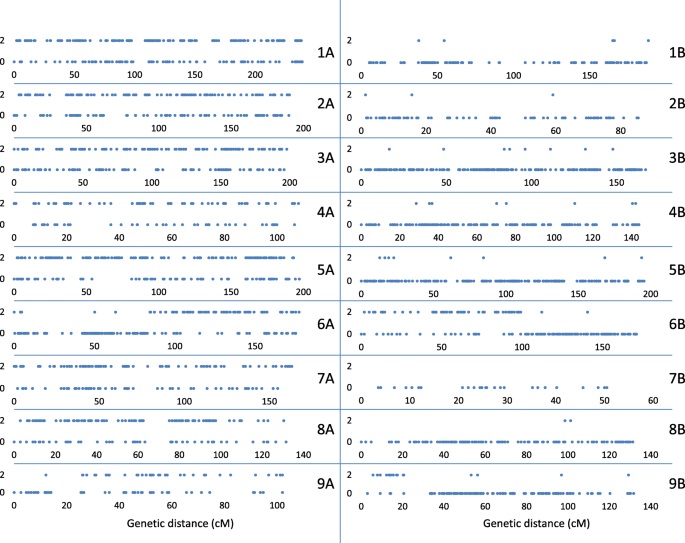
由于一些扭曲的标记导致了假键,我们最初删除了偏析比偏离1:2:1 (A:H:B)的所有标记。我们还删除了同隔离标记,以减少地图构建过程中的标记负载。在最初的地图生成之后,三组以高LOD分数连接在一起并扩展初步地图的扭曲标记被重新添加到数据集中,以生成最终的地图。这些图谱共包含3772个SNP标记,分布在18个连锁群中,每个连锁群的标记数量从39 (51 cM)到301 (240 cM)不等(图2)。3.,4而且5、表4,附加文件8:表S5)。对共分离标记进行积分后,映射的标记总数为4453(附加文件9:表S6)。每个染色体的重组箱数量从25到120不等(附加文件)8:表S5和附加文件9:表S6)。所映射的GBS标签的序列以及这些标签中的SNP位置在附加文件中提供9:表S6。
指谷子的高密度遗传图谱(同源类群1、2、3),右侧为标记名称,左侧为centiMorgan (Kosambi)距离。为了可读性,在地图上只表示每个标记库的第一个标记。所有标记的位置可从附加文件9表S6
指谷子(4、5、6同源类群)的高密度遗传图谱,右侧为标记名称,左侧为centiMorgan (Kosambi)距离。为了可读性,在地图上只表示每个标记库的第一个标记。所有标记的位置可从附加文件9表S6
指谷子的高密度遗传图谱(同源类群7、8、9),右侧为标记名称,左侧为centiMorgan (Kosambi)距离。为了可读性,在地图上只表示每个标记库的第一个标记。所有标记的位置可从附加文件9表S6
将连锁基团分配到a和B亚基因组
大约13%的地图大肠coracanaGBS标签,相应的GBS读数在三者中都被识别出来大肠籼登记入册分析。另外14%的映射标签在三种分析中的两种中都有体现大肠籼而18%的人只出现在一个单一的大肠籼加入。Excel散点图显示了所有三种GBS标签的分布大肠籼至少在三个中有两个出现大肠籼18所大学均有上榜大肠coracana连杆群如图所示。6.9个同源组中的7个染色体被明确地分配到A或B亚基因组。在同源组6和9中鉴定了A/B易位。
讨论
GBS工艺优化
我们测试了Elshire等人开发的实验性GBS协议的几个修改。[2]和波兰等。[8].目的是减少测序的片段池,并在池中的样本中提供更均匀的读取覆盖,以增加每个位点的读取深度和样本中的SNP代表性。通过在PCR步骤中防止将Illumina测序适配器添加到DNA片段的子集,可以减少将要测序的片段数量。这可以使用具有一个或多个选择性碱基的引物来实现,如在可调GBS (tGBS®,Data2Bio, ISU Research Park, Iowa)中应用的引物,或通过使用限制性内切酶切割pcr扩增片段来实现。我们在指谷(一种近交四倍体物种)中率先采用了后一种方法。对3份材料的DNA进行双酶解太平洋标准时间我/Msp我或太平洋标准时间我/濒死经历I,或者说通过加法来消化三倍猿KI到太平洋标准时间我/Msp我消化。太平洋标准时间我和濒死经历我是6个bp的切割机,MspI是一个4-bp切割机和猿KI是一个5 bp的切割机。太平洋标准时间我,濒死经历我和猿KI对CNG甲基化敏感。第三种酶产生的位点没有连接。虽然使用第三种酶确实显著减少了片段池,但在适配器-连接步骤中,起源于基因组不同部分的片段之间的随机连接导致嵌合片段。这些片段已测序,但在系内或系间均未对齐。因此,使用三种酶的混合并不比双酶消化有优势。在连接适配器之后,第三种酶可能会更有效地使用,但这将需要GBS协议中的一个额外步骤。使用濒死经历双摘要中的6 bp切割机I更适合低读数(50万或更少),因为较小的片段池允许测序更多样本中常见的片段,深度至少为8倍。对于每个样本的目标读取数在1 M到2 M范围内,并且SNP评分在最小读取深度为8×时,我们建议使用太平洋标准时间我/Mspi。太平洋标准时间我/MspI组合生成的片段池比太平洋标准时间我/濒死经历但是这个片段池仍然足够小,以至于许多样本中常见的片段被测序到所需的8×深度。对于生成的片段池来说,获得超过2m的读操作是不划算的太平洋标准时间我/ MspI(附加文件3.:表S1)。
我们在协议中添加了两个大小选择步骤。使用Sera-Mag SpeedBeads将适配器连接到DNA片段后,去除小于300 bp的片段。在PCR步骤中,引物延伸时间保持在15 s,这在很大程度上限制了扩增到小于800 bp的片段。此外,我们加入了一个修改,以获得更均匀的读取数跨池样本。在原协议中[2],在PCR步骤前将样品混合。Elshire和同事们最初注意到样品之间的读数有很大的变化,他们认为这是由于液体处理系统的移液不一致造成的,并通过调整系统来纠正[2].我们还看到了一个池内不同样本之间读取数的显著差异。虽然我们的目标是每个样本获得200万次阅读,但在我们早期的GBS实验中,阅读数从103,000到接近1300万不等(图2)。7)。人工移液的变化可能是一个影响因素,但样本之间在适配器连接效率方面的变化也可能起作用。因此,我们分别对样本进行PCR扩增,并在量子比特荧光计上测量DNA浓度,然后将等量的DNA汇集在一起。DNA浓度低于5 ng/μl的样品被丢弃,因为即使调整了加入池的样品量,也会导致低读数。此外,这些样本通常不能仅通过重做PCR步骤来挽救,而必须从头开始重做,这表明问题在于消化或适配器-连接步骤。这种修改的引入大大缩小了读取数字的范围(图。7)。虽然这种方法增加了样品制备的成本,但它允许在测序前识别“坏样品”,因此,在测序端节省了成本。
生成GBS引用
我们想要一个GBS方案和分析管道,可以在各种物种中提供数千个强大的SNP标记,而不考虑育种系统、倍性水平、多态性水平和全基因组序列的可用性。为了最大限度地提高在未知或低变异水平的物种中发现SNP变异的机会,我们选择了从两端(一个NextSeq上2 × 150 bp)对减少的表示库进行测序。此外,较长的配对末端reads更有可能识别相关物种的同源序列,比较信息的可用性是研究遗传和基因组资源较少的孤儿作物的关键。而一些管道(例如tasser - gbs [10],堆栈[12]),当我们开始从GBS标签构建基因组参考的分析时,没有一个可以处理成对末端读取,也没有一个可以用于多倍体物种的数据.最近,GBS-SNP-CROP管道的发布解决了这些缺陷[13].我们提出了一种替代管道UGbS-Flex,它可以从近交二倍体和异交异体多倍体的配对端数据生成GBS参考。这个直观而灵活的管道由公开可用的软件包和内部perl和python脚本组成,涵盖从读取处理到SNP评分的数据分析(图2)。1)。每个UGbS-Flex模块都可以独立运行。
UGbS-Flex管道开发的大部分工作都是使用F2指谷子种群。然而,我们已经成功地使用UGbS-Flex管道分析了指谷子和狐尾百合多样性面板生成的GBS数据,并绘制了海滨雀稗(一种异交二倍体)和柳枝稷(一种异交四倍体)的种群图。为了方便处理成对端数据,我们合并了重叠序列,并将非重叠的成对端读取视为连续序列。然后,我们使用STACKs包中的' ustacks '模块,根据样本内的相似度对读取进行分组[12],并在样本之间使用“cstacks”或新颖的“ASustacks”方法。后者将“ustacks”应用于包含所有样本中每个样本读取集群的共识序列的文件。由于“ASustacks”方法在计算资源和已识别的GBS参考标签数量方面更有效,因此已集成到UGbS-Flex管道中,用于分析使用甲基化敏感限制性内切酶生成的简化表示测序数据,从而丰富了低复制序列。
最佳的“ustacks”和“cstacks/“ASustacks”参数取决于所研究物种的育种系统、倍性和多态性水平。例如,在一个近交物种中,大多数位点是纯合的,并且在一个附加序列中应该建立不包含或仅包含一个不匹配的reads的堆栈。在近交系个体中使用非常严格的堆叠构建条件将促进准亲缘系的分离,在多倍体中,将促进同源位点的分离。然而,在异交多倍体物种中,需要在亲本内产生包含同源等位基因而不是同源等位基因的堆栈。找到实现这一目标的最佳参数可能具有挑战性,特别是在高度多态的多倍体中,需要根据经验来完成。在跨访问生成堆栈时,需要考虑类似的考虑。对于指谷子,我们允许样本内和样本间的聚类不匹配为1 bp。手指小米的多态性水平相对较低,因为我们研究的是F2总体上,样本内和样本间的等位基因变异相似。“cstacks”/“ASustacks”输出被过滤,只保留那些在特定比例的样本中存在的一致序列,这些序列被用作参考。这一步限制了具有大量缺失数据的snp的下游识别。
SNP打电话
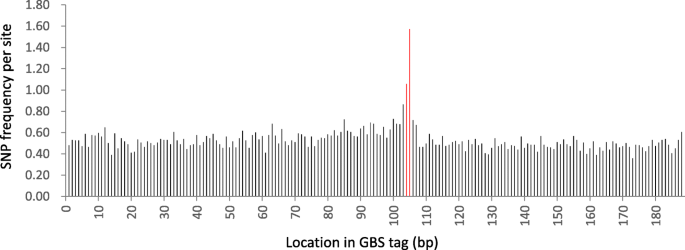
使用Bowtie 2将质量修剪过的读与GBS引用对齐[27].使用GATK调用SNPs [28].事实上,GBS参考由人工连接但在基因组中物理分离的成对末端reads组成,并不影响蝴蝶结排列或SNP呼叫。唯一的例外是当一个等位基因相对于GBS引用中使用的等位基因存在缺失时。因为在Illumina平台上,等位基因的测序长度是固定的(例如150 bp),例如,在测序区域中存在2-bp的缺失多态性,实际上意味着与未缺失的等位基因相比,缺失等位基因在读取的3 '端会多生成2 bp的序列信息。如果相应的GBS参考标记缺少缺失,则在缺失的位置会有一个对齐间隙,并且测序的“额外”2 bps将超出GBS参考中正向和反向读取被人为连接的结合点。这将导致SNP工件(附加文件10:图S3)。因为相邻的SNP作为SNP过滤协议的一部分被删除了,所以只有由单个碱基对删除引起的伪SNP保留在我们的数据集中。在正向读取和反向读取之间的交界处,SNP频率比其余读取的SNP频率高出两到三倍(图2)。8)。丢弃这两个位置的SNP可以减少大约2.5%的SNP总数。
我们测试了GATK的单倍型调用者和统一基因型调用SNPs。统一基因型比单倍型Caller多鉴定出17%的SNPs(表3.)。平均来说,用统一基因型识别的snp中有25%没有被单倍型Caller识别(图2)。2,附加文件6:图S2)。相反,约12%的单倍型Caller识别的snp没有被统一基因型识别。这些比较是在过滤后的数据集上进行的,因此涉及到稳健的snp。由统一基因型和单倍型调用者唯一识别的snp被纳入遗传图谱并因此得到验证的百分比非常相似(41.3%)vsRef50_98为40.1%,47.5%vsRef70_98为40.8%)。这表明,至少在我们的数据集中,统一基因型在调用snp时的高敏感性/侵略性并没有增加假阳性率。相比之下,由两个SNP调用者识别的纳入遗传图谱的SNP的百分比为Ref50_98的56%和Ref70_50的55.4%。Freebayes [29],一种基于单倍型的SNP调用者,在对映射群体的父母进行测试时,也产生了比统一基因型更低的SNP数量。虽然我们没有验证Freebayes识别的SNP,但统一基因型识别的共同SNP的数量和共同SNP映射的百分比表明,在Freebayes中较低的SNP检出率将导致较低的可映射多态性百分比。
每个具有代表性的GBS标记必须存在的样本的阈值数量,以便被包括在GBS参考中,也极大地影响了已识别的snp的数量,尽管趋势与我们预期的相反(表2)3.)。令我们惊讶的是,在筛选后,当选择一个代表性标签出现在至少50%的样本中(Ref50)而不是至少70%的样本中(Ref70)时,我们识别出更少的snp,尽管与Ref70参考文献相比,Ref50参考文献中出现更多的参考标签。我们假设,尽管Ref70参考位点携带较少的标记,但更多的位点仅由单个等位基因表示。如果来自同一位点的等位基因(例如,亲本1等位基因和亲本2等位基因)在“ustacks”进行聚类时形成了单独的堆栈,则两者都将包含在GBS引用中。在领结比对过程中,来自杂合个体的同一位点的Reads将与等位基因1 GBS参考标记或等位基因2 GBS参考标记进行比对,并在GATK中被标记为两个纯合位点。为了验证这一假设,我们删除了可能与另一个标签等位的标签。当一个GBS标签与另一个标签的同源性超过98%时,在对所有标签进行爆破分析后,只有一个标签被包含在GBS引用中。从Ref50中去除等位基因标签(称为Ref50_98)导致136%的新snp的调用,而不到2%的snp丢失(附加文件)6:图S2)。正如预期的那样,当从Ref70(称为Ref70_98)中去除等位基因标签时,SNP的增益明显较小(15%),但仍大大超过SNP的损失(1%)(附加文件)6:图S2)。现在,SNP的发现遵循了预期的趋势,与Ref70_98相比,Ref50_98识别出的SNP数大约高出24%(图2)。2)。有趣的是,只有75%用Ref70_98鉴定的snp被Ref50_98检测到。Ref70_98和Ref50_98被单一参考文献唯一发现的snp的百分比分别为25%和39%。这表明,通过结合两个GBS引用中的标签,可以获得一个显著改进的引用,并且SNP调用最大化。
UGbS-Flex管道比GBS-SNP-CROP更优越[13],它也可以使用成对的末端读取作为输入。使用相同的过滤条件,UGbS-Flex检测到的snp数量是GBS-SNP-CROP检测到的snp数量的3倍多。有趣的是,GBS-SNP-CROP比UGbS-Flex鉴定出更多的杂合子。而GBS-SNP-CROP识别出的SNPs比例高于UGbS-Flex (99.9%)vs73.7%)的读深≥20,读深似乎不能解释纯合子的召唤差异vs由于在SNP呼叫中使用严格的标准,两个管道之间的杂合子。根据甜瓜和他的同事[13],当次要等位基因数为0时,最小读深为11;当次要等位基因数为1时,最小读深为48。由于猕猴桃是四倍体,很可能某些杂合子SNPs是同源等位基因之间的变异,而不是同源等位基因之间的变异。用于聚类读取的参数可能在UGbS-Flex中比GBS-SNP-CROP中以更高的频率将同源物从同源物中分离出来。
构建高密度遗传图谱
我们之前在F2由野生动物杂交产生的种群大肠coracana无性系种群。非洲象acc。MD-20和栽培的大肠coracana无性系种群。coracanaacc。利用限制性片段长度多态性(RFLP)、简单序列重复序列(SSR)和表达序列标记(EST) [15,16].同样的种群被用于生成高密度遗传图谱。使用三种酶组合的决定(太平洋标准时间我/Msp我加猿KI)用于定位群体的GBS,早于不同酶组合产生多态标记的相对效率的数据可用性。酶组合的选择不影响图谱质量,只影响标记数量。因为个体F的DNA2植物不再可用,我们使用的DNA从膨大的F2:3用于映射的族。这样做的缺点是映射数据不像实际F基因分型时那么干净2杂合植物:植物,尤指在杂合区域发生重组或显示分离变形的植物绘图程序,例如MSTmap [21]及Lep-Map [22基于旅行推销员原理(TSP),可以非常快速地生成带有大量标记的地图。然而,由于标记排序依赖于两点连锁信息,TSP绘图程序更容易受到缺失数据的影响,并且提供的遗传图谱不如MAPMAKER等程序健壮。17]使用多点分析[30.](附加文件11:图S4)。此外,C(不明确的B或H)和D(不明确的A或H)值可以合并到MAPMAKER中,但必须转换为MSTmap中缺失的数据点。因此,我们使用了一种混合方法,在这种方法中,我们识别了链接组,并使用MSTmap进行了初始标记排序。使用MSTmap标记顺序,我们在MAPMAKER中生成带有“错误检测”选项的地图,以识别具有高水平基因型错误的标记。这些标记从进一步的分析中删除。然后,我们使用MSTmap标记顺序在MAPMAKER中使用三点/多点分析选择重叠标记组进行精细排序。使用的MAPMAKER版本(可从http://research.franklin.uga.edu/devoslab/)已经过修改,以在Windows命令提示符环境中有效运行,并比原来的MAPMAKER版本处理更多数量的标记[18].尽管进行了修改,但由于固有的软件限制,标记排序仍然仅限于大约100个标记的组。MAPMAKER中的“try”和“ripple”命令是通过内部开发的python脚本实现半自动的。因为重叠段是根据MSTmap定义的初始标记顺序选择的,所以MST映射中一个或多个标记的错误放置可能会影响mapmaker生成的最终映射。因此,最终地图中的重组事件会被手动检查,如果有必要,可能有问题的地图区域会被重新分析。两个标记块,一个在连锁群2A上,一个在连锁群2B上,因为它们两侧有大量的重组事件而被移除。在人工将标记分配到重组箱和添加共分离标记之后,我们获得了一个由18个连锁组中的4453个SNP标记组成的鲁棒图谱(图2)。3.,4而且5;额外的文件9:表S6)。
指谷的遗传图谱及其特征
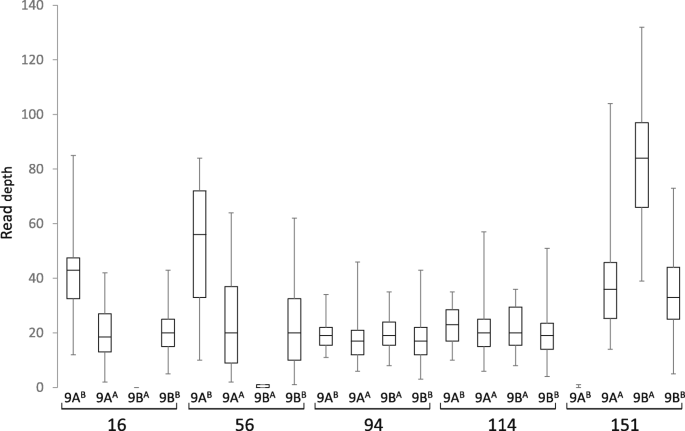
18大肠coracana连锁组被标记为1 - 9,后缀为A或B,以表明它们起源于A亚基因组还是B亚基因组(图2)。3.,4而且5;额外的文件9:表S6)。连锁群的命名与Dida等人的研究相同。[15],这是通过将迪达及其同事生成的地图中的标记子集合并到高密度GBS地图中确定的。每个连锁群到一个亚基因组的分配以前是通过为每个连锁群识别少量的RFLP标记来实现的,这些标记之间的大小是保守的大肠籼A基因组的祖先大肠coracana的A基因组大肠coracana[15].生成的GBS数据大肠籼为我们提供了一个扫描每个连锁基团的整个长度以寻找a基因组标记的机会。a基因组连锁群有望携带大肠籼GBS标签沿着它们的整个长度,而b基因组连锁群应该没有这种标签。我们在9个同源连锁组中的7个观察到预期的模式(图。6)。有趣的是,在同源组6和9中,的存在/缺失模式大肠籼GBS标记表明同源染色体之间存在互易易位。目视检查第9组重组数据(附加文件8:表S5)显示了许多a和B同源体在假定的易位断点处携带重组事件的子代,导致在我们的映射分析中以高LOD评分分裂的十字形映射。这表明第9组易位只存在于两个作图亲本中的一个。为易位而杂合的染色体以交叉型配置进行配对。根据染色体如何分离(并假设没有交叉发生),后代既可以携带A和B基因组染色体的完整副本,也可以携带携带易位染色体区域缺失/重复的染色体补体。一个子代(附加文件中的子代151)8:表S5)确实缺乏位于易位区域内的所有a基因组标记,并且有5个子代(16、56、125、131、148)缺乏b基因组标记,表明这些区域在这些子代中被删除了。进一步的分析表明,在第151代相应的b基因组区域和第16代、第56代、第125代、第131代和第148代相应的a基因组区域中,读取数大约是两倍,这表明在这些后代中,一个区域的缺失被额外的同源区域副本所弥补(图2)。9)。虽然染色体区域的缺失在二倍体物种中可能是有害的,但异体多倍体中同源染色体的存在在很大程度上缓冲了染色体缺失引起的负面影响。在同源A和B染色体上有明显重组事件的子代,或在6A和6B染色体上有缺失区域的子代未被鉴定出来。6A/6B重排可能发生在多倍体进化的早期,并且在双亲中都存在。通过比较鉴定杂合子易位大肠籼取决于十字形状的图谱是被分裂成两条非易位染色体还是两条易位染色体。因此,我们仔细检查了其他同源组的交叉形连接和缺失,并在同源组2中发现了间质重排。我们在地图构建过程中删除的两个间质标记块,因为它们的两侧都有大量的重组事件,是在亲本之一中经历了间质易位的区域。在这一点上,我们不知道9A/9B和2A/2B重排是发生在栽培亲本还是野生亲本。在异源多倍体中,配对通常由基因控制,并仅限于同源染色体[31,32].配对控制位点的移除可导致染色体重排,包括同源易位[33,34].据报道,指谷子显示二组遗传[35,36]但配对控制的机制尚不清楚。需要对配对行为进行分析,以确定亲本中的任何一方是否抑制了同源配对控制。
结论
我们详细分析了GBS方法、参考生成和SNP调用中参数和模块变化的影响,旨在最大限度地发现高置信度SNP,并将缺失数据最小化。新开发的UGbS-Flex管道为目前可用的GBS数据分析工具提供了一个有用的补充,特别是在缺乏全基因组参考的情况下,通过配对端测序reads对杂合和多倍体个体进行基因分型。UGbS-Flex管道被用于鉴定高置信度的snp,随后用于生成第一张指谷子的高密度遗传图谱,Eleusine coracana.采用多点标记排序实现了地图鲁棒性。我们证明了GBS在亲缘关系密切的物种之间的跨物种应用是可行的,当应用于一个异体多倍体和一个二倍体基因组供体之间时,可以用来识别连锁基团的亚基因组起源和同源染色体之间易位的发生。指谷子是一种基因组资源很少的近交系异体多倍体物种,本研究以其为例进行研究,但UGbS-Flex管道已成功应用于其他物种,包括异交四倍体柳枝稷。
参考文献
- 1.
Luikart G, England PR, Tallmon D, Jordan S, Taberlet P.人口基因组学的力量和前景:从基因分型到基因组分型。中国农业科学。2003;4(12):981-94。
- 2.
Elshire RJ, Glaubitz JC, Sun Q, Poland JA, Kawamoto K, Buckler ES, Mitchell SE。一种用于高多样性物种的强大、简单的基因测序(GBS)方法。5 .公共科学图书馆,2011
- 3.
Baird NA, Etter PD, Atwood TS, Currey MC, Shiver AL, Lewis ZA, Selker EU, Cresko WA, Johnson EA.使用测序RAD标记快速发现SNP和遗传图谱。2 .公共科学图书馆,2008
- 4.
阿特舒勒D,波拉拉VJ,考尔斯CR,范埃顿WJ,鲍德温J,林顿L,兰德ES。由简化表示霰弹枪测序生成的人类基因组SNP图谱。大自然。2000;407(6803):513 - 6。
- 5.
陆飞,Lipka AE, Glaubitz J, Elshire R, Cherney JH, Casler MD, Buckler ES, Costich DE.柳枝稷基因组多样性、倍性和进化:基于网络的SNP发现协议的新见解。PLoS Genet, 2013;9:e1003215。
- 6.
Gardner KM, Brown P, Cooke TF, Cann S, Costa F, Bustamante C, Velasco R, Troggio M, Myles S.使用下一代测序在苹果中快速和经济有效的基因定位。G3: |基因组的基因|.遗传学。2014;4(9):1681 - 7。
- 7.
Davey JW, Hohenlohe PA, Etter PD, Boone JQ, Catchen JM, Blaxter ML.利用下一代测序发现全基因组遗传标记并进行基因分型。植物学报。2011;12(7):499-510。
- 8.
Poland JA, Brown PJ, Sorrells ME, Jannink J-L。利用一种新的双酶基因测序方法开发大麦和小麦的高密度遗传图谱。科学通报。2012;7:e32253。
- 9.
白伯利,张哲,张文杰,张文杰,张文杰,张文杰。TASSEL:用于不同样本中复杂性状关联映射的软件。生物信息学。2007;23(19):2633 - 5。
- 10.
格劳比茨JC, Casstevens TM,陆飞,Harriman J, Elshire RJ,孙Q, Buckler ES。tasser - gbs:高容量基因分型测序分析管道。公共科学图书馆,2014;9(2):e90346。
- 11.
Torkamaneh D, Laroche J, Bastien M, Abed A, Belzile F. Fast-GBS:一种从测序基因分型数据中高效、高精度调用snp的新管道。BMC生物信息学。2017;18:5。
- 12.
Catchen JM, Amores A, Hohenlohe P, Cresko W, Postlethwait JH。堆栈:从短读序列构建和基因分型新位点。G3: |基因组的基因|.遗传学。2011;1(3):171 - 82。
- 13.
Melo ATO, Bartaula R, Hale I. GBS-SNP-CROP:使用可变长度、配对端测序数据进行SNP发现和植物种质鉴定的参考可选管道。BMC生物信息学。2016;17(1):29。
- 14.
王晓明,王晓明,王晓明,等。基于激光流式细胞仪对禾草属植物核DNA含量的重新评价。植物科学进展。1997;23(1):1 - 11。
- 15.
Dida MM, Srinivasachary RS, Bennetzen JL, Gale MD, Devos KM。指谷子的遗传图谱。应用理论与实践,2007,34(3):326 - 326。
- 16.
Srinivasachary DMM, Gale MD, Devos KM。比较分析表明,手指小米和水稻基因组之间存在高度保守共线性。应用理论,2007;115:489-99。
- 17.
Lander ES, Green P, Abrahamson J, Barlow A, Daly MJ, Lincoln SE, Newburg L. MAPMAKER:一个交互式计算机包,用于构建实验和自然群体的主要遗传连锁图。基因组学。1987;1:174 - 81。
- 18.
李国强,李国强,李国强:利用MAPMAKER/EXP 3.0构建基因图谱。剑桥,质量。,美国。:Whitehead Institute for Biomed Res; 1993.
- 19.
Van Ooijen JW: Joinmap®4,用于计算实验人群遗传连锁图的软件。在.荷兰瓦赫宁根:Kyazma B.V.;2006.
- 20.
利用一种新的计算机程序:JoinMap构建综合遗传连锁图谱。植物学报1993;3:739-44。
- 21.
吴勇,Bhat PR, Close TJ, Lonardi S.基于最小生成树的遗传连锁图谱的高效和准确构建。公共科学学报,2008;4(10):e1000212。
- 22.
Rastas P, Paulin L, Hanski I, Lehtonen R, Auvinen P. Lep-MAP:大型SNP数据集快速准确的链接映射构建。生物信息学)。2013;29(24):3128 - 34。
- 23.
道尔JJ,道尔JL。一种用于小数量新鲜叶片组织的快速DNA分离程序。植物化学通报,1987;19:11-5。
- 24.
Rohland N, Reich D.用于多目标捕获的高成本效益,高通量DNA测序文库。中国生物工程学报,2012;22(5):939-46。
- 25.
Andrews S: FastQC:高通量序列数据的质量控制工具。2010.网上订购地址:http://www.bioinformatics.babraham.ac.uk/projects/fastqc.
- 26.
Magoc T, Salzberg S. FLASH:快速调整短读长以改善基因组组装。生物信息学。2011;27
- 27.
朗米德B,萨尔茨堡SL.快速间隙阅读对齐领结2。南京大学学报:自然科学版,2012;9:357-9。
- 28.
McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M,等。基因组分析工具包:用于分析下一代DNA测序数据的MapReduce框架。基因组学报2010;20(9):1297-303。
- 29.
Garrison E, Marth G.基于单倍型的短读测序变异检测。: arXiv: 12073907;2012.
- 30.
阿福克D,伍德I,斯蒂芬S,卡瓦纳CR,黄斌。多亲本群体高密度图谱的不确定性特征。遗传学。2014;198(1):117 - 28。
- 31.
六倍体小麦细胞学二倍体行为的遗传控制。大自然。1958;182:713-5。
- 32.
Jenczewski E, Eber F, Grimaud A, Huet S, Lucas MO, Monod H, Chevre AM。PrBn是控制油菜同源配对的主要基因(芸苔属植物显著迄今己。遗传学。2003;164(2):645 - 53年。
- 33.
异体多倍体中的同源重组:多倍体棘轮。植物学报,2010;18(1):18-28。
- 34.
Devos KM, Sorrells ME, Anderson JA, Miller TE, Reader SM, Lukaszewski AJ, Dubcovsky J, Sharp PJ, Faris J, Gale MD.小麦零体-四体和二体系的染色体畸变。谷物决议,1999;27:231-9。
- 35.
陈纳维莱雅MS,何瑞梅SC.龙尾蛇属植物基因组分析。Euphytica。1974;23:489 - 95。
- 36.
Hiremath SC, Salimath SS.禾本科Eleusine coracana Gaertn基因组供体。应用理论。1992;84:747-54。
确认
该研究部分由非洲生物创新项目、佐治亚大学和美国能源部资助DE-SC0010743资助。DS由富布赖特-尼赫鲁高级研究奖学金资助(Award 1865/FN-SR/2013),来自美国印度教育基金会,印度新德里。
数据和材料的可用性
所有使用的数据集和生成的脚本都包含在本手稿中作为补充数据和/或可通过Devos实验室网站(http://research.franklin.uga.edu/devoslab/)。
作者信息
从属关系
贡献
PQ开发了大部分脚本并主导了数据分析。DG对测绘人群进行了GBS,并协助进行数据分析。DC和XW贡献了脚本。DS和SS参与GBS实验方案的优化。MMD生成映射种群并收集大肠籼种质。RLM生成MAPMAKER的更新版本。KMD构思研究,参与分析并主导文稿准备。所有作者都参与了稿件的准备工作,并通过了稿件。
相应的作者
道德声明
伦理批准并同意参与
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
附加文件
附加文件1:
数据S1。用于GBS引用生成、SNP调用和进一步SNP处理的系统要求和UGbS-Flex命令。(PDF 165kb)
附加文件2:
图S1。“Bcraw”文件夹包含单个样本的原始测序文件。修剪后,修剪的序列文件被放置在' BCpc '文件夹中。使用' BCpc '文件夹中的文件作为输入,所有读取长度相等的文件都放在' BCfin '文件夹中。“ASU”文件夹保存了“BCfin”文件夹中所有文件的ASU方法结果。ASU结果用于生成参考;过滤后的引用被放在' Ref '文件夹中。' BCpc '文件夹中的修剪序列与' Ref '文件夹中的引用文件对齐(与Bowtie对齐);校准用于SNP调用(使用GATK);所有结果都存储在“SNP”文件夹中。“流程”编号对应于附加文件中的步骤编号1:数据S1。(pptx52 kb)
附加文件3:
表S1A。对reads子集的三种酶组合进行了汇总统计;条目按读数分组。表印地。对reads子集的三种酶组合进行了汇总统计;参赛作品按酶组合进行分组。(xlsx29 kb)
附加文件4:
表S2。数据库中所有三个访问所共有的GBS参考标签的平均读取深度(在测试的三个访问中)太平洋标准时间我/濒死经历我是碎片池。(docx13kb)
附加文件5:
表S3。数目及百分比猿KI站点存在于太平洋标准时间我/Msp我和太平洋标准时间我/Msp我+猿KI摘要。(docx12kb)
附加文件6:
图S2。使用不同SNP调用者识别的SNP数量的比较(UG =统一基因型;HC=Haplotype Caller)和不同的GBS参考文献(Ref50:标签出现在≥50%的样品中;Ref70:标签在≥70%的样品中存在;Ref50_98:标签在≥50%的样品中存在,同源性≥98%的标签仅保留1个标签;Ref70_98:标签在≥70%的样品中存在,同源性≥98%的标签仅保留1个标签。(pptx7261 kb)
附加文件7:
表S4。UGbS-Flex和GBS-SNP-CROP识别snp的比较。(xlsx9kb)
附加文件8:
表S5。MD-20 × Okhale-1群体的基因型数据。(xlsx2444kb)
附加文件9:
表S6。包含所有标记的遗传图谱。(xlsx717kb)
附加文件10:
图S3。样本中相对于GBS参考等位基因缺失的影响。形成一个间隙对齐,并且3 '端延伸到GBS参考中的正向和反向读的交界处之外,导致在该位置调用SNP。整合基因组学观察者(Robinson等人,2011,自然生物技术29:24-26;Thorvaldsdóttir等人。2013,生物信息学简报14:178-192)用于可视化。(PPTX 75kb)
附加文件11:
图S4。使用MSTmap(左边)和MAPMAKER(右边)生成的遗传图谱的比较。与MSTmap地图相比,近65%的标记在MAPMAKER中重新排序。在两幅地图中占据不同相对位置的标记用一条线连接起来。(PPTX 17kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
齐,P.,吉摩德,D.,萨哈,D。et al。UGbS-Flex,一种新的生物信息学管道,用于在没有参考基因组的多倍体中发现无推断的SNP:指谷子作为案例研究。BMC植物生物学18,117(2018)。https://doi.org/10.1186/s12870-018-1316-3
收到了:
接受:
发表:
关键字
- 染色体重组
- Eleusine coracana
- 大肠籼
- 手指小米
- 基因映射
- Genotyping-by-sequencing (GBS)
- GBS-pipeline
- Paired-end读取
- 多倍体
- SNP打电话