抽象的
背景
转录因子(TFS)是可以与DNA序列结合并调节基因表达的蛋白质。许多TFS是细胞中的母体调节剂,其有助于真核生物中的组织特异性和细胞类型特异性基因表达模式。玉米一直是一百年的模型生物,但对其组织特异性基因调节几乎没有人通过TFS。在这项研究中,我们利用网络方法在玉米中的四种组织(叶,根,SAM和种子)中阐明基因调节网络(GRNS)。我们利用Genie3,一种机器学习算法与大量的RNA-SEQ表达数据组合,以构建四个组织特异性GRN。与其他一些技术不同,这种方法不受高质量位置称重矩阵(PWM)的限制,因此可以预测GRNS在玉米中超过2000个TFS。
结果
尽管在多种组织中表达了许多TFS,但是多层分析预测许多转录因子的组织特异性调节功能。在四个组织特异性GRN内出现的一些良好的TFS,并且GRN预测基于许多这些示例的出版结果匹配期望。我们的GRNS也被Chip-SEQ数据集(KN1,FEA4和O2)验证。针对每个组织鉴定关键TFS,并对每个组织中的关键调节剂进行匹配期望,包括富集和具有已知该组织的调控因子的富集和身份。我们还通过使用MCL算法进行分析来找到每个网络中的功能模块。
结论
通过组合公开的基因组表达数据和网络分析,我们可以在玉米中发现GRNS。由于芯片SEQ和PWMS仍然有限于多种模型生物,我们的研究提供了统一的平台,可以适应具有基因组表达数据的任何物种以构建GRN。我们还提供了公开的数据库,玉米组织特异性GN(MGRN,https://www.bio.fsu.edu/mcginnislab/mgrn/),以方便查询。所有源代码和数据可在Github (https://github.com/timedreamer/maize_tissue-specific_GRN).
背景
基因表达调控是生物学中最重要、最复杂的问题之一。这在真核生物中尤其有趣和复杂,因为它们的大基因组和高阶核组织。从孟德尔到芭芭拉·麦克林托克,植物生物学家是基因调控基因研究的先驱,他们的工作构成了当前理解的基础。
玉米(玉米)是一百多年来的模范生物,而且具有重大的经济意义。新一代测序技术的发展使研究全基因组表达变化变得更容易,大大加强了玉米研究。这些数据可用于构建基因调控网络(GRNs),以系统的方式阐明基因调控相互作用[1,2].尽管所有细胞携带相同的遗传密码,但可能是由不同的GRN引导的细胞分化。玉米有限的研究来破译组织特异性GRN [3.,4].
尽管存在许多不同类型的遗传调节蛋白,但转录因子特别感兴趣,因为它们代表了蛋白质和染色体之间的相对直观的调节相互作用,可能导致转录活性的直接变化。玉米的TF资源之一是草地性信息服务(Grassius),玉米中有2587个注释TFS [5]和2034 TF开放阅读框架(ORF)克隆的矢量[6]促进TF目标相互作用分析。在这项研究中,我们专注于来自Grassius注释的TFS,我们的GRN是指Grassius TFS与其受管制目标之间的相互作用。其他类型的调节,例如蛋白质 - 蛋白质相互作用和表观遗传调节,超出了本研究的范围,但可以通过GRN分析可获得的方法的变化分析[7,8].
解开TF调控性相互作用,使用染色质免疫沉淀(ChIP)体内方法是金标准。基本上,芯片实验隔离TF-DNA复合物的体内。加上PCR(芯片的qPCR),微阵列(芯片芯片)或测序(芯片起),这种方法可确定TF结合的位置,并允许高可信度的TF调节区的预测。然而,在植物中有公布的ChIP数据集的数量相对较少,可能是由于与植物转化,抗体效,以及其他实验上的困难限制。即使在充分研究的对象模型像拟南芥蒂利亚纳,只有46个TFs拥有ChIP-chip/Seq数据,收集自三个主要数据库,包括jasper [9], CIS-BP [10.]和CressInt [11.].在玉米中,只有五个TFS发布的芯片SEQ数据。作为比较,人类编码项目生成的芯片-SEQ数据630 TFS [12.].
作为一种替代或互补的方法,体外方法可用于构建大规模grn。拟南芥TF-DNA筛选已建立几种酵母- 1杂交(Y1H)体系[13.,14.,15.].在玉米,TFOME项目[6]提供超过2000米玉米TF克隆的宝贵资源,以促进高通量研究,包括最近的Y1H筛网,其鉴定在玉米酚醛代谢途径中的一千TF靶相互作用中[16.].通过指数富集配体的系统进化(SELEX)可以生成grn的其他潜在数据[17.、蛋白结合微阵列(PBM) [18.或DNA亲和纯化测序(DAP-seq) [19.].这些方法可以发现CIS.-Elements为数百个TFS帮助解码复杂的转录网络。DAP-SEQ还可以包含DNA甲基化信息,该信息已被证明是在拟南芥中影响TF-TAIGIN结合[19.]和人类[20.].大多数这些方法尚未用于玉米。
PlantRegMap及其他工具[21,22,23]预测TF结合位点,基于不同相关物种的同源TF可能识别相同motif或CIS.监管元素(CRE)。这些序列用位置加权矩阵(PWMs)表示,可以用来预测TF靶标。这种方法依赖于由ChIP-Seq、PBM或DAP-Seq数据生成的高质量pwm。PlantRegMap收集了674个高质量motif,这些motif只能预测GRASSIUS预测的2587个TFs中的229个[5].体外和基于同源性的方法的另一个限制是,在组织和细胞类型水平上的调节相互作用不能被检测或从这个数据单独推断。
推断监管网络的替代方法是使用应用于基因表达数据的统计推理算法。一种特别有效的算法是Genie3 [24],这是基于对对话的逆向工程评估和方法(梦想)挑战4和5的对话进行比较的最高评分25].GENIE3已成功应用于拟南芥[26]和玉米[27] GRN建筑。此方法使用回归树[28[因此,对每个基因的模型调节剂,因此可以预测TF目标相互作用。Genie3它不需要特定的实验设计,因此可以应用于大量公共可用的基因组表达式数据集。此外,Genie3可以揭示非线性关系。它利用随机森林算法[29],并发现可以通过劈开树来减少模型方差的调控器。GENIE3有Python、Matlab和R语言的实现,便于研究人员使用。同样,类似的方法也适用于时间序列[30.],单细胞[31],以及综合[32grn显示出广泛的适用性。GENIE3利用了并行计算的优势,可以在多核桌面上生成大型网络。通过网络分析,构建了4种不同组织(叶、根、茎尖分生组织和种子)的2241个转录因子的grn。我们发现TF相互作用可以被ChIP-Seq数据集证实,这表明这种方法在预测真实的相互作用方面是有效的。不同组织中的grn显示了组织特异性TF调节相互作用,可能与不同的生物学功能相关。我们发现TF的中心性与其表达无关,每个组织都使用不同的TF作为主调控因子。方便使用的入门网站(http://www.bio.fsu.edu/mcginnislab/mgrn.)开发。所有的源代码都可以在Github上找到,可以很容易地应用到其他生物体上。
结果
玉米转录因子具有组织特异性表达模式
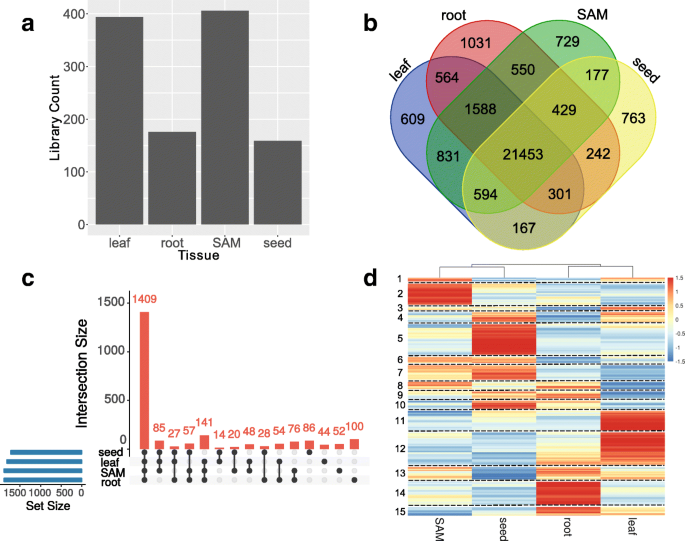
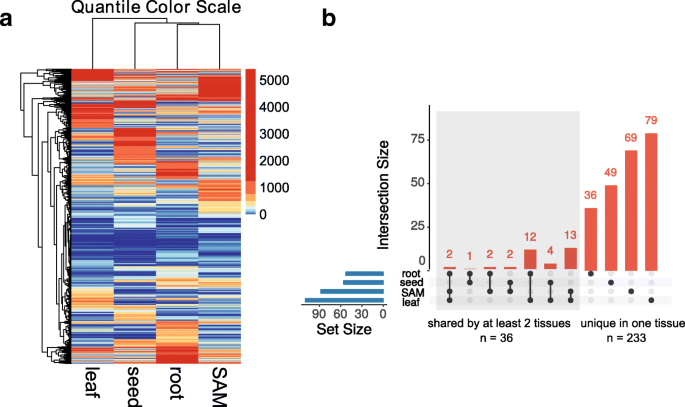
以前,我们在各种玉米组织中重新分析了1266个高质量的RNA-SEQ文库,并产生了基因共抑制网络[33].选择来自该表达基质的100多种文库的组织以构建组织特异性GRNS(图。1).还有四种组织包括:叶,根,拍摄顶端商品(SAM)和种子(附加文件1).在每种组织中,在该组织中表达了超过10%组织文库中百万分之一百万(CPM)的基因。我们发现在至少一种组织中表达了76.06%(30,028/39479)的玉米基因,并且在所有四个组织中表达了54.34%(21,453 / 39479)基因(图。1 b).这些数字与前一项研究相当[34尽管我们的分析使用了较少的组织和更新的基因注释(AGPv3),但报告91.4%的基因在至少一个组织中表达,44.5%的基因在所有组织中表达。一小部分基因表现出组织特异性表达(图。1 b),并对这些基因进行基因本体论(GO)富集分析(附加文件2).我们发现富含叶特异性基因用于光合作用活动(p-Value = 2.40e-06)和种子特异性基因富含营养储层活性(p-value = 1.39E-37),包括20个玉米醇溶蛋白基因。
玉米基因调控网络(的GRNs)四个组织。一个用于在每个组织中构建GRN的RNA-SEQ库的数量。基于SRA元数据数据和/或公布的论文将文库分为组织(补充表1中的细节)。山姆:拍摄顶端公司。b维恩图显示了在叶子,根,SAM和种子中表达的基因重叠。如果一个基因在超过10%的文库中每百万计数(CPM)值大于1,则该基因被指定为在组织中表达。c一个UpSet图显示了每个组织中表达的转录因子(TFs)的重叠。条形图(橙色)表示每个个体或组织组合中表达的tf数量。交叉点用连在一起的黑点表示。每个组织中表达的TFs总数用蓝色条形图表示。d1409个转录因子在所有四种组织中均有表达。根据转录因子的表达模式,将转录因子分成15个子组(以虚线分隔)。对每个组织的基因表达值取平均值并进行z变换,结果显示每个基因的表达值在- 1.5到+ 1.5之间。层次聚类是通过R中的hclust()函数来计算的
接下来,我们检查四种组织中的转录因子的表达模式。通过GRASSIUS [注释所有2587级的TFS5]在玉米AGPv3中,86.63%的表达在至少一个组织中,54.46%(1409/2587)在所有四个组织中表达(图。1 c).这表明相当数量的转录因子存在于多个植物器官中。10.90%(282/2587)的转录因子仅在一个组织中表达(叶中44个,根中100个,SAM中52个,种子中86个),包括一些研究充分的例子:缩小护套1在叶(NS1);冠根和种子根无根1(RTCS1)中的;蜀黍Branched1(TB1)在SAM;viviparous1.(VP1)1).这些tf的突变体已被证明在相关组织中表现出表型。例如,ns.突变植株表现出下叶缘缺失[35];的rtcs1突变体完全失去冠根和侧根[36];TB1突变体由于失去顶端优势而高度分枝[37];vp1突变种子早期发芽在未成熟的玉米棒上[38].尽管有部分转录因子在所有四种组织中均有表达,但这1409个基因的表达模式不同(图1)。1 d),其可以有助于组织特定功能。因为转录因子是关键的基因表达调节剂,它们的模式可以代表不同的和组织特异性基因调控网络。
四种组织的基因调控网络构建
为了构建组织特异性grn,我们使用了基于树集成的基因网络推理(GENIE3)算法[24],梦中的最佳表演者4和5挑战,使用基于树的集合机学习方法来预测基因监管关系[25].尽管之前已经报道过该算法与其他算法之间的广泛基准比较[24,25,我们比较了GENIE3和其他两个技术发展水平算法:最小冗余网络(MRNET)[39]和上下文相关性似然(CLR)算法[40].通过将2587个TFs设置为“候选调控因子”,从每个组织的表达基质中推断出组织特异性grn。这导致了4种组织类型的预测GRN。
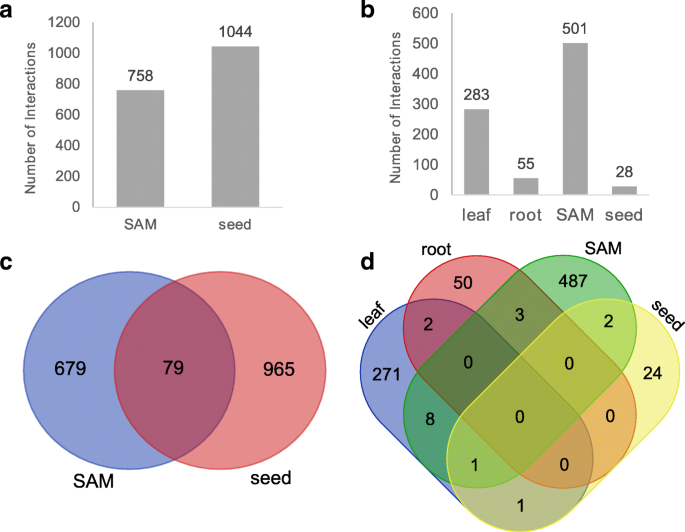
首先,我们利用已发表的TF ChIP-Seq数据评估了这四个网络的质量,这些数据是使用与Knotted1 (KN1)相互作用的抗体沉淀的DNA [41],迷人的EAR4(FEA4)[42]和不透明的endosperm2(O2)[43].这些3种蛋白质是已知的TFS,具有SAM和耳朵发育(KN1和FEA4)或种子发育(O2)的专用功能。FEA4在所有四种组织中表达,而KN1仅在SAM和种子中表达,并且O2仅以种子表示。通过在精密召回曲线(AUPR)下的接收器操作员特征曲线(AUROC)和面积下,通过TF芯片-SEQ数据评估GRN的性能。这些是广泛使用的二进制分类问题的汇总统计,使得高于使用随机样本获得的值表明分类算法检测到随机子集的预期比预期更多的模式。从每个芯片-SEQ数据集中,10千克地区内具有高置信峰的基因被认为是该转录因子的阳性靶标(见方法有关详细信息)。KN1,FEA4和O2的表达模式与已发表的基因表达式ATLAS一致[44,除O2外,所有grn的汇总统计均优于随机样本(表1)2).已经表明,O2结合的基因和误解的基因之间存在非常低的重叠O2.突变体[43].因此,可能难以为纯粹是基于表达数据的任何算法来推断对于O2调控性相互作用。除了从O2 GRN中,AUROC和AUPR值表明,我们的组织特异性的GRNs预测,用芯片起的数据是一致的监管作用。虽然通常GENIE3导致类似的AUROC和AUPR值相比MRNET和CLR,用于通过GENIE3产生FEA4 SAM网络AUROC和AUPR值比MRNET和CLR网络更高(附加文件3.).由于我们已经知道FEA4在SAM中是一个重要的调节器,所以我们选择GENIE3作为我们额外实验的网络构建方法。
在接下来的一组分析中,出于规范化的目的,四个grn被限制只包括GENIE3计算的前100万个交互作用(边)。这是一种常用的网络切断[27]并允许我们将网络之间的网络与不同的边缘总数进行比较。除非具体说明,否则所有剩余结果,否则用于分析的GRS以这种方式约束。网络的边缘被视为“指导”,其中TFS被建模为调节剂和在该组织中表达的所有基因作为靶标。我们将边缘重叠与四个组织之间进行比较(附加文件4).对于成对比较,叶子和SAM GRS为两种组织之间的7.12%(71,190 / 100000)共享大部分边缘,其次是种子和Sam 5.07%(50,664 / 1000000)。令我们惊讶的是,总共有40万边缘,约80%的边缘对组织是独一无二的,只有0.268%(2679/1000000)边缘在所有四种组织之间共用。该结果表明,即使在四种组织中表达超过50%的TFS,也有可能在不同的特异性组织中具有不同的调节靶标。我们调查了由353个TFS和1657个目标基因组成的四个GRN的2679个共享边缘(附加档案4和附加文件5).靶基因的GO分析揭示了包括细胞周期、DNA复制、细胞分裂和染色体组织在内的多个必要的生物学过程(补充文件)6).有趣的是,有30个基因注释为组蛋白H3K9甲基化(p-value = 1.04E-21),表明表观遗传调控,特别是异染色质形成和基因沉默的重要性[45,46,47].这些相互作用可能是植物生长所必需的。
GRN分析可用于预测TFs的组织特异性调控
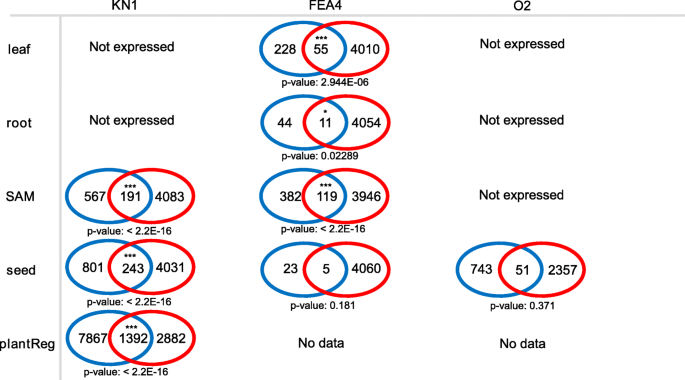
在探索整体网络质量之后,我们进一步分析了KN1,FEA4和O2的组织特异性相互作用。与其表达模式一致,O2仅预测了种子的相互作用,KN1仅预测了SAM和种子的相互作用,而FEA4已经预测了所有四种组织中的相互作用(图2和3.).对于KN1和FEA4,SAM GNS具有超过500个预测的相互作用。这与SAM开发中的KN1和FEA4的重要调节作用一致,并且已经向这些TFS报告了这种功能[41,42].对于KN1和FEA4,预测的91.23%(1644/1802)和95.96%(832/867)相互作用仅局限于一个组织(图。2摄氏度,d).有趣的是,我们从KN1的SAM靶标和O2种子靶标中分别发现了两个独特的GO术语:“梢系发育(p-值:1.31E-02)”和“营养库活性(p-值:6.31E-25)”7)表明组织特异性GRNS鉴定了相关功能的基因。
如果为芯片-SEQ确认的目标富集了GRN预测,那么该目标将建议GRN可以可靠地识别没有芯片SEQ数据的其他TFS的推定目标。从KN1,FEA4和O2的预测靶标,我们将它们中有多少由芯片-SEQ数据确认(图。3.和附加文件8).采用单尾Fisher精确检验来检验重叠的显著性。除了FEA4种子和O2种子预测(p值> 0.05;无花果。3.).这可能是因为FEA4在种子中具有有限的功能。尽管O2种子预测靶未富集在O2-结合的基因中,但我们的网络预测了最多10个下调基因中的7种的相互作用O2.突变体,包括6个特征明确的O2靶点玉米醇溶蛋白基因[43].
另一种预测方法是搜索基因启动子区以进行TF特异性CIS.- 调节元素[21,22,48,49].该方法依赖于高质量的位置重量矩阵(PWM),该矩阵仅适用于玉米中有限的TFS。我们将我们的预测与PlantRegMap数据库进行了比较[21]包含KN1,但FEA4和O2没有数据。PlantRegMap对KN1 chip结合靶点的预测也显著富集(p值< 2.2E-16)。
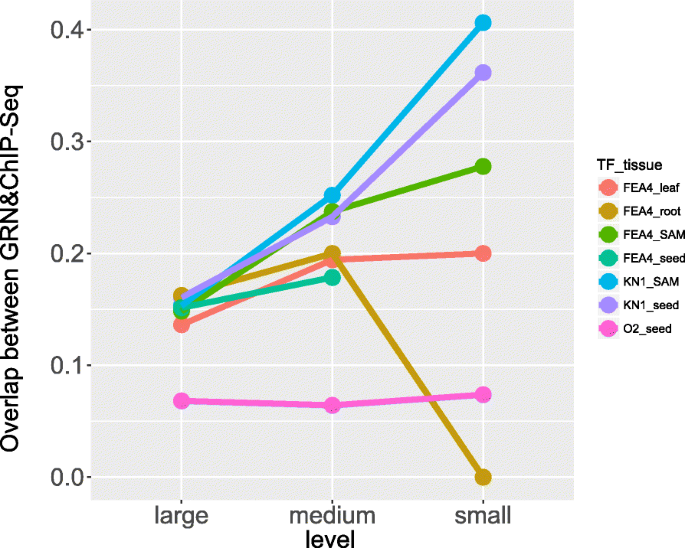
此外,我们还比较了不同网络规模下GRN预测目标与ChIP识别目标的重叠率。对于KN1、FEA4和O2,在1000万条边内的预测被纳入“大”网络,只有前100万条边内的预测被纳入“中等”网络,只有前10万条边内的预测被纳入“小”网络。重叠百分比递增的模式(图。4为大多数TFS观察到的TFS作为更严格的网络,选择具有更少边缘的网络,除了root中的FEA4。在FEA4根GRN中,无法找到重叠目标(附加文件8),但这可能与少量的相互作用有关(n= 2)。此外,我们还比较了组织grn和发育图谱grn(附加文件)8)[27].也使用Genie3创建了AtLASGRN,但使用了不同的mRNA和蛋白表达数据集。分别在mRNA和蛋白质GRN中包含2200 TFS和545 TFS。KN1和O2都在GRNS中,但仅在MRNA GRN中(附加文件)9).我们发现我们的组织特异性GRNS在预测目标和芯片识别的目标与ATLAS GRNS之间具有可比或更好的重叠百分比(附加文件9).除了FEA4-mRNA GRN之外,使用小型网络时,重叠百分比也增加。总之,这些结果表明,组织特异性GRN可以预测不同组织中的TF结合相互作用。
GRN分析可用于识别不同组织中转录因子的中心功能
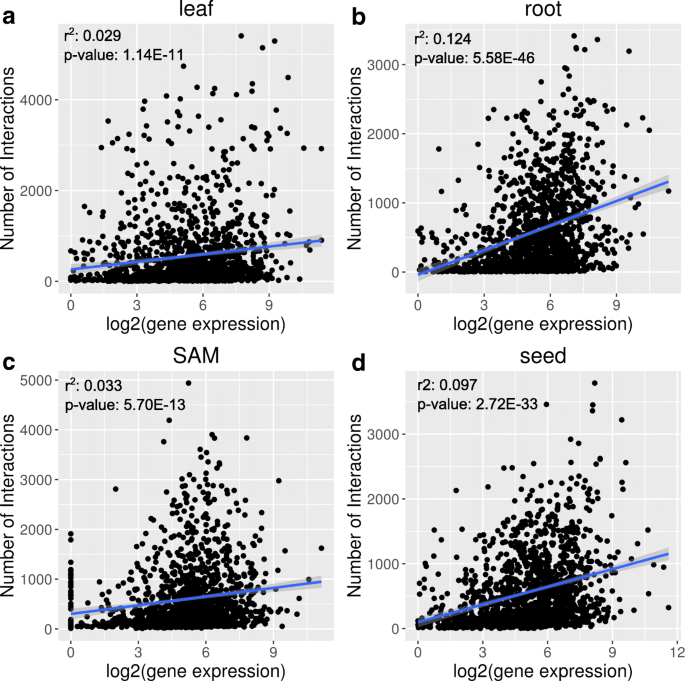
如我们从KN1和FEA4发现的,TFS可能在不同组织中具有各种相互作用(度中心)。我们想知道这是否可能与TF基因表达的差异相关。例如,TFS可以在组织中具有更多的相互作用,其中TF是最表达的。为了测试这一点,我们将每个TF的相互作用绘制在每个组织中的表达水平(图。5和附加文件10.).这项分析包括1406个在所有四种组织grn中至少有一种相互作用的转录因子。对于所有四种组织,r平方值都在0.0012到0.124之间(图4)。5和附加文件10.)的线性回归模型(由CPM或log2(CPM + 1)测量)。说明TF表达与程度中心性之间不存在线性关系,相互作用数量的差异不太可能是由TF基因表达差异引起的。反之,中心性程度的差异可能揭示了不同组织中转录因子的不同生物学功能或活性。
TF程度中心广泛变化,对于组织之间的特异性TFS(图。6).我们计算了变异系数(CV),测量相对变异性,四种组织之间的程度中心。CV为9.583至186.376,平均值等于88.444。要专注于具有大量预测相互作用的TFS,500的程度中心的最小差异被认为是可接受的,以便进一步分析(附加文件11.).然后,我们分析了具有前100个最大CV的TFS,并发现它们的12(叶),28(根),28(SAM)和32(种子)在每个组织中具有最高程度的中心。叶片中具有大量相互作用的TFS包括基因基于对拟南芥的同源性来调节叶片开发的不同方面,如SPL9 / 15 [50]和tcp2 / 24 [51].例如,GRMZM2G126018(SPL9 / 15同源)在叶中具有765个相互作用,但其他三种组织中不超过80。我们还发现GRMZM2G171912(HY5),GRMZM2G028438(SCL8)和GRMZM2G146020(VIP1)与其他组织有更多的循环相互作用(附加文件11.).这些表明,基于我们的分析,这些TFS具有暗示每个组织中具有独特作用的其他功能。
具有程度中心地位> 2000的TFS被定义为每个组织中的键TFS,并且每个组织中存在相对较少的这些(图。6和附加文件12.).叶片中有110个关键TFS,root 53,sam和56 in种子中的88个(其他文件13.).一些研究的实例包括叶片,Homobox3(HOX3,GRMZM2G314546)中的粗SHEAP2(RS2,GRMZM2G403620),SAM和PROVAMIN盒结合因子1(PBF1,GRMZM2G146283)中的种子。重叠可视化(图。6 b)显示75.90%(233/307)关键转录因子为单一组织所特有。我们还发现了至少两个组织共享的36个关键tf(附加文件14.).深入挖掘利用BioMart(http://plants.ensembl.org)显示,这36种TFS的同源物对拟南芥的发展至关重要(附加档案14.).一个例子是BZIP113(GRMZM2G445575),其与拟南芥中的TGACG基序系数(TGA)家族同源。已经表明,参与开花的TGA基因[52],生物应激[53]和氮信号[54].这36个转录因子是玉米广泛转录调控因子的候选因子。简而言之,我们的数据表明,每个组织可能使用独特的转录因子作为关键调控因子,可以通过网络分析加以识别。
拓扑和聚类分析
为了描述组织特异性grn的拓扑结构,每个网络的拓扑特征由NetworkAnalyzer计算[55].已经证明,健壮的生物网络往往具有符合幂律分布的无标度结构[55,56].由于GRNS是针对网络(TFS调节目标基因),因此仅计算出外节点度分布。对于所有四个组织GRN,Out-Node分布的连接性适用于幂律,R线值范围为0.398至0.601(附加文件15.),建议我们的GRNS是强大的。这些R线值低于针对我们之前优化的玉米GCN确定的值。这可能与使用TFS作为监管机构的GRN的事实有关,这往往具有比权力分布所预测的相互作用更高。
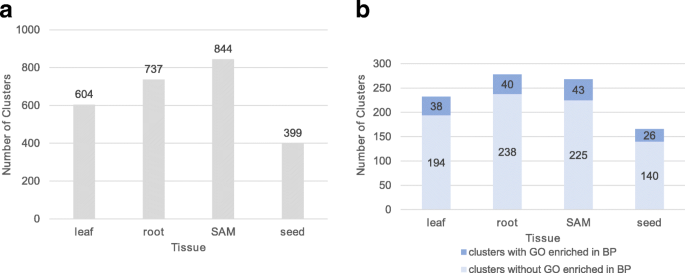
其次,为寻找功能模块,采用Markov聚类算法(MCL)对grn进行分区[57[基于网络拓扑的无监督聚类算法。该方法已成功应用于玉米和其他植物物种[58,59,60,61].叶片,根,SAM和种子中的MCL分化的604,737,844,399模块,表明这些组织中是功能性模块(图。7).其中,232个(叶片GRN)、278个(根GRN)、268个(SAM GRN)和166个(种子GRN)具有10个以上的基因(图2)。7),因此易于进行氧化石墨烯分析。我们使用g:profile对这些944个模块进行了GO富集分析[62].我们发现被富集用于至少一个生物处理工艺(BP)GO术语,156个模块。在各组织中,最大模块是富集的基因,将有可能以支持组织的生物学:光合作用(叶),翻译(根),蛋白质分解过程(SAM)和蜂窝氨基酸代谢过程(种子)。在叶最大的模块从深层次看在发电前体代谢物和能源的富集表现(p-value = 7.65E-08)、类胡萝卜素生物合成过程(p-value = 1.59E-04)和蓝光响应(p-value = 5.87E-04)(附加文件16.).这表明MCL可以恢复各组织中生物相关的模块。每个组织模块的基因列表及其氧化石墨烯富集可以从我们的网站下载。
网站设计
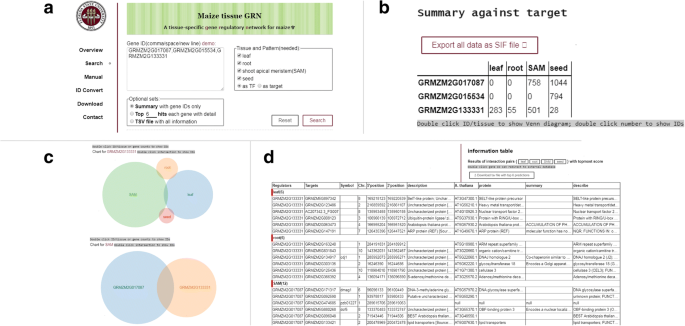
要分享我们的结果,我们构建了一个用户友好的网站,玉米组织特异性基因监管网络(MGRN,http://www.bio.fsu.edu/mcginnislab/mgrn.)使用MySQL和PHP。它提供搜索,可视化和下载服务(图。8).用户可以在特定于组织特定GRNS中搜索用于查询基因的TF调节目标(AS TF)或TF调节剂(作为靶标)(图。8).默认情况下,提供一个预测调控相互作用数量的汇总表(图。8 b).双击数字将链接到每个类别中的基因ID。如果有两到四个交叉点,则双击组织或基因将发射交互式Venn图(图。8 c).重叠基因id和基因数量可以从图中检索。详细的教程提供了如何在Cytoscape和R中可视化我们的网络结果(附加文件17.).
可以在网上捕获具有详细信息的顶部目标基因(图。8 d).具有预测目标的TF调节剂是前两列。为了更好地理解基因功能,本文基于AGPv3.31提供了靶标或调控因子的基因位置和描述,以及来自BLASTP的最佳匹配拟南芥基因注释。双击“调节器”、“目标”或“A。thaliana "基因id重定向到GRASSIUS [5], MaizeGDB [63]或Araport [64],以便于挖掘。所有搜索结果以及整个网络都可以从网站上下载以作进一步分析。到目前为止,我们的数据库只接受玉米版本3的基因id。一个“ID转换”工具用于在玉米版本4基因ID和版本3基因ID之间转换。
讨论
玉米基因表达阿特拉斯[34[描述了一半的组织特异性表达,对玉米中的一半基因。这表明基因表达的组织特异性是生物学上重要的。在这项研究中,我们使用机器学习算法,Genie3构建了来自RNA-SEQ表达数据的玉米GRN,用于叶,根,SAM和种子组织。还有其他GRN施工方法,但选择Genie3进行分析,因为它只需要基因表达数据,并且不需要对数据分布的假设。基于贝叶斯的方法,如BNFinder [65,需要精细的遗传扰动或时间序列设计。两者都很难从公共数据中获得。GENIE3还具有揭示TF与靶点之间非线性关系的能力,其中基于Pearson相关系数(PCC)和Spearman相关系数(SCC)的方法检测线性关系[66].第三,与贝叶斯方法不同,GENIE3可以发现生物网络中普遍存在的反馈回路。例如,反馈回路CLAVATA和Wuschel.在拟南芥和玉米中都有关于控制SAM发育的报道[67,68,69].与基于相关和互信息(MI)的方法不同,GENIE3预测了调控的方向,因为它生成了两个用于基因建模的值我来j和j来我.GENIE3的作者表明,考虑到方向性,他们的方法明显优于MRNET和CLR [24],根据一组独立评估者的公正评估,GENIE3是DREAM5挑战中表现最好的[25].当我们将GENIE3与两种基于mi的方法MRNET和CLR进行比较时,AUROC和AUPR值有微小的差异(附加文件3.)这并没有超过Genie3的优势。
使用公共可用的RNA-SEQ数据,我们以类似的阳性率预测组织特异性TF相互作用与阿特拉斯GRN研究[27].我们的GRNS基于使用TF芯片SEQ数据的评估来表现良好。该研究用2241 TFS生成GRN,提供足够高的分辨率,以揭示基因调控的空间变化。
在我们的分析中,我们发现相互作用的80%,是唯一的一个组织在玉米基因虽然超过80%和TFS的50%以上在所有四个组织中表达。此外,TF表达与互动的次数有关。这表明基因之间的相互作用可能提供可以不与变异的基因表达单独解释组织特异性功能的机构。这个良好的相关性与一个最近的研究在人类表明转录因子具有均匀的表达,但差异结合的目标,以支持组织特异性功能[70].此前已有报道称,人类的转录调节网络可以是细胞类型特异性的[71].
与以往的玉米大规模研究相比,我们利用网络在多种组织中阐明基因调节,并确认先前已发表过一些转录因子的功能模式[3.,72].例如,TF myb相关蛋白-1 (MRP-1)只包含在我们的种子特异性GRN中,而我们的网络包含了另一项研究中预测由MRP-1调控的93个基因中的51个[4].这证实了网络分析可以用于发现组织特异性TF相互作用。我们还将我们的网络预测与PlantRegMap进行了比较,发现它们都对chip确认的目标富集。通过从表达数据推断grn,我们不像PlantRegMap那样依赖高质量的pwm,因此预测可以扩展到更多的tf。此外,通过使用来自多个库的表达数据,我们可以关注空间和/或时间特定的调控相互作用。
结论
在这项研究中,我们为玉米构建了四种组织特异性GRN,包括叶,根,山姆和种子。使用与实验数据和生物功能富集的比较评估这些GRN的质量。这些评估表明组织特异性GRN预测高置信TF监管目标。我们提供了预测的TF-Target相互作用的例子,以具有组织特异性功能。还鉴定了功能模块,可以在组织水平提供基因簇信息。为了我们的知识,这是玉米TF调控网络中的玉米系统研究,表明使用统计方法推断GRN可以扩大我们对基因调控的知识并避免植物中某些基因组技术的局限性。让我们的发现更可访问,一个MGRN Web数据库(http://www.bio.fsu.edu/mcginnislab/mgrn.)建,包括基因功能,并链接到其他门户网站。我们希望我们的研究结果可以进一步促进基因调控的研究。此外,我们的构建组织特异性的GRNs框架也可以被应用到具有丰富的全基因组表达数据的其它生物体。
方法
RNA-Seq数据收集和处理
RNA-Seq文库的处理如前所述[33].简而言之,RNA-Seq样本从NCBI SRA下载[73,在SRA Toolkit 2.5.2中通过fastq-dump命令转换为fastq格式。适配器被Cutadapt 1.8.1修剪[74].然后通过HISAT2 v2.0.4比对玉米基因组AGPv3 (Ensembl Plant release 31) [75].采用featurecots 1.5.0检测基因表达[76],然后用m值修剪均值(TMM)归一化[77]并在log2中报告据标准化计数每百万(CPM)。总之,针对每个组织分析394(叶),176(根),406(SAM)和159(种子)文库。
网络建设
到过滤器低表达基因,与在每个组织文库的10%以上的小于1个CPM基因被排除在外。基因网络推理的树木合奏(GENIE3)算法[24]用于构建组织特异性基因调节网络(GRNS),更具体地说,在R和C中实施的版本在G Grassius中在玉米基因组中实现的(2587 TF)[5]被指定为候选监管机构。
CHIP-SEQ确定的目标和网络评估
对于KN1和FEA4,芯片-SEQ目标从原始文件下载[41,42].对于O2, ChIP-Seq summit文件从Gene Expression Omnibus下载(http://www.ncbi.nlm.nih.gov/geo.),加入编号为GSE39161。为了与KN1和FEA4的标准保持一致,我们将峰峰10 kb以内的基因定义为ChIP-Seq鉴定靶标。
我们使用ROC曲线下面积(AUROC)和Precision-Recall曲线下面积(AUPR)来评价网络质量。以KN1、FEA4和O2 ChIP-Seq鉴定的靶标作为阳性集。值由R [78].为了产生随机网络,允许靶基因10,000次次数,并且对应于AuroC和AUPR值进行平均。使用Fisher.Test()函数在R中计算单尾Fisher的确切测试。从原文中下载了Atlas mRNA和蛋白质GRN [27].PlantRegMap预测的KN1指标名单已从其网站(http://plantregmap.cbi.pku.edu.cn.).Venn图被Venn(http://bioinformatics.psb.ugent.be/webtools/Venn/).通过干预绘制镦锻图[79].
基因本体论丰富和同源性分析
GO富集由g:Profiler (version Ensembl Genomes 31)的基因组功能分析(GOST)工具进行分析[62].P- 由Fisher的单尾测试计算和通过设定的计数和大小(SCS)方法来计算,用于多次测试。报告了P值的术语只有小于0.05。从Biomart检索拟南芥同源物(Ensembl Genomes 31)。
模块检测和表征
用于组织的GRNs(顶部百万边缘)网络拓扑的表征通过NetworkAnalyzer [计算55]在Cytocscape [80].Markov Cluster算法(MCL)检测模块[57[通货膨胀值设置为2.5。基因列表被读入r并由GPROFILER分析(https://cran.r-project.org/web/packages/gProfileR)包富集。
网站设计
Web数据库(https://www.bio.fsu.edu/mcginnislab/mgrn/)使用MySQL和PHP构建。从Biomart上检索玉米基因描述,在Ensembl植物版本31中。基因符号基于MaizeGDB的注释。拟南芥基因描述从Tair10下载。BLASTP使用当地BLAST + 2.2.28完成[81].玉米基因ID转换从甘草(ftp://ftp.grachene.org/pub/gramene/rampes/past_releases/release-56/gff3/zea_mays/gene_id_mapping_v3_to_v4/).所有数据和源代码可在Github (https://github.com/timedreamer/maize_tissue-specific_GRN).
缩写
- AUPR:
-
精密召回曲线下的区域
- 奥克托克:
-
接收算子特征曲线下的面积
- ChIP-Seq:
-
染色质免疫沉淀测序
- CLR:
-
相关性的情境可能性
- CPM:
-
每百万计数
- 简历:
-
变异系数
- DAP-seq:
-
DNA亲和纯化测序
- Genie3:
-
基因网络推论树木的集合
- 去:
-
基因本体论
- GRASSIUS:
-
草监管信息服务
- grn:
-
基因调控网络
- MCL:
-
马尔可夫聚类算法
- MGRN:
-
玉米组织特异性基因监管网络
- 小姐:
-
相互信息
- MRNET:
-
最小冗余网络
- PBM:
-
蛋白质结合微阵列
- PCC:
-
皮尔森相关系数
- PWM:
-
位置称重矩阵
- 山姆:
-
拍摄顶端公司
- 鳞状细胞癌:
-
斯皮尔曼相关系数
- SELEX:
-
指数富集的配体系统演化
参考
- 1。
Basso K,Margolin AA,Stolovitzky G,Klein U,Dalla-Favera R,Califano A.人类B细胞中调节网络的逆向工程。NAT Genet。2005; 37:382-90。自然出版集团
- 2.
Marbach D, Prill RJ, Schaffter T, Mattiussi C, Floreano D, Stolovitzky G.揭示基因网络推断方法的优缺点。acta Natl academy Sci . 2010; 107:6286-91。
- 3.
Li P,Ponnala L,Gandotra N,Wang L,Si Y,Tausta Sl,等。玉米叶片转录组的发育动态。NAT Genet自然研究。2010; 42:1060-7。
- 4.
Zhan J,Thakare D,Ma C,Lloyd A,Nixon Nm,Arakaki Am,等。激光捕获的MAIZE核的激光捕获微小隔室的RNA测序识别与胚乳细胞分化相关的调节模块。植物细胞。2015; 27:513-31。可以从:http://www.ncbi.nlm.nih.gov/pubmed/25783031.
- 5.
Yilmaz A,Nishiyama My,Fuentes BG,Souza Gm,Janies D,Gray J等人。Grassius:草地上对比较监管基因组学的平台。植物Physiol AM Soc植物Biol。2009; 149:171-80。
- 6.
李涛,王志强,李志强,等。玉米tome -功能基因组学中转录因子开放阅读框的开发。植物j . 2014; 80:356 - 66。
- 7.
朱刚,吴安,徐晓军,肖鹏,陆璐,刘军,等。玉米蛋白质相互作用数据库。植物杂志。2016;170:15.01821。可以从:http://www.plantphysphysiol.org/content/early/2015/11/30/pp.15.01821.Abstract..
- 8.
王丽丽,王鹏,李明杰,秦军,王旭,张明敏,等。EpiRegNet:利用人类高通量基因表达数据构建表观遗传调控网络。表观遗传学。Taylor & Francis. 2011; 6:1505-12。
- 9.
可汗,Fornes O, Stigliani A, Gheorghe M, Castro-Mondragon JA, van der Lee R, et al.;JASPAR 2018:转录因子结合概况开放存取数据库及其web框架的更新。核酸Res. 2017;46; 260-66。https://www.ncbi.nlm.nih.gov/pubmed/29140473.
- 10。
杨志强,杨志强,杨志强,等。真核生物转录因子序列特异性的测定和推断。细胞。2014;158:1431-43。elewsvier。
- 11.
Chen X,Ernst K,Soman F,Borowczak M,Weirauch Mt。Cressint:用于拟南芥基因调控的基因组规范的用户友好的网站资源。Curr植物BIOL。2015; 3:48-55。elewsvier。
- 12.
财团EP。人类基因组中DNA元素的完整百科全书。《美国国立卫生研究院公共通道》2012;489:57。
- 13.
Pruneda-Paz JL,Breton G,Nagel DH,Kang Se,Bonaldi K,Doherty CJ等。Arabidopsis转录因子功能表征的基因组规模资源。细胞批准。2014; 8:622-32。elewsvier。
- 14.
欧波,尹克强,刘世宁,杨勇,顾婷,惠建伟,等。拟南芥转录因子的高通量筛选系统及其在med25依赖的转录调控中的应用摩尔。2011;4:546-55。elewsvier。
- 15.
Omranian N,Eloundou-MBEBI JMO,Mueller-roeber B,Nikoloski Z.基因调节网络推论在多个数据集上使用熔融套索。SCI REP自然。2016; 6:20533。出版集团
- 16.
阳楼,李伟,江N,俞H,诸桥K,欧码WZ等。玉米基因调控网络的酚类物质代谢。莫尔植物。2017; 10:498-515。
- 17.
柴志强,谢志强,谢志强。基于指数富集的配体系统进化研究(SELEX),作为蛋白质- dna相互作用空间破译的有力工具。植物转录因子方法2011:249-58。施普林格。https://link.springer.com/protocol/10.1007/978-1-61779-154-3_14.
- 18.
Franco-Zorrilla JM,López-Vidriero I,Carrasco JL,Godoy M,Vera P,Solano R.植物转录因子的DNA结合特异性及其定义靶基因的潜力。PROC NATL ACAD SCI国家舆论科学。2014; 111:2367-72。
- 19.
黄绍山,宋丽,刘志强,等。顺向和Epicistrome的特征塑造了调控DNA的景观。细胞。2016;166:1598。可以从:http://dx.doi.org/10.1016/j.cell.2016.04.038爱思唯尔有限公司
- 20.
Yin Y,Morgunova E,Jolma A,Kaasinen E,Sahu B,Khund-Sayeed S等人。胞嘧啶甲基化对人转录因子DNA结合特异性的影响。科学。2017; 356:EAAJ2239。(80-。)。美国科学进步协会
- 21.
金杰,田F,杨立德,孟悉,孔L,罗杰,等。Planttfdb 4.0:朝向植物中的转录因子和监管相互作用的中心集线器。核酸RES。2017; 45:D1040-5。可以从:https://academer.oup.com/nar/article/45/d1/d1040/2290936.
- 22.
周志宁,郑华强,吴宁勇,钱志华,黄宏东,李天勇,等。PlantPAN 2.0:重建植物转录调控网络的植物启动子分析导航仪的更新。核酸Res牛津大学出版社,2016;44:D1154-60。
- 23.
植物顺式作用调控DNA元件(PLACE)数据库:1999。核酸Res. 1999; 27:297-300。牛津大学出版社
- 24.
黄长发至周四VA,Irrthum A,Wehenkel L,使用基于树的方法表达数据Geurts P.推断的调控网络。Plos一个。2010; 5:1-10。
- 25.
Marbach D,Costello JC,KüffnerR,Vega NNM,Prill RJ,Camacho DM等。鲁棒基因网络推理的人群智慧。NAT方法。2012; 9:796-804。
- 26.
Ezer D,Shepherd Sjk,Brestovitsky A,Dickinson P,Cortijo S,Charoensawan v等。拟南芥的G字箱转录规范代码。植物Physiol AM Soc植物Biol。2017年:01086。
- 27.
Walley JW,Sartor Rc,Shen Z,Schmitz RJ,Wu KJ,Urich Ma,等。在湄化网络中的群体集成群体(80-。)。美国泰国科学局部协会。2016; 353:814-8。可以从:http://science.sciencemag.org/content/353/6301/814.
- 28.
刘志强。分类与回归树。Wiley跨学科Rev Data Min know Discov. 2011; 1:14-23。威利在线图书馆。
- 29.
Hastie T,Robert Tibshirani JF。统计学习的元素:数据挖掘,推理和预测,第二版(Springer系列统计数据)。2埃德:斯普林克;2009年。https://www.springer.com/us/book/9780387848570.
- 30.
Geurts P. Dyngenie3:动态Genie3从时间序列表达数据中获取基因网络的推断。SCI REP自然出版集团。2018; 8:3384。
- 31。
Aibar S,González-Blas CB,Moerman T,Imrichova H,Hulselmans G,Rambow F等人。风景:单细胞调节网络推论和聚类。NAT方法。2017; 14:1083。自然出版集团
- 32。
Banf男,李承晚SY。通过与马尔科夫随机字段中的数据集成增强的基因调控网络的推理。SCI代表2017; 7:41174。自然出版集团。
- 33。
黄Ĵ,文德拉明S,施L,麦金尼斯KM。建设与玉米使用RNA-Seq的数据的大基因共表达网络的优化。植物杂志。2017; 175:568 LP-583。可以从:http://www.plantphysphysiol.org/content/175/1/568.Abstract..
- 34。
王志强,王志强,王志强,等。玉米发育过程中转录的全基因组图谱。植物j . 2011; 66:553 - 63。
- 35。
Nardmann J, Ji J, Werr W, Scanlon MJ。玉米的重复基因narrow sheath1和narrow sheath2在茎尖分生组织的侧域编码一个保守的同源盒基因功能。生物科技有限公司2004;131:2827-39。
- 36。
黄志强,王志强,王志强,等。玉米节状根形成缺陷突变体rtc的分离与鉴定。植物j . 1996; 10:845-57。威利在线图书馆。
- 37.
黄志强,王志强,王志强,等。大刍草分支1基因的表达与玉米和大刍草的生长抑制相关。遗传学报。2002;42(4):429 - 434。
- 38.
McCarty Dr,Carson CB,Stinard PS,Robertson DS。ViviParous-1的分子分析:玉米的脱落酸不敏感突变体。植物细胞AM SOC植物BIOL。1989; 1:523-32。
- 39.
Meyer Pe,Kontos K,Lafitte F,Bontempi G.大转录监管网络的信息理论推断。EuraSip J Bioinforma Syst Biol。2007; 8-8。https://www.ncbi.nlm.nih.gov/pubmed/18354736..
- 40。
信仰JJ,Hayete B,Thaden JT,Mogno I,Wierzbowski J,Cottarel G,等。大规模映射和验证表达型材概要的大肠杆菌转录规范。Plos Biol。2007; 5:0054-66。
- 41。
Bolduc N,耶尔马兹A,希亚-格拉MK,诸桥K,O'Connor的d,Grotewold E,等人。揭开玉米分生组织的KNOTTED1调控网络。基因开发。2012; 26:1685至90年。[引用的2014 2月5日]
- 42。
杨凤英,杨凤英,杨凤英,周志强,等。FASCIATED EAR4编码调控玉米茎分生组织大小的bZIP转录因子。植物学报。2015;2 tpc.114.132506。可以从:http://www.plantcell.org/lookup/doi/10.1105/tpc.114.132506.
- 43。
李超,乔志,齐伟,王强,袁勇,杨旭,等。顺式作用DNA靶点的全基因组特征揭示了玉米中Opaque2的转录调控框架。植物细胞。2015;27:1-15。
- 44.
Stelpflug sc,Rajandeep s,Vaillancourt b,hirsch cn,buell cr,de ln,等。基于RNA测序的扩展玉米基因表达式地图集及其用于探索根部发育。植物基因组。2015:314-62。
- 45.
贾斯,贾S.重新审议了杂草。NAT Rev Genet。2007; 8:35-46。自然出版集团
- 46.
法律J A,Jacobsen SE。建立,维持和修饰植物和动物的DNA甲基化模式。NAT Rev Genet。自然出版集团;2010年[引用2014年7月9]; 11:204-220。
- 47.
Huang J,Lynn JS,Schulte L,Vendramin S,McGinnis K.玉米基因表达的两端表达控制。INT Rev Cell Mol Biol。2017; 328:25-48。elewsvier。
- 48.
Sullivan AM, Arsovski AA, Lempe J, Bubb KL, Weirauch MT, Sabo PJ,等。调控DNA和转录因子网络的定位和动态答:芥.细胞众议员2014;8:2015-30。爱思唯尔可以从:http://dx.doi.org/10.1016/j.celrep.2014.08.019
- 49.
De Witte D,Van de Velde J,Decap D,Van Bel M,Audenaert P,Demeester P等人。BLSSPELLER:保守的CIS-MOVICETION元素的详尽比较发现。生物信息学。2015; 31:3758-66。
- 50.
在拟南芥中,调控SBP-box基因SPL9和SPL15的microRNA调控茎成熟。acta botanica sinica(云南植物学报)2008;
- 51.
李Z,李斌,沉W,黄鹤,董A.TCP转录因子与AS2相互作用,征征在拟南芥中瘤瘤基因的抑制。工厂J. 2012; 71:99-107。Wiley在线图书馆
- 52。
宋耶,宋纽约,胫骨,金HJ,云D-J,LIM CO等。作为TGA4 / OBF4相互作用蛋白的致致硫的分离。Mol细胞(Springer SCI。总线。媒体BV)。2008; 25:559-65。
- 53。
Després C, Chubak C, Rochon A, Clark R, Bethune T, Desveaux D, et al.;拟南芥NPR1抗病蛋白是一种新型的辅助因子,它将DNA结合活性的氧化还原调节作用赋予基本结构域/亮氨酸拉链转录因子TGA1。acta botanica sinica(云南植物学报)2003;15:2181-91。
- 54。
Alvarez JM,Riveras E,Vidal EA,Gras de,Contreras-Lópezo,Tamayo Kp等人。系统方法识别TGA1和TGA4转录因子作为拟南芥根系硝酸盐反应的重要调节组分。工厂J. 2014; 80:1-13。
- 55。
生物网络和蛋白质结构的拓扑分析和交互可视化。Nat Protoc。2012;7:670 - 85。自然出版集团;可于以下地点索取:http://www.ncbi.nlm.nih.gov/pubmed/22422314.
- 56。
Barabasi A-L, Oltvai ZNZN, Barabási A-L。网络生物学:了解细胞的功能组织。Nat Genet.2004牧师;5:101-13。可以从:http://www.ncbi.nlm.nih.gov/pubmed/14735121.
- 57。
Van Dongen S.通过流动模拟绘制图形聚类。博士论文。乌得勒支大学;2000年。https://micans.org/mcl/index.html?sec_thesisetc.
- 58。
Li L,Briskine R,Schaefer R,Schnable PS,Myers Cl,鞭毛Le等。玉米(Zea mays L.)中的重复基因的共表达网络分析揭示了没有亚基因组偏差。BMC基因组学。2016; 17:875。可以从:http://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-016-3194-0.
- 59。
Guerin C, Joët T, Serret J, Lashermes P, Vaissayre V, Agbessi MDT,等。油棕种间回交油脂生物合成的基因共表达网络分析。j . 2016:1-19植物。可以从:http://www.ncbi.nlm.nih.gov/pubmed/27145323.
- 60.
Baute J,Herman D,Coppens F,De Block J,Slabbinck B,Dell'Acqua M等。玉米RIL群中成熟叶片参数生长叶片转录组的相关分析。基因组Biol。2015; 16:168。可以从:http://genomebiology.com/2015/16/1/168.
- 61。
Wong DCJ,Sweetman C,Ford Cm。基因表达信息和共表达网络注释柑橘中基因函数。BMC植物BIOL。2014; 14:186。可以从:http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=4108274&tool=pmcentrez&rendertype=abstract.
- 62。
Reimand J,Arak T,Adler P,Kolberg L,Reisberg S,Peterson H等人。g:profiler-a Web服务器,用于基因列表的功能解释(2016年更新)。核酸RES。2016; 44:W83-9。牛津大学出版社
- 63。
Andorf CM, Cannon EK, Portwood JL, Gardiner JM, Harper LC, Schaeffer ML,等。MaizeGDB更新:玉米模型生物数据库的新工具、数据和接口。核酸学报2015;44:D1195-201。牛津大学出版社。
- 64。
拟南芥参考基因组的一个完整的重新注释。植物j . 2017; 89:789 - 804。
- 65。
Wilczyński B, Dojer N. BNFinder:学习贝叶斯网络的精确和有效的方法。生物信息学。2008;25:286-7。牛津大学出版社
- 66。
德Siqueira桑托斯S,高桥DY,中田A,用于鉴定基因表达的信号之间的相关性的统计方法藤田A.比较研究。简短生物形式。2013; 15:906-18。牛津大学出版社
- 67。
somsich M, Je B. Il, Simon R, Jackson D. CLAVATA-WUSCHEL信号在茎分生组织中的作用。发展。2016;143:3238-48。牛津大学出版社生物学家有限公司。
- 68.
品牌U,Fletcher JC,Hobe M,Meyerowitz Em,Simon R.干细胞命运在拟南芥中的依赖性对CLV3活性调节的反馈回路。科学(80-。)。2000; 289:617-9。美国科学进步协会。
- 69.
Schoof H,Lenhard M,Haecker A,Mayer Kfx,JürgensG,Laux T.拟南芥芽分泌的干细胞群由Clavata和WUSchel基因之间的调节环保持。elthingvier。2000; 100:635-44。
- 70。
陈志勇,陈志勇,陈志勇,等。了解组织特异性基因调控。细胞众议员21:1077 2017;88年。ElsevierCompany。可以从:http://linkinghub.elsevier.com/retrieve/pii/S2211124717314183.
- 71.
尼弗S,Stergachis Ab,Reynolds A,Sandstrom R,Borenstein E,Stamatoyannopoulos Ja。人转录因子监管网络的电路和动力学。细胞。2012; 150:1274-86。elsevier公司;可以从:http://dx.doi.org/10.1016/j.cell.2012.04.040.
- 72.
陈继,曾B,张米,谢S,王g,哈克A等。玉米胚胎和胚乳发育的动态转录景观。植物杂志。2014; 166:252-64。可以从:http://www.pubmedcentral.nih.gov/articlerender.fcgi:artid=4149711&Tool=Pmcentrez&rendertype=abstract..
- 73.
Leinonen R,Sugawara H,Shumway M.序列读取存档。核酸RES。2010; GKQ1019牛津大学新闻
- 74.
Martin M.Cutadapt从高吞吐量测序读取中删除适配器序列。嵌入j11111; 17:10。
- 75.
Kim D,Langmead B,Salzberg Slhisat。具有低内存要求的快速拼接对齐器。NAT方法。2015; 12:357-60。自然出版集团。
- 76。
Liao Y,Smyth GK,Shi W. FeatureCounts:用于分配序列的有效通用程序读到基因组特征。生物信息学。2014; 30:923-30。牛津大学出版社。
- 77。
罗宾逊医学博士,麦卡锡DJ,史密斯GK。edgeR: Bioconductor包,用于数字基因表达数据的差异表达分析。生物信息学。2010;26:139-40。牛津大学出版社
- 78。
[J] .生物信息学杂志,2015,31(6):641 - 646。牛津大学出版社
- 79。
干预:交叉和可视化多基因或基因组区域集的工具。BMC生物信息学。生物医学。2017;18:287。
- 80.
Shannon P,Markiel A,Ozier O,Baliga NS,Wang JT,Ramage D等人。Cytoscape:用于生物分子交互网络的集成模型的软件环境。基因组res cold。2003; 13:2498-504。春天港口实验室。
- 81.
卡马乔C,库鲁利斯G, Avagyan V, Ma N, Papadopoulos J, Bealer K,等。BLAST+:架构和应用程序。BMC生物信息学。2009; 10:421。
- 82.
王志强,王志强,王志强,等。玉米种子根原基形成过程中非同同基因对胚胎转录组的调控作用。J Exp Bot. 2016; 68:403-14。牛津大学出版社
- 83.
Doebley J,Stec A,Hubbard L.玉米顶级优势的演变。自然。1997年; 386:485。自然出版集团。
- 84.
李启波,张立新,张立新,等。胎生菌1通过调控脱落酸信号改变基因表达模式。植物杂志。植物生物学杂志。2003;132:1664-77。
- 85。
张Z,郑X,杨姬,乱七八宫玉米胚乳特异性转录因子O2和PBF网络调节蛋白质和淀粉合成的调节。Proc。Natl。阿卡。SCI。国家院科学院。2016; 201613721。
- 86。
Muñiz LM, Royo J, Gómez E, Barrero C, Bergareche D, Hueros G.玉米转移细胞特异性a型反应调节剂ZmTCRR-1似乎参与细胞间信号传导。植物j . 2006; 48:17-27。威利在线图书馆。
确认
我们感谢亚历山大学习和Joshua M. McCoy从生物科学系的计算机支持设施,用于建立计算和Web服务器环境。
资金
这项工作是由美国国家科学基金会,BIO-MCB-035919卡伦·M.·麦金尼斯成为可能的资金。
数据和材料的可用性
本研究中生成和/或分析的所有数据都包含在本文中,或者可以从MGRN网站下载(https://www.bio.fsu.edu/mcginnislab/mgrn/).源代码和数据可在GitHub中获得(https://github.com/timedreamer/maize_tissue-specific_GRN).
作者信息
从属关系
贡献
JH和KMM设计了实验;JH进行实验。JH和YH分析了数据;JH、KMM和YH对数据进行了解释;JZ和JH制作了网站;JH和KMM写了这篇文章。所有作者均已阅读并批准本稿件。
通讯作者
道德声明
伦理批准和同意参与
不适用
利益争夺
Karen M. McGinnis是BMC植物生物学的助理编辑。其他提交人声明没有竞争利益。
附加文件
附加文件1:
在此分析中使用的RNA-SEQ库。(XLSX 71 KB)
附加文件2:
四种组织GRNS中组织特异性基因进行富集分析。(XLSX 23 KB)
附加文件3:
评估MRNET和CLR产生的组织GRN。(XLSX 9 KB)
附加文件4:
维恩图显示了每个组织特异性GRN的顶部100万条边之间的重叠。(PDF 55 kb)
附加文件5:
四个组织grn共享的2679条边中包含的353个TFs和1657个靶点。(XLSX 70 kb)
额外的文件6:
在四个组织GRNS中进行1657个保守靶标的浓缩分析。(XLSX 22 KB)
额外的文件7:
GO富集分析KN1, FEA4和O2靶标在四个组织grn。(XLSX 19 kb)
额外的文件8:
预测与芯片-SEQ的TF靶重叠与KN1,FEA4和O2的确认结合基因。叶子,根,山姆和种子是指我们的组织GRN。“蛋白质”和“RNA”是指来自Walley等人的蛋白质Grn和RNA。(2016)数据集。“大型网络”在KN1,FEA4和O2网络中使用前1000万边。“中网络”使用前100万边,而“小网络”使用前100,000个边缘。“阿特拉斯Grn媒体”从Walley等人使用前100万根边缘。(2016)数据集,而“atlas grn small”使用前100,000个边缘。(XLSX 13 KB)
额外的文件9:
比较组织特异性GRN和图谱GRN预测靶点与ChIP-Seq识别靶点重叠百分比。在本研究中,叶、根、SAM和种子grn是网络。Walley等人构建了mRNA和蛋白质网络。中等网络(浅灰色)是在前100万边缘内的目标。小网络(深灰色)的目标在10万边缘之内。(PDF 77 kb)
附加文件10:
基因表达(CPM计算)对(a)叶GRN,(B)根GRN,(C)SAM GRN,(D)种子GRN的TFS相互作用的影响。线性回归用蓝线绘制,灰色带作为95%置信区间。R2和p-值由R.的lm()函数从线性模型计算得到(pdf914kb)
附加文件11:
从每个组织的前100万个边缘预测TF靶点。“max _tissue”是指哪些组织具有最多的交互作用。CV是方差系数。(XLSX 76 kb)
额外的文件12:
(a)叶grn,(b)根grn,(c)sam grn和(d)种子grm中,TFS中TFS的前100万边缘的程度中心(靶数)。显示TF的红线与程度中心> 2000.(PDF 338 KB)
额外的文件13:
关键的tf在四个组织。(XLSX 12 kb)
额外的文件14:
拟南芥中两个以上组织共享TF的同源物。未被BioMart鉴定的同源基因为空白。(XLSX 11 kb)
额外的文件15:
四个组织grn的平均邻域连通性。将所有tf的平均邻域连通性分布与邻居数进行对比。在每个网络中,选取排名前100万的边。红色曲线为幂律拟合分布。R2值表明具有幂律模型的适应性。(PDF 2647 KB)
额外的文件16:
MCL检测到最大模块的浓缩分析。(XLSX 17 KB)
额外的文件17:
在Cytoscape和R中可视化MGRN数据的简短教程(HTML 3228 KB)
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
黄军,郑军,袁华。等等。基因调控网络在玉米中揭示的不同组织特异性转录调节。BMC植物杂志18,111(2018)。https://doi.org/10.1186/s12870-018-1329-y
已收到:
接受:
发表:
关键词
- 玉米
- 基因表达
- 转录调控
- 转录因子
- 网络
- 生物信息学
- 系统生物学
- 机器学习
- 数据库