摘要
背景
替代剪接(AS)表示基因后转录后调节的真核生物广泛使用的机制。对花生的详细探索尚未记录。
结果
利用链特异性RNA-Seq技术分析了AS在花生4个样品(FH1-seed1、FH1-seed2、FH1-root和FH1-leaf)中的分布。AS影响了37.2%的多外显子基因。其中一些基因在整个植物中都经历了AS,而其他基因的影响是器官特异性的。总的来说,AS在种子中比在根或叶中更常见。AS的主要形式为内含子保留,且在转录起始位点和转录末端位点的AS在4个样本中均有发现。有趣的是,在受AS影响的基因中,大多数只经历了一种类型的事件。并不是所有的硅预测转录本都被翻译,这意味着这些转录本要么被降解,要么从翻译机制中分离出来。在参与脂肪酸代谢的基因中,约61.6%表现为AS。
结论
我们的报告一般来说,对花生基因的分析有显着贡献,这些结果尚未提及。与表格不同的具体职能需要进一步调查。
背景
在真核生物中,作为从单个编码序列产生多于一个转录物的过程,已经发展为普遍的转录后基因调节模式[1,2].与许多临床状况相关的有缺陷[3.,4遗传疾病[5,6,7]和老化[8].在植物中,正如已显示的调节生长,发育,信号转导,开花,昼夜节日功能和对各种环境提示的响应[9,10.,11.,12.,13.],也与物种形成有关[14.,15.,16.].如已认识到的五种形式,即转录起始网站(TSS),转录终端位点(TTS),外显子跳跃(ES),内含子保持(IR),替代外显子结束(5',3'或两者; AE)[17.].
高通量测序数据集为研究AS的全基因组分布提供了一个重要机会。迄今为止的印象是,AS的程度随着器官和物种的复杂性而增加[18.,19.].在动物基因组中,含有内含子的基因的比例在20-95%范围内的谎言中的体验[20.,21.,22.,23.,24.]但虽然植物基因组中的等效比例很大[25.,26.,27.,28.,29.,30.,31,32,33].在两个动物和酵母中,ES是最普遍的形式,而IR是最不常见的[22.,24.,34,35].相比之下,在植物中,大多数AS事件涉及IR,在单子叶/双子叶分裂中,五种AS形式的相对频率不同[25.,36,37,38].大约60-75%的AS事件导致基因产物的结合特性、磷酸化状态、稳定性、细胞内定位、酶活性或信号转导活性的变化[1,38,39,40].许多产生的额外的转录物由废义介导的腐烂处理或由microRNA(miRNA)调节[41,42,43,44].至少13%的内含子基因拟南芥蒂利亚纳可能受到无意义介导的衰变的调控[45].
花生(arachis hypogaea.l.),领先的油和蛋白质作物,是同种异体一体化(2n= 4×= 40)从两个二倍体野生物种之间的杂交开发A. Duranensis.和答:ipaensis.对这两个祖先物种的测序已经覆盖了约96%的栽培花生基因组[46,从而为基因研究提供了坚实的基础。到目前为止,花生中As的全基因组发生情况还没有被探索。在这里,利用链特异性RNA-Seq方法来描述AS发生在种子、幼苗根系和叶片两个不同发育阶段的特征。
结果
花生四个器官来源库的测序
从花生中提取了4个样本(FH1-seed1, FH1-seed2, FH1-root和FH1-leaf)进行链特异性rna测序。RNA-Seq平台实现了51.3 × 109基础,Q30值为88.90%,GC含量范围为44.48-50.48%(附加文件1:表S1)。编辑后,剩余的高质量序列用27.25 × 10表示9FH1-Seey1和FH1-Seey2的基础组合,12.42×109FH1-root和11.67 × 10 .9在FH1-leaf基地。原始序列数据已提交至NCBI BioProject数据库,登录号为PRJNA354652。当干净的基因序列与A. Duranensis.和答:ipaensis,匹配比率从79.91到87.49%(附加文件2:表S2);FH1-leaf库的比例最高,但唯一映射读的数量在该库中最低。在这四个库中,读更频繁地映射到' + '链而不是' - '链。
花生转录组
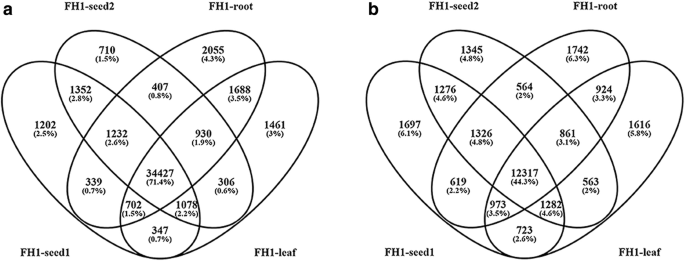
共有431,596个独特的转录物在FH1-Seef2,11.0910的四个样品中拼凑在四个样品,107,102,分别来自FH1-Root的109,210,以及来自FH1-叶的105,279(表1).检测到54,047个基因的转录物(表2),占两种野生祖细胞基因总数的68.8% [47].在将外显子的碎片映射到每百万片段(FPKM)阈值0.1的阈值后,检测到的基因数量降至48,236(表2).这些基因从FH1-Seey2,41,780的FH1-Seey1,40,442中突破了40,679,来自FH1根和40,939来自FH1-叶。在所有四种文库中检测到这些基因的34,427的转录,将1202个特异于FH1-Seef1,710至FH1-Seey2,2055至FH1-根和1461-叶(图。1A).
如在花生转录组
将FPKM阈值施加为0.1至48,236个转录基因的组揭示了27,829个基因,如表现为AS的证据(表2):这个数字代表了转录组的57.69%和全基因组序列预测的多外显子基因的37.2%。4个文库分别含有20,213 (FH1-seed1)、19,534 (FH1-seed2)、19,326 (FH1-root)和19,259 (FH1-leaf)受AS影响的基因。所有4个文库中AS基因共12317个,而文库特异性AS基因的数量分别为1697、1345、1742和1616(图1)。1B.).共有4318个基因被限制在从种子发育准备的两个文库中。总的来说,AS事件在种子中比在根或叶中更频繁3.),在种子中检测到的剪接亚型数量也多于根和叶(附加文件3.:表S3),暗示了种子开发中的特定重要性。
在FH1-seed1中检测到的AS事件数为92,483次,在FH1-seed2中检测到的AS事件数为85,562次,在FH1-root中检测到的AS事件数为79,763次,在FH1-leaf中检测到的AS事件数为83,290次3.).根据 [17.],可以识别出五种不同类型的AS事件,即TSS、TTS、ES、IR和AE(表1)2).在花生中观察到的事件(26.84-33.45%)涉及IR的最常见,频繁频繁(11.68-14.17%)。AE事件分别占,分别为20.06-22.07%。这些估计与以往的其他植物的研究一致。TSS和TTS事件分别占19.01-20.78%和15.70-17.25%。
TSS和TTS事件通常在四个样本中识别。在FH1种子的组合中,17,584个TSS事件影响了14,314个基因,14,523个事件影响了12,680个基因,每种基因产生的TSS和TTS事件的数量分别为1.22和1.15(表4).在四个样本内,每个基因产生的TS和TTS事件的数量分别为1.18-1.22和1.12-1.15(表4),每个基因产生的TSS事件数多于TTS事件数。
ES'事件'被定义为“开”和“关闭”之间的配对,在同一外显子和相同的侧面内含子。相同的外显子或内含子可能涉及多个外显子跳跃(MSKIP)事件,因此估计的ES事件数量超过其实数。在es事件中,Skip_off数量比SKIP_ON事件更高(表5).IR'事件'也被定义为配对(IR_ON,IR_OFF)。有趣的是,IR_ON事件的数量超过IR_OFF ONE(表6).
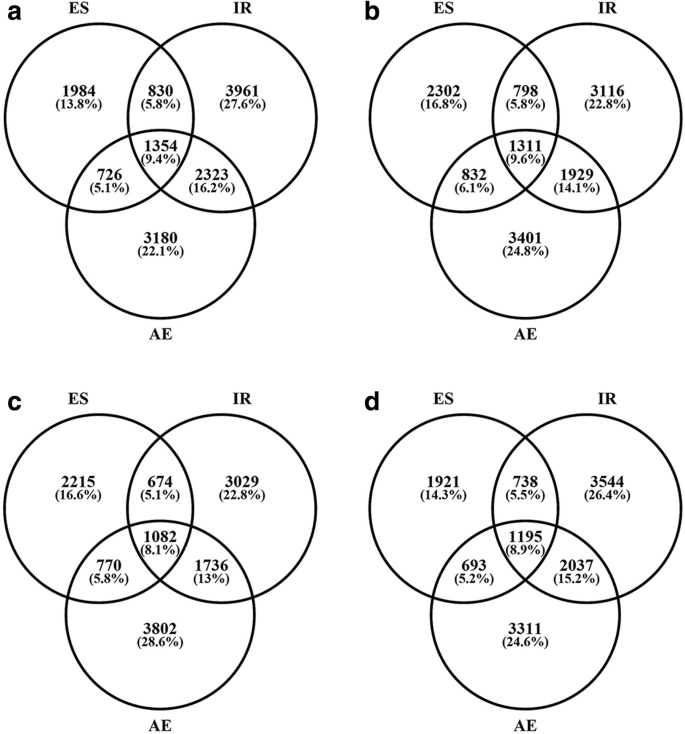
在一组fh1种子AS基因中,10891个ES事件影响4894个基因,30931个IR事件影响8468个基因,18554个AE事件影响7583个基因(表)2, 图。2).单独受ES、IR和AE影响的基因数分别为1984、3961和3180。共有9125个(63.55%)基因经历单一AS型,3879个(27.02%)基因经历两种AS型,只有1354个(9.43%)基因经历三种AS型。同时经历IR和AE的AS基因多于同时经历IR和ES、AE和ES的AS基因。其他三个文库的转录组显示了类似的AS事件分布(图。两个罪犯).总体而言,在受到影响影响的基因中,大多数人只经历了一系列事件。
大多数作为基因产生的主要转录物具有相当高的表达表达水平,在所有四个文库中检测到,以及以基本上更低的频率发生的附加情况(附加文件4:表S4)。例如,Aradu.000JC生成了四个转录本,其中两个在所有四个文库中检测到,一个在FH1-seed1和FH1-seed2中(但在FH1-root和FH1-leaf中均未检测到),第四个在FH1-root和FH1-leaf中(但在FH1-seed1和FH1-seed2中均未检测到)。在第二个例子中,Araip。GAW68生成了20份转录本,其中5份出现在所有4个图书馆,只有1份出现在3个图书馆,7份出现在2个图书馆,7份出现在一个图书馆。
花生AS基因外显子数的统计分析
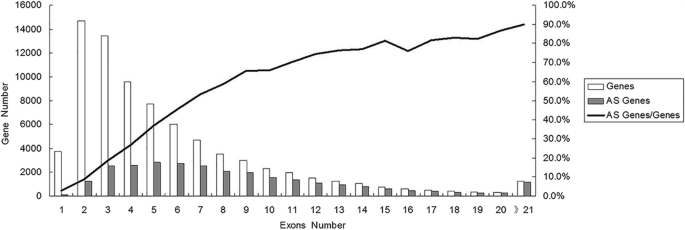
栽培花生含有两种亚因子(A和B)。虽然栽培花生的基因组测序尚未完成,但是花生两种祖细胞的基因组测序(A. Duranensis.和答:ipaensis),包含78,574个基因(分别为36,734和41,840个基因)[46].因此,我们使用两个野生花生基因组作为参考分析我们的转录组数据。我们分析了78,574个基因的外显子数(图。3.),发现内含内基因的数量为3740,占所有基因的4.76%;含有2个或更多个外显子的基因数量占总基因的约95%。47.26%基因含有≥5外显子,15.56%基因含有≥10个外显子。我们分析了外显子数为27,829(图。3.).结果表明,AS基因的外显子数主要分布在3 ~ 10个区间,占AS基因总数的67.33%。AS基因的发生率随外显子数的增加而增加。这与其他报告一致[48].
GO注释和对基因的花生分类分析
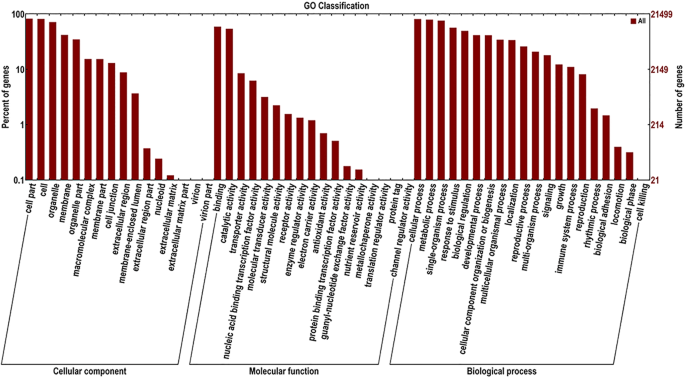
我们将基因函数注释进行了几个不同的数据库,例如NCBI非冗余蛋白序列(NR),蛋白质群(Kog / Cog),基因和基因组(Kegg)和基因本体(GO)的蛋白质群组群(GO).在这里,我们详细分析了Go注释,并作为基因的花生分类。作为基因的27,829次进行分类,导致21,499分配给GO类别(图。4).在“细胞成分”类别中,基因主要集中在“细胞部分”(22.3%)、“细胞”(22.1%)和“细胞器”(19.4%)三类。在“分子功能”类别中,主要的基团是“结合”(38.9%)和“催化活性”(42.5%);最后,在“生物过程”这一类别中,除了“细胞杀死过程”这一未被充分代表的类别外,基因在不同组之间的分布相当均匀。这意味着AS对于广泛的生物过程的调控是很重要的。
基因结构的新基因鉴定和优化
大多数序列在一个或两个祖先基因组中都有,而1776个新基因没有5:表S5);其中1250个可以通过引用不同的序列数据库(附加文件)进行功能性分配5:表S5)。根据COG分析,在几个类别中浓缩这些新基因的优势,例如一般函数预测,复制,重组和修复,转录等。
的二倍体A. Duranensis.和答:ipaensis基因(http://www.peanutbase.org.)[46]仅使用开放阅读框架(open reading frames, ORFs)进行注释,因此大多数5 ' -和3 ' -非翻译区域(utr)尚未定义。这里通过全局比较完整的文本和参考A. Duranensis.和答:ipaensis我们成功地延长了24,717个基因的utr,其中有9177个基因分别是5 ' -和3 ' - utr(附加文件6:表S6)。
与与脂肪酸代谢相关的基因有关
酰基-脂质代谢是一个非常重要的代谢过程,在所有植物中发挥着多种多样的功能。拟南芥酰基脂质代谢需要600多个基因参与至少120种酶反应。总共有2275个花生基因(附加文件7表S7)在前体基因组(不包括转录因子和ABC转运体)中鉴定出与脂肪酸代谢相关的脂类,可分为12类脂类,包括脂肪酸合成和输出、质体甘油脂合成、真核磷脂合成等。2275个基因中,FPKM≥0.1的基因有1724个。其中超过半数(1062人,占61.6%)受AS影响(附加文件)7:表S7)。高达比例表明了在调节脂肪酸代谢中的重要性。与脂肪酸合成相关的基因形成了最大的基团(422名成员,其中226人经历过)。3-酮酰基CoA合酶基因家族包含49个成员,但只有九(<20%)受到如此。相反,在酰基-Coa硫代酯酶基因家族中,频率为75%。
将聚合酶链反应(PCR)和排序应用于编码脂肪酸去饱和酶(FAD)(Aradu.5N10F,Araip.84LR8和Araip.92Q2X)的三个基因,以验证与序列数据中预测的存在(附加文件4:表S4)。PCR扩增产物的测序表明,这些AS转录本中存在新的外显子(图)。5).与Aradu.5N10F相比,Aradu.5N10F01获得了新的第5外显子;与Araip.84LR8相比,Araip.84LR801获得了新的第二外显子;与Araip.92Q2X相比,Araip.92Q2X01和Araip.92Q2X02均获得了新的第二外显子。Araip.92Q2X01和Araip.92Q2X02的差异是由于影响第7外显子的3 ' -AE事件,但这些AS事件不影响翻译;所有的异构体都能转化成完整的蛋白质。将这些编码蛋白序列在NCBI Nr数据库中进行blast分析,发现它们都属于FAD基因家族。
讨论
由于基因可以产生多个转录物的原始发现[47, AS现象在真核生物基因组中普遍存在[8,12.,13.,28.,29.,30.,36,38,49,50,51].高通量测序技术的发展允许进行的基因组扫描,导致估计至少42%的内含子的基因答:芥经验[31].根据张等人。[49],每个基因产生的转录物的平均数量为2.4。在植物中经验的基因比例变化大大变化了[26.].如此受影响的花生基因的比例约为37.2%,低于番茄,拟南芥和大豆的37.2%[25.,37,50].有以下几种原因:首先,我们使用的样品的数量较少,(种子1,种子2,根和叶),许多基因或特异性在茎,花和其他组织中表达的基因。许多报道表明,随着比例与样品数量有关,样品越多,比率越大[25.,32,50].其次,我们的样本来自不同的发育阶段,没有包括来自不同环境的样本,因此很多基因或AS基因在应激条件下特异表达没有被检测到。据报道,许多AS事件仅由非生物和生物胁迫引起[25.,39].另外,由于使用的预测算法,同一组织和生长条件中发现的AS比例是不同的[52,53,54].与拟南芥和其他植物相比,随着花生事件的研究得到了大幅滞后。在这里,我们确定了27,829个花生基因作为事件,作为基因的大约一半是组成型在四种样品中的替代,其他物质显示出显着的差异组织表达模式。
尽管许多AS事件是由组织特异性信号调控的,但AS似乎在种子发育中发挥着特殊的重要作用,因为本研究中在种子中检测到的AS事件和AS亚型比在根和叶中检测到的更多(表)1和2,附加文件4:表S4)。种子是一个非常重要的生殖器官,它混合了许多不同的组织。以前的研究也报告了类似的结果[25.,50].撒切尔等人。发现玉米种子比胚乳和胚胎多于同种型,并且只有在种子中发现的同种型30.].在番茄中,果实中每个基因产生的AS亚型比花和其他器官产生的多[50].Shen等人。发现更多随着年龄较小的发展阶段发生的事件多于同一类型组织的较旧的发育阶段[25.].
有趣的是,只有一半的花生,因为基因只经历了单一类型的事件(图。2),这意味着随着基因的更倾向于一种作为类型来调节其表达模式的更加花生。Potenza等。据报道,葡萄葡萄酒中的这种现象,约49.7%的葡萄葡萄酒,因为主要经历一次或两次的基因作为事件[51].有报道称基因结构对AS事件类型和AS频率有重要影响,如内含子长度、外显子数目、基因表达水平等[25.].随着内含子长度的增加,ES所占比例增加,IR所占比例降低;随着基因表达量的增加,IR的比例增加,ES的比例降低[25.].
TSS和TTS在花生不同器官中普遍存在。核糖体上的蛋白质合成是由信使RNA (mRNA)模板引导的,因此二级信使RNA的结构可能会影响翻译起始。目前积累的研究表明,由于TSS的使用,信使rna可以产生蛋白质异构体,特别是在人类和小鼠[55,56,57,58,59].在哺乳动物中,TSS转录本以组织特异性的方式和/或发育阶段特异性的方式调控[60.].N-术语数据显示,在较高的真核中,约占所有鉴定的蛋白质N Termini的20%,指向此类TSS [59].在植物中,对TSS的研究已经取得了一些进展。Kitagawa等人鉴定了水稻中许多推定的TSSs,并使用逆转录聚合酶链反应(RT-PCR)对其进行了验证,结果表明水稻的TSSs比小鼠的多样性更少,其中一些是按组织特异性方式调控的[61.].另一个例子是拟南芥的SYN1基因,它利用不同的启动子和剪接产生两个具有不同5 '端亚型[62.].成千上万的人和小鼠基因产生mRNA同种型,其3'UTR在包含许多涉及许多细胞过程中的许多监管要素[63.,64.].到目前为止,TTS如此丰富和保存的原因仍然是个问题。有研究报道TTS与RNA定位、转录稳定和蛋白质生产有关[65.]但最近的全基因组分析显示了显着的结果[66.].TTS还可以增加转录蛋白质的多样性。Fontana等。找到一种新的梵天调节机制(BRM),氧化应激通过Brahma-BRCA1-CSTF途径选择TTS的选择[67.].Potenza等研究了多个品种AS事件的位置,发现86% AS事件落在编码外显子中,其余发生在UTR或UTR- cds [51].Vitulo等发现,在葡萄中,18和11%的AS事件发生在5'UTR和3'UTR区域,约1%的AS事件发生在UTR-CDS区域[27.].在该研究中,鉴定了许多TSS和TTS剪接事件,并且它们以组织特异性方式表达(图。5).我们认为这个比率可能被高估了。主要原因是野生型花生基因组中的大部分基因都是预测的,其utr未被实验鉴定,因此utr是未知的。第二个原因是种植的花生是异源四倍体,它们的基因型环境将比它们的祖先更加复杂。据报道,全基因组复制在AS中起着重要的倍性依赖作用[28.].这些研究表明,转录物的所有区域易于没有任何显着偏好。
脂肪酸代谢是油籽植物的一个关键代谢过程,但迄今很少有人明确AS在这方面的贡献。Thambugala等人在亚麻中鉴定了6个去饱和酶基因,发现一些SAD和FAD亚型对脂肪酸组成、油脂含量和碘值有显著影响[68.].后来,罗维丹诺维奇等人。发现所有FAD2同种型都是活性的,两个FAD3A和三种FAD3B同种型不起作用,其中一些是由过早的止脚密码子的存在引起的[69.].在这里,我们发现花生基因组患者与脂肪酸合成/代谢相关的一些1062基因(FPKM≥0.1),其中预计将预计约为61.6%的经验;这种高比例表明,这可能对脂肪酸代谢的主要影响。三种时的实验验证表明,花生脂肪酸代谢相关基因中的同种型确实存在许多。但同种型的功能需要进一步研究。
结论
应用链特异性RNA-Seq技术在花生转录组中鉴定了27,829个AS基因。AS基因的发生率随外显子数的增加而增加。AS在种子中出现的频率较高,部分AS基因具有器官特异性,以内含子保留的形式占优势。详细分析了花生AS基因的GO注释和GO分类。我们克隆了部分基因,验证了序列数据中预测的AS基因的存在。这一鉴定将对花生研究领域产生重大影响。
方法
RNA分离、定量、鉴定
从花生品种丰花1号(Fenghua1)中获得4个RNA库:“FH1-seed1”取自开花后30天收获的种子(DAF),“FH1-seed2”取自开花后50天收获的种子,“FH1-root”和“FH1-leaf”取自12天幼苗。每个文库都是基于从至少三种植物中收集的组织,这些组织在RNA分离之前被快速冷冻在液氮中。使用TRIzol试剂(Invitrogen, Carlsbad, CA, USA)提取RNA,然后用RNase-free DNase I (New England Biolabs, USA)在37°C下处理30分钟,以降解存在的任何污染DNA。使用NanoDrop 2000分光光度计(Thermo Fisher Scientific, Wilmington, DE, USA)评估所得RNA制剂的浓度和纯度,使用RNA Nano 6000检测试剂盒(Agilent Technologies, CA, USA)检测其完整性。
cDNA图书馆建设和测序
用Ribo-of rRNA去除试剂盒(Epicenter,Madison,Wi,USA)加工1.5μg等分试样的RNA,以除去RRNA组分,以及随后基于残留的RNA的序列文库,用NEBNext®ultAT治疗后处理Illumina®(新英格兰Biolabs,USA)的定向RNA库预备套件。添加索引代码以使每个序列能够归因于其器官出处。配对端序列由Illumina Hiseq2500平台产生。
基因和转录本丰度的量化和AS事件的预测
去除低质量reads和适配器序列后,将序列映射到参考花生基因组(https://www.peanutbase.org/)[46]在TopHat2软件的协助下(http://ccb.jhu.edu/software/tophat/index.shtml).Cufflinks软件中的cuffdiff程序(http://cufflinks.cbcb.umd.edu/)用于基于FPKM来定量基因和转录物丰度[70].选择与在给定文库内大于0.1的FPKM相关的转录物作为表达指示剂。通过总结成分转录物的FPKMS来计算总体基因FPKMS。将组装的转录物使用袖扣包装中的袖扣模块映射到它们的相应基因模型。由于使用ASProfile软件识别事件(http://ccb.jhu.edu/software/ASprofile/).
功能注释
基因函数注释是使用软件,工具和数据库的组合进行的。Artoroman用于搜索蛋白质领域和功能站点,整合几个不同的数据库(Prosite,Prints,PFAM,Prodom和Smart)[71.].Nr (72.), KOG /齿轮[73.],瑞士人[74.],kegg [75.]和GO [76.数据库在花生基因的蛋白质水平上进行了大量的分析。
由编码FAD的基因产生的替代转录本的实验验证
来自FH1籽1文库的5μg等分试样的RNA在使用cDNA合成试剂盒(Invitrogen,Carlsbad,Ca,USA)中以20μl反应中转录。设计了两个引物对,一个靶向araip.92Q2x和其他Aradu.5N10F和Araip.84LR8。前成对(92Q-F / -R)的序列为5'-TGTGCGTGTTTCATCACCTCCTCTCTCTCTCTCTCTCTCTGTGCCTCAT,而后者对(5N10F-F / -R)的那些是5'-cattttctccacacacactaacttg和5'-tgatcatttagacttgtcgtcgaag。每种50μlrt-PCR含有100ng /μlcDNA,每个引物(10μm),5μl10×PCR缓冲液(Transgen Biotech,Peking,China),2.5μl2.5mM DNTP和1u反式Taqhifi DNA聚合酶(北京北京州生物科技)。将反应(94℃/ 4分钟)变性(94℃/ 4分钟),通过94℃/ 30 s,58℃/ 30 s,72℃(120秒)循环30次,最后在72℃下保持10分钟。使用Minelute TM凝胶萃取试剂盒(Qiagen,Hilden,德国)纯化PCR产物,并插入Peasy-T3载体(Transgen Biotech,Peking,China)进行测序。使用DNAMAN软件进行比较序列(http://www.lynnon.com/index.html),并且使用基因结构显示服务器在线绘制基因结构(http://gsds.cbi.pku.edu.cn/).
缩写
- AE:
-
替代外显子结束
- 作为:
-
可变剪接
- BRM:
-
梵天
- DAF:
-
几天后开花
- ES:
-
外显子跳跃
- FAD:
-
脂肪酸desaturase
- FPKM:
-
每千碱基外显子的片段,每百万片段的图谱
- 走:
-
基因本体论
- 红外光谱:
-
基因内区保留
- KEGG:
-
京都基因和基因组百科全书
- Kog / Cog:
-
同源组的蛋白质簇
- mirnas:
-
microRNA.
- 信使rna:
-
信使RNA
- MSKIP:
-
多个外显子跳跃
- NR:
-
非冗余蛋白质序列
- 子:
-
开放阅读框
- 聚合酶链反应:
-
聚合酶链反应
- RT-PCR:
-
逆转录 - 聚合酶链反应
- TSS:
-
转录起始点
- TTS:
-
转录终端站点
- UTRS:
-
5 ' -和3 ' -非翻译区域
参考文献
- 1。
Reddy As,Marquez Y,Kalyna M,Barta A.植物中替代拼接景观的复杂性。植物细胞。2013; 25(10):3657-83。
- 2。
STAMM S,Ben-Ari S,Rafalska I,Tang Y,Zhang Z,Toiber D,Thanaraj Ta,Soreq H.替代拼接的功能。基因。2005; 344:1-20。
- 3.
Scotti MM,Swanson MS。RNA在疾病中错误拼接。NAT Rev Genet。2016; 17(1):19-32。
- 4.
Le KQ,Prabhakar BS,Hong WJ,Li LC。作为生物标志物的替代剪接和药物发现的潜在目标。acta pharmacol sin。2015; 36(10):1212-8。
- 5.
kornblhtt AR, Schor IE, Allo M, Dujardin G, Petrillo E, Munoz MJ。选择性剪接:真核生物转录和翻译之间的关键步骤。细胞生物学杂志。2013;14(3):153-65。
- 6。
李志刚,李志刚。RNA剪接在人类疾病中的应用。中国细胞生物学杂志2016;212(1):13-27。
- 7。
选择性剪接:多种控制机制与人类疾病的关系。趋势麝猫。2002;18(4):186 - 93。
- 8。
Rodriguez SA,Grochova D,McKenna T,Borate B,Trivedini,Erdos Mr,Eriksson M.全局基因组剪接分析显示出随老化的替代性转化基因的数量增加。老化细胞。2016; 15(2):267-78。
- 9。
杨某,唐F,朱H.植物免疫替代剪接。int j mol sci。2014; 15(6):10424-45。
- 10。
Remy E,TR C,Batista Ra,Hussein Ma,Teixeira MC,Athanasiadis A,Sa-Correia I,Duque P. Intron Intron保留在新型ZIF2转运蛋白的5'UTR中,增强了促进拟南芥锌耐受的翻译。Plos Genet。2014; 10(5):E1004375。
- 11.
布兰考BJ。另类拼接:来自全球分析的新见解。细胞。2006;126(1):37-47。
- 12.
张强,张旭,王胜,谭超,周刚,李超。大麦种子萌发过程中选择性剪接的参与。《公共科学图书馆•综合》。2016;11 (3):e0152824。
- 13。
唐伟,郑勇,董建军,于建军,刘飞,郭旭,黄胜,Wisniewski M,孙建军,等。综合转录组分析揭示了猕猴桃(猕猴桃)果实发育和成熟过程中的长链非编码RNA表达和可变剪接调控。植物学报。2016;7:335
- 14.
XING Y,LEE C.替代剪接和RNA选择真核基因组的压力进化后果。NAT Rev Genet。2006; 7:499-509。
- 15.
Keren H,Levmaor G,AST G.替代拼接和进化:多样化,外显子定义和功能。NAT Rev Genet。2010; 11(5):1031-34。
- 16.
陈志强,陈志强,陈志强,等。可变剪接与进化。Bioessays。2003;25(11):1031 - 4。
- 17。
在16个人体组织中,数千个外显子跳过事件区分了不同的剪接模式。F1000Research。2013; 2:188。
- 18。
Barbosa-Morais NL,Irimia M,Pan Q,Xiong Hy,Guorysov S,Lee LJ,Slobodeniuc V,Kutter C,Watt S,Colak R等。脊椎动物物种替代拼接的进化景观。科学。2012; 338(6114):1587-93。
- 19。
Merkin J, Russell C, Chen P, Burge CB。哺乳动物组织中基因和亚型调控的进化动力学。科学。2012;338(6114):1593 - 9。
- 20。
Sultan M, Schulz MH, Richard H, Magen A, Klingenhoff A, Scherf M, Seifert M, Borodina T, Soldatov A, Parkhomchuk D,等。通过人类转录组的深度测序对基因活性和选择性剪接的全局观点。科学。2008;321(5891):956 - 60。
- 21。
潘琪,夏欧,李LJ, Frey BJ, Blencowe BJ。通过高通量测序对人类转录组中可变剪接复杂性的深入调查。Nat麝猫。2008;40(12):1413 - 5。
- 22。
Wang ET, Sandberg R, Luo S, Khrebtukova I, Zhang L, Mayr C, Kingsmore SF, Schroth GP, Burge CB。人类组织转录组中的替代亚型调控。大自然。2008;456(7221):470 - 6。
- 23。
Graveley B, Brooks AN, Carlson JW, Duff MO, Landolin JM, Yang L, Artieri CG, Baren MJ, Boley N, Booth BW,等。黑腹果蝇的发育转录组。大自然。2011;471(7339):473 - 9。
- 24。
徐Q,Modrek B,Lee C.全基因组检测人体转录组中的组织特异性替代剪接。核酸RES。2002; 30(17):3754-66。
- 25。
沉Y,周Z,王Z,李W,方c,吴M,Ma Y,Liu T,Kong La,Peng DL等。古代聚倍性大豆替代拼接的全局解剖。植物细胞。2014; 26(3):996-1008。
- 26.
黄杰,高益,贾H,刘L,张D,张Z。比较转录组织揭示了玉米改善的替代剪接变化和选择签名。BMC基因组学。2015; 16:363。
- 27.
Vitulo N,Forcato C,Carpinelli Ec,Telatin A,Campagna D,D'Angelo M,Zimbello R,Corso M,Vannozzi A,Bonghi C等人。对葡萄中的替代拼接的深度调查显示了与组织,应力条件和基因型相关的拼接机械的变化。BMC植物BIOL。2014; 14:99。
- 28.
Saminathan T, Nimmakayala P, Manohar S, Malkaram S, Almeida A, Cantrell R, Tomason Y, Abburi L, Rahman MA, Vajja VG,等。二倍体和四倍体西瓜基因表达差异与选择性剪接。J Exp Bot. 2015;66(5): 1369-85。
- 29。
Du Q,Li C,Li D,Lu S.全基因组分析,分子克隆和表达分析揭示了组织 - 特异性表达,反馈调节,应激响应性,综合剪接的新药基因,涉及丹贝林代谢在Salvia Miltiorrhiza。BMC基因组学。2015; 16:1087。
- 30.
王志刚,王志刚,李斌。干旱胁迫和玉米发育过程中选择性剪接的全基因组分析[J]。植物杂志。2016;170(1):586 - 99。
- 31。
Filichkin SA, Priest HD, Givan SA, Shen R, Bryant DW, Fox SE, Wong WK, Mockler TC。拟南芥可变剪接的全基因组定位。基因组研究》2010;20(1):45-58。
- 32。
撒切尔SR,周W,伦纳德A,王B-B,Beatty M,Zastrow-Hayes G,Zastrow-Hayes G,Zastrow-Baumgarten A,Li B. Zea中替代剪接的基因组分析:景观和遗传调节。植物细胞。2014; 26:3472-87。
- 33。
李琦,萧克,朱yx。Gossypium Raimondii转录组的单核苷酸分辨率映射显示了内含子的替代剪接的新机制。莫尔植物。2014; 7(5):829-40。
- 34。
Calarco Ja,Xing Y,Caceres M,Calarco JP,Xiao X,Pan Q,Lee C,Preuss TM,Blencowe BJ。全球分析人与黑猩猩之间的替代拼接差异。基因开发。2007; 21(22):2963-75。
- 35。
王志刚,王志刚,王志刚,等。水稻团藻选择性剪接的全基因组分析。BMC基因组学。2014;15:1117。
- 36。
曼迪克,学者KB。基因组分析植物病毒相互作用替代拼接景观景观。植物细胞。2015; 27(1):71-85。
- 37。
Marquez Y,Brown JW,Simpson C,Barta A,Kalyna M.转录组调查显示拟南芥中替代剪接景观的复杂性增加。Genome Res。2012; 22(6):1184-95。
- 38.
Min XJ,Powell B,Braessler J,Meinken J,Yu F,Sablok G.基因组宽的编目和分析谷物作物中的可拼接基因。BMC基因组学。2015; 16:721。
- 39.
Staiger D,棕色JW。生物定时,发展和应力响应交叉处的替代拼接。植物细胞。2013; 25(10):3640-56。
- 40.
Remy E,Cabrito TR,Baster P,Batista Ra,Teixeira MC,Friml J,Sa-Correia I,Duque P.一个主要的促进剂超家族运输司满在极地养蛋白运输和拟南芥中的干旱胁迫耐受性中起着双重作用。植物细胞。2013; 25(3):901-26。
- 41.
Lewis BP,Green Re,Brenner SE。人类替代剪接和废话介导的mRNA腐蚀的迹象。Proc Natl Acad Sci U S A. 2003; 100(1):189-92。
- 42.
奥斯F,Gehring NH。废话介导的mRNA腐烂的生理和病理生理学作用。Pflugers arch - eur j physiol。2016年; 468(6):1013-28。
- 43。
王BB,Brendel V. Genomewide植物替代剪接的比较分析。Proc Natl Acad Sci U S A. 2006; 103(18):7175-80。
- 44。
Drechsel G, Kahles A, Kesarwani AK, Stauffer E, Behr J, Drewe P, Ratsch G, Wachter A. nonsense介导的替代前体mRNA剪接变异的衰减是拟南芥稳态转录组的一个主要决定因素。植物细胞。2013;25(10):3726 - 42。
- 45。
Kalyna M,Simpson CG,Syed NH,Lewandowska D,Marquadz Y,Kusenda B,Marshall J,Fuller J,Cardle L,McNicol J等人。替代拼接和废话介导的衰减调节拟南芥中重要调控基因的表达。核酸RES。2012; 40(6):2454-69。
- 46。
Bertioli DJ,Cannon SB,Froenicke L,Huang G,Farmer Ad,Cannon Ek,Liu X,Gao D,Clevenger J,Dash S等人。栽培花生的二倍体祖先的Arachis Duranensis和Arachis iPaensis的基因组序列。NAT Genet。2016; 48(4):438-46。
- 47。
选择性剪接与基因复制是一种负相关的进化机制。37 Nat麝猫。2005;(6):588 - 9。
- 48。
Modrek B,Lee C:替代拼接的基因组视图。自然遗传学2002,30(1):13-9。
- 49。
张R,Calixto Cpg,Marquez Y,Venhuizen P,Tzioutziou Na,Guo W,Spensley M,Entizne JC,Lewandowska D,ST H等。一种高质量的拟南芥转录组,用于准确转录级别分析的替代剪接。核酸RES。2017; 45(9):5061-73。
- 50。
关键词:番茄,剪接,RNA测序,选择性基因组学。2015;16(1):948。
- 51。
Potenza E, Racchi ML, Sterck L, Coller E, Asquini E, Tosatto SCE, Velasco R, Peer YV, cesaro A.十个不同葡萄品种的选择性剪接事件探讨。BMC基因组学。2015;16 (1):1 - 9
- 52.
Trapnell C, Pachter L, Salzberg SL. TopHat:用RNA-Seq发现剪接连接。生物信息学。2009;25:1105-11。
- 53.
Foissac S,Sammeth M. Astalavista:自定义基因数据集中的替代拼接事件的动态和灵活分析。核酸RES。2007; 35(Web服务器问题):W297-9。
- 54.
Trapnell C,Roberts A,Goff L,Pertea G,Kim D,Kelley Dr,Pimentel H,Salzberg SL,RINN JL,Pachter L.差分基因和RNA-SEQ实验与Tophat和袖扣的实验分析。NAT PROTOC。2012; 7(3):562-78。
- 55。
Gandre S, Bercovich Z, Kahana C.抗酶的线粒体定位是通过上下文依赖的两个AUG起始密码子的替代利用来确定的。线粒体。2003;2:245-56。
- 56。
Leissring MA, Farris W, Wu X, Christodoulou DC, Haigis MC, Guarente L, Selkoe DJ。替代翻译起始产生一种新的针对线粒体的胰岛素降解酶亚型。j . 2004; 383:439-46。
- 57。
人神经肽Y基因线粒体靶向信号的研究。生物化学学报。2005;33:633 - 40。
- 58。
Kochetov AV.真核mrna的可选翻译起始位点和隐藏编码潜力。Bioessays。2008;30:683 - 91。
- 59。
Damme PV,Gawron D,Criekinge Wv,Menschaert G. n-末端蛋白质组学和核糖体剖面提供了小鼠和男性替代翻译启动景观的综合观点。Mol细胞蛋白质组学。2014; 13:1245-61。
- 60。
杰克逊rj。哺乳动物MRNA翻译的替代机制。Biochem Soc Trans。2005; 33:1231-41。
- 61。
Kitagawa N, Washio T, Kosugi S, Yamashita T, Higashi K, Yanagawa H, Higo K, Satoh K, Ohtomo Y, Sunako T,等。计算分析表明,不同的第一外显子参与了水稻(Oryza sativa)组织特异性转录。生物信息学。2005;21(9):1758 - 63。
- 62。
白X,Peirson BN,Dong F,薛C,Makaroff Ca.Syn1的分离和表征,拟南芥中的Meiosis是必不可少的Rad21的基因。植物细胞。1999; 11:417-30。
- 63。
Taliaferro JM,Vidaki M,Oliveira R,Olson S,Zhan L,Saxena T,Wang et,Graveley Br,Gertler FB,Swanson Ms,等。远端替代的最后外显子将MRNA定位为神经预测。Mol细胞。2016; 61(6):821-33。
- 64。
Miura P, Shenker S, Andreu-Agullo C, westtholm JO, Lai EC。哺乳动物大脑中3' utr广泛和广泛的延长。基因组研究》2013;23(5):812 - 25。
- 65.
定位或不定位:mRNA命运在3'UTR末端。《细胞生物学趋势》2009;19:465-74。
- 66.
间谍N, Burge CB, Bartel DP。3’utr亚型的选择对小鼠成纤维细胞中大多数mrna的稳定性和翻译效率影响有限。基因组研究》2013;23:2078 - 90。
- 67。
Fontana Ga,Rigamonti A,Lenzken SC,Filosa G,Alvarez R,Calogero R,Bianchi Me,Barabino Sml。氧化应激通过Brahma-BRCA1-CSTF途径控制替代最后一个外显子的选择。核酸res斜体。2017; 45(2):902-14。
- 68。
Thambugala D, Duguid S, Loewen E, Rowland G, Booker H, You FM, Cloutier S.亚麻中6个去饱和酶基因的遗传变异及其对脂肪酸组成的影响。acta photonica sinica . 2013;126(10): 2627-41。
- 69。
在酵母中表达的亚麻脂肪酸去饱和酶FAD2和FAD3亚型的功能特征显示了广泛的活性多样性。生物科技摩尔》。2014;56(7):609 - 20。
- 70.
Trapnell C, Williams BA, Pertea G, Mortazavi A, Kwan G, Baren MJ, Salzberg SL, Wold BJ, Pachter L.通过RNA-Seq转录本组装和定量揭示了细胞分化过程中未注释的转录本和亚型转换。生物科技Nat》。2010;28:511-5。
- 71。
ZDOBNOV EM,APWEILER R.InterCan-ARTRAGES-识别方法中的一个集成平台。Bioinforma Oxf Engl。2001; 17(9):847-8。
- 72。
邓媛媛,李建强,吴顺峰,朱艳萍,陈永文,何福成。蛋白质注释系统中NR数据库的集成及其定位。计算机工程学报。2006;32(5):71-4。
- 73。
Tatusov RL, Galperin MY, Natale DA。COG数据库:用于蛋白质功能和进化的基因组规模分析的工具。核酸学报2000;28(1):33-6。
- 74。
Apweiler R,Bairoch A,Wu Ch,Barker Wc,Boeckmann B,Ferro S,Gasteiger E,Huang H,Lopez R,Magrane M等。UNIPROT:通用蛋白质知识库。核酸res斜体。2004; 32(数据库问题):D115-9。
- 75。
Kanehisa M,Goto S,Kawashima S,Okuno Y,Hattori M.用于解密基因组的Kegg资源。核酸res斜体。2004; 32(数据库问题):D277-80。
- 76。
Ashburner M, Ball CA, Blake JA, Botstein D, Butler H, Cherry JM, Davis AP, Dolinski K, Dwight SS, Eppig JT,等。基因本体论:生物学统一的工具。中国科学:地球科学,2000;25(1):25 - 9。
确认
不适用。
资金
中国天然科学基金(2015-2017),中国国际科技合作计划(2015DFA31190),中国国家科学与技术支持计划(2014Bad11b04)和现代农业工业技术研究系统(Cars-14)的专用基金。创始人在研究设计,数据收集和分析中没有作用,决定发布或准备手稿。
可用性数据和材料
cDNA文库和其他分析数据集的测序数据可在NCBI BioProject数据库中获得,登录号为PRJNA354652。所有支持数据都包含在附加文件中。
作者信息
隶属关系
贡献
JR和FG获取的数据,YW和XL分析和解释的数据,LS进行了PCR放大和序列,LS,SW和ZP参与了手稿的设计和起草,修订了稿件并获得了最终批准的发布版本。所有作者阅读并认可的终稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
阮军,郭飞,王永强。等等。花生选择性剪接的转录组分析(arachis hypogaea.L.)。BMC植物杂志18,139(2018)。https://doi.org/10.1186/s12870-018-1339-9
已收到:
接受:
发表:
关键词
- arachis hypogaea.l
- 可变剪接
- 转录组分析
- 器官特异性表达
- 脂肪酸新陈代谢