- 研究文章gydF4y2Ba
- 开放访问gydF4y2Ba
- 发布:gydF4y2Ba
在结合MADS结构域蛋白的图案的比较分析gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba18.gydF4y2Ba, 文章编号:gydF4y2Ba131.gydF4y2Ba(gydF4y2Ba2018年gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
正确的花形成需要高度特异的基因表达时间和空间调控。在gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba决定花器官身份的大多数主监管机构属于疯子域转录因子家庭。该转录因子家族的规范DNA结合基序是碳盒,具有共识CC(A / T)gydF4y2Ba6gydF4y2Ba然而,到目前为止,尚未对mads结构域结合模式进行全面的分析。gydF4y2Ba
结果gydF4y2Ba
我们分析了8个公开的mads结构域蛋白的ChIP-seq数据集,这些蛋白调控花的转变和形成。令人惊讶的是,每个蛋白质的首选DNA结合基序是一个带有NAA延伸的CArG-box。此外,在mads结构域转录因子结合位点附近发现了其他转录因子的基序,表明mads结构域蛋白与其他转录因子的相互作用对靶基因的调控具有重要意义。最后,车厢之间的保护gydF4y2Ba拟南芥gydF4y2Ba对生态型进行评估以获得关于它们进化重要性的信息。完全符合共识的CArG-box比其他CArG-box更保守,这表明完美的CArG-box在进化中比其他CArG-box变体更重要。gydF4y2Ba
结论gydF4y2Ba
我们的分析提供了对mads结构域蛋白结合模式的详细了解。这一结果强调了CArG-box扩展版本的重要性,并为mads结构域蛋白结合位点的进化保护提供了第一个观点gydF4y2Ba拟南芥gydF4y2Ba生态型。gydF4y2Ba
背景gydF4y2Ba
正确的开花时间和空间规划对植物繁殖至关重要。这个程序的主要部分是由一类被称为mads结构域转录因子的转录因子调控的。该转录因子家族的成员在发育的不同方面发挥关键作用,并在植物、真菌和动物界的许多其他生物中具有同源物[gydF4y2Ba1gydF4y2Ba].gydF4y2Ba
在gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba,疯子域蛋白可以分为基于保守域的两个主要曲线。I型MADS-域蛋白只有他们的DNA结合MADS域名。相比之下,表征型II型疯子域蛋白质的更好有四个共同点;疯子域参与DNA结合,中间结构域具有二聚化的作用,异蛋白样结构域,也称为k盒,在二聚化以及其他蛋白质 - 蛋白质相互作用中具有作用,以及C-终端域具有不同的功能,例如稳定蛋白质复合物和激活转录[gydF4y2Ba2gydF4y2Ba].gydF4y2Ba
在过去的几十年MADS结构域蛋白中的作用gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba花卉开发已被广泛研究。这导致了所谓的ABC(d)e型号的开发。根据这种模型,一朵花可以被视为四轮螺纹的集合。从外面到内部,这些螺纹分别由萼片,花瓣,雄蕊和卡皮组成。这些螺纹的同一性是通过表达特定基因的表达来确定,其可分为四种不同的类(A,B,C和E)。通过表达来确定萼片同一性gydF4y2Ba拟南芥gydF4y2Ba阶级基因gydF4y2BaApetala1.gydF4y2Ba(gydF4y2BaAP1.gydF4y2Ba),gydF4y2BaApetala2.gydF4y2Ba(gydF4y2BaAP2.gydF4y2Ba).花瓣发育需要这些A类基因和B类基因的联合表达,包括gydF4y2BaApetala3.gydF4y2Ba(gydF4y2BaAP3.gydF4y2Ba),gydF4y2BaPISTILLATAgydF4y2Ba(gydF4y2BaPI.gydF4y2Ba).雄蕊发育需要这些B类基因和C类基因的联合表达gydF4y2Ba无性生殖的gydF4y2Ba(gydF4y2BaAG)gydF4y2Ba).最后,心皮的同一性由C类基因的表达决定gydF4y2BaAG)gydF4y2Ba一个人。D类基因的活性决定了胚珠的特性。此外,E类基因的表达,包括gydF4y2BaSEPALLATA1 2 3gydF4y2Ba和gydF4y2Ba4gydF4y2Ba(gydF4y2BaSEP1-4gydF4y2Ba),在每一个螺纹中都需要适当的花朵发展。除了gydF4y2BaAP2.gydF4y2Ba,所有花器官类的基因都编码疯狂域蛋白质。gydF4y2Ba
植物MODS-域蛋白质将DNA与同性恋或异二聚体结合[gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba].另外,它们可以形成可以在多个位点结合DNA的高阶络合物,导致位点之间的DNA环[gydF4y2Ba5gydF4y2Ba,gydF4y2Ba6gydF4y2Ba,gydF4y2Ba7gydF4y2Ba].我们认为决定花特性的mads结构域蛋白形成了四聚体复合物,也被称为“花四聚体”,它由相应的A、B和/或C类蛋白以及SEP蛋白组成。这种SEP蛋白可以被看作是一种将四聚体结合在一起的分子胶[gydF4y2Ba8gydF4y2Ba].gydF4y2Ba
除了调节花器官的身份,MADS域转录因子也有开花,从营养到生殖生长过渡的调节作用。例如,CONSTANS1的过度表达(SOC1)抑制物已被确定为在一个基因调控网络的主要枢纽调节开花的时间[gydF4y2Ba9gydF4y2Ba].其他开花调控因子的例子还有mads结构域蛋白开花位点C (FLC)和短营养期(SVP),它们通过结合两个蛋白的复合物或单独结合到不同的启动子序列来抑制开花[gydF4y2Ba10.gydF4y2Ba].gydF4y2Ba
每个mads结构域TF,无论是作为二聚体还是更高阶复合物,都应该调控特定的靶基因集合,以控制它们参与的不同发育过程。这需要这些蛋白质对某些DNA序列的特异性和亲和力。mads结构域蛋白与一个叫做CArG-box的DNA基序结合,其一致序列为CC(a /T)。gydF4y2Ba6gydF4y2BaGG [gydF4y2Ba11.gydF4y2Ba].除了这一共识,这将被称为完美的CArG-box,变体CC(A/T)gydF4y2Ba7gydF4y2Bag和c(a / t)gydF4y2Ba8gydF4y2BaG也被认为对一些mads结构域蛋白的结合很重要[gydF4y2Ba11.gydF4y2Ba,gydF4y2Ba12.gydF4y2Ba].gydF4y2Ba
一种研究转录因子与体内特定DNA序列结合的流行技术是染色质免疫沉淀,然后深序(芯片-SEQ)。芯片SEQ已被用于研究与花发育有关的不同蛋白质的DNA结合,其中是AG [gydF4y2Ba13.gydF4y2Ba], AP1 [gydF4y2Ba14.gydF4y2Ba,gydF4y2Ba15.gydF4y2Ba],ap3 [gydF4y2Ba16.gydF4y2Ba],FLC [gydF4y2Ba10.gydF4y2Ba],π[gydF4y2Ba16.gydF4y2Ba],SEP3 [gydF4y2Ba15.gydF4y2Ba,gydF4y2Ba17.gydF4y2Ba],SoC1 [gydF4y2Ba9gydF4y2Ba]和SVP [gydF4y2Ba10.gydF4y2Ba].在每一项研究中,对蛋白质结合的DNA序列进行分析,发现了一个与经典CArG-box相似的基序。当比较不同研究中发现的CArG-box样motif时,可以观察到不同蛋白质结合的motif之间的差异。例如,west等人发现AP3/PI二聚体主要与一个motif结合,该motif类似于典型的3 '侧有三个腺嘌呤的CArG-box [gydF4y2Ba16.gydF4y2Ba],而Pajoro等人发现SEP3与一个类似的motif结合,在motif的第一个胞嘧啶之前的−3和−2位置有两个额外的胸腺嘧啶[gydF4y2Ba15.gydF4y2Ba].基于这些观察,我们很容易推测,观察到的结合基序变化解释了不同mads结构域转录因子的结合特异性。然而,这些研究中用于分析ChIP-seq数据集的方法并不相同,因此无法得出可靠的结论。gydF4y2Ba
在本研究中,AG的芯片起数据集[gydF4y2Ba13.gydF4y2Ba], AP1 [gydF4y2Ba15.gydF4y2Ba],ap3 [gydF4y2Ba16.gydF4y2Ba],FLC [gydF4y2Ba10.gydF4y2Ba],π[gydF4y2Ba16.gydF4y2Ba],SoC1 [gydF4y2Ba9gydF4y2Ba],SVP [gydF4y2Ba10.gydF4y2Ba]及SEP3 [gydF4y2Ba15.gydF4y2Ba以统一的方式重新分析。分析表明,蛋白质的结合基序高度相似,但不完全相同。此外,研究人员还确定了不同特征的基序对蛋白质结合的相对重要性,结果表明,对于所有蛋白质来说,带有两到三个附加腺嘌呤的完美CArG-box可能是最重要的基序。此外,发现了非carg -box基序,并发现了de novo基序,表明一些mads结构域蛋白通过与其他类型转录因子的相互作用间接调控其靶标。最后,通过分析不同生境中CArG-boxes的保存情况,评价了CArG-boxes的进化重要性gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba生态型。gydF4y2Ba
方法gydF4y2Ba
ChIP-seq数据处理gydF4y2Ba
从NCBI基因表达综合数据库(gydF4y2Bahttps://www.ncbi.nlm.nih.gov/geo/gydF4y2Ba).GSE45938 (AGAMOUS)、GSE46986 (APETALA1和SEPALLATA3)、GSE38358 (APETALA3和PISTILLATA)、GSE54881 (FLOWERING LOCUS C and SHORT VEGETATIVE PHASE)和GSE45846 (SUPPRESSOR OF OVEREXPRESSION OF CONSTANS1)。对于AP1和SEP3,选择诱导后4天的数据集进行分析。读取被映射到未掩蔽的gydF4y2Ba拟南芥蒂利亚纳gydF4y2BaTAIR10基因组(TAIR10_chr_all.fas;gydF4y2Baftp.arabidopsis.orggydF4y2Ba)使用SOAPaligner 2.21版本(gydF4y2Bahttp://soap.genomics.org.cn/soapaligner.html.gydF4y2Ba)的默认设置,除了设置-r 0。这种设置的选择是为了让重复的命中,不能可靠地分配到基因组的特定部分,被忽略的分析。gydF4y2Ba
使用R软件包CSAR对每个生物复制(如果可用)进行峰值呼叫[gydF4y2Ba18.gydF4y2Ba,gydF4y2Ba19.gydF4y2Ba].如果生物学重复组成的几个技术复制,技术重复进行了合并。默认设置使用,除了在mappedReads2Nhits功能,其中uniquePosition被设置为TRUE。选择该设置,以确保仅读取并不完全重合与其他读取(暗示它们实际上是相同的读取通过PCR扩增)用于分析。在ChIPseqScore功能使用泊松分布富集的得分。在sigWin起作用的阈值gydF4y2BatgydF4y2Ba= 1.3(对应于agydF4y2BapgydF4y2Ba-值约为0.05),并采用FDR阈值0.001来选择显著峰。峰数最多的生物复制被用于进一步分析。gydF4y2Ba
为了进一步分析,使用峰值中心(定义为围绕峰峰峰值的500bp)。写入自定义Python脚本以查看这些峰中心是否在数据集之间显示任何重叠。gydF4y2Ba
De novo motif discoverygydF4y2Ba
使用MEME-ChIP进行De novo motif发现[gydF4y2Ba20.gydF4y2Ba].这个程序由几个子程序组成,每个子程序执行一个特定的分析。本研究使用了模因、FIMO和CentriMO。MEME [gydF4y2Ba21.gydF4y2Ba与那些序列中的核苷酸频率的背景模型相比,在一组序列中寻找超人所谓的图案。在本研究中,对应于每个数据集的峰中心的500bp序列作为输入。节目中央100bp用于寻找超人效果的基序并确定核苷酸频率。找到主题后,FIMO [gydF4y2Ba22.gydF4y2Ba,用于查找所提供的500bp序列中每个motif的所有出现情况。最后,CentriMO [gydF4y2Ba23.gydF4y2Ba用来测试发现的图案是否集中富集。在目前的研究中,研究主题是否像一个CArG-box,或者是否在模因芯片定义的前三名。使用WebLogo 2.8.2可视化Motif序列logo [gydF4y2Ba24.gydF4y2Ba].gydF4y2Ba
在用作MEME芯片的输入的序列中,使用reffmasker掩盖散射的重复和低复杂性DNA(gydF4y2Bawww.repeatmasker.orggydF4y2Ba)扣除发动机,默认速度和gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba作为DNA源。使用MEME的默认设置,使用以下例外:由于载体箱通常彼此靠近,因此使用设置--MEME-MOD ANR,该设置为零,一个或多个每个序列的主题出现。此外, - MEME-NMOTIFS,MEME MEME尝试发现的数量设置为10,以确保将找到所有可能的重要主题。-meme-MaxSites,MEME考虑的主题的最大发生量被设置为5000,以确保找到给定主题的所有出现。-nmeme,即MEME分析序列的量,设定为10,000,000和-meme-MAXSIZE,总数据集大小MEME分析,被设定为1,000,000,000,000,使得整个数据集将被分析。gydF4y2Ba
在另一种方法中,编写了一个定制的Python脚本,用于分析每个可能的10个字符长的字符串的出现情况,其中每个位置要么定义为四个核苷酸(“a”、“C”、“G”或“T”)中的一个,要么定义为四个核苷酸中的任何一个(“N”)。在与MEME-ChIP相同的DNA序列中搜索这些字符串的匹配。分析这些正则表达式匹配到最近山峰的平均距离,并将正则表达式从最低到最高的平均距离进行排序。只有到峰顶的平均距离小于或等于90 bp且匹配量大于或等于80的正则表达式被选择进行进一步分析。我们对这个数据集进行了手工分析,以发现与模因芯片分析中发现的CArG-box或次级模因不同的模因。gydF4y2Ba
Motif频率和中心富集分析gydF4y2Ba
为了比较相对频率,计算包含某个基序的峰中心的脉冲频率,并除以背景频率。使用了两个不同的背景定义:(i)从整个整个100,000个随机选择的500bp的100,000次计算后台频率gydF4y2Ba拟南芥gydF4y2BaTair10基因组;或(ii)背景频率从100,000个随机选择的500 bp的延伸计算gydF4y2Ba拟南芥gydF4y2Ba启动子区域。这些区域是通过连接每个转录起始位点上游500bp而形成的,因为这些区域是功能基序最丰富的[gydF4y2Ba25.gydF4y2Ba以及分析的大部分峰值中心与这些区域重叠(附加文件gydF4y2Ba1gydF4y2Ba:表S1)。此外,它计算在每个数据集的许多峰如何包含在所述基序CC(A / T)gydF4y2Ba6gydF4y2BaGG, CC (A / T)gydF4y2Ba7gydF4y2BaG、C (A / T)gydF4y2Ba8gydF4y2BaG一次或多次,以及有多少个峰包含这三个主题中的两个的组合。对于最后一个数字,通过将包含每个motif的峰值频率相乘,并将该数字与每个数据集中的峰值总数相乘,也可以计算出一个期望值。gydF4y2Ba
为了分析中央富集,计算每个匹配与峰值和该峰值的峰值峰值的匹配之间的平均距离。为了可视化中央富集,使用R函数'密度'获得包含含有Al匹配的数据集的核密度估计。gydF4y2Ba
CArG-box样图案的可拓分析gydF4y2Ba
编写了两个不同的定制Python脚本来分析carg -box周围的核苷酸。第一个脚本查看由MEME-ChIP定义的CArG-box两侧三个核苷酸不同组合的频率。首先,给定MEME-ChIP提供的链,通过比较类似CArG-box的序列和规范的CArG-box定义(CC(a /T))来定义CArG-box的位置1和10。gydF4y2Ba6gydF4y2BaGG)。位置1被定义为对应于典型碳盒箱的第一C的核苷酸,并且定位10被定义为对应于对应于典型G盒的最后G的核苷酸。在位置1的5'侧发生三个核苷酸的不同组合(从附近5'延伸部)和位置10的3'侧(从附近3'延伸)和5'和3'延伸的组合。对于组合,还通过将5'和3'延伸的发生和将其划分为克箱的总量来计算预期值。请注意,这里有5'和3'在此处定义为由MEME芯片定义的碳盒方向定向,这导致以尽可能多的腺嘌呤对齐。gydF4y2Ba
编写了第二个自定义Python脚本,用于分析围绕序列NCC(A/T)的具体扩展。gydF4y2Ba6gydF4y2BaGGN(因此,与第一次分析相比,使用更严格的CArG-box定义作为开始)。该脚本确定是否有回文定义的主题(如NCC(a /T))gydF4y2Ba6gydF4y2BaGGN)在3 '端有三个核苷酸(从今以后称为三聚体)的特定组合,这种组合出现的频率比预期的更频繁。该脚本在一组序列中查找回文基序的所有出现,并统计三聚体-3 '的5 ' -母序-三聚体-3 '和5 ' -反补(在反补中查找也是如此)。然后,它将计数除以两倍基序出现的总数,给出一个观察到的三聚体频率。然后,通过在给定序列中乘以三聚核苷酸的频率来计算期望的三聚核苷酸频率。然后用公式计算相对三聚体频率gydF4y2Ba
接下来,输入序列,除了母题本身,被洗牌和一个新的延伸分析完成。这样做了1,000,000次,计算洗牌序列中某三聚体的相对频率高于实际序列中某三聚体的相对频率的次数,得到agydF4y2BapgydF4y2Ba价值。gydF4y2Ba
主题保护分析gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba生态型gydF4y2Ba
含有SNP的位置和1-3个碱基缺失的数据集gydF4y2BaArabdidopsisgydF4y2Ba从1001基因组计划网站(gydF4y2Ba1001 genomes.orggydF4y2Ba)[gydF4y2Ba26.gydF4y2Ba,gydF4y2Ba27.gydF4y2Ba].使用的数据集为“MPICao2010”、“Salk”和“MPICWang2013”。为了保证分析的质量,只使用snp和质量评分大于等于25的小缺失进行分析。共分析了包括Col-0在内的595个生态型。gydF4y2Ba
对于保守性分析,匹配AG的CArG-box motif(图。gydF4y2Ba1AgydF4y2Ba)使用FIMO在所有数据集中搜索[gydF4y2Ba22.gydF4y2Ba,因为这个主题代表了所有数据集。对每个基序出现的每个位置,计算不同生态型中该位置的每个核苷酸的相对频率。根据这些频率,用公式H =−∑计算每个位置的香农熵(H)gydF4y2BapgydF4y2Ba我gydF4y2Ba日志gydF4y2Ba2gydF4y2BapgydF4y2Ba我gydF4y2Ba, 和gydF4y2BapgydF4y2Ba我gydF4y2Ba在该位置的所有生态型中核苷酸的相对频率;gydF4y2Ba我gydF4y2Ba标记在给定位置的四种不同的核苷酸。gydF4y2Ba
熵为所有主题出现的每个位置平均,并由背景平均熵除以每个位置的突变指数;这允许将给定主题位置的熵与随机的熵值进行比较。为了定义计算突变指数所需的位置的平均熵,分析了芯片-SEQ峰的随机位置,以便它们的熵gydF4y2Ba拟南芥gydF4y2Ba生态型以类似的方式作为图案位置的熵分析。由于腺嘌呤和胸腺嘧啶的自发性突变率较慢,而不是胞嘧啶和鸟嘌呤[gydF4y2Ba28.gydF4y2Ba,在一个特定位置上出现的相同数量的核苷酸被作为背景分析。该背景一共计算了10,000次,并使用每个位置的平均值来计算突变指数:给定位置的所有motif出现的平均熵除以背景中该位置的平均熵。此外,计算一个位置的背景平均熵比实际平均熵高或低的次数。平均熵高于或低于95%或高于背景集的位置分别被认为比背景具有更少或更大的保守性。gydF4y2Ba
为了确定观察到的所有carg -box和完美carg -box子集之间的保存差异是否显著,以每个子集25个背景集的平均值,确定428个CArG-boxes(与完美CArG-boxes数量相同)的10,000个随机子集的突变指数,计算突变频率。在完美的carg -box中,突变指数高于或低于95%或超过10,000个随机集的位置分别被认为比完整的carg -box集更少或更保守。gydF4y2Ba
对于CArG盒中的每个位置,计算其突变指数(基于生态型的保守性)与该位置在所有ChIP-seq峰(Col-0基因组)中与基序匹配的熵之间的相关性。用公式H =−∑计算各位置的香农熵gydF4y2BapgydF4y2Ba我gydF4y2Ba日志gydF4y2Ba2gydF4y2BapgydF4y2Ba我gydF4y2Ba,在那里gydF4y2BapgydF4y2Ba我gydF4y2Ba是每个核苷酸的相对频率gydF4y2Ba我gydF4y2Ba在Col-0中的特定位置。gydF4y2Ba
结果gydF4y2Ba
类似CArG-box的motif在所有数据集中都得到了丰富gydF4y2Ba
来自芯片SEQ实验的原始数据AG [gydF4y2Ba13.gydF4y2Ba], AP1 [gydF4y2Ba15.gydF4y2Ba],ap3 [gydF4y2Ba16.gydF4y2Ba],FLC [gydF4y2Ba10.gydF4y2Ba],π[gydF4y2Ba16.gydF4y2Ba],SoC1 [gydF4y2Ba9gydF4y2Ba],SVP [gydF4y2Ba10.gydF4y2Ba]及SEP3 [gydF4y2Ba15.gydF4y2Ba]重新分析。一般数据集特性如表所示gydF4y2Ba1gydF4y2Ba.再分析后的结合位点数与原始文献中结合位点数的Pearson相关性为0.97。这表明,对于大多数数据集,推断的绑定位点的数量与原始出版物的数量相对相似。然而,对于两个数据集(AG和FLC),存在较大的差异(附加文件gydF4y2Ba2gydF4y2Ba:表S2)。基于峰位相似度对数据集进行聚类,除AG和FLC外,所有数据集的原始峰集和我们重新分析的峰集是最相似的(附加文件gydF4y2Ba3.gydF4y2Ba:图S1)。总的来说,这些结果强调了各种数据集的统一重新分析的重要性。gydF4y2Ba
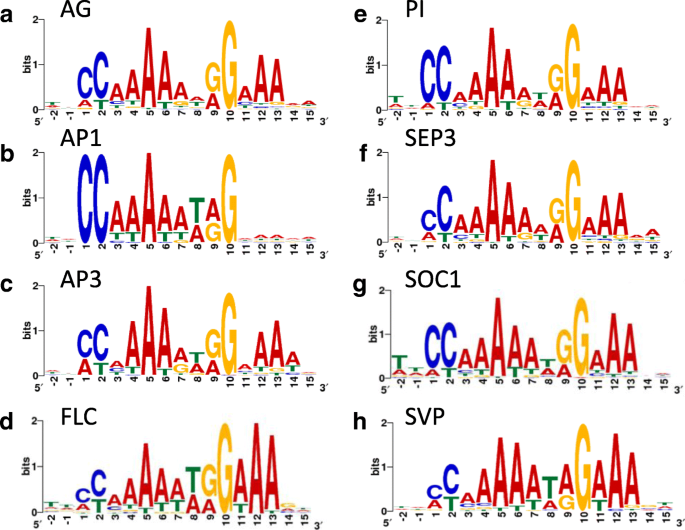
为了在每个数据集中找到丰富的motif,我们使用MEME-ChIP进行de novo motif发现[gydF4y2Ba20.gydF4y2Ba].此从头motif发现程序过表达的基序在中央100bp的一组给定的序列的搜索,并将其与背景,这是从在所提供的序列中的核苷酸的频率上进行。在所有数据集的基序相似于典型的CArG盒(CC(A / T)gydF4y2Ba6gydF4y2BaGG)(图。gydF4y2Ba1 a -gydF4y2Ba;额外的文件gydF4y2Ba4gydF4y2Ba:表S3)。gydF4y2Ba
在描述以下部分中的图案时,每个位置将以与规范碳盒箱匹配的方式编号。这个规范的碎片盒由10个位置组成。在这里描述的图案中,第一C与典型碳盒箱匹配的第一C将具有位置1,并且与典型碳盒匹配的最后一个G匹配的最后G将有位置10.在第一个C之前的位置在-1开始的负值和最后G后的位置将具有从11开始的正值。gydF4y2Ba
在不同的数据集中发现了类似于CArG-box的图案的相似和不同的特征。例如,所有主题的A/ t核心主要由A组成,特别是在位置5。此外,除AP1外的所有基序在12和13位均有较高的A 's发生。有趣的是,CArG-box的9号位置在所有的主题中几乎都是A和g。此外,CArG-box的1号和2号位置上的C通常分别有A或T作为替代。这种效应在AP3中最强,在AP1中最弱。gydF4y2Ba
主题在单个数据集的特点是缺乏一个主题后,近100%发生C在AP1位置1和2,相对较高的发生在位置7 G AP3, T在位置2−π和SOC1和高的T在AP1位置8,AP3,方法和高级副总裁。最后,对于11位的FLC和SVP, T是相对常见的替代a。值得注意的是,许多靶标同时被FLC和SVP结合[gydF4y2Ba10.gydF4y2Ba,它们能够形成异质二聚体,因此得到的基序高度相似。gydF4y2Ba
因为主题是如此相似,所以计算有多少碳乳盒,贡献了对整个图案的疾病,因为克盒只存在于单个TF的峰值(请注意,唯一性是指的这到了gydF4y2Ba位置gydF4y2Ba基因组中;这样的CArG-box可能有也可能没有独特的gydF4y2Ba序列gydF4y2Ba功能)。对于除SEP3和SVP以外的所有数据集,一个数据集的绝大多数carg -box发生在一个或多个其他数据集(附加文件gydF4y2Ba5gydF4y2Ba:表S4)。我们还计算了唯一峰的百分比(意思是一个峰只存在于一个TF的数据集中;见附加文件gydF4y2Ba6gydF4y2Ba:表S5)。De Novo Motif在这些独特的峰值中发现导致了像PI,SEP3和SOC1特定的图案这样的碎屑盒发现。这些图案与全套中的主题类似,具有一些微小的差异(附加文件gydF4y2Ba7gydF4y2Ba:图S2)。由所有8个TFS(全重叠)限定的特定碎片箱的百分比非常低,表明疯子域TFS对特定的碳盒图案具有结合特异性。gydF4y2Ba
完美的CArG-box是最相关的CArG-box变体gydF4y2Ba
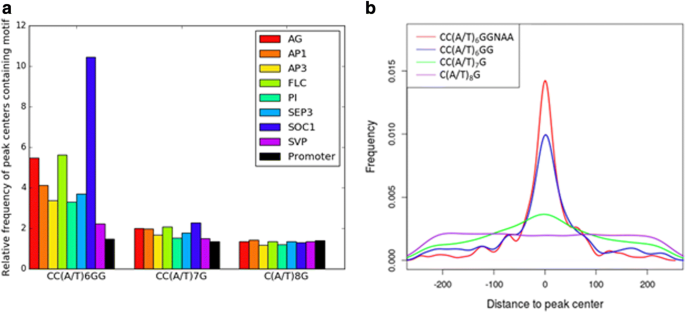
基于序列标志,Carg-Box像不同的MADS TFS的图案似乎有很多类似的功能。因此,进一步检查了所有数据集中的套件箱的一般特征。首先,完美的碎片箱的相对富集(CC(A / T)gydF4y2Ba6gydF4y2BaGG)和文献中已知的两种常见变异(CC(A/T))gydF4y2Ba7gydF4y2Bag和c(a / t)gydF4y2Ba8gydF4y2BaG)峰中心与背景启动子区域的比较被确定。峰中心定义为峰顶上游和下游的250 bp。gydF4y2Ba
理想的CArG-box富集程度最高,SVP的富集倍数约为2倍,AP3和PI的富集倍数约为3倍,SOC1的富集倍数超过10倍。gydF4y2Ba2gydF4y2Ba).CC变体的相对富集(A/T)gydF4y2Ba7gydF4y2BaG值要低得多,PI的G值约为1.5倍,其他数据集的G值约为2倍,SVP除外(图2)。gydF4y2Ba2gydF4y2Ba).在所有数据集中,变量C(A/T)gydF4y2Ba8gydF4y2Ba与启动子背景相比,G未富集(图。gydF4y2Ba2gydF4y2Ba).gydF4y2Ba
在ChIP-seq数据中,预期感兴趣的蛋白质直接结合到DNA的峰顶下。因此,大多数直接结合基序出现在峰顶附近,而未结合基序则均匀分布在整个峰。因此,相关的主题将被集中丰富。为了确定CArG-box不同变体的相关性,在不同的数据集中确定了它们的集中富集。数字gydF4y2Ba2 bgydF4y2Ba显示SEP3的基序变体的中央富集。其他蛋白质数据集中的主题中的中央富集显示了SEP3数据集中的主题与图案相同的趋势(附加文件gydF4y2Ba8gydF4y2Ba:图S3)。gydF4y2Ba
在所有数据集中,完美的碎片盒是三个碎片盒式变体中最集中的富集的主题。变体CC(A / T)gydF4y2Ba7gydF4y2BaG的集中浓缩程度要低得多而母题变型C(A/T)gydF4y2Ba8gydF4y2Ba根本不是集中浓缩的gydF4y2Ba
因为在序列标识中,A的位置12和13似乎是重要的,中央富集与扩展-NAA的完美CArG-box进行了测试。这个CArG-box变种,CC(A/T)gydF4y2Ba6gydF4y2BaGGNAA甚至比没有扩展的完美CArG-box更集中富集(图。gydF4y2Ba2 bgydF4y2Ba).中央浓缩的CC(A/T)gydF4y2Ba7gydF4y2BaG motif在5 ' -NAA-3 '延伸时也较高,但仍低于完美的CArG-box。相反,母题C(A/T)gydF4y2Ba8gydF4y2BaGnaa仍然没有居中富集(数据未显示)。gydF4y2Ba
确保结果主题浓缩浓缩和中部没有偏见的存在弱峰,分析重复只使用每个契尼的前500的峰值蛋白质(除了方法之外,SOC1和高级,只有59岁,分别在301年和445年的峰值,并留下相同的分析)。使用该分析得到的结果与上述报告的结果在定性上相似。此外,我们还检查了测试结果是否会受到一些测试基序比其他基序更退化这一事实的人为影响。作为阴性对照,motif CC(a /T)gydF4y2Ba6gydF4y2Ba用GGNTT代替CC(A/T)gydF4y2Ba6gydF4y2Baggnaa,和图案cc(a / t)gydF4y2Ba6gydF4y2BaCG代替CC(A/T)gydF4y2Ba6gydF4y2BaGG;阴性对照确实远远不如符合主题的富集。最后,我们测试了中央富集可能受到更加简并基序在给定峰区域中多次发生的事实的影响。我们发现变体CC(A / T)gydF4y2Ba7gydF4y2Bag和c(a / t)gydF4y2Ba8gydF4y2BaG实际上比规范图谱CC(A / T)更常见在一起gydF4y2Ba6gydF4y2BaGG。但是,绝大多数克隆盒式变体仍然只在峰值中心内发生一次(附加文件gydF4y2Ba9gydF4y2Ba:表S6,面板A)。因此,这不大可能影响集中富集的分析。同样,当比较不同CArG-box变体在峰值中的共现组合时,也没有很强的趋势(附加文件gydF4y2Ba9gydF4y2Ba:表S6,面板B-D);共现的频率总是与包含每个母题变体的峰值频率的预期相同。gydF4y2Ba
其他主题也在数据集中丰富gydF4y2Ba
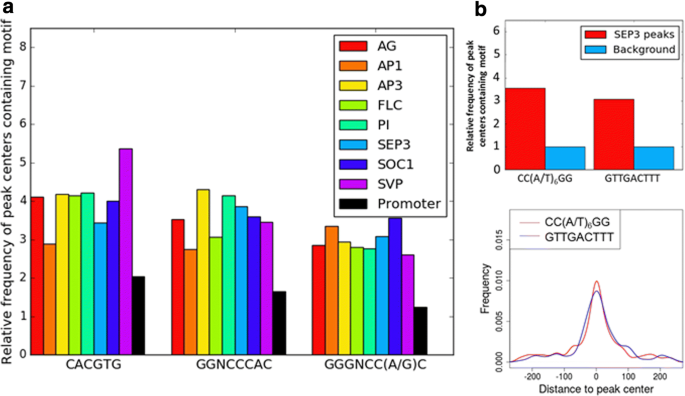
使用MEME芯片的De Novo Motif发现也导致了其他丰富的主题的发现(概述在表格中)gydF4y2Ba2gydF4y2Ba).在除FLC和SVP以外的所有数据集中,几乎完全由a和G组成的motif被富集(附加文件)gydF4y2Ba10.gydF4y2Ba:图S4)。与启动子背景相比,该motif几乎不富集(小于1.5倍);它也不是集中富集的,因此,很可能与MADS域TF绑定无关。文学中的三个主题[gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba29.gydF4y2Ba]在一些数据集中也得到了丰富:除了AP1、FLC和SOC1数据集之外,其他数据集中都发现了类似于G-box的motif(附加文件gydF4y2Ba11.gydF4y2Ba:图S5),而类似于TCP类型II的motif在AP1和SOC1中发现,而TCP类I和类II的motif在SEP3数据集中发现(附加文件gydF4y2Ba12.gydF4y2Ba:图S6)。gydF4y2Ba
与用作背景模型的核苷酸频率相比,MEME-CHIP可能忽略不强烈富集的较弱的基序,但与基因组的其余部分中的频率相比,仍然富集。为了克服这个问题,还进行了与启动子背景相比,G字幕框架和两个TCP-MOTIF的超重分析。与所有数据集中的背景相比,所有三个图案都在峰中心中富集(图。gydF4y2Ba3.gydF4y2Ba).在所有数据集中,三个基模都显示富集向峰值中心方向,尽管这种富集强度低于CArG盒(附加文件gydF4y2Ba13.gydF4y2Ba:图S7)。所有的主题在数据集中都差不多集中丰富。carg -box的出现与峰中的其他图案之间没有明显的相关性。gydF4y2Ba
以上,通过与背景相比,通过分析富集来发现图案,然后分析中央富集。要自行分析中央浓缩,写入了一个旨在找到集中富集的主题的自定义Python脚本。通过此脚本,丰富低的新图案(GTTGACTTT)(4447峰的89例),但在SEP3数据集中发现了完美的碎片盒中的集中富集(图。gydF4y2Ba3 b, cgydF4y2Ba).该motif在SEP3的ChIP-seq峰的相对富集也可与完美的CArG-box相媲美(图。gydF4y2Ba3 b, cgydF4y2Ba).基序只在48个病例中出现在与CArG-box相同的峰值,比预期的概率低25%。该基序与WRKY转录因子的W-box基序相似,具有一致的TTGACC/T [gydF4y2Ba30.gydF4y2Ba].gydF4y2Ba
不同MADS蛋白结合位点的单个CArG-box序列gydF4y2Ba
如上所述,一个完美的CArG-box (CC(a /T))gydF4y2Ba6gydF4y2BaGG)在不同的MADS转录因子中均显著富集。为了进一步研究不同MADS转录因子结合位点之间的潜在差异,我们对结合位点上出现的完美CArG-box的单个序列进行了分析。关注CArG-box序列本身时,最引人注目的模式是一个偏爱马德斯TFs连续延伸的特别是,不同的完美CArG-box包含至少连续三核苷酸序列占79%(高级)- 94%(方法)的序列。这些数字比随机预期的要高得多:在所有不同的完美CArG-box变种中,36个中有20个,即56%包含至少三个连续的a。当需要一个更特异的变异,至少有四个连续的A核苷酸时,仍然有45% (SVP) - 61% (SOC1)的个体序列包含这样的AAAA延伸。这里随机期望的差异甚至更大:在所有不同的完美CArG-box变体中,只有8 / 36,即22%包含至少4个连续的a。gydF4y2Ba
对完美CArG-box中单个序列的分析并没有表明不同的MADS转录因子之间存在明显的差异。当对完美CArG-boxes的3 '扩展进行类似的分析时,得到了不同的tf之间的一些区别。如附加文件所示gydF4y2Ba14.gydF4y2Ba图S8,虽然SEP3出现了大量的序列,但对于其他MADS转录因子,观察到更多的限制性集合。基于优选的扩展序列对TFs进行聚类,发现相关的MADS TFs对:PI和AP3聚在一起,FLC和SVP也聚在一起(附加文件gydF4y2Ba14.gydF4y2Ba:图S8)。然而,在几乎所有的情况下,PI和AP3或FLC和SVP同时发生的序列都是这两个tf发生相同结合位点的情况。除了MADS TFs之间的这些差异之外,对个体序列的分析再次表明了对NAA扩展名的明显偏好(附加文件gydF4y2Ba14.gydF4y2Ba:图S8)。gydF4y2Ba
Carg-Box Extensions的超级陈述表明存在混合套装 - TCP绑定站点gydF4y2Ba
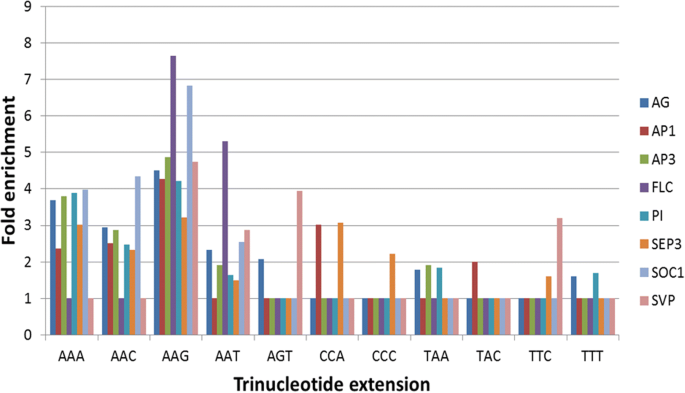
为了进一步研究CArG-boxes的3 '扩展的本质,我们分析了特定扩展的过度代表性。首先,研究了与MEME-ChIP定义的CArG-box样序列直接相邻的核苷酸。我们用5 '和3 '表示这些核苷酸的位置,相对于被MEME-ChIP防卫的CArG-box方向,这是面向对齐尽可能多的腺嘌呤在CArG-box核心。统计了3个核苷酸在1号位置的5 '侧(来自5 '延伸)和10号位置的3 '侧(来自3 '延伸)以及5 '和3 '延伸组合的不同序列的出现情况。可以看到一个惊人的模式(附加文件gydF4y2Ba15.gydF4y2Ba:表S7)。虽然在3'侧面,但对延伸5'-AAA-3'后跟5'-NAA-3'的变体,对5'侧的某些序列的偏好进行了清晰的偏好,较弱。通常,序列5'-AAA-3'和5'-TTT-3'最常发生。gydF4y2Ba
接下来,我们专注于人工定义的完美CArG-boxes的3 '侧的核苷酸。对于每个数据集,CC发生后三种核苷酸的所有组合的频率(A/T)gydF4y2Ba6gydF4y2Ba根据分析的ChIP-seq峰的核苷酸分布,对ChIP-seq峰中的GGN进行计数并除以预期频率。通过与随机排列比较(方法)评估统计学意义。在所有数据集中,不同的A的组合显著富集(图。gydF4y2Ba4gydF4y2Ba).这符合上面讨论的含A扩展的重要性。然而,在SEP3的数据集中,显着富集了两种额外的核苷酸组合,即5'-CCC-3'和5'-CCA-3'(图。gydF4y2Ba4gydF4y2Ba).将这些扩展添加到该分析中使用的Carg-Box核心给出了MOTIF CC(A / T)gydF4y2Ba6gydF4y2BaGGNCCC或CC (A / T)gydF4y2Ba6gydF4y2BaGGNCCA,这是一个CArG-box和核心的I类(GGNCCC)和II类(GGNCCA) TCP绑定motif的组合。然而,需要注意的是,扩展CCA和扩展CCC的发生频率仅为31,但由于ChIP-seq峰中C的频率较低,这仍然是一个显著的富集。gydF4y2Ba
显着超越的3'克盒核心的延伸。将3个核苷酸的延伸的浓缩计算为CC之后的延伸频率(A / T)gydF4y2Ba6gydF4y2Ba基于芯片-SEQ峰的核苷酸的频率,Chink-SEQ峰的GGN核差分除以延伸的预期频率。描绘了所有扩展,至少一个数据集是重要的gydF4y2BapgydF4y2Ba< 0.05。为了可视化的目的,相对于预期的核苷酸频率,但不显著的所有扩展都被设置为1。请注意,在附加文件中有一个类似的分析,但是针对MEME-ChIP获得的类似CArG-box的序列gydF4y2Ba15.gydF4y2Ba:表S7gydF4y2Ba
除了结合的CArG盒 - TCP的主题,当芯片起峰分析TCP图案的主题主场迎战的CArG盒的TCP的位置更普遍的趋势观察,特别是对完美的CArG盒。相比于MADS芯片起峰所有的CArG盒(完美和不完美的),有对TCP图案被定位3'的CArG盒(261×TCP图案3'的CArG盒与轻微的偏好217×TCP基序5'到的CArG盒)。然而,只用完美的CArG盒时,这种偏好是强多了,有68例在TCP图案3'的CArG盒,只有两例的TCP图案5'到的CArG盒。请注意,只有在一个完美的CArG盒一个TCP主题的情况下,可形成3'的CArG盒作为混合MADS-TCP结合位点。gydF4y2Ba
完美的CArG-boxes比全套的CArG-boxes保存得更好gydF4y2Ba
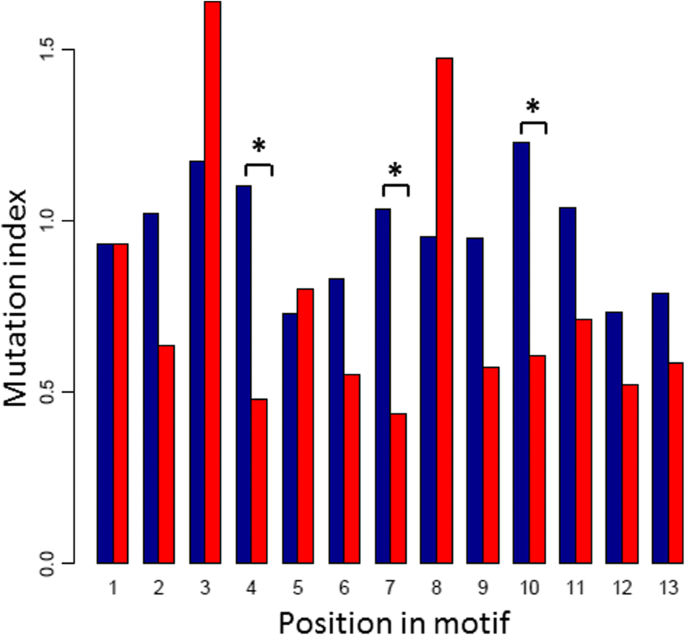
保存在595个货柜内gydF4y2Ba拟南芥gydF4y2Ba对生态型进行评估,以研究CArG-boxes的进化重要性。几乎三分之二的分析carg -box(从Col-0 ChIP-seq数据获得)在595个生态型中至少有一个突变(附加文件gydF4y2Ba16.gydF4y2Ba:表S8;额外的文件gydF4y2Ba17.gydF4y2Ba:图S9,面板A)。使用所有生态型的所有carg -box的每个位置计算突变指数。简而言之,该突变指数表明了所有生态型之间该位置的突变变异性与ChIP-seq峰值的背景突变变异性相比,考虑到一些核苷酸比其他的突变更快。突变指数的计算方法是将motif中给定位置的平均熵除以每个位置计算的平均背景熵(方法)。突变指数为1表示位置和背景一样保守,突变指数越低表示位置比背景更保守。gydF4y2Ba
对于全套carg -box,总体平均突变指数为0.96(+/−0.15)。相对保守的位置是5、6、12和13号位点,而分析生态型时变化最大的位置是3和10号位点(图5)。gydF4y2Ba5gydF4y2Ba).总的来说,趋势是基于ChIP-seq峰匹配的motif标识中变化较小的位置(图2)。gydF4y2Ba1gydF4y2Ba)之间的可变变量较小gydF4y2Ba拟南芥gydF4y2Ba突变指数较低,但10号位置除外(附加文件)gydF4y2Ba17.gydF4y2Ba换句话说,尽管在CArG-box motif中,位置10被强烈定义为“G”(图。gydF4y2Ba1gydF4y2Ba)在不同生态型之间的保守性相对较差。该“G”在生态型中观察到明显的突变趋势。在522个独特的变化(发生在一个或多个生态型)中,超过一半(282个)发生在a上,54个发生在C上,120个发生在T上,有66个缺失。在所有这些情况下,CArG-box会根据图中所示的标志被销毁。gydF4y2Ba1gydF4y2Ba.gydF4y2Ba
有趣的是,当只从完整集中对完美的carg -box进行子抽样时,平均突变指数大大降低,为0.76(+/−0.36)。这意味着完美的CArG-boxes比非完美的CArG-boxes保存得更好。在perfect carg -box中,除3和8位置外,所有位置的突变指数都小于1,即它们比背景更保守(图3)。gydF4y2Ba5gydF4y2Ba).值得注意的是,当考虑到所有的carg -box时,位置10是不保守的,在完美carg -box中是高度保守的(突变指数0.61)。gydF4y2Ba
分析完美和不完美的CArG盒之间的观察差异显着,万个随机子样本是从全套的CArG盒取出,并计算在每个子样本每个位置的突变指数。为位置4,7和10,发现在95%或更多的情况下,突变指数是在完美的CArG盒的子样本比在随机子样本低,这表明在突变索引的子集之间的观察到的差异完善的CArG盒和全套是显著(在图中标有星号。gydF4y2Ba5gydF4y2Ba).对于类似的分析,发现完美克隆箱的所观察到的位置3和8的较高突变指标并不重要。gydF4y2Ba
讨论gydF4y2Ba
以前关于MADS-域蛋白的研究表明,该蛋白质类的优选DNA结合基质是碳盒,其具有CC(A / T)gydF4y2Ba6gydF4y2BaGG.一些使用ChIP-seq对不同蛋白质的研究揭示了在gydF4y2Ba拟南芥gydF4y2Ba.然而,由于分析方法的不同,无法根据这些文章中描述的数据来比较mads结构域蛋白首选结合基序的异同。在本研究中,我们收集了8组mads结构域蛋白的ChIP-seq数据集gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba重新分析。通过观察内部carg -box的保存,从进化的角度分析了carg -box的重要性gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba生态型。gydF4y2Ba
使用全新motif发现工具MEME-ChIP,在所有数据集中都发现了高度相似的CArG-box样motif。这可能是因为mads结构域蛋白主要以异源二聚体或(异源)四聚体的形式结合DNA,而且在特定的ChIP样品中,mads结构域蛋白可以形成不同的异源二聚体(专用AP3-PI异源二聚体除外)。因此,结合基序实际上代表了一个“平均”基序,它是由多个异质二聚体组合的基序组成的。通过这种分析,特定于蛋白质的基序可能会被大量的其他序列所掩盖,而这些序列对该蛋白质的特异性较低,甚至根本无法检测到。我们不能完全排除的另一种解释是,不同的CArG-box样motif,尽管高度相似,但包含足够的变异,至少在某种程度上区分不同的MADS tf。使用更有预测性的计算方法而不是描述性主题搜索的进一步研究可能会更清楚地说明这一点。gydF4y2Ba
接下来,我们研究了有多少CArG-boxes是特定数据集所特有的(这意味着它们出现的位置只是该特定数据集中峰值的一部分)。注意,这样的CArG-boxes可能有也可能没有独特的序列特征。除了SEP3和SVP,这个数量与CArG-boxes的总量相比非常低,甚至与一个数据集重叠的CArG-boxes数量也相对较低。这表明,许多carg -box对特定的蛋白质不是特异性的,但也可以与其他mads结构域复合物结合。然而,在不同mads域的tf之间,carg -box和扩展存在微小的变化。这些偏好是否反映了靶基因调控的选择性结合和特异性仍有待观察。gydF4y2Ba
为了找到额外的类似CArG-box的序列,我们在每个数据集的独特峰值中进行了从头motif发现。我们推测,通过这种方式,我们可能不仅会发现已经发现的CArG-box,而且可能还会发现看起来像CArG-box的序列,但在使用数据集的所有峰值进行分析时,这些序列并不被认为是基序的一部分。然而,在这些独特的峰中只发现了PI、SEP3和SOC1的CArG-box样motif。在PI、SEP3和SOC1的独特峰中的carboxes形成的基模与各自数据集的所有峰的基模略有不同。这表明,这些峰值的一部分是由蛋白质与特定的CArG-box结合引起的。gydF4y2Ba
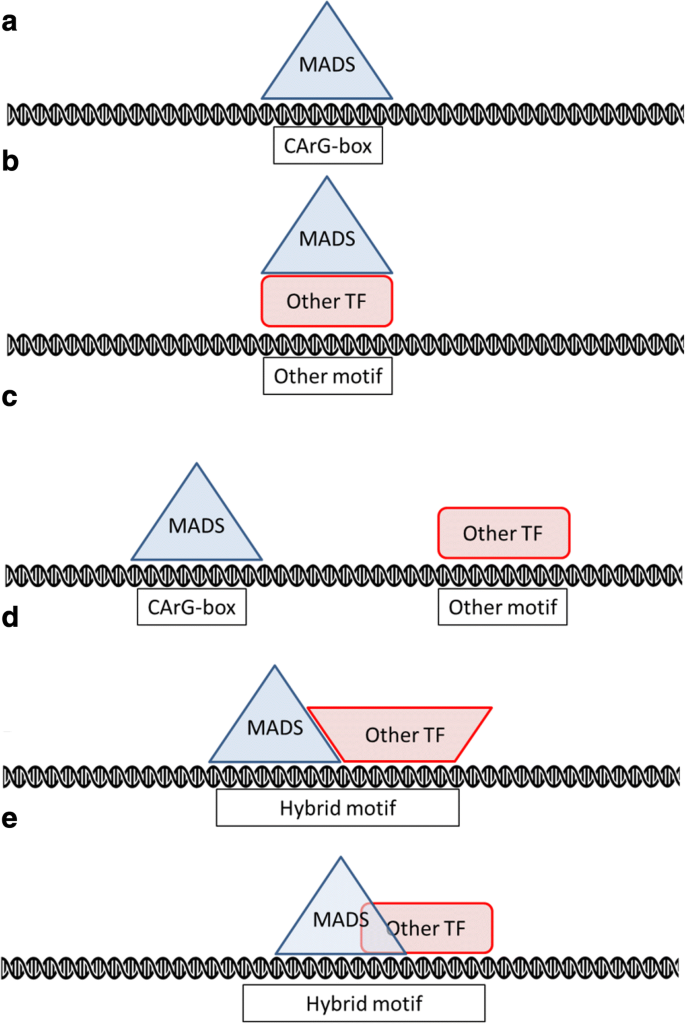
综上所述,CArG-box的一般motif在不同的数据集分析中相当相似。这就提出了一个问题:除了CArG-box序列,是否还有其他特性使mads结构域蛋白能够与特定的基因组序列结合,并调节不同的生理和发育过程。当然,不同蛋白质的基序变化有限可能是由于模因所使用的位置权重矩阵(PWM)基序模型的局限性。例如,PWM不考虑依赖关系,例如,在CArG-box motif中,位置2的基座依赖于位置9的基座。然而,其他的解释也是可能的(图。gydF4y2Ba6gydF4y2Ba).可能的情况是不同的蛋白质结合到相同的序列,但调节相同的基因差异[gydF4y2Ba12.gydF4y2Ba].另一种可能性是它们与某些共同因素或其他促进合作的TF互动,这对特异性可能很重要(图。gydF4y2Ba6 bgydF4y2Ba和gydF4y2BadgydF4y2Ba).尽管在不同的数据集中发现的母题基本相似,但在本研究中对其他几个母题的识别支持了这一假设。此外,CArG-boxes之间的间距也可能是重要的,因为MADS-domain tf能够以四聚体的形式结合到两个相邻的carg -box上,这在quarte -model中被提出[gydF4y2Ba31.gydF4y2Ba].当四聚体与两个carg -box结合时,协同性可能会促进TF与“弱”结合位点的结合,从而模糊整体基元。gydF4y2Ba
ChIP-seq数据分析了5种解释mads结构域蛋白中不同结合基序出现的模型。gydF4y2Ba一个gydF4y2Bamads结构域蛋白与CArG-box结合。gydF4y2BabgydF4y2Bamads结构域蛋白与另一个转录因子结合,该转录因子在该转录因子特异的基序上结合DNA。gydF4y2BacgydF4y2Ba一样(gydF4y2Ba一个gydF4y2Ba),但由于偶然或作为增强体的一部分,附近有另一个转录因子的结合位点,CArG-box和其他motif均出现在ChIP-seq峰。gydF4y2BadgydF4y2BaMADS-域蛋白需要另一种转录因子,用于结合在碎片箱和其他转录因子的基序之间的杂交之间的基序。gydF4y2BaegydF4y2Ba该基序被mads结构域蛋白和另一个蛋白竞争性结合,因此是CArG-box和其他转录因子基序之间的混合gydF4y2Ba
可以将浊域蛋白与特异性DNA的结合的另一方面是DNA的结构性质。最近证明,考虑DNA形状描述符描述各种转录因子的DNA结合位点的可预测性,包括疯子域TFS可以改善[gydF4y2Ba32.gydF4y2Ba].更具体地说,我们以前注意到SEP3结合峰的A / T芯的盒式盒类似于称为A-TRACT的结构基质[gydF4y2Ba33.gydF4y2Ba,与DNA向小槽弯曲有关的基序[gydF4y2Ba34.gydF4y2Ba].由TF结合与A-TRACT产生的DNA弯曲程度被假设以有助于特异性。在本研究中,我们确认了我们分析的各种MADS TFS的结合位点中连续延伸的存在。此外,规范套装的3'侧的序列大部分包括A的。注意,显然对于一个人也可以在此处读取T,具体取决于股线;重点是凯盒盒的核心和延伸,不是A和T的随机组合。gydF4y2Ba
因为CArG-boxes在数据集之间高度相似,所以我们接下来关注的是在所有数据集中CArG-boxes的相似方面。基于相对富集和集中富集,完美的CArG-box似乎是文献中描述的三种通用CArG-box变体中最重要的母题[gydF4y2Ba11.gydF4y2Ba,gydF4y2Ba12.gydF4y2Ba].变异体C(A/T)gydF4y2Ba8gydF4y2BaG仅相对富集而非集中富集,说明该motif对于本研究分析的mads结构域蛋白的结合并不重要,尽管不能排除该motif的部分序列仍然被这些蛋白结合。gydF4y2Ba
在八个数据集中学习,具有延伸NAA的完美碎片箱的中央富集以及变体CC(A / T)gydF4y2Ba7gydF4y2BaG加NAA的扩展比单独的每个核心基序的高。此外,扩展NAA出现在MEME-ChIP在所有数据集中发现的CArG-boxes的很大比例中。这强调了12和13位腺嘌呤的重要性,并为将完美CArG-box的共识motif扩展到CC(a /T)提供了强有力的理由。gydF4y2Ba6gydF4y2BaGGNAA。此外,我们惊奇地看到,存在用于单侧扩展偏好。这是特别有趣的是考虑到MADS-box蛋白结合的二聚体,因此预期结合或多或少回文图案。的概念,即一个的CArG盒实际上不是回文是一个事实,即A / T的核心主要是由A和A和T的不是随机组合的,如前面所讨论的支持。对于此可能的解释是,这种非回文基序导致促进MADS-box蛋白结合(如上文所讨论的)的DNA结构,或MADS-box蛋白通常结合作为异二聚体和所述的CArG盒的每一侧为的两个结合配偶一个优化。gydF4y2Ba
有趣的是,根据对SEP3中完美CArG-box的3 '扩展的分析,扩展5 ' -NCCC-3 '和5 ' -NCCA-3 '被过度代表。具有此类扩展的motif除了包含一个完美的CArG-box外,还包含序列GGNCCC或GGNCCA,分别是TCP I类motif和II类motif的核心[gydF4y2Ba29.gydF4y2Ba].一个有趣的假设是,这些基元是SEP3和TCP蛋白之间的混合结合位点(图。gydF4y2Ba6 dgydF4y2Ba).虽然还没有测试这种混合复合物是否可以在物理上如此接近地结合两段DNA,但最近已经证明,两个转录因子可以在体外结合到“混合”基元,这一模型被称为潜在特异性[gydF4y2Ba35.gydF4y2Ba].在该模型中,结合可以是合作的,需要SEP3和TCP蛋白以获得最佳结合。另一个解释是,该基序是TCP蛋白和SEP3之间与促进剂结合的竞争的部位(图。gydF4y2Ba6 egydF4y2Ba).在这种情况下,预期两种蛋白质会差异调节靶基因。gydF4y2Ba
除了这些混合网站之外,发现了来自疯狂家庭以外的已知转录因子家族的四个不同的图案。这些是G盒,其受BHLH和BZIP蛋白的约束[gydF4y2Ba36.gydF4y2Ba,gydF4y2Ba37.gydF4y2Ba, TCP I类和II类蛋白质的基序[gydF4y2Ba29.gydF4y2Ba[类似于Wrky Proteins [gydF4y2Ba38.gydF4y2Ba].一种假设的解释模型是mads结构域蛋白与另一类转录因子相互作用,这些转录因子与特定的DNA序列结合,而不依赖于CArG-box基序。正如最近回顾的,不同家族的转录因子之间的这种相互作用越来越被认为是重要的[gydF4y2Ba39.gydF4y2Ba].当MADS结构域蛋白进行免疫沉淀,与其他TF和DNA复合物也被沉淀,最终导致在其他基序(图读富集。gydF4y2Ba6 bgydF4y2Ba).或者,也可以富集图案,因为它们出现在相同的启动子区域中,其中碎屑盒定位并形成具有定义的多个TF结合位点的所谓的增强体[gydF4y2Ba39.gydF4y2Ba(图。gydF4y2Ba6摄氏度gydF4y2Ba).所有基序的集中富集表明,至少有一些基序被发现,因为它们与mads结构域蛋白一起免疫沉淀,如模型6b (mads结构域蛋白与其他转录因子之间的相互作用)中解释的那样。同样,类似wrky的母题脱颖而出,因为它比其他母题更集中丰富。这说明在SEP3数据集中出现的WRKY-like motif主要是由MADS-WRKY相互作用造成的。最近的一项分析gydF4y2Ba拟南芥gydF4y2BaChIP-seq数据(gydF4y2Ba40gydF4y2Ba[还分析了多种转录因子的共结合;具体而言,观察到G盒与浊域蛋白相关,但在该研究中未发现TCP结合位点和WRKY选项。gydF4y2Ba
有趣的是,虽然mads结构域蛋白和TCP蛋白或bHLH蛋白之间的相互作用已经在一些生物体中被描述[gydF4y2Ba39.gydF4y2Ba,gydF4y2Ba41gydF4y2Ba,gydF4y2Ba42gydF4y2Ba据作者了解,mads结构域蛋白与WRKY转录因子之间的相互作用尚未见报道。然而,WRKY转录因子与开花时间和生长有关[gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba],表明Mads Wrky相互作用可能与花发育有关。gydF4y2Ba
除了已知的转录因子基序外,在每个数据集中还发现了一个可变长度的基序,主要由G′s和a′s组成。这个motif很可能是GA元素的一部分,GA是一种启动子元素,被描述为TATA-box的替代[gydF4y2Ba47gydF4y2Ba].由于mads结构域蛋白主要与启动子区域结合,与基因组背景相比,mads结构域蛋白的ChIP-seq峰中通常富含该元素,这是有道理的。这个主题几乎浓缩与启动子的背景和比较时也不是集中浓缩符合假设这个元素出现在数据仅仅是因为它位于接近绑定网站,但不直接或间接地受MADS-domain蛋白质。gydF4y2Ba
为了更好地了解CArG-box的进化重要性和一个CArG-box内的不同核苷酸,我们调查了不同CArG-box之间的保护gydF4y2Ba拟南芥gydF4y2Ba生态型。令人惊讶的是,当考虑到所有的carg -box时,大多数位置在ChIP-seq峰中并没有比随机位置更好的保存。这包括位置1和2的C和位置9和10的G。然而,当只分析完美的CArG-boxes时,大多数位置比背景更保守。这表明这种类型的CArG-box通常比非完美的CArG-box更重要。gydF4y2Ba
对于车箱的保存情况通常并不比背景好多少这一事实,有几种解释。首先,没有考虑到突变的性质。这意味着可能存在无害突变。在未来,观察某些突变是否比其他突变发生得更频繁将是一件有趣的事情。如果编码重要调节蛋白的基因在其启动子中只有带有非有害突变的carg -box,那将是一项有趣的研究。对于CArG-box没有很好的保存这一事实的另一个解释可能是,CArG-box中的一个突变被CArG-box中的另一个突变所补偿。这在本研究中没有考虑。最后,由于许多启动子包含多个CArG-box,其他的CArG-box有可能取代一个突变的CArG-box,并维持相应基因的调控。在任何情况下,完美的CArG-boxes更加保守的特性强调了它们的重要性。gydF4y2Ba
结论gydF4y2Ba
本文通过重新分析ChIP-seq数据,分析了八个不同mads结构域蛋白与DNA结合的几个方面。mads结构域蛋白结合到一个叫做CArG-box的DNA基序上。根据本文的分析,可以得出结论,CArG-box的确切定义比大多数以前的论文所建议的更加灵活。确切的顺序通常与经典的定义CC(A/T)不同gydF4y2Ba6gydF4y2Ba此外,在相同的DNA区域内,大多数位置几乎与其他核苷酸一样经常发生突变gydF4y2Ba拟南芥蒂利亚纳gydF4y2Baevotypes,特别是当碎片箱不符合经典定义时。然而,克隆盒的一些一般特征似乎对疯狂域转录因子的结合很重要。例如,符合Carg-Box的规范定义的碎屑盒比其他碎片盒更富有相对且集中的富有富集和更好的保守。此外,带有3'延伸5'-NAA-3'的碎片盒比没有这种延伸的碎屑盒更富有富有碎屑盒。在所有数据集中,还富集了其他已知的转录因子家族的几个图案,表明intercamily转录因子相互作用的重要性。发现在不同的疯狂域芯片-SEQ数据集中富有富有疯狂的盒式图案是相似的,但对于每个MAD域TF也不相同。这些小差是否足以解释仍有待确定的靶基因调节的提出的特异性。对特异性和转录因子的更具决定性,如何调节特定过程,芯片数据应与更多的定量DNA结合数据(亲和力)互补,更多关于TF复合物组成的更多信息,包括共同因素,以及合作的作用DNA结合和基因调控。gydF4y2Ba
缩写gydF4y2Ba
- AG:gydF4y2Ba
-
无性生殖的gydF4y2Ba
- AP1:gydF4y2Ba
-
Apetala1.gydF4y2Ba
- AP3:gydF4y2Ba
-
Apetala3.gydF4y2Ba
- bHLH:gydF4y2Ba
-
碱性螺旋-环-螺旋(TF族)gydF4y2Ba
- 英国石油公司:gydF4y2Ba
-
完全gydF4y2Ba
- bzip:gydF4y2Ba
-
基本亮氨酸拉链(TF系列)gydF4y2Ba
- CArG:gydF4y2Ba
-
c - a / T-rich-GgydF4y2Ba
- 芯片SEQ:gydF4y2Ba
-
染色质immunoprecipitation-sequencinggydF4y2Ba
- 罗斯福:gydF4y2Ba
-
错误发现率gydF4y2Ba
- FLC:gydF4y2Ba
-
开花基因座C.gydF4y2Ba
- 麦斯:gydF4y2Ba
-
MCM1,静静,缺乏症和SRF(TF家族)gydF4y2Ba
- PI:gydF4y2Ba
-
PISTILLATAgydF4y2Ba
- PWM:gydF4y2Ba
-
位置权重矩阵gydF4y2Ba
- SEP3:gydF4y2Ba
-
Sepallata3.gydF4y2Ba
- SNP:gydF4y2Ba
-
单核苷酸多态性gydF4y2Ba
- SOC 1:gydF4y2Ba
-
constans过表达抑制因子1gydF4y2Ba
- SVP:gydF4y2Ba
-
营养期短gydF4y2Ba
- TCP:gydF4y2Ba
-
TB1,CYC和PCF(TF家族)gydF4y2Ba
- TF:gydF4y2Ba
-
转录因子gydF4y2Ba
- WRKY:gydF4y2Ba
-
核心氨基酸基序的名字命名(WRKYGQK)TF系列gydF4y2Ba
参考文献gydF4y2Ba
- 1。gydF4y2Ba
Smaczniak C,Immink Rg,Angenent GC,Kaufmann K.植物疯子域因素的发育和进化多样性:最近研究的见解。发展。2012; 139(17):3081-98。gydF4y2Ba
- 2。gydF4y2Ba
陆生植物mads结构域蛋白的结构模块化、蛋白质相互作用和网络进化。基因。2005;347(2):183 - 98。gydF4y2Ba
- 3.gydF4y2Ba
德Folter S,Immink RG,基弗尔男,Parenicova L,Henz SR,威格尔d,等。拟南芥MADS综合互动地图框的转录因子。植物细胞。2005; 17(5):1424至1433年。gydF4y2Ba
- 4.gydF4y2Ba
许志强,王志强,王志强,等。金鱼草花的同源性MADS-box基因缺陷的特征:DNA结合和其在花发育过程中持续表达的自动调节的证据。EMBO j . 1992; 11(1): 251 - 63。gydF4y2Ba
- 5.gydF4y2Ba
E类花同源性蛋白SEPALLATA3在离体的“花四边形”复合物中足够环DNA。核酸学报2009;37(1):144-57。gydF4y2Ba
- 6.gydF4y2Ba
Mendes MA, Guerra RF, Berns MC, Manzo C, Masiero S, Finzi L,等。在拟南芥中,MADS结构域转录因子介导了靶基因表达所必需的DNA短链。植物细胞。2013;25(7):2560 - 72。gydF4y2Ba
- 7.gydF4y2Ba
Theissen G,Saedler H.植物生物学。花卉四重奏。自然。2001; 409(6819):469-71。gydF4y2Ba
- 8.gydF4y2Ba
等。关键词:人工神经网络,神经网络,神经网络SEPALLATA3: MADS盒转录因子复合物形成的“胶水”。基因组医学杂志。2009;10 (2)::R24。gydF4y2Ba
- 9.gydF4y2Ba
等。通过识别其上游和下游的调控因子来描述SOC1在开花过程中的中心作用。植物杂志。2012;160(1):433 - 49。gydF4y2Ba
- 10。gydF4y2Ba
马特奥斯等。短营养期和开花位点C的组合活动确定了拟南芥不同的开花调控方式。基因组医学杂志。2015;16:31。gydF4y2Ba
- 11.gydF4y2Ba
De Folter S,Angenent GC。在Mads Science中遇见CIS。趋势植物SCI。2006; 11(5):224-31。gydF4y2Ba
- 12.gydF4y2Ba
严伟,陈德华,陈德华。MADS结构域蛋白调控花器官发育的分子机制。植物学报。2016;29:154-62。gydF4y2Ba
- 13。gydF4y2Ba
O'Maoileidigh DS, west SE, Rae L, Raganelli A, Ryan PT, Kwasniewska K, et al.;C功能调节剂AGAMOUS对拟南芥生殖花器官识别规范的控制植物细胞。2013;25(7):2482 - 503。gydF4y2Ba
- 14。gydF4y2Ba
等。无瓣花起始的配器1。科学。2010;328(5974):85 - 9。gydF4y2Ba
- 15.gydF4y2Ba
Pajoro A,Madrigal P,Muino JM,Matus JT,Jin J,Mecchia Ma,等。花卉发育中浊域转录因子的染色质可用性和基因调控的动态。基因组Biol。2014; 15(3):R41。gydF4y2Ba
- 16.gydF4y2Ba
west SE, O’maoileidigh DS, Rae L, Kwasniewska K, Raganelli A, hanzaryk K, et al.;aptala3和PISTILLATA鉴定花器官的分子基础。中国科学院院刊2012;109(33):13452-7。gydF4y2Ba
- 17.gydF4y2Ba
Kaufmann K, Muino JM, Jauregui R, Airoldi CA, Smaczniak C, Krajewski P, et al.;MADS转录因子SEPALLATA3的靶基因:拟南芥花发育和激素途径的整合。公共科学图书馆杂志。2009;7 (4):e1000090。gydF4y2Ba
- 18.gydF4y2Ba
Muino JM, Hoogstraat M, van Ham RC, van Dijk AD。PRI-CAT:一个用于分析、存储和可视化工厂ChIP-seq实验的网络工具。核酸学报2011;39(Web服务器版):W524-7。gydF4y2Ba
- 19.gydF4y2Ba
Muino JM,Kaufmann K,Van Ham RC,Angenent GC,Krajewski P.芯片-SEQ分析R(CSAR):蛋白质结合基因组区域的统计检测的R包。植物方法。2011; 7:11。gydF4y2Ba
- 20.gydF4y2Ba
MEME-ChIP:大型DNA数据集的motif分析。生物信息学。2011;27(12):1696 - 7。gydF4y2Ba
- 21.gydF4y2Ba
通过期望最大化拟合混合模型发现生物聚合物中的基序。Proc Int Conf Intell Syst Mol Biol. 1994; 2:28-36。gydF4y2Ba
- 22。gydF4y2Ba
Grant CE, Bailey TL, Noble WS。FIMO:扫描给定主题的出现。生物信息学。2011;27(7):1017 - 8。gydF4y2Ba
- 23。gydF4y2Ba
从ChIP-seq推断DNA直接结合。核酸学报2012;40(17):e128。gydF4y2Ba
- 24。gydF4y2Ba
GE, Hon G, Chandonia JM, Brenner SE。WebLogo:一个序列logo生成器。基因组研究》2004;14(6):1188 - 90。gydF4y2Ba
- 25。gydF4y2Ba
于长平,林俊杰,李文华。拟南芥中转录因子结合位点的位置分布。Sci众议员2016;6:25164。gydF4y2Ba
- 26。gydF4y2Ba
曹Ĵ,SCHNEEBERGER K,Ossowski S,冈瑟T,弯机S,菲茨J,等。多个拟南芥种群的全基因组测序。NAT Genet。2011; 43(10):956-63。gydF4y2Ba
- 27.gydF4y2Ba
Schmitz RJ,Schultz Md,Urich Ma,Nery Jr,Pelizzola M,Libiger O等人。人口表观簇多样性的模式。自然。2013; 495(7440):193-8。gydF4y2Ba
- 28.gydF4y2Ba
Ossowski S, Schneeberger K, Lucas-Lledo JI, Warthmann N, Clark RM, Shaw RG,等。拟南芥自发突变的速率和分子谱。科学。2010;327(5961):92 - 4。gydF4y2Ba
- 29.gydF4y2Ba
TCP基因:十年后的家庭快照。植物学报,2010;15(1):31-9。gydF4y2Ba
- 30.gydF4y2Ba
罗斯顿PJ,Somssich IE,Ringler P,Shen QJ。腕骨转录因子。趋势植物SCI。2010; 15(5):247-58。gydF4y2Ba
- 31。gydF4y2Ba
拟南芥SEPALLATA蛋白在花类四元复合物形成过程中协同dna结合的差异。核酸学报2014;42(17):10927-42。gydF4y2Ba
- 32.gydF4y2Ba
Mathelier A,Xin B,Chiu TP,Yang L,RoHS R,Wasserman WW。DNA形状特征改善了体内转录因子结合位点预测。细胞系统。2016; 3:278-86。gydF4y2Ba
- 33.gydF4y2Ba
Muino JM,Smaczniak C,Angenent GC,Kaufmann K,Van Dijk广告。植物浊域转录因子的DNA识别结构决定因素。核酸RES。2014; 42(4):2138-46。gydF4y2Ba
- 34。gydF4y2Ba
Stefl R,Wu H,Ravindranathan S,Sklenar V,Feigon J. DNA A-Tract三维弯曲:求解DA4T4与DT4A4难题。Proc Natl Acad SCI U S A. 2004; 101(5):1177-82。gydF4y2Ba
- 35。gydF4y2Ba
Jolma A,Yin Y,Nitta Kr,Dave K,Popov A,Taipale M等。转录因子对的DNA依赖性形成改变其结合特异性。自然。2015; 527(7578):384-8。gydF4y2Ba
- 36。gydF4y2Ba
杨志强,王志强,王志强,等。拟南芥bZIP转录因子的克隆及表达分析。植物科学进展,2002;7(3):106-11。gydF4y2Ba
- 37。gydF4y2Ba
拟南芥碱性/螺旋-环-螺旋转录因子家族。植物细胞。2003;15(8):1749 - 70。gydF4y2Ba
- 38。gydF4y2Ba
Franco-Zorrilla JM,Lopez-Vidriero I,Carrasco JL,Godoy M,Vera P,Solano R.植物转录因子的DNA结合特异性及其定义靶基因的潜力。Proc Natl Acad Sci U S A. 2014; 111(6):2367-72。gydF4y2Ba
- 39.gydF4y2Ba
范迪克AD,伊明克RG,安捷安特GC。跨家族转录因子的相互作用:基因调控的另一层。植物学报,2017;22(1):66-80。gydF4y2Ba
- 40。gydF4y2Ba
Heyndrickx Ks,Van de Velde J,Wang C,Weigel D,Vandepoele K.在拟南芥中转录因子结合的一种功能和进化视角。植物细胞。2014; 26(10):3894-910。gydF4y2Ba
- 41.gydF4y2Ba
Dornelas MC, Patreze CM, Angenent GC, Immink RG。身份认同与成长之间缺失的环节?植物科学进展。2011;16(2):89-97。gydF4y2Ba
- 42。gydF4y2Ba
郭某,徐y,刘H,毛泽东,张c,ma y等。OSMADS57与OSTB1之间的相互作用通过DWARF14调制米粉分蘖。NAT Communce。2013; 4:1566。gydF4y2Ba
- 43。gydF4y2Ba
WRKY转录因子WRKY12和WRKY13对拟南芥短日照开花的调控作用。摩尔。2016;9(11):1492 - 503。gydF4y2Ba
- 44.gydF4y2Ba
罗旭,孙旭,刘波,朱东,白旭,蔡辉,等。大豆WRKY同源基因异位表达改变拟南芥开花时间。《公共科学图书馆•综合》。2013;8 (8):e73295。gydF4y2Ba
- 45.gydF4y2Ba
Cai Y,Chen X,Xie K,Xing Q,Wu Y,Li J等。DLF1,一种衣衫转录因子,参与水稻开花时间和植物高度的控制。Plos一个。2014; 9(7):E102529。gydF4y2Ba
- 46。gydF4y2Ba
于勇,刘铮,王磊,金圣成,徐pj,乔敏,等。WRKY71通过直接激活拟南芥开花位点T和叶片加速开花。植物j . 2016; 85(1): 96 - 106。gydF4y2Ba
- 47。gydF4y2Ba
Yamamoto YY, Yoshitsugu T, Sakurai T, Seki M, Shinozaki K, Obokata J.高密度TSS分析揭示拟南芥核心启动子的异质性。植物j . 2009; 60(2): 350 - 62。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
在这篇文章中分析主要数据集是可用的(gydF4y2Bahttps://www.ncbi.nlm.nih.gov/geo/gydF4y2Ba),加入号码GSE45938,GSE46986,GSE38358,GSE54881和GSE45846。支持结论的所有其他数据集都包含在文章(及其附加文件)中。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
ADJvD和GCA监督该项目;NA和ADJvD进行分析;SdB和HvM参与解释结果;NA撰写手稿,由GCA和ADJvD编辑;所有作者编辑并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
伦理宣言gydF4y2Ba
伦理批准和同意参与gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
两位作者宣称他们没有相互竞争的利益。gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。gydF4y2Ba
附加文件gydF4y2Ba
额外的文件1:gydF4y2Ba
表S1。gydF4y2Ba峰中心的百分比至少有一半的峰中心落在启动子内。(PDF 45 KB)gydF4y2Ba
额外的文件2:gydF4y2Ba
表S2。gydF4y2Ba原始分析中的峰值数和再分析后。(PDF 45 KB)gydF4y2Ba
附加文件3:gydF4y2Ba
图S1。gydF4y2Ba聚类原始峰集和重新分析后的峰集。使用两个峰集之间的距离度量一个峰集和另一个峰集之间的平均距离来进行具有完全链接的层次聚类(函数hclust在R中)。重新分析后的峰集以转录因子的名称表示,原始峰集以后缀“orig”表示。(PDF 38 kb)gydF4y2Ba
附加文件4:gydF4y2Ba
表S3。gydF4y2Ba母题出现的峰值百分比。对于CArG-box motif,除了在所有峰中观察到的百分比,在前500个峰中观察到的百分比也被列出(括号之间)。(PDF 55 KB)gydF4y2Ba
附加文件5:gydF4y2Ba
表S4。gydF4y2Ba每个数据集中的唯一和重叠的碎片箱。套件箱被定义为八个蛋白中的每一个发现的所有非重叠叉箱(见图)(见图。gydF4y2Ba1gydF4y2Ba相应的主题)。注意,在所有数据集中出现的CArG-boxes的数量对于每个数据集是不一样的。这是由于通过从头motif搜索得到的CArG-box定义在数据集之间是不同的。(PDF 50 kb)gydF4y2Ba
额外的文件6:gydF4y2Ba
表S5。gydF4y2Ba每个数据集中的唯一和重叠的高峰中心。峰值中心定义为峰顶和下游的区域250bp。(PDF 52 KB)gydF4y2Ba
附加文件7:gydF4y2Ba
图S2。gydF4y2Ba在PI, SEP3和SOC1数据集的独特峰值中CArG-box类似的图案。标志代表所有符合MEME发现的主题。标识来自(A) PI (B) SEP3 (C) SOC1。(PDF 126 kb)gydF4y2Ba
额外的文件8:gydF4y2Ba
图S3。gydF4y2Ba在不同的蛋白质数据集集中富集不同的CArG-box变体。核密度图与母题在峰中心相对于峰顶。图来自(A) AG (B) AP1 (C) AP3 (D) FLC (E) PI (F) SEP3 (G) SOC1 (H) SVP。(PDF 251 kb)gydF4y2Ba
额外的文件9:gydF4y2Ba
表S6。gydF4y2BaChIP-seq峰中几个CArG-box变体的共现。(A)预先定义的CArG-box变体单次出现和多次出现的峰值数量。(B)只含CC的峰量(A/T)gydF4y2Ba6gydF4y2Bagg,只有cc(a / t)gydF4y2Ba7gydF4y2BaG或两者都有。(C)只含CC的峰量(A/T)gydF4y2Ba7gydF4y2BaG、C (A / T)gydF4y2Ba8gydF4y2BaG或两者都有。(D)只含CC的峰值量(A/T)gydF4y2Ba6gydF4y2BaGG,只有C (A / T)gydF4y2Ba8gydF4y2BaG或两者都有。B、C、D的期望值是通过与每个motif的峰频率相乘,再与数据集的峰总数相乘得到的。(PDF 71 kb)gydF4y2Ba
附加文件10:gydF4y2Ba
图S4。gydF4y2BaGA / CT丰富的主题。MEME发现的主题的标志表示所有匹配。数据集来自(a)ag(b)ap1(c)ap3(d)pi(e)sep3(f)soc1。(PDF 122 KB)gydF4y2Ba
附加文件11:gydF4y2Ba
图S5。gydF4y2Bag-box等图案。MEME发现的主题的标志表示所有匹配。数据集来自(a)ag(b)ap3(c)pi(d)sep3(e)sep3(e)svp。(PDF 81 KB)gydF4y2Ba
额外的文件12:gydF4y2Ba
图S6。gydF4y2BaTCP样图案。MEME发现的主题的标志表示所有匹配。数据集来自(a)ap1(类似于TCP II型图案的图案);(b)SEP3(类似于TCP I型MOTIF的基序);(c)SEP3(类似于TCP II型图案的基序)和(D)SOC1(类似于TCP II型图案的图案)。(PDF 65 KB)gydF4y2Ba
附加文件13:gydF4y2Ba
图S7。gydF4y2BaG-Boxes和TCP类I和II的中央丰富。在(a)Ag中,在(a)AG中的峰值与G-Box I(GGNCCCCAC)和II类(GGGNCC(A / G)C)和II类(GGGNCC(GGGNCC(A / G)C)中的峰值中心的核心密度绘制相对于G-Box(CaCGTG)的峰值峰值和II类(GGGNCC(A / G)C)的位置;(b)ap1;(c)AP3;(d)FLC;(e)pi;(f)SEP3;(g)SOC1和(H)SVP。(PDF 232 KB)gydF4y2Ba
额外的文件14:gydF4y2Ba
图S8。gydF4y2Ba热图的出现个别完美的CArG箱的扩展。对于每个MADS TF,颜色表示特定子序列出现的百分比(每行)。红色,零发生;yello越多gydF4y2BawgydF4y2Ba/白色,百分比越高。(PDF 70 KB)gydF4y2Ba
额外的文件15:gydF4y2Ba
表S7gydF4y2Ba.在CArG-box的5 '侧和3 '侧最常见的5个扩展,以及5 '和3 '扩展的前5个组合。CArG-boxes是由MEME-ChIP重新定义的,如材料和方法部分所述。对于每个类似CArG-box的序列,通过将序列与规范CArG-box (CC(A/T))进行比较来定义位置1和10。gydF4y2Ba6gydF4y2BaGG.位置1被定义为对应规范CArG-box中第一个C的位置,位置10被定义为对应规范CArG-box中最后一个G的位置。采用MEME-ChIP定义的链来区分5 '侧和3 '侧。随后,统计每个CArG-box在1号位置5 '侧和10号位置3 '侧的三个核苷酸。(A) AG, (B) AP1, (C) AP3, (D) FLC, (E) PI, (F) SEP3, (G) SOC1和(H) SVP。(PDF 95 kb)gydF4y2Ba
额外的文件16:gydF4y2Ba
表S8。gydF4y2Ba有和没有突变的叉箱数量。请注意,此表中的碎片箱都与AG的主题匹配,因为这些碳盒用于所有突变分析。因此,套件箱的总数与附加文件中的总数不同gydF4y2Ba5gydF4y2Ba:表S4。(PDF 47 kb)gydF4y2Ba
额外的文件17:gydF4y2Ba
图S9。gydF4y2Ba在ChIP-seq峰之间的CArG-boxes的保存gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba生态型。(a)对于每次出现碳水盒箱中的每个位置,颜色表示熵作为保护在生态型之间该位置的衡量标准。图例表示用于熵值的对数标度,在0.0(完美的保护)和最大观察到的值0.4之间。套件盒出现被命令,使得具有类似熵值的人在一起。在底部〜三分之一的底部观察到的白色块表示完全保守的碎片箱出现。(b)主题位置熵与生态型中的突变指数之间的关系。每个点代表包括3个核苷酸延伸的碳盒的一个位置。使用Col-0的主题徽标底层的所有碎片箱获得了图案位置的熵。位置10分开绘制,因为它是一个主要的异常值;这是通过在完美的叉箱中获得这种位置的更强大的保护来解释。 (PDF 55 kb)
权利和权限gydF4y2Ba
开放访问gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条提供的数据,除非另有说明。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
阿尔茨,N,德·布鲁因,S,范·莫里克,H。gydF4y2Baet al。gydF4y2Ba在结合MADS结构域蛋白的图案的比较分析gydF4y2Ba拟南芥蒂利亚纳gydF4y2Ba.gydF4y2BaBMC植物杂志gydF4y2Ba18,gydF4y2Ba131(2018)。https://doi.org/10.1186/s12870-018-1348-8gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
迪伊gydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-018-1348-8gydF4y2Ba
关键字gydF4y2Ba
- 疯狂域蛋白gydF4y2Ba
- CArG-boxgydF4y2Ba
- 芯片SEQ.gydF4y2Ba
- 转录因子结合特异性gydF4y2Ba
- 序列保护gydF4y2Ba