- 研究文章GyD.F4.y2Ba
- 开放获取GyD.F4.y2Ba
- 发布:GyD.F4.y2Ba
与分化群体中生物能量相关的切换曲率性的基因组预测精度GyD.F4.y2Ba
BMC植物生物学GyD.F4.y2Ba体积GyD.F4.y2Ba18GyD.F4.y2Ba文章编号:GyD.F4.y2Ba142GyD.F4.y2Ba(GyD.F4.y2Ba2018年GyD.F4.y2Ba)GyD.F4.y2Ba
抽象的GyD.F4.y2Ba
背景GyD.F4.y2Ba
柳枝稷育种者需要提高许多与生物能相关性状的遗传增益率,以创造产量更高、生物量组成最优的改良品种。实现这一目标的一种方法是通过基因组选择。然而,为了确定是否有可能进行有效的选择,除了预测的准确性外,还需要确定性状的遗传力。GyD.F4.y2Ba
结果GyD.F4.y2Ba
使用三个低地组成的五个不同的交叉口群体,一个普通和一个混合加入,在不同交叉验证策略和预测方法下进行了基因组预测的准确性。使用GBS收集单个基因型,而在Kin-Blup,局部最小二乘,稀疏的部分最小二乘和贝贝布方法中,用于预测2012 - 2013年收集的产量,形态学和基于NIRS的组成数据,从复制的内布拉斯加州现场试验中试验。通过批准人口结构GyD.F4.y2BaFGyD.F4.y2Ba统计数据在低地和高地院校之间的0.3952范围为0.0131,在低地加入中。预测精度分别为细胞壁可溶性葡萄糖和果糖的0.57-0.52分别为具有低重复性的性状的微不足道。遗传学比率在群体内部范围为15至0.6。GyD.F4.y2Ba
结论GyD.F4.y2Ba
结果表明,交叉验证策略和性状对准确性均有显著影响。使用受加入约束的交叉验证策略来考虑种群结构,其准确率比使用无约束交叉验证的表观准确率低69%。当大多数表型变异存在于种群之间(如春季返青和产量表型)时,预计基因组选择的准确性会降低。GyD.F4.y2Ba
背景GyD.F4.y2Ba
在没有表型数据的情况下,基于大量遗传标记的大量遗传标记准确地预测作物和牲畜的性能和育种的能力一直是激烈的研究努力的重点,现在正在应用于高级育种计划[GyD.F4.y2Ba18GyD.F4.y2Ba那GyD.F4.y2Ba35GyD.F4.y2Ba那GyD.F4.y2Ba56GyD.F4.y2Ba].这种被称为基因组选择(GS)的方法在理论上对像柳枝稷这样需要2-3个季节才能确定其全部产量潜力的多年生禾草很有用。在奶牛中,代距已从4 - 7年减少到大约2.5年,根据性状的遗传力,每年的遗传增益率可从~ 50-400%提高[GyD.F4.y2Ba18GyD.F4.y2Ba].在玉米和小麦等作物中,预测显示,GS精度低至0.2,就能比标记辅助选择育种方案获得更大的年度遗传收益[GyD.F4.y2Ba24GyD.F4.y2Ba].在多年生植物中,每单位收益的年遗传收益和成本也预计较高,GS [GyD.F4.y2Ba20.GyD.F4.y2Ba那GyD.F4.y2Ba48GyD.F4.y2Ba那GyD.F4.y2Ba64GyD.F4.y2Ba].这可能是switchgrass(GyD.F4.y2Ba柳枝稷GyD.F4.y2Ba)如果育种值(GEBV)的基因组估计足够准确,并且可以缩短生成间隔。GyD.F4.y2Ba
Switchgrass是美国的有吸引力的多年生生物量作物,由于其有利的净能源,以及几个环境和经济优势[GyD.F4.y2Ba51GyD.F4.y2Ba].柳枝稷是开放授粉的,高度自交不亲和,具有丰富的遗传多样性[GyD.F4.y2Ba53GyD.F4.y2Ba].品种之间的遗传变异可以为生物量的3-5倍[GyD.F4.y2Ba54GyD.F4.y2Ba],Lignin含量的10%[GyD.F4.y2Ba4.GyD.F4.y2Ba]和85%的水使用效率[GyD.F4.y2Ba28GyD.F4.y2Ba].有些柳枝稷的高度可达3米以上,根系旺盛,深度可达3.5米以上[GyD.F4.y2Ba41GyD.F4.y2Ba].许多性状的表型变异足以进行遗传分析和育种。育种计划主要集中在从野生柳枝稷群体中循环选择合成品种,针对诸如(1)高生物量产量、(2)改善育苗和(3)提高原料质量等性状[GyD.F4.y2Ba3.GyD.F4.y2Ba].在美国上东南部,Switchgrass生物量产量平均超过14毫克HAGyD.F4.y2Ba−1GyD.F4.y2Ba[GyD.F4.y2Ba17GyD.F4.y2Ba],在某些地方超过25 mg haGyD.F4.y2Ba−1GyD.F4.y2Ba,表明通过繁殖进一步改善,提高产量潜力。GyD.F4.y2Ba
通过对选择方案的类型和在牧草中实施遗传选择的相对优势的分析,发现在某些情况下,遗传选择可能优于表型选择。对于像柳枝稷这样的异源物种来说,GS优势可导致其数量增加2.6倍[GyD.F4.y2Ba52GyD.F4.y2Ba].GyD.F4.y2Ba
迄今为止,柳枝稷的基因组预测已经在适于北方的旱地和低地种质的面板中进行了尝试,并在两个半同胞家系的群体中进行了尝试,一个是旱地的,另一个来自Liberty品种[GyD.F4.y2Ba5.GyD.F4.y2Ba那GyD.F4.y2Ba31GyD.F4.y2Ba那GyD.F4.y2Ba47GyD.F4.y2Ba].这些报告采用各种不同的建模和基因分型方法,代表不同的人口结构和独特的环境。报告的准确度从超过0.5到负范围,这取决于校准集(CS)和验证集(VS)的特征,环境和组成。GyD.F4.y2Ba
CS的组成已被证明对提高GS的准确性至关重要,特别是在存在人口结构的情况下[GyD.F4.y2Ba26GyD.F4.y2Ba].人口结构可以在基因组关联研究中产生虚假关联[GyD.F4.y2Ba45GyD.F4.y2Ba那GyD.F4.y2Ba66GyD.F4.y2Ba].它可以向向上偏见引入遗传性估算[GyD.F4.y2Ba58GyD.F4.y2Ba,并可能在基因组预测中引入偏差[GyD.F4.y2Ba32GyD.F4.y2Ba那GyD.F4.y2Ba49GyD.F4.y2Ba那GyD.F4.y2Ba65GyD.F4.y2Ba].在CS中捕获尽可能多的表型变化是理想的[GyD.F4.y2Ba26GyD.F4.y2Ba].然而,当评估CS的性能时,应该考虑到显著的群体结构,随着CS和VS之间的关系变得较弱,基因组预测的准确性下降[GyD.F4.y2Ba23GyD.F4.y2Ba].GyD.F4.y2Ba
在这项研究中,人口结构的基因组遗传的估计和对基因组预测的影响,利用与生物能源的柳枝稷两种成分和形态特征进行了评估。遗传试验是与未知的血统关系的空间种植人群中进行。这些人口从两个高地和低地生态型衍生的,因此代表高多样性。基因组预测四种不同的方法进行测试,其性能相比。遗传分配到可能有预测精度的具体贡献的对面,可人口成分。GyD.F4.y2Ba
方法GyD.F4.y2Ba
植物种群GyD.F4.y2Ba
实验温室生长的苗于2009年在该领域在内布拉斯加州东部研究与推广中心(ENREC)米德,NE附近建立。一百25苗5行25个苗木种植为每5个群体对1.1米中心。人群被随机分配到地块和基因型范围内的人口被随机分配到各行和位置的地块内。没有化肥在建立一年被应用,而在建立后几年112千克公顷GyD.F4.y2Ba−1GyD.F4.y2Ba在春季以nhh的形式施氮GyD.F4.y2Ba4.GyD.F4.y2Ba不GyD.F4.y2Ba3.GyD.F4.y2Ba.在建立和建立后几年除草剂和手绘用于控制年度杂草。阿特拉津(6-氯 -GyD.F4.y2BaNGyD.F4.y2Ba乙基-GyD.F4.y2BaNGyD.F4.y2Ba- 丙基-1,3,5, - 三嗪-2,4-二胺),2,4-D(2,4-二氯酸乙酸)和S-氯化甲酸(2-氯)GyD.F4.y2BaNGyD.F4.y2Ba(2-ethyl-6-methylphenyl)GyD.F4.y2BaNGyD.F4.y2Ba- (2-甲氧基-1-甲基)乙酰胺]以1.96,1.48和1.26kg HA的速率施用GyD.F4.y2Ba−1GyD.F4.y2Ba分别在春季或初夏次。2011年,两个振动从原始地块中的每种基因型挖出,并用于每个人口建立两个额外的随机地块,保持群体单独的块。在2011年4月和2012年4月再次出现春天再生之前,剩余地块茬被烧毁。GyD.F4.y2Ba
本研究中使用的五种种群由NRCS植物物质中心获得,或者从Mead附近的米德,NE附近的隔离的多边形块收获,从其他开关场保持至少500米。所有群体都被认为是基于育种史的四倍体。填充群体的摘要GyD.F4.y2Ba1GyD.F4.y2Ba.Kanlow (K)是一个高产的释放型四倍体低产品种。Kanlow N1 (N1)先前已被描述[GyD.F4.y2Ba59GyD.F4.y2Ba]代表在内布拉斯加州没有冬季杀死4年的人口。夏季是一种四倍体,高地品种适应更北部纬度,冬季生存良好。用于建立我们夏季人口的种子批次代表了一个由1000名具有高产和良好活力组成的选择托儿所的批量收获。Kanlow N1-EM(N1EM)人口是选自Kanlow N1基底的早期开花植物。Kanlow X夏季F2(KXS)人口代表了两代人口从最初的随机交互的父母种群中移除,其中Kanlow被用作雄性父母。GyD.F4.y2Ba
表型分析GyD.F4.y2Ba
春季出现GyD.F4.y2Ba
2013年春出(GRN)被确定为绿芽从冬季休眠中明显出现的那一天(DOY)。GyD.F4.y2Ba
收益率GyD.F4.y2Ba
在2012年11月2日和7日期间的粉碎后,所有地块都是在单一克隆的基础上使用卡特收割机(卡特制造有限公司,Brookston,In)在10厘米的切割高度。确定单个克隆的新鲜重量,并采取归档以确定百分比水分并提供干重的估计。报告产量(YLD)作为干重校正了子样本的重量。GyD.F4.y2Ba
开花GyD.F4.y2Ba
花期(ANT)确定为每个无性系50%的成熟分蘖达到花药出苗/开花R4期的天数[GyD.F4.y2Ba39GyD.F4.y2Ba].GyD.F4.y2Ba
近红外光谱数据GyD.F4.y2Ba
将收获的样品在60℃的强制空气烘箱中干燥。然后将这些样品研磨在Wiley磨机中以通过2mm试验筛,后来在旋风磨机中以通过1mm试验筛。使用6500型近红外光谱仪(FOSS NIRSYSTEMS,INC.,LAUREL,MD)扫描研磨地样品。在宽的近红外光谱(NIRS)校准中获得超过20种生物质组分的估计,包括简单糖和乙醇的理论产率。可以在表中找到特征的描述GyD.F4.y2Ba2GyD.F4.y2Ba并更详细地在[GyD.F4.y2Ba61GyD.F4.y2Ba].GyD.F4.y2Ba
表型数据分析GyD.F4.y2Ba
最佳线性无偏预测(BLUP)值的估计跨重复每个性状,基因型方差(GyD.F4.y2BaσGyD.F4.y2BaGGyD.F4.y2Ba2GyD.F4.y2Ba),误差方差(GyD.F4.y2BaσGyD.F4.y2BaE.GyD.F4.y2Ba2GyD.F4.y2Ba)和重复性(GyD.F4.y2BaHGyD.F4.y2Ba2GyD.F4.y2Ba)采用“lme4”软件包中的限制最大似然法[GyD.F4.y2Ba2GyD.F4.y2Ba]在r(r核心团队)。估计使用Blup模型获得的方差分量估计了广泛的遗传性,相当于基于线的基于线的重复性:GyD.F4.y2Ba
在哪里GyD.F4.y2BaN.GyD.F4.y2Ba指的是平均每基因型重复的。数据为中心,由标准偏差分析,以允许的方法和性状和一些异常值之间简单的比较被拆除之前缩放。GyD.F4.y2Ba
基因分型GyD.F4.y2Ba
从建立复制前的原始小区采集叶片组织样本,采用CTAB法提取基因组DNA [GyD.F4.y2Ba6.GyD.F4.y2Ba].基因分型的分测序文库的生产,接着先前建立的协议[GyD.F4.y2Ba13GyD.F4.y2Ba]有一些修改。简而言之,GDNA被消化了GyD.F4.y2Ba太平洋标准时间GyD.F4.y2Ba我并连接到Illumina的适配器与一组自定义序列索引的条形码标签。条形码DNA随后适于与Illumina测序引物和用Qiagen PCR净化试剂盒纯化。DNA文库汇集(每流动池土地96个样品)和单端读段上的Illumina公司HiSeq 2000在文森特J.科茨基因组测序实验室,美国加州大学伯克利分校的生成。GyD.F4.y2Ba
测序产生1.25×10GyD.F4.y2Ba9.GyD.F4.y2Ba100-BP单端读取到TASSEL-GBSV2管道[GyD.F4.y2Ba19GyD.F4.y2Ba],共生成8.1亿个64-bp序列标签,其中7.21亿个可以与柳枝稷基因组装配4.0版本的主标签数据库相匹配(GyD.F4.y2Bahttps://www.phytozome.jgi.doe.govGyD.F4.y2Ba)使用BWA [GyD.F4.y2Ba30.GyD.F4.y2Ba].用于对准,只使用18组主要的染色体。预测和单核苷酸多态性的注释使用SNPEff [进行(SNP)的影响GyD.F4.y2Ba9.GyD.F4.y2Ba].横跨各个分类群标签数为13777至5104802,平均为1229285。TASSEL总共70215个SNP位点检测到与本潜在基因型的呼叫的46%。GyD.F4.y2Ba
然后进行SNP滤波。首先,将深度为1的基因型被设定为缺失,其消除了2.33%的单个SNP呼叫。其次,如果它们包含缺失的SNP位置的信息,则筛选出缺失的信息。这导致了104个个体的损失(16%的人口)。第三,消除了较小等位基因频率和较少于50%的基因型频率的次要等位基因频率的遗址。第四,修剪完整LD的网站被修剪。在施加这些过滤器后,总共19,342个SNP留在483个个体上,具有71%的潜在基因型呼叫。GyD.F4.y2Ba
遗传分化和基因组遗传力估计GyD.F4.y2Ba
为了评估基因组选择方法,并且需要使用LD-KNNI算法进行缺失基因型的遗传性归力。GyD.F4.y2BaL.GyD.F4.y2BaLD中最多的网站的网站用于确定抵御时要使用的最接近的邻居和加权[GyD.F4.y2Ba38GyD.F4.y2Ba].在Imprue DataSet中的参数中GyD.F4.y2BaK.GyD.F4.y2Ba = 13,L.GyD.F4.y2Ba = 8 were applied. Accuracy was determined by masking and imputing 10,000 known genotypes and was estimated at 88.5%.
在避税数据集上测量人口分化GyD.F4.y2BaFGyD.F4.y2Ba英石GyD.F4.y2Ba统计使用堰和科克勒姆[方法GyD.F4.y2Ba62GyD.F4.y2Ba].利用基因型数据进行主成分分析的个体聚类,以可视化群体结构[GyD.F4.y2Ba33GyD.F4.y2Ba].GyD.F4.y2Ba
为了预测基因组育种值(GEBV)和估计群体内和群体间的基因组遗传力,加性遗传方差(GyD.F4.y2BaσGyD.F4.y2BaGGyD.F4.y2Ba2GyD.F4.y2Ba)残差方差(GyD.F4.y2BaσGyD.F4.y2BaE.GyD.F4.y2Ba2GyD.F4.y2Ba)来自应用Ridge回归混合建模亲属 - Blup使用了在R包RRBLUP 4.5中实现的受限制的最大可能性(REML)方法[GyD.F4.y2Ba14GyD.F4.y2Ba那GyD.F4.y2Ba46GyD.F4.y2Ba].计算实现的添加剂关系矩阵的估计计算为:GyD.F4.y2Ba
其中w是中心的(GyD.F4.y2Ban x m)GyD.F4.y2Ba基因型矩阵和GyD.F4.y2BaP.GyD.F4.y2Ba等位基因频率是[GyD.F4.y2Ba15GyD.F4.y2Ba].基本模型GyD.F4.y2Ba
被用在哪里GyD.F4.y2BaXGyD.F4.y2Ba是固定效果(β)的全级设计矩阵,GyD.F4.y2BaZ.GyD.F4.y2Ba是随机效果的设计矩阵GyD.F4.y2Ba你GyD.F4.y2Ba那GyD.F4.y2BaK.GyD.F4.y2Ba是加法关系矩阵吗GyD.F4.y2Ba一种,GyD.F4.y2Ba和GyD.F4.y2BaE.GyD.F4.y2Ba对于残差(e ~GyD.F4.y2BaNGyD.F4.y2Ba(0,GyD.F4.y2Ba一世GyD.F4.y2BaσGyD.F4.y2BaE.GyD.F4.y2Ba2GyD.F4.y2Ba)) 和GyD.F4.y2Ba一世GyD.F4.y2Ba是身份矩阵。GyD.F4.y2Ba
矩阵GyD.F4.y2BaK.GyD.F4.y2Ba由于混合物,遗传漂移和选择历史以及血缘关系,捕获人口结构。在这里,基于标记的遗传性估计和群体内的遗传性(GyD.F4.y2BaHGyD.F4.y2BaGGyD.F4.y2Ba2GyD.F4.y2Ba那GyD.F4.y2BaHGyD.F4.y2BaGA.GyD.F4.y2Ba2GyD.F4.y2Ba, HGyD.F4.y2BaGW.GyD.F4.y2Ba2GyD.F4.y2Ba),通过对上述基本模型的重新参数化,基于BLUP表型和遗传方差分量的正交分解得到。这是以类似于Guo等人的方式完成的[GyD.F4.y2Ba21GyD.F4.y2Ba]基于[GyD.F4.y2Ba12GyD.F4.y2Ba那GyD.F4.y2Ba27GyD.F4.y2Ba].在特征值后k作为GyD.F4.y2Ba
在哪里GyD.F4.y2Ba你GyD.F4.y2Ba是一个GyD.F4.y2BaN.GyD.F4.y2Bax (GyD.F4.y2BaN.GyD.F4.y2Ba-1)特征向量的矩阵GyD.F4.y2BaK.GyD.F4.y2Ba和GyD.F4.y2Ba你GyD.F4.y2Ba一世GyD.F4.y2Ba列I(GyD.F4.y2Ba一世GyD.F4.y2Ba = 1,2, …, n-1) representing the individual principal component loadings, andD.GyD.F4.y2Ba是一个(GyD.F4.y2BaN.GyD.F4.y2Ba-1)x(GyD.F4.y2BaN.GyD.F4.y2Ba的对角矩阵,每个对角元素表示特征值GyD.F4.y2BaK.GyD.F4.y2Ba,排列在减小值λGyD.F4.y2Ba1GyD.F4.y2Ba,λ.GyD.F4.y2Ba2GyD.F4.y2Ba。λ.GyD.F4.y2Ban - 1GyD.F4.y2Ba.然后将基本模型重写为GyD.F4.y2Ba
和GyD.F4.y2BaαGyD.F4.y2Ba成为(GyD.F4.y2BaN.GyD.F4.y2Ba-1)×1载体具有正态分布的随机遗传效果的载体GyD.F4.y2BaNGyD.F4.y2Ba(GyD.F4.y2Ba0.GyD.F4.y2Ba那GyD.F4.y2BaD.GyD.F4.y2BaσGyD.F4.y2BaGGyD.F4.y2Ba2GyD.F4.y2Ba)主要组件用作随机变量,该组件具有与基本模型相同的分布,但允许分离主导主组件解释的子潜水结构(GyD.F4.y2BaD.GyD.F4.y2Ba)进入群体的遗传方差,GyD.F4.y2Ba
种群内的遗传变异,GyD.F4.y2Ba
计算总遗传方差计算为GyD.F4.y2Ba
相应的基于标记的特性可遗传性获得为[GyD.F4.y2Ba27GyD.F4.y2Ba]:GyD.F4.y2Ba
基因组预测GyD.F4.y2Ba
采用四种方法对数据进行建模:(1)亲属- blup法(kin-BLUP);(2)偏最小二乘回归方法(PLS),(3)稀疏偏最小二乘方法(SPLS), (4) BayesB (BB)。GyD.F4.y2Ba
预测精度GyD.F4.y2BaR.GyD.F4.y2Ba(VS GEBV与其结合值的相关性)被评估了使用修改的5倍交叉验证(CV)的所有方法,其中VS由特定群体或群体组合构成,并分为5套,每个套件包括单一折叠.然后,校准集(CS)由所有人群的剩余个体组成。在整个数据集中使用5倍CV的所有特征的初始筛选所有特征(“全部”)使我们能够专注于具有更高预测精度的特征。通常,分析仅限于具有Kin-Blup方法的预测精度的22个特征,预测精度大于0.5。GyD.F4.y2Ba
除Bayesb外,每次分析使用5倍CV方法的20个复制来进行,以估计单个人口或人群组合的预测精度。受试者随机分为五套。对于每组,表型结合值(GyD.F4.y2BayGyD.F4.y2Ba)通过屏蔽和预测值依次用作验证集(VS)GyD.F4.y2Ba\ (\ widehat {y} \)GyD.F4.y2Ba基于与其它四组,并与从投放作为VS.受试者组外面包容其它基因型的估计模型参数在CS总是包含不包含在VS.所有个人GyD.F4.y2Ba
从平均表型相关性确定精度GyD.F4.y2Ba\(r \ left({\ widehat {y}} ^ 2,{y} ^ 2 \右)\)GyD.F4.y2Ba在真正的表型之间(GyD.F4.y2BayGyD.F4.y2Ba)和gebv.GyD.F4.y2Ba左(\ \ (\ widehat {y} \) \)GyD.F4.y2Ba在折中和复制。均方误差(MSE)从估计GyD.F4.y2Ba\(\ frac {1} {n} \ sum {\ left(y- \ widehat {y} \ revent)} ^ 2 \)GyD.F4.y2Ba在哪里GyD.F4.y2BaN.GyD.F4.y2Ba是每个折叠使用的个体vs基因型的数量。报告的MSE值在折叠和重复上取平均值。GyD.F4.y2Ba
偏最小二乘法GyD.F4.y2Ba
由Wold引入的部分最小二乘法(PL)[GyD.F4.y2Ba63GyD.F4.y2Ba]分解基因型数据GyD.F4.y2BaW.GyD.F4.y2Ba通过将预测变量和可观察变量投影到新的空间和回归来获得的正交分数T称为潜在变量和加载P.GyD.F4.y2BayGyD.F4.y2Ba而不是在GyD.F4.y2BaW.GyD.F4.y2Ba但第一GyD.F4.y2Ba一种GyD.F4.y2Ba得分列GyD.F4.y2BaT.GyD.F4.y2Ba.在迭代过程中分别获得潜在变量,然后作为线性回归的新变量使用。这允许解释预测变量之间的共线性,以及预测变量比观测值多的实例。PLS方法是通过R包' PLS '实现的[GyD.F4.y2Ba36GyD.F4.y2Ba].在该特定实例中,前15个潜在变量用于预测。GyD.F4.y2Ba
稀疏偏最小二乘法GyD.F4.y2Ba
稀疏偏最小二乘方法(SPLS) [GyD.F4.y2Ba29GyD.F4.y2Ba[就像PLS一样,允许在保持良好的预测能力的同时选择稀疏的解释变量和维度降低。在SPLS案例中,这是通过将约束施加到加载中包括的解释性变量上来完成的。通过阈值参数λ施加约束GyD.F4.y2Ba1.GyD.F4.y2Ba通过交叉验证方法获得该参数的值以及为每个特征使用的最佳变量的最佳数量。使用“SPLS”包来实现SPLS预测[GyD.F4.y2Ba7.GyD.F4.y2Ba那GyD.F4.y2Ba8.GyD.F4.y2Ba].GyD.F4.y2Ba
BayesB方法GyD.F4.y2Ba
该方法允许标记的一部分(π)具有很大的效果,而大多数标记没有效果。标记方差对于每个标记是唯一的。用蒙特卡罗马尔可夫陈模拟估计标记效应。贝贝布模型与采用GIBBS采样算法的R中的“BGLR”包运行[GyD.F4.y2Ba11GyD.F4.y2Ba那GyD.F4.y2Ba35GyD.F4.y2Ba].π的值为0.05,用于5000次迭代和1500次迭代烧坏周期,采样频率为5。GyD.F4.y2Ba
结果GyD.F4.y2Ba
阅读分布和SNP多样性GyD.F4.y2Ba
序列标签共与148,557个独特的基因组位点对齐,约为104 MbpGyD.F4.y2Ba−1GyD.F4.y2Ba,其中19,769(13%)与注释的基因区域重叠或与之交叉。这与作为基因区域注释的总切换基因组的15%相当,并且使用GBS表明很少确定的偏差。在这些序列中,过滤后鉴定了19,342个SNP。这些主要是双腿,总结在表中GyD.F4.y2Ba3.GyD.F4.y2Ba.在19,342,52.1%的转变中,44%是横向(TSTV = 1.183),0.8%是单核苷酸吲哚,3.6%是试验性,0.1%是四等位基因。平均而言,有14.84个SNP MBPGyD.F4.y2Ba−1GyD.F4.y2Ba以染色体的总数和染色体的注释长度之间检测到强烈的重要关系(GyD.F4.y2BaP.GyD.F4.y2Ba < 0.001, AdjustedR.GyD.F4.y2Ba2GyD.F4.y2Ba = 0.7791), and between SNP density and distance to the end of the chromosomes (P.GyD.F4.y2Ba < 0.001, AdjustedR.GyD.F4.y2Ba2GyD.F4.y2Ba= 0.7147)。编码序列共注释了5118个SNPs,其中65%的非参考等位基因是由误义或无义突变引起的,2216个snp位于基因区域的内含子中,786个snp位于3 ' UTR区域,670个snp位于5 ' UTR区域。GyD.F4.y2Ba
分化群体GyD.F4.y2Ba
表格中显示了全植物测量和NIR特征的人口手段和可重复性值GyD.F4.y2Ba2GyD.F4.y2Ba.MANOVA检验表明,种群起源对形态学和nir组成均有显著影响。群体平均产量在夏季干物质YLD 0.6 kg至坎洛1.47 kg之间。各居群开花时间和春出时间不同。Kanlow的开花期为夏季7月20日(202年)至9月2日(246年),春季返青期为夏季5月10日(130年)至5月17日(137年)。分别为皮尔逊积矩相关系数GyD.F4.y2BaR.GyD.F4.y2Ba在YLD和GRN之间为0.22GyD.F4.y2BaR.GyD.F4.y2Ba在YLD和Ant之间是0.43(附加文件GyD.F4.y2Ba1GyD.F4.y2Ba:图S1)。两组内的NIR估计的细胞壁属性中也存在显着的正相关(附加文件GyD.F4.y2Ba1GyD.F4.y2Ba:图S2)。这两组是:(1)GLC,KL,ADL,NDF,DM,FUC;(2)N,IVDMD,MAN,UA,RHA,ARA,GAL。这两组性质之间存在显着的负相关性。在这种情况下,这种可重复性表示广义遗传性的上限范围为0.94,对于醚化阿魏酸盐(FETH)。GyD.F4.y2Ba
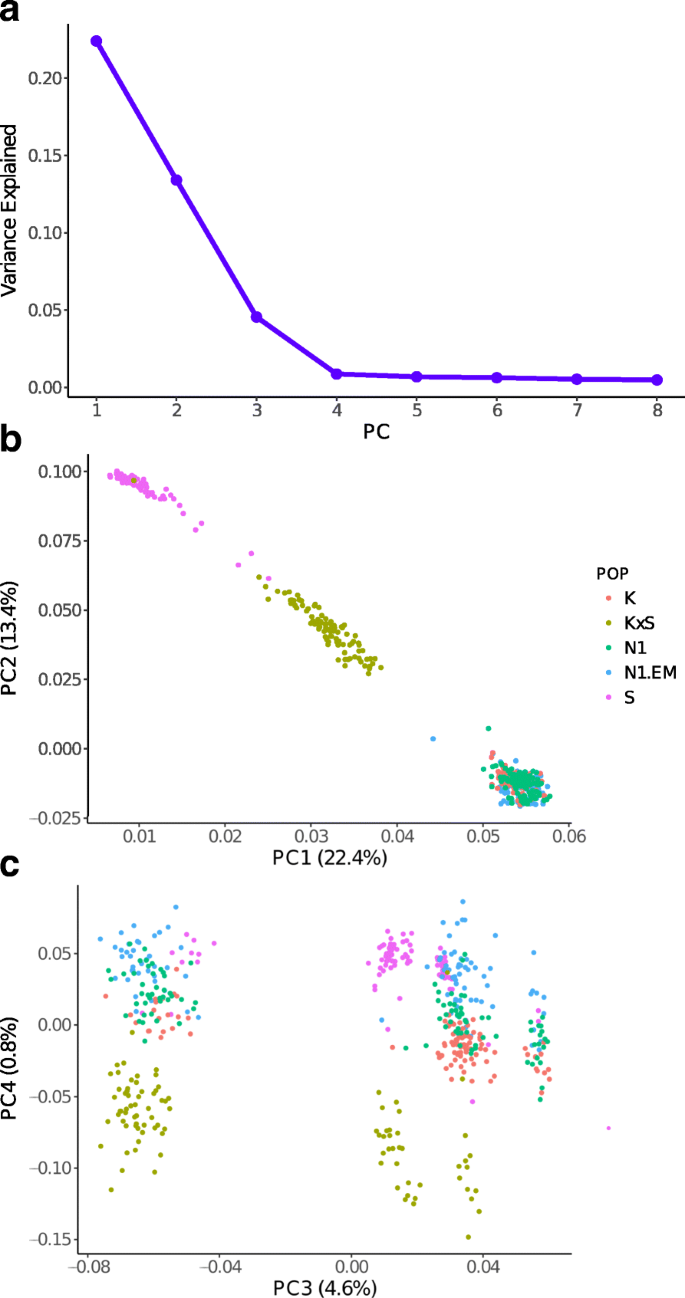
群体固定统计(GyD.F4.y2BaFGyD.F4.y2Ba英石GyD.F4.y2Ba)通过群体大小加权的所有SNP进行评估,并在表格中报告GyD.F4.y2Ba4.GyD.F4.y2Ba.可以通过人口结构解释的大多数遗传变异是由于K,N1和N1EM群体(作为群体)和S种群之间的差异(GyD.F4.y2BaFGyD.F4.y2Ba英石GyD.F4.y2Ba〜0.39-0.40)。K,N1和N1EM人群均密切相关(GyD.F4.y2BaFGyD.F4.y2Ba英石GyD.F4.y2Ba〜0.01-0.03)彼此。杂交的KXS人群与基于K和K的人群共享更多相似之处(GyD.F4.y2BaFGyD.F4.y2Ba英石GyD.F4.y2Ba〜0.13-0.16)比这些相互彼此。这表明低地衍生(K,N1,N1EM)和高地群体之间存在显着的遗传分化。GyD.F4.y2Ba
将K,N1和N1EM人群与群体的群体进行比较,没有固定差异(GyD.F4.y2BaD.GyD.F4.y2BaFGyD.F4.y2Ba发现,低地群体将纯合的一个等位基因和普满群体纯合。然而,3715个位点在任一组中固定在另一个组中并占据另一个。GyD.F4.y2Ba
在进行基因组预测之前,对无序标记使用k-最近邻估算法(LD-kNNi)估算缺失数据。这种方法使用来自基因型中连接最紧密的标记(即LD)的成对LD信息。归罪结果增加了3139,549个基因型,占数据集的28%。基于掩蔽和输入10,000个已知基因型,缺失数据的预测准确率估计为88.5%。GyD.F4.y2Ba
基于分子数据PCA的五个群体的人口结构推理表明,前三个主要成分占共同解释40.4%的4.6-22.4%(图。GyD.F4.y2Ba1GyD.F4.y2Ba)。群体可以通过第一PC完全解决(图。GyD.F4.y2Ba1b,cGyD.F4.y2Ba)被分为3组,分别代表低地种质(K、N1和N1EM)、高地种质(S)和位于其他两组之间的杂种种质(KxS)。这一结果与表明这三个群体之间显著差异的许多表型的组均值一致(见表)GyD.F4.y2Ba2GyD.F4.y2Ba和附加文件GyD.F4.y2Ba1GyD.F4.y2Ba:图S3)以及群体固定统计数据。以前的研究还报告了普满和低地群体之间的遗传分化[GyD.F4.y2Ba16GyD.F4.y2Ba那GyD.F4.y2Ba37GyD.F4.y2Ba那GyD.F4.y2Ba40GyD.F4.y2Ba].GyD.F4.y2Ba
前三个PC用于评估群体结构对基因组遗传率的影响(GyD.F4.y2BaHGyD.F4.y2BaGGyD.F4.y2Ba2GyD.F4.y2Ba),用于整个数据由遗传方差分量的正交分解设定。独立基因遗传率(GyD.F4.y2BaHGyD.F4.y2BaGGyD.F4.y2Ba2GyD.F4.y2Ba那GyD.F4.y2BaHGyD.F4.y2BaGA.GyD.F4.y2Ba2GyD.F4.y2Ba, 和GyD.F4.y2BaHGyD.F4.y2BaGW.GyD.F4.y2Ba2GyD.F4.y2Ba)所选性状列于表中GyD.F4.y2Ba5.GyD.F4.y2Ba并且差异估计是在附加文件中提供的GyD.F4.y2Ba1GyD.F4.y2Ba:表S5。估计GyD.F4.y2BaHGyD.F4.y2BaGGyD.F4.y2Ba2GyD.F4.y2Ba范围从ANT和SC的0.57到YLD的0.15。最高估计GyD.F4.y2BaHGyD.F4.y2BaGA.GyD.F4.y2Ba2GyD.F4.y2Ba蚂蚁为0.43,而Ash和YLD的最低时间为0.14。更重要的是基因组选择应用的准确性,GyD.F4.y2BaHGyD.F4.y2BaGW.GyD.F4.y2Ba2GyD.F4.y2Ba对于NSC,估计范围为0.01至0.31。这GyD.F4.y2BaHGyD.F4.y2BaGW.GyD.F4.y2Ba2GyD.F4.y2Ba对于灰烬占总数的62%GyD.F4.y2BaHGyD.F4.y2BaGW.GyD.F4.y2Ba2GyD.F4.y2Ba对于NSCE表示总数的60%。统称,诸如群体结构的酚类特征(蚂蚁和GN)和YLD受到群体结构的影响,而NIR组成性状受到可变程度的影响。这GyD.F4.y2BaHGyD.F4.y2BaGA.GyD.F4.y2Ba2GyD.F4.y2Ba近红外组成性状的遗传率为38 ~ 74%。GyD.F4.y2Ba
基因组的连锁不平衡模式GyD.F4.y2Ba
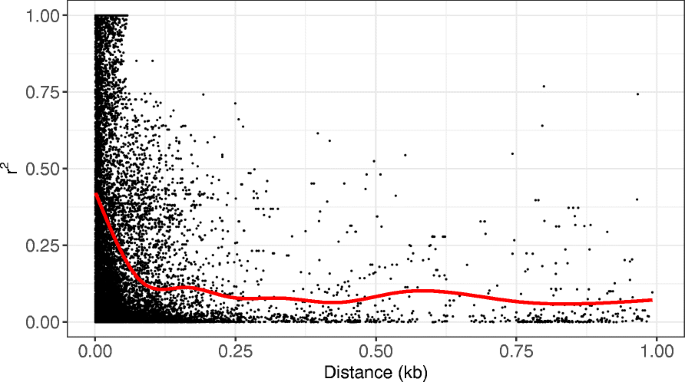
基因组选择的准确性取决于相关性状的QTL的标记不平衡程度。利用标记数据对潜在QTL位点的不平衡进行建模,并进行两两计算GyD.F4.y2BaR.GyD.F4.y2Ba2GyD.F4.y2Ba定位于同一染色体上的所有标记。然后按距离排序,0-1 kb距离区间的结果绘制在图中。GyD.F4.y2Ba2GyD.F4.y2Ba.的意思是GyD.F4.y2BaR.GyD.F4.y2Ba2GyD.F4.y2Ba在整个数据集的染色体中是0.178,并且似乎衰变相当迅速,在0.25 kb内下降到名义水平。GyD.F4.y2Ba
根据每个基因型所需的标准过滤数据,包括每个基因型所需的序列覆盖的深度,数据缺失或次要等位基因频率,具有标称效果GyD.F4.y2BaR.GyD.F4.y2Ba2GyD.F4.y2Ba作为SNP网站被过滤出来的价值并导致信息丢失。使用指数分布的非线性曲线拟合用于估计距离阈值GyD.F4.y2BaR.GyD.F4.y2Ba2GyD.F4.y2Ba = 0.2. Changing the sequence depth required to call a genotype from 2 to 8 resulted in an approximate 6% increase in the threshold distance. Changing the amount of missing data allowed from 25 to 50% resulted in a 19% increase in threshold distance, while filtering sites based on minimum minor allele frequency from 0.05 to 0.20 resulted in a 380% increase in threshold distance. However, these changes only resulted in a small overall increase in threshold to 76 bp. The threshold for the S population was 69 bp while that for the combined lowland accessions (Low) was 58 bp. The KxS population had a lower threshold than either the S or K populations.
因为GyD.F4.y2BaR.GyD.F4.y2Ba2GyD.F4.y2Ba众所周知,我们试图在单一染色体上看待趋势的基因组,但数据太稀疏,无法可靠地检测这些变化。总体而言,阈值范围从所有18个染色体上的33bp(chr7n)到82bp(chr3n)。阈值在n和k亚组之间没有显着差异。GyD.F4.y2Ba
基因组预测GyD.F4.y2Ba
预测精度结果列于表GyD.F4.y2Ba6.GyD.F4.y2Ba和附加文件GyD.F4.y2Ba1GyD.F4.y2Ba:表S1-S4。在所有情况下,由于人口结构,当在整个数据集(不受约束)上采样VS时,GEBV更准确。使用Kin-Blup(蚂蚁)为开花DOY(蚂蚁)获得了使用该CV方法的最高精度(GyD.F4.y2BaR.GyD.F4.y2Ba = 0.88) while the following traits (FEST, HEX, PCA, PPEN, GRN, IVDMD, FAT, SUC, YLD, SC, GLCS, ETOH, HEXE, UA, NSC, FRU, ASH, AX, HEXEP, PSOL) were above 0.5 under the same conditions. When using a CV method where the VS was constrained to consist exclusively within one of either K, N1, N1EM, S, or KxS populations, ANT accuracy ranged from 0.46 in the N1 population to 0.06 in the K population and across different traitsR.GyD.F4.y2Ba在KXS人群中,在KXS人群中,果糖(FRU)(MSE = 0.18)的0.57〜0.23(MSE = 0.05),用于KXS群体。平均而言,在所有特征和群体中,使用Kin-Blup的无约束的CV策略,准确度降低了69.6%,并且类似于其他预测方法。当使用“低”种群作为VS而不是单独的K,N1或N1EM群体和准确度,从未约有的CV策略减少了66.9%时,GEBV略微更准确。GyD.F4.y2Ba
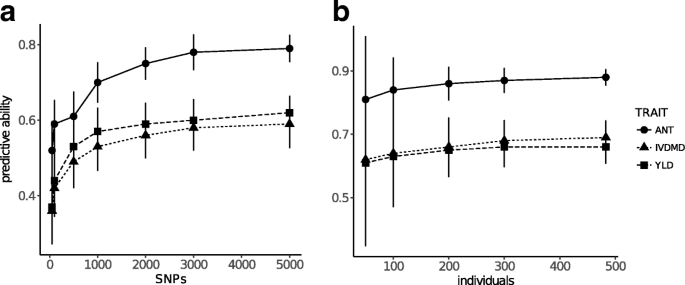
增加了SNP密度以及培训人口大小对预测准确性的影响,以便在所有单个中用5倍的CV进行三个测试特征:IVDMD,YLD和ANT。结果如图1所示。GyD.F4.y2Ba3GyD.F4.y2Ba和GyD.F4.y2BaB.GyD.F4.y2Ba.对于这些性状,增加CS中包含50个以上SNP的数量将提高准确率50.0% - 62.2%,而超过3000个SNP的准确率几乎没有提高。将CS的个体数量从40增加到386,预测精度提高了7.6-10.1%,在重复和折叠中降低了标准差。GyD.F4.y2Ba
当CV方法被人口不同的回归技术不受约束,除了GRN之外的所有特征产生了非常相似的预测精度。对于GRN,SPLS方法产生了最高的精度(GyD.F4.y2BaR.GyD.F4.y2Ba= 0.74, MSE = 0.31), PLS最小(GyD.F4.y2BaR.GyD.F4.y2Ba= 0.65, mse = 0.34)。当CV策略被个体种群限制或在低地(“低”)种群范围内时,各回归方法的结果更有变数。各方法间变异最大的是性状·种群组合,其预测精度在0.3以下。部分性状·群体组合的变异系数大于20%,精度在0.3以上。这包括NSCE·S、PPEN·KxS、NSC·S、PCA·Low、GRN·S和SC·S组合。在这些情况下,SPLS或PLS是最可靠的预测方法,而BB是最不可靠的。GyD.F4.y2Ba
利用方差分析量化性状、种群和预测方法对GEBV测量精度和偏差的影响(附加文件)GyD.F4.y2Ba1GyD.F4.y2Ba:表S6)。GEBV预测精度受CV策略和特征的严重影响及其与预测方法的二阶相互作用。仅预测方法的影响仅暗示了显着效果。偏差也受到群体和特征的较大程度,而其相互作用效应,而Trait X预测方法的二阶相互作用仅暗示了一些影响。GyD.F4.y2Ba
讨论GyD.F4.y2Ba
GBSGyD.F4.y2Ba
通过测序方法的基因分型已被用于不同数量的基因座和不同种类的基因组覆盖范围在许多物种中。以前在SheckGrass中,两种基于参考和非参考的SNP呼叫方法已经应用于GBS数据,这些方法提供了证据,即标记发现和GS育种方法可能是成本效益和准确的[GyD.F4.y2Ba5.GyD.F4.y2Ba那GyD.F4.y2Ba31GyD.F4.y2Ba那GyD.F4.y2Ba44GyD.F4.y2Ba].在这项研究中,TASSEL-GBSv2管道促进了准确调用SNP的能力。该方法获得的标记数和snp数与油棕(GyD.F4.y2BaElaeis Guineensis.GyD.F4.y2Ba;19,432 snps)和小麦(GyD.F4.y2BaTriticum Aestivum.GyD.F4.y2Ba;34,749)[GyD.F4.y2Ba10GyD.F4.y2Ba那GyD.F4.y2Ba43GyD.F4.y2Ba].标签和SNP在参考基因组上不均匀地对齐,其朝向每种染色体的末端,并且对亚基因组没有显着偏见。使用GyD.F4.y2BaPSTI.GyD.F4.y2Ba以“CTGCAT”识别序列组装文库可能导致较少的标记与低GC含量的重复元件对齐,而更多的标记与位于亚端粒区参考序列的编码或调节区域相关。这种不均匀的分布很难从现有的柳枝稷参考基因组的碎片状态中分离出来,但与其他草类物种如高粱(GyD.F4.y2Ba高粱二色的GyD.F4.y2Ba;[GyD.F4.y2Ba42GyD.F4.y2Ba])。GyD.F4.y2Ba
人口结构GyD.F4.y2Ba
许多特征表型和分子变异显示出中度至严重的人口结构。这是预期的,鉴于这些线的繁殖历史和起源。低地群体K,N1和N1EM在它们之间不区分。根据人口和特征,发生基因组遗传性估计的巨大差异。除了几个特征(PSOL,NSC,ASH,FAT)外,大多数遗传性都存在于群体中而不是群体内。这种对比玉米中的一些结果(GyD.F4.y2BaZea Mays.GyD.F4.y2Ba)及米(GyD.F4.y2Ba栽培稻GyD.F4.y2Ba)最遗传性可以在人群中解释的群体[GyD.F4.y2Ba21GyD.F4.y2Ba].GyD.F4.y2Ba
基因组预测的准确性和可靠性已被证明随着验证和训练集之间的距离增加而下降[GyD.F4.y2Ba26GyD.F4.y2Ba].为了避免这种情况,随机采样与大型CS相结合,使得跨越群体的CS中的大多数现有的多样性。其他人通过分层采样处理了这个问题,以优化校准组或使用诸如可靠性的标准[GyD.F4.y2Ba57GyD.F4.y2Ba],预测误差方差或确定系数[GyD.F4.y2Ba1GyD.F4.y2Ba那GyD.F4.y2Ba26GyD.F4.y2Ba那GyD.F4.y2Ba50GyD.F4.y2Ba].这些选择育种程序内特别相关,并取决于性状架构和本结构的程度的相互作用。GyD.F4.y2Ba
基因组预测GyD.F4.y2Ba
测量性状的可重复性在0.34和0.94之间。强烈的积极关系(RGyD.F4.y2Ba2GyD.F4.y2Ba= 0.7;(未显示数据),重复度与GEBV精度之间存在较大差异,对重复度合理且精度在0.5以上的21个性状进行了重点分析。这些性状包括总己糖和乙醇潜能,这是生物能量的重要参数,以及IVDMD,这是预测牛日增重的指标。导致GEBV准确性较差的一些性状包括CAL、GAL、N、STA、MAN、FUC、GLC和DM。使用受限vs,这些性状中的一个或多个可能具有更好的准确性。然而,由于整体准确性较差,这些性状没有被检测。尽管本研究包含了产量,但产量数据是在间隔种植试验中收集的,这并不是植物竞争和死亡率较高的密集种植(草地)地块产量的准确指标[GyD.F4.y2Ba5.GyD.F4.y2Ba那GyD.F4.y2Ba44GyD.F4.y2Ba].这一事实使基因组选择策略复杂化包括产量,但甚至包括在地图中的后代测试,GS育种策略仍然可以缩短生成间隔。据报道,随着基因分型平台,据报道,据报道,对草原产量的预测准确性在0.145和0.237之间,这取决于基因分型平台[GyD.F4.y2Ba5.GyD.F4.y2Ba].GyD.F4.y2Ba
类似于基于GBS的基因组预测的先前示例[GyD.F4.y2Ba10GyD.F4.y2Ba,当VS在所有人群中不受限制时,减少用于分析的SNP标记的数量(在本例中约为3000个)并不会对预测能力产生不利影响。使用kin-BLUP检测gebv的准确性提高到包括3000个YLD、ANT和IVDMD SNP标记。这可能是由于相对较少的标记能够充分捕捉种群之间的关系,以及每个种群内相对密切的个体关系。为了充分捕获与群体间共同QTL相关的遗传变异,需要更高的标记覆盖率。事实上,在许多情况下,个体人群的预测能力较差,平均标记间间隔远远大于LD的距离阈值(GyD.F4.y2BaR.GyD.F4.y2Ba2GyD.F4.y2Ba≤0.2)。本研究发现LD在小于1000 bp的短距离内迅速下降。这种快速的LD衰变可以用柳枝稷的异交特性来解释[GyD.F4.y2Ba34GyD.F4.y2Ba],有效的人口大小,重组率高或其他机制[GyD.F4.y2Ba22GyD.F4.y2Ba].这些利率类似于美国近交易员和地铁的人GyD.F4.y2BaZea Mays.GyD.F4.y2Ba[GyD.F4.y2Ba55GyD.F4.y2Ba]和在GyD.F4.y2Ba挪威云杉GyD.F4.y2Ba[GyD.F4.y2Ba25GyD.F4.y2Ba].增加包括50到483的Kin-Blup分析中的个体数量导致中等,线性的平均预测精度从平均预测精度增加,并且使用5倍交叉验证的重复之间的标准偏差较低。GyD.F4.y2Ba
预测方法之间的差异是感兴趣的,因为使用实际数据而不是模拟数据的这些方法之间的直接比较的实证例子非常少。PLS方法采用维度降低,而稀疏的PLS组合了维度降低和可变选择。Bayesb还使用变量选择,因为它允许一些标记具有大量效果,并且大多数是遗传方差为0.以前,SwitchGrass的结果显示出没有三种不同预测方法的可辨别性能差异[GyD.F4.y2Ba31GyD.F4.y2Ba].该研究表明,这些方法在不同特征的预测能力中总体上非常相似,而VS.相对于具有较低精度的特征·种群组合观察方法之间的变化。这些组合在复制和跨折叠之间具有更高的标准偏差,因此在很大程度上,该变化可能代表随机误差组件。然而,几个特质·人口组合突出了。这些包括来自非结构碳水化合物(NSCE),可溶性葡萄糖(GLC),蔗糖(SUC)和非结构碳水化合物(NSC)的估计的乙醇,特别是在夏季群体中,PLS方法比其他方法显着更好。在这种情况下,这些特征应该与这些表型相似,因为这些表型是正相关的,并且直接从STA,GLC,FRU和SUC的NIR估计计算NSCE和NSC [GyD.F4.y2Ba60GyD.F4.y2Ba].有趣的另一组合是对P-香豆素酯(PCA)和酯与酯连接的阿魏酸盐(FETH)的估计值。这些特征是肯定的相关性,并且在夏季人口中具有最低的值。当VS组成的任何人口组成时,GEBV的准确性相对较高,除了Kanlow和Kanlow N1中的PCA的特定情况。这只是因为这两个特征,似乎没有强大的理由,为什么在不同群体中应该存在这样一系列的预测精度,在低地群体中的相同特征中,鉴于它们如此密切相关PCA和GyD.F4.y2BaFGyD.F4.y2Ba统计数据。GyD.F4.y2Ba
这组数据的预测精度可与在北部高地多样性小组中发现的形态和组成特征相媲美[GyD.F4.y2Ba31GyD.F4.y2Ba].在该研究中,分析了一些相同的性状,Cs和Vs来自所有线,并且PCA的前两个主要成分被用作解释变量,以考虑人口结构。在本研究中,对于在交叉验证步骤中独立或组合处理群体的前三个原理组分仅考虑用于计算群体的遗传性,以评估模型性能而不尝试使用PC作为解释性变量。GyD.F4.y2Ba
结论GyD.F4.y2Ba
本研究强调了在SwitchGrass中使用基因组选择的可能益处。仍然可以在表型和基因分型方法中进行许多改进(例如预测高竞争环境中的产量的有效间接方法),以在SheckGrass中更快地提高遗传衰退[GyD.F4.y2Ba5.GyD.F4.y2Ba].然而,很明显,形态学和组成性状可以有效地选择使用间接的方法,仅基于基因型数据和全基因组回归技术。考虑到生物能源生产过程的不确定性和复杂性,未来的关键将是评估这些技术在柳枝稷上的成本效益。GyD.F4.y2Ba
缩写GyD.F4.y2Ba
- ADF:GyD.F4.y2Ba
-
酸性洗涤剂纤维GyD.F4.y2Ba
- ADL:GyD.F4.y2Ba
-
酸性洗涤木质素GyD.F4.y2Ba
- 蚂蚁:GyD.F4.y2Ba
-
花分动日期GyD.F4.y2Ba
- 蚂蚁:GyD.F4.y2Ba
-
开花期GyD.F4.y2Ba
- 阿拉:GyD.F4.y2Ba
-
阿拉伯糖GyD.F4.y2Ba
- 灰:GyD.F4.y2Ba
-
矿物质(总灰分)GyD.F4.y2Ba
- AX:GyD.F4.y2Ba
-
ARA + XYL.GyD.F4.y2Ba
- AXMG:GyD.F4.y2Ba
-
ARA + XYL + MAN + GALGyD.F4.y2Ba
- BB:GyD.F4.y2Ba
-
贝叶斯GyD.F4.y2Ba
- 结影:GyD.F4.y2Ba
-
最佳线性无偏预测器GyD.F4.y2Ba
- C:GyD.F4.y2Ba
-
碳GyD.F4.y2Ba
- cal:GyD.F4.y2Ba
-
总能量内容GyD.F4.y2Ba
- CS:GyD.F4.y2Ba
-
校准集合GyD.F4.y2Ba
- CTAB:GyD.F4.y2Ba
-
十六烷基三甲基溴化物GyD.F4.y2Ba
- 简历:GyD.F4.y2Ba
-
交叉验证GyD.F4.y2Ba
- 《禁止化学武器公约》:GyD.F4.y2Ba
-
细胞壁浓度GyD.F4.y2Ba
- CWE:GyD.F4.y2Ba
-
细胞壁乙醇GyD.F4.y2Ba
- CWEP:GyD.F4.y2Ba
-
从细胞壁己糖转化乙醇的理论效率GyD.F4.y2Ba
- DM:GyD.F4.y2Ba
-
干物质GyD.F4.y2Ba
- doy:GyD.F4.y2Ba
-
一天的一天GyD.F4.y2Ba
- Etoh:GyD.F4.y2Ba
-
乙醇/ g干饲料GyD.F4.y2Ba
- 胖的:GyD.F4.y2Ba
-
提取脂肪GyD.F4.y2Ba
- 节日:GyD.F4.y2Ba
-
酯化ferulatesGyD.F4.y2Ba
- Feth:GyD.F4.y2Ba
-
醚化的刺激GyD.F4.y2Ba
- FRU:GyD.F4.y2Ba
-
果糖GyD.F4.y2Ba
- FUC:GyD.F4.y2Ba
-
岩藻糖GyD.F4.y2Ba
- 加:GyD.F4.y2Ba
-
半乳糖GyD.F4.y2Ba
- GBS:GyD.F4.y2Ba
-
基因分型结果进行排序GyD.F4.y2Ba
- gDNA:GyD.F4.y2Ba
-
基因组DNAGyD.F4.y2Ba
- GEBV:GyD.F4.y2Ba
-
基因组育种值GyD.F4.y2Ba
- 相关:GyD.F4.y2Ba
-
葡萄糖GyD.F4.y2Ba
- GLCS:GyD.F4.y2Ba
-
可溶性葡萄糖GyD.F4.y2Ba
- 入库单:GyD.F4.y2Ba
-
春季出现日期GyD.F4.y2Ba
- 入库单:GyD.F4.y2Ba
-
春季出现日期GyD.F4.y2Ba
- GS:GyD.F4.y2Ba
-
基因组选择GyD.F4.y2Ba
- 十六进制:GyD.F4.y2Ba
-
总六半GyD.F4.y2Ba
- 六角:GyD.F4.y2Ba
-
从己糖理论乙醇(不含淀粉)GyD.F4.y2Ba
- HEXEP:GyD.F4.y2Ba
-
己糖乙醇提取效率GyD.F4.y2Ba
- IVDMD:GyD.F4.y2Ba
-
体外干物质消化率GyD.F4.y2Ba
- k:GyD.F4.y2Ba
-
kanlowGyD.F4.y2Ba
- kin-BLUP:GyD.F4.y2Ba
-
亲属性最佳线性无偏见预测GyD.F4.y2Ba
- KL:GyD.F4.y2Ba
-
克拉森木质素GyD.F4.y2Ba
- KXS:GyD.F4.y2Ba
-
Kanlow x夏季F2GyD.F4.y2Ba
- LD:GyD.F4.y2Ba
-
连锁不平衡GyD.F4.y2Ba
- 低的:GyD.F4.y2Ba
-
低地GyD.F4.y2Ba
- 男人:GyD.F4.y2Ba
-
曼诺斯GyD.F4.y2Ba
- 马诺瓦:GyD.F4.y2Ba
-
多元差异分析GyD.F4.y2Ba
- MSE:GyD.F4.y2Ba
-
均方误差GyD.F4.y2Ba
- n:GyD.F4.y2Ba
-
氮GyD.F4.y2Ba
- n1:GyD.F4.y2Ba
-
Kanlow N1GyD.F4.y2Ba
- n1em:GyD.F4.y2Ba
-
Kanlow N1-早期成熟GyD.F4.y2Ba
- NDF:GyD.F4.y2Ba
-
中性洗涤剂纤维GyD.F4.y2Ba
- 德尼尔斯:GyD.F4.y2Ba
-
近红外光谱GyD.F4.y2Ba
- NSC:GyD.F4.y2Ba
-
非结构碳水化合物(淀粉+ SC)GyD.F4.y2Ba
- NSCE:GyD.F4.y2Ba
-
来自非结构碳水化合物的估计乙醇GyD.F4.y2Ba
- 个人电脑 :GyD.F4.y2Ba
-
主成分GyD.F4.y2Ba
- PCA:GyD.F4.y2Ba
-
p-Coumarate酯GyD.F4.y2Ba
- PCA:GyD.F4.y2Ba
-
原理成分分析,p-香豆酸GyD.F4.y2Ba
- PCR:GyD.F4.y2Ba
-
聚合酶链反应GyD.F4.y2Ba
- 浮:GyD.F4.y2Ba
-
pentose糖relased / g干法GyD.F4.y2Ba
- PENTP:GyD.F4.y2Ba
-
pentose提取效率GyD.F4.y2Ba
- 请:GyD.F4.y2Ba
-
部分最小二乘GyD.F4.y2Ba
- PPEN:GyD.F4.y2Ba
-
总碳水化合物的pentose比例GyD.F4.y2Ba
- PSOL:GyD.F4.y2Ba
-
非结构性或可溶性的六角体比例GyD.F4.y2Ba
- QTL:GyD.F4.y2Ba
-
数量性状位点GyD.F4.y2Ba
- REML:GyD.F4.y2Ba
-
限制最大可能性GyD.F4.y2Ba
- rha:GyD.F4.y2Ba
-
rhamnose.GyD.F4.y2Ba
- S:GyD.F4.y2Ba
-
夏天GyD.F4.y2Ba
- SC:GyD.F4.y2Ba
-
总可溶性碳水化合物GyD.F4.y2Ba
- SNP:GyD.F4.y2Ba
-
单核苷酸多态性GyD.F4.y2Ba
- SPLS:GyD.F4.y2Ba
-
稀疏的部分最小二乘GyD.F4.y2Ba
- STA:GyD.F4.y2Ba
-
淀粉GyD.F4.y2Ba
- 成功:GyD.F4.y2Ba
-
蔗糖GyD.F4.y2Ba
- 见sub:GyD.F4.y2Ba
-
总糖GyD.F4.y2Ba
- TSTV:GyD.F4.y2Ba
-
过渡:颠换比GyD.F4.y2Ba
- UA:GyD.F4.y2Ba
-
糖酸GyD.F4.y2Ba
- UTR:GyD.F4.y2Ba
-
翻译区GyD.F4.y2Ba
- vs:GyD.F4.y2Ba
-
验证集GyD.F4.y2Ba
- XYL:GyD.F4.y2Ba
-
木糖GyD.F4.y2Ba
- 收获率:GyD.F4.y2Ba
-
干重产量GyD.F4.y2Ba
- 收获率:GyD.F4.y2Ba
-
干物质收益率GyD.F4.y2Ba
参考GyD.F4.y2Ba
- 1.GyD.F4.y2Ba
Akdemir D,Sanchez Ji,Jannink J-L(2015)用遗传算法优化基因组选择培训群体。Genet Sel Evol GSE 47:38。https://GyD.F4.y2Badoi.org/10.1186/s12711 - 015 - 0116 - 6GyD.F4.y2Ba.GyD.F4.y2Ba
- 2.GyD.F4.y2Ba
Bates D,MächlerM,Bolker BM,Walker SC(2015)使用LME4拟合线性混合效果模型。j stat softw 67:1-48。https://GyD.F4.y2Badoi.org/10.18637/jss.v067.i01.GyD.F4.y2Ba.GyD.F4.y2Ba
- 3.GyD.F4.y2Ba
Bouton J.美国牧草改善的经济效益。Euphytica Neth J植物品种。2007; 154:263-70 ..GyD.F4.y2Ba
- 4.GyD.F4.y2Ba
Casler M.北方北部开关种群的生态型变异。农作物科学。2005; 45:388-98 ..GyD.F4.y2Ba
- 5.GyD.F4.y2Ba
Casler MD, Ramstein GP(2017)利用产量替代措施进行柳枝稷生物量产量的育种。生物能源Res 1 - 12。https://GyD.F4.y2Badoi.org/10.1007/s12155 - 017 - 9867 - yGyD.F4.y2Ba.GyD.F4.y2Ba
- 6.GyD.F4.y2Ba
Chen D-H, Ronald P.一种适用于AFLP及其它PCR应用的快速DNA微制备方法。Plant Mol Biol rep 1999; 17:53-7 ..GyD.F4.y2Ba
- 7.GyD.F4.y2Ba
基于稀疏偏最小二乘回归的同步降维和变量选择。J R Stat Soc Ser B Stat方法72:3-25。https://GyD.F4.y2Badoi.org/10.1111/j.1467-9868.2009.00723.xGyD.F4.y2Ba.GyD.F4.y2Ba
- 8.GyD.F4.y2Ba
钟德华,田华,Keleş S(2013)稀疏偏最小二乘(spls)回归与分类。GyD.F4.y2Bahttps://CRAN.R-project.org/package=splsGyD.F4.y2Ba.GyD.F4.y2Ba
- 9。GyD.F4.y2Ba
Cingolani P,Platts A,Wang Ll等)(2012)一种注释和预测单核苷酸多态性,SNPEFF:SNP在基因组中的效果的程序GyD.F4.y2Ba果蝇黑胶基GyD.F4.y2Ba应变W1118;ISO-2;ISO-3。飞(奥斯汀)6:80-92。https://GyD.F4.y2Badoi.org/10.4161/fly.19695GyD.F4.y2Ba.GyD.F4.y2Ba
- 10。GyD.F4.y2Ba
CROS D,BOCS S,RIOU V,等(2017)基因组预选与逐个测序的基因分序列增加了商业油棕榈杂交交流的性能。BMC基因组学18:839。https://GyD.F4.y2Badoi.org/10.1186/s12864 - 017 - 4179 - 3GyD.F4.y2Ba.GyD.F4.y2Ba
- 11.GyD.F4.y2Ba
De Los Campos G,Rodriguez PP(2016)BGLR:贝叶斯广义线性回归。GyD.F4.y2Bahttps://CRAN.R-project.org/package=BGLRGyD.F4.y2Ba.GyD.F4.y2Ba
- 12.GyD.F4.y2Ba
De Los Campos G,Gianola D,Rosa GJM,Weigel Ka,Crossa J(2010)使用再现核Hilbert空间方法的遗传值的启用遗传值的半导体基因组的预测。Genet Res 92:295-308。https://GyD.F4.y2Badoi.org/10.1017/S001667231000000285.GyD.F4.y2Ba.GyD.F4.y2Ba
- 13.GyD.F4.y2Ba
Elshire RJ,Glaubitz JC,孙Q,等人(2011)一个强大的,简单的基因分型,通过测序(GBS)的高度多样性的物种的办法。公共科学图书馆·一六:19379。https://GyD.F4.y2Badoi.org/10.1371/journal.pone.0019379GyD.F4.y2Ba.GyD.F4.y2Ba
- 14.GyD.F4.y2Ba
Endelman JB(2011)利用R包rrBLUP进行基因组选择的Ridge回归和其他核。植物基因组,4:250 - 255。https://GyD.F4.y2Badoi.org/10.3835/plantgenome2011.08.0024GyD.F4.y2Ba.。GyD.F4.y2Ba
- 15.GyD.F4.y2Ba
Endelman JB, Jannink J-L(2013)实现关系矩阵的收缩估计。G3 2:1405 - 1413。https://GyD.F4.y2Badoi.org/10.1534/g3.112.004259GyD.F4.y2Ba.GyD.F4.y2Ba
- 16.GyD.F4.y2Ba
埃文斯J,Crisovan E,Barry K,Et(2015)北方交叉口的多样性和人口结构,通过Exome捕获测序揭示。工厂J 84:800-815。https://GyD.F4.y2Badoi.org/10.1111/tpj.13041GyD.F4.y2Ba.GyD.F4.y2Ba
- 17.GyD.F4.y2Ba
Fair J,Parrish D,Wolf D等人。美国上东南部的切换生成:品种和切割频率对生物质产量的影响。生物量生物能源。2006; 30:207-13 ..GyD.F4.y2Ba
- 18.GyD.F4.y2Ba
García-Ruiz A,Cole JB,Vanraden PM,等人(2016)由于基因组选择,遗传选择差异和发电间隔的变化。Proc Natl Acad SCI U S 113:E3995-E4004。https://GyD.F4.y2Badoi.org/10.1073/pnas.1519061113GyD.F4.y2Ba.GyD.F4.y2Ba
- 19。GyD.F4.y2Ba
Glaubitz JC,Casstevens TM,Lu F等人(2014)Tassel-GBS:通过测序分析管道进行高容量基因分型。Plos一个9:E90346。https:/doi.org/10.1371/journal.pone.0090346GyD.F4.y2Ba
- 20。GyD.F4.y2Ba
Gois IB, Borém A, Cristofani-Yaly M,等(2016)柑橘育种中的全基因组选择。基因摩尔Res 15:gmr15048863。https://GyD.F4.y2Badoi.org/10.4238/gmr15048863GyD.F4.y2Ba.GyD.F4.y2Ba
- 21。GyD.F4.y2Ba
过Z,塔克DM,巴斯滕CJ,等人,(2014)群体结构的基因组中预测分层群的影响。理论应用遗传学127:749-762。https://GyD.F4.y2Badoi.org/10.1007/s00122 - 013 - 2255 - xGyD.F4.y2Ba.GyD.F4.y2Ba
- 22。GyD.F4.y2Ba
古普塔PK,Rustgi S,Kulwal PL。连锁不平衡和关联研究高等植物:现状和未来前景。植物mol biol。2005; 57:461-85 ..GyD.F4.y2Ba
- 23。GyD.F4.y2Ba
Habier D,Tetens J,Seatfried FR,Lichtner P,Thaller G.遗传关系信息对德国Holstein牛基因组育种价值的影响。Genet Sel Evol。2010; 42:5 ..GyD.F4.y2Ba
- 24。GyD.F4.y2Ba
Heffner El,Lorenz Aj,Jannink J-L,Sorrells Me(2010)植物育种与基因组选择:每单位时间和成本获得。作物SCI 50:1681-1690。https://GyD.F4.y2Badoi.org/10.2135/cropsci2009.11.0662GyD.F4.y2Ba.GyD.F4.y2Ba
- 25。GyD.F4.y2Ba
Heuertz男,德保利E,卡尔曼T,等人(2006)核苷酸多样性,连锁不平衡和挪威云杉人口史[中多位模式GyD.F4.y2Ba挪威云杉GyD.F4.y2Ba(L.)喀斯特]。遗传学174:2095-2105。https://GyD.F4.y2Badoi.org/10.1534/genetics.106.065102GyD.F4.y2Ba.GyD.F4.y2Ba
- 26.GyD.F4.y2Ba
ISIDRO J,Jannink J-L,Akdemir D等人(2015)在基因组选择中培训群体结构下的优化。Al Appl Genet 128:145-158。https://GyD.F4.y2Badoi.org/10.1007/S00122-014-2418-4GyD.F4.y2Ba.GyD.F4.y2Ba
- 27.GyD.F4.y2Ba
Janss L,De Los Campos G,Sheehan N,Sorensen D.(2012)分层群体基因组模型推断。遗传学192:693-704。https://GyD.F4.y2Badoi.org/10.1534/Genetics.112.141143GyD.F4.y2Ba.GyD.F4.y2Ba
- 28.GyD.F4.y2Ba
Koshi Pt,StubBendieck J,ECK HV,McCully WG。切换:饲料产量,饲料质量和水利用效率。j范围管理。1982; 35:623-7 ..GyD.F4.y2Ba
- 29.GyD.F4.y2Ba
LêCAOK-A,Rossouw D,Robert-Graniéc,Besse P(2008)在整合OMICS数据时为变量选择的稀疏PLS。STAT APPL GEAT MOL BIOL 7:第35条HTTPS://GyD.F4.y2Badoi.org/10.22022/1544-6115.1390GyD.F4.y2Ba.GyD.F4.y2Ba
- 30.GyD.F4.y2Ba
Li H,Durbin R(2010)快速准确地与挖掘机轮转变换的长读对齐。Bioinforma Oxf Engl 26:589-595。https://GyD.F4.y2Badoi.org/10.1093/bioinformatics/btp698GyD.F4.y2Ba.GyD.F4.y2Ba
- 31。GyD.F4.y2Ba
Lipka Ae,Lu F,Cherney JH,等(2014)加速了SwitchGrass(GyD.F4.y2BaPanicum Virgatum.GyD.F4.y2BaL.)使用基因组选择方法进行繁殖循环。PLO一个9:E112227。https://GyD.F4.y2Badoi.org/10.1371/journal.pone.0112227.GyD.F4.y2Ba.GyD.F4.y2Ba
- 32。GyD.F4.y2Ba
Makowsky r,Pajewski nm,Klimentidis Yc,等(2011)超出了遗失的遗传性:复杂性状的预测。Plos Genet 7:E1002051。https://GyD.F4.y2Badoi.org/10.1371/journal.pgen1002051GyD.F4.y2Ba.GyD.F4.y2Ba
- 33。GyD.F4.y2Ba
Mardia kv,肯特J,Bibby J.多变量分析。伦敦:学术出版社;1979年..GyD.F4.y2Ba
- 34。GyD.F4.y2Ba
Martínez-reyna j,Vogel K.在SwitchGrass中的不兼容系统。农作物科学。2002; 42:1800-5 ..GyD.F4.y2Ba
- 35.GyD.F4.y2Ba
Meuwissen T,Hayes B,戈达德M.使用基因组密集标记图预测总遗传价值。遗传学。2001; 157:1819-29 ..GyD.F4.y2Ba
- 36.GyD.F4.y2Ba
Mevik B-H,Wehrens R.PLS包:R. J Stat软件中的主成分和偏最小二乘性回归。2007; 1((2)):2007 ..GyD.F4.y2Ba
- 37.GyD.F4.y2Ba
Missaoui A,Paterson A,Bouton J(2006)分子标记为SheckGrass分类(GyD.F4.y2BaPanicum Virgatum.GyD.F4.y2BaL.)种质和评估三种合成交叉草群中的遗传多样性。Genet Resour Reach Evol 53:1291-1302。https://GyD.F4.y2Badoi.org/10.1007/S10722-005-3878-9.GyD.F4.y2Ba.GyD.F4.y2Ba
- 38.GyD.F4.y2Ba
钱学森,王志强,王志强,王志强:基于基因型的非模式生物基因型分析。3:基因,基因组,遗传学。2015;5:2383-90。GyD.F4.y2Bahttps://doi.org/10.1534/g3.115.021667GyD.F4.y2Ba.。GyD.F4.y2Ba
- 39.GyD.F4.y2Ba
作者:王志强,王志强,王志强,王志强。描述和量化多年生牧草的生长阶段。阿格龙j . 1991; 83:1073-7 . .GyD.F4.y2Ba
- 40。GyD.F4.y2Ba
okada m,lanzatella c,tobias cm(2010)单轨道EST-SSR标记,用于表征群体遗传多样性和结构跨越股份(GyD.F4.y2BaPanicum Virgatum.GyD.F4.y2Bal .)。Genet Resour Crop Evol 58:919-931。https://GyD.F4.y2Badoi.org/10.1007/S10722-010-9631-Z.GyD.F4.y2Ba.GyD.F4.y2Ba
- 41。GyD.F4.y2Ba
Parrish D,Fike J.生物燃料的Switchgrass生物学和农学。CRIT Rev Plant Sci。2005; 24:423-59 ..GyD.F4.y2Ba
- 42。GyD.F4.y2Ba
等。这GyD.F4.y2Ba高粱二色的GyD.F4.y2Ba基因组和草的多样化。自然。2009; 457:551-6 ..GyD.F4.y2Ba
- 43。GyD.F4.y2Ba
波兰JA, Brown PJ, Sorrells ME, Jannink J-L(2012)利用新型双酶基因分型测序方法构建大麦和小麦高密度遗传图谱。《公共科学图书馆•综合》7:e32253。https://GyD.F4.y2Badoi.org/10.1371/journal.pone.0032253GyD.F4.y2Ba.GyD.F4.y2Ba
- 44。GyD.F4.y2Ba
(5)陆生四倍体柳枝稷二级性状的分化选择及其对草地生物量的影响。生物能源Res 7:329 - 337。https://GyD.F4.y2Badoi.org/10.1007/S12155-013-9374-8GyD.F4.y2Ba.GyD.F4.y2Ba
- 45。GyD.F4.y2Ba
价格Al,Zaitlen Na,Reich D,Patterson N(2010)基因组 - 范围协会研究人口分层的新方法。NAT Rev Genet 11:459-463。https://GyD.F4.y2Badoi.org/10.1038/nrg2813GyD.F4.y2Ba.GyD.F4.y2Ba
- 46。GyD.F4.y2Ba
R开发核心团队。R:统计计算的语言和环境。统计计算基础。维也纳,奥地利;2017年:GyD.F4.y2Bahttps://www.r-project.org.GyD.F4.y2Ba.GyD.F4.y2Ba
- 47。GyD.F4.y2Ba
Ramstein GP, Evans J, Kaeppler SM,等(2016)柳枝稷基因组预测的准确性(GyD.F4.y2BaPanicum Virgatum.GyD.F4.y2BaL.)通过考虑连锁不平衡而得到改善。G3 Bethesda Md 6:1049-1062。https://GyD.F4.y2Badoi.org/10.1534/g3.115.024950GyD.F4.y2Ba.GyD.F4.y2Ba
- 48。GyD.F4.y2Ba
Resende MFR,MuñozP,Acosta JJ,et al(2012)使用基因组选择加速树木的驯化:跨老年和环境预测模型的准确性。新的Phytol 193:617-624。https://GyD.F4.y2Badoi.org/10.1111/j.1469-8137.2011.03895.x.GyD.F4.y2Ba.GyD.F4.y2Ba
- 49。GyD.F4.y2Ba
Riedelsheimer C, Czedik-Eysenberg A, Grieder C, et al .(2012)杂交玉米复杂杂种优势性状的基因组和代谢预测。Nat麝猫44:217 - 220。https://GyD.F4.y2Badoi.org/10.1038/ng.1033GyD.F4.y2Ba.GyD.F4.y2Ba
- 50。GyD.F4.y2Ba
Rincent R,Laloëd,尼古拉斯S,等人(2012)通过优化参考个体的校准组来最大化基因组选择的可靠性:两种多种玉米血统中的方法比较(GyD.F4.y2BaZea Mays.GyD.F4.y2Bal .)。遗传学192:715 - 728。https://GyD.F4.y2Badoi.org/10.1534/genetics.112.141473GyD.F4.y2Ba.GyD.F4.y2Ba
- 51。GyD.F4.y2Ba
Schmer M,Vogel K,Mitchell R,Perrin R.来自SwitchGrass的纤维素乙醇的净能量。Proc Natl Acad Sci U S A. 2008; 105:464-9 ..GyD.F4.y2Ba
- 52。GyD.F4.y2Ba
SimeãoResendeRM,Casler Md,Vilela de Resende MD(2014)饲料育种中的基因组选择:准确性和方法。作物SCI 54:143。https://GyD.F4.y2Badoi.org/10.2135/cropsci2013.05.0353GyD.F4.y2Ba.GyD.F4.y2Ba
- 53。GyD.F4.y2Ba
Talbert Le,Timothy DH,Burns Jc等。expressGrass中遗传参数的估计GyD.F4.y2Ba1GyD.F4.y2Ba.农作物科学。1983; 23:725-8 ..GyD.F4.y2Ba
- 54。GyD.F4.y2Ba
塔利亚弗罗C.育种和选择新型Estropgrass品种,以增加生物质生产。ORNLSUB-02-19XSY162C01。俄克拉荷马州立大学:Stillwater;2002年..GyD.F4.y2Ba
- 55。GyD.F4.y2Ba
Tenaillon Mi,Sawkins MC,Long Ad,Gaut RL,Doebley JF,Gaut BS。DNA序列多态性的模式兼染色体1的玉米(GyD.F4.y2BaZea Mays.GyD.F4.y2Bassp。GyD.F4.y2Ba梅斯GyD.F4.y2Bal .)。Proc Natl Acad SCI U S A. 2001; 98:9161-6 ..GyD.F4.y2Ba
- 56。GyD.F4.y2Ba
Vallejo RL, Leeds TD, Gao G,等(2017)在虹鳟鱼养殖中,基因组选择模型比传统的基于系谱的模型的细菌冷水病抗性育种预测值的准确性提高了一倍。Genet Sel Evol 49:17。https://GyD.F4.y2Badoi.org/10.1186/s12711-017-0293-6GyD.F4.y2Ba.GyD.F4.y2Ba
- 57。GyD.F4.y2Ba
Vanraden P.有效地计算基因组预测的方法。j乳制品sci。2008; 91:4414-23 ..GyD.F4.y2Ba
- 58。GyD.F4.y2Ba
Visscher PM, Yang J, Goddard ME (2010) Yang等人对“常见SNPs解释了人类身高遗传力的很大一部分”的评论(2010)。Twin Res Hum Genet 13:517-524。https://GyD.F4.y2Badoi.org/10.1375/twin.13.6.517GyD.F4.y2Ba.GyD.F4.y2Ba
- 59.GyD.F4.y2Ba
Vogel Kp,Mitchell RB(2008)杂种中的杂种优势在SwitchGrass中:草原中的生物质产量。作物SCI 48:2159。https://GyD.F4.y2Badoi.org/10.2135/cropsci2008.02.0117.GyD.F4.y2Ba.GyD.F4.y2Ba
- 60。GyD.F4.y2Ba
Vogel KP, Dien BS, Jung HG, Casler MD, matson SD, Mitchell RB(2010)利用近红外光谱分析柳枝稷株的实际和理论乙醇产量bioenergy Res 4:96-110。https://GyD.F4.y2Badoi.org/10.1007/s12155 - 010 - 90104 - 4GyD.F4.y2Ba.GyD.F4.y2Ba
- 61。GyD.F4.y2Ba
Vogel Kp,Dien Bs,Jung Hg,等(2011)使用NIR分析量化了Shiftgrass菌株的实际和理论乙醇产量。生物能源4:96-110。https://GyD.F4.y2Badoi.org/10.1007/S12155-010-9104-4GyD.F4.y2Ba.GyD.F4.y2Ba
- 62。GyD.F4.y2Ba
Weir B, Cockerham C(1984)种群结构分析的f统计量估计。另一个星球38:1358 - 1370。https://GyD.F4.y2Badoi.org/10.2307/2408641GyD.F4.y2Ba.GyD.F4.y2Ba
- 63。GyD.F4.y2Ba
基于最小二乘法的主成分和相关模型估计。纽约:学术出版社;1966 . .GyD.F4.y2Ba
- 64。GyD.F4.y2Ba
黄志强(2008)油棕的全基因组选择:在小种群条件下增加单位时间和成本的选择增益。Theor Appl Genet 116:815-824。https://GyD.F4.y2Badoi.org/10.1007/S00122-008-0715-5.GyD.F4.y2Ba.GyD.F4.y2Ba
- 65。GyD.F4.y2Ba
·雷NR,杨洁,海斯BJ,等人(2013)从预测的SNP复杂性状的陷阱。纳特启遗传学14:507-515。https://GyD.F4.y2Badoi.org/10.1038/nrg3457GyD.F4.y2Ba.GyD.F4.y2Ba
- 66。GyD.F4.y2Ba
余建军,王志强。玉米遗传关联作图与基因组组织。生物技术。2006;17:155-60 ..GyD.F4.y2Ba
确认GyD.F4.y2Ba
在本出版物中提及商品名或商业产品仅为特定信息的目的,并不意味着美国农业部的推荐或认可。这项研究使用了美国农业部农业研究服务的Scinet项目提供的资源,ARS项目编号0500-00093-001-00-D。GyD.F4.y2Ba
资金GyD.F4.y2Ba
本材料基于美国能源部、科学办公室、生物与环境研究办公室支持的研究成果,获奖编号为DE-AI02-09ER64829。本研究还得到了美国农业部、农业研究局(USDA-ARS)当前研究信息系统(CRIS) 2030-21000-023D和5440-21000-030-00D的支持。资助方在研究设计、数据收集和分析、决定发表或手稿准备方面没有作用。GyD.F4.y2Ba
可用性数据和材料GyD.F4.y2Ba
在当前研究期间使用和/或分析的数据集可在补充信息文件或合理的请求中获得相应的作者。GyD.F4.y2Ba
作者信息GyD.F4.y2Ba
隶属关系GyD.F4.y2Ba
贡献GyD.F4.y2Ba
CT,GS,NP和JF构思和设计了该研究。CT,JF,GS,NP,CC,SE生成和分析数据。所有作者都写了并批准了稿件。GyD.F4.y2Ba
通讯作者GyD.F4.y2Ba
道德声明GyD.F4.y2Ba
伦理批准和同意参与GyD.F4.y2Ba
不适用。GyD.F4.y2Ba
同意出版物GyD.F4.y2Ba
不适用。GyD.F4.y2Ba
相互竞争的利益GyD.F4.y2Ba
两位作者宣称他们没有相互竞争的利益。GyD.F4.y2Ba
出版商的注意事项GyD.F4.y2Ba
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。GyD.F4.y2Ba
附加文件GyD.F4.y2Ba
附加文件1:GyD.F4.y2Ba
图S1。GyD.F4.y2Ba对整个植物性状的阳性(蓝色)和负(红色)相关性的刻探性。右侧的颜色刻度表示Pearson相关系数GyD.F4.y2BaR.GyD.F4.y2Ba.GyD.F4.y2Ba图S2。GyD.F4.y2Ba近红外光谱测定的墙成分性状之间的正相关(蓝色)和负相关(红色)的相关四边形。右侧的颜色刻度表示Pearson相关系数GyD.F4.y2Bar。GyD.F4.y2Ba图S3。GyD.F4.y2Ba(a)蚂蚁,(b)IVDMD和(C)YLD的Boxplots为每种人口。每个框的底部和顶部代表第一和第三个四分位数。水平线代表中位数,晶须延伸到最极端的数据点,这些数据点不超过框中的四分位数范围的1.5倍。GyD.F4.y2Ba表S1。GyD.F4.y2Ba健BLUP回归统计从5倍CV的20次重复。GyD.F4.y2Ba表S2。GyD.F4.y2Ba部分最小二乘从20个复制的5倍CV的回归统计。GyD.F4.y2Ba表S3。GyD.F4.y2Ba稀疏的部分最小二乘从20个复制的5倍CV复制的回归统计。GyD.F4.y2Ba表S4。GyD.F4.y2BaBayesb回归统计5倍CV。使用5000次迭代和1500次迭代刻录期(参见方法部分)。GyD.F4.y2Ba表S5。GyD.F4.y2Ba基于主导主成分1-3的分区后选择特征的方差分量。表S6:影响预测准确性的因素的ANOVA。(docx 442 kb)GyD.F4.y2Ba
权利和权限GyD.F4.y2Ba
开放获取GyD.F4.y2Ba本文根据创意公约署署署的条款分发了4.0国际许可证(GyD.F4.y2Bahttp://creativecommons.org/licenses/by/4.0/GyD.F4.y2Ba)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(GyD.F4.y2Bahttp://creativecommons.org/publicdomain/zero/1.0/GyD.F4.y2Ba)除非另有说明,否则适用于本文中提供的数据。GyD.F4.y2Ba
关于这篇文章GyD.F4.y2Ba
引用这篇文章GyD.F4.y2Ba
Fiedler,J.D.,Lanzatella,C.,Edmé,S.J.GyD.F4.y2Ba等等。GyD.F4.y2Ba鉴别群体中生物能量相关的基因组预测精度。GyD.F4.y2BaBMC植物杂志GyD.F4.y2Ba18,GyD.F4.y2Ba142(2018)。https://doi.org/10.1186/s12870-018-1360-z.GyD.F4.y2Ba
已收到GyD.F4.y2Ba:GyD.F4.y2Ba
公认GyD.F4.y2Ba:GyD.F4.y2Ba
发表GyD.F4.y2Ba:GyD.F4.y2Ba
迪伊GyD.F4.y2Ba:GyD.F4.y2Bahttps://doi.org/10.1186/s12870-018-1360-z.GyD.F4.y2Ba
关键词GyD.F4.y2Ba
- 多年生GyD.F4.y2Ba
- 生物量GyD.F4.y2Ba
- 生物燃料GyD.F4.y2Ba
- panicum.GyD.F4.y2Ba
- 多向杂交GyD.F4.y2Ba