- 研究文章gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
大豆泛素系统核心成分基因的全基因组分析(gydF4y2Ba大豆gydF4y2Ba)揭示了泛素化在大豆囊线虫宿主免疫中的潜在作用gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba18gydF4y2Ba,文章号:gydF4y2Ba149gydF4y2Ba(gydF4y2Ba2018gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
泛素化是一种主要的翻译后蛋白质修饰,基本上调节真核生物中的所有细胞和生理途径。泛素化过程通常涉及三种不同类型的酶,泛素激活酶(E1),泛素结合酶(E2)和泛素连接酶(E3)。迄今为止,对整个大豆的核心成分进行了全面的鉴定和分析(gydF4y2Ba大豆gydF4y2Ba)泛素系统(UBS)未见报道。gydF4y2Ba
结果gydF4y2Ba
在这项研究中,我们对编码大豆UBS核心成员的基因进行了系统的全基因组分析。共鉴定出1431个编码大豆UBS组分的高信度基因,其中E1s编码基因4个,e2编码基因71个,e3相关基因1356个。在编码e3的基因中,编码ring型E3s的基因760个,编码U-box结构域E3s的基因124个,编码F-box蛋白的基因472个。为了确定所鉴定的大豆UBS基因是否编码活性酶,随机选取一组基因,并对其重组蛋白的酶活性进行检测。硫酯分析表明蛋白由大豆E1基因编码gydF4y2BaGmUBA1gydF4y2Ba所选的E2基因大多为活性E1酶或E2酶。同时,体外泛素化实验显示大部分纯化的RING和U-box结构域蛋白具有E3活性。此外,在14个大豆组织中,有1034个已鉴定的大豆UBS基因至少在其中一个组织中表达,338个大豆USB基因在非生物或生物处理后转录水平发生了显著变化(gydF4y2Ba尖孢镰刀菌gydF4y2Ba而且gydF4y2Ba根瘤菌gydF4y2Ba压力治疗。最后,我们发现大量已鉴定的大豆UBS相关基因在大豆囊线虫(SCN)处理后表达水平发生了显著变化,这表明大豆UBS可能在大豆抗SCN免疫中发挥重要作用。gydF4y2Ba
结论gydF4y2Ba
我们的研究结果表明,大豆基因组中存在大量不同种类的核心UBS蛋白,这表明泛素靶特异性修饰是大豆细胞和生理调控的一个复杂而重要的部分。我们还发现大豆UBS的某些成员可能参与对大豆囊肿线虫(SCN)的免疫。本研究为进一步研究大豆UBS在宿主抗SCN免疫等多种生理过程中的功能特性奠定了基础。gydF4y2Ba
背景gydF4y2Ba
泛素化是一种主要的翻译后蛋白质修饰,在真核生物的许多细胞和生理过程中起着重要作用[gydF4y2Ba1gydF4y2Ba].它涉及到通过三类酶催化的连续反应,将一种高度保守的小蛋白质泛素共价连接到底物上:泛素激活酶(E1)、泛素偶联酶(E2)和泛素连接酶(E3) [gydF4y2Ba2gydF4y2Ba].在酶级联过程中,E1酶首先在ATP水解作用下激活游离泛素,形成硫酯连接,泛素分子的c端甘氨酸与E1活性中心的半胱氨酸残基相连。激活的泛素然后转移到E2酶的保守半胱氨酸残基上。在最后一步中,泛素分子在E3连接酶的帮助下从e2 -泛素中间体转移到底物蛋白。泛素分子通常附着在底物赖氨酸残基的ε-氨基上[gydF4y2Ba3.gydF4y2Ba].在第一个泛素与底物蛋白连接后,酶级联可以重复,从而形成与底物蛋白连接的聚合泛素链,其中泛素部分之间的连接决定了底物在细胞中的命运[gydF4y2Ba3.gydF4y2Ba].gydF4y2Ba
E1s作为泛素偶联级联第一步的催化酶,调控泛素化速率,从而调控细胞内的整体泛素功能[gydF4y2Ba4gydF4y2Ba].到目前为止,已从家兔中分离并鉴定了E1基因及其蛋白。gydF4y2Ba5gydF4y2Ba],酵母[gydF4y2Ba6gydF4y2Ba],小麦[gydF4y2Ba7gydF4y2Ba],老鼠[gydF4y2Ba8gydF4y2Ba], human [gydF4y2Ba9gydF4y2Ba),gydF4y2Ba拟南芥gydF4y2Ba[gydF4y2Ba4gydF4y2Ba]和烟草[gydF4y2Ba10gydF4y2Ba].在所分析的每种植物和动物基因组中都鉴定出了多个E1基因,而酵母基因组只包含一个E1基因。来自所有王国的E1蛋白具有相似的大小,从110到125 kDa,共享高同源性区域,通常包含四个不同的特征结构单元:由两个thif同源基元组成的腺苷酸化结构域[gydF4y2Ba11gydF4y2Ba];由FCCH和SCCH半结构域组成的催化半胱氨酸结构域(分别为“第一”和“第二”催化半胱氨酸半结构域)[gydF4y2Ba12gydF4y2Ba];紧接FCCH的四螺旋束(4HB);c端泛素折叠结构域(UFD) [gydF4y2Ba11gydF4y2Ba,gydF4y2Ba13gydF4y2Ba].E1对e2的特异性部分取决于负责招募同源e2的UFD [gydF4y2Ba14gydF4y2Ba].gydF4y2Ba
E2酶最初被定义为能够通过与半胱氨酸巯基的硫酯连接从E1上接受泛素的蛋白质[gydF4y2Ba15gydF4y2Ba].所有e2都具有一个高度保守的约140-150个氨基酸的结构域,称为泛素偶联结构域(UBC),活性位点的半胱氨酸残基位于该结构域[gydF4y2Ba16gydF4y2Ba].目前,已知酵母中存在11、50和40个泛素E2蛋白(gydF4y2Ba酿酒酵母gydF4y2Ba)、人类(gydF4y2Ba智人gydF4y2Ba)和番茄(gydF4y2Ba茄属植物lycopersicumgydF4y2Ba),分别[gydF4y2Ba17gydF4y2Ba,gydF4y2Ba18gydF4y2Ba,gydF4y2Ba19gydF4y2Ba].除37种泛素E2蛋白外[gydF4y2Ba17gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba],在两种rubb偶联酶(RCE1, At4g36800和RCE2, At2g18600)和一种sumo偶联酶(SCE1, At3g57870)中也发现了UBC结构域gydF4y2Ba拟南芥gydF4y2Ba[gydF4y2Ba21gydF4y2Ba].此外,还有8种拟南芥UBC蛋白缺乏硫酯形成所需的活性位点半胱氨酸残基[gydF4y2Ba2gydF4y2Ba].以前,e2通常被认为是具有辅助作用的“泛素载体”。然而,最近的研究表明,e2控制着从链起始到延伸的切换,并控制着所形成的泛素链的拓扑结构,从而决定了被修饰的底物蛋白的命运[gydF4y2Ba22gydF4y2Ba].gydF4y2Ba
E3泛素连接酶是催化泛素级联的三类酶中最大和最多样化的一组。它们招募目标蛋白进行泛素化,是决定泛素化特异性的主要因素[gydF4y2Ba23gydF4y2Ba].在拟南芥和人类基因组中,预计分别有1300多个基因和600多个基因编码e3相关成分[gydF4y2Ba24gydF4y2Ba,gydF4y2Ba25gydF4y2Ba].E3连接酶可以是单多肽蛋白,也可以是多亚基复合物。根据E3连接酶的作用机制和不同蛋白结构域的存在,单多肽泛素连接酶可分为三种明确的类型:泛素连接酶gydF4y2BahgydF4y2BaOmology to thegydF4y2BaEgydF4y2Ba6-associated蛋白质gydF4y2BaCgydF4y2Ba-gydF4y2BatgydF4y2Baerminus (HECT),gydF4y2BargydF4y2Ba真的gydF4y2Ba在gydF4y2Bateresting新gydF4y2BaggydF4y2Ba烯(环)-,或u盒域包含E3s。he赫特型E3s是单亚基蛋白,其特征是具有一个c端约350个氨基酸的HECT结构域,该结构域参与接受E2蛋白的泛素并将其转移到底物蛋白[gydF4y2Ba26gydF4y2Ba].heht型E3连接酶的一个独特特征是一个保守的催化半胱氨酸残基,作为形成硫酯连接泛素-E3中间体的位点。在这些E3连接酶中,E2在泛素转移到底物之前将半胱氨酸残基与泛素结合。迄今为止,植物hect型E3s已在gydF4y2Ba拟南芥gydF4y2Ba仅含有7个HECT基因命名gydF4y2BaUPL1gydF4y2Ba-gydF4y2BaUPL7gydF4y2Ba[gydF4y2Ba27gydF4y2Ba].进化分析表明,在不同的植物基因组中,HECT基因的数量一直保持相当稳定[gydF4y2Ba28gydF4y2Ba].与heht型E3s不同,RING和U-box蛋白通过保守的RING或U-box结构域与携带硫酯连接泛素的E2发生非共价相互作用,以促进泛素向底物的转移[gydF4y2Ba2gydF4y2Ba].RING和U-box连接酶在结构上相互关联,分别利用锌螯合结构域和氢键/盐桥传递泛素[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba29gydF4y2Ba].环型E3s是单亚基泛素连接酶中含量最多的[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba].U-box结构域是一个经过修饰的RING结构域,缺乏保守的zn配位残基[gydF4y2Ba32gydF4y2Ba].U-box型E3泛素连接酶的特征是最初在酵母UFD2蛋白中发现的保守的~ 70个氨基酸U-box结构域[gydF4y2Ba33gydF4y2Ba].除了典型的单多肽E3s外,含有RING结构域的蛋白质也可以是复杂的多亚基E3s的亚基,包括Skp1-Cullin-F-box (SCF),后期促进复合物/环体(APC/C)和Cullin-Elongin-BC-VHL (CBC VHL)型E3连接酶[gydF4y2Ba34gydF4y2Ba].在研究充分的scf型多亚基连接酶中,含有RING结构域的蛋白RBX/ROC/HRT负责与E2结合,而F-box蛋白则赋予底物识别能力[gydF4y2Ba35gydF4y2Ba].一个F-box蛋白至少包含一个F-box结构域,在其n端包含约40个氨基酸,与SKP1结合形成SCF复合体[gydF4y2Ba36gydF4y2Ba].F-box蛋白已在两种原核生物中被发现[gydF4y2Ba37gydF4y2Ba]和真核生物。在植物中,F-box基因家族也是最大的基因家族之一,这表明它们可能调节许多重要的生物过程[gydF4y2Ba38gydF4y2Ba,gydF4y2Ba39gydF4y2Ba].gydF4y2Ba
泛素化最初被认为是细胞中选择性蛋白质降解的主要信号。然而,自30多年前发现泛素化以来,它的功能已经远远超出了这一范围。在过去的三十年中,泛素化在动物、人类和植物中调控无数细胞和生理过程中的重要性日益得到重视[gydF4y2Ba31gydF4y2Ba,gydF4y2Ba40gydF4y2Ba].大豆(gydF4y2Ba大豆gydF4y2Ba)是世界上主要的作物和主要的油籽。病害一直是大豆产量的主要制约因素。gydF4y2Ba41gydF4y2Ba].大豆囊线虫gydF4y2Ba异皮线虫属甘氨酸gydF4y2Ba一病病(Ichinohe)一直是全球大豆最重要的经济病原体,在美国每年造成约10亿美元的产量损失[gydF4y2Ba42gydF4y2Ba].虽然已经在大豆中单独研究了E3连接酶的几个亚家族[gydF4y2Ba43gydF4y2Ba,gydF4y2Ba44gydF4y2Ba,gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba],对整个泛素系统(UBS)核心组成部分的全面认识尚未见报道。在本研究中,对大豆UBS的核心成分进行了全基因组分析。通过一系列生物信息学分析,从大豆基因组中鉴定出4个泛素E1基因,71个泛素E2基因,1356个编码泛素E3s的基因,包括760个RING结构域,124个U-box结构域和472个F-box结构域[gydF4y2Ba47gydF4y2Ba].克隆了大豆UBS的数十个组分,并对其酶活性进行了测定。此外,RNA-seq数据分析和实时定量PCR (real time qPCR)结果表明,大豆UBS中许多成分的表达模式在大豆囊肿线虫(SCN)处理下发生了显著变化,这支持了大豆UBS可能在宿主对SCN的免疫中发挥作用的观点。这些结果为进一步研究大豆UBS关键成分在大豆抗SCN免疫过程中的功能特性提供了有价值的基础。gydF4y2Ba
结果gydF4y2Ba
大豆基因组中有4个泛素E1基因gydF4y2Ba
所有真核生物E1s都含有一个由两个ThiF同源基序组成的腺苷酸化结构域,这些基序来源于细菌ThiF蛋白[gydF4y2Ba48gydF4y2Ba].ThiF基序被认为是泛素和类泛素蛋白(UBL)- e1识别和腺苷酸化活性的最小模块[gydF4y2Ba49gydF4y2Ba].因此,以NCBI保守域数据库(CDD)中ThiF基序的一致序列(PF00899)为查询对象,对大豆基因组Phytozome v.12.1数据库(gydF4y2Ba大豆gydF4y2BaWm82.a2.v1)。共鉴定出来自20个编码ThiF基序的蛋白质基因的37个转录本,显然是由于一些基因具有多个注释转录本(附加文件)gydF4y2Ba1gydF4y2Ba:表S1)。其中,来自4个基因的7个转录本编码超过1000个氨基酸和分子量(Mw)约120 kD的蛋白质(附加文件)gydF4y2Ba1gydF4y2Ba:表S1),与已在其他植物中鉴定的泛素E1蛋白相似[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba7gydF4y2Ba].gydF4y2Ba
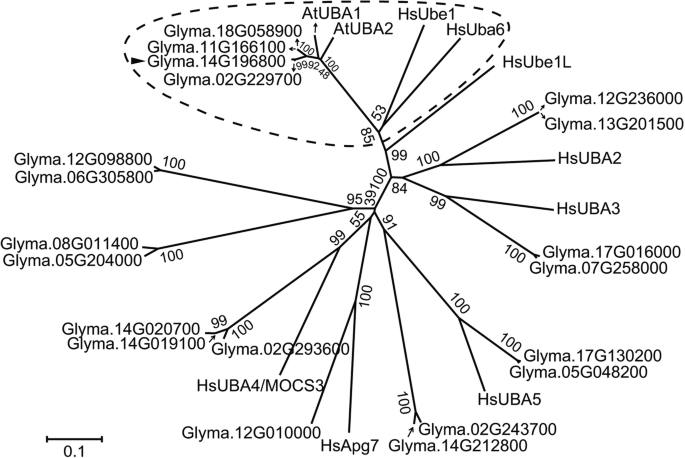
在人类中,已知有8个E1s负责初始的泛素和UBL识别以及酰基腺苷化,而只有两个不同的E1s, UBE1和UBA6,专门启动泛素的偶联,而不是其他UBLs [gydF4y2Ba14gydF4y2Ba].为了研究大豆和人类中含有ThiF基序的蛋白质之间的进化关系,我们生成了一个邻居连接(NJ)系统发育树(图2)。gydF4y2Ba1gydF4y2Ba).对于那些有多个注释转录本的基因,只有数据库指定的主要转录本的蛋白质被包括在分析中。由基因座编码的四个ThiF基序蛋白质gydF4y2BaGlyma.02G229700gydF4y2Ba,gydF4y2BaGlyma.11G166100gydF4y2Ba,gydF4y2BaGlyma.14G196800gydF4y2Ba而且gydF4y2BaGlyma.18G058900gydF4y2Ba,分别与人类UBE1和UBA6亲缘关系更密切,在树中与拟南芥泛素E1 AtUBA1和AtUBA2聚在同一分支中(图;gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
利用Pfam数据库对所有含ThiF基序的大豆蛋白进行结构域分析[gydF4y2Ba50gydF4y2Ba].在系统发育分析中,与拟南芥泛素E1s聚类的四个蛋白质中,每个蛋白质中都含有两个ThiF基序(PF00899),一个UFD (PF09358),一个FCCH (PF16190)和4HB (PF16191)插入到第一个ThiF基序中,一个UBA_e1_thiolCys (SCCH) (PF10585)插入到第二个ThiF基序中(图10585)。gydF4y2Ba2gydF4y2Ba),这类似于泛素E1酶的典型结构域组织。其他含有ThiF基序的大豆蛋白在c端要么只有一个ThiF基序,要么没有UFD结构域。c端UFD在不同王国的e1中都是保守的,在招募特定e2中起着重要作用[gydF4y2Ba13gydF4y2Ba].序列比对显示了四个由基因座编码的含ThiF基序的大豆蛋白的ufdgydF4y2BaGlyma.02G229700gydF4y2Ba,gydF4y2BaGlyma.11G166100gydF4y2Ba,gydF4y2BaGlyma.14G196800gydF4y2Ba而且gydF4y2BaGlyma.18G058900gydF4y2Ba与其他植物和人体内相似(图2)。gydF4y2Ba2 bgydF4y2Ba).综上所述,我们认为大豆基因组中含有4个编码泛素E1蛋白的基因。gydF4y2Ba
大豆含ThiF基序蛋白的序列分析。gydF4y2Ba一个gydF4y2Ba大豆含ThiF基序蛋白的结构,以glyma . 14g1968002为代表。大豆含ThiF基序的蛋白序列用Pfam (gydF4y2Bahttp://pfam.xfam.org/gydF4y2Ba).FCCH:第一催化半胱氨酸半畴;4HB:四螺旋束;UFD:泛素折叠域。UBA_e1_thiolCys结构域也被称为SCCH,包含一个负责泛素硫酯连接的半胱氨酸残基。gydF4y2BabgydF4y2Ba大豆E1蛋白UFD(泛素折叠结构域)的多序列定位gydF4y2Ba大豆,gydF4y2BaGlyma), Arabidopsis (gydF4y2Ba拟南芥gydF4y2Ba, At),小麦(gydF4y2Ba小麦,gydF4y2BaTa)、烟草(gydF4y2Ba烟草gydF4y2Ba, Nt)和human (gydF4y2Ba智人gydF4y2BaHs)。用Pfam分析UFD。多序列比对由MUSCLE程序使用MEGA6实现,图像生成由BoxShade [gydF4y2Ba91gydF4y2Ba].保守残基和相似残基用黑色和灰色表示。排列下方的序列表示排列ufd的一致序列gydF4y2Ba
大豆基因组编码的71种泛素e2被分为11类gydF4y2Ba
为了精确定位编码泛素E2的大豆基因,泛素偶联(UBC)结构域(PF00179)的隐马尔可夫模型(HMM)轮廓(附加文件)gydF4y2Ba2gydF4y2Ba:表S2)作为查询,采用HMMER 3.1程序对大豆蛋白数据库进行检索[gydF4y2Ba51gydF4y2Ba].与E1基因类似,我们发现许多假定的E2基因具有多个注释转录本(异构体),并且仅使用数据库中为这些基因指定的主要转录本(即主转录本)进行后续分析。通过HMMER分析,共鉴定出107个编码含UBC结构域蛋白的基因。进一步利用Pfam数据库和NCBI CCD数据库对107个蛋白进行验证。BLAST分别在99个和106个蛋白质中鉴定出一个假定的UBC结构域(表2)gydF4y2Ba1gydF4y2Ba).结合这些结果,我们初步预测了大豆基因组中编码假定含UBC结构域蛋白的106个基因(表2)gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
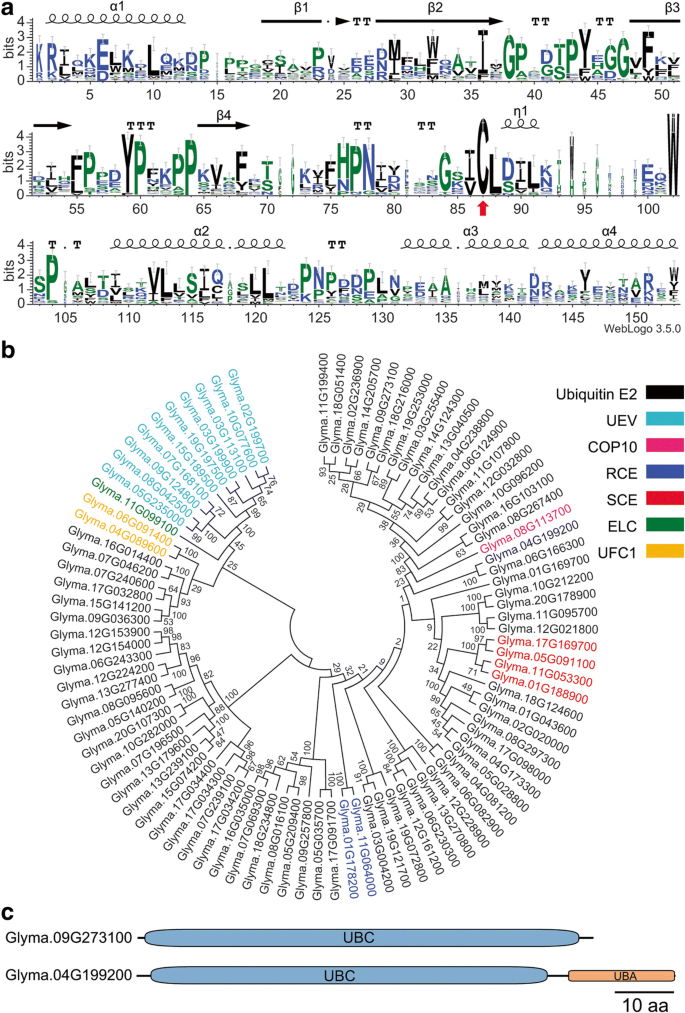
为了进一步研究这些假定的UBC结构域蛋白编码基因,我们进行了手动验证。UBC结构域折叠的核心形成了一个四链的β-薄片[gydF4y2Ba52gydF4y2Ba].在这个基本核心之外,UBC结构域褶皱在c端瓣状结构中包含两个小元素,在n端也有一个螺旋,在c端也有1-2个螺旋[gydF4y2Ba53gydF4y2Ba].此外,活性中心的催化半胱氨酸残基通常位于皮瓣的c端,在UBC结构域中高度保守[gydF4y2Ba16gydF4y2Ba,gydF4y2Ba53gydF4y2Ba].序列分析最终从106个候选基因中确定了91个编码典型UBC结构域蛋白的基因(附加文件)gydF4y2Ba3.gydF4y2Ba:表S3)。使用Weblogo3在线生成了一个图形序列标识,表示基于上述91个蛋白质的UBC结构域对齐的序列模式。gydF4y2Ba3gydF4y2Ba) [gydF4y2Ba54gydF4y2Ba],完整的对齐信息显示在附加文件中gydF4y2Ba4gydF4y2Ba:图S1。gydF4y2Ba
大豆含UBC结构域蛋白的结构及系统发育分析。gydF4y2Ba一个gydF4y2Ba图形序列标志表示91个大豆含UBC结构域蛋白的UBC结构域序列。每个堆栈的整体高度表示序列的保守程度,而每个堆栈中字母的高度表示相应氨基酸在该位置的相对频率。字母的宽度与该位置有效字母的比例成正比。误差条表示贝叶斯95%置信区间。蓝色、绿色和黑色字母分别表示亲水性、中性和疏水性残留。y轴的比特分数表示给定序列位置的信息量。红色箭头表示活性半胱氨酸残留。二级结构元素的位置用α表示α-螺旋,β表示β-薄片,η表示3gydF4y2Ba10gydF4y2Ba-helix, TTT表示α-转弯,TT表示α-和β-转弯。gydF4y2BabgydF4y2Ba基于全长蛋白序列的91个含UBC结构域蛋白的系统发育树。不同类型的E2蛋白用不同的颜色表示。UEV:泛素偶联酶变体,RCE: rub -偶联酶,SCE: sumo -偶联酶,ELC: ELCH, UFC1:泛素-折叠修饰剂1偶联酶。gydF4y2BacgydF4y2Ba71种大豆泛素E2蛋白的两个代表性结构域组织。UBC:泛素偶联结构域,UBA:泛素相关结构域gydF4y2Ba
除泛素E2外,其他蛋白质如泛素偶联酶变异蛋白(UEV)、相关泛素(RUB)偶联酶(RCE)、小泛素样修饰物(SUMO)偶联酶(SCE)、ELCH (ELC同源物)和泛素折叠修饰物1偶联酶(UFC1)也含有UBC结构域[gydF4y2Ba19gydF4y2Ba].为了区分这些泛素E2蛋白,我们生成了大豆和拟南芥UBC结构域蛋白的系统发育(附加文件)gydF4y2Ba5gydF4y2Ba:图S2)。系统发育分析表明,在91个基因中,71个编码泛素E2蛋白,11个编码UEV蛋白(包括AtCOP10同源物),2个编码RUB E2蛋白(RCE), 4个编码推定的SUMO E2蛋白,1个编码ELC, 2个编码UFC1 E2蛋白(图。gydF4y2Ba3 bgydF4y2Ba).gydF4y2Ba
拟南芥泛素E2蛋白主要被细分为12组[gydF4y2Ba55gydF4y2Ba].此外,由于AtUBC37与番茄UBC37同源,被归为第XIII类[gydF4y2Ba19gydF4y2Ba].根据拟南芥和大豆中含UBC结构域蛋白的系统发育分析,大豆与拟南芥中的V E2s和AtUBC37族没有密切的同源物(附加文件)gydF4y2Ba5gydF4y2Ba:图S2)gydF4y2Ba.gydF4y2Ba因此,71个大豆泛素E2蛋白被分为11个组(附加文件gydF4y2Ba6gydF4y2Ba:图S3)。利用Pfam和NCBI数据库进行结构域组织分析表明,除Glyma.04G199200和Glyma.06G166300外,71个泛素E2蛋白仅含有一个UBC结构域(图2)。gydF4y2Ba3 cgydF4y2Ba).Glyma.04G199200和Glyma.06G166300在其c端还含有一个称为泛素相关(UBA)结构域(图2)。gydF4y2Ba3 cgydF4y2Ba).UBA结构域已被发现通过泛素分子的结合介导蛋白质-蛋白质相互作用[gydF4y2Ba56gydF4y2Ba].gydF4y2Ba
大豆基因组中含有E3s的RING-、U-box-和f -box结构域基因的鉴定gydF4y2Ba
为了鉴定大豆中编码RING-、U-box-和f -box型E3连接酶的基因,研究了这些结构域的HMM谱图(附加文件)gydF4y2Ba2gydF4y2Ba:表S2)从Pfam作为查询文件。通过HMMER分析,在大豆基因组中分别鉴定出RING、U-box和F-box蛋白的1234个、158个和579个同源蛋白gydF4y2Ba1gydF4y2Ba).为了验证这些已识别的蛋白质,所有FASTA格式的序列都被上传并在Pfam和NCBI数据库中进行搜索,分别检测RING、U-box和F-box结构域。将BLAST结果与Pfam和NCBI数据库相结合,在去除冗余序列后,获得了1034、145和547个编码假定的RING结构域-、U-box结构域-和F-box结构域蛋白质的基因gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
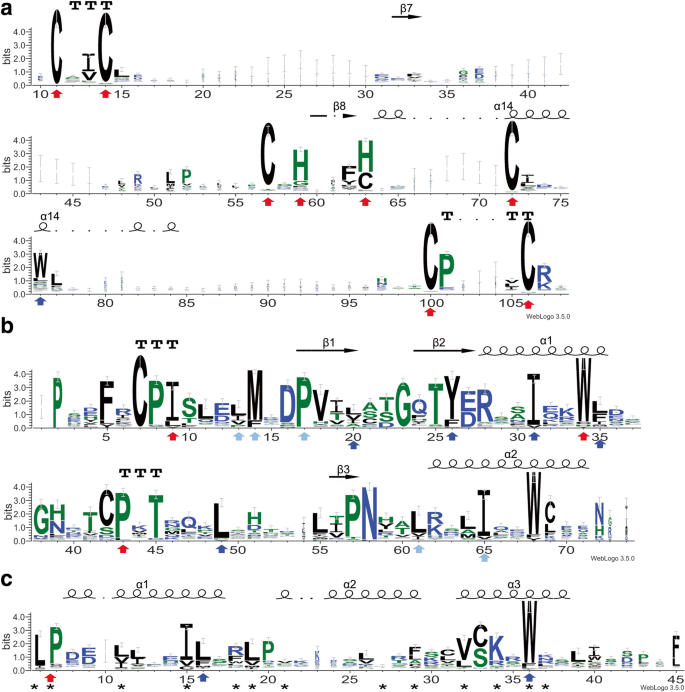
之前的结构和生化研究已经确定了RING的关键氨基酸残基和相应的二级结构[gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba,gydF4y2Ba59gydF4y2Ba], u盒[gydF4y2Ba32gydF4y2Ba,gydF4y2Ba60gydF4y2Ba]和F-box [gydF4y2Ba61gydF4y2Ba,gydF4y2Ba62gydF4y2Ba域。这些信息被用于进一步验证分别由1034个RING、145个U-box和547个F-box基因编码的每个蛋白质。通过这种人工验证,在大豆基因组中鉴定出760个RING基因、124个U-box基因和472个F-box基因,具有较高的可信度(表2)gydF4y2Ba1gydF4y2Ba).这些基因的详细信息列在附加文件中gydF4y2Ba3.gydF4y2Ba:表S3。各自域的代表序列被对齐(附加文件gydF4y2Ba7gydF4y2Ba:图S4,附加文件gydF4y2Ba8gydF4y2Ba:图S5和附加文件gydF4y2Ba9gydF4y2Ba:图S6)和使用Weblogo3在线生成的图形序列共识标识[gydF4y2Ba54gydF4y2Ba(图。gydF4y2Ba4gydF4y2Ba,gydF4y2BabgydF4y2Ba而且gydF4y2BacgydF4y2Ba).gydF4y2Ba
图形序列标识,表示已识别的相应类型大豆E3蛋白中RING、U-box和F-box结构域的序列模式。web logo是基于RING (gydF4y2Ba一个gydF4y2Ba)、u盒(gydF4y2BabgydF4y2Ba)及f盒(gydF4y2BacgydF4y2Ba)域。红色和蓝色箭头(gydF4y2Ba一个gydF4y2Ba)表示环结构域的锌结合残基和与同源E2s相互作用的保守疏水残基。红色箭头(gydF4y2BabgydF4y2Ba)表示疏水e2结合残基,蓝色和浅蓝色表示疏水相互作用两个核中的保守残基。红色和蓝色箭头(gydF4y2BacgydF4y2Ba)标记了有助于α1螺旋形成和f盒螺旋排列的保守残基,而标记有星号的残基则是人类Skp2与Skp1接触的重要位置gydF4y2Ba
一个典型的环具有一致的40-60个氨基酸C-X线性序列gydF4y2Ba2gydF4y2BacxgydF4y2Ba(9-39)gydF4y2BacxgydF4y2Ba(1 - 3)gydF4y2Ba-H-XgydF4y2Ba(2 - 3)gydF4y2BacxgydF4y2Ba2gydF4y2BacxgydF4y2Ba(4-48)gydF4y2BacxgydF4y2Ba2gydF4y2Ba-C,高度保守的半胱氨酸(C)和组氨酸(H)残基形成两个交叉支撑结构,结合两个锌离子,X可以是20个氨基酸中的任何一个[gydF4y2Ba57gydF4y2Ba].两种典型环型(C3H2C3和C3HC4)在第5个Cys残基上存在Cys或His,得到了很好的表征[gydF4y2Ba58gydF4y2Ba].我们从所有人工验证的大豆环结构域蛋白中提取了环结构域的序列。然后,我们使用Weblogo3算法在线对序列进行对齐并生成图形序列共识标识(图2)。gydF4y2Ba4gydF4y2Ba) [gydF4y2Ba54gydF4y2Ba].保守的Cys和His残基通过配位两个锌离子来稳定两个环区域,以及连接第一和第二环的中心保守α-螺旋,在序列consensus logos [gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba59gydF4y2Ba(图。gydF4y2Ba4gydF4y2Ba).还发现了常在α-螺旋区域发现的色氨酸或其他疏水残基,并与e2相互作用有关[gydF4y2Ba30.gydF4y2Ba(图。gydF4y2Ba4gydF4y2Ba).与RING结构域不同,U-box结构域缺乏锌结合位点。提出了含有疏水氨基酸和极性氨基酸的氢键网络来维持U-box支架[gydF4y2Ba32gydF4y2Ba].Weblogo3算法利用已识别的大豆U-box结构域序列生成的一致性序列在二级结构上显示出2个α-螺旋和3条β-链,这与已知植物U-box结构域的一致性结构一致,如拟南芥U-box蛋白AtPUB14 [gydF4y2Ba60gydF4y2Ba(图。gydF4y2Ba4 bgydF4y2Ba).在U-box结构域中,3个疏水E2结合位点和2个疏水核对于U-box结构域的功能至关重要[gydF4y2Ba60gydF4y2Ba].这些氨基酸残基也在Weblogo3生成的大豆U-box结构域一致序列中被识别出来(图2)。gydF4y2Ba4 bgydF4y2Ba).gydF4y2Ba
F-box结构域是F-box蛋白的特征结构,通过与Skp1相互作用作为SCF催化核心的亚基[gydF4y2Ba62gydF4y2Ba].一些已知有助于蛋白质相互作用和结构稳定性的保守残基被用于验证大豆F-box蛋白。在人类Skp2蛋白中,F-box结构域的标志性氨基酸残基Pro113有助于α-螺旋的启动,而Leu124和Try139有助于F-box螺旋的包装[gydF4y2Ba61gydF4y2Ba].这些氨基酸残基在大豆F-box蛋白中高度保守。gydF4y2Ba4摄氏度gydF4y2Ba).此外,人类Skp2蛋白的Skp1结合残基在大豆和其他植物含有F-box结构域的蛋白中是保守的[gydF4y2Ba63gydF4y2Ba,gydF4y2Ba64gydF4y2Ba,gydF4y2Ba65gydF4y2Ba(图。gydF4y2Ba4摄氏度gydF4y2Ba).gydF4y2Ba
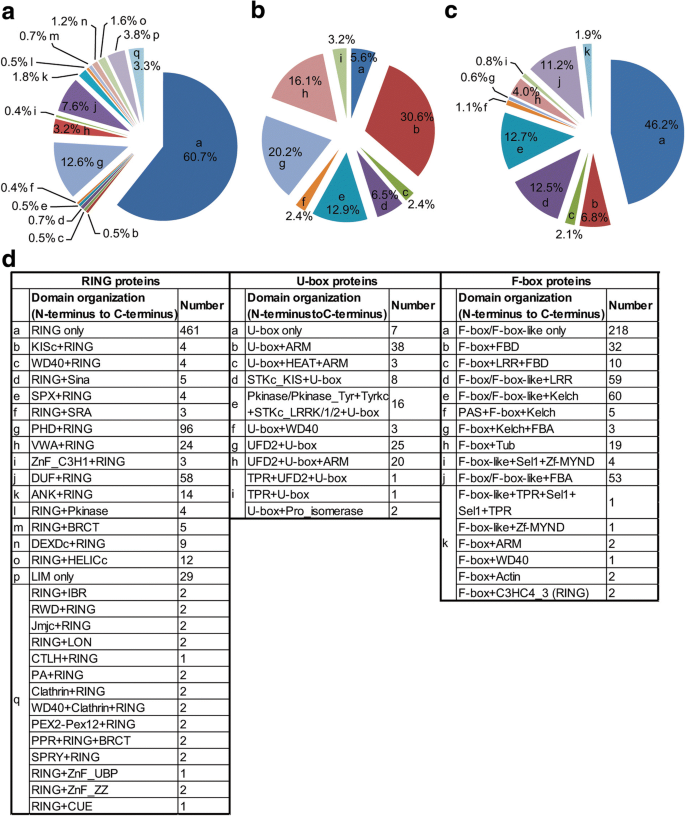
蛋白质结构域是一种特定蛋白质的保守结构,它可以独立于蛋白质的其他部分进化、发挥功能和存在。因此,分析结构域组织将为预测蛋白质的假定功能提供重要信息。为了进一步了解RING、U-box和F-box基因在大豆中的多样性,还对这些基因的对应蛋白进行BLAST比对Pfam和NCBI数据库,以鉴定这些蛋白中存在的其他结构域。结果表明,大约一半的RING蛋白(60.7%)和F-box蛋白(46.2%)不包含其他已知结构域,而U-box蛋白仅为5.6%(图2)。gydF4y2Ba5gydF4y2Ba).除了RING、U-box和F-box结构域外,在含有RING、U-box和F-box结构域的蛋白质中分别发现了28、10和13种其他类型的蛋白质结构域(图2)。gydF4y2Ba5 dgydF4y2Ba).我们的分析显示,一个额外的已知结构域可能出现在不同类型的E3蛋白中,而一个特定的E3蛋白可能包含多个已知结构域。例如,WD40重复序列域在RING、U-box和F-box蛋白中被发现(图2)。gydF4y2Ba5 dgydF4y2Ba).为了了解所鉴定的E3蛋白的进化关系/同源性,利用大豆RING、U-box和F-box蛋白的全长序列分别构建了系统发育树(补充文件)gydF4y2Ba10gydF4y2Ba:图S7,附加文件gydF4y2Ba11gydF4y2Ba:图S8和附加文件gydF4y2Ba12gydF4y2Ba:图S9)。接下来,分别用MCScanX分析编码RING-和F-box-type E3连接酶的基因家族中的基因复制事件[gydF4y2Ba66gydF4y2Ba].分析结果显示,543个RING基因(占总RING基因的71.4%)来自全基因组复制/节段复制的同源染色体区域,而只有24个(占总RING基因的3.2%)来自串联复制。对于F-box基因,发现181个(占F-box基因总数的38.3%)为WGD/片段重复,而86个(占F-box基因总数的18.2%)为串联重复(附加文件)gydF4y2Ba13gydF4y2Ba:图S10和附加文件gydF4y2Ba14gydF4y2Ba:图S11和附加文件gydF4y2Ba15gydF4y2Ba:表S4)。这些结果表明,在这些大豆基因家族中,WGD/片段重复主要参与了基因扩增。gydF4y2Ba
大豆泛素E1蛋白和大多数检测的泛素e2具有酶活性gydF4y2Ba
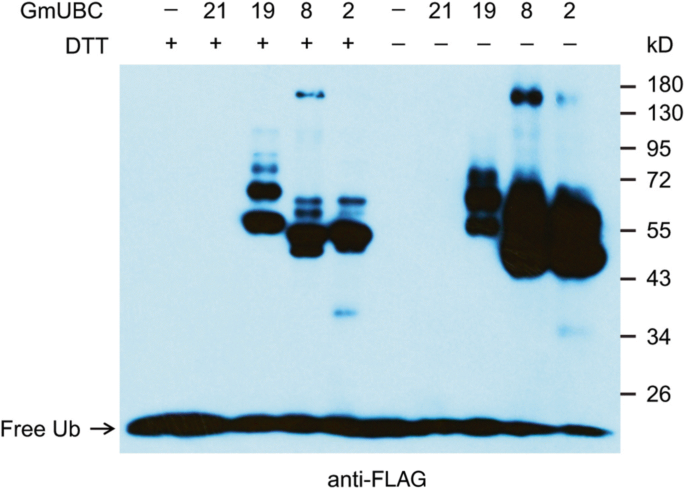
为了确定确定的大豆E1和E2基因是否编码活性泛素激活酶和泛素偶联酶,我们克隆了一个E1基因,gydF4y2BaGlyma.14G196800gydF4y2Ba,以及4个E2基因,gydF4y2BaGlyma.17G098000gydF4y2Ba,gydF4y2BaGlyma.09G273100gydF4y2Ba,gydF4y2BaGlyma.12G021800gydF4y2Ba而且gydF4y2BaGlyma.04G081200gydF4y2Ba(无花果。gydF4y2Ba1gydF4y2Ba;额外的文件gydF4y2Ba5gydF4y2Ba:图S2),表达重组蛋白gydF4y2Ba大肠杆菌gydF4y2Ba(gydF4y2Ba大肠杆菌gydF4y2Ba).我们将E1基因命名为gydF4y2BaGmUBA1gydF4y2Ba因为它是第一个在大豆中被鉴定和鉴定的泛素E1。E2基因被命名gydF4y2BaGmUBC2gydF4y2Ba(gydF4y2BaGlyma.17G098000gydF4y2Ba),gydF4y2BaGmUBC8gydF4y2Ba(gydF4y2BaGlyma.09G273100gydF4y2Ba),gydF4y2BaGmUBC19gydF4y2Ba(gydF4y2BaGlyma.12G021800gydF4y2Ba),gydF4y2BaGmUBC21gydF4y2Ba(gydF4y2BaGlyma.04G081200gydF4y2Ba)基于它们与拟南芥的同源性gydF4y2BaUBC2gydF4y2Ba,gydF4y2BaUBC8gydF4y2Ba,gydF4y2BaUBC19gydF4y2Ba而且gydF4y2BaUBC21gydF4y2Ba基因,分别[gydF4y2Ba20.gydF4y2Ba](附加文件gydF4y2Ba5gydF4y2Ba:图S2)。我们成功纯化了所有克隆基因的重组蛋白(附加文件)gydF4y2Ba16gydF4y2Ba:图S12)。然后将纯化的E1和E2蛋白用于体外硫酯试验,以检测其酶活性[gydF4y2Ba19gydF4y2Ba].在硫酯分析中,E1酶激活游离的泛素分子,以依赖于atp的方式形成硫酯连接的泛素。然后,硫酯连接的泛素转移到活性E2酶上,形成E2-泛素加合物,该加合物对还原剂二硫苏糖醇(DTT)敏感[gydF4y2Ba20.gydF4y2Ba].如图所示。gydF4y2Ba6gydF4y2Ba,除GmUBC21外,其余GmUBC2、8和19均与泛素形成了对100 mM DTT敏感的加合物,表明在GmUBA1存在下形成了硫酯连接。这些结果表明,GmUBA1是活性泛素E1酶,GmUBC2、8和19具有泛素偶联活性。gydF4y2Ba
随机选择大豆RING和U-box基因编码具有E3泛素连接酶活性的蛋白质gydF4y2Ba
先前的研究表明,含有RING和U-box结构域的蛋白一般具有E3泛素连接酶的功能[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba29gydF4y2Ba,gydF4y2Ba67gydF4y2Ba].F-box蛋白作为SCF (Skp1-Cullin-F-box)泛素连接酶复合物的一个亚基。不幸的是,目前尚无体外检测SCF复合物E3连接酶活性的方法。为了确定假定的大豆RING和U-box蛋白是否能够催化蛋白质泛素化,我们进行了体外泛素化实验。为此,4个RING蛋白编码基因,gydF4y2BaGlyma.04G235700gydF4y2Ba,gydF4y2BaGlyma.17G094000gydF4y2Ba,gydF4y2BaGlyma.15G001100gydF4y2Ba而且gydF4y2BaGlyma.10G24100gydF4y2Ba和4个U-box蛋白编码基因,gydF4y2BaGlyma.20G013200gydF4y2Ba,gydF4y2BaGlyma.11G140100gydF4y2Ba,gydF4y2BaGlyma.19G199300gydF4y2Ba,gydF4y2BaGlyma.04G179300gydF4y2Ba随机选择克隆,表达并纯化gydF4y2Ba大肠杆菌gydF4y2Ba.u型盒基因被命名gydF4y2BaGmPUB10gydF4y2Ba(gydF4y2BaGlyma.20G013200gydF4y2Ba),gydF4y2BaGmPUB13gydF4y2Ba(gydF4y2BaGlyma.11G140100gydF4y2Ba),gydF4y2BaGmPUB22gydF4y2Ba(gydF4y2BaGlyma.19G199300gydF4y2Ba),gydF4y2BaGmPUB38gydF4y2Ba(gydF4y2BaGlyma.04G179300gydF4y2Ba)基于他们的同源性gydF4y2Ba拟南芥PUB10gydF4y2Ba[gydF4y2Ba68gydF4y2Ba),gydF4y2BaPUB13gydF4y2Ba[gydF4y2Ba69gydF4y2Ba),gydF4y2BaPUB22gydF4y2Ba[gydF4y2Ba70gydF4y2Ba),gydF4y2BaPUB38gydF4y2Ba[gydF4y2Ba67gydF4y2Ba基因,分别(附加文件gydF4y2Ba17gydF4y2Ba:图S13)。通过大豆E1 GmUBA1、大豆E2 (GmUBC8、GmUBC2或GmUBC19)和泛素组成的体外泛素化试验检测克隆的每个RING或U-box蛋白。如图所示。gydF4y2Ba7gydF4y2Ba,除GmPUB38外,所有被测的RING和U-box E3连接酶均能在含有大豆E1 GmUBA1、E2 GmUBC8、游离泛素和缓冲液中必要辅因子的完整反应中催化形成高分子量的多泛素链,而在缺乏E1、E2、E3和泛素的对照反应中未检测到信号。此外,U-box蛋白GmPUB10也能够与GmUBC2协同作用,催化泛素化,这表明GmUBC2与GmPUB10之间存在E2-E3特异性(图2)。gydF4y2Ba7gydF4y2Ba).然而,在任何GmUBC19作为E2的完全反应中都没有检测到多泛素链,这可能是因为所测试的E3s都不能与E2酶协同催化泛素化(图2)。gydF4y2Ba7gydF4y2Ba).同样,GmPUB38在体外泛素化中催化失败可能是由于没有GmUBC8, GmUBC2或GmUBC19是其E3活性的真正同源E2。这些结果表明,我们检测的大多数RING和U-box蛋白都表现出E3泛素连接酶活性,这验证了我们在基因组规模上鉴定大豆UBS核心成分的算法和协议。gydF4y2Ba
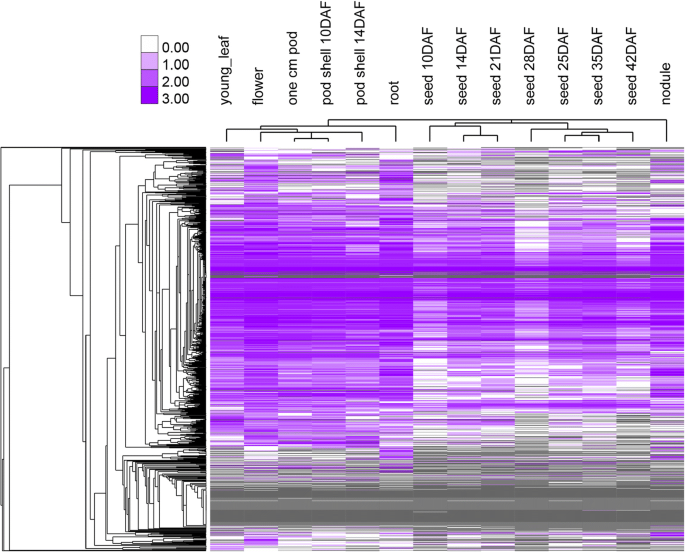
大豆UBS基因在植株发育和胁迫处理后的表达谱gydF4y2Ba
基因表达模式可以为基因功能提供重要信息。因此,我们利用之前在Soybase公开的RNA-seq数据探索了大豆UBS基因的表达模式[gydF4y2Ba71gydF4y2Ba].基于该数据集,我们发现1431个大豆UBS基因中有1034个(72.3%)的转录本在被检测的14个大豆植物组织中至少一种被检测到。我们的分析还显示在所有14个组织中有564个UBS基因组成性表达,其中UBA基因2个(占UBA基因总数的50%),UBC基因50个(占UBC基因总数的70.4%),RING基因305个(占RING基因总数的40.1%),U-box基因30个(占U-box基因总数的24.2%),F-box基因177个(占F-box基因总数的37.5%)(图5)。gydF4y2Ba8gydF4y2Ba和附加文件gydF4y2Ba18gydF4y2Ba:表S5)。这些结果表明,许多UBS基因可能参与了大豆的多个发育过程。gydF4y2Ba
大豆UBS基因在14个组织中的表达谱热图。RNA-seq数据下载自Soybase数据库[gydF4y2Ba71gydF4y2Ba].热图中的颜色刻度表示log-RPKM(每千碱基百万读数)值,在SoyBase中规范化。UBS基因表达模式的完整列表包含在附加文件中gydF4y2Ba18gydF4y2Ba表S5gydF4y2Ba
为了阐明大豆UBS基因在生物或非生物胁迫下的潜在作用,我们使用以前的RNA-seq数据集分析了基因表达,这些数据来自大豆植物受到各种胁迫处理的实验[gydF4y2Ba72gydF4y2Ba,gydF4y2Ba73gydF4y2Ba,gydF4y2Ba74gydF4y2Ba].如果处理株与对照株之间的褶皱变化≥2或≤0.5,则认为基因存在差异表达gydF4y2BaPgydF4y2Ba-值小于0.05(或接种根瘤菌菌株的数据集中错误发现率≤0.001)。鉴定出的上调和下调基因见附加文件gydF4y2Ba19gydF4y2Ba:图S14和附加文件gydF4y2Ba20.gydF4y2Ba:表S6。在非生物胁迫(干旱和盐胁迫)处理后,196、45和112个大豆USB基因的转录水平显著改变。gydF4y2Ba尖孢镰刀菌gydF4y2Ba和根瘤菌株分别接种。其中,41个基因在干旱和盐胁迫下表达均受到显著影响,12个基因对干旱和盐胁迫均有响应gydF4y2Ba病圃gydF4y2Ba还有根瘤菌菌株。进一步分析生物和非生物胁迫之间的重叠,发现3环型E3连接酶的转录水平(gydF4y2BaGlyma.03G215500gydF4y2Ba,gydF4y2BaGlyma.06G150400gydF4y2Ba而且gydF4y2BaGlyma.12G112000gydF4y2Ba)在所有压力测试下都发生了显著变化。gydF4y2Ba
经SCN处理后,大豆许多UBS基因的表达水平发生了显著变化gydF4y2Ba
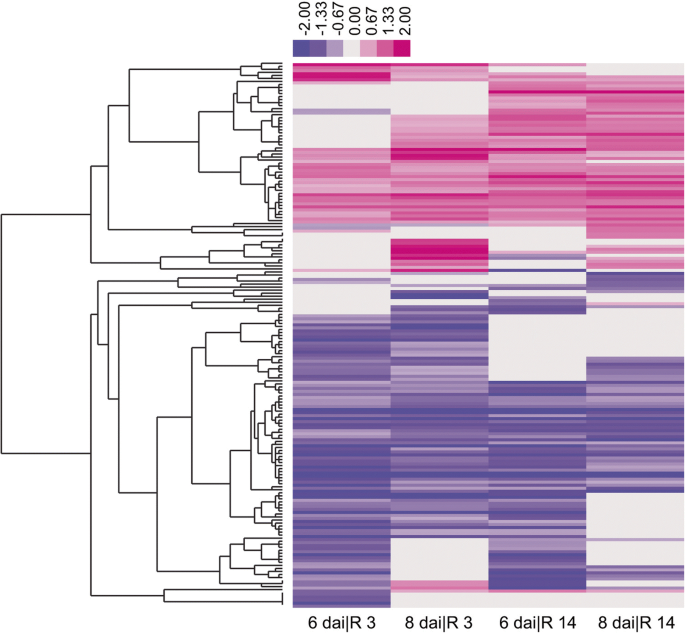
近年来,泛素化已成为植物对许多不同病原体免疫的关键调控机制[gydF4y2Ba75gydF4y2Ba,gydF4y2Ba76gydF4y2Ba,gydF4y2Ba77gydF4y2Ba].为了阐明大豆UBS在调节宿主免疫中的可能作用和机制基础,我们探索了公开的RNA-seq数据集,以检测大豆UBS基因在SCN处理下的转录谱[gydF4y2Ba78gydF4y2Ba].这种分析将有助于识别大豆UBS的关键候选基因,这些候选基因与宿主免疫有关。在产生RNA-seq数据的研究中[gydF4y2Ba78gydF4y2Ba],分别对导致宿主敏感和耐药反应的毒SCN (Race 3, R3)和无毒SCN (Race 14, R14)接种(dai)后6天和8天的大豆根系转录组进行测序,随后将其与未接种SCN的大豆根系的转录组进行比较(作为基线对照)。因此,通过使用参数每千碱基百万(RPKM)比较实验和未接种的样品,得到了四组不同处理的数据(6组|R3, 8组|R3, 6组|R14, 8组|R14)。gydF4y2Ba78gydF4y2Ba].我们将具有显著差异表达的大豆UBS基因定义为具有agydF4y2Ba日志gydF4y2Ba2gydF4y2Ba在任何数据集中RPKM≥1或≤- 1(即RPKM变化超过2倍)。用截断点2gydF4y2Ba-gydF4y2Ba在RPKM中,我们发现接种SCN后180个大豆UBS基因的转录水平显著改变,约占大豆UBS基因的12.6%(图5)。gydF4y2Ba9gydF4y2Ba;额外的文件gydF4y2Ba21gydF4y2Ba:表S7)。这些基因包括22个UBC基因(占UBC基因总数的31.0%)、91个RING基因(占RING基因总数的12.0%)、36个U-box基因(占U-box基因总数的29.0%)和31个F-box基因(占F-box基因总数的6.6%)。gydF4y2Ba
SCN处理后差异表达UBS基因的热图。利用之前的RNA-seq数据集分析大豆UBS基因的表达[gydF4y2Ba78gydF4y2Ba].大豆全根独立接种SCN群体NH1-RHg (Race 3)和TN8 (Race 14)后6、8天分别记为6代| r3、8代| r3、6代| r14和8代| r14。色标表示将接种样品中基因的表达值与未接种样品中基因的表达值进行比较计算得到的对数比。这些差异表达的UBS基因的完整列表包含在附加文件中gydF4y2Ba21gydF4y2Ba表S7gydF4y2Ba
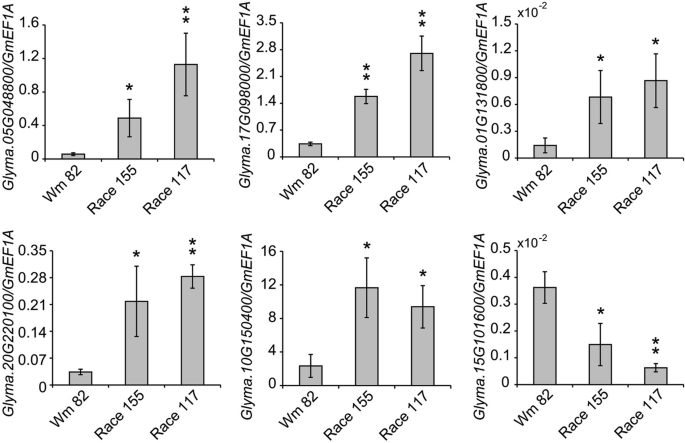
为了确认RNA-seq结果的可靠性,我们从180个大豆UBS基因中随机选取了10个基因,并通过实时定量PCR (real time qPCR)分析进一步检测了SCN处理后的表达情况。总RNA来源于scn敏感大豆。两种大豆囊线虫(SCN,gydF4y2Ba异皮线虫属甘氨酸gydF4y2BaIchinohe)居群,种族155 (HG型2.5.7)和种族117 (HG型1.2.3.5.6.7)。如图所示。gydF4y2Ba10gydF4y2Ba,实时qPCR检测6个大豆UBS基因在SCN处理后表达水平显著改变。它们表达的变化趋势(即增加或减少)与RNA-seq数据一致,尽管确切的变化幅度不同。然而,在SCN处理后,我们通过实时qPCR观察到其他四个基因的表达水平没有明显变化(附加文件gydF4y2Ba22gydF4y2Ba:图S15)。基于这些结果,我们推测在大豆与scn相互作用过程中,约有100个大豆UBS基因(约180个基因中的60%)的表达水平可能发生显著改变。综合RNA-seq和实时qPCR分析的结果,可以得出这样的结论:大豆的数十个UBS基因可能参与了宿主免疫或SCN感染易感性的调节。gydF4y2Ba
通过实时定量PCR (qPCR)对RNA-seq分析显示的大豆UBS基因差异表达模式进行了验证。用两种大豆囊线虫(SCN)分别接种3周龄大豆威廉姆斯82 (wm82)的根系,gydF4y2Ba异皮线虫属甘氨酸gydF4y2BaIchinohe)居群,155种(HG型2.5.7)和117种(HG型1.2.3.5.6.7)进行1周,然后收集RNA提取。以未接种的Wm82植株根部为对照。用大豆进行实时qPCR实验gydF4y2BaEF1agydF4y2Ba基因(gydF4y2BaGmEF1agydF4y2Ba)为内控,并以三个独立实验的平均值±SD表示。采用Student 's t检验(*gydF4y2BapgydF4y2Ba< 0.05, **gydF4y2BapgydF4y2Ba< 0.01)gydF4y2Ba
讨论gydF4y2Ba
对编码大豆UBS核心成分的基因进行全基因组鉴定将是进一步研究这些基因在大豆中的功能特征的重要一步。先前的研究报道了大豆中几个单独的泛素E3连接酶基因家族,包括HECT [gydF4y2Ba45gydF4y2Ba], RBR (RING的子集)[gydF4y2Ba46gydF4y2Ba], u型盒[gydF4y2Ba44gydF4y2Ba和F-box [gydF4y2Ba43gydF4y2Ba)基因。然而,系统地鉴定和分析编码整个大豆核心成分的基因(gydF4y2Ba大豆gydF4y2Ba)泛素系统(UBS)迄今尚未在大豆中进行过研究。为了弥补这一知识空白,本研究对大豆UBS基因进行了全面的鉴定和分析。我们利用最新的大豆基因组数据库Wm82.a2.v1,从大豆基因组中确定了3个泛素E3连接酶家族的4个泛素e1编码基因、71个泛素e2编码基因和1356个编码基因(包括760个RING基因、124个U-box基因和472个F-box基因),具有较高的可信度。gydF4y2Ba
一般来说,泛素E1酶是分子量为110-125 kDa的单体蛋白,包含两个参与腺苷酸化的ThiF基体[gydF4y2Ba79gydF4y2Ba].在本研究中,我们从大豆基因组中鉴定出20个编码含ThiF基序蛋白的基因。然而,这些基因中只有4个编码的蛋白质预测分子量超过110 kDa,并且存在e1特异性催化Cys结构域(UBA_e1_thiolCys)和泛素折叠结构域(UFD)。UBA_e1_thiolCys结构域,也称为SCCH [gydF4y2Ba12gydF4y2Ba]含有一个负责泛素硫酯连接的半胱氨酸残基,而UFD赋予E1招募泛素e2的特异性[gydF4y2Ba20.gydF4y2Ba].系统发育分析表明,4个大豆基因编码的ThiF基序- ccd - ufd结构域蛋白在拟南芥和人体内也属于已知泛素E1s的同一分支。我们还从大豆中91个含UBC结构域的基因中鉴定出71个泛素E2基因。根据拟南芥对应物的分组,将71个泛素e2分为11组,除V外,I-XII [gydF4y2Ba55gydF4y2Ba].我们没有在大豆基因组中发现AtUBC37的同源物。在本研究鉴定的大豆e2列表中,UBC37和V族e2的缺失可能是由于基因组未完全测序,导致这些基因的序列未在大豆基因组数据库中显示,或者基因组中确实不存在这类e2。大豆泛素E1基因编码的蛋白质(gydF4y2BaGmUBA1gydF4y2Ba)和四个泛素E2基因(gydF4y2BaGmUBC2gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba19gydF4y2Ba而且gydF4y2Ba21gydF4y2Ba)进行体外硫酯含量测定。结果证明GmUBA1作为活性E1酶激活游离泛素形成硫酯连接的泛素。然后将硫酯连接的泛素转移到活性E2酶(GmUBC2, 8和19)上,生成硫酯连接的E2-泛素加合物。gydF4y2Ba
泛素E3连接酶是泛素系统中最多样化的一类,负责泛素化的底物特异性。根据作用机制和结构特征,E3连接酶可分为HECT、RING和u -box型等单亚基[gydF4y2Ba31gydF4y2Ba]和多亚基包括SCF (Skp1-Cullin-F-box)、Cullin-Elongin-BC-VHL (CBC VHL)和APC (anphase Promoting Complex)类型[gydF4y2Ba38gydF4y2Ba].我们没有在本研究中鉴定大豆E3s的HECT型,因为之前的进化分析表明,HECT基因的数量在不同的植物基因组中保持相当稳定,在大豆基因组中预测有19个[gydF4y2Ba28gydF4y2Ba,gydF4y2Ba45gydF4y2Ba].相反,我们专注于构成植物基因组中E3最大群的三种类型的E3 (RING, U-box和F-box)。在本研究中,大豆基因组中鉴定出760个RING基因,这几乎是拟南芥、人类和小鼠等真核生物基因组中分别编码469、385和305个RING蛋白的2倍[gydF4y2Ba80gydF4y2Ba,gydF4y2Ba81gydF4y2Ba].最近,有24个基因编码RBR (RING1-IBR-RING2;从大豆基因组中鉴定出含有结构域的蛋白[gydF4y2Ba46gydF4y2Ba].这些RBR基因在我们的HMMER3.1分析中鉴定出的1234个RING基因中gydF4y2Ba3.gydF4y2Ba:表S3)。但是这些RBR基因中只有5个在BLAST比对NCBI和Pfam数据库生成的列表中,由于其RING结构域高度非典型,人工验证后均未进入列表。作为culling -RING-like (CRL)型多亚基泛素E3s的亚基的RING蛋白和RBR蛋白在人工验证后也被排除在我们最终的大豆RING E3s列表中。拟南芥和水稻(gydF4y2BaOryzae漂白亚麻纤维卷gydF4y2Ba)基因组分别包含64个和77个预测的U-box蛋白[gydF4y2Ba29gydF4y2Ba,gydF4y2Ba82gydF4y2Ba].最近,在大豆基因组中发现了125个U-box基因[gydF4y2Ba44gydF4y2Ba].我们的分析确定了124个U-box基因,其中119个(96%)在该研究中报道的列表中[gydF4y2Ba44gydF4y2Ba],我们的研究发现了5个额外的U-box基因,但在前一项研究中没有发现,该研究报告的另外3个U-box基因在人工验证后从我们的列表中删除gydF4y2Ba3.gydF4y2Ba:表S3) [gydF4y2Ba44gydF4y2Ba].仔细观察三个被淘汰的U-box基因,发现这些基因编码的蛋白质中缺失了U-box结构域n端高度保守的氨基酸残基(数据未显示)。在植物中,已在拟南芥、水稻、流行的(gydF4y2Ba杨树trichocarpagydF4y2Ba)和葡萄(gydF4y2Ba葡萄gydF4y2Ba),分别[gydF4y2Ba39gydF4y2Ba,gydF4y2Ba83gydF4y2Ba].在本研究中,鉴定出472个F-box基因,其中440个(93%)也在最近的一项研究中被另一组鉴定出[gydF4y2Ba43gydF4y2Ba].然而,本研究中发现的32个F-box基因在该研究中被遗漏,而该研究中报道的64个F-box基因在本研究中经过人工验证后从我们的最终列表中删除,尽管它们在HMMER 3.1分析后实际上在列表中(附加文件)gydF4y2Ba3.gydF4y2Ba:表S3)。我们在本研究中鉴定的绝大多数E3基因与先前报告的重叠表明了我们用于全基因组基因鉴定的算法的有效性。另一方面,我们在本研究中结合了HMMER 3.1分析、NCBI和Pfam数据库中的蛋白质结构域检测工具,以及人工验证来鉴定感兴趣的基因,这比以前的研究更严格,这些研究只涉及HMMER分析和/或BLAST,这可能解释了为什么这些研究中确定的一些基因不在我们的最终列表中。gydF4y2Ba
到目前为止,一些大豆环蛋白和U-box蛋白已被证明具有E3泛素连接酶活性[gydF4y2Ba44gydF4y2Ba,gydF4y2Ba84gydF4y2Ba,gydF4y2Ba85gydF4y2Ba].然而,在这些研究中,商业上可用的非大豆E1和e2用于体外泛素化测定,以检查E3活性。在本研究中,一个泛素E1基因gydF4y2BaGmUBA1gydF4y2Ba和三个E2基因gydF4y2BaGmUBC2gydF4y2Ba,gydF4y2Ba8gydF4y2Ba而且gydF4y2Ba19gydF4y2Ba硫酯实验证明GmUBC21能编码活性的泛素E1和E2酶,但不能与泛素形成加合物。类似地,拟南芥GmUBC2, 8和19的同源物,AtUBC2, 8和19也被证明携带硫酯连接的泛素[gydF4y2Ba20.gydF4y2Ba]但AtUBC21在硫酯检测中未显示E2活性[gydF4y2Ba20.gydF4y2Ba].利用大豆泛素系统的组分,用GmUBC8作为同源泛素E2酶,测试了4个RING蛋白和3个U-box蛋白是真正的E3泛素连接酶。与拟南芥AtPUB10相似,AtPUB10能够使用AtUBC2作为同源E2酶进行自泛素化[gydF4y2Ba68gydF4y2Ba],在大豆E2 GmUBC2存在的情况下,大豆GmPUB10也表现出E3活性。这些随机选择的大豆UBS蛋白具有酶活性的演示验证了我们用于在基因组规模上鉴定构成大豆UBS的组分的算法。gydF4y2Ba
基因表达分析可以为了解大豆UBS基因的潜在功能提供关键信息。因此,我们利用公开的RNA-seq数据集分析了UBS基因在植物发育过程中以及在非生物和生物胁迫下的表达谱。在被检测的14个大豆组织中,至少有一个可以检测到1034个UBS基因的转录本,这进一步表明了我们用于全基因组基因鉴定的算法的有效性。同时,338个大豆USB基因在非生物(干旱和盐)或生物(gydF4y2Ba病圃gydF4y2Ba和根瘤菌菌株)的胁迫处理,表明它们可能在这些过程中发挥作用。在生物胁迫中,SCN (gydF4y2Ba异皮线虫属甘氨酸gydF4y2Ba一虫病(Ichinohe)一直是全球大豆的主要害虫,每年造成大豆产量损失15-30%。育种和种植抗SCN品种是控制SCN最有效的策略[gydF4y2Ba86gydF4y2Ba].迄今为止,关于大豆抗SCN和其他病原体免疫中泛素系统(UBS)的研究非常有限。为了扩大我们对大豆免疫中泛素化相关基因功能的理解,我们利用公开的RNA-seq数据集检测了SCN处理后它们的表达谱[gydF4y2Ba78gydF4y2Ba].通过对RNA-seq数据的分析,发现大豆UBS基因在SCN孵育后转录本丰度发生了显著变化,其中包括22个E2基因和158个E3基因。在RNA-seq分析鉴定的基因中,随机抽取10个基因中的6个,用scn敏感大豆cv进行实时qPCR验证。与SCN孵育后威廉斯82。这些结果支持了UBS可能在大豆抗SCN免疫中发挥重要作用的观点。迄今为止,大多数抗SCN的大豆品种都来自有限的抗性来源,SCN品种已开始进化以克服抗性[gydF4y2Ba86gydF4y2Ba].因此,工程新型SCN耐药可能是管理SCN感染的一个有趣的策略。为此,下一步的工作应该是确定本研究确定的大豆UBS中在大豆免疫中起关键作用的成员。鉴于泛素化在植物生长、发育以及生物和非生物胁迫反应的调控中无处不在,本研究中确定的大豆UBS组分的进一步功能表征也将有助于深入了解许多其他植物生理过程。gydF4y2Ba
结论gydF4y2Ba
本研究采用生物信息学方法系统地鉴定了大豆泛素系统(UBS)核心组分的编码基因。从大豆基因组中共鉴定出4个泛素E1基因、71个泛素E2基因和1356个E3连接酶基因。UBS蛋白数量如此之多、种类如此之多,说明泛素靶特异性修饰是大豆细胞和生理调控中复杂而重要的组成部分。随机选取大豆E1、E2和E3基因编码的十多个蛋白进行生化测试,并对其中大部分蛋白的酶活性进行了验证。结合RNA-seq数据和实时qPCR结果分析表明,在SCN处理后,大豆大量UBS基因的表达水平发生了显著变化,这表明UBS组分参与了大豆-SCN的相互作用。本研究为进一步研究大豆UBS基因在多种生理过程中的功能特性,包括其在大豆抗SCN免疫调控中的作用和潜在的分子机制,奠定了基础和基本框架。gydF4y2Ba
方法gydF4y2Ba
大豆UBS基因的鉴定gydF4y2Ba
以ThiF基序的一致性序列为查询对象,结合BLASTP算法,利用最新的大豆蛋白质组数据库(Phytozome 12.1,gydF4y2Bahttps://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_GmaxgydF4y2Ba).ThiF基序的一致序列(PF00899)从NCBI CDD数据库(gydF4y2Bahttp://www.ncbi.nlm.nih.gov/cdd/gydF4y2Ba).为确认所获得的蛋白质,Pfam数据库(gydF4y2Bahttp://pfam.xfam.orggydF4y2Ba/) (gydF4y2Ba50gydF4y2Ba]用于进一步检测候选蛋白中ThiF基序的存在。gydF4y2Ba
为了鉴定大豆中泛素E2酶和E3连接酶的潜在成员,HMM profile(附加文件)gydF4y2Ba2gydF4y2Ba:表S2)对应域从Pfam数据库中下载。HMMER3.1 [gydF4y2Ba87gydF4y2Ba程序在大豆蛋白质组数据库(Wm82.a2.v1)中进行检索。gydF4y2Bahttp://www.soybase.org/gydF4y2Ba) [gydF4y2Ba47gydF4y2Ba,gydF4y2Ba71gydF4y2Ba使用这些HMM配置文件作为查询。根据HMMER搜索结果从Soybase中提取完整的蛋白质序列,然后提交到Pfam和NCBI CDD数据库以验证感兴趣的结构域的存在。为了最终确定这些预测蛋白,我们对候选蛋白的感兴趣域序列和从CDD数据库下载的相应一致序列进行了手工验证。那些缺乏高度保守的关键氨基酸或二级结构的蛋白质被排除在最终的数据集中。gydF4y2Ba
根据在Phytozome的大豆基因组数据库中BLASTP搜索的结果,我们获得了染色体位置、cDNA序列、CDS序列、蛋白质序列和替代剪接事件的信息。用ProtParam计算分子量(gydF4y2Bahttp://web.expasy.org/protparam/gydF4y2Ba).表达序列标签(EST)通过NCBI blast进行识别。如果一个基因在Soybase中存在多个转录本,则所有后续分析都使用主转录本。gydF4y2Ba
系统发育,序列保存和基因重复分析gydF4y2Ba
采用MEGA6程序中的Neighbor-joining (NJ)方法构建了系统发育树,参数为gydF4y2BapgydF4y2Ba-距离,部分删除处理的间隙,1000次自举重复[gydF4y2Ba88gydF4y2Ba].gydF4y2Ba
为了分析目标结构域的序列特征,基于NCBI blast结果提取预测蛋白中相应结构域的序列,并从CDD数据库中下载UBC、RING、U-box和F-box结构域的一致序列。多序列比对采用CLUSTAL2.1 [gydF4y2Ba89gydF4y2Ba],并使用ESPript3 (gydF4y2Bahttp://espript.ibcp.fr/ESPript/cgi-bin/ESPript.cgigydF4y2Ba) [gydF4y2Ba90gydF4y2Ba]和BoxShade (gydF4y2Bahttp://www.ch.embnet.org/software/BOX_form.htmlgydF4y2Ba) [gydF4y2Ba91gydF4y2Ba].二级结构也由ESPript3根据参考序列生成。序列标识是使用在线程序WebLogo3 (gydF4y2Bahttp://weblogo.threeplusone.com/create.cgigydF4y2Ba) [gydF4y2Ba54gydF4y2Ba],使用默认参数。gydF4y2Ba
为了检测所鉴定蛋白的结构域组织,FASTA格式的蛋白氨基酸序列在Pfam和CDD数据库中进行检索。提取保守域信息用于分析域组织。gydF4y2Ba
为了分析RING和F-box共线对偶,采用MCScanX,如前所述[gydF4y2Ba92gydF4y2Ba].通过标准设置的动态规划确定最高评分路径。根据匹配命中的数量以及在染色体和支架中的位置,将基因位点分为全基因组复制(WGD) /节段复制、串联复制、近端复制或分散复制。gydF4y2Ba
蛋白表达及纯化gydF4y2Ba
将所选基因的全长编码序列利用Gateway克隆系统(Invitrogen)克隆到pDEST15载体上,并转化到gydF4y2Ba大肠杆菌gydF4y2Ba菌株BL21 (DE3)。用于本试验的引物列在附加文件中gydF4y2Ba18gydF4y2Ba:表S5。gst标记的融合蛋白在BL21中表达,并使用Glutathione Sepharose 4 Fast Flow beads (GE Healthcare)按照制造商提供的方案进行纯化。简单来讲,gydF4y2Ba大肠杆菌gydF4y2Ba离心收集细胞,用裂解缓冲液(50 mM Tris-HCl (pH 7.5), 100 mM NaCl, 1 mM EDTA, 1% Triton X-100, 1 mg/mL溶菌酶和鸡尾酒)悬浮,用超声波仪破坏。纯化时,将200 μL谷胱甘肽Sepharose 4 Fast Flow珠加入清除的上清液中,在旋转器上4℃孵育过夜。珠粒用10 mL洗涤缓冲液(1 × PBS, 1 mM EDTA和0.5% Triton X-100)洗涤3次,然后用4 mL洗脱缓冲液(10 mM还原谷胱甘肽在50 mM Tris-HCl pH 8.8中)洗脱。使用Amicon离心过滤器(Millipore)将纯化的蛋白质进一步脱盐并浓缩在蛋白质存储缓冲液(50 mM Tris-HCl, pH 8, 50 mM KCl, 0.1 mM EDTA, 1 mM DTT和0.5 mM PMSF)中。将甘油添加到重组蛋白中,最终浓度为40%,在−80°C保存,直到使用为止。用蛋白测定剂(Bio-Rad)测定纯化蛋白的浓度。gydF4y2Ba
硫代酸酯测定gydF4y2Ba
如前所述,E1泛素激活活性和E2泛素偶联活性由体外硫酯测定[gydF4y2Ba19gydF4y2Ba].反应总体积为20 μL,由20 mM Tris-HCl (pH 7.5), 10 mM MgCl组成gydF4y2Ba2gydF4y2Ba,和1mm ATP。将40 ng大豆E1 (GST-GmUBA1)与2 μg flagu -ubiquitin在20 μL的反应中预孵育10分钟,加入约100 ng gst -融合E2蛋白,持续15分钟。孵育后将反应分成两个半体积,加入含有100 mM二硫苏糖醇(DTT)的SDS样品缓冲液或不含DTT的4 M尿素样品缓冲液(−)终止反应。在使用ECL试剂盒(Pierce,现为赛默飞世尔)检测之前,用小鼠单克隆抗flag m2 -过氧化物酶偶联抗体(Sigma-Aldrich)检测这些反应。gydF4y2Ba
体外泛素化试验gydF4y2Ba
体外泛素化实验如前所述[gydF4y2Ba19gydF4y2Ba].简单地说,在泛素化缓冲液(50 mM Tris-HCl (pH 7.5)、5 mM ATP、5 mM MgCl)中,以30 μL、40 ng大豆E1 (GST-GmUBA1)、约100 ng gst -融合E2、2 μg GST-E3连接酶和2 μg泛素结合gydF4y2Ba2gydF4y2Ba、2 mM DTT、3 mM磷酸肌酸、5 μg/mL磷酸肌酸激酶)。30℃1.5 h后,加入100 mM DTT的SDS装样缓冲液,100℃煮沸5 min,反应产物用10% SDS- page凝胶分离,用小鼠单克隆抗泛素m2 -过氧化物酶偶联抗体(Sigma-Aldrich)免疫印迹检测。gydF4y2Ba
SCN接种后的植物材料及基因表达分析gydF4y2Ba
用两种大豆囊线虫(SCN,gydF4y2Ba异皮线虫属甘氨酸gydF4y2BaIchinohe)居群,种族155 (HG型2.5.7)和种族117 (HG型1.2.3.5.6.7)。接种SCN一周后,收集3株大豆的根系,立即在液氮中冷冻,研磨成细粉,用于RNA提取。收集3株未接种的Williams 82植株的根系作为阴性对照。gydF4y2Ba
总RNA使用RNeasy Plant Mini Kit与DNase处理(Qiagen)按照制造商的程序提取。然后使用2微克总RNA作为模板,在SuperScript III逆转录酶和oligo (dT)引物(Life Technologies)的存在下合成第一链cDNA。用消毒过的ddH将cDNA群体稀释10倍gydF4y2Ba2gydF4y2BaO,然后用于实时定量PCR (qPCR)。实时qpcr在LightCycler 480仪器II (Roche)上进行,使用SYBR Green (Life Technologies)和基因特异性引物。大豆gydF4y2BaEF1agydF4y2Ba的基因,gydF4y2BaGmEF1agydF4y2Ba(gydF4y2BaGlyma.19G052400gydF4y2Ba)被用作内部控制(附加文件gydF4y2Ba23gydF4y2Ba:表gydF4y2Ba年代gydF4y2Ba8)。gydF4y2Ba
参考文献gydF4y2Ba
- 1.gydF4y2Ba
叶烨,蔡敏。构建泛素链:E2酶的作用。中国生物医学工程学报。2009;10(11):755-64。gydF4y2Ba
- 2.gydF4y2Ba
泛素系统的泛素化机制。拟南芥图书。2014;12:e0174。gydF4y2Ba
- 3.gydF4y2Ba
Komander D, Rape m,泛素密码。生物化学,2012;81:203-29。gydF4y2Ba
- 4.gydF4y2Ba
Hatfield PM, Gosink MM, Carpenter TB, Vierstra RD.拟南芥泛素活化酶(E1)基因家族。植物学报,1997;11(2):213-26。gydF4y2Ba
- 5.gydF4y2Ba
蔡哈诺沃A, Elias S,海勒H, Hershko A.“共价亲和”纯化泛素活化酶。中国生物化学杂志,1982;27(5):344 - 344。gydF4y2Ba
- 6.gydF4y2Ba
黄志刚,张志刚,张志刚。酵母泛素活化酶基因uba1的研究进展。中国科学d辑,2001;10(1):227-36。gydF4y2Ba
- 7.gydF4y2Ba
王晓明,王晓明。小麦中泛素活化酶E1的研究进展。中国生物医学工程学报。1992;26(4):369 - 369。gydF4y2Ba
- 8.gydF4y2Ba
米切尔MJ,伍兹DR,塔克PK, Opp JS,主教CE。从小鼠Y染色体到泛素激活酶E1的候选生精基因的同源性。大自然。1991;354(6353):483 - 6。gydF4y2Ba
- 9.gydF4y2Ba
刘志刚,王晓明,王晓明,王晓明,等。人泛素激活酶E1的克隆、序列分析及组织分布。中国科学院学报(自然科学版),2001;29(1):1 - 6。gydF4y2Ba
- 10.gydF4y2Ba
Takizawa M, Goto A, Watanabe Y.烟草泛素激活酶NtE1A和NtE1B是由烟草花叶病毒、损伤和应激激素诱导的。生物化学学报。2005;19(2):228-31。gydF4y2Ba
- 11.gydF4y2Ba
Walden H, Podgorski MS, Schulman BA。从NEDD8激活酶的结构深入了解泛素转移级联。大自然。2003;422(6929):330 - 4。gydF4y2Ba
- 12.gydF4y2Ba
王晓明,李志强,李志强,等。小鼠泛素激活酶的晶体结构研究。中国生物医学工程学报,2005;29(3):344 - 344。gydF4y2Ba
- 13.gydF4y2Ba
黄dt, Hunt HW,庄m, Ohi MD, Holton JM, Schulman BA。类泛素蛋白硫酯开关E1-E2亲和力的基础。大自然。2007;445(7126):394 - 8。gydF4y2Ba
- 14.gydF4y2Ba
金杰,李旭,Gygi SP, Harper JW。泛素的双E1激活系统差异调节E2酶充电。大自然。2007;447(7148):1135 - 8。gydF4y2Ba
- 15.gydF4y2Ba
Glickman MH, Ciechanover A.泛素-蛋白酶体蛋白水解途径:为了构建而破坏。物理学报,2002;32(2):373-428。gydF4y2Ba
- 16.gydF4y2Ba
泛素和类泛素修饰物的扩展宇宙。植物科学进展。2012;30(1):1 - 14。gydF4y2Ba
- 17.gydF4y2Ba
李文杰,李志强,李志强,等。植物泛素化的研究进展。植物科学进展,2001;6(10):463-70。gydF4y2Ba
- 18.gydF4y2Ba
蒋永华,蒋永华。泛素化与蛋白酶体降解的研究进展。儿科儿科杂志,2004;16(4):419-26。gydF4y2Ba
- 19.gydF4y2Ba
周斌,陈旭,陈晓明,陈晓明,陈晓明,陈晓明,陈晓明。泛素偶联酶在植物免疫中的作用。中国生物医学工程学报,2017;29(2):379 - 379。gydF4y2Ba
- 20.gydF4y2Ba
Kraft E, Stone SL, Ma L, Su N, Gao Y, Lau OS,邓晓伟,Callis J.拟南芥E2和E3型连接酶泛素化酶的基因组分析及功能表征。植物科学进展。2005;39(4):344 - 344。gydF4y2Ba
- 21.gydF4y2Ba
Kurepa J, Walker JM, small J, Gosink MM, Davis SJ, Durham TL, Sung DY, Vierstra RD.拟南芥小泛素样修饰物(SUMO)蛋白修饰体系。应力增加了SUMO1和−2缀合物的积累。中国生物医学工程学报,2003;29(3):344 - 344。gydF4y2Ba
- 22.gydF4y2Ba
Windheim M, Peggie M, Cohen P.两类不同的E2泛素偶联酶用于蛋白质的单泛素化和具有特定拓扑结构的多泛素链的延伸。中国生物化学杂志,2008;29(3):344 - 344。gydF4y2Ba
- 23.gydF4y2Ba
王志刚,王志刚,王志刚。泛素-蛋白酶体系统在植物信号转导中的作用。植物学报。2012;196(1):13-28。gydF4y2Ba
- 24.gydF4y2Ba
small J, Vierstra RD.泛素26S蛋白酶体蛋白水解途径。植物学报2004;55:55 - 90。gydF4y2Ba
- 25.gydF4y2Ba
施瓦茨AL, Ciechanover A.靶向蛋白的破坏泛素系统:对人类病理生物学的影响。中国医药科学。2009;49:73-96。gydF4y2Ba
- 26.gydF4y2Ba
Rotin D, Kumar S.泛素连接酶HECT家族的生理功能。中国生物医学工程学报。2009;10(6):398-409。gydF4y2Ba
- 27.gydF4y2Ba
Downes BP, Stupar RM, Gingerich DJ, Vierstra RD.拟南芥HECT泛素蛋白连接酶(UPL)家族:UPL3在毛状体发育中具有特定作用。植物学报,2003;35(6):729-42。gydF4y2Ba
- 28.gydF4y2Ba
植物HECT泛素连接酶的进化。公共科学学报,2013;8(7):e68536。gydF4y2Ba
- 29.gydF4y2Ba
Yee D, Goring DR.植物U-box E3泛素连接酶的多样性:从上游激活物到下游靶底物。中国机械工程学报,2009;30(4):369 - 369。gydF4y2Ba
- 30.gydF4y2Ba
Deshaies RJ, Joazeiro CA. RING结构域E3泛素连接酶。生物化学。2009;78:399-434。gydF4y2Ba
- 31.gydF4y2Ba
植物生物学关系中的泛素- 26s蛋白酶体系统。中国生物医学工程学报。2009;10(6):385-97。gydF4y2Ba
- 32.gydF4y2Ba
Aravind L, Koonin EV。U盒是一个改良的无名指——泛素化中的一个常见区域。中国生物医学杂志,2000;10(4):R132-4。gydF4y2Ba
- 33.gydF4y2Ba
Koegl M, Hoppe T, Schlenker S, Ulrich HD, Mayer TU, Jentsch S.一种新型泛素化因子E4参与多泛素链组装。细胞。1999;96(5):635 - 44。gydF4y2Ba
- 34.gydF4y2Ba
通过基于蛋白质组学的底物鉴定了解culin - ring E3生物学。蛋白质组学学报。2012;11(12):1541-50。gydF4y2Ba
- 35.gydF4y2Ba
张华,李志强,李志强。筛选环泛素蛋白连接酶的研究。植物学报,2011;gydF4y2Ba
- 36.gydF4y2Ba
王志刚,王志刚,王志刚。F-box蛋白募集底物的机制与功能。中国生物医学工程学报。2013;14(6):369-81。gydF4y2Ba
- 37.gydF4y2Ba
Angot A, peters N, Lechner E, Vailleau F, Baud C, Gentzbittel L, Sartorel E, Genschik P, Boucher C, Genin S. Ralstonia solanacearum需要含有F-box样结构域的III型效应子来促进几种寄主植物的病害。中国科学(d辑:自然科学),2006;29(3):344 - 344。gydF4y2Ba
- 38.gydF4y2Ba
Lechner E, Achard P, Vansiri A, Potuschak T, Genschik P, F-box蛋白。植物学报。2006;9(6):631-8。gydF4y2Ba
- 39.gydF4y2Ba
徐刚,马红,内敏,孔慧。植物F-box基因序列分化的不同模式及其与功能多样化的关系。中国科学(d辑:自然科学),2009;26(3):339 - 344。gydF4y2Ba
- 40.gydF4y2Ba
王晓峰,王晓峰,王晓峰,等。泛素类蛋白修饰蛋白的研究进展。细胞发育生物学,2006;22:159-80。gydF4y2Ba
- 41.gydF4y2Ba
Koenning SR, Wrather JA: 2006 - 2009年植物病害对美国大陆大豆产量潜力的抑制。植物健康进展2010:在线,doi:gydF4y2Bahttps://doi.org/10.1094/PHP-2010-1122-1001-RSgydF4y2Ba.gydF4y2Ba
- 42.gydF4y2Ba
田b,王s, Todd TC, Johnson CD,唐g, Trick HN。通过深度测序鉴定大豆囊线虫感染反应的microRNA。BMC基因组学杂志,2017;18(1):572。gydF4y2Ba
- 43.gydF4y2Ba
贾强,肖志祥,王福林,孙松,梁建军,林敏敏。大豆F-box基因家族对盐胁迫响应的全基因组分析。国际分子化学杂志,2017;18(4):818。gydF4y2Ba
- 44.gydF4y2Ba
王宁,刘艳,丛艳,王涛,钟霞,杨松,李艳,盖杰。大豆U-box E3泛素连接酶的全基因组鉴定及GmPUB8在拟南芥干旱胁迫负调控中的作用。植物物理学报。2016;57(6):1189-209。gydF4y2Ba
- 45.gydF4y2Ba
孟X,王超,苏拉曼,王勇,王安,陶山。大豆HECT基因的全基因组鉴定与进化。国际分子化学杂志,2015;16(4):8517-35。gydF4y2Ba
- 46.gydF4y2Ba
陈萍,张旭,赵涛,李艳,盖杰。大豆RBR泛素连接酶基因的全基因组鉴定与鉴定。公共科学学报,2014;9(1):e87282。gydF4y2Ba
- 47.gydF4y2Ba
Schmutz J, Cannon SB, Schlueter J, Ma J, Mitros T, Nelson W, Hyten DL,宋q, Thelen JJ,程杰,等。古多倍体大豆基因组序列。自然。2010;463(7278):178 - 83。gydF4y2Ba
- 48.gydF4y2Ba
舒尔曼BA,哈珀JW。泛素样蛋白被E1酶激活:下游信号通路的顶点。中国生物医学工程学报。2009;10(5):319-31。gydF4y2Ba
- 49.gydF4y2Ba
泛素样蛋白偶联系统的进化与功能。中国生物医学工程学报。2000;2(8):E153-7。gydF4y2Ba
- 50.gydF4y2Ba
Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, Heger A, Hetherington K, Holm L, Mistry J,等。Pfam:蛋白质家族数据库。Nucleic Acids Res. 2014;42(数据库issue): D222-30。gydF4y2Ba
- 51.gydF4y2Ba
Finn RD, Clements J, Arndt W, Miller BL, Wheeler TJ, Schreiber F, Bateman A, Eddy SR. HMMER web服务器:2015年更新。核酸研究,2015;43(W1): W30-8。gydF4y2Ba
- 52.gydF4y2Ba
Aravind L, Iyer LM, Koonin EV。真核生物分子创新的比较基因组学和结构生物学。中国生物医学工程学报。2006;16(3):409-19。gydF4y2Ba
- 53.gydF4y2Ba
Burroughs AM, Jaffee M, Iyer LM, Aravind L. E2连接酶折叠的解剖:泛素/ ub样蛋白偶联的酶学和进化的意义。中国生物医学工程杂志,2008;32(2):344 - 344。gydF4y2Ba
- 54.gydF4y2Ba
Crooks GE, Hon G, Chandonia JM, Brenner SE。WebLogo:一个序列logo生成器。基因组学报,2004;14(6):1188-90。gydF4y2Ba
- 55.gydF4y2Ba
赵强,田敏,李强,崔峰,刘林,尹波,谢强。植物特异性体外泛素化分析系统。植物学报,2013;36(3):344 - 344。gydF4y2Ba
- 56.gydF4y2Ba
狄基奇I,若月S,沃尔特斯KJ。泛素结合域-从结构到功能。中国生物医学工程学报。2009;10(10):659-71。gydF4y2Ba
- 57.gydF4y2Ba
Borden KL, Freemont PS.无名指结构域:序列结构家族的一个最新例子。中国生物医学工程学报。1996;6(3):395-401。gydF4y2Ba
- 58.gydF4y2Ba
费蒙PS:无名指。一种与锌指相关的新蛋白质序列motif。南京大学学报(自然科学版),2001;gydF4y2Ba
- 59.gydF4y2Ba
Hanzawa H, de Ruwe MJ, Albert TK, van Der Vliet PC, Timmers HT, Boelens R.人类NOT4的C4C4无名指结构显示出与C3HC4无名指不同的特征。中国生物医学工程学报,2001;26(3):339 - 339。gydF4y2Ba
- 60.gydF4y2Ba
安徒生P, Kragelund BB, Olsen AN, Larsen FH, Chua NH, Poulsen FM, Skriver K.拟南芥U-box结构域的结构和生化功能。中国生物医学工程学报,2004;29(3):344 - 344。gydF4y2Ba
- 61.gydF4y2Ba
Schulman BA, Carrano AC, Jeffrey PD, Bowen Z, Kinnucan ER, Finnin MS, Elledge SJ, Harper JW, Pagano M, Pavletich NP。从Skp1-Skp2复合物的结构了解SCF泛素连接酶。大自然。2000;408(6810):381 - 6。gydF4y2Ba
- 62.gydF4y2Ba
白春,Sen P, Hofmann K, Ma L, Goebl M, Harper JW, Elledge SJ。SKP1通过一个新颖的motif - F-box将细胞周期调节因子与泛素蛋白水解机制连接起来。细胞。1996;86(2):263 - 74。gydF4y2Ba
- 63.gydF4y2Ba
Gagne JM, Downes BP, Shiu SH, Durski AM, Vierstra RD.拟南芥SCF E3复合体的F-box亚基由一个不同的超家族基因编码。中国科学(d辑),2002,26(3):326 - 326。gydF4y2Ba
- 64.gydF4y2Ba
贾峰,吴斌,李华,黄娟,郑超。玉米F-box家族的全基因组鉴定与特征分析。中国生物医学工程学报。2013;26(11):559-77。gydF4y2Ba
- 65.gydF4y2Ba
崔海仁,张志荣,吕伟,徐金娟,王晓宇。家苹果基因组F-box蛋白编码基因的全基因组特征与分析。中国生物医学工程学报,2015;29(4):457 - 457。gydF4y2Ba
- 66.gydF4y2Ba
王Y,唐H, Debarry JD,谭X,李J,王X,李TH,金H,马勒B,郭H, et al。MCScanX:一个用于检测和进化分析基因同步性和共线性的工具包。中国生物医学工程学报,2012;40(7):e49。gydF4y2Ba
- 67.gydF4y2Ba
Mudgil Y, Shiu SH, Stone SL, Salt JN, Goring DR.预测的拟南芥ARM重复蛋白大部分是U-box E3泛素连接酶家族成员。植物科学进展。2004;34(1):59-66。gydF4y2Ba
- 68.gydF4y2Ba
郑超,赵鹏,徐杰,三田N,邓松,蔡新华。植物U-BOX蛋白10调控拟南芥MYC2稳定性植物细胞,2015;27(7):2016-31。gydF4y2Ba
- 69.gydF4y2Ba
孔林,程杰,朱勇,丁勇,孟J,陈震,谢强,郭勇,李娟,杨松,等。由PUB12/13 U-box E3连接酶降解ABA共受体ABI1。Nat Commun. 2015;6:8630。gydF4y2Ba
- 70.gydF4y2Ba
Furlan G, Nakagami H, Eschen-Lippold L, Jiang X, Majovsky P, Kowarschik K, Hoehenwarter W, Lee J, Trujillo M.丝裂原激活蛋白激酶3引发PUB22泛素化模式的改变抑制免疫应答。植物学报。2017;29(4):726-45。gydF4y2Ba
- 71.gydF4y2Ba
格兰特D,纳尔逊RT,大炮SB,鞋匠RC。美国农业部大豆基因和基因组数据库。Nucleic Acids Res. 2010;38(数据库issue): D843-6。gydF4y2Ba
- 72.gydF4y2Ba
Belamkar V, Weeks NT, Bharti AK, Farmer AD, Graham MA, Cannon SB.大豆脱水和盐胁迫下HD-zip转录因子家族的综合表征和RNA-Seq分析。BMC Genomics, 2014;15:950。gydF4y2Ba
- 73.gydF4y2Ba
Lanubile A, Muppirala UK, Severin AJ, Marocco A, Munkvold GP。大豆(Glycine max)根系受致病性和非致病性枯萎菌侵染的转录组分析。BMC Genomics. 2015;16:1089。gydF4y2Ba
- 74.gydF4y2Ba
袁松,李锐,陈松,陈宏,张超,陈林,郝强,单志,杨震,邱东,等。大豆(Glycine max)根系对不同根瘤菌菌株响应差异基因表达的RNA-Seq分析前沿植物科学,2016;7:721。gydF4y2Ba
- 75.gydF4y2Ba
程燕婷,李霞。nb - lrr介导的泛素化免疫。植物学报。2012;15(4):392-9。gydF4y2Ba
- 76.gydF4y2Ba
wall RV, Liu Y, Rosebrock TR, Brady JJ, Hamera S, Connor RA, Martin GB, Zeng L.番茄Fni3赖氨酸-63特异性泛素结合酶和suv泛素E2变异体正向调控植物免疫。植物学报。2013;25(9):3615-31。gydF4y2Ba
- 77.gydF4y2Ba
朴超,陈森,Shirsekar G,周波,Khang CH, Songkumarn P, Afzal AJ,宁勇,王锐,Bellizzi M,等。稻瘟病菌效应子avrpi -t靶向RING E3泛素连接酶APIP6抑制水稻病原体相关分子模式引发的免疫。植物学报。2012;24(11):4748-62。gydF4y2Ba
- 78.gydF4y2Ba
胡塞尼P,马修斯BF。大豆根与大豆囊线虫在抗性和敏感反应中的调控相互作用。BMC植物生物学2014;14:300。gydF4y2Ba
- 79.gydF4y2Ba
Lee I, Schindelin H. e1催化泛素活化和转移到偶联酶的结构见解。细胞。2008;134(2):268 - 78。gydF4y2Ba
- 80.gydF4y2Ba
Stone SL, Hauksdottir H, Troy A, Herschleb J, Kraft E, Callis J.拟南芥环型泛素连接酶家族功能分析。植物科学进展。2005;37(1):13-30。gydF4y2Ba
- 81.gydF4y2Ba
简单CA,组RG,成员GSL。小鼠泛素化的比较蛋白质组学研究。基因组学报2003;13(6B): 1389-94。gydF4y2Ba
- 82.gydF4y2Ba
曾丽玲,朴春霞,王丽丽,高夫J。水稻u -box蛋白的分类、表达模式及E3连接酶活性测定。植物学报。2008;1(5):800-15。gydF4y2Ba
- 83.gydF4y2Ba
Yang X, Kalluri UC, Jawdy S, Gunter LE, Yin T, Tschaplinski TJ, Weston DJ, Ranjan P, Tuskan GA。相对于多年生木本植物,F-box基因家族在一年生草本植物中扩展。植物科学进展。2008;32(3):339 - 344。gydF4y2Ba
- 84.gydF4y2Ba
张霞,王楠,陈萍,高敏,刘娟,王艳,赵涛,李艳,盖杰。大豆泛素连接酶基因GmARI1的过表达增强拟南芥铝耐受性。公共科学图书馆,2014;9(11):e111120。gydF4y2Ba
- 85.gydF4y2Ba
杜清良,崔文忠,张春春,于达元。GmRFP1编码大豆环型E3泛素连接酶(Glycine max)。Mol生物学杂志2010;37(2):685-93。gydF4y2Ba
- 86.gydF4y2Ba
张宏,宋bh。野生大豆对大豆囊肿线虫(Heterodera glycines)基因型的RNA-seq数据比较。基因数据。2017;14:36-9。gydF4y2Ba
- 87.gydF4y2Ba
Eddy SR.加速廓线HMM搜索。科学通报。2011;7(10):e1002195。gydF4y2Ba
- 88.gydF4y2Ba
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析版本6.0。中国生物医学杂志,2013;30(12):2725-9。gydF4y2Ba
- 89.gydF4y2Ba
Larkin MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWilliam H, Valentin F, Wallace IM, Wilm A, Lopez R,等。Clustal W和Clustal X 2.0版本。生物信息学。2007;23(21):2947 - 8。gydF4y2Ba
- 90.gydF4y2Ba
Robert X, Gouet P.用新的ENDscript服务器破译蛋白质结构的关键特征。Nucleic Acids Res. 2014;42(Web Server issue): W320-4。gydF4y2Ba
- 91.gydF4y2Ba
Artimo P, Jonnalagedda M, Arnold K, Baratin D, Csardi G, de Castro E, Duvaud S, Flegel V, Fortier A, Gasteiger E,等。ExPASy: SIB生物信息学资源门户。Nucleic Acids Res. 2012;40(Web Server issue): W597-603。gydF4y2Ba
- 92.gydF4y2Ba
Ariani P, Regaiolo A, Lovato A, Giorgetti A, Porceddu A, Camiolo S, Wong D, Castellarin S, Vandelle E, Polverari A.葡萄ATL泛素连接酶家族的全基因组特征和表达谱揭示了生物和非生物胁迫响应和发育相关成员。科学代表2016;6:38260。gydF4y2Ba
资金gydF4y2Ba
这项工作得到了内布拉斯加州大豆委员会(1719号基金)对LZ的支持。gydF4y2Ba
数据和材料的可用性gydF4y2Ba
在本研究过程中产生或分析的所有数据都包含在本文及其补充数据文件中。gydF4y2Ba
作者信息gydF4y2Ba
从属关系gydF4y2Ba
贡献gydF4y2Ba
LS、BZ和CZ对大豆UBS基因进行了生物信息学分析。MC克隆并纯化了部分大豆E2和E3基因的重组蛋白。GS对RING和F-box基因家族进行了全基因组基因重复分析。KB和LG种植大豆植株,用SCN接种大豆根系。CZ完成了大部分的实验,分析了数据,并撰写了文章。LZ设计实验,分析数据,撰写和编辑文章。所有作者都阅读并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
道德声明gydF4y2Ba
伦理批准并同意参与gydF4y2Ba
不适用gydF4y2Ba
发表同意书gydF4y2Ba
不适用gydF4y2Ba
相互竞争的利益gydF4y2Ba
作者宣称他们之间没有利益冲突。gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。gydF4y2Ba
附加文件gydF4y2Ba
附加文件1:gydF4y2Ba
表S1。gydF4y2Ba大豆含ThiF基序蛋白列表。(docx124kb)gydF4y2Ba
附加文件2:gydF4y2Ba
表S2。gydF4y2BaHMM剖面用于目前的研究。(docx191kb)gydF4y2Ba
附加文件3:gydF4y2Ba
表S3。gydF4y2Ba大豆UBC、RING、U-box和F-box结构域编码基因鉴定列表。(xlsx62 kb)gydF4y2Ba
附加文件4:gydF4y2Ba
图S1。gydF4y2Ba大豆含UBC结构域蛋白的多序列比对。(PDF 28kb)gydF4y2Ba
附加文件5:gydF4y2Ba
图S2。gydF4y2Ba拟南芥和大豆含UBC结构域蛋白的系统发育树。(JPG 1759 kb)gydF4y2Ba
附加文件6:gydF4y2Ba
图S3。gydF4y2Ba大豆泛素E2蛋白的系统发育树。(JPG 905kb)gydF4y2Ba
附加文件7:gydF4y2Ba
图S4。gydF4y2Ba大豆环结构域含蛋白环结构域序列的比对。(PDF 88kb)gydF4y2Ba
附加文件8:gydF4y2Ba
图S5。gydF4y2Ba大豆U-box结构域含蛋白序列的比对。(PDF 27kb)gydF4y2Ba
附加文件9:gydF4y2Ba
图S6。gydF4y2Ba大豆含F-box结构域蛋白的F-box结构域的多个序列比对。(PDF 524kb)gydF4y2Ba
附加文件10:gydF4y2Ba
图S7。gydF4y2Ba大豆含环结构域蛋白的系统发育分析。(PDF 188kb)gydF4y2Ba
附加文件11:gydF4y2Ba
图S8。gydF4y2Ba大豆U-box结构域蛋白的系统发育分析。(PDF 30kb)gydF4y2Ba
附加文件12:gydF4y2Ba
图S9。gydF4y2Ba大豆含F-box结构域蛋白的系统发育分析。(PDF 118kb)gydF4y2Ba
附加文件13:gydF4y2Ba
图S10。gydF4y2Ba大豆RING基因的复制事件。(PPTX 1668 kb)gydF4y2Ba
附加文件14:gydF4y2Ba
图S11。gydF4y2Ba大豆F-box基因的复制事件。(PPTX 1064 kb)gydF4y2Ba
附加文件15:gydF4y2Ba
表S4。gydF4y2Ba总结了大豆UBS基因进行不同类型复制的数量。(xlsx9kb)gydF4y2Ba
附加文件16:gydF4y2Ba
图S12。gydF4y2Ba纯化大豆E1和E2蛋白的SDS-PAGE检测(JPG 992 kb)gydF4y2Ba
附加文件17:gydF4y2Ba
图向。gydF4y2Ba拟南芥大豆U-box结构域蛋白克隆及其同源物的系统发育分析。(JPG 1220kb)gydF4y2Ba
附加文件18:gydF4y2Ba
表S5。gydF4y2BaRNA-seq分析显示UBS基因的表达谱。(xlsx213kb)gydF4y2Ba
附加文件19:gydF4y2Ba
图S14系列。gydF4y2Ba非生物和生物胁迫处理后大豆UBS基因差异表达的热图。(JPG 1112 kb)gydF4y2Ba
附加文件20:gydF4y2Ba
表S6。gydF4y2BaRNA-seq分析显示大豆UBS基因在胁迫下的差异表达。(xlsx34kb)gydF4y2Ba
附加文件21:gydF4y2Ba
表S7。gydF4y2BaRNA-seq分析显示,SCN处理后UBS基因表达差异。(xlsx29 kb)gydF4y2Ba
附加文件22:gydF4y2Ba
图S15。gydF4y2Ba经SCN处理后,经RNA-seq分析鉴定的大豆UBS基因的表达水平未发生变化。(JPG 1032kb)gydF4y2Ba
附加文件23:gydF4y2Ba
表S8。gydF4y2Ba本研究使用的PCR引物。(docx18kb)gydF4y2Ba
权利和权限gydF4y2Ba
开放获取gydF4y2Ba本文根据创作共用属性4.0国际许可协议(gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)除另有说明外,适用于本条所提供的资料。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
张春,宋,L.,乔杜里,M.K.gydF4y2Baet al。gydF4y2Ba大豆泛素系统核心成分基因的全基因组分析(gydF4y2Ba大豆gydF4y2Ba)揭示了泛素化在大豆囊线虫宿主免疫中的潜在作用。gydF4y2BaBMC植物生物学gydF4y2Ba18日,gydF4y2Ba149(2018)。https://doi.org/10.1186/s12870-018-1365-7gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-018-1365-7gydF4y2Ba
关键字gydF4y2Ba
- 大豆gydF4y2Ba
- 泛素系统(UBS)gydF4y2Ba
- 泛素激活酶(E1)gydF4y2Ba
- 泛素结合酶(E2)gydF4y2Ba
- 环域gydF4y2Ba
- U-box域gydF4y2Ba
- 盒域gydF4y2Ba
- 大豆囊肿线虫gydF4y2Ba
- 免疫力gydF4y2Ba