摘要
背景
DNA条形码即将到来,使用标准基因区域的物种歧视的通用标准的产生,该标准基因区域可以准确地在短时间内测序。在这项研究中,我们在开发有效的条形码基因座中取得了成功猪笼草属。从NCBI的GenBank中检索到317份植物资源,代表140个不同的物种猪笼草采用条形码间距、应用距离相似度和基于树的方法评价ITS、rbcl和matK候选条形码的有效性。
结果
我们的结果表明,与塑体区(MATK)的单个轨迹显示了具有独特条形码间隙的最佳物种歧视。因此,我们暂时提出了其+ MATK作为核心条码的组合猪笼草属。
结论
本研究为独特食虫动物的DNA条形码研究提供了一份报告。猪笼草属。作为不同的物种猪笼草是higly地方病和危害,这将是一个有益的研究,以了解的进化关系,勾勒移民,贴错标签,可以作为其生物多样性可能的评估。
背景
分类是一个生态系统中物种的准确命名的根本基础。在分类学的知识差距是没有适当的遗传和生物分布由于分类学专家不足,到今天数以百万计的物种的增加仍然不明。因此,对于许多理论研究和现实应用[各个品种划定明确的分类和分类的迫切要求1].传统的基于形态学的分类学提供了大量多样化植物属的模糊系统发育证据[2].为了克服分类学中的这一问题,基因组DNA测序可以作为物种鉴定的标准化方法,因为亲缘关系越近的物种相对于亲缘关系越远的物种拥有更多的同源DNA序列[3.].DNA条形码被认为是一种有效利用特定DNA序列的短区域进行物种识别的有前途的方法[2那4.].在动物基因组中,线粒体细胞色素氧化酶I (mitochondrial cytochrome oxidase I, COI)基因是被普遍接受的DNA条形码,而植物中的这一区域由于突变率低,变异性不足,因此需要替代条形码区域[5.那6.那7.].因此,一些叶绿体基因座和这些基因座的组合被认为是一种很有前景的植物DNA条形码[8.].除了质体DNA序列,核糖体内部转录间隔区(ITS)也被用于植物[9.那10.].然而,它在放大过程中经受了复杂的考验,这使得它成为陆生植物的通用条码成为可能。尽管存在这些并发症,但许多研究人员证明,与其他编码或非编码质体标志物相比,ITS可以实现更好的扩增[11.那12.那13.].由于有限的研究是在不同的Anviosperm和猪笼草作为高度濒危的属之一,对其分类学分类和多样性的研究十分必要。
猪笼草(Caryophyllales:Nepenthaceae),其中包括世界各地的170种,从澳大利亚北部到中国南部到中国北部[14.新的喀尔多尼亚和向西延伸到塞舌尔和马尔加什里。它们在其生长形式,栖息地,猎物和投手形式中表现出巨大的多样性。猪笼草sp.受第《生物资源和生态系统的保护》(1990),并符合《濒危物种国际贸易公约》(CITES)的规定N. Rajah.和N. Khasiana载于附录i,其余则载于附录ii [15.那16.].这使得限制了该属的交易活动。在人类的利益猪笼草从利用率到其治疗效果。它的栖息地和各种投手形式的独特特征使属成为迷恋和时尚朝向人类的对象。此外,投手室内的高潮蜡表面还鼓励工程师根据此功能开发许多独特的产品。由于过度开采,这种属的人口正在迅速下降,并且如果这种拒绝继续,那么它将导致多样性降低并导致其灭绝。分类学猪笼草主要基于形态学如形状,颜色,大小和纹饰[17.那18.].关于植物史的记录猪笼草结果显示,有分类混乱的各种情况,如N. Pilosa.与n chaniana直到2006年,类似的N. talangensis与n bongso和N. Lamii.与N. vieillardii[18.那19.].此外,属的进化也是一个挑战,因为它们没有近亲/祖先类型或过渡物种。但猪笼草有远房亲戚,可提供约属的起源的线索。此前,分子系统发育研究中猪笼草是基于叶绿体(trnK和matK基因)和核(PRT1)序列[20.那21.];然而,最近的研究基于RAPD,ISSR等的分子标记[22.那23.].在判别的种类的适用性和DNA条形码的有效性猪笼草这项研究中的第一次进行了。另一方面,难以在整个大型地理区域中收集该属的所有物种。因此,这项研究侧重于序列猪笼草国家生物技术信息中心(NCBI)数据库报告的物种。在这里,我们评估了三种潜在的条形码,抽样140种猪笼草目的是提出一个实用和通用的标准条形码区域,必须保存和区分物种与其他属。
方法
分类群抽样
在本研究中选择了其,RBCL和MATK的基因座作为条形码候选者。所有可用的序列猪笼草从NCBI的GenBank下载。序列的选择是基于两个标准:1 .适当的凭证标本;长度超过300 bp。本研究使用的分类群、作者和GenBank登录号见附加文件1:表S1。
数据分析
每个区域的下载的序列使用克利亚尔XV1.8.7对齐[24.在BioEdit v7.1.3.0中手动同步[25.].对于它,我们在5.8S rDNA的两端调整区(ITS1和ITS2)基于简约原则[14.].剖视原则指出,在一套可能的解释中,预计最简单的解释是准确的。在系统发生的基础上,定义意味着在最可能是正确的最可能是正确的影响中的关系的假设。因此,所有其序列都基于定义原理对齐和安排,以避免错误结果。
遗传对明智距离用Mega 7中的Kimura-2参数(K2P)距离计算.K2P是非常小距离的最佳模型之一[2].使用MEGA 7软件的一对明智距离进行比较每对三个单个条形码的特定间隔和特定距离之间的差异。条形码差距是当最小K2P间距距离大于最大内距距离时存在有效条形码轨迹的量度[26.].使用具有“两两汇总功能”的Taxon DNA,比较每个候选条形码的两两种种内和种间距离的分布,以0.05的间隔距离估计条形码间隙。
为了准确地分析物种,测量每个条形码候选者使用具有最佳匹配,“最佳亲密匹配”和“所有物种条形码”功能的分类仪DNA测量正确的识别比例。“最佳匹配”分析确定给定序列最接近的匹配。如果比较序列来自相同的物种,则认为鉴定被认为是正确的,而如果序列不属于相同的物种,则不正确[27.].
为了进入标记歧视性能的有效性,我们通过进行基于树的分析评估了单声道的起源[26.那28.那29.].在MEGA 7中使用Neighbor-joining (NJ)估计系统发育树,通过bootstrap测试评估节点支持[30.[1000伪复制与K2P距离的运行作为替换模型。盾叶穗叶藤属用作小组。
结果
基于筛选序列的两个标准,我们获得了从NCBI 317个序列,其包括ITS,分别rbcL基因和matK基因,183,33和101个序列(附加文件1:表S1)。
遗传分解分析
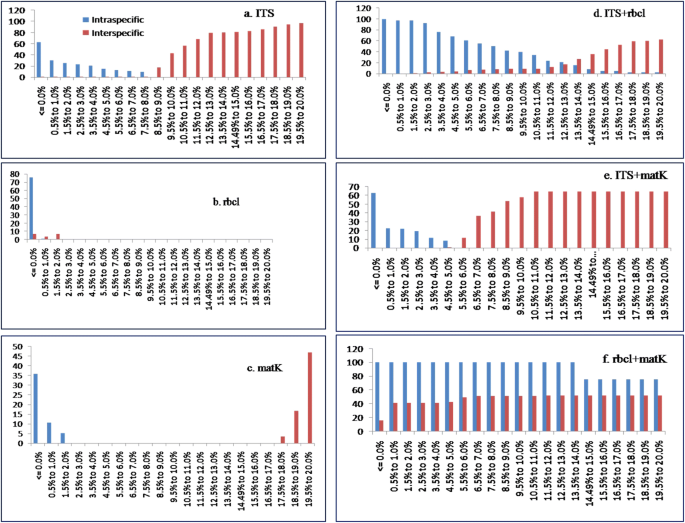
rbcl的序列长度为1251 bp, ITS的序列长度为951 bp1)。ITS的变异位点最大,其次是matK。6种条形码的种内距离范围为0.0 ~ 0.9%,rbcl+matK的种内距离均值最小(0.02%),ITS的种内距离均值最高(1.31%)。ITS+rbcl的种间距离最小(0.16%),ITS的种间距离最大(0.84%)。综上所述,ITS揭示了最高的种内和种间距离平均值(表1)2)。
条形码差距分析
利用Taxon DNA软件中的K2P距离计算3种候选条形码的种内和种间遗传距离的相对分布。所有亚属的种间距离较高,且与种内距离不完全重叠。因此,我们对所有的数据集和子属进行了条形码差距分析。三个条形码,即ITS(图。1A), matK(无花果。1C)及其+ matk(图。1E)显示较明显的条码缺口。所有其他条形码在种内和种间距离之间存在重叠,没有明显的条形码缝隙(图。1)。
歧视物种
使用分类群DNA进行鉴别物种的分析,其具有最高的成功率进行正确鉴定物种(最佳匹配:78.12%;最佳近距离:77.67%;所有物种条形码:80.76%)随后是其+ MATK和最少的歧视在其+ RBCL中观察到成功率(表3.)。
基于树的分析
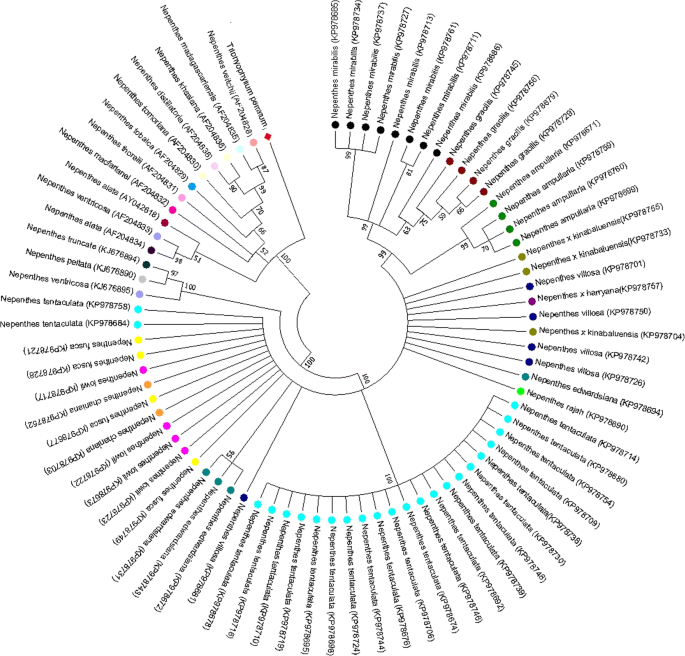
通过评估使用NJ树基于NJ树的分析来估计基于系统发育树的六个条形码候选六个条形发作树木的六个条形候候选物估计(图。2)。我们观察到,所有的单基因条形码已经从11.76物种歧视变水平低30.68%(表1)。在多点条形码中,其+ MATK显示出最大成功率(83.33%),然后是其+ RBCL(50.00%)。因此,可以得出结论,当其包括三种组合中包含物种歧视更高。我们完成了我们的结果表明它的+ MATK在所有核心条形码中都是卓越的。
讨论
进行了几项研究以发现不同植物的合适的条形码,但到目前为止实现了所需的共识[31.那32.].在本研究中,我们包括猪笼草sp.通过他们的GenBank记录,从不同的研究中获得的序列。因此,我们强烈假设所有报告的序列猪笼草sp.是基于正确识别植物物种。质区域最初提议作为植物条形码的核心,但他们都没有成功在植物的所有属。此外,许多研究人员发现其作为在植物中一个具有挑战性的条形码,从而拒绝用于掺入植物[核心条形码区域9.那33.那34.那35.].通过高级研究,我们观察到其广泛用于恢复正确分配物种的高率,因为它具有较少的特定内部变异,但更高的特异性分歧[36.].此外,发现座位ITS和质的组合在一些植物属的最佳选择。根据我们的结果,ITS和matK基因有中所提出的条形码位点即ITS,rbcL基因和matK基因更好的简约信息位点和区分能力这同样涉及到以前的研究结果[14.那37.那38.].如果种间距离大于种内距离,则以成对距离为基础进行鉴别的物种是多的[8.]最后,我们观察到其基于距离分析方法具有最高的特异性序列分歧。本研究中使用了“最佳匹配”,“最佳匹配”和“所有物种条形码”选项的统计数据,并以高物种歧视率,其再次观察其+ MATK。基于NJ树,其+ MATK条形码POSSE的最大值和RBCL含有最小物种对该属的分辨率。另一方面,在植物中提出了两种或三个条形码的几种组合,包括其+ Trnh-PSBA [12.],ITS + rbcL基因[39.],matK基因rbcL基因+ [8.]和ITS + matK序列+ rbcL基因[28.,但对其用途尚未达成共识。matK+rbcl被认为是所有陆生植物的通用条码猪笼草sp。MATK + RBCL在三个条形码组合中具有低物种分辨率,因为编码基因中的替代率低,其+ MATK与具有明确定义的条形码间隙的条形码候选的其他单一或组合相比,其+ MATK占物种鉴定的最高百分比。但是,所有物种猪笼草是特定的或局限于不同的地理区域。因此,利用DNA条形码技术对非法转移的物种和地理信息进行鉴定是一种潜在的解决方案。在未来,这些发现将有可能有助于描绘物种猪笼草因此,他们可能最有可能成为成功的条形码此属。
结论
本研究评估了非非法交易的分类危害和地方植物的分类学征收/鉴定的DNA条形码技术。从本研究中,我们可以得出结论,通过依赖于包括先前和后验日期的综合方法,可以更真实地进行DNA条形码识别。在这项研究中,它描绘了六个条形码候选单个基因座,其+ MATK其+ MATK可以进一步进入Nepenthes属的核心条形码的高鉴别功率率。由于这种属在世界的不同地区是独一无二的,因此需要一种不可挽回的系统,如DNA条形码,以保护生物多样性和控制在物种的非法贸易中。
参考文献
- 1。
Dayrat B.迈向综合分类。Biol J Linn Soc。2005; 85:407-15。
- 2。
赫伯特PDN,Cywinska A,球SL,deWaard JR。通过DNA条形码生物标识。PROCř志林斯顿B. 2003; 270:313-21。
- 3.
美国巴布森大学植物研究基金工作论文;2010.75年p。纸。
- 4.
DNA条形码:基于全面抽样的错误率。公共科学图书馆杂志。2005;3(12):422。
- 5。
Kress WJ, Wurdack KJ, Zimmer EA, weight LA, Janzen DH。利用DNA条形码识别开花植物。美国国家科学研究院2005;102:8369-74。
- 6。
Chase MW,Salamin N,Wilkinson M,Dunwell JM,Kesanakurthi RP,Haider N,Savolainen V.陆地植物和DNA条形码:短期和长期目标。Philos Trans R SoC Lond B. 2005; 360:1889-95。
- 7。
Fazekas AJ, Kesanakurti PR, Burgess KS, Percy DM, Graham SW, Barrett SC, Newmaster SG, Hajibabaei M, Husband BC。使用DNA条形码标记,植物是否天生就比动物物种更难区分?摩尔生态资源。2009;9:130-9。
- 8.
Cbol工厂工作组。土地植物的DNA条形码。Proc Natl Acad Sci U S A. 2009; 106:12794-7。
- 9.
Chase MW, Cowan RS, Hollingsworth PM, Berg CVD, Madriñán S, Petersen G, Seberg O, Jørgsensen T, Cameron KM, Carine M, Pedersen N, Hedderson TAJ, Conrad F, Gerardo GA, Richardson JE, Hollingsworth ML, Barraclough TG, Kelly L, Wilkinson M。分类单元。2007;56:295-9。
- 10。
Giudicelli GC,马德尔G,弗雷塔斯LBD。ITS序列的效率为DNA条形码中西番莲(西番莲)。中华医学杂志。2015;16:7289-303。
- 11.
Muellner An,Schaefer H,Lahaye R.候选DNA条形码基因座的经济上重要的木材种类评估桃花心木家庭(楝科)。摩尔生态资源。2011;11:450-60。
- 12.
杨建斌,王艳萍,Möller M,高利明,吴丹。植物DNA条形码在植物物种鉴定中的应用帕尼亚西亚(parnacciaceae)。Mol Ecol Resour。2012; 12:267-75。
- 13。
张达,段L,周N. DNA条形码的应用Roscoea姜科植物的分类地位及其初步探讨Roscooea Cauteoides.var。pubescens..生物化学学报。2014;52:14-9。
- 14。
徐胜,李丹,李军,向旭,金伟,黄伟,金鑫,黄磊芽筋(Orchidaceae)来自大陆亚洲。Plos一个。2015; 10(1):E0115168。
- 15.
克拉克CM,鲍尔U,李CC,屯AA,Rembold K,莫兰JA。在热带食虫植物一种新型的氮气封存策略:树鼩厕所。生物学快报。2009; 5(5):632-5。
- 16。
N,Setyaningsih D.热带投手植物的探索性研究(猪笼草sp在加里曼丹Palangka Raya中加里曼丹的Sebangau国家公园,被困在里面的种类和昆虫。J Phys Conf Ser. 2017;795:012062。
- 17。
菲利浦斯A,婆罗洲羔羊A.猪笼草。1 ed。Kota Kinabalu:自然历史出版物;1996. p。12-31。
- 18.
麦克弗森SR。旧世界2卷的猪笼草。1 ed。普尔:雷德芬自然历史制作公司;2009年,P。768 ISBN 9780955891830。
- 19.
克拉克厘米。苏门答腊岛和马来西亚半岛的猪笼草。Kota Kinabalu:自然历史出版物;2002.p。336。
- 20。
soltis ps,soltis de。同种异体中的多个起源Tragopogon Mirus.(菊科):rDNA证据。系统机器人。1991;16:407-13。
- 21。
Meimberg H,Thalhammer S,Brachmann A,的易位拷贝的Heubl G.比较分析trnK内含子在食肉属猪笼草(Nepenthaceae)。mol phylocyet evol。2006; 39:478-90。
- 22。
Bhau BS, Medhi K, Sarkar T, Saikia SP. PCR的分子表征猪笼草khasianaHook.f.猪笼草。遗传资源作物evol。2009; 56:1183-93。
- 23。
Bunawan H, Yen CC, Yaakop S, Noor NM。系统发育推断的猪笼草叶绿体(TRNL内含子)和核(ITS)DNA序列揭示的半岛马来西亚种类。BMC RES笔记。2017; 10:67。
- 24。
汤普森JD,Gibson TJ,Plewniak F,Jeanmougin F,Higgins DG。Clustal_x Windows接口:通过质量分析工具提供多个序列对齐的灵活策略。核酸研究。1997; 25:4876-82。
- 25。
大厅TA。BIOEDIT:对于Windows 95/98 / NT用户友好的生物序列比对编辑器和分析程序。核酸序列SYMP。1999; 41:95-8。
- 26。
王志强,王志强,王志强,等。不同基因型的DNA条形码鉴定Lamium。L.(唇形科)。Mol Ecol Resour。2014; 14:272-85。
- 27。
王志强,吴世扬,王志强。双翅目的DNA条形码与分类:种内变异高而鉴定成功低的故事。系统医学杂志。2006;55:715-28。
- 28。
张春媛,王凤英,严海峰,郝刚,胡明明,葛晓军。在密切相关的群体中测试DNA条形码Lysiaachia。l .(紫金牛科)。生态学报。2012;12:98-108。
- 29。
Alves TLS, Chauveau O, Eggers L, de Souza-Chies TT。物种歧视Sisyrinchium鸢尾科:一个具有分类挑战性的属的DNA条形码的评估。生态学报。2013;14:324-35。
- 30.
Felsenstein J.来自分子序列的系统发育:推理和可靠性。Annu Rev Genet。1988; 22:521-65。
- 31.
Lahaye R, Van der Bank M, Bogarin D, Warner J, Pupulin F, Gigot G, Maurin O, Duthoit S, Barraclough TG, Savolainen V.生物多样性热点区系的DNA条形码。美国国家科学学院学报2008;105:2923-8。
- 32。
陈志强,陈志强,陈志强,等。DNA条形码技术在DNA条形码鉴定中的应用卡拉迪尼亚(兰科)物种。Aust J Bot. 2009; 57:276-86。
- 33。
阿尔瓦雷斯一世,温德尔。核糖体ITS序列与植物系统发育推断。acta physologica sinica, 2003; 29:417-34。
- 34。
斯塔尔JR,Naczi RFC,乔伊纳德BN。在莎草植物DNA条形码和物种分辨率(苔草,莎草科)。Mol Ecol Resour。2009; 9(1):151-63。
- 35。
霍林沃思总理,克拉克AA,福雷斯特LL,理查森J,彭宁顿RT,朗DG,考恩R,蔡斯MW,高德尔M。植物条形码基因座的选择:在3个不同的陆生植物类群中,用种水平抽样法评价7个候选基因座。生态学报2009;9:439-57。
- 36。
朱兰,李叶,钟dl,张jq。建立最全面的ITS2条形码数据库到传统药用植物的日期红景天(Crassulacaee)。Sci众议员2017;7:10051。
- 37。
李德志,高利明,李海涛,王浩,葛晓军,刘建强,陈志东,周少林,陈少林,杨建平,付彩霞,曾彩霞,闫长发,朱永杰,孙永生,陈少云,赵林,王凯,杨涛,段国伟。大数据集的比较分析表明,内部转录间隔器(ITS)应纳入种子植物核心条码。美国国家科学研究院2011;108:19641-6。
- 38。
Cabelin VL, Alejandro GJ。matK、rbcL、trnH-psbA和trnL-F (cpDNA)对菲律宾民族药用夹竹桃科分子鉴定的有效性。Pharmacogn杂志。2016;12 (3):S384-8。
- 39。
俞WB,黄PH,稀土元素RH,刘ML中,L1 DZ,王H. DNA条形码Pedicularis(Orobanchaceae):在一个大的和Hemiparasitic属。J Syst Evol. 2011; 49:425-37。
致谢
作者感谢印度科学院东北科学技术研究所所长,该研究所位于印度阿萨姆邦乔哈特。
资金
这项工作是由印度阿萨姆邦Jorhat的csir -东北科学技术研究所在MLP1000内部项目下支持的。
可用性数据和材料
从CSIR-NEIST种质收集收集植物材料。
作者信息
隶属关系
贡献
BG和BSB进行了实验设计。BG采集植物材料,进行DNA提取、DNA测序和序列比对。BG和BSB参与了稿件的编写,对稿件的初稿进行了审核,最终审定稿件。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
额外的文件
附加文件1:
表S1。本研究使用的猪笼草种类样本信息一览表。(PDF 255 kb)
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Gogoi, B., Bhau, B. s .属的DNA条形码猪笼草(投手植物):对其识别的初步评估。BMC植物BIOL.18,153(2018)。https://doi.org/10.1186/s12870-018-1375-5
已收到:
公认:
发表:
关键词
- 猪笼草
- 猪笼草
- DNA条码技术
- 系统发育
- 它的
- 分类