摘要
背景
禾本科(禾本科),约12075种,是最近的许多研究旨在使用完整plastomes揭示和加强家庭内部关系的焦点。利用新一代测序技术已经揭示了许多禾本科plastomes错综复杂的细节;特别是TRNI.-TRNL.基因间的隔离区域。这项研究调查了这一区域和假定的线粒体插入其中的完整的质体雀稗和其他禾本科。
结果
9个新测序的质体,其中7个含有插入物TRNI.-TRNL.将基因间间隔体与其他52种植物的质体系统发育和差异数据进行了分析。一个健壮的雀稗拓扑恢复,始于10.6 Ma,插入物产生于8.7 Ma。对插入的横向对齐雀稗在19中揭示了21个与成对同源性的次区域。在分析四核苷酸频率的紧急自组织地图中,雀稗插入物与线粒体DNA分组。
结论
发现了一个假设的祖先插入,17,685英镑的大小,发现了TRNI.-TRNL.代表间隔物雀稗血统。在同一区域发现一个不同的插入物,2808 bpParaneurachne Muelleri..在其中发现了七种不同的娱乐事件雀稗谱系,表明选择性压力去除大部分非码DNA。最后,使用四核苷酸频率分析来确定插入物的起源雀稗谱系就是线粒体DNA。
背景
草地(Poaceae)包含12个含有Ca的亚壳。12,075种物种[1]。近年来,以完整质体基因组(plas质体)作为基因组标记,深入研究了禾本科植物的系统基因组结构;其中许多已经通过下一代测序(NGS)方法获得了质体。NGS技术允许在草亚科中进行更广泛和更深层次的取样[2,3.,4,5,6,7,8,9,10.,11.,12.,13.,14.,15.],部落[16.,17.,18.,19.]甚至属(Alloteropsis.:[20.,21.];Zea.:[22.])。这种深入的抽样策略使研究人员能够发现Poaceous Plastomes的独特功能。
一种这样的特征是将线粒体DNA(MTDNA)转移到塑性DNA(PTDNA)的转移。这种类型的突变事件最初被认为是不存在的或极其罕见的[23.,24.,25.]。发现了第一个报告的MTDNA转移到PTDNA的情况胡萝卜胡萝卜[26.然后第二个发现马利syriaca[27.]。由于这两个发现都发生在双双子体中,直到Wysocki等人[14.,即在单子叶植物中发现了mtDNA到ptDNA的转移。
单子叶植物的第一个发现与两种竹子的转移有关,eRemitis.sp。和Pariana radiciflora,来自同一个子条件,帕里安娜。插入内部eRemitis.sp。和Pariana radiciflora分别为5千比西对(KBP)和2.7 kBp,以及2.7 kbp部分的高度相似的顺序[14.]。后来又增加了两个ParianinaeParianasp。和p .定[9],它还包含在同一区域的同源插入物TRNI.-TRNL.基因间的间隔(IGS)。虽然在草中发现的ptDNA插入物中有mtDNA的其他发现[Triticum Monococcum.:11]其他edicots [薰衣草花angustifolia,Orobanche californica,黄芩: 28]中插入的位置TRNI.-TRNL.IGS一直备受关注。
最近一项研究的作者[3.研究了这个区域,发现了假定的mtDNA插入TRNI.-TRNL.两种雀稗.这些插入物彼此之间没有相似之处,也与同一地区的Parianinae物种没有任何相似之处。他们的结论是要么是内部的多次插入雀稗彼此独立地出现,或者两个事件是较大插入物的一部分。如果已知这种嵌入式DNA转移的机制,则此类事件的分类分类分布可能更明显。但是,没有找到这样的机制[25.]。本研究将在Burke等人的前一项研究中建立。[3.],通过在物种的增加的采样雀稗,确定在塑料中发现的插入的起源,机制和时间雀稗.
方法
采样
物种雀稗根据另一项研究[29.发现它们与那些有或没有mtDNA插入的细胞密切相关[3.选择在本文中进行进一步分析。根据这些标准,八个新的雀稗塑料(表格1)被选中进行测序,这是在之前发表的三篇文章的基础上增加的。外类群雀稗被选中以适应Poaceae中的可用化石[4]。因此,分析包括九个新的质体,八个雀稗物种和Paraneurachne Muelleri.和来自所有亚属的45种其他淘eae塑料,其中包括分类日期分析的分类群。除了在塑料中发现mtDNA的样品,还包括其中的分类群。TRNI.- - - - - -TRNL.区域[Parianinae: 9,14]或在其他地区[小麦属植物:11]。
提取和图书馆制剂
从物种的DNA提取雀稗使用DNeasy Plant Mini试剂盒(Qiagen, Valencia, CA, USA)根据制造商的说明,在液氮均质步骤后,在二氧化硅或植物标本室标本中干燥的嫩绿叶材料上进行。然后使用Illumina Nextera协议制备NGS样品。DNA样品稀释至2.5 ng/ul(共50 ng),使用Nextera DNA样品制备试剂盒制备配对端文库。所有文库在爱荷华州立大学(艾姆斯,爱荷华,美国)的核心DNA设施上使用Illumina HiSEq 3000进行测序。序列读取,来自Washburn等人[19.], 为了单纯雀稗(SRR2162764),Paspalum vaginatum.(SRR2163016),和Paraneurachne Muelleri.(SRR2163452)从NCBI的SRA档案馆(https://www.ncbi.nlm.nih.gov/sra.)对于整个塑料组装。
NGS塑料组装,验证和注释
在Burke等人的方法中过滤并组装了Illumina和SRA读数。[3.]。在默认设置下,低质量的读取被删除(DynamicTrim, SolexaQA++;[30.]),仍然附着在读数(CENCADAPT; [31.])然后丢弃任何读数少于25个碱基对(BP)Long [LenceSort,SolexaQa ++;30]。黑桃v3.6.1 [32.]用于从头装配k-mers区间为19 ~ 73 bp,区间为6。CD-Hit v4.6 [33.删除了contig文件中的冗余序列。英亩(34.是用来把contigs架在一起的。
将ACRE支架和clean reads导入geneipro v9.1.6 [35.]。对于每一个新加入的物种,选择一个在NCBI储存的密切相关的参考质体。然后使用MAFFT v7.222将支架的contigs与参考质体对齐[36.]插件在诅咒中。通过使用地图在硅基因组步行中的佐动中引用功能来关闭Contig之间的间隙[3.]。通过将reads映射到最终确定的质体,对每个质体进行最终验证,当reads和质体之间发生不一致时,进行手动调整。
然后将已验证的塑料载玻片与其参考塑料成对对齐,并使用佐剂Pro中的转移注释特征施用注释。检查并手动调整编码序列边界以保护读数帧。使用Burke等人的方法定位倒置重复(IRS)。[37.]。blastn [38.通过将新的塑料对准,并寻找从加/加上的加上/加上/减去的段来定位IR边界。然后使用葛辩中的主题特征标记这些边界,并进行IR的注释。
系统核发生组分析和发散日期估计
组装了含有九个新塑性的Poaceae Plastid DNA(PTDNA)的61个分类群矩阵和11雀稗总体上物种。使用MAFFT v7.222对这61个完整的质体(不包括一个IR拷贝)进行比对[36.]插件,默认设置。
的TRNI.- - - - - -TRNL.物种的地区雀稗从MaFFT比赛中提取并手动对齐。对于共享或独特的稀有基因组变化,通过BLASTN搜索鉴定出对共享或独特的稀有基因组变化和序列进行了预先检查的。在该区域中也进行了手动搜索,以查找分子机制的序列证据,其指示插入,缺失或特定于特别特异性重组。还在所有成对比对(55,总计)上进行了这种搜索TRNI.-TRNL.插入区域雀稗特别对串联重复序列、分散重复序列和倒置重复序列的存在进行了研究。中间结果表明,存在显著放置的分散重复trnI——trnL插入区域。将分散的重复定位为次区域的终点TRNI.-TRNL.通过注释和预测菜单中的图案搜索选项插入区域,并在佐释序列的序列查看器中。从5bp的最小序列长度进行搜索,用于分散的重复,介绍格拉特等人所建议的缺点缺陷。[39.]。
在对61个分类单元基质进行质体系统基因组学分析之前,将排列产生的任何缝隙从基质中剥离。使用jModelTest v2.1.10为剥离的核苷酸比对选择一个模型[40]并且在Akaike信息标准下选择GTR + I + G型号[41.]。最大可能性(ml)和贝叶斯MC3推理(BI)分析在对准上进行。ML分析通过Xsede V8.2.9上的RaxM1-HPC2进行[42]有关CIPRES科学门户[43]。Bootstrap Reparicates的数量设置为1000,并将所有其他参数设置为默认值。BI分析使用MRBAYES在XSEDE v3.2.3上[44]在Cipres科学门户。指定了两个具有四个链条的独立分析的设置,每个分析每代都有四支链,默认烧伤值为25%。该模型设置为“invgamma”和“nst = 6”,默认情况下所有其他参数。
在野兽V2.4.3中的一个IR中对61个征集汽提对准进行分歧时间的估计[45]。参数包括一个不相关的轻松时钟模型[46[GTR替换模型,具有六个γ的六个,估计形状参数为0.92,初始值为0.52,估计不变量位点比例。从ML分析获得形状参数的初始值和不变位点的比例。
选择为校准点的七种化石基于最近在发散日期分析中使用[4],其年代可靠,并确信与现存的同源物种有关[47,48,49]。给出了这些化石的细节(附加文件1:表S1)。化石校准放在指定的节点上(附加文件)1:表S1)与最小节点年龄设置为化石的地层或辐射性日期,最大年龄设置为淘汰淘汰的最古老的化石[110 mA;49]通常遵循Christin等人的方法。[46]。
BEAST分析是在CIPRES科学门户上进行的,历经4000万代,每1万棵树就被砍伐一次。示踪剂v1.6 [50]用于评估收敛性。TreeAnnotator [51]用于总结树木,燃烧值为25%。随后用封装“带子”来观察具有支撑值的塑料编程图[52]和" ips " [53]在r [54];(无花果。1).
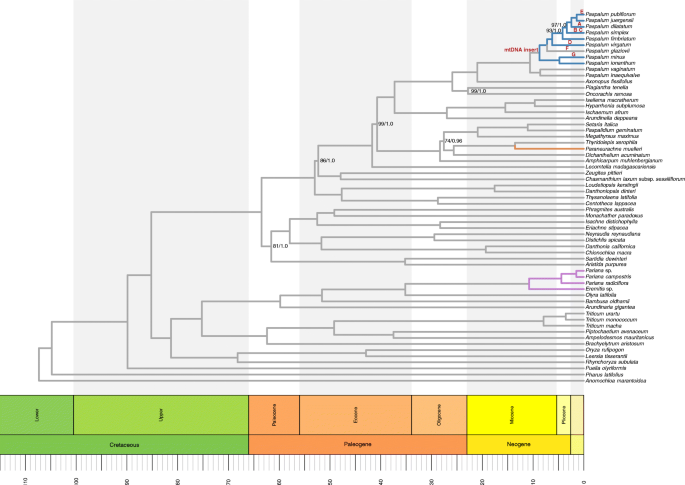
计时图的Poaceae.未恢复最大支持的节点的支持值(ML / BI)物种。最大限度支持所有其他节点。突出显示的分支描述了具有相似插入的不同分类组TRNI.-TRNL.IGS。信中的信件雀稗谱系指定IDE(表2)
四聚物的身份分析
在黑桃中组装的contigs雀稗以已公布的11条Poaceae线粒体序列(NC_007579.1、NC_008362.1、NC_011033.1、NC_007886.1、NC_007982.1、NC_029816.1、NC_013816.1、NC_008360.1、NC_008333.1、NC_008332.1和NC_008331.1)为数据库,在默认参数下对BLASTn进行过滤。使用BBmap“filterbyname.sh”可执行脚本提取具有较高线粒体身份的contigs [55]。使用Binning-Master对五个分区进行了分类[56]以四个核苷酸的滑动窗确定四聚体同一性,前进一,对于大单拷贝(LSC),小单拷贝(SSC),IR没有插入的IR,从BLASTN滤波中检索的线粒体CONTIGS,以及雀稗插入IR。然后将文件导入紧急自组织地图(ESOM)软件[57]为了可视化每个分区的四聚体。用20个时期的K批算法进行训练,具有50的起始半径和150×150块的尺寸。其他参数设置为默认值。
与成对比较相比,ESOM方法提供了更彻底和精确的插入原点估计。ESOM方法统计统计四聚体序列区域并确定它们是否具有相似的标识。我们的假设如下。如果是雀稗插入物为质体来源,则四聚体在其他质体区域(LSC、SSC和IR)周围表现出强烈的上升边界;否则它们就会分布在线粒体序列中。
结果
塑料特色
本研究获得了9个新的完整质体的GenBank登录号(见表)1).不包括TRNI.-TRNL.IGS插入11的区域雀稗物种,整体成对标识为98.3%,而手动对齐TRNI.-TRNL.IGS插入区域相同的占28.6%,这是由于大量的缺口位置。的插入雀稗从976年的BP中等P. DilataTum.至11,393 bpP.Ionanthum..由于这个插入的长度,P.Ionanthum.是目前已知最长的禾本科质体,长度为162086 bp。其他值得注意的品种雀稗是一个776 bp删除psbe-petlIGS在所有雀稗除了p . inaequivalve和P. vaginatum..所有Paspaleae都分享了222家BP删除petA-psbJIGS。
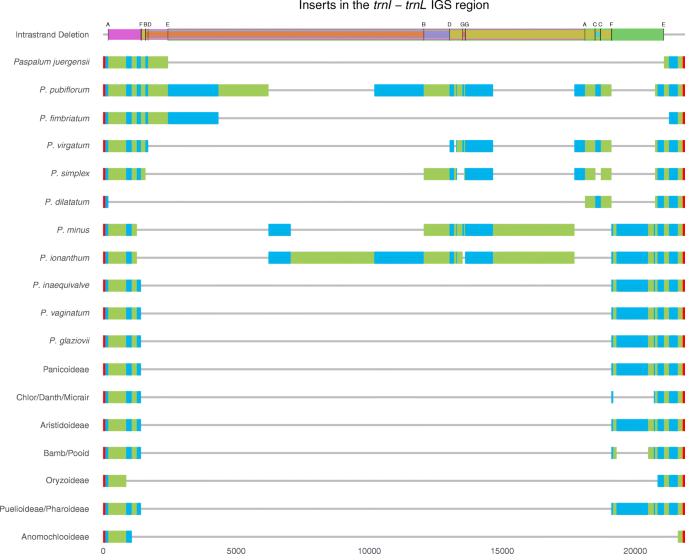
基于手动对齐,21个单独的子区域TRNI.-TRNL.IGS插入被识别。其中有19个在两个或两个以上之间有相同的同源性雀稗物种,另外两个,发现p . publiforum1878 bpP.Ionanthum.长度分别为3146 bp,仅为这些物种所特有。这是可视化的(图。2)使用函数" geom_linerange "在ggplot2 [58],并在tidyverse中使用“选择”和“收集”功能[59在R统计组中强制获取数据。基于对注释区域的广泛搜索,确定了7对离散重复序列,它们立即被推测为链内缺失事件(IDE)的侧翼。分散重复序列大小在8 ~ 17 bp之间,基于比对的缺失区域在112 bp(“IDE G”)到18,645 bp(“IDE E”)之间2).注意,由于通过重组之间的重组介导的通过筛选缺失的自切除,消除了任何给定物种中重复的一个拷贝,仅在比较之间的同源塑性区时识别出分散的重复的存在雀稗sp.最后,一个blast搜索串雀稗插入序列代表祖插入件,17,685bp的长度,返回完全线粒体基因组的顶部遭受Tripsacum dactyloides.(DQ984517)在99%的同一性为33%的序列。
插入比较雀稗物种和跨越普通的亚科。对齐的子区域与交替的蓝色和绿色棒差异化。这些次区域是由代表的红色条形侧翼TRNI.(左)和TRNL.(右)。ide用重叠的彩色条和它们指定的字母表示(表2)
的TRNI.-TRNL.IGS中插入Paraneurachne Muelleri.2808 bp长,与雀稗或Parianinae。blast结果显示92%的人有相同的身份高粱二色的线粒体基因组(DQ984518.1)序列的68%。
质体系统基因组学和差异数据分析
同样的拓扑检索为ML和BI分析。除了ML分析中的7个节点和BI分析中的1个Panicinae节点外,大多数节点都得到了最大支持(图)。1).在分析中,Panicidoideae是Pacmad的其余部分的姐姐,其自举值81%和后概率(PP)为1.0。的雀稗物种被检索为单系,只有两个自引导值小于最大值和所有具有1.0的PP。
相同的拓扑结构是在由发生在v2.4.3 BEAST进行发散日期分析检索。所述PACMAD进化枝的冠节点在63.5马25.9 Ma和Paspalinae在21.0马日与禾本科发散在53.0马,Paspaleae的冠节点。皇冠上的节点雀稗分散在10.6 mA,最近的多元化事件为1.4 mA,差异P. pubiflorum.和P. juergensii从他们的共同祖先。种分歧之间的最短时间是0.9马云,从中单P.从姊妹分支中分离出来的分支包括:P. DilataTum.,P. pubiflorum., 和P. juergensii.发散事件间最长时间为8.6 Ma,分岔为P. vaginatum.和p . inaequivalve最早的世系雀稗在此采样。
四聚物的身份分析
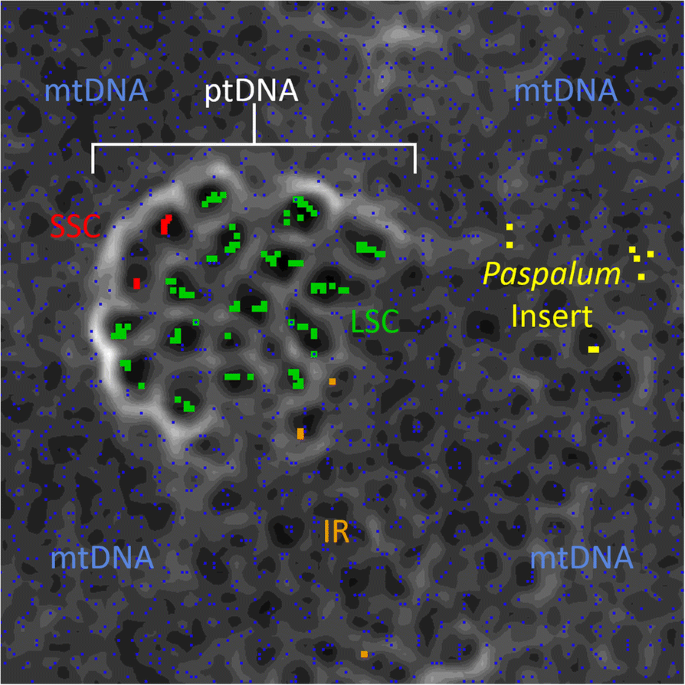
分别为五个分区的产生四聚体基质。将这些文件进行到U形矩阵,这是数据点之间的四聚体频率的距离的可视化,并且表示为映射高度。这些点之间的这些距离在充当截止的升高屏障中进一步可视化,从而表示数据集之间的大差异[56,57]。这用于解释起源雀稗插入,取决于它们发生的地方以及它们在地图上是如何分离的。
检查ESOM结果表明,大多数塑料组合在一起。LSC和SSC集群在大部分点周围具有高“海拔”(白色和浅灰色区域),并且具有较低的“高度”(深灰色和黑色区域)在该屏障内的LSC和SSC的群组合。IR沿着高度不那么高的区域接近LSC / SSC区域。LSC / SSC区域的一组IRS不靠近LSC / SSC区域,位于MTDNA之间,附近没有高“海拔”壁垒。的雀稗插入也位于MTDNA区域内,也在一个具有极少的高“升高”屏障中的区域内。其中一些插入数据点集群,两组两个和一组四。在整个ESOM中,MTDNA点位于eSOM中,但位于LSC / SSC区域内的数据点较少(图。3.).
讨论
塑料特色
在八个新组装的雀稗种,六种含有插入物TRNI.-TRNL.IGS。我们调查了插入的原点是一个或多个事件。来自不同物种的插入物质很少,并且只有一个物种,P.Fimbriatum.,有一个次级插入,它与草的黑穗病寄生虫具有同源性[3.]。但是,虽然每个物种没有序列都完全相同,但这六个新插入物包含在新及以前发现的部分之间共享的高标识的多个子区域雀稗插入[3.]。因此,在21个子区域中的19个(图。2)包含整个抽样的共享序列相似性雀稗的某个点上的单个插入事件雀稗血统(无花果。1).根据目前的取样,假设的祖先插入长度估计为17685 bp左右,而且只发生过一次。这与之前对线粒体在质体中插入的描述一致,这表明虽然这些插入事件发生,但它们是罕见的[25.]。
同源IGS还包含用于新测序的插入物Paraneurachne Muelleri..而这个2808 bp的插入不匹配任何子区域雀稗插入或Parianinae插入,这确实说明质体中的IGS是indel突变的热点。剔除可能有线粒体插入的物种,所有禾本科植物质体IGSs的平均长度为422 bp,而IGSs的平均长度为422 bpTRNI-TRNL.IGS平均为3071基点。这IGS的更大的长度可能是由于大的解体YCF2.CDS,这是Poaceous Plastomes的定义特征之一[60]。塑性Ig的较大尺寸可能在与较大线粒体插入件的重组期间提供较少的空间障碍。未来发现更多草属,其中插入TRNI.-TRNL.IGS将支持该间隔物作为重组突变的突变热点的想法。
解释这些插入长度不同的最可能的突变机制是基因转换后的重复链内缺失。当同源区域的序列替换不均匀时,就会发生基因转换或非互反重组,从而导致一个变异序列的丢失[39.]。为了帮助将这种机制置于语境中,有必要了解植物线粒体基因组。
线粒体已知积极承担,并纳入外源DNA,其中包括ptDNA [61]。一项研究[62,发现22,593 bp的ptDNA位于一个线粒体基因组(AB076665-6)。这只占线粒体DNA的6.3%,但几乎占供体质体的20% (NC_001320)。另一项研究[63[发现两个主要PTDNA插入物,为12.6kb和4.1kb,主要是IR序列,其它插入物总计23.9kb。这仅占线粒体基因组(AY506529)的4.2%,但占各种血浆的17.0%(X86563)。在这些塑料插入中,可以发现完整的基因TRNI.(NC_007982在326704-327726)和TRNL.(NC_007982在336020 - 336100年)。由于线粒体基因组以重排著称,以至于属甚至同属物种之间的基因顺序都不保守[64,65,66,67],可能在不同的禾草类群中,线粒体基因组中与质体同源基因的基因间距离在长度和序列上存在差异。
因此,含有含有PTDNA的保守区域的MTDNA的线粒体基因组中的序列(TRNI.和TRNL.)将有基因转换的潜力。保守的PTDNA将作为在线粒体基因组中的塑性基因座和同源基因座之间的重组点以产生MTDNA插入。不同一般观察到的不同长度也是通过特定于分类水平特异的线粒体基因组内的重排来解释,例如在其中雀稗或帕里亚纳血统。这将创建不同的插入长度以及不同分类水平的单独插入事件的序列内容(雀稗祖:17685基点,Paraneurachne.:2808 BP,Parianina:4920 BP)。
一旦插入件建立的原质体系,该机构用于差分降解被内看到雀稗很容易解释。对这些区域端点的分散重复序列的识别清楚地表明IDE是致病机制。这里有七个ide示例(表2),其具有不同的长度的序列去除,从112个碱基至18,645个碱基,在整个插入物的子区域中雀稗和侧翼ptDNA,精确地在短的分散重复中终止。这些ide,以及其他可能由于抽样的分类子集而无法检测的ide,创建了看似独特的序列,但这只是从原始插入中删除序列的各种方法的结果。
IDE还可以解释一个物种的单一情况,P. glaziovii,它不包含mtDNA插入物。有证据表明其中有一个IDE雀稗可以删除整个插入件。在序列附近的序列中可以在早期找到上游重复(16bp的长度)TRNI., 为了P. juergensii,P.Fimbriatum.,P. pubiflorum.,p . virgatum, 和单P.,而下游的精确重复更接近TRNL.基因P. minus, P. ionanthum, P. glaziovii, P. inaequivalve,和P. vaginatum..在这一点上没有雀稗种具有此重复的两个副本。在包含一个重复复印和插入序列的物种,很可能是一个小的IDE删除重复的一个。这样做将保留原始插入的较大部分,和什么其他的IDE可以发生基于第一或第二个重复是否删除了潜在的创造突发。
通过移除其中一个重复保留插入件的假设较小的ides也已经消除了其他IDE的可能性。这是一个例子在开始时p -和P.Ionanthum,在“IDE F”之前,这缺少一个次区域,可能是由于IDE较小的。这种身份不明的IDE最有可能删除“IDE F”的第一个重复,而且还可以删除“B”,“D”和“E”的第一个分散重复。因此,基于先前IDE的未来IDE的差欲和理解IDE的优先级将更好地确定进一步的抽样帕斯普拉姆谱系(例如piot等人。[10.]和未来的研究)。
系统基因组学和差异数据分析
拓扑雀稗本研究的物种很好地支持ML分析中的所有节点,并且在BI分析中最大地支持的所有节点。对于代表物种的分歧毫无疑问,我们发现没有刀片证据的物种是非单态的。p . inaequivalve和P. vaginatum.缺两种插入件,都在本研究中最早的分歧的疏水板,但是P. Glaziovii,其中也缺乏插入,是更近分歧的内部雀稗血统。这表明缺少插入不是祖先的特征。根据目前的取样,插入物很可能出现在包含所有进化枝的地方雀稗除了p . inaequivalve和P. vaginatum..这可能会解释原因P.Ionanthum.有最长的插入(11,393 bp),因为它靠近插入事件的原点。随着插入起始点的确定,从外源DNA插入到ide的时间将进一步了解质体如何与插入的非ptdna相互作用。
在计时图系统发育,对于形态选择发散日期有效地代表了插入事件和花费的IDE成为固定在质体时的假想时间。如果插入出现8.7左右马,插入在随后的损失P. Glaziovii发生在7.2 Ma,然后“IDE F”固定发生在大约1.5 Ma的时间内。其他IDE (A, B, D, E, F)也在相对较短的时间内固定,从最小的1.4 Ma(“IDE E”)到3.3 Ma,除了“IDE D”在6.2 Ma。这些更长的时间估计可能是由于缺乏采样,并不能分解较长的分支与物种丰富的属雀稗(〜350 spp。)[29.]。因此,ide,特别是较大的ide,在相对较短的时间内被固定,可能意味着对原始插入进行了选择。
祖先的假设插入尺寸超过17,000bp,不包含可识别的编码序列。插入物越长,塑料的复制过程中需要越多的能量和材料。这可能对整个系统发育中看到的各种不同的IDE。虽然某些物种雀稗缺少一个或两个可以删除插入的重复,“IDE F”,它们包含其他IDE,可以缩短插入或用侧翼的PTDNA删除大部分插件(P. juergensii,P.Fimbriatum., 和P. DilataTum.).因此,较小的质体,很少或没有插入,可能比较大的质体更有利于质体的整体。
四聚物的身份分析
如果源不是来自MTDNA,则确定插入的原点也可能具有其他含义也很重要。为此,ESOM分析(参见视觉矩阵输出,图。3.)进行了比较四核苷酸的频率来评估我们的起源假说的支持。的雀稗在ESOM矩阵内插入并未与其他PTDNA集群,但相当聚集到MTDNA之间的较小组中。来自ESOM矩阵(图。3.),有一个明显的高“海拔”围绕ptDNA。ptDNA的这个界限靠近ptDNA簇的顶部和左侧部分更加明显,与“标高”,因为它更接近与IR序列的部分的损失。这可以通过有,它们位于许多禾本科物种的线粒体DNA [IR的大部分进行说明62,63]。因此,存在一些相似性,从而围绕IR点左右创建较低的“高度”。将pTDNA掺入MTDNA也是为什么PTDNA簇中的MTDNA点数有点[62,63]。还有一个IR点明显远离ptDNA簇的其他部分。这很可能是在反向重复序列中发现的rrna。这些区域富含GC (雀稗在本研究中的其他Poaceae物种:GC%= 54.7%),因此看起来更像MTDNA,这整体也更多GC富含(AY506529:GC%= 43.9%,AB076665-6:GC%= 43.9%)比总体在富人的ptdna(雀稗:GC%= 37.8%,本研究中的POACEAE物种:GC%= 37.6%)。
我们对这些结果的解释是,插入物是原点的MTDNA,然后通过先前所提出的机制转移到PTDNA中。这种可能性,曾经认为是不可能的或不太可能的[23.,24.),直到现在才变得明显,因为更多的NGS工作已经完成[3.,9,10.,14.,28.]。
结论
总之,雀稗插入TRNI.-TRNL.在11个银币塑料中的八个中观察到IGS。原始插入物被确定为具有暗示MTDNA起源的MTDNA的相似性。与其他线粒体塑料相比,这是由高对同一性的支持,以及在四核苷酸分析中的插入物的放置。这个MTDNA在包含全部的人工间都会出现雀稗除了可能p . inaequivalve和P. vaginatum.大约8.7 mA,随后在某些物种中丢失了MTDNA序列。插入中的删除事件并不总是删除,但是删除了序列的大次区域的一般趋势。这是在整个过程中看到的雀稗谱系,确定了七种不同的IDE。这些IDE在给定的物种谱系中在相对较小的时间内除去大部分序列,其中最短的是在1.4 mA时注意到。因此,这表明可能存在进化压力以从塑料中除去这种过量的mTDNA。
缩写
- 英亩:
-
锚定节约区延期
- 野兽:
-
贝叶斯进化分析采样树木
- 双:
-
贝叶斯MC3推断推断
- BLASTN:
-
核苷酸基本局部比对搜索工具
- BP:
-
碱基对
- Cipres:
-
系统发育研究的网络基础设施
- Contigs:
-
连续序列
- esom:
-
紧急自组织映射
- gtr + g + i:
-
一般时间可逆加上伽玛分配加上不变网站的比例
- IDE:
-
链内删除事件
- IGS:
-
代际垫片
- 红外光谱:
-
反向重复
- KBP:
-
千碱基对
- LSC:
-
大单拷贝
- 嘛:
-
Megaannus或百万年
- Mafft:
-
快速傅里叶变换的多重对齐
- ml:
-
最大似然
- MTDNA:
-
线粒体DNA
- NCBI:
-
国家生物技术信息中心
- ngs:
-
下一代测序
- PACMAD:
-
青藤亚科、青藤亚科、绿藤亚科、小藤亚科、青藤亚科
- PP:
-
后验概率
- PTDNA:
-
质体DNA
- SSC:
-
小单副本
参考文献
- 1。
Judd WS, Campbell CS, Kellogg EA, Stevens PF, Donoghue MJ。植物系统学:系统发育学方法。北美杜鹃花。2016;118(976):418 - 20。
- 2。
Burke SV,Clark LG,Triplett JK,Grannan CP,Duvall Mr。新世界Bambusoideae(Poaceae)的生物地理和系统核科学检测,重新审视。我是J机器人。2014; 101(5):886-91。
- 3.
Burke SV,Wysocki WP,Zuloaga Fo,Craine JM,Pires JC,Edger PP等。基于血糖植物核糖核生物学(胰腺炎; POACEAE)的胰腺进化关系。BMC植物BIOL。2016; 16(1):140。
- 4.
Burke SV,Lin CS,Wysocki WP,Clark LG,Duvall Mr。热带森林草(Leptaspis,Streptochaeta:Poaceae)的系统核糖学和塑料演变。前植物SCI。2016; 7:1-12。
- 5.
棉jl,wysocki wp,clark lg,kelchner sa,pires jc,edger pp等。解决Pacmad草的深层关系:一种系统染色方法。BMC植物BIOL。2015; 15(1):178。
- 6.
Duvall MR, Fisher AE, Columbus JT, Ingram AL, Wysocki WP, Burke SV等。绿甲草(chloridoid grass: Poaceae)的系统基因组学和质体进化。acta botanica sinica(云南植物学报),2017;
- 7.
Duvall先生,Yadav SR, Burke SV, Wysocki WP。草质体揭示了与印度特有种Micrairoideae和Arundinoideae的新的单倍型标记。acta botanica yunnanica(云南植物研究). 2017;104(2):286-95。
- 8.
从禾本科的系统发育学、分子进化和谱系年龄的估计。acta botanica sinica(云南植物学报). 2014;40(6):1421 - 1436。
- 9.
马鹏飞,张应祥,郭振华,李德志。竹子属中线粒体DNA水平转移到质体基因组的证据。Sci众议员2015;5:1-9。
- 10。
PIOT A,Hackel J,Christin Pa,BESNARD G.三分之一的塑体基因在Pacmad草的阳性选择下进化。Planta。2018; 247(1):255-66。
- 11.
Saarela JM, Wysocki WP, Barrett CF, Soreng RJ, Davis JI, Clark LG, Duvall MR. Plastid systems genomics:澄清早期分化部落之间的关系。AoB植物。2015;7:1-27。
- 12.
Teisher JK,Mckain Mr,Schaal Ba,Kellogg EA。芳宁肌益叶植物(Poaceae)的多重和扭曲胰腺引理的演化。anghars botany。2017; 120(5):725-38。
- 13。
吴ZQ,GE S.草地上的BEP思工的系统发育重新判断:来自叶绿体的全基因组序列的证据。mol phylocyet evol。2012; 62(1):573-8。
- 14。
Wysocki WP, Clark LG, Attigala L, Ruiz-Sanchez E, Duvall先生竹子的进化(竹科;一个完整的质体系统基因组分析。BMC Evol Biol. 2015;15(1):50。
- 15.
张YJ,MA PF,LI DZ。六竹叶绿体基因组的高通量测序:温带木质竹子(Poaceae:Bambusoideae)的系统发育意义。Plos一个。2011; 6(5):E20596。
- 16.
Arthan, W., M. R. mccain, P. Traiperm, C. A. D. Welker, J. K. Teisher, E. A. Kellogg。(2017)。东南亚矮嵩草科(禾本科)的亲缘关系。Syst Bot:出版中。
- 17.
Attigala L, Wysocki WP, Duvall MR, Clark LG。基于胞浆体系统发育分析的青竹科系统发育及形态进化研究。分子系统学进展。2016;101:111-21
- 18.
中国科学院植物分类与系统基因组学重点实验室,中国科学院植物分类与系统基因组学重点实验室。安机器人。2013;112(6):1057 - 66。
- 19.
Washburn JD, Schnable JC, Davidse G, Pires JC。草族Paniceae的系统发育和光合作用。acta botanica sinica(云南植物学报). 2015;40(9):1493-505。
- 20。
Lundgren Mr,Besnard G,Ripley BS,Lehmann Ce,Chatelet DS,Kynast Rg等。光合创新在单一物种中拓宽了利基。ECOL LETT。2015; 18(10):1021-9。
- 21。
olofsson jk,bianconi m,besnard g,迪瑞宁Lt,Lundgren Mr,Holota H等人。基因组生物地理揭示揭示了复杂性状的适应性突变的内部涂抹。Mol Ecol。2016; 25(24):6107-23。
- 22。
玉米属(Zea)物种的Plastid系统基因组学研究:3类微观结构变化的速率和模式。咕咕叫麝猫。2017;63(2):311 - 23所示。
- 23。
米DW,Palmer JD。塑体中的特殊水平基因转移:遥远的细菌鉴定性替代的基因,证据表明患有耳咽和加密体积血液的证据。BMC BIOL。2006; 4(1):31。
- 24。
Richardson Ao,Palmer JD。植物水平基因转移。J Exp Bot。2006; 58(1):1-9。
- 25。
线粒体到质体的DNA转移:确实发生了。新植醇。2014;202(3):736 - 8。
- 26。
Goremykin VV,Salamini F,Velasco R,Viola R. vitis Vinifera的线粒体DNA和猖獗的水平基因转移问题。mol Biol Evol。2009; 26(1):99-110。
- 27.
斯特劳布SC,Cronn RC,爱德华兹C,菲斯男,从线粒体DNA的利斯顿A.水平转移到质体基因组和在其马利筋后续演进(夹竹桃科)。基因组Biol Evol。2013; 5(10):1872至1885年。
- 28.
Gandini Cl,Sanchez-Puerta MV。植物线粒体中的外塑序列经常通过线粒体到线粒体水平转移获得。SCI REP。2017; 7:1-8。
- 29.
Scataglini MA,Zuloaga Fo,Giussani LM,Denham SS,Morrone O.新世界Paspalum(Poaceae,Panicoideae,Paspaleae)的系统发育基于塑性和核标志物。植物系统中的Evol。2014; 300(5):1051-70。
- 30.
考克斯议员,彼得森检察官,比格斯律师。SolexaQA:对Illumina第二代测序数据进行一目了然的质量评估。BMC生物信息学。2010; 11(1): 485。
- 31。
Martin M.Cutadapt从高吞吐量测序读取中删除适配器序列。embnet.journal。2011; 17(1):10-2。https://doi.org/10.14806/ej.17.1.200.
- 32。
王志强,王志强,王志强,等。一个新的基因组组装算法及其在单细胞测序中的应用。中国生物医学工程学报。2012;19(5):455-77。
- 33。
李伟,高德泽。Cd-hit:一种用于聚类和比较大组蛋白质或核苷酸序列的快速程序。生物信息学。2006;22(13):1658 - 9。
- 34。
Wysocki WP,Clark Lg,Kelchner SA,Burke SV,Pires JC,Edger PP,Duvall Mr。基层短读全塑性序列组装方法的多步比较。分类。2014; 63(4):899-910。
- 35。
Kearse M,Moir R,Wilson A,Stones-Havas S,Cheung M,Sturrock S等人。佐芙基本:用于组织的集成和可扩展的桌面软件平台和序列数据分析。生物信息学。2012; 28(12):1647-9。
- 36。
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30(4):772-80。
- 37。
两种新世界竹(arundinaria gigantea和Cryptochloa strictiflora (Poaceae))的质体序列扩展了对竹亚科(Bambusoideae)的系统基因组学理解。acta botanica sinica(云南植物学报),2012;
- 38。
Altschul SF, Madden TL, Schäffer AA, Zhang J, Zhang Z, Miller W, Lipman DJ。gap BLAST和PSI-BLAST:新一代蛋白质数据库搜索程序。核酸学报1997;25(17):3389-402。
- 39.
Graur D, Sater AK, Cooper TF。分子和基因组进化。马萨诸塞州:西纳奥尔联合公司;2016.
- 40。
Darriba D,Taboada GL,Doallo R,Posada D. JModeltest 2:更多型号,新启发式和并行计算。NAT方法。2012; 9(8):772。
- 41。
Akaike H.新看统计模型识别。IEEE Trans自动控制。1974; 19(6):716-23。
- 42。
Stamatakis A. Raxml版本8:一种系统发育分析和大脑发育后的分析。生物信息学。2014; 30(9):1312-3。
- 43。
基于CIPRES的大型系统发育树推断的科学门户。在:网关计算环境研讨会(GCE), 2010;2010.1 - 8页。Ieee。
- 44。
Ronquist F, Teslenko M, Van Der Mark P, Ayres DL, Darling A, Höhna S, et al.;MrBayes 3.2:跨大模型空间的高效贝叶斯系统发育推理和模型选择。系统医学杂志。2012;61(3):539 - 42。
- 45。
Bouckaert R,Heled J,Kühnertd,沃恩T,Wu Ch,谢D等人。BEAST 2:一个软件平台贝叶斯进化分析。PLOS计算BIOL。2014; 10(4):e1003537。
- 46。
Christin PA, Spriggs E, Osborne CP, Strömberg CA, Salamin N, Edwards EJ。分子年代测定,进化速率和草类的年龄。系统医学杂志。2014;63(2):153 - 65。
- 47。
Vicentini A,Barber Jc,Aliscioni SS,Giussani LM,Kellogg EA。草地的年龄和C4光合作用的起源簇。Glob Chang Biol。2008; 14(12):2963-77。
- 48。
史密斯Sy,Gandolfo Ma,Graham Sw。单子叶化石适用于分子约会分析。Bot J Linn Soc。2015; 178(3):346-74。
- 49。
波伊纳尔,斯蒂芬市议员,温德里奇。一亿年前的麦角草:白垩纪的精神药物。Palaeodiversity。2015;8:13-9。
- 50。
谢丹丹,张晓东,张晓东。(2015)。示踪剂v1。6.2014.
- 51。
兰博,德拉蒙德,AJ。TreeAnnotator v2。1.2.爱丁堡:爱丁堡大学进化生物学研究所;2014.
- 52。
贝尔马克,A.,&Lloyd,G.T.(2014)。表带:古生物学的地层树分析。R包版本1.4。
- 53。
Heibl, c(2008)。起。系统发育:R语言树绘图工具和各种系统发育软件包的接口。
- 54。
R核心团队(2017)。R:统计计算的语言和环境。R基金会统计计算,维也纳,奥地利。URL.https://www.r-project.org/.
- 55。
布什内尔B. BBMap短读对齐。加利福尼亚州:加州大学伯克利分校;2016.http://sourceforge.net/projects/bbmap.
- 56。
Dick GJ, Andersson A, Baker BJ, Simmons SS, Thomas BC, Yelton AP, Banfield JF。全社区微生物基因组序列特征分析。基因组医学杂志。2009;10:R85。
- 57。
吴志强,吴志强(2005)。ESOM-maps:使用紧急SOM进行聚类、可视化和分类的工具。
- 58。
韦翰H. GGPLOT2:优雅的图形进行数据分析。纽约:Springer-Verlag;2009年。
- 59。
威克姆,H.(2017)。TidyVerse:轻松安装和加载'Tidyverse'软件包:R包版本1.1。0。
- 60。
MAIER RM,Reckermann K,Igloi GL,KösselH。玉米叶绿体基因组的完整顺序:基因含量,发散热点和转录信息的遗传信息的微调。J Mol Biol。1995年; 251(5):614-28。
- 61。
Koulintchenko M,Konstantinov Y,Dietrich A.植物线粒体通过渗透性过渡孔隙复合物积极进口DNA。禁止J. 2003; 22(6):1245-54。
- 62。
野津Y,马苏德S,西川T,久保N,Akiduki G,Nakazono M,等。频繁的DNA序列采集和损失开花植物的进化过程:将水稻(Oryza sativa L.)线粒体基因组的完整序列。Mol Gen Genomics。2002; 268(4):434-45。
- 63。
等。玉米NB线粒体基因组序列及比较分析。植物杂志。2004;136(3):3486 - 503。
- 64。
努普五,陆生植物的线粒体DNA:在进化的角度特殊性。Curr Genet。2004; 46(3):123-39。
- 65。
Palmer JD, Herbon LA。植物线粒体DNA在结构上进化迅速,但在序列上进化缓慢。中国科学(d辑:地球科学)1988;28(1-2):87-97。
- 66。
Richardson AO,米DW,年轻GJ,Alverson AJ,Palmer JD。LiriDendron Tulipifera的“僵化”线粒体基因组:祖先基因含量和秩序,祖先编辑位点,突变均低突变率。BMC BIOL。2013; 11(1):29。
- 67。
Satoh M,Kubo T,Nishizawa S,estiati a,itchoda n,mikami t。糖甜菜的细胞质雄性无菌型和正常型线粒体基因组共享相同的已知功能基因,但表达ORF的含量不同。Mol Gen Genomics。2004; 272(3):247-56。
- 68。
Prasad V,StrömbergCae,LeachéAd,Samant B,Patnaik R,Tang L等。稻米部落的后期白垩纪起源为淘汰淘汰的初期多样化提供了证据。NAT Communce。2011; 2:480。
- 69。
MacGinitie高清。科罗拉多州弗洛里森特床层的植物化石。卡内基研究所华盛顿出版559。198 p。1962。Kilgore植物群,内布拉斯加州北部的晚中新世植物群。中国地质大学学报(自然科学版)。1953;3(5):67-158。
- 70.
Walther H.ErgänzungenZur Flora Von Seifhennersdorf(Sachsen),I.Teil.-Abh。staatl。亩。矿物。Geol。1967年; 12:259-77。
- 71.
Dugas DP, Retallack GJ。中中新世肯尼亚特尔南堡的草化石。J Paleontol。1993;67(1):113 - 28。
- 72。
王志强。晚新生代隐杆草外稃和外稃的特征。Am J Bot. 1978:34-9。
- 73。
来自高地平原的第三纪草原草和其他草本植物。陆军特种部队1942;41:7-171。
确认
我们要感谢L. Attigala提交订单到ISU DNA设施。特别感谢J. Craine谁在这项研究中使用提供的样品。我们也感谢基础的筛选在分子进化类的2017年初步PCR学生雀稗插入。
资金
这项工作得到了植物分子生物学中心、北伊利诺伊大学生物科学系和美国国家科学基金会的部分支持,MCU和MRD的授权编号为DEB-1120856, MRD的授权编号为DEB-1120761。本材料中表达的任何意见、发现、结论或建议都是作者的观点,并不一定反映美国国家科学基金会的观点。
可用性数据和材料
支持我们结论的数据可从通信作者在合理的要求。所有核苷酸序列均保存在NCBI Genbank库中,登录号见表1.
作者信息
隶属关系
贡献
SVB进行了数据采集、Nextera文库准备、质体组装、比对、系统基因组分析并起草了手稿。MCU提供了来自美国农业部国家种质库的植物材料用于测序。MRD协助数据采集,Nextera图书馆的准备工作,对研究的设计做出了贡献,并促进了合著者之间的互动。所有作者以编辑的方式阅读并协助手稿的起草。所有作者阅读并批准了最终的手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Burke,S.V.,Ungerer,M.C.&duvall,m.r.对线粒体衍生的塑料序列的研究雀稗谱系(胰腺炎; Poaceae)。BMC植物杂志18,152(2018)。https://doi.org/10.1186/s12870-018-1379.1.
已收到:
接受:
发表:
关键词
- 草
- 插入
- MTDNA.
- 下一代测序
- 雀稗
- Poaceae.
- PTDNA