摘要
背景
地瓜(番薯甘薯(L.)林)是从家庭中最重要的粮食作物之一旋花科植物.据广泛报道,栽培甘薯起源于中国番薯trifida.然而,二倍体,四倍体和六倍体即trifida都存在于自然界中。它们之间,以及它们与红薯之间的关系尚不清楚。
结果
本研究检测了甘薯与不同多倍体类型的基因组多样性及其相互关系即trifida使用限制位点相关DNA测序(RAD-seq)。共鉴定出38605个rad5标签,包含832,204个snp。这些标签使用5个公共数据库进行注释,约有11519个标签与不同途径的功能基因进行了比对。基于SNP基因型,系统发育关系分析结果证实栽培甘薯与即trifida6×比即trifida4 x和即trifida2×。共检测到5042条SSRs即trifida共获得3202对优质SSR引物。随机选取并合成68条引物,成功扩增61条。
结论
这些结果为栽培甘薯的起源提供了新的证据即trifida6×,即trifida6×进化而来即trifida4 x和即trifida2×。因此,使用即trifida6×作为甘薯模式植物的研究应该比使用更实用即trifida2×在未来。同时,本研究所获得的序列信息和标记也将为甘薯种质资源的研究提供参考即trifida未来的研究。
关键信息
本文报道了利用radseq技术发现的全基因组SNP&SSR和SSR标记的开发,并对甘薯和不同多倍体的系统发育进行了分析番薯trifida.
背景
番薯,约600-700种旋花科植物[1].该属野生种约有13种番薯部分甘薯.地瓜(番薯甘薯(l)是唯一一种在100多个国家广泛种植的主要作物[2].它在许多非洲国家的粮食安全中发挥着重要作用[1,3.].然而,在遗传学和基因组学甘薯的研究远远落后于它的复杂的基因组结构等主要农作物杂交不亲和性[中4].
到目前为止,甘薯的祖先和驯化尚不清楚。目前关于甘薯与野生种关系的研究还很少[5,6,但关于甘薯的起源仍然存在一些争论。人们普遍认为即trifida是最近的野生甘薯品种[7,8].然而,甘薯与野生种之间的亲缘关系尚未完全阐明。
甘薯野生近缘种具有耐旱、耐盐、抗病、淀粉含量高等优良性状。它们为甘薯育种和品种改良提供了宝贵的基因库,为甘薯的遗传研究提供了新的途径。因此,研究甘薯与近缘种的亲缘关系是今后甘薯遗传研究的重要内容。
几种假说已被应用到揭示甘薯的起源。最近的研究主要集中在两个方面对种植甘薯的起源[5,9,10].第一个假说提出甘薯的祖先来自于两者的杂交即trifida和即triloba.另一种假说提出了二倍体之间的杂交即leucantha和它的多倍体四倍体即littoralis抱生成的三倍体番薯trifida(H.B.K.)堂。,which spontaneous polyploidy originated wild ancestor of hexaploid一、甘薯.虽然有两种假说支持红薯起源模型的不同,但它们都承认这一点即trifida是甘薯的祖先之一。
即trifida(h . b . k .)g .堂。具有复杂的多倍体形式,包括二倍体(2N= 2× = 30),三倍体(2N= 3× = 45),四倍体(2N= 4× = 60)和六倍体(2N= 6× = 90),与栽培甘薯杂交亲和性好[5].此外,一些分子遗传学和细胞遗传学的研究表明即trifida是栽培甘薯最近的野生亲缘种[7,被认为是甘薯最可能的候选祖先。
随着下一代测序(NGS)技术的应用,基因组学近年来得到了迅速发展。越来越多的植物和动物物种的全基因组序列被公布[11].然而,许多农作物和动物仍然没有因为基因组的复杂基因组参考。这种情况将很难在短期内改变。对于这些品种,测序(RRLS)减少,表示库被认为是最有效和最经济的选择在基因组学研究[12,13,14].限制位点相关的DNA测序(RAD-SEQ)是最广泛使用的RRLS技术之一,它结合了低成本和高吞吐量[优点15,对于缺乏参考基因组的物种的基因组研究尤其有用。现在,RAD测序被广泛应用于遗传多样性、生态和进化遗传学、遗传作图和分子标记发展研究[16,17].
在本研究中,我们使用RAD-seq(1)评估该方法的应用价值,(2)在甘薯及其近缘野生物种中检测到大量的SNPs即trifida(3)评价甘薯种质与不同多倍体形态的系统发育关系即trifida,(4)检测SSR位点即trifida6×,开发SSR标记。这些研究结果将有助于我们更好地理解植物的系统发育关系和遗传多样性一、甘薯种及其假定的祖先即trifida.
结果
RAD-seq和SNP发现
通过对包括甘薯品种在内的27个样本的基因组测序,即trifida(2×, 4X, 6×)和合成接入,共获得37.29 Gb高质量序列数据,包含100,507,572对端读和7,843,552单端读。每次访问的原始读取数量从6,453,618到23,422,064不等,平均为9,320,100。尽管甘薯和即trifida6×的尺寸比即trifida2×和即trifida4X,原始读的总数没有显著差异。总原始读取数为45236943。红薯的原始读数为254.5682万~ 999.4361万,平均为383.1194万。不同的多倍性类型即trifida,即trifida2×的平均原始读取为3,965,810,平均原始读取为3,511,641即trifida4X, 2,975,540个原始读取来自即trifida6×。在过滤原始读取之后,生成了8,585,422个高质量的读取标签。对于每个样本,读取标签的数量从209,850到608,047不等,平均为317,978(表1和2).
在将RAD-seq reads分组到rad -tag后,发现测序深度在不同位点之间存在显著差异,这在许多其他基于RAD-seq的研究中已经观察到。大部分位点的覆盖率在8.6 ~ 21.98之间。多态性位点的平均覆盖率为12.151:图S1),处于中等水平(表2).
所有候选等位基因在栽培甘薯和不同多倍体类型之间进行了鉴定即trifida采用SNP基因型聚类。最终,27个样品中有38605个可比较的rad标签,鉴定出832,204个snp。A/G型、T/C型和A/C型的SNPs分别占32.87%、29.17%和13.34%。
甘薯与即trifida
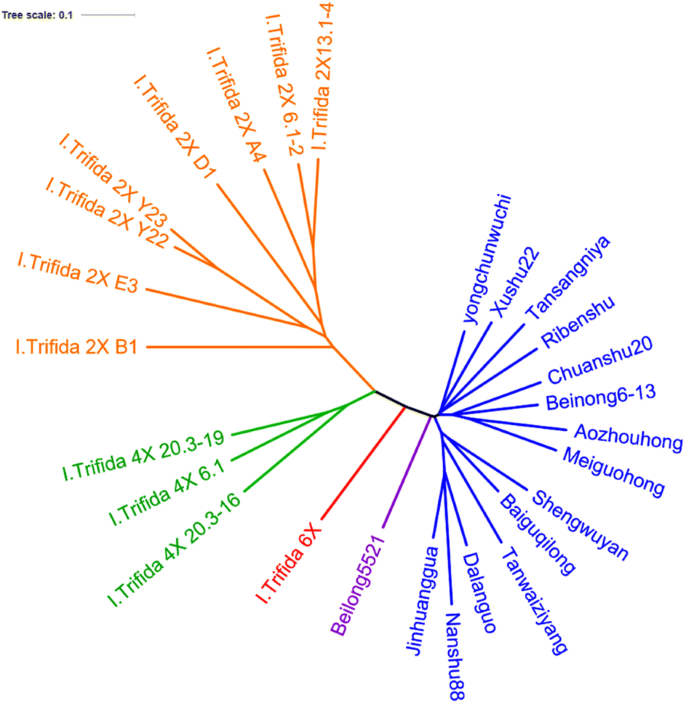
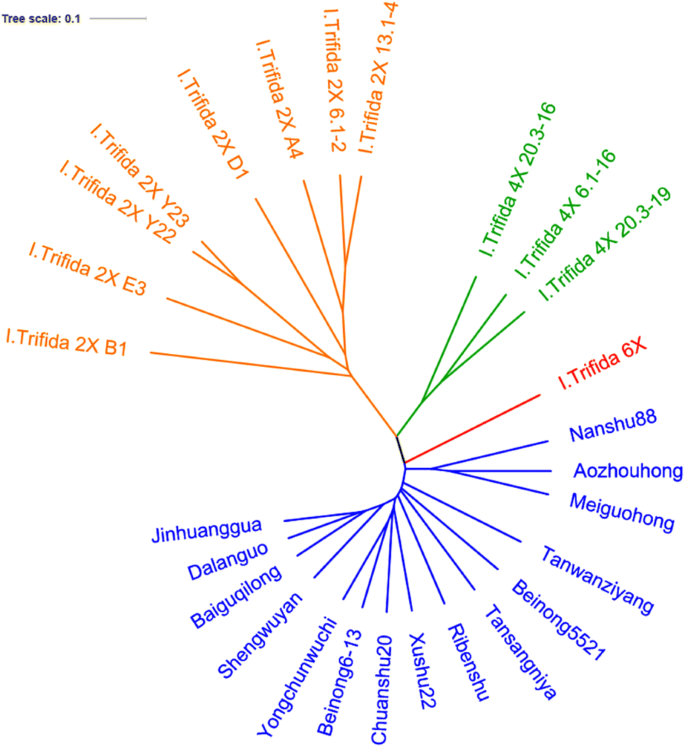
利用Phylip、PLINK和fastSTRUCTURE等方法,揭示了27种植物的进化历史和种群结构一、甘薯和即trifida.结合所有SNPs的数据矩阵进行系统发育分析表明,以简约遗传距离和邻居连接构建的树具有几乎相同的拓扑结构(图2)。1和2).本研究将27份被试材料按0.02884的距离分为3个主要组。第1类群包括13个甘薯品系和一个合成品系北农6-13,可以清晰地划分为3个类群。白谷奇龙、圣武岩、大国、金黄瓜、潭万紫阳等5个地方甘薯品种与现代品种南薯88聚在一起。另一个集群包含一个日本品种,一个坦桑尼亚品种和两个中国品种。最后一个聚类包括美国的梅国红和澳大利亚的敖州红,中国现代品种川树20和合成系北龙6-13(图6-13)。1).第二组有8人即trifida2 x登记入册。这些材料具有明显的特征即trifida2X,如茎细,叶小,种子小。此外,根据细胞学观察,该组家蚕均有30条染色体。第三组位于第一组和第二组之间,由三个人组成即trifida4 x登记入册,一个即trifida6× accession and 1个合成系北龙5521(图。1).系统进化树显示即trifida6×与即trifida4 x比即trifida2 x。
利用SNP数据集进行遗传相似度计算,构建系统发育关系。UPGMA的遗传相似性为0.1779 ~ 0.06699。基于简单匹配系数构造了树状图。在距离0.02295处,树状图上聚为两个类群,一个类群中存在两个截然不同的亚类群,这与系统发育分析结果基本一致(图2)。2).
第1组包含所有八个即trifida2X,但他们之间的基因相似性不同。即trifida2X B1与其他基因型差异最大。即trifida2 x和每个即trifida2X Y22的亲缘关系最接近,遗传相似度为0.05087,其次为即trifida2 x 13.1 - 4即trifida2X 6.1-2,遗传相似性为0.08058。各品种间存在丰富的遗传多样性即trifida这组是2X。第二组包括三个人即trifida4 x登记入册,一个即trifida6X和所有甘薯品种。在这组中可以识别出两个不同的亚组。一个子群只包含三个子群即trifida4 x登记入册。有趣的是,另一组只有一个人即trifida6X和所有甘薯品种。它显示即trifida6X与一、甘薯比,即trifida4 x和即trifida2 x。除了即trifida6X,在这个亚组中有一点遗传差异。南薯88、美国红和敖州红在甘薯品种上表现出差异。其中两种分别来自美国和澳大利亚,南薯88是一种广泛种植的中国红薯。其他甘薯种质在树状图上具有较高的遗传相似性。此外,人工合成种北农5521和北农6 - 13与其他野生种的亲缘关系较近(图5)。2).
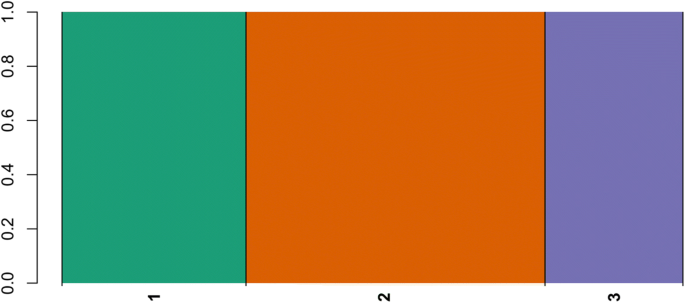
为了进一步揭示遗传结构,利用fastSTRUCTURE软件对所有有效SNP标记的K值从1到10进行分析。采用基于模型的聚类分析,描述数据的最优聚类数为K = 3,这区分了即trifida2 x,培养一、甘薯登记入册,即trifida6 x,培养一、甘薯登记入册,即trifida4 x。这一分组结果也基本证实了系统发育分析结果和遗传相似性。即trifida2个加入由集群K1表示。三种登记入册的即trifida4X聚类在K3中,K3中含有3个甘薯品种,即南薯88、丹蓝果和金黄瓜。一、甘薯研究结果将其分为K2和K3类。除了三个一、甘薯入库被归因于即trifida4X集群(K3),大多数一、甘薯研究结果表明,该品种为K2,包括地方品种和现代品种。有趣的是,即trifida6X和两个合成物种也被分为K2组。因此,聚类结果表明了两者之间的遗传关系一、甘薯和即trifida2X与。的系统发育关系相去甚远即trifida4 x和即trifida6 x。相比之下,遗传距离揭示了这一点即trifida6X与甘薯的遗传关系比与甘薯更近即trifida4 x和即trifida2 x(图。3.).
SSR位点检测即trifida6x.
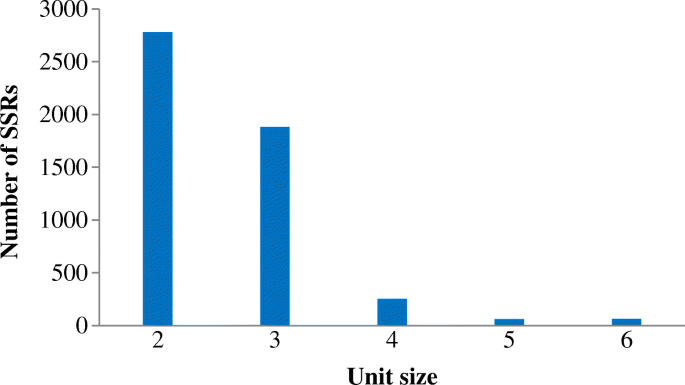
为了进一步检测SSR位点,MISA脚本(http://pgrc.ipk-gatersleben.de/misa/),并使用默认设置进行分析即trifida6 x。标准包括50 bp的最小匹配,95%的最小同一性重叠区域和20 bp的最大未匹配悬垂。结果表明,从180,286 reads in的55,167,516 bp中共鉴定出5042个SSRs即trifida6X,平均每3.6 kb有1个SSR。2951个SSR包含序列(见表)3.).综上所述,SSR单元大小所占比例分布不均匀。2个单位大小型2779个(55.12%),其次为三核苷酸重复基序(trinucleotide repeat motif) 1882个(37.33%)。总共381个(7.56%)ssr的单位大小在3到6之间(附加文件2:表S1)4).共包含1个以上SSR的序列1812条,有694条以上重复类型的复合序列(见表)3.).
5042个SSRs中,4172个SSRs大小在10 ~ 20 bp之间,高度分散,占82.74%,其次为21 ~ 30 bp(715个,14.18%)2:表S1)。三核苷酸重复序列(Tri-nucleotide repeats, TAA)最长为69 bp。共鉴定出224个SSR基序,其中二、三、四、五、六核苷酸重复序列分别有4、10、31、67和112个类型。RAD序列中以AT/TA二核苷酸重复序列最为丰富(779,15.45%),其次为GA/AG(721, 14.30%)、TC/CT(696, 13.80%)、TG/GT(298, 5.91%)、AC/CA(279, 5.53%)、AAT/TAA(93, 4.01%)和TTC/CTT(166, 3.29%)。其余161种母题的频率占3.19%(图2)。4).
发展和SSR标记的评价
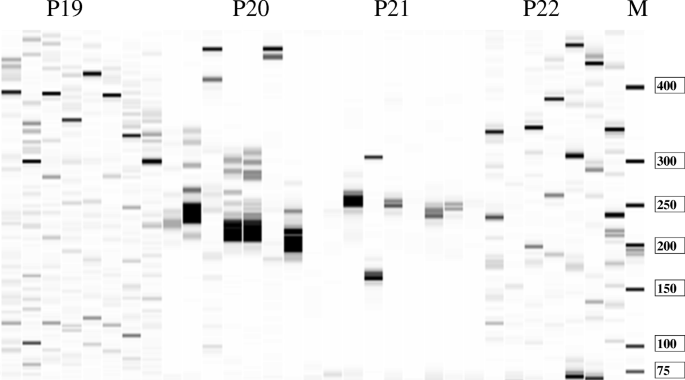
基于5042个SSR序列,采用Primer Premier 5.0 (Premier Biosoft International, Palo Alto CA)进行严格筛选,成功设计出3202对高质量SSR引物。为进一步评价SSR标记的多态性,随机合成了68个SSR标记。经过3即trifida共扩增出61对引物(89.71%)。在不同的退火温度下,仍有7条(10.29%)SSR引物不能产生PCR产物。这61对工作引物中,除11对SSR引物扩增出的条带大于预期外,大部分引物扩增出的条带几乎达到预期大小。
对61对SSR引物进行了多态性分析即trifida(无花果。5).结果表明,所有引物均扩增出2个以上的多态性条带。PCR产物大小在100 ~ 480 bp之间。优化引物的多态位点数量在2 ~ 11个之间,平均为6个。
序列的SNPs注释
利用Blast2GO软件对38,605条多态性SNP位点序列进行blast分析,其中有11,519条(29.84%)符合显著命中(E = 10)−5)。共11541个(29.90%)与UnProtKB数据库中的序列对齐,其中2516个reads仅带blast hit, 2551个hits未加GO Annotation映射。只有6474个(16.77%)命中被成功映射和注释。27064(70.10%)单身者没有显著的命中。与转录研究相比,本研究中具有显著命中的序列频率较低。这可能是因为大多数没有命中的解读来自基因组的非编码区域。
48种植物中有50余种被杀,大部分为一年生或多年生植物。1726个读数被输入番薯零.866号和722号被击中烟草的抗旱性和烟草tomentosiformis,分别。有趣的是,只有200个点击来自番薯甘薯,有127个点击率来自番薯trifida.此外,只有少数点击率来自其他番薯物种,包括番薯紫竹,番薯pes-caprae等(附加文件1:图S2)。
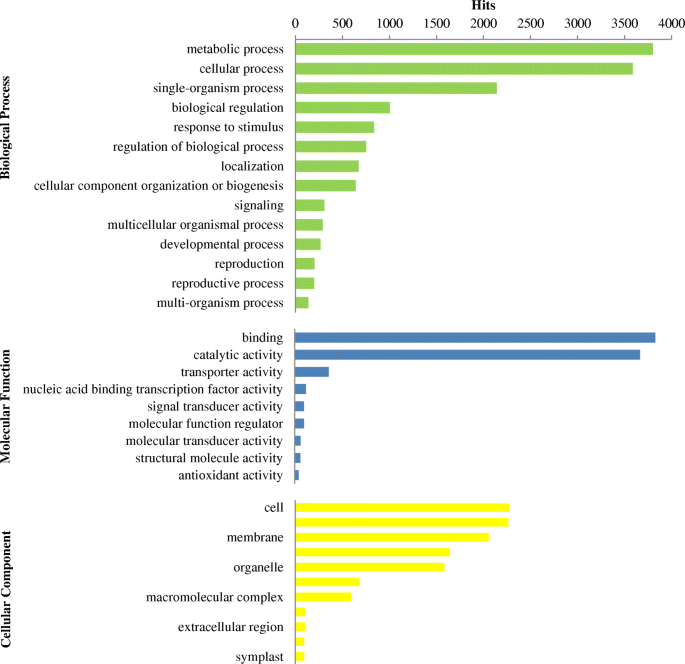
在38,605个blastx注释的序列中,共有6474个(16.77%)与GO项相关。共注释35384个ungenes。通过注释分析,将blast hits基因分为2级分子功能(MF)、细胞成分(CC)和生物过程(BP)三类。15163个(42.85%)与生物过程相关的注释,8328个(23.54%)与分子功能相关的注释,11,893个(33.61%)与细胞成分相关的注释。在生物学过程中,以代谢过程(3800)、细胞过程(3588)和单生物过程(2142)最为突出。在分子功能类别中,“结合”(3803)、“催化活性”(3641)和“转运体活性”(358)是最具代表性的类别。在细胞组分类别中,基因的比例最大的是“细胞”(2334)、“细胞部分”(2324)和“膜”(2110)(图2)。6).此外,只有7个序列被分配到“营养库活动”项。
通过对GO证据码的分析,共检测到电子注释(Electronic Annotation, EA)中推测的基因8836个(72.47%)。这些注释没有被手动检查。1749个推测基因来源于祖先生物学方面(BA),占14.34%。其余1148个(9.42%)推定基因来自表达模式(EP)、直接分析(DA)、突变表型(MP)、序列模型(SM)和序列或结构相似性(SS)(附加文件)1:图S3)。
同时,使用Blast2GO将总共8587个rad标签与OG数据库对齐。有趣的是,2025例(23.58%)患者的功能未知。这可能与本研究的reads来自基因组,具有非编码区有关,也可能是因为目前对甘薯的研究较少即trifida.6562个序列OG类别被注释。‘Signal transduction mechanisms’ and ‘Posttranslational modification, protein turnover, chaperones’ had the largest amount sequences of 992 (11.55%) and 703 (8.19%), respectively, followed by ‘Energy production and conversion’, ‘Replication, recombination and repair’ and ‘Carbohydrate transport and metabolism’, with 671 (7.81%), 558 (6.50%) and 495 (5.76) sequence, respectively (Additional file1:图S4)。其中有164个(1.91%)序列被注释为“防御机制”,这在甘薯和油菜的抗病基因研究中具有特殊的潜力即trifida.
当e值<=1e时,将推定蛋白的基因映射到KEGG数据库中−5和等级< = 5。在KEGG数据库中分类为“代谢”的通路中,共有1727个unigenes被映射到349个代谢通路上(附加文件)1:图S5)。这些途径包括“淀粉和蔗糖代谢”、“类胡萝卜素生物合成”、“油菜素内酯生物合成”、“光合作用-天线蛋白”、“吲哚生物碱生物合成”、“光合作用”、“玉米素生物合成”、“氨基酸生物合成”、“植物激素信号转导”、这将为未来的研究提供有价值的信息。
讨论
RAD-seq在甘薯和即trifida
下一代(NGS)测序可以用来推断物种之间的进化关系,以及识别大量分散在基因组中的snp。在本研究中,我们对甘薯和不同的多倍体类型进行了测序即trifida共发现832204个SNP位点和5042个SSRs位点。这些标记可广泛应用于甘薯和油菜的农艺基因定位和遗传图谱的构建即trifida.
没有参考基因组的物种的遗传复杂性仍然难以利用NGS的优势进行遗传研究,这种情况在近期内难以改变。一些研究人员试图首先组装初步参考基因组,然后重新测序以确定一组SNPs。然而,这种方法成本太高,无法在大多数物种中实施。在降低SNP质量和降低成本的问题上,尝试了多种方法。最后,建立了简化表示库排序(RRLS),该方法大大简化了库的生成过程,更适合用于大量的个体/行[18].目前,已经发展了几种RRLS方法,包括限制性位点相关DNA (RAD)测序[16,19]和基因分型测序(GBS) [20.,21),等。利用RRLS方法在无参考的情况下研究复杂物种的遗传多样性,效率高,成本低。
大量报道表明,RAD-seq数据可以在没有参考基因组的情况下准确确定SNP基因型[22,23].但是,准确的结果是建立在相对提升测序深度。然而,较高的测序深度意味着更高的成本。更重要的是,提高测序覆盖并不总是有效的,必要的。先锋的研究已经表明,测序深度至少有四个读取都必须从纯合和杂位点的基因型调用,以确保95%的置信度,并被推荐的6-89深度,以平衡成本和数据的质量。在这点上,平均测序深度为12.15在本研究中,这远高于下限。因此,用于分析的SNP基因型可以提供最保守的结果。
到目前为止,SNP基因分型仍然难以完成。虽然毛细管电泳、高分辨熔解等方法可以用于SNP等位基因的鉴别,但它们需要专门的仪器和检测试剂盒。相比之下,降低基因组复杂性和RAD-seq是SNP基因分型的最佳选择[24].然而,现在它仍然很贵。在水稻、小麦和玉米上的应用证明,DNA芯片是SNP基因分型的最佳选择,具有高通量、高精度、快速和廉价的特点。在本研究中,我们利用RAD-seq获得了38,605个rad标签,共83,204个多态性SNP,这为今后开发SNP芯片提供了有用的信息。
甘薯及其野生种的起源中心即trifida
目前关于甘薯起源的假说主要有两种。一种设想是,根据遗传多样性分析,甘薯的驯化可能起源于中美洲[25,26].另一种观点认为,两个驯化事件发生在中美洲和南美洲西北部,这是由南北基因库的明显差异支持的[6,26,27].相反,起源即trifida很清楚,但它的分类一直存在争议。Tamari和Kobayashi在墨西哥的阿卡普尔科发现了这种野生二倍体物种,这被认为是一个典型的物种即trifida现在(28,29].Shiotani等人在墨西哥阿卡普尔科采集了三倍体的Ipomoes种子,在采集二倍体的同一地区[30.].类似甘薯的四倍体番薯,也在墨西哥、危地马拉、哥伦比亚和厄瓜多尔采集[31,32].从安第斯山脉到墨西哥观察到的四倍体被认为是来自二倍体的同源多倍体即trifida[29].1955年,西山在墨西哥的Fortin采集到一株野生六倍体(6X, 2n = 90),并将其命名为即trifida(H.B.K.)g .(不33,可以与甘薯杂交。除此之外,还有几个社区即trifida包括二倍体、四倍体和六倍体即trifida它们在圣玛尔塔、马拉凯和哥伦比亚海岸被观察到。这些植物广泛分布在海拔5至20米的地方,也可在海拔1000米左右的地区发现[29].
所有这些报告可以简单总结一下即trifida和它的亲戚主要位于从太平洋到墨西哥湾的带,大约17o-20oN纬度(34,而这片广阔的地理区域最有可能是甘薯的驯养地之一。此外,根据形态学、细胞学和遗传学的证据,这是合乎逻辑的假设即trifida是甘薯的直接祖先之一[33,35,36,37].近年来,众多的遗传,进化和细胞遗传学研究已进一步证实之间的密切关系一、甘薯和即trifida[3.,7,38].然而,还需要进一步的研究来精确地确定甘薯驯化发生的时间、地点和方式。
即trifida6X曾与甘薯的关系更密切
关于起源的两个主要假设番薯甘薯提出了。一个提议一、甘薯同源多倍化或异源多倍化来自哪里即trifida.另一个人认为即trifida和即triloba是野生动物的祖先吗一、甘薯.Austin(1988)指出即trifida和即triloba最有可能是栽培甘薯的祖先[10].Roullier等人对此并不支持一、甘薯是源于即triloba[6].他们认为即trifida和一、甘薯是密切相关的。大多数研究人员接受了这一点即trifida是谁的祖先之一一、甘薯.然而,关于该组织的成立还没有达成一致一、甘薯多倍体基因组是否同源多倍体起源即trifida或异源多倍体从即trifida和远亲物种。Kobayashi等人指出甘薯的同源多倍体来自于其祖先。它和即trifida[29].
四种不同的多倍体类型即trifida被发现,包括2 x (2 n = 2 x = 30), 3 x (2 n = 3 x = 45), 4 x (2 n = 4 x = 60)和6 x (2 n = 6 x = 90)。揭示甘薯与不同多倍体类型的关系具有重要意义即trifida.利用SNP基因型对甘薯的全基因组进行了分布分析即trifida,应比传统的分子标记更可靠和准确。三种不同的多倍体类型即trifida和培育一、甘薯形成了分离良好的集群。在群集内即trifida,3种不同类型的多倍体被分为三个不同的谱系。这两个嵌套Beinong5521和即trifida6X种质介于栽培甘薯和栽培甘薯之间即trifida2 x集群。在多倍体即trifida在我们的进化树中,虽然它们具有不同的多倍体和不同的区域,但它们的遗传关系密切。它可以提出至少一个多倍体或杂交事件即trifida2 x生成即trifida4 x。在此基础上,又产生两个或两个以上的多倍体化或杂交事件即trifida6 x。
有趣的是,地点即trifida6X远非即trifida2 x,和之间即trifida4X和甘薯在进化树上。它与甘薯的遗传关系比与甘薯更密切即trifida4 x和即trifida2 x。这一结果支持了西山的假设[34],而且应该是有价值的,以进一步澄清甘薯的起源。Kobayashi等。报道称,野生多倍体一、甘薯从墨西哥到秘鲁北部采集[29].此外,我们进行了3年多的杂交研究,表明甘薯与即trifida6X比with更容易成功即trifida2 x或即trifida4X,支持甘薯与白薯有更密切的遗传关系即trifida是二倍体的6倍即trifida.然而,需要使用全基因组测序和其他先进的生物技术来揭示甘薯的起源进一步研究。
序列的snp功能注释
共有11519个带有snp位点的rad -tag与公共数据库中的唯一基因相匹配,仅占总数的29.90%。其中大部分被划分为不同功能类型的基因本体分类和COG分类,包括“信号转导机制”、“翻译后修饰、蛋白质转换、伴侣”和“防御机制”等。从KEGG数据库的爆破中,共命中1727个相关基因,这些基因连接到349个表征良好的途径,如“淀粉和蔗糖代谢”、“类胡萝卜素生物合成”、“油菜素内酯生物合成”和“植物激素信号转导”等。因此,这些结果初步表明,这些rad -tag与不同的功能基因相关,在功能基因研究中有一定的应用价值。与转录组测序相比,RAD-seq可以产生更多的SNP基因型,获得更丰富的基因组信息。此外,高度相似的物种一、甘薯,即零和即trifida在NCBI数据集中发现,表明甘薯基因组中存在同源基因。
结论
综上所述,本文的研究结果表明,RAD-seq在甘薯的进化和遗传研究中应该比传统的分子方法更有效和可靠即trifida.从RAD-seq中检测到数千个SNP,并使用公共数据集进行注释。基于SNP基因型,研究甘薯与不同多倍性野生种的进化关系即trifida研究首次揭示了栽培甘薯与甘薯的亲缘关系最为密切即trifida6X,紧随其后的是即trifida4 x。相比之下,即trifida2X有进一步的遗传关系。这一结果为研究人员提供了有价值的线索即trifida6X作为甘薯研究的模式植物,应该比使用更实用即trifida未来是2X。同时,SSR引物,由即trifida6X时,应有助于解决SSR标记缺乏的问题即trifida研究。
方法
植物材料
13份甘薯种质,包括4个外来品种、4个中国品种和5个中国地方品种,12份不同的多倍体即trifida本研究使用了来自原生质体融合的包含二倍体型、四倍体型和六倍体型的材料(见表)4).
DNA提取
采用CTAB(十六烷基三甲基溴化铵)法从所有样品中分离基因组DNA [39].所有DNA样品的浓度采用NonoDrop ND-2000 (Thermo Scientific, Wilmington, DE, USA)定量,并通过1%凝胶电泳确认。最后用1 × TE缓冲液(10 mM Tris-HCl和1 mM EDTA, pH 8.0)溶解DNA,用ddH稀释至50 ng/ul2O用于快速应用系统分析。
RAD库的构建和测序
按照Baird等人描述的方法共制备了27个RAD库[19].首先,酶EcoRI和NlaIII通过比较不同酶重复标签的数量和酶标签的分布来选择。其次,利用预先选择的酶构建RAD库。然后,将含有Illumina序列引物、PCR正向引物和条形码的P1接头连接的所有样品的酶解基因组DNA汇集在一起。利用琼脂糖凝胶电泳分离得到300 ~ 400 bp的片段并进行纯化。将获得的DNA片段与另一个适配器(P2)连接,该适配器包括PCR反引物和分叉端。最后,通过PCR反应选择性扩增带有两个适配器(P1和P2)的DNA片段。PCR产物经纯化和聚合,然后在2%琼脂糖凝胶上分离。全长375 ~ 400 bp(带索引和适配器)的片段分离纯化。使用Illumina HiSeq4000平台(http://www.illumina.com/)。利用Stacks v1.40进行质量过滤和位点组装[40].每个样本的序列都根据条形码进行排序。
To ensure the nucleotides quality value above Q30 (< 0.1% sequencing error) and more than 99% above Q20 (< 1% sequencing error), raw reads with ≥10% unidentified nucleotides (N), > 50% bases having phred quality < 5, with > 10 nt aligned to the adapter, containing enzyme sequence, were discarded. Raw sequence reads with RAD-tag were divided following the order of sequence depth. Heterozygous loci were tested through comparison in each sample. The comparison among RAD-tag of discrete samples produced SNPs. Finally, combine the frequency data and comparison results, low confidence SNPs from repeat regions were filtered.
SNP检测与注释
单核苷酸多态性(SNPs)用最大似然模型进行标记,在Stacks v1.40中实现[40].对于SNPs调用,使用Stacks v1.40基于序列相似性将所有修剪过的读压缩到聚类中[40在默认参数下。Vcftools v 0.1.1.12b [41]用于质量控制。最后,以SNP位点检测到22个以上的个体,等位基因频率大于0.1的个体进行进一步分析。
为了避免错误,排除了深度超过500的rad标签。将所有样品的rad标记序列进行Blast处理。在比对结果中发现了snp,当每个等位基因至少观察4次时,视为真正的多态性。所有样品的基因型均由包含单核苷酸多态性位点的序列序列决定。
为了评估SNP位点的潜在功能,使用本地基因发现软件Blast2GO v2.4.2进行相似性搜索[42, E值截止值为10−5对非冗余(NR)数据库,基因本体论(GO)数据库,蛋白质的直系同源群的集群(COG)(http://www.ncb我。nlm.nih.gov /齿轮)数据库、京都基因与基因组百科全书(KEGG)数据库(http://www.genome.jp/kegg)和unprott知识库(UnProtKB)蛋白数据库。将包含SNP位点的RAD序列与这5个数据库进行比对,预测和分类可能的功能。
SSR标记的发展
使用MIcroSAtellite (MISA, http:// pgrc.ipk-gatersleben.de/ MISA /)脚本检测带有默认参数的rad标签中的微卫星。保留重复单元为2-6个核苷酸,每个重复单元至少5个重复的SSR位点,用于引物设计。然后,使用primer premier 5.0 (premier Biosoft International, Palo Alto, CA)对包含微卫星的标签进行引物设计,采用默认设置,不包括三个标准[43,44],(1)引物长度在18 ~ 25个碱基之间,(2)引物GC含量在40 ~ 60%之间,退火温度在50℃~ 60℃之间,(3)设计引物PCR产物大小为100 bp ~ 400 bp。
为了确定这些引物是否有效,我们随机合成了68条引物,并在8条引物中进行扩增即trifida登记入册。PCR扩增和电泳条件如Ban等[45].使用Fragment Analyzer INFINITY™(Advanced Analytical Technologies (AATI), Ames, Iowa, USA)电泳系统和DNF-900 dsDNA试剂试剂盒分离PCR产物,结果由PROSize 2.0软件版本1.3 (AATI)进行分析。
甘薯和白薯的进化分析即trifida
使用Phylip (v 3.695)进行Neighbor-joining系统发育树[46基于SNP数据集进行质量控制。用APE和iTOL v3的R-package (http://itol.embl.de) [47,48].在子群数K值为0 ~ 10的前提下,使用fastSTRUCTURE (v1.2)和默认参数[49].从每个K分析中选取边际最大似然的K作为最优子群数。使用PLINK (v1.90)计算每个样本之间的遗传相似度矩阵[50,51基于]上过滤数据集。然后,算术平均值(UPGMA)聚类的不加权配对组方法的树用PHYLIP(V 3.695)[计算46],用APE的r -包提取[48]及iTOL v3 (http://itol.embl.de) [47].
缩写
- 芭:
-
祖先的生物学方面
- 英国石油公司:
-
生物过程
- 答:
-
蜂窝组件
- 齿轮:
-
蛋白质的同源组的簇
- CTAB:
-
Cetyltrimethyl溴化铵
- 大卫·爱登堡:
-
直接法
- 背景:
-
脱氧核糖核酸
- EA:
-
电子注释
- EP:
-
表达式模式
- GBS:
-
Genotyping-by-sequencing
- 走:
-
基因本体论
- KEGG:
-
京都基因和基因组途径百科全书
- MF:
-
分子功能
- 对剧中:
-
微卫星
- MP:
-
突变表型
- NCBI:
-
国家生物技术信息中心
- 门店:
-
新一代测序
- Nr:
-
冗余
- 聚合酶链反应:
-
聚合酶链反应
- 体育:
-
Pair-end
- RAD-seq:
-
限制性位点相关DNA测序
- RRLS:
-
Reduced-representation库测序
- SM:
-
序列模式
- SNP:
-
单核苷酸多态性
- SS:
-
序列或结构相似性
- 苏维埃社会主义共和国:
-
简单序列重复
- UnProtKB:
-
UniProt知识库
- UPGMA:
-
算术均值聚类的非加权对组方法
参考文献
- 1.
花蜜、花蜜生产动态及化学成分分析番薯物种(旋花科植物)与传粉者的关系。安机器人。2004;94:269 - 80。
- 2.
Woolfe农协。红薯:一种未开发的食物资源。纽约:剑桥大学出版社;1992.
- 3.
黄娟,孙敏。甘薯及其野生近缘种的遗传多样性及其亲缘关系番薯系列甘薯(旋花科植物叶绿体DNA的ISSR和限制性内切分析结果表明。2000; 100:1050-60。
- 4.
Hirakawa H, Okada Y, Tabuchi H, Shirasawa K, Watanabe A, Tsuruoka H, Minami C, Nakayama S, Sasamoto S, Kohara M.野生甘薯基因组序列调查,番薯trifida(HBK) g .堂。DNA研究》2015;22(2):171 - 9。
- 5.
甘薯的同源倍性(番薯甘薯(林)及其前身。Euphytica。1975;24:197 - 208。
- 6.
Roullier C, Duputié A, Wennekes P, Benoit L, Bringas VMF, Rossel G, Tay D, McKey D, Lebot V.解析栽培甘薯的起源(番薯甘薯(l) lam)。《公共科学图书馆•综合》。2013;8:e62707。
- 7.
甘薯的起源和进化(番薯甘薯和野生近缘种的细胞遗传学方法。植物科学。2006;171:424-33。
- 8。
胡建军,中谷俊,刘志强,等。基于ISSRs的甘薯与野生近缘种的遗传分析。育种科学。2003;53:297 - 304。
- 9.
O ' brien PJ。红薯:它的起源和传播。Anthropol。1972;74(3):342 - 65。
- 10.
奥斯汀DF。红薯及其近缘野生种的分类、进化及遗传多样性。甘薯遗传资源开发、维护和利用规划会议,秘鲁利马,1987年2月23-27日。CIP 1988:27-60。
- 11.
Pruitt KD, Tatusova T, Maglott DR. NCBI reference sequences (RefSeq):一个非冗余序列数据库的基因组,转录本和蛋白质。核酸Res. 2007; 35:61-5。
- 12.
Altshuler D, Pollara VJ, Cowles CR, Van Etten WJ, Baldwin J, Linton L, Lander ES。由简化代表霰弹枪测序生成的人类基因组SNP图。大自然。2000;407:513-6。
- 13.
Young AL, Abaan HO, Zerbino D, Mullikin JC, Birney E, Margulies EH。一种利用短序列读取和简化的表达库进行基因组组装的新策略。基因组研究》2010;20:249-56。
- 14.
Deschamps S, la Rota M, Ratashak JP, Biddle P, Thureen D, Farmer A, Luck S, Beatty M, Nagasawa N, Michael L. Rapid全基因组单核苷酸多态性发现,通过Illumina基因组分析仪的简化代表库的深度重测序。植物基因组。2010;3 - 68。
- 15.
Varshney RK, Song C, Saxena RK, Azam S, Yu S, Sharpe AG, Cannon S, Baek J, Rosen BD, Tar an B.鹰嘴豆基因组测序(中投arietinum)为性状改良提供了资源。生物科技Nat》。2013;31:240-6。
- 16.
Baxter SW, Davey JW, Johnston JS, Shelton AM, Heckel DG, Jiggins CD, Blaxter ML。《公共科学图书馆•综合》。2011;6:e19315。
- 17.
Cromie GA,Hyma KE,勒德洛CL,迪亚托雷斯C,吉尔伯特TL,五月P,黄AA,达德利上午,费伊JC。基因组序列多样性和酿酒酵母的群体结构评估通过RAD-起。G3:基因|基因组|。遗传学。2013; 3:2163-71。
- 18。
Davey JW, Hohenlohe PA, Etter PD, Boone JQ, Catchen JM, Blaxter ML.全基因组遗传标记的发现和下一代测序的基因分型。Nat Rev Genet, 2011; 12:499-510。
- 19。
Baird的NA,埃特尔PD,阿特伍德TS,Currey提供MC,寒战AL,刘易斯ZA,Selker EU,Cresko WA,约翰逊EA。快速SNP发现和遗传学图谱测序使用RAD标志。Plos一个。2008; 3:e3376。
- 20.
Elshire RJ, Glaubitz JC, Sun Q, Poland JA, Kawamoto K, Buckler ES, Mitchell SE。一个强大的,简单的基因分型测序(GBS)方法的高多样性物种。《公共科学图书馆•综合》。2011;6:e19379。
- 21.
Glaubitz JC, Casstevens TM, Lu F, Harriman J, Elshire RJ, Sun Q, Buckler ES。TASSEL-GBS:一个高容量的基因分型测序分析管道。《公共科学图书馆•综合》。2014;9:e90346。
- 22.
利用RAD(限制性位点相关DNA)标记快速定位黑麦草茎秆锈病抗性QTL。Theor app Genet. 2011; 122:1467-80。
- 23.
Lamour KH, Mudge J, Gobena D, Hurtado-Gonzales OP, Schmutz J, Kuo A, Miller NA, Rice BJ, Raffaele S, Cano LM。基因组测序和定位揭示了杂合度的损失是蔬菜病害辣椒疫霉快速适应的一种机制。acta botanica sinica(云南植物研究);2012;25:1350-60。
- 24.
张斌斌,薛德兴,王健,李玉玲,刘伯杰,刘建新。小黄鱼全基因组单核苷酸多态性资源的开发与初步评价(Larimichthys确定).Mol Ecol Resour. 2016; 16:55 - 68。
- 25.
张DP,塞万提斯Ĵ,华曼Z,凯里E,Ghislain的M.评估甘薯的遗传多样性(番薯甘薯利用AFLP从热带美洲的栽培品种。转基因资源作物Ev. 2000; 47:659-65。
- 26.
Gichuki ST, Berenyi M, Zhang DP, Hermann M, Schmidt J, Glössl J, Burg K.甘薯遗传多样性研究进展[J]番薯甘薯(l)林。与地理来源相关的RAPD标记。植物资源学报2003;50:429-37。
- 27.
利用叶绿体和核微卫星技术研究新大陆甘薯地方品种的起源和传播。摩尔生态。2011;20(19):3963 - 77。
- 28.
Teramura T, Shiotani, Kawase T.在墨西哥采集的番薯三倍体植物。日本J品种。1967;17:187-8。
- 29.
小林m .的番薯trifida与红薯关系密切。国际热带根作物学会第六次专题讨论会论文集/国际热带根作物学会于1983年2月21-26日在秘鲁利马主办。秘鲁利马:国际马铃薯中心(CIP), 1984;页:561 - 68。
- 30.
甘薯的起源与分化。基因组构成和驯化。日本J品种1980;30:306-9。
- 31.
Martin FW, Ruberté RM, Jones A. A wild番薯与甘薯密切相关的物种。经济学机器人。1974;28(3):287 - 92。
- 32.
村松男,盐谷一密切相关的红薯在墨西哥和危地马拉的野生种蕹菜。京都大学科学考察到拉丁美洲的初步报告。1974; 1:9-30。
- 33.
西山一。甘薯的起源。见:夏威夷第十太平洋科学大会大学,檀香山;1961.119 - 28页。
- 34.
西山。甘薯的进化与驯化。Shokubutsugaku Zasshi。1971;84(996):377 - 87。
- 35.
西山I, Fujise K,寺村T,宫崎T.甘薯及其近缘种的研究:I.甘薯染色体数目和主要植株性状的比较研究番薯speclesin部分薯。日本Ĵ品种。1961; 11(1):37-43。
- 36.
西山一,Teramura T.墨西哥野生甘薯。经济学机器人。1962;16(4):305 - 14所示。
- 37.
利用野生近缘种进行甘薯育种。在:Proc IV Symp。Int Soc太多。块根作物;1976.53-7页。
- 38.
jared RL, Austin DF。甘薯的遗传多样性和系统关系(番薯甘薯和近缘种的RAPD分析。植物遗传资源学报1994;41(3):165-73。
- 39.
穆雷,汤普森WF。快速分离高分子量植物DNA。核酸Res. 1980; 8:4321-6。
- 40.
Catchen J, Hohenlohe PA, Bassham S, Amores A, Cresko WA。人口基因组学的分析工具集。摩尔生态。2013;22:3124-40。
- 41.
Danecek P, Auton A, Abecasis G, Albers CA, Banks E, DePristo MA, Handsaker RE, Lunter G, Marth GT, Sherry ST.不同的呼叫格式和VCFtools。生物信息学。2011;27:2156-8。
- 42.
Blast2GO:功能基因组学研究中用于注释、可视化和分析的通用工具。生物信息学。2005;21:3674-6。
- 43.
Barchi L, Lanteri S, Portis E, Acquadro A, Valè G, Toppino L, Rotino GL.利用RAD标签测序技术鉴定茄子SNP和SSR标记。BMC基因组学。2011;12:304。
- 44.
杜塔S,Kumawat G,辛格BP,古普塔DK,辛格S,多格拉V,Gaikwad K,夏尔马TR,Raje RS,Bandhopadhya TK。在木豆的深转录组测序核不-SSR标记的开发[Cajanus毛竹(l)Millspaugh].BMC Plant Biol. 2011;11:17。
- 45.
潘树华,尹文华,金金华,权思,崔灿。基于简单序列重复标记的韩国苹果品种遗传鉴定。园艺。2014;55:531-9。
- 46.
Felsenstein J. PHYLIP(系统发生推理包)3.6版。由作者分发。西雅图:华盛顿副基因组科学大学;2005.
- 47.
交互式生命树(iTOL) v3:用于显示和注释系统发育和其他树的在线工具。核酸Res. 2016;44(W1): 242-5。
- 48.
猿(APE): R语言的系统发育和进化分析。生物信息学。2004;20:289 - 90。
- 49.
Raj A, Stephens M, Pritchard JK。fastSTRUCTURE:大SNP数据集中群体结构的变异推断。遗传学。2014;197(2):573 - 89。
- 50.
张CC,周CC,泰利尔LC,瓦提库提S,珀塞尔SM,李JJ。第二代PLINK:迎接更大、更丰富的数据集的挑战。Giga科学。2015;4。
- 51.
Purcell S, Neale B, Todd-Brown K, Thomas L, Ferreira MA, Bender D, Maller J, Sklar P, De Bakker PI, Daly MJ。PLINK:全基因组关联和基于群体的连锁分析的工具集。Am J Hum Genet. 2007; 81:559-75。
确认
本研究由四川省金融创新项目(No. 2015JSCX-003, No. 2016LWJJ-002, No. 2016ZYPZ-005, No. 2017QNJJ-001, No. 2017QNJJ-021),国家自然科学基金项目(No. 31101119),四川省育种合作项目(甘薯)资助。中国农业研究系统专项资金(CARS-10-B5)。我们非常感谢Majorbio生物制药技术有限公司(中国上海)对RAD-Seq技术的帮助。
资金
四川省金融创新项目(No. 2015JSCX-003, No. 2016LWJJ-002, No. 2016ZYPZ-005, No. 2017QNJJ-001, No. 2017QNJJ-021),国家自然科学基金项目(No. 31101119),四川省育种合作项目(甘薯),中国农业科研体系专项资金(CARS-10-B5)资助。
数据和材料的可用性
在当前研究期间生成和分析的数据集不公开,因为这些数据集将用于其他研究,但可从通信作者在合理要求下获得。
作者信息
从属关系
贡献
JYF对数据进行分析并起草文稿。ML选取甘薯品种并进行数据分析。SZ分析了数据并起草了手稿。CZ指导SSR和SNP标记的分析,并对手稿进行了修改。选用麦粒肿红薯品种。SQ分析了SNP数据并对手稿进行了修改。WFT和HJQ指导了甘薯多样性分析,并参与了手稿的起草。DYW指导数据分析,修改稿件。ZGP指导了整个研究,并对手稿进行了修改。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
冯建勇,李敏,赵胜。et al。甘薯及其相关不同多倍体野生种的进化及遗传多样性分析即trifida使用RAD-seq。BMC植物杂志18,181(2018)。https://doi.org/10.1186/s12870-018-1399-x
收到了:
接受:
发表:
关键字
- 地瓜
- RAD-SEQ
- 单核苷酸多态性
- 苏维埃社会主义共和国
- 进化分析