摘要
背景
基因在不同组织/细胞类型和发育阶段的全基因组表达谱是许多功能基因组研究的重要组成部分。通过rna测序(RNA-Seq)获得的转录组数据通常保存在公共数据库中,通过数据门户提供。数据可视化是评估和假设生成的第一步。然而,这些数据库通常不包括可视化工具,对于非计算专家的用户来说,建立一个可视化工具并非易事。这一点,以及通常存储数据的各种格式,使得数据访问、共享和使用的过程更加困难。我们的目标是提供一个简单和用户友好的存储库,以满足这些主要农业作物数据集的需求。
描述
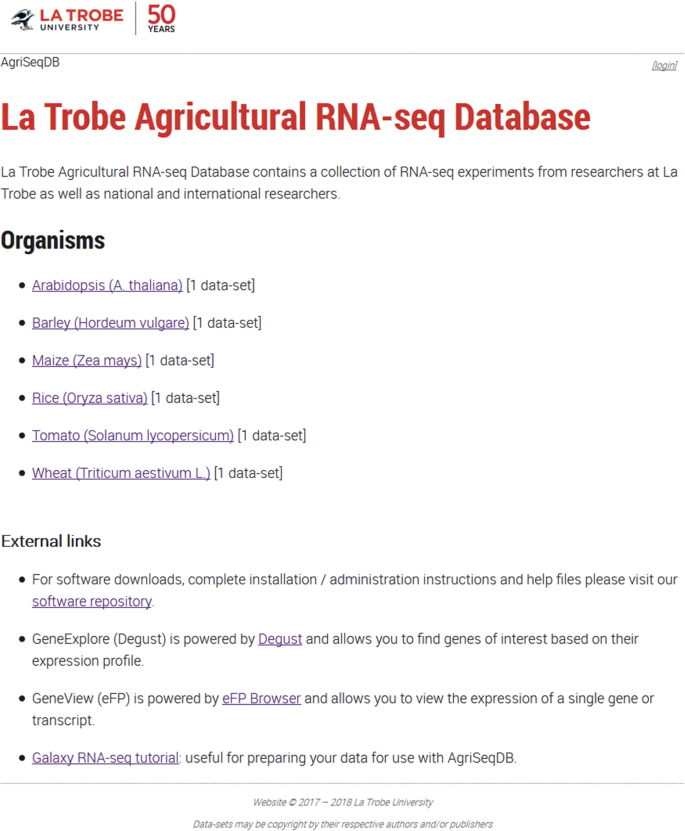
AgriSeqDB (https://expression.latrobe.edu.au/agriseqdb)是一个数据库,用于查看、分析和解释来自几个物种的发育和组织/细胞特异性转录组数据,包括主要农业作物,如小麦、水稻、玉米、大麦和番茄。公共转录组数据的不同存储方式和可视化原始数据的挑战都是数据重用的主要障碍。流行的eFP浏览器在以易于解释的视图呈现转录组数据方面做得很好,但以前的实现大多是基于具体情况的。在这里,我们提供了一个整合的可视化数据库,该数据库包含了六个物种的转录组数据集,这些物种以前没有面向公众的可视化。我们将eFP浏览器与Degust浏览器相结合,用于逐基因调查,它可以在多个样本中显示所有转录本。两个可视化界面从同一点启动,使用户可以轻松地在分析模式之间切换。这些工具允许用户,即使是那些没有生物信息学专业知识的用户,挖掘数据集,并了解不同样本和时间中感兴趣的转录本的行为。我们还加入了一个额外的图形下载选项,以简化合并到演示文稿或出版物。
结论
基于eFP和Degust浏览器,AgriSeqDB是一个快速易用的数据分析和可视化平台,在五种作物和拟南芥。此外,它提供了一个工具,使研究人员可以轻松地共享他们的数据集,促进研究协作和数据集重用。
背景
rna -测序(RNA-Seq)目前是全基因组转录分析的首选技术,因为它结合了易于使用、数据质量和适用于多种应用的适用性[1那2].下一代测序(NGS)技术的最新进展,加上测序成本的降低,已经从许多物种收集了大量的RNA-Seq数据[3.那4.].这些数据通常以文本和/或基于表格的格式存储在在线存储库中。对许多生物学家来说,数据可视化是转录组分析的关键早期步骤,它允许检查数据质量,以及快速询问线索和假设生成。许多希望调查公共转录组数据的研究人员不是计算专家,对他们来说,将数据从在线存储库格式转换到可视化工具是一个挑战。这对数据重用造成了障碍。eFP浏览器,这是第一次开发在拟南芥的硅基因表达分析,是一个优秀的软件,以可视化显示转录组数据[5.].在撰写本文时,已有20个植物转录组数据集在专用eFP浏览器中公开可用(http://bar.utoronto.ca, (5.那6.那7.那8.那9.那10.那11.那12.那13.那14.那15.那16.那17.])。Degust是一个基于web的数据可视化工具,提供不同于eFP功能(https://github.com/drpowell/degust).它允许用户查看实验中所有样本的所有转录本,检查样本之间的趋势,可视化质量控制指标,并深入到具有感兴趣的表达模式的转录本子集。这两个数据浏览器可以集成在一起,为用户提供一个易于使用的工具来访问和分析多个数据集,通过开发一些增强的功能,它们可以用于数据下载和为演示文稿或出版物生成高质量的图像。
RNA-Seq通常在整个植物或器官水平上进行,使用的样本由不同的组织和细胞类型组成。这种方法掩盖了有关转录本的细胞或组织特异性信息,这对理解基因的空间调节和功能非常重要[18.那19.].空间分辨率对于捕获在特定细胞类型中以极低水平表达的转录本也很重要,因此在组织的大量样本中低于检测限度[2].时间基因表达数据也是一个重要的工具,可以用来研究基因组调控机制,了解发育与基因功能之间的关系[20.].这些方法都旨在破译不同植物物种的监管和结构基因网络,包括林木和主要农作物,如小麦(功能研究中使用小麦)、水稻(栽培稻),玉米(Zea Mays.),大麦(大麦芽)和番茄(茄属植物lycopersicum)[21.那22.那23.那24.那25.那26.那27.].
这里我们展示了AgriSeqDB (https://expression.latrobe.edu.au/agriseqdb),这是一个基于网络的资源,可以作为一个公共门户,访问、分析和可视化来自多个物种的组织和细胞特异性转录组数据集。我们在这方面的重点主要是农业相关物种种子和果实发育期间的转录组数据集。该数据库集成了两个现有的开源浏览器,并增强了它们的功能。这个浏览器提供了通过样本和数据集获取全基因组表达的信息,帮助发现有助于作物改良的新基因。它还提供质量控制信息。eFP浏览器允许用户在不同的样本之间可视化由感兴趣的基因编码的个体转录本的丰度。
结构和内容
数据库/网站架构
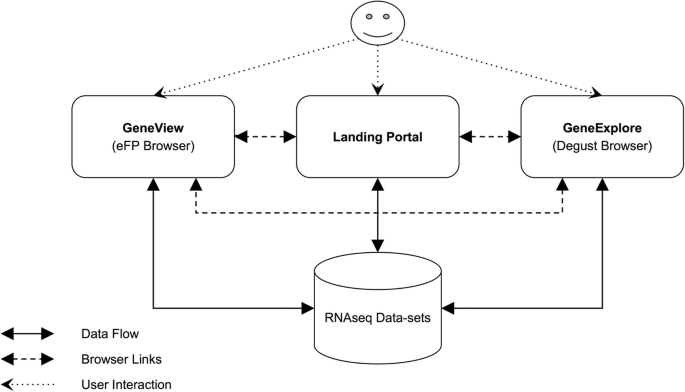
AgriSeqDB的主要结构如图所示。1.它包括被实现使用着陆门户的HTML前端和Python / Django的后端以呈现所有的数据集和相关联的元数据给用户。登陆门户网站,用户可以发现数据集,并导航到感兴趣的数据观众。现有的浏览器EFP,其具有HTML(前端)和Python(后端)工具,以允许用户查看表达数据在基因 - 基因基础选择了[5.].此外,包括现有的工具Degust,以允许查看所有(或一个子集的)基因的表达概况一次[28.].Degust使用HTML / JavaScript的前端和后端的Haskell。这两个工具都挂和着陆门户内包装,保证用户获得了一致的外观和使用门户和每个观众(图时感觉。2).对于着陆门户和整合与观众的源代码是可以重复使用(https://bitbucket.org/arobinson/agribiohvc).该仓库利用git的子模块的链接的EFP和Degust浏览器,其中的每一个从原始版本略作修改,以确保它们干净链接的源代码;的修改版本的源代码可在https://bitbucket.org/arobinson/efp和https://github.com/andrewjrobinson/degust,分别。Landing Portal和eFP浏览器使用MySQL数据库服务器来存储设置和数据/元数据,而Degust使用文件系统上的文件。增加了一个中央配置门户,以简化数据集到数据库和登陆门户文档的加载,允许组织注释上传、数据集上传、数据集配置(如私有/公开、提供外部链接和摘要等),并将数据集部署到我们所说的GeneView (eFP)或GeneExplore (Degust)。
数据源
目前在AgriSeqDB中显示的所有数据集都是最近发表并保存在公共数据库中的转录组(图)。2, 桌子1).AgriSeqDB的用户可以直接从数据库服务器查看数据,而不需要下载数据,然后安装/配置查看器来可视化数据。这些数据集是在对种子或水果的六项研究中生成的。第一个是我们对整个拟南芥种子在萌发过程中转录组变化的研究,该研究为该物种高质量的基因组序列和注释提供了有用的参考[20.].此外,我们还展示了来自主要农业作物的5个数据集。研究了大麦不同组织(糊粉、含淀粉胚乳、胚、盾片、果皮、种皮、壳和破碎细胞层)在不同萌发阶段的转录组变化[21.];玉米胚乳不同细胞类型授粉后转录组变化的研究[22.];水稻种子萌发和胚芽鞘生长相关转录组变化的研究[23.];番茄果实发育相关转录组变化的研究[26.];面包小麦籽粒/胚乳转录组的研究[24.].AgriSeqDB的GeneExplore (Degust)组件需要原始计数格式的RNA-seq数据(即每个基因或转录本的读取数,未归一化),用于后续分析。我们使用了原始作者在各自的GEO/SRA库中提供的拟南芥、小麦、水稻和大麦研究的原始计数数据(见表1)1).在这些情况下,映射和读计数被因而如先前所述[20.那21.那23.].在玉米和番茄数据集的情况下,原始计数数据无法从GEO/SRA存储库获得,但原始序列读取可以。为此,我们使用Kallisto将计数数据与参考番茄或玉米转录组进行了比对和量化(茄属植物lycopersicumSL2.50或Zea Mays.AGPv4),使用所得到的数据作为输入到AgriSeqDB [29.].
效用和讨论
我们的目标是开发一个公开可访问的转录组数据库,提供简单且易于提供的工具,以对单个靶基因或基因组进行功能分析。AGRISEQDB是一种高度交互式和多视图数据库,可用于各种目的,包括发现感兴趣基因。AgriSeqDB的用户可以直接从数据库服务器查看数据,而不需要下载数据,然后安装/配置查看器来可视化数据。但是,我们为高级用户提供了下载并安装自己的本地AgriseQDB的选项,以获取自定义数据集。
GeneView (eFP)
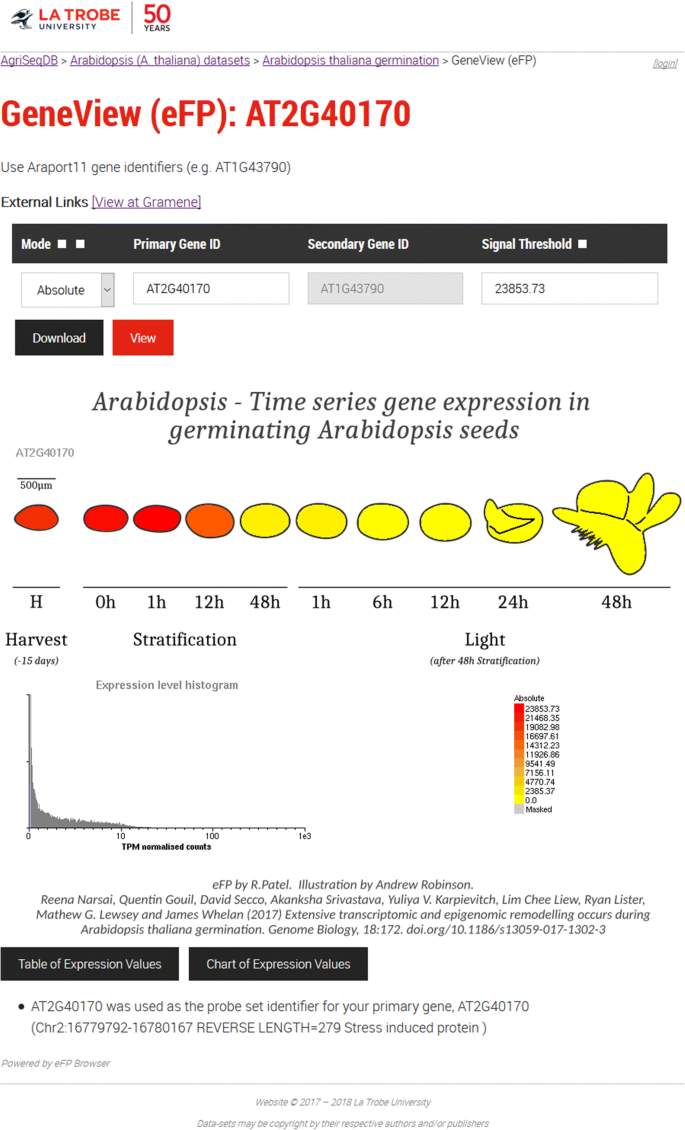
AgriSeqDB也可以让用户更好地了解个人感兴趣的基因,由内GeneView(EFP)(图检查它们。3.).这项计划包括EFP的全部现有功能[5.].用户可以在所有样品中可视化转录物的表达,以便他们可以考虑样品之间的关系(即生长阶段,组织类型,各种治疗)。此外,我们还包含一个额外的图像下载功能,以前没有可用。可以以高分辨率的.png格式下载图像以进行演示或出版物。这是通过单击下载按钮来完成的(图。3.).我们还可以直接从GeneView (eFP)记录进行跨物种比较。当用户在GeneView (eFP)中查看他们感兴趣的基因时,他们可以点击一个按钮,直接返回Gramene数据库的搜索结果(http://www.gramene.org).这返回了来自53种作物和模式植物的2076020个基因的同源、直系和副同源,以及一个比较系统发生树。
显示AT2G40170基因在GeneView (eFP)浏览器中的表达的完整截图。用户使用顶部的搜索表单选择感兴趣的基因并选择操作方式包括:(1)绝对的,显示的是存储在数据库中主要基因,(2)相对的,展示了主要的数量相对于控制基因,和(3)比较,数量之间的比率主要和次要的基因。单击视图按钮更新下图,通过用红-黄(表示绝对值)和红-灰-蓝(表示相对值和比较值)的比例对填充区域进行颜色编码,来显示每个示例的表达式级别。或者,用户可以单击下载按钮(由绿色箭头指示)以两倍于屏幕上显示的分辨率下载表达式图像(准备发布)。数据来自拟南芥种子萌发期间的转录组[20.]
GeneExplore(Degust)
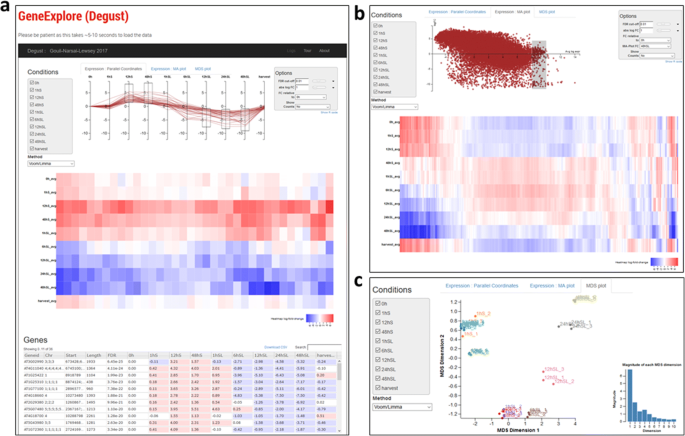
使用GeneExplore (Degust)为用户提供了一个简单的查询所有基因的界面(图。4A).丰富现有的功能是提供给用户内Degust [28.].可以根据个体样本中的表达水平、错误发现率(FDR)和Log2倍变化截止值在数据上创建过滤器。可以选择样本或转录本的子集进行分析,也可以选择用于参考折叠变化的样本。可以显示成对样品之间的MA比较图(图。4 b).数据质量度量可以通过检查每个样本组的重复是否在多维尺度(MDS)图中(图2)来评估。4摄氏度).数据表也可以在.csv格式下载选定转录物下游分析。
GeneExplore (Degust)浏览器和后续结果页面的屏幕截图。一种拟南芥时间序列数据集如此显示为示例,显示在S样品中上调的转录物,但在SL样品(顶部面板)中下调。用户可以选择要在屏幕左上左上角的复选框中选择哪些样本以及分析方法(变速/利马,编辑或变速器)。在右上方,用户可以控制使用“选项”对话框的渲染和阈值。匹配上述过滤器的所有基因在热图中示出,该热图簇具有相似水平的表达(中间板)。运行鼠标鼠标 - 每个基因在上面的图中突出显示它。表显示所有匹配基因的表格格式,表达水平为每个样本,虚假发现率和数据集中提供的任何额外注释列(下面的面板)。在顶部中心,用户可以通过使用3个交互式图中的1限制基因,并且并联坐标曲线允许用户通过它们的日志折叠基因表达(每个样品)来限制基因。B.MA图的例子。用户可以通过在MA图上的基因周围画一个框来限制基因;用于MA图的两个样本在选项对话框(右上角)中指定。C一个MDS情节表示各样品的每次重复的分组。数据是来自拟南芥的种子发芽转录期间[20.]
数据集管理(高级用例)
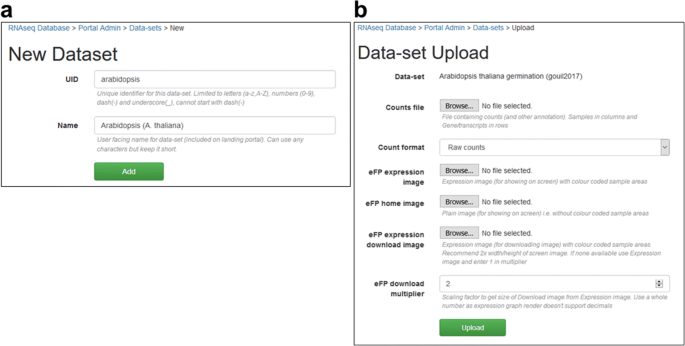
AgriSeqDB的一个关键功能是数据集管理工具。此工具仅在用户下载并安装他们自己的本地AgriSeqDB时可用,原因如下所述。eFP浏览器不包含上传数据的接口,配置时需要大量的手工交互。虽然Degust包含自己的管理工具,但它不够灵活,无法容纳eFP和登陆门户。因此,我们开发了一个新的数据上传接口,包括eFP和Degust。这允许用户(由用户名/密码保护)上传新的数据集,并将它们部署到每个查看者。该工具为用户提供了在主机服务器上执行自定义代码的能力,应该将访问权限限制在本地数据库管理员和受信任的用户。图中包含一个示例配置。5.和额外的文件1:图S1。提供一个链接在我们AgriSeqDB登陆门户网站,需要用户从所有AgriSeqDB代码可以下载的存储库,从那里安装/管理工作的指示和帮助文件可以看到(图2).代码和帮助存储库的直接地址是https://bitbucket.org/arobinson/agribiohvc.存储库包括来自其的用户可以访问的银河项目RNA-SEQ分析教程的链接(https://galaxyproject.org/tutorials/rb_rnaseq/),其用户可能会发现建立自己的本地AgriSeqDB时准备数据是有用的。
将数据集上传到本地安装AgriSeqDB的过程包括3个步骤。首先,用户选择数据集的唯一标识符和显示名称(一种).第二,用户从他们的PC中选择计数文件和用于显示表达式值的各种eFP图像来上传(B.).最后,用户可以改变许多设置,如图附加文件控制的数据集是如何显示在着陆门户和每个数据查看器1(见补充图1)大多数设置有合理的默认值,用户需要输入,节省时间的工具(如颜色选择器和点击图像的eFP设置)或电子表格导入/导出(示例设置)
结论
我们认为AGRISEQDB将成为植物生物学家的重要资源和数据重用工具,他们寻求更大了解生物过程中个体基因或基因组基因的作用,包括在重大农业重要性的作物种类中的比较研究。数据库将定期使用更多查看器和数据集更新,专注于从裁剪种类的附加组织和特定于特定于特定的数据集。该数据库目前包含来自不同组织和细胞类型的RNA-SEQ的结果,计划在未来添加来自单个细胞RNA-SEQ的转录组数据。从长远来看,预计用户将提供与Geo自动下载和视图的链接,以及允许至少临时上传数据集。所有源代码都可以通过高级用户自由使用可用于重用。
缩写
- EFP:
-
电子荧光象形文字
- 罗斯福:
-
错误发现率
- 地理:
-
基因表达综合
- HTML:
-
超文本标记语言
- MDS:
-
多维缩放
- NGS:
-
下一代测序
- RNA-Seq:
-
RNA序列
参考文献
- 1。
Wang Z,Gerstein M,Snyder M.RNA-SEQ:转录组织的革命性工具。自然rev genet。2009; 10:5-63。
- 2。
范志飞,乔凡诺尼JJ,罗丝JK。用RNA-Seq催化植物科学研究。植物科学学报2013;4:66。
- 3.
Petryszak R, Fonseca NA, Füllgrabe A, Huerta L, Keays M, Tang YA, Brazma A. RNASeq-er api -系统更新公共RNA-Seq数据分析的门户。生物信息学。2017;33:2218-20。
- 4.
基于云计算的基因组数据分析与协作。Nature Rev Genet. 2018; 19:208-19。
- 5。
冬天D,醋B,Nahal H,Ammar R,Wilson GV,荣誉NJ。一个“电子荧光象形文字”浏览器,用于探索和分析大规模生物数据集。Plos一个。2007; 2:E718。
- 6.
王志强,范建军,王志强,等。植物假说生成过程中数据的可视化和可视化研究。植物科学进展,2012,30(4):429 - 434。植物细胞。2017;doi.org/10.1105/tpc.17.00073.
- 7.
王志强,王志强,王志强,等。ePlant和3D数据显示技术:集成系统生物学在互联网上的应用。《公共科学图书馆•综合》。2011;6:e15237。
- 8.
曹亚平,王志强,王志强,等。拟南芥种皮发育过程中的基因表达模式分析。摩尔。2011;4:1074 - 91。
- 9.
Mustroph A, Zanetti ME, Jang CJ, Holtan HE, Repetti PP, Galbraith DW, et al.;在拟南芥中,离散细胞群体的图谱翻译组解决了缺氧时细胞优先级的改变。《美国国家科学院学报》上。2009; 106:18843-8。
- 10。
Wilkins O, Nahal H, fong J, Provart NJ, Campbell MM. Populus R2R3-MYB家族转录因子的扩展和多样化。植物杂志。2009;149:981 - 93。
- 11.
Tran F,Penniket C,Patel RV,Provart NJ,Laroche A,Rowland O,Robert LS。发育转录分析揭示了对麦芽果生殖发展的关键见解。工厂J. 2013; 74:971-88。
- 12.
陈志强,陈志强,陈志强,等。表达图谱和比较共表达网络分析揭示了短叶木化细胞壁形成的重要基因。新植醇。2017;215:1009-25。
- 13。
CHAMPIGNY MJ,宋WW,卡塔纳V,Salwan R,夏天PS,达力SA。,等人。在山嵛菜salsugineum植物在极端的自然栖息地生长和在受控生长柜条件RNA测序有效监视器基因表达。BMC基因组学。2013; 14:578。
- 14。
Kagale S, Nixon J, Khedikar Y, Pasha A, Provart NJ, Clarke WE,等。生物燃料作物亚麻荠的发育转录组图谱。植物j . 2016; 88:879 - 94。
- 15.
陈志强,王志强,王志强,等。花生异源四倍体的发育转录组图谱。植物学报2016;7:1446。
- 16。
Fasoli M, Dal Santo S, Zenoni S, Tornielli GB, Farina L, Zamboni A, et al.;葡萄藤表达图谱揭示了一个深层的转录组转变,推动整个植物进入成熟程序。植物细胞。2012;24:3489 - 505。
- 17.
李鹏,彭娜娜。玉米叶片转录组的发育动态。Nat麝猫。2010;42:1060。
- 18.
张志强,张志强,张志强,等。早期拟南芥胚细胞类型特异性转录组分析。发展。2014;41:4831-40。
- 19.
陈杰,曾B,张米,谢S,王G,Hauck A,Lai J.动态转录玉米胚胎和胚乳发育的景观。植物理性。2014; 166:252-64。
- 20.
王志强,王志强,王志强,等。广泛的转录组和表观基因组重塑发生在拟南芥萌发。基因组医学杂志。2017;18:172。
- 21。
Betts NS, Berkowitz O, Liu R, Collins HM, Skadhauge B, Dockter C, et al.;从完整的、发芽的大麦粒中分离组织和保存RNA。植物j . 2017; 91:754 - 65。
- 22。
Zhan J,Thakare D,Ma C,Lloyd A,Nixon Nm,Arakaki Am,等。激光捕获的MAIZE核的激光捕获微小隔室的RNA测序识别与胚乳细胞分化相关的调节模块。植物细胞。2015; 27:513-31。
- 23。
Narsai R, Secco D, Schultz MD, Ecker JR, Lister R, Whelan j栽培稻)胚芽鞘在缺氧和复氧条件下。植物j . 2017; 89:805-24。
- 24。
张志刚,张志刚,张志刚,等。六倍体面包小麦的谷物转录组中的基因组相互作用。科学。2014;345:1250091。
- 25。
白云杉细胞类型和组织特异性转录组的研究(Picea glauca.)树皮UNMASK精细尺度构和诱导针叶树防御的空间格局。植物杂志2017; 92:710-26。
- 26。
D’esposito D, Ferriello F, Dal Molin A, Diretto G, Sacco A, Minio A, et al.;揭示番茄果实转录组学、代谢组学和品质环境响应的复杂性。BMC Plant Biol. 2017;17:66。
- 27。
Shinozaki Y, Nicolas P, Fernandez-Pozo N, Ma Q, Evanich DJ, Shi Y, et al.;番茄果实发育和成熟的高分辨率时空转录组定位。Nat Commun。2018;9:364。
- 28。
鲍威尔博士。https://github.com/drpowell/degust, 2013年。
- 29.
Bray NL, Pimentel H, Melsted P, Pachter L.近最优概率RNA-seq定量。生物科技Nat》。2016;34:525。
致谢
我们非常感谢Nicholas Provart(U. Toronto)和EFP团队过去和现在的优秀工作,以及DaviD Powell(Monash University)的Devust Devust开发商,我们在这里使用了其软件。我们还要感谢他们在建立数据库时为他们的建议。我们感谢所有生成我们在AgriseQDB中显示的数据的团队的所有成员,他们在这里都有太多的清单。我们感谢毛山陈(La Trowe University)为他的帮助数据处理。
资金
这项工作以及由实物拉筹伯大学信息与通信技术和拉筹伯大学基因组学平台捐款澳大利亚国立数据服务(ANDS)补助金的资助。
数据和材料的可用性
该数据库可通过https://expression.latrobe.edu.au/agriseqdb.它与所有现代流行的Web浏览器通过平板电脑和移动/手机兼容使用,并可能。数据库的源代码是可以重复使用的https://bitbucket.org/arobinson/agribiohvc.在这个项目中使用的修改的Degust和eFP源代码可以在https://bitbucket.org/arobinson/efp和https://github.com/andrewjrobinson/degust.
作者信息
从属关系
贡献
AJR开发了数据门户,创建了插图(用于eFP)和上传数据。MT为项目开发做出了贡献。MGL和JW共同构思并提供科学指导。MGL进行了工具研究。CB注释元数据和RDA记录。RS、AW、SH、EF、KU参与项目管理。手稿由AJR, MT, MGL和JW撰写。所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
KU宣称她是资助者澳大利亚国家数据服务中心的雇员。两位作者宣称他们没有其他相互竞争的利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
额外的文件
附加文件1:
图S1许多设定的用户的可上传数据集到AgriSeqDB到控制该数据组是如何显示在着陆门户和每个数据查看器的过程中改变。(PNG 165 KB)
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Robinson, a.j., Tamiru, M, Salby, R。et al。AgriSeqDB:对于农业相关的植物物种的功能研究在线RNA-Seq的数据库。BMC植物生物学18,200(2018)。https://doi.org/10.1186/s12870-018-1406-2
收到了:
公认:
发表:
关键字
- RNA-Seq
- 转录组
- 基因表达
- 可视化
- 数据库
- 农业
- 大麦
- 玉米
- 大米
- 小麦
- 番茄