抽象的
背景
c型细胞质雄性不育(CMS- c)是玉米细胞质雄性不育(CMS)的三大类型之一。Rf4是CMS-C的显性恢复基因,并在杂交玉米育种中具有很大的价值,但是有关其功能机制的信息很少。
结果
揭示功能机制Rf4在此基础上,建立了一对玉米近等基因系(NILs)Rf4轨迹,包括尼尔币rf4男性无菌线和尼尔币Rf4男性fertility-restored线。遗传分析和分子标记检测表明NIL_Rf4被控制的Rf4.全基因组测序显示两个NIL之间的基因组差异聚集在Rf4映射区域。未映射的NIL读数进一步组装以发现Rf4候选基因。然后对NILs发育中的花药进行RNA Seq分析,以确定与育性恢复相关的关键基因和途径。共鉴定了7125个差异表达基因(DEG)。这些DEG在242个基因本体(GO)中显著富集类别,其中100个DEG参与花粉管发育、花粉管生长、花粉发育和配子体发育。同源性分析显示198个雄性育性相关DEG,途径富集分析显示58个DEG富集在参与糖酵解的细胞能量代谢过程中通过查询植物反应体途径数据库,我们发现14个DEG参与线粒体三羧酸(TCA)循环,其中大部分属于异柠檬酸脱氢酶(IDH)和氧谷氨酸脱氢酶(OGDH)转录组测序和实时定量PCR(qPCR)显示上述所有TCA循环相关基因在NIL中上调_Rf4.随后的酶联免疫吸附试验(ELISA)结果表明,无球茎小穗中IDH和OGDH酶的含量积累较多_RF4.比NIL_RF4。
结论
本研究为深入了解CMS-C雄性不育恢复的分子机制提供了宝贵的基因组资源。重要的是,我们的研究结果表明,能量代谢相关基因,特别是线粒体TCA周期相关基因与玉米CMS-C雄性生殖恢复有关。
背景
植物细胞质雄性不育是指功能性花粉粒缺乏,而雌性配子仍然正常。CMS是由自发线粒体基因组重排产生的嵌合开放阅读框(ORF)引起的[1.].一些被称为生育恢复剂的核基因(射频)基因可以在不同水平挽救CMS,如基因组、mRNA、蛋白质和代谢水平[2.,3.,4.].CMS/射频该系统不仅为研究细胞核与线粒体之间的通讯提供了理想的模型,而且是利用杂种制种优势的有价值的工具[2.,5.].目前,对植物CMS育性恢复的深入了解有助于其在杂交制种中的广泛应用[6.].
在玉米中,雄性无菌细胞质可根据F中显示的生育恢复模式分为三类1.混合动力车:德克萨斯州(T),USDA和Charrua(C)[7.].在这些类别的植物中,C型细胞质雄性不育(CMS-C)呈现稳定的雄性不育性,积极影响谷物产量,有助于其在玉米杂交种子生产中的应用价值[8.,9].然而,CMS-C的生育恢复机制似乎非常复杂。以前的研究表明,除了两个主导之外Rf4和Rf5数量性状位点(qtl)也参与玉米CMS-C育性恢复[10,11,12].这些QTL总是部分恢复CMS-C的男性生育能力,这使得它们难以实现。而且,射频-我在染色体7上可以抑制功能Rf5但不是Rf4[13].此外,不育系的遗传背景也可能影响其功能射频CMS-C中的基因,以及功能的数量射频在面对不同的雄性无菌线时,基因可能发散[14].综上所述,CMS-C复育机制复杂,给其在杂交种生产中的应用带来了严峻的挑战。Rf4能完全挽救大部分玉米CMS-C系,在玉米CMS-C杂交推广利用中具有很大的潜在价值。已经进行了许多工作Rf4,预先映射到染色体的短臂8 [15,16,17].Rf4进一步定位到包含12个候选基因的1.5 cM区间(520 kb) [18].Rf4最终缩小到8号染色体末端的12kb区域;该区域包含两个核靶向基因:GRMZM2G021276和GRMZM2G582028[19].然而,还需要更多的实验证据来证实Rf4候选基因并阐明其功能机制。
大多数恢复基因编码不同的五磷酸肽重复序列(pentatricoptide repeat, PPR)蛋白,使它们都能靶向CMS基因和产物所在的线粒体,并结合CMS转录本[20.,21].令人惊讶的是,在最终的图谱区域没有发现ppr编码的基因Rf4,这表明Rf4可能是一种新的恢复基因[18].阐明了Rf4这可能为植物CMS的育性恢复提供新的思路。然而,发生的概率Rf4不能排除编码PPR蛋白,因为B73的参考基因组仍然不完整,因为B73甚至可能缺乏Rf4.在本研究中,比较转录组测序Rf4近代源线(尼尔)揭示功能机制Rf4.
方法
植物材料
在这项研究中,在Rf4轨迹(NIL_Rf4)是为对比基因组和转录组分析而开发的。零_Rf4是从玉米近交捐赠捐助线A619开发的(Rf4Rf4)和CMS-C男性无菌受体线C698-3(rf4rf4)(零_rf4).将雄性可育植株> 5与保持系698-3 (rf4rf4)制作后交人群的后续生成。简而言之,来自BC的植物4.和公元前5.群体用于表型和基因型表征,来自BC的雄性肥沃的植物5.群体和品系C698-3进行比较基因组和转录组分析,以及来自BC的雄性可育植株6.人群用于测量异柠檬酸脱氢酶(IDH)和氧基氟化酸脱氢酶(OGDH)酶丰富。具体来说,从三个尼尔的新鲜叶子Rf4或NIL_rf4单独混合个体以进行基因组测序。此外,从三个单独的肥沃植物中收集了长度为0.7-0.8厘米的花粉成熟阶段的尖峰(NIL_Rf4_r1, NIL_Rf4_r2, NIL_Rf4_r3)和3个单独的不育植株(NIL_rf4_r1, NIL_rf4_r2, NIL_rf4_r3)用于转录组测序。样品直接在液氮中冷冻,然后保存在−70℃。
表型和基因型特征
描述NIL_Rf4,则为回交居群BC中每个个体的雄性生育能力4.和公元前5.被调查。对于雄性的育性,花药的发挥和花粉的育性的评价如我们前面所述[14].此外,在该报告中,我们制定了一个紧密联系的Rf4标记(5′-CGCACCTACCGTCC-3′,5′-GCGCAAGTACGCCGTAC-3′),用于确认BC中的所有植物基因型5.回交群体。用改良的十六烷基三甲基溴化铵(CTAB)法从每一株植物的新鲜叶子中提取基因组DNA [22].采用青岛PCR Master Mix (Tsingke, China)按其规范进行PCR扩增。
基因组序列定位,单核苷酸多态性(SNP)调用和注释
基因组DNA从NIL_的新鲜叶子中分离出来Rf4(公元前5.)和零rf4使用植物基因组DNA试剂盒(天根,中国)。使用1%琼脂糖凝胶、NanoDrop 2000分光光度计和量子位荧光计(Thermo Fisher Scientific, USA)检测DNA样品的浓度、纯度和完整性。随后,两种DNA复合物被送往中国北京诺金科技有限公司进行文库建设和测序。在Illumina HiSeq™2500分析仪(Illumina,美国)上准备了对端测序库,用于基因组测序。reads长度为150 bp,得到配对的末端。在映射前,我们去除了适配器片段、低质量序列(质量分数Q≤5的碱基≥50%)和杂质(N> 10%)。利用BWA 0.7.8(参数:mem -t 4 -k 32 -M -R)对B73参考基因组序列(ftp://ftp.ensemblgenomes.org/pub/release-31/plants/fasta/zea_mays/dna/).使用SAMTOOLS 0.1.19软件进行SNP呼叫[23].排除了碱质质量值<20或读取深度<4或> 1000的低质量SNP被排除在外。基于其位置,SNP被分类为外源,内肠,代际和拼接以及未转换区域(UTR)。此外,平均SNP指数[24,25使用滑动窗口分析来计算两个样品,使用100 kB窗口尺寸和10kb增量。
非内参基因的鉴定与鉴定
未映射的NIL读数用于通过SOAPDenovo与默认参数组装Contigs。使用遗传和奥古斯图来预测新的基因模型。然后,使用Acrigro注释每种新的基因推定功能(http://www.ebi.ac.uk/interpro/),KEGG(https://www.kegg.jp/), Swissprot (https://www.uniprot.org./),TrEMBL(https://www.uniprot.org./)和GO (http://www.geneontology.org/)数据库。采用HMMER 3.0软件,利用PPR结构域隐马尔科夫(HMM)谱(PF01535)对新基因中的PPR基因进行鉴定。TargetP (http://www.cbs.dtu.dk/services/TargetP/)来预测每个新基因的亚细胞位置。
RNA-SEQ映射
6个样本的总RNA,包括3个NIL_rf4样本(无)_rf4_r1, NIL_rf4_r2, NIL_rf4_r3)和三个nil_Rf4(公元前5.)样品(尼尔_Rf4_r1, NIL_Rf4_r2, NIL_Rf4_R3)分别使用QIAGENRNEASY套件(USA)提取。用1%琼脂糖凝胶进行研究浓度,纯度和RNA样品的完整性,使用NanoDrop 2000分光光度计(Thermo Fisher Scientific公司,USA),和安捷伦生物2100系统(安捷伦科技,USA)。DNase I用于消化残留的基因组DNA。随后将样品送到成都BaseBio生物技术公司(中国),用于Illumina Hiseq™2500平台(Illumina,USA)的图书馆建设和转录组测序。根据用于基因组-SEQ的相同方法过滤原始读取(150bp成对端)。从RNA-SEQ的清洁读数通过TOPHAT V2.0.9映射到与基因组-SEQ的B73参考基因组的相同版本。仅读取唯一映射到参考基因组的进一步分析。
差异表达基因的鉴定与功能分析
使用每百万转录本(TPM)计算每个转录本的表达[26].基于标准化的转录表达式,P_根据Benjamini-Hochberg方法对数值进行校正,以及Q_Value(纠正P_Value)<0.01用作筛选DEG的阈值。然后将所有的DEG进行基因本体(GO)分析(http://bioinfo.cau.edu.cn/agrigo/analysis.php.“基因和基因组(Kegg)途径分析(Kyoto)京都百科全书(http://www.genome.jp/kegg/)通过超几何测试。在上述富集分析中P_values用网站上的默认方法进行了修正,并且Q以_value≤0.05作为选择显著富集的GO项和KEGG通路的阈值。
为了鉴定涉及改变男性生育能力的潜在基因,所有的e_value <1e的eg- 30在植物雄性生殖数据库(PMRD)中查询(http://202.120.45.92/ADDB/),其中含有548个雄性生育能力相关基因拟南芥.玉米基因序列从MaizeGDB (ftp://ftp.ensemblgenomes.org/pub/plants/release-31/fasta/zea_mays).之后,从Qteller获得各种组织中的所有靶基因表达数据(每百万片映射的每百万片段(FPKM))的碎片(www.qteller.com).日志中记录FPKM值2.标准化(FPKM + 1),之后计算每个基因的Z分数以评估其组织表达特异性。此外,从最近的报告中获得了玉米中PPR基因的相应信息[27].
酶联免疫吸附试验(ELISA)检测小穗IDH和OGDH丰度
共有100毫克的尖峰,其长度在花粉成熟阶段(与转录组测序的相同阶段)的长度范围为0.7-0.8厘米的三个单独的无菌液滴rf4植物和三个个人肥沃尼尔_Rf4植物(BC.6.收集)被收集为IDH和OGDH内容测量。将每个样品在900μLPBS(0.01mol / L,pH = 7.2-7.4)中完全造粒,在冰上冷却并用300W的功率超声处理30分钟,以获得线粒体IDH和OGDH。然后离心匀浆(5000g,10min,4℃),之后,上清液最终通过ELISA试剂盒(南京尖峰生物技术有限公司)的IDH和OGDH含量测量,如此manufacturer’s protocol. Each sample was tested three times, and statistical differences were tested by independent Student’sT测试。
实时定量PCR(QPCR)分析
为了测量三羧酸(TCA)循环相关基因的表达水平并验证转录组测序的准确性,分别选择12和9个基因以通过QPCR分析它们的表达(见附加文件中的引物信息1.).18S RNA作为内参对照。RNA样本的制备方法与RNA- seq相同。从每个样本中提取1微克总RNA,通过PrimeScript™RT Reagent Kit (TaKaRa,中国)与RT- primer Mix合成cDNA。qPCR在CFX96™Real Time系统(Bio-Rad,美国)和SYBR Green Real- Time PCR Master Mix (TaKaRa,中国)上进行。比较阈值循环(CT)方法(2-ΔΔct),用以评估每个基因的相对表达水平[28].
结果
尼尔的特征Rf4
在BC中只观察到两种表型,完全雄性不育和完全雄性不育4.和公元前5.人群,男性肥沃的植物可以形成正常的花粉并正常开花(见附加文件2.).我们在产生正常花粉的植物的数量方面观察到1:1的比例和在BC中显示中止花粉的植物数量4.和公元前5.群体,表明男性生育能力由一个单一核基因控制(见附加文件3.).此外,我们观察到BC的男性生育能力5.人口与紧密联系在一起的Rf4标记(参见附加文件4.).以上结果表明Rf4已成功转移到NIL_Rf4.
NILs的基因组图谱
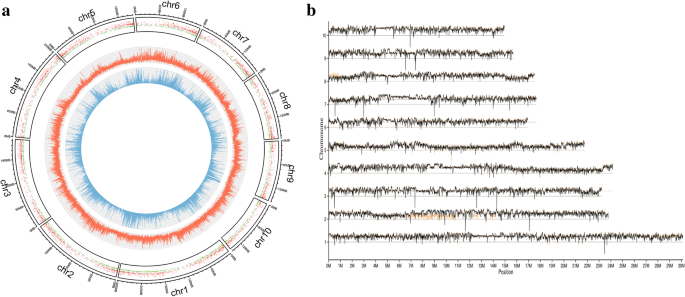
为了揭示NILs之间的基因组差异,利用全基因组测序检测多态SNPs。从NIL_中获得了98,331,340和83,494,528个短readsRf4(7.44×深度覆盖)和NIL_rf4(6.48×深度覆盖率)。然后将这些读数与B73参考基因组对齐。Nil_之间总共1,216,260个SNPRf4和NIL_rf4在整个基因组中鉴定出,其中26,383个SNP位于外显子,位于内含子中,58,671个SNP位于内含子,位于可变剪接位点,41,150个SNPS位于UTRS中,位于非基因区域中,位于1,089,887个SNP中,位于非基因区域中(图.1).上述结果表明,这些SNP主要位于非编码区。基因组测序结果表明GRMZM2G122850是含有氨基酸变化的独特基因Rf4映射区域。GRMZM2G122850编码一个含390个氨基酸残基的硒结合蛋白;通过TargetP进行的亚细胞定位预测表明,该蛋白可能靶向线粒体。在其编码序列中发现4个SNPs,导致R316H和D359N两个错义突变。通过双脱氧链终止法证实了这些snp(见附加文件)5.),并且需要进一步研究它们的潜在效果。
接下来,计算每个已识别SNP的SNP指数,并将两个样本的所有SNP指数绘制到整个玉米基因组上(图。1 b).我们发现,NILS在大多数玉米基因组中共享类似的SNP指标。然而,在染色体的短臂上观察到一系列偏置的SNP指数从0到1 MB的短臂,这与之一致Rf4映射区域[19].这些结果表明,NILS之间的基因组变化主要位于附近Rf4发生地
新基因的推定功能
如前所述,B73的参考基因组可能不完整,甚至可能缺失序列Rf4.在这项研究中,这些未映射的reads被进一步组装和注释。我们在NILs中获得了2890个推测基因(见附加文件)6.)以及它们的大部分功能(> 90%)可以注释(参见附加文件7.).在新发现的基因中,有645个基因可以靶向线粒体,并发现1个PPR蛋白(Contig.4402.1)。这些新基因可能有助于揭开谜底Rf4未来的候选人。
NILs的转录组谱
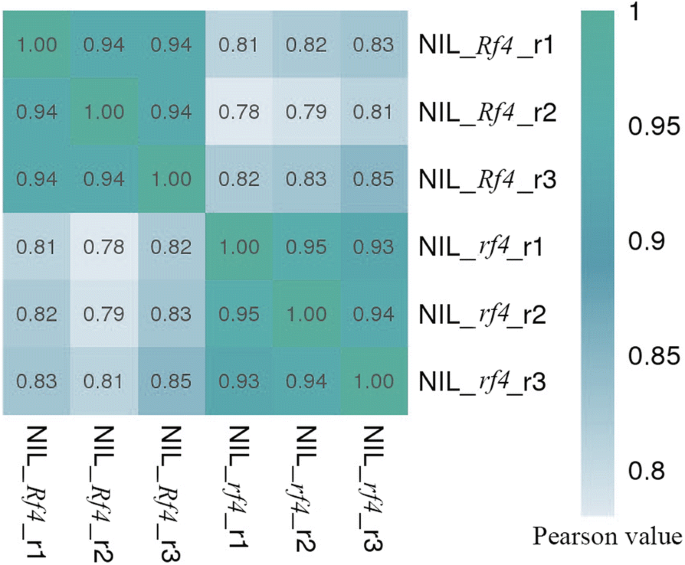
为RNA-Seq制备了6个文库,每个文库包含3个生物复制。6个样本共获得2.65亿个150 bp的配对端序列;每个样本的读数在3700万到5400万之间。所有样本的读长为> 37gb,约为玉米基因组的15倍。大约71%的短reads可以被唯一地映射到参考基因组(见表)1.)皮尔逊相关系数分析显示2.两个组的值大于0.93,并揭示了生物重复之间的高度相关性和略有可变性,显示转录组测序数据的可靠性(图。2.).
二基因的验证和功能表征
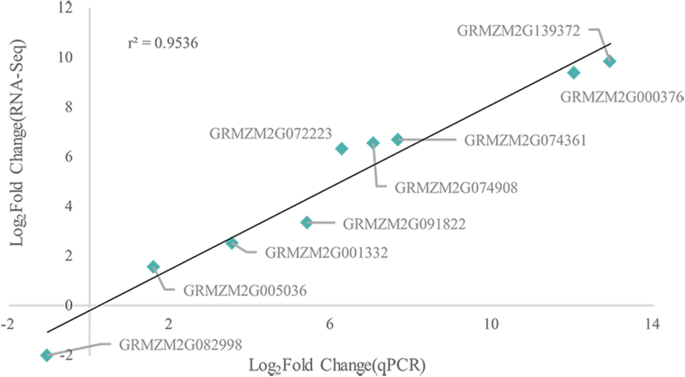
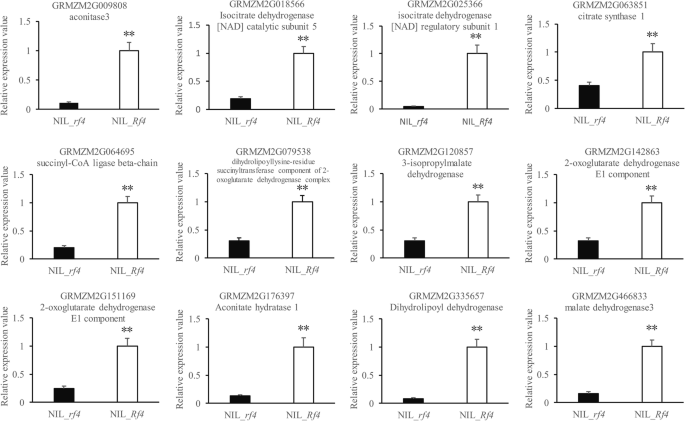
为了检测雄性育性转换过程中的转录组学变化,将发育小穗中表达的所有基因的表达水平与NIL进行比较_Rf4和NIL_rf4(无花果。1).共鉴定出7125个deg, 8125个转录本,其中91.22%(7740/8125)上调或下调≥2.0倍。在7125个基因中,有3873个基因表达上调Rf4, 3252个基因表达下调。deg的丰度表明,在CMS-C玉米育性恢复过程中,基因表达的变化是活跃的。通过结合我们的genome - seq数据,我们共鉴定了1679个在整个基因组中具有外显子SNPs的DEGs(图)。1和附加文件8.).这些DEG的额外分析可能有助于揭示生育恢复机制Rf4.为了验证本研究中RNA-Seq所揭示的转录组变化的准确性,我们随后通过qPCR检测9个随机选择的deg的转录水平(图)。3.).qPCR得到的9个基因的表达谱与RNA-Seq (r2. = 0.9536),证实了转录组测序结果的可靠性。
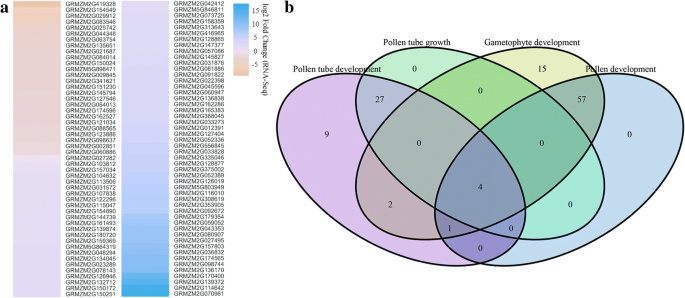
为了确定其推定功能,对DEGS进行分析。共有5269次涨幅为4810级。在生物过程类别中,代谢过程,细胞过程和单有机物过程是前三个丰富的簇。在细胞组分类别下,有451个富集的术语,大多数基因被分类为细胞和细胞部分。关于分子函数类别,结合和催化活性是最丰富的子类别。此外,超高度测试显示242显着丰富的GO条款(参见附加文件9).在242个GO项中,有4个与100个DEGs直接参与花粉形成:花粉管发育(GO0048868)、花粉管生长(GO0009860)、花粉发育(GO0009855)和配子体发育(GO0048229)。RNA-Seq结果显示,与上述四个GO项相关的100个基因中,有74个在雄性可育的NIL_中表达上调Rf4植物(图。4),表明大多数这些基因在玉米花粉形成中发挥了正调节作用。此外,欧拉图分析显示,大多数这些基因在花粉发育中具有各种功能(图。4 b).重要的是,其中四个,GRMZM2G170400(磷乙醇胺N-甲基转移酶3),GRMZM2G060886(S-腺苷-1-甲硫氨酸依赖性甲基转移酶),GRMZM2G122296(磷乙醇胺N-甲基转移酶1)和GRMZM2G113506(半乳糖核糖基转移酶)与所有人有关与花粉发育有关的术语,暗示了对花粉发育的关键影响。
鉴定雄性生育率相关的次数
为了鉴定与雄蕊发育相关的关键基因,DEGs与548只雄性进行了比对拟南芥从PMRD检索到生殖基因,共有198个基因与这些基因高度同源拟南芥男性生殖基因;截止e_value是<1e- 30(请参阅附加文件10).其中63个靶基因在玉米花药和穗状花序中优先表达(见附加文件)11),建议他们可能在玉米男性复制中发挥关键作用。大多数RF蛋白由PPR基因家族编码。为了揭示参与玉米CMS-C生育恢复的潜在PPR基因,观察到56个差异表达的PPR蛋白,其中分别位于3和28中,分别位于线粒体和叶绿体中(参见附加文件12).然而,在染色体8上发现只有两个差异表达的PPR基因,ZMPPR432(GRMZM5G851564)和ZMPPR443(GRMZM2G050485),但两者都远离映射区域Rf4并且不能是Rf4候选基因。
能量代谢相关DEGs的鉴定
植物雄性不育转化与能量代谢密切相关,能量不足可导致雄性不育[2.,29].为了深入了解CMS-C玉米育性恢复的能量机制,通过KEGG分析发现58个DEGs参与了糖酵解、戊糖磷酸途径和丙酮酸代谢等细胞能量代谢过程(表)2.).在58个能量代谢相关基因中,NIL_基因表达上调和下调分别为31和27个Rf4,分别。编码乙醇脱氢酶样2蛋白的GRMZM2G154007是上调最多的基因(fold change = 4096),编码果糖二磷酸醛缩酶7蛋白的GRMZM2G345493是下调最多的基因(fold change = - 2048)。有趣的是,NIL_中两个糖酵解相关基因(GRMZM5G824600和GRMZM2G071630)未表达Rf4或NIL_rf4,分别。能量代谢相关基因表达水平的不同表明NIL_之间存在潜在的能量水平差异rf4和NIL_Rf4能量代谢过程可能在CMS-C育性恢复中起关键作用。
线粒体TCA循环中IDH和OGDH的丰度与生育能力的恢复有关
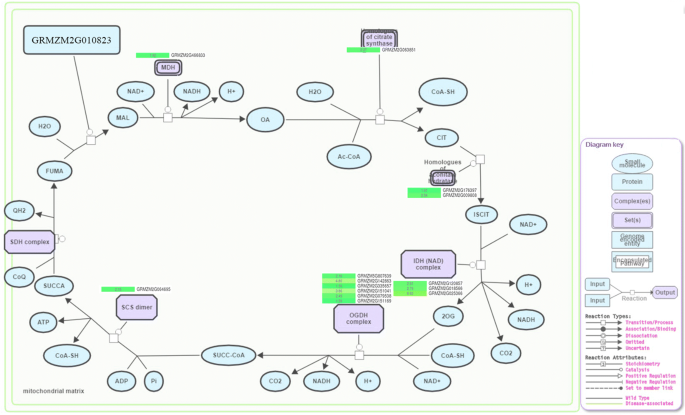
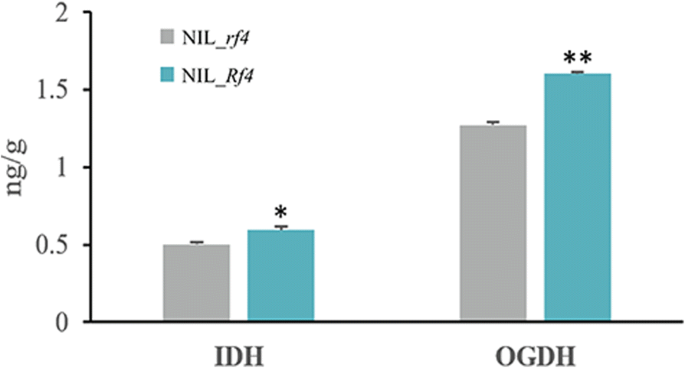
以上KEGG分析结果表明NIL_之间可能存在不同的能量代谢水平Rf4和NIL_rf4.线粒体TCA循环在能量代谢中起着至关重要的作用[30.].因此,在Plant Reactome Pathway数据库中查询所有的deg (http://plantreactome.oicr.on.ca/),鉴定出14个参与线粒体TCA循环的DEGs。其中,GRMZM2G079538/GRMZM5G807639和GRMZM2G142863/GRMZM2G151041是重复基因。RNA-Seq数据显示,这些基因在NIL_中均表达上调Rf4(无花果。5.), qPCR结果证实了它们在NIL_Rf4和NIL_rf4(无花果。6.).表达谱分析表明,这些基因主要在花药和穗状花序中表达13).我们还使用qPCR来研究这些基因在花药和叶片发育中的表达水平(见附加文件)13).对于维护系(HZS),这些基因的表达水平在叶片和显影化学物之间相似;而对于CMS-C的雄性无菌线(CHZ),其中大多数在减数分裂期间在花药中高度表达。与维护系列相比,所有这些基因都倾向于在雄性无菌中表现出Meiosis I的更高的转录水平线。三(GRMZM2G120857,GRMZM2G018566,GRMZM2G025366)和六(GRMZM2G807636,GRMZM2G142865,GRMZM2G142865,GRMZM2G33565,GRMZM2G33565,GRMZM2G33565,GRMZM2G151169)基因分别参与IDH和OGDH复合物,两者都是TCA循环中的关键酶。我们的额外ELISA实验表明,IDH和OGDH的丰度在尼尔_明显高于Rf4比在尼尔_rf4(无花果。7.).以上结果表明,与TCA周期相关的基因与CMS-C雄性育性有关。
讨论
作为CMS-C的显性恢复基因,Rf4在杂交玉米制种中具有很大的潜在价值。理解的函数方法Rf4还将有利于揭示玉米CMS-C的流动机制。到目前为止,Rf4候选基因尚未被证实,其作用机制尚不清楚。在这项研究中,在Rf4建立基因座用于比较转录组分析以揭示其功能。遗传分析和基因型鉴定结果表明,NIL_Rf4男性生育力恢复Rf4.Genome-SEQ用于揭示尼尔的基因组变异,结果证实,大多数SNP都挤在附近Rf4的位置。然而,许多SNPs也聚集在2号染色体的中间附近。其他研究也报道了NILs非目标区域的各种基因组差异聚类[31].事实上,由于基因组组成和重组的复杂性,nill总是呈现出广泛的残差杂合性[32,33].此外,以前使用SNP芯片的研究普遍报道了NILs之间存在SNP [34,35,36].与SNP芯片相比,下一代测序具有更广泛和更敏感的检测能力,这也可能解释为什么我们检测到比以前的研究人员更多的SNP。
能量供应不足会损害雄性配子的发育,但不会损害营养器官,最终导致雄性不育[2.].前人的研究也表明,CMS-C中花粉功能障碍与能量代谢密切相关。三个atp合成的嵌合orf,atp9-c,COX2-C.,atp6-c,首先在CMS-C植物中鉴定[37].尽管如此,Meyer并未观察到任何嵌合蛋白,但确实报道了几种ATP合酶复合物蛋白在CMS-C流苏中的表达减少[38].可以推断,由于低ATP9-C翻译效率,CMS-C植物中的花粉流产是由ATP生产不足引起的。此外,我们的组分析了雄性无菌线(C48-2)及其维护系N48-2之间的差异,并且我们发现一些可脱果和差异表达的蛋白质与能量代谢有关[39,40].在代谢水平上,与保持系花药相比,CMS-C系花药在小孢子发育过程中呼吸强度、抗氰化物呼吸、细胞色素氧化酶活性和ATP含量均较低[41,42].在本研究中,在雄性生育率恢复的线中检测到升高的IDH和OGDH酶水平。IDH和OGDH都是NADH合成酶,并在线粒体TCA循环中发挥关键作用,这决定了细胞中的能量供应[29,30.].线粒体柠檬酸盐合酶酶的下调最终导致雄性不育性[43]我们的结果为线粒体TCA循环与植物雄性育性之间的关系提供了新的证据。有趣的是,与CMS-C雄性不育系相比,这些TCA循环相关基因在NIL的花药中上调_Rf4而在保持系花药中下调。这些结果表明,维持系与NIL_之间可能存在不同的能量代谢相关基因表达水平Rf4尽管它们都是雄性的。这一假设还需要进一步验证,因为这种差异也可能是由不同的花粉发育阶段造成的。
除了玉米中的那些,许多关于其他物种中CMS的研究表明,与正常线相比,CMS线始终似乎具有较低的能量代谢水平[44,45,46].线粒体在提供蜂窝能量方面发挥着重要作用。大多数线粒体蛋白质由核基因编码;这些蛋白质在细胞质中合成,然后进入线粒体[47].最近,已经增加了这些核编码的线粒体蛋白质和CMS生育恢复之间的联系所关注的增加。Rf2编码线粒体醛脱氢酶(ALDH)酶,其表达可挽救玉米t型细胞质雄性不育(CMS-T) [48,49].GRP162和RFC3蛋白都在线粒体内积累,可以直接相互作用Rf5拯救红莲细胞质雄性不育(HL-CMS)在水稻中[50,51].六酮酶6是一种线粒体局部化蛋白,其相互作用Rf6是恢复HL-CMS生育能力所必需的[52].该研究为能量代谢相关基因直接参与植物CMS育性恢复提供了直接证据。在我们的实验中,鉴定了14个参与TCA循环的上调基因;但其在恢复玉米CMS-C肥力中的具体作用尚需进一步研究。
结论
几乎是均匀的线条Rf4通过基因分析、分子标记检测和全基因组测序等方法对该位点进行了鉴定。雄性可育NIL_的比较转录组分析Rf4植物和男性无菌尼尔_rf4植物在CMS-C雄性生殖恢复过程中发现了几个关键基因和代谢途径。有趣的是,所有参与线粒体TCA周期的DEGs在雄性可育NIL_中均上调Rf4我们的ELISA结果表明,在NIL中,IDH和OGDH酶的丰度都较高_Rf4比在尼尔_rf4,表明IDH和OGDH酶可能与玉米CMS-C肥力恢复有关。这些结果扩展了目前对玉米CMS-C雄性不育恢复的认识。
缩写
- ALDH:
-
醛脱氢酶
- CMS:
-
细胞质雄性不育
- CMS-C:
-
c型细胞质雄性不育
- CMS-T:
-
t型细胞质雄性不育
- 可见:
-
差异表达基因
- ELISA:
-
酶联免疫吸附试验
- FPKM:
-
每千碱基外显子的片段,每百万片段的图谱
- 去:
-
基因本体论
- HL-CMS:
-
红莲细胞质雄性不育
- 嗯:
-
隐马尔可夫模体
- IDH:
-
异柠檬酸脱氢酶
- Kegg:
-
京都基因和基因组百科全书
- 零:
-
near-isogenic线
- OGDH:
-
酮戊二酸脱氢酶
- ORF:
-
开放阅读框
- PMRD:
-
植物男性再现数据库
- 警察公共关系科:
-
戊庚二肽重复
- QPCR:
-
实时定量PCR
- QTL:
-
数量性状位点
- 射频:
-
生育恢复者
- SNP:
-
单核苷酸多态性
- 柠檬酸:
-
三羧酸
- TPM:
-
记录每百万
- UTR:
-
翻译区
参考文献
- 1.
陈志强,赵宁,李松,聂辉,温德尔JF,华军。植物线粒体基因组进化与细胞质雄性不育。植物学报。2017;36(1):55-69。
- 2.
陈林,刘彦。作物雄性不育与育性恢复。植物学报。2014;65:579-606。
- 3.
胡俊,黄虎,黄q,秦x,yu c,王l,李思,朱克,朱玉粒细胞和植物细胞质雄性不育。线粒体。2014; 19:282-8。
- 4.
touzet p,meyer eh。植物中细胞质雄性不育和线粒体代谢。线粒体。2014; 19:166-71。
- 5.
Kim Y,Zhang D.作物杂交育种中雄性育性的分子控制.趋势植物科学.2017;23(1):53-65。
- 6.
Bohra A,Jha UC,Adhimoolam P,Bisht D,Singh NP.大田作物杂交育种中的细胞质雄性不育(CMS).植物细胞代表2016;35(5):967-93。
- 7.
Beckett JB。玉米雄性无菌细胞质的分类(玉米l .)。作物科学。1971;11(5):724 - 7。
- 8.
玉米细胞质雄性不育的稳定性研究。作物科学。2009;49(1):77 - 84。
- 9。
研究了蛋白质标记在玉米自交系转化为细胞质雄性不育基础中的应用。Genetika。2016;48:691-8。
- 10。
秦TC,徐曼丽,盾DX。细胞质雄性不育:恢复系基因数量的鉴定。玉米遗传学鸡笼新闻Lett. 1990;64:124。
- 11.
陈树生,陈伟。玉米c质雄性不育恢复基因的染色体定位。河南农业大学学报,1992;26(2):125-30。
- 12.
Kohls S,邮票P,KNAAK C,Messmer R. QTL参与玉米C型细胞质雄性不育的男性生育率的部分恢复。Al Appl Genet。2011; 123(2):327-38。
- 13.
胡玉明,唐建辉,杨辉,谢海林,卢晓明,牛建辉,陈伟伟。识别和映射RF-I.抑制剂Rf5玉米cms-c恢复基因(玉米l .)。应用电子学报。2006;113(2):357-60。
- 14.
刘y,赵z,鲁y,李c,王继,东b,梁b,邱t,zeng w,cao m。玉米CMS-C新型恢复基因的初步鉴定(Zea Mays L.)。peerj。2016; 4:E2719。
- 15.
复制使基因作图复杂化Rf4玉米cms-C细胞质雄性不育恢复基因。作物科学。1991;31(5):1263 - 6。
- 16.
唐j,ji h,黄z,ji l,王c。玉米C-细胞雄性不育的主要恢复基因(RF4)的映射。Acta Tryanturaue Univ Henanensis。2000; 35(2):99-102。
- 17.
唐j,胡y,ji h,陈文,ji l,zhen y。玉米CMS-C细胞质雄性不育的恢复基因RF4的SSR标记。J河南农业大学。2001; 35(1):1-03。
- 18.
玉米c型细胞质雄性不育恢复的遗传因素分析.2010https://doi.org/10.3929/ethz-a-006391548.访问2017年6月13日。
- 19.
ren r,nagel ba,kumpatla sp,zheng p,cutter g,greene tw,thompson sa。玉米细胞质雄性不育(CMS)C型恢复剂RF4基因,分子标记及其使用。2011. HTTPS://www.google.com/专利/ US20120090047。2012年12月12日。
- 20。
acta botanica sinica(云南植物研究中心);RNA杂志。2013;10(9):1469 - 76。
- 21。
Gaborieau L, Brown GG, Mireau H. pentatricop肽重复基因进化为细胞质雄性不育恢复体的倾向。前面。植物科学。2016;7:e1002910。
- 22。
Porebski S, Bailey LG, Baum BR。高多糖和多酚成分植物CTAB DNA提取工艺的改进。植物学报1997;15(1):8-15。
- 23.
李洪波,李洪波。基于盲轮变换的短读快速定位。生物信息学。2010;25(5):1754 - 60。
- 24.
利用MutMap对水稻进行基因组测序,揭示了水稻中重要的农艺位点。生物科技Nat》。2012;30(2):174 - 8。
- 25.
Takagi H,Abe A,Yoshida K,Kosugi S,Natsume S,Mitsuoka C,Uemura A,Utsushi H,Tamiru M,Takuno S. QTL-SEQ:通过两种基因组重构DNA从两种DNA中的定量特质基因座的快速映射膨胀的人口。工厂J. 2013; 74(1):174-83。
- 26.
Wagner GP, Kin K, Lynch VJ。使用RNA-seq数据测量mRNA丰度:RPKM测量在样本之间是不一致的。定理Biosci。2012;131(4):281 - 5。
- 27.
Wei K,Han P.玉米戊肽重复蛋白质。mol繁殖。2016; 36:170。
- 28.
Livak KJ,Schmittgen TD。使用实时定量PCR和2的相对基因表达数据分析(-Delta Delta C(T))方法。方法。2001; 25(4):402-8。
- 29.
张颖,王志强。三羧酸循环对植物生产力的影响。2018;https://doi.org/10.1111/jipb.12690..
- 30.
Nunes Nesi A,Araújo WL,Obata T,Fernie AR.线粒体三羧酸循环的调节。Curr Opin Plant Biol.2013;16(3):335-43。
- 31.
梁J,Ma Y,Wu J,Cheng F,Liu B,王X.基于地图的卷心病患者卷心菜(Brassica Oleracea)中的主要基因雄性无菌MS-CD1基因克隆。Al Appl Genet。2017; 130(1):71-9。
- 32。
重组:植物基因组进化中一个被低估的因素。(1)中国科学(d辑:地球科学)2007;
- 33。
Eichten SR, Foerster JM, de Leon N, Kai Y, Yeh C, Liu S, Jeddeloh JA, Schnable PS, Kaeppler SM,施普林格NM。B73-Mo17近等基因系在玉米中表现出分散的结构变异。植物杂志。2011;156(4):1679 - 90。
- 34。
Pea G,Aung Hh,FraScaroli E,Landi P,Pème。玉米(Zea mays L.)中的一组近代近代线的广泛基因组特征。BMC基因组学。2013; 14(1):61。
- 35。
Brinton J, Simmonds J, Minter F, Leverington Waite M, Snape J, Uauy C.增加果皮细胞长度是六倍体小麦粒重的主要数量性状位点。新植醇。2017;215(3):1026 - 38。
- 36.
关键词:水稻,BPH,近等基因系,抗性谱,分子标记应用电子学报。2017;130(11):2345-60。
- 37.
作者简介:杜威(1996 -),男,硕士研究生。玉米C雄性不育细胞质中嵌合线粒体基因的表达。咕咕叫麝猫。1991;20(6):475 - 82。
- 38.
梅尔LJ。玉米CMS-C花粉败育原因的研究.2009.http://hdl.handle.net/10355/9563。2009年5月查阅。
- 39.
黄磊,向军,刘军,荣涛,王军,陆艳,唐强,文伟,曹明。玉米CMS-C基因的表达特征。原生质。2012;249(4):1119 - 27所示。
- 40.
李春华,赵志强,刘永强,梁波,关松,兰华,王军,陆勇,曹明。玉米CMS-C不育性关键转录因子基因及其代谢途径的比较转录组分析。PeerJ。2017; 5: e3408。
- 41.
夏涛,刘军。玉米细胞质雄性不育与抗氮呼吸关系的研究。中国农业科学,1988;21(5):39-43。
- 42.
夏T,刘杰.玉米雄性不育细胞质的细胞色素氧化酶活性和ATP含量.农业学报.1994;9(4):33-7。
- 43.
Landschutze V, Willmitzer L, Muller robber B.通过反义抑制线粒体柠檬酸合酶来抑制花的形成,导致特定的花子房组织的瓦解。Embo j . 1995; 14(4): 660 - 6。
- 44。
雄性不育烟草线粒体atp1转录本积累异常,花期ATP/ADP比值降低。植物学报。2000;42(3):531-44。
- 45。
Teixeira Rt,Knorpp C,Glimelius K.修饰的蔗糖,淀粉和ATP水平在两种异质雄性无菌线中B. Napus.《实验机器人》2005;56(414):1245-53。
- 46。
关键词:大豆,细胞质雄性不育系,NJCMS1A,保持系,转录组分析稳定)。《公共科学图书馆•综合》。2015;10 (5):e126771。
- 47。
线粒体基因组:结构、转录、翻译和复制。生物化学与生物物理学报-能量学。1999年,1410(2):103 - 23所示。
- 48.
关键词:玉米,t细胞质雄性不育,rf2核恢复系科学。1996;272(5266):1334 - 6。
- 49.
刘F,Cui X,Horner HT,Weiner H,Schnable PS。Mitochondrial醛脱氢酶活性是玉米雄性生育所必需的。植物细胞。2001; 13(5):1063-78。
- 50。
胡建军,王凯,黄伟,刘刚,高艳,王军,黄强,季勇,秦旭,万磊,等。水稻pentatricoptide repeat protein RF5通过与富含甘氨酸的GRP162的复合物在红连细胞质雄性不育系中恢复育性。植物细胞,2012;24(1):109 - 22所示。
- 51。
秦X,黄Q,萧H,张Q,Ni C,Xu Y,Liu G,杨D,朱y,Hu J.组装恢复的含水稻DUF1620和WD40样重复蛋白生育复杂。新植物。2016; 210(3):934-45。
- 52.
黄武,俞c,胡j,王l,丹z,周w,他c,zeng y,姚g,齐联肽 - 重复家庭蛋白质蛋白RF6用六酮酶6拯救水稻细胞质雄性不育。PROC NATL ACAD SCI。2015; 112(48):14984-9。
- 53.
翟建军,张华,黄轲,南广,Walbot V, Meyers BC。玉米花药中细胞类型依赖的减数分裂前和减数分裂相rna的时空动态。中国科学院院刊。2015;112(10):3146-51。
确认
我们感谢Novogene(中国北京)和Basebio(中国成都)的同事,感谢他们在解释基因组和转录组数据方面的帮助。
资金
国家重点研发计划项目(no . 2016YFD0101206);四川省科技厅项目(no . 2016NZ0106)。
数据和材料的可用性
在当前研究中生成和分析的基因组测序和转录组测序原始数据可在Fig SHARE存储库中获得(https://doi.org/10.6084/m9.figshare.5709637.v1,https://doi.org/10.6084/m9.figshare.5709646.v1,https://doi.org/10.6084/m9.figshare.5709658.v1).
作者信息
隶属关系
贡献
YML设计了该研究,进行了大部分实验,并写了这篇论文。GW大多数实验并分析结果。YYX,XWL和JT执行了部分实验。YLL,HL,SZZ和CL为实验提供了建议。MJC设计了该研究并修改了论文。所有作者审查并批准了最终手稿。
通讯作者
道德声明
道德认可和参与同意
不适用。
同意出版
不适用。
竞争利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意
Springer Nature在公布的地图和机构附属机构的管辖权主张方面保持中立。
附加文件
附加文件1:
本研究用于qPCR的引物列表。18S RNA作为内参对照。(XLSX 12 kb)
额外的文件2:
雄性可育穗子的比较(一),花药(b)和花粉(c)零和零之间_Rf4和NIL_rf4.在图中。(c),花粉粒用1% (W/v) KI-I2.(TIF 787 kb)
额外的文件3:
不列颠哥伦比亚省的生育隔离4.和公元前5.人群。采用卡方检验评价育性分离率。(XLSX 11 kb)
附加文件4:
BC的基因型分析5.人口通过主导Rf4紧密连接标记(5 ' -CGCACCTAACCGTCTCC-3 ', 5 ' -GCGCAAGTACGCCGTAC-3 ')。F,肥沃的个人;年代,无菌的个人。(TIF 129 kb)
额外的文件5:
GRMZM2G122850编码序列中SNP的验证。(TIF 191 KB)
额外的文件6:
新型基因的编码序列。(TXT 1323 KB)
额外的文件7:
新型基因的功能注释。(XLSX 315 KB)
附加文件8:
所有带有外显子SNPs的deg。(XLSX 504 kb)
额外的文件9:
由Fisher的测试获得二百四十二个显着丰富的术语(Q≤0.05)。(XLSX 20 kb)
额外的文件10:
对同源的待同源拟南芥雄性不育(MS)/生殖(MR)基因(XLSX 35 kb)
额外的文件11:
男性再现相关的DEG的表达模式。从Qteller网站检索它们的表达值(FPKM)(www.qteller.com),并且FPKM值是日志2.由(FPKM + 1)变换;然后计算每个基因的z值。(TIF 298 kb)
额外的文件12:
DEG中PPR基因的鉴定。“方向”是指相对于NIL中的表达水平_Rf4.(XLSX 13 kb)
额外的文件13:
(一)14个TCA周期相关基因的组织表达谱。表达数据(FPKM值)来自qTeller (www.qteller.com).日志中记录FPKM值2.转换(FPKM)+ 1) ,并计算每个基因的Z分数。(b)雄性不育系CHZS与保持系HZS的tca相关DEGs表达水平比较A1、A2和A3分别为发育的花药,花药长度分别为1.5~ 2.0 mm(减数分裂I)、2.0~ 2.5 mm(减数分裂II)和2.5~ 3.0 mm(单核小孢子)。这些数据是作为至少三个生物重复的平均值±标准差给出的。玉米花粉发育阶段与花药长度的关系在另一篇报道中也有发现[53](TIF 466 kb)
权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
刘彦,魏刚,夏彦。et al。比较转录组分析表明,三羧酸循环相关基因与玉米CMS-C的育性恢复有关。BMC植物杂志18,190(2018)。https://doi.org/10.1186/s12870-018-1409-z
收到了:
认可的:
出版:
关键字
- 玉米
- 细胞质雄性不育
- 生育恢复
- 氧化戊二酸脱氢酶
- 异柠檬酸脱氢酶
- 转录组测序