抽象的
背景
圣Augustinegrass [Stenotaphrum secundatum(沃尔特。)Kuntze]是一个暖季,常年草坪草种,适用于家庭草坪和经济生态价值的商业景观。然而,圣奥古斯丁斯缺乏基因组资源妨碍了遗传方差充分利用遗传方差,以最大限度地提高遗传利益,并限制了我们对物种演变的理解。
结果
在本研究中,我们使用基因分型测序(GBS)方法构建了首个St. Augustinegrass高密度连锁图谱。该综合连锁图谱由2871个单核苷酸多态性(SNP)和81个简单序列重复(SSR)标记组成,全长1241.7 cM,标记间平均距离0.4 cM,是迄今为止最密集的圣奥古斯特草遗传图谱。比较基因组分析显示,在圣奥古斯汀草、谷子、高粱和水稻从一个共同祖先分化出来之后,发生了染色体间排列和独立的嵌套染色体融合事件。共检测到48个与草坪质量相关性状的QTL候选位点,包括草坪总体质量、叶片质地、遗传颜色和草坪密度。在LG3和LG8连锁群上鉴定出3个热点区域,不同性状的多个qtl重叠。这些QTL区域包含多个叶片发育相关基因。
结论
本研究开发了第一个高密度遗传图谱,并确定假定的QTL与草坪质量,这对于圣Augustinegrass标记辅助选择(MAS)提供了宝贵的遗传资源。
背景
圣Augustinegrass(Stenotaphrum secundatum[华] Kuntze)是暖季型草坪草被很好地适应世界的热带和亚热带地区[1].这种草原产于墨西哥湾、西印度群岛和西非,在美国的墨西哥湾沿岸、墨西哥南部、整个加勒比地区、南美洲、南非、西非、澳大利亚、南太平洋和夏威夷群岛被广泛使用。与其他草坪品种相比,圣奥古斯汀草表现出优越的耐阴性和中等低的投入要求[2].它已经在美国南部在其宽广叶片流行的草坪,和匍匐茎迅速伸长,从而使草非常适合草皮生产[3.].
圣奥古斯汀草属于Panicoideae亚科Paniceae部落,是禾本科(禾本科)中最大的亚科之一。这个大亚科包含许多具有重要经济价值的物种,包括草坪草蜈蚣草(假俭草)和圣Augustinegrass(Stenotaphrum secundatum)[4],生物燃料储备柳枝稷(柳枝稷)[5]等重要作物,如粟(Setaria italica)、高粱(高粱二色的)和玉米(Zea Mays.)[6].该亚家族包括巨大的形态学,生理和细胞学多样性和几种基本染色体数。据报道了这一点X = 9 andX = 10 predominate the basic chromosome number in Panicoideae [6].了解染色体数目的进化机制是成功破译草类基因组进化的关键组成部分。因此,人们对了解St. Augustinegrass和其他Panicoideae成员之间的比较基因组关系有很大的兴趣。
圣奥古斯丁的基本染色体数量是X= 9,带有二倍体(2N = 2X = 18), triploids (2N = 3X = 27), and tetraploids (2N = 4X = 36) reported [7],虽然非整倍体(2N = 28, 2N = 32) genotypes have also been identified more recently [3.].分子标记可用于鉴定品种和表征种质多样性。更重要的是,分子标记可用于构建遗传图谱,并与表型数据一起,可用于绘制控制感兴趣性状的基因组区域。近年来,圣奥古斯汀草分子标记的研究和连锁图谱的构建取得了一定的进展。SSR (Simple sequence repeats, SSR)标记已被开发并用于评价St. Augustinegrass [8,9].最近,Kimball等报道了第一张St. Augustinegrass连锁图谱,共160个SSR标记,由9个连锁基团组成,跨越1176.24 cM [10.].利用该连锁图谱还鉴定出多个与冬季生存相关性状相关的QTL。然而,这是一个低标记密度的地图,使得它不足以精细地绘制感兴趣的特征。此外,以pcr为基础的标记,如SSRs,使基因分型的时间和劳力需要很高。为了进行大规模的关联作图,提高标记辅助选择(MAS)在育种工作中的效率,利用单核苷酸多态性(SNP)等新型分子标记开发高密度连锁图谱至关重要。
高吞吐量排序技术为开发大量SNP标记提供新的工具,探索物种多样性,构建联系地图和进行基因组 - 宽协会研究(GWAS)[11.,12.].基因分型通过测序(GBS),一个简单的高度多路系统,用于构建遗传分析和基因分型的简化代表性库,作为一系列植物物种中MAS育种的成本效益和独特工具,正变得越来越重要[13.,14.,15.,16.,17.].最重要的是,即使在没有参考基因组的情况下,GBS也是植物育种应用的优秀方法[13.].
高密度连锁图谱是定量性状位点定位和基因图谱克隆的重要遗传工具。本研究旨在1)利用GBS方法构建结合SNP和SSR标记的高密度连锁图谱;2)与其他草种进行基因组比较研究;3)鉴定与草坪品质性状相关的QTL。
结果
通过测序和SNP发现基因型
“伪f2”作图策略使我们能够生成非近交系物种的连锁图谱[18.].GBS文库构建的两个亲本(“罗利”和“塞维利亚”)和115 04-0030-03-F2杂交和测序上的Illumina HiSeq 2500共236600000原始单端的读出是从测序获得。通过识别条形码和切割位点,低质量的读取被拆除,并211500000高质量读取被保留。读取数从048万范围为330万每个混合,平均为160万。亲本系在比F相对更高的深度进行测序1以最大限度地检测和分离亲本的snp。“罗利”和“塞维利亚”分别获得了760万和650万reads,大约是杂交平均值的4.7倍和4倍(附加文件1:表S2)。
使用非引用GBS SNP调用管道UNEAK发现SNP标记。共鉴定出19,810个双等位基因多态性位点。在丢弃低基因分型率(< 90%)和明显扭曲的snp (P. < 0.01) segregation ratios, 2871 high quality SNPs were obtained for linkage map construction. These consisted of 1100 ‘lm x ll’, 1571 ‘nn x np’, and 200 ‘hk x hk’ type SNPs (For definition of allele scoring refer to Methods section.).
连锁图施工
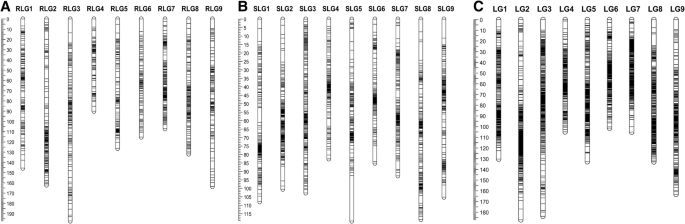
过滤后的标记被加载到JoinMap 4.0中[19.,用于联动图建设。最初,我们使用' lm xll '和' nn xnp '型snp为每个亲本构建单独的连锁图谱。在‘Raleigh’的连锁图谱中,1100个SNPs被定位为9个连锁群(LGs)(图2)。1a)中,名为RLG1-RLG9,将其基于“罗利”地图和谷子基因组之间的同源性指定。每个LG SNP标记的数量与上RLG7最多151和上RLG4最小的78(表改变1).一个ll nine LGs spanned a total distance of 1238.7 cM, with an average distance of 1.1 cM between markers. While RLG3 was the longest with 197.2 cM, RLG4 was the shortest with 90.0 cM. The same LG number was observed on the linkage map of ‘Seville’, named SLG1-SLG9 based on homology with the genome of foxtail millet (Fig.1b)。Total length for all the ‘Seville’ LGs was 914.2 cM, with average distance 0.6 cM between markers (Table1).对于每个LG,尺寸范围从82.6 cM的SLG4到119.3 cM的SLG5。SLG3包含的SNPs最多(223个),SLG6最少(128个)。与“Raleigh”图谱相比,“Seville”图谱包含了更多的SNP标记,但覆盖的长度更短。
' lm x ll '和' nn x np '型以及' hk x hk '型SNP和先前已鉴定的SSR标记[10.用于构建St. AugustineGrass的集成图。集成映射由2871个SNP和81个SSR标记组成,其分布在九个LG上(图。1C).综合图谱的遗传长度为1241.7 cM,标记间的平均距离为0.4 cM1).最长的LG2 (187.1 cM),最短的LG6 (101.4 cM)。“罗利”、“塞维利亚”标记和综合地图的完整信息可在附加文件中找到1:表S3,S4和S5。通过与由金博尔等构成的前面的连锁图谱锚固SSR标记。[10., LG1-kLG6、LG2-kLG1、LG3-kLG3、LG4-kLG9、LG5-kLG7、LG6-kLG4、LG7-kLG8、LG8-kLG5和LG9-kLG2对应。(桌子1出现在本段之后)。
草类比较基因组学分析
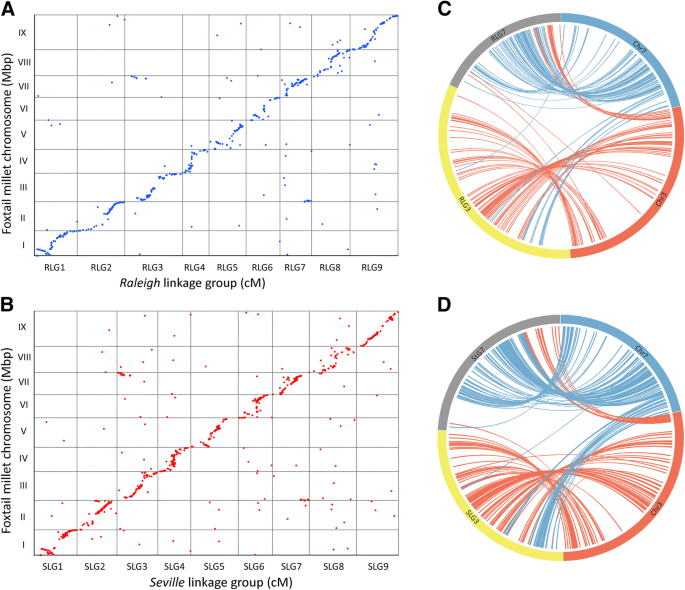
采用比较基因组学方法,对三种模式禾草(谷子、高粱和水稻)进行了同源性保存和染色体重排的研究。圣奥古斯汀草和谷子都属于禾本科Panicoideae亚科,基本染色体数相等X = 9. The blastn search against the foxtail millet genome revealed both ‘Raleigh’ and ‘Seville’ LGs showed orthologous relationships with foxtail millet chromosomes. St. Augustinegrass LGs were numbered in order based on the orthology to foxtail millet chromosomes. Among 1100 SNPs on ‘Raleigh’ LGs, 603 SNPs (54.8%) could be positioned on foxtail millet chromosomes, 544 of which could be mapped on the orthologous chromosomes (Table2和附加文件1:表S6)。同时,873(55.6%)SNP映射在'塞维利亚'LG上,位于Foxtail Millet染色体上,其中794个可以放在直晶染色体上(表2和附加文件1:表S7)。此外,点线图显示,St. Augustinegrass和谷子的基因组之间存在高度共线性(图2)。2a,b)。
尽管圣奥古斯汀草和谷子之间高度共线性和共线性,一些染色体重排被观察到区分两个物种。这些染色体间重排发生在ChrIII和ChrVII之间,导致RLG3和RLG7, SLG3和SLG7(图。2c, d)。谷子RLG3和SLG3的一端与ChrVII端同源,而RLG7和SLG7的一端位于chrii端(图2)。2C,D)。此外,有染色体倒位的是邻近RLG1,RLG5,RLG6和同源SLG1,SLG5,SLG6(图2的端部发生。2a,b)。
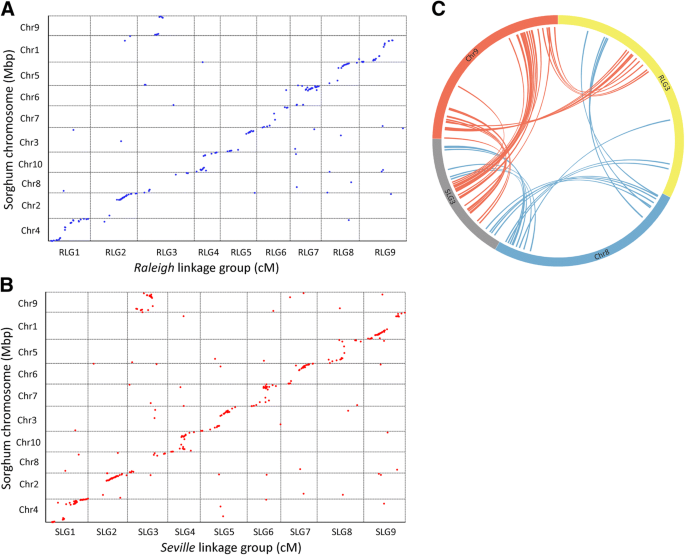
高粱是胰腺炎群的另一个成员,但具有比圣奥古斯丁的基本染色体数更高(X= 10)。比较基因组学分析发现,在“Raleigh”和“Seville”LGs上有199个和338个SNPs可以定位在高粱染色体上(见表)2,附加文件1:表S8和S9)。点图显示图圣Augustinegrass和高粱基因组之间的高共线性(。3.除R(S)LG3与高粱中的Chr8和Chr9同源外,St. Augustinegrass与高粱的同源染色体存在一一对应关系(图8)。3.C)。这种关系表明,在高粱中的Chr8和Chr9之间发生巢染色体融合事件,以在圣奥古斯丁草中形成R(S)LG3。
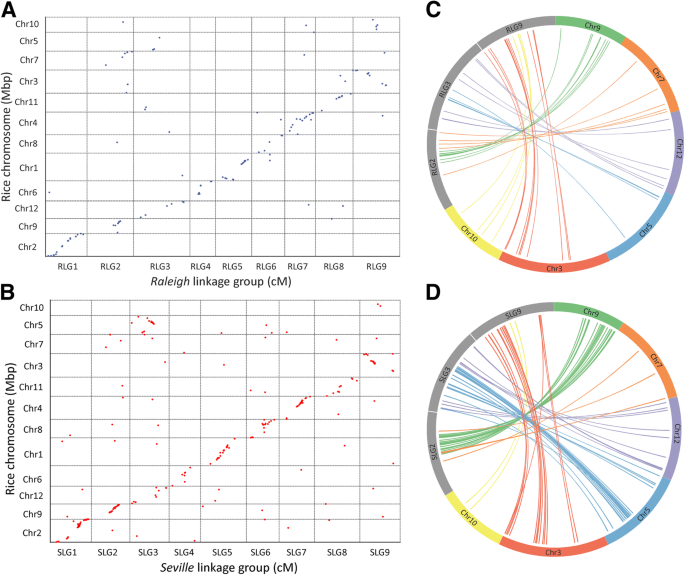
水稻基因组(奥雅萨苜蓿)已被普遍用作用于禾本科基因组分析的参考对照,因为它保留了12对基本染色体从草的共同祖先。没有关于“罗利”和“塞维利亚”连锁群128点220的SNP可能对水稻染色体被映射(表2,附加文件1:表S10和S11)。R(S)LG2、R(S)LG3和R(S)LG9分别与水稻Chr7和Chr9、Chr12和Chr5、Chr3和Chr10同源。Rice Chr9、Chr5和Chr10分别与Chr7、Chr12和Chr3的中间区域融合形成R(S)LG2、R(S)LG3和R(S)LG9(图)。4C,D)。这些结果表明,染色体的三个独立的双融合禾本科的进化过程中,形成在圣Augustinegrass 3条染色体。(桌子2出现在本段之后)。
草地品质相关性状的QTL鉴定
四对草皮质量相关性状(整体草皮质量,叶片质地,遗传颜色,和草皮密度)在金博尔等下多个环境评价。被选择用于QTL分析[10.].在所有环境中,共检测到48名候选QTL(表3.).其中,11 QTL,确定了整体草坪质量,11 QTL叶纹理,16 QTL遗传颜色和10 QTL为草坪密度。所有48个QTL都分布在所有连锁群LG6和LG7除外。
多个QTL在不同的环境中被重复鉴定。从总体质量来看,LW2013、LS2013和Across在LG3上的位置均为38.27 cM,而LW2013和Across在LG3上的位置均为53.57 cM(表2)3.).在LS2013、LW2015和cross中,在LG8的40.44 cM上发现了草坪整体质量的重叠区域。在遗传颜色方面,在所有环境中LG8的遗传颜色区间均为41.44-42.44 cM,而在LW2013和Across中LG5的遗传颜色区间均为81.61-82.01 cM(表2)3.).鉴定出四个重叠区域,用于草皮密度,包括53.57厘米和37.55-40.15cm,LG4上的45.22-56.52cm,LG8上的38.08厘米(表3.).叶片纹理在LG3 (38.27 cM和54.94 cM)和LG5 (90.54 cM)上存在3个重叠区域(表2)3.).
此外,我们发现QTL为共同位于同一区域,特别是在LG3和LG8不同性状。的re were seven QTL that overlapped in the interval of 35.66–40.15 cM on LG3, which included overall turf quality, turf density and leaf texture. On the same linkage group, there was another hot spot region (47.03–54.93 cM) that contained seven QTL for overall turf quality, turf density and leaf texture (Table3.).在LG8上,38.08 ~ 50.27 cM范围内,10个草坪总体质量、密度和遗传颜色的QTL重叠(见表2)3.).
QTL区域内的标记序列随后用于基因注释分析。结果表明:与叶的形成和发展,包括数种基因:叶毛状体形态发生,花青素合成,叶衰老,生长素的生物合成,细胞壁代谢,和蜡/脂质生物合成(附加文件1表S12)被纳入这些区域。基因本体论(GO)分析表明,这些基因可能与草叶形态相关性状有关。(桌子3.出现在本段之后)。
讨论
圣奥古斯汀草的高密度基因图谱
养殖已作出努力,以提高草坪质量及其容限对生物和非生物胁迫。虽然常规育种方法在大多数草坪草育种计划中,分子育种等方法MAS正变得越来越受欢迎[17.,20.].MAS依赖于标记-性状关联,因此包含大量分子标记的高密度遗传图谱将最大化我们检测这些关联的能力。Kimball等之前生成的St. Augustinegrass连锁图谱仅包含160个SSR标记,标记间平均距离为8.2 cM [10.].在本研究中,我们构建了包含2871个SNP和81个SSR标记的9个连锁群的整合连锁图谱。图宽1241.7 cM,标记间平均距离0.4 cM1).该地图高度改善的标记密度,因此代表了迄今为止圣奥古斯丁草的最密度遗传图谱。此外,我们的地图还集成了两种类型的分子标记,SNP和SSR。虽然基于SNP的高密度连杆地图已成功用于草坪草中的比较基因组学分析和QTL映射[17.,21.[SSR标记通常提供高水平的多态性信息。然而,SSRS更多的劳动密集型,而SNP标记是高度丰富和高吞吐量。因此,高密度遗传图谱同时包含标记类型是有利的,并且可以是用于比较基因组学和QTL分析非常丰富。梨和小麦(如梨和小麦)的许多物种都报道了具有SNP和SSR标记的联系地图[22.,23.].
比较基因组学研究
草科可以说是农业中最重要的一科。由于基本染色体数目的变异和多倍体的高频率,它为植物进化研究提供了丰富的资源[24.,25.,26.,27.,28.,29.].可用于几种草种的基因组信息,如高粱,小麦,玉米和稻米,促进了草家庭成员的许多比较基因组学研究[24.,25.,26.].人们普遍认为,草的基因组是从一个共同祖先进化而来的,该共同祖先经历了一系列的全基因组复制、染色体融合和重排,产生了一个具有12条基本染色体的中间祖先,尽管对于这个共同祖先的基本染色体数目仍存在争议(X = 5 orX = 7) [27.,28.,29.].这种假设的12染色体中间祖先与现在的水稻有着非常相似的染色体排列(2N = 2X = 24). Most grass genomes are hypothesized to have formed from this intermediate ancestor through chromosome fusions, leading to reduction in chromosome numbers and additional rearrangements [29.].
胰腺炎具有主要的基础染色体数量X = 9 andX = 10 [6].这是以前假设嵌套染色体融合(NCF)是在禾本科减少染色体数目的主要机制[25.,26.].Panicoideae祖先基因组与X = 9 andX = 10 may have evolved from theX = 12 intermediate ancestor through three and two, respectively, NCFs. Comparative genomic analysis between sorghum (X = 10) and rice (X = 12) determined that sorghum chromosome Sb 1 originated from the insertion of the entire rice chromosome Os 10 to the centromeric region of Os 3, while Sb 2 was formed by insertion of Os 9 into Os 7 [25.].In addition, comparison between foxtail millet (x = 9) and rice found that foxtail millet chromosomes 2, 3 and 9 were collinear with rice chromosomes 7 and 9, 5 and 12, and 3 and 10 respectively, which indicates that another single NCF occurred in the evolution of the foxtail millet genome in addition to the two NCFs that happened in sorghum [30.].在我们的研究中,我们发现ST.AugustineGrass R(S)LG2,R(S)LG3和R(S)LG3分别与水稻CHR7和CHR9,CHR12和CHR5,CHR3和CHR10分别是直向性的。将米CHR9,CHR5和CHR10融合到CHR7,CHR12和CHR3的中间区域,以形成R(S)LG2,R(S)LG3和R(S)LG9(图。4).与以前的假设一致,我们的结果表明St. AugustineGrass基因组通过三个NCF从中间祖先演变。通过比较高粱和狐尾小米的基因组,张等人。发现NCF融合染色体8和9在FOXTAIL Millet中的3中的高粱[30.].在本研究中观察到类似的结果,其中发现ST.AugustineGrass R(S)LG3与高粱中的CHR8和CHR9都是直向性的(图。3.).这些结果表明,该NCF事件很可能发生在谷子和圣奥古斯汀草分化之前。在我们的结果中观察到的圣奥古斯汀草和谷子之间的高度共线性和共线性表明两个物种之间的进化关系非常密切(图。2a,b).然而,St. Augustinegrass LG3和LG7与谷子3和7染色体之间存在染色体间重排(图2)。2C,D)。这样的染色体重排事件可能引入的遗传变异,并促成了这些物种之间的分歧。
QTL鉴定草坪质量相关性状
在这项研究中所产生的高密度连锁图谱提供了在圣Augustinegrass的重要农艺性状相关性状QTL定位的平台。草皮草品质被定义为其中草皮符合一个标准时商定的程度。通过NTEP通过的草皮草品质的组分包括均匀性,枝条密度,叶质地,叶取向,光滑度和颜色(NTEP,2017)。在本研究中,叶片质地,草坪密度,遗传颜色和整体草坪质量选择评价圣Augustinegrass的美学性能。Kimball等人。检测到8个QTL分布在四个LG的用于使用基于SSR-连锁图谱[这些性状10.].在本研究中,成功确定了与这些特征相关的48个拟给QTL区域。这些QTL区域分布在九个LG中的七个(表3.).与以前的基于SSR的地图相比,高密度连杆地图显着改善了QTL映射的检测功率和分辨率。在这些QTL中,在LG3(35.66-40.15cm和47.03-54.93厘米)和LG8(38.08-50.27厘米)上观察到叶形纹理,草皮密度,遗传颜色和总体草皮质量重叠QTL的许多发生量。(38.08-50.27厘米)(表3.).协同定位于不同性状的QTL的可指示这些性状共同遗传机制,这表明这些区域的重要性为精细定位以及MAS [31.].QTL的重叠也可能表明这些区域含有控制叶和芽发育和形态的基因。通过爆破这些区域内的标记序列,几个与叶片形成和发育相关的同源基因,包括:叶片毛状体形态发生、花青素生物合成、叶片衰老、生长素生物合成、细胞壁代谢和蜡质/脂质生物合成(附加文件)1:表S12)被发现。例如,的同源基因pnh1.在拟南芥(PNH / ZLL)和米饭(ospnh1.)均报道了从那里树叶产生的形成茎尖分生组织(SAM)中发挥重要作用[32.,33.].DTX,据报道,其中编码解毒蛋白,也称为多药和有毒化合物挤出(配偶)转运蛋白通过养羊酸和ABA途径影响植物结构[34.,35.].据推测,所识别的QTL区域可能是通过在ST.AugustineGrass基因组上的这些基因的正陈来控制草皮质量性状。然而,需要实施进一步的实验以验证我们的结果并提高推定QTL的规模和质量,并鉴定控制草皮质量的功能基因。
进一步分析与草坪品质相关的QTL区域,有助于阐明这些复杂性状的遗传机制,并提高我们在育种周期中对其进行筛选的能力。高密度St. Augustinegrass遗传图谱是首个此类物种,有潜力帮助鉴定许多具有经济和农艺重要性的定性和定量性状的标记-性状关联,如草坪质量和对环境胁迫的耐耐性。这些关联可以随后用于MAS,从而提高圣奥古斯汀草育种的选择效率。
结论
总体而言,我们发现数千采用GBS的做法在圣Augustinegrass SNP标记和构建高密度遗传图谱,包括SNP和SSR标记。迄今为止,这是为此物种开发的最全面的遗传图谱。使用这种遗传图,我们在圣奥斯汀·米特和福克斯丘陵,高粱和稻米之间进行了比较基因组学分析,这揭示了草家族进化历史中发生的染色体重排事件。这些结果为未来的功能基因克隆和基因组组装提供了遗传和基因组。此外,鉴定了几种与不同相关的QTL有关的QTL,其分布在不同的联动组上。高密度遗传图和鉴定的QTL将增强草坪草改善计划。
方法
植物材料和DNA提取
一个pseudo-F2由115个杂交种群组成的人口来自圣奥古斯丁格拉斯品种“罗利”和“塞维利亚”(父母两家二倍体),2N = 2X = 18) following artificial hybridization methods [10.].来自北卡罗莱纳州立大学中心获得这类人群对草坪环境研究和教育。每个人在含Fafard盆栽混合物(康拉德Fafard公司,阿格瓦姆,MA)塑料容器无性繁殖,并保持在北卡罗莱纳州立大学,北卡罗来纳州,美国的温室。收集的基因组DNA提取与父母一起每辆混合动力的嫩叶。的DNA的质量首先通过琼脂糖凝胶电泳可视化并使用NanoPhotometer(Implen,München,德国)进一步测试。使用Hoefer说DQ 300荧光计(Hoefer说,霍利斯顿,美国)的DNA浓度进行定量。
GBS图书馆建设
根据Poland等人详述的方法制备测序文库。微小修改[36.].每个样本(115个杂种和2个亲本)约200 ng的基因组DNA被消化PSTI.和MSPI.(新英格兰生物实验室公司;(Ipswich, MA)在37°C、20 μL体积下酶切2 h。65℃孵育20 min,终止反应。条形码适配器(包含独特的条形码序列,详情见附加文件1:表S1)和一种Common-Y adapter were ligated to digested genomic DNA fragments at 22 °C overnight in 40 μL volume and stopped at 65 °C for 20 min. Then, 10 μL of each sample was pooled and cleaned up using QIAquick PCR Purification Kit (QIAGEN, Hilden, Germany). After that, purified DNA was amplified using NEB MasterMix (New England BioLabs, Inc.; Ipswich, MA). PCR products were purified and size-selected using GeneRead Size Selection Kit (QIAGEN, Hilden, Germany) to remove adapter dimers and small fragments (< 150 bp). The library was size-selected at a range of 250–400 bp using D1000 ScreenTape assay (Agilent, Waldbronn, Germany) and sequenced on Illumina HiSeq 2500 (Illumina, San Diego, United States).
SNP鉴定和基因分型
在本研究中,非参考UNEAK管道被用于进行SNP发现和基因分型[15.,37.].GBS原始读取被处理以保持包含条形码和限制站点的读取。将高质量的读数修剪到64bp,并且相同的读数折叠成标签。成对对准标识标签对具有单个碱基对不匹配,其可以被认为是候选SNP。采用网络滤波器(错误公差率= 0.03)来丢弃重复,副歌曲和错误标签。然后可以将其余的倒数标签对被识别为SNP。最后,通过测序深度(≥10),次要等位基因频率(≤0.05)和呼叫率(≥90%)来过滤SNP,以获得高质量的SNP。
连锁图施工
JoinMap 4.0 [19.用来构建联动地图。SNP markers were assigned to three categories according to segregation type: heterozygous in parent ‘Raleigh’ and homozygous in parent ‘Seville’ (‘lm x ll’ type), homozygous in parent ‘Raleigh’ and heterozygous in parent ‘Seville’ (‘nn x np’ type), heterozygous in both parents (‘hk x hk’ type). Markers that showed abnormal segregation ratios (chi-squared test, df = 2, cut off value = 9.21,P. < 0.01) were excluded from map construction. The ‘lm x ll’ type and ‘nn x np’ type SNP markers were used to construct separate parental linkage maps for parent ‘Raleigh’ and ‘Seville’, respectively. Meanwhile, ‘hk x hk’ type SNP markers along with previously identified SSR markers from Kimball et al. [10.,将亲本连锁群整合成一致图谱。所有的连锁图谱均采用回归映射算法构建,最小LOD为9.0,最大重组率为0.4(拟合好度跳跃值3.0,纹波值1)。图谱距离采用Kosambi映射函数计算。地图质量由' N.N. '检查fit '函数在JoinMap 4.0。MapChart 2.32用于可视化联动图[38.].
比较基因组学分析
映射SNP标签的序列进行比对模型草种的基因组序列:小米(Setaria italica)、高粱(高粱二色的)和米饭(奥雅萨苜蓿)using the blastn program in BLAST+ 2.6.0 [39.]带有1×10的电子值截止值- 5.参考基因组Setaria斜体版本,高粱二色的ncbiv3,奥雅萨苜蓿粳稻构建4.0是从NCBI基因组数据库下载。该显示命中参照基因组的标记序列被用于进一步的比较分析。使用R中包GGPLOT2和Circos情节点图在Circos包比较的结果显现[40,41.].
草坪质量相关性状的QTL定位
草坪质量相关性状由金博尔等评估。被用于QTL作图[10.].将所有杂交种与父母线一起种植在随机完成的块设计(RCBD)中,两个地点(Raleigh和Laurel Springs,NC,美国)进行了三次重复,并评估了两年(2013年和2015年)。Turf quality-related traits, including overall turf quality, leaf texture, genetic color, and turf density were evaluated visually on a 1 to 9 scale according to the National Turfgrass Evaluation Program’s (NTEP) guidelines as follows: turf quality, 1 = poor quality and 9 = excellent quality; leaf texture, 1 = coarsest texture and 9 = finest texture; genetic color, 1 = light green/yellow and 9 = dark green; turf density, 1 = sparsest density and 9 = densest turf. Each year by each location combination was considered as a separate environment. An analysis of variance (ANOVA) and least square (LS) means were generated using the GLM procedure in SAS statistical software version 9.4 (SAS Inst. Inc., 2017) for each trait. QTL analysis was performed using LS mean values both for individual environments and across environments against the integrated linkage map using MapQTL 6.0 [42.].采用区间作图法(IM)和多重QTL方法(MQM),利用回归分析方法检测标记与表型性状之间的显著相关性。LOD阈值(P. < 0.05) for genome-wide were determined for each trait using a permutation test with 10,000 iterations. Regions with a LOD score above threshold values were considered as potential QTL intervals. Allelic effects were estimated as AF = [(μ交流 + μ广告) - (μ公元前 + μ双相障碍)]/4为雌性(Raleigh)可加性;一个m = [(μ交流 + μ公元前) - (μ广告 + μ双相障碍)] / 4对于雄性(塞维利亚)添加性和D = [(μ交流 + μ双相障碍) - (μ广告 + μ公元前)] / 4主导权其中μ交流,μ.广告,μ.公元前和μ.双相障碍估计了与ac, bc, AD和bd 4种可能基因型各相关的表型平均数,由此衍生出ab × CD杂交[43.].此外,利用blastn/blastp工具在NCBI NR数据库中搜索感兴趣区域内的标记序列,以获得它们的同源性。利用UniProt数据库进行基因本体(GO)注释,预测QTL区域的基因功能。
缩写
- CHR:
-
染色体
- 厘米:
-
Centimorgan.
- GBS:
-
通过测序进行基因分型
- 去:
-
基因本体论
- GWAS:
-
全基因组关联研究
- 我是:
-
区间映射
- LG:
-
连锁群
- LOD:
-
赔率的对数
- MAS:
-
标记辅助选择
- MQM:
-
多个QTL的方法
- NTEP:
-
国家草坪评估计划
- QTL:
-
定量特质基因座
- SNP:
-
单核苷酸多态性
- SSR:
-
简单序列重复
参考文献
- 1。
绍尔JD。修订StenotaPraprum.(禾本科:Paniceae),并注意其历史地理位置。Brittonia。1972;24:202-22。
- 2。
Busey P,Davis EH。草坪草丛环境。Proc Fla状态座位SoC。1991; 104:353-8。
- 3.
米拉 - 刘易斯SR,苏莱塔MC,凡Esbroeck GA,Quesenberry KH,肯沃西KE。在遗传多样性的细胞学和分子鉴定StenotaPraprum..农作物科学。2013; 53(1):296-308。
- 4.
凯洛格ea。开花植物。单古氏体:Poaceae(第13卷)。纽约:斯普林克;2015年。
- 5。
Parrish DJ,Fike Jh。生物燃料切换的生物学与农业。CRIT Rev Plant Sci。2005; 24(5-6):423-59。
- 6.
作者简介:Giussani LM, Cota-Sánchez JH, Zuloaga FO, Kellogg EA. C4光合作用的多重起源显示了禾本科Panicoideae亚科(禾本科)的分子系统发育。植物学报。2001;88(11):1993-2012。
- 7.
龙Ja,Bashaw Ec。圣奥古斯丁斯孢子发生和染色体数字。农作物科学。1961; 1:41-3。
- 8.
Mulkey SE。圣奥古斯汀草抗灰叶斑病基因的SSR开发与定位(硕士论文,北卡罗来纳州立大学,罗利)2012.从检索http://www.lib.ncsu.edu/resolver/1840.16/8590/.
- 9。
Mulkey SE,苏莱塔MC,奇宝JE,沙夫JE。米拉 - 刘易斯SRDevelopment和简单重复序列标记为St. Augustinegrass作物科学表征。2014; 54:401-12。
- 10。
金博尔JA,TUONG TD,阿雷利亚诺C,利文斯顿DP III,米拉 - 路易斯SR。连锁分析和冻结宽容和圣Augustinegrass草坪质量性状相关的数量性状位点的识别。mol繁殖。2018; 38:67。
- 11.
陈志强,陈志强,陈志强,等。利用Solexa测序技术对植物叶绿体基因组进行多重测序。核酸学报2008;36:e122。
- 12.
黄宇烽,波兰JA,怀特CP,杰克逊EW,补锅匠NA。使用于种植燕麦基因组基因分型发现,通过测序(GBS)。Plos一个。2014; 9:e102448。
- 13。
Elshire RJ,Glaubitz JC,Sun Q,Poland Ja,Kawamoto K,Buckler Es,等。一种高分辨率物种的鲁棒,简单的基因分序列(GBS)方法。Plos一个。2011; 6:e19379。
- 14。
Beissinger TM, Hirsch CN, Sekhon RS, Foerster JM, Johnson JM, Muttoni G,等。利用基因分型法测定基因分型人群的标记密度和阅读深度。遗传学。2013;193:1073 - 81。
- 15.
路楼Lipka的AE,GlaubitzĴ,Elshire R,查尼JH,Casler MD,圆盾ES,Costich DE。柳枝稷的基因组多样性,倍性,并且演化:从基于网络的SNP发现协议新的见解。Plos Genet。2013; 9(1):e1003215。
- 16。
SONAH H,巴斯蒂安男,伊基拉E,Tardivel A,Legare G,博伊尔B,等人。通过测序(GBS)的方法提供一种改进的基因分型增加的多功能性和SNP发现和基因分型的效率。Plos一个。2013; 8:e54603。
- 17.
霍洛威HM,于晓伟,邓恩JC, Schwartz BM, Patton AJ, Arellano C,等。结缕草的SNP高密度连锁图谱(结缕草Steud。)及其用于鉴定与冬季耐核性相关的QTL。mol繁殖。2018; 38:10。
- 18.
Rouppe Van der Voort J,Van Eck H,Van Zandvoort P,Overmars H,Helder J,Bakker J.通过兄弟姐妹的基因分型进行基因分型:使用“伪F2”测绘策略构建的土豆囊肿线性的遗传图谱。Mol Gen Genet。1999年; 261:1021-31。
- 19。
范Ooijen JW。JoinMap 4,用于计算实验群体遗传连锁图的软件。瓦赫宁根:Kyazma BV;2006.
- 20.
Bonos SA,克拉克BB,迈耶WA。育种的主要冷季型草坪草的抗病能力。Annu Rev phytopathol。2006; 44:213-34。
- 21.
王楼,辛格R,杰诺韦西AD,围CM,黄X,钱德拉A,于Q.序列标记的高密度的遗传的图结缕草提供了对绿甲亚科基因组进化的深入了解。植物j . 2015; 82(5): 744 - 57。
- 22。
吴俊,李立涛,李敏,马可汗,李晓刚,陈辉,等。利用SNP和SSR标记构建梨果实相关qtl的高密度遗传图谱。J Exp Bot. 2014; 65:5771-81。
- 23。
吴Qh,陈yx,周舍,傅立l,陈俊,小伊等。小密度遗传联系地图建设和QTL晶粒粒度曲线绘制yanda1817×beinong6。Plos一个。2015; 10:E0118144。
- 24。
Srinivasachary DMM,Gale Md,Devos Km。比较分析揭示了手指小米和水稻基因组之间的高水平保守的水分性。Al Appl Genet。2007; 115(4):489-99。
- 25。
罗MC,新政KR,Akhunov ED,Akhunova AR,安德森OD,安德森JA等。基因组的比较揭示染色体数目减少的草的主导机制,加快了小麦基因组进化。P Natl Acad Sci USA。2009; 106:15780-5。
- 26。
王旭,金东,王震,郭华,张磊,王磊,李俊,Paterson AH。在真核生物的进化过程中,以端粒为中心的基因组重模式决定了染色体数目的重复减少。新植醇。2015;205:378 - 89。
- 27。
SalseĴ,Bolot S,Throude男,Jouffe V,Piegu B,古莱氏UM,Calcagno T,库克R,Delseny男,Feuillet C.鉴定与水稻和小麦之间共享重复的表征提供新的见解草基因组进化。植物细胞。2008; 20:11-24。
- 28。
麦类全基因组复制在水稻分化之前的证据与进化分析。BMC Evol Biol. 2009; 9:209-26。
- 29.
狄维士公里。草基因组的组织与进化。植物学报。2010;13:139-45。
- 30.
张刚,刘旭,全志,程胜,徐旭,潘胜,等。谷子基因组序列(Setaria italica)提供了深入了解基层进化和生物燃料的潜力。NAT BIOTECHNOL。2012; 30:549-54。
- 31。
Cavagnaro PF, Iorizzo M, Yildiz M, Senalik D, Parsons J, Ellison S, Simon PW。基于snp的胡萝卜高分辨率连锁图谱,包括调控根和叶花青素色素沉着的QTL定位。BMC基因组学。2014;16(15):1118。
- 32.
Lynn K, Fernandez A, Aida M, Sedbrook J, Tasaka M, Masson P, et al.;的PINHEAD / ZWILLE.基因在拟南芥发育过程中起多效性作用,与拟南芥基因具有重叠功能ARGONAUTE1基因。发展。1999; 126(3):469-81。
- 33.
西村A,伊藤男,神谷N,佐藤Y,松冈M.ospnh1.调节水稻叶片的发育和茎尖分生组织的维持。植物j . 2002; 30(2): 189 - 201。
- 34。
李锐,李军,李胜,秦刚,Novák O, Pěnčík A,等。ADP1通过调节植物生长素的生物合成来影响植物的构型。公共科学图书馆麝猫。2014;10 (1):e1003954。
- 35。
张H,朱H,潘Y,俞Y,栾S,李L.一个DTX / MATE型转运方便脱落酸流出和调制ABA的敏感性和耐旱拟南芥.摩尔。2014;7(10):1522 - 32。
- 36。
波兰J,Brown PJ,Sorrells Me,Jannink J.通过测序方法使用新型双酶基因分型的大米和小麦的高多数遗传地图的开发。Plos一个。2012; 7(2):E32253。
- 37。
Glaubitz JC,Casstevens TM,吕男,哈里曼Ĵ,Elshire RJ,孙Q,圆盾ES。流苏GBS:高容量的测序分析流水线基因分型。Plos一个。2014; 9:e90346。
- 38。
vooorres重新。MapChart:用于链接地图和QTL的图形演示软件。j hered。2002; 93(1):77-8。
- 39。
Camacho C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K, Madden TL. BLAST+:建筑与应用。BMC生物信息学。2009;10:421。
- 40。
Wickham H. ggplot2:用于数据分析的优雅图形。纽约:斯普林格出版社;2016.ISBN 978-3-319-24277-4。http://ggplot2.org/.
- 41。
Krzywinski男,沙因Ĵ,比罗尔I,康纳斯Ĵ,加斯科因R,霍斯曼d,琼斯SJ,马拉MA。电讯:对比较基因组学的信息审美。Genome Res。2009; 19:1639至1645年。
- 42。
范Ooijen JW。MapQTL 6:用于绘制二倍体物种实验居群数量性状位点的软件。瓦赫宁根:Kyazma BV;2009.
- 43。
Ben Sadok I,Celton JM,Essalouh L,Zine El Aabidine A,Garcia G,Martinez S等人。橄榄花和果实性状的QTL映射。Plos一个。2013; 8:E62831。
致谢
作者要感谢NC州立大学基因组科学实验室(Ralaleigh,NC,USA)为下一代测序服务和Jared Smith以及USDA-ARS东部区域小谷物基因分型实验室(ERSGL)(Raleigh,NC,USA)寻求GBS图书馆建设的帮助。
资金
这项研究得到了北卡罗来纳作物改良协会、北卡罗来纳州立大学草坪草环境研究和教育中心以及美国农业部国家食品和农业研究所特种作物研究计划[2015-51181-24291]提供的部分资金支持。作者声明,没有任何资助机构在研究的设计、收集、分析和数据解释以及手稿的写作中有任何作用。
可用性数据和材料
在SRA ID SRP149564下,所有原始测序数据都存入国家生物技术信息中心(NCBI)中的序列读取档案(SRA)数据库。在合理的请求时,当前研究中使用的其他数据集可从相应的作者获得。
作者信息
从属关系
贡献
XY负责实验的设计和执行,数据分析和手稿撰写。JK进行了表型评估。SML构思了项目,维护了人口,对建议的工作提供了反馈,并在提交之前修改了手稿。所有的作者阅读,编辑,并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
额外的文件1:
表S1到S12.表S1。在圣Augustinegrass GBS图书馆条形码和适配器的顺序。表S2。圣奥古斯汀草亲本和杂交种的测序阅读数。表S3。的“罗利”圣Augustinegrass遗传图谱连锁群和标记序列的细节。表S4。“塞维利亚”圣奥古斯汀草遗传图谱的连锁群和标记序列的细节。表S5。联动小组和标记分离细节在集成的圣奥古斯丁遗传地图中。表S6。“罗利”圣Augustinegrass连锁群之间的比较基因组学和谷子基因组。表S7。“塞维利亚”圣奥古斯汀草连锁组和谷子基因组的基因组学比较。表S8。“罗利”圣奥古斯丁胶层锁定群体与高粱基因组之间的基因组学比较。表S9。“塞维利亚圣奥古斯丁联动群体与高粱基因组之间的基因组学比较。表S10。“罗利”圣奥古斯汀纺血基与水稻基因组之间的基因组学比较。表S11。“塞维利亚”圣Augustinegrass连锁群,水稻基因组之间基因组的比较。表S12。QTL区域中序列序列的基因本体论分析。(XLSX 346 KB)
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
玉,X.,金博尔,J.A.&米拉刘易斯,S.R.圣Augustinegrass的高密度遗传图谱和应用,为草坪质量性状的比较基因组分析和QTL定位。BMC植物BIOL.18,346(2018)。https://doi.org/10.1186/s12870-018-1554-4
收到了:
接受:
发表:
关键词
- 圣奥古斯丁
- 联系地图
- 比较基因组分析
- QTL.