- 研究文章
- 开放访问
- 发表:
细胞细胞基因组的测序Gymnomitrion concinnatum(Jungermanniales)揭示了进化稳定的苔类有丝分裂基因组在结构和基因顺序上的第一个例外
BMC植物生物学体积18., 文章编号:321.(2018)
抽象的
背景
叶绿体和线粒体基因组的比较分析表明,苔藓植物的细胞器基因组进化缓慢。然而,与种子植物相比,苔藓植物的细胞器基因组研究尚不充分,特别是在苔类植物中。通过测序发现了苔藓植物的另一个细胞器基因组,为苔藓植物的进化提供了新的结论。
结果
在这项工作中,细胞器基因组Gymnomitrion concinnatum第一次测序,组装和注释肝脏。叶绿体基因组显示器,典型的大多数植物,含有大单拷贝区域(81,701bp)的四脚石结构,两个倒置重复区域(每个8704bp)和小单拷贝区域(20,179bp)。叶绿体的基因阶数和含量与观察到的较小差异的其他肝脏非常相似。在叶绿体和线粒体基因中预测了739和222个RNA编辑位点的总数g . concinnatum.线粒体基因组基因含量也符合Liverworts,但除了几个改变之外,例如:内含子损失COX1.和atp1基因。尽管如此,分析显示g . concinnatum与Liverworts的其他毒蛛相比,重新排列了促滤蛋白结构和基因令。研究了这种含有含有丝链质重排的原因,并提出了重组模型。
结论
这项研究提供的线粒体和叶绿体基因组结构和基因顺序多样性的概述Gymnomitrion concinnatum针对肝脏的已知细胞甲基因组的背景。所获得的结果对早期研究的促进型植物促进植物的构造受到疑虑的观点,令人疑问。事实上,在肝曲线的进化稳定,促进的态度素内的第一种重组的案例。
背景
在进化研究中,细胞器基因组被广泛用作遗传信息的来源,这主要是由于其单倍体特性以及每个细胞中存在成百上千个拷贝[1那2].在大多数已知的生物体中,毒蛛和塑料是母体遗传的,导致在生物体中仅产生单个基因组的单倍型。几项研究描述了塑性基因组的异质性[3.然而,大多数研究没有揭示内在的特异性多态性[4.那5.支持细胞内基因组作为进化研究的资源。
线粒体全基因组序列主要用于动物和真菌的系统发育、系统地理学和种群遗传学[6.那7.那8.[虽然在植物中,水资源基因组主要用于这些目的。
与种子植物相比,苔藓植物的细胞器基因组尤其是质体基因组的研究较少。迄今为止,已知的苔藓植物属的质体全基因组序列只有15个,线粒体全基因组序列只有48个。此外,大多数测序的基因组只属于四个苔藓科真菌[9.],grimmiaceae [10.那11.],Orthotrichaceae [12.那13.那14.那15.]和sphagnaceae [16.].
与种子植物相比,早期陆生植物的线粒体基因组结构稳定[12.那17.].苔类植物是孢子体植物中最古老的进化谱系,也是遗传多样性最高的物种。然而,尽管在基因间和基因内水平上有高的核苷酸变异,基因的含量和顺序几乎保持不变,因为肝苔多样化的最深处节点[18.那19.那20.].唯一观察到的变化是内含子损失ATP1和COX.叶状肝曲线组中的1个基因[20.和nad7基因的假殖化treubia lacunosa.[18.].
这种稳定性似乎与早期陆地植物的有丝分裂基因组中缺乏重复序列有关,这在种子植物中很常见[12.].然而,即使与苔藓相比,肝脏的毒蛛探索差,其中已知6属的最新完全线粒体基因组序列。
现有数据是即使在叶绿体基因组中的情况下更加稀少,限定于属地钱那佩利亚那Aneura和Ptilidium..
肝徽Gymnomitrion concinnatum(Lightf。)Corda属于Gymnomitriveae H. Klinggr的家庭。该组包括十属(Acrolophozia那apomarsupella.那Gendnomitrion那Herzogobryum.那Marsupella那Nanomarsupella.那Nothogymnomitrion那Paramomitrion那Poeltia, 和Prasanthus.),其中最多的是Gendnomitrion(27种)和Marsupella(26种)[21.].从历史上看,只有两个广泛的属(Gendnomitrion和Marsupella)被认为是Gymnomitriveae的一部分。基于属属的周围Gendnomitrionvářa等人提出。[21.],有七种记录在波兰和斯洛伐克。他们大多生长在塔特拉山(西喀尔巴阡山脉)。Gymnomitrion concinnatum是酸痛,脱毛管和Epigeic Liverwort,在岩浆(花岗岩)和变质(大多是神社)岩石和晶体板岩上生长。最常见的是,它发生在岩壁的架子和缝隙上,较少往往在松散的高山(和街道)草原和雪地床上,具有苔藓植物的优势[22.].从中欧,植物植物从植物植物到高发生g . concinnatum被描述为GymnomiTietum Concinnati.赫尔佐格1943年前女友腓立比1956(类:Grimmietea Alpestris.哈迪奇等Vondráček.在杰伊克等Vondráček1962)(比较[22.那23.那24.那25.])。Gymnomiraceae的已知遗传资源仅限于其其三个叶绿体基因座的序列[26.那27.]属于Gendnomitrion那Herzogobryum.那Marsupella和Prasanthus..
在本研究中,我们测序,组装,注释和分析的细胞细胞基因组Gymnomitrion concinnatum这为研究苔类植物有丝染色体组和质体的进化提供了新的思路。
成绩与讨论
叶绿体基因组的特征Gymnomitrion concinnatum
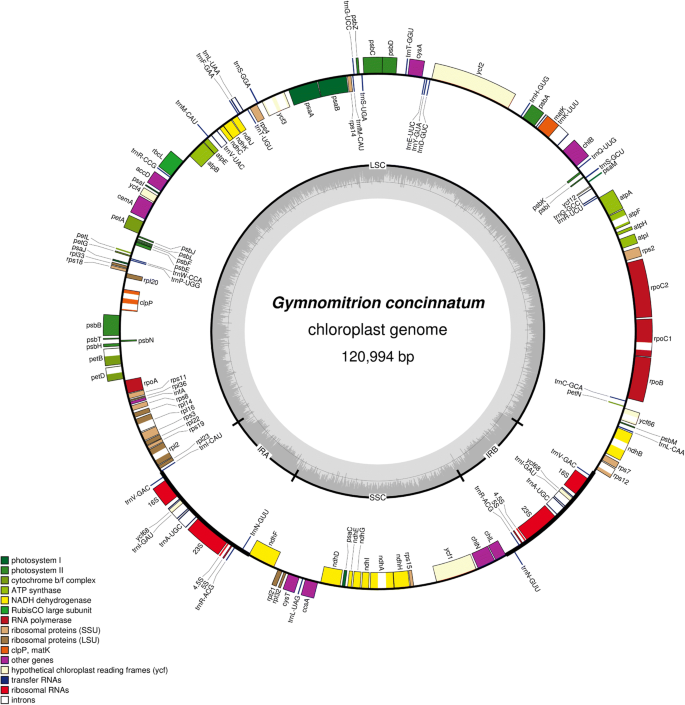
的质体Gymnomitrion concinnatumis 120,994 bp long with a structure typical for most plants, including a pair of IR regions (each of 8704 bp) separated by LSC (81,701 bp long) and SSC (20,179 bp) regions (Fig.1).塑料近2000年,比第二最长已知的叶状植物鳞状基因组Ptilidium pulcherrimum然而,长度似乎是在属级别的变化。六叶片叶绿体基因组的比较分析Aneura pinguis.隐秘物种显示叶绿体基因组的长度范围为120.698至121,140bp [19.].第一测序苔质体的地钱paleacea[28.稍后将相同物种的稍后测序长度为390 bp。塑料的长度的变化可以具有进化的意义,但是具有Liverworts血液序列的可用性有限,因此得知叶状Liverworts谱系中的塑料大小的可能变化。
GC含量的含量g . concinnatum塑料量为34.5%,落在其他已知的肝曲线范围内,GC含量范围为26.6%Marchantia polymorpha.到40.6%Aneura mirabilis.[29.],但比在较高Ptilidium pulcherrimum(33.2%),排序完全塑性基因组的第一个叶状肝曲[30.].
就像已知的最接近的有质体序列的亲戚,p . pulcherrimum,塑料Gendnomitrion由121个独特的基因组成,包括81个蛋白质编码基因,6个未知功能的6个基因(ycf.基因),4个核糖体RNA和30转移RNA(附加文件1:表S2)。基因令和内容似乎在叶子中稳定(Gymnomitrion concinnatum那Ptilidium pulcherrimum)和简单的脂肪肝曲线(Aneura pinguis.那薄层终极玉米糖尿病).复杂叶状体苔类(Marchantia Paleacea,M.多晶型)有两个基因:半胱氨酸一个和半胱氨酸T. offerotcophicAneura mirabilis.具有降低的原质体系由于丢失或pseudogenization参与光合作用的过程的基因的[29.].
通过对两种已知叶苔质体的比较分析,除发现了微小的差异外,还发现了基因大小和功能上的相似性。的质体Gymnomitrion concinnatum1987年的bp比吗Ptilidium pulcherrimum主要是由于超过500bp的插入TRN.H-GUC和ycf.2基因。第二个插入位于所提及的内部ycf.2基因,这是598英镑的时间更长Gendnomitrion而不是Ptilidium.(5828 bp vs 5242 bp)。
特点Gymnomitrion concinnatum线粒体基因组
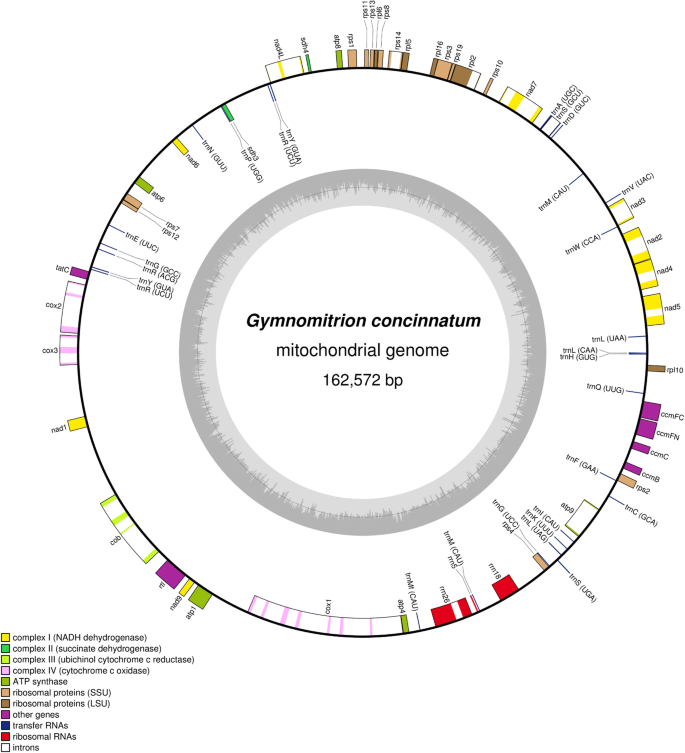
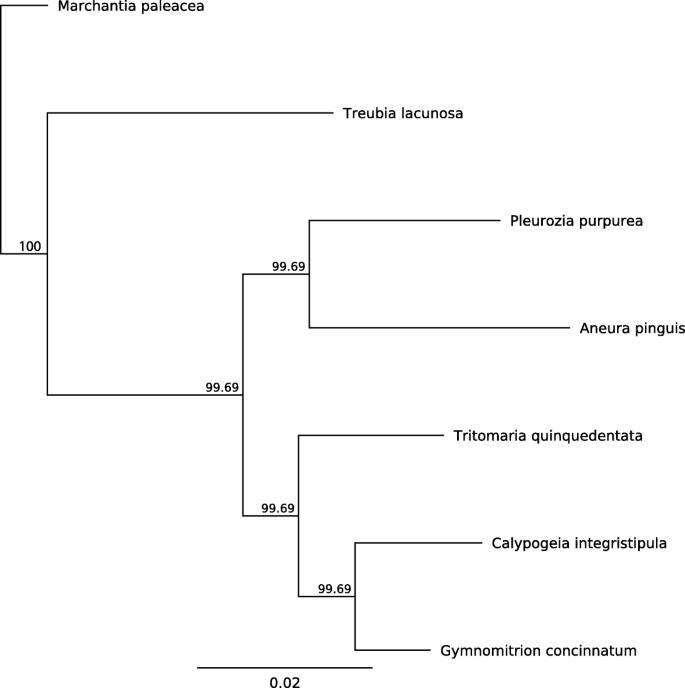
的线粒体全基因组g . concinnatum长度为162,574 bp(图。2),符合其他报告的Markantiophyta毒蛛,即,即Aneura pinguis.[19.],四Calypogeia物种(20.],地钱paleacea[31.],Pleurozia菊[17.],treubia lacunosa.[18.],Tritomaria Quinquedentata.[20.].上述线粒体基因组长度为142,510 (t . quinquedentata)至186,609 bp (M.Paleacea.).线粒体DNA的总体GC含量是44.7%,这类似于其他已知苔类植物线粒体基因组(42.2-47.4%)。的线粒体基因g . concinnatum含有70个独特的基因,包括42个蛋白质编码基因,3个核糖体RNA和25转移RNA(附加文件1:表S2),这是一种典型的线粒体蛋白质编码基因,参与呼吸和蛋白质合成。基于38个蛋白质编码序列构建的7个肝曲面的38个蛋白质编码序列的系统发育树符合Markantioophyta的思考[32.] (图。3.).
这nad7鉴定蛋白质编码序列g . concinnatum促滤蛋白酶作为假基因。的缺席河畔鉴于此前对苔类植物的分析表明,CDS并不奇怪河畔7发生在Marchantiopsida和Jungermanniopsida的伪基因[17.那19.那20.那31.那33.].唯一保留功能的苔类植物nad7属于Haplomitriopsida分支:treubia lacunosa.和Haplomitrium mnioides.[18.那33.].有趣的是,第一个外显子的ORFG. Concinnatum Nad.序列同源性分别为84.8和82.8%t . lacunosa和h . mnioides, 分别。没有其他已知的肝脏线粒体基因组保存在第一个外显子的ORFnad7基因[17.那19.那20.那31.那33.].
观察到的其他基因结构改变g . concinnatum有丝分裂基因组内含子丢失COX.1和ATP1基因。G. concinnatum COX1含有7个内含子,而其他的苔类植物,早前报道[17.那18.那19.那31.],包含9个内含子(附加文件2:图S1)。COX.最近在另一个Jungermanniales中报告了1个内含子损失 -Tritomaria Quinquedentata.和Calypogeia物种(分别为4和3内含子)[20.].所有这些观察表明内含子丢失COX.1是特定于Jungermanniales种类的特征。此外,这是ATP1基因失去了两个内含子g . concinnatum线粒体基因组,无内含子。这种现象在以前的研究中也有报道Calypogeia物种,以及Tritomaria Quinquedentata.和treubia lacunosa.物种(18.那20.].
叶绿体基因RNA编辑位点的预测
RNA编辑是改变RNA序列中核苷酸的身份或加入或删除核苷酸的过程,使得成熟的RNA序列与基因组中定义的不同[34.那35.那36.].为了分析蛋白质编码序列中可能的RNA转录后修饰,我们使用PREPACT 2.0对87个质体CDSs进行了研究[37.].预计在叶绿体基因中预测了739个RNA编辑位点的总数g . concinnatum.C - U取代位点占总RNA编辑位点的60.1%(444个),U - C取代位点占总RNA编辑位点的39.9%(295个)。三种替换影响两种cds的ORF:CCSA和佩德,其中谷氨酰胺密码子被改变为终止密码子,以及atpF,其中苏氨酸密码子变更为甲硫氨酸密码导致在该基因的ORF的恢复。在替代atpF似乎对提供蛋白质产品至关重要。考虑到上述取代,两种基因的CDS与其他肝脏均一致。RNA编辑位点含量最高的是在佩尔基因(4.2%的CD核苷酸被改变),但是基因的编码序列仅为96bp长度。值得一提的是,在光照系统II的8个CDS的8个CDS中没有找到RNA编辑网站(附加文件3.:表S3)。
线粒体基因RNA编辑位点预测
在42个cds中预测了222个RNA编辑位点g . concinnatummitogenome。C - U取代位点占总RNA编辑位点的76.6%(170个),U - C取代位点占总RNA编辑位点的24.4%(52个)。质粒cds(739个替换,71,379 bp)所包含的RNA编辑位点是有丝分裂基因组cds(222个替换,33,534 bp)的1.6倍。有丝分裂基因组中发现的两个苏氨酸密码子与蛋氨酸密码子的替换导致起始密码子的出现(nad2和nad6基因)和一种取代改变了谷氨酰胺密码子来阻止密码子(atp9基因)。尽管进行了上述三种替换,但预测的cds翻译产物与相应的其他苔类cds是一致的。事实上,由于RNA的编辑修改,适当的ORFnad2和nad6基因是恢复。RNA编辑位点含量最高的是在CCMFC基因序列(2.3%的CD核苷酸被改变)以及平均RNA编辑位点含量在细胞色素C成熟编码基因中最高(1.5%)。另一方面,六个CDS中没有发现替代品:COX2.那nad1那rpl6.那rpl16那rps4那rps13和rps19(附加文件4.:表S4)。
基因令和重复序列
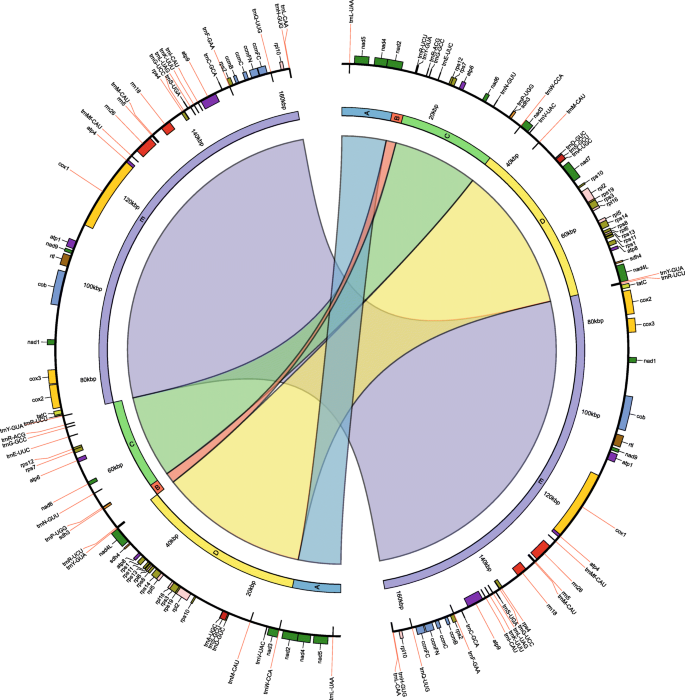
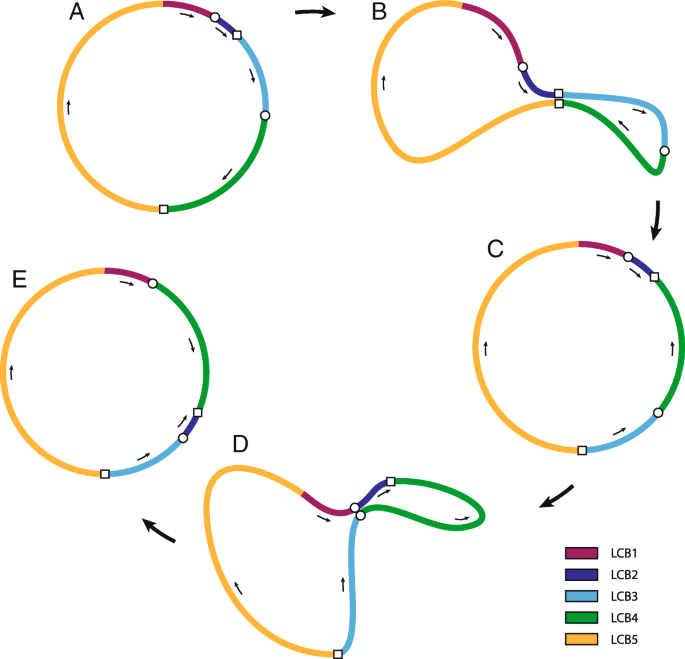
就线粒体基因顺序进化而言,苔类被认为是保守的[12.那17.那18.].所有已公布的完整的苔类线粒体基因组序列表明,其基因顺序在苔类中保留了下来。这些分析包括6种苔类植物,分别来自Treubiales, Marchantiales, Pleuroziales, Metzgeriales和Jungermanniales,跨越Marchantiophyta [17.那18.那19.那20.那31.].所以Gymnomitrion concinnatum预计将保持相同的线粒体基因令。为了验证结构的同源性g . concinnatum线粒体基因与苔类植物的已知物种,的线粒体基因序列g . concinnatum和c . integristipula使用紫红质软件对齐[38.[然后使用电机的基因组图相比可视化[39.].分析显示g . concinnatum与其他Markantiophyta诱导物相比,促滤皮组结构被重新排列。和。。比较C. IntegrIsipula.,在这两个线粒体基因组中鉴定了五个局部共线嵌段(LCB)(图。4.).第一和最后一个LCB(A和E)位于两种诱导物的同一区域内。然而中间三个LCBg . concinnatum(B,C和D)以不同的顺序排列。这三个LCB中的两个相反c . integristipulamitogenome。所有的lcb都在基因间间隔内开始和结束。有丝分裂基因组的结构符合mapping to reference和de novo组装方法,LCB连接处的覆盖率较高。另外,上述有丝分裂基因组的结构通过PCR方法独立确认(附加文件)5.:图S2)。上述观察结果对以往研究认为的苔类甚至整个苔藓植物的有丝分裂基因组结构高度保守的观点提出了质疑[12.那17.那18.].
种子植物的线粒体基因组结构的重排通常与反复序列的发生相连[40那41.那42.].在较高植物诱导型毒蛛中观察到不同尺寸的重复:大(> 500bp),中间体(50-500bp)和小(<50bp)。大型和中间重复被认为参与线粒体基因组结构的重组[41.那43.].这种重复的序列可以配对,重组和形成线粒体基因的不同的配置,结果[17.].考虑到上述情况以及前面尚未报道Marchantiophyta和Bryophyta之间的促使结构重排,在本研究中进行了进一步研究序列重复和重组的进一步研究。
因此,在样本中发现了超过100 bp的重复序列g . concinnatum促催化序列。该分析分析了十对序列重复,从107bp到566bp长度不同,总体占线粒体基因组序列的3.1%。结果证明,在上述LCBS之间的交叉点上,两个最长的重复,566bp(97.7%序列同一性)和435bp长度(95.9%序列同一性)位于上述LCB之间的连接。因此,第一对(R1)的两次重复序列位于B-C和D-E LCB的边缘上,而第二对(R2)的两个重复序列位于A-B和D-C LCB的边缘上(图。4.).
考虑不寻常的配置g . concinnatum提出了重复序列的促催化和位置,提出了重组模型(图。5.).促丝杆菌组中的两个重排已经发生在另一个之后。First, starting with LCBs arranged in order common for liverworts i.e. A, B, C, D, E, the rearrangement within R1 repeats has taken place resulting with following order of LCBs: A, B, D (inverted), C (inverted) and E. Next, the second rearrangement within R2 repeats has occurred resulting with following order of LCBs: A, D, B (inverted), C (inverted) and E, which is mitochondrial genome configuration identified ing . concinnatum(图。4.).
为了研究在同一划分的其他物种的线粒体基因组内发生相同的重复序列模式,进行了上述肝曲线的6种完全促进的促进肝序列中的序列重复的分析。在每个线粒体基因组序列中,除了A. Pinguis,发现了超过400bp和90%的身份的两对重复序列。考虑到诱导毒蛛的重复序列的位置,区分了三对不同的对(图。6.).6个物种中有4个具有相同的重复序列模式:R2和R3重复序列,其中R2重复序列位于其中nad2-rps12和NAD4L-TATCr3重复的代际区域位于内部nad5-nad4代际间隔和第二内含子结实的矮.有趣的是,M.Paleacea.促滤皮组仅含有上述重复序列对 - R3,而且还含有R1 - 鉴定g . concinnatum线粒体序列。似乎线粒体基因组g . concinnatum在共享一对(R2)来识别c . integristipula那p .紫竹那t . quinquedentata和t . lacunosa和另一对(R1)识别M.Paleacea..然而,进一步的分析表明,在g . concinnatum在该基因组中没有完全缺失促催化剂。R3重复序列对的第二次交配位于第二内含子内结实的矮在缺失超过600 bp的情况下仍然存在基因nad5-nad4基因间间隔导致R3对的第一配偶消失。
考虑到重复序列的独特模式g . concinnatum促使,似乎合理地称为重复序列的模式可能是对维持基因阶的至关重要,并且特定模式的变化可能导致线粒体基因组的重组,因此基因令重新排列。到目前为止,仅在种子植物中鉴定了由同源重组引起的线粒体基因组内基因的相对放置的变化。在这些植物的不同水平水平的不同水平上发现了线粒体基因阶排雷[8.那41.那44.].本文的研究结果首次证实了苔类线粒体基因组在结构和基因顺序上的重排。
结论
本研究中获得的结果提供了线粒体和叶绿体基因组结构和基因序列的概述Gymnomitrion concinnatum针对肝脏的已知细胞甲基因组的背景。完整的细胞细胞内基因组序列g . concinnatum一度完全测序,首次延长了博福素的较差的细胞细胞基因组的知识。几乎所有细胞细胞基因组进化的各个方面都是基因含量的多样性,基因组大小和序列相似性似乎相当保守g . concinnatum与其他利物套相似。
尽管如此,研究的最相关的结论,相反,是重排的结构和基因序列的发现g . concinnatum线粒体基因组。它是Liverworts之间的第一种线粒体重组的案例。植物线粒体的重组活性在线粒体基因组的演变中发挥着重要作用[45.].以前的研究得出结论,利物套的促使表现出保守的进化与种子植物中的高度动态演变相反[17.].本研究提供的调查结果强烈支持对线粒体基因组结构的基因令重排的假设不仅限于与普遍接受的陈述常数促进催化剂序命令相反的种子植物的序列12.].考虑到目前只有7个苔类有丝分裂基因组,包括g . concinnatum,这一植物群的其他线粒体基因组很可能也会按其结构和基因顺序重新排列。然而,为了验证上述假设,还需要进一步研究其他苔藓植物的细胞器基因组。
方法
植物材料
样本细节如下:Gymnomitrion concinnatum(Lightf.)Corda,斯洛伐克,高Tatra Mountains,VõsřéKôprovickéSedloPass(LiptovskéKopy),49.19040°N,19.9641°E,Alt。1910米A.S.L.,细粒度筛选,高度参与地衣,腿部。,DET。P.Górski,4.09.2014。使用Zymo植物/种子DNA试剂盒(Zymo Research,Irvine,Ca,USA)提取DNA。从一岁的母标标本中有一个人用碎石母线均化器用二氧化硅床进行研磨50秒,随后根据制造商协议加工。使用Qubit荧光计和Qubit™DSDNA Br测定试剂盒(initrogen,Carbad,NM,USA)估计DNA量。用0.5%琼脂糖凝胶中的电泳检查DNA质量,用Euryx简单拯救(EURX,GDAńSK,波兰)染色。
基因组测序,组装和注释
基因组文库由TruSeq Nano DNA试剂盒(Illumina, San Diego, CA, USA)构建,并使用HiSeqX (Illumina)进行测序,在Macrogen公司(Seoul, Korea)生成150 bp的配对端序列,配对端之间插入长度为350 bp。随后,使用Trimmomatic v0.36去除适配器序列和低质量reads,清理20,166,426个测序reads [46.].使用SPAdes 3.12.0对过滤后的读数进行组装[47.].参考的有丝分裂基因组序列Calypogeia integristipula(NC035977.1)和塑料序列Ptilidium pulcherrimum(NC015402.1)用于鉴定细胞细胞基因组g . concinnatum在生成的contig中。为了验证De Novo组件,使用佐埃R8软件独立地对每个基因组进行迭代映射[48.].叶绿体和线粒体基因组序列g . concinnatum有1440倍和264倍的覆盖深度。
基因被鉴定和注释基于最近已知的器官基因组的相关物种g . concinnatumIE。,Calypogeia integristipula那Tritomaria Quinquedentata.那Pleurozia菊那Ptilidium pulcherrimum和Aneura pinguis..使用佐埃利R8软件进行预测[48.]和BLAST+ 2.8.0工具[49.].带注释的序列g . concinnatum叶绿体基因组和线粒体基因组分别以MH705066和MH705065登录号提交到GenBank。使用OGDraw软件绘制了圆形基因组图[50.].验证基因顺序g . concinnatum促催化促滤蛋白序列g . concinnatum和c . integristipula使用Mauve 2.3.1对齐[38.].两个有丝分裂基因组的比较分析使用Circos图显示[39.].
PCR分析
重新排列的LCB之间的接线区g . concinnatum用PCR确定有丝分裂基因组。根据4对LCBs边缘重叠的核苷酸序列设计了4对引物。引物预期扩增长度的序列在附加文件中6.:表S1。在含有20ng DNA的25μl反应混合物中进行PCR反应,1x PCR缓冲液,1.5mM MgCl2, 200 μM dNTP (dATP, dGTP, dCTP, dTTP),每个引物1.0 μM, 1u RUN聚合酶(A&A Biotech, Gdańsk, Poland)。反应在以下热条件下进行:(1)94°C下初始变性4分钟;(2) 94°C下变性- 45 s;(3) 53°C退火- 50 s,(4) 72°C拉伸- 60 s;(5)最终延伸- 72°C下7min。2-4阶段重复30次。PCR产物在QIAxcel毛细管电泳系统中分离,使用QIAxcel高分辨率试剂盒;15-3000 bp序列标记(Qiagen)和100-2500 DNA大小标记。电泳程序采用标准OM500设置。扩增产物的大小用BioCalculator软件(Qiagen)测定。
系统染色剂重建
七种肝曲面的毒素序列I.。Aneura pinguis.那Calypogeia integristipula那地钱paleacea那Pleurozia菊那Treubia Lacunosa,Titomaria Quinquedentata在Genbank和Gymnomitrion concinnatum本研究介绍,用于系统发育分析。首先,一组38个蛋白质编码序列(附加文件1:表S2),提取,连接和比对gener8 [48.]和mafft [51.].接下来,基于对准,使用MRBAYES 3.2.1进行贝叶斯分析[52.),包括M.Paleacea.作为外群。所述MCMC算法2,000,000世代(采样每1000)具有四个递增加热链运行。第1000种树木被丢弃残影滞留。剩下的树木被用来产生一致树。
预测RNA编辑网站
预测蛋白质编码序列中的编辑位点87叶片和42个线粒体基因,Prepact 2.0 [37.]工具,e值截止值为0.001。
缩写
- CD:
-
编码序列
- 红外:
-
反向重复区域
- LCB:
-
当地共线块
- LSC:
-
大单拷贝区
- ORF:
-
开放阅读框架
- SSC:
-
小单拷贝区域
参考
- 1。
帕尔默JD。叶绿体基因组的比较组织。Annu Rev Genet。1985; 19:325-54。
- 2。
·派克KA。质体分裂和发育。植物细胞。1999;11:549-56。
- 3.
Sabir JSM, Arasappan D, Bahieldin A, aboa - aba S, Bafeel S, Zari TA, et al.(2014) 9个枣椰树品种线粒体和质体基因组SNP分析揭示了质体间的异质性和亲缘关系。《公共科学图书馆•综合》2014;九4。https://doi.org/10.1371/journal.pone.0094158
- 4.
库尔奇PL,德保拉d,掸子d,文德拉明GG。Sonnate G(2015)多官能作物朝鲜蓟和与其他比较菊科的完整叶绿体基因组。Plos一个。2015; 10:3。https://doi.org/10.1371/journal.pone.0120589..
- 5。
Krawczyk K,Nobis M,Myszczyńskik,Klichowska E,Sawicki J.Plastid超级条形码作为羽毛草(Poaceae:Stipa)的物种歧视的工具。SCI批准。2018; 8(1924年)。https://doi.org/10.1038/s41598-018-20399 -2198-018-20399-W..
- 6.
Duchene S,Frederick Ia,Vilstrup J,Caballero S,Morin PA。促滤皮瘤:使用单个区域和分配方案对拓扑,替代率和发散时间估计的影响。Plos一个。2011; 6(11)。https://doi.org/10.1371/journal.pone.0027138.
- 7.
Fabbre P,upham ns,emmons lh,justy f,leite ylr,损失Ac,等。南非刺鼠的毒素内植物学,多样化和生物地理。mol Biol Evol。2017; 34(3):613-33。https://doi.org/10.1093/molbev/msw26..
- 8.
Dietrich A, Wallet C, Janicka S, Gualberto JM。线粒体DNA重组、修复和分离:最新的科学数据和展望。WMS杂志。2017;2(2)。https://doi.org/10.18143/JWMS_v2i2_2023.
- 9.
王志强,刘永强,王志强,等。苔藓植物的进化动力:系统发育学推断证实了蕈藓科的快速辐射。分子系统学进展。2018;120:240-7。https://doi.org/10.1016/j.ympev.2017.12.002.
- 10。
Savicki J,Szczecińskam,Bednarek-Ochyra H,Ochyra R.线粒体系统支持Mitococondralocomics支持传统上构思的属racomatium(Broyophyta:Grimmiaceae)。Nova Hedwigia。2015; 100(3-4):293-317。https://doi.org/10.1127/nova_hedwigia/2015/0248.
- 11.
Szczecińskam,sramko g,Wołoszk,Sawicki J.稀有和濒危植物物种Pulsatilla Pulsatibla Pulsatibla Patens(L.)磨机的遗传多样性和人口结构。Plos一个。2016; 11(3)。https://doi.org/10.1371/journal.pone.0151730.
- 12.
刘Y,梅迪纳R,Goffinet B. 350苔藓线粒体基因组化瘀,早期的陆生植物系的我。mol Biol Evol。2014; 31(10):2586-91。https://doi.org/10.1093/molbev/msu199.
- 13。
张志强,张志强,张志强,等。有丝分裂基因组分析支持了藓属(藓科,苔藓植物门)最近的划分。Sci众议员2017;7。https://doi.org/10.1038/s41598-017-04833-z.
- 14。
陈志强,陈志强,陈志强,等。植物线粒体基因组的初步研究进展。线粒体DNA。2016; 27(3): 1709 - 10。https://doi.org/10.3109/19401736.2014.961133.
- 15.
Vigalondo B,刘Y,Draper的I,拉拉楼Garillwti R,马津帕卡V,等人。比较苔属灵藓藓三个完整的线粒体基因组。线粒体DNA部分B 2016; 1(1):168-70。https://doi.org/10.1080 / 23802359.2016.1149784.
- 16。
Shaw Aj,Devos N,Liu Y,Cox Cj,Goffinet B,Flatberg Ki,等。新兴模型系统的细胞内系统:SpHagnum(Peatmoss)。Ann Bot。2016; 118:185-96。https://doi.org/10.1093/aob/mcw086.
- 17.
王B,薛俊,李L,刘y,邱yl。Liverwort Pleurozia Purpurea的完全线粒体基因组序列揭示了Liverworts中极度保守的线粒体基因组进化。Curr Genet。2009; 55(6):601-9。https://doi.org/10.1007/s00294-009-0273-7.
- 18.
刘艳,薛建勇,王斌,李丽,邱玉玲。早期陆生植物Treubia lacunosa和Anomodon rugelii的线粒体基因组:动态和保守进化。《公共科学图书馆•综合》。2011;6(10)。https://doi.org/10.1371/journal.pone.0025836..
- 19.
Myszczyńskik,bączkiewicza,buczkowska,ślipikom,szczecińskam,Savicki J.Aneura Pinguis的细胞细胞基因组的非凡变化揭示了早期陆地植物的先进隐秘形态。SCI批准。2017; 7(1)。https://doi.org/10.1038/s41598-017-10434-7.
- 20。
Ślipiko M, Myszczyński K, Buczkowska-Chmielewska K, Bączkiewicz A, Szczecińska M, Sawicki j。基因。2017;8(12):395。https://doi.org/10.3390/genes8120395.
- 21。
Vana J, Söderström L, Hagborg A, von Konrat M, Engel JJ。今日早期陆生植物:分类学。裸子蕨科植物分类与命名。2010;11:1-80。https://doi.org/10.11646/phytotaxa.11.1.1.
- 22。
HübschmannVA。Prodromus DER Moosgesellschaften Zentraleuropas。Bryophytorum bibliotheca乐队。J. Cramer。1986;(32):I-413。
- 23。
Górskip.Tatra山脉的白雪丛白桦植被(西喀尔巴阡西部,波兰和斯洛伐克)。Nova Hedwigia。2016; 102(1-2):9-67。https://doi.org/10.1127/nova_hedwigia/2015/0286.
- 24。
DIESSEN K.欧洲苔藓植物的分布,生态振幅和植物脑病特征。Bryophytorum bibliotheca乐队。J. Cramer。2001; 56:3-289。
- 25。
Marstaller R. Syntaxonomischer Konspekt der Moosgesellschaften Europas and angrenzender Gebiete。Thuringische Botanische充电器。Haussknechtia。2006年,十三1 - 192。
- 26。
De RoO Rt,Hedderson Ta,SöderströmL.分子见解叶状肝曲族植物的植物植物的系统发育。分类。2007; 56:310-4。
- 27。
Vilnet AA,Konstantinova Na,Troitsky AV。在Gymnomitriveae H. Klingr的分子系统发育。(肝脏)。计算系统发育和分子系统“CPMS 2007”。莫斯科:会议诉讼。KMK。p。24-6。
- 28。
Ohyama K,Fukuzawa H,Kohchi T,Shirai H,Sano T,Sano S等人。从Liverwort Markantia多晶晶叶绿体DNA的完整序列推导出叶绿体基因组织。自然。1986; 322:572-4。
- 29.
WICKETT NJ,GOFFINET B. MYCO-exerootcophic Liverwort Cryptothallus Mirabilis Malb的起源和关系。(Metzgeriales,Marchantiophyta)。Bot J Linn Soc。2008; 156:1-12。https://doi.org/10.1111/j.1095-8339.2007.00743.x.
- 30.
Forrest LL,Wideett NJ,Cox CJ,Goffinet B. Ptilidium(Ptilidiaceae)的深度测序表明了Liverwort Plastid基因组结构中的进化瘀滞。植物ECOL EVOL。2011; 144:29-43。https://doi.org/10.5091/plecevo.2011.535.
- 31。
小田,大和K,大田E,中村Y,竹村M,等。一种苔类植物线粒体基因组中的转移RNA基因:类似叶绿体的tRNAs的缺失。核酸学报1992;20(14):3773-7。
- 32。
Shaw J,Renzaglia K.系统发生和苔藓植物的多样化。我是J机器人。2004; 91(10):1557-81。https://doi.org/10.3732/AJB.91.10.1557.
- 33。
Groth-Malonek M,Wahrmund U,Polsakiewicz M,Knopp V.伪核苷酸的进化:功能性线粒体NAD7基因的独家存活支持Haplomitium作为最早的Liverwort谱系,并提出了Marchantiidae的次级RNA编辑的二次丧失。mol Biol Evol。2007; 24(4):1068-74。https://doi.org/10.1093/molbev/msm026..
- 34。
割草机jp。预备套件:用于植物线粒体基因,叶绿体基因和用户定义的对准的预测RNA编辑器。核酸RES。2009; 37:253-9。https://doi.org/10.1093/nar/gkp337.
- 35。
南极苔藓植物Sanionia uncinata (hew .)的完整质体序列Loeske。中华医学杂志。2018;19(3)。https://doi.org/10.3390/ijms1903070.
- 36。
张志强,张志强,张志强,等。植物RNA编辑的研究进展。Annu Rev Genet. 2013; 47:335-52。https://doi.org/10.1146/annurev-genet-111212-133519..
- 37。
preact 2.0:预测细胞器基因组序列中的c - u和u - c RNA编辑。生物信息学报。2013;7:1-19。https://doi.org/10.4137/bbi.s11059.
- 38。
Darling Ac,Mau B,Blattner Fr,Perna nt。紫红色:重排的保守基因组序列的多次对准。Genome Res。2004; 14(7):1394-403。https://doi.org/10.1101/gr.2289704.
- 39。
Krzywinski M,Schein J,Birol I,Connors J,Gascoyne R,Horsman D等人。电讯:对比较基因组学的信息审美。Genome Res。2009; 19(9):1639-45。https://doi.org/10.1101/gr.092759.109.
- 40.
古布尔托JM,Mileshina D,钱包C,NIAZI AK,Weber-Lotfi F,Dietrich A.植物线粒体基因组:动力学和维护。生物chimie。2014; 100:107-20。https://doi.org/10.1016/j.biochi.2013.09.016.
- 41.
戈伯托·杰姆,牛顿kj。植物线粒体基因组:突变动力学和机制。Annu Rev植物Biol。2017; 68:225-52。https://doi.org/10.1146/annurev-arplant-043015-112232.
- 42.
陈泽,赵n,李某,格罗弗ce,nie h,wnedel jf等。植物线粒体基因组进化和细胞膜雄性不育。CRIT Rev Plant Sci。36(1):55-69。https://doi.org/10.1080/07352689.2017.1327762.
- 43。
阿列塔-Montiel的MP,Shedge V,达维拉Ĵ,Christensen的AC,麦肯齐SA。拟南芥线粒体基因组的多样性经由核控制重组活性发生。遗传学。2009; 183:1261-8。https://doi.org/10.1534/Genetics.109.108514.
- 44。
Aguileta G,德维也纳DM,罗斯ON,排ME,吉罗T,珀蒂E,菌类中的线粒体基因顺序的Gabaldon T.高的可变性。基因组Biol Evol。2014; 6(2):451-65。https://doi.org/10.1093/gbe/evu028..
- 45。
Kühnk,gualberto jm。在线粒体基因组的稳定性,修复和演化中重组。ADV BOT RES。2012; 63:215-52。https://doi.org/10.1016/B978-0-12-394279-1.00009-0.
- 46。
Bolger Am,Lohse M,Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014; 30(15):2114-20。https://doi.org/10.1093/bioinformatics/btu170.
- 47。
Bankievich A,Nurk S,d安季波,古列维奇AA,德沃尔金男,库利科夫AS,等人。黑桃:一种新的基因组装配算法及其在单细胞排序的应用。j计算biol。2012; 19(5):455-77。https://doi.org/10.1089/cmb.2012.0021.
- 48。
等。Geneious basic:一个集成的、可扩展的用于序列数据组织和分析的桌面软件平台。生物信息学。2012;28(12):1647 - 9。https://doi.org/10.1093/bioinformatics/bts199.
- 49。
Johnson M, Zaretskaya I, Raytselis Y, Merezhuk Y, McGinnis S, Madden TL. NCBI BLAST:更好的网页界面。核酸学报2008;36。https://doi.org/10.1093/nar/gkn201.
- 50.
Lohse M, Drechsel O, Kahlau S, Bock R. OrganellarGenomeDRAW——一套用于生成质体和线粒体基因组物理图和可视化表达数据集的工具。核酸学报2013;41。https://doi.org/10.1093/nar/gkt289.
- 51.
Katoh K,Standley DM。Mafft多序列对齐软件版本7:性能和可用性的提高。mol Biol Evol。2013; 30(4):772-80。https://doi.org/10.1093/molbev/mst010.
- 52.
Ronquist F, Huelsenbeck JP。贝叶斯先生3:混合模型下的贝叶斯系统发育推断。生物信息学。2003;19(12):1572 - 4。https://doi.org/10.1093/bioinformatics/btg180.
致谢
不适用。
资金
liverwort有丝分裂基因组测序由波兰国家科学中心Kraków资助:Calypogeia线粒体基因,批准号19分之2015/ B / NZ8 / 03970,Aneura pinguis.有丝分裂基因组,批准号2016/21/B/NZ8/03325,Tritomaria Quinquedentata.和Gymnomitrion concinnatum促滤膜,授予No.2017 / 01 / X / NZ8 / 01094。
数据和材料的可用性
本研究期间生成或分析的所有数据都包含在本发布的文章及其补充信息文件中。带注释的序列g . concinnatum叶绿体基因组和线粒体基因组分别以MH705066和MH705065登录号提交到GenBank。
作者信息
隶属关系
贡献
KM组装基因组序列,分析的数据,写到主原稿文本和制备图PG收集,识别和分析研究材料以及精细的手稿。MS进行DNA提取和质量检查以及修改和编辑手稿。JS组装基因组序列,写并审查了原稿,并在整个研究提供了指导。所有作者阅读并认可的终稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
附加文件1:
表S2。细胞器基因组的基因含量Gymnomitrion concinnatum。(医生50 kb)
额外的文件2:
图S1。结构的结构COX1.Liverworts中的基因。浅灰色块描绘内含子,而深灰色块描绘了基因的外显子。这m . paleacea cox1基因是代表苔类植物的其他部分。这四个序列中的共有图形呈现的序列同一性(更环保区域的更高的同一性)。(PDF 580 KB)
附加文件3:
表S3。预测叶绿体基因中的RNA编辑位点Gymnomitrion concinnatum。(Doc 149 KB)
额外的文件4:
表S4。预测线粒体基因内的RNA编辑位点Gymnomitrion concinnatum。(Doc 84 KB)
额外的文件5:
图S2。促滤布组结构的PCR验证。由于PCR分析而获得的扩增子的四个电泳图。X轴表示y轴表示相对荧光的时间。(PDF 209 KB)
额外的文件6:
表S1。在本研究中所用的引物序列。(DOC 37 KB)
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
myszczyński,k。,górski,p.,ślipiko,m。et al。细胞细胞基因组的测序Gymnomitrion concinnatum(Jungermanniales)揭示了进化稳定的苔类有丝分裂基因组在结构和基因顺序上的第一个例外。BMC植物BIOL.18,321(2018)。https://doi.org/10.1186/s12870-018-1558-0
已收到:
公认:
发表:
关键字
- 基因组重排
- 基因订单
- 苔类
- plastid基因组
- 线粒体基因组
- 地钱门