摘要
背景
大豆是经历了两次全基因组复制事件paleopolyploid。基因重复是一个型基因组变化,可导致预先存在的基因新颖功能。查耳酮合酶(CHS)是植物特异性III型聚酮化合物合酶,其催化在(异)在植物中类黄酮合成的第一个关键步骤。
结果
在这里,我们进行了全基因组搜索CHS.大豆的基因,并确定了21种GMCHS含有14个独特的基因座格克斯(GmCHS1-GmCHS14)包括5个新发现的格克斯s(GmCHS10-GmCHS14).此外,3份GMCHS3和2份GMCHS4在大豆中发现。基因结构分析格克斯S显示在蛋白编码区存在一个内含子GmCHS12包含3个内含子。虽然格克斯基因位于8种不同的染色体上,存在大量这些基因在染色体8上存在,其中它们形成3个不同的簇。表达式的分析格克斯基因显示了组织特异性的表达模式,一些GmCHS亚型定位于细胞质和细胞核,而其他亚型仅局限于细胞质。
结论
总的来说,我们已经鉴定出21个GmCHS基因座,其中14个是唯一的格克斯大豆基因组中的基因。它们的基因结构和基因组组织与时空表达和蛋白质定位以及在下游代谢产物的重要性,例如(ISO)类黄酮和它们衍生的植物脂蛋素。
背景
在过去2000万年的植物演进中发生了全基因组重复,导致基因重复。大量植物物种的全基因组序列的可用性表明,大约64.5%的植物基因被重复(在[1])。基因复制事件随后导致基因组大小和整个基因的增加,从而影响许多基因组的结构和功能[2那3.].在适应或在减压选择性约束进化的过程中,重复基因获得的预先存在的基因新颖功能[4.那5.].新的基因也可以从基因间空间从头产生[6.或者启动子的新转录调节位点,其改变基因表达[7.].这P.otentialCIS.-启动子区域的元件也可能因发育阶段和环境的变化而在序列和特异性上发生变化[8.].尽管基因家族的成员含有非常高的序列同一性,其时间和空间的表达水平可以不同。
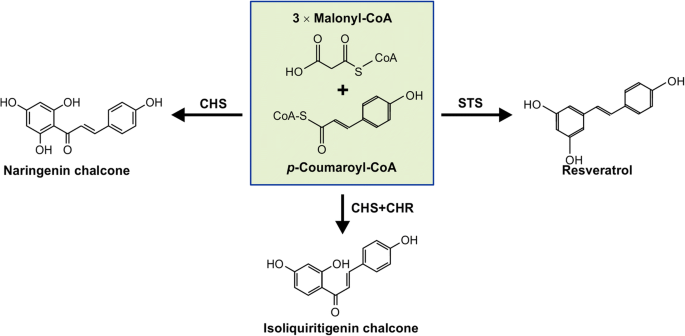
聚酮合成酶(PKS)通过催化二碳乙酸酯单元的顺序缩合形成一个不断生长的聚酮链,在植物的初级和二级代谢中起关键作用。根据PKS酶的催化机理、结构域和亚基组织,将其分为I型、II型和III型。I型和II型PKSs在细菌和真菌中发现,III型PKSs主要是植物特异性的。III型PKSs作用于同型二聚体,活性位点包含一个Cys-His-Asn催化四分体[9.那10那11],与I型和II PKS不同,它们不需要酰基载体进行其功能[12].这些酶已知为查耳酮合酶(CHS)样酶包括CHS,芪合酶(STS),2- pyronesynthase,吖啶酮合成酶,二苯甲酮合酶,bibenzyle合酶,phlorisovalerophenone合酶,亚苄基丙酮合酶,C-甲基查尔酮合成酶,高绒毛膜二醇/绒毛膜二醇合成酶,芦荟素合成酶,香豆素三乙酸合成酶,己酸合成酶,联苯合成酶,二苯乙烯羧酸合成酶,八酸合成酶,五酮色素素合成酶,花药特异性的chs样[9.].在这些PKS中,CHS和STS在结构上相似[11那13,植物特异性和催化凝结反应P.-香豆蔻酰辅酶a和丙二酰辅酶a的3个乙酰基分子生成一种常见的四羧酸中间体,在CHS催化下发生克莱森缩合反应[11]或由STS催化的醛醇环化[14]分别产生Naringenin Chalcone和白藜芦醇(图。1).在豆科植物中,CHS与豆科植物特有的查尔酮还原酶共同作用,产生同质查尔酮。这些查尔酮的生产是大量类黄酮生物合成的第一步,这些类黄酮已被证明在抵御各种生物和非生物胁迫、花色素、固氮、花粉育性和种皮颜色等方面发挥重要作用。
在大豆中,种子涂色是各种发展的重要性状之一。这CHS.基因家族已被广泛研究,因为它们的表达造成种子涂层色素沉着的变化[15].大豆是一个古代聚宝脂,经过了两个全部基因组重复事件[16那17那18] 75%的多基因家族存在存在[19].早些时候,CHS.Superfamily有9名成员在大豆中报道[20.].在这里,我们进行了全基因组搜索CHS.基因大豆和确定了21个格克斯基因组中有14个独特基因的位点。除了已知的9格克斯,我们报告5个新格克斯S在大豆和它们的基因建筑,系统发育,基因表达和蛋白质定位。结果提供了对功能分歧的证据格克斯这参与了大豆中许多重要化合物的生产。
结果
这格克斯基因家族包含14个假定成员
确定成员的第一步格克斯基因家族,我们在注释中使用了关键字搜索“Chalcone Synthase”G. Max.Wm82.a2。Phytozome的v1基因组。这导致1516个基因和2635个本体匹配。如此大量的基因和本体论匹配是由于在大豆基因组数据库中包含了“查尔酮”和/或“合酶”的所有注释。在2635个本体论的列表中,识别出了一个单词为“chalcone”和“synthase”的本体论(PANTHER IDPTHR11877:SF27),并选择该本体论寻找其他相关的本体论格克斯s在植物血统中使用“共享注释”功能。这个过程确定了总共19格克斯这包括先前识别的9个基因GMCHS[15].确保所有CHS.基因在大豆中鉴定出来格克斯被用作BLAST搜索的查询,确定了两个额外的格克斯S(Glyma.09g074900和Glyma.13g034300),共21岁CHS.大豆基因组的基因座。基于公共领域中可用的RNASEQ数据,表达分析格克斯进行基因。没有成绩单4格克斯在任何组织中检测到基因(Glyma.05G153100,Glyma.09G074900,Glyma.11G097900和Glyma.13G034300)在任何组织中检测到伪原的任何组织中。序列比较格克斯基因家族成员透露,有三个副本GMCHS3(Glyma.08G109300,Glyma.08G110900和Glyma.08G110300)和两个副本GMCHS4(Glyma.08G110700和Glyma.08G110500)。总共,我们发现共14个独特格克斯大豆基因组中的基因。这些格克斯基因编码蛋白质,计算分子量从37到45 kDa不等。详细的特点格克斯基因如表所示1.
推导的GMCHS蛋白质序列的对准在整个区域中显示出非常高的序列同一性。在GMCHS中,GMCHS14最多样化,并且在氨基酸水平下仅显示43至52.9%的序列同一性,其它GMCHS同种型。氨基酸和核苷酸水平在氨基酸和核苷酸水平的其他GMCH的成对百分比同一性分别从73.4%到100%和67.7%至100%变化(附加文件1:表S1)。由于有3份GMCHS3和2份GMCHS4,我们分析了所有人的启动子地区(1000 bp上游的翻译网站上游)格克斯s。所有候选基因启动子之间的成对序列比较显示序列同一性范围为0.4至100%(附加文件2:表S2)。即使编码区域DNA序列序列,3份)GMCHS3范围为99.9至100%,其启动子序列显着不同(2.6〜48.9%的身份)。因此,我们命名为3份GMCHS3作为GMCHS3A(Glyma.08G109300),gmchs3b.(Glyma.08G110900),和GMCHS3C.(Glyma.08G110300)。同样,两份拷贝的启动子序列GMCHS481.6%是相同的,被命名为GMCHS4A(Glyma.08G110700)和GMCHS4B.(Glyma.08G110500)。尽管如此GMCHS5和GmCHS12编码区序列同源性仅为87.4%,启动子区域(翻译起始位点上游1000 bp处)同源性为100%。
GMCHS的序列比较和系统发育分析
CHS的晶体结构来源于Medicago Sativa.(MSCHS2)阐明了四个活性位点残留物(Cys 164,PHE 215,His 303和ASN 336)的重要性,其中Cys-His-Asn Triads对底物结合至关重要[11].为了评估GMCHSS是否包含有源站点残留物和CHS / STS签名主题(WGVLFGFGPGLT),我们使用其推导的氨基酸序列与MSCHS2进行所有推定的GMCHS的序列对准。结果表明,所有14个GMCH的酶III型活性位点被保守(图。2).除了发现四个氨基酸取代(V369i,F371L,L377 V和T278A)外,除了GMCHS14外,CHS / STS签名基序在所有GMCHS中都是保守的。除了GMCHS14之外的所有GMCHS蛋白中也存在产物和丙酰基-CoA结合位点。这些发现表明GMCHS14可以具有比其同种型不同的功能不同。此外,GMCHS12包含CHS所需的所有关键残留物,但它在其序列中具有3个大缺失。
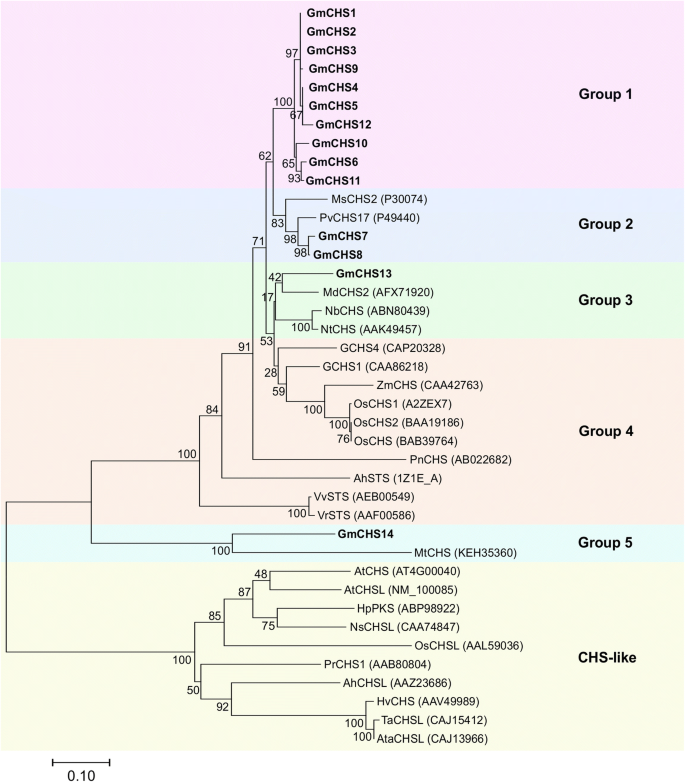
为了阐明GmCHS同亚型的进化关系以及与其他植物CHS的进化关系,我们将14个推测的GmCHS的氨基酸序列与其他植物CHS、CHS样蛋白和STS蛋白进行了系统发育分析。如图1所示。3., GmCHSs聚集成4个不同的组。第1组由10个GmCHSs组成,其中6个(GmCHS1、GmCHS2、GmCHS3、GmCHS9、GmCHS4和GmCHS5)紧密聚集,除GmCHS2外,其余5个GmCHSs位于第8染色体上。第2类群包含GmCHS7和GmCHS8,这两个类群形成了一个紧密的演化支,其中包括之前已经确定的豆科特有的CHSs、PvCHS17和MsCHS2。组3和组5分别含有GmCHS13和GmCHS14。GmCHS14更接近STS葡萄属锐利那V. Vinifera., 和arachis hypogaea.在进化树上。来自单码的CHSS,如米饭(OSCHS1,OSCHS2和OSCHS3)和玉米(ZMCH)形成单独的疏水板(第4组).来自不同物种的CHS样蛋白,包括rapidopsis chs形成了从系统发育树中大多数已知的chs形成了不同的腕表,证明了Chs超级家庭在植物中的发散。
GMCHS推导氨基酸的分子系统发育分析。来自大豆的GMCHSS的推导的氨基酸序列与来自其他植物物种的表征CHS和CHS样蛋白与其他植物物种的表达,使用MEGA7中的邻近方法产生进化树[45]。在Bootstrap测试中聚集在一起的相关分类卡的复制树的百分比显示在分支旁边。秤条表示每个网站代表残留物替换的分支长度。GMCHS以粗体表示。在,拟南芥蒂利亚纳;操作系统,奥雅萨苜蓿;女士,Medicago Sativa.;公吨。,Medicago Truncatula.;马里兰州,Malus Domestica.;PV,phoudolusulus vulgaris.;VR,葡萄属锐利;vv,葡萄;啊,arachis hypogaea.;PR., Pinu sradiata;生命值,金刚素孔化物质;ns,烟草的抗旱性;HV,Hordeum Vulgare.;助教,Triticum Aestivum.;Ata,Aegilops Tauschii.;Ahchl,拟南芥halleri;PN,松叶蕨;NB,尼古利亚娜·宾夕法尼亚州;NT,烟草tobaccum;Zm评选,Zea Mays.
确定对差异的选择性进化压力格克斯基因,我们获得了来自植物基因组中的每个重复基因对的非同义(KA)和同义词(KS)值的重复基因组区域,从植物基因组重复数据库(附加文件)3.:表S3)。提取重复格克斯来自40,972个基因列表的基因导致3重复格克斯基因对:i)GMCHS5和glyma.05g153100.(假基因),(二)GMCHS7.和GMCHS8和III)GmCHS10和GmCHS11(桌子2).在进化过程中有纯化选择的基因Ka/Ks值小于1。复制的Ka/Ks值格克斯基因对范围为0.065至0.549(表2)表示它们可能在复制事件后获得有限的功能分歧。
染色体排列和基因结构GMCHS
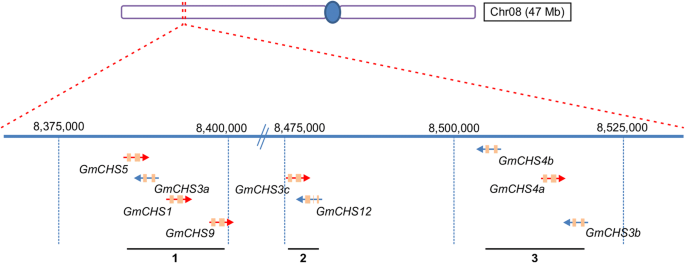
这21格克斯在大豆中的8种不同的染色体上分布包括14个独特基因和3个重复副本。除了染色体1和8的染色体中,这些染色体中的基因密度均匀(每种染色体的一个基因),含有2和9的染色体格克斯基因,分别(表1).9.格克斯染色体8上的基因位于富含135kb基因的区域内,含有总共18个基因。如图1所示。4., 9GMCHS在染色体8上形成3个不同的簇,每个簇包含副本GmCHS3。群集1包含GMCHS5,GMCHS3A,GMCHS1和GMCHS9.在15 kB的区域内,它们以尾部布置成尾部,头部到头部或头部到尾部方向。群集2包含GMCHS3C.和GmCHS12安排尾巴到尾巴。最后,在8,504,479..8519303上的染色体8形成的14.8 kB区域形成群体3,其中包含2份GMCHS4(GMCHS4B.和GMCHS4A)排列在头部到头向方向和GmCHS3b。关于染色体8上的135 kB区域内的所有18个基因的详细信息在附加文件中显示4.:表S4。
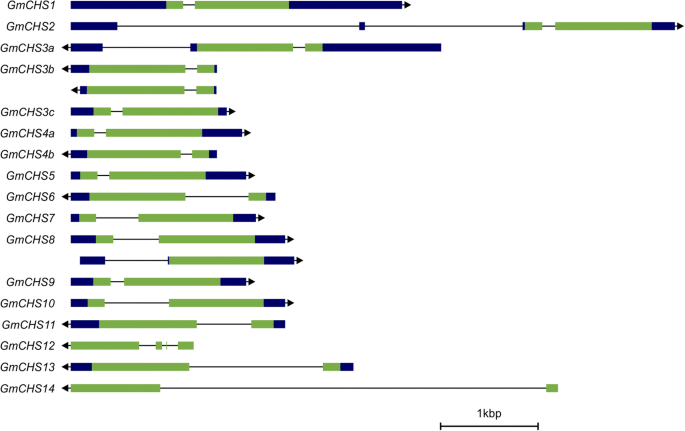
基因结构分析格克斯基因显示2个外显子和1个内含子GmCHS12包含4个外显子和3个内含子(图。5.).即使大多数格克斯小号包含在单个内含子,它们的内含子大小家庭成员范围从121到4347个核苷酸内变化。另外,存在单个内含子GMCHS3A3'UTR和2个内含子GMCHS2找到了5'UTR。
示意图格克斯基因结构。格克斯从Phytozome数据库(https:///phytozome.jgi.doe.gov/pz/portal.html# !info?alias=org_gmax.).黑色和绿色的盒子分别代表utr和外显子,线条代表内含子。相对于基因组序列,右箭头表示“+”链,左箭头表示“-”链。如图所示,按比例绘制基因结构图像
表达式的分析格克斯基因家族
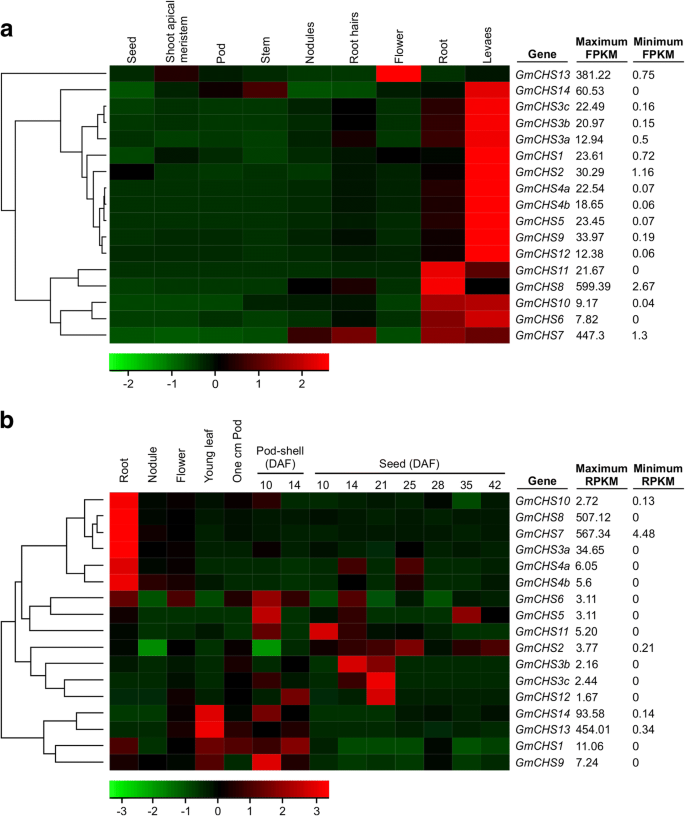
以确定组织特异性基因表达模式格克斯基因家族,我们使用了两套公开可用的基因组宽转录物分析大豆组织作为资源[21那22那23].Libault等人[21]数据集由大豆组织中的转录性丰度组成,如花,拍摄顶端单位,种子,豆荚,茎,根,结节,叶和根毛。碎片perkilobase的成绩单每百万映射读取读数(fpkm)值格克斯在他们的高表达组织中,从7.82到599.39(附加文件)不同5.:表S5)。如图1所示。6.一、多数格克斯S在叶子中高度表达。成绩单GMCHS6,GMCHS7,GMCHS8,GMCHS10和GmCHS11与其他组织相比,根中有丰富的。积累GmCHS13与其他组织相比,成绩单较高。三个副本的表达式模式GMCHS3在大豆组织中显示的差异表达模式,同时的两个副本GMCHS4表现出几乎相似的表达模式。根据转录组数据,没有GmCHS14在结节中观察到,而GMCHS6那GmCHS11和GmCHS14在种子组织中观察到。Severin等人的第二个数据集。[23]在几种不同的发育阶段的根,花,幼叶,结节和豆荚和种子如根,花,幼叶,结节和种子中的转录性丰富。每百万映射读取(RPKM)值的数千碱基读数格克斯S在高表达组织中的差异为1.673 (GmCHS12种子21-DAF)至567.342 (GMCHS7.在根目录中)(附加文件5.:表S5)。由于本研究包括多个阶段的豆荚和种子组织样本,它提供了更好地评估表达水平格克斯种子组织基因与早期的研究相比(比较图。6.A和B)。在数据集中,成绩单GMCHS7,GMCHS8,和GmCHS10与其他组织相比,根中有丰富的。同样的,GMCHS1,GMCHS9和GmCHS14转录本在叶片组织中以较高水平积累。但是,在表达模式上存在一些差异格克斯S在这两组研究中被观察到。例如,相对转录本丰度GMCHS3A,GMCHS4A和GMCHS4B.在根系组织中在这两个研究中不匹配(图。6.A和B)。
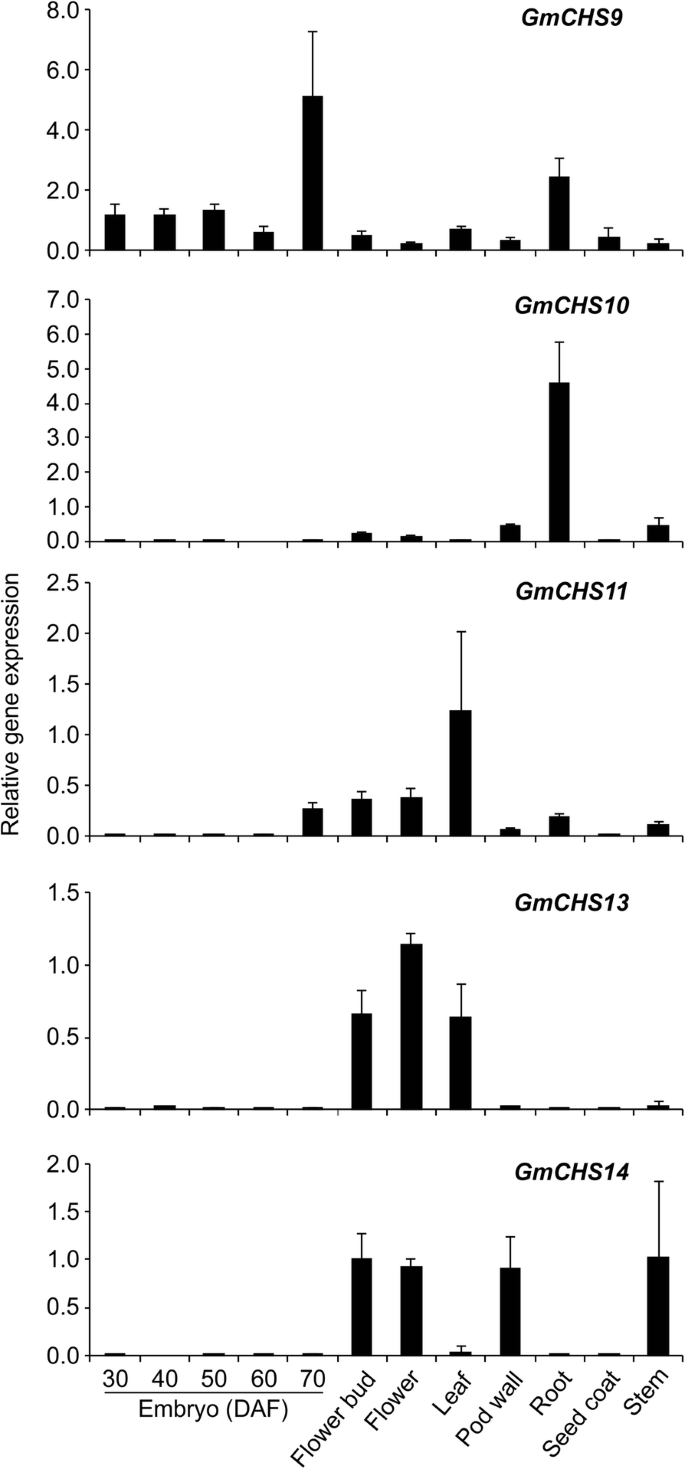
为了验证RNASEQ表达数据,我们研究了新发现的组织特异性表达格克斯年代随着GMCHS9.通过qRT-PCR。RNA从大豆的营养生长和生殖组织中分离在开发进行qRT-PCR分析。我们的试验结果与两个以前报道的RNA测序研究。如图1所示。7.,的表达GmCHS10有大量的根和GmCHS13在与RNASEQ数据相关的花朵中(图。6.).低表达GmCHS10、GmCHS11 GmCHS13,和GmCHS14在胚胎组织中观察到(30至70dAf)(图。7.),结果与RNAseq研究一致。尽管,无花果。6.b包含表达格克斯种子组织中的基因,只有两个种子(28和42个DAF)的发育阶段更接近我们研究中使用的胚胎(30和40个DAF)。为了确定重复基因的表达分歧,两个公开的RNASEQ数据集中常见的样品(根,结节和花)的基因表达值[21那23通过II型单向ANOVA分析,然后进行多次比较HOC Tukey的测试。结果表明表达模式GMCHS7.和GMCHS8重复组与其他组差异显著格克斯根,结节和花组织中的基因。但是,没有鉴定出其他两个重复的这种差异格克斯基因对。
GMCHS同种型的亚细胞定位
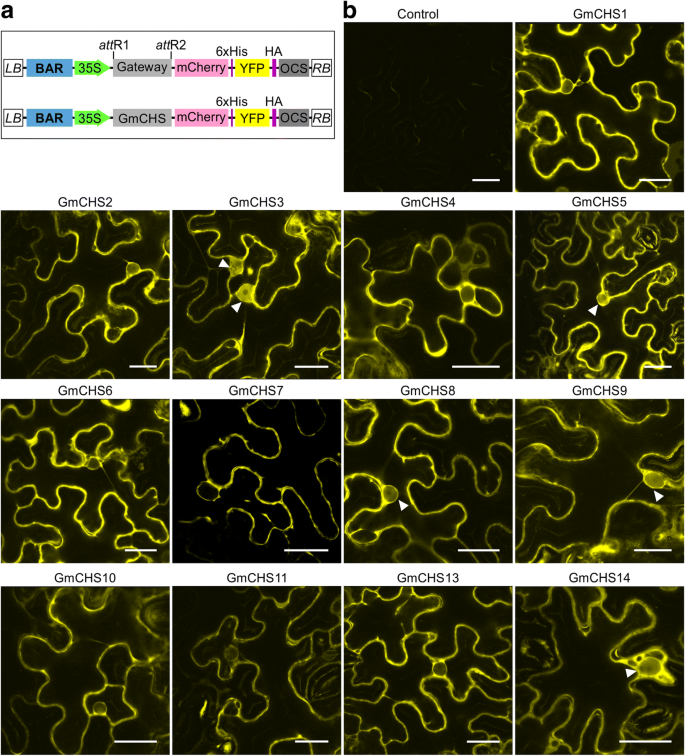
以前我们报道了GMCHS8的双亚细胞定位(细胞质和细胞核)[24].尽管预测所有GMCHS同种型是细胞溶症,但我们确定了他们的本地化在足底.全长的翻译融合格克斯被创建的双重报道基因的mCherry和YFP(图的上游。8.一个),和瞬时在叶表皮细胞中表达n benthamiana.试图克隆GmCHS12由于其表达水平低,并不成功。因此,GMCHS12不包括在亚细胞定位研究中。为了避免GMCHS蛋白的被动扩散到核中,通过在载体珍珠化物体101中添加MCHERRY来产生双重报道载体,其增加了融合蛋白的尺寸。如图1所示。8.B,在细胞质中观察到所有13种GMCHS蛋白。此外,还发现了5种GMCHSS(GMCHS3,GMCHS5,GMCHS8,GMCHS9和GMCHS14)。
讨论
植物基因组往往比哺乳动物进化得更快,从而产生更多的动态和更高的基因组多样性[25].具有多基因家族的大植物基因组是由多种因素,如基因重复,全基因组重复和驯化。大多数植物种类包含小型CHS.基因家庭。例如,拟南芥基因组含有一个单一的CHS.基因(26] 尽管矮牵牛织布达[27],Ipomea purpurea[28],非洲菊矮牵牛[29] 和Pisum sativum.[30.]包含8,6,3和8CHS.成员分别。最近,A.CHS.在玉米中鉴定出含有14个成员的基因家族[31].在这里,我们总共确定了14个独特的CHS.基因(GmCHS1-GmCHS14)在大豆基因组中。我们在大豆基因组中发现了21个CHS位点,其中包括3个拷贝GMCHS3,2份GMCHS4和4个伪原。这一世先前描述了在大豆中控制种子涂层颜色的基因座,以含有两个相同的簇(串联反转重复)CHS1.那CHS3.和CHS4.[20.].在我们的分析中没有找到这种串联重复Glycine Max WM82.A2.v1.然而,CHS.富含染色体8的基因富含区域9CHS.在义丝股线上的基因座,5位上的反义链上的4(图。4.).许多人中很多格克斯基因家族成员包含非常高的序列同一性。例如GMCHS4和GMCHS5在核苷酸水平分享99.7%的序列同一性(附加文件2:表S2)。有可能具有如此高的序列同一性格克斯基因在染色体上的组织,这可能导致染色体上的反向重复而引起突变一世基因座[32].
的成员CHS.其他物种的基因家族表现出功能变异和组织特异性表达模式,如3个物种CHS.基因表现出不同的时空调节非洲菊矮牵牛,只有GCHS1促进黄酮类生物合成[29].由于CHS和STS使用相同的底物,并且这些蛋白质之间的催化活性位点是一致的,所以这些酶在类黄酮或二苯乙烯生物合成中的参与要等到酶活性测定进行之后才能知道。
CHS形成其酶活性的同态二聚体。CHS同源二聚体包含两个功能独立的活性位点。CoA-ThioSters和产品类似物占据CHS复杂结构中同源体的两个活性位点。这些结构鉴定了在每种单体的下部和上部结构域之间的裂隙处的活性位点的位置,其中有少量的化学反应残基存在于活性位点中[11那33].四个保守的氨基酸残基,特别是Cys 164, Phe 215, His 303和Asn 336(根据MsCHS2编号),构成所有chs相关酶的活性位点[11]在GMCHS同种型中保存(图。2).Cys 164用作亲核试剂,作为CHS和STS中的聚酮化酮中间体的附着部位。His 303的氮电子是Cys 164的硫原子的氢键距离,他的303最有可能作为一代来自Cys 164的亲核硫醇化合物期间作为一般碱基。CHS的活跃点体系结构由三个组成与这四个残基相交的互连腔和这些腔包括香豆酰基粘合口袋,COA结合隧道和环化口袋[11].由于GmCHS14序列与其他gmchs序列主要不同,且缺少影响结合和环化囊袋的重要残基,因此其功能可能不同,催化的反应也可能不同。GmCHS7和GmCHS8可能是大豆中活性的CHSs,因为在系统发育树中,它们与PvCHS17和MsCHS2属于同一分支(图2)。3.).GmCHS12序列中存在若干缺失,产生的蛋白质与其他亚型相比分子质量更低(见表)1).然而,在GMCHS12中保存其活性所需的关键残留情况表明它可能是功能性的。
基因家族成员的变异CIS.- 启动子DNA区域的建筑构理导致物种内的差异表达模式。的成员CHS.基因家族在非洲菊矮牵牛显示出功能差异和组织和发育特异性的表达模式[29].尽管两者都有GCHS1和GCHS4在格伯拉花瓣中表达,只有Gchs1负责非洲菊花瓣中的黄酮类生物合成,而GCHS4在营养组织中具有颜料生产中的作用。大部分的格克斯转录物在大豆叶片和根部积累,表明他们在这些组织中的重要性。大豆根中这些基因的表达非常重要,因为CHS催化步骤的下游是参与植物防御机制的异黄酮,以及在大豆和细菌的共生关系中进行氮固定。高表达GMCHS7.和GMCHS8在大豆组织中已经研究过[34]与此处报告的表达分析一致(图。6.).最多格克斯小号是在大豆叶子和根这可以解释为在(异)类黄酮合成的各组织中这些基因的表达要求。的多样化表达格克斯除了它们的不同启动子区域之外,大豆组织中的基因可能是由于它们不同GMCHS5和GmCHS12因为他们的发起人是100%相同的(附加文件3.:表S3)。相同的启动子区域与保守CIS.- 调节元素可能是节段性重复的结果,并且已经在某些重复的基因中观察到这一点[34].已经记录了显示在大豆中不同基因表达的基因家族成员。例如,大豆14-3-3蛋白(SGF14s)[35],GMCHR.[36] 和Chalcone异构酶(GMCHIS.) [37家族成员在大豆组织中也表现出不同的表达模式。
我们的发现是GmCHS的异构体定位于细胞质和细胞核,粘附于其他(iso)类黄酮酶的共同定位[36那37那38]异黄酮代谢蛋白[24].由于(ISO)的类黄酮生物合成涉及多种细胞色素P450S,其局部化并且不在细胞核中,一些GMCHS家族成员的存在提出了这些酶在细胞核中的额外作用的可能性。
结论
总的来说,我们对此进行了全面的分析CHS.在大豆基因组中发现14个独特的基因格克斯■其中6个以及副本GMCHS3和GMCHS4在3个单独的簇中纳入染色体8。基于系统发育分析,GMCHS13和GMCHS14远处与其他角色不同的GMCHS相关。此外,时间和空间表达格克斯成员和GmCHS在子细胞水平同种型特异性的一些同种型的功能的替代揭示。
方法
植物材料
尼古利亚娜·宾夕法尼亚州种子从Rima Menassa博士(伦敦研究和发展中心,农业和农业食品加拿大)获得。种子在25°C/20°C、相对湿度60-70%、光照16 h /暗8 h的生长室中生长。瞬时表达为6 ~ 8周龄的完整叶片n benthamiana使用植物。
在硅和系统发育分析中
来确定的格克斯大豆的基因,植物血统数据库(https:///phytozome.jgi.doe.gov/pz/portal.html.) [22]用于在注释中使用“Chalcone Synthase”的关键字搜索G. Max.wm82.a2.v1基因组。每个CHS.在大豆基因组中进行BLAST (BLASTn)搜索。检索所有GmCHSs的蛋白序列,并使用基于网络的工具ExPASy (https://web.expasy.org/translate/).使用TABRETP进行亚细胞定位的预测(http://www.cbs.dtu.dk/services/targetp/),使用默认参数。大豆基因组重复区域及每个重复基因的Ka/Ks值来自植物基因组重复数据库(http://chibba.agtec.uga.edu/duplication/)。重复格克斯从大豆基因组复制基因中手工提取基因对。
对于系统发育分析,氨基酸序列在Clustral中对齐,并且通过使用Mega7,用1000个引导复制构建邻近的树木[39].使用BIOEDIT序列比对编辑器版本7.5的序列同一性矩阵函数进行成对核苷酸和氨基酸比较。活性位点,丙二酰辅酶A的结合位点和产品的结合位点上GmCHSs的序列使用NCBI保守结构域搜索(标识https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi).
生成热图
来自不同大豆组织的两组来自不同大豆组织的RNA族数据,并且表达值以每百万映射的读数(FPKM)的数百万映射的读数(FPKM)或每百万映射读数(RPKM)的数千碱基读取表达值。所有的FPKM值格克斯从植物血红素中检索大豆组织中的s(https:///phytozome.jgi.doe.gov/pz/portal.html.) [22].下载第二组RNAseq实验的原始数据https://www.soybase.org/ [23].读取进行修整,映射到大豆参照基因组和RPKM值CLC基因组工作台计算(Qiagen公司,USA)用于.Heatmaps的表达水平格克斯在r中使用来自手术库的Heatmap.2功能在r中产生大豆组织。来自两组数据的根,豆荚和花组织的基因表达值用于通过II型单向ANOVA进行表达分解分析,然后进行多次比较后HOC Tukey的测试。
定量RT-PCR分析
在qRT-PCR研究中,Wang和Vodkin从12个不同的大豆组织中分离出RNA [40].使用ThermoScript TM RT-PCR系统(Invitrogen,USA),总RNA(1μg)逆转录。QPCR的基因特异性引物序列在附加文件中列出6.:表S6。所有反应均在三个技术重复进行,并且表达归一化至参考基因CON4[41].实验包括两个生物学重复。使用CFX管理器(BioRad,美国)对数据进行分析。
质粒构建和亚细胞定位
对于亚细胞本地化研究,GMCHS通过PCR使用基因特异性引物从大豆cDNA中扩增。用于用于的底漆格克斯文件的放大部分列在附加文件中6.:表S6。利用BP克隆酶(Invitrogen)将PCR产物克隆到入口载体pDONR-Zeo (Invitrogen)中,然后转化为大肠杆菌DH5α。重组质粒pdOnz-gmchs是证实和与目的载体peg克利101 using的LR克隆酶反应混合物(Invitrogen)重组的序列。将重组质粒转化为农杆菌肿瘤术GV3101通过电穿孔。利用AvrII-mCherry-F和XbaI-6His-mCherry-R引物(附加文件),通过PCR扩增mCherry片段,构建pEGmCherry1016.:表S6)。将所得的PCR产物用AvrII位和XbaⅠ消化,并插入到在YFP在pEarleyGate101 [的N-末端的位点AvrII位42].
pEGmCherry-GmCHS在A. Tumefaciens.gv3101被转化为尼古利亚娜·宾夕法尼亚州浸润叶子[43],通过徕卡TCS SP2倒置共聚焦显微镜观察瞬时表达。共聚焦显微镜使用63X水浸物镜,激发波长为514 nm, YFP发射光谱为530-560 nm。
缩写
- CHS:
-
Chalcone合成酶
- FPKM:
-
每百万映射reads中转录本的超千碱基片段
- PKS:
-
聚酮合酶
- rpkm:
-
每百万次映射读数每千碱基读数
参考文献
- 1.
Panchy N,Lehti-肇MD,肇S-H。基因重复在植物中的演变。植物理性。2016; 171:2294-16。
- 2.
Renny-Byfield, Wendel JF。基因组加倍:多倍体和作物植物。[J] .中国生物医学工程学报,2014;
- 3.
李t-h,唐h,王x,帕特森啊。PGDD:植物中基因和基因组重复数据库。核酸RES。2013; 41(D1):D1152-8。
- 4.
生理和形态进化中的基因共选。《细胞发展生物学》2002;18:53-80。
- 5.
Magadum S,Banerjee U,Murugan P,Gangapur D,Ravikesavan R.基因复制作为演变的主要力量。J Genet。2013; 92(1):155-61。
- 6.
Schlotterer C.来自划痕的基因 - 进化命运德诺维基因。趋势麝猫。2015;31(4):215 - 9。
- 7.
Wray GA, Hahn MW, Abouheif E, Balhoff JP, Pizer M, Rockman MV, Romano LA。真核生物中转录调控的进化。中国生物医学工程学报。2003;20(9):1377-419。
- 8.
Dey N,Sarkar S,Acharya S,Maiti IB。植物中的合成促进剂。Planta。2015; 242(5):1077-94。
- 9.
奥斯汀MB,Noel JP。III型聚酮合成酶的Chalcone合成酶超家族。NAT产品批准。2003; 20(1):79-110。
- 10.
ABE I,Morita H.植物III型聚酮合成酶的Chalcone合成酶超家族的结构和功能。Nat Prod Rep。2010; 27(6):809-38。
- 11.
费雷尔J-L,Jez的JM,鲍曼ME,狄克逊RA,诺尔JP。查尔酮合成酶和植物聚酮的生物合成的分子基础的结构。纳特结构生物学。1999; 6:775。
- 12.
Hopwood DA, Sherman DH。多酮的分子遗传学及其与脂肪酸生物合成的比较。Annu Rev Genet, 1990; 24:37-66。
- 13.
陈志强,陈志强,陈志强,等。花生二苯乙烯合酶的晶体结构。蛋白质。2005;60(4):803 - 6。
- 14.
Tropf S,驰B,宝源G,同型二聚体植物聚酮化合物合酶(芪和查耳酮合酶)的宝源J.反应的机制。用于冷凝反应的单一活性位点是足够的均二苯乙烯,查耳酮,和6'- deoxychalcones的合成。J Biol Chem。1995; 270(14):7922-8。
- 15.
Tuteja JH,Chough SJ,Chan W-C,Vodkin Lo。由天然发生的组织特异性基因沉默介导Chalcone合成酶基因群集甘氨酸最大.植物细胞。2004; 16(4):819-35。
- 16.
Shoemaker RC, Polzin K, Labate J, Specht J, Brummer EC, Olson T, Young N, Concibido V, Wilcox J, Tamulonis JP,等。大豆基因组复制(甘氨酸亚属Soja.).遗传学。1996; 144(1):329-38。
- 17.
Blanc G,Wolfe Kh。植物模型植物物种广泛古代聚倍性,从二份基因的年龄分布推断。植物细胞。2004; 16(7):1667-78。
- 18.
吉尔N,芬德利S,Wallling JG,汉斯C,马静,道尔Ĵ,斯泰西G,杰克逊SA。分子和大豆allopolyploidy chromosmal证据。植物理性。2009; 151:1167至1174年。
- 19.
Schmutz J,Cannon SB,Schlueter J,MA J,Mitros T,Nelson W,Hyten DL,Song Q,Thelen JJ,Cheng J,等。古代聚丙烯大豆的基因组序列。自然。2010; 463(7278):178-83。
- 20。
Tuteja JH, Vodkin LO。大豆基因组中内源性CHS沉默的结构特征及靶位点。作物科学。2008;48:s49 - 68。
- 21。
Libault M, Farmer A, Joshi T, Takahashi K, Langley RJ, Franklin LD, He J, Xu D, May G, Stacey G.一个作物模型Glycine max的整合转录组图谱及其在植物比较分析中的应用。植物j . 2010; 63(1): 86 - 99。
- 22。
Goodstein DM,Shu S,Howson R,Neupane R,Hayes Rd,Fazo J,Mitros T,Dirks W,Hellsten U,Putnam N等人。植物血统:绿色植物基因组学的比较平台。核酸RES。2012; 40(数据库问题):D1178-86。
- 23。
Severin A, Woody J, Bolon Y-T, Joseph B, Diers B, Farmer A, Muehlbauer G, Nelson R, Grant D, Specht J,等。RNA-Seq阿特拉斯的甘氨酸最大:指导,以大豆转录。BMC植物BIOL。2010; 10(1):160。
- 24。
Dastmalchi男,Bernards旅馆MA,大豆异黄酮metabolon的Dhaubhadel S.双锚:对于复杂的绑定,即可通过IFS和C4H内质网的证据。植物杂志2016; 85(6):689-706。
- 25。
Murat F,Van de Peer Y,Salse J.解码植物和动物基因组可塑性,来自差分古进化模式和过程。基因组Biol Evol。2012; 4(9):917-28。
- 26。
Burbulis Ie,Iacobucci M,Shirley BW。黄酮类生物合成的第一种酶中的零突变不会影响拟南芥的男性生育能力。植物细胞。1996; 8(6):1013-25。
- 27。
Koes RE, Spelt CE, van den Elzen PJM, Mol JNM。查尔酮合酶多基因家族的克隆及分子特征矮牵牛织布达.基因。1989; 81:245-57。
- 28。
德宾ml,mccaig b,clegg mt。辉煌基因组在荨麻酮合成酶多核家族的分子演变。植物mol biol。2000; 42(1):79-92。
- 29。
邓X,Bashandy H,Ainasoja M,Kontturi J,Pietiainen M,Laitinen Ra,Albert VA,Valkonen JP,Elomaa P,Teeri Th。重复的功能多样化Chalcone合成酶花青素生物合成的基因非洲菊矮牵牛.新植物。2014; 201(4):1469-83。
- 30.
伊藤男,濑Y,加藤H,白石T,山田T.分子进化和功能相关Chalcone合成酶豌豆的基因。Mol Gen Genet。1997; 255(1):28-37。
- 31。
汉y,丁T,苏B,江河玉米基因组型鉴定,表征及表达及表达分析。int j mol sci。2016:17(2)。
- 32。
Tuteja JH,Zabala G,Varala K,Hudson M,Vodkin Lo。内源性,组织特异性短干扰RNAs沉默在甘氨酸Max 种子涂层中的Chalcone合成酶基因家族。植物细胞。2009; 21(10):3063-77。
- 33。
Austin MB, Bowman ME, Ferrer JL, Schroder J, Noel JP。在二苯乙烯合酶中发现的一个醛醇开关介导了III型聚酮合酶的环化特异性。化学杂志。2004;11(9):1179 - 94。
- 34.
易J,Derynck先生,陈L,Dhaubhadel S.差异表达CHS7.和CHS8.基因大豆。足底。2010;231(3):741 - 53年。
- 35.
Li X,Dhaubhadel S.大豆14-3-3基因家族:鉴定和分子表征。Planta。2011; 233(3):569-82。
- 36.
Sepiol CJ,Yu J,Dhaubhadel S.基因组 - 范围内识别Chalcone还原酶大豆的基因家庭:洞察根特异性GMCHR.沙Phytophthora sojae.阻力。植物学报2017;8.:2073。
- 37.
Dastmalchi M,Dhaubhadel S. Soybean Chalcone异构酶:折叠的演变,以及基因家族的差异表达和定位。Planta。2015; 241(2):507-23。
- 38.
大豆异黄酮特异性糖基转移酶和丙二酰基转移酶的鉴定与表征。中国生物医学工程学报。2008;59(4):981-94。
- 39.
MEGA7:用于更大数据集的分子进化遗传学分析7.0版本。生物化学学报。2016;33(7):1870-4。
- 40。
Wang CS, Vodkin LO。从含有大量结合RNA的原花青素的组织中提取RNA。植物学报1994;12(2):132-45。
- 41。
Libault M,Thibivilliers S,Bilgin DD,Radwan O,Benitez M,Clouge SJ,Stacey G.鉴定四种大豆参考基因的基因表达标准化。植物基因组。2008; 1(1):44-54。
- 42。
Earley Kw,Haag Jr,Pontes O,Opper K,Juehne T,Song K,Pikaard CS。植物功能基因组和蛋白质组学的网关兼容载体。工厂J. 2006; 45(4):616-29。
- 43。
荧光融合蛋白在烟草植株中的快速瞬时表达和稳定转化植株的生成。自然Prot。2006;1(4):2019 - 25所示。
致谢
作者感谢Ling Chen,Shaomin Bian,Tim McDowell和Alex Molnar的技术援助。
资金
这项研究是由加拿大农业及农业食品部的ABASE赠款,以支持SD。这些资金的身体已经在研究和收集,分析和解释数据的设计没有任何作用,并以书面稿件。
可用性数据和材料
支持本文结论的数据集包含在文章及其附加文件中。
作者信息
隶属关系
贡献
AKAV收集和分析数据,进行了亚细胞定位实验,准备了稿件草案。KK进行了基因克隆和亚细胞定位实验。GT构建了在亚细胞定位研究中使用的改进的双重记者向量,以及SD构思和设计的实验,分析了数据,写了稿件的最终草案。所有作者阅读并认可的终稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
作者声明他们没有竞争利益。
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
安古拉·瓦迪维尔,克里西亚克,田国华。等等。大豆含有Chalcone合成酶系列的基因组鉴定与定位(甘氨酸最大[l] merr)。BMC植物杂志18,325(2018)。https://doi.org/10.1186/s12870-018-1569-x
已收到:
公认:
发表:
关键词
- Chalcone合成酶
- Isoflavonoid
- 黄酮类动物
- 基因复制
- 基因表达
- 大豆
- 基因家族