摘要
背景
玫瑰是人类的重要植物,具有枢轴经济和生物学性状,如连续开花,花卉建筑,颜色和香味。由于频繁的杂交和高基因组杂合子,玫瑰和他们的亲属的分类仍然是一个很大的挑战。
结果
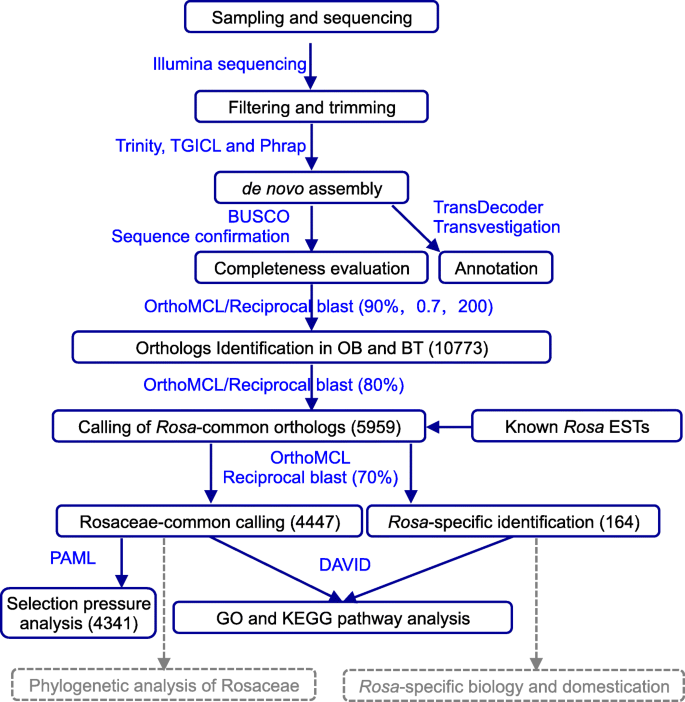
在此,为了识别蔷薇系统发育重建的潜在标记,揭示蔷薇的自然选择模式,我们生成了一套高质量和全面的参考转录组罗莎对《老脸红》(Old Blush, OB)和r . wichuriana'Basye's Thornless'(BT),两种物种表现出对比的高度经济性的特征。组装的参考转录om显示成绩调节N50以上BP。两朵玫瑰共用约10,073份转录物(N50 = 2282 bp),其中一套5959名转录物在属内被保守罗莎.进一步与蔷薇科物种进行比较,发现共有4447个转录本(蔷薇科-共有)罗莎,马鲁斯,李属,rub, 和草莓属,而164名成绩单的游泳池是针对玫瑰的特异性(罗莎特殊技能)。在蔷薇科常见转录本中,有409个转录本表现出阳性选择的特征,并在不同组织中呈聚集表达。有趣的是,这些快速进化的基因中有9个与DNA损伤修复和对环境刺激的反应有关,这可能与杂交后基因组冲突有关。与这种快速进化模式相一致的是,24个F-box和4个TMV抗性蛋白在玫瑰基因中显著富集罗莎特殊的基因。
结论
我们期望这些蔷薇科常见和罗莎特异性转录物应促进蔷薇科植物的系统发育分析以及研究罗莎特殊生物学。这里报告的数据可以提供基本的基因组工具和知识,对理解生物学和玫瑰的驯化和玫瑰育种至关重要。
背景
了解木质植物适应局部环境条件的分子机制仍然是生物学仍然是由于他们的长期和多年生的生命史上的生物学挑战。然而,木质植物代表地球上的大部分生物多样性,涉及草本植物不具有特征的许多不同的酚类特征(https://www.worldwildlife.org).其中一个特征就是玫瑰连续开花的行为,玫瑰是人类社会中具有高度重要性的重要作物。为了保证切花及相关产品的原料供应,连续开花成为玫瑰最重要的生物学和经济性状之一[1].因此,持续调控花期的遗传控制和相关基因调控网络不仅是科学家们多年来的研究课题,也是育种家们多年来的研究课题[1,2].调节连续开花的QTL数量仍然是争议的[1,2,3.].RoKSN,拟南芥的同源物TFL1是唯一已知的负责持续开花的基因[4,5].
耕种玫瑰的驯化主要涉及十几种物种中的杂交[2,6,7,8].频繁的不同人物交叉/回复和玫瑰的多倍化已经使玫瑰的分类非常困难[9,10,11,12].一套高质量和特色的基因组工具/资源是理解现代玫瑰的生物学和驯化所必需的,包括超过30,000种品种[13].最近,已经开发了几个基因图谱群体(参见综述[1,2]),并进行玫瑰基因组的测定[14]序列最近发布了一倍的单倍体罗莎对'旧腮红'[15].然而,由于种间杂交和多倍体化极有可能导致高水平的杂合度,要获得一个准确和完整的玫瑰基因组似乎并不容易。另外,还可以利用不同物种的多个组织构建一个全面的基因表达图谱。
利用含有350个四倍体的微阵列构建了第一套基因表达图谱r .矮牵牛) [16],其后约有4800个选定的无害环境技术(r对,r . wichuriana, 和r .矮牵牛) [17].一个更全面的数据库,包含大约80,714个抄本集群r对“Old Blush”是利用Illumina和454测序平台从13个不同发育阶段或不同非生物和生物胁迫下的组织/器官构建而成的[18].一些最近的研究也对各种罗莎不同目的的物种[19,20.,21,22即使对于单核苷酸多态性(SNP)阵列[23,24].尽管所有这些研究都促进了对玫瑰生物学的理解,但这些转录om的质量通常相对较差,低N50值,完整性差和平均长度短。最近,发表了一倍的单倍体的基因组序列r对“老脸红”为玫瑰研究树立了里程碑[15,25].然而,缺少玫瑰的半基因组信息,其中大多数通常具有频繁的杂交杂交和多倍化引起的高杂合子,可能导致缺乏能力在理解与隐性标记相关的玫瑰族特征的遗传基础时[3.].
为了鉴定支撑蔷薇特性的分子组成,并找到阐明蔷薇科系统发育关系的分子标记,我们为两个蔷薇种生成了一套高质量的参考转录组,其中至少有6对对比性状[3.[,通过在不同的发育阶段测序三个组织,并通过整合公开的数据集。我们鉴定了在Rosaceae植物中保守的约4447份转录物,其中405在显着的选择压力下,只有164个转录物仅在玫瑰中存在。
结果
转录组测序和组装
利用高通量测序技术对茎叶和幼叶的RNA样本进行了分析。1和2).这导致了4.218亿和4.275亿的干净读数罗莎对《老脸红》(Old Blush, OB)和r . wichuriana《巴斯耶的无刺》(Basye’s Thornless) (BT),分别为1).原始序列文件已上载至国家生物技术信息中心序列读取档案(http://www.ncbi.nlm.nih.gov/sra/),注册号码为SAMN07808857-07808870。
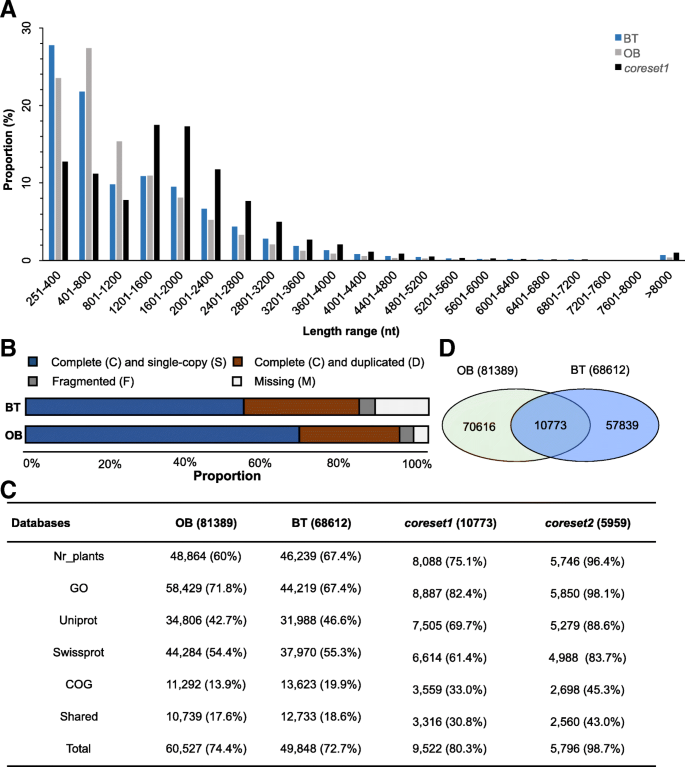
然后我们生成了BT(68,612个转录本)和OB(74,975个转录本)的组装体,其中转录本N50约为2099/1732 bp (BT/OB),平均长度约为1338/1170 bp (BT/OB)。这比之前公布的这两个物种的数据要好得多(见表)2额外的文件1:表S1,附加文件2:表S2;无花果。3.) (18,20.,26,27和其他物种/材料罗莎[19,21,22,28].进一步结合OB的所有数据进行组装,得到了更好的转录本N50 (2092 bp)和平均长度(1359 bp)。将组装的OB转录本与包含45,469个编码基因和4918个非编码基因的基因组组装(v2.0_a1)进行比较[15发现这些转录本几乎100%都可以被绘制出来。另一方面,通过在组装的转录本中寻找包含在基因组组装中的基因,我们在OB组装中鉴定了大约97%的这些基因。各组份的平均GC含量(44.2 ~ 46.4%)与已发表的(45.8 ~ 46.5%)蔷薇具有可比性2).BUSCO分析显示,完整(C)和单拷贝(S)的比例从54.4 ~ 68.8%,完整(C)和重复(D)的比例从24.5 ~ 28.2%。碎片化(F)和缺失(M) BUSCO项目约占5.6-17.4%(图4)。3.b;额外的文件1:表S1)。预测高数量的转录本可能与玫瑰种内/种间杂交和多倍体化的高频率有关。综上所述,这些结果表明组装的BT和OB转录组具有高质量、完整和覆盖率。
玫瑰高质量转录组的组装。一个两种单基因组合的长度分布比例,罗莎对“Old Blush”(OB,灰色条),还有r . wichuriana巴斯耶斯(Basyes)的《无刺》(Thornless)。黑色的条形图标记了两个物种之间共享转录本的长度分布(coreset1;见下面和主要文本)。bBUSCO分析显示了装配和coreset1.c合成的unigenes和核心集年代罗莎.的coreset1是在两个物种之间,而coreset2是共同的基因吗罗莎(见图。2)。对于每个类别(Nr_plants, GO, Uniprot, Swissprot和COG数据库),除了给出比例(括号中)外,还给出了不同数据库中注释的总unigene计数。给出了所有数据库标注的共享和总unigenes。d维恩图显示了结果coreset1识别。在95%的序列同源性水平上,共鉴定出10773个转录本
功能注释
探讨了五个数据库,包括NR,Go,Uniport,Swissprot和Cog数据库,以注释这些组件的功能。大约72.7%(BT)至74.4%(OB)转录物可以用任何一个数据库注释,而只有约17.6%(OB)到18.6%(BT)成绩单,为所有数据库共享注释(表2).有趣的是,大约67.4% (BT)到71.8% (OB)的转录本具有GO注释特征,使GO富集分析在以下步骤中可行。图中包含了详细的标注信息。3.c以及附加文件2:表S2和附加文件3.:图S1。对于两个物种,范畴相关草莓属呈现了最大比例的文本(附加文件2:表S2)。
鉴定在OB和BT之间的保守的正非转录元件组(coreset1),而对于罗莎(coreset2)
为了鉴定所有蔷薇科植物共享的转录本,我们首先鉴定了两个物种之间共享的转录本。采用相对严格的阈值(identity = 90%, minimum length coverage =0.7, minimum alignment length = 200 bp),我们鉴定了两个物种共有的10073个独特转录本(coreset1;无花果。3.D和表2).有趣的是,coreset1显示超过80%的转录本带有注释(图。3.c和附加文件4:表S3)。
接下来我们筛选了所有罗莎具有80%序列一致性的植物。在这个阈值下,共有5959个转录本(N50为2326 bp,平均长度为2161 bp)罗莎物种(coreset2),其中大多数都有很好的注释(98.7%;表格2;无花果。3.c)。
蔷薇科植物的鉴定罗莎您记录
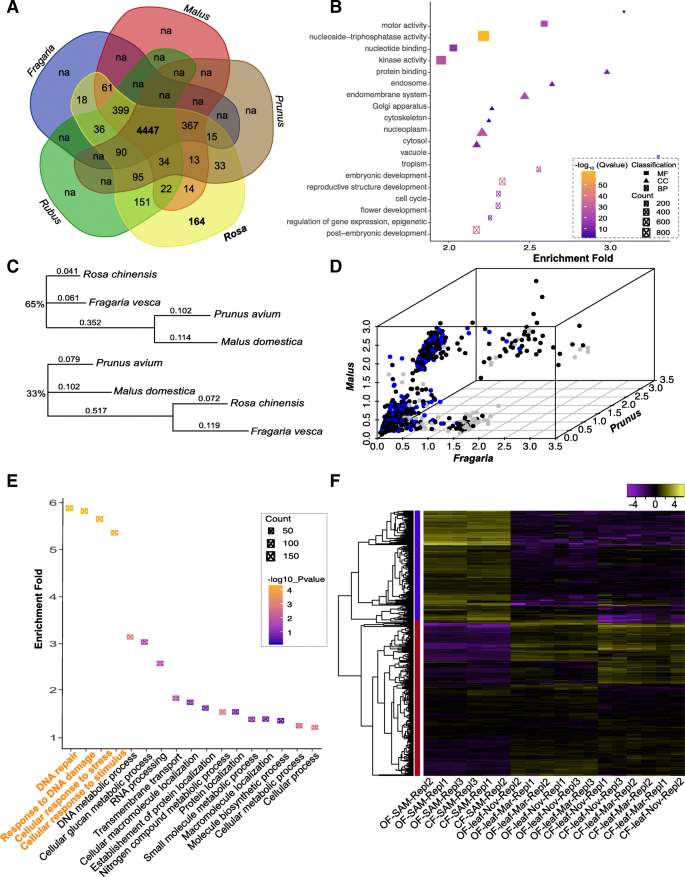
本研究的目的之一是寻找可能用于蔷薇科植物系统发育重建的转录本/标记。然后我们比较了coreset2来自基因的转录物M. domestica,r。orcidentalis,P. Avium,和Fragaria veseca.的近亲罗莎(无花果。4a).在所有五个属中发现了大约4447个转录本(蔷薇科共有;74.6%的coreset2而只有164份转录本是特定的罗莎植物(罗莎-具体的;2.75%coreset2).大多数rosaceae-常见的转录物被注释(4228或约96%;表2和额外的文件5:表S4),并能编码蛋白质(4341;97.6%)。大约504个蔷薇科常见转录本属于1440 BUSCO单拷贝基因(35%;额外的文件5:表S4,附加文件6:表S5)。氧化石墨烯富集试验显示,这些转录本中的大多数都参与了非常重要和基本的功能(图)。4b,额外的文件5:表S4)。系统发育分析表明,大多数Rosaceae-常见的转录物支持聚类p .鸟结核与M. Domestica.(2812或65%;无花果。4c上面板和图。4D黑点)或r对与f . veseca(1436或33%;图。4C下板和图。4d蓝点)(附加文件7:图S2)。
蔷薇科共同潜在编码基因的鉴定与鉴定。一个Venn图显示了蔷薇科 - 常见和罗莎您记录。注意,除了罗莎,未鉴定对其他属的转录物(标有Na)。为此,我们对其他共享集不感兴趣。b致富集分析4447葡萄园常见的成绩单(http://bioinfo.cau.edu.cn/agriGO).与背景相比,X轴显示特定GO术语的丰富折叠。BP,CC和MF平均生物过程,细胞组分和分子功能分别。该区域表示基因计数。c基于4447 rosaceae-常见基因的代表性文学拓扑。上面面板表示大约65%的拓扑(2812)支持聚类李属与马鲁斯,而下面板中的拓扑结构有33%的基因(1436个)支持。树枝上的数字表示距离。d用于4447转录物之间的遗传距离的三维图罗莎和草莓属(轴)/马鲁斯(y轴)/李属(z轴)。黑色和蓝色的点表示支持C中拓扑的基因(黑色表示上面板,蓝色表示下面板),灰色的点表示支持其他拓扑的基因。e409个蔷薇科常见转录本的分布和GO富集分析。y轴表示特定GO项相对于背景的富集倍数。只有4个GO项目被显著富集(用橙色标记)。f聚集的热图比较409所选择的Rosaceae-常见记录的缩放表达值。黄色表示紫色表示更低的表达。蓝色和红色条表示已识别的转录集群中的成员资格
蔷薇科常见转录本的特征
在4447个蔷薇科常见转录本中,有编码潜能的有4341个(97.6%),用于选择压力分析dN(非同义)与dS(同义)变化。马吕斯/李属/草莓属近似序列作为背景(附加文件8:表S6)。该分析确定了409种显着选择的转录物P< 0.05(在Bonferroni纠正).令人惊讶的是,约42%(173)的这些基因在茎尖分生组织(SAM;无花果。4f,蓝色条),而其他基因在两种植物的叶片中表达量均较高,在SAM和叶片材料中表达量均较低(图2)。4F,红条)。这些显着选择的基因显示了DNA修复的富集样品(GO:0006281),对DNA损伤的反应刺激(GO:0006974),对应激(GO:0033554)的细胞反应和对刺激的细胞响应(GO:0051716)(GO:0051716)(图.4e,额外的文件9:表S7)。有趣的是,所有四个GO项目都有相同的9个基因(附加文件10:表S8) [29,30.,31].虽然绝对表达水平不同,但9个基因中有8个在茎尖分生组织材料中表达增加11图S3),表明这些基因可能在玫瑰的发育或环境适应中发挥重要作用。
的特征罗莎您记录
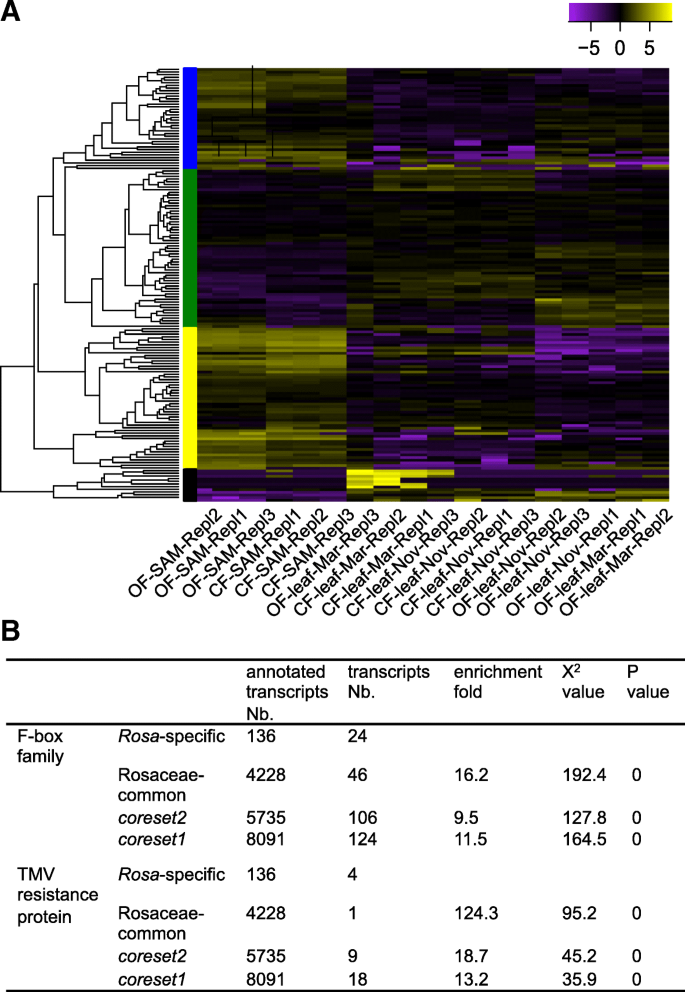
在164罗莎-特异性分子中,147个具有蛋白编码潜能,其中136个具有注释(65个以前未被描述;额外的文件12:表S9)。约72其中罗莎-特异基因在叶片和SAM材料中均有较低水平的表达。5其中约有92个(~ 56.1%)在SAMs中表达量高于叶片材料(图2)。5A,蓝黄条)。无GO项显著富集。然而,假定F-box家族成员的转录本编码强烈富集(24x;无花果。5b;额外的文件12:表S9)。F-box蛋白是真核生物中最大的超家族之一,含有F-box motif,用于识别泛素-蛋白酶体途径中的底物蛋白[32,33].F-box蛋白在细胞周期进程、转录调控、激素信号、程序性细胞死亡和细胞信号转导等生理活动中发挥着关键作用。F-box蛋白可分为三种类型:F-box/kelch-repeat型、F-box/LRR-repeat蛋白和其他F-box蛋白。至少7罗莎实惠的成绩单属于F-Box / LRR重复类型,这可能在自动应力适应方面发挥重要作用(附加文件12:表S9)。巧合的是,四个TMV抗性基因对防御各种植物病毒至关重要[34,35,这些物质中也显著富集罗莎特殊基因(图。5b).这些表明玫瑰可能已经进化出新的防御相关蛋白来对抗其特定的生物胁迫。还需要进一步的实验来研究这些物质的生物学作用罗莎您记录。
识别和描述罗莎您记录。一个热图比较比例表达值为164罗莎您记录。黄色表示紫色表示更低的表达。蓝色,绿色,黄色和黑条表示已识别的转录集群的成员资格。bF-box和TMV抗性蛋白显著富集罗莎您记录。X2在线执行测试(http://www.quantpsy.org/chisq/chisq.htm)通过比较罗莎- 特定的转录数与来自rosaceae-common的数量,coreset1和coreset2基因。P值用Bonferroni校正
讨论
作为最重要的园艺植物之一,罗斯有其特殊的生物学。连续开花,香味,花形,刺和许多没有呈现的特征杨树在玫瑰中还可以找到其他木本模型植物,因此玫瑰现在正成为了解这些特性的分子机制的木本模型物种。有趣的是,现代玫瑰的选育往往涉及品种间频繁的杂交和多倍体化,往往具有更强的抗病性和抗寒性,更好的香味和无刺[36,37,38].另一方面,追踪现代玫瑰驯化和育种的过程和历史仍然是一个挑战[2].
在本报告中,我们生产了高质量的转录组组装r对《老脸红》(Old Blush, OB)和r . wichuriana贝叶斯' Thornless ' (BT),转录本N50大于2 kb,平均长度约1.3 kb。通过合并已发表的OB数据,我们生成了一个平均转录长度超过1.3 kb的改进组装。我们鉴定了10073个高度保守的转录本(coreset1)之间的OB和BT与BUSCO检测,证实了这些组装的高度完整性。作为coreset1转录本基于相对严格的序列一致性(90%),它们可以直接用于评估物种间同源基因的差异表达。
然后探讨这些组件以确定共享的约5959份转录物罗莎sp.最后我们检测到4447个转录本共享于蔷薇科,其中约164个转录本仅存在于蔷薇科罗莎转录组。由于比较选择压力分析为理解形态性状变异和驯化背后的分子信号提供了一种有效的方法[39,40,41,然后我们在这些蔷薇科常见转录本中鉴定了蔷薇中正选择的基因。该分析检测到409个显著筛选的玫瑰基因,其中约40%的基因在SAM组织中高表达,表明这些基因可能在玫瑰幼器官/组织的发育中发挥重要作用。值得注意的是,这些积极选择的基因中有9个属于与DNA修复和应激适应相关的GO项目(图)。4) [29,30.,31,42].这种强富集似乎与生物过程中由于环境条件转换和/或频繁的种内和种间杂交而导致的基因组损伤/冲突的强烈要求有关。rosaceae -公共数据集包含约504个与BUSCO基因集重叠的单拷贝转录本。这些单拷贝转录本可以直接用于阐明蔷薇科的系统发育关系(图。4),这可能是由频繁杂交、快速辐射、多倍体化和驯化引起的挑战[43,44,45,46,47].
与蔷薇科常见的转录本相比,鉴定罗莎-特定的转录本似乎非常有趣。尽管驯化的过程已经有文献记载[2],其进化历史和控制玫瑰特殊性状的分子机制尚不清楚[1].大约有164名转录物中的一半是无声的或没有已知的GO注释。这些罗莎特异性转录物可能与尚未以其他物种特征的表型有关。这些转录物的序列可能显着分歧但仍然存在类似的功能,如拟南芥和玫瑰中的同源物。有趣的是,我们观察到F盒和TMV抗性蛋白质的强烈富集。这些蛋白质可能在涉及玫瑰的生物应力调整的过程中发挥作用。
结论
在本研究中,我们提供了更好的玫瑰转录组组合,并筛选出了可能使玫瑰特殊的基因。此外,我们还鉴定了蔷薇科植物中常见的转录本,这将有助于我们澄清蔷薇科植物的系统发育关系。
方法
植物材料和数据的生成
r对《老脸红》(Old Blush, OB)和r . wichuriana“巴斯耶无刺”(BT)植物在云南省农业科学院花卉研究所(中国云南昆明)的温室中种植。叶材料约4厘米长(从花梗基部到叶尖3.5-4.5厘米,所有小叶刚刚变平;在这个阶段,叶子被认为完全具备光合作用的功能;无花果。1),分别于秋季(2015年11月21日)和春季(2016年3月21日)从开花的OB植株和未开花的BT植株中采集。1).拍摄尖端材料,其中大多数叶片材料被取样于2016年3月21日。为每个发育阶段和每个物种进行至少3个生物重复,组成至少3个单独植物。
用RNAprep Pure Plant Kit(天根,北京)分离总RNA,用poly-T寡聚附着磁珠纯化mRNA(见图)。2对于工作流程)。对于图书馆建设,使用大约1μgmRNA具有RNA完整性数(rin)得分大于8的得分。在Illumina专有碎裂缓冲液中的温度下使用二价阳离子进行碎片。使用Truseq RNA样品制备试剂盒(Illumina,San Diego,Ca,USA)产生测序文库。使用随机寡核苷酸和上标II合成第一链cDNA。随后使用DNA聚合酶I和RNase H进行第二链cDNA合成。通过外切核酸酶/聚合酶活性将剩余的悬垂末端转化为钝端,除去酶。在DNA片段的3'末端腺苷酸化后,将illumina PE衔接子寡核苷酸连接以制备杂交。为了长度选择优选的380bp的cDNA片段,使用AMPure XP系统(Beckman Coulter,Ca,USA)纯化文库片段。在15个循环PCR反应中使用Illumina PCR引物混合物选择性富集两端具有连接衔接子分子的DNA片段。将产品纯化(Ampure XP系统)并在生物分析仪2100系统(Agilent)上使用Agilent高灵敏度DNA测定量化。 Sequencing was carried out on either Illumina NextSeq 500 or Hiseq2000 platform.
数据过滤
所有样本大约生成了105Gb对端数据(BT约52.3Gb, OB约52.8Gb;表格1).OB的最终数据量约为116.1Gb(包括已发布的数据)。其他物种/材料的数据信息见表2.使用FastQC (V0.10.1;http://www.bioinformatics.babraham.ac.uk/projects/fastqc)和Trimmomatic(V0.36; Illuminaclip:Truseq3-Pe.Fa:2:45:10 /领先:10 /尾随:10 /滑动窗口:4:25 / Minlen:48)[48].适配器序列,读取PHRED质量低于92%,PCR重复全部用自定义perl脚本(https://github.com/ckenkel/annotatingtranscriptomes.).所有数据的短读归档(SRA)访问可以在表中找到1.
OB和BT的转录om的组装和功能注释
在组装之前,每个物种的数据被连接(SAM, leaf_Nov和leaf_Mar),并使用Trinity中的in silicon标准化工具将读取丰度归一化为50倍覆盖范围[49]备用时间和最小化内存要求。BT的组装是由本研究生成的数据构建的,而首先使用新生的数据建立两个组件,并稍后与已发布的数据组合(见表1和2).经过滤波和归一化后的数据约为157 Gb,包含约13亿个归一化读对,然后使用优化的参数(Kmer = 2, min_glue = 5, SS_lib RF)在三位一体(r2014_07-17) [49].TGICL工具箱聚集了Trinity组装,以去除已识别的重复[50].使用开放阅读框架(ORF)使用Transdecoder.(https://github.com/TransDecoder/TransDecoder/wiki).hmmer3.用于识别额外的ORFS匹配PFAM-A.域(51].
这些装配体的完整性通过使用1440个近乎通用的单拷贝同源物的RNA模式的Benchmarking Universal Single-Copy Orthologs (BUSCO)策略进行了进一步评估[52].使用定义蛋白质产品的定制流水线对肽水平的每个转录om进行功能注释,并针对多个数据库分配转录名称(图。2).预测蛋白/多肽使用InterProScan5分析[53检索所有可用数据库,包括Gene Ontology (GO:201605)。利用NCBI非冗余蛋白序列(NR)数据库、真核生物同源组(KOG)数据库、KEGG同源组(KO)数据库、Swiss-Prot蛋白数据库、基因本体(GO)数据库和蛋白家族(PFAM)数据库进行BLASTx分析。由此产生的.gff3和.tbl文件进一步注释了转胶中的功能描述符(https://doi.org/10.5281/zenodo.10471).
调用OB和BT之间的保守同源转录元件(coreset1),而对于罗莎(coreset2)
找出两人共享的文字记录罗莎物种,我们确定了coreset1在OB和BT之间使用orthoMCL [54]和优化的反向爆破方法[55]序列同一性为90%,覆盖率高于0.7且超过200bp长度。
来筛选内部共享的记录罗莎(coreset2相比),我们coreset1其他已发表的RNA-seq数据r .野蔷薇,R. Roxburghii.等(见表2为本分析所使用的数据)。序列一致性设置为80%。
蔷薇科的鉴定罗莎您记录
我们进一步比较了coreset2记录到已知的cd马吕斯有明显(v3.0.a1),李属鸟结核(v1.0.a1),悬钩子属植物occidentalis(v1.0.a1),Fragaria veseca.(v4.0),以找出所有蔷薇科中仅在蔷薇科中存在的转录本罗莎植物。在序列一致性为70%的情况下进行反向blast分析,其他参数与coreset1相同。随着coreset2转录本被用来对来自其他植物的CDS进行blast,该分析没有检测到其他属特异性转录本/基因。随后,在超几何检验的基础上对两组转录本进行GO和KEGG Orthology (KO)富集分析(FDR < 0.05)。
蔷薇科常见转录本的选择分析
如前所述,对蔷薇科共有的一组分子中具有编码潜能的4341个转录本进行了选择压力分析[39,40,41].推定的正非基因家蝇,禽亚种和f . veseca作为背景资料,而罗莎转录本被认为是选择分析的前景。对氨基酸进行比对ClustalO(带有默认参数)[56].系统发育分析Fasttree.(−gtrγ;http://tree.bio.ed.ac.uk.无花果树/软件/ /)。的codeml计划PAML软件包被用来估计同义的(dS)和非同义(dN)的比率,根据杨[57].费舍尔的确切测试使用Bonferroni校正来比较H0和H1模型之间的选择压力的重要性。
缩写
- 英国电信(BT):
-
罗莎WichurianaBasye的无刺的
- 车身:
-
基准通用单拷贝Orthologs
- Coreset:
-
保守的同源转录本元素集
- DN:
-
非同义变化
- DS:
-
的变化
- 走:
-
基因本体论
- OB:
-
罗莎对“老脸红”
- RIN:
-
RNA完整性
- 山姆:
-
技条顶端分生组织
参考
- 1.
董旭,姜旭,匡刚,王强,钟敏,金东,胡建宇。木本植物开花时间的遗传控制:玫瑰作为一个新兴模式。植物多样性。2017;39:104-10。
- 2.
Bendahmane M,Dubois A,Raymond O,Bris ML。玫瑰花开发与发展的遗传和基因组学。J Exp Bot。2013; 64(4):847-57。
- 3.
李胜,周宁,周强,严华,简辉,王强,陈明,邱鑫,张辉,王胜,等。继承的永恒在绽放罗莎对'旧腮红'。Horticult Plant J. 2015; 1(2):108-12。
- 4.
Randoux M, Daviere JM, Jeauffre J, Thouroude T, Pierre S, Toualbia Y, Perrotte J, reynnoird JP, james MJ, Oyant LHS等。RoKSN是一种花抑制因子,与RoFD和RoFT形成蛋白复合物,调控玫瑰的营养和生殖发育。新植醇。2014;202(1):161 - 73。
- 5.
iwata H,Gaston A,Remay A,Thouroude T,Jeauffre J,Kawamura K,Oyant LH-S,Araaki T,Denoyes B,Foucher F. TFL1同性恋ksn是玫瑰和草莓连续开花的调节器。工厂J. 2012; 69(1):116-25。
- 6.
de Vries DP, Dubois LAM。玫瑰育种:过去,现在,前景。鲁汶:国际园艺科学学会(ISHS);1996.241 - 8页。
- 7.
Debener T,Linde M.探索复杂装饰基因组:玫瑰作为模型植物。CRIT Rev Plant Sci。2009; 28(4):267-80。
- 8.
Vukosavljev M, Zhang J, Esselink GD, van ' t Westende WPC, Cox P, Visser RGF, Arens P, Smulders MJM。玫瑰的遗传多样性和分化:一个花园玫瑰的视角。Sci Hortic。2013;162:320-32。
- 9.
李丹桂,赵辉,王旭。月季种质资源亲缘关系研究进展。J杂志。2016;33(2):103 - 5。
- 10.
Zhu Z-M,Gao X-F,Fougère-danezan M.罗莎段中的系统发育,基于叶绿体和核标志物的芦荟骨膜和Synstylae(Rosaceae)。mol phylocyet evol。2015; 87:50-64。
- 11.
张立波。野生玫瑰的系统发育和生物地理学,特别是多倍体。安机器人。2015;115(2):275 - 91。
- 12.
Koopman WJ, Volker W,Katrien DC, Johan VH, Jan DR, JH SG, Dirk V, Ben V, M RC, Bert M等。AFLP标记作为重建复杂关系的工具:以蔷薇(蔷薇科)为例。[J] .植物学报。2008;49(3):353-66。
- 13.
凯恩斯T.现代玫瑰纽约:学术出版社;2007.
- 14.
Foucher F, hibrandt - saint Oyant L, Hamama L, Sakr S, Nybom H, Baudino S, Caissard JP, Hokanson SC, Byrne DM, Smulder JMS,等。玫瑰基因组序列及其在研究和育种中的应用。在:Debener T, Linde M,编辑。第六届国际玫瑰研究与栽培研讨会,第1064卷。Leuven 1: Int Soc园艺学;2015.p . 167 - 75。
- 15.
Raymond O,Gouzy J,Just J,Badouin H,Verdenaud M,Lemainque A,Vergne P,Moja S,Choisne N,Pont C,等。ROSA Genome为现代玫瑰的驯化提供了新的见解。NAT Genet。2018; 50:772-77。
- 16.
Guterman I, Shalit M, Menda N, Piestun D, Dafny-Yelin M, Shalev G, Bar E, Davydov O, Ovadis M, Emanuel M,等。玫瑰香味:发现与花香味相关的新基因的基因组学方法。植物细胞。2002;14(10):2325 - 38。
- 17.
杜波依斯,雷梅,雷蒙。6 .李志刚,李志刚,李志刚。蔷薇花发育基因的基因组学研究。植物学报。2011;6(12):e28455
- 18.
Dubois A, Carrere S, Raymond O, Pouvreau B, Cottret L, Roccia A, Onesto J-P, Sakr S, Atanassova R, Baudino S, et al.;玫瑰的转录组数据库资源和基因表达图谱。BMC基因组学。2012;13:638。
- 19.
严华,郝志强,闵超,简华,Baudino S, Caissard JC, Bendahmane M,李松,张涛,周宁。淡紫月季(Rosa chinensis ' pallida ')开花转录组和基因表达分析。基因。2014;540(1):96 - 103。
- 20。
严浩,张辉,王强,邱旭东,张辉,王军,等。月季(Rosa chinensis)。病毒的表型与花器官识别基因的错误表达有关。植物科学学报,2016;
- 21。
张XY,张吉兹,张力,杨t,熊y,che dd。冷应力下ROSA Multiflora的转录组测序和DE Novo分析。Acta physiol植物。2016; 38(7):13。
- 22.
Gao Y, Liu C, Li X, Xu H, Liang Y, Ma N, Fei Z, Gao J, Jiang C-Z, Ma C. a Aux/IAA家族基因RhIAA16在玫瑰花瓣脱落过程中的功能分析。植物科学学报2016;7:1375。
- 23.
Vukosavljev M, Arens P, Voorrips RE, van ' t Westende WPC, Esselink GD, Bourke PM, Cox P, van de Weg WE, Visser RGF, Maliepaard C,等。一个异交和一个自交四倍体花园玫瑰杂交亲本的高密度SNP遗传图谱,利用68k玫瑰SNP阵列从混合后代推断。Horticult杂志2016;3:16052。
- 24.
Koning-Boucoiran CFS,Esselink Gd,Vukosavljev M,Van't Westende WPC,Gitonga VW,Krens Fa,Voorrips Re,Van de Weg We,Schulz D,Debener T,等。使用RNA-SEQ以超过13,000个全长表达基因组装玫瑰转录组,并开发用于玫瑰(ROSA L.)的Wagrhsnp 68k Axiom SnP阵列。前植物SCI。2015; 6:249。
- 25.
Hibrand Saint-Oyant L,Ruttink T,Hamama L,Kirov I,Lakhwani D,周NN,Bourke Pm,Daccord N,Leus L,Schulz D等人。Rosa Chinensis的高质量基因组序列,以阐明装饰性状。Nat植物。2018; 4:1-16。
- 26.
郭X,yu c,罗l,万h,zhen,xu t,谭j,潘h,张q.罗莎中华民族'旧腮红的花卉过渡的转录组。BMC基因组学。2017; 18(1):199。
- 27.
韩y,万H,Cheng T,Wang J,Yang W,Pan H,Zhang Q.罗莎中华民族病毒发育过程中转录组动力学的比较RNA-SEQ分析。SCI REP。2017; 7:43382。
- 28.
关键词:刺梨,抗坏血酸,转录组,EST-SSR标记基因。2015;561(1):54 - 62。
- 29.
Rohde A, Morreel K, Ralph J, Goeminne G, Hostyn V, De Rycke R, Kushnir S, Van Doorsselaere J, Joseleau J- p, Vuylsteke M, et al.; 拟南芥pal1和pal2突变体的分子表型揭示了对苯基丙氨酸、氨基酸和碳水化合物代谢的深远影响。植物细胞。2004;16(10):2749 - 71。
- 30.
Rowan BA, Oldenburg DJ, Bendich AJ。RecA维持拟南芥叶绿体DNA分子的完整性。中国农业科学。2010;61(10):2575-88。
- 31。
通过对拟南芥甲基组沉默突变体的综合分析,揭示了拟南芥甲基组的复杂调控。细胞。2013;152(1):352 - 64。
- 32。
魏超,李华,田敏,余旭,刘东。拟南芥F-box蛋白家族功能的研究进展。博坦西北植物学报,2017;37(11):2300-8。
- 33。
史蒂芬诺维茨K,兰奴N,云顿EJM。植物F-box蛋白-判断生死。植物学报。2015;34(6):523-52。
- 34.
威尔逊TMA。保护作物植物免受病毒的策略 - 病原体衍生的抗性开花。美国国立科学院。1993; 90(8):3134-41。
- 35.
Simon-Mateo C, Garcia JA。基于RNA沉默的植物抗病毒策略。Bba-Gene Regul Mech. 2011;1809(11-12): 722-31。
- 36.
刘春英,王国良,王浩,夏涛,张淑珍,王庆刚,方玉梅。基于rpl16、trnL-F和atpB-rbcL序列对蔷梅属植物的系统发育关系进行了研究。50 Hortscience。2015;(11):1618 - 24。
- 37.
刘义,刘Q.玫瑰遗传资源的评估与利用。j植物类型resour。2004; 5:87-90。
- 38.
邱旭,张华,李树华,简辉,唐凯。云南玫瑰种质资源SSR标记的亲缘关系分析。植物学报2009;29:1764-71。
- 39.
Khan Mr,Hu J-Y,Riss S,He C,Saedler H.MPF2样的疯狂箱基因控制嗜酸剂(Solanaceae)的充气钙霉素综合征:达尔文选择的角色。mol Biol Evol。2009; 26(11):2463-73。
- 40。
彭志鹏,卢勇,李丽,赵强,冯强,高志,陆华,胡涛,姚宁,刘凯,等。速生非用材林毛竹(毛竹属杂环竹)的基因组草案。Nat麝猫。2013;45:456。
- 41。
Koenig D, Jimenez-Gomez JM, Kimura S, Fulop D, Chitwood DH, Headland LR, Kumar R, Covington MF,设计etti UK, Tat AV等。比较转录组学揭示了驯化和野生番茄的选择模式。美国国立科学院。2013; 110 (28): e2655 - 62。
- 42。
李建涛,高永东,谢林,邓超,石鹏,管茂林,黄松,任建林,吴丹东,丁磊,等。异温蛇高海拔适应的比较基因组研究。美国国立科学院。2018; 115(33): 8406 - 11所示。
- 43。
向勇,黄春华,胡勇,文杰,李树华,易涛,陈辉,向俊,马辉。地理时代背景下蔷薇科果实类型的核系统发育。生物化学学报。2017;34(2):262-81。
- 44。
张世东,金建军,陈世勇,Chase MW, Soltis DE, Li H-T, Yang J-B, Li D-Z, Yi T-S。基于质体系统基因组学的晚白垩世以来酒渣鼻科的多样性。新植醇。2017;214(3):1355 - 67。
- 45。
Potter D, Eriksson T, Evans RC, Oh S, Smedmark JEE, Morgan DR, Kerr M, Robertson KR, Arsenault M, Dickinson TA,等。蔷薇科的系统发育与分类。acta botanica sinica(云南植物学报),2012;
- 46.
Campbell CS,Evans Rc,Morgan Dr,Dickinson Ta,Arsenault MP。吡喃吡喃(Pertribe Pylinae)(以前是Maloideae,Rosaceae):有限的分辨率的复杂进化史。植物系统中的Evol。2007; 266(1):119-45。
- 47.
David RM, Douglas ES, Kenneth RR。蔷薇科rbcL序列变异的系统和进化意义。acta botanica sinica(云南植物学报)1994;
- 48.
Bolger Am,Lohse M,Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014; 30(15):2114-20。
- 49.
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q, et al.;没有参考基因组的RNA-Seq数据的全长度转录组组装。Nat生物技术。2011;29(7):644 - 52。
- 50.
黄雪梅,梁飞,黄雪梅,等。tiger基因指数聚类工具(TGICL):一个用于大型EST数据集快速聚类的软件系统。生物信息学。2003;19(5):651 - 2。
- 51。
Mistry J, Finn RD, Eddy SR, Bateman A, Punta M.同源性搜索的挑战:HMMER3和线圈-线圈区域的收敛进化。核酸学报2013;41(12):e121。
- 52。
Simão FA, Waterhouse RM, Ioannidis P, Kriventseva EV, Zdobnov EM. BUSCO:用单拷贝同源性评估基因组组装和注释完整性。生物信息学,2015,31(19):3210 - 2。
- 53。
张海英,张海英,李伟,张海英,等。InterProScan 5:基因组级蛋白质功能分类。生物信息学。2014;30(9):1236 - 40。
- 54。
Li L, Stoeckert CJ, Roos DS。OrthoMCL:真核生物基因组同源组的鉴定。基因组研究》2003;13(9):2178 - 89。
- 55。
Moreno-Hagelsieb G, Latimer k。生物信息学,2008;24(3):319 - 24。
- 56。
Sievers F,Wilm A,Dineen D,Gibson TJ,Karplus K,Li Wz,Lopez R,McWilliam H,Remmert M,Soding J等。快速,可伸缩的高质量蛋白质多序列比对使用簇生ωω。MOL SYST BIOL。2011; 7:6。
- 57。
杨志。PAML 4:最大似然法的系统发育分析。中国生物医学工程学报。2007;24(8):1586-91。
确认
感谢张成军教授和他的同事们访问高性能计算机。
资金
国家自然科学基金项目(no . 31660583, no . 31570311, no . 31501034);中国科学院先锋百人计划项目(no . 292015312D11035);云南省应用基础研究计划项目(no . 2016FB040);云南省科技人才招聘计划项目。本工作在中国野生物种种质资源库的支持下进行。
数据和材料的可用性
支持本研究结果的数据集包括在本文(及其附加文件)中。
作者信息
隶属关系
贡献
JH、XD和SL与DL、SD、SW和KT共同构思了这项研究。SL, MZ, XJ, YS, FC进行了实验并帮助进行了数据分析。MZ、XD、YX和JH分析了数据。JH撰写的手稿由所有作者贡献。所有作者都已阅读并批准了最终稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
附加文件1:
表S1。组件和组件的BUSCO分析coreset1.C、S、D、F、M分别表示完整、完整和单拷贝、完整和重复、片段和缺失BUSCO转录本的比例。(XLSX 128 kb)
附加文件2:
表S2。两种基因型的注释细节。注释过程中使用COG、GO、KEGG、KOG、Pfam、Swissprot、TrEMBL、NR和NT数据库。(XLS 24958 kb)
附加文件3:
图S1。注释结果到NR数据库。一个bt和b for ob。BT和OB的成绩单使用默认参数爆炸到NR数据库,并保持最佳击中。颜色表示每个壁橱种类的注释比例。两种基因型最佳击中的比例最高Fragaria Vesca.(大约75%),而李属mune占有第二高的比例。(PDF 251 kb)
附加文件4:
表S3。KEGG和GO富集分析coreset1转录物。请注意,每个成绩单都可以映射到几个GO术语和KEGG类别。(XLSX 306 KB)
附加文件5:
表S4。4447个蔷薇科共有基因的KEGG和GO聚类信息。(XLSX 274 kb)
额外的文件6:
表S5。蔷薇科常见的单副本成绩单列表。(XLSX 15 kb)
额外的文件7:
图S2。基于4447蔷薇科共有转录本的4种植物系统发育聚类研究。对于每个转录本,构建Neighbor-Joining树。A、B和C的拓扑结构显示了65%和33% (A), 0.7和0.4% (B), 0.5和0.5% (C)的转录本支持的系统发育关系。34个kb (PDF)
额外的文件8:
表S6。蔷薇科常见转录本中阳性选择基因的原始数据。(XLSX 185 kb)
额外的文件9:
表S7。对409个积极选择基因的GO注释。(XLSX 22 kb)
额外的文件10:
表S8。9个go富集显著的正选基因的注释和表达水平。(XLSX 15 kb)
额外的文件12:
表S9。详细信息为164罗莎特殊的基因。(XLSX 20 KB)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
李淑娟,钟敏,董旭。et al。比较转录组学鉴定了玫瑰的选择模式。BMC植物杂志18,371(2018)。https://doi.org/10.1186/s12870-018-1585-x.
已收到:
接受:
发表:
关键字
- 罗莎sp.
- 选择模式
- 比较转录组学
- Rosaceae-common
- 罗莎特殊技能