抽象的
背景
山药(薯蓣属对生活在潮湿和半湿润热带地区的数百万人来说,它们是经济上重要的食物。薯蓣属dumetorum(Kunth)是八种山药中最有营养的一种,通常在西非和中非种植和食用。尽管有这些特性,储存能力D. Dumetorum.受采后块茎严重硬化的限制,可以通过协调一致的育种努力来解决。任何育种计划的第一步必然是研究遗传多样性。本研究采用单核苷酸多态性基因分型法(GBS-SNP),对44份紫穗槐属植物的遗传多样性和群体结构进行了研究D. Dumetorum.在喀麦隆。使用流式细胞术和GBS2倍性推断倍增倍性。
结果
在75%的遗传资源中,平均有6371个位点含有至少信息。基于6457个未连接的SNPs,我们的结果表明D. Dumetorum.被划分为四个群体。我们明确地识别出了一个西部/西北、西部和西南种群,表明海拔高度和农民-消费者偏好是这些种群差异适应和分离的决定性因素。贝叶斯聚类和邻居聚类检测到最高的遗传变异D. Dumetorum.来自西南地区的植物。这种变异可能是由于在该区域的更大的育种努力,如之间的基因流动所示D. Dumetorum.最大似然法推断西南地区的资源。倍性分析显示二倍体和三倍体水平D. Dumetorum.主要是二倍体遗传(77%)。雄性和雌性分别以三倍体(75%)和二倍体(69%)居多。1C基因组大小值为D. Dumetorum.分别平均为0.333±0.009 pg和0.519±0.004 pg,分别用于二倍体和三倍体。
结论
种质表征,人口结构和倍增性是育种计划中的基本信息,以及保护内部多样性。因此,本研究中获得的结果为改善和保护提供了有价值的信息D. Dumetorum..此外,GBS显示为一个有效的强大的工具,以检测种内变异。
背景
山药(薯蓣属构成了潮湿和半湿润热带地区3亿多人的主食。约有600个品种,广泛分布于热带地区[1].薯蓣属dumetorum已之中等八个品种山药最高的营养价值普遍增长,在西非和中非[消耗2].该品种原产于热带非洲和发生在野生和栽培形式。其种植仅限于西非和中非[3.],并在喀麦隆西部普遍。块茎D. Dumetorum.富含蛋白质的(9.6%),具有相当平衡的必需氨基酸,其淀粉易消化[4,5,6].性状,D. Dumetorum.是高产,用40吨的产量/公顷记录在农业电台[7].薯蓣属dumetorum还可识别其药物性质。已经确定了一种新型的生物活性化合物二磷灰石D. Dumetorum.[8],可作为抗糖尿病药物的降糖药使用[9].
尽管有这些特性,储存能力D. Dumetorum.受采后块茎严重硬化的限制,在采后24小时内开始硬化,使其不适合人类食用[2].根据特雷切和德尔珀赫[10],西非的常用储存条件(在Airy仓库,遮阳棚下)储存4个月后造成100%的损失。由于常规但无意中有害反应导致纹理变化的因素的组合,因此由于烹饪质量的组合而表现出烹饪质量的表现出来[11].所以,D. Dumetorum.在其有限的收获期间专门消耗,只有新鲜收获的块茎煮沸并销往消费者。添加更多价值D. Dumetorum.作为一种重要的食物和能量来源,硬化块茎被转化成速溶面粉[12].然而,从硬化块茎直接获得的面粉具有差的感官素质,例如口腔粗糙[4].因此,使用了其他技术,如盐浸泡处理[13]和发酵[14],但硬化现象尚未克服的。因此,分子育种D. Dumetorum.似乎是克服这种现象的合适方法。
遗传多样性的研究是植物育种的重要,早一步。突出这种变化是种质资源调查中的特征的一部分。在我们最近的表型多样性研究D. Dumetorum.我们发现了相对较高的形态特征多样性,表明其潜在的遗传多样性较高[15].实际上,形态特征的表达受到农业气候变化的影响,从而提供有限的遗传信息。因此,不受环境变化的分子标记对于估计遗传多样性是必要的。过去30年的分子标记的发展使研究了多样性和演化以及种质表征[16].在这些标记中,单核苷酸多态性(Single Nucleotide Polymorphisms, SNPs)已经成为最广泛使用的基因分型标记,因为它们在基因组中的丰度不仅允许对种质进行表征,而且还可以量化来自当前生长的品种的各种创始人基因型的祖先的相对比例[16].此外,传统标记如SSRs、RFLPs和aflp的开发是一个昂贵的、迭代的过程,要么涉及耗时的克隆和酶测试,要么涉及不容易并行的引物设计步骤[17].
基因分型测序(GBS)已成为缓解这些限制的一种新方法。该方法已被证明适用于各种生物的群体研究、种质鉴定、遗传改良、性状定位,因此,多个体的SNP发现和基因分型具有成本效益和效率[18].GBS是通过限制性内切酶对样本DNA进行初步消化,降低基因组复杂性,然后进行一轮PCR,生成高通量测序库[19].通过限制酶降低基因组复杂性是快速的,极定的和高度可重复的[19].与其他使用限制性内切酶的类似方法不同,GBS在技术上很简单[20.].此外,生物信息学管道是公开的[21]和GBS可以容易地应用到具有有限的基因组信息的非模型物种[20.].这种方法已经成功用于木薯(木薯耐Crantz) [22],几内亚山药[23]和水山药[24,证明了GBS-SNP基因分型技术是一种适用于山药高通量基因分型的技术。
薯蓣的遗传学是人们了解最少的,而且由于一些生物学限制和研究上的忽视,在主要的粮食作物中仍然很大程度上被忽视[25].在种质鉴定和基因组分析分子标记研究方面取得了一些进展。各种优势分子标记(AFLP, RAPD)已被用于山药,但很少成功(例如,[9])。此外,已经为山药种开发了基因组微卫星标记[24,25,26,27,28,29,30.,31.,32.].然而,目前还没有针对D. Dumetorum.尽管培养山药中的品质,其遗传学是最不名人的。到目前为止,使用SNP基因分型没有任何信息可以评估人口结构,遗传多样性和关系中的关系D. Dumetorum.品种。
影响种群结构和遗传多样性的一个可能的附加因素是多倍性。多倍体在植物育种中有几个优势,如植物器官的增加(“gigas”效应)、缓冲有害突变、增加杂合度和杂种优势(杂交活力)[33.].在山药中,倍增性增加与生长活力,更高和更稳定的块茎产量和对非生物和生物应激的耐受性增加[33.,34.].使用流式细胞仪的最近的研究显示了二倍体和三倍体水平D. Dumetorum.具有二倍体细胞型的主要突出[35.,36.].因此,本研究的目的是了解遗传多样性和人口结构D. Dumetorum.利用基因分型测序(GBS)来研究倍性信息。
方法
植物材料
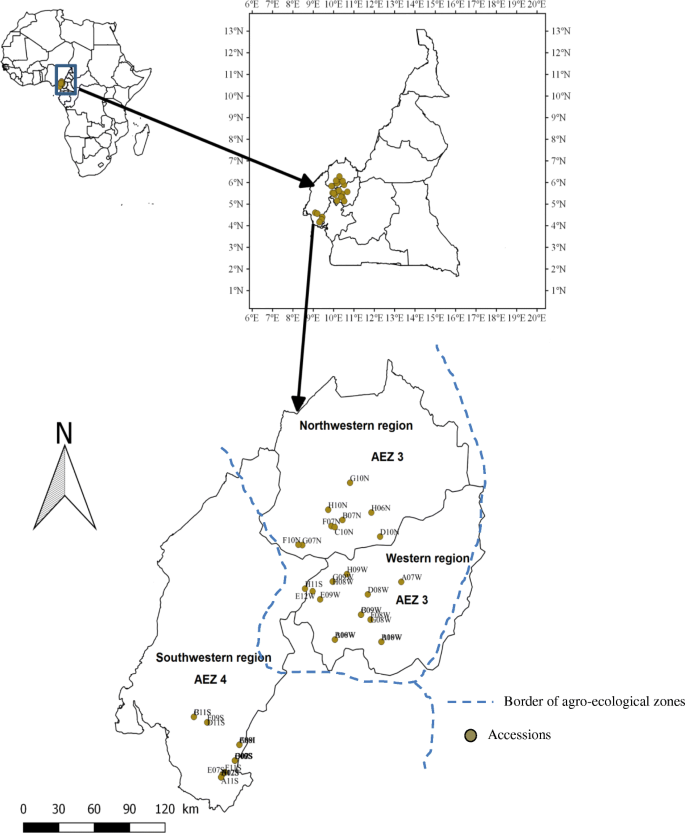
总体而言,44名换乘D. Dumetorum.在本研究中使用(表1).所有这些种质均采自喀麦隆主要山药产区(西部、西南部和西北部)的不同地点,另有3个种质D. Dumetorum.来自尼日利亚补充数据集(图。1).西部和西北部地区属于喀麦隆农业生态区3,西南部至喀麦隆农业生态区4 [38.].大多数这些种质的以前用于形态学特征[15]及硬度评估[39.].在这里,我们选择了一些与块茎相关的性状(图。2).2015年4月,在喀麦隆西部地区(北纬05°20.040′,东经010°22.572 E)的“Ferme Ecole de Bokué”种植了这些山药块茎,经干燥后的幼叶运往德国Oldenburg进行分子分析。基因组DNA提取使用innuPREP植物DNA试剂盒(Analytik Jena, Jena,德国)。
抽样的地图D. Dumetorum.在喀麦隆登记入册。农业生态区(AEZ)的边界根据[38.]
下一代测序库的准备
将每种样品的总共200k个基因组DNA用1单位MSLI(新英格兰Biolabs,NEB)在1×NEB4缓冲液中在30μl体积的37℃下以1×NEB4缓冲液中消化。限制酶通过在80℃下孵育20分钟而灭活。然后,将15μl消化的DNA转移到新的96PR PCR板上,混合并储存在冰上,用3μl的192L2连接适配器(Ovation Rapid Dr Multiplex System,Nugen Technologies,Leek,Netherland),然后用12μL主混合物(4.6μLD1水/6μLL1连接缓冲液混合物/1.5μLL3结扎酶混合物)。将连接反应在25℃下孵育15分钟,然后在65℃下灭活酶10分钟。然后,将20μL套件的套件的最终修复'主混合物加入每个管中,并将反应物在72℃温育3分钟。对于库纯化,将反应用50μLTE10/50(10mM Tris / HCl,50mM EDTA,pH:8.0)稀释,并与80μL磁珠混合,在室温下孵育10分钟并置于5分钟磁铁收集珠子。弃去上清液,将珠子用200μL80%乙醇洗涤两次。将珠子风干10分钟,并在20μLTRIS缓冲液(5mM Tris / HCl,pH 9)中洗脱文库。 Each of the 45 libraries (including one technical repeat) were amplified with 10 μl of the purified restriction product in 20 μl PCR reactions using 4 μl MyTaq (Bioline) 5x buffer, 0.2 μl polymerase and 1 μl (10 pmol/μl) of standard Illumina TrueSeq amplification primers. Cycle number was limited to ten cycles. Then, 5 μl from each of the 48 amplified libraries were pooled. PCR primers and small amplicons were removed by magnetic bead purification using 0.6 volume of beads. The PCR polymerase was removed by an additional purification on Qiagen MinElute Columns. The pooled library was eluted in a final volume of 20 μl Tris buffer (5 mM Tris/HCl, pH 9). The final library pool was sent to LGC genomics (Berlin, Germany) and sequenced on an Illumina NextSeq with 1.5 million 150 bp paired-end reads for each sample. Additional steps at LGC for the sequencing preparation were normalization, reamplification and size selection. Normalization was conducted using Trimmer Kit (Evrogen). For this 1 μg pooled GBS library in 12 μl was mixed with 4 μl 4x hybridization buffer, denatured for 3 min at 98 °C and incubated for 5 h at 68 °C to allow re-association of DNA fragments. 20 μl of 2 x DSN master buffer was added and the samples were incubated for 10 min at 68 °C. One unit of DSN enzyme (1 U/μl) was added and the reaction was incubated for another 30 min. The reaction was terminated by the addition of 20 μl DSN Stop Solution, purified on a Qiagen MinElute Column and eluted in 10 μl Tris Buffer (5 mM Tris/HCl pH 9).The normalized library pools were re-amplified in 100 μl PCR reactions using MyTaq (Bioline). Primer i5-Adaptors were used to include i5-indices into the libraries, allowing parallel sequencing of multiple libraries on the Illumina NextSeq 500 sequencer. Cycle number was limited to 14 cycles. The nGBS libraries were size selected using Blue Pippin, followed by a second size selection on a LMP-Agarose gel, removing fragments smaller than 300 bp and those larger than 400 bp. Libraries were sequenced on an Illumina NextSeq 500 using Illumina V2 Chemistry.
GBS数据分析
使用Eaton和Ree开发的定制软件管道iPyrad(版本:0.7.19和0.7.28)分析GBS数据[21]对于人群遗传和系统发育研究。它包括解复用和质量过滤的七个步骤,群集基因座与共识对齐和SNP与SNP滤波调用到最终SNP矩阵,可以转移成各种输出格式。我们分别进行了多路分解和QC,以检索FASTQ序列作为IPyrad的输入。为每个序列修剪限制性位点和条形码,将质量得分小于捕获20的碱基被改变为N的N,丢弃超过5%的序列。Ipyrad的第3步在我们的de-novo snp分析vsearch [40用于消除复制和配对合并读取和对每个样品聚类读取与85%的序列相似性的推定基因座。假定位点的共有序列的比对与肌肉建[41.].在估计测序误差(Π)和杂合度(ɛ)后,利用这些估计参数估计一致等位基因,并记录等位基因数量。结果一致等位基因再次与VSEARCH聚类,并与MUSCLE对齐。在75%以上的样本中观察到碱基SNPs,在50%的样本中不超过20个SNPs,有8个插入位点和杂合子位点,但所有样本都被处理为二倍体,因此每个多态位点有两个单倍型。
系统发育推断
使用SpletStree中的邻近网络(版本4.14.6)生成一个大型树[42.基于]关于级联GBS数据。要控制引进三倍体种质与否影响到我们的系统发育分析,我们构建树状图有和无三倍体种质。
历史关系(TreeMix)
历史关系D. Dumetorum.通过TreeMix (version 1.13)中实现的最大似然法评估包括可能的基因流动事件的入库[43.].TreeMix基于基因组数据的等位基因频率重建了可能的种群间迁移。它使用了一种既允许种群分裂又允许基因流动的方法。我们将总体参数定义为0,因为我们是在个体层面上工作的。在25541个SNPs位点中,筛选157个SNPs,得到无间隙矩阵,用于确定各位点之间的关系。该树的构建具有1000个引导复制的可信度,并使用toytree(版本0.1.4)和toyplot(版本0.16.0)进行可视化。
人口结构分析
使用structure软件对种群结构进行分析[44.]及MavericK [45.].Structure使用基于贝叶斯模型的聚类方法和启发式方法进行估计,而MavericK使用一种称为热力学积分(TI)的计算技术。然而,这两个程序中的混合建模框架是相同的[45.].使用跨越10个复制(K为2至5个)的采样未链接的SNP(每次IPyrad-Cluster的SNP的一个随机选择的SNP),在结构中进行分析。运行了10,000次迭代和100,000马尔可夫链蒙特卡罗(MCMC)复制的烧坏。使用Evanno的方法检测到真正的簇(k)[46.]在结构收割机中实施[47.].Maverick的MCMC实现略微不同,尽管假定的核心模型与结构中使用的核心模型相同[45.].因此,在2000次迭代和10,000次MCMC的老化周期中,运行了跨越5个重复(K为2到5)的混合模型。在默认设置的K(2 ~ 5)范围内,25个TI梯级检测到最佳K值。
倍增性/基因组大小估计
每次加入,约1厘米2在含有1.1 mL冰点Otto I缓冲液(0.1 M柠檬酸一水和5% Triton X-100)的培养皿中,用剃须刀片与标准叶片共同切碎。我们使用Solanum lycopersicum.L. ' Stupicke ' (1C = 0.98 pg;[48.]作为内部标准。切碎的材料和缓冲液通过Cell-Tric 30 μm滤器过滤到塑料管中,加入50 μL RNase。在37℃水浴中孵育30分钟后,将450 μL的溶液转移到另一管中,其中2 mL Otto II(碘化丙啶+钠)2HPO4)补充道。将此溶液置于4°C 1 h。使用CyFlow流式细胞仪(Partec GmbH, Münster, Germany)对样品进行分析。对于每个添加,测量3个重复,包含5000个计数。我们测量了44个基因组中的17个D. Dumetorum.由于某些丢失而导致的进入,其中已识别性别。使用R包GBS2PLOIDY评估剩余种植(27)的倍增水平[49.].该方法根据每个个体在变异呼叫过程中所识别的杂合子SNPs的等位基因比率推断细胞类型。数据通过获取a *来准备。使用VCFConverter2.py从iPyrad的所有样本的vcf输出文件(https://github.com/dandewaters/VCF-File-Converter)如[50.].通过两种方法估计细胞类型:1)不参考已知倍性的材料,2)参考17份已知倍性的材料,从流式细胞术中以三倍体和二倍体为一组来估计剩余的27份材料。
结果
GBS数据分析总结
我们平均生成了220万次原始读取D. Dumetorum.Illumina测序分析2).过滤后,我们平均获得1.3×104读取集群为85%,平均每次读取深度为53。杂合度的最大似然平均估计(ɛ= 1.1 × 10- 2)大于序列错误率(Π = 6 × 10- 3.).共有序列被称为对于每个群集,得到平均32532每加入读取。我们记录了平均6371种质中至少75%的基因位点恢复。加入D09S有丢失数据的显着比例较高。
系统发育推断
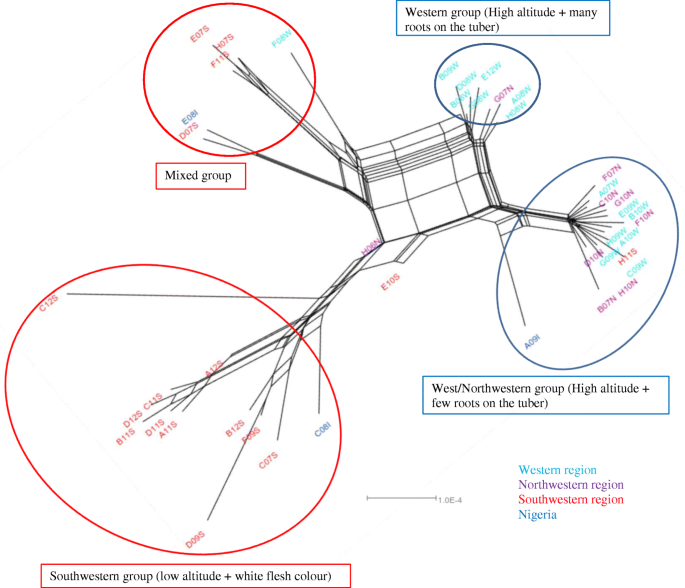
无根的邻居网集群的44个种质D. Dumetorum.分为四组:西部/西北组,西部组,西南组和混合组(图。3.).然而,E10S和H06N在这些组中不聚类。三倍体加入没有影响网络的拓扑结构(附加文件1:图S1)。
西部/西北部组有16种,88%来自西北部和西北地区(50%来自西部,距西北50%)。剩余的豁免(12%)来自西南地区(H11s)和尼日利亚(A09i)。在这一组中,除了块茎上的黄色肉颜色,块茎几个根部,均为0.09i以外的高海拔地区。在这里,除了来自尼日利亚的A09i之外,所有的牧区都会硬化。
西方组织包括八个豁免;几乎所有人都来自西部地区,其中一个来自西北部(G07 N)。该小组由带有黄色肉颜色和块茎的许多根源构成。它们来自高海拔地区,并在收获后硬化。西方团体与西北部小组密切相关,并在块茎上的根部数量不同。
除尼日利亚的C08I除外,西南部集团始于西南部地区。与西北部和西部小组和西方团体不同,所有换乘来自低空地区,都有白色肉体颜色。然而,收获后所有豁免都硬化。第四组是一个由六名加入的混合组,其中四个是来自西南部,一个来自西部(F08 W)和来自尼日利亚(E08I)的群体。与其他人相比,该组是关于块茎字符的变化。再次,收获后,所有牧区都会变硬。
人口结构
我们确定了的人口结构D. Dumetorum.使用贝叶斯方法和热力学积分(TI),分别在STRUCTURE和MavericK中实现。STRUCTURE和MavericK的结果表明D. Dumetorum.种质可聚成群体。The delta K (∆K) of Evanno’s method and TI estimator of the evidence for K showed strong peaks at K = 4 and K = 2 respectively (Additional file2:图S2)。K值(K = 4)是最可能的种群数量(图。4),因为邻近净方法也支持四组的存在(图。3.).总共,33个样本(75%)被分配到前三个种群中的一个,至少60%的推断祖先来自这三个种群中的一个。第四组人口中至少有60人的推断祖先没有归属。P1、P2和P3群体分别有16个、8个和9个物种。其余(11份)均为种群间混合的结果。
在人口P1中,除了加入A09I(尼日利亚)和H11s(西南部地区)之外的西北部和西北地区。这里,将三个载体分配100%至P1,12个作为P1和P4之间的混合物,一个版本A09I作为P1XP2XP3XP4的混合物。相比之下,除H06N(西北)外,人口P2的所有含义都来自南方区域。将四个载体分配100%至P2,两种载体作为掺合P2xP4,而两个均为掺合P1XP2XP4和P1XP2。关于P3,几乎所有的换乘(8)都来自西部地区,除西北地区G07 N之外。相反,没有将100%分配给P3的加入。五个被分配为混合P1XP3,三个分类为P1XP2XP3和一个作为P1XP2XP3XP4。此外,人口结构不会随着k = 5的增加而变化(附加文件3.:图S3)。比较从邻居网结构分析的结果,我们发现一般类似的结果。因此,P1对应于西/西北人口,P2到西南人口和P3西部人口。不属于P4种质进行了鉴定。
倍增性/基因组大小估计
我们发现,13(76%)的种质D. Dumetorum.是二倍体(2x)和四(24%)为三倍体(3×)(表3.).的1C基因组大小值D. Dumetorum.二倍体为0.333±0.009 pg,三倍体为0.519±0.004 pg。所有测试的标准变异系数(CV)均小于5%(附加文件)4:表S1)。通过与性别的比较,我们发现二倍体遗传中女性占69%,男性占31%。在三倍体材料中,雄性占75%,雌性占25%。就地理来源而言,所有三倍体材料均来自西南地区。
使用与已知倍性(17)种质将R包gbs2ploidy,我们评估了在我们的GBS数据的gbs2ploidy灵敏度。流式细胞术和gbs2ploidy之间同意的概率为35%,与被分配给相对细胞型和三个(A09I,B09W,E08I)是不确定的17 8的种质。正确二倍体和三倍体指配的概率为38和25%之间。参照种质从流式细胞仪上的剩余种质(27)训练gbs2ploidy,我们发现,21(78%)份材料二倍体并用74%和73%分别平均分配概率,6(22%)三倍体。对于二倍体加入,七,五和九个能分别起源于西部,东北,西部和西南部地区。对于三倍体,3人来自西北,两名来自西方,一个来自西南地区。总之,34个种质D. Dumetorum.(77%)为二倍体(2x), 10(23%)为三倍体(3x)。三倍体材料主要来源于西南地区(70%)。
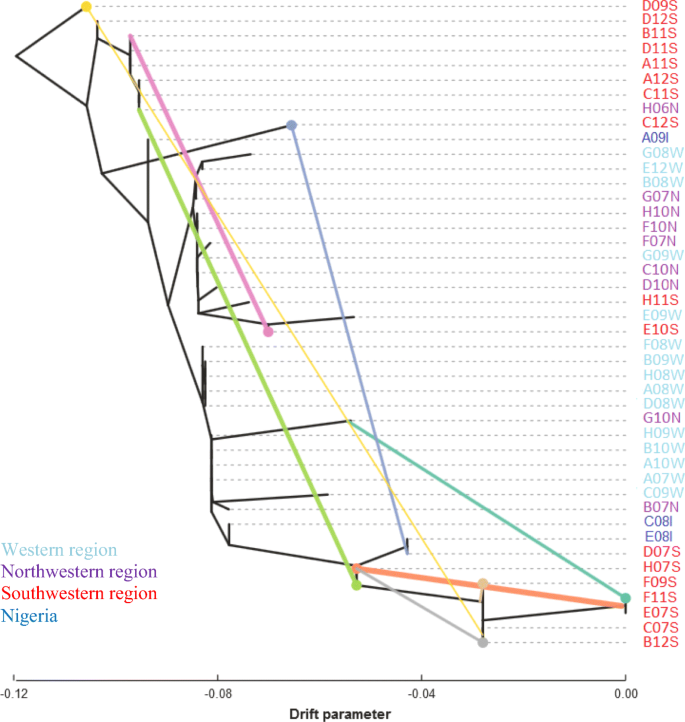
加入之间的历史关系
我们使用Treemix以确定分裂和基因流动D. Dumetorum.加入。我们构建的树没有迁移和十个迁移事件之间允许。我们发现之间的八个基因流事件D. Dumetorum.accessions(图。5).尽管有9个迁移事件的树可能是最高的(但几乎类似于8个迁移),但我们选择了有8个事件的树,因为第9个迁移是冗余的(附加文件5).迁移事件涉及西南部地区的十一种加入,从西北部地区和两(G10 N和H06N)。我们没有找到涉及A08的迁移事件,在收获后不会淬火,以及源自西部地区和尼日利亚的过程。C12s(2x,少量根和白肉)可能是D07s(2x,雌性,少量根和白色肉)和D09s(3x,雄性,少量根和白色肉体)或其祖先之间的基因流动的结果;C07s(3x,雄性,少数根和白色肉)和E07s(2x,雄性,许多根和黄色肉)可能是H06N(2x,少数根和黄色肉)和H07s(2x,男性,2次)之间引入的结果许多根和黄色肉体)。此外,允许迁移与没有迁移事件的树相比改变了树的拓扑结构(附加文件6:图S4)。
讨论
基因分型逐序列是一种创新,强大,经济效益的方法,允许在一个库中复用个体,以在各种各样的物种中产生数千万的SNP [51.].在我们的研究中,我们平均识别了每次加入30,698读。过滤后,避免缺失数据的效果,保留5054个基因座进行分析。总共调查了26,325个SNP。这些数字类似于在另一种非模型物种中使用相同管道的先前研究[21].
大邻居净树(图。3.)集群D. Dumetorum.种质分为四组:A西部/西北部组,一组西,南,西部组和混合组。西部和西北部属于农业生态区III(西方高地)和西南属于农业生态区IV。这与使用形态特征[先前的结果结果如有异议15],没有明确的分离D. Dumetorum.按农业生态区分类。然而,形态学标记受环境条件的影响,因此提供的遗传信息有限。此外,Sonibare等人[9使用AFLPD. Dumetorum.来自三个国家种质根据收集方面并没有找到一个明确的分离。然而,SNP标记物是与用于分析的最丰富的在基因组中合适的就广泛的基因组尺度[52.,53.].结合高通量测序,使用GBS生成成千上万的SNP [54.相比AFLP]允许评估更有效的遗传多样性。这已经由萨斯基等建议。[24他指出,GBS是山药高通量基因分型的有力工具。
我们基于STRUCTURE的分配测试结果也是分离的D. Dumetorum.4个种群,其中3个已明确确定,即西部/西北人口,西部人口和西南人口。相反,MavericK透露D. Dumetorum.按照已知的农业生态区分为两个种群(附加文件2,图S2)。然而,研究的基因座数量大(超过数百个基因座)。在这种情况下,结构中实现的启发式近似似乎更好[45.].西部和西北地区所有品种的块茎果肉颜色均为黄色,而西南地区大部分品种的块茎果肉颜色为白色。研究结果表明,海拔高度和农民-消费者偏好之间存在一定的障碍D. Dumetorum.人口。事实上,AEZ 3对应于覆盖西北地区的西部高地。它的特点是高海拔(1000-2740米),低温(年平均19°C)和年降雨量为1500至2000毫米。相比之下,AEZ 4主要包括涵盖南方西南和沿海地区的潮湿林。它的特点是低空(除了几山外<700米),年降雨量为2500至4000毫米,平均温度为26°C [38.].喀麦隆的所有三个区域都属于山药带,那里的物种发生在野生和栽培形式。尽管如此,它的起源的中心仍然是未知的,到目前为止不包括人口在喀麦隆分离的起源的解释。块茎品质是农民和消费者[采纳的山药品种的重要标准55.].因此,西部/西北和西南地区块茎肉色的差异可以解释为这些地区消费者偏好的不同,这也取决于山药的食物形式。在西部和西北部地区,山药块茎几乎只作为煮熟的块茎来食用,而在西南部地区,山药块茎可以煮熟或捣碎食用。喀麦隆的消费者可能更喜欢煮熟的黄色块茎和捣碎的白色块茎。的确,Egesi等人[56.证明肉的颜色决定了人们对煮山药或捣碎山药的偏好d . alata.假设白肉是基于其其他山药种类的主要发生的祖传人物状态,我们认为黄色肉颜色已经发展了几次(可能四次),因为它存在于我们的四组推断中,但随后的单一起源无法排除内部杂交或损失。山药有许多根源的可能在西部地区发展,可能是由于高地的环境条件,偶尔水的稀缺。根系系统具有营养和吸水性的重要生理功能。众所周知,若干根系特征被认为是在干旱胁迫下维持植物生产率的重要性[57.].与黄肉的颜色和许多根在西南地区(混合组)的块茎突变的发生可能是由该地区的遗传多样性种质的人工杂交造成的。
基因流动在我们的四个主要群体内和之间的重要性D. Dumetorum.从掺合料的高比例可以看出。这种看法可以用过去在喀麦隆,特别是在西南部为改进所作的努力来解释D. Dumetorum.[7].事实上,遗传多样性可以通过育种活动来增加[58.].特别值得注意的是,第四个组的所有个体都被混合在一起,这表明喀麦隆没有属于这个组的基因上明确的归属(图。3.).这一群体的基因明确的个体可能不是在喀麦隆采集的,也可能已经灭绝了,但我们更倾向于假设,这些植物起源于尼日利亚。这一发现进一步证实了两者之间的密切关系D. Dumetorum.尼日利亚和喀麦隆的进入。喀麦隆西南和西北地区与尼日利亚共同分享了一个共同的边界。交流D. Dumetorum.边境两侧的农民之间的进入是众所周知的,提供基因流动和杂交。的确,Sonibare等。[9报道了介绍D. Dumetorum.中非国家的种质受到尼日利亚农民活动的影响。
在我们的研究中获得的Treemix结果还表明,西南部地区的牧区之间存在更多的基因流,而不是西部/西北部地区。这些发现支持上面讨论的结构的混合物结果,并允许改进我们对过去交叉的基因型的理解。然而,关于具有非前季度硬化的样品,我们没有检测到任何基因流动。这表明在喀麦隆的任何繁殖中,还没有使用样品,并且尚未受到限制D. Dumetorum.到尼日利亚。因此,需要对涉及物种分布范围的样品的遗传多样性进行更广泛的研究,以跟踪该样本的这种特征和血统的起源。
倍增性是可能与人口结构和育种导致杂化活力(杂种优势)和有害突变缓冲的另一个因素。我们的分析显示77%的D. Dumetorum.材料为二倍体,23%为三倍体。这一结果与之前的研究结果大致一致,其中83%是二倍体,17%是三倍体[36.]及60%二倍体及40%三倍体[37.].然而,流式细胞术和gbs2倍体的一致性的概率很低(35%)。事实上,gbs2倍性方法的一个局限性是覆盖率低,特别是当物种可能的倍性水平未知时[49.].作者报告说,这个问题可以通过在分析中加入已知细胞类型的有效参考样本来解决,就像我们在研究中做的那样。
性和倍性之间的关联显示了用于女性牧草的三倍体的三倍体和女性accive。这些发现部分与Adaramola等人进行了矛盾。[37.其中,二倍体的雄性遗传优势已被报道。然而,Adaramola等人[37.]概述了确保相等数量的更系统的抽样方法D. Dumetorum.加入可能会改变他们的结果,我们的研究就是这样。1C基因组大小值为D. Dumetorum.分别为10.33至0.52 pg分别为二倍体和三倍体的平均值。这支持Obidiegwu等人的结果。[36.],谁发现有五个二倍体和三倍体一个的1C基因组D. Dumetorum.无性系为0.35 ~ 0.53 pg。因此,D. Dumetorum.似乎有一个非常小的基因组(1c值≤1.4 pg),以下类别[59.].TreeMix结果表明不同倍性之间的一些种质的混合物。三倍体种质可以是三倍体(3×)或二倍体(2×)雄性与二倍体(2×)女性之间的一个可能的混合的结果,虽然种质H06N的性别和C12S尚未确定。类似的结果报告中d . alata[60.].这表明三倍体遗传发生在D. Dumetorum.很可能是由于未减数(2N)配子的花粉,而不是卵细胞的参与。这是由三倍体的人工杂交(3次)的男性和二倍体(2X),女,我们在现场进行(Siadjeu未公布的数据,其他文件证实7:图S5)。最后,西南地区三倍体遗传的优势发生与该地区更密集的育种计划相吻合,这可能是由于已知的一个物种的遗传多样性的遗传之间的杂交可以增加未减少配子的数量[61.].
结论
在这项研究中,我们报道了人口结构,遗传多样性和套数/基因组大小D. Dumetorum.在喀麦隆使用GBS。我们证明了D. Dumetorum.是按人口结构划分的。有很高的遗传变异性D. Dumetorum.在喀麦隆登记入册。我们发现了种内杂交,并提供了有关倍性/基因组大小的有用信息D. Dumetorum..所有这些信息都与保护和繁殖计划有关D. Dumetorum..然而,我们没有推断出样品与采后硬化之间的牢固关系,这是未来育种工作中最重要的特性,这意味着需要在西非和中非对这一特性进行广泛的研究,以阐明其起源。最后,GBS作为一种有效的研究番薯系统地理的工具出现了。
缩写
- AEZ:
-
农业生态区域
- AFLP:
-
扩增片段长度多态性
- 简历:
-
变异系数
- EDTA:
-
Ethylenediaminetetra-acetic酸
- GBS:
-
基因分型,通过测序
- 密度:
-
马尔可夫链蒙特卡罗
- 病人:
-
人口
- PCR:
-
聚合酶链反应
- RAPD:
-
随机扩增多态性DNA
- RFLP:
-
限制性片段长度多态性
- SNP:
-
单核苷酸多态性
- 苏维埃社会主义共和国:
-
单重复序列
- TI:
-
热力学整合
参考文献
- 1.
王志强,王志强,王志强,等。薯蓣核Xdh分析(薯蓣属,薯蓣科)与塑植树的一致性揭示了一种新的新媒体谱系。Bot J Linn Soc。2018;在新闻。
- 2.
Sefa-Dedeh S, Afoakwa EO。三叶山药的生化和结构变化薯蓣属dumetorum块茎收获之后。食品化学。2002;79:27-40。
- 3.
降级L.山药:热带根系作物。山药一个。根作作物。伦敦:Macmilan Press Ltd .;1993年。
- 4.
Medoua GN,Mbome IL,Agbor-EGBE T,Mbofung CMF。枳,山药采后硬化期间发生的物理化学变化(薯蓣属dumetorum块茎。食品化学。2005;90:597 - 601。
- 5。
Mbome LAPE我,山药Treche S.营养品质(薯蓣属dumetorum和D rotundata)用来喂养老鼠的面粉。中国农业科学。1994;66:447-55。
- 6。
Afoakwa EO, Sefa-Dedeh S.化学成分和质量变化薯蓣属dumetorum收获后PAX块茎。食品化学。2001; 75:85-91。
- 7。
Treche S. Potentialités nutritionnelles des ignames (Dioscorea spp.) cultivées au Cameroun。《奥尔斯托姆CE和T版》,编辑。巴黎;1989.
- 8。
iwu mm,okunji c,阿卡普,Corley d,Tempesta S.来自块茎的二十次的低血糖活性薯蓣属dumetorum在正常和四氧嘧啶糖尿病兔。Planta Med。1990; 56:264-7。
- 9。
Sonibare MA, Asiedu R, Albach DC。遗传多样性的薯蓣属dumetorum(Kunth)大同使用扩增片段长度多态性(AFLP)和叶绿体。生物学习系统系统。2010; 38:320-34。
- 10.
Treche S,Delpeuch F.证据膜增厚的块茎的实质发展薯蓣属dumetorum在存储。巴黎学院1979;288:67-70。
- 11.
Medoua GN。结核的营养与技术潜力薯蓣属dumetorum(kunth)。喀麦隆:博士论文,Ngaoundere大学;2005年。
- 12.
营养价值的变化和产品的准备工作的变化是一种栽培。Rev Sci Tech (Sci Santé)。1984:7-22。
- 13.
Medoua GN, mome IL, Egbe TA, Mbofung CMF。盐渍处理改善三叶山药的结构和功能特性(薯蓣属dumetorum)硬化块茎。J Food Sci。2007; 72:E464-E469。
- 14。
Medoua GN,Mbome IL,Agbor-EGBE T,Mbofung CMF。发酵对枳山药的一些质量特性的影响(薯蓣属dumetorum)硬化块茎。食品化学。2008; 107:1180-6。
- 15.
Siadjeu C,Mahbousomotoukam G,Bell JM,NKWATE S.甜玉米遗传多样性薯蓣属dumetorum(kunth)pax在喀麦隆两种农业生态区域中的形态特征揭示。非洲J Biotechnol。2015; 14:781-93。
- 16。
德尚S, Llaca V, May GD。Genotyping-by-sequencing于植物。生物学(巴塞尔)。2012; 1:460 - 83。
- 17。
Davey JW,Hohenlohe Pa,Etter Pd,Boone JQ,Catchen JM,Blaxter ML。基因组遗传标记使用下一代测序发现和基因分型。NAT Rev Genet。2011; 12:499-510。
- 18。
De Donato M, Peters SO, Mitchell SE, Hussain T, Imumorin IG。基因分型-测序(GBS):一种新型、高效、经济的牛基因分型方法。《公共科学图书馆•综合》。2013;8:1-8。
- 19。
Elshire RJ, Glaubitz JC, Sun Q, Poland JA, Kawamoto K, Buckler ES, et al.;一个强大的,简单的基因分型测序(GBS)方法的高多样性物种。《公共科学图书馆•综合》。2011;6:1-10。
- 20。
Wickland DP,Battu G,Hudson Ka,Diers Bw,Hudson Me。低覆盖作物数据集对基因分序分析方法的比较显示了新的工作流程,GB的优点。BMC生物信息学。2017; 18:586。
- 21.
eeton DAR, Ree RH。利用RADseq数据推断系统发育和渐深:以开花植物为例(马先蒿:列当科).《公共科学图书馆•综合》。2013;62:689 - 706。
- 22.
拉比IY,库拉克PA,马努 - Aduening JA,Dankyi AA,Asibuo JY,帕克斯EY等。跟踪使用基因分型,通过测序标记的作物品种:使用木薯案例研究(木薯耐Crantz)。BMC Genet。2015; 16:1-11。
- 23.
基于基因分型、流式细胞术和表型分析的下一代测序技术研究几内亚山药的多样性和进化。Theor Appl Genet. 2014;127。
- 24.
Saski Ca,Bhattacharjee R,Scheffler,Asiedu R.水山药的基因组资源(参薯L.):estsequences,de novo测序和gbs库分析。Plos一个。2015; 10:1-14。
- 25.
Mignouna HD,Abang MM,Asiedu R.利用热带块茎作物改进的现代生物技术:山药(薯蓣属spp)分子育种。非洲生物技术学报2003;2:475-85。
- 26。
Terauchi R,Konuma A.微卫星多态性萆山老,一个野生山药物种-基因组。基因组。1994;37:794 - 801。
- 27。
Mizuki I,Tani N,Ishida K,Tsumura Y.克隆植物微卫星标志物的开发和表征,Dioscorea japonica研究。摩尔生态学笔记2005;5:721-3。
- 28。
Tostain S, Scarcelli N, Brottier P, Marchand JL, Pham JL, Noyer JL。热带山药DNA微卫星标记的发展(薯蓣属SP。)。Mol Ecol Notes。2006; 6:173-5。
- 29。
Hochu I,Santoni S,Bousalem M.热带美国山药中微卫星DNA基因座的分离,表征和跨物种扩增Dioscorea Trifida..Mol Ecol Notes. 2006; 6:137-40。
- 30.
Siqueira MVBM, Marconi TG, Bonatelli ML, Zucchi MI, Veasey EA.新的水山药微卫星位点(参薯,薯蓣科)和其他的交叉放大薯蓣属物种。我是J机器人。2011; 98:E144-E146。
- 31。
席尔瓦LRG,Bajay MM,蒙泰罗男,Mezette TF,纳西门托WF,祖奇MI,皮涅罗JB,Veasey EA。分离和鉴定微的山药Dioscorea Cayenensis.(薯蓣科)和交叉扩增D. Rotundata..genet mol res。2014; 13(2):2766-71。
- 32。
Tamiru男,山中S,光冈C,巴比P,高木H,洛佩兹蒙达斯A,等人。基因组简单重复序列标记山药发展。农作物科学。2015; 55:2191-200。
- 33.
Sattler MC, Carvalho CR, Clarindo WR。多倍体及其在植物育种中的关键作用。足底。2016;243:281 - 96。
- 34.
(1)大山药的遗传多样性(参薯L.)和相关性D. nummularia我是。和d . transversaBr。AFLP标记显示。植物资源学报。2005;52:919-29。
- 35.
Lebot V.热带根和块茎作物。在:编辑器Wh。油,植物生长作物刺激。encyclopei。牛津:Eloss出版商;2010. p。9。
- 36.
Obidiegwu Je,Rodriguez e,Loureiro J,Muoneke Co,Asiedu R.在某些代表性中核DNA含量的估算薯蓣属.Sci Res Easay. 2009; 4:448-52。
- 37.
Adaramola TF, Sonibare MA, Sartie A, Lopez-Montes A, Franco J, Albach DC。结合倍性水平、次生代谢产物和形态性状分析确定三叶山药(薯蓣属dumetorum(肯)罗马帝国)。植物遗传资源。2014;14:1-10。
- 38。
IRAD。植物遗传资源在喀麦隆粮食和农业状况的第二次报告。雅温得:农业研究与发展研究所(IRAD);2008.可从:http://www.fao.org/docrep/013/i1500e/Cameroun.pdf
- 39。
Siadjeu C, pananyoo EA, Mahbou Somo Toukam G, Bell M, Nono B, Medoua GN。品种对三叶山药采后硬化的影响薯蓣属dumetorum块茎。阿阿格利司。2016;2016:1-18。
- 40.
Rognes T, Flouri T, Nichols B, Quince C, Mahé FVSEARCH。一个多功能的元基因组学开源工具。PeerJ。2016; 4: e2584。
- 41。
埃德加RCMUSCLE。一种降低时间和空间复杂度的多序列比对方法。BMC生物信息学。2004;5:1-19。
- 42。
Huson DH,Bryant D.系统发育网络在进化研究中的应用。mol Biol Evol。2006; 23:254-67。
- 43。
Pickrell JK, Pritchard JK。从全基因组等位基因频率数据推断群体分裂和混合。公共科学图书馆麝猫。2012;8:1-17。
- 44。
基于多位点基因型数据的群体结构推断。遗传学。2000;155:945-59。
- 45.
Verity R, Nichols RA。估计结构居群的亚居群数(K)。遗传学。2016;203:1827-35。
- 46.
利用结构化软件检测个体聚类数量:一项仿真研究。摩尔生态。2005;14:2611-20。
- 47.
Earl DA, vonHoldt BM。结构收获:一个用于可视化结构输出和实现埃万诺方法的网站和程序。conservation Genet Resour. 2012; 4:359-61。
- 48.
利用流式细胞术估算植物基因组大小:实验室间比较。安机器人。1998;82:17-26。
- 49.
Gompert Z,Mock Ke。用基因分序(GBS)分析检测单个倍性水平的分析。Mol Ecol Resour。2017; 17:1156-67。
- 50.
烧伤男,赫定男,鹤崎N.人口基因组学和地理孤雌生殖在日本harvestmen(盲蛛目,Sclerosomatidae,Leiobunum).生态另一个星球。2018;8:36-52。
- 51.
Torkamaneh d,拉罗奇Ĵ,Belzile F.全基因组SNP从通过测序基因分型(GBS)数据调用:七个管道和两个测序技术的比较。Plos一个。2016; 11:1-14。
- 52.
李志强。单核苷酸多态性在作物遗传研究中的应用。植物学报2002;5:94-100。
- 53。
Zhu YL, Song QJ, Hyten DL, Van Tassell CP, Matukumalli LK, Grimm DR, et al.;大豆单核苷酸多态性研究。遗传学。2003;163:1123-34。
- 54。
基于核苷酸、indel和存在-缺失多态性的DdRAD-seq系统发育研究:两个鸟类属具有对比历史的分析。分子系统学进展。2016;94:122-35。
- 55。
Wanyera NM NS和NQ。山药。在:Fuccillo, Dominic, Sears, Linda, Stapleton P,编辑。生物潜水员相信conservation利用CGIAR中心的植物基因资源。剑桥:剑桥大学出版社;1997.p。371。
- 56。
水山药感官性状的遗传多样性(参薯l)。J SCI食物农业。2003; 83:858-65。
- 57.
陈志强,王志强,王志强,等。干旱胁迫下水稻根系基因表达调控的研究进展。J Exp Bot. 2016; 67:1003-14。
- 58.
Arnau G,Bhattacharjee R,Mn S,Malapa R,Lebot v,亚伯拉罕K,等。了解山药的遗传多样性和人口结构(参薯升。)利用微。马克理论。2017; 12:1-17。
- 59.
利奇IJ,大通兆瓦,班尼特博士。DNA C-值的系统发生分析提供的证据在开花植物小祖基因组大小。Ann Bot。1998; 82:85-94。
- 60.
Nemorin A, David J, Maledon E, Nudol E, Dalon J, Arnau G.微卫星和流式细胞术分析有助于理解起源参薯多倍体植株。安机器人。2013;112:811-9。
- 61.
Ramsey J, Schemske DW。开花植物多倍体形成的途径、机制和速率。大学学报1998;29:467-501。
致谢
我们感谢西尔维娅·肯本,感谢她对基因组大小分析的支持。我们也感谢Robert Verity博士在对MavericK进行分析后对处理TI的不良等级的支持,以及两位帮助改进这篇论文的匿名审稿人。
资金
本研究由德国学术交流服务处(DAAD)和非洲适当发展基金会(ADAF)资助,用于甘薯的收藏和在喀麦隆的实地工作。此外,资助方在研究的设计、数据的收集、分析和解释、决定发表或手稿的准备方面没有作用。
数据和材料的可用性
GBS数据保存在欧洲核苷酸档案库(ENA)中(访问号:PRJEB27526;https://www.ebi.ac.uk/ena/data/search?query=prejeb27526.).目前的研究过程中产生的所有数据都包含在此发表的文章以及其作为附加文件的补充资料1,2,3.,4,5,6, 和7.
作者信息
隶属关系
贡献
CS促成设计实验,山药加入,数据分析和解释,并手稿写作的集合。EMQ:DNA提取和测序,DCA:数据的解释和稿件写作。所有作者都已经阅读并赞成最终的手稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
附加文件1:
图S1。文学内的系统发育关系D. Dumetorum.基于多程度连接的SNP序列,从34个二倍体中的GBS数据进行对齐。(PDF 259 KB)
附加文件2:
图S2。使用TI估计器估计K = 2:5的模型证据一个)日志证据和b)证据和证据构成推定三角洲ķΔKc) (PDF 95kb)
附加文件3:
图S3。的44个种质结构阴谋D. Dumetorum.with K = 2, 3, 5 clusters based on 6457 unlinked SNPs. (PDF 133 kb)
额外的文件4:
表S1。利用流式细胞仪测定倍性的变异系数和用gbs2ploidy估计的每个材料的倍性水平。*通过gbs2ploidy评估倍性水平(pd47kb)
额外的文件5:
边缘,迁移的权重(8和9)和迁移0:9的可能性。(TREEOUT 1 kb)
额外的文件6:
图S4。中推断基因流的最大似然树D. Dumetorum.没有基因流动事件的物种。(PDF 125 KB)
额外的文件7:
图S5。流动与果实D. Dumetorum..a)男性花,b)女性花。条形秤= 3厘米。c)水果,d)种子。条形秤= 2厘米(PDF 288 KB)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
三叶山药的遗传多样性和群体结构研究(薯蓣属dumetorumKunth)在喀麦隆的基因分型逐序列(GBS)揭示。BMC植物BIOL.18,359(2018)。https://doi.org/10.1186/s12870-018-1593-x
已收到:
接受:
发表:
关键字
- 喀麦隆
- D. Dumetorum.
- 山药
- 遗传多样性
- 人口结构
- GBS.
- 倍增