摘要
背景
大豆(大豆L. merril)作物是食用油和人类和动物蛋白质的主要来源,除了其各种工业用途,包括生物燃料。植物诱导的花芽畸变综合征(FBD),也称为女巫的扫帚综合征(WBS)是主要的生物强调的一个,对其生产率产生不利影响。转录组种方法可用于通过晶体生理关键途径来用于该疾病表现的知识发现。
结果
利用Illumina HiSeq NGS数据对大豆FBD进行转录组学研究,揭示了17454个差异表达基因、5561个转录因子、139条通路和176,029个基因区域推测标记的单序列重复、单核苷酸多态性和插入缺失。本文阐述了PmbA、锌依赖蛋白酶、SAP家族和生长素应答系统的作用,揭示了花粉、柱头发育中花芽畸变的机制。随机选取10个基因进行qPCR验证。本研究揭示了FBD发病的基本机制,即寄主植物感知植物原体感染,触发分子信号,导致碳水化合物和蛋白质的动员,叶状体,花粉发育异常,昆虫在寄主植物体内的定植从而传播疾病。研究揭示了植物原体如何劫持大豆表现FBD的代谢机制。
结论
这是大豆或WBS疾病的转录组签名的第一个报告,揭示了吸引昆虫的形态学和代谢变化。所有基因区域推定标记物可用作各种改善和新农业化学发育的基因组资源,以提高大豆生产率。
背景
大豆(大豆L. Merril)迎合牲畜,家禽和鱼类食用食用油(25%)和蛋白质浓缩物(67%)的全球需求。它有助于肉,牛奶,奶酪,面包和油。每单位面积,这种作物产生的蛋白质是最高的,因此这种作物具有各种绰号,如“牛的牛”或“来自土壤的黄金”,“黄色宝石”,“大宝”,“大自然的奇迹蛋白”[1].全球五大大豆生产国分别是美国、巴西、阿根廷、中国和印度,贡献率超过92% [2].它也在涵盖20多个国家,包括印度的四大大陆。大豆是一种很有希望的作物,因为它在降低胆固醇,抗致癌物质和肥胖,糖尿病,消化道,骨骼和肾脏疾病的肥胖,刺激性症的保护作用中的潜在有益作用3.].作为生物燃料生产的生物能源作物,大豆及其副产品作为牲畜饲料也更为相关[4.].大豆的生产力受到各种生物和非生物胁迫的不利影响,如盐分、病原体、热、干旱、热、土壤重金属和压实[5.].在生物胁迫中,植物原体和病毒是造成生产力损失2 - 90%的主要障碍[6.].根据早期的报告,已经在宿主中发现了一些导致花粉活力下降的生物因素[7.]种子质量变体[8.]、花畸形、萼片肥大、绿色、叶状、生殖器官流产[9.,增强营养生长[10,过早的花败[11)等。植物生殖器官的细胞学检查显示,有症状的植物花粉粒畸变,妨碍授粉和受精[12].
大豆巫婆帚状病的症状和传播在印度有充分的记录[13].这种紊乱导致豆荚不能形成,直到季末才衰老成熟。症状也是不可预测的。大豆巫婆病的花芽形成异常发育,甚至导致生产力的严重损失[12].它在印度被称为“花蕾扭曲”,由植物原体引起[14那15].在美国和伊朗,这被称为芽增殖综合征[16, pod集故障[17那18], 分别。
早期花组织的基因表达研究局限于基于微阵列的有限差异基因表达[19].虽然大豆的RNA-Seq图谱有14种不同的组织,包括花,但没有任何非生物和生物胁迫的表达数据可用[20.那21].已经对FBD进行了细胞学和有限分子研究,发现27 DEG具有ARF9转录因子和调节蛋白FHA以及它们的蛋白-蛋白相互作用网络[12].虽然大豆的全基因组序列已经在2010年完成[22但如果没有RNA-Seq方法,就无法解释FBD的详细机制。这种知识发现不仅是了解病害的基本机制所必需的,而且对大豆生产中病害防治策略的设计也有重要意义。通过RNA-Seq方法,单个作物基因型的胁迫响应主要形态和生理途径已经成功破译了像茶叶这样的作物[23,番茄24cassava [25].对于像大豆这样的农业作物,应激反应被发现是特定于品种/种质的[26].
本研究旨在揭示大豆巫婆病花芽扭曲的关键候选基因及其分子机制。本研究还旨在描述与FBD相关的生化途径,以及基因区域标记(SSR、SNP和InDels)的发现和预测与FBD相关的转录因子。
结果与讨论
鉴定FBD的症状和无症状植物
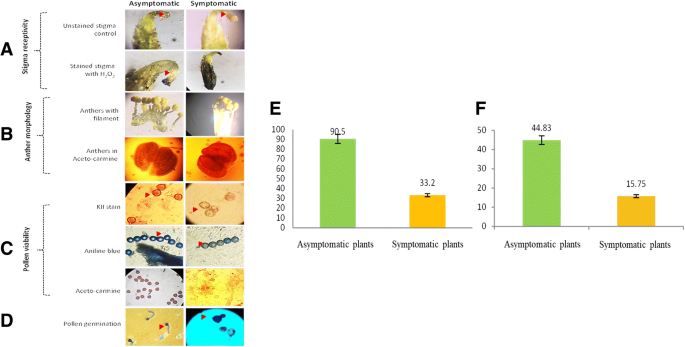
通过花粉活力,形态,耻辱应接受,花药形态和萌发测定成功地通过花粉生殖器官的细胞学研究成功进行症状和无症状植物的歧视(图。1).花器官、花粉和花药的扫描电镜(SEM)也证实了这一点。2).随后,通过扩增子大小及阳性和阴性对照的PCR检测,进一步成功证实了它们(图)。3.).由于不容易排除无症状植物支原体感染,因此需要基于PCR的诊断[27].
RNA-Seq数据生成
感染组和对照组分别产生30669 583和3667 121对端读。从两个样本中共删除156,054和198,931个低质量reads,其余高质量reads用于转录组分析。Trinity汇编器生成的de novo汇编具有258,427份默认抄本k-mer值251).然后用CAP3汇编器去除冗余序列,共获得211343个转录本用于进一步分析。最终组装的GC含量为41.96%,N50为1081 bp。最小和最大转录本长度分别为190 bp和50081 bp。
虽然大豆基因组具有66,210个预测的基因模型[20.[所构造的转录组组件似乎由相对较高数量的转录物(211343)表示。观察到的转录物数量相对较高的潜在原因可以是(1)大豆基因组的古基多倍素来源,其具有两次的重复事件(59 mEA和13 mERA),其中预期变体转录物由于基因组的同源物的差异半衰期/保留时间是预期的它们之间的变化。这些变化是由于重组,由于插入,缺失,反演,不等交叉,假性生成和背景突变而导致的结构变异[22那28],(2)应激诱导的同种型由于免疫相关基因的替代剪接[29(3)(3)较高数量的基因(> 66 k)和每个基因的平均外显子数量,这些基因不同于3-5,也有望产生更多数量的转录物(196至330万)[20.].
差异表达基因的鉴定
DEG的分析是通过基于转录组组装的方法进行的,因为它有望发现“额外的基因”[30.和异构体[31.也保留了植物原体转录本。然而,通过大豆参考基因组组装的转录本作图来评估获得的DEG2).这表明转录组组装的准确性以及覆盖率和深度的均匀性,因为有< 5%的变异。
通过转录组件方法,我们发现35,725,1487和4490°,分别在三个不同的q值参数中。q = 0.9,q = 0.95和q = 0.99。通过方法,即Edger和噪音问题发现了Degs。通过jeckq发现的Deg与Edger进行了比较。通过edger获得的17,454℃,发现总共12,182和12,053次分别在Q = 0.9和0.95中常见。在Q = 0.99,两种方法的比较显示3879次常见的次数。我们的比较分析通过噪音验证了该DEG。与不同Q值的edger的比较呈现在表中3..在更严格的条件下(q = 0.99),有更高表达(>±8倍变化值)的基因是常见的,超过85%。这些组中上调和下调基因的图示在热图中描述(附加文件)1).
注释和功能表征
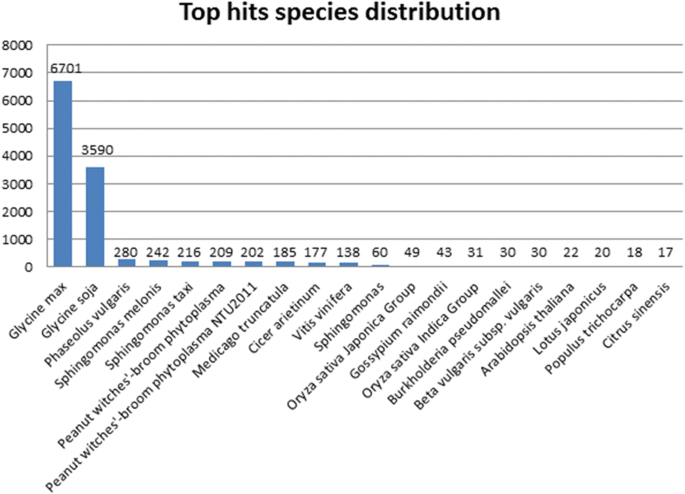
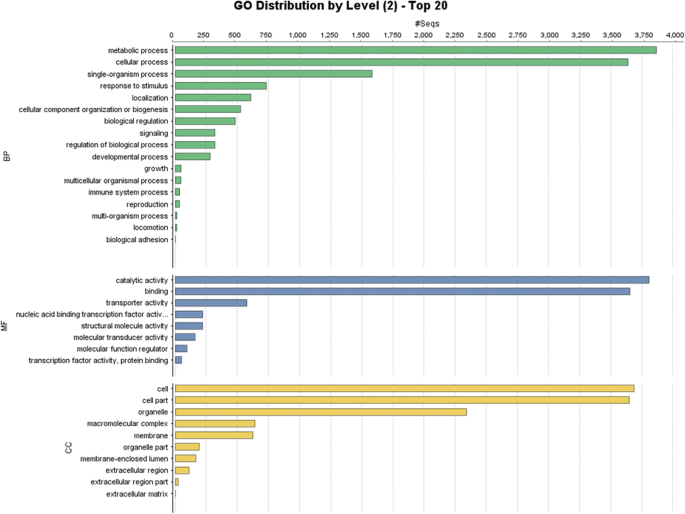
对大豆差异表达基因进行同源性搜索,结果显示,在数据库中17454个与其他已知基因相似的基因中,共有12900个与其他已知基因相似。最大匹配次数(6701)大豆, 其次是甘氨酸Soja.和菜豆即3590和280。4.额外的文件2).植物原体特异性转录本仅为411份,占总数的2.35%。通过基因本体论分析,将基因分为分子功能、生物功能和细胞功能三个子类(图1)。5.).
在对PlantTFDB v4.0进行blast分析的17454个差异表达基因中,5561个ungenes与转录因子具有相似性。与MYB相似的unigenes最多为449个,其次为bHLH、ERF、NAC和FAR1,分别为439、363、339和296个(附加文件)3.).KEGG通路分析显示,有6041个unigenes参与了139个通路(附加文件)4.).
在大豆花芽组织转录组数据中,共发现296个与植原体相关的基因序列。FBD转录组是宿主和寄生虫的混合转录。这是由于在有症状的大豆样品中存在植物原体。寄主植物的许多转录本都是响应植物原体的需要。植物原体的生存依赖于宿主的蛋白质合成机制[32.由于其较小的基因组大小(500-1500kbp),它含有非常有限的基因组[33.].类似的更高丰度的植物浆转录本在其他作物中也有报道,巫婆的扫帚病也包括泡桐[34.].植物浆体通过抑制水杨酸途径降低宿主植物的系统抗性,增强其生长以进一步入侵、增殖和扩散[35.].
其中发现了DNA旋回酶的调节基因PmbA的调控。这是加快细菌复制速度所必需的,随着解旋酶双向DNA解开叉[36.].由于其在基因组大小可塑性中的作用,发现了多组被称为“逆转录酶”的转录本,这显然是在植物浆中预期的[37.].其编码基因内含子II逆转录酶分散在植物原体基因组上,通过产生变异性实现基因的横向转移。由于这些基因的存在,同一种植物原体的不同品系大小不同[37.].
我们发现Zn依赖性蛋白酶转录物的差异表达,所述蛋白酶转录物已知用于毒力触发基因的作物苹果。已发现该基因的多态性与中度至严重的疾病病症有关。该依赖性蛋白酶基因也称为HFLB(同义词FTSH),其用于膜相关ATP和Zn2 +依赖性蛋白酶控制组件,蛋白质的蛋白质和细胞质的蛋白质的劣化和稳定性[38.].
毒力蛋白SAP54上调,也是一种效应体,它通过26S蛋白酶体穿梭蛋白RAD23介导MADS-box转录因子的降解,导致叶状花发育过程中花芽扭曲(FBD)。这是植物原体通过扩大营养组织(叶状花)来吸引昆虫载体的适应性反应。这进一步增强了昆虫定植,这是植物原体通过昆虫载体更广泛传播的一种战略适应[39.].
植物生长素反应因子、植物生长素转运蛋白、植物生长素诱导蛋白的各种转录本均有差异表达。它们在花粉的壁纹和花粉发育中发挥作用[40].生长素响应启动子元件与环境信号一起参与营养向生殖阶段的转换。植物原体扰乱花组织的正常生殖发育,导致FBD [12].
在女巫的扫帚综合征(WBS)中,碳水化合物蛋白质和叶绿素增加> 2倍[41.].这是由于激素平衡受损,导致氨基酸和碳水化合物易位随着衰老而受损。营养期随着植物的延长,但豆荚没有适当的发展。
在芸苔属植物中也有这种花毒力和畸变,并伴有异常的枝分枝和发育不良[42.].糖的运输和积累导致FBD表现的遥远信号[9.].
在SAP11基因中观察到上调,已知用于通过衰减植物发育和免疫力来增强昆虫载体繁殖的植物蛋白效应器[43.].据报道,植物等离子体效应剂可以改变花的发育,从而诱导巫婆的扫帚和改变叶子的形状,以促进植物和昆虫的相互作用。这些效应体促进植物原体在生命周期中的优势进入两个不同的领域,即植物(大豆)和动物(昆虫)[44.].这些效应剂可用于进一步研究开发防治疾病战略所需的新农药[45.].
挖掘推定的分子标记
通过De Novo转录组合组件获得的211,344个转录物中断了总共27,925个SSR标记物大豆.表格4.提供单核苷酸,二核苷酸,三核苷酸,四核苷酸,戊核苷酸和己核苷酸标记物中发现的所有重复单元的信息。没有多态性发现,因为这些SSRS从单个基因型中开采,所生成的信息(附加文件5.)有基因座的详细信息以及用于基因分型的计算机引物。通过e-PCR对SSR位点进行硅分型,对这些基因组资源的未来利用进行了评价。为此,利用已发掘的具有双重复和三重复的SSR位点进行e-PCR验证。共使用1874个转录本(大小为> 1000 bps)进行e-PCR。排除“非特异性”e-PCR产物(有一个以上的命中,相似度100%)后,共发现193个特异性SSR位点,可用于未来的基因分型(附加文件)6.).在大豆基因组中预期由多次命中表示的非特异性E-PCR产物,因为它含有重复事件引起的> 75%重复基因[22也与异构体有关。我们的e-PCR结果清楚地表明,对于像大豆这样的古二倍体基因组物种,该方法可以成功地在SSR位点的湿实验室验证之前使用,从而在基因分型中排除多个拷贝转录本,减少时间和成本。这些SSR位点可用于多样性、QTL定位和关联定位的基因分型。
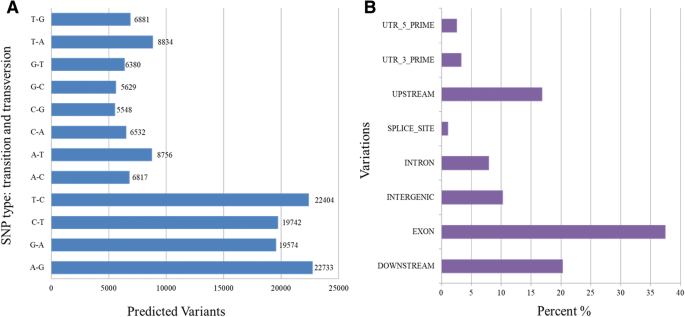
虽然转录组数据属于单基因型,JS- 335印度大豆品种,但SNP和InDels的挖掘已经成功地进行了比对,以代表美国Williams 82品种的可用参考基因组。大豆RNA-seq共检测到146,065个变异,包含139,461个SNPs和6604个InDels。18号染色体的变异量最大,为9582,其次是6号染色体的8748和2号染色体的7489。在11号染色体中发现的变异最少,为4739个。SNP的Ts/Tv比值为1.53,A-G和T-C是转化中的最大预测变量,而T-A和A-T是转化中的最大预测变量(图)。6.a).此外,注释是针对大豆基因组,其中最大变体(37.48%)在封面区域中发现,其次分别在下游(20.32%)和上游(16.88%)区域(图。6.b;额外的文件7.).
所有176,029个推定标记(SSR,SNP和Indels)可用作未来研究的基因组资源,特别是对于QTL,基因测绘和连杆分析。这种DNA标志物用于柔软植物抗肌型QTL映射,如苦贝里(李古兰弗吉尼亚州)[46.].使用DNA标记物的品种改善计划中的植物抗性性状迟发已在苹果中成功报道[47.].在椰子中,由植物性DNA标记引起的致致死的致黄性疾病(LYD)已用于杂交品种发展方案,以改善Lyd抗性[48.].
QPCR验证和表达分析
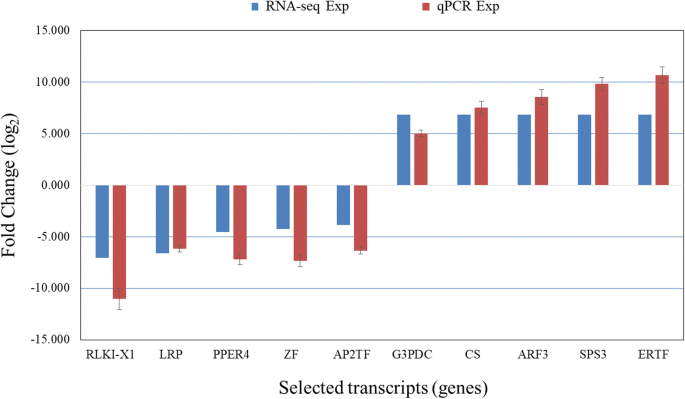
对所有10个上调和下调基因进行qPCR分析,得到的相对基因表达值与计算得到的DEGs的对数倍变化值一致(图2)。7.).qPCR所用基因及引物的详细信息见表5..
数据提交
该研究使用的测序数据集可在NCBI知识库中获得,生物项目:PRJNA472133、生物样本:SAMN09227257(大豆对照无症状花芽组织)和SAMN09227258(植物原体感染大豆有症状花芽组织)。
结论
这是大豆中FBD或WBS疾病的第一次转录组研究。我们报告了17,454只涉及疾病的139例途径的转录组签名。该研究在分子水平方面表现出异常的花朵发育,碳水化合物和蛋白质,植物异常,花粉发育,改善宿主植物中昆虫的定植。报告了总共176,029个基因区域推定标记物(SSR,SNP和吲哚),可用作未来分子育种计划的基因组资源,用于转移大豆种类改善的植物抗性。不仅需要在分子水平上理解疾病,因此不仅需要进行这些研究,但它们也需要种质改善大豆生产率的致力。
方法
鉴定FBD的症状和无症状植物
在目前的调查中,选择了印度最受欢迎的广泛种植的大豆品种JS- 335,该品种最近发生的FBD疫情对其产量产生了不利影响。实验数据和组织采集自农业植物学系Panjabrao Deshmukh Krishi Vidyapeeth博士的实验田,位于热带,平均海平面307.4米。地理位置为北纬20.42°,东经77.02°,黑色中等,土质,地势较平,地形均匀,水系适宜。为了区分有症状和无症状的植物,在R2生殖期进行了花粉活力、扫描电镜(SEM)、柱头接受度、花药形态和发芽试验。12].
基于PCR的植物诊断测试
对采集的植物叶片组织样本进行PCR检测,诊断有症状和无症状的植物。DNA分离使用MO BIO PowerPlant Pro DNA分离试剂盒(Carlsbad, CA),按照制造商的协议进行。采用1X PCR缓冲液、1.5 mM MgCl2、50 ng基因组DNA、dNTPs 200 μM、Taq酶(德国Invitrogen公司)1个单位、0.5% DMSO、2 μg/μl BSA、P1和P7引物1 μM [49.].随后使用引物R16F2N和R16R 2进行嵌套的PCR [50.其中使用10倍稀释的初始PCR产物作为模板。
组织收集和RNA提取
三组R5期(1/8英寸)有症状和无症状的植物组织(冷冻芽和节组织)。收集主茎最上节之一荚果中的长种子,用RNAlater处理,-80℃保存。
对于RNA提取,进行了10种生物学重复的症状和无症状植物组织的样品,以最小化每个集合中的样品变异性[51.].根据制造商的协议使用Purelink_RNA Mini Kit(Invitrogen,San Diego,USA)。
使用Agilent RNA芯片检查BioAnalyzer 2100(Agilent Technologies)上检查RNA质量。使用> = 8 RNA完整性数(rin)值的样品用于进一步分析。使用4微克总RNA通过Truseq RNA样品制备试剂盒(Illumina)制备RNA-SEQ库。根据制造商的协议,通过Poly-T oligo附着的磁珠分离含有聚-α的mRNA分子。使用二价阳离子在升高的温度下进行mRNA的破碎化。使用逆转录酶和随机引物和随机引物和第二链CDNA合成进行CDNA合成,使用DNA POL I和RNase H进行第二链CDNA合成。通过终止修复过程,加入单个“A”以连接适配器。最终cDNA文库是在纯化和富集后创建的。为了评估mRNA质量,富集,片段大小和图书馆尺寸生物分析仪。在测序之前,使用量子位和QPCR测量文库的数量。在Illumina Hiseq 2000平台上测序了单组的构建图书馆,并生成了6000万读(6GB)数据(2 X100BP PE读数)。
预处理和从头转录组组装
配对的读数大豆(大豆)使用Illumina Hiseq2000技术产生。使用FASTQC工具进行评估和可视化原始读取[52].用于修剪和去除低质量读取(从3 '和5 '端基;Phred-score≤20,trimmomatic tool version 0.38 [53使用了)。使用Trinity进行从头转录组组装[54CAP3除去冗余序列[55].作为互乱的数据,具有来自两个基因组的转录物,即大豆和柔软,对准/过滤参考基因组上的读取的标准方法。这样做是为了获得“额外基因”以及同种型以及在随后的分析中保留植物转录物。
差异表达基因的鉴定
de novo转录组组件用于鉴定次数。使用Bowtie进行映射和原始读取的原始读取的对齐[56].通过期望最大化(RSEM)工具的RNA-Seq [57]用来计算每个转录本的表达值,即每千个外显子的片段/百万图读。随后,为鉴别差异表达基因,采用edgeR (Empirical analysis of Digital Gene Expression in R) [58,采用TMM (trim Mean of M-values)方法对数据进行归一化[59].为了降低噪声和计算再现性,使用噪声技术,其具有非参数和数据 - 自适应方法,用于识别差异表达基因是基于计数矩阵[60].对于表达式分析,阈值方差和P.-Value被设定为0.05以鉴定显着基因[61那62].在三个不同的q值下,即q = 0.9, q = 0.95和q = 0.99,用这两种工具对DEG进行了比较识别。
注释和功能表征
使用BLASTX算法(独立本地NCBI-BLAST-2.2.31 +;电子值1E-5)对NCBI非冗余数据库进行同源性表达基因。63].BLAST2GO PRO 3.1软件[64用于注释和功能表征的次数。使用PlantTFDB V4.0数据库进行转录因子预测[65].
挖掘推定的分子标记
从转录组数据中提取了可能的分子标记,即简单序列重复(SSRs)、单核苷酸多态性(SNPs)和InDels。MISA-MIcroSAtellite识别工具[66],利用PERL脚本挖掘假定的SSR标记。单核苷酸和双核苷酸的重复单位分别为10和6。对于三、四、五和六核苷酸,使用5个重复单元。随后利用PRIMER3工具对SSR位点进行引物筛选[67].具有DI和TRI重复的挖掘SSR用于Silico PCR验证。选择具有超过1000bp的转录物,设计了基于E-PCR基因分型的引物。排除了在整个大豆基因组中具有多于一个击中的E-PCR产物。
由于转录组数据是从单印度基因型(JS-335)的变化,因此美国品种的参考基因组,威廉姆斯82可用于SNP采矿。为了使用基于参考的映射来发现SNP。SNP(单核苷酸多态性)呼叫是针对参考基因组进行的大豆V2.0使用几个过滤器,如最小读取深度覆盖> = 15 [68],质量分数> 30 [69那70]并考虑来自两侧最小50bp剥落区域的所有变体。参考大豆基因组组装(大豆在登录号GCA_000004515.3下,从NCBI检索用于使用挖掘机轮式对准器从大豆转录组中采集SNP和Indels [71]和Samtools [72].此外,通过使用SNPeff工具进行所识别的变体的注释[73].
QPCR验证和表达分析
使用REVERTAID第一链CDNA合成试剂盒(GEATIX,USA)来获得CDNA合成,以获得QPCR的模板。从DEG,10种转录物被随机选择(5升高,下调),用于通过PRIMER3设计底漆[67].PCR在实时PCR机(ABI-7300,Applied Biosystem)上使用Quantifast Sybr Green PCR主混合物套件(Genetix,USA)进行了一式三份进行了40个循环的熔融曲线步骤。对于归一化,使用家务基因,actin用作参考。完成PCR优化以获得每组引物对的线性关系。最后,ΔΔCT折叠变化值[74计算出获得差分基因表达的大小。它与LOGFC值进行了比较的验证。
缩写
- cDNA:
-
互补DNA
- DEG:
-
差异表达基因
- 背景:
-
脱氧核糖核酸
- 磨边机:
-
数字基因在R
- 定期监测:
-
植物的芽失真
- InDel:
-
Insertion-Deletion
- LYD:
-
致命的泛黄的疾病
- NCBI:
-
国家生物技术信息中心
- 聚合酶链反应:
-
聚合酶链反应
- PERL:
-
实用提取和报告语言
- PlantTFDB:
-
植物转录因子数据库
- RIN:
-
RNA完整性
- RNA:
-
核糖核酸
- RSEM:
-
RNA-SEQ通过期望 - 最大化
- 扫描电镜:
-
扫描电子显微镜
- SNP:
-
单核苷酸多态性
- SSR:
-
简单序列重复
- TF:
-
转录因子
- WBS:
-
女巫扫帚综合症
参考
- 1.
Horvath AA。在原始大豆饲喂兔子血液组成的变化。J Biol Chem。1926; 68:343-55。
- 2.
Masuda T,Goldsmith PD。世界大豆生产:面积收获,产量和长期预测。国际食品和农业企业管理审查。2009; 12(4):143-62。
- 3.
Friedman M,Brandon DL。大豆蛋白的营养和健康益处。JAgric Food Chem。2001; 49(3):1069-86。
- 4.
Popp J,Harangi-RákosM,Gabnai Z,Balogh P,Antal G,Bai A. Biofuels及其共同产品作为牲畜饲料:全球经济和环境影响。分子。2016; 21(3):285。
- 5。
Miransari M,编辑。大豆生产中的非生物和生物胁迫:大豆生产。学术出版社;2015年。
- 6.
Jadhav Pravin V,Mane Ss,Nandanwar Rs,Kale Pb,Dudhare Ms,Moharil Mp,等。在大豆和印度中部的大豆和发病率的花芽畸变。埃及J BIOL。2013; 15:59-65。
- 7.
Subekti NA。病害对植物繁殖的影响,作为抗病育种的基础。In the Proceedings of the scientific seminar and annual meeting of the regional commissioner PEI PFI XIX South Sulawesi. 2008; 5:167-80。
- 8.
3种水叶植物(水叶科)的花大小和种子质量变化的真菌病原作用。植物学报。2006;33(3):389-98。
- 9.
Pracros P, Renaudin J, Eveillard S, Mouras A, Hernould M.番茄stolbur植物原体侵染诱导的花异常与花发育基因表达的变化有关。植物与微生物的相互作用。2006;19(1):62-8。
- 10.
Singh AK, Bhatt BP。比哈尔邦蚕豆的植原体叶栅病和女巫帚病的发生。环境生物学杂志,2013;34(5):837。
- 11.
引用本文Sugano J, Melzer M, Pant A, Radovich T, Fukuda S, Migita S, et al.。番茄黄曲叶抗病品种的田间评价。工厂说》2011。
- 12.
Kale PB, Jadhav PV, Wakekar RS, Moharil MP, Deshmukh AG, Dudhare MS, et al.;大豆花器官的细胞学行为和与“花芽扭曲”相关的差异表达转录源片段的硅特性。J麝猫。2016;95(4):787 - 99。
- 13.
丁格拉吉隆坡,Chenulu VV。印度大豆巫婆病的症状学和传播。在:当代科学(印度);1983.
- 14。
Thorat V,更多V,Jadhav P,Mane Ss,Nandanwar Rs,Surayavanshi M,等。第一次报告与印度马哈拉施特拉大豆(甘氨酸最大甘氨酸)的巫婆 - 扫帚病相关的16SRII-D组植物。植物DIS。2016; 100(12):2521。
- 15.
Kumar S, Sharma P, Sharma S, Rao GP。豇豆黄花叶印度病毒与‘Candidatus Phytoplasma asteris’混合侵染和自然传播对印度大豆作物的影响。J Phytopathol。2015;163(5):395 - 406。
- 16.
k . Subramanya Sastry。种子传播的植物病毒病。施普林格印度2013,施普林格科学和商业媒体。ISBN 978-81-322-0812-9。http://www.soybeancheckoffresearch.org/DetailsbyPaperid.php?id_Paper=1144.2014年8月访问。
- 17.
Golnaraghi AR,Shahraeen N,Pourahim R,Farzadfar S,Ghasemi A.感染伊朗大豆病毒的发生和相对发病率。植物DIS。2004; 88(10):1069-74。
- 18.
伊朗与大豆荚果集败综合征相关的病毒。第12届伊朗植物保护大会论文集,1995年9月2日至7日。Karadj(伊朗伊斯兰共和国)。1995.
- 19.
黄志强,黄志强,黄志强。大豆茎尖分生组织成花转变的分子机制。植物j . 2009; 57(5): 832 - 45。
- 20.
Severin AJ, Woody JL, Bolon YT, Joseph B, Diers BW, Farmer AD等。Glycine max的RNA-Seq图谱:大豆转录组的指南。植物学报。2010;10(1):160。
- 21.
郑昌华,黄志强,黄志强。大豆开花基因的基因组学分析。《公共科学图书馆•综合》。2012;7 (6):e38250。
- 22.
徐志强,王志强,王志强,等。古多倍体大豆的基因组序列。大自然。2010;463(7278):178。
- 23.
刘长生,金建强,马建强,姚明志,马春林,李春发,等。茶树对干旱胁迫和恢复的转录组分析。《公共科学图书馆•综合》。2016;11 (1):e0147306。
- 24.
刘志强,刘志强,刘志强,等。转录组变化驱动番茄对渐进性干旱胁迫和复水的生理反应。植物科学学报2016;7:371。
- 25.
傅L,丁Z,汉B,胡w,李y,张建国聚乙二醇(PEG)诱导脱水应力的生理学研究和转录分析。int j mol sci。2016; 17(3):283。
- 26。
Shin JH, Vaughn JN, Abdel-Haleem H, Chavarro C, Abernathy B, Do Kim K,等。水分亏缺引起的转录组变化定义了大豆的一般反应和抗旱的特异性通路。植物学报。2015;15(1):26。
- 27。
博夫JM。植物的支原体感染。ISR J MED SCI。1981; 17(7):572-85。
- 28.
Panchy n,Lehti-shiu m,shiu sh。基因重复在植物中的演变。植物理性。2016; 171(4):2294-316。
- 29.
杨树华,唐芳,朱红梅。植物免疫的选择性剪接。中华医学杂志。2014;15(6):10424-45。
- 30.
任敏,林建新,廖伟,Spolski R, Leonard WJ。来自未定位的人类RNA-Seq数据的新转录本的综合组装及其与癌症的关联。中国生物医学工程学报。2015;11(8):826。
- 31.
Palmieri N,Nolte V,Suvorov A,Kosiol C,SchlöttererC.使用RNA-SEQ的不同基于参考的注释策略的评估 - 以甲卓奥齐氏菌伪科学案例研究。Plos一个。2012; 7(10):E46415。
- 32.
白X,张j,ewing a,miller sa,radek aj,shevchenko dv等。生活在基因组不稳定性:植物的适应对昆虫和植物主持人的不同环境。J细菌。2006; 188(10):3682-96。
- 33.
Marcone C, Neimark H, Ragozzino A, Lauer U, Seemüller E.构成主要系统发育群和亚群的植物浆体的染色体大小。植物病理学。1999;89(9):805 - 10。
- 34.
MOU HQ,LU J,朱SF,林CL,Tian GZ,Xu X等。泡桐巫婆的浮游生浮肿的转录组分分析。Plos一个。2013; 8(10):E77217。
- 35.
吕伊,李我,程克,谭厘米,苏lw,lin wy等。表达植物效应器SAP11的转基因植物显示出改变的磷酸盐饥饿和防御反应。植物理性。2014; 164(3):1456-69。
- 36.
Kube M,Mitrovic J,Duduk B,Rabus R,Seewüllere.目前关于植物基因组和编码代谢的目前的观点。SCI世界J. 2012; 2012年。
- 37.
比较两个密切相关的“Candidatus Phytoplasma austrense”分离株的全基因组序列,揭示了基因组的可塑性。BMC基因组学。2013;14(1):529。
- 38。
Seemüllere,Sule S,Kube M,Jelkmann W,Schneider B. AAA + ATP酶和“Candidatus植物植物Mali”的HFLB / FTSH蛋白酶:系统发育多样性,膜拓扑和与应变毒力的关系。Mol植物微生物相互作用。2013; 26(3):367-76。
- 39。
作者简介:陈志刚,男,博士,主要研究方向:植物浆体效应蛋白在植物发育和植物-昆虫相互作用中的作用。2017.
- 40。
杨军,田丽,孙明霞,黄晓燕,朱军,管玉峰,等。生长素反应因子17在拟南芥花粉壁图案形成过程中起重要作用。植物杂志。2013;162(2):720 - 31所示。
- 41。
Wakekar RS, Jadhav PV, Kale PB, Moharil MP, Nandanwar RS, Mane SS, et al.;花粉功能障碍导致印度大豆(Glycine max)花芽扭曲。农业研究。2018;7(1):10 - 24。
- 42。
王志强,王志强,王志强,等。植物原体侵染对芸薹属植物花芽形成的影响。中国农业科学,2012;4(2):219。
- 43.
Sugio A,Maclane Am,Greive VM,Hogenhout Sa。植物蛋白效应器SAP11通过操纵植物发育和防御激素生物合成来增强昆虫载体繁殖。PROC NATL ACAD SCI。2011; 108(48):E1254-63。
- 44.
Sugio A, MacLean AM, Kingdom HN, Grieve VM, Manimekalai R, Hogenhout SA。植物浆体效应剂的不同靶点:从植物发育到昆虫防御。植物病理学杂志2011;49:175-95。
- 45.
anouard R, DiRita VJ。应用于研究细菌病原体的化学生物学。感染Immun。2015;83(2):456 - 69。
- 46.
李志刚,戴伟。植物抗x -病害的研究进展。植物学报2017;8。
- 47.
Jarausch W,Bisognin C,Schneider B,Grando Ms,Velasco R,Seemüllere.植物养殖抗康登斯植物马里的植物植物。昆虫学公告。2007; 60(2):299-300。
- 48.
Gurr Gm,Johnson AC,Ash GJ,Wilson Ba,Ero MM,Pilotti Ca等。椰子致命黄色疾病:对全球经济和社会意义的棕榈树威胁的植物威胁。前植物SCI。2016; 7:1521。
- 49.
Smart CD,Schneider B,Blomquist Cl,Guerra LJ,Harrison Na,Ahrens U等人。基于16S-23S rRNA间隔区的序列的植物特异性PCR引物。申请环境微生物。1996年; 62(8):2988-93。
- 50.
张志强,张志强,张志强,等。植物原体菌株16S rRNA组I(紫菀及相关植物原体)和III (X-disease及相关植物原体)的基因组多样性和分化中国微生物学杂志。1996;46(1):64-75。
- 51。
邹超,王鹏,徐勇。遗传、基因组学与作物改良的大样本分析。中国生物技术学报;2016;14:41 - 55。
- 52。
FastQC:高通量序列数据的质量控制工具;2010.
- 53。
Trimmomatic:用于Illumina序列数据的灵活微调器。生物信息学。2014;30(15):2114 - 20。
- 54。
Haas BJ,Papanicolaou A,Yassour M,Grabherr M,血液PD,Bowden J,等人。从RNA-SEQ使用Trinity平台进行参考生成和分析的DE Novo转录序列重建。NAT PROTOC。2013; 8(8):1494。
- 55.
黄旭,马丹。CAP3:一种DNA序列组装程序。基因组研究》1999;9(9):868 - 77。
- 56.
Langmead B,Trapnell C,Pop M,Salzberg SL。超快和记忆高DNA序列对人类基因组的对准。基因组Biol。2009; 10(3):R25。
- 57.
Li B,杜威CN。RSEM:具有或不具有参考基因组的RNA-SEQ数据的准确转录物定量。BMC生物信息学。2011; 12(1):323。
- 58.
罗宾逊MD,麦卡锡DJ,SMYTH GK。编辑:用于数字基因表达数据的差异表达分析的生物导体包。生物信息学。2010; 26(1):139-40。
- 59.
一种用于RNA-seq数据差异表达分析的标准化方法。基因组医学杂志。2010;11 (3):R25。
- 60.
Tarazona S,García-Alcalde F,Dopazo J,Ferrer A,Conesa A. RNA-SEQ中的差异表达:深度问题。Genome Res。2011; 21(12):2213-23。
- 61.
李丽萍,李丽萍,李丽萍,李丽萍。4种金丝桃属植物转录组结构的比较研究。植物学报2016;7:1039。
- 62.
王y,guo zy,sun x,lu sb,xu wj,zhao q等。感应和动力神经损伤后大鼠基因表达的变化。SCI批准。2016; 6:26579。
- 63。
卡马乔C,库鲁利斯G, Avagyan V, Ma N, Papadopoulos J, Bealer K,等。BLAST+:架构和应用程序。BMC生物信息学。2009; 10(1): 421。
- 64。
Conesa A,Götzs,García-gómezJM,Terol J,TalónM,Robles M.Bast2Go:功能基因组学研究中的注释,可视化和分析的通用工具。生物信息学。2005; 21(18):3674-6。
- 65。
金军,田芳,杨大川,孔林,罗军,等。PlantTFDB 4.0:植物转录因子和调控相互作用的中心枢纽核酸Res 2016:gkw982。
- 66。
Thiel T,Michalek W,Varshney R,Graner A.利用EST数据库进行大麦(Hordeum Vulgare L.)的基因衍生的SSR标记的开发和表征。Al Appl Genet。2003; 106(3):411-22。
- 67.
Untergasser A,Cutcutache I,Koressaar T,Ye J,Faircloth Bc,Remm M等。Primer3-新功能和接口。核酸RES。2012; 40(15):E115。
- 68.
uitdigilligen jg,wolters am,bjorn b,borm tj,visser rg,van eck hj。高杂合子自动四倍体马铃薯的基因分序测序的下一代测序方法。Plos一个。2013; 8(5):E62355。
- 69.
刘建军,刘建军,刘建军,等。整合外显子组和转录组测序揭示ZAK亚型在胃癌中的应用。Nat Commun。2014;5:3830。
- 70。
Yu X, Sun S.基于低覆盖率测序数据的SNP呼叫算法比较。BMC生物信息学。2013; 14(1): 274。
- 71。
李H,Durbin R.快速准确的短读对齐与挖洞轮转变换。生物信息学。2009; 25(14):1754-60。
- 72.
李H,手机B,Wysoker A,Fennell T,Ruan J,Homer N等人。序列对齐/地图格式和samtools。生物信息学。2009年; 25(16):2078-9。
- 73.
王丽萍,王丽萍,王丽萍,等。一个用于注释和预测单核苷酸多态性影响的程序,SnpEff:果蝇黑腹菌株w1118基因组中的SNPs;iso-2;iso-3。飞(奥斯丁)。2012; 6(2): 80 - 92。
- 74.
Livak KJ,Schmittgen TD。实时定量PCR的相对基因表达数据分析及2ΔΔCT方法。方法。2001; 25(4):402。
致谢
我们衷心感谢印度政府孟买核科学研究委员会原子能部(DAE)提供的财政支持(批准号:2013/37B / 44 / BRNS / 1904)。作者谨向印度马哈拉施特拉邦阿克拉444104的Panjabrao Deshmukh Krishi Vidyapeeth博士(国立农业大学)表示诚挚的感谢和感谢,感谢他提供开展调查所需的设施。作者感谢印度农业研究委员会、农业和农民福利部、印度政府提供的CABin赠款形式的财政援助。Agril。Edn.4-1/2013-A&P),以及印度新德里ICAR-IASRI农业组学知识高级超级计算中心(ASHOKA)设施。
资金
印度政府孟买核科学研究委员会原子能部(DAE)(批准号:2013/37B/44/BRNS/1904)和CABin补助金,印度农业研究委员会,农业和农民福利部,印度政府(F. no. 1)。Agril。Edn.4-1/2013-A&P)。
数据和材料的可用性
该研究使用的测序数据集可在NCBI知识库中获得,生物项目:PRJNA472133、生物样本:SAMN09227257(大豆对照无症状花芽组织)和SAMN09227258(植物原体感染大豆有症状花芽组织)。这些将在出版后公开。
作者信息
从属关系
贡献
PVJ,SJ,AGD,RSN,SP,JGM,SSM和DK:构思了研究的主题,SJ,RSJ,MAI,MPM和MSD:计算工作;PBK,PVJ和SKK:样品收集和湿实验室炒面;JK,RST,PGK和MPM:验证工作;SJ,PVJ,MAI,RSJ和DK:起草稿件;AR,JGM,RST和DK编辑了稿件;所有作者都阅读并完成了稿件。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Jaiswal, S., Jadhav, p.v., Jasrotia, R.S.et al。转录组签名揭示了大豆扫帚疾病中花蕾畸变的机制(大豆).BMC植物BIOL.19,26(2019)。https://doi.org/10.1186/s12870-018-1601-1.
收到了:
公认:
发表:
关键字
- 大豆
- 巫婆扫帚
- 转录组
- 组装
- 微分表达式