摘要
背景
CCCH核酸结合基序广泛存在于多种生物体中。CCCH基因参与植物的发育过程以及生物和非生物胁迫反应。Brassica Rapa.是重要的经济作物和多倍体进化的经典模式植物,但CCCH基因在b·拉伯尚不清楚。
结果
在这项研究中,103个CCCH基因b·拉伯被确定。对二者染色体位置、基因结构、结构域组织及重复事件进行了比较分析b·拉伯和拟南芥被执行。结果表明,CCCH基因可分为18个亚家族,其中节段复制可能是该家族扩展的主要原因。cx7/8cx5.- c3.-H是最常见的基序,但也发现了一些新的CCCH基序,以及一些在其他植物中普遍存在的典型的CCCH基序缺失。CCCH基因结构和结构域的多样性与CCCH基因的功能多样性有关b·拉伯.证据还表明,由于成员之间的高度保守,至少在一个亚家族中存在功能冗余。最后,亚家族ix基因的表达谱表明,它们可能参与了各种胁迫反应。
结论
这项研究首次提供了CCCH基因的全基因组特征b·拉伯.结果表明b·拉伯CCCH基因可能在功能上存在分歧,但主要参与植物发育和胁迫反应。这些结果有望促进该潜在rna结合蛋白家族未来的功能表征芸苔属植物作物。
背景
转录因子(Transcription factor, TF),又称反式作用因子,是指一种DNA结合蛋白,通过与特异性分子相互作用,激活或抑制遗传信息从DNA到mRNA的转录顺式元件DNA靶标[1那2].植物生长因子调节植物营养和生殖组织的发育,包括根[3.,茎木纤维[4.)、水果(5.]和种子[6.],以及叶片脱落[7.],开花[8.那9.和植物免疫[10].TFs在应对细菌、真菌和病毒攻击等生物压力方面发挥着重要作用[11],以及非生物胁迫,包括干旱,盐,高渗透压,热或冷[12那13那14那15].
锌指motif最早发现于非洲爪蟾蜍转录因子IIIa和锌指蛋白现在被认为是真核基因组中最丰富的蛋白质家族之一。锌指基序基于锌结合氨基酸和锌离子的征用以稳定其结构。锌指图案是一个小型,功能,独立折叠的域[16那17那18].锌指蛋白在许多生物过程中发挥多面体作用,包括DNA识别,RNA包装,转录激活或抑制,细胞凋亡调节,蛋白质折叠和组装,以及脂质结合[18那19].在植物中发现了十四个锌手指家庭,其中DNA或蛋白质结合蛋白已过度代表[20.].
Cys2His2(C2H2)锌指是在真核转录因子中发现的最常见的DNA结合基序[21].相比之下,普遍存在的CCCH基序优先用于rna结合和加工。在哺乳动物中,一个典型的串联CCCH锌指蛋白三乙酰丙酮蛋白(TTP)结合到TNFα.(肿瘤坏死因子α)(AU富含元件)在3’ UTR区域动摇TNFα.信使rna (22].在拟南芥和水稻,不同CCCH基序的特征在于每个半胱氨酸和半胱氨酸 - 组氨酸(C-X之间的氨基酸间隔的可变数量4-15cx4 - 6cx3.-H) [23].CCCH锌指蛋白通常含有1-6个CCCH重复基序和C-X7/8cx5.cx3.-H是拟南芥和水稻CCCH蛋白中最丰富的基序。拟南芥有68个CCCH蛋白基因,分为11个亚科[23].ATC3H14(AT1G66810)和ATC3H15(AT1G68200)含有C-X.8.cx5.cx3.-H-X18cx8.cx5.cx3.-H motif是拟南芥中仅有的两个串联CCCH锌指(TZF)蛋白,其保守的TZF motif与动物相同[24].AtC3H48 (AT4G25440), AtC3H59 (AT5G40880), AtC3H62 (AT5G49200), AtC3H63 (AT5G51980),含有一或两个C-X的CCCH基序7.cx4/5cx3.-H和另外7个WD40 (WD或beta-转导蛋白重复)结构域,是植物独有的[23那25那26].水稻是CCCH锌指蛋白的另一个研究的模式植物。大米CCCH锌指蛋白包含在8个亚家族67名成员。水稻CCCH蛋白主要参与非生物胁迫响应和发展[23].
同样,拟南芥的CCCH基因也参与了发育过程[27那28那29那30.那31和各种应激反应[32].拟南芥精氨酸丰富的图案串联CCCH锌指(RR-TZF)蛋白,其特征在于两个相同的C-x7 - 8cx5.cx3.- h和cx5.cx4.cx3.-H基序由16-18个氨基酸分开,已被相当深入地研究(由[33那34].AtTZF脱落酸(ABA)、赤霉素(Gibberellin acid, GA)、盐、冷、H2O.2,渗透胁迫,营养缺乏,主要参与生长和胁迫反应。例如,AtTZF1(ATC3H23,AT2G25900)局部化细胞质加工体(PBS)和应激颗粒(SGS),并通过ABA,糖耗尽和盐胁迫阳性调节,但是通过GA负调节。attzf1在体外结合RNA和DNA [34那35那36那37].
白菜(Brassica Rapa.l . ssp.pekinensis.)是一个亚种b·拉伯(AA, 2n = 20),是亚洲最重要的蔬菜之一[38].CCCH基因的基因组组织和功能芸苔属植物品种仍然未鉴定。AtC3H36(AT3G12130)的同源基因芸苔栗鸟CL1Contig3630在花粉、小孢子、胚珠和合子胚中共表达[39].全基因组转录组分析显示,2个CCCH基因参与了干旱胁迫反应b·拉伯[40].随着基因组测序技术的出现,Gene家族的全球鉴定成为众多植物物种的现实。CCCH基因家族的基因组特征已在拟南芥中进行,稻米进行[23)、玉米(20.],柑橘[41,番茄42],葡萄[43],杨树[44],苜蓿[45],鹰嘴豆[46],山羊草属tauschii[47]和switchgrass [48].在这里,我们报告了CCCH基因的基因组鉴定b·拉伯[49]和它们的表达响应于ABA,高的温度,干旱和盐胁迫。
结果
CCCH基因的鉴定b·拉伯
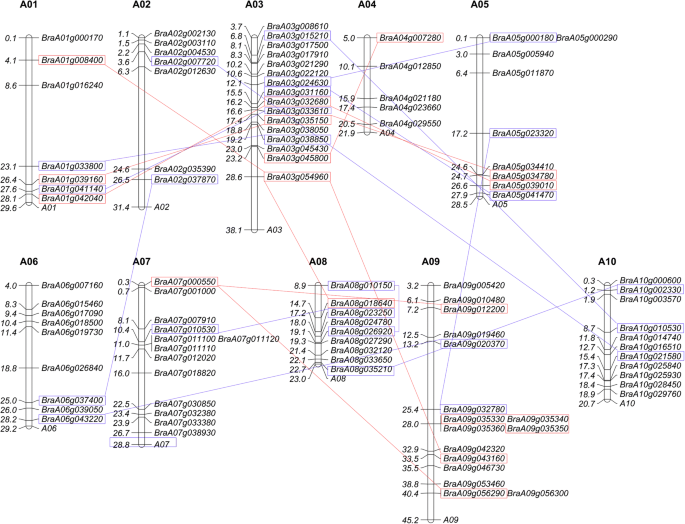
使用Arabidopsis CCCH锌指蛋白作为查询,我们筛选了b·拉伯基因组的BLASTp工具(http://brassicadb.org/brad/).我们通过SMART和NCBI对103个CCCH锌指蛋白进行了鉴定和确认b·拉伯(附加文件1).在103个相应的CCCH基因中,有102个被定位到A01-A10染色体上(图1)。1).第一长染色体A09和第二长染色体A03的CCCH基因数量最多(16个),其次是ChrA07,共有13个成员。ChrA10染色体最短,为20.72 M,是11个CCCH基因中密度最高的。ChrA02是第三长染色体,携带CCCH基因的密度最低。ChrA01和ChrA02的中部(长度大于10 M)未发现CCCH基因。每条染色体上CCCH基因的平均密度低于拟南芥,而高于水稻和玉米(图2)。2).
CCCH基因的重复b·拉伯
基因复制在新基因功能的进化中起着至关重要的作用,是适应性进化的主要驱动因素之一[50].根据之前建立的标准,一些b·拉伯CCCH基因显然已经复制[51].例如,复制了11对基因:BRAA03G024630 / BRAA05G000180那BraA03g031160 / BraA05g041470那BraA03g015210 / BraA10g010530那Braa05G023320 / BRAA09G032780那BraA01g033800 / BraA03g038850那BraA09g020370 / BraA06g043220那BraA10g002330 / BraA08g035210那BraA07g010530 / BraA08g023250那BraA03G033610 / BraA01g041140那BraA08g026920 / BraA08g010150那BraA06g037400 / BraA02g037870,虽然六个基因组一式三份显示:BraA03g008610/BraA02g007720/BraA10G021580那BRAA03G035150/BraA05g034780/BraA01g039160那BraA03g054960/BraA08g018640/BraA01g008400那BraA03g045800/BRAA09G043160/Braa04g007280那braa09g035330./braa09g035340./Braa09g035350.那BRAA09G056290 / BRAA07G000550./Braa09g012200一个基因组四倍体BraA05g039010 / BraAnng001760 / BraA03g032680/BraA01g042040。令人惊讶的是,除了ChrA09上的三个基因外,所有这些基因都是节段复制(图)。1;额外的文件2).CCCH基因片段重复事件发生频率最高的发生在ChrA01和ChrA03之间,其中包含5个片段重复事件,其次是ChrA03和ChrA05之间的4个片段重复事件。大多数重复的基因对是相连的,提示ChrA01、ChrA03和ChrA05之间可能发生染色体或片段复制(图5)。1).另外6对位于ChrA03和ChrA05上的CCCH基因对没有被认为是重复基因,因为CDS覆盖率或蛋白质鉴定不符合Yang的标准[51]和Sun [52,尽管它们与相应的拟南芥基因同源(附加文件)2).所有Ka/Ks比值均显著< 0.5,为阴性选择结果。基因复制时间估计约为0.29-19.58年(百万年前)(附加文件2).
CCCH锌指蛋白的系统发育分析b·拉伯
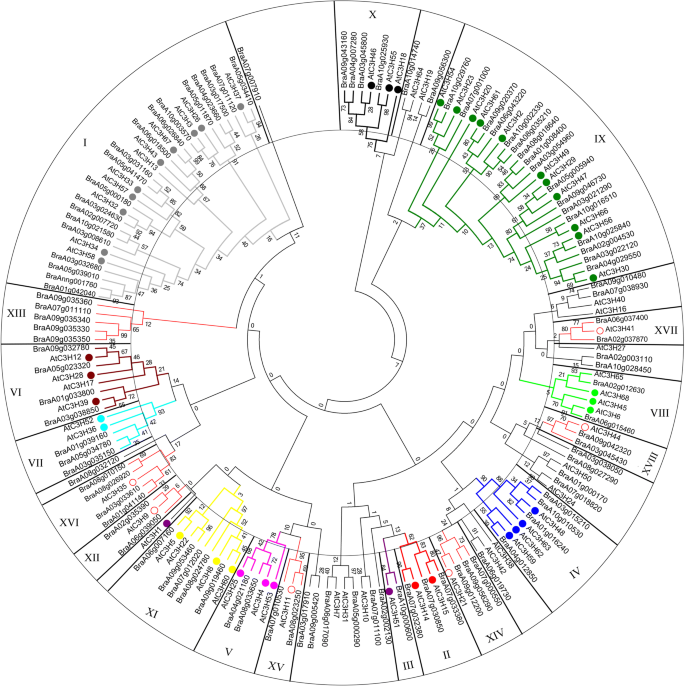
进一步研究CCCH基因的特征b·拉伯,采用最大似然法(Maximum Likelihood, ML)对103条CCCH锌指蛋白序列进行系统发育分析(图3)。3.;附加文件1和3.;[23])。基于拟南芥分类系统[23, 103个CCCH锌指蛋白中的84个可分为18个不同的亚家族。亚科i有19个成员是最大的,其次是亚科IX,有17个成员,而亚科iii只有一个成员。亚家族xviii包含具有最长序列的蛋白质(图。4.).CCCH锌指蛋白的分布b·拉伯由最大似然法构造的系统发育树类似于邻近拟南芥的邻近(NJ)制作的拟南芥 -b·拉伯(附加文件4.)或ML系统发育树拟南芥-b·拉伯大米(附加文件5.).
CCCH蛋白的系统发育树拉伯。这棵树包括来自芥蓝和拟南芥68。84个CCCH蛋白b·拉伯可分为18个亚科,亚科I-XI是根据参考文献[23],亚科XI-XVIII为新组。实圈显示拟南芥CCCH蛋白在I-XI亚家族中。蛋白序列用MUSCLE比对,系统发育树用MEGA 7.0分析。构建树的设置如下:统计方法为最大似然;处理:部分删除;替代模型:泊松模型;以及1000个内部分支可靠性的Bootstrap测试
CCCH蛋白的结构域组织拉伯。通过SMART和NCBI检测CCCH蛋白的结构域组织(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),关闭低复杂度过滤器,并将期望值设置为10。利用IBS1.0对结构域的位点信息进行分析,构建蛋白质组织结构图
的结构b·拉伯CCCH基因
为了了解基因多样化的进化,我们对基因结构进行了分析(图。5.).结果表明,CCCH基因的内含子/外显子数量变化范围较大,从1 ~ 16个不等,但每个亚家族的基因结构相对保守。例如,亚家族i基因总是包含5到7个外显子BraA05g011870, BraA05g034410和BraA07g011120.亚家族6和亚家族7基因分别包含2个和3个外显子。ix亚家族的所有基因结构都只有一个外显子,没有内含子。有趣的是,一些未分组的基因有更复杂的基因结构。重复基因对主要分布在亚家族i和IX中,但亚家族vii /XIV成员均为3倍基因。大多数复制基因在姐妹基因之间或姐妹基因之间表现出相似的基因结构。
基因的结构CCCH基因在拉伯。利用基因结构显示服务器(gene structure Display Server,http://gsds.cbi.pku.edu.cn./)
域的组织b·拉伯锌指蛋白
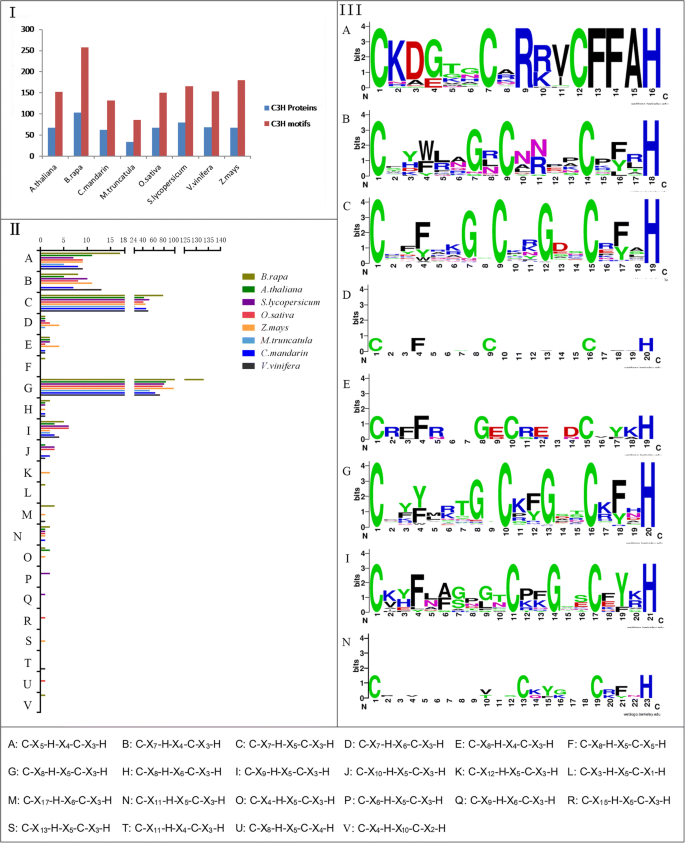
为了确定CCCH锌指蛋白的进化和保守性,利用全长蛋白对结构域进行了解析。亚家族间结构域的组织结构存在显著差异,甚至CCCH基序的类型和数量也存在显著差异b·拉伯.总共有15种类型和257种CCCH图案(C-X3 - 17cx4-10cx1 - 5- h)。其中,cx7/8cx5.- c3.-H是最常见的基序(Figs。2和4.).一般来说,每个CCCH锌指蛋白携带1-6个CCCH基序副本(图。4.).cx7/8cx5.- c3.-H motif主要存在于亚家族i、VI、VII、VIII、XI和XIII。与拟南芥相反,C-X7.cx6.cx3.- h和cx10cx5.cx3.-H主题没有出现在b·拉伯虽然小说图案c-x17cx6.cx3.-h在Braa07g000550和Subfamily-xiv的Braa09g012200中找到。c-x17cx6.- c3.-H基序在拟南芥中不存在,但在玉米基因组中存在[20.].这部小说的主题是C-X3.cx5.- c1- h, cx17cx6.- c3.- h, cx8.cx5.- c5.- h和cx4.cx10- c2-H在未分类蛋白BraA10g028450、BraA09g056300、BraA01g000170和BraA02g003110中分别存在。除CCCH基序外,WD40 (WD或beta-转导蛋白重复序列)、ANK(锚蛋白重复序列)、RRM (RNA识别基序)和RING(真正有趣的新基因)域分别存在于- IV、IX、X/XI、XVI亚家族中。
保守的应激反应亚家族ix成员
拟南芥RR-TZF基因(Subfamily-IX)在植物生长、发育和胁迫反应中发挥关键作用,可能通过靶向3 ' UTR富含au的RNA元件,并召集分解机制触发mRNA降解[34].中相应的ix亚家族同源物的特征b·拉伯,利用蛋白质序列构建系统发育树和结构域,并测定其对ABA、干旱和盐胁迫的转录反应(fig .;6.和7.).
拟南芥和b·拉伯RR-TZF蛋白可分为两组(图。6.a),由于高度保守的ankyin重复序列(ANK域),它通过介导蛋白-蛋白相互作用对所有已知的Notch信号通路都是必不可少的[53].Group-2成员包含两个富含精氨酸的motif (RR)和TZF域,而group-1成员包含另外两个或三个ANK重复序列。b·拉伯RR-TZF蛋白含有保守的C-X7 - 8cx5.cx3.- h和cx5.cx4.cx3.-H基序和一个RR基序,其中包含一个保守的C-X基序5.-H-X4.cx3.(图- h的主题。6.b).我们使用了Wang等人的程序[23]检测核导出信号(NES)b·拉伯RR-TZF蛋白,发现所有成员均含有推定的NES序列,提示它们可能是参与信号转导的核质穿梭蛋白(图。6.c)。
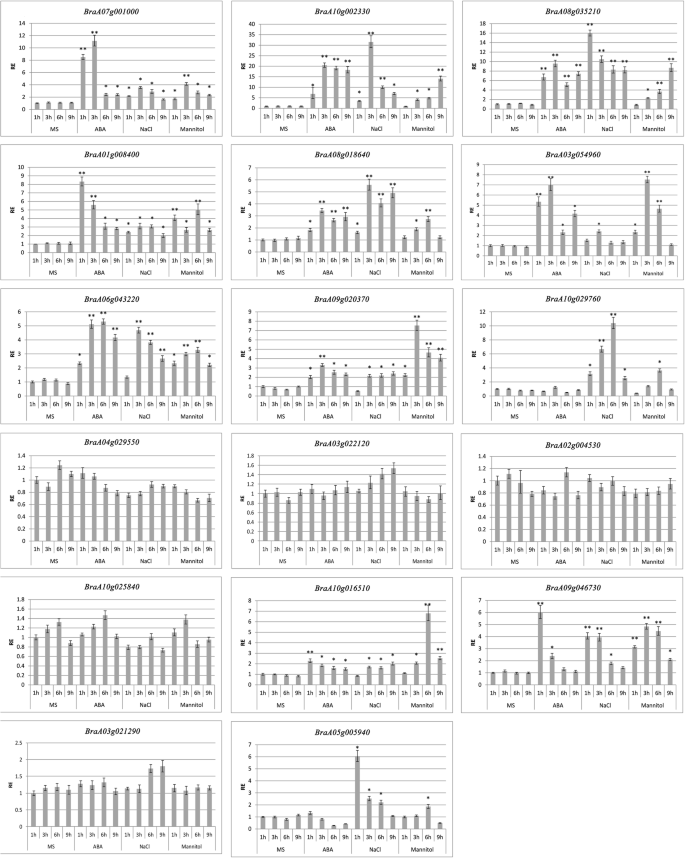
的表达式概况RR-TZF基因在b·拉伯利用实时荧光定量PCR检测4个不同发育阶段的叶片。7.).在NaCl、ABA和甘露醇处理下,17个RR-TZF基因中有12个基因的表达量显著升高。然而,BraA03g021290, BraA03g022120, BraA04g029550, BraA10g025840没有明显的表达变化,所以它们可能没有参与对这些压力的反应。除上述5个基因和2个基因外,大多数基因在ABA条件下的表达量均显著高于对照(MS)BraA10g029760和BraA05g005940。在ABA响应基因中,Braa07g001000.那BraA01g008400那BraA03g054960和BraA09g046730表达量显著高于NaCl和甘露醇胁迫下的表达量。BraA10g029760和BraA05g005940NaCl易于诱导,而BRAA09G020370是由甘露醇胁迫引起的。大多数b·拉伯RR-TZF基因在3 ~ 6 h内表达量增加,3 ~ 6 h后表达量下降BraA01g008400和BraA09g046730在ABA胁迫下BraA08g035210和BraA05g005940under NaCl stress, which showed expression peaks after only 1 h.
讨论
识别和分类b·拉伯锌指蛋白
芸薹属作物是重要的经济作物,也是多倍体进化的经典模式植物。b·拉伯,是最重要的蔬菜作物和基因组模式生物之一,可能是a亚基因组的假定贡献者(b . oleracea提供C-subgenome,甘蓝型油菜,混合后代b·拉伯和b . oleracea,包含A和c亚基因组)[49].以往的研究表明转录因子b·拉伯被显着过度保留[49].CCCH基因作为多种生物中普遍存在的调节因子,通过与DNA、RNA或蛋白质相互作用,在植物发育和胁迫反应中发挥作用[22那23].Rameneni等人刚刚鉴定出63个CCCH基因b·拉伯[54,比预期的数量少得多[49].在这里,我们识别并确认了103个CCCH基因b·拉伯(无花果。1额外的文件1),比拟南芥中的68丰富得多(N= 5, [23]), 67米(N= 12,23], 68在玉米(N= 10,20.]甚至认为植物物种与近两倍染色体数目,如在91杨树(N(= 19日44]),葡萄为69 (n = 19, [43]),以及类似的编号,以四倍体柳枝[48].
植物CCCH锌指图案显示一个宽间距模式,如C-X4-15cx4 - 6cx3.- h (23].在本研究中发现15种类型的CCCH基序b·拉伯(图2和4.).类似于其他植物物种,c-x7/8cx5.cx3.-H motif是最丰富的模式(82.10%),与拟南芥(82.24%)、玉米(79.44%)和水稻(78.67%)相似6.).相比于其他植物物种,一些CCCH图案缺失,而其他新的CCCH基序在进化b·拉伯.例如,与拟南芥相比,C-X10cx5.cx3.- h和cx7.cx6.cx3.-h没有检测到motifb·拉伯,而小说C-X17cx6.cx3.- h, cx8.cx5.cx5.- h, cx4.cx10cx2- h和cx3.cx5.- c -1-H基序;与水稻相比,b·拉伯有额外的cx/ 8/17cx6.cx3.-h,c-4.cx5.cx3.- h cx8.cx5.cx5.- h, cx4.cx10cx2- h和cx3.cx5.- c1-h主题,但缺乏c-x10/15cx5.cx3.- h cx8.cx5.cx4.- h和cx7.cx6.cx3.-h motif [23];与玉米,C-X相比8.cx5.cx5.- h, cx4.cx10cx2- h和cx3.cx5.- c1-H基序是新颖的在b·拉伯,但cx12/13cx5.cx3.- h和cx7.cx6.cx3.-h没有找到图案[20.].这些结果提示保守的C-X7/8cx5.cx3.-H基序在CCCH蛋白家族中起着重要的功能作用。除CCCH基序外,还发现了大量其他调控域,如RING、WD40、RRM和KH (K同源基序)b·拉伯CCCH蛋白(无花果。4.).有趣的是,大多数这些额外的结构域都涉及到与RNA、DNA或蛋白质的相互作用[28那55那56那57那58那59那60].
b·拉伯基因平均转录本长度为2015 bp,编码序列长度为1172 bp,平均每个基因有5.03个外显子[49].b·拉伯CCCH基因的内含子/外显子数目变化很大,从1 - 16个不等。大多数序列的外显子少于10个,平均每个基因有5.72个外显子(图1)。5.).此外,与玉米相似,玉米的外显子长度范围b·拉伯CCCH基因非常广泛。综上所述,这些结果表明CCCH基因之间的基因结构和功能高度多样化[43].然而,基因结构和结构域组织在每个亚家族中相对保守,表明亚家族成员的功能冗余。
复制和进化b·拉伯CCCH基因
CCCH家族似乎经历了复杂的进化过程,成为植物中最大的基因家族之一[23那44].芸苔属植物由于其与拟南芥谱系的分歧至少13-17 mya,基因组经历了额外的全基因组三次(WGT)。超过90%的人b·拉伯基因组是同位的拟南芥基因组[49].以前的研究还表明三重一起芸苔属植物基因组片段从一个共同的祖先分化出来芸苔属植物谱系分化,大约35%的这些基因已经丢失,最有可能是通过一种分散模式中的删除机制[61].然而,编码信号转导或转录调节中涉及的蛋白质的基因在很大程度上保留了[49那61].环境因素可能在转录因子的保留中发挥重要作用[49].同样,CCCH基因家族的b·拉伯可能是因为基因组的三倍扩增。在此之后,基因家族可能仍然保留了大多数关键成员,因为它们在应对非生物或生物胁迫方面具有全面和重要的功能,尽管一些成员总体上有所减少,因为总数量仅为拟南芥的约1.5倍([49),无花果。2).芸苔属植物物种b . oleracea(2n = 20)和b .黑质(2n = 16) CCCH家族也有所扩展,分别拥有75,92个序列(附加文件3.那7.和8.).
令人惊讶的是,除了ChrA 09上的一个组外,所有17个重复的CCCH基因对都是分段重复,所有的姐妹基因都是拟南芥的同源基因,除了一对(图)。1;额外的文件2).计算重复基因的Ka、Ks和Ka/Ks比值,探索选择模式[20.那62].通常,比率<1表示阴性选择,比率= 1表示中性选择,而Ka / ks比例> 1表示正选择[20.].大多数of the resultant Ka/Ks ratios were significantly < 0.5 (Additional file2).这些结果与之前的报道高度一致,强烈提示大多数复制的CCCH基因都经历了提纯选择,复制事件发生后复制基因的功能并没有太大的差异[20.那44].复制基因对姊妹基因之间的相对保守基因结构和绝对保守结构域进一步证明了其保留功能(图1)。4.和5.).
估计的重复日期表明,在额外的整个基因组三份(WGT)之前可能发生9个基因对复制事件,并且在此之后可能发生其他基因对(附加文件)2;[49])。
领域组织多样性和假定功能分析
与蛋白质功能直接相关的结构域[63].拟南芥和南芥之间的结构域是保守的拉伯。大多数亚家族i蛋白高度保守,有5个C-X8.cx5.cx3.-H基元,如拟南芥AtC3H37 (HUA1, AT3G12680),通过优先结合聚rU和聚rG调控雄蕊、心皮和花的发育[27].亚家族ii保存在植物和动物之间,如拟南芥AtC3H14 (At1G66810)和AtC3H15 (At1G68200)含有典型的C-X8.cx5.cx3.-H-X18cx8.cx5.cx3.-H在动物TZFS中发现的主题。ATC3H14参与次壁生物合成[24].亚家族iii如拟南芥AtC3H1 (AT1G01350)与C-X8.cx5.cx3.-H基序也属于RING-finger家族,参与木质素生物合成[28那29].的b·拉伯Subfamily-III Homolog Braa10G000600具有保守的CCCH和环形域。所有亚家族IV蛋白,包括拟南芥和b·拉伯同源物,包含C-X7.cx4.cx3.- h或cx7.cx5.cx3.-H基序,加上另外的六种Wd40重复,这是蛋白质 - 蛋白质或蛋白质-DNA相互作用域[56].亚家族iv蛋白是植物特异性蛋白,可能参与叶绿素生物合成和光反应[23那25].亚科-V,在拟南芥中X和XI蛋白含有一个C-X7.cx5.cx3.-h主题和RNA识别基序(RRM)。RRM是真核生物中最常见的RNA结合结构域。含植物RRM的蛋白质参与了开花和适应热应激的调节[57那58那64].subfamily vi, BraA05g023320和BraA09g032780有一个C-X7.cx5.cx3.-H和两个C-X8.cx5.cx3.-H图案,而BraA01g033820和BraA03g038850有两个C-X7.cx5.cx3.h和一个C-X8.cx5.cx3.-h主题。亚家族 - vii蛋白,含有c-x7.cx5.cx3.cx - h,8.cx5.cx3.-H和一个保守的rna结合K同源基序(KH),在结构域上是保守的,可能具有反式激活和rna结合活性,这也被认为在拟南芥开花和衰老的调控中具有冗余作用[31那59].亚家族ix成员的特征是两个相同的C-X7 - 8cx5.cx3.- h和cx5.cx4.cx3.-H基序由16-18个氨基酸分开。ix亚家族蛋白参与植物生长、发育和胁迫反应[33那34].Subfamily-X和subfamily-XI具有相似的域组织。亚家族xv蛋白有两个C-X7.cx5.cx3.h和一个C-X8.cx4.cx3.- h。亚家族xvi蛋白有三个C-X7.cx5.cx3.- h, cx9.cx5.cx3.-H和蛋白结合的RING结构域。亚家族xvii蛋白有一个C-X7.cx5.cx3.-H和生长素抑制基序。除C-X外,还有两个亚家族xviii蛋白携带PHD(植物同源结构域)、SWIB (SWI/SNF复合物B)和GYF(甘氨酸-酪氨酸-苯丙氨酸)基序7.cx5.cx3.- h。除CCCH外,RING、PHD、RRM、ANK基序还负责蛋白质-蛋白质或蛋白质- dna或RNA的结合,提示CCCH蛋白可能参与了多种功能[53那57那58那64那65].
的反应b·拉伯RR-TZF基因对压力的影响
拟南芥和b·拉伯RR-TZF亚家族蛋白可能是参与信号转导的核质穿梭蛋白(图。6.c;[23])。拟南芥RR-TZF蛋白通过ABA、盐、冷、H等信号转导参与生长和胁迫响应2O.2,渗透,及糖类枯竭应力[34那66].AtTZF1、AtTZF2 (AtC3H20/AtOZF1, AT2G19810)和AtTZF3 (AtC3H49/AtOZF2, AT4G29190)在不同植物中高度保守,调控种子萌发及对ABA、茉莉酸甲酯(Methyl jasmonate, MeJA)、GA、氧化和盐胁迫的响应[34那35那36那37].AtTZF4 (AtC3H2/SOM, At1g03790)、AtTZF5 (AtC3H61, At5g44260)和AtTZF6 (AtC3H54/PEI1, AT5g07500)参与光、ABA、GA反应,调节种子萌发[66那67那68].AtTZF7 (AtC3H30, AT2G41900)、AtTZF8 (AtC3H56, AT5G12850)、AtTZF9 (AtC3H66, AT5G58620)、AtTZF10 (AtC3H29/AtSZF2, AT2G40140)和AtTZF11 (AtC3H47/AtSZF1, AT3G55980)参与非生物或生物胁迫耐受反应和胁迫诱导的开花过渡[34那69].有AtTZF4和AtTZF5两个拷贝,AtTZF3三个拷贝,但在b·拉伯(无花果。6.一个,附加文件2).在很多相似的基因结构和拟南芥之间TZF基因的域组织b·拉伯表明其保守的生物学功能(Figs。4.和5.).12个RR-TZF基因的表达谱b·拉伯牵连,它们可能参与ABA,干旱或盐胁迫应答(图7.).然而,即使在同源基因之间,它们对这些应激刺激的转录反应也是不同的。例如,响应BraA10g002330和BraA08g035210与同源基因相似吗AtTZF4,但BraA08g018640表现远远超过同源基因AtTZF3在NaCl压力条件下。这些结果表明一些同源基因功能可能分歧b·拉伯.ABA是植物响应非生物胁迫的关键信使,参与诱导ja依赖的防御反应和植物免疫反应的各种信号传导过程[70].表达分析结果表明,RR-TZF基因的响应于NaCl和甘露醇压力的速度快,但除外BraA08g035210那BraA08g018640那BraA10g029760和BraA05g005940NaCl胁迫Braa10G01510甘露醇压力。
结论
在本研究中,我们鉴定了103个CCCH基因b·拉伯.八八这些基因分为基于系统发育,基因结构和结构域组织分析结果18个亚科。基因的结构和结构域组织结果表明,CCCH基因的功能发散,但亚科成员中高度保守。有九个二倍体基因对七三倍体基因对,所有重复的基因是由于节段性重复。此外,表达谱分析的结果表明,亚家族-IX的成员可能在ABA,干旱和盐胁迫应答有关。
方法
的CCCH锌指蛋白基因和染色体图谱的构建鉴定
拟南芥中CCCH锌指蛋白的序列分析23作为查询来搜索Brassica Rapa.基因组(http://brassicadb.org/brad/)使用BLASTp工具。所有假定的CCCH锌指蛋白在SMART (http://smart.embl-heidelberg.de/)及NCBI (https://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)的SMART数据库(版本6.0)。关闭低复杂度过滤器,期望值设置为10 [71].CCCH基因座信息b·拉伯(染色体版本3.0,2018,http://brassicadb.org/brad/datasets/pub/Genomes/Brassica_rapa/V3.0/)用Mapchart 2.2程序生成染色体图谱[72].
基因结构、结构域和系统发育关系的分析
使用基因结构显示服务器(http://gsds.cbi.pku.edu.cn./)。利用结构域组织的位点信息,利用IBS1.0构建蛋白质组织示意图[73].
CCCH基序序列是从推定的CCCH锌指蛋白萃取,用ClustalW比(对齐http://www.genome.jp/tools/clustalw/),并将得到的文件被用来创建标识映射(http://weblogo.berkeley.edu/logo.cgi).
采用MUSCLE (Multiple sequence Comparison by Log- Expectation)程序对CCCH锌指蛋白进行多序列比对[74],结果文件使用MEGA 7.0程序进行系统发育分析[75].基于蛋白质全长序列,采用含有部分缺失和泊松模型的最大似然法(ML)构建树,并采用Bootstrap方法对内部分支的可靠性进行1000次重复检验。
编码CCCH锌指蛋白的重复基因
复制基因是根据Yang等人定义的[51:短全长cds序列覆盖及其编码氨基酸> 70%的基因对被认为是重复基因。串联重复是根据Sun等人定义的[52]:由10个或更少的非同系物分开的位于100 kb以内的重复基因被定义为串联重复基因。利用NCBI网站上的Blastn/Blastp确定CDS全长序列覆盖率和氨基酸特性[71].
通过DnaSP 6.0计算复制基因的非同义突变(Ka)和同义替换(Ks)的数量[62].通过分析重复基因之间的Ka/Ks比值,确定选择方式。复制时间(T,百万年前,MYA)计算为T = Ks/2λ × 10−6λ = 1.5 × 10−8为b·拉伯[76].
植物材料和胁迫处理
b·拉伯幼苗在25°C、16 h光照/8 h暗光周期下在1/2 MS板上生长。将3周龄、2-3片真叶的幼苗置于生长室内3 d进行驯化,然后再进行ABA、NaCl和甘露醇处理。应力处理是按照Lee等人的方法进行的[77].采收整株幼苗,将其置于含有250 mM NaCl、100 μM ABA或300 mM甘露醇的1/2 MS液体培养基中。以同一时间点未处理的幼苗为对照,分别在胁迫1、3、6和9 h取样检测基因表达响应。采集3个重复的幼苗样本。材料在液氮中迅速冷冻,并储存在−80°C,以供进一步分析。
RNA提取和实时定量RT-PCR
如前所述进行RNA提取和实时定量RT-PCR [71].根据制造商的规格,使用Trizol Reagent Kit(Invitrogen,Carlsbad,CA)从样品中提取总RNA。使用琼脂糖凝胶电泳和溴化乙烯染色来评估RNA完整性。然后用DNA酶I处理RNA制剂,通过使用寡核苷酸(DT)底漆和RT酶(Thermo Fisher,USA)进行第一次CDNA的链合成。
与SYBR-green荧光定量实时PCR进行了使用CF×96实时系统(BIORAD) 20μl PCR反应混合物,包括8.8μl(稀释cDNA、2×10μl由于“快速上手”项目普遍SYBR绿色大师(火箭)(瑞士罗氏公司)和0.6μl(正向和反向引物(附加文件9.).的BraA02g003190基因作为参照基因。每个样品都进行了三份分析。在PCR循环结束时,进行熔化曲线分析,以验证预期PCR产物的具体生成。用2−法计算RR-TZF基因的表达量ΔΔct.方法(78].
缩写
- 阿坝:
-
脱落酸
- ANK:
-
锚蛋白重复
- 是:
-
富裕的元素
- b .黑质:
-
芸苔属植物黑质
- b . oleracea:
-
芸苔属植物oleracea
- b·拉伯:
-
Brassica Rapa.
- 遗传算法:
-
赤霉素酸
- GYF:
-
Glycine-tyrosine-phenylalanine
- KH:
-
K同源性主题
- 惩罚:
-
甲基jasmonate
- ML:
-
最大似然
- 米娅:
-
几百万年前
- NES:
-
核导出信号
- NJ:
-
Neighbor-joining
- PBs:
-
处理尸体
- 博士:
-
植物Homeo域
- 戒指:
-
有趣的新基因
- RR:
-
Arginine-rich
- RRM:
-
RNA识别主题
- RR-TZF:
-
富精氨酸图案串联CCCH锌指
- SGS:
-
压力颗粒
- SWIB:
-
瑞士/ SNF复杂B
- TF:
-
转录因子
- TNFα:
-
肿瘤坏死因子α
- TTP:
-
Tristetraprolin
- TZF:
-
串联CCCH锌指
- WD40:
-
WD或β -转导蛋白重复
- 重量:
-
全基因组三倍
参考文献
- 1.
Latchman DS。转录因子:综述。国际生物化学杂志。1998;29(12):1305-12。
- 2.
Bakthisaran R, Tangirala R, Rao CM。小热休克蛋白:在细胞功能和病理中的作用。生物物理学报。2015;1854(4):291-319。
- 3.
Devaiah Bn,Karthikeyan As,Raghothama Kg。Wrky75转录因子是拟南芥磷酸盐采集和根部发育的调节剂。植物理性。2007; 143(4):1789-801。
- 4.
高志华,杨树华,朴雅,勒鲁塞尔·欧,韩国汉。ANAC012是植物特异性NAC转录因子家族的成员,负调控木质素纤维的发育拟南芥.植物j . 2007;(6): 1035 - 48。
- 5.
Giovannoni JJ。果实发育和成熟的遗传调控。植物细胞。2003:s170 - 80。
- 6.
Le BH,Cheng C,Bui AQ,Vagmaister Ja,Henry Kf,Pelletier J,Kwong L,Belmonte M,Kirkbride R,Horvath S,Drews GN,Fischer RL,Okamuro JK,Harada JJ,Goldberg RB。拟南芥种子发展基因活性的全局分析及种子特异性转录因子鉴定。Proc Natl Acad Sci U S A. 2010; 107(18):8063-70。
- 7.
Hinderhofer K,转录因子的Zentgraf酒店U.鉴定在叶子衰老的开始特异性表达。Planta。2001; 213(3):469-73。
- 8.
在拟南芥中,转录因子FLC通过抑制分生组织能力和系统信号转导来调控开花对春化的响应。基因Dev。2006;20(7):898 - 912。
- 9.
花形态发生的关键发育转变及其调控。Curr Opin Genet Dev. 2017; 45:44-50。
- 10.
Tsuda K,Somssich Ie。植物免疫中的转录网络。新植物。2015; 206(3):932-47。
- 11.
刘志强,刘志强,杨志强。定义植物免疫反应的转录事件。植物学报。2017;38:1-9。
- 12.
郭艳红,于亚平,王东,吴彩霞,杨国光,黄建刚,郑春成。棉花ccch型锌指蛋白GhZFP1与转基因烟草GZIRD21A和GZIPR5互作,增强烟草耐盐胁迫和抗真菌病能力。新植醇。2009;183(1):62 - 75。
- 13.
Lindemose S,O'Shea C,Jensen MK,Skriver K.结构,功能和转录因子的结构和网络涉及非生物应激反应。int j mol sci。2013; 14(3):5842-78。
- 14.
Kovalchuk N, Chew W, Sornaraj P, Borisjuk N, Yang N, Singh R, Bazanova N, Shavrukov Y, Guendel A, Munz E, Borisjuk L, Langridge P, Hrmova M, Lopato S.小麦同源结构域转录因子TaHDZipI-2调控转基因大麦的耐霜性、开花时间和穗部发育。新植醇。2016;211(2):671 - 87。
- 15.
王凯,丁玉英,蔡超,陈志强,朱超。C2H2锌指蛋白在植物对非生物胁迫响应中的作用。杂志。2018。https://doi.org/10.1111/ppl.12728.
- 16.
米勒Ĵ,McLachlan的AD,在从爪蟾卵母蛋白转录因子IIIA克鲁格A.重复锌结合结构域。EMBO J. 1985; 4(6):1609至1614年。
- 17.
克鲁格A,罗德D.锌指:一种新的蛋白质基序核酸识别。趋势Biochem SCI。1987; 12(4):464-9。
- 18.
Laity JH, Lee BM, Wright PE。锌指蛋白:结构和功能多样性的新见解。中国生物医学工程学报。2001;11(1):39-46。
- 19.
台湾海陆运输公司。锌指蛋白RNA识别的多种模式。中国生物医学工程学报。2005;15(3):367-73。
- 20。
张赵彭X, Y,曹J, W,江H,李X,妈妈问,朱年代,程b CCCH-type锌指家庭在玉米基因组鉴定、分类和表达分析下脱落酸和干旱的治疗方法。《公共科学图书馆•综合》。2012;7 (7):e40120。
- 21。
Cys2His2锌指蛋白的DNA识别。生物物理学报。2000;29(1):183-212。
- 22。
赖WS,卡瓦略E,索恩JM,肯宁顿EA,布莱克希尔PJ。CCCH锌与基因指蛋白的相互作用。tristetraprolin相关的锌指蛋白结合丰富的AU-元素和mRNA的不稳定。生物化学杂志。2000; 275(23):17827-37。
- 23。
王东,郭艳,吴超,杨国刚,李勇,郑春华。拟南芥和水稻CCCH锌指家族的全基因组分析。BMC基因组学,2008;9(1):44 - 63。
- 24.
Kim WC, Kim JY, Ko JH, Kang H, Kim J, Han KH。AtC3H14是一种植物特异性串联CCCH锌指蛋白,以序列特异性的方式与靶mrna结合并影响细胞的伸长拟南芥.工厂j . 2014, 80(5): 772 - 84。
- 25.
ZFWD:含有一个C3H锌指和7个WD40重复序列的植物蛋白的一个新亚家族。基因。2000;260(1 - 2):45-53。
- 26.
Tsukumo Y, Tsukahara S, Furuno A, Iemura S, Natsume T, Tomida A. TBL2通过其WD40域与ATF4 mRNA结合并调节其在内质网应激中的翻译。细胞生物学杂志。2016;117(2):500-9。
- 27.
李军,贾东,陈晓华。拟南芥雄蕊和心皮特性的调控因子。核RNA结合蛋白的编码。植物细胞。2001;13(10):2269 - 81。
- 28.
Franke R,Hemm Mr,Denault JW,Ruegger Mo,Humphreys JM,Chapple C.在拟南芥Ref8突变体中次生新陈代谢和沉积异常木质素的变化。工厂J. 2002; 30(1):47-59。
- 29.
Kosarev P,Mayer Kf,Hardtke CS。拟南芥基因组编码的戒指域的评价与分类。基因组Biol。2002; 3(4):1-12。
- 30.
关键词:拟南芥,根毛密度,根毛长度,多样性,磷响应《公共科学图书馆•综合》。2015;10 (3):e0120604。
- 31.
关键词:拟南芥,拟南芥,锌指结构域,k -同源蛋白,调控,开花,衰老植物学报。2017;95(6):1-17。
- 32.
徐飞,张永强,李晓霞。mos4相关复合物中两种rna结合蛋白的非等量遗传冗余功能。植物杂志。2010;154(4):1783 - 93。
- 33.
Bogamuwa SP, Jang JC。串联CCCH锌指蛋白在植物生长,发育和应力反应中。植物细胞生理。2014; 55(8):1367-75。
- 34.
jang jc。富有精氨酸的主题串联CCCH锌指蛋白在植物应激反应和基因表达后的转录后调节。植物SCI。2016; 252:118-24。
- 35.
Lin PC, Pomeranz MC, Jikumaru Y, Kang SG, Hah C, Fujioka S, Kamiya Y, Jang JC。拟南芥锌指串联蛋白AtTZF1影响ABA和ga介导的生长、胁迫和基因表达响应。植物j . 2011; 65(2): 253 - 68。
- 36.
李世杰,郑海杰,姜赫,金诗。拟南芥锌指蛋白AtC3H49/AtTZF3和AtC3H20/AtTZF2参与ABA和JA反应。植物生理学杂志。2012;53(4):673-86。
- 37.
D'Orso F, De Leonardis AM, Salvi S, Gadaleta A, Ruberti I, Cattivelli L, Morelli G, Mastrangelo AM。保护AtTZF1那AtTZF2,AtTZF3同源基因在盐胁迫下的调控。植物学报。2015;6(1):394-410。
- 38.
邱嗯,刘谦,李江,张Y,王楼,高J.生理和中国白菜转录反应(Brassica Rapa.l . ssp。对盐胁迫的影响。中华医学杂志。2017;18(9):1953-71。
- 39.
李锐。胚珠、小孢子和成熟花粉的比较转录分析芸苔栗鸟.植物学报。2010;72(3):279-99。
- 40.
(Yu S ., Zhang F ., Yu Y ., Zhang D ., Zhao X ., Wang W. Chinese cabbage in water stress的翻译结果:Brassica Rapa.l . ssp。通过标签测序。植物学报2012;30(1):17-28。
- 41.
柑橘ccch型锌指基因家族的综合分析克莱门泰普通话)通过全基因组表征。Mol Gen Genomics。2014; 289(5):855-72。
- 42.
徐瑞。茄CCCH锌指家族胁迫响应基因的全基因组分析与鉴定。中国生物医学工程学报。2014;29(5):945 - 948。
- 43。
王小林,钟毅,程志明。CCCH锌指基因家族的进化及表达分析葡萄.植物基因组。2014;第16 - 25(3):7。
- 44。
柴刚,胡锐,张东,齐刚,左锐,曹勇,陈鹏,孔勇,周刚杨树trichocarpa).BMC基因组学。2012; 13(1):253-74。
- 45。
张C,张H,赵义,江H,朱S,程B,Xiang Y.CCCH锌指基因家族在Medicago truncatula的基因组分析。植物细胞批准。2013; 32(10):1543-55。
- 46。
Pradhan S, Kant C, Verma S, Bhatia S.对鹰嘴豆中CCCH锌指家族的全基因组分析确定了组织特异性和应激反应候选基因(中投arietinumL.)。Plos一个。2017; 12(7):E0180469。
- 47。
姜爱玲,徐志松,赵国英,崔学勇,陈敏,李凌龙,马永忠。C3H锌指转录因子家族的全基因组分析及其成员的干旱响应山羊草属tauschii.植物学报2014;32(6):1241-56。
- 48.
袁胜,徐斌,张军,谢志成,杨志强,蔡强,黄斌。四倍体柳枝稷中ccch型锌指家族基因的综合分析有助于功能基因的发现和最近异源多倍体化事件的发生。BMC基因组学。2015;16(1):129 - 44。
- 49.
王X,王H,王建,孙R,吴Ĵ,刘S,佰Y,门JH,班克罗夫特我,程楼黄S,李X,华W,王静,王X,弗里男,皮雷JC那Paterson AH, Chalhoub B, Wang B, Hayward A, Sharpe AG, Park BS, Weisshaar B, Liu B, Li B, Liu B, Tong C, Song C, Duran C, Peng C, Geng C, Koh C, Lin C, Edwards D, Mu D, Shen D, Soumpourou E, Li F, Fraser F, Conant G, Lassalle G, King GJ, Bonnema G, Tang H, Wang H, Belcram H, Zhou H, Hirakawa H, Abe H, Guo H, Wang H, Jin H, Parkin IA, Batley J, Kim JS, Just J, Li J, Xu J, Deng J, Kim JA, Li J, Yu J, Meng J, Wang J, Min J, Poulain J, Wang J, Hatakeyama K, Wu K, Wang L, Fang L, Trick M, Links MG, Zhao M, Jin M, Ramchiary N, Drou N, Berkman PJ, Cai Q, Huang Q, Li R, Tabata S, Cheng S, Zhang S, Zhang S, Huang S, Sato S, Sun S, Kwon SJ, Choi SR, Lee TH, Fan W, Zhao X, Tan X, Xu X, Wang Y, Qiu Y, Yin Y, Li Y, Du Y, Liao Y, Lim Y, Narusaka Y, Wang Y, Wang Z, Li Z, Wang Z, Xiong Z, Zhang Z. The genome of the mesopolyploid crop speciesBrassica Rapa..Nat麝猫。2011;43:1035-9。
- 50.
Panchy n,Lehti-shiu m,shiu sh。基因重复在植物中的演变。植物理性。2016; 171(4):2294-316。
- 51.
杨S,张X,岳JX,田d,陈JQ。最近重复主宰编码NBS-两个木本物种基因扩张。Mol Gen Genomics。2008; 280(3):187-98。
- 52.
孙锐,王凯,郭涛,张斌,王强。植物生长素响应因子(auxin response factor, ARF)基因的全基因组鉴定及其在植物组织中的显著表达Gossypium raimondii就.Funct综合基因组学。2015; 15(4):481-93。
- 53.
锚定位点遗传与生物化学;对锚定域的结构分析揭示了现有的数据。摩尔细胞。2004;13(5):619 - 26所示。
- 54.
Rameneni JJ, Dhandapani V, Paul P, Devaraj SP, Choi SR, Yi SY, Kim M, Hong S, Oh SH, Oh MH, Lim YP。CCCH锌指型转录因子的综合分析Brassica Rapa.基因组。环境科学学报。2018;59(5):729-47。
- 55.
王志强,程凯,万丽,姜洪涛,刘树华,雷勇,廖波。豆科植物基本亮氨酸拉链(bZIP)转录因子基因家族的全基因组分析。BMC基因组学。2015;16(1):1053 - 67。
- 56.
Xu C,Min J.WD40域蛋白的结构和功能。蛋白质细胞。2011; 2(3):202-14。
- 57.
P34蛋白的RNA识别基元(RRM)域的非典型结合作用调节了5S核糖核蛋白颗粒(5S RNP)内的结合。《公共科学图书馆•综合》。2017;12 (5):e0177890。
- 58.
Lee Kc,Jang Yh,Kim Sk,Park Hy,Thu MP,Lee Jh,Kim JK。拟南芥剪接因子SF1的RRM结构域对于特定基因组的前mRNA剪接是重要的。植物细胞批准。2017; 36(7):1083-95。
- 59.
格兰汀NV。kh域:一个图案,两倍。核酸RES。2001; 29(3):638-43。
- 60。
环指结构域:序列结构族的一个最近的例子。中国生物医学工程学报。1996;6(3):395-401。
- 61.
城市CD,张F, Maiti R, Crabtree J, Haas BJ, Wortman JR, Hine EE, Althoff R, Arbogast TS, Tallon LJ, Vigouroux M, Trick M, Bancroft .比较基因组学芸苔属植物oleracea和拟南芥揭示多倍体后的基因丢失、碎片化和分散。植物细胞。2006;18(6):1348 - 59。
- 62.
DnaSP 6:大数据集的DNA序列多态性分析。生物医学进展。2017;34(12):3299-302。
- 63.
奥伦戈C A。利用结构域家族预测蛋白质功能。Bmc生物信息学。2013;14 (S3): S5。
- 64.
Martínez-Lumbreras S, Taverniti V, Zorrilla S, Séraphin B, Pérez-Cañadillas JM。Gbp2通过一种新型的RRM域与THO/TREX相互作用。核酸学报2016;44(1):437-48。
- 65.
德赛RJ,洋枣CA.RING结构域E3泛素连接酶。Annu启生物化学。2009; 78:399-434。
- 66.
Bogamuwa SP, Jang JC。拟南芥CCCH锌指串联蛋白AtTZF4、5和6参与光、脱落酸和赤霉素介导的种子萌发调控。植物学报。2013;36(8):1507-19。
- 67。
李Z,托马斯TL。PEI1,用于在拟南芥心脏期胚胎形成所需胚特异性锌指蛋白基因。植物细胞。1998; 10(3):383-98。
- 68。
Kim DH, Yamaguchi S, Lim S, Oh E, Park J, Hanada A, Kamiya Y, Choi G.拟南芥CCCH-type锌指蛋白SOMNUS负调控PIL5下游光依赖种子萌发。植物细胞。2008;20(5):1260 - 77。
- 69。
魏胜,魏平,金建华,欧伟。植物对逆境逃逸的耐受性:OXS2锌指转录因子家族的作用。EMBO j . 2011; 30(18): 3812 - 22所示。
- 70.
Nguyen D, Rieu I, Mariani C, van Dam NM。植物如何处理多重胁迫:非生物胁迫和昆虫草食反应的激素相互作用。acta botanica sinica(云南植物学报). 2016;40(6):727-40。
- 71.
黄y,莫y,陈p,袁x,孟f,朱S,刘z.鉴定含域蛋白质的鉴定Gossypium raimondii就及其对高温胁迫的反应。SCI批准。2016; 6:32729。
- 72.
Voorrips RE. MapChart:图形化表达连锁图和qtl的软件。J在这里。2002;93(1):77 - 8。
- 73.
马谢刘W, Y, J,罗X,聂P,左Z, Lahrmann U,赵问,郑Y, Y,雪Y,任J .肠易激综合症:一个插画家生物序列的表示和可视化。生物信息学,2015,31(20):3359 - 61。
- 74.
Li W, Cowley A, Uludag M, Gur T, McWilliam H, Squizzato S, Park YM, Buso N, Lopez R. EMBL-EBI生物信息学网络和编程工具框架。核酸学报2015;43(1):580-4。
- 75.
田村K,施特歇尔G,彼得森d,Filipski A,库马尔S. EGA6:分子进化遗传学分析版本6.0。mol Biol Evol。2013; 30(12):2725-9。
- 76.
关键词:拟南芥,拟南芥属,查尔酮合成酶,醇脱氢酶,进化分析中国生物医学工程学报。2000;17(10):1483-98。
- 77.
Lee SC, Lim MH, Kim JA, Lee SI, Kim JS, Jin M, Kwon SJ, Mun JH, Kim YK, Kim HU, Hur Y, Park BS。转录组分析Brassica Rapa.在非生物胁迫下应用芸苔属24K寡核苷酸芯片。摩尔细胞。2008;26(6):595 - 605。
- 78.
Livak KJ, Schmittgen TD。利用实时荧光定量PCR技术对相关基因表达数据进行分析——ΔΔCT方法。方法。2001;25:402-8。
确认
感谢来自中国Edanz集团(中国)Liwen Bianji的Robbie Lewis MScwww.liwenbianji.cn/ac),编辑本手稿的草稿。
资金
本研究由湖南大学青年骨干教师培训项目(黄勇)资助(水稻抗性与表观遗传机制)芸苔属植物2015年)。
数据和材料的可用性
本研究期间生成或分析的所有数据都包含在此已发布的文章中及其附加文件中。在当前研究期间生成和分析的数据集可从相应的作者获得合理的请求。
作者信息
隶属关系
贡献
BY和XH贡献了大部分实验并起草了手稿;RY和HY构思并指导了这项研究;HY和JC撰写稿件的最终版本;所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
种子b·拉伯均来自湖南省植物遗传与分子生物学重点实验室。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
额外的文件1:
CCCH基因家族的详细信息B.rapa.CCCH基因和蛋白质的序列和信息来源http://brassicadb.org/brad/ (xlsx51 kb)
额外的文件2:
CCCH复制的基因b·拉伯.拟南芥与拟南芥之间的同步分析b·拉伯被发现在http://brassicadb.org/brad/searchSyntenytPCK.php;KA和KS由DNASP 6.0计算;CD覆盖并通过NCBI检查了蛋白质的鉴定。(XLSX 15 KB)
额外的文件4:
NJ系统发育树拟南芥-b·拉伯.用ClustalX(1.83)对蛋白序列进行比对,用MEGA 7.0进行系统发育树分析。构建树的设置如下:统计方法为Neighbor-joining;包括位点作为部分删除选项进行全序列分析;替换模型:p-distance;以及1000个内部分支可靠性的Bootstrap测试。(JPG 1481 kb)
额外的文件5:
ML的系统发育树Arabidopsis-B。拉伯大米。238个CCCH蛋白序列(拟南芥68,b·拉伯103那大米那67)用MUSCLE进行比对,MEGA 7.0进行系统发育树分析。构建树的设置如下:统计方法为最大似然;包括位点作为部分删除选项进行全序列分析;替代模型:泊松模型;以及500个内部分支可靠性的Bootstrap测试。(JPG 7157 kb)
额外的文件6:
植物中不同CCCH类型的数量。(XLSX 10 kb)
额外的文件7:
NJ系统发育树拟南芥- b . oleracea。143个蛋白质的序列(拟南芥68,b . oleracea75)用MUSCLE进行比对,MEGA 7.0进行系统发育树分析。构建树的设置如下:统计方法为Neighbor-joining;包括位点作为部分删除选项进行全序列分析;替换模型:p-distance;以及1000个内部分支可靠性的Bootstrap测试。(JPG 623 kb)
额外的文件9:
中IX亚家族的引物列表拉伯。(XLSX 12 kb)
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Pi, B, He, X, Ruan, Y。et al。CCCH锌指家族基因的全基因组分析及胁迫响应表达Brassica Rapa..BMC植物杂志18,373(2018)。https://doi.org/10.1186/s12870-018-1608-7
收到了:
公认:
发表:
关键字
- CCCH锌指族
- 串联CCCH锌指
- 进化
- 非生物胁迫
- Brassica Rapa.