抽象的
背景
叶绿体是地球上最重要的细胞器之一,它通过光合作用使生命形式成为可能。这些细胞器拥有一个带有许多负责自我调节的编码基因的环状基因组。trna是一个重要的进化保守基因家族,负责蛋白质翻译。然而,在叶绿体基因组中,tRNA的机制知之甚少。
结果
在本研究中,六种单子叶植物的叶绿体基因组,oryza nivara.(NC_005973),栽培稻(NC_001320),Sachharum OfficinArum.(NC_006084),高粱二色的(nc_008602),Triticum aestivum.(nc_002762),和Zea Mays.(NC_001666)下载并分析以鉴定TRNA序列。进一步分析单子叶植物的叶绿体基因组中的TRNA序列导致鉴定几种新功能。单子叶植物叶绿体基因组中的TrNA的长度范围为59至155个核苷酸。配对序列对齐揭示了保守的A-C-X-U-A-X-U-A-X-U-X的存在5.-u-a-a核苷酸共有序列。另外,单子叶植物的叶绿体基因组中的TrNA在基因组中含有21-28个抗密码子。他们还含有I内含子和用于TRNA的C-A-U抗密码子伊尔,是tRNA常见的反密码子遇见.进化分析表明,叶绿体基因组中的tRNA是从多个共同祖先和tRNA进化而来的遇见似乎是经过重复和多样化的祖先TRNA,以产生其他TRNA。
结论
从叶绿体TRNA的研究中获得的结果将大大有助于增加我们对新水平的TRNA生物学的理解。报告的单胞片植物叶绿体TRNA的新颖方面的功能研究将极大地帮助破译其在多种细胞过程中的作用。
背景
叶绿体是多拷贝细胞细胞器[1]这责对调节我们生物圈的光合营养植物中的光合作用和碳水化合物代谢[2那3.].它们是一个活跃的代谢中心,通过光合作用将太阳能转化为碳水化合物,负责维持地球上的生命[4.那5.那6.].除了光合作用的主要过程外,叶绿体还在其他各种分子过程中发挥重要作用;包括核苷酸、氨基酸、脂肪酸、维生素、植物激素和其他几种代谢物的合成[7.那8.那9.那10.那11.那12.].此外,它们还有助于氮和硫的同化[13.那14.那15.].在植物中,这些代谢物已被证明在生理学,生长和发展的调节中起着关键作用;以及压力反应。因此,叶绿体可以被视为细胞反应的“代谢中心”。进化研究表明,叶绿体通过真核细胞内的内化而产生的叶绿体祖先,并在植物细胞内保持了一个独立的基因组[16.那17.那18.那19.那20.].叶绿体基因组(CPDNA)是含有TRNA,RRNA和许多蛋白质编码基因的双链圆形分子[21.].大多数蛋白质编码基因与光合作用和生物能学有关[22.那23.].叶绿体基因组包含两个大的6 - 76kb的反向重复序列(IRs),分为大单拷贝(LSC)和小单拷贝区域(SSC) [24.那25.那26.].叶绿体基因组是非重组的,通过母体遗传来分娩[27.那28.].因此,叶绿体基因组是基因组和进化研究的优异工具。然而,由于低水平的替换,这是非常困难的,以检测CPDNA中的多态性[29.那30.].近年来,高通量基因组测序技术的发展使得叶绿体基因组的测序和分析取得了快速进展。具体来说,这些技术成果使我们能够获得和分析几种植物的完整叶绿体基因组,以便更好地了解它们的分子和基因组特征。
由于叶绿体编码一个完整而独立的基因组,因此研究叶绿体基因组具有重要意义;尤其是负责蛋白质翻译的叶绿体tRNAs。由于叶绿体基因组参与核苷酸、氨基酸和蛋白质的合成,了解其组织结构以确定这些过程在叶绿体基因组中是如何调控的是很重要的。叶绿体内的蛋白质翻译受tRNA和其他相关基因的调控。因此,对叶绿体trna的详细分析可以为了解蓝藻trna的基因组学和进化提供依据。单子叶植物基因组比双子叶植物基因组更保守,它们是从双子叶植物谱系进化而来的[31.那32.那33.].此外,一些重要的农艺作物品种是单子叶植物。因此,在本研究中,我们考虑对6种单子叶植物的叶绿体基因组进行研究,以便更好地了解叶绿体tRNA的基因组和进化特征,为今后的功能研究提供依据。
TRNA是负责维持和维持蛋白翻译机械的最重要和最通用的分子之一。它们的特点是存在由Robert Holley提出的三叶草叶状结构[34.].该结构包含受体臂,D形臂,D圈,抗密码架,防密码子环,可变臂,假核臂和假核环的特征。TRNA在核基因组内和亚细胞细胞器的基因组内编码,包括塑性体和线粒体。多年来,与核手裂性表征有关的详细研究已经增强了相当大的关注[35.那36.那37.].Mareachal-Drouard等人之前描述了叶绿体基因组TrNA和TRNA基因的结构和功能,(1991)[38.].然而,由于缺乏叶绿体基因组的完整基因组序列,本研究缺乏质体基因组tRNAs的完整基因组细节。因此,我们试图了解植物叶绿体tRNA的详细基因组和分子方面。考虑到单子叶植物保守的进化谱系,本研究对6个具有重要经济意义的单子叶植物进行了调查和报道。
结果
叶绿体tRNA基因组
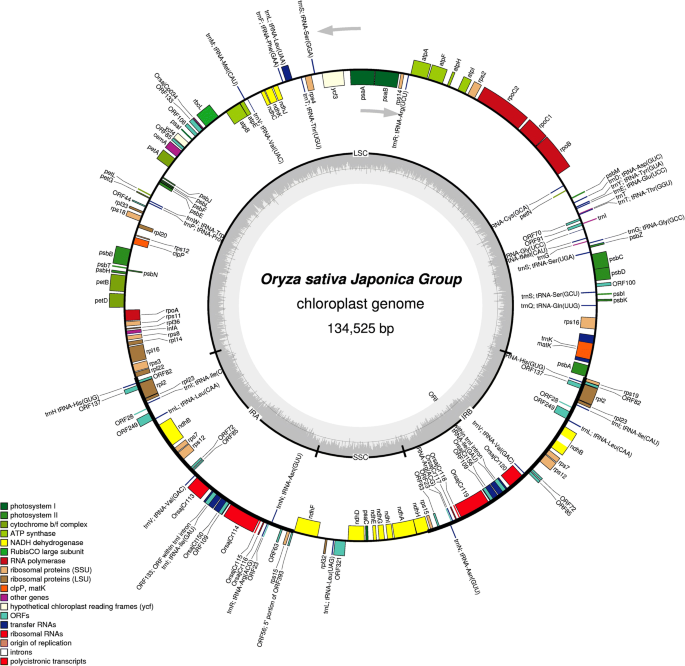
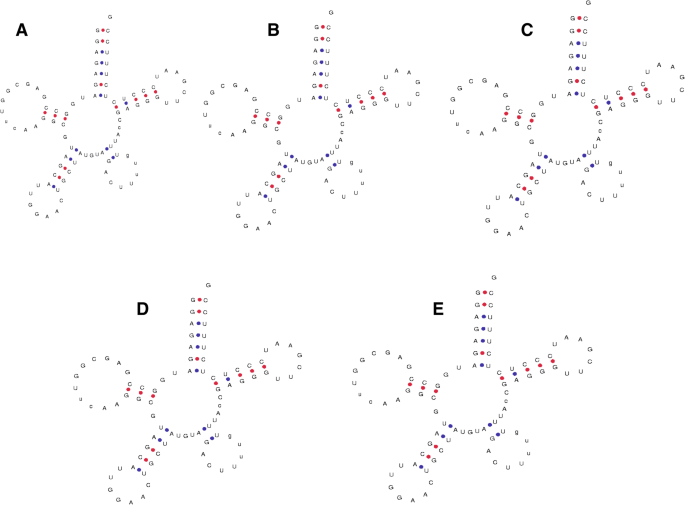
六种单子叶植物的整个叶绿体基因组序列,oryza nivara.(NC_005973),栽培稻(NC_001320),Sachharum OfficinArum.(NC_006084),高粱二色的(nc_008602),Triticum aestivum.(nc_002762),和Zea Mays.(NC_001666),从国家生物技术信息(NCBI)数据库中下载。随后,序列被注释以鉴定这些基因组中的基因组TRNA序列(图。1).使用Trnascan-SE服务器进一步分析所获得的基因组TRNA序列以确认它们作为TRNA的身份。结果表示O. Nivara.那o.苜蓿那S. Officinarum.那S. Bicolor.那T. Aestivum., 和Z. Mays.分别编码38,35,37,29,39和39个TrNA(表1).叶绿体TrNA的长度范围为59 nt [trnathrGGU,高粱二色的,(20385)]至155 nt [trnaLys.某某,T. Aestivum., (4982 _traect095)]。TRNA.gUCC的O. Nivara.(6129)只含有65 nt,而tRNAGLN.Uug.T. Aestivum.(4985)和TRNA鲁UAG的T. Aestivum.(5086_TraeCt128)分别包含118 nt和100 nt。在tRNA中,tRNAGLN.UUG(4985_Traect096),TRNA从46 nT和TRNA开始鲁UAG(5086_traect128),它从21 nt开始。这两个TrNA的5'核苷酸序列的成对序列比对显示出22.2%相似性(55.6%间隙)和保守的A-C-X-U-A-X-U-A-X-U-X的存在5.-u-a-a consensus序列。平均而言,检查的单焦植物中的叶绿体TrNAs含有76个核苷酸。TRNA.半胱氨酸, tRNAAsn, tRNA阿拉巴马州, tRNAAsp, tRNA板式换热器和trna.Trp发现分别含有71,72,73,74,73和74个核苷酸。所有序列的tRNA鲁和tRNA塞被发现包含80个或更多。TRNA.Lys.被发现在叶绿体基因组中缺失o.苜蓿和S. Bicolor.(桌子1).此外,TRNA.阿拉巴马州和tRNA伊尔也被发现缺席S. Bicolor.(桌子1).
单子叶植物的叶绿体TrNA仅编码21-28个抗密码子
然而,单子叶植物叶绿体基因组仅编码21-28个反密码子(见表)2).叶绿体基因组O. Nivara.那o.苜蓿那S. Officinarum.那S. Bicolor.那T. Aestivum., 和Z. Mays.分别编码28、25、28、21、28和28个反密码子2).叶绿体基因组tRNA中最常见的反密码子是UGC (tRNA)阿拉巴马州),GCC(TRNAg), UCC (tRNAg),UGG(trna箴),ggu(trnathr), UGU (tRNAthr),gac(trna瓦尔), UAC (tRNA瓦尔), GGA (tRNA塞),UGA(tRNA塞),GCU(TRNA塞),acg(trnaarg),ucu(trnaarg),uag(trna鲁),caa(trna鲁),UAA(trna鲁),Gaa(trna板式换热器),瓜(trnaAsn),uuu(trnaLys.), GUC (tRNAAsp),UUC(tRNAglu.),GUG(TRNA他的),Uug(TRNAGLN.),Cau(trna伊尔),高斯(tRNA伊尔)标出(tRNA遇见),GUA(tRNA酪氨酸),GCA(tRNA半胱氨酸)和CCA(tRNATrp) (桌子2).UCC (tRNAg)和UAC(tRNA瓦尔的基因组中存在的反密码子O. Nivara.在相关物种的叶绿体基因组中缺失,o.苜蓿(桌子2).同样,抗密码子UCC(TRNAg)和UAC(tRNA瓦尔的基因组中存在的O. Nivara.那S. Officinarum.那T. Aestivum,和Z. Mays.被发现在S. Bicolor.(桌子2).此外,抗密码子GGU(tRNAthr)和UAA (tRNA鲁)也没有出现S. Bicolor.;而他们被发现O. Nivara.那o.苜蓿那S. Officinarum.那T. Aestivum.和Z. Mays..在上述28个抗密码子之外,在研究的单焦叶类叶绿体基因组的任何TRNA中未发现33个抗密码子的其余部分(表2).
叶绿体tRNA序列的保存具有家族特异性
对所研究的单子叶植物所有20个tRNA基因家族成员进行多序列比对分析,发现在伪尿嘧啶环(Ψ)中存在小的、高度保守的一致序列,而在tRNA的其他部分则不存在(表)3.).发现ψ循环具有保守的U-U-C-X-A共有核苷酸序列(表3.).大部分TrNAS在第一位置含有G核苷酸。TRNA.瓦尔, tRNA遇见和trna.箴,但在第一个位置有一个A核苷酸而不是G(表3.).TRNA.GLN.和tRNAAsn发现在受体臂的第一个位置有一个U核苷酸。虽然在5 ' -受体臂中没有观察到一致的序列保守,但d -臂在臂的第4位(tRNA的第13位)包含一个保守的C核苷酸。相比之下,tRNAglu., tRNAg, tRNA遇见, tRNA塞, tRNA酪氨酸和所有其他TrNA在D形臂的第4位上具有C核苷酸。典型TRNA的核苷酸7至16形成盒子,据报道含有两个保守的共识序列,7.Guggcnnagu.16.- 和-ggu-agngc15.( - 代表Gap&N代表任何核苷酸)[39.].我们的分析显示,在分析的20个trna中,只有6个trna在第7位具有保守的G核苷酸(见表)3.).tRNA的第7位被A、U或C核苷酸占据(见表)3.).在大多数tRNA中发现第14位(D-loop的第1核苷酸)是保守的。除了tRNAarg, tRNAAsn, tRNAg和trna.遇见,发现所有其他TrNA被发现在第14位含有保守的核苷酸(表3.).同样,d -环的最后一个核苷酸被发现是一个保守的a核苷酸,除了tRNA酪氨酸(桌子3.).共识序列52Gguuganucc.62从第52个位置开始,从第52个位置开始,在第62个TRNA位置,形成B盒[40].我们的分析表明,TRNA中盒A和B核苷酸序列的保护以家庭特异性的方式发生。除了TRNA之外,发现52ND和53RD位置处的G-G核苷酸在大多数TRNA中被保守glu., tRNALys.和trna.瓦尔;虽然,发现核苷酸序列U-U-C-X-A-X-U在第54,55,56,58和第60个位置保守(表3.).TRNA.遇见U-U-C-x-A-U-C在第54位、第55位、第56位、第58位、第59位和第60位的共识序列为保守的U-U-C-x-A-x-U(表3.).同样,TRNA.Asp具有保守的U-U-C-G-A-G-C共识序列,而tRNA瓦尔含有U-U-C-G-A-X-X保守核苷酸。在TRNA的第59和第60次位置中发现了没有保守的核苷酸瓦尔.第32位和第33位的反密码子环中含有保守的C-U或U-U核苷酸。TRNA.GLN., tRNAg, tRNA他的, tRNA箴和trna.瓦尔含有保守的U-U核苷酸代替C-U核苷酸。此外,在大多数情况下,第38位的抗密码子环路具有保守的核苷酸。TRNA.GLN., tRNA箴和trna.瓦尔然而,在第38位而不是核苷酸A(表格)在第38次位置上具有保守的U核苷酸(表3.).叶绿体基因组在tRNA基因中编码一个预定义的C-C-A尾巴。当tRNA基因转录时,包括一个C-C-A尾巴。本研究发现tRNA阿拉巴马州, tRNAarg, tRNA伊尔, tRNALys.和trna.酪氨酸3 '端含有C-C-A核苷酸。一些编码的tRNA鲁单子叶叶叶绿体基因组中的基因还含有C-C-A尾部,然而,剩余的TRNA在其3'末端不具有C-C-A共有序列。
tRNA臂和环的核苷酸变异
在本研究中,揭示了叶绿体TRNA的受体臂以含有1-7个核苷酸。在代表六种单焦植物的213个TRNA序列中,发现只有两个含有一个核苷酸,一个有五个核苷酸,一个含有六个核苷酸;虽然209(98.12%)的其余部分有七个核苷酸。发现D形臂含有3和4个核苷酸,并且在D臂中没有少于三个或多于四个核苷酸的TRNA。共有73个(34.25%)有三个核苷酸,而140(65.73%)含有四个核苷酸。形成一个盒子的一部分的D圈有7至11核苷酸。在213个TrNAs中,45(21.12%)含有7,38(17.84%)含有8,75(35.21%)含有九个,22(10.32%)含有10和33(15.49%)含有11核苷酸。叶绿体TrNA的抗密码子臂具有4-5个核苷酸。在213个TrNAs中,23个(10.79%)抗密码子臂含有四个核苷酸,而190(89.20%)含有五个核苷酸(附加文件1:表S1)。除了一个外,所有TRNA都有七个核苷酸在抗密码子环中。trna 6160_ornict018O. Nivara.含有九个核苷酸而不是七个(附加文件1:表S1)。发现可变环具有不同多数量的核苷酸,其不同的TRNA具有4(9.38%),5(59.62%),6(3.75%),7(5.63%),11(2.34%),12(0.46%)),13(6.1%),14(0.46%),15(1.87%),16(2.34%),18(2.34%)或19(5.63%)核苷酸。没有发现叶绿体TrNAs在可变循环中具有8,9,10,17,20或更多个核苷酸(附加文件1:表S1)。TRNA.鲁, tRNA塞和trna.酪氨酸分别有10个或更多个核苷酸,而其他TrNAs在可变循环中具有少于10个核苷酸(附加文件1:表S1)。在213个检查的TRNA序列中,只有三个TRNAg基因Ψ-arm中有4个核苷酸,而其余tRNA序列中有5个核苷酸。同样,213个trna中的Ψ-loop区域拥有7个核苷酸。我们的研究发现受体臂为7 bp, d臂为3-4 bp,其他部位也存在较大差异。反密码子臂有4 - 5bp,反密码子环有7或9个核苷酸。组成可变环的核苷酸数量从4个到19个不等,在可变环中没有trna的核苷酸超过19个。与之前的报道相似,Ψ-arm含有4-5个核苷酸。
叶绿体tRNA包含I组内含子
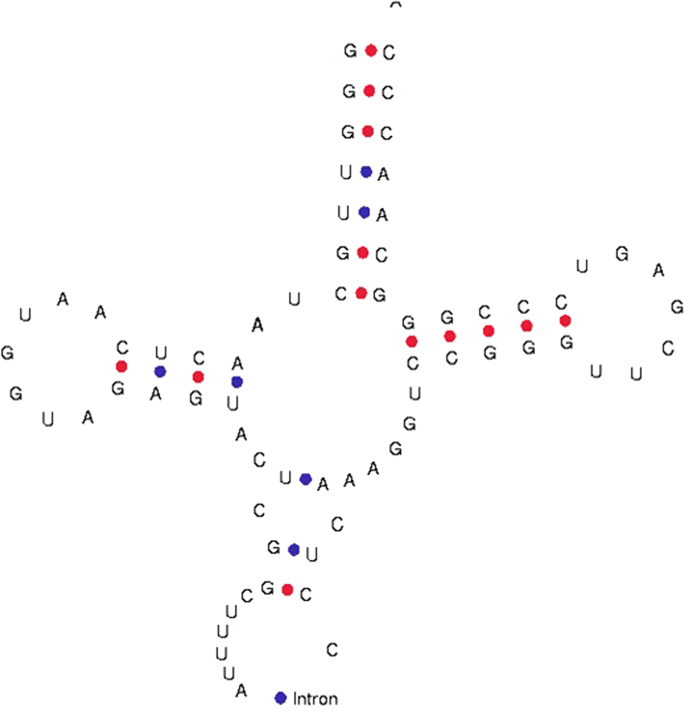
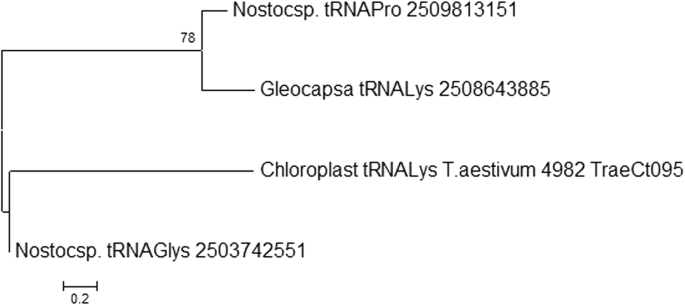
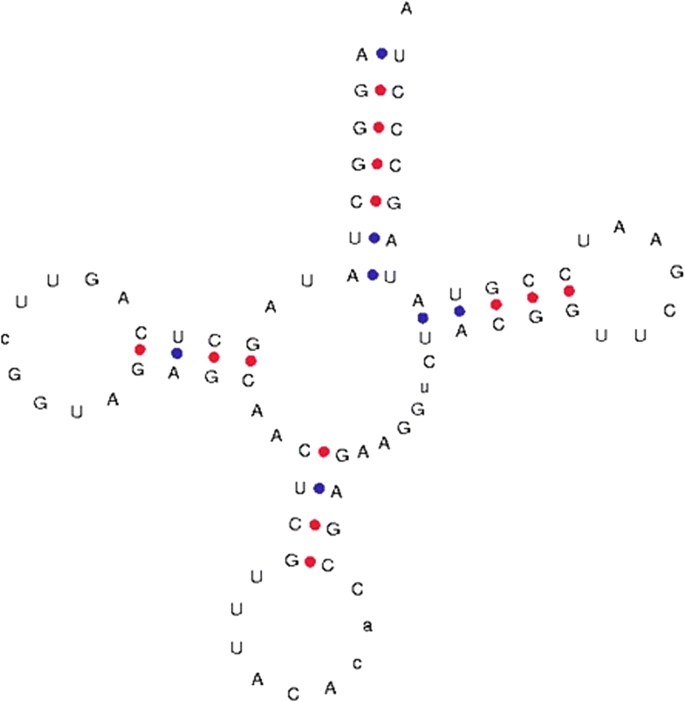
然而,在我们的研究中,发现叶绿体TRNA含有内含子。TRNA.Lys.的T. Aestivum.(4982_traect095)被发现含有I的Intron位于TRNA的抗密码子环路区域中Lys.(无花果。2).内含子长度为84个核苷酸,从37个核苷酸开始,到120个核苷酸结束Lys.基因。叶绿体tRNA的I组内含子中含有保守的U-U-x2-c和a-g-x2-U一致序列(图。3.).构建了系统发育树以阐明Intron群体的演变。系统发育分析表明,用氯蛋白血管基团与乙型子系统的内含子进行分组(图。4.).
叶绿体tRNA编码假定的新tRNA
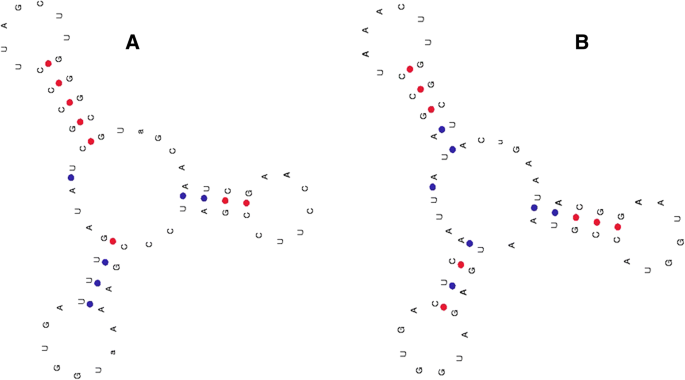
在本研究中,一些推测的新trna被发现是由叶绿体基因组编码的(图。5.).TRNA.g(UCC)O. Nivara.(6129_OrniCt007, ΔG =−18.10)和tRNAthr(GGU)的S. Bicolor.(20,385_trnm-cau sobict011,Δg= 14.7)在5'端不含受体臂(图。5.).此外,还有一些tRNA塞在O. Nivara.(6152_OrniCt014, ΔG =−34.13),o.苜蓿(3720_OrsajCt137, ΔG =−34.13),S. Bicolor.(20,407_trnS-GGA SobiCt019, ΔG =−34.13),S. Officinarum.(6593),和T. Aestivum.(5020_traect112,Δg= - 34.13)在可变环区域中含有七核苷酸环结构,类似于TRNA的抗密码子环(图。6.).所有构成可变环区的环状结构均由a - c - u - u - u - g核苷酸组成。的tRNA瓦尔的O. Nivara.(6160_ornict018,Δg= - 25.20)被发现仅在抗密码子臂中仅含有四个核苷酸和抗密码子环中的九个核苷酸(图。7.).在蓝藻和植物的基因组tRNA中发现了许多类似的tRNA结构(未发表的数据)。
tRNA的C-A-U反密码子伊尔在叶绿体trnas.
C-A-U反密码子是tRNA的一个特征遇见并且只有一个同位受体。除了在tRNA中存在一个C-A-U反密码子遇见,我们也发现tRNA伊尔叶绿体TRNA还编码C-A-U抗密码子。的tRNA伊尔在O. Nivara.(6206_ornicp049,6270_ornict035),o.苜蓿3828 (3774 _orsajct146 _orsajct160),S. Officinarum.(officinarum_6644,officinarum_6710),S. Bicolor.(20,460,20,502),T. Aestivum.(5069, 5108)Z. Mays.(2069_trnI ZemaCt144, 2131_trnI ZemaCt154)叶绿体基因组编码一个C-A-U反密码子。据我们所知,这可能是首个证明叶绿体tRNA中存在C-A-U反密码子的报告伊尔.
叶绿体trna是由多个共同祖先进化而来的
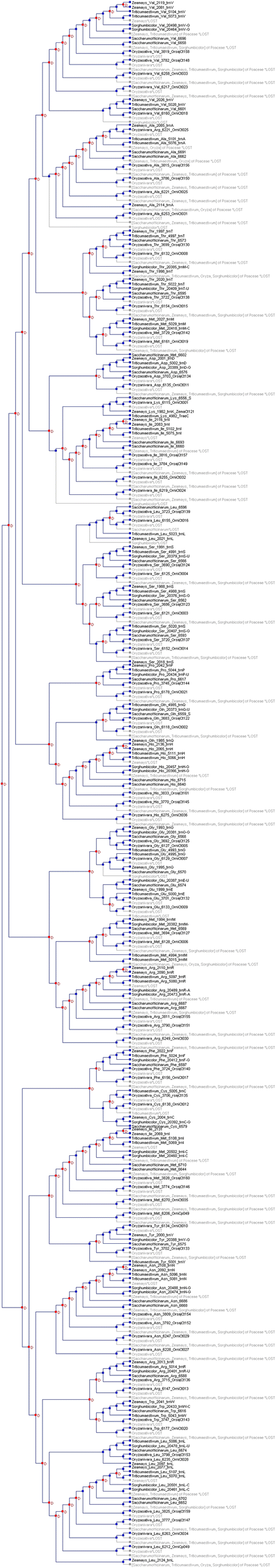
利用所有单子叶植物叶绿体基因组的tRNA序列构建系统发育树。系统发育分析显示存在两个主要的聚类,包括30组。集群I包含tRNA瓦尔, tRNA阿拉巴马州, tRNAarg, tRNAthr, tRNA遇见, tRNAAsp, tRNALys., tRNA伊尔, tRNA鲁, tRNA塞, tRNA箴, tRNAGLN., tRNA他的, tRNAg, tRNAglu.和trna.arg.簇II包含tRNA板式换热器, tRNA半胱氨酸, tRNA伊尔, tRNA遇见, tRNA酪氨酸, tRNAAsn, tRNAarg, tRNATrp和trna.鲁.集群I和集群II中的9组中有21组(图。8.).在簇I中,是tRNAarg分组两次;一旦与tRNA阿拉巴马州并靠近trna遇见.同样,TRNA.遇见也分为两次;靠近含有TRNA的组thr一旦靠近含有tRNA的基团arg(无花果。8.).TRNA.arg, tRNA伊尔, tRNA鲁和trna.遇见在系统发生树的聚类II中也有。带有反密码子G-A-C和U-A-C的tRNA瓦尔,g-g-u和trna的u-g-uthr,U-G-A,G-C-U和TRNA的G-G-A塞, G-C-C和U-C-CgU-A-A, U-A-G和C-A-A鲁;c-a-u的trna伊尔、U-G-C、U-C-U和A-C-Garg,分别分组(图。8.).TRNA.Trp(CCA)与tRNA紧密结合arg(UCU),提示tRNA的进化Trp来自trna.arg(无花果。8.).同样,TRNA.酪氨酸(GUA)与TRNA紧密分组遇见(CAU)和TRNA伊尔(CAU),提示tRNA的进化酪氨酸(GUA)和TRNA伊尔从tRNA(标出)遇见(CAU)。分组tRNA遇见(CAU)具有TRNA伊尔(CAU)和它们相似的反密码子核苷酸强烈提示tRNA伊尔直接从tRNA进化而来遇见.此外,tRNA的紧密分组遇见(CAU)具有TRNAarg(ACG)进一步提示tRNAarg是从tRNA遇见也分组tRNAglu.(UUC)与tRNAg(GCC),TRNA他的(GUG)用tRNAGLN.(uug)和trna箴(UGG)提示这些trna可能是由共同祖先进化而来的,或者是通过基因复制事件进化而来的。TRNA.塞(GGA,GCU,UGA)与TRNA分组鲁(UAA);这表明tRNA塞从trna演变出来鲁.值得注意的是,trna.鲁含有C-A-A抗密码子,而TRNA鲁那哪个与tRNA结合塞,包含一个U-A-A反密码子。这表明tRNA鲁(CAA)经历了基础替代,以产生TRNA鲁(uaa)并且进一步重复和多样化导致TRNA塞(GGA,GCU,UGA)。分组tRNA伊尔(高斯),tRNALys.(下面),tRNAAsp(GUC)在一起建议他们的常见进化谱系。此外,分组TRNA遇见与tRNAthr(UGU和GGU)提示tRNAthr(UGU和GGU)由tRNA进化而来遇见.同样,tRNA的亲缘关系也很密切遇见与tRNA阿拉巴马州和tRNA瓦尔在群集中,我表示该TRNA阿拉巴马州和tRNA瓦尔也从trna演变遇见.使用蒙特卡罗复制进行替代模式均匀性的差异指数试验,以确定所有取代和核苷酸的取代率是否是均匀的。结果表明,零假设被拒绝用于TRNAarg, tRNAGLN., tRNA阿拉巴马州, tRNA遇见, tRNAthr和trna.瓦尔;暗示这些基团中核苷酸的替代率是均匀的。在这六个trna同学之外,14个未显示模式均匀性,因此替代核苷酸和TRNA的演变g, tRNA箴, tRNA塞, tRNA鲁, tRNA板式换热器, tRNAAsn, tRNALys., tRNAAsp, tRNAglu., tRNA他的, tRNA伊尔, tRNA酪氨酸, tRNA半胱氨酸和trna.Trp不是同质。为了更好地了解叶绿体tRNA与古菌的关系,我们将两种古菌的tRNA合并,并以三种蓝藻的tRNA序列为类群。两个互补的DNA序列拟南芥蒂利亚纳NAC转录因子(ATNAC1和ATNAC2)被用作组(附加档案2:图S1)。系统发育分析显示古藻与叶绿体TRNA的一些重叠关系。然而,与古代相比,叶绿体TrNas与蓝藻TRNA更接近蓝藻。
过渡和转化率是特定的isoacceptor
TRNA是进化的保守分子,并且经历主要过渡或横向事件的可能性非常小。TRNA的过渡率(8.33)和转烷(8.34)阿拉巴马州, tRNAAsn, tRNAAsp, tRNA他的, tRNA板式换热器和trna.箴几乎是相等的。这表明,尽管转位率略高于转位率,但这些trna在转位和转位方面的进化速度几乎相同(见表)4.).此外,tRNA的转化速率(25.00)和转化速率(0.00)半胱氨酸, tRNAGLN., tRNATrp和trna.酪氨酸也与彼此相似(表)。但值得注意的是,TRNA半胱氨酸, tRNAGLN., tRNATrp和trna.酪氨酸在单子叶植物的叶绿体基因组中,发生了高速率的转变,但没有发生任何转化。相反,tRNA的转换率伊尔(8.60), tRNALys.(10.09),trna塞(9.15),相对于TRNA的过渡速率,被发现更高伊尔(7.80),TRNALys.(4.82),和tRNA塞分别为(6.70)(表4.).在TRNA中也观察到较高的过渡率arg(12.40),TRNAglu.(12.53),TRNAg(17.39),TRNA鲁(11.88), tRNA遇见(16.87),TRNAthr和trna.瓦尔(桌子4.).tRNA的过渡取代率最高(25.00)半胱氨酸, tRNAGLN., tRNATrp和trna.酪氨酸.然而,当所有TRNA被共同检查时,过渡的平均速率(14.71)大于横向的平均速率(5.15)(表4.).
叶绿体tRNA的复制先于缺失
植物基因组包含更丰富的复制基因,整个基因组复制事件在过去2亿年间发生了多次[41.那42.那43.那44.].考虑到叶绿体基因组的蓝藻起源,复制和损失事件的速率可能与核编码基因组中的基因不同。本研究通过对单子叶植物叶绿体tRNA的重复/损失分析发现,101个基因发生了重复,139个基因发生了损失;然而,有80个基因经历了条件复制。大部分叶绿体trna在进化过程中发生了损失。尽管所有的trna都来自同一谱系(单子叶),但基因的丢失仍然大于复制的基因(图。9.).
讨论
trna是一种保守的家族基因,负责蛋白质翻译事件的传导。它们在叶绿体基因组中的存在是对基因组的补充,使其半自主。叶绿体trna的多序列比对揭示了一些基本的保守基因组特征。一些trna在5 '端含有扩展的核苷酸序列。然而,tRNAscan-SE服务器无法确认这些5 '端核苷酸序列是否为内含子。因此,这些序列极有可能是tRNAs的内含子。之前的一项研究报道了在蓝藻tRNA中存在一组I内含子[45.].鉴于来自蓝藻谱系的叶绿体基因组的起源,需要考虑这些序列最有可能是叶绿体TRNA的内含子[45.].对每个tRNA序列进行分析,发现了tRNA鲁和tRNA塞编码的最长tRNA序列。之前的一项研究也报道了tRNA中存在80个或更多的核苷酸鲁和tRNA塞的栽培稻[45.].这说明tRNA鲁和tRNA塞编码更长的tRNA序列。这项研究也揭示了tRNA的缺失Lys., tRNA阿拉巴马州和trna.伊尔这些单子植物叶绿体基因组中的基因。在叶绿体基因组中没有重要的TRNA编码基因非常有趣,并且可以了解在没有重要的TRNA的情况下进行这些单胞植物的蛋白质翻译。最有可能的是基因组TRNA补偿没有塑性的TrNAs,或者可以从细胞细胞基因组中的其他TrNA进行传导蛋白翻译的多种功能。这是关于没有TRNA的第一个报告Lys., tRNA阿拉巴马州和trna.伊尔在叶绿体基因组中。除了tRNA的缺失Lys., tRNA阿拉巴马州和trna.伊尔,单子叶植物的叶绿体基因组也缺乏Selenocystein,吡咯醇素和抑制器TRNA(表1).我们的分析还显示单子叶叶绿体基因组含有编码TRNA的最高数量基因鲁和tRNA遇见;(4)其次是tRNAarg和trna.塞(3)。通用基因表包含64个密码子;其中,61是有道感的,3是反感密码子。因此,可以有61个独特的防密码子的TRNA可以编码61个感测密码子。发现大约33个抗密码子免受叶绿体基因组的TRNA不存在。但是,没有UCC抗密码子的tRNAg通过TRNA的GCC抗密码子的存在来补偿g,而tRNA的反密码子UAC缺失瓦尔是由tRNA的GAC反密码子补偿的瓦尔.同样,TRNA的抗密码子GGUthr通过TRNA的UGG抗密码子存在补偿thr和TRNA的抗密码子UAA鲁被反密码子UAG和CAA的存在所补偿。tRNA基因完全缺失Lys.(uuu,cuu)在o.苜蓿和S. Bicolor,和tRNA阿拉巴马州(AGC、GGC、CGC和UGC)很难理解。然而,我们可以推测,叶绿体基因组中这些trna的缺失可能会被基因组trna或叶绿体或细胞核来源的其他trna弥补。反密码子CAU由tRNA编码遇见和tRNAfMet.分析表明,单子叶植物叶绿体基因组编码tRNA遇见和tRNAfMet也以前,Howe(1985)和Hiratsuka等人,(1989)报道了TRNA的存在fMet在叶绿体基因组中[46.那47.].所有的物种都含有至少一个tRNAfMet和一个trna遇见.O. Nivara.(6128 _ornict006),o.苜蓿(3694_orsajct127),S. Officinarum.(6569),S. Bicolor.(20382),T. Aestivum.(4994),和Z. Mays.(1994)每个编码一个tRNAfMet.在原核基因组中,蛋白质翻译的起始是由tRNA介导的fMet,而随后向多肽链加入甲硫氨酸是通过TRNA介导的遇见[48.那49.那50].tRNA的存在遇见和tRNAfMet是原核基因和细胞器基因的一个特征[51和tRNA的存在fMet单子叶植物叶绿体基因组显示其原核起源。
由于许多物种的功能相似性,TrNA是一种进化的多岛家族。TRNA的核苷酸组合物负责维持平移的TRNA的三级结构。因此,TRNA的常见保守功能也应该反映在保守的编码序列中。先前的研究报告说,在TRNA中存在保守的核苷酸共有序列,其仅限于ψ循环[45.].在我们的研究中,我们发现在Ψ-loop中存在U-U-C-x-A核苷酸一致序列。但在其他部分未发现保守的一致序列。相反,他们被发现含有一些保守的核苷酸。核编码tRNAGLN.和tRNAAsn在第一位置含有U核苷酸(表3.)[45.].然而,一项多序列比对研究表明,存在于叶绿体tRNAs中的序列保守是家族特异性的(见表)3.).在蛋白质翻译期间,聚合酶与TRNA的促进剂结合,该TRNA称为A和B盒。这两个盒子包含保守的共识序列。框A从成熟TRNA的+ 8个核苷酸开始,而BOX B包含保守52Gguuganucc.62核苷酸共有,其构成ψ臂和整个ψ圈的一部分。凸形蛋白盒TRNA没有那么保守,而Box B很高度保守。盒A和B被认为是RNA聚合酶III的腺瘤转录促进剂信号序列[52].转录活化的信号序列在叶绿体基因组的TrNA中不受普通方式保守。据报道,抗密码子环在第32个位置被保存[52].然而,在本研究中,在大多数病例中的32nd和33个位置中发现了核苷酸的保护。此外,发现几种TRNA序列含有3'-C-C-C尾。通过TRNA核苷酸转移酶促促进将C-C-A尾部添加到TRNA的3'-末端。然而,叶绿体基因组不编码TRNA核苷酸转移酶。因此,在没有核苷酰亚酰基转移酶的情况下,将C-C-A尾部添加到TRNA的3'-末端。在少数TrNA的3'末端没有C-C-A尾部反映了它们最近的演变,因为大多数核网络缺乏3'C-C-A尾巴。
鉴于叶绿体基因组的蓝藻来源,它应该是原核,一般来说,应该是无内部的。然而,我们发现在叶绿体TrNA中的I族内含子存在。以前的研究还报告了在TRNA中存在内含子鲁(uaa)和trnafMet(UAC)蓝藻TRNA [53那54].此外,在我们的实验室进行的最近进行的一项研究还报告了蓝藻TRNA中的内含子arg, tRNAg和trna.Lys.[45.].虽然已经通过几项研究报告了含有蓝细菌基因组中内含子的内含子,但是本研究似乎是第一个报告叶绿体TRNA中内含子的存在的研究。In Introns缺乏显着的序列保护,然而,本分析表明它们含有短暂的保守共识序列。叶绿体TRNA的内含子与乙型内含子细胞的内含子(图。4.),从而提供额外的证据表明他们从常见的蓝藻谱系中演变。
由罗伯特·霍利提出[34.[Trnas的特征在于三叶腰状结构,尽管在其二级结构中几个TrNA也会变化[35.].trna包含在蛋白质翻译中起作用的各种臂和环。每个手臂和环都有自己独特的核苷酸组成。先前的研究报道,受体臂包含7个碱基对7 bp, D-stem 3-4 bp, D-loop 4-12核苷酸,反密码子臂5 bp,反密码子环7核苷酸,可变区4-23核苷酸,Ψ-arm 5 bp, Ψ-loop 7核苷酸[37.].先前的报告和目前的研究表明,叶绿体trna的臂和环存在显著的变异。受体臂包含trna -核苷酸转移酶的不同信息。然而,tRNA中缺少受体臂g(UCC)O. Nivara.和tRNAthr(GGU)的S. Bicolor.很有趣的。在蛋白质翻译过程中,一个没有受体臂的tRNA是如何参与携带氨基酸的?一些trna包含新环路具有a - c - u - u - u - g核苷酸。新环路的主干允许A与U和G与U核苷酸成键。本研究中发现的新环结构提出了一个问题,即这些环是否模仿tRNA的反密码子环,并在叶绿体内的蛋白质翻译机制中发挥关键作用。一些tRNA在反密码子环中还含有9个核苷酸;这可能是tRNA的一种新现象。反密码子环中有9个核苷酸对功能的影响仍有待确定。除了存在一些推测的新tRNA结构外,叶绿体tRNA还包含一个C-A-U反密码子,编码tRNA伊尔也然而,在TRNA中存在C-A-U抗密码子伊尔以前报道过枯草芽孢杆菌[55].
叶绿体tRNA的系统发育分析显示出两个不同的聚类和多个类群。我还发现集群中的一些tRNA成员也存在于集群II中;表明它们是通过复制和分化进化的。而反密码子GAC、UAC、GGU、UGU、UGA、GCU、GGA、GCC、UCC、UAA、UAG、CAA、CAU、UGC、UCU、ACG分别独立存在于系统发育树中;表明它们是由多个共同祖先进化而来的。tRNA家族成员的重叠分组表明,具有这些反密码子群的tRNA可能是从不同的共同祖先进化而来的,也可能是由重复事件产生的。tRNA的存在遇见在群中两次,在群中II中的一次表明TRNA遇见是一个经过重复的重复事件的TRNA系列之一,以产生其他TRNA。系统发育分析进一步揭示了TRNA鲁(CAA) tRNATrp(CCA), tRNAarg(UCU) tRNAAsn(郭),trna酪氨酸(卦),tRNA遇见(标出),tRNA半胱氨酸(GCA)和TRNA板式换热器(Gaa)存在于聚类II中是最原始的TrNA与TRNA的形式鲁作为最基本的进化祖先。分组tRNA遇见(CAU)具有TRNA伊尔(CAU)和它们相似的反密码子核苷酸强烈提示tRNA伊尔直接从tRNA进化而来遇见.整体分析清楚地表明TRNA遇见是叶绿体基因组中trna进化的主要参与者。tRNA的分布遇见在两个不同的群集中,强烈表明TRNA遇见经历了几次主要的取代和复制事件,产生了不同的tRNA家族,具有不同的反密码子。叶绿体trna的转化速率高于转化速率。TRNA.半胱氨酸, tRNAGLN., tRNATrp和trna.酪氨酸属于极性R组,携带极性氨基酸的TRNA中的横转化率为零。极性氨基酸易溶于水并形成具有相互作用分子的强氢键。这表明叶绿体TRNA的演变半胱氨酸, tRNAGLN., tRNATrp和trna.酪氨酸强烈地有利于过渡取代而不是横转化取代,并且一些TRNA异抗受体剂比横转化更容易过渡。然而,几个TrNAS经历了比过渡更高的横向率;表明,进化速率和TrNA的转变率和转化率是异抗体特异性,并且该TRNA没有经历相等的进化速度。
除了突变事件,基因复制也是进化的主要力量,是物种获得新基因的重要机制[56].大多数新的基因功能是通过基因复制事件进化而来的,基因复制事件可以通过基因组复制、反转录转座子和不平等交叉发生[57那58].古老的复制事件加上现存的复制基因对的保留极大地促进了基因家族的进化和功能多样化[59].植物基因组倾向于以高速率发展,导致相对于其他生物的更大的基因组多样性[60].对叶绿体tRNA的研究表明,tRNA的缺失率高于复制率。这表明蓝藻来源的叶绿体基因组的母体遗传比核编码的植物基因组更完整。因此,尽管物种是同一谱系的一部分,但每个物种仍然丢失了一些基因。这进一步证明了蓝藻trna来源于多系共同祖先,因此损失事件比重复事件更明显。几乎所有的trna都经历了损失事件在任何一个被研究的物种(表)5.).
结论
我们对六种单子叶植物的叶绿体基因组进行了TRNA分析,发现这些植物物种中的叶绿体基因组编码了28至39个TRNA基因。图23至29的TRNA异抗菌物的数量和大多数TRNA仅与一种异征感剂相关。还发现叶绿体基因组中的TrNA在抗密码区区域中含有Intron,并且系统发育分析显示单子植物中的叶绿体TrNA从多个常见的祖先演变。还发现所研究的单子叶植物物种的叶绿体基因组含有推定的新型TRNA,需要进一步研究以了解其生物学意义。基因重复和损失事件的分析表明,基因损失事件比叶绿体TRNA中的重复事件更明显。
方法
单子叶植物叶绿体tRNA的鉴定与分析
单子叶种类的叶绿体基因组,O. Nivara.(NC_005973),o.苜蓿(NC_001320),S. Officinarum.(NC_006084),S. Bicolor.(nc_008602),T. Aestivum.(nc_002762),和Z. Mays.(NC_001666)从国家生物技术信息中心提供的公共数据库下载(NCBI,https://www.ncbi.nlm.nih.gov/)[46.那61那62].该序列以Fasta格式下载(附加文件1:表S1,附加文件3.:数据S1),随后对所有叶绿体基因组进行注释。使用Geseell Genomes的Geseq注释进行所有叶绿体基因组的注释(https://chlorobox.mpimp-golm.mpg.de/geseq.html)[63].用于执行注释过程的参数是圆形序列;序列源,叶绿体;生成多个fasta;注释塑体IR,Blat蛋白质搜索身份25%;Blat RRNA,TRNA和DNA搜索85%的身份;HMMER配置文件搜索;胚胎叶绿体(CDS + RRNA);第三方Trna Annotator Aragron V1.2.38,Arwen V1.2.3,Trnascan-SE V2.0;没有使用Refseq选择。 Annotated nucleotide sequences of the chloroplast tRNA genes in the six-monocot species were collected and used in the further sections of this study. The free energy calculation of predicted novel tRNAs were performed using the RNAalifold webserver with default parameters [64].
单子叶植物叶绿体TRNA分析
利用ARAGRON和tRNAscan-Se服务器对采集到的单子叶植物叶绿体tRNA基因组序列进行进一步分析[65].默认参数用于分析阿拉罗朗的基因组TRNA序列。在Trnascan-SE服务器中,使用以下参数来分析基因组TRNA;序列源,细菌;搜索模式,默认;查询序列,格式化(FASTA);和TRNA同种型预测,通用的遗传密码。使用相同的参数分析所有TRNA,并且单独记录不同臂和环中的核苷酸的数量和组成。发现与TRNA的规范三叶草叶状结构具有不同结构的TRNA被认为是推定的新型TRNA。
多序列比对
为了鉴定和分析tRNA同型的保守核苷酸序列,将20个同型的核苷酸序列分别进行分组。随后,利用Multalin服务器对tRNA亚型进行多序列比对。所有序列均采用FASTA格式进行比对分析,参数如下:序列输入格式,自动;显示序列对齐,彩色;一致性矩阵,Blosum61-12-2;开球和延期罚球,违约;极限间隙惩罚,没有,只有一次迭代,没有。最高的对齐共识值保持在90%(默认);然而,最低的共识值保持在50%(默认)。 In the displayed alignments, red indicates a similarity/conservation of 90% or more; whereas, blue indicates a sequence conservation less than 90%. Alignments displayed in black indicates no conservation.
系统发育树的构建
为了分析单胞菌植物中叶绿体TrNA的演变,使用Mega6.0软件构建了系统发育树[66].在构建系统发育树之前,使用Clustal omega服务器创建了所有tRNAs的Clustal文件。利用MEGA6软件将生成的tRNAs Clustal文件转换为MEGA文件格式。在构建系统发育树之前进行模型选择。采用MEGA6软件进行模型选择,统计参数如下:分析、模型选择(ML);使用树,自动(邻居连接);统计方法,最大似然;核苷酸替换类型;空白/缺失数据处理、部分删除和站点覆盖率截断率为95%。基于最小贝叶斯信息准则(BIC)的模型选择分析被认为是构建系统发育树的最佳模型。 The lowest BIC score was found to be 7785.682 for the Kimura2+ G + I model; as a result, the latter model was used to construct a phylogenetic tree. Other statistical parameters within the Kimura2+ G + I model were: analysis, phylogeny reconstruction; statistical model, maximum likelihood; test of phylogeny, bootstrap method; no. of bootstrap replicates, 1000; substitution type, nucleotides; rates among sites, Gamma distributed with invariant sites (G + I), no of discrete Gamma categories, 5; gaps/missing data treatment, partial deletion; site coverage cutoff, 95%; and branch swap filter, very strong.
转型和横转化分析
用于构建系统发育树的TRNA的MEGA文件格式用于分析所有TRNA的过渡/横转率。另外,分别研究了所有20个TRNA同种型的过渡/转变速率。使用Clustal Omega服务器还经过多个序列对齐,为每个单独的同学生成群集文件。TRNA同学的生成的群集文件被转换为MEGA文件格式,使用MEGA6软件估计替换速率。以下统计参数用于研究单子叶植物的叶绿体TrNA中的过渡/转化率:分析,替代模式估计(mL);树使用,自动(邻居加入树);统计方法,最大似然;核苷酸替换类型;模型/方法,Kimura2参数模型;网站之间的价格,伽玛分布(g); no. of discrete Gamma categories, 5; gaps/missing data treatment, partial deletion, site coverage cutoff 95%, and branch swap filter, very strong.
差异指数分析
为了确定在进化期间发生均匀(等速率)的所有核苷酸的所有取代,进行了模式异质性的差异指数试验以确定核苷酸取代的均匀性。用于分析均匀性模式的统计参数是:分析,替代模式均匀性的差异指数试验;范围,顺序对;不。蒙特卡罗复制,1000;核苷酸替换类型;空白/缺失数据处理,部分删除;和现场覆盖截止值为95%。
基因重复和丢失的分析
首先使用NCBI分类浏览器构建所有物种树(https://www.ncbi.nlm.nih.gov/taxonomy/commontree/wwwcmt.cgi.)来分析tRNA基因的重复和丢失事件。用来建造种树的种数是O. Nivara.那o.苜蓿那S. Officinarum.那S. Bicolor.那T. Aestivum., 和Z. Mays..进化分析用系统发生树作为基因树。利用no钨2.6软件研究基因重复/丢失事件。在分析过程中,将基因树与种树进行调和,得到基因的复制和丢失节点。
缩写
- A:
-
腺嘌呤
- C:
-
胞嘧啶
- 旅客:
-
鸟嘌呤
- TRNA:
-
转移RNA.
- 你:
-
尿嘧啶
- Ψ:
-
伪心
参考
- 1。
Wise Rr,Hoob J.Plastids的结构和功能。潜水员。奏奏。in:表格功能;2006年。
- 2。
王志强,王志强。基于光合作用和能量平衡模型的全球陆地生物圈模拟。全球生物地球化学循环。2001; 15:207-25。
- 3.
des marais dj。光合作用何时出现在地球上?科学。2000; 289:1703 LP-1705。
- 4.
Stern DB, Goldschmidt-Clermont M, Hanson MR.叶绿体RNA代谢。植物生物学进展。2010;61:125-55。
- 5.
小博尔顿,霍尔。太阳能光化学转换与储存。《年度能源回顾》1979;4:353-401。
- 6.
詹森佤邦。叶绿体和光合作用。In: Jensen WA,编辑。植物细胞。伦敦:麦克米伦教育英国;1973.25-45页。
- 7.
Spetea C,Hundal T,Lundin B,Heddad M,Adamska I,Andersson B.在叶绿体囊体内腔中的核苷酸代谢的多种证据。Proc Natl Acad Sci U S a。国家科学院。2004; 101:1409-14。
- 8.
STITT M,Lilley RM,Holdt HW。小麦叶片原生质体的细胞溶胶,叶绿体和线粒体中的腺嘌呤核苷酸水平。植物理性。1982; 70:971 LP-977。
- 9.
Noctor G,Arisi A-CM,Jouanin L,Foyer Ch。叶绿藻谷胱甘肽和氨基酸生物合成的操纵。植物理性。1998; 118:471 LP-482。
- 10。
Schulze-Siebert D,Heineke D,Scharf H,Schultz G.刺瓜替代丙二酸衍生的氨基酸。植物理性。1984; 76:465 LP-471。
- 11.
Blee E,Joyard J.来自菠菜叶绿体的封套膜是脂肪酸氢过氧化物的代谢位点。植物理性。1996; 110:445 LP-454。
- 12.
维克学士,齐默尔曼DC。菠菜叶片叶绿体中脂肪酸氢过氧化物代谢途径的研究。植物生理学。1987;85:1073 LP-1078。
- 13。
Wallsgrove RM, Keys A, Lea PJ, Miflin BJ。光合作用、光呼吸和氮代谢。植物细胞环境。1983;6:301-9。
- 14。
Pilon-Smits EAH, Garifullina GF, Abdel-Ghany S, Kato S- i, Mihara H, Hale KL, et al.;拟南芥nifs样叶绿体蛋白的特性研究。它在硫和硒代谢中的作用。植物生理学。2002;130:1309 LP-1318。
- 15.
引用本文:王旭。高等植物硫代谢:IV.叶绿体中硫酸盐还原的机制。生物物理学报,1964;82:58-66。
- 16.
王志强,王志强,王志强,等。拟南芥、蓝藻和叶绿体基因组的进化分析揭示了质体系统发育和细胞核中数千个蓝藻基因。美国国家科学院。2002;99:12246-51。
- 17.
灰色mw。细胞器的进化起源。趋势类型。1989; 5:294-9。
- 18.
王志强,王志强,王志强,等。基因转移到细胞核和叶绿体的进化[j]。大自然。出版社1998;393:162。
- 19.
FalcónLi,Magallóns,Castillo A.约会叶绿体的蓝藻祖先。ISME J.国际微生物生态学协会。2010; 4:777。
- 20。
Raven Ja,Allen JF。基因组学和叶绿体进化:蓝藻对植物做了什么?基因组Biol。伦敦:生物化的中央。2003; 4:209。
- 21。
Kolodner R,Tewari KK。来自高等植物的叶绿体DNA中的倒置重复。PROC NATL ACAD SCI。1979; 76:41 LP-45。
- 22。
Shinozaki K,Ohme M,Tanaka M,Wakasugi T,Hayashida N,Matsubayashi T.烟草叶绿体基因组的完全核苷酸序列:其基因组织和表达。Embo J. 1986; 5。
- 23。
玉米叶绿体基因组全序列:基因含量、分化热点和遗传信息的转录编辑微调。中华生物学杂志。1995;251。
- 24。
王荣杰,程春林,张春春,吴春林,苏天明,周淑敏。单子叶植物叶绿体基因组中反向重复-大单拷贝连接的动态和进化。BMC Evol Biol. 2008;8:36。
- 25。
甘蓝和烟草叶绿体DNA中大单拷贝区和反向重复序列的连接:tRNA his和核糖体蛋白S19和L2的基因序列。核酸Res. 1984; 12:6547-58。
- 26.
在这里,JP,Werth Ja,Thornby DF,Keenan M,Chauhan BS,Walter GH。从澳大利亚的草甘膦抗性Sonchus Oleraceus L的完全叶绿体基因组,关于小单拷贝(SSC)区域定位的笔记。线粒体DNA第B部分B. 2018; 3:363-4。
- 27.
Kuo L-Y,Tang T-Y,Li F-W,Su H-J,Chiou W-L,Huang Y-M等。Senselle基因组遗传在Deparia Ferns(Athyriadeae,Aspleniineae,Polypodales)。前植物SCI。2018; 9:486。
- 28.
Neale DB,Wheeler NC,Allard RW。Douglas-FIR中叶绿体DNA的患者遗传。可以J for Res NRC Research Press。1986; 16:1152-4。
- 29.
KH,李万辉,夏普总理。在植物线粒体、叶绿体和核dna中,核苷酸替代率差异很大。acta Natl academy Sci. 1987;84:9054 LP-9058。
- 30.
Provan J, Soranzo N, Wilson NJ, Goldstein DB, Powell WA。叶绿体微卫星突变率低。遗传学。1999;153:943 lp - 947。
- 31。
Leitch IJ,Beaulieu JM,Chase MW,Leitch Ar,Fay MF。单焦度的基因组大小动态和演化。j bot。2010; 2010:1-18。
- 32。
(1)被子植物的起源和早期进化。Ann N Y Acad Sci John Wiley & Sons, Ltd. 2008; 1133:3-25。
- 33。
单子叶植物早期进化的各个方面。机器人学报。John Wiley & Sons, Ltd;2018; 24:421 - 436。
- 34。
霍利,阿普加J,埃Everett GA, Madison JT, Marquisee M, Merrill SH,等。核糖核酸的结构科学美国科学促进会1965;147:1462-5。
- 35。
水稻基因组trna分析表明,水稻中存在新的trna。麝猫。2017;8。
- 36。
Goodenbour JM,泛T.真核生物中TRNA基因的多样性。核酸RES。2006; 34:6137-46。
- 37。
Kirchner S,Ignatova Z. TRNA在自适应翻译中的作用作用,信号动态和疾病。NAT Rev Genet自然出版集团。2014; 16:98-112。
- 38。
Maréchal-Drouard L, guilleaut P, Pfitzingzer H, Weil JH。叶绿体tRNA和tRNA基因的结构和功能。在:Mache R, Stutz E, Subramanian AR,编辑。光合作用细胞器。柏林,海德堡:施普林格柏林海德堡;1991.45-57页。
- 39。
Laslett D,CanbäckBaragorn。在核苷酸序列中检测TRNA基因和TMRNA基因的程序。核酸RES。2004; 32(1):11-6。
- 40.
扩展RNA聚合酶III转录组。趋势麝猫。23日(12):614 - 22所示。
- 41.
Lyons E,Pedersen B,Kane J,Alam M,Ming R,Tang H等。在拟南芥和小组番木瓜中发现和比较了同族地区,杨树和葡萄:COGE与ROSID。植物理性。2008; 148:1772 LP-1781。
- 42.
作者相关文章(1)多倍体和被子植物多样化。我是J·伯特·威利·布莱克威尔。2009; 96:336-48。
- 43.
李天华,唐华,王旭,AH Paterson。PGDD:植物基因和基因组复制数据库。核酸Res。牛津大学出版社;2013年,41:D1152-D1158。
- 44。
Simon R,WJ F.在基因组上加倍:多倍体和作物植物。我是J·伯特·威利·布莱克威尔。2014; 101:1711-25。
- 45。
Mohanta Tk,Syed As,Feen F,Bae H.新的基因组和肌瘤TrNA的进化视角。麝猫。2017;8。
- 46。
Hiratsuka J, Shimada H, Whittier R, Ishibashi T, Sakamoto M, Mori M.水稻叶绿体基因组的完整序列:不同tRNA基因之间的分子间重组解释了在谷物进化过程中主要的质体DNA倒置。Mol Gen Genet, 1989;217。
- 47。
豪CJ。小麦叶绿体DNA逆转的终点与短的重复序列有关,包含同源的toatt-lambda。咕咕叫麝猫。1985;10:139-45。
- 48。
Kozak M.在原核生物和真核生物中发起翻译。基因。1999; 234:187-208。
- 49。
Guillon JM,Mechulam Y,Schmitter JM,Blanquet S,Fayat G. Met-Trna(F)/(Met)甲基转移酶的破坏严重损害大肠杆菌的生长。J细菌。1992年; 174:4294-301。
- 50。
沃尔斯特u,李cp,seong bl,拉杰赫卡里ul。引发剂TRNA的突变体,其作为引发剂和延长器起作用。J Biol Chem。1991年; 266:18018-24。
- 51。
Salinas-Giegé T, Giegé R, Giegé P.线粒体中的tRNA生物学。Ibba M,编辑Int J Mol Sci MDPI。2015; 16:4518-59。
- 52。
关键词:tRNA,真核生物,基因结构,转录生物化学批评家泰勒和弗朗西斯1985;19:107-44。
- 53。
Paquin B,Kathe SD,Shub Da,Paquin B,Kathe SD,Nierzwicki-Bauer SA。C组中Incanobacterial TRNA基因的原点和演变。C组中Incanobacterial TRNA基因的原点和演变。J细菌。1997年; 179:6798-806。
- 54。
Rudi K,Jacobsen Ks。睾丸细胞TRNA(LEU)(UAA)I基团Introns具有多骨来源。有限元微生物吧。1997年; 156(2):293-8。
- 55。
Köhrer C, Mandal D, Gaston KW, Grosjean H, Limbach PA, RajBhandary UL。没有tRNA(Ile)-赖氨酸合成酶的生命:在缺乏典型tRNA(2)(Ile)的枯草芽孢杆菌中异亮氨酸密码子AUA的翻译。核酸学报2014;42:1904-915。
- 56。
Magadum S,Banerjee U,Murugan P,Gangapur D,Ravikesavan R.基因复制作为演变的主要力量。J Genet。2013; 92:155-61。
- 57。
基因家族的进化。在:Genet E,编辑。G.纽约:学术出版社;2001. p。666.–9.
- 58。
Carroll D,复制G.在:Genet E,编辑器中。G.纽约:学术出版社;2001. p。778-80。
- 59。
Panchy n,Lehti-shiu m,shiu s-h。基因重复在植物中的演变。植物生理美国植物生物学家学会。2016; 171:2294-316。
- 60.
kejnovsky e,leitch ij,leitch ar。对比在高原培养物和哺乳动物基因组之间的进化动态。趋势ECOL EVOL EXTELVIER。2009; 24:572-82。
- 61.
Shahid Masood M,Nishikawa T,福冈S,Njenga PK,Tsudzuki T,Kadowaki K.野生稻(Oryza Nivara)叶绿体基因组的完整核苷酸序列:野生栽培水稻的第一基因组宽比较分析。基因。2004; 340:133-9。
- 62.
Saski C,Lee Sb,Fjellheim S,Guda C,Jansen Rk,Luo H.完全叶绿体基因组序列的Hordeum Vulgare,高粱双子和Agrostis stolonifera,以及与其他草内基因组的比较分析。Al Appl Genet。2007; 115。
- 63.
Tillich M,Lehwark P,Pellizzer T,Ulbricht-Jones ES,Fischer A,Bock R等人。Geseq - 多功能和准确的细胞器基因组的注释。核酸RES。2017; 45:W6-11。
- 64.
Bernhart SH, Hofacker IL, Will S, Gruber AR, Stadler PF. RNAalifold:改进的RNA序列一致结构预测。BMC生物信息学。2008;9:474。
- 65.
Lowe TM,CHAN PP。Trnascan-SE在线:集成搜索和上下文以分析转移RNA基因。核酸Res。牛津大学出版社;2016; 44:W54-W57。
- 66.
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析版本6.0。生物学杂志。2013;30:2725-9。
确认
作者谨此感谢Nizwa大学自然和医学研究中心的主席,为携带这项研究提供必要的支持。作者还希望对其在沙特大学国王科学研究的院长致以诚挚的赞赏,以获得这项研究组NO(RGP-271)。
数据材料的可用性
本研究中使用的所有基因组TRNA序列作为附加文件提供3.:数据S1。
竞争竞争
两位作者宣称他们没有相互竞争的利益。
作者信息
隶属关系
贡献
TKM:构思构思,进行实验和分析,起草和修改稿件,ALK:修改稿件,DY:修改稿件;AH:草稿修改稿,EFA:草稿修改稿,AH:修改稿所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
附加文件1:
表S1。叶绿体tRNA的受体臂、d臂、d环、反密码子臂、可变环、伪尿嘧啶臂、伪尿嘧啶环的核苷酸组成。(多克斯26 kb)
附加文件2:
图S1。蓝藻tRNAs的系统发育树项圈藻cyalindrica那Methanococcus maripludis.那Methanospirillum Hungatei.那颤藻渐尖, 和热电偶Sibiricus..这些物种的TrNA被包括为InGroup,而AtNAC1和AtNAc2(NAC转录因子)包括拟南芥蒂利亚纳作为外群体使用。采用Neighbor-joining法构建系统发育树,MEGA6软件进行1000个bootstrap重复。(PDF 114 kb)
附加文件3:
数据S1。单胞植物研究叶绿体基因组的TRNA序列。(TXT 24 KB)
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
莫汉塔,t.k.,可汗,a.l.,哈什姆,A。et al。单子叶植物叶绿体tRNA的基因组和进化。BMC植物BIOL.19,39(2019)。https://doi.org/10.1186/s12870-018-1625-6
已收到:
公认:
发表:
关键字
- TRNA.
- 叶绿体
- 反密码子
- 进化
- 过渡
- 颠换
- 系统发育学报