摘要
背景
转录组图是多种生物学研究的有力工具;转录组图谱包含了至少30种植物的不同器官、组织、细胞和发育阶段。其中一些包括经过环境或生物胁迫处理的样品。然而,大多数研究只探讨了有限的器官和发育阶段(叶片或幼苗)。为了更广泛地了解冷胁迫反应的器官特异性策略,我们利用RNA-seq技术研究了低温(+ 4°C)下植物不同地上部位子叶、下胚轴、叶片、幼花、成熟花和种子的表达变化。
结果
叶片中差异表达的结果与目前对胁迫反应途径的认识是一致的,特别是CBF基因。在其他器官中,基因表达变化的本质和动态是不同的。我们展示了在非应激条件下局限于狭窄表达模式的基因参与应激反应。特别是,控制花粉细胞壁修饰的基因在叶片中被激活。在种子中,主要模式是脂质代谢的变化。
结论
应激反应是高度器官特异性的;在不同类型的器官中,这一过程涉及不同的途径。结果与先前发表的转录组图谱相结合拟南芥并用于公共数据库TraVa的更新:http://travadb.org/browse/Species=AthStress。
背景
自第一个植物转录组图谱构建以来[1],许多植物的基因表达图谱被发表,并成为植物研究中广泛使用的工具。根据定义,转录组图或基因表达图谱是所有基因在不同器官、组织或细胞中在各种环境条件下的表达谱的集合[1]。目前,这样的收集覆盖了从苔藓到植物分类群[2]和松树[3.]到许多种类的小行星和小行星,包括模型[4,5],农业的[6,7]和观赏植物[8]。
在过去的十年中,转录组图被应用于各种研究,包括基因调控网络推断、进化分析和某些发育过程的解剖[3.,9,10]。然而,尽管定义了基因表达图谱,但只有少数论文将胁迫条件下样本的转录组图谱作为主要数据集的一部分[8,11,12]或用于验证地图的完整性[5]。
提高抗冻性的过程-冷驯化-在中国得到了很好的研究拟南芥[13]。主要的调控基因在答:芥是在冷反应研究中用经典遗传学方法发现的。CBF通路在冷驯化中起重要作用[j]14]。CBF(CBF1,CBF2,CBF3也被称为DREB1b,DREB1c,DREB1a,分别)在15分钟内诱导基因[15,16,17,18]。这些基因编码AP2/ERF转录因子(TF)家族,该家族识别靶基因启动子中的C-repeat (CRT)/脱水反应元件(DRE) DNA调控元件[17,19]。在本构性下过度表达CBF基因,许多基因,被称为CBF调控,被激活;这些基因与转录组研究发现的一组冷调控基因部分重叠[16,17,20.,21]。除CBF抗寒途径外,其他tf也参与冷驯化。特别是,ZAT12表达与并行增加CBFS,其过表达导致高抗冻性[22]。
在过去的20年里,人们做了大量的工作来全面表征低温处理后的转录组变化答:芥[13]。在微阵列研究中,数千个基因已被确定为冷诱导基因,这使得对控制驯化过程的调控级联有了更全面的了解[22,23,24,25]。这些工作研究了耐寒性的各个方面。例如,Kilian等人的论文[24]提供了一个时间过程实验中基因表达谱的详细描述。Vogel等人的研究[22],分析了TFs组成型过表达的转基因系。最后,Rasmussen等人[26研究了10的冷反应的变化答:芥生态型。这些研究强调了不同生态型的冷胁迫反应调控网络的巨大差异。
然而,大多数聚焦于转录组学研究的论文都是在植物的整个地上部分进行的[23,24]或树叶[25,26]。据我们所知,目前还没有人尝试分析和比较植物不同部位在冷暴露期间的表达谱。此外,缺乏将应激数据与发育转录组图相结合的研究。本文旨在探讨这一问题,并利用RNA-seq分析了冷处理3和27 h后拟南芥植株六个部位的表达情况。我们鉴定了胁迫和对照样品之间的差异表达基因,并在植物器官之间进行了比较。这一比较揭示了DE基因的数量和组成以及受影响的生物过程的显著差异。我们还观察到CBF和其他已知冷反应基因在样品之间的行为差异,并在拟南芥基因表达图谱中发现了这些基因的独特转录组特征。基因表达谱数据上传到数据库TraVa (travadb.org) [5],并集成了详细的转录组图答:芥。
结果与讨论
正常条件下转录组图中与冷相关的氧化石墨烯富集
分析过程中基于RNA-Seq拟南芥转录组图[5我们检测了所有对样本之间的差异表达(DE)基因,以表征基因表达谱的一般变异性。对于每个比较,我们都计算了GO的富集程度,并发现在3081个比较中,有598个比较中GO:0009631~冷驯化或GO:0009409~对冷的响应中GO:0009409~的过度代表。在许多情况下,这种富集出现在同一器官的不同部分之间,如叶柄和嫩叶的薄片,因此这种效应的起源不太可能是由于植物生长过程中的扰动。
我们引入了一个度量标准“DE Score”答:芥转录组图,对应于基因为DE的两两比较的数量,反映了基因表达模式的宽度和表达水平的差异。带有GO:0009631和GO:0009409类别注释的基因的中位DE Score为1326。这一结果表明,在正常条件下,胁迫应答基因参与了植物的各种过程,并在植物的不同器官中以不同水平表达。
因此,冷相关基因在其他生物过程中的差异参与提出了一个问题,即这些基因在植物不同器官中参与冷驯化的差异。
样品的选择和测序
由于我们的研究旨在分析和比较植物不同部位的胁迫反应,我们使用最近构建的转录组图选择了最不同的生物样本答:芥[5]。在样本聚类的基础上,我们选择了幼苗的子叶和下胚轴、成熟叶、花期初花、第9期花[27]和绿色成熟种子(样本在附加文件中描述)1:表1)。从另一个角度来看,我们关注的是在接近自然的条件下生长的植物;因此,植物是在长白天(16小时光照/8小时黑暗)-光照循环的土壤中种植的,而不是之前使用恒定(24小时)光照的许多研究。众所周知,昼夜节律对应激反应基因的表达有很大的影响[28,29];因此,在光周期内,每个样品都在同一时间收集。对于每个样本,获得了2000万至2500万次读取。皮尔逊平均值r2重复之间的相关性为0.97(附加文件)1表2)。种子、花和幼花按生物样品聚类,而叶片、子叶和下胚轴彼此非常接近,并根据胁迫处理时间部分聚类(附加文件)2)。这可能是由于所有这些器官都是光合作用的,因此具有高度相似的表达谱和相似的应激反应动态。
不同样本间差异表达基因的数量差异很大
为了研究不同器官对寒冷反应的差异,我们对每个样品(特定器官分别在冷处理3 h和27 h后分别称为“样品名称3”和“样品名称27”)在冷处理3和27 h时的DE基因进行了鉴定。我们观察到DE基因上调和下调的数量有显著差异(花3为1022个,叶27为6867个)。不同器官同时的DE基因下调与上调比例(叶3为0.57,下胚轴3为1.32)和同一器官在两个时间点的DE基因下调与上调比例(花3为0.52,花27为1.4)不同(表2)1)。
总的来说,在12例病例中,至少有1例的15,459个基因是DE(附加文件)1表S3)。所有的后续分析仅针对这15459个应激反应基因进行。
CBF调节基因在应激条件下被激活
如前所述,早期对冷响应的大规模研究主要集中在叶片或整株幼苗上。当考虑叶片、子叶和下胚轴时,我们的结果与这些研究结果高度一致。特别是Park等人。[21]通过对过表达的转基因株系进行表达分析,剖析了CBF调控CBF组成型花椰菜花叶病毒(CaMV) 35S启动子下的基因对于叶片、子叶和下胚轴,在我们的数据集中,大多数DE基因在转基因系和冷处理下的表达变化方向匹配(匹配百分比从下胚轴3的50%到叶27的82%)(附加文件)1:表S4和表S5)。匹配率最低的是种子3(32%)。这表明我们的分析为先前探索的系统提供了一致的结果,并有可能为应激反应中的器官特异性模式提供新的见解。
所有器官共有的基因都与压力有关
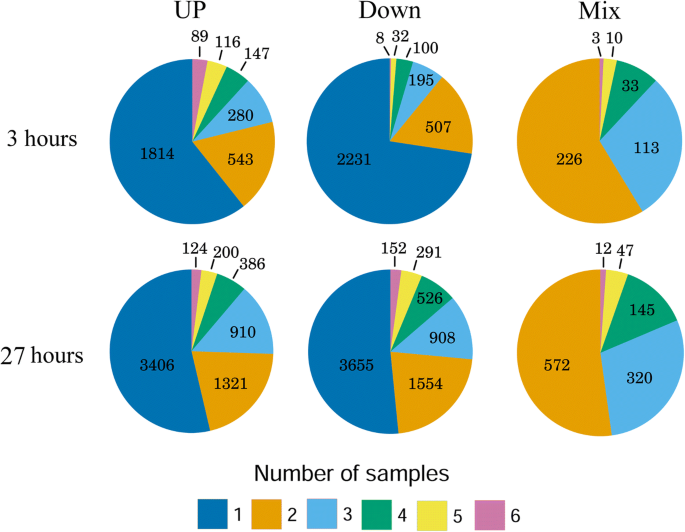
我们将DE基因分为以下三类:在至少一个样本中下调且在任何样本中均未上调的基因(“Down”),在至少一个样本中上调且在任何样本中均未下调的基因(“Up”)以及在至少一个样本中下调且在至少一个样本中上调的基因(“Mix”)。在3 h时,3073个DE基因属于Down类,2989个属于Up类,只有385个DE基因属于Mix类。在27 h时,7086个基因属于Down类别,6347个基因属于Up类别,令人惊讶的是,大量DE基因属于Mix类别,1096个,揭示了不同器官中基因参与冷反应的差异(附加文件)1表6)。我们发现,在所有类别的所有器官中,共有的基因数量极低。在3 h时,只有8个常见基因下调,89个基因在所有器官中上调,3个基因呈混合模式(即在几个样本中上调,在其他样本中下调)。27 h后,Up、Down和Mix类别共有基因数量分别增加到152个、154个和12个(图2)。1、附加文件1:表S7和附加文件3.)。冷处理3 h和27 h的Up类别中常见DE基因在与应激相关的基因本体(GO:0009631~冷驯化)类别中都有强烈的富集(附加文件)1表S8和附加文件4)。器官中常见的少量基因及其显著的氧化石墨烯富集表明,这些基因是抗寒最普遍的“核心”基因。共同基因数量在27 h的增加表明植物器官对胁迫的早期反应具有相当的特异性,在胁迫反应的后期阶段变得更加普遍。
在冷处理3小时后,6个样品中的5个或所有样品中共有的下调基因数量非常少,并且它们没有任何显著的氧化石墨烯富集。低温处理27 h后下调的基因在光合条件下富集,这与低温对光合作用的抑制作用是一致的[30.[附加文件1表S8和附加文件4)。
样本特有的基因参与了器官特异性过程
以器官特异性方式改变冷响应表达水平的基因数量从花3和幼花3中上调基因的90个到叶27中下调基因的1115个不等(附加文件)1:表S7和附加文件3.)。冷处理3 h后,共有4045个基因为器官特异性基因,冷处理27 h后,共有9780个基因为器官特异性基因。
虽然普通基因有应激相关的富集,但器官特异性DE基因以特定的方式富集(附加文件)1(表S9和S10)。在子叶3中下调的基因富含含有五肽重复序列(pentatricopeptide repeat, PPR)基序的基因,这些基因已被证明在细胞器生物发生和RNA编辑中发挥重要作用[qh]31,32]。这类基因在子叶27的上调基因中被过度表达。在这些基因中,PPR蛋白是由SOAR1已被证明调节寒冷、干旱和盐胁迫反应的基因[33]。低温处理27 h后,叶片中表达下调的基因被富集用于光合作用。在种子3号和27号中,与RNA剪接、真菌响应和脂质转运相关的下调基因种类丰富,而在种子3号和27号中,与脂质储存相关的上调基因数量过多。已知导致膜稳定的细胞脂质组成的变化在抗冻机制中起作用[34]并且可能特别重要,因为在种子发育过程中脂质的积累是植物繁殖的关键过程[35]。下胚轴27在上调基因中表现出光合富集。幼花27的上调基因在分解代谢过程和花粉相关术语中富集(附加文件)1(表S9和S10)。
不同器官冷适应过程的显著多样性为通过器官特异性反应调节一般应激反应提供了证据。这一事实可能会限制将一个器官(如叶片)的冷反应知识应用于另一个器官(如种子)。
非叶器官的应激反应不涉及许多已知的调节因子和应激反应基因
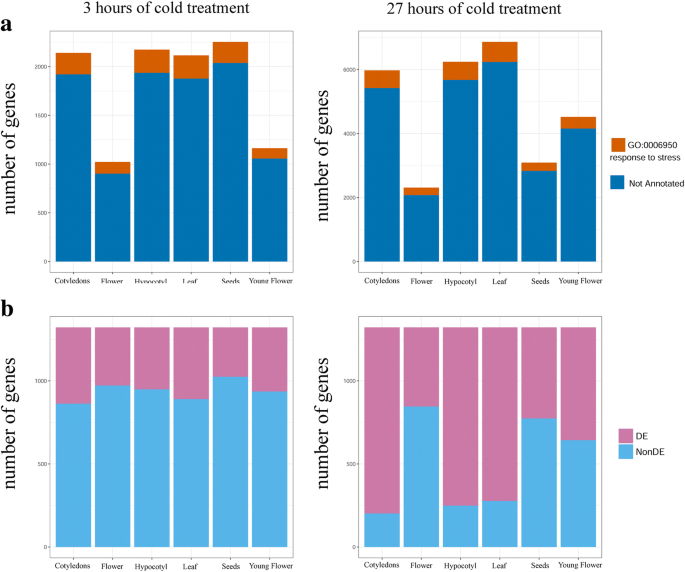
我们发现令人惊讶的低比例DE基因被注释为应激反应(GO类别“GO:0006950~对应激的反应”和下游类别)。在3 h和27 h的样本中,这类基因的比例在8%到12%之间变化;胁迫注释基因的绝对数量以3号花最少(106个),27号叶最多(628个)。这些结果表明,GO的注释拟南芥基因组严重低估了应激反应基因的数量。这在通常不是应激反应研究重点的器官中尤其明显,例如花和种子(图2)。2a). GO注释有几个缺点,已知是不完整的;特别是,大约50%答:芥基因没有生物学过程注释。此外,许多GO注释仅基于计算预测,不受实验数据的支持[36]。
搜索由实验数据确定参与冷应激反应的基因列表(1322 COR基因)[21],我们发现经过3小时的冷处理后,这些基因中有64%在至少一个器官中存在DE(26%属于Down类,34%属于Up类,4%属于Mix类)(附加文件)1(表S11)。从各器官基因来看,DE基因所占比例从种子3的22%到子叶3的35%不等(图2)。2b). 27小时后,情况更加明显。虽然99%的COR基因在至少一个器官中为DE,但基因在各器官中的分布差异很大;在27号子叶、27号叶和27号下胚轴中,79-85%的COR基因为DE,而在27号花、27号幼花和27号种子中,DE的比例分别为36%、51%和41%(图2)。2b、附加文件1:表S12)。这些结果表明,非光合器官的冷胁迫反应不仅涉及以前与胁迫无关的额外基因,而且还没有招募许多已知的调节因子。
的CBF1,CBF2和CBF3基因在接触寒冷后几分钟内就会被激活[15,18]。尽管这些基因在寒冷适应中至关重要,但在对压力的早期反应中并不是唯一的。采用Kilian等人的时程实验。[24], Park等。[21],确定了27个转录因子与cbf编码基因(称为“第一波”基因)具有相同的行为。在我们的数据中,第一次表达测量时间是在低温条件开始后3小时,这不是最早的时间点,尽管我们仍然能够在至少一个器官中识别出除一个外的所有第一波基因为DE。冷处理3 h后,4个基因在各器官中均失活(包括CBF3),而其他人在表达模式上存在明显差异(附加文件1(表S13)。对于5个基因,我们观察到不同器官中相反的表达变化,其中折叠变化差异很大(例如ZAT12下胚轴3和叶3分别为0.22和2.96)。27 h后,表达变化的多样性更加显著;来自大多数器官的第一波基因表达下调或已恢复到控制值。然而,在叶片27中,仍有19个基因上调(附加文件)1(表S14)。早期激活的转录因子对寒冷反应的多样性证实了简单转移或从一个器官到另一个器官的结果的不充分。
应激反应基因的表达特征:香农熵
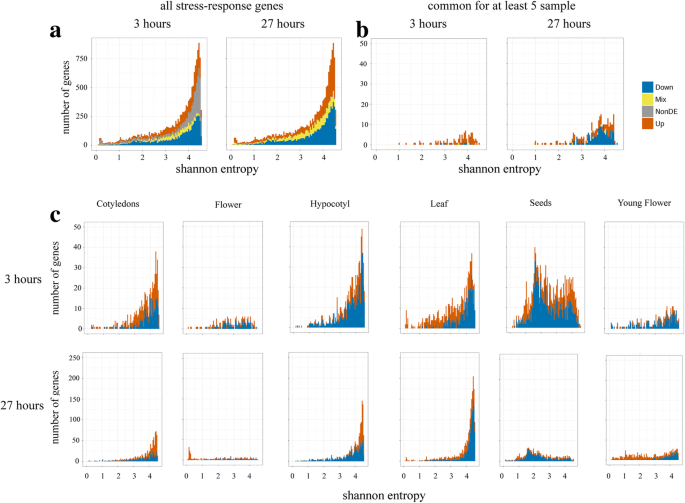
基于RNA-seq转录组图,我们评估了与器官特异性应激反应基因相关的几个参数答:芥[5]。我们首先利用Shannon熵H [37,38]。高H值的基因普遍表达,低H值的基因表达模式较窄。所有表达基因的熵分布明显向右倾斜,表明大量基因具有广泛的表达模式[5]。第二个小峰出现在非常低的H值,对应于以器官、组织或阶段特异性方式高度表达的基因。15459个因应激而发生表达变化的基因分布与总体分布相似,而至少5个器官基因中常见的基因缺乏低H峰(图2)。3.a和b)。大多数样本的独特基因熵分布与常见DE基因相似。然而,一些样本在H分布上有明显的特征。具体来说,对于叶片3中上调的基因,在低熵值(0-0.3)处有一个峰值(图2)。3.c).我们分析了这些基因在转录组图中的表达模式。令人惊讶的是,在非胁迫条件下,所有这些基因的表达(除了一个例外)仅限于成熟花药和在同一阶段含有花药的整朵花1:表S15)。冷处理3 h后,这些基因在叶片中差异表达,其中一半(44%)在叶片27中也为DEs。在花27中只有33%的基因是DE,在花3中没有基因表达转移。对于这些基因中的大多数,它们的功能与控制细胞壁条件和花粉管生长有关1:表S15)。其中,编码果胶甲基酯酶的基因变化最为明显。果胶是细胞壁的重要组成部分,是其他多糖(纤维素和半纤维素)嵌入的基质。果胶在高尔基体中以高度甲基化的形式产生,然后被果胶甲基化酯酶修饰,催化去酯化[39]。酯化果胶与去酯化果胶的比例决定了许多细胞壁性质,如刚性、渗透性和内聚性。在其他植物中也发现了在冷胁迫下果胶甲基酯酶活性的增加[40,41]。它被认为是一种冷驯化机制,因为细胞壁硬度的增加提供了更高的抗脱水能力和抑制器官生长。我们的研究结果表明,该机制的实施答:芥通过招募花粉特异性基因而发生。
与叶片3类似,花27中唯一上调的DE基因的H分布在低值处有一个峰值(图2)。3.c).所有这些基因也是花药特异性的。氧化石墨烯富集分析显示,与花粉管生长和细胞壁修饰相关的术语代表性过高1(表S16)。这些基因揭示了一幅复杂的画面。特别是,我们观察到果胶甲基酯酶抑制剂(PMEI5(AT2G31430)等),而果胶甲基酯酶(PPME1,VGD1,VGDH2(AT1G69940,AT2G47040,AT3G62170)也有所增加。果胶甲基化酯酶PPME1已被证明能线性地对花粉管壁上的果胶链进行去甲基化。PPME1突变体中PPME1活性降低导致细胞壁硬度降低[42]。VGD1是另一种编码果胶甲基酯酶的基因。VGD1也具有线性去甲基化活性,并修饰花粉壁果胶[43]。与PPME1类似,VGD1功能的丧失会导致花粉管壁强度的降低。这两个基因在花27中均上调了2 - 3倍。我们发现增加了2倍CWINV2表达式(附加文件1(表S16)。CWINV2是细胞壁转化酶基因家族(CWINs)的一员,该家族编码将蔗糖降解为葡萄糖和果糖的酶。CWIN的活性对花药发育和活花粉的产生至关重要[44]。在许多抗寒植物中,冷胁迫降低了花粉不育性[45,46]。对低温反应不同的水稻品种的比较表明,花粉具有特异性CWIN在感冷品种中下调,在耐寒品种中不变[46]。增加CWINV2因此可能是冷驯化途径中的一种机制,通常伴随着单糖转运体活性的增加,从而为花药提供糖[46]。然而,我们没有观察到STP编码单糖转运蛋白的基因拟南芥。我们认为,我们在花27中观察到的基因表达景观不是一个稳定的状态,如果回到非胁迫条件下,可能会逆转到正常的发育状态,或者由于冷驯化反应而进一步展开。
如上所述,我们还观察到参与花粉管生长的基因。极性生长是正常花粉管发育的基本特征,需要f -肌动蛋白排列的时空调节。植物Rho GTPases (ROPs)家族参与一系列生物过程,包括花粉管和根尖生长、激素反应和生物胁迫反应[j]。47]。rop受阳性调节因子鸟嘌呤核苷酸交换因子(gef)的控制[48]。对于ROPGEFs家族(含有植物特异性ROP核苷酸交换结构域的GEFs),已经证明参与花粉管生长[49,50]。ROP1已知激活两种相互竞争的f -肌动蛋白组装途径,这两种途径由RIC4, f -肌动蛋白的分解由RIC3[51]。f -肌动蛋白的组装导致细胞尖端囊泡的积累,而f -肌动蛋白的分解则需要胞吐作用,这两个过程的适当平衡对于极性生长是必要的[52]。在《花》第27章中,ROPGEF8 (AT3G24620)和RIC3 (AT1G04450)是双重上调的(附加文件1(表S16)。此外,两个基因-PRF4和PRF5-增加了两倍。Profilin是花粉管顶膜肌动蛋白聚合的调节剂;prf5和prf4突变体的花粉管生长速度减慢。花的样本对应于花期的花,那里已经发生了授粉。负责花粉管生长的基因表达的增加可能表明受精率的增强。
在种子3中,上调和下调基因的香农熵分布都是双峰的(图2)。3.c).种子27缺少高H基因,中等H基因列表与种子3的基因有强烈重叠。对转录组图中含有中等H的基因的表达模式分析显示了它们对种子的特异性。这两种情况下的大多数基因在F-box、植物防御GO和其他类别术语中都被下调和富集(附加文件)1(表S17)。植物F-box是一个大的蛋白质家族,在植物中有897个成员答:芥,而这些基因中的大多数都没有得到充分的研究[53]。的救赎(AT2G17310)是一种含F-box结构域的基因,它编码系统获得性抗病原体的成分[54]。SCRL5 (AT1G60987),LCR73 (AT2G02147),RALFL3 (AT1G23147)其他基因被称为富含半胱氨酸的小型抗菌肽,包括真核生物中常见的防御素[55]。这类基因是防御病原体的已知部分[56]。在种子3中(在大多数情况下,种子27),这些基因被下调(附加文件)1(表S17)。此外,如前所述,种子3和27的上调基因富集于脂质代谢和储存。
应激反应基因的表达特征:DE评分
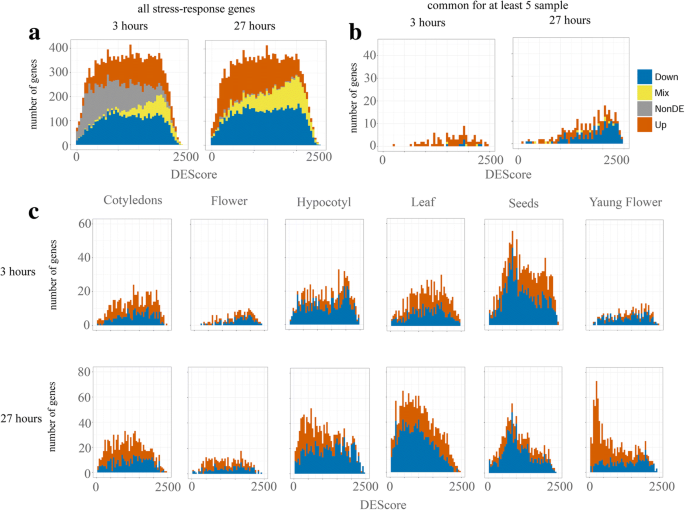
基因表达的另一个参数是DE Score。Klepikova等。[5],分析各对转录组图样本之间的差异表达;DE Score是该基因为DE的配对比较的数量。在转录组图中的79个样本中,最大可能的DE Score为3081。我们分析了在胁迫下表达发生变化的基因的DE分数分布(图2)。4)。与所有表达基因的分布相比,低DE得分的基因在应激数据的DE得分分布中代表性不足(图2)。4a). mix3和mix27基因偏高DE得分。DE Score反映了基因表达水平的变化以及表达模式的宽度。DE评分高表明该基因在多个组织中表达,且在这些组织中的表达水平有显著差异。因此,在冷反应中具有相反表达变化的基因(Mix基因)可能参与不同组织的各种过程。
DE基因在至少5个器官中常见,DE评分分布偏右(图2)。4b). Shannon熵H表明了常见的冷反应基因的广泛表达模式,并支持了这些基因在不同器官的应激反应中的差异参与的观点。
各器官特有DE基因的DE Score分布偏左(图2)。4c).大多数器官在分布上没有明显的峰值,而幼花27的DE基因大多呈上调和下调的峰值,DE评分小于500(图5)。4c)。根据转录组图,这些基因中的大多数是幼小花药所特有的(根据Smyth等人的说法,来自花的第9阶段)。[27])和相似阶段的幼花。这些基因具有较强的氧化石墨烯和其他类别富集,与泛素依赖性蛋白分解代谢过程、转录调控和F-box相关1(表S18)。
泛素化是植物发育过程中重要的翻译后修饰调控机制。泛素26S蛋白酶体系统在蛋白质调节和去除缺陷蛋白中起着重要作用。一些编码泛素26S蛋白酶体系统组分的基因参与了非生物应激反应[57]。
许多F-box蛋白家族成员是SCF (SKP1, CULLIN, FBX和RBX1)复合体的亚基,其中包括E3泛素连接酶的类型[58]。总共有37个被注释为F-box结构域编码的基因在Young Flower 27(附加文件)中上调了2- 13倍1(表S18)。对于一些拟南芥SKP-LIKE (ASK)蛋白,SCF复合体的另一个参与者,显示与F-box蛋白直接相互作用[59]。在《小花》27,8问基因显著上调。
在格莱德曼等人最近发表的一篇论文中[60],结果表明,来自NO APICAL MERISTEM/ARABIDOPSIS转录激活因子-1/CUP-SHAPED COTYLEDONS-2 (NAC)家族的两个蛋白NAC78和NAC56是泛素26S蛋白酶体系统基因的正调控因子。虽然我们没有观察到这些基因在Young Flower 27中的表达变化,但DE基因列表中还包括其他几个基因南汽基因,比如NAC023或NAC063这可能与花发育过程中的冷应激反应有关。
器官特有的基因:ERF/AP2家族外转录因子调控元件的过度表达
为了更深入地了解不同器官中参与冷驯化的基因网络,我们分析了来自不同基因列表(例如,在所有或至少五个器官中常见的转录因子,某些样品特有的DE基因,或具有特定香农熵H的基因)的调控元件的过度代表性;有关测试基因组的完整列表,请参阅附加文件1(表S19)。
正如预期的那样,在所有或至少5个器官中上调的DE基因的启动子区域富含CBF调节元件。具有这些元素的基因,因此可能受到CBF1-3转录因子的调控,显示出应激氧化石墨烯富集条款(附加文件)1(表S20)。正如预期的那样,在所有或至少5个器官中上调的DE基因的启动子区域富含CBF调节元件。导致这种过度表达并受CBF1-3转录因子调控的基因具有应激氧化石墨烯富集。在这些基因中,有12个转录因子在DE基因中也具有代表性,至少在5个器官中常见,并可能受这些基因调控,这些转录因子也富含应激氧化石墨烯术语。其中四个转录因子属于ERF/AP2家族,被描述为生物和非生物应激反应的参与者[61]。
关于每个样本的独特基因,我们没有发现CBF调节元件的任何过度代表,这表明CBF以外的因素控制器官特异性应激反应。特别地,我们发现9个NAC转录因子的调控元件在幼花27的上调DE基因的启动子中过度代表(附加文件)1(表S21)。
数据库
为了使植物科学界能够获得这些数据,我们将它们纳入了我们的数据库TraVA (https://travadb.org)。界面和选项类似于Klepikova等人的数据集。[5]和Kasianov等人。[10]。在对照和冷处理条件下,子叶、下胚轴、叶片、幼花、成熟花和种子中每个基因的谱以相对于对照的reads数和表达水平的倍数变化来表示(图2)。5)。
结论
我们利用RNA-seq分析了冷处理3和27 h后6个拟南芥器官和组织的基因表达。我们发现至少在一个样本中有15459个基因存在差异表达。经过充分研究的器官(叶、子叶和下胚轴)显示出与其他研究相似的结果,而种子、花和幼花则显示出明显的差异。只有少数基因在所有样本中是共同的。这些核心基因在与应激相关的氧化石墨烯类别中富集。独特的样本基因与每个器官的不同过程有关。其中一些基因表现出表达特异性,如香农熵或DE分数分布的峰值。因此,虽然冷胁迫反应的机制在所有植物中都是共同的,但在每个器官中,它们都以一种独特的方式进行修饰,包括在非胁迫条件下在其他器官中表达的基因的募集。
方法
植物生长、冷处理和样品采集
Col-0答:芥(accession CS70000)的植株生长情况与Klepikova等人的描述一致。[62],春化除外。附加文件中描述了收集的示例1表S1。对于每个样品,获得两个生物重复,15个单株植物,并在RNAlater (Qiagen, USA)固定。
对照样品从zt8至9收集,第二天在+ 4°C的气候室中以zt5温度收集。冷处理3 h的样品在zt8采集,第二天在zt8采集27 h。
RNA提取与测序
用RNeasy Plant Kit (Qiagen, USA)按照制造商的方案提取总RNA。测序用TruSeq RNA Sample Prep Kits v2 (Illumina)按照制造商的协议构建cDNA文库。测序使用Illumina HiSeq2000,读取长度为50 bp,序列深度为2000万唯一映射读取。
修剪和映射读取和表达水平的确定
对于读段修剪,使用CLC Genomics Workbench 6.5.1,参数如下:“质量分数- 0.005;修剪歧义核苷酸- 2;去除5 '端核苷酸- 1;去除3 '端核苷酸- 1;和丢弃读数低于25英寸的长度。使用CLC Genomics Workbench将修剪后的reads映射到参考文献答:芥只有唯一定位(长度分数= 1,相似分数= 0.95)的基因组(TAIR10基因组释放)对于每个基因,总基因reads (TGR)被确定为该基因上所有reads的总和。排序和映射统计信息显示在附加文件中1表S22。在附加文件中提供了所有样品的总基因读数和RPKM1:表S23和表S24。
差异表达基因的鉴定
使用R包“DESeq2”鉴定差异表达(DE)基因[63]。错误发现率(FDR)为0.05,倍数变化为2作为显著差异表达的初始阈值。
基因本体富集分析
通过基因本体(gene Ontology, GO)和其他注释(作为关键词或作为蛋白质结构域)富集,使用DAVID基因功能注释工具分析下调和上调的DE基因列表,FDR值为0.05,fold change category representation为2作为显著性阈值[64,65]。
分层聚类
使用R包stats中的" hclust "函数获得了一个层次树[66]。
关键转录因子的鉴定
为了确定参与调控观察到的差异基因表达的转录因子,我们使用了基于ampDAP-seq的转录因子靶标注释[67]。对于每组DE基因,我们考虑了所有转录因子的数据。我们估计差异表达基因中靶基因的相对富集量为%靶基因(DE) / %靶基因(非DE)的log2。使用2 × 2列联表(靶标与非靶标,DE与非DE)的右尾Fisher精确检验评估统计显著性,并对多个测试的转录因子(219个tf)进行FDR校正。
加入数据
Illumina序列读取已存入NCBI序列读取档案,项目ID为PRJNA411947。
缩写
- 林后:
-
积极响应
- 德:
-
差异表达
- 走:
-
基因本体论
- PPR:
-
Pentatricopeptide重复
- TF:
-
转录因子
参考文献
Schmid M, Davison TS, Henz SR, Pape UJ, Demar M, Vingron M,等。的基因表达图谱拟南芥发展。生物医学学报。2005;37:501-6。
Ortiz-Ramırez C, Hernandez-Coronado M, Thamm A, Catarino B, Wang M, Dolan L,等。的转录组图谱Physcomitrella金属盘为陆地植物的进化和发展提供了见解。植物化学学报,2016;9(1):205 - 20。
Rafael A, Canas RA, Li Z, Pascual MB, Castro-Rodrıguez V, Avila C,等。松树幼苗组织基因表达景观。植物科学学报,2017;41(1):464 - 467。
李建军,李建军,李建军,等。豌豆RNA-seq数据的全长从头组装(英文)Pisum一L.)提供了一个基因表达图谱,并对该物种的根瘤形成有了深入的了解。植物学报,2015;44(4):1145 - 1145。
Klepikova AV, Kasianov AS, Gerasimov ES, Logacheva MD. Penin AA。高分辨率的地图拟南芥基于RNA-seq分析的发育转录组。植物学报,2016;33(2):444 - 444。
王磊,谢伟,陈勇,唐伟,杨杰,叶睿,等。覆盖水稻全生命周期的动态基因表达图谱。植物科学学报,2010;31(1):591 - 591。
麦考密克RF, Truong SK, Sreedasyam A, Jenkins J, Shu S, Sims D,等。的高粱二色的参考基因组:改进组装、基因注释、转录组图谱和基因组组织的特征。植物学报,2018;33(2):368 - 368。
杜波依斯A, Carrere S, Raymond O, Pouvreau B, Cottret L, Roccia A,等。玫瑰转录组数据库资源及基因表达图谱。生物医学工程学报,2012;13:638。
Edwards KD, Bombarely A, Story GW, Allen F, Mueller LA, Steve A, Coates SA, Jones L. TobEA:烟草种子到衰老的基因表达图谱。生物医学工程学报,2010;11(1):1 - 4。
Kasianov AS, Klepikova AV, Kulakovskiy IV, Gerasimov ES, Fedotova AV, Besedina EG等人通过高质量基因组组装法氏囊-pastoris揭示了多倍体基因组进化早期调控元件的不对称性。植物科学学报,2017;31(1):278 - 291。
Nobuta K, Venu RC, Lu C, Belo A, Vemaraju K, Kulkarni K,等。水稻mrna和小rna的表达图谱。生物工程学报,2007;24(4):473 - 779。
马萨·安,蔡氏,林海,Bryan GJ, Giuliano G,等。参考马铃薯基因组的转录组茄属植物tuberosumPhureja克隆DM1-3 516R44组。科学通报,2011;6(10):e26801。
李建军,李建军。低温感知对高等植物基因表达和抗寒性的影响。植物学报,2012;19(5):737 - 751。
托马斯休曼氏金融。植物冷驯化:抗冻基因及其调控机制。植物生理学报。1999;50:571 - 599。
Gilmour SJ, Zarka DG, Stockinger EJ, Salazar MP, Houghton JM, Thomashow MF。拟南芥CBF家族AP2转录激活因子的低温调控是冷诱导COR基因表达的早期步骤。植物学报。1998;16(4):433 - 442。
Jaglo-Ottosen KR, Gilmour SJ, Zarka DG, Schabenberger O, Thomashow MF。拟南芥CBF1过表达诱导COR基因,增强抗冻能力。科学1998:280:104 - 106。
刘强,Kasuga M, Sakuma Y, Abe H, Miura S, Yamaguchi-Shinozaki K,等。在拟南芥中,DREB1和DREB2两个转录因子与EREBP/AP2 DNA结合域分别分离了干旱和低温响应基因表达的两条细胞信号转导途径。植物学报。1998;10:1391-406。
Medina J, Bargues M, Terol J, psamuz - alonso M, Salinas J.拟南芥CBF基因家族由3个编码AP2结构域蛋白的基因组成,其表达受低温调控,而不受脱落酸和脱水的调控。植物生理学报。1999;19(1):463 - 467。
刘建军,刘建军,刘建军。拟南芥CBF1编码一个含有AP2结构域的转录激活子,该转录激活子与C-repeat/DRE结合,后者是一种顺式作用的DNA调控元件,在低温和缺水条件下刺激转录。科学通报,1997;19(4):344 - 344。
张建军,张建军,张建军。拟南芥转录激活因子CBF1、CBF2和CBF3具有匹配的功能活性。植物化学学报,2004;44(4):767 - 781。
李建军,李建军,李建军,李建军,李建军。拟南芥CBF调控的复杂低温调控网络。植物学报,2015;32(2):391 - 391。
沃格尔JT,扎尔卡DG,范布斯柯克HA,福勒SG,托马斯MF。CBF2和ZAT12转录因子在拟南芥低温转录组配置中的作用植物学报,2005;41(1):1 - 6。
Fowler S, Thomashow MF。拟南芥转录组分析表明,在冷驯化过程中,除了CBF冷响应途径外,还激活了多种调节途径。植物学报。2002;14:16 - 16。
Kilian J, Whitehead D, Horak J, Wanke D, Weinl S, Batistic O,等。AtGenExpress全球胁迫表达数据集:UV-B光、干旱和冷胁迫响应的协议、评估和模型数据分析。植物科学学报,2007;35(2):347 - 356。
Robinson SJ, Parkin IAP。拟南芥的差异SAGE分析揭示了低温下转录组复杂性的增加。生物医学工程学报,2008;9:434。
Rasmussen S, Barah P, Suarez-Rodriguez MC, Bressendorff S, Friis P, constantino P,等。拟南芥转录组对胁迫组合的响应。植物生理学报,2013;31(1):393 - 394。
Smyth DR, Bowman JL, Meyerowitz EM.拟南芥早期花发育。植物细胞。1990;2:755-67。
张建军,李建军,李建军。拟南芥CBF1、2和3的低温诱导是由生物钟控制的。植物生理学报。2005;37:961 - 968。
李春华,郝根内施,张洪生,韩波,朱涛,等。生物钟调控拟南芥关键通路的转录。科学。2000;290:2110-3。
四季植物:拟南芥冷驯化过程中光合碳代谢的变化。植物学报,2002;5(5):596 - 596。
Kotera E, Tasaka M. Shikanai T.一个五肽重复蛋白在叶绿体RNA编辑中的重要作用。大自然。2005;433:326-30。
刘建军,刘建军,刘建军,等。拟南芥五肽重复蛋白的全基因组分析揭示了它们在细胞器生物发生中的重要作用。植物学报。2004;16(6):2089 - 103。
蒋顺生,梅超,梁生,于玉云,陆凯,吴忠,等。五肽重复蛋白SOAR1在拟南芥干旱响应中的关键作用。植物生理学报,2015;33(2):369 - 369。
李强,郑强,沈伟,Cram D, Fowler DB,魏勇,等。了解植物温度诱导脂质通路调节的生化基础。植物学报。2015;27:86-103。
Penfield S, Rylott EL, Gilday AD, Graham S, Larson TR, Graham IA。拟南芥胚乳中的储备动员为黑暗中下胚轴的伸长提供燃料,不依赖于脱落酸,需要磷酸烯醇丙酮酸羧基激酶1。植物细胞。2004;16:2705-18。
李建军,李建军,李建军,等。植物基因组和转录组注释:从误解到简单的解决方案。生物通报,2018;19(3):437-49。
林绍平,林勇,李建平,李建平,等。人与小鼠组织间转录格局的比较。中国科学d辑,2014;31(1):391 - 391。
李建军,李建军,李建军,李建军,李建军。启动子特征与Shannon熵测量的组织特异性相关。中国生物医学工程学报,2005;6:533。
果胶甲基酯酶:在植物生理中起重要作用的细胞壁酶。植物科学,2001;6:414-9。
鲍baldwin L, Domon JM, Klimek JF, Fournet F, Sellier H, Gillet F,等。豌豆发育过程中细胞壁果胶的结构变化是对寒冷的反应。植物化学。2014;104:37-47。
soecka D, Żebrowski J, Kacperska A.果胶是否参与了冬季油菜植物的冷驯化和脱驯化?中国生物医学工程学报。2008;31(1):521 - 531。
田国文,陈明明,Zaltsman A, Citovsky V.花粉特异性果胶甲基酯酶与花粉管生长的关系。生物医学进展。2006;29(4):83 - 91。
姜林,杨少林,谢立林,普春春,张晓青,杨万成,等。VANGUARD1编码一种果胶甲基酯酶,该酶能促进拟南芥花柱和传递束中花粉管的生长。植物学报。2005;17:584-96。
王晓明,王晓明,王晓明,等。低温胁迫下花粉发育的调控网络。植物科学,2016;7:402。
Ohnishi S, Miyoshi T, Shirai S.低温胁迫对大豆花粉发育、授粉和结荚的影响。环境科学学报,2010;39(6):562 - 562。
Oliver SN, Van Dongen JT, Alfred SC, Mamun EA, Zhao X, Saini HS,等。水稻花药特异性细胞壁转化酶基因OSINV4的低温抑制与蔗糖积累和花粉不育有关。植物生态学报。2005;28(3):344 - 344。
倪宝成,吴宏,张怡。RAC/ROP GTPases:植物信号整合和多样化的“集线器”。植物科学,2006;11:309-15。
李志,刘东。ROPGEF1和ROPGEF4在aba介导的拟南芥气孔关闭中的功能调控因子。生物工程学报。2012;56:1253 - 1258。
顾云,李生,Lord EM,杨震。一类新的拟南芥rho鸟嘌呤核苷酸交换因子控制rho gtpase依赖的极性生长。植物学报。2006;18:366-81。
张颖,张晓明,张晓明,等。拟南芥花粉特异性鸟嘌呤核苷酸交换因子GTPase Rop的调控机制。科学通报,2007;44(4):593 - 593。
顾莹,傅莹,Dowd P,李松,Vernoud V, Gilroy S,等。rho家族GTPase通过花粉管的两个反向下游途径控制肌动蛋白动力学和尖端生长。中国生物医学工程学报(英文版);2009;32(1):391 - 391。
李玉军,杨志强,李建军,杨志强。rho - gtpase依赖的丝状肌动蛋白动力学协调尖端生长的囊泡靶向和胞外分泌。中国生物医学工程学报,2008;31(1):444 - 444。
华忠,邹晨,邵诗,Vierstra,等。植物界F-box FBX基因超家族的系统发育比较:基因组漂移的不同进化历史。科学通报,2011;6:e16219。
Kim HS, Delaney TP。拟南芥SON1是一种F-Box蛋白,它调节一种新的诱导防御反应,不依赖于水杨酸和全身获得性抗性。植物学报。2002;14:14 . 69 - 82。
Silverstein KAT, Graham MA, Paape TD, VandenBosch KA。拟南芥300多个防御素样基因的基因组组织。植物生理学报,2005;38(2):391 - 391。
Silverstein KAT, Moskal WA, Wu HC, Underwood BA, Graham MA, Town CD等。富含半胱氨酸的小肽类抗菌肽在植物中被低估。植物科学学报,2007;31(1):391 - 391。
李建军,李建军,李建军,等。蛋白质泛素化介导的非生物胁迫抗性研究。[J] .中国生物医学工程学报,2012;33(3):591 - 596。
J.泛素系统的泛素化机制。见:拟南芥丛书,美国植物生物学家学会;2014.p . e0174。
黄春华,杨春华,李春华,等。拟南芥SKP1-LIKE ASK和F-box FBX蛋白的基因定位分析。科学通报,2012;7:50009。
Gladman NP, Marshall RS, Lee KH, Vierstra RD.拟南芥NAC转录因子对蛋白酶体胁迫调控的研究。植物学报,2016;28:1279-96。
Tsutsui T, Kato W, Asada Y, Sako K, Sato T, Sonoda Y,等。DREB蛋白转录抑制因子DEAR1介导拟南芥植物防御和冰冻胁迫反应。植物学报,2009;22(2):633 - 633。
Klepikova AV, Logacheva MD, Dmitriev SE, Penin AA。一个顶端分生组织时间序列的RNA-seq分析揭示了拟南芥花起始的一个临界点。生物医学工程学报,2015;16:466。
Love MI, Huber W, Anders S.使用DESeq2对RNA-seq数据的折叠变化和离散度进行调节估计。基因组生物学。2014;15:550。
黄d W, Sherman BT, Lempicki RA。利用David生物信息学资源对大型基因列表进行系统综合分析。Nat protocol . 2009; 4:44-57。
黄d W, Sherman BT, Lempicki RA。生物信息学富集工具:通往大型基因列表的综合功能分析的路径。核酸学报,2009;37:1-13。
R核心团队。R:用于统计计算的语言和环境。维也纳,奥地利:R统计计算基金会;2017.
黄春华,宋丽丽,黄春华,李春华,等。漩涡和漩涡特征塑造了调控DNA的景观。细胞。2016;165:1280 - 92。
致谢
作者感谢美国杂志专家(https://www.aje.com/)编辑此稿。
资金
该研究得到了俄罗斯科学基金会(项目# 14-50-00150)的支持。植物生长是在莫斯科国立大学罗蒙诺索夫遗传学系的设备上进行的。调控要素分析是2013-2020年国家科学院基础研究计划(01201363825)资助的。数据库TraVA在IITP RAS(植物基因组学实验室)预算补贴的支持下进行了更新。TraVA数据库在IITP RAS(植物基因组学实验室)预算补贴的支持下进行了更新。
数据和材料的可用性
在当前研究中生成和分析的数据集可在NCBI序列读取存档库中获得,项目ID为PRJNA411947。
关于本品
这篇文章已经作为《植物生物学》2018年第19卷第1期:BGRS\SB-2018:植物生物学论文选集。该补充的全部内容可在网上获得//www.cinefiend.com/articles/supplements/volume-19-supplement-1。
作者信息
从属关系
贡献
AVK收集植物材料,生成图像,执行大部分计算分析,并参与撰写手稿。IVK进行转录因子分析。ASK为转录因子分析准备了数据。MDL设计和协调研究,并参与测序和撰写手稿。AAP构思和协调研究,构建转录组文库,设计最终图形,并参与测序和计算分析。所有的作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们没有竞争利益
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
Klepikova, a.v., Kulakovskiy, i.v., Kasianov, A.S.et al。数据库TraVA的更新:器官特异性冷应激反应拟南芥。BMC Plant Biol19,49(2019)。https://doi.org/10.1186/s12870-019-1636-y
发表:
DOI:https://doi.org/10.1186/s12870-019-1636-y
关键字
- 冷应激
- 拟南芥
- RNA-seq
- 转录组地图
- 器官特异性应激反应