摘要
背景
参与生物发育的基因调节网络(GRNs)结构和功能的变化是理解物种特异性进化策略的关键。即使是发育GRN最微小的修饰也可能导致复杂形态发生模式的实质性改变。毛状体种类繁多,可接近性强,是研究细胞命运决定、细胞周期控制和细胞形态发生等分子过程的有用模型。目前,有大量的基因调控着植物的形态发生答:芥描述了毛状体。在这里,我们旨在研究定义毛状体形成的GRN的进化,并评估其在其他发育过程中的重要性。
结果
在毛状体形成GRN的演化研究中,我们将经典系统发育分析与主要植物类群中GRN的拓扑结构和组成信息相结合。这种方法使我们能够估计GRN组分(主要是蛋白质)的进化出现时间,以及它们分子进化的相对速度。蛋白质结构的各种简化(基于蛋白质球中氨基酸残基的位置、二级结构类型和结构紊乱)使我们能够证明蛋白质球的变化与物种形成/复制事件之间的进化关联。我们讨论了它们在蛋白质-蛋白质相互作用和GRN功能中的潜在参与。
结论
我们假设形成三毛体的GRN的分化和/或专门化与植物类群的出现有关。关于GRN中蛋白质进化的结构目标的信息可以预测进化过程中基因网络功能的开关点。我们还提出了一个候选基因的名单,负责发展的毛状体在广泛的植物物种。
背景
为了理解生物体的发育和进化过程,“基因调节网络”(grn)必须被考虑在内。这种网络的可变性决定了动植物器官形态和功能的多样性[1,2].特化毛状细胞可作为研究细胞命运决定、细胞周期控制和细胞形态发生等分子过程的模型[3.].特别是,该模型有助于解剖模型植物表皮形态发生的机制拟南芥[4].决定具有毛状体的细胞命运的核心作用是组装毛状体起始MBW复合物- (GL3/EGL3-GL1-TTG1),它启动编码转录因子的基因GLABRA2 (GL2)的表达,以启动细胞向毛状体分化的转变[5].除了GL2, MBW复合物诱导抑制基因(TRY/CPC)的表达,这些基因可以在细胞之间移动并组装成复合物(GL3/EGL3-CPC/TRY- ttg1),而这种复合物不能启动毛状体的形成。在拟南芥,发现7个MBW复合物抑制剂的R3-MYB蛋白:TRIPTYCHON (TRY) [6,7],反复无常(cpc) [8, TRY和CPC的增强子1,2 eth3 (ETC1, ETC2 eth3) [9,10,11],以及TRICHOMELESS 1 2 (TCL1 2 TCL2) [12,13].显示了它们之间函数的不同效率[11,14].TCL1很可能是GL1表达的负调节因子[12]以及毛状体的发育,影响GL1的表达,并与GL1竞争与GL3的结合[15,16].值得注意的是,毛状体形成的模式是由广泛的横向抑制机制描述的,已知存在于各种植物和动物生物体中。此外,它还负责蓝藻杂囊的发育[17,18].
除了MBW复合物外,在叶片和花中还发现了一些增加启动子复合物基因表达的基因:无毛花序茎(GLABROUS infrescence stem, GIS), [19] gis2,锌指蛋白8 (zfp8)20.,21], zfp5 [22,23], zfp6 [24].研究表明,MBW复合体中的关键转录因子GL1和GL3在被GIS2和ZFP8激活后发挥作用[24].
许多研究表明,基因同源于拟南芥毛状体相关基因参与棉毛的形成[25,26,27,28,29].然而,早前有人提出,在系统发育较远的物种中,毛状体可以通过其他遗传机制以收敛的方式发展[30.].此外,某些数据也支持毛状体发育的个体调控途径的功能多样化。结果表明,水稻R3 MYB转录因子OsTCL1在水稻中异位表达拟南芥基因组影响毛状体的形成;然而,水稻中OsTCL1表达的改变不会导致任何与三毛体相关的表型变化[31].此外,烟草中GL1基因的过表达对毛状体的发育没有影响。一种解释是含有GL1基因的基因网络首先出现在Rosids中。此外,烟草有五种类型的毛状体,这应该也没有反映出遗传机制的差异[30.].
需要注意的是,MBW复合体与其调控因子直接参与根毛形态发生的抑制[7,9,10,32].因此,有理由认为,一个基因网络的变异是叶表皮和根毛的毛状体模式形成的原因答:芥[33].通过RNA-seq数据,Huang表明,负责根毛的主要基因组在进化距离中保存了长达2亿年或更长时间[34].然而,这些基因的表达模式在不同物种之间差异很大[35].
我们也知道表皮细胞的生长是广泛的,并且是非常古老的形成。在藻类中发现了简单的生长产物轮藻(Charophytales),水绵(双星藻目) [36].藓类植物中的类虫有其特有的模式,并在基质中发挥固定作用,参与吸收水分和养分[37].据透露,Physcomitrella金属盘基因PpRSL1和PpRSL2影响植物的根茎数量[38,39].突变体的拟南芥缺乏RHD6(头发发育的关键基因之一)功能的人如果被PpRSL1基因转化,就会产生根毛Physcomitrella.这表明在4.2亿年的物种分化过程中,RSL家族蛋白的功能并没有丧失[38].
因此,为了理解GRN毛状体形态发生的发育和进化过程,我们需要将蛋白质及其功能的数据结合到与每个主要植物类群分化相关的GRN拓扑中,然后将GRN拓扑的变化与GRN组分(单个蛋白质)的变化联系起来。
任何蛋白质的功能都是其化学和物理性质的直接结果,而化学和物理性质又是由在三维空间中折叠成蛋白质球的空间和物理化学要求所定义的。因此,可以预计,蛋白球中残基与其他氨基酸相互作用的变化与残基上位相互作用的变化密切相关。换句话说,蛋白质进化是崎岖不平的,而这种不均衡性是由最优三维蛋白质空间拓扑结构(如吉布斯能)的突变所驱动的,这反过来又导致了蛋白质空间和进化时间上的崎岖选择。蛋白质进化的计算研究发现了几个众所周知的主要上位特征。这些是(1)在不同谱系中导致蛋白质功能障碍(或疾病)的氨基酸状态的变异性[40];(2)突变耐受性在蛋白质进化过程中切换,换句话说,在一次进化中有害的突变变成无害的,反之亦然[41];(3)任何蛋白质和任何谱系中普遍存在的共变特征[42,43,44].此外,限制性上位相互作用被证明是在蛋白质进化过程中逐渐出现的[45,46].这些相互作用反过来又使祖先的状态变得有害或不可逆[45]或蛋白质进化中的“斯托克斯位移”[46].尽管如此,到目前为止,绝大多数现有的祖先序列重建程序[47,48]基于单一经验氨基酸替代矩阵的可逆性(适用于所有蛋白质位点)。因此,新的祖先蛋白质重建软件工具(例如ProtASR) [49]是最适合蛋白质结构和折叠稳定性的。然而,仍然缺乏实验解决的3D蛋白质结构,特别是在植物科学中。另一种解释标准祖先蛋白质重建中蛋白质上位性的方法是构建祖先文库,以解决由于祖先序列重建不完善而导致的序列不确定性[50].这种方法考虑了一个众所周知的缺陷,即不能保证祖先序列是正确的生物学功能蛋白质,并且在研究深层进化事件时最有用。最近对mRFP1蛋白人工进化的实验研究表明,最大似然法获得的祖先序列与天然祖先mRFP1蛋白的亲缘关系最密切,而采用系统进化树感知贝叶斯方法重构的最佳蛋白质与原生祖先的亲缘关系不太相似[51].然而,这种方法只能重建一个最好的祖先蛋白质,而不能用于祖先文库的生成。为了使祖先库生成足够准确,最近有人建议使用“AltAll”重建方法。这种方法将所有可能的替代状态结合到单个蛋白质中,然后通过这些状态的集合在功能上表征该蛋白质[52,53].结果表明,该方法显著地纠正了贝叶斯后验概率探测生成的祖先序列的不完备性。因此,在缺乏3D蛋白质结构的情况下,我们所能做的最好的事情就是使用“AltAll”衍生方法来构建后续进化研究的祖先库,并进行进化蛋白质功能推断。
因此,有两个总体目标与我们的研究高度相关:(1)为了填补对毛状体形态发生GRN拓扑结构的进化动力学认识的空白,我们需要结合类群特异性GRN并分析其差异;(2)为了填补对类群特异性GRN蛋白质相互作用的分子基础认识的空白,以及对类群特异性GRN之间差异的分子基础认识的空白,需要分析GRN蛋白质结构和功能的进化。在这项工作中,我们结合了与毛状体形态发生相关的GRN拓扑结构的定性信息,并对其组成部分进行了详细的系统发育分析。原始的系统发育分析让我们找到了一个简单的答案,即核心基因亚网络的起源点何时形成。此外,利用蛋白质序列结构类/特征的详细信息,我们研究了与各种物种形成或复制点相关的蛋白质球的进化变异以及潜在的蛋白质-蛋白质相互作用。这允许假设与植物分类群起源相关的GRN函数的分化和/或专门化。关于GRN中蛋白质进化的结构靶点的信息也对未来基因网络功能中进化开关点的识别起着预测作用。
结果与讨论
入库单重建
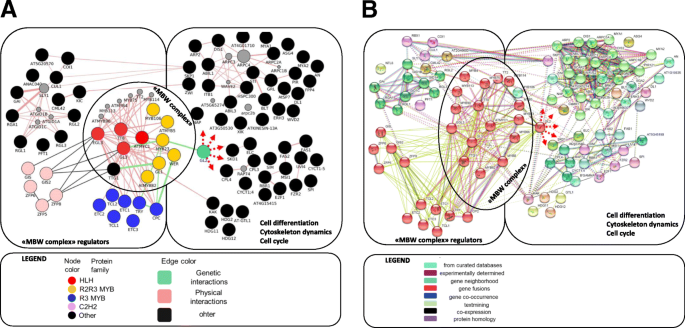
基于多位专家对功能注释的分析,列出了90个与毛状体形成相关的基因答:芥创建.在丰富的过程中,我们使用了来自STRING数据库和Cytoscape (GeneMania插件)系统的文章和信息的手动分析。结果加入了与我们的基因样本连接得分最高的基因。得到的富集基因网络包含123个节点。
在节点中,检测到51个转录因子和6个细胞周期基因(附加文件1).在样本集中,109个超家族域被揭示(附加文件1).其中,MYB, SANT, Homeobox, C2H2这些结构域是蛋白质的特征,其功能包括调节转录。此外,我们发现蛋白质中含有hlh结构域,它介导蛋白质-蛋白质相互作用。然后通过go术语的共现分析基因网络(详情见附加文件)1).
不与其他基因相互作用的基因以及没有实验证实的直接参与毛状体起始和/或发育的信息的基因已从网络中删除。该网络在验证和缩减前后的统计数据在附加文件中提供2.利用STRING和Cytoscape数据对基因网络进行图形表示,如图所示。1.在该网络中,与大量蛋白-蛋白相互作用相关的区域对应于MBW复合体的组成部分(11个节点,如图所示)。1),以及其7个抑制剂TRY, CPC, ETC1, ETC2, ETC3, TCL1, TCL2。在网络的左侧,代表了许多MBW复合物成分表达的调控因子:这是21个基因,其中一些显示了毛状体启动复合物基因的表达,其直接调控或参与激素信号的传递(其中6个对GA和细胞分裂素敏感,6个对茉莉酸敏感,见附加文件1.在网络的右侧有调节级联,其工作可能是在启动复合物的控制下,这部分网络包含与毛状体细胞的生长和分化相关的基因。它们调节细胞分化、细胞骨架动力学和细胞周期等过程。
对于绝大多数基因,如SPI, SIM, FAS, KAK, STI,直接参与调节毛状体的细胞分化过程,分支的形成,以及控制毛状体的细胞内还原(附加文件)1).我们在图中标记信使GL2。1,因为它是选择毛胚细胞命运的关键蛋白质。
MBW毛状体起始复合物组分的系统发育分析
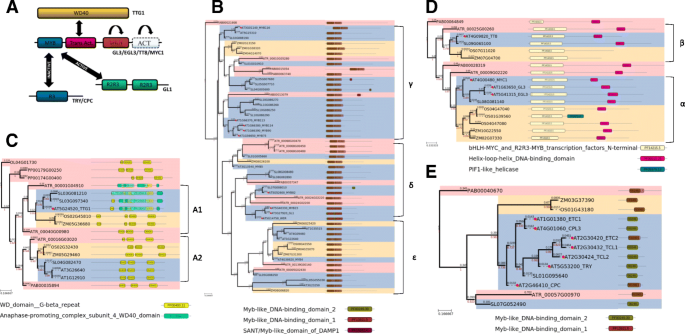
MBW复合体包含11种蛋白质,它们的功能部分重复。这些蛋白分属于四个家族:HLH(4个蛋白:TT8、EGL3、GL3、MYC1)、MYB-R2R3(5个蛋白:GL1、WER、MYB82、MYB5、MYB23)和WD40(1个代表-TTG1)。MBW复合体组装方案和最近同源物的简化系统发育树如图所示。2.完整的树在(附加文件3.,4,5而且6:图S1A-S3A)。蛋白质结构变化的数据也在相应的(附加文件3.,4,5而且6:图S1B-S3B)。
GL3 (AT1G63650)和EGL3 (AT5G41315)序列聚在一起。他们的分歧发生在共同的祖先十字花科.转录因子的MYC/MYB n端结构域(IPR025610 (PF00010))大约有180个氨基酸长。对包含α1支的双子叶植物的所有同源序列进行了预测。2b和附加文件3.),而长度约为45个氨基酸的基本螺旋-环-螺旋结构域(bHLH, IPR011598 (PF00010))介导蛋白质二聚化并表征蛋白质转录因子。在双子叶植物的主要进化谱系分化之前,双子叶植物的共同祖先Gl3/EGL3和MYC1的进化谱系已经发生了多样化。值得注意的是,在α2支序列中,bHLH结构域仅在部分序列中处于可靠的预测状态,而在单子叶子的外层组中,两个结构域都被强烈预测。在双子叶植物和单子叶植物中,与TT8同源的蛋白序列形成了一个独立的分支β,具有相同的结构域组成。这些世系的分离发生在双子叶和单子叶植物分化之前。值得注意的是,苔藓和裸子植物中最近外群的同源序列揭示了bHLH和MYC/MYB n端结构域(图2)。2b和(附加文件3.: S1A)。除了Gl3和EGL3外,有迹象表明闭合基因(TT8 AT4G09820和MYC1 AT4G00480)也影响毛状体的发育[54,55,56].结合保守的结构域组织,这表明参与调控形态发生的复合物的组装是其主要功能,并且可能出现在早期陆生植物中。
通过分析蛋白质结构的进化变化(见方法),发现最大的变化发生在同源基因EGL3和GL3的长基支上十字花科序列在彼此分离之前(附加文件3.:图S1B)。同样的观察到的基枝十字花科MYC1进化枝。然而,含有EGL3、GL3和MYC1的开花植物的巨大分支的共同祖先具有低蛋白质结构变化率的特点;其他研究的双子叶植物类群亦是如此(番木瓜;草莓属vesca;木薯耐糖;杨树trichocarpa;碧桃).EGL3/GL3蛋白的结构在单子叶植物的基枝、双子叶和单子叶的分化以及单子叶内的分化中都有显著的变化。同样,在茄科的共同祖先中也观察到显著的变化,但这可能是由于它们在进化过程中很早就发生了分化。
的TTG1蛋白(AT5G24520)答:芥参与组装MBW复合体[57,58,59与TTG1同源的序列形成α支(图1)。2c和(附加文件4: S2A))在双子叶和单子叶中观察到。在所有属于相邻支系的植物同源序列中(α In(附加文件4(: S2)),预测WD40块,根据[60],使蛋白质能够介导蛋白质复合物的组装。与进化支α一起,一个额外的β进化支被聚在一起,包括AT3G26640 (LWD2)和AT1G12910 (ATAN11)蛋白,它们与昼夜节律的功能有关。这表明,双子叶植物和单子叶植物的祖先经历了祖先WD40的多样化,形成了两个生物学功能不同的进化谱系。针叶植物序列(PAB00042769、PAB00049457、PAB00035894、PAB00018188)和苔藓植物序列(PP00290G00030、PP00092G00020、PP00179G00250、PP00174G00400)也具有类似TTG1的结构域组织A.thaliana.在单子叶植物TTG1序列的祖先进化谱系中,该蛋白的结构经历了本质的变化(附加文件)4:图S2B)。在十字花科分支,结果各不相同。用三态模型标注的蛋白失序区和二级结构发生了剧烈变化。同时,8态模型描述的二级结构和氨基酸替代率(相对于蛋白质特异性氨基酸替代率模型)显示出较高的结构保守性。茄科也有显著的变化,但这可能是由于它们在进化的早期就分化了。其他双子叶植物没有显示出任何特定的蛋白质变化模式。
Gl1蛋白是MBW复合体的重要蛋白质,属于R2R3-MYB蛋白家族。这个家族含有126种蛋白质答:芥并根据c端动机分为25个亚组[61].第15亚组R2R3 MYB的代表(例如MYB0/GLABROUS1 (GL1), MYB23 [62], myb5 [63], myb82 [64], wer [65]参与毛状体的形态发生。这些系统发育关系显示在(附加文件5:图S3A)及2D。所有蛋白质在n端都含有两个MYB-DNA结合结构域。蛋白质的系统发育关系被弱分解,这与MYB因子进化的特异性及其蛋白长度小有关。MYB23、WER、GL1的分化发生在祖先十字花科.它们的谱系与MYB82谱系的分歧似乎发生在双子叶植物的祖先身上。对于AT3G13540 (MYB5),在双子叶植物和单子叶植物中都发现了同源物。AT4G38620 - MYB4也是如此。然而,与MYB4一起,基因AT4G09460 (MYB6)和AT1G22640 (MYB3)是聚类的。这些特殊的基因并不知道参与毛状体的发育。基因MYB113、MYB114、MYB90、MYB75、MYB116聚集在一起,被认为参与花青素的合成[66,67].因此,广泛的R2R3-MYB蛋白拟南芥是观察,并为他们中的一些组同源序列一直到开花植物的共同祖先被发现。在R2R3-MYB同源系统发育树中,有3个进化支(MYB75/MYB90/MYB113/MYB114, WER/MYB23/GL1)和一个不含基因序列的进化支十字花科).MYB75/MYB90/MYB113/MYB114及其附近的双子叶蛋白的蛋白结构改变比来自枝WER/MYB23/GL1的蛋白更强烈(附加文件)5图S3B)。在双子叶组中不含十字花科与WER/MYB23/GL1分支相比,其二级结构的变化也有所加快。在单子叶的进化枝中,观察到相对保守的蛋白质(与所有双子叶相比)。值得注意的是,分支中二级结构保存相对较好(与存储结构相比),而树枝相当长,表明氨基酸取代的积累率很高。
R3-MYB家族的蛋白质在拟南芥AT2G46410 (CPC)、AT1G01380 (ETC1)、AT4G01060 (CPL3)、AT2G30420 (ETC2)、AT2G30432 (TCL1)、AT2G30424 (TCL2)、AT5G53200 (TRY)为7个副逻辑基因。在其他双子叶植物中,同源体的数量从1个(草莓)到4-5个(EG和MD,分别)(附加文件)6:图S4和图2E)所有单子叶基因均为1个拷贝。所有的R3-MYB蛋白中间都含有一个MYB-DNA结合域。在R3-MYB蛋白的系统发育树中,有三个进化支。第一种包括单子叶植物和双子叶植物。第二种包括双子叶植物和裸子植物。我们发现,从开花植物的祖先到单子叶植物,二级结构发生了加速变化(单子叶植物中的二级结构发生了变化,参见(附加文件)6:图S4B))在第二个分支中,在双子叶植物和裸子植物中都可以观察到替代积累的减缓。
网络中其他节点的系统发育分析在附加文件中给出7,8,9,10,11而且12.
毛状体起始复合物组分的系统发育分析
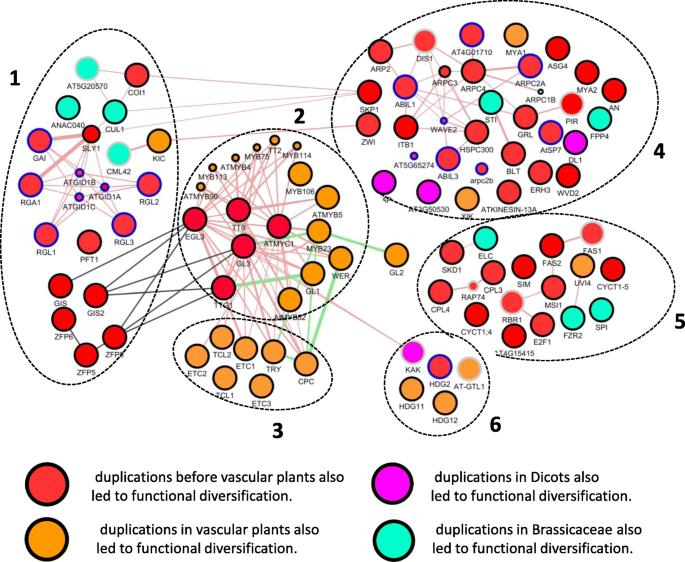
大规模的进化分析使得估计基因网络中每个节点的功能出现的时间成为可能。最广泛的骨科组包括P.patens蛋白质和不含早期重复的蛋白质对应40个基因网络节点,如图所示。3..对于这些蛋白质,我们可以假设在维管植物共同祖先的水平上较早地分离出功能。
表皮形态发生网络的每个节点的功能出现的估计时间答:talyana。配色方案如下。根据节点的功能相似性来区分聚类:1 -与激素信号转导相关的节点。这个集群包含激素反应调节和调节负责感知激素信号。2 - MBW毛状体引发组分的复合物。这个簇包含转录因子和因子负责启动细胞发育沿毛状体的路径。3 - MBW毛状体起始抑制剂的复合物。该簇包含R3MYB基因,并负责抑制启动器复合物(详见简介)。4 -与细胞骨架结构和功能动力学相关的节点。该簇包含细胞内肌动蛋白和肌凝蛋白丝的相关基因和调节组装。5 -与细胞周期动态相关的节点。 This cluster contains genes regulating the cell cycle, in particular G1/S transition of the mitotic cell cycle; the endomitotic cell cycle; and regulation of DNA endoreduplication. 6 - Specific trichome differentiation genes. This cluster contains genes for which participation in the development of trichomes has been shown, but their role is currently not fully understood
这些节点相对均匀地分布在基因网络的不同部分,包括21个与细胞骨架功能相关的蛋白质,12个介导激素调节的蛋白质,以及6个不属于这些组的蛋白质(见附加文件)2).这些蛋白质在图中被标记为红色节点。3..
在双子叶植物或十字花科植物的共同祖先的树形拓扑中具有基础重复的许多蛋白质,并且在两个子分支中都具有重复(参见附加文件)2).这些蛋白质在图中用蓝色圈标出。3..
维管植物中大量的重复导致了功能的多样化。对于这样的蛋白质,我们只能在维管植物共同祖先的水平上断言存在与基因网络相对应的基础功能。细胞骨架功能相关基因(AT1G17580 (MYA1);AT5G20490 (XIK)),细胞周期的功能(AT2G42260 (UVI4)),参与激素信号的传递(AT2G46600 (KIC);AT4G24210 (SLY1))以及三种功能尚未完全建立的蛋白质(见。额外的文件2).这些蛋白质被标记为图中的橙色节点。3..
双子叶植物中大量的重复导致了功能的多样化。对于这样的蛋白质,我们可以断言在双子叶植物共同祖先的水平上存在一种功能。这是与细胞骨架工作相关的2个基因(AT5G42080 (DL1);AT3G50530)和两个功能尚未完全阐明的基因(AT1G69490;AT4G38600(谷湖))。对于KAK基因,在单子叶植物的祖先中也发现了重复。这些蛋白质被标记为图中的粉红色节点。3..
大量的重复十字花科也导致了功能多样化。对于这样的蛋白质,我们可以断言在的共同祖先水平上存在感兴趣的函数十字花科或者只在拟南芥物种。这些蛋白质的功能被认为是“年轻的”。这些节点包括3种与细胞骨架功能相关的蛋白质(AT2G31300 (ARPC1B);AT1G19835), 2个与细胞周期动力学相关的蛋白(AT4G22910 (FZR2);AT3G12400 (ELC)), 6个介导激素调节的蛋白质(AT4G20780 (CML42);AT5G20570 (RBX1);AT4G02570 (CUL1);AT2G27300 (ANAC040);AT5G20570 (RBX1)),以及6个不属于这些基团的蛋白(AT2G02480(STI);AT1G03060 (SPI))。这些蛋白质被标记为图中的绿色节点。3..
在单子叶植物中有相对早期重复的蛋白质被注意到。这是5个与细胞骨架功能相关的基因(AT1G13180 (TMM);AT5G18410 (PIR121);AT3G12280 (RBR1);AT1G65470 (FAS1)), 2个激素调节途径基因(AT4G20780 (CML42);AT5G20570 (RBX1))和6个不属于这些基团的蛋白质(AT4G38600 (KAK);AT1G33240 (AT-GTL1);AT4G12610 (RAP74))。这些蛋白质被标记在图中的灰色圈中。3..
许多基因不适合简单的分类,需要单独提及。
AT5G28646 WVD2是一个与细胞骨架工作相关的基因。双子叶植物的祖先在与单子叶植物分化后发生了重复。谷类植物的祖先已被确认有两个副本。
另一个与细胞骨架工作相关的基因AT5G43900 MYA2,在十字花科植物的祖先中也有两个重复,在谷类植物的祖先中也有一个重复。
与细胞周期进程相关的AT4G15415有两个独立的双子叶分支和一个单子叶分支,其中发生了两次重复。
在开花植物的祖先中出现了环素(AT1G47870 (E2F2)、AT5G22220 (E2F1)、AT2G36010 (E2F3)的分化。
AT1G01520 (ASG4)和AT4G01280(它不是所研究的网络的一个节点)是从双子叶植物的祖先分化出来的。para功能(AT4G01280)没有澄清。在单cots中,复制也被注意到。
进化谱系AT5G06650 (GIS2)、AT3G58070 (GIS)和AT2G41940 (ZFP8)与开花植物的祖先不同,有一个外群PAB00021121。我们在进化支中发现了两个重复的十字花科单子叶(一个在香蕉分化之前,一个在香蕉分化之后)。
AT5G06650 (GIS2)、AT3G58070 (GIS)和AT2G41940 (ZFP8)的进化谱系在开花植物的祖先(PAB00021121外群)中相互分化。我们在进化支中发现了两个重复的十字花科单子叶(一个在香蕉分化之前,一个在香蕉分化之后)。
在下一阶段,对基因网络的定量组成进行了评估(附加文件)2).在大多数既有毛状体又有根毛的开花植物中,基因的数量从120到170不等。在最近进行全基因组复制的物种(最近10 MYA)中,有超过190个基因(芸苔属植物拉伯,大豆,Gossypium raimondii就,马吕斯有明显,木薯耐,杨树trichocarpa).在甜菜属在美国,我们发现了相对较少的基因——95个。裸子植物的典型代表,其表皮组织的分化和根毛的存在被显示出来,挪威云杉在研究中有150个与GRN节点同源的基因。
低等植物的代表携带着各种各样的分枝,约有100个与GRN节点相对应的基因(Amborella trichopoda- 108;Physcomitrella金属盘- 119)。甚至是解剖学上更简单的生物体卷柏moellendorffii而且Marchantia polymorpha分别有57和48个基因,和单细胞莱茵衣单胞菌lucimarinus-分别有19个和22个基因。
为了更详细地评估GRN的定量组成对形态发生复杂性的依赖性,应用了主成分分析。
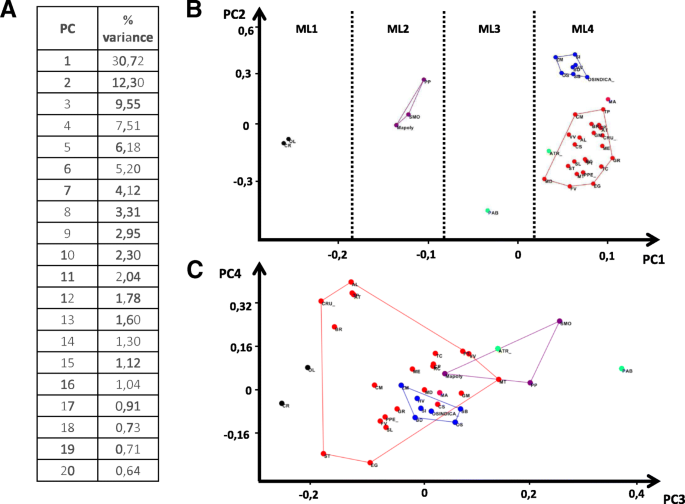
数字4C表示前两个主成分的物种散射,它们描述了43%的方差(分别为30.7和12.3%)。可以注意到,根据形态发生的复杂性的增加,植物分类单元沿着第一个成分排列(图。4ML1-ML4)。
基因网络定量组成的主成分分析与形态发生复杂性相对应。给定各组成部分对应的方差百分比(一个)和1-2 (B)和3-4 (C)分量的散点图。点代表物种,标签根据附加文件2.形态发生复杂性组(b)。MC1 -没有任何组织的单细胞植物,MC2 -有一些组织的多细胞植物。表皮生长表现为根状,在某些情况下为乳头状,MC3 -(裸子植物)多细胞植物,具有不同的组织类型。表皮组织包含多种细胞类型。他们有一个模式的根毛,MC4 -(开花植物)多细胞植物与不同的组织类型。表皮组织包含多种细胞类型。他们有一个模式的根毛和广泛的毛状体类型
因此,第一个组成部分区分形态发生模式的一般并发症。沿着这一组分,发现了多种同源基因的代表,包括MBW毛状体起始复合物的MYB组分(AT4G38620 (MYB4)、AT2G16720、AT4G34990 (MYB7))、毛状体起始主基因AT1G79840 (GL2)和激素信号转导相关基因片段(AT1G66350 (RGL1);AT3G03450 (RGL2);AT5G17490 (RGL3);AT2G01570 (RGA1);AT1G14920(盖);AT2G27300 (ANAC040);AT4G24210 (SLY1))和细胞骨架结构(AT3G50530 (CRK);AT5G28646 (WVD2);AT5G43900 (MYA2); AT1G19835 (FPP4); AT2G35110 (GRL); AT2G46225 (ABIL1); AT5G42030 (ABIL4); AT5G24310 (ABIL3); AT4G01710 (CRK); AT5G65274; AT5G42080 (DL1)). We detected also 2 genes related to cell cycle dynamics (AT2G42260 (UVI4); AT4G15415) and 5 specific trichome differentiation genes (AT1G64690 (BLT); AT1G69490 (NAP); AT1G05230 (HDG2); AT4G21750 (ATML1); AT4G04890 (PDF2)).
第二部分不允许这样一个明确的生物学解释,但它清楚地区分了双子叶和单子叶的进化谱系。沿着这一组分,发现了各种同源基因的某些代表。这些是MBW毛状体起始复合物的抑制剂)。已知启动复合物的抑制剂在毛状体形态的形成中起关键作用拟南芥[6,7,8,9,10,11,12,13].这反映了单子叶和双子叶外部生长的不同模式复杂性。双褶子中更多的MBW抑制剂很可能会增加双褶子中不同器官上形成二维图案的自由度。单子叶有叶表皮(像根一样),呈细胞行或文件的形式。也许,这种图案的相似性质使得使用不那么灵活的分子系统来形成它成为可能。另一种解释是,在单子叶植物中存在大量具有结构域组成变化的TTG1直系同源物,它们可以以类似于一组抑制剂的方式起作用。
沿着这一组成部分的数量的各种同源基因的代表被发现。其中值得注意的是3个基因- MBW毛体起始复合物的不同组成部分(AT4G00480 (ATMYC1), AT4G09820 (TT8), AT5G41315(GL3)), 9个激素信号转导相关基因(AT2G46600;AT5G27320 (GID1C);AT3G05120 (GID1A);AT3G63010 (GID1B);AT4G02570 (CUL1);AT4G20780 (CML42);AT5G06650 (GIS2), AT2G41940 (ZFP8), AT3G58070 (GIS)), 7个与细胞骨架结构和动力学相关的基因(AT1G29170 (WAVE2), AT2G34150 (ATRANGAP2), AT2G38440 (SCAR2);AT1G80350 (ERH3);AT3G16630 (KINESIN-13A); AT5G20490 (XIK), AT1G17580 (MYA1)) and 4 specific trichome differentiation genes (AT1G01520 (ASG4); AT1G33240 (GTL1); AT2G02480 (STI); AT4G12610 (RAP74)).
数字4B表示物种对第三和第四个主成分的散射,两个主成分描述了17%的方差(分别为9.5和7.5%)。第三种成分分离裸子植物、开花植物和藻类。第四个组成部分不区分开花植物和藻类。最大的PC4值有十字花科,茄属植物lycopersicum而且桉树茅.然而,这些数据的生物学解释是困难的。
特异性毛状体分化基因(AT1G6949、AT1G01510)、激素信号转导相关基因(AT2G46600、AT2G39940、AT2G39940)和细胞骨架结构相关基因(AT5G43900)的数量对第三成分的影响最大。对第三组分影响最大的有含有MBW毛状体起始复合物的基因数量(AT1G01380、AT2G30420、AT2G30424、AT2G30432、AT2G46410、AT4G01060、AT5G53200)、激素信号转导相关基因(AT2G27300、AT2G27300)、细胞周期动力学相关基因(AT5G45190、AT4G19600)、细胞骨架结构相关基因(AT2G46225、AT5G42030、AT5G24310)、特异性毛状体分化基因(AT1G05230、AT4G21750、AT4G04890、AT1G79840)。
由此可见,MBW毛状体起始复合体主要成分的同源基因在双子叶植物和单子叶植物中均存在,表明所研究的基因网络在开花植物的共同祖先中已经存在。因此,关于毛状体形成的独特机制的假说答:芥鉴于现代数据需要修订[30.].在裸子植物和更早的共同祖先的水平上检测到复合物组分的特征域结构(图2)。2,其他文件3.,4,5而且6).这使我们可以推断MBW复合体是一个相对古老的结构。表皮细胞的表皮生长是广泛的,也是古老的形成。在藻类中发现了简单的生长产物- Chara (Charophytales),水绵(双星藻目) [36].苔藓中的根茎有其特有的形态,在基质中起固定作用,参与水分和养分的吸收[37].据透露,Physcomitrella金属盘基因PpRSL1和PpRSL2影响植物的根茎数量[38,39].突变体的拟南芥由于缺乏RHD6(头发发育的关键基因之一)的功能,如果它们被PpRSL1基因转化,就会产生根毛Physcomitrella.这表明在4.2亿年的物种分化过程中,RSL家族蛋白的功能并没有丧失[38].同时,在双子系和单子系分化之前,HLH基因发生了大量的重复。这与水稻R3 MYB转录因子OsTCL1在水稻中异位表达的信息相对应拟南芥基因组影响毛状体的形成[31].同时,水稻中OsTCL1表达的变化不会导致任何与三毛体相关的表型变化,这表明这些物种中相应grn的操作之间存在重要的功能差异[31].在十字花科,我们观察到含有HLH结构域和一些MYB因子的蛋白质中取代积累的加速(附加文件)4,5而且6).与此同时,我们在水稻中观察到TTG1的一系列重复事件和结构域重排(图1)。2d,(其他文件3.,4,5而且6).因此,在谷物植物中HLH调控的分子水平上,GRN复合物具有更多的自由度,可以潜在地执行更广泛的特定功能。
蛋白质的结构和功能演化细节可以通过三维结构构建和研究直接解决,也可以通过基于蛋白质相似性的简单结构残基类型预测间接解决。最后,我们选择后一种方法来分析所研究蛋白质的结构进化。这一选择在计算上大大降低了成本,使我们能够证明植物蛋白在MBW复合物中至少存在两种相反的进化趋势:一种是改变蛋白表面(TTG1和EGL3),使蛋白球的内部结构保持保守;另一种是改变蛋白球的内部结构(CPC和GLABRA),主要是通过优化无序区域。
需要注意的是,MBW复合体与其调控因子直接参与根毛形态发生的抑制[7,9,10,32].因此,有理由认为一个基因网络的变异是叶表皮和根毛的毛状体模式形成的原因答:芥[33].通过RNA-seq数据,Huang表明,负责根毛的主要基因组在进化距离中保存了长达2亿年或更长时间[34].然而,这些基因的表达模式在不同物种之间差异很大[35].使用主成分法,我们建立了18个主要的骨科组(GRN中的30个独立节点)答:芥),对应于进化方面的表皮形态发生复杂性。
结论
启动复合体的主要参与者(基因)是古老的,可能在所有维管植物的祖先中具有类似的功能,以形成简单的一维模式。在各种进化系中都有重复和基因丢失的现象。鉴定了单子叶植物和双子叶植物的各种趋势。具有更复杂模式的生物的基因网络经历了大量的单个基因复制事件,这些事件可能在复杂模式的形成中发挥了作用。然而,单子叶和双子叶模式在复杂性上是由相同的基因网络形成的。
在发育的基因网络中,下列功能块以毛状体(激素反应调节因子、启动复合物及其抑制剂、细胞骨架基因、细胞周期基因等)区分;
所有维管植物的祖先已经具备了所有这些元素,但缺乏这些元素;
在广泛的物种中,预测了负责毛状体发育的候选基因的数量。
方法
入库单重建
根据GeneOntology对GO相关术语的分析[68], tair [69], PLAZA数据库[70], 90个与毛状体形成相关的基因答:芥发现(毛状体形态的负调控,毛状体分支的调控,毛状体形态发生的调控,毛状体形态发生的调控,毛状体分化的调控,毛状体形态的调控)。富集的基因/蛋白质集是基于Cytoscape数据库中托管的蛋白质-蛋白质相互作用数据获得的。基因及其相互作用的初步列表通过额外的相互作用(调节,蛋白质-蛋白质等)而丰富起来,这些相互作用使用了存储调节和其他(STRING [71],细胞膜[72]和Andsystem [73])。该列表包括100个基因(表1)。此外,我们使用GeneMania数据集检查基因与基因网络的相互关系,并丰富基因集。另外,从STRING数据库中添加基因,并使用Cytoscape程序(GeneMania插件)。与我们的样本有最高的连接得分的基因被选择。考虑到功能注释、同行评审文章中的提及和基因网络中的连通性,对基因进行了专家士绅化。
序列数据库和系统发育分析
为了阐明三毛体相关基因网络的进化途径,我们在PLAZA 3.0中研究了全测序基因组中所有同源物之间的系统发育关系(http://bioinformatics.psb.ugent.be/plaza/[70].爆炸声+ [74,75],我们进行了序列检索,得到了与拟南芥GRN成分具有显著相似性(E值<1e-5)的序列列表。利用搜索的倒数爆破,得到了最完整的同源组。蛋白质结构域的鉴定使用包HMMER v.3的hmmsearch程序进行(http://hmmer.org/) [76]和隐马尔可夫模型(隐马尔可夫模型- HMM)从pfam数据库(https://pfam.xfam.org/)使用阈值e-value = 1e-7。不包含与查询相对应的任何结构域的蛋白质被排除在分析之外。使用mafft 7对蛋白质进行多次比对[77参数"——add " "——auto "和"——keeplength "。使用Python编写的内部脚本自动清除无信息站点(超过80%的蛋白质有间隙的站点)的多个对齐。此外,清洗后在排列中有超过75%间隙的蛋白质从分析中去除。进行了专家评估,以确定结构域中有显著缺失的蛋白质。在找到这种树的情况下,通过构造没有检测到缺陷序列的树来验证拓扑。借助SAMEM v. 0.82管道进行分子进化分析[78].基于多重比对的氨基酸取代模型的构建采用算法Modelestimator [79].FastTree 2.1.1 [80]用于估计主拓扑结构。在先前生成的替代模型的基础上,由Phyml [81]通过优化主树拓扑结构和分支长度。为了测试树分支点的稳定性,我们使用了aLRT程序。在FigTree v.1.4.2程序中进行树可视化和拓扑分析[82],始祖鸟(https://sites.google.com/site/cmzmasek/home/software/archaeopteryx)及ETE工具包[83].在树中,根据拓扑结构和OTU (Operational Taxonomic Units)进行了物种评价,明确了蛋白质功能和结构域组成的信息。估计了同源序列的演化支和相应功能的多样化时间。从进化的角度来看,通过计算各开花植物单系群的序列数量,可以估计开花植物科目分化后出现的重复。
主成分分析结果被纳入程序,以减少数量的测量和形态变量。通过主成分分析(PCA)对不同进化系中不同数量的基因进行排序。为了描述形态变异,我们使用PAST统计程序(版本2)计算主成分[84].在这种情况下,符号是orthologic组中的基因数量,而对象是物种本身。在分析之前,对每个正交组中的基因数进行归一化以消除噪声。每一种的同源体数目在附档中给出2.
深入分析GRN蛋白的进化
为了深入分析GRN组分的进化,我们选择了4个研究对象(蛋白质家族):EGL3, GLABRA, CPC, TTG1。先前由MAFFT构建的CPC蛋白的多蛋白比对,由PROMALS进一步细化。系统发育分析的对准区选择由GBlocks依次完成[85]和手动对齐检查(在核心对齐块中选择缺口丰富的序列)。通过IQTree 1.5.4筛选氨基酸取代率的最佳矩阵(模型)。对于所有四种蛋白比对,JTT + G4被发现是最好的模型。该模型用于初始树拓扑结构的重构。使用TreeFix v1.1.10软件对timmetree DB中的Viridiplantae物种树进行初始蛋白质树拓扑校正,然后使用IQTree 1.5.4和JTT + G4模型对分支长度进行重新优化。
使用PhyloBayes 4.1、CAT进化模型和6个不同类别的站点进化速率,对四棵树的每个内节点的祖先序列进行MCMC系统发育树感知贝叶斯采样。MCMC采样用于完整的和隔离的(使用称为“AltAll*N”的改进方法)祖先库生成。我们的' AltAll*N '过程是在每个内部树节点上迭代重写祖先序列中所有合理的(后视概率> 0.1)替代状态。例如,如果有3替代状态在网站和4替代site B的祖先节点X我们应该重写祖先序列4次获得4替代祖先节点X: A)最好的州组成的一个序列A和B站点,B)与第二序列可能的A和B, c)与第三序列可能的A和B, d)序列和第三个可能状态的A和B的提出可能的状态。
为了找到上位转换特征或进化的“Stokes位移”,我们分析了蛋白质进化与蛋白质特定矩阵中每个蛋白质树分支上氨基酸取代的相对速率的偏差(1),并简单比较了基于蛋白质结构数据的内分支长度计算(2)。
(1)为了比较分支特异性氨基酸替换率与作为相对氨基酸替换率矩阵的全树特异性氨基酸替换率,我们使用了完整的祖先库。为此,我们连续采取了以下步骤:a)重建蛋白质特异性时间可逆的氨基酸替换相对速率模型(模型估计软件),以对所分析的4棵树的现有蛋白质序列进行比对;b)d对于每个内部树节点的每种可能替换的度量计算,d=页一个*页b * 2 *数控,在那里页一个和页B是a和B氨基酸的后验概率,a不等于B,数控= 1/(1 + e的200*射频ab)),射频Ab为蛋白质特异性时间可逆氨基酸取代矩阵中Ab取代的相对速率;C)总结d在每个内树节点上测量所有站点,并计算这些和的自然对数;D)对所有树的对数和进行非参数比较(按百分位数),以识别具有最大对数和的分支(具有上位转换签名的分支)。
(2)为了比较分支结构变化的特定速率,我们使用了隔离的' AltAll*N '祖先库。为此,我们连续采取了以下步骤:a)在每个内部树节点中,使用RaptorX_Property快速管道推导每个备选祖先序列的每个残差的二级结构、表面状态和无序签名[86];B)计算相邻内树节点的所有可选祖先序列之间的二级结构、表面状态和无序特征的变化频率;C)在所有树中对上述变化频率进行非参数比较(按百分位数),以确定结构变化最大的分支。
缩写
- bHLH:
-
基本helix-loop-helix
- 乙炔:
-
锌指c2h2型
- 入库单:
-
基因调控网络
- MBW:
-
一种特殊的蛋白质复合物含有MYB-bHLH-WDR蛋白
- 米娅:
-
百万年前
- OTU:
-
操作分类单位
- PC:
-
主成分
参考文献
Ó’Maoiléidigh DS, Graciet E, Wellmer F.控制拟南芥花发育的基因网络。植物学报。2014;201(1):16-30。
对基因调控网络进化的展望。植物学报,2017;33(7):436-47。
杨超,叶哲。植物细胞分化研究的毛状体模型。细胞生物学与生物工程学报,2013;29(11):344 - 344。
杜兰,李志强,李志强,等。植物中介体结构与功能的保护与分化。中国生物医学工程学报,2017;29(1):344 - 344。
赵m, Morohashi K, Hatlestad G, Grotewold E, Lloyd A. TTG1-bHLH-MYB复合物通过直接靶向调控位点控制毛状体细胞命运和模式。发展。2008;135(11):1991 - 9。
Schnittger A, Folkers U, Schwab B, Jürgens G, Hülskamp M.拟南芥间隔模式的生成:TRIPTYCHON在拟南芥毛状体模式中的作用。植物学报。1999;11(6):1105-16。
Schellmann S, Schnittger A, Kirik V, Wada T, Okada K, Beermann A, Thumfahrt J, Jürgens G, Hülskamp M. TRIPTYCHON和CAPRICE介导拟南芥毛状体和根毛形态的横向抑制。中国生物医学工程学报。2002;21(19):5036-46。
拟南芥表皮细胞分化的Myb同源物分析。中国科学(英文版)。1997;277(5329):1113-6。
Kirik V, Simon M, Huelskamp M, Schiefelbein J. TRY AND CPC1基因增强子与tritychon和CAPRICE在拟南芥毛状体和根毛细胞模式中的冗余作用。中国生物医学工程学报,2004;26(2):339 - 339。
Kirik V, Simon M, Wester K, Schiefelbein J, Hulskamp M.拟南芥试和CPC 2 (ETC2)的增强子揭示了拟南芥毛状体发育的区域特异性控制中的冗余。植物分子生物学杂志,2004;55(3):389-98。
Wester K, Digiuni S, Geier F, Timmer J, Fleck C, Hülskamp M. R3单重复基因在毛状体发育中的功能多样性。发展。2009;136(9):1487 - 96。
王松,郭绍升,曾强,陈晓霞,陈建国,陈建国。拟南芥TRICHOMELESS1通过抑制GLABRA1调控毛状体模式。发展。2007;134(21):3873 - 82。
甘兰,夏凯,陈建国,王舒。拟南芥毛状体模式调控的新单重复R3 MYB转录因子TRICHOMELESS2的功能特征。植物学报。2011;11(1):176。
王松,Hubbard L,常勇,郭洁,Schiefelbein J,陈建国。拟南芥表皮细胞模式中单重复序列R3 MYB蛋白的综合分析及其转录调控。植物学报。2008;8(1):81。
Morohashi K, Zhao M, Yang M, Read B, Lloyd A, Lamb R, Grotewold E.拟南芥bHLH因子GL3参与毛状体启动调控事件的研究。植物科学进展。2007;32(3):344 - 344。
Morohashi K, Grotewold E.一种系统方法揭示了由GL3和GL1选择子启动拟南芥毛状体的调节电路。公共科学学报,2009;5(2):e1000396。
Brown AI, Rutenberg AD。蓝藻中基于存储的杂囊承诺和模式模型。物理学报,2014;11(1):016001。
Corrales-Guerrero L, Mariscal V, Flores E, Herrero A.杂囊分化PatS形态发生的功能解剖和细胞间转移的证据。中国生物医学工程学报,2013;29(6):1093-105。
安玲,周志,苏松,闫安,甘艳。拟南芥毛状体通过赤霉素信号通路进行分枝需要无毛花序茎(GIS)。植物生理学报。2011;32(2):457-69。
甘y, Kumimoto R,刘c, Ratcliffe O, Yu H, Broun P.无毛花序茎对拟南芥表皮分化和芽成熟的调控。植物学报。2006;18(6):1383-95。
甘艳,刘超,于红,Broun P.拟南芥转录因子GIS、ZFP8和GIS2整合细胞分裂素和赤霉素信号在表皮细胞命运调控中的作用。发展。2007;134(11):2073 - 81。
周忠,安磊,孙玲,朱松,奚伟,Broun P,余华,甘勇。锌指蛋白5 (Zinc Finger Protein 5, ZFP5)通过作用于ZFP8的上游来控制毛状体形成拟南芥.植物杂志。2011, 157(2): 673 - 82。
周震,安磊,孙玲,甘艳。ZFP5编码一种功能等效的GIS蛋白控制毛状体形成。植物信号行为研究。2012;7(1):28-30。
周泽,孙玲,赵艳,安玲,闫安,孟X,甘艳。锌指蛋白6 (ZFP6)通过整合赤霉素和细胞分裂素信号调控拟南芥毛状体启动。环境科学学报,2013;32(3):379 - 379。
李艳,单霞,高瑞,杨松,王松,高霞,王磊。小兰IIIf分支中的两个bhlhs在拟南芥中异位表达时,在类黄酮生物合成和毛状体形成中发挥不同的作用。科学报告2016;6:30514。
Jaffé FW, Tattersall A, Glover BJ。一种截断的MYB转录因子调节表皮细胞生长。应用物理学报,2007;58(6):1515-24。
濮玲,李强,范霞,杨伟,薛艳。R2R3 MYB转录因子GhMYB109是棉纤维发育所必需的。遗传学。2008;180(2):811 - 20。
关旭,余娜,上官霞,王松,陆松,王玲,陈霞。拟南芥毛状体研究揭示棉纤维发育机制。科学通报,2007;52(13):1734-41。
关晓阳,李庆军,单春明,王松,毛一斌,王丽娟,陈晓阳。来自棉花的HD-zip IV基因GaHOX1是拟南芥GLABRA2的功能同源物。中国生物医学工程学报。2008;29(1):344 - 344。
瑟娜L,马丁C.特里霍姆斯:不同的调控网络导致收敛结构。植物科学进展,2006;11(6):274-80。
郑凯,田红,胡青,郭红,杨玲,蔡林,王霞,刘波,王松。拟南芥R3 MYB转录因子OsTCL1基因异位表达影响毛状体和根毛形成,水稻不影响。科学代表2016;6:19254。
Salazar-Henao JE, Vélez-Bermúdez IC, Schmidt W.根毛图案和形态发生的调控和可塑性。发展。2016;143(11):1848 - 58。
琼斯VA,多兰L.根毛和根茎的进化。学报学报。2012;11(2):205-12。
黄玲,史晓霞,王伟,刘克勤,等。维管植物根毛发育基因的多样性。植物杂志。2017, 174(3): 1697 - 712。
刘志强,李志强,李志强,等。表皮顶端生长细胞的生长调控。植物学报。2016;34:77-83。
Lewis LA, McCourt RM。绿藻和陆生植物的起源。中国科学(d辑),2004;29(4):357 - 357。
Sakakibara K, Nishiyama T, Sumikawa N, Kofuji R, Murata T, Hasebe M.生长素和同源结构域亮氨酸拉链I基因在小立穗藓(Physcomitrella patens)根茎发育中的参与。发展。2003;130(20):4835 - 46所示。
梅兰B,伊K, Jouannic S, Hoffmann L, Ryan E, Linstead P, Schaefer DG, Dolan L.一种古老的机制控制着陆生植物具有生根功能的细胞发育。科学。2007;316(5830):1477 - 80。
Jang G, Yi K, Pires ND, Menand B, Dolan L. RSL基因对早期分化陆生植物的根茎系统发育是充分的。发展。2011;138(11):2273 - 81。
徐娟,张娟。为什么人类疾病相关残基在其他物种中以野生型出现:补偿假说的基因组尺度结构证据。分子生物学杂志,2014;31(7):1787-92。
乌斯马诺娃DR,法拉蒂L,波沃洛茨卡娅IS,弗拉索夫PK,康德拉绍夫FA。序列空间和长期蛋白质进化的替代轨迹模型。分子生物学杂志,2014;32(2):542-54。
Morcos F, Pagnani A, Lunt B, Bertolino A, Marks DS, Sander C, Zecchina R, Onuchic JN, Hwa T,权重M.残基协同进化的直接耦合分析捕获了许多蛋白质家族的原生接触。中国生物工程学报,2011;29(3):344 - 344。
Ovchinnikov S, Kamisetty H, Baker D.利用进化信息对蛋白质界面上的残基-残基相互作用进行稳健和准确的预测。elife。2014; 3: e02030。
邬晓峰,陈晓峰,陈晓峰,陈晓峰,陈晓峰,陈晓峰。基于进化信息的蛋白质结构研究进展。elife。2015; 4: e09248。
斯塔尔TN,桑顿JW。蛋白质进化中的上位现象。中国生物工程学报,2016;25(7):1204-18。
郭德华,李志强,李志强。氨基酸的绝对取代率与折叠的序列熵。自然生态与进化。2017; 1(12): 1923。
Joy JB,梁rh, McCloskey RM, Nguyen T, Poon AF.祖先重建。中国生物医学工程学报。2016;12(7):e1004763。
王晓明,王晓明。祖先蛋白重建技术及其应用。生物化学,2016;397(1):1 - 21。
Arenas M, Weber CC, Liberles DA, Bastolla U. ProtASR:祖先蛋白质重建与折叠稳定性选择的进化框架。中国生物医学工程学报,2017;26(6):1054-64。
用祖先序列重建探索蛋白质进化的过去和未来:蛋白质工程的“复古”方法。生物化学学报,2017;29(1):1 - 9。
Randall RN, Radford CE, Roof KA, Natarajan DK, Gaucher EA.祖先序列重建的实验系统发育。Nat Commun. 2016;7:12847。
Anderson DP, Whitney DS, Hanson-Smith V, Woznica, campodon尼科- burnett W, Volkman BF, King N, Thornton JW, Prehoda KE。动物中有组织多细胞的一种古老蛋白质功能的进化。elife。2016; 5: e10147。
Eick GN, Bridgham JT, Anderson DP, Harms MJ, Thornton JW。重建祖先蛋白函数对统计不确定性的鲁棒性。中国生物医学杂志,2017;34(2):247-61。
Maes L, Inzé D, Goossens A.透明TESTA GLABRA1网络的功能特化允许不同激素控制拟南芥莲座丛叶片的层状和边缘毛状体起始。植物科学进展。2008;32(3):344 - 344。
西蒙兹VV,哈特斯塔德G,劳埃德AM。自然等位基因变异定义了ATMYC1的作用:毛状体细胞命运的决定。公共科学图书馆,2011;7(6):e1002069。
赵红,王霞,朱东,崔松,李霞,曹勇,马磊。拟南芥基本螺旋-环-螺旋转录因子AtMYC1 IIIf亚家族的单氨基酸取代通过取消其与伙伴蛋白的相互作用导致毛状体和根毛模式缺陷。中国生物医学工程学报,2012;
张峰,张立军,张立军。GL3编码bHLH蛋白,通过与GL1和TTG1相互作用调节拟南芥的毛状体发育。遗传学。2000;156(3):1349 - 62。
张峰,赵敏,张志刚,张志刚,张志刚。拟南芥ttg1依赖通路中冗余bHLH蛋白的表达。发展。2003;130(20):4859 - 69。
Kirik V, Lee MM, Wester K, Herrmann U, Zheng Z, Oppenheimer D, Schiefelbein J, Hulskamp M. MYB23和GL1基因在毛状体形态发生和启动中的功能多样化。发展。2005;132(7):1477 - 85。
李文杰,李志刚,李志刚。WD重复:不同功能的通用架构。生物化学,1999;24(5):181-5。
Dubos C, Stracke R, Grotewold E, Weisshaar B, Martin C, Lepiniec L.拟南芥MYB转录因子研究。植物科学进展,2010;15(10):573-81。
Kirik V, Schnittger A, Radchuk V, Adler K, Hülskamp M, Bäumlein H.拟南芥AtMYB23基因异位表达诱导毛状体细胞分化。生物工程学报。2001;35(2):366-77。
李顺丰,Milliken ON, Pham H, Seyit R, Napoli R, Preston J, Koltunow AM, Parish RW。拟南芥MYB5转录因子调节粘液合成、种皮发育和毛状体形态发生。植物学报。2009;21(1):72-89。
梁刚,何辉,李勇,艾强,于东。MYB82在拟南芥毛状体发育调控中的作用。中国机械工程学报,2014;26(3):344 - 344。
Tominaga-Wada R, Nukumizu Y, Sato S, Kato T, Tabata S, Wada T.拟南芥myb相关基因wolf和AtMYB23的功能分化。中国生物工程学报。2012;26(5):344 - 344。
赵敏,李伟,李志刚。拟南芥幼苗TTG1/bHLH/Myb转录复合物对花青素生物合成途径的调控植物学报,2008;36(5):344 - 344。
戚涛,宋松,任强,吴东,黄红,陈艳,范敏,彭伟,任超,谢东。茉莉酸- zimm结构域蛋白与WD-Repeat/bHLH/MYB复合物相互作用调控茉莉酸介导的花青素积累和毛状体启动拟南芥.植物科学进展。2011;23(5):1795-814。
基因本体联盟。基因本体论联盟:前进。中国生物医学工程学报,2014;43(D1): D1049-56。
Lamesch P, Berardini TZ, Li D, Swarbreck D, Wilks C, Sasidharan R, Muller R, Dreher K, Alexander DL, Garcia-Hernandez M, Karthikeyan AS。拟南芥信息资源:改进的基因注释和新工具。核酸研究,2011;40(D1): D1202-10。
Proost S, Van Bel M, Vaneechoutte D, Vande Peer Y, Inzé D, muller - roeber B, Vandepoele K. PLAZA 3.0:植物比较基因组学的访问点。中国生物医学工程学报,2014;43(D1): D974-81。
Szklarczyk D, Morris JH, Cook H, Kuhn M, Wyder S, Simonovic M, Santos A, Doncheva NT, Roth A, Bork P, Jensen LJ。2017年的STRING数据库:质量控制的蛋白质-蛋白质关联网络,广泛可访问。Nucleic acids res. 2016;45(D1): D362-68。
王俊杰,王俊杰,王俊杰,等。细胞景观:生物分子相互作用网络集成模型的软件环境。基因组学报,2003;13(11):2498-504。
Ivanisenko VA, Saik OV, Ivanisenko NV, Tiys ES, Ivanisenko TV, Demenkov PS, Kolchanov NA。ANDSystem:一个用于生物学领域自动文献挖掘的关联网络发现系统。中国生物医学工程学报,2015;9(2):S2。
阿特舒尔SF,吉什W,米勒W,迈尔斯EW,李普曼DJ。基本的本地对齐搜索工具。中华分子生物学杂志,1990;215(3):403-10。
卡马乔C, Coulouris G, Avagyan V, Ma N, Papadopoulos J, Bealer K, Madden TL. BLAST+:结构与应用。BMC生物信息学。2009; 10(1): 421。
Mistry J, Finn RD, Eddy SR, Bateman A, Punta M.同源性搜索的挑战:HMMER3和卷曲线圈区域的收敛演化。核酸研究,2013;41(12):e121。
Katoh K, Standley DM. MAFFT多序列比对软件版本7:性能和可用性的改进。中国生物医学杂志,2013;30(4):772-80。
Gunbin KV, Suslov VV, Genaev MA, Afonnikov DA。分子进化模式分析计算机系统(SAMEM):分析系统发生树深层内部分支的分子进化模式。在硅生物中。2012; 11(3, 4): 109 - 23所示。
估计氨基酸替代率的有效方法。中华生物医学杂志,2006;26(6):663-73。
Price MN, Dehal PS, Arkin AP. FastTree 2 -用于大对齐的近似最大似然树。公共科学学报,2010;5(3):e9490。
Guindon S, Dufayard JF, Lefort V, Anisimova M, Hordijk W, Gascuel O.估计最大似然系统发生的新算法和方法:评估PhyML 3.0的性能。中国生物医学工程学报。2010;59(3):307-21。
Rambaut A, Drummond A. FigTree:树图形绘制工具,版本1.2.2。爱丁堡大学进化生物学研究所,2008。http://tree.bio.ed.ac.uk/software/figtree/.
胡尔塔- cepas J, Serra F, Bork P. ETE 3:系统基因组数据的重建、分析和可视化。中国生物医学杂志,2016;33(6):1635-8。
汉默Ø,哈珀检察官,瑞安警察。古生物统计软件:教育和数据分析软件包。古生物电子,2001;4:1-9。
从蛋白质序列比对中去除分歧和不明确的排列块后的系统发育的改善。中国生物医学工程学报。2007;36(4):564-77。
马军,王松,赵峰,徐娟。基于上下文特异性对齐势的蛋白质穿线。生物信息学)。2013;29 (13):i257 - 65。
确认
这项工作和出版费用由俄罗斯科学基金会资助(RSF资助18-14-00293,蛋白质序列分析和系统发育重建,基因网络分析)。计算使用国家预算项目0324-2018-0017支持的生物信息学共享访问中心和新西伯利亚州立大学高性能计算中心的设备进行。
资金
研究工作和出版费用由俄罗斯科学基金会资助(RSF资助18-14-00293,蛋白质序列分析和系统发育重建,基因网络分析)。
数据和材料的可用性
所有支持数据可在附加文件。
关于这个补充剂
本文已作为BMC植物生物学第19卷增刊1,2018:摘自BGRS\SB-2018:植物生物学。该补充的全部内容可在网上查阅//www.cinefiend.com/articles/supplements/volume-19-supplement-1.
作者信息
从属关系
贡献
所有作者对这项研究的贡献相同。所有作者都阅读并认可了最终的手稿。作者声明没有利益冲突。
相应的作者
道德声明
伦理批准并同意参与
不适用
发表同意书
不适用
相互竞争的利益
作者宣称他们之间没有利益冲突。
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
附加文件
附加文件1:
表S1:表包含以下表:GRN_statistics -这个表包含网络中节点和边的数量的信息。go_enrichment -该表包含了关于网络中所有节点的GO富集信息(根据AgriGO,上诉日期为11/07/2018)。GO_terms_associated_GRN_nodes -该表包含了网络中分配给每个节点的术语信息(根据TAIR,上诉日期为11/07/2018)。GO术语的GRN集群-这张表包含关于哪个术语属于基因网络的哪个集群的信息。(xlsx64kb)
附加文件2:
表S2:表中包含以下表格:同源基因数量-此表格包含物种研究中基因网络中每个基因的同源基因数量的信息。GRN节点的进化特征-这张表包含了基因网络中每个基因的不同进化系的重复事件的信息。(xlsx26 kb)
附加文件3:
图S1: A。代表性植物EGL3 (AT1G63650)、GL3 (AT5G41315)、MYC1 (AT4G00480)和TT8 (AT4G09820)同源物的PhyML系统发育关系和结构域组成。B。EGL3 (AT1G63650)、GL3 (AT5G41315)、MYC1 (AT4G00480)和TT8 (AT4G09820)同源蛋白的蛋白质结构进化变化从右至左依次为:无序(2种残基型)、二级结构(3种残基型)、二级结构(8种)、球表面(3种残基型)、罕见(与蛋白质特异性模型相比)氨基酸取代。配色方案:黑色-外部分支(未分析);颜色定义分支长度四分位数:蓝色- Q1;green - Q2;橙色第三季度;红色的第四季度。(PDF 502kb)
附加文件4:
图S2: A。代表性植物TTG1 (AT5G24520)同源物的PhyML系统发育关系及结构域组成B。所研究的TTG1 (AT5G24520)同源蛋白的蛋白质结构进化变化从右至左依次为:无序(2种残基型)、二级结构(3种残基型)、二级结构(8种)、球表面(3种残基型)、罕见(与蛋白质特异性模型相比)氨基酸取代。配色方案:黑色-外支(未分析);颜色定义分支长度四分位数:蓝色- Q1;green - Q2;橙色第三季度;红色的第四季度。(PDF 406kb)
附加文件5:
图S3: A。R2R3-MYB的PhyML系统发育关系及结构域组成(AT1G66370;AT1G566650;AT1G66390;AT1G66380;AT5G35550;AT5G14750;AT5G40330;AT3G27920;AT5G52600)代表性植物物种的同源。B。R2R3-MYB蛋白结构的进化变化(AT1G66370;AT1G566650;AT1G66390;AT1G66380;AT5G35550;AT5G14750;AT5G40330;AT3G27920;AT5G52600)被研究的同源蛋白从右至左分支反映:无序(2种残基类型)、二级结构(3种)、二级结构(8种)、球表面(3种残基类型)、罕见(与蛋白质特异性模型相比)氨基酸取代。配色方案:黑色-外支(未分析); colours define branch lengths quartile: blue – Q1; green – Q2; orange –Q3; red –Q4. (PDF 5149 kb)
附加文件6:
图S4: A。代表性植物R3-MYB同源物的PhyML系统发育关系及结构域组成。B。所研究的R3-MYB同源蛋白的蛋白质结构进化变化从右至左依次为:无序(2种残基型)、二级结构(3种残基型)、二级结构(8种残基型)、球表面(3种残基型)、罕见(与蛋白质特异性模型相比)氨基酸取代。配色方案:黑色-外支(未分析);颜色定义分支长度四分位数:蓝色- Q1;green - Q2;橙色第三季度;红色的第四季度。(PDF 385kb)
附加文件7:
图S5:代表性植物AT1G01510同源物的PhyML系统发育关系及结构域组成。图S6:代表性植物AT1G01520同源物的PhyML系统发育关系及结构域组成。图七:代表性植物AT1G03060同源物的PhyML系统发育关系及结构域组成。图S8:AT1G05230的PhyML系统发育关系及结构域组成AT4G21750;AT4G04890;AT1G79840代表植物的同源物。图S9: AT1G67030的PhyML系统发育关系及结构域组成;AT3G58070;AT2G41940;AT5G06650;AT1G10480;AT1G68360代表植物的同源物。图S10:代表性植物AT1G13180同源物的PhyML系统发育关系及结构域组成。图S11AT1G66350的PhyML系统发育关系及结构域组成;AT5G17490;AT3G03450;AT2G01570;AT1G14920代表植物的同源物。图S12AT1G17920的PhyML系统发育关系及结构域组成;代表性植物的AT1G73360同源物。图向:代表性植物AT1G19835同源体的PhyML系统发育关系及结构域组成。图S14系列:代表性植物AT1G25540同源物的PhyML系统发育关系及结构域组成。(PDF 10138kb)
附加文件8:
图S15AT1G29170的PhyML系统发育关系及结构域组成;AT2G34150;AT2G38440代表植物的同源物。图S16:AT2G33385的PhyML系统发育关系及结构域组成AT1G30825代表植物的同源物。图肌力表现:代表性植物AT1G33240同源物的PhyML系统发育关系及结构域组成。S18图美国:代表性植物AT1G60430同源物的PhyML系统发育关系及结构域组成。图S19:代表性植物AT1G64690同源物的PhyML系统发育关系及结构域组成。图S20:代表性植物AT1G65470同源物的PhyML系统发育关系及结构域组成。图S21: AT5G38110的PhyML系统发育关系及结构域组成;AT1G66740代表植物的同源物。图S22:代表性植物AT1G69490同源物的PhyML系统发育关系及结构域组成。图S23:代表性植物AT1G75950同源物的PhyML系统发育关系及结构域组成。图S24:代表性植物AT1G80350同源物的PhyML系统发育关系及结构域组成。(PDF 798kb)
附加文件9;
图S25:代表性植物AT2G02480同源物的PhyML系统发育关系及结构域组成。图S26:代表性植物AT2G22640同源物的PhyML系统发育关系及结构域组成。图S27:代表性植物AT2G27300同源物的PhyML系统发育关系及结构域组成。图S28:代表性植物AT2G27600同源物的PhyML系统发育关系及结构域组成。图S29:代表性植物AT2G31300同源物的PhyML系统发育关系及结构域组成。图S30:代表性植物AT2G33540同源物的PhyML系统发育关系及结构域组成。图S31:代表性植物AT2G35110同源物的PhyML系统发育关系及结构域组成。图S32:代表性植物AT2G39940同源物的PhyML系统发育关系及结构域组成。图S33:代表性植物AT2G42260同源物的PhyML系统发育关系及结构域组成。图S34AT2G46225的PhyML系统发育关系及结构域组成;AT5G42030;AT5G24310代表植物的同源物。(PDF 6990kb)
附加文件10:
图S35:代表性植物AT2G46600同源物的PhyML系统发育关系及结构域组成。图S36:代表性植物AT3G12280同源物的PhyML系统发育关系及结构域组成。图S37:代表性植物AT3G12400同源物的PhyML系统发育关系及结构域组成。图S38:代表性植物AT3G16630同源物的PhyML系统发育关系及结构域组成。图S39:代表性植物AT3G27000同源物的PhyML系统发育关系及结构域组成。图S40:代表性植物AT3G50530同源物的PhyML系统发育关系及结构域组成。图S41AT5G27320的PhyML系统发育关系及结构域组成;AT3G05120;AT3G63010代表植物的同源物。图S42:AT4G01710的PhyML系统发育关系及结构域组成AT5G65274代表植物的同源物。图S43:代表性植物AT4G02570同源物的PhyML系统发育关系及结构域组成。图S44:代表性植物AT4G12610同源物的PhyML系统发育关系及结构域组成。(PDF 7692 kb)
附加文件11:
图S45:代表性植物AT4G14147同源物的PhyML系统发育关系及结构域组成。图S46:代表性植物AT4G15415同源物的PhyML系统发育关系及结构域组成。图S47:代表性植物AT4G20780同源物的PhyML系统发育关系及结构域组成。图S48:代表性植物AT4G22910同源物的PhyML系统发育关系及结构域组成。图S49:代表性植物AT4G24210同源物的PhyML系统发育关系及结构域组成。图S50:代表性植物AT4G38600同源物的PhyML系统发育关系及结构域组成。图S51:代表性植物AT4G04470同源物的PhyML系统发育关系及结构域组成。图S52: AT5G06650的PhyML系统发育关系及结构域组成;AT2G41940;AT3G58070代表植物的同源物。图S53:代表性植物AT5G18410同源物的PhyML系统发育关系及结构域组成。图S54AT1G17580的PhyML系统发育关系及结构域组成;AT5G20490代表植物的同源物。(PDF 7225kb)
附加文件12:
图S55:代表性植物AT5G20570同源物的PhyML系统发育关系及结构域组成。图S56: AT2G36010的PhyML系统发育关系及结构域组成;AT1G47870;AT5G22220代表植物的同源物。图S57AT5G24310的PhyML系统发育关系及结构域组成;AT5G42030;AT2G46225代表植物的同源物。图S58:代表性植物AT5G28646同源物的PhyML系统发育关系及结构域组成。图S59:代表性植物AT5G42080同源物的PhyML系统发育关系及结构域组成。图S60:代表性植物AT5G43900同源物的PhyML系统发育关系及结构域组成。图S61AT5G45190的PhyML系统发育关系及结构域组成;AT4G19600代表植物的同源物。图S62:代表性植物AT2G33540同源物的PhyML系统发育关系及结构域组成。图S63:代表性植物AT5G58230同源物的PhyML系统发育关系及结构域组成。图S64:代表性植物AT5G64630同源物的PhyML系统发育关系及结构域组成。图S65车型:代表性植物AT5G65930同源物的PhyML系统发育关系及结构域组成。(PDF 9760kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
多罗什科夫,a。v。康斯坦丁诺夫,d。ket al。基因调控网络的进化控制拟南芥L.毛状体发育。BMC植物生物学19日,53(2019)。https://doi.org/10.1186/s12870-019-1640-2
发表:
DOI:https://doi.org/10.1186/s12870-019-1640-2
关键字
- 叶表皮
- 基因调控网络
- 蛋白质进化
- 基因组合调控
- 毛状体