摘要
背景
NLP转录因子是高等植物硝酸盐诱导基因表达的主要调控因子。NLP转录因子在氨基端有一个硝酸盐信号响应结构域,在中间有一个rwp - rk型dna结合结构域,在羧基端有一个Phox和Bem1 (PB1)结构域。尽管NLP转录因子的PB1结构域在高等植物中介导与硝酸盐诱导基因表达相关的蛋白质-蛋白质相互作用,但其在硝酸盐诱导基因表达中的确切作用尚未被阐明。
结果
与拟南芥转录因子NLP7的PB1结构域的酵母双杂交分析显示,NLP-NLP相互作用需要PB1结构域内的核心氨基酸残基(K867、D909、D911和E913)。与之前推测的不同拟南芥NLP转录因子之间的功能冗余和重叠一致,在不同NLP转录因子的多种组合之间观察到NLP-NLP相互作用。此外,在K867、D909、D911和E913位点存在氨基酸取代的突变型NLP7需要比野生型NLP7更高的表达水平才能恢复硝酸盐反应基因的表达和生长nlp6 nlp7-1双突变体。然而,令人惊讶的是,在原生质体瞬时表达实验中,野生型和突变型NLP7的硝化反应启动子转激活能力相似,这表明PB1结构域对于裸DNA的转录并不需要。
结论
NLP转录因子PB1结构域介导的蛋白-蛋白相互作用是充分诱导靶基因的硝酸盐依赖表达所必需的在足底.NLP转录因子的PB1结构域可能在硝酸盐存在时通过同源和异源寡聚作用于染色体DNA的基因表达。
背景
植物从土壤中以硝酸盐和铵离子的形式获取氮(N),这些氮被同化为氨基酸,然后用于生成含氮的有机分子,如叶绿素、核苷酸和蛋白质。硝酸盐是山地氧化土壤中无机氮的主要形式,而大部分高等植物都生长在山地氧化土壤中,硝酸盐的供应通常是植物生长的限制因素[1].硝酸盐离子作为肥料施用于土壤或由微生物的硝化活动产生,但由于其带负电荷的性质,它们不能长时间停留在土壤颗粒上,因此很容易从土壤中滤出[2].因此,硝酸盐对植物的有效性是波动的,并受到降雨、灌溉和施肥的强烈影响。
植物具有感知硝酸盐和/或氮状态的调节机制,然后调节基因表达,以适应硝酸盐可用性和内部氮需求,微调代谢和生长[3.,4,5,6,7,8].硝酸盐是调控基因表达以优化硝酸盐利用并协调生长与硝酸盐有效性的关键信号分子之一[3.,9,10,11,12].近年来,硝酸盐诱导的高等植物基因表达的分子框架已被阐明。该框架的最新模型表明,硝酸盐信号在III组钙依赖蛋白激酶(包括CPK10、CPK30和CPK32)的帮助下诱导磷酸化和NLP转录因子的激活[13,14,15,16,17].
单个植物物种可拥有多个编码NLP转录因子的基因[17,18,19,20.];例如,拟南芥基因组包含9个NLP基因(NLP1-9).不同NLP转录因子氨基酸序列的高度相似性表明它们在硝酸盐诱导基因表达中具有冗余和重叠的功能,尽管某些NLP转录因子可能具有不同的生理作用。NLP7似乎在拟南芥中起主导作用NLP7导致部分硝酸盐诱导基因表达水平显著降低,降低硝酸盐依赖性生长促进[14,21,22].关于另一个NLP的基因,NLP6就像NLP7最接近,但破坏NLP6单独使用不会造成明显缺陷;然而,突变NLP6结合nlp7-1突变加剧基因表达变化和生长缺陷[23],证明了NLP6和NLP7的冗余作用。另一方面,NLP8调节硝酸盐促进拟南芥种子萌发[24].
利用NLP6-SUPRD转基因拟南芥植物对NLP转录因子的生理功能进行了分析,这种方法绕过了之间冗余造成的问题NLP基因(25,26].NLP6- suprd是NLP6的嵌合阻遏形式,由NLP6融合到转录阻遏结构域组成;它在NLP6-SUPRD植物中高度表达,因此与内源性NLP转录因子竞争。当硝酸盐或硝酸铵是唯一的氮源时,NLP6-SUPRD植株表现出严重的生长抑制[13].此外,在NLP6-SUPRD植物中,大多数硝酸盐诱导基因的表达也受到抑制,这些基因参与了各种细胞过程,包括硝酸盐运输和同化、碳水化合物代谢和激素反应。26].因此,NLP转录因子可能通过作为硝酸盐诱导基因表达的主要调节因子来协调合成代谢和生长。与得到的结果一致nlp7在突变体和NLP6-SUPRD植株中,过表达NLP7可以改善拟南芥的生长,而过表达NLP7也可以改善拟南芥的生长Zm评选NLP6或Zm评选NLP8来自玉米[20.,27],表明NLP转录因子调节植物对氮的利用。最近,也有报道称,在豆科植物中,一些NLP转录因子调节硝酸盐反应基因的表达,以促进硝酸盐的吸收和同化,并调节结瘤[16,17].
NLP转录因子包含三个保守的结构域:氨基末端的硝酸盐信号响应结构域,中间的rwp - rk型dna结合结构域,以及羧基末端的Phox和Bem1 (PB1)结构域[18,26].硝酸盐信号响应域介导NLP转录因子的激活以响应硝酸盐[13,24].该区域内保守的丝氨酸残基(NLP7中的丝氨酸205)在感知硝酸盐信号后被CPK10、CPK30和CPK32磷酸化[15].rwp - rk型dna结合域,因其包含保守的氨基酸序列Arg-Trp-Pro-X-Arg-Lys(其中X表示任何氨基酸)而得名,与硝酸盐反应性结合独联体-元件(NREs),最初在拟南芥亚硝酸盐还原酶基因的启动子中被发现NIR1和其他硝酸盐诱导基因[13,28,29,30.].单独的RWP-RK dna结合域可以独立于硝酸盐与NREs结合[13].PB1结构域被认为参与蛋白质-蛋白质相互作用,如下所述。Guan等人(2017)最近利用拟南芥的NLP6和NLP7表明,NLP-NLP相互作用以及NLP与TCP20转录因子之间的相互作用是由PB1结构域介导的[23].他们还提出,基于NLP6/7和TCP20在核内的相互作用,以及在核内的作用tcp20基因表达突变NRT1.1,NIA1,NIA2研究表明,NLP6/7-TCP20复合物参与了硝酸盐转运蛋白基因的控制表达NRT1.1还有硝酸盐还原酶基因NIA1而且NIA2在缺氮条件下[23].因此,PB1结构域是否参与调节硝酸盐激活基因的表达仍然是难以捉摸的。
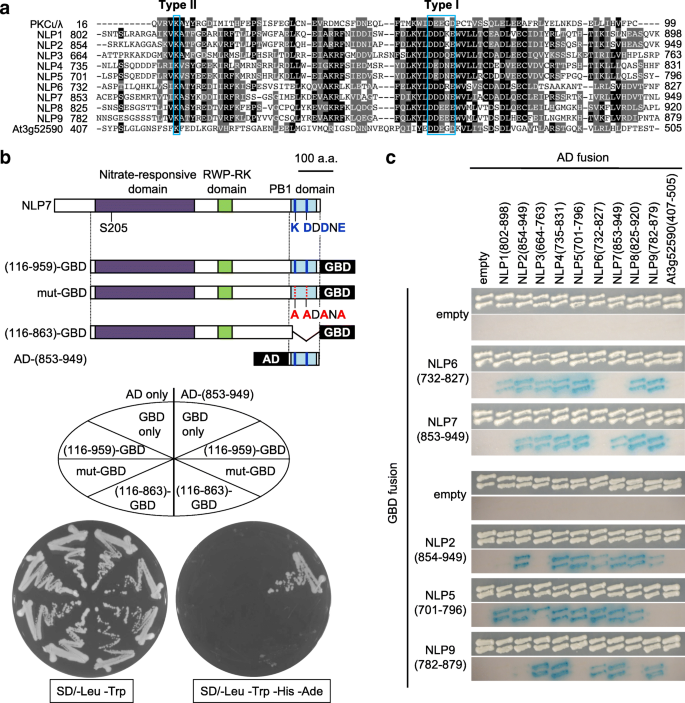
PB1结构域在动物、真菌和植物的多种蛋白质中作为蛋白质-蛋白质相互作用结构域;其中包括Cdc24和Bem1,在萌芽酵母中建立细胞极性所必需的,p40phox和p67phox,它们参与人类吞噬细胞的过氧化物形成,以及哺乳动物和植物自噬所必需的p62 [31,32,33,34].PB1结构域由大约80个氨基酸残基组成,包含I型和II型基序中的一种或两种。1一个);因此,它们被分为I型、II型和I/II型PB1域。I型基序包含三个谷氨酸或天冬氨酸残基,位于PB1结构域的后表面,而II型基序包含一个不变的赖氨酸残基,位于结构域的前表面。因此,两个PB1结构域之间的相互作用以从前到后的方式发生,其中一个PB1结构域中的碱性赖氨酸残基与另一个PB1结构域中的酸性谷氨酸/天冬氨酸残基之间存在静电相互作用[31].含有I型PB1结构域的蛋白质与含有同源的II型PB1结构域的蛋白质异二聚,而含有I/II型PB1结构域的蛋白质可与I型、II型和I/II型PB1结构域相互作用,也可同二聚[31,32].此外,一些PB1结构域介导了与缺乏PB1结构域的蛋白质的相互作用[31].NLP转录因子具有I/II型PB1结构域(图。1一个)。
酵母中PB1结构域介导的NLP-NLP相互作用。一个拟南芥NLP转录因子、人PKC1/λ和一种不相关的拟南芥蛋白(At3g52590) PB1结构域氨基酸序列的比对。黑色和灰色背景分别表示相同和相似的残差。蓝色框标记I型和II型基序中的核心残基。bY2H以NLP7的PB1结构域(残基853-949)为猎物,以野生型NLP7(氨基酸残基116-959)、含有PB1结构域突变的NLP7 (K867、D909、D111和E913被丙氨酸残基取代)或缺乏PB1结构域的NLP7(残基116-863)为诱饵进行分析。GBD和AD分别是酵母Gal4的dna结合域和活化域;AD还包含一个核定位信号。cY2H β-半乳糖苷酶测定NLP PB1结构域之间的相互作用。括号中的数字表示在GBD和AD融合结构中使用的氨基酸残基。对于每个GBD融合结构,上面和下面的图像分别显示细胞生长和β-半乳糖苷酶活性
在本研究中,为了鉴定与NLP转录因子相互作用的蛋白,我们用NLP7进行了酵母双杂交(Y2H)筛选。我们发现PB1结构域介导的相互作用发生在多种NLP转录因子组合之间。使用突变形式的NLP7,其中PB1结构域介导的蛋白质-蛋白质相互作用所必需的氨基酸被丙氨酸取代,我们表明PB1结构域对NLP7的全部活性很重要在足底.然而,在原生质体瞬时表达实验中,PB1结构域的破坏并不影响NLP7从质粒转激活硝酸盐诱导基因的能力。因此,我们的结果表明,由NLP转录因子PB1结构域介导的蛋白-蛋白相互作用对于充分促进靶基因在染色体中的表达是必要的,而不是促进裸DNA的转录。本文报道的依赖于PB1结构域的硝酸盐诱导基因表达的影响不同于NLP-TCP20相互作用,后者仅发生在n饥饿条件下。
结果
PB1域介导的NLP转录因子间的相互作用
为了鉴定与NLP转录因子相互作用的蛋白,我们以NLP7为诱饵进行了Y2H筛选。由于NLP7的氨基末端区域被发现是一个转录激活域(附加文件)1:图S1),我们使用缺少氨基端(氨基酸116-959)的NLP7来避免酵母中的人工转活化。我们获得了多个包含NLP2、NLP3、NLP4、NLP7和NLP9的PB1结构域的cDNA片段,以及编码伴侣相关蛋白的cDNA片段(附加文件)1:图S2);然而,在我们的筛选中没有分离到编码TCP20的cDNA。因此,蛋白质-蛋白质相互作用可能涉及多种NLP转录因子的组合。
为了验证NLP-NLP相互作用依赖于PB1结构域,我们首先使用NLP7的野生型和突变形式进行了Y2H实验。为了产生突变的NLP7蛋白,我们要么删除PB1结构域,要么用丙氨酸替换PB1结构域(K867、D909、D911和E913)的核心氨基酸残基(图9)。1b). Y2H实验表明原生PB1结构域对于NLP-NLP相互作用是必要的(图。1b).接下来,我们使用来自其他NLP转录因子的PB1结构域和来自不相关蛋白At3g52590的PB1结构域作为阴性对照,重复Y2H实验(图2)。1a和c).这揭示了PB1结构域介导的多种NLP转录因子组合之间的相互作用。然而,我们没有检测到NLP6和NLP7之间预期的相互作用,也没有观察到一些其他蛋白质组合之间的相互作用;这可能是由PB1结构域与酵母Gal4结构域融合引起的实验性人工产物。或者,这可能是由于在酵母中难以共表达这些蛋白质。
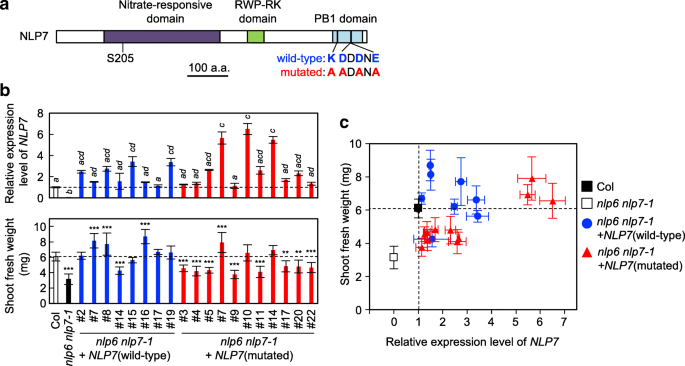
互补nlp6 nlp7-1双突变体,其中NLP7在PB1结构域存在突变
评价NLP转录因子PB1结构域的作用在足底,我们生成了表达NLP7突变形式的转基因拟南芥nlp6 nlp7-1背景。双突变体,nlp6 nlp7-1,在本分析中使用,因为它表现出更强和更明显的表型nlp7-1单突变体[35],从而更容易评估重新引入functional的效果NLP7.突变的NLP7含有一个PB1结构域,其中K867、D909、D111和E913被丙氨酸残基取代(图2)。2A),基因在控制下表达NLP7启动子。表达野生型的转基因植物NLP7从NLP7促进者nlp6 nlp7-1背景生成作为控制。
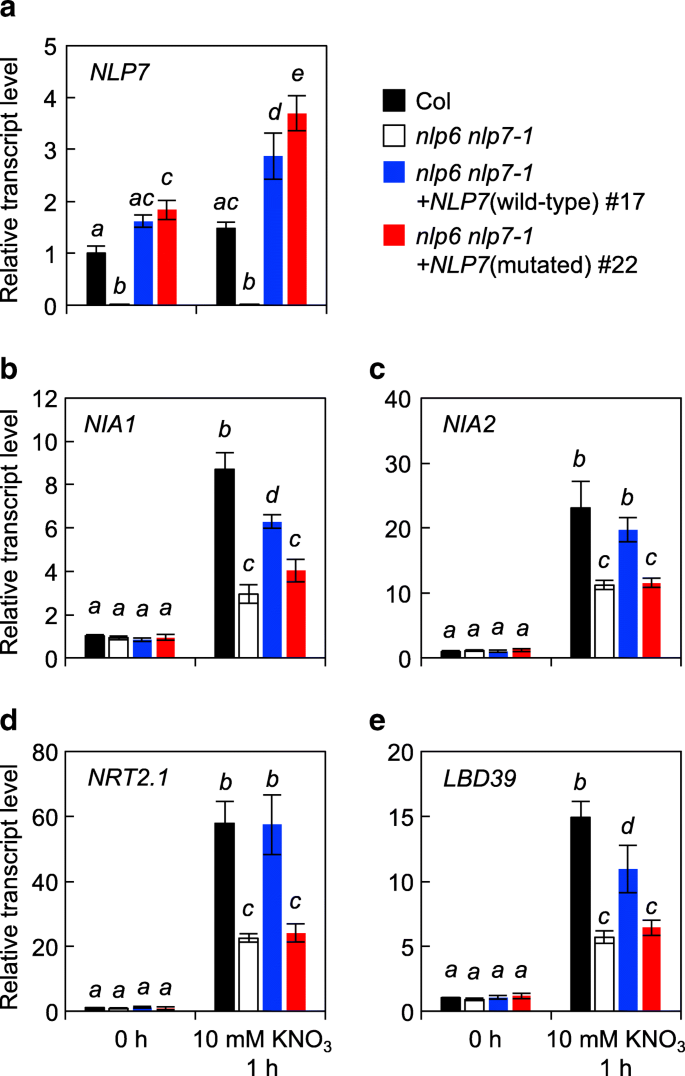
互补nlp6 nlp7-1NLP7野生型和突变型双突变。一个NLP7蛋白的结构域结构。PB1结构域内的核心氨基酸残基以蓝色字体标记;在突变蛋白中,这些氨基酸残基被丙氨酸残基(红色字体)所取代。b介绍表达NLP7(上图)和新梢重(下图)。nlp6 nlp7-1双突变体与野生型或突变型转化NLP7在编码序列的控制下NLP7启动子。种子萌发后,在0.5× MS琼脂板上培养4天,然后转移到含有10 mM KNO的琼脂板上3.作为氮源5天。Col:野生型哥伦比亚幼苗。NLP7转录水平被标准化UBIQUITIN10(UBQ10)表达式,表示±SD (n= 3)。用不同字母标记的柱形之间存在显著差异[Tukey 's诚实显著差异(HSD)测试,P< 0.05)。笋鲜重量值为平均值±SD (n= 9-10株植物)。星号表示通过Dunnett事后检验进行多重比较的单向方差分析与Col有显著差异;* *:P< 0.01和***:P< 0.001。c之间的相关性NLP7表达和拍摄新鲜的重量。误差条为SD
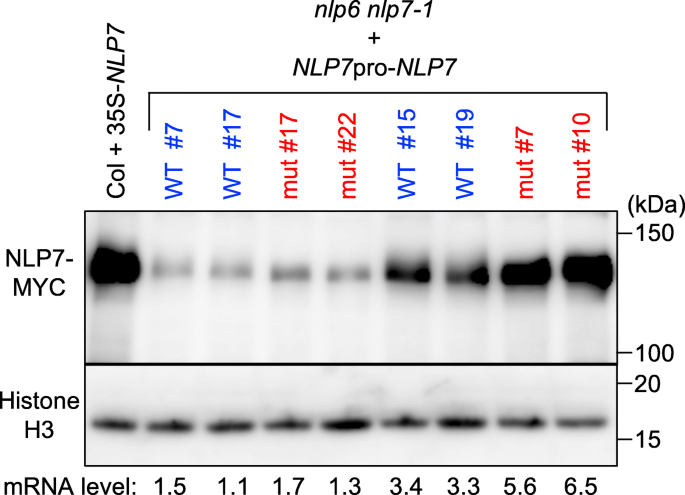
我们首先测量了NLP7在多个转基因系中的转录本(图;2b).可以忽略不计的水平NLP7转录本被检测到nlp6 nlp7-1双突变体,说明NLP7在转基因系中检测到的转录本来源于引种NLP7基因。转录水平NLP7不同的行之间,可能是由于位置的影响。携带野生型的转基因系的转录本水平NLP7是野生型哥伦比亚(Col)植株的1.1 ~ 3.4倍,除1个株系外,其余株系均恢复完全。野生型的表达NLP7在原生启动子的控制下,因而足以弥补缺失的缺失NLP6而且NLP7.只有三个转基因株系发生了突变NLP7,但表达了高水平的NLP7转录本(是Col的5.4 ~ 6.5倍);同一3个品系的鲜重完全恢复,而其他8个品系的鲜重恢复水平一般NLP7转录本(是Col的1.2 ~ 2.6倍),而没有(图;2b和c).为了消除突变NLP7不稳定的可能性,因此需要更高水平的表达来挽救nlp6 nlp7-1突变后,我们使用免疫印迹分析检测这些转基因株系中的NLP7水平(图2)。3.).该分析是使用抗MYC抗体进行的,因为转基因系中的NLP7蛋白被MYC标记。在转基因株系中检测到的蛋白质含量与株系的含量基本成正比NLP7mRNA表达,无论它们是否含有野生型或突变蛋白。因此,我们得出结论,突变形式的NLP7不具有恢复细胞生长所需的全部活性nlp6 nlp7-1双突变体。
互补系NLP7蛋白水平。WT和mut分别表示表达野生型和突变型NLP7的转基因株系。种子萌发后,在0.5× MS琼脂板上培养4天,然后转移到含有10 mM KNO的琼脂板上3.作为N源5天。使用抗MYC抗体检测NLP7,该抗体识别引入NLP7羧基末端的MYC标签。组蛋白H3作为加载对照。车道下面的数字显示了学生的相对成绩水平NLP7在这些线中(图;2b).过度表达野生型的转基因系NLP7在控制之下35个年代启动子作为对照,用于MYC标记的检测
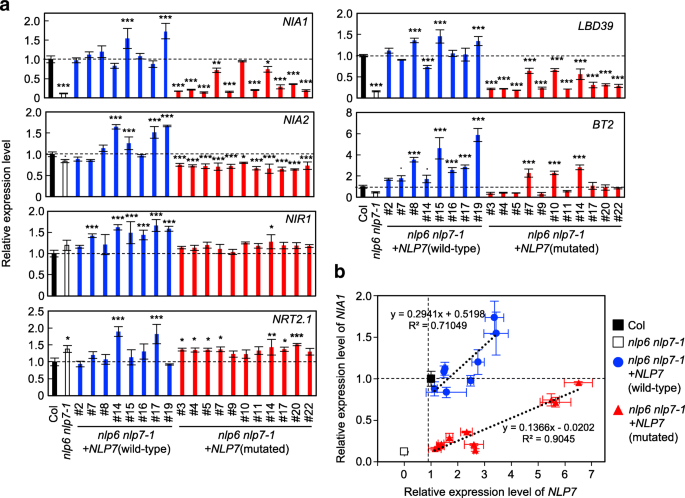
接下来,我们比较了NLP靶基因、硝酸盐还原酶基因转录本的稳态水平NIA1而且NIA2,LBD39(编码一种侧器官边界结构域(LBD)蛋白[4]),BT2(编码BTB-POZ结构域蛋白[30.]),NIR1,NRT2.1(编码高亲和力硝酸盐转运蛋白)。在野生型Col中测定了转录水平,nlp6 nlp7-1和在硝酸盐充足条件下生长9天的转基因幼苗(图2)。4).水平的NIA1,LBD39,BT2转录本在nlp6 nlp7-1幼苗,但对稳态水平的影响有限NIA2,NIR1,NRT2.1观察转录本(图;4a).的表达式NIA1,LBD39,BT2在表达野生型NLP7的转基因株系中得到恢复。的表达NIA1而且LBD39在表达高水平突变NLP7转录本(第7、第10和第14行),但在其他8行中没有,它们只表达了适度的突变水平NLP7成绩单(无花果。4a).的转录水平BT2也表现出相似的模式(图;4a).曲线之间的相关回归线的梯度NLP7而且NIA1表达野生型NLP7和突变型NLP7的转基因株系的转录水平明显不同。4b).从回归分析中得到的最佳拟合线在表达野生型NLP7的系中比表达突变蛋白的系中更陡峭,这表明突变NLP7的活性受到严重损害。
PB1结构域突变对硝酸盐诱导基因表达的影响在足底
为了评估突变NLP7激活硝酸盐诱导基因表达的能力,我们使用qRT-PCR方法分析了在无硝酸盐条件下和添加硝酸盐后1 h生长的幼苗的转录水平。一对转基因株系表达相当水平的NLP7从携带NLP7野生型和突变型的株系中选择Col幼苗的转录本(图2)。5a).添加硝酸盐诱导NIA1,NIA2,NRT2.1,LBD39在nlp6 nlp7-1低至Col幼苗的一半(图2)。5抵扣)。这种表达上的差异在年几乎消失了nlp6 nlp7-1表达野生型NLP7的幼苗;相比之下,施用硝酸盐后,这些基因的表达水平相似nlp6 nlp7-1幼苗和表达突变体NLP7的转基因株系(图;5抵扣)。从另一对独立的转基因株系中获得了非常相似的结果(附加文件1:图S3)。添加硝酸盐增加了野生型和突变型的水平NLP7仅在有限的范围内转录(图;5a).因此,硝酸盐诱导基因表达的差异并不是由于硝酸盐处理导致的NLP7突变转录水平的人为降低。突变的NLP7不能促进硝酸盐诱导基因的表达。值得注意的是,表达NRT2.1更低nlp6 nlp7-1在监测硝酸盐短期反应的条件下,突变体的数量是Col幼苗的两倍(图2)。5D),而减少NRT2.1表达不明显nlp6 nlp7-1在持续的硝酸盐供应下生长的突变体(图;4a).与硝酸盐长期孵育可能导致硝酸盐信号通路的下调和N代谢产物的增加,这两者都掩盖了硝酸盐的直接作用nlp突变。
原生质体瞬时表达分析中PB1结构域突变对NLP7转激活的影响
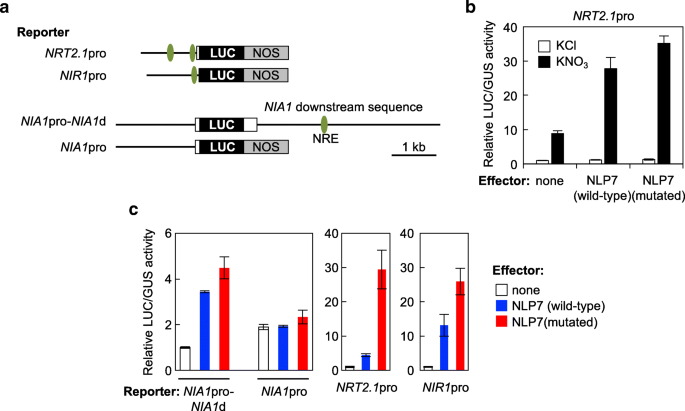
为了研究PB1结构域在靶基因转激活中的作用,我们进行了原生质体瞬时表达测定。我们首先证实了PB1结构域单独不能诱导转录激活(附加文件)1:图S1d),然后评估野生型NLP7和突变型NLP7在PB1结构域携带氨基酸取代的转激活靶基因的能力。
含有野生型或突变型的质粒NLP7在控制之下35个年代将本构启动子与含有该启动子的质粒一起导入从缺氮Col植物分离的叶肉原生质体中卢克在记者的控制下NRT2.1启动子(无花果。6a).转染的原生质体在硝酸盐存在或不存在的条件下孵育。野生型和突变形式的NLP7增强了硝酸盐依赖的活化NRT2.1启动子达到类似程度(图;6b).这是一个意想不到的结果,因为这意味着由PB1结构域介导的蛋白质-蛋白质相互作用对于原生质体中目标启动子的转激活是不必要的。
原生质体瞬态分析系统中突变NLP7的活性。一个中使用的报告器构造的示意图表示b而且c;LUC:萤火虫荧光素酶基因;NOS:诺帕林合酶基因的转录终止序列。白色方框表示5 '或3 '未翻译区域,水平线表示序列上游(“启动子”)或下游NRT2.1,NIR1,或NIA1编码区域。绿色椭圆标记实验验证的nlp结合位点。b原生质体瞬态分析使用n饥饿Col原生质体。原生质体与NRT2.1原报告质粒,表达野生型或突变NLP7的效应质粒,以及表达β-葡萄糖醛酸酶(GUS)的对照质粒UBQ10启动子(UBQ10-GUS),在添加1mm KCl或KNO的培养基中孵育过夜3..c原生质体瞬态分析使用原生质体从叶片分离nlp6 nlp7-1植物提供氮。将原生质体与报告质粒、NLP7表达质粒和对照质粒共转染,在原生质体培养培养基中孵育一夜。荧光素酶活性值根据GUS活性进行归一化。平均值±SD (n= 3)b而且c
所有的在足底分析如图所示。2,3.,4而且5涉及到nlp6 nlp7-1表达野生型或突变型NLP7的植物。为了理解图中结果之间的差异。5而且6B,我们使用从叶片中分离的原生质体进行了进一步的瞬态分析nlp6 nlp7-1在充足氮条件下生长的植物。突变的NLP7蛋白强烈激活了该蛋白的表达卢克在记者的控制下NIR1而且NRT2.1启动子的nlp6 nlp7-1原生质体的增加卢克与突变型NLP7蛋白相关的活性略高于与野生型NLP7相关的活性(图2)。6c).突变体NLP7也促进了卢克记者通过与位于下游的nlp绑定站点的交互NIA1(无花果。6c).如NRE中的NIA1位于下游3 '的侧翼区域NIA1[13,36,37], NLP7(野生型和突变型)激活NIA1pro- nia1d报告基因,但不激活NIA1pro报告基因(图2)。6c).这些结果表明突变体NLP7蛋白本身保留了dna结合和转激活活性。
讨论
我们在Y2H筛选中分离出编码各种NLP转录因子的cDNA克隆,旨在识别与NLP7相互作用的蛋白质。此外,我们发现在NLP转录因子中发现的大多数PB1结构域以各种组合相互作用(图2)。1c).这一结果强烈表明,除了之前报道的NLP6与NLP6、NLP6与NLP7、NLP7与NLP7之间的相互作用外,[23],涉及其他具有重叠表达模式的NLP转录因子组合的相互作用可能发生在植物细胞中。NLP转录因子之间的相互作用是典型的PB1结构域介导的相互作用,因为它们被PB1结构域核心氨基酸残基的突变所消除(图2)。1b).然而,我们的Y2H分析并没有检测到NLP6和NLP6之间或NLP6和NLP7之间的相互作用,这些相互作用之前是通过烟草细胞识别出来的[23],可能是因为实验人工产物是由PB1结构域与酵母Gal4结构域的融合引起的,或者因为酵母中这些蛋白质的表达水平太低。
我们的互补实验揭示了PB1结构域的突变强烈地损害了nlp7依赖的硝酸盐诱导基因对硝酸盐的响应激活(图2)。5;额外的文件1:图S3)。我们还表明,PB1结构域对于一些硝酸盐诱导基因的稳态表达水平是必要的(图2)。4).这些结果提供了直接证据,证明在硝酸盐存在的情况下,由PB1结构域介导的相互作用是NLP7发挥全部功能所必需的。此前,由于NLP6/7-TCP20相互作用(依赖于N饥饿)与植物的表型之间存在相关性tcp20NLP与TCP20的相互作用被认为仅在n饥饿条件下才调控基因表达[23].然而,我们发现,在硝酸盐充足的条件下,硝酸盐诱导基因以PB1结构域依赖的方式被激活,因此PB1结构域的这种调控应该与依赖NLP-TCP20相互作用的调控区别开来。
出乎意料和有趣的是,在原生质体瞬态实验中,野生型和突变型NLP7诱导的转录水平相似(图2)。6),表明突变体NLP7可以被硝酸盐信号激活,与DNA结合,并以与野生型NLP7相当的水平激活转录,从而完全保留了作为转录因子的能力。尽管有这个发现,在足底转基因系中的基因表达受到NLP7野生型和突变型的差异调控(图2)。4而且5).虽然原生质体瞬态分析系统是评估转录因子对目标启动子影响的有力工具[38],它并不完全模仿染色体基因的转录。报告质粒中的DNA瞬间导入原生质体时处于裸态,而染色体DNA则包裹在组蛋白周围,形成核小体。由于核小体是对抗转录的一般屏障[39],由PB1结构域介导的NLP蛋白的寡聚可能是诱导染色质重塑从而实现转录所必需的。
最近,一种pb1介导的相互作用Medicago truncatulaNLP1具有结节特异性转录因子NIN (NODULE INCEPTION)m . truncatula[17].正如它们的全名“NIN- like蛋白”所示,来自NLP家族的蛋白质与NIN有很大的相似之处。豆科NIN蛋白含有RWP-RK dna结合和PB1结构域,以及一个类似于NLP转录因子中发现的硝酸盐反应区域的区域。Lotus对虾NIN作为一种转录激活因子,具有与NLP转录因子相似的dna结合特异性,但是Lj由于NIN的硝酸盐反应区发生突变,NIN失去了对硝酸盐的反应能力[19,26].因此,pb1介导的NIN和NLP转录因子之间的相互作用是非常合理的。之间的相互作用太NLP1和太NIN抑制NIN活性,从而抑制硝酸盐对结瘤的反应[17].这表明PB1结构域介导的相互作用可能对NLP转录因子有积极作用,但对NIN活性有抑制作用。因此,有必要进一步分析,以准确理解NLP-NLP和NLP-NIN相互作用如何产生不同的输出。
结论
NLP转录因子的PB1结构域介导了不同NLP转录因子之间的多种相互作用。由PB1结构域介导的蛋白-蛋白相互作用对于硝酸盐诱导基因的充分表达是必要的在足底.这种相互作用可能通过PB1结构域介导的NLP转录因子的自低聚化来招募染色质重塑因子。对这些蛋白-蛋白相互作用的进一步分析可能会为充分理解调节硝酸盐相关生理过程的NLP/NIN网络提供线索。
方法
质粒构建
对于Y2H分析,pGBT9K-MCS-GBD能够表达羧基端Gal4 dna结合域(GBD)融合蛋白,使用pGBT9 (Clontech, Mountain View, CA, USA)生成,pGBT9是一种能够产生氨基端GBD融合蛋白的质粒载体。的欣dIII站点之间ADH1启动子和pGBT9中的GBD编码序列被包含BamH1,以区域我和斯图I位点,以及GBD序列和克隆位点之间的多重克隆位点(MCS)ADH1终结者被移除。为了得到用于Y2H分析的pNLP7(116-959)-GBD和pNLP7(116-863)-GBD,使用合适的PCR引物分别连接到nnlp7的116-959和116-863氨基酸,通过PCR扩增得到pNLP7(116-959)-GBD以区域我或斯图我的网站。将PCR产物克隆到pGBT9K-MCS-GBD中以区域我和斯图I网站之间的ADH1启动子和GBD编码序列。为了生成质粒pNLP7(PB1mut)-GBD,使用突变引物组通过5轮PCR将突变引入PB1结构域(附加文件)1:表S1)。突变片段也被插入以区域我和斯图I网站在pGBT9K-MCS-GBD。为了获得用于分析PB1结构域之间相互作用的质粒,采用PCR扩增PB1结构域周围的DNA片段,PCR引物分别与生态国际扶轮或BamHI,并将PCR产物插入pGBT9或pGADT7 (Clontech)中。
报告质粒pNRT2.1pro-LUC [40]和pNIR1pro-LUC [13],效应质粒p35SC4PPDK-NLP7-MYC6 [13在共转染实验中使用已在前面描述过。质粒pNIA1pro-LUC-NOS被替换35个年代pJD301中的启动子[41]中含有1.9 kb的DNA片段NIA1启动子(36].pNIApro-LUC-NIA1d由替换号pNIA1pro-LUC-NOS的终止子区域,其终止密码子下游的4.5 kb DNA片段NIA1[36].为了产生能够表达融合到LexA的NLP7特定区域的效应质粒,LexA-MYC质粒中的MYC标签编码序列被替换为编码NLP7特定区域的DNA片段,这些DNA片段通过PCR扩增得到[42].所有使用的引物列在附加文件中1:表S1。所有PCR扩增得到的克隆片段均经DNA测序确认。
酵母转化和β-半乳糖苷酶测定
酿酒酵母菌株AH109和Y187分别进行生长试验和β-半乳糖苷酶试验。以pGBT9和pGADT7为载体,采用醋酸锂/聚乙二醇介导的质粒转化酵母,在缺乏色氨酸和亮氨酸的合成定义(Synthetic Defined, SD)培养基上选择转化菌落。为了进行生长试验,菌落被划到缺乏色氨酸、亮氨酸、组氨酸和腺嘌呤的SD培养基上。对于β-半乳糖苷酶测定,菌落被划到缺乏色氨酸和亮氨酸的SD培养基上,培养2天后按照酵母规程手册的方法进行菌落提升过滤器测定[43].
植物材料和生长条件
拟南芥从拟南芥生物资源中心(ABRC)获得的ecotype Columbia (Col-0)在本研究中作为野生型对照。的nlp6而且nlp7-1还从ABRC中获得突变体,并进行杂交以生成nlp6 nlp7-1双突变体[40].拟南芥的繁殖植物生长在含营养的泥炭上(Jiffy-7, Sakatanotane,横滨,日本),并用水灌溉。的nlp6 nlp7-1用含1mm氯化钙的水灌溉突变体2.
为了测定幼苗鲜重,并测定转录物和蛋白质水平,种子播种在含有0.5× MS[一半强度Murashige和Skoog (MS)盐[44], 0.5 g L−12- morpholinoethanonic acid, one - hydrate (MES)-KOH, pH 5.7, 1%蔗糖,0.8%琼脂(Sigma A1296)],在4℃下分层3-4天。将平板水平放置于23°C、持续光照(60 μmol m)的生长室中−2年代−1)四天。幼苗转移到垂直试验板(0.5× MS无氮盐,10 mM KNO3., 0.5 g L−1MES-KOH, pH 5.7, 1%蔗糖,0.8%琼脂)孵育5天。避免嫩枝与培养基直接接触[45]时,孵育前将琼脂培养基从测试板上部2cm处移除。为分析硝酸盐诱导基因的表达,将100粒种子表面消毒后,播种于20 ml(无氮0.1× MS盐,0.1 g L−1MES-KOH, pH 5.7, 0.5%蔗糖,0.5 mM琥珀酸铵)在一个塑料盘子里。4°C分层3-4 d后转移至23°C连续光照(60 μmol m−2年代−1).4 d后,用10 mM KNO处理幼苗3.提取RNA,进行RT-qPCR。
二元质粒的构建与转基因植物的生成
一个3kb的DNA片段NLP7启动子(碱基对- 3018到- 1,相对于翻译起始位点)通过PCR扩增,用酶切酶切欣dIII和以区域I,克隆到pJD301中,得到质粒pNLP7pro-LUC。所有使用的引物列在附加文件中1:表S1。然后从pNLP7pro-LUC中切除启动子片段NLP7的编码序列,以及编码MYC标记的六个副本的序列号从p35SC4PPDK-NLP7-MYC6中切除[13].这两个序列一起被插入到pCB302HYG-35SΩ-GUS [46,以取代35SΩ启动子格斯吉恩,还有号终结者。将得到的二元质粒导入根癌土壤杆菌菌株GV3101 (pMP90) [47],用于转换nlp6 nlp7-1双突变体。选择T2代具有3:1偏析抗潮霉素的品系,用于生成T3代的纯合子品系。
RT-qPCR
RNA提取,逆转录,定量PCR (qPCR),如前所述[28].qPCR分析中使用的引物在附加文件中列出1:表S1。
原生质体共转染试验
原生质体共转染试验采用水培法种植,缺氮Col植物,如前所述[40],每2 × 10添加0.1 μg效应质粒4本研究中的原生质体。nlp6 nlp7-1种子放在0.1× MS溶液加2 mM氯化钙浸湿的聚氨酯海绵上2(0.1× MS盐,0.1 g L−1MES-KOH, pH 5.7, 2 mM氯化钙2),在连续光照下孵育1天,然后在黑暗中孵育2天。缺乏活力的nlp6 nlp7-1在添加2 mM氯化钙的0.1× MS溶液中水培2在阳光下呆了20天。原生质体分离和转染2 × 104含有报告质粒(6 μg)、效应质粒(0.1 μg)、内控质粒UBQ10-GUS (2 μg)和空质粒(12 μg)的原生质体均如前文所述。转染的原生质体在黑暗的WI培养基中孵育一夜。
免疫印迹分析
样品制备、SDS-PAGE和免疫印迹分析均如Liu等所述[15].
缩写
- 广告:
-
Gal4转活化域
- GBD:
-
Gal4 dna结合域
- 格斯:
-
β葡萄糖醛酸酶
- 小黑裙:
-
外侧器官边界域
- 主持人:
-
多个克隆网站
- 护士:
-
氮
- NLP:
-
NIN-LIKE蛋白质
- 号《终结者》:
-
诺帕林合酶的转录终止序列
- 负阻元件:
-
Nitrate-responsive独联体元
- PB1:
-
Phox和be1
- Y2H:
-
酵母2台混合动力
参考文献
Vitousek PM, Howarth RW。陆地和海洋的氮限制:它是如何发生的?生物地球化学。1991;13:87 - 115。
约翰逊DW,科尔DW。土壤中的阴离子迁移:与森林生态系统的养分运输相关。环境科学,1980;3:79-90。
王锐,Tischner R, Gutierrez RA, Hoffman M,邢X,陈敏,等。拟南芥硝酸盐还原酶缺失突变体硝酸盐反应的基因组分析。植物物理学报。2004;136:2512-22。
鲁宾G, Tohge T,松田F,齐藤K, Scheible WR。转录因子LBD家族成员抑制拟南芥花青素合成并影响额外的氮反应。植物学报。2009;21:3567-84。
Tabata R, Sumida K, Yoshii T, Ohyama K, Shinohara H, Matsubayashi Y.根源多肽通过芽LRR-RKs介导系统性n -需求信号。科学。2014;346:343-6。
木叶,稻叶J,工藤T,上田N,小西M,光田N,等。拟南芥garp型转录因子AtNIGT1/HRS1亚家族成员对氮饥饿反应的抑制植物学报。2018;30:925-45。
Widiez T, El Kafafi El S, Girin T, Berr A, Ruffel S, Krouk G,等。高氮不敏感9 (HNI9)介导的根系NO全身性抑制3.−摄取与组蛋白甲基化变化有关。中国自然科学学报(英文版);2011;
鲁菲尔,克鲁克,李志刚,李志刚,李志刚。植物根系觅食的氮经济学:硝酸盐-细胞分裂素传递关闭和氮素供需的系统信号传递。自然科学学报(英文版);2011;
王锐,冈本M,邢X,克劳福德NM。对拟南芥根和芽中硝酸盐反应的微阵列分析揭示了超过1000个快速反应基因,以及与葡萄糖、海藻糖-6-磷酸盐、铁和硫酸盐代谢的新联系。植物学报。2003;32:556 - 67。
谢伟林,陈志伟,陈志伟,等。全基因组重编程的初级和次级代谢,蛋白质合成,细胞生长过程,以及拟南芥的调节基础设施对氮的响应。植物物理学报。2004;136:2483-99。
O 'Brien JAA, Vega A, Bouguyon E, Krouk G, Gojon A, Coruzzi G,等。硝酸盐在植物中的运输、感应和反应。Mol Plant, 2016; 9:837-56。
Gras DE, Vidal EA, Undurraga SF, Riveras E, Moreno S, Dominguez-Figueroa J,等。SMZ/SNZ信号通路和赤霉素信号通路是硝酸盐诱导的油菜开花时间延迟所必需的拟南芥.中国机械工程学报,2018;49(6):619 - 31。
拟南芥n -like转录因子在硝酸盐信号传导中的核心作用。Nat Commun. 2013;4:1617。
Marchive C, Roudier F, Castaings L, Brehaut V, Blondet E, Colot V,等。转录因子NLP7的核保留调控植物对硝酸盐的早期反应。Nat Commun. 2013;4:1713。
刘克勤,牛勇,小西明,吴勇,杜华,孙忠华,等。中枢营养生长网络中硝酸盐- cpk - nlp信号的发现。大自然。2017;545:311-6。
西田H,田中S,韩田Y,伊藤M,坂本Y,松永S,等。一种nine - like蛋白介导硝酸盐诱导的根结节共生控制Lotus对虾.Nat Commun. 2018;9:499。
林娟,李霞,罗志林,Mysore KS,温娟,谢峰。NIN与nps相互作用介导硝酸盐对植物结瘤的抑制作用Medicago truncatula.植物学报。2018;4:942-52。
李志强,李志强,李志强,等。拟南芥、水稻和水稻中NIN-like蛋白的进化Lotus对虾.中华药理学杂志,2005;
铃木W, Konishi M, Yanagisawa S.豆科植物共生固氮的进化过程可能与NIN转录因子对硝酸盐反应性的丧失有关。植物学报。2013;8:e25975。
曹浩,齐松,孙敏,李忠,杨勇,Crawford NM,等。过表达玉米ZmNLP6和ZmNLP8可以补充拟南芥硝酸盐调控突变体nlp7通过恢复硝酸盐信号和同化。前沿植物科学,2017;8:1703。
Castaings L, Camargo A, Pocholle D, Gaudon V, Texier Y, boutete - mercey S,等。拟南芥中结节起始样蛋白7调节硝酸盐的感知和代谢。植物J. 2009; 57:426-35。
王锐,邢霞,王勇,陈安,郭伟强NM。硝酸盐调节突变体的基因筛选捕获了硝酸盐转运蛋白基因NRT1.1.植物科学进展。2009;
关鹏,Ripoll J-J,王锐,Vuong L, Bailey-Steinitz LJ,叶东,等。TCP和NLP转录因子相互作用控制植物对硝酸盐有效性的反应。中国科学院学报(自然科学版),2017;
闫迪,Easwaran V, Chau V,冈本M, Ierullo M,木村M,等。nin样蛋白8是硝酸盐促进种子萌发的主要调节因子拟南芥.7. Nat Commun. 2016;
Hiratsu K, Matsui K, Koyama T, Ohme-Takagi M.拟南芥嵌合抑制子对靶基因的显性抑制,包括EAR基序,一个抑制域。植物J. 2003; 34:733-9。
Konishi M, Yanagisawa S.在理解硝酸盐调控基因表达的分子机制方面迈出了新的一步。中国机械工程学报(自然科学版),2014;
于汉林,吴娟,唐辉,袁勇,王明民,王一平,等。过度的拟南芥NLP7通过加强氮和碳同化,在限制氮和充足氮条件下促进植物生长。科学代表2016;6:27795。
Konishi M, Yanagisawa S.硝酸盐反应的鉴定独联体-元素在拟南芥NIR1启动子定义了氮反应中多个顺式调控元件的存在。植物学报,2010;
Maeda S, Konishi M, Yanagisawa S, Omata T.一种新型HPP家族蛋白在蓝藻和叶绿体中的亚硝酸盐转运活性。中国生物医学工程学报。2014;
佐藤T,前川S,小西M,吉冈N,佐佐木Y,前田H,等。的直接转录激活英国电信NLP转录因子介导的基因是拟南芥硝酸盐反应的关键组成部分。生物化学与生物物理学报,2017;
Sumimoto H, Kamakura S, Ito T. PB1结构域的结构和功能,这是动物、真菌、变形虫和植物中保守的蛋白质相互作用模块。Sci抽烟可以。2007: re6。
林霞,李松,赵颖,马霞,张凯,何霞,等。p62相互作用域:p62与选择性自噬之间的桥梁。中国生物医学工程学报。2013;32:20 20 - 7。
Svenning S, Lamark T, Krause K, Johansen T.植物NBR1是一种选择性自噬底物,是哺乳动物自噬适配体NBR1和p62/SQSTM1的功能性杂交产物。自噬。2011;7:993 - 1010。
Zientara-Rytter K, Sirko a . PB1和UBA结构域在烟草选择性自噬货物受体Joka2多聚化中的重要作用。中国植物科学,2014;5:13。
关鹏,王锐,Nacry P, Breton G, Kay SA, Pruneda-Paz JL,等。采食硝酸盐拟南芥根是由转录因子TCP20通过系统信号通路介导的。中国科学院学报(自然科学版),2014;
Konishi M, Yanagisawa S.硝酸盐还原酶基因调控区对硝酸盐反应表达的控制,NIA1在拟南芥中。中国生物医学工程学报,2011;
硝酸盐还原酶基因3 '侧区nlp结合位点与硝酸盐诱导表达有关拟南芥(l)Heynh。土壤科学与工程,2013;29(3):344 - 344。
刘世德,赵永华,辛俊。拟南芥叶肉原生质体:用于瞬时基因表达分析的多功能细胞系统。Nat protocol . 2007; 2:1565-72。
Lorch Y, Kornberg RD.染色质重塑转录。Q Rev biophysical . 2017;50:e5。
前田Y,小西M,木叶T,樱叶Y,泽木N,仓井T,等。以nigt1为中心的转录级联调节硝酸盐信号,并将磷饥饿信号纳入拟南芥.Nat Commun. 2018;9:1376。
吕赫森KR, de Wet JR, Walbot .萤火虫荧光素酶报告基因在植物中的瞬时表达分析。方法酶促化学,1992;216:397-414。
Sawaki N, Tsujimoto R, Shigyo M, Konishi M, Toki S,藤原T,等。水稻中硝酸盐诱导的GARP家族基因编码一种自抑制转录抑制因子。中国生物医学工程学报,2013;
Clontech实验室公司克隆技术-酵母协议手册。2009。
一种改进的烟草组织培养快速生长和生物测定的培养基。物理植物。1962;15:473-97。
杜brovsky J, Forde B.侧根发育的定量分析:陷阱和如何避免它们。《植物细胞》2012;24:4-14。
Kato Y, Konishi M, Shigyo M, Yoneyama T, Yanagisawa S.植物真核翻译起始因子6 (eIF6)基因在胚胎发生中的作用及其差异表达拟南芥和米饭。生物化学与生物物理学报,2010;
康兹C,谢尔jl-DNA基因5控制嵌合基因的组织特异性表达的一种新型农杆菌属二进制向量。MGG Mol Gen Genet, 1986; 204:383-96。
致谢
作者感谢拟南芥生物资源中心提供的种子nlp6而且nlp7-1.
资金
这项工作部分由JST CREST拨款号JPMJCR 15O5支持SY和JSPS KAKENHI拨款号26221103和25252014支持SY, 15H05616支持MK。JST和JSPS没有参与研究的设计和数据的收集、分析和解释,也没有参与撰写手稿。
数据和材料的可用性
本研究中使用的数据集可根据合理要求从通讯作者处获得。
作者信息
从属关系
贡献
MK和SY设计了研究;MK进行实验;MK和SY撰写了手稿。两位作者都已阅读并批准了手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
额外的文件
附加文件1:
图S1。NLP7的非保守氨基末端区域是一个转活化域。图S2。用NLP7 (aa。116-959)作为诱饵。图S3。PB1结构域突变对硝酸盐诱导基因表达的影响。表S1。研究中使用的引物列表。(PDF 66kb)
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
Konishi, M., Yanagisawa, S. NLP转录因子PB1域介导的蛋白-蛋白相互作用在硝酸盐诱导基因表达中的作用。BMC植物生物学19日,90(2019)。https://doi.org/10.1186/s12870-019-1692-3
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-019-1692-3
关键字
- 类蛋白(nlp)
- 硝酸反应
- PB1域
- 蛋白质相互作用