- 研究文章gydF4y2Ba
- 开放访问gydF4y2Ba
- 发布:gydF4y2Ba
野生甘薯(gydF4y2BaIpomoea Trifida.gydF4y2Ba)基因组提供了存储根系开发的见解gydF4y2Ba
BMC植物生物学gydF4y2Ba体积gydF4y2Ba19.gydF4y2Ba, 文章编号:gydF4y2Ba119.gydF4y2Ba(gydF4y2Ba2019gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
地瓜(gydF4y2Ba番薯甘薯gydF4y2Ba(l)是世界上第七重要的作物,主要种植其地下贮藏根(SR)。由于该物种基因组结构复杂,缺乏高质量的参考序列,阻碍了该物种的遗传研究。二倍体gydF4y2BaIpomoea Trifida.gydF4y2Ba是甘薯最近的亲属和假定的祖先,被认为是甘薯的模式种,包括遗传学,细胞学和生理学分析。gydF4y2Ba
结果gydF4y2Ba
在这里,我们产生了SR形成二倍体的染色体级基因组序列gydF4y2Ba即trifidagydF4y2Bavar。Y22与高杂合度(2.20%)。虽然基于染色体同线性分析表明,gydF4y2Ba即trifidagydF4y2Ba与。共享保守核型gydF4y2BaIpomoea nil.gydF4y2Ba分离后,gydF4y2Ba即trifidagydF4y2Ba有一个比更小的基因组gydF4y2Ba一,零gydF4y2Ba由于LTR-RODRONSPOSONS更有效地消除了LTR-RTS的物种特异性放大爆发。与四种非SR形成物种的比较表明,β-淀粉酶基因家族的演化可能与SR形成有关。我们进一步调查了关键基因的关系gydF4y2BaBMY11gydF4y2Ba(身份47.12%至gydF4y2Baβ-淀粉酶1gydF4y2Ba通过基因表达谱和数量性状位点(QTL)的定位,对该重要农艺性状进行了分析。并结合SR形态结构、基因表达谱和qPCR结果,推测产物的活性gydF4y2BaBMY11gydF4y2Ba裂解淀粉颗粒,再循环合成更大的颗粒,促进淀粉积累和SR溶胀。此外,我们发现了的表达模式gydF4y2BaBMY11gydF4y2Ba糖代谢和石柱木质素化的关键基因与甘薯相似。gydF4y2Ba
结论gydF4y2Ba
我们构建了高质量的高杂合子基因组参考gydF4y2Ba即trifidagydF4y2Ba通过一种联合的方法和该基因组使该物种的基因组特征和基因组进化的更好的分辨率。甘薯SR发育基因可以在gydF4y2Ba即trifidagydF4y2Ba这些基因具有相似的功能和模式,显示出二倍体gydF4y2Ba即trifidagydF4y2Bavar. Y22具有典型的SR,是研究甘薯SR发育的理想模型。gydF4y2Ba
背景gydF4y2Ba
地瓜(gydF4y2Ba番薯甘薯gydF4y2Ba(l)Lam.),主要是为了其地下贮藏根(SR)而种植,大约在8000 - 10000年前在美洲被发现,至少在4000年前被驯化[gydF4y2Ba1gydF4y2Ba].这种植物随后遍布世界各地,这可以追溯到前哥伦比亚时代的开始[gydF4y2Ba2gydF4y2Ba].由于它易于种植,产量高,淀粉和营养丰富,已成为世界各地饮食的重要组成部分。近年来,全球甘薯产量超过1亿吨(gydF4y2Bahttp://www.fao.org/faostat/en/#homegydF4y2Ba);它已成为三个主要的根和块茎作物之一,以及世界上第一个最重要的食物作物[gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba4gydF4y2Ba,gydF4y2Ba5gydF4y2Ba].了解SR的形成和发育机理对进一步提高甘薯产量具有至关重要的意义[gydF4y2Ba6gydF4y2Ba].迄今为止,sporamin蛋白蛋白质,Dof结构型锌指蛋白,MADS盒蛋白和KNOX蛋白已被证明是与SR发展相关联gydF4y2Ba7gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba].基因组信息的缺乏减缓了SR发展的研究[gydF4y2Ba7gydF4y2Ba].对应于许多甘薯定量特质基因座(QTL)的候选基因仍然是难以捉摸的[gydF4y2Ba10.gydF4y2Ba].新发布的单倍型甘薯基因组[gydF4y2Ba11.gydF4y2Ba提供了一种额外的资源来帮助实现这一目标,但是染色体规模的组装是根据gydF4y2BaIpomoea nil.gydF4y2Ba基因组,可能不适合基因组学研究,如QTL调查。甘薯有大量的小染色体(2gydF4y2BangydF4y2Ba = 6xgydF4y2Ba = 90, B1gydF4y2BaBgydF4y2Ba1gydF4y2BaBgydF4y2Ba2gydF4y2BaBgydF4y2Ba2gydF4y2BaBgydF4y2Ba2gydF4y2BaBgydF4y2Ba2gydF4y2Ba),具有高度重复的元素和高杂合性,它含有均质B.gydF4y2Ba2gydF4y2Ba亚血管组[gydF4y2Ba11.gydF4y2Ba,gydF4y2Ba12.gydF4y2Ba,这使得在染色体水平上产生高质量的序列变得困难[gydF4y2Ba13.gydF4y2Ba].gydF4y2Ba
在该属的大约700种中gydF4y2Ba番薯gydF4y2Ba[gydF4y2Ba14.gydF4y2Ba],gydF4y2Ba即trifidagydF4y2Ba是甘薯最近的野生亲戚[gydF4y2Ba12.gydF4y2Ba,gydF4y2Ba15.gydF4y2Ba,gydF4y2Ba16.gydF4y2Ba].人工交叉和细胞遗传学研究表明六倍体gydF4y2Ba即trifidagydF4y2Ba来自二倍体gydF4y2Ba即leucanthagydF4y2Ba和四倍体gydF4y2BaI. Littoralis.gydF4y2Ba,而四倍体gydF4y2BaI. Littoralis.gydF4y2Ba同源多倍体来自gydF4y2Ba即leucanthagydF4y2BaB基因(gydF4y2Ba17.gydF4y2Ba,gydF4y2Ba18.gydF4y2Ba]gydF4y2Ba.即leucanthagydF4y2Ba和gydF4y2BaI. Littoralis.gydF4y2Ba应该考虑二倍体和四倍体的形式gydF4y2Ba即trifidagydF4y2Ba分别基于种间杂交和细胞遗传学[gydF4y2Ba18.gydF4y2Ba,gydF4y2Ba19.gydF4y2Ba].人工六倍体gydF4y2Ba即trifidagydF4y2Ba从二倍体和四倍体产生gydF4y2Ba即trifidagydF4y2Ba具有相同的染色体类型作为甘薯,并且因此,甘薯可以从六倍体导出gydF4y2Ba即trifidagydF4y2Ba[gydF4y2Ba18.gydF4y2Ba,gydF4y2Ba19.gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba].此外,来自非编码叶绿体、核ITS序列和核SSRs的数据支持甘薯同源多倍体起源,来自共享二倍体的祖先gydF4y2Ba即trifidagydF4y2Ba基因组[gydF4y2Ba16.gydF4y2Ba].三倍体gydF4y2Ba即trifidagydF4y2Ba可能已经为二倍体和四倍体提供给六倍体的桥梁gydF4y2Ba即trifidagydF4y2Ba[gydF4y2Ba16.gydF4y2Ba,gydF4y2Ba19.gydF4y2Ba,gydF4y2Ba20.gydF4y2Ba].我们最近的限制性位点相关DNA测序(RAD-seq)结果表明甘薯来源于六倍体gydF4y2Ba即trifidagydF4y2Ba和那个六倍体gydF4y2Ba即trifidagydF4y2Ba从四倍体gydF4y2Ba即trifidagydF4y2Ba和二倍体gydF4y2Ba即trifidagydF4y2Ba[gydF4y2Ba21.gydF4y2Ba].然而,gydF4y2BaWX.gydF4y2Ba内含子变化支持SweetPotato的Allohexaploid起源gydF4y2BaI. Tenuissima.gydF4y2Ba和四倍体gydF4y2BaI. Littoralis.gydF4y2BaBlume或Tetraploid.gydF4y2Ba即tabascanagydF4y2Ba,gydF4y2BaI. Tenuissima.gydF4y2Ba早期由二倍体派生的gydF4y2Ba即trifidagydF4y2Ba和一个身份不明的物种[gydF4y2Ba22.gydF4y2Ba].此外,新公布的单倍型解析甘薯基因组的结果也表明甘薯可能是四倍体祖先与二倍体之间的初始杂交的结果gydF4y2Ba即trifidagydF4y2Ba祖(gydF4y2Ba11.gydF4y2Ba].它要么是甘薯最可能的祖先,要么它的一部分基因组已经渗入甘薯的基因组[gydF4y2Ba23.gydF4y2Ba].总之是二倍体gydF4y2Ba即trifidagydF4y2Ba在甘薯复杂的进化史上处于一个临界点,甘薯的起源仍然存在争议。因此,许多研究都集中在二倍体上gydF4y2Ba即trifidagydF4y2Ba;据报道,一项基因组调查[gydF4y2Ba24.gydF4y2Ba),另一个基因组组合可以在线查看(gydF4y2Bahttp://sweetpotato.plantbiology.msu.edu/new.shtml.gydF4y2Ba).以往的研究主要集中在须根(FR)、铅笔根(PR)或粗根(TR)gydF4y2Ba即trifidagydF4y2Ba[gydF4y2Ba20.gydF4y2Ba,gydF4y2Ba24.gydF4y2Ba,gydF4y2Ba25.gydF4y2Ba,gydF4y2Ba26.gydF4y2Ba,gydF4y2Ba27.gydF4y2Ba,gydF4y2Ba28.gydF4y2Ba].在SR上报告了很少的研究,可能是因为缺乏具有SR的二倍体材料,并且尚未报告使用基因组进行SR发展。gydF4y2Ba
在这里,我们报告了二倍体的高质量,染色体锚定参考基因组gydF4y2Ba即trifidagydF4y2Bavar。Y22 [gydF4y2Ba21.gydF4y2Ba],具有典型的SR,类似于SweetPotato(附加文件gydF4y2Ba1gydF4y2Ba:图S1)。采用组合策略重新组装高杂合子基因组(2.20%),预测了30227个基因模型。我们发现全基因组三倍扩增(WGT)发生在它的物种形成之前,大约7450万年前(MYA)。通过基因家族进化、根比较转录组、QTL定位、qPCR等综合分析,发现了该新基因gydF4y2BaBMY11gydF4y2Ba(身份47.12%至gydF4y2Baβ-淀粉酶1gydF4y2Ba)可能在SR开发过程中发挥关键作用。Y22和甘薯之间的关键基因具有类似的表达模式,并且可以通过二倍体的QTL映射来识别甘薯的SR开发基因gydF4y2Ba即trifidagydF4y2Ba人口遗传。这一工作将有助于进一步了解复杂的演化历史和SR的发展机制gydF4y2Ba即trifidagydF4y2Ba和地瓜。gydF4y2Ba
结果gydF4y2Ba
基因组装配,验证和注释gydF4y2Ba
我们构建了一个180 bp的PE文库,并利用Illumina PE125对Y22基因组进行了测序,获得了62.27 Gb的数据用于基因组调查分析。K-mer频率(gydF4y2Ba29.gydF4y2Ba]的分布分析显示Y22的基因组为476.6 Mb,与基于流式细胞仪的估计一致[gydF4y2Ba30.gydF4y2Ba],和杂合性和重复序列的比例分别为2.20和48.42%,分别为(附加文件gydF4y2Ba2gydF4y2Ba:表S1,附加文件gydF4y2Ba1gydF4y2Ba:图S2和S3)。为了达到高质量和染色体级参考基因组,125.6 GB的清洁illumina霰弹枪读取(约264倍的基因组覆盖率)和537 MB的Illumina分子合成长读取(基因组的约1倍)(附加文件gydF4y2Ba2gydF4y2Ba:表S2)最初组装成431.57 Mb的序列,contig N50为26.50 Kb, scaffold N50为580.68 Kb(附加文件gydF4y2Ba2gydF4y2Ba:表S3)。然后,我们加入10.05 Gb(组装覆盖率的21倍)的PacBio RS II数据,N50读取长度为17.03 Kb,以增加序列的连续性。最终组装的contig和scaffold N50分别为54.49 Kb和607.92 Kb(附加文件gydF4y2Ba2gydF4y2Ba:表S4)。此外,96.71%的gydF4y2Ba即trifidagydF4y2Ba基因组was covered by the assembled 460.93 Mb scaffolds. A genotype-by-sequencing (GBS) [31.gydF4y2Ba,gydF4y2Ba32.gydF4y2Ba利用200个真实F1个体(Y25 × Y22)构建遗传图谱(附加文件)gydF4y2Ba1gydF4y2Ba:数字S1和S4;附加文件gydF4y2Ba2gydF4y2Ba:表S5和S6), 400.44 Mb的序列(最终支架的86.88%)成功固定在15条染色体(附加文件gydF4y2Ba1gydF4y2Ba:图S5和S6),可以被认为是更好的参考gydF4y2Ba即trifidagydF4y2Ba(373.4 Mb支架固定在15个连锁组上)[gydF4y2Ba28.gydF4y2Ba].gydF4y2Ba
通过不同的数据集对装配的质量和完整性进行评估。首先,我们将短插入大小的文库读取映射到组装的基因组;图谱作图率为95.2,95.76%的基因组覆盖率超过20倍(附加文件)gydF4y2Ba2gydF4y2Ba:表S7)。通过对装配基因组10 k不重叠滑动窗口的GC含量和序列深度分布计算,装配基因组未被污染(附加文件)gydF4y2Ba1gydF4y2Ba:图S7和S8)。第二,由Trinity从7.2 Gb叶片RNA-seq读取的全长转录本组装而成[gydF4y2Ba33.gydF4y2Ba[将组装的基因组排列。结果表明,超过98.80%的全长转录序列可以映射回一个脚手架,序列覆盖超过50%(附加文件gydF4y2Ba2gydF4y2Ba表S8),与高杂合子组装的结果相当[gydF4y2Ba34.gydF4y2Ba].第三,248个核心真核基因(基本基因簇数据库)中98.39% [gydF4y2Ba35.gydF4y2Ba]可以高可信度地重新排列回基因组组装(附加文件gydF4y2Ba2gydF4y2Ba:表S9)。和93.9%的BUSCO [gydF4y2Ba36.gydF4y2Ba保守的基因在组装中是完整的。这些结果表明,我们组装的基因组实现了保守基因的完全覆盖。gydF4y2Ba
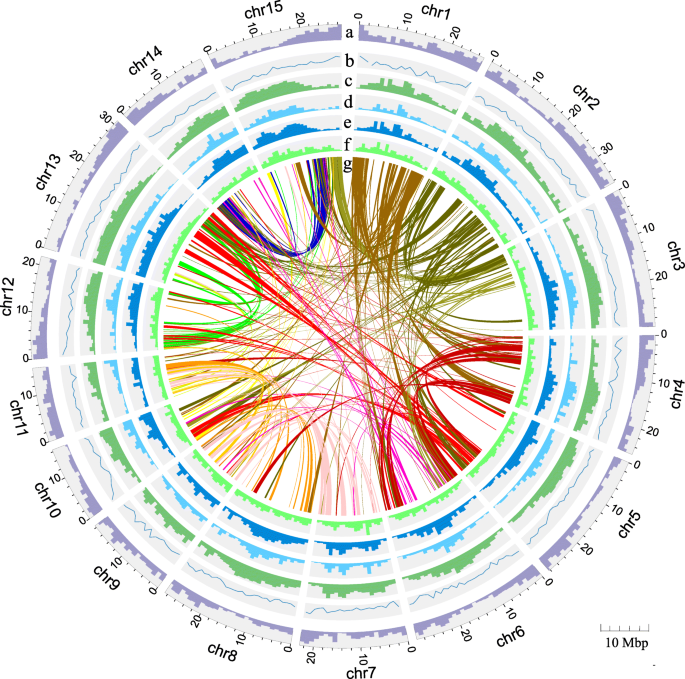
在组装的基因组中,共预测了30227个基因模型gydF4y2Ba即trifidagydF4y2Ba其中79.76%的基因来自7个不同组织(包括叶、花、柱头、花粉、茎、根和种子)的表达证据(RNA-seq reads)gydF4y2Ba2gydF4y2Ba:表S10)。此外,所有基因模型的84.75%有同源性偏见,七种物种序列中具有> 50%的高分分段对覆盖,包括gydF4y2Ba拟南芥gydF4y2Ba,gydF4y2Ba甜菜属gydF4y2Ba,gydF4y2BaCapsicum Annuum.gydF4y2Ba,gydF4y2Ba芝麻纪录gydF4y2Ba,gydF4y2Ba茄属植物lycopersicumgydF4y2Ba,gydF4y2Ba茄属植物tuberosumgydF4y2Ba和gydF4y2Ba葡萄gydF4y2Ba(附加文件gydF4y2Ba1gydF4y2Ba:图S9)。基于与京都基因和基因组百科全书(KEGG)、Swiss-Prot、TrEMBL和基因本体论(GO)公共数据库中已知蛋白的同源性,共注释28456个基因(占全部基因的94.14%)gydF4y2Ba2gydF4y2Ba:表S11)。我们鉴定了候选非编码RNA (ncRNA)序列gydF4y2Ba即trifidagydF4y2Ba通过与已知的ncRNA文库比较和结构预测,基因组中miRNA和tRNA的比例分别为0.0310和0.0423%(附加文件gydF4y2Ba2gydF4y2Ba:表S12)。基因组中有50.86%的重复序列,其中串联重复序列为5.92%。长末端重复(Long terminal repeat, LTR)逆转录转座子是最丰富的元素,占基因组的30.41%,而DNA转座子、长核杂散元件(Long - spersed nuclear elements, LINEs)和短核杂散元件(short interspersed nuclear elements, SINEs)分别占基因组的13.15、4.86和0.68% (Additional file)gydF4y2Ba2gydF4y2Ba:表S13)。我们映射的基因,GC含量,重复序列的分布,gydF4y2Ba吉普赛gydF4y2Ba,gydF4y2BaCopiagydF4y2Ba和DNA重复的gydF4y2Ba即trifidagydF4y2Ba以获得基因组特征的概述(图。gydF4y2Ba1gydF4y2Ba).我们发现,转座因子主要位于染色体中端区,而大多数基因位于染色体臂上。gydF4y2Ba
二倍体的基因组特征gydF4y2Ba即trifidagydF4y2Ba.gydF4y2Ba一个gydF4y2Ba基因密度每Mb。gydF4y2BabgydF4y2Ba每Mb的GC内容。gydF4y2BacgydF4y2Ba每MB的重复内容。gydF4y2BadgydF4y2Ba吉普赛gydF4y2Ba内容/ Mb。gydF4y2BaegydF4y2BaCopiagydF4y2Ba内容/ Mb。gydF4y2BafgydF4y2BaDNA每兆重复含量。gydF4y2BaggydF4y2Ba用MCScanX鉴定了不同染色体间的同线区。显示了大于350 Kb的同序带。每条染色体都被赋予一种颜色,染色体之间连接的颜色由染色体中数目较小的染色体的颜色决定gydF4y2Ba
比较基因组学分析gydF4y2Ba
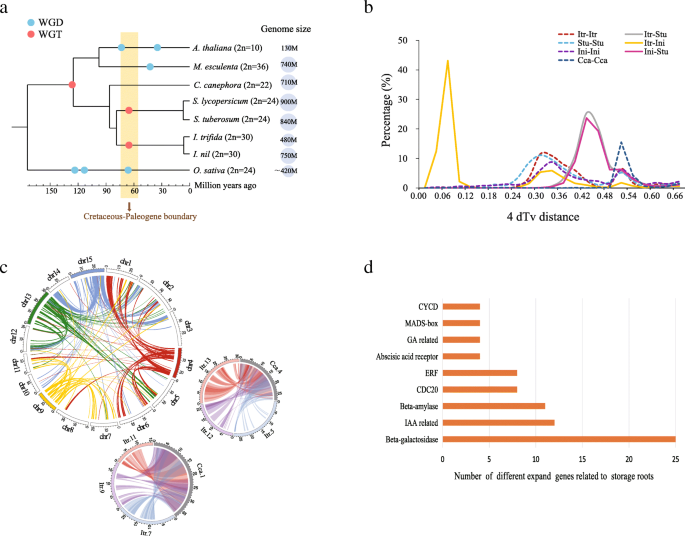
来自八个植物基因组进化的推论(附加文件gydF4y2Ba2gydF4y2Ba表S14)用1930个单拷贝基因家族说明了这一点gydF4y2Ba即trifidagydF4y2Ba和gydF4y2Ba一,零gydF4y2Ba有一个共同的祖先,与共同的祖先分开gydF4y2Ba马铃薯gydF4y2Ba和gydF4y2Ba番薯gydF4y2Ba约78.8 MYA和gydF4y2Ba即trifidagydF4y2Ba和gydF4y2Ba一,零gydF4y2Ba从6.4 mya的共同祖先分歧(图。gydF4y2Ba2gydF4y2Ba一种)。尽管他们密切关系,估计的基因组大小gydF4y2Ba即trifidagydF4y2Ba比……小得多gydF4y2Ba一,零gydF4y2Ba(476.6 Mb vs 750mb) [gydF4y2Ba37.gydF4y2Ba].对这两个络甲状腺结构基因组的重复含量的比较表明gydF4y2Ba一,零gydF4y2Ba基因组含有较高丰富的重复元素,尤其是LTR回收器的家庭,而不是gydF4y2Ba即trifidagydF4y2Ba(额外的文件gydF4y2Ba2gydF4y2Ba:表S15)。单独的LTR /完整的LTR比例gydF4y2Ba即trifidagydF4y2Ba(0.96:1)特别高于gydF4y2Ba一,零gydF4y2Ba(0.38:1),表明复合频率较高gydF4y2Ba即trifidagydF4y2Ba基因组,可以消除可转换元素(附加文件gydF4y2Ba2gydF4y2Ba:表S16)。此外,我们确定了共线之间的区域的基因组gydF4y2Ba即trifidagydF4y2Ba和gydF4y2Ba一,零gydF4y2Ba基于MCScanX检测到的基因块[gydF4y2Ba38.gydF4y2Ba].在该同线区块的总大小gydF4y2Ba即trifidagydF4y2Ba基因组大小为248.3 Mb,小于gydF4y2Ba一,零gydF4y2Ba基因组(339.9 Mb)。在同序块中,重复序列的总长度为gydF4y2Ba即trifidagydF4y2Ba是97.1 Mb,比gydF4y2Ba一,零gydF4y2Ba(165.0 Mb)。重复层序占同序块体的39.1%gydF4y2Ba即trifidagydF4y2Ba的比率小于gydF4y2Ba一,零gydF4y2Ba(48.5%)(附加文件gydF4y2Ba2gydF4y2Ba:表肌力表现)。这些结果表明,重复因子是造成基因组大小差异的主要因素gydF4y2Ba即trifidagydF4y2Ba和gydF4y2Ba一,零gydF4y2Ba,可能是由于LTR-RTs的物种特异性扩增爆发gydF4y2Ba一,零gydF4y2Ba(附加文件gydF4y2Ba1gydF4y2Ba图S10)和更有效的消除ltr -反转录转座子在gydF4y2Ba即trifidagydF4y2Ba.虽然可转换元素gydF4y2Ba一,零gydF4y2Ba比gydF4y2Ba即trifidagydF4y2Ba,两个物种的染色体数目相同,表明它们形成后没有发生大规模的染色体分裂或融合(附文件)gydF4y2Ba1gydF4y2Ba:图S11)。新释出的单倍体解析甘薯组合具有相似的染色体同位性gydF4y2Ba即trifidagydF4y2Ba(附加文件gydF4y2Ba1gydF4y2Ba图S12),这是因为甘薯伪染色体是根据甘薯和甘薯之间的基因和序列共同产生的gydF4y2Ba一,零gydF4y2Ba[gydF4y2Ba11.gydF4y2Ba].虽然Yang等人开发了一种新的单倍分型方法,可以有效地解决多倍体基因组组装问题,但单倍体组装由于其高多态性水平和有限的测序深度(根据六倍体基因组大小约为67倍),支架N50 (~ 201 Kb)较低。因此,更详细的比较gydF4y2Ba即trifidagydF4y2Ba结合长读测序技术、Hi-C测序和多倍体基因组组装算法,一旦获得甘薯基因组更高质量的染色体参考,就应该开展甘薯研究[gydF4y2Ba39.gydF4y2Ba].gydF4y2Ba
演变的gydF4y2Ba即trifidagydF4y2Ba基因组和基因家族。gydF4y2Ba一个gydF4y2Ba系统发育树显示了8个物种的分化时间。蓝点代表WGD事件,红点代表WGT事件。灰色圆圈表示这8个物种的基因组大小。gydF4y2BabgydF4y2Ba同基因4dTv值的分布。虚线表示每个物种与其自身序列的比较,峰值表示基因组复制/三倍事件。固体峰表示物种间的差异事件。Itr:gydF4y2Ba即trifidagydF4y2Ba斯图:gydF4y2Ba马铃薯gydF4y2Ba,CCA:gydF4y2Bac . canephoragydF4y2BaIni:gydF4y2Ba一,零gydF4y2Ba.gydF4y2BacgydF4y2Ba大圆表示gydF4y2Ba即trifidagydF4y2Ba-gydF4y2Ba即trifidagydF4y2Ba这是由一系列旁系同源基因的检测推定从WGT衍生基因内同线区域,(仅染色体4,9,13和15示出)。底部圆示出共线的1号染色体之间的块gydF4y2Bac . canephoragydF4y2Ba第6,7和11号染色体gydF4y2Ba即trifidagydF4y2Ba.右圆圈显示染色体4之间的共线块gydF4y2Bac . canephoragydF4y2Ba染色体5 12 13gydF4y2Ba即trifidagydF4y2Ba.每个链接的颜色对应于染色体的颜色。gydF4y2BadgydF4y2Ba基因家族与SR在相关的扩展gydF4y2Ba即trifidagydF4y2Ba
全基因组复制(WGD)事件gydF4y2Ba即trifidagydF4y2Ba使用蛋白质序列相似检测到的副骨区域来研究其在基因组进化中的重要性[gydF4y2Ba40gydF4y2Ba].我们确定了1856年内部二倍体gydF4y2Ba即trifidagydF4y2Ba共基因块(每块至少5个基因),共包含17630个基因,占总基因的58.33%。四重转换(4dTv)分析表明,WGD发生在0.31附近的峰值,约74.5 MYA(图4)。gydF4y2Ba2gydF4y2BaA, b)与报告的结果相似[gydF4y2Ba37.gydF4y2Ba].值得注意的是,这些历史时期的WGD/WGT事件可能使这些物种最近的共同祖先得以跨越白垩纪-古近纪界线(图。gydF4y2Ba2gydF4y2BaA),正如在许多其他被子植物中证明的那样[gydF4y2Ba41gydF4y2Ba].的基因块比较gydF4y2Ba即trifidagydF4y2Ba与本身也表现出了许多重复的基因对(图gydF4y2Ba1gydF4y2Ba),这是WGT事件的明显结构性证据gydF4y2Ba.gydF4y2Ba我们发现第4、9、13和15号染色体gydF4y2Ba即trifidagydF4y2Ba每个染色体显然都有来自其他染色体的两个额外的副同源片段(图。gydF4y2Ba2gydF4y2Bac).这一结果表明,发生了一个WGT事件gydF4y2Ba番薯gydF4y2Ba基因组,而不是报告的WGD [gydF4y2Ba37.gydF4y2Ba].为了证实这一WGT事件,我们进一步比较了gydF4y2Ba即trifidagydF4y2Ba与gydF4y2BaCoffea canephoragydF4y2Ba,除年龄较大者外,没有谱系特异性WGD/WGT事件gydF4y2BaγgydF4y2Ba最犹太病的共同祖先的基因组三倍[gydF4y2Ba42gydF4y2Ba].正如所料,结构分析表明gydF4y2Bac . canephoragydF4y2Ba区域倾向于对应三个部分gydF4y2Ba即trifida。gydF4y2Ba更具体地说,是1号染色体gydF4y2Bac . canephoragydF4y2Ba有,9染色体7和11同线一关系gydF4y2Ba即trifidagydF4y2Ba的第4号染色体gydF4y2Bac . canephoragydF4y2Ba具有与染色体5,12和13的同步关系gydF4y2Ba即trifidagydF4y2Ba(无花果。gydF4y2Ba2gydF4y2Bac)gydF4y2Ba.gydF4y2Ba此外,共同承保gydF4y2Bac . canephoragydF4y2Ba和gydF4y2Ba即trifidagydF4y2Ba在1:3对应关系下达到最大值。同样的,比较gydF4y2Ba即trifidagydF4y2Ba和gydF4y2Ba葡萄gydF4y2Ba,基因组覆盖率最高,为93.2%gydF4y2Ba即trifidagydF4y2Ba95.8%的人gydF4y2Ba诉酿酒用葡萄gydF4y2Ba在3:1的比率,这也支持上述WGT分析的结果(附加文件gydF4y2Ba2gydF4y2Ba: S18表美国)。gydF4y2Ba
基因家族的扩大和缩小gydF4y2Ba即trifidagydF4y2Ba使用CAFE识别[gydF4y2Ba43gydF4y2Ba中)gydF4y2Ba即trifidagydF4y2Ba和四种非SR形成物种,包括gydF4y2Ba答:芥gydF4y2Ba,gydF4y2Ba美国lycopersicumgydF4y2Ba,gydF4y2Bac . canephoragydF4y2Ba和gydF4y2Ba即零。gydF4y2Ba在22个途径中富集了910个扩增基因家族(gydF4y2BapgydF4y2Bavalue< 0.05). Notably, some of these families have been reported to have functions in carbohydrate metabolism, for example, glycosaminoglycan degradation, carbon metabolism, and carbon fixation in photosynthetic organisms (Additional file2gydF4y2Ba:表S19)。在这些扩增的基因家族中,基于功能注释,我们发现其中一些基因可能对SR的发展很重要,如gydF4y2Baβ-淀粉酶gydF4y2Ba基因(gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba44gydF4y2Ba],gydF4y2BaMADS盒gydF4y2Ba基因(gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba],乙烯响应转录因子(gydF4y2Ba小块土地gydF4y2Ba)基因[gydF4y2Ba47gydF4y2Ba],gydF4y2Ba苷酶gydF4y2Ba基因(gydF4y2Ba48gydF4y2Ba,与iaa有关的基因[gydF4y2Ba49gydF4y2Ba,gydF4y2Ba50gydF4y2Ba和赤霉酸(GA)相关基因gydF4y2BaGA20oxgydF4y2Ba赤霉素调节蛋白[gydF4y2Ba51gydF4y2Ba] (无花果。gydF4y2Ba2gydF4y2Bad).此外,还有100个浓缩基因家族富集在15个KEGG通路中,如植物与病原体的相互作用、ABC转运体、抗坏血酸和醛达酸代谢(Additional file)gydF4y2Ba2gydF4y2Ba:表S20)。gydF4y2Ba
SR发育关键基因的鉴定gydF4y2Ba
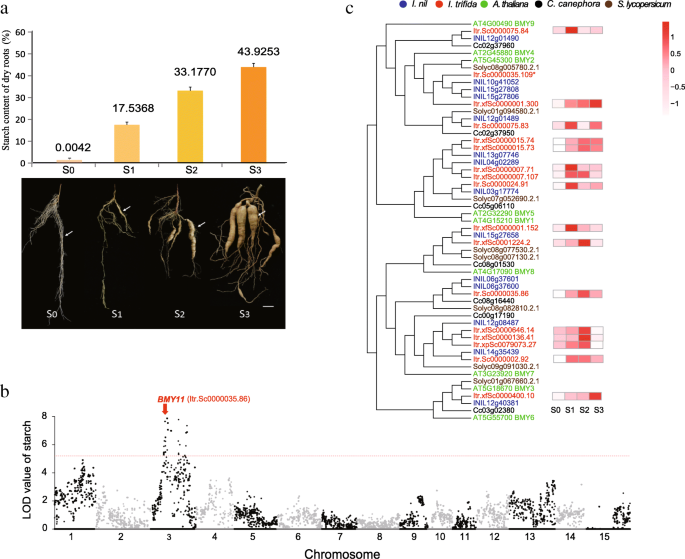
测定了4个典型时期的根直径和淀粉含量gydF4y2Ba即trifidagydF4y2Ba老的发展。不定根(AR, S0)淀粉含量为0.0042%;在初始贮藏根(ISR, S1)中迅速增长至17.54%,在成熟贮藏根(MSR, S3)中迅速增长至43.93%(图4)。gydF4y2Ba3.gydF4y2Baa).根直径从AR增加到20 mm以上(附加文件)gydF4y2Ba1gydF4y2Ba:图S1)。这些结果表明gydF4y2Ba即trifidagydF4y2BaSR发展紧密联系在一起淀粉积累有关。因此,我们使用RNA-seq的数据进行比较基因表达(附加文件gydF4y2Ba2gydF4y2Ba:从S0,S1,S2和S3样本中的表S21)以识别SR开发中的关键基因(附加文件gydF4y2Ba1gydF4y2Ba:图S13A)。比较S0,我们分别识别了S1,S2和S3中的211,718和791个上调基因(附加文件gydF4y2Ba1gydF4y2Ba:图S13c)。值得注意的是,淀粉代谢KEGG途径和蔗糖代谢KEGG途径在三个上调基因组中分别富集最多(附加文件)gydF4y2Ba2gydF4y2Ba:表S22)。我们发现涉及淀粉新陈代谢的几个关键基因,包括gydF4y2Baβ-淀粉酶gydF4y2Ba,gydF4y2BaAGPasegydF4y2Ba(ADP葡萄糖焦磷酸化酶),gydF4y2Ba瑞士gydF4y2Ba(可溶性淀粉合酶),gydF4y2BaSBEgydF4y2Ba(淀粉分支酶)和gydF4y2BagbsgydF4y2Ba(颗粒结合淀粉合酶),与S0相比,在S1,S2和S3之间的一组或两组中上调(附加文件gydF4y2Ba1gydF4y2Ba:图S14系列)。此外,在SR发育过程中有109个常见的上调基因(附加文件)gydF4y2Ba1gydF4y2Ba:图S13c)。在这些基因中,属于淀粉代谢途径的八个基因[gydF4y2Ba52gydF4y2Ba,gydF4y2Ba53gydF4y2Ba),包括gydF4y2BaGPTgydF4y2Ba(Glucose-6-phosphate转运蛋白)gydF4y2Ba53gydF4y2Ba,gydF4y2Ba54gydF4y2Ba,gydF4y2Ba55gydF4y2Ba],gydF4y2Ba的PGMgydF4y2Ba(Phosphoglucomutase),gydF4y2BaISAgydF4y2Ba(异淀粉酶)gydF4y2BaSPgydF4y2Ba(淀粉磷酸化酶),gydF4y2BaDPEPgydF4y2Ba(4-α-葡聚糖转移酶)和3gydF4y2BaBeta-amylasegydF4y2Ba基因(额外的文件gydF4y2Ba1gydF4y2Ba:图S14系列)。gydF4y2Ba诺克斯gydF4y2Ba和gydF4y2BaMIKC般的gydF4y2Ba(II型gydF4y2BaMADS盒gydF4y2Ba)基因[gydF4y2Ba45gydF4y2Ba,gydF4y2Ba46gydF4y2Ba,gydF4y2Ba56gydF4y2Ba在109个基因中也发现了。值得注意的是,在这109个基因中,我们还发现了两种特异性蛋白sporamin的高表达(RPKM = 32145 ~ 113739)gydF4y2Ba1gydF4y2Ba:图S15,附加文件gydF4y2Ba2gydF4y2Ba:表S23)。孢粉素是甘薯SR中主要的贮藏蛋白,占可溶性总蛋白的60 ~ 80%,其表达已被证明主要与SR有关[gydF4y2Ba57gydF4y2Ba,gydF4y2Ba58gydF4y2Ba].该甘薯贮藏蛋白的高表达gydF4y2Ba即trifidagydF4y2Ba表明甘薯孢子素可能遗传自gydF4y2Ba即trifida。gydF4y2Ba此外,差异下调基因的KEGG富集显示,与S0相比,S1、S2和S3富集的前两个途径是苯丙氨酸代谢和苯丙氨酸生物合成(Additional file)gydF4y2Ba1gydF4y2Ba:图S13b);我们发现木质素生物合成的关键基因,gydF4y2BaC4HgydF4y2Ba(coumarate 4-hydroxylase)和gydF4y2BaF5HgydF4y2Ba(Ferulate - 5-羟基化酶)[gydF4y2Ba59gydF4y2Ba],从ARs肿胀到ISRs表达下调,并在SRs增大过程中保持低表达(附加文件gydF4y2Ba1gydF4y2Ba:图S16,附加文件gydF4y2Ba2gydF4y2Ba:表S24)。这些结果表明,淀粉积累是负木质素形成相关,并且可以通过一些代谢途径来调节。gydF4y2Ba
鉴定gydF4y2BaBMY11gydF4y2Ba.gydF4y2Ba一个gydF4y2Ba上图显示了SR发育的四个典型阶段的淀粉含量。下图显示了SR发展的四个典型阶段(白色箭头)。S0,不定根(AR);S1,初始化存储根(ISR);S2:幼嫩存储根(YSR);S3,成熟存储根(MSR)。条= 2厘米。gydF4y2BabgydF4y2BaBMY11gydF4y2Ba(gydF4y2BaItr.Sc0000035.86gydF4y2Ba)位于3号染色体上。gydF4y2BacgydF4y2Ba系统发育树gydF4y2Baβ-淀粉酶gydF4y2Ba基因从五个品种。不同的颜色代表不同的物种。的17的右侧示出了表达热图gydF4y2Baβ-淀粉酶gydF4y2Ba基因的gydF4y2Ba即trifidagydF4y2Ba.* The heatmaps of genes with RPKM = 0 were not shown due to invalid calculation
为了进一步研究响应SR淀粉积累的关键基因,我们使用了200个真正的F1个体的干燥根部的淀粉含量和两个父母进行了GBS遗传图谱的QTL分析(附加文件gydF4y2Ba1gydF4y2Ba中:图S17和S18)。我们得到5点的QTL,其均位于3号染色体,包括39个基因(附加文件gydF4y2Ba2gydF4y2Ba:表S25)。其中四个上调了,一个是gydF4y2BaBMY11gydF4y2Ba(Itr.Sc0000035.86,同一性47.12%至gydF4y2Baβ-淀粉酶1gydF4y2Ba,命名gydF4y2BaBMY11gydF4y2Ba) (无花果。gydF4y2Ba3.gydF4y2Bab).值得注意的是,在4个上调基因中,gydF4y2Baβ-淀粉酶gydF4y2Ba在基因组扩增基因家族和差异上调基因中均发现上述基因。我们检查了这个基因家族的所有成员gydF4y2Ba即trifidagydF4y2Ba和其他四种植物。gydF4y2Ba一,零gydF4y2Ba,gydF4y2Ba美国lycopersicumgydF4y2Ba,gydF4y2Bac . canephoragydF4y2Ba,gydF4y2Ba答:芥gydF4y2Ba,并进行比较。我们发现gydF4y2Baβ-淀粉酶gydF4y2Ba基因数量gydF4y2Ba即trifidagydF4y2Ba高于非sr -形成植物(附加文件gydF4y2Ba1gydF4y2Ba:图S19)。在gydF4y2Baβ-淀粉酶gydF4y2Ba系统发育树,gydF4y2Ba即trifidagydF4y2Ba拥有相同或更多数量的gydF4y2Baβ-淀粉酶gydF4y2Ba基因成员比gydF4y2Ba一,零gydF4y2Ba人希望两个分支,这是进化枝gydF4y2BaBMY11gydF4y2Ba包含两个gydF4y2Ba一,零gydF4y2Ba基因(INIL06g37601和INIL06g37600),由于其特定的串联重复(这两个基因之间的距离是3350个碱基),并且含有三个进化枝gydF4y2Ba一,零gydF4y2Ba基因(INIL15g27806, INIL15g27808和INIL10g41052)gydF4y2Ba3.gydF4y2BaC)。gydF4y2Ba
BMY11gydF4y2Ba和老的发展gydF4y2Ba
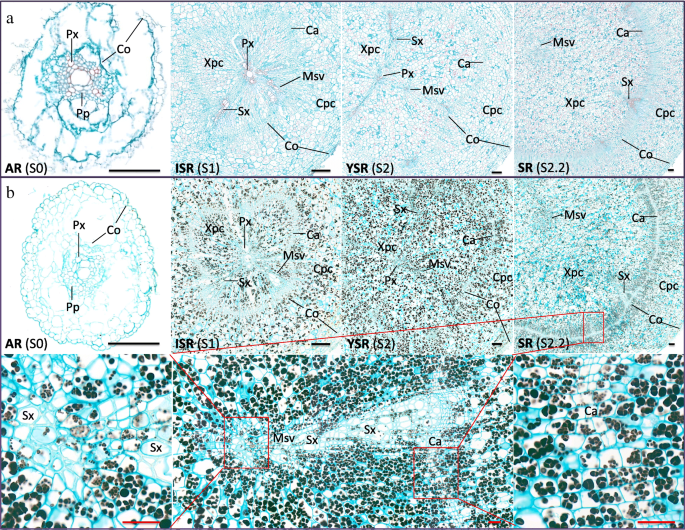
β -淀粉酶的作用是分解淀粉,促进谷物萌发、幼苗生长、胚乳发育和对非生物胁迫的响应[gydF4y2Ba60gydF4y2Ba,所以令人惊讶的是gydF4y2Baβ-淀粉酶gydF4y2Ba基因,包括gydF4y2BaBMY11gydF4y2Ba,在Y22 SR发育过程中上调(图。gydF4y2Ba3.gydF4y2Bab, c)虽然淀粉最终在SR中积累(图。gydF4y2Ba3.gydF4y2Ba我们推测gydF4y2BaBMY11gydF4y2Ba可能在SR开发中发挥特殊作用。为了调查这种作用,我们采样了Y22 SR,将它们切成横向切片并进一步将它们分成五个部分。这五部分的QPCR结果表明gydF4y2BaBMY11gydF4y2Ba在整个横向SR部分中表达(附加文件gydF4y2Ba1gydF4y2Ba:图S20)。据报道,β -淀粉酶位于薄壁细胞中,并伴有淀粉颗粒[gydF4y2Ba60gydF4y2Ba,gydF4y2Ba61gydF4y2Ba].为了进一步研究gydF4y2BaBMY11gydF4y2Ba在淀粉合成中,我们使用S0,S1,S2和S2.2的根(S2和S3之间的阶段2.2,更靠近S2),用于淀粉染色。我们发现,在粘顶鸟和围绕血管(MSV)附近的新细胞中的淀粉颗粒较小,并且比远离这两个组织的细胞中的那些较小,并且更加众多(图。gydF4y2Ba4gydF4y2Ba).结合以上结果,我们推导出gydF4y2BaBMY11gydF4y2Ba可以分裂细胞中较小的淀粉颗粒,然后淀粉合酶可以立即回收它们合成较大的淀粉颗粒。此外,这种分裂还可能以降解产物的形式促进淀粉在细胞之间的转运,再次循环这些降解产物形成更大的淀粉颗粒。这些降解和回收过程是同时发生的;因此,淀粉含量在SR发育过程中逐渐增加,并逐渐促进SR溶胀(图2)。gydF4y2Ba3.gydF4y2Ba然而,的表达gydF4y2BaBMY11gydF4y2Ba大豆的淀粉积累量明显低于大豆,表明大豆的淀粉积累量明显低于大豆gydF4y2BaBMY11gydF4y2Ba主要是降解淀粉,在SR中不能高度表达;如果没有,过度降解将减少淀粉累积并引导根部形成到PR或FR(附加文件gydF4y2Ba1gydF4y2Ba:图S20)。gydF4y2Ba
SR发育不同阶段的解剖结构。gydF4y2Ba一个gydF4y2Ba不同发育阶段SR的解剖结构。藏红花素-不褪色绿染色显示角质细胞壁为红色,纤维素细胞壁为绿色。AR、ISR、YSR和SR (S2.2)的直径分别为0.7 mm、2.6 mm、5.4 mm和11.2 mm。Px,原生木质部;有限公司,皮层;Pp、初生韧皮部;Xpc:木质部薄壁组织细胞;Sx,次生木质部;Msv:围绕导管的分生组织;Ca,形成层; Cpc, cortex parenchyma cell.bgydF4y2Ba上面的图像显示了淀粉颗粒在SR生长的不同阶段的碘-钾染色。蓝黑色和棕黑色的圆点代表淀粉颗粒。下面的图像显示部分放大显微图:中间显示一个图像形成层附近的组织和血管周围分生组织在SR,左边显示了一个放大的图像周围分生组织血管的中心,和右边显示了形成层的放大图像。黑色条= 200 μm,红色条= 50 μmgydF4y2Ba
我们发现使用微阵列杂交在SWERPOTATO VAR的SR发育过程中发现了一种同源转录物(同一性= 98.85%)。广舒87(附加档案gydF4y2Ba2gydF4y2Ba:表S26) [gydF4y2Ba6gydF4y2Ba].一个gydF4y2BaBMY11gydF4y2Ba(gene ID: TU52177, identity = 99.39%)在单倍型解析甘薯组装中[gydF4y2Ba11.gydF4y2Ba].通过对甘薯品种徐薯18的全长转录本鉴定,我们发现gydF4y2BaBMY11gydF4y2Ba可能存在于栽培甘薯基因组的多个拷贝中(附加文件gydF4y2Ba2gydF4y2Ba:表S27)。与Y22类似,的表达式gydF4y2BaBMY11gydF4y2BaPR和FR均高于SR,说明保持适当的活性有利于SR的肿胀。根据qPCR结果,表达gydF4y2BaBMY11gydF4y2Ba徐书18的横向SR剖面上明显低于Y22 SR(附加文件)gydF4y2Ba1gydF4y2Ba:图S20)。这些表达差异表明gydF4y2BaBMY11gydF4y2Ba可能有利于增加淀粉的积累,有助于栽培甘薯的快速SR膨大和更大的块根形成。在马铃薯中也发现了类似的表达模式,即gydF4y2Baβ-淀粉酶gydF4y2Ba一倍的单倍体(来自原始南美品种)块茎的活性比二倍体养殖线(更紧密地类似于商业栽培的四倍体马铃薯)块茎为5至10倍。gydF4y2Ba3.gydF4y2Ba].类似的现象可能发生由于β-淀粉酶基因家族自然进化和该基因的相对较低的表达可能是块根作物至关重要。gydF4y2Ba
讨论gydF4y2Ba
在这项研究中,我们利用了一种综合策略来克服高杂合子的问题,并提供比以前的调查结果更高的质量更高的染色体级参考基因组序列[gydF4y2Ba24.gydF4y2Ba]最近释放的基因组[gydF4y2Ba28.gydF4y2Ba,可作为二倍体的较好参考gydF4y2Ba即trifidagydF4y2Ba.组装的基因组使我们能够在该物种中表征基因组特征,并将基因组与其他公开的植物基因组进行比较,例如培养的甘薯及其密切相关的物种,gydF4y2Ba一,零gydF4y2Ba[gydF4y2Ba11.gydF4y2Ba,gydF4y2Ba37.gydF4y2Ba].例如,gydF4y2Ba即trifidagydF4y2Ba重复序列比gydF4y2Ba一,零gydF4y2Ba并显示出更有效地消除LTR反转录转座子,导致更小的基因组。利用新的证据,我们澄清了两者gydF4y2Ba即trifidagydF4y2Ba和gydF4y2Ba一,零gydF4y2Ba经历了WGT,而不是报道WGD [gydF4y2Ba37.gydF4y2Ba早在他们分道扬镳之前。此外,植物中自然发生的水平基因转移(HGT)的报道很少,但有二倍体gydF4y2Ba即trifidagydF4y2Ba(包括Y22, Contig_2131, identity = 92.66%)通常包含gydF4y2BaIB.gydF4y2BaT-DNA2 [gydF4y2Ba62gydF4y2Ba].这结果表明二倍体gydF4y2Ba即trifidagydF4y2Ba作为一种天然转基因植物,可能已经存在至少近1.3 MYA [gydF4y2Ba11.gydF4y2Ba].甘薯还包含gydF4y2BaIB.gydF4y2BaT-DNA2 [gydF4y2Ba62gydF4y2Ba,gydF4y2Ba63gydF4y2Ba];因此,该序列可能是从二倍体遗传而来gydF4y2Ba即trifidagydF4y2Ba.所有这些发现使我们能够更好地理解基因组的进化gydF4y2Ba即trifidagydF4y2BaY22的高品质基因组对甘薯和甘薯属的基因组进化研究具有重要意义gydF4y2Ba番薯gydF4y2Ba一般来说。gydF4y2Ba
在属gydF4y2Ba番薯gydF4y2Ba,甘普托是最重要的作物之一,全球年产量超过1亿吨。通过将基于基因和甜味育素之间的基因和序列联合锚定,通过将75.7%的支架锚固至15个伪染色体产生释放的单倍型分离基因组。gydF4y2Ba一,零gydF4y2Ba[gydF4y2Ba11.gydF4y2Ba].该压缩单倍体(~ 836 Mb)也被认为是研究甘薯染色体序列组成复杂性的宝贵资源[gydF4y2Ba39.gydF4y2Ba],利用长读测序技术、Hi-C测序结合新型装配算法对甘薯参考序列进行改进[gydF4y2Ba11.gydF4y2Ba,gydF4y2Ba39.gydF4y2Ba,gydF4y2Ba64gydF4y2Ba].六倍体基因组很难组装,阻碍了基因组水平上的遗传研究;找到一个合适的基因研究模型可能是一条捷径。作为最近的野生亲缘和假定的祖先,二倍体gydF4y2Ba即trifidagydF4y2Ba已被认为是SweetPOTATO的模型物种,包括遗传,细胞学和生理分析[gydF4y2Ba30.gydF4y2Ba].拥有高质量的基因组gydF4y2Ba即trifidagydF4y2Ba(1) Y22可以像甘薯一样移植,并发育一个重要农艺性状(图2)。gydF4y2Ba3.gydF4y2Ba一个,附加文件gydF4y2Ba1gydF4y2Ba:图S1),两个物种的SRs都含有表达的高度特异的蛋白sporamin [gydF4y2Ba57gydF4y2Ba](附加文件gydF4y2Ba1gydF4y2Ba:图S15,附加文件gydF4y2Ba2gydF4y2Ba:表S23);(2)通过对4种典型根的转录组比较分析,发现与甘薯表达模式相似的糖类代谢相关基因的上调和柱状木质素化相关基因的下调在Y22 SRs的发育过程中发挥了关键作用[gydF4y2Ba4gydF4y2Ba,gydF4y2Ba59gydF4y2Ba,gydF4y2Ba65gydF4y2Ba](附加文件gydF4y2Ba1gydF4y2Ba:图S14和S16;附加文件gydF4y2Ba2gydF4y2Ba:表S24);(3)的关键基因gydF4y2BaBMY11gydF4y2Ba通过比较转录组学和QTL定位,发现与SR肿胀有关gydF4y2BaBMY11gydF4y2Ba在sweetpotato中有一个类似的表达式模式(附加文件gydF4y2Ba1gydF4y2Ba:图S20)[gydF4y2Ba61gydF4y2Ba].这些研究成果有助于促进该属植物淀粉生物合成的研究gydF4y2Ba番薯gydF4y2Ba和二倍体gydF4y2Ba即trifidagydF4y2Bavar. Y22可以作为甘薯SR发育研究的理想模型。gydF4y2Ba
结论gydF4y2Ba
我们获得了sr形成二倍体的染色体级基因组序列gydF4y2Ba即trifidagydF4y2Ba通过整合全基因组shotgun reads、单分子长reads (PacBio RS II)和GBS基因图谱,获得高杂合度(2.20%)的var. Y22;这个基因组提供了一个更好的基因组特征的分辨率。比较基因组学分析表明,在二倍体中发生了全基因组三倍增殖事件gydF4y2Ba番薯gydF4y2Ba基因组,可以更好地了解这个物种的基因组进化。我们发现了关键基因gydF4y2BaBMY11gydF4y2Ba(身份47.12%至gydF4y2Baβ-淀粉酶1gydF4y2Ba结合基因家族扩展分析、QTL定位、基因差异表达谱分析、SRs形态结构分析和qPCR分析,推测SRs可能有助于淀粉积累和SR发育gydF4y2BaBMY11gydF4y2Ba.甘薯SR发育基因可以在gydF4y2Ba即trifidagydF4y2Ba这些基因具有相似的功能和模式,显示出二倍体gydF4y2Ba即trifidagydF4y2Bavar. Y22是研究甘薯SR发育的理想模型。gydF4y2Ba
方法gydF4y2Ba
植物材料和测序gydF4y2Ba
Y22(附加文件gydF4y2Ba1gydF4y2Ba:图S1a)是二倍体的克隆gydF4y2Ba即trifidagydF4y2Ba(CIP没有种子。:PC98_1 (698014), female parent 2X P96124.5, male parent PC) with good SR-forming characteristics. Y25 (Additional file1gydF4y2Ba:图S1b中)是二倍体的克隆gydF4y2Ba即trifidagydF4y2Ba(CIP没有种子。:69615.3.,female parent 2X 6.1, male parent OP) that does not form SR (Additional file1gydF4y2Ba:图S1)。徐薯18是我国著名的栽培甘薯品种。材料在四川省农业科学院生物技术与核技术研究所实验温室内种植,采用蚊帐和黄粘纸严格防治。二倍体F1遗传群体gydF4y2Ba即trifidagydF4y2Ba由Y25(母本)和Y22(父本)构建。gydF4y2Ba
用Y22进行全基因组测序和组装。利用Qiagen DNeasy Plant Mini Kit (Qiagen, Valencia, CA, USA)从叶片中分离出高质量的基因组DNA。首先,按照制造商的说明,使用NEBNext Ultra DNA Library Prep Kit构建PE文库,插入尺寸分别为230 bp、350 bp和500 bp。然后,通过creo - lox重组循环生成插入大小为2 Kb、5 Kb、10 Kb和20 Kb的配对文库[gydF4y2Ba66gydF4y2Ba].合成长读是由分子化学产生的。以上文库均置于Illumina HiSeq 2500平台上,生成pe2 × 125 bp reads。其次,使用带有PacBio P6-C4化学的RS II平台来生成单分子长reads。gydF4y2Ba
对于淀粉含量的检测和转录组测序,我们把岩屑Y22和四川AAS,海南省三亚市,中国南方试验站(SES)移植他们的沙质土壤。The four typical stages of SR development were AR, ISR (diameter = ~ 2 mm), YSR (5–8 mm) and MSR (>20 mm). To examine these stages, the roots were sampled at approximately 25 (S0), 50 (S1), 85 (S2) and 120 (S3) days after transplantation (DAT) (Fig.3.gydF4y2Ba一种)。每个样本由三个重复组成。根据中文测试标准NY / T11-1985测试淀粉含量。使用Trizol试剂(Invitrogen)提取总RNA,并用无RNase的DNase I(USA)处理。使用NEBNext Ultra RNA Library Prep Kit (NEB, USA)生成相应的RNA-seq文库。然后,所有库都被Illumina Hiseq Xten平台对测序。gydF4y2Ba
对于基于标签的测序,FgydF4y2Ba1gydF4y2Ba种植人口(Y25×Y22)种植在中国三亚的四川AAS的SES。从个体F的新鲜叶子中提取基因组DNAgydF4y2Ba1gydF4y2Ba使用DNA提取试剂盒(天根生物技术有限公司,北京)和溶解在1× TE缓冲液(10 mM Tris-HCl和1 mM EDTA, pH 8.0)中。这些人是从FgydF4y2Ba1gydF4y2Ba利用SSR (simple sequence repeat, SSR)标记进行群体鉴定;SSRs是根据组装的Y22基因组用Perl脚本设计的(附加文件gydF4y2Ba1gydF4y2Ba:图S4和附加文件gydF4y2Ba2gydF4y2Ba:表S5)。根据参考方法共制备了202个GBS库,包括2个亲本库和200个F1个体库[gydF4y2Ba32.gydF4y2Ba].最后,利用Illumina HiSeq Xten平台对所选标签进行PE测序。gydF4y2Ba
为了进行徐书18转录组测序,我们采集了花、茎、叶和SRs样本,用TRIzol试剂(Invitrogen)提取总RNA,用RNase-free DNase I (Promega, USA)处理。RNA浓度由Nanodrop (Thermo Scientific)测定,将不同的RNA按相同比例混合,构建RNA-seq库。使用NEBNext Ultra RNA Library Prep Kit (NEB, USA)生成相应的RNA-seq文库。然后,在Illumina HiSeq 2500和PacBio RS II平台上对文库进行测序。gydF4y2Ba
基因组组装和假染色体的构建gydF4y2Ba
为了获得高质量的reads,我们首先过滤了以下所有reads:(a)包含' N '的reads占核苷酸的10%以上;(b)读取包含适配器序列的数据;(c) PCR扩增产生的重复序列。在质量控制之后,所有的PE和配对读数都用Platanus软件组装起来[gydF4y2Ba67gydF4y2Ba],使用默认参数。其次,Illumina Moleculo合成长读取被用来执行间隙填充程序PBJelly [gydF4y2Ba68gydF4y2Ba],使用默认参数。第三,使用PacBio单分子长reads进一步延伸序列连续性。第四,HaploMerger [gydF4y2Ba69gydF4y2Ba]的使用以减少由杂合序列的冗余。gydF4y2Ba
以组装好的Y22支架作为参考基因组,对Y22、Y25及其子代进行snp(单核苷酸多态性)鉴定。使用GATK对所有示例执行变体调用[gydF4y2Ba70gydF4y2Ba] 软件。The parental polymorphic markers were classified into eight segregation patterns (ab × cd, ef × eg, hk × hk, lm × ll, nn × np, aa × bb, ab × cc and cc × ab). For the F1gydF4y2Ba人口,隔离模式被选择为遗传图谱[gydF4y2Ba71gydF4y2Ba].前映射结构,用隔离失真的标记(gydF4y2BapgydF4y2Ba< 0.05)、完整性(> 75%)或异常碱基被过滤。使用内部脚本将其余标记转换为bin标记。使用JoinMap 4.0软件根据物理位置构建连杆组[gydF4y2Ba72gydF4y2Ba,最小LOD值为5.0,最大复合量为45%。由于目前的连锁分析涉及到大量分离的SNP位点,因此利用Kosambi作图功能将重组频率转换为作图距离。每个连杆组的最终标记顺序由软件程序RECORD[验证gydF4y2Ba73gydF4y2Ba].亲本Y25和Y22的平均测序深度分别为29.11×和24.68×,高于后代(平均11.77×)。Y25和Y22分别检测到489,692和178,112个SNP位点。最终图谱由15个连锁群的6306个bin标记组成,包含15526个SNPs,跨越3156.55 cM(附加文件)gydF4y2Ba1gydF4y2Ba:图S5和附加文件gydF4y2Ba2gydF4y2Ba:表S6)。SNP标记间的平均遗传距离为0.50 cM。然后,利用构建的连锁图谱,根据标记的位置将支架固定在假染色体上(附加文件)gydF4y2Ba1gydF4y2Ba:图S6)。gydF4y2Ba
装配验证gydF4y2Ba
为了评估组装的基因组序列的准确性,我们选择了小片段文库reads,并使用BWA软件[gydF4y2Ba74gydF4y2Ba将读取到的信息映射到组装好的基因组上。随后,我们计算了作图率、覆盖度和基因组深度。为了评估组装中的基因结构完整性,转录本由Trinity组装[gydF4y2Ba33.gydF4y2Ba-ss 0.5 -jc 0,−minkmercov 2 -minglue 2。利用BLAT将EST序列与组装的基因组进行比对[gydF4y2Ba75gydF4y2Ba],使用默认参数。核心真核基因定位方法[gydF4y2Ba35.gydF4y2Ba]被用来定义也被用来评估在最后组装的基因序列的完整性248个保守的基因。gydF4y2Ba
基因组注释gydF4y2Ba
重复序列注释方法分为同源序列比对和从头计算两种。同源序列比对方法基于重复序列数据库Repbase [gydF4y2Ba76gydF4y2Ba].我们使用了RepeatMasker和RepeatProteinMask [gydF4y2Ba77gydF4y2Ba]鉴定与已知的重复序列类似的序列。我们也使用了ltr_finder [gydF4y2Ba78gydF4y2Ba],堆垛机[gydF4y2Ba79gydF4y2Ba], RepeatScout [gydF4y2Ba80gydF4y2Ba]和RepeatModeler (gydF4y2Bahttp://www.repeatmasker.org/RepeatModeler/gydF4y2Ba)构建de novo重复数据库。然后,我们使用了重复播放器[gydF4y2Ba77gydF4y2Ba],根据已建立的重复序列数据库来识别重复序列。gydF4y2Ba
基因预测是基于从头预测,基于同源性的预测和RNA-seq的预测的整合。基因结构从头预测进行了使用奥古斯[gydF4y2Ba81gydF4y2Ba], GlimmerHMM [gydF4y2Ba82gydF4y2Ba基因检测),(gydF4y2Ba83gydF4y2Ba],基因[gydF4y2Ba84gydF4y2Ba]和SNAP [gydF4y2Ba85gydF4y2Ba] 软件。基于同源性的预测包括来自密切相关或模型物种的基于蛋白质的同源性。总共使用11种同源物种,包括gydF4y2Ba拟南芥gydF4y2Ba,gydF4y2Ba茄属植物tuberosumgydF4y2Ba,gydF4y2Ba茄属植物lycopersicumgydF4y2Ba,gydF4y2BaCapsicum Annuum.gydF4y2Ba,gydF4y2Ba芝麻纪录gydF4y2Ba,gydF4y2Ba甜菜属gydF4y2Ba,gydF4y2Ba葡萄gydF4y2Ba,gydF4y2Ba木薯耐gydF4y2Ba,gydF4y2Ba莲属椰子。gydF4y2Ba,gydF4y2Ba萝卜gydF4y2Ba和gydF4y2Ba茄属植物pennellii。gydF4y2Ba支持该预测的其他证据包括我们用于将预测的基因结构对准的同源EST或cDNA数据[gydF4y2Ba75gydF4y2Ba].gydF4y2Ba
RNA-seq预测使用实验RNA-seq数据来预测基因。基于上述预测结果,结合转录组比较数据,利用EVM将每种方法预测的基因集整合到一个非冗余系统中[gydF4y2Ba86gydF4y2Ba].我们使用过pasa [gydF4y2Ba86gydF4y2Ba]和转录组装配结果,以修正EVM注释的结果,并添加utr和剪接变异等信息,以获得最终的基因集。利用BLASTP (e值≤1e-5)对InterPro、KEGG、Swiss-Prot和TrEMBL 4个数据库进行最佳匹配,确定基因功能。我们在组装物中识别候选ncrnagydF4y2Ba即trifidagydF4y2Ba通过与已知的ncRNA文库进行比较或结构预测。gydF4y2Ba
系统发育分析gydF4y2Ba
七种植物的蛋白质序列,包括gydF4y2Ba答:芥gydF4y2Ba,gydF4y2Ba羊肚菌gydF4y2Ba,gydF4y2Ba一,零gydF4y2Ba,gydF4y2Bac . canephoragydF4y2Ba,gydF4y2Bao .漂白亚麻纤维卷gydF4y2Ba,gydF4y2Ba美国lycopersicumgydF4y2Ba和gydF4y2Ba马铃薯gydF4y2Ba下载。然后,对每个物种的基因集进行筛选:(a)当一个基因中存在多个转录本时,仅使用编码区域中最长的转录本进行进一步分析;(b)筛选出编码少于30个氨基酸的蛋白质的基因。然后,通过BLASTP对所有物种的蛋白序列进行相似性评价,e值为1e-5。使用OrthoMCL程序将所有7个物种的蛋白质数据集和Y22蛋白质数据集聚为平行和同源数据集[gydF4y2Ba87gydF4y2Ba,其中充气参数为1.5。gydF4y2Ba
在基因家族聚类之后,我们用MUSCLE比对了所有1930个单拷贝基因蛋白序列[gydF4y2Ba88gydF4y2Ba并将所有对齐结果组合起来创建一个对齐超矩阵。然后,利用RAxML构建了8个物种的系统发育树[gydF4y2Ba89gydF4y2Ba]具有最大似然方法,并将引导样本的数量设置为100。gydF4y2Bao .漂白亚麻纤维卷gydF4y2Ba被指定为系统发育树的外群。利用单拷贝基因家族,在PAML中使用MCMCtree进行分化时间估计[gydF4y2Ba90gydF4y2Ba,gydF4y2Ba91gydF4y2Ba)包。MCMCtree运行参数为:burn-in: 5,000,000, sample-number: 1,000,000, sample-frequency: 50。时间修正点为gydF4y2Bao .漂白亚麻纤维卷gydF4y2Ba和gydF4y2Ba答:芥gydF4y2Ba, 140 - 200米娅;gydF4y2Ba答:芥gydF4y2Ba和gydF4y2Ba马铃薯gydF4y2Ba,101-156 mya;gydF4y2Bac . canephoragydF4y2Ba和gydF4y2Ba美国lycopersicumgydF4y2Ba, 83 - 89米娅;gydF4y2Ba马铃薯gydF4y2Ba和gydF4y2Ba美国lycopersicumgydF4y2Ba, 7.2 - -7.4米娅。所有时间校正点均来自时间树网站(gydF4y2Bahttp://www.timetree.org/gydF4y2Ba).gydF4y2Ba
基因组共同和全基因组复制gydF4y2Ba
BLASTP用1E-5的不同物种,包括之间的E值实现gydF4y2Ba即trifidagydF4y2Ba来gydF4y2Ba即trifidagydF4y2Ba,gydF4y2Ba马铃薯gydF4y2Ba来gydF4y2Ba马铃薯gydF4y2Ba,gydF4y2Ba一,零gydF4y2Ba来gydF4y2Ba一,零gydF4y2Ba,gydF4y2Bac . canephoragydF4y2Ba来gydF4y2Bac . canephoragydF4y2Ba,gydF4y2Ba即trifidagydF4y2Ba来gydF4y2Ba马铃薯gydF4y2Ba,gydF4y2Ba即trifidagydF4y2Ba来gydF4y2Ba一,零gydF4y2Ba和gydF4y2Ba一,零gydF4y2Ba来gydF4y2Ba马铃薯gydF4y2Ba.MCscan [gydF4y2Ba92gydF4y2Ba]用于在每个比较对中搜索共线段。然后,从与MUSCLE连接的核苷酸序列中计算出每个比较组的区段中每个基因对的四倍转位(4dTv)比率[gydF4y2Ba88gydF4y2Ba].使用4DTV比分布估计WGD。染色体之间的同胞菌被通过电池可视化。gydF4y2Ba
根系的基因表达剖析gydF4y2Ba
在测序之后,原始的RNA读取被过滤和修剪,以产生干净的读取。所有的RNA序列都被TopHat2定位到Y22基因组[gydF4y2Ba93gydF4y2Ba使用如下参数:——max-intron-length 500,000,−-read-gap-length 10,−-read-edit-dist 15,−-max-insert -length 5和——max- delete -length 5。每个样本的读取计数由HTSeq计算[gydF4y2Ba94gydF4y2Ba].然后,RPKM通过其定义计算(每百万次映射读数读取每千碱基)。使用Deseq定义不同比较组的差异表达基因[gydF4y2Ba95gydF4y2Ba],调整后Pvalue(Padj) < 0.05。gydF4y2Ba
Y22淀粉代谢基因的定义与鉴定gydF4y2Ba
我们用一种联合的方法定义和鉴定了淀粉代谢基因。淀粉代谢基因被定义为参与淀粉合成或淀粉合成途径的上游或下游基因。本研究的关键酶基因是通过以下方法鉴定的:首先,这些基因的蛋白序列gydF4y2Ba答:芥gydF4y2Ba从NCBI下载(gydF4y2Bahttps://www.ncbi.nlm.nih.gov/gydF4y2Ba).经BLASTP鉴定候选基因,e值为1e-5。其次,通过身份筛选候选基因,并去除重叠部分。利用PFAM预测同源物种和Y22候选基因的蛋白结构域(gydF4y2Bahttp://pfam.xfam.org/gydF4y2Ba).只有与同源物种具有相同蛋白质结构域的候选基因被保留下来。通过iTAK软件鉴定转录因子[gydF4y2Ba96gydF4y2Ba].gydF4y2Ba
β -淀粉酶(BMY)基因家族的系统发育重建gydF4y2Ba
用于识别gydF4y2Ba宝宝gydF4y2Ba基因家族成员gydF4y2Ba即trifidagydF4y2Ba,gydF4y2BaI.nil.gydF4y2Ba基因组和其他基因组,这些物种的蛋白质组与已知的β -淀粉酶蛋白质相一致gydF4y2Ba答:芥gydF4y2BaBLASTp。对于每个蛋白,只有e值为1e-5和> 50%的序列被保留。这些基因保留了对反应的排列gydF4y2Ba答:芥gydF4y2Ba进一步需要蛋白质以具有PF01373 PFAM结构域,然后被认为是初始候选者gydF4y2Ba宝宝gydF4y2Ba这些基因组中的基因。为了消除这种人工选择条件的不准确性(识别> 50%可能过于严格gydF4y2Ba即trifidagydF4y2Ba和gydF4y2BaI.nil.gydF4y2Ba),我们重做gydF4y2Ba宝宝gydF4y2Ba对这些物种进行基因鉴定,以取代已知的物种gydF4y2Ba答:芥gydF4y2Baβ -淀粉酶蛋白及其各自的蛋白质序列的初步检测gydF4y2Ba宝宝gydF4y2Ba基因,然后按照同样的过程。然后,所有的保留gydF4y2Ba宝宝gydF4y2Ba基因排列互相用肌肉和进化树gydF4y2Ba宝宝gydF4y2Ba利用RAxML最大似然法构建基因。gydF4y2Ba
QTL映射gydF4y2Ba
200 f1个体的整根挖出并在130日下挖出并洗涤(附加文件gydF4y2Ba1gydF4y2Ba:图肌力表现)。整根切成3mm片或3cm片,80℃干燥至稳定重量。整个根被粉碎,直到所有碎片通过一个0.17 mm的筛网。然后将所有样品送到四川省原子吸收系统分析测定中心,按照中国检测标准NY/ T11-1985进行淀粉检测。干根淀粉含量为13.91 ~ 47.32%gydF4y2Ba1gydF4y2Ba:图S18)。根淀粉内容数据被构建为数据矩阵,用于通过Windows QTL Cartographer V2.5软件用于QTL分析[gydF4y2Ba97gydF4y2Ba].表型变异和QTL检测用多QTL作图模型(MQM)来计算。使用1000个排列的阈值被确定LOD值的用于评估的QTL效应的统计显着性的表型的阈值gydF4y2BapgydF4y2Ba = 0.05. LOD values 3.0 and above were considered to indicate QTL loci.
qPCRgydF4y2Ba
用RNAprep Pure Plant Kit(天根生物技术,北京)分离的总RNA,用oligo (dT)18和随机六聚体引物,用日本Toyobo公司的ReverTra Ace qPCR RT Master Mix合成第一链cDNA。在Bio-Rad iCycler MyiQ real-time PCR系统中,使用稀释的cDNA和SYBR®Green real-time PCR Master Mix (Toyobo, Japan)进行实时定量反转录PCR (qPCR)。qPCR的循环曲线包括一个95°C为30 s的循环,接着是40个95°C为5 s, 58°C为30 s的循环,最后一个熔化曲线曲线(65-95°C)。用2gydF4y2Ba−ΔΔCtgydF4y2Ba方法(附加文件gydF4y2Ba2gydF4y2Ba:表S28)。每个数据点代表平均值的三个重复。gydF4y2Ba
根解剖结构gydF4y2Ba
新鲜根在4%中性缓冲福尔马林中固定48 h,然后脱水,石蜡包埋。8微米的切片被切割并放置在硅烷涂层的载玻片上以固定样品。切片在60℃干燥后脱蜡再水合。切片进行淀粉染色和藏红花耐晒绿染色。淀粉染色用I-KI染色。藏红花素-耐晒绿染色:用藏红花素溶液染色60-120分钟,梯度乙醇染色。切片放入快绿溶液中30-60秒,脱水后用树脂固定。使用3DHISTECH扫描仪(Pannoramic MIDI)收集所有图像,使用CaseViewer软件查看图像数据(gydF4y2Bahttps://www.3dhistech.com/caseviewergydF4y2Ba).gydF4y2Ba
参考gydF4y2Ba
- 1。gydF4y2Ba
Woolfe农协。甘薯:一种未开发的食物资源。剑桥:剑桥大学出版社;1992.gydF4y2Ba
- 2。gydF4y2Ba
Roullier C, Benoit L, McKey DB, Lebot V.历史收集揭示了红薯在大洋洲的扩散模式被现代植物运动和重组所掩盖。中国科学(d辑:地球科学)2013;110(6):2205-10。gydF4y2Ba
- 3.gydF4y2Ba
马铃薯基因组测序联盟。块茎作物马铃薯的基因组序列分析。自然。2011;475(7355):189 - 95。gydF4y2Ba
- 4.gydF4y2Ba
Wang H, Yang J, Zhang M, Fan W, et al ., et al ., Yuan L ., Zhang P., et al ., et al ., et al ., Zhang P., et al ., et al ., et al ., Zhang P., et al ., et al ., et al ., et al ., Zhang P., et al ., et al ., et al ., et al ., et al .。Sci众议员2016;6:18645。gydF4y2Ba
- 5.gydF4y2Ba
关键词:无性系作物,传统育种,标记辅助选择,基因组选择,近亲繁殖,木薯acta photonica sinica . 2015;128(9): 1647-67。gydF4y2Ba
- 6.gydF4y2Ba
王志强,方斌,陈旭,廖敏,张旭,黄磊,罗振华,姚振华,李艳艳。甘薯块根形成与发育相关基因表达的时间模式。BMC Plant Biol. 2015;15:180。gydF4y2Ba
- 7.gydF4y2Ba
Tanaka M.甘薯(Ipomoea Batatas贮藏根部储存根系分子研究的最新进展。JPN Agric Res Q. 2016; 50(4):293-9。gydF4y2Ba
- 8.gydF4y2Ba
叶伟明,陈建军,林密,陈玉梅,林春英。一种具有胰蛋白酶抑制活性的甘薯贮藏蛋白的功能活性。植物学报。1997;33(3):565-70。gydF4y2Ba
- 9.gydF4y2Ba
杨胜,刘旭,乔胜,谭伟,李敏,冯军,张超,康旭,黄涛,朱勇,等。两个甘薯品种间淀粉含量的差异与基因表达的特定变化有关。功能整合基因组学。2018;18(6):613-25。gydF4y2Ba
- 10.gydF4y2Ba
李辉,赵宁,于旭,刘艳,翟辉,何松,李强,马东,刘强。甘薯贮藏根产量qtl的鉴定。Sci Hortic。2014;170:182-8。gydF4y2Ba
- 11.gydF4y2Ba
杨建军,莫因扎德H H H,肖鹏,刘刚,郑军,孙振东,范伟,等。单倍型分解甘薯基因组可追溯其六倍化历史。Nat植物。2017;3(9):696 - 703。gydF4y2Ba
- 12.gydF4y2Ba
关键词:甘薯,bac末端序列,全基因组gydF4y2Ba番薯甘薯gydF4y2Ba(l)Lam)基因组组成。BMC基因组学。2016;17(1):945。gydF4y2Ba
- 13。gydF4y2Ba
甘薯基因组序列分析的挑战。Sci繁殖。2017;67(1):35 - 40。gydF4y2Ba
- 14。gydF4y2Ba
Padma Nimmakayala GV,Reddy UK。CHARPET 7:IPOMOEA。在:Kole C,编辑器。野生作物亲属:基因组和育种资源。DONDRECHT:SPRINGER HEIDELBERG;纽约:斯普林克;2011. p。123。gydF4y2Ba
- 15.gydF4y2Ba
Kriegner A,Cervantes JC,Burg K,Mwanga Ro,张D.甘薯的遗传联系地图[Ipomoea Batatas(L.)林]基于AFLP标记。mol品种。2003; 11(3):169-85。gydF4y2Ba
- 16.gydF4y2Ba
Roullier C, Duputie A, Wennekes P, Benoit L, Fernandez Bringas VM, Rossel G, Tay D, McKey D, Lebot V.解析栽培甘薯的起源(gydF4y2Ba番薯甘薯gydF4y2Ba(l)Lam)。8。2013;(5):e62707。gydF4y2Ba
- 17.gydF4y2Ba
西山。甘薯的进化与驯化。Bot Mag Tokyo. 1971; 84:377-87。gydF4y2Ba
- 18.gydF4y2Ba
王志刚,王志刚。甘薯(Ipomoea batatas (L.) lam)及其亲本的同源倍性研究。Euphytica。1975;24(1975):197 - 208。gydF4y2Ba
- 19.gydF4y2Ba
盐谷I,红薯和六倍体在番薯三裂叶豚草(H. B. K.)唐濑T.基因组结构。日本Ĵ品种。1989; 39:57-66。gydF4y2Ba
- 20。gydF4y2Ba
卢胜,刘强,李伟。甘薯育种。北京:中国农业出版社;1998.gydF4y2Ba
- 21。gydF4y2Ba
冯JY,李毅男,赵S,张成,杨ST,巧S,谭WF,曲HJ,王DY,浦ZG。甘薯的进化和遗传多样性分析及其相关不同的多倍体野生物种gydF4y2Ba即trifidagydF4y2Ba使用rad-seq。BMC植物BIOL。2018; 18(1):181。gydF4y2Ba
- 22.gydF4y2Ba
郜呒,阿苏GM,斯图尔特L,Akwe WA,Njiti V,巴恩斯S.蜡质内含子的变化支持甘薯的原点异源六倍体[gydF4y2Ba番薯甘薯gydF4y2Balam) (l)。Euphytica。2011;177(1):111 - 33所示。gydF4y2Ba
- 23.gydF4y2Ba
Munoz-Rodriguez P, Carruthers T, Wood JRI, Williams BRM, Weitemier K, Kronmiller B, Ellis D, Anglin NL, Longway L, Harris SA,等。在甘薯起源和传播到波利尼西亚的过程中协调相互冲突的系统发育。咕咕叫医学杂志。2018;28 (8):1246 - 1256. - e1212。gydF4y2Ba
- 24.gydF4y2Ba
Hirakawa H,Okada Y,Tabuchi H,Shirasawa K,Watanabe A,Tsuruoka H,Minami C,Nakayama S,Sasamoto S,Kohara M等人。野生马铃薯基因组序列调查,Ipomoea Trifida(H.B.K.)G. Don。DNA Res。2015; 22(2):171-9。gydF4y2Ba
- 25.gydF4y2Ba
小牧K,二倍体番薯三裂叶豚草(H. B. K.)G.唐的片山K.根厚度以及从与甘薯的杂交衍生的子代的表现。品种sci。1999; 49:123-9。gydF4y2Ba
- 26.gydF4y2Ba
奥斯汀DF。番薯综合体-我。分类法。[j] . acta photonica sinica, 2012, 37(2): 429 - 434。gydF4y2Ba
- 27.gydF4y2Ba
Eserman LA, jarrett RL, Leebens-Mack JH。牵牛花(旋花科)贮藏根的平行进化。植物生态学报。2018;18(1):95。gydF4y2Ba
- 28.gydF4y2Ba
吴胜,刘国强,曹强,Hamilton JP,孙辉,周超,Eserman L, Gemenet DC, Olukolu BA, Wang H,等。栽培甘薯的两个二倍体野生近缘种的基因组序列揭示了遗传改良的目标。Nat Commun。2018;9(1):4580。gydF4y2Ba
- 29.gydF4y2Ba
李锐,朱洪,阮建军,方旭东,史志华,李胜,单刚,K K,等。大规模平行短读测序的人类基因组从头组装。基因组研究》2010;20(2):265 - 72。gydF4y2Ba
- 30.gydF4y2Ba
李安,吴志明,周志林,张安,马东风,李亚东,赵大东,唐杰,曹庆华。三叶甘薯基因组Fosmid文库的构建及文库PCR筛选系统的建立。Acta Agriculturae Boreali-Sinica。2014; 29(2): 45 - 50。gydF4y2Ba
- 31.gydF4y2Ba
周Z,张c,周y,郝z,王z,zeng x,di h,li m,张d,yong h等。基于重组自交系的超高密度箱地图玉米植物架构的遗传解剖。BMC基因组学。2016; 17:178。gydF4y2Ba
- 32.gydF4y2Ba
张震,魏涛,钟勇,李旭,黄俊gydF4y2BaZiziphus jujubagydF4y2Ba轧机。通过测序技术进行基因分型。果树基因组。2016;12(4):76。gydF4y2Ba
- 33.gydF4y2Ba
Grabherr MG, Haas BJ, Yassour M, Levin JZ, Thompson DA, Amit I, Adiconis X, Fan L, Raychowdhury R, Zeng Q, et al.;没有参考基因组的RNA-Seq数据的全长度转录组组装。生物科技Nat》。2011;29(7):644 - 52。gydF4y2Ba
- 34.gydF4y2Ba
张克,方X,郭X,李l,罗河,徐f,杨p,张l,王x,qi h等。牡蛎基因组揭示了壳体形成的应力适应和复杂性。自然。2012; 490(7418):49-54。gydF4y2Ba
- 35.gydF4y2Ba
CEGMA:在真核生物基因组中准确注释核心基因的途径。生物信息学。2007;23(9):1061 - 7。gydF4y2Ba
- 36。gydF4y2Ba
Simao FA, Waterhouse RM, Ioannidis P, Kriventseva EV, Zdobnov EM. BUSCO:用单拷贝同源性评估基因组组装和注释完整性。生物信息学,2015,31(19):3210 - 2。gydF4y2Ba
- 37。gydF4y2Ba
星野A, Jayakumar V, Nitasaka E, Toyoda A, Noguchi H, Itoh T, Shin IT, Minakuchi Y, Koda Y, Nagano AJ,等。日本牵牛花的基因组序列与分析。Nat Commun。2016;7:13295。gydF4y2Ba
- 38。gydF4y2Ba
Wang Y, Tang H, Debarry JD, Tan X, Li J, Wang X, Lee TH, Jin H, Marler B, Guo H, et al. .MCScanX:基因共线性和共线性的检测和进化分析工具包。核酸学报2012;40(7):e49。gydF4y2Ba
- 39.gydF4y2Ba
张j,张x,唐h,张q,hua x,ma x,zhu f,琼斯t,zhu x,鲍德·j等。等位基因定义的自动聚合物甘蔗Saccharum Spontaneum L. Nat Genet的基因组。2018; 50(11):1565-73。gydF4y2Ba
- 40。gydF4y2Ba
古代基因组复制的进化意义。[j] .自然科学进展,2009;10(10):725-32。gydF4y2Ba
- 41。gydF4y2Ba
Vanneste K,Maere S,Van de Peer Y.纠结在两者:白垩纪尽头的基因组重复突发以及植物演化的后果。Philos Trans R SoC Lond Ser B Biol Sci。2014; 369(1648年)。gydF4y2Bahttps://doi.org/10.1098/rstb.2013.0353gydF4y2Ba.gydF4y2Ba
- 42。gydF4y2Ba
denoeud f,carretero-paulet l,dereether a,droc g,guyot r,pietrella m,zheng c,alberti a,Anthony F,Aprea G,等。咖啡基因组可以深入了解咖啡因生物合成的会聚演化。科学。2014; 345(6201):1181-4。gydF4y2Ba
- 43。gydF4y2Ba
De Bie T, Cristianini N, Demuth JP, Hahn MW。CAFE:研究基因家族进化的计算工具。生物信息学。2006;22(10):1269 - 71。gydF4y2Ba
- 44。gydF4y2Ba
Monroe JD,Storm Ar。综述:rabidopsisβ-淀粉酶(Bam)基因家族:形式和功能的多样性。植物SCI。2018; 276:163-70。gydF4y2Ba
- 45。gydF4y2Ba
关键词:甘薯,马铃薯,MADS-box 1基因,块根分化安机器人。2008;102(1):57 - 67。gydF4y2Ba
- 46.gydF4y2Ba
Noh Sa,Lee Hs,Huh Ej,Huh Gh,Paek Kh,Shin Js,Bae JM。SRD1通过增强甘薯(Ipomoea Batatas)的甲状腺细胞和钩环细胞的增殖,涉及养猪介导的储存初始增厚生长。J Exp Bot。2010; 61(5):1337-49。gydF4y2Ba
- 47.gydF4y2Ba
徐志生,陈敏,李凌龙,马永忠。AP2/ERF转录因子家族在作物改良中的作用及应用植物学报,2011;53(7):570-85。gydF4y2Ba
- 48.gydF4y2Ba
Goujon T, Minic Z, El Amrani A, Lerouxel O, Aletti E, Lapierre C, Joseleau JP, Jouanin L. AtBXL1是高等植物(拟南芥)推测的β -木糖苷酶基因,参与了次生细胞壁代谢和植物发育。植物j . 2003; 33(4): 677 - 90。gydF4y2Ba
- 49.gydF4y2Ba
Chapman EJ,Estelle M.植物中疾病调控基因表达的机制。Annu Rev Genet。2009; 43:265-85。gydF4y2Ba
- 50.gydF4y2Ba
沙洛QL,刘S,皮尼亚斯NM,田H,OsIAA9的王S.组成型表达影响淀粉颗粒的积累和在拟南芥根向地响应。前植物SCI。2015; 6:1156。gydF4y2Ba
- 51.gydF4y2Ba
王光良,熊芳,阙芳,徐志生,王芳,熊安生。形态特征、解剖结构和基因表达:胡萝卜生长发育过程中赤霉素生物合成和感知的新见解。Hortic杂志2015;2:15028。gydF4y2Ba
- 52.gydF4y2Ba
Nakkanong K, Yang JH, Zhang MF。新型种间自交系及其亲本果实发育过程中淀粉积累和淀粉相关基因表达的研究Sci Hortic。2012;136:1-8。gydF4y2Ba
- 53.gydF4y2Ba
McGregor CE, He L, Ali RM, Sosinski B, Jankowicz J, Burg K, LaBonte DR. .复制数和图像分析方法对甘薯的影响[J]gydF4y2Ba番薯甘薯gydF4y2Ba(l)林。]cDNA microarray results. Plant Mol. Biol. Rep. 2005;23: 367-381.
- 54.gydF4y2Ba
karabian - masouleh A, Waters DL, Reinke RF, Ward R, Henry RJ。与水稻营养和功能特性相关的淀粉生物合成基因的SNP。Sci众议员2012;2:557。gydF4y2Ba
- 55.gydF4y2Ba
Dyson BC, Allwood JW, Feil R, Xu Y, Miller M, Bowsher CG, Goodacre R, Lunn JE, Johnson GN。拟南芥代谢对光的适应:葡萄糖6-磷酸/磷酸转运子GPT2指导代谢适应。植物学报。2015;38(7):1404-17。gydF4y2Ba
- 56.gydF4y2Ba
田中,加藤,中山,中谷,高田。甘薯贮藏根中I类结1样同源盒基因的表达。acta botanica sinica(云南植物学报),2008;gydF4y2Ba
- 57.gydF4y2Ba
陈志强,陈志强,陈志强,等。甘薯芽孢素与抗逆境相关的生物学功能研究。Biotechnol放置2012;30(6):1309 - 17所示。gydF4y2Ba
- 58.gydF4y2Ba
块茎贮藏蛋白。安机器人。2003;91(7):755 - 69。gydF4y2Ba
- 59.gydF4y2Ba
史锐,孙永华,李强,何柏生,塞德罗夫,蒋维林。杨木素生物合成的系统方法:单木质素生物合成基因的转录丰度和特异性。植物生理学报。2010;51(1):144-63。gydF4y2Ba
- 60.gydF4y2Ba
Srivastava G,Kayastha am。来自三角形Foenum-Graecum的淀粉种子的β-淀粉酶及其发芽种子的定位。Plos一个。2014; 9(2):E88697。gydF4y2Ba
- 61。gydF4y2Ba
秦勇,王勇,段春青,张德平。在甘薯块根发育过程中,β-淀粉酶主要定位于质体。植物学报,2003;45(5):581-8。gydF4y2Ba
- 62。gydF4y2Ba
金恩德特,翟海,刘强。栽培甘薯的基因组包含有表达基因的农杆菌t - dna:自然转基因粮食作物的一个例子。中国科学院院刊2015;112(18):5844-9。gydF4y2Ba
- 63。gydF4y2Ba
Quispe-Huamanquispe DG, geysen G, Kreuze JF。水平基因转移有助于植物进化:以农杆菌t - dna为例。植物学报2017;8:2015。gydF4y2Ba
- 64。gydF4y2Ba
Zimin AV, Puiu D, Luo MC, Zhu T, Koren S, Marcais G, Yorke JA, Dvorak J, Salzberg SL.基于MaSuRCA超读算法的面包小麦先祖Aegilops tauschii大重复基因组杂交组装。基因组研究》2017;27(5):787 - 92。gydF4y2Ba
- 65。gydF4y2Ba
张凯,吴志刚,唐东,罗凯,陆辉,董军,王昕,吕超,王军,等。比较转录组分析揭示了蔗糖代谢相关酶在甘薯贮藏根淀粉积累中的关键作用。植物学报2017;8:914。gydF4y2Ba
- 66。gydF4y2Ba
陈志强,陈志强,陈志强,等。重组腺病毒载体的构建。J微生物学报。1997;7(3):1842 - 9。gydF4y2Ba
- 67。gydF4y2Ba
Kajitani R, Toshimoto K, Noguchi H, Toyoda A, Ogura Y, Okuno M, Yabana M, Harada M, Nagayasu E, Maruyama H,等。高杂合子基因组的高效从头组装全基因组猎枪短读。基因组研究》2014;24(8):1384 - 95。gydF4y2Ba
- 68。gydF4y2Ba
英语交流,理查兹S,韩Y,王梅,V型V,瞿Ĵ,秦X,Muzny DM,里德JG,沃利KC等。注意差距:与太平洋生物科学RS长读测序技术升级的基因组。Plos一个。2012; 7(11):e47768。gydF4y2Ba
- 69。gydF4y2Ba
黄松,陈志,黄国光,杨鹏,李军,傅莹,袁胜,陈松,徐安华。HaploMerger:重组多态性二倍体基因组的等位基因。基因组研究》2012;22(8):1581 - 8。gydF4y2Ba
- 70.gydF4y2Ba
Van Tassell CP, Smith TP, Matukumalli LK, Taylor JF, Schnabel RD, Lawley CT, Haudenschild CD, Moore SS, Warren WC, Sonstegard TS.通过深度测序来发现SNP和估计等位基因频率。Nat方法。2008;5(3):247 - 52。gydF4y2Ba
- 71.gydF4y2Ba
王志强,王志强,王志强,等。基于radon的海胆连锁图谱及比较基因组学研究。BMC基因组学。2013;14:32。gydF4y2Ba
- 72.gydF4y2Ba
凡Ooijen JW。在全同胞家系的异交种的多点最大似然映射。遗传学水库。2011; 93(5):343-9。gydF4y2Ba
- 73.gydF4y2Ba
Van Os H, Stam P, Visser RG, Van Eck HJ。记录:一种在遗传连锁图上排序位点的新方法。应用计算机学报。2005;112(1):30-40。gydF4y2Ba
- 74.gydF4y2Ba
李H,德宾R.快速并用Burrows-Wheeler变换精确的短读对齐。生物信息学。2009; 25(14):1754至1760年。gydF4y2Ba
- 75.gydF4y2Ba
与核功能相关的新蛋白结构域家族的系统鉴定。基因组研究》2002;12(1):47-56。gydF4y2Ba
- 76.gydF4y2Ba
Jurka J, Kapitonov VV, Pavlicek A, Klonowski P, Kohany O, Walichiewicz J. Repbase update,真核重复元素数据库。细胞基因组Res. 2005;110(1-4): 462-7。gydF4y2Ba
- 77.gydF4y2Ba
Tarailo-Graovac M,Chen N.使用regectmasker识别基因组序列中的重复元素。Curr Protoc生物信息学。2009;第4章:4.10单元。gydF4y2Ba
- 78.gydF4y2Ba
XU Z,Wang H.LTR_Finder:一种有效的工具,用于预测全长LTR回收输送。核酸RES。2007; 35(Web服务器):W265-8。gydF4y2Ba
- 79.gydF4y2Ba
作者简介:王志强,男,博士,主要研究方向为基因组重复序列的识别与分类。生物信息学。2005;21 (1):i152。gydF4y2Ba
- 80.gydF4y2Ba
Price AL, Jones NC, Pevzner PA。大基因组中重复家族的从头鉴定。生物信息学。2005;21 (1):i351-8。gydF4y2Ba
- 81.gydF4y2Ba
基于广义隐马尔可夫模型的真核生物基因预测。BMC生物信息学。2006;7:62。gydF4y2Ba
- 82.gydF4y2Ba
Majoros WH, Pertea M, Salzberg SL. TigrScan和GlimmerHMM:两个开源从头开始的真核生物基因发现者。生物信息学,2004;20(16):2878 - 9。gydF4y2Ba
- 83.gydF4y2Ba
Salamov AA, Solovyev VV。在果蝇基因组DNA中发现从头基因。基因组研究》2000;10(4):516 - 22所示。gydF4y2Ba
- 84.gydF4y2Ba
Blanco E,Parra G,Guigór.使用基因鉴定基因。Curr Protoc生物信息学。2007;第4章:第4.3章。gydF4y2Ba
- 85。gydF4y2Ba
Korf I.基因在新型基因组中发现。BMC生物信息学。2004; 5:59。gydF4y2Ba
- 86。gydF4y2Ba
哈斯BJ,Salzberg SL,朱W,Pertea男,艾伦JE,奥维斯Ĵ,白O,比尔CR,Wortman JR。自动使用EVidenceModeler和装配拼接比对的程序的真核基因结构注释。基因组Biol。2008; 9(1):R7。gydF4y2Ba
- 87。gydF4y2Ba
李L, Stoeckert CJ Jr, Roos DS。OrthoMCL:真核生物基因组同源组的鉴定。基因组研究》2003;13(9):2178 - 89。gydF4y2Ba
- 88。gydF4y2Ba
Edgar Rc。肌肉:高精度和高吞吐量的多个序列对齐。核酸RES。2004; 32(5):1792-7。gydF4y2Ba
- 89。gydF4y2Ba
Stamatakis A. RAxML-VI-HPC:成千上万的类群和混合模型的最大可能基于-系统发育分析。生物信息学。2006; 22(21):2688-90。gydF4y2Ba
- 90.gydF4y2Ba
杨志。PAML:基于最大似然法的系统发育分析程序包。计算机应用生物科学。1997;13(5):555-6。gydF4y2Ba
- 91.gydF4y2Ba
杨志。PAML 4:最大似然法的系统发育分析。中国生物医学工程学报。2007;24(8):1586-91。gydF4y2Ba
- 92.gydF4y2Ba
唐华,王旭,Bowers JE, Ming R, Alam M, Paterson AH。通过多重排列的被子植物基因图谱揭示古代的六倍性。基因组研究》2008;18(12):1944 - 54。gydF4y2Ba
- 93.gydF4y2Ba
金d,Pertea G,Trapnell C,皮门特尔H,凯利R,Salzberg SL。TopHat2:在插入,缺失和基因融合的存在转录组的准确的对准。基因组Biol。2013; 14(4):R36。gydF4y2Ba
- 94.gydF4y2Ba
Anders S, Pyl PT, Huber W. HTSeq—一个用于处理高通量测序数据的Python框架。生物信息学,2015,31(2):166 - 9。gydF4y2Ba
- 95.gydF4y2Ba
安德斯S,用于顺序计数数据胡伯W.差异表达分析。基因组Biol。2010; 11(10):R106。gydF4y2Ba
- 96.gydF4y2Ba
Zheng Y Y, Jiao C, Sun H, Rosli HG, Pombo MA, Zhang P, Banf M, Dai X, Martin GB, Giovannoni JJ, et al. iTAK:一种植物转录因子、转录调节因子和蛋白激酶的全基因组预测和分类程序。摩尔。2016;9(12):1667 - 70。gydF4y2Ba
- 97.gydF4y2Ba
王S,巴斯滕CJ,曾ZB。的Windows QTL制图V2.5_011。罗利:统计,北卡罗莱纳州立大学的系;2012年(gydF4y2Bahttp://statgen.ncsu.edu/qtlcart/wqtlcart.htm.gydF4y2Ba)gydF4y2Ba
确认gydF4y2Ba
我们非常感谢裴燕教授和向苏琼教授(西南大学,中国)分享gydF4y2Ba即trifidagydF4y2Ba感谢万建民教授(中国农业科学院)、任航兴副教授(中国重庆畜牧科学院)和蔡浩阳教授(四川大学生命科学学院)的宝贵意见。gydF4y2Ba
资金gydF4y2Ba
这项工作是由中国(31471448,31101119,91417305,31470342),国家自然科学基金,为中国农业研究系统(CARS-11-B04,CARS-10-B5),中国四川金融创新工程,专项资金支持(2015LWJJ-001,2016QNJJ-001,2016ZYPZ-005,2015KXJJ-002,2015JSCX-007),四川养殖合作项目,中国(2016NYZ0032,2016NZ0103)和中国国家重点基础研究发展计划(973计划)(2015CB150100).gydF4y2Ba
数据和材料的可用性gydF4y2Ba
在当前研究期间生成和分析的数据集可在BioProject Number下的国家生物技术信息(NCBI)数据库中提供:Prjna362521。植物材料可从通讯作者获得。生成的数据集和从GB和F的表型分析gydF4y2Ba1gydF4y2Ba由于这些数据在进行另一项研究的过程中,目前的研究期间的人口还不能公开获得,但可以通过通讯作者的合理要求获得。gydF4y2Ba
作者信息gydF4y2Ba
隶属关系gydF4y2Ba
贡献gydF4y2Ba
ML和HL设计了这项工作。ML管理这个项目。YL、HY、AL、WS和SQ制备植株,构建遗传群体,检测表型。WT、FL、SY和HZ分离核酸并鉴定FgydF4y2Ba1gydF4y2Ba人群。LZ和JL进行测序。JF、ZP、ZW和ZZ对其遗传图谱和qtl进行分析。WT、WX、ZP和CW的基因组序列组装分析。WX、MP、CD和KW进行了基因预测、功能注释和比较基因组学分析。ML、MP、WX、YZ进行比较转录组分析。CZ、YL、ZP、HZ进行基因家族分析、基因表达分析。ML撰写稿件,HL和JZ修改稿件。所有作者阅读并批准了最终的手稿。gydF4y2Ba
相应的作者gydF4y2Ba
伦理宣言gydF4y2Ba
伦理批准和同意参与gydF4y2Ba
不适用。gydF4y2Ba
同意出版gydF4y2Ba
不适用。gydF4y2Ba
相互竞争的利益gydF4y2Ba
两位作者宣称他们没有相互竞争的利益。gydF4y2Ba
出版商的注意gydF4y2Ba
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。gydF4y2Ba
附加文件gydF4y2Ba
额外的文件1:gydF4y2Ba
图S1。gydF4y2Ba二倍体gydF4y2BaIpomoea Trifida.gydF4y2Ba和地瓜。(a) Y22整株,显示贮藏根(SR)形成。Y22是一个二倍体无性系gydF4y2Ba即trifidagydF4y2Ba种子(CIP编号PC98_1(698014),母本2X P96124.5,父本PC)。(b) Y25整株,不形成SRs。Y25是一个二倍体无性系gydF4y2Ba即trifidagydF4y2Ba种子(CIP编号:696153,母本2X 6.1,父本OP)。(c) Y25的铅笔根(PR)和Y22的铅笔根(SR)的横向和纵向切片;Y22 SR. (d) F1代(克隆3-11)也具有较强的SR发育。(e)甘薯品种徐薯22的SR。秤栏:2厘米。gydF4y2Ba图S2。gydF4y2Ba利用K-mer分析方法估算基因组大小gydF4y2Ba即trifidagydF4y2Ba.K = 17。x轴表示深度,y轴表示每个深度的频率。gydF4y2Ba图S3。gydF4y2Ba使用拟合曲线进行杂合子评估。浅蓝色曲线与基因组的杂合子一致。因此,杂合子为2.20%。gydF4y2Ba图S4。gydF4y2Ba真F1个体的SSR鉴定。M代表记号笔。Y25为母本,Y22为父本。3-11、1-6、2-6、4-1、2-3、2-7为F1个体。在电泳谱带中,在Y25谱带的基础上存在的Y22谱带均为真正的杂种。gydF4y2Ba图S5。gydF4y2Ba高密度的基因图谱gydF4y2Ba即trifidagydF4y2Ba.gydF4y2Ba图S6。gydF4y2Ba的15个假染色体gydF4y2Ba即trifidagydF4y2Ba.根据构建的连锁图谱中的标记位置,将支架固定在假染色体上。蓝色的柱子代表染色体,每个染色体都由多个支架组成。绿色的柱子代表15个连锁组,灰色的线将连锁组的标记与染色体上的物理位置连接起来。gydF4y2Ba图S7。gydF4y2Ba用10k不重叠滑动窗口计算GC含量和平均层序深度。x轴表示GC内容,y轴表示10-kb非重叠滑动窗口的平均深度。右边的直方图表示平均深度分布,上面的直方图表示GC含量分布gydF4y2Ba即trifidagydF4y2Ba基因组。gydF4y2Ba图S8。gydF4y2Ba的GC含量和平均序列深度gydF4y2Ba即trifidagydF4y2Ba基因组用10k非重叠滑动窗口计算。X轴表示GC含量,Y轴表示GC含量的密度。gydF4y2Ba图S9。gydF4y2Ba基因组证据支持的统计数据。的蓝色圆圈代表29728个基因的预测从头,淡蓝色圆圈表示24109组的基因通过RNA证据预测,红色圆圈表示25618种的基因来自七个物种,包括通过同源性预测gydF4y2Ba拟南芥gydF4y2Ba,gydF4y2Ba甜菜属gydF4y2Ba,gydF4y2BaCapsicum Annuum.gydF4y2Ba,gydF4y2Ba芝麻纪录gydF4y2Ba,gydF4y2Ba茄属植物lycopersicumgydF4y2Ba,gydF4y2Ba茄属植物tuberosumgydF4y2Ba和gydF4y2Ba葡萄gydF4y2Ba.gydF4y2Ba图S10。gydF4y2BaLTR-RTs的插入时间分布gydF4y2Ba即trifidagydF4y2Ba和gydF4y2Ba一,零gydF4y2Ba.Y轴表示LTR-RTS的副本数,X轴表示LTR-RTS的插入时间。我们使用肌肉在5'和3'LTR之间进行对齐(V3.8.31,gydF4y2Bahttp://www.drive5.com/muscle.gydF4y2Ba).LTR插入时间(T)计算公式为T = k/2r(每年每个位点LTR /替代之间的差异,r = 1.05E-8)。gydF4y2Ba图S11。gydF4y2Ba共线块之间gydF4y2Ba即trifidagydF4y2Ba和gydF4y2Ba一,零gydF4y2Ba.不同的颜色代表不同的染色体。gydF4y2Ba图S12。gydF4y2Ba共线块之间gydF4y2Ba即trifidagydF4y2Ba和haplotype-resolvedgydF4y2Ba一、甘薯gydF4y2Ba基因组。不同的颜色代表不同的染色体。Itr:gydF4y2Ba即trifidagydF4y2Ba,IB:gydF4y2Ba甘薯。gydF4y2Ba图向。gydF4y2Ba当比较组S1 VS S0,S2 VS S0和S3 VS S0时差异表达基因编号的VENN图。(a)所有差异表达基因的Venn图,包括上调和下调的基因。(b)差异下调基因的Venn图。(c)三个比较组中的差异上调基因的Venn图。使用DESEQ具有乳头J <0.05的差异上调基因。第109号表示所有组共同的差异上调的基因。gydF4y2Ba图S14系列。gydF4y2BaSR开发和响应基因调控gydF4y2Ba即trifidagydF4y2Ba.淀粉合成途径(a)的模型表示109倍通常上调的基因。每个基因旁边的小维恩图表示在这三个比较组的不同基因上调号码。gydF4y2BaGPTgydF4y2Ba, Glucose-6-phosphate转运蛋白;gydF4y2Ba的PGMgydF4y2BaPhosphoglucomutase;gydF4y2BaSBEgydF4y2Ba,淀粉分支酶;gydF4y2BaISAgydF4y2Ba异淀粉酶;gydF4y2BaAGPasegydF4y2BaADP葡萄糖焦磷酸化酶;SSS:可溶性淀粉合成酶;gydF4y2BaSPgydF4y2Ba、淀粉磷酸化酶;gydF4y2BagbsgydF4y2Ba,颗粒结合淀粉合酶;gydF4y2BaSuSgydF4y2Ba蔗糖合酶;gydF4y2BaDPEPgydF4y2Ba,4-α-葡聚糖转移酶;gydF4y2Baβ-淀粉酶gydF4y2Ba,gydF4y2Baβ-淀粉酶。gydF4y2Ba对四种典型根源的比较转录组分析提供了强有力的证据表明,涉及碳水化合物代谢的基因的上调和涉及体内斑点的人的下调,一种类似于甘薯的表达模式,在Y22 SR的发育中发挥了关键作用。(B)淀粉合成途径基因的表达热图。gydF4y2Ba图S15。gydF4y2Ba在Y22的SR 2个sporamins的RPKM值。gydF4y2Ba图S16。gydF4y2Ba的木质素合成的基因的热图gydF4y2Ba.gydF4y2Ba在木质素合成途径中鉴定了10个基因gydF4y2Ba即trifidagydF4y2Ba.gydF4y2Ba朋友gydF4y2Ba苯丙氨酸ammonia-lyase;gydF4y2Ba摘要:gydF4y2Ba, 4-Coumarate 3-hydroxylase;gydF4y2BaC4HgydF4y2BaCoumarate-4-hydroxylase;gydF4y2BaCAD.gydF4y2Ba,肉桂醇脱氢酶;gydF4y2BaCCRgydF4y2BaCinnamoyl-CoA还原酶;gydF4y2BaCCoAOMTgydF4y2Ba, Caffeoyl-CoAgydF4y2BaOgydF4y2Ba甲基转移酶;gydF4y2BaCOMT的gydF4y2Ba,咖啡酸/ 5- hydroxyconiferaldehydeö-methyltransferase;gydF4y2BaF5HgydF4y2Ba,配合5-羟化酶;gydF4y2Ba4CL.gydF4y2Ba, 4-香豆酸:辅酶a连接酶;gydF4y2BaHCTgydF4y2Ba,gydF4y2BapgydF4y2Ba-hydroxycinnamoyl-CoA:奎尼酸shikimategydF4y2BapgydF4y2Ba-hydroxycinnamoyltransferase。gydF4y2Ba图S17。gydF4y2Ba代表F1个人。3-11有典型的SR。1-6,2-6和4-1具有增厚的根(Sr或Sr样;一些根的Xylem是部分萎靡的)。2-3有铅笔根(PR)。2-7有纤维根(FR)。秤栏:2厘米。gydF4y2Ba图S18。gydF4y2Ba干根淀粉含量的频率分布。13≤16表示淀粉含量高于13%且小于或等于16%;在整个X轴标签中类似地使用≤符号。gydF4y2Ba图S19。gydF4y2BaWbeta-淀粉酶gydF4y2Ba五个物种的基因数量。gydF4y2Ba图S20。gydF4y2Ba表达gydF4y2BaBMY11gydF4y2Ba在SR。(a)SR的横截面gydF4y2Ba即trifidagydF4y2Bavar。Y22。所述SR进行切片横向并进一步分成五个部分:部分1是皮质包括表皮(SC1)的外部分,部分2包括在木质部(SC2)的皮质和最外部分的内部部分,部分3为木质部(SC3)的外侧部分,部分4是木质部(SC4)的中间部分,和部分5是木质部(SC5)的内部部分。PR和FR作为对照。(b)由甘薯VAR的SR的横切面。Xushu18。该SR被切开的(a)。B一个r = 10 mm.(c)gydF4y2Ba中存在的结果gydF4y2BaBMY11gydF4y2Ba在Y22和徐书18的横断面上。多克斯(5745 kb)gydF4y2Ba
额外的文件2:gydF4y2Ba
表S1。gydF4y2Ba调查统计结果。gydF4y2Ba表S2。gydF4y2Ba测序数据统计。gydF4y2Ba表S3。gydF4y2Ba仅使用Illumina测序数据统计装配结果。gydF4y2Ba表S4。gydF4y2Ba使用PacBio RS II数据进行扩展后装配结果的统计。gydF4y2Ba表S5。gydF4y2BaSSR引物用于真F1杂种的鉴定。gydF4y2Ba表S6。gydF4y2Ba遗传连锁群的统计信息。gydF4y2Ba表S7。gydF4y2Ba保险覆盖范围的统计数字gydF4y2Ba即trifidagydF4y2Ba基因组。gydF4y2Ba表S8。gydF4y2BaEST序列评价结果。gydF4y2Ba表S9。gydF4y2Ba毛皮评估结果。gydF4y2Ba表S10。gydF4y2Ba用于注释的RNA-SEQ数据。gydF4y2Ba表S11。gydF4y2Ba基因功能注释的统计结果。gydF4y2Ba表S12。gydF4y2Ba非编码RNA的统计结果。gydF4y2Ba表S13。gydF4y2Ba重复分类的统计结果。gydF4y2Ba表S14系列。gydF4y2Ba本研究中使用的植物物种和组装/基因模型的概述。gydF4y2Ba表S15。gydF4y2Ba比较之间的重复内容gydF4y2Ba即trifidagydF4y2Ba和gydF4y2Ba一,零gydF4y2Ba.gydF4y2Ba表S16。gydF4y2BaLTR号码的统计数据。gydF4y2Ba表S17。gydF4y2Ba同步块的长度和块重复序列。gydF4y2Ba表S18。gydF4y2BaY22染色体重复试验结果。gydF4y2Ba表S19。gydF4y2BaKEGG富集结果为基因扩增gydF4y2Ba即trifidagydF4y2Ba.gydF4y2BaS20表。gydF4y2BaKEGG富集结果在签约的基因gydF4y2Ba即trifidagydF4y2Ba.gydF4y2Ba表S21。gydF4y2BaS0 ~ S3 RNA测序数据统计gydF4y2BaS22表。gydF4y2BaKegg对上调基因的途径。gydF4y2Ba表S23。gydF4y2Ba芽孢素是一种特异的蛋白gydF4y2Ba即trifidagydF4y2Ba集会。gydF4y2Ba表S24。gydF4y2Ba下调基因的KEGG通路。gydF4y2Ba表S25。gydF4y2BaQTL定位的结果。gydF4y2Ba表S26。gydF4y2BaSWERPORATION瓦斯塔托瓦斯的SR发育过程中BMY11微阵列杂交的结果。广舒87。gydF4y2Ba表S27。gydF4y2Ba爆炸的结果gydF4y2BaBMY11gydF4y2Ba在甘薯的全长转录物中。Xushu18。gydF4y2Ba表S28。gydF4y2Ba用于放大的QPCR引物gydF4y2BaBMY11gydF4y2Ba.多克斯(75 kb)gydF4y2Ba
权利和权限gydF4y2Ba
开放访问gydF4y2Ba本文遵循知识共享署名4.0国际许可协议(gydF4y2Bahttp://creativecommons.org/licenses/by/4.0/gydF4y2Ba),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(gydF4y2Bahttp://creativecommons.org/publicdomain/zero/1.0/gydF4y2Ba)适用于本条提供的数据,除非另有说明。gydF4y2Ba
关于这篇文章gydF4y2Ba
引用这篇文章gydF4y2Ba
李,M.,杨,S.,徐伟。gydF4y2Baet al。gydF4y2Ba野生甘薯(gydF4y2BaIpomoea Trifida.gydF4y2Ba)基因组提供了对贮藏根发育的深入了解。gydF4y2BaBMC植物杂志gydF4y2Ba19,gydF4y2Ba119(2019)。https://doi.org/10.1186/s12870-019-1708-z.gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
迪伊gydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/s12870-019-1708-z.gydF4y2Ba
关键字gydF4y2Ba
- Ipomoea Trifida.gydF4y2Ba基因组gydF4y2Ba
- 地瓜gydF4y2Ba
- 进化gydF4y2Ba
- 存储根发展gydF4y2Ba
- QTLgydF4y2Ba
- BMY11gydF4y2Ba(gydF4y2Baβ-淀粉酶gydF4y2Ba)gydF4y2Ba