摘要
背景
荞麦(Fagopyrum esculentumMoench。)是源于中国南方的年度作物。营养种子用于烹饪,如谷物谷物。荞麦是一种具有异常自我不相容性的突出物种,由于其二态(即短和长型)花和变形内的不孕症。花卉形态和变形内的不相容性都是由单一的S.轨迹。植物短风花是杂合的(S / S)和长风花的植物是纯合的隐性(s / s)在这个地方,和S / S未发现基因型。最近,我们建立了荞麦基因组组装草案,并确定了5.4 mb长S.- 短方形植物覆盖的植物。
在这项研究中,关于荞麦基因组多样性的第一报告,我们使用了基因分型逐个测序(GBS)DataSet来评估全球耕种荞麦体型内的基因组核苷酸多样性。我们还调查了该效用S.荞麦系统发育分析的-等位基因区。
结果
根据全基因组简单核苷酸多态性(SNP)分析,荞麦具有0.0065的核苷酸多样性,可与其他异交植物相媲美。基于全基因组SNPs的系统发育分析表明,栽培荞麦分为亚洲组和欧洲组,欧洲组的核苷酸多样性较低(0.0055),亚洲组和欧洲组之间的分化较低。
单核苷酸多态性(0.0039)S.-等位基因区域低于全基因组SNPs。基于该区域的系统发育分析发现了三个分离的群体,S-1、S-2和S-3。
结论
利用GBS数据集检测到的SNPs可以有效地阐明荞麦的进化史,并得出以下结论:(1)欧洲类群全基因组核苷酸多样性低,亚洲类群和欧洲类群间分化低,提示了亚洲向欧洲扩散和/或欧洲近期强化栽培和选择相关的遗传瓶颈;(2)企业多元化程度高S.-等位基因区域表明基因从野生荞麦流向栽培荞麦,表明栽培荞麦可能有多个起源。
背景
荞麦(Fagopyrum esculentumMoench。2N= 2X = 16), a member of the Polygonaceae family, is an annual crop that originated in southern China [1那2].种子(严格的酸味)用作米饭(奥雅萨苜蓿)及小麦(小麦);因为荞麦不属于Poaceae家族,所以它通常被称为伪谷物。荞麦具有优异的栽培性能,具有较短的日益增长的时期和凉爽的气候和高海拔的耐受性。因此,荞麦在欧亚亚洲的温带区中被广泛栽培,并且用于许多传统食品,如荞麦面(日本面条),Memic Guksu(韩国面条),Pizzoccheri(意大利面食)和Galettes(法国煎饼)。荞麦种子在淀粉和高质量的蛋白质中致密,具有良好平衡的氨基酸组合物[3.],也是膳食纤维、微量元素和酚类化合物的重要来源[4.那5.].由于其高营养含量和缺乏麸质,荞麦现在在欧亚大陆的地区广泛种植,包括在美国,加拿大,澳大利亚和新西兰。
为了鉴定农艺上有用的基因,已经使用各种分子标记构建了荞麦键,包括同工酶变异[6.],简单的序列重复(SSR)[7.[扩增的片段长度多态性(AFLP)[8.].近年来,人们建立了普通荞麦的微阵列标记系统和全基因组连锁图谱[9.].他们后来证实,利用全基因组微阵列标记进行基因组选择是提高荞麦产量的有效方法[10.].此外,我们课题组构建了荞麦基因组数据库(BGDB) [11.].使用BGDB,已经确定了各种农艺上有用的基因,例如控制类黄酮生物合成和编码2S白蛋白型过敏原和颗粒结合淀粉合酶(GBSSS)的那些。11.那12.那13.那14.].因此,荞麦育种的遗传工具已经高度发达。
遗传多样性评价是探索作物中农艺有用基因的关键步骤。荞麦是一种异态自交不亲和(SI)的异态种,这是由于其花的二态性(即短型和长型花),每个花与同一形态的花不亲和,但跨形态的花亲和[15.那16.].花卉形态和变形内的不相容性既通过单一遗传轨迹决定,S..植物短风花是杂合的(S / S),长花柱的则为纯合隐性(s / s)在这个地点[15.那17.].因此,像其他异交作物如玉米(Zea Mays.),荞麦预计将保持大量的内饰多样性。实际上,同工酶分析表明荞麦具有良好的遗传多样性,与野生植物物种的卵巢相当18.];栽培群体的杂合度高于原始物种的野生群体,f . esculentumSSP。抱怨产于中国南方。随后的AFLP和SSR分析表明f . esculentumSSP。抱怨由两种远端相关的系统发育组组成,藏族和云南 - 四川群体,栽培荞麦地区属于西藏组[1那19.].然而,这些分析仅基于DNA条带状图案的相似性,以及每个核苷酸位点的平均核苷酸差异(即核苷酸多样性)[20.]没有估计。迄今为止,没有关于基因组核苷酸多样性的信息f . esculentum可用。因此,对研究核苷酸多样性很有兴趣f . esculentum使用较新的和卓越的基因组工具。
最近,我们建立了荞麦基因组组装草案,然后应用基因分型测序技术(GBS) [21.到一群来自世界各地的荞麦地方品种[11.].使用受限制酶分散的扩增基因组DNA序列的GBS方法变得越来越受到检测大量的SNP [22.].我们使用GRAF GRAZEMOMES总成作为GBS标记的参考,并成功确定了S-等位基因地区,由332个脚手架组成,包括5.4 MBP [11.].该区域包含了在短型植物中绘制GBS读数而在长型植物中未绘制GBS读数的位置,并且有两个位置S.-allele-specific基因,S-ELF3和SSG2这种基因只存在于短株植物的基因组中。因此,短花柱和长花柱的基因型分别为半合子和零纯合子[15.].这类似于人类性染色体基因的情况;y-染色体基因的嗜态态度证明了一种用于澄清人文发育结构的有用特征[23.].因此,我们预测DNA序列的比较S.-等位基因区是研究荞麦地方品种间系统发育关系的有力工具。
在这项研究中,我们利用已发表的GBS数据评估了世界各地栽培荞麦地方品种的全基因组核苷酸多样性。这是第一个关于全基因组核苷酸多样性的报告f . esculentum.我们讨论的效用S.- 荞麦物种的系统发育分析的区域,并展示了从野生植物中流出的可能基因流动从多样化S.等位基因区域。
结果
整体SNP检测和核苷酸多样性
我们平均每株植物获得720万读(对应于726.1 MBP)。过滤由GBS检测到46个荞麦植物检测到的位点后,我们保留了7,154,454个位点,对应于参考基因组的0.61%,以进一步分析(表1).在7154,454个位点中,我们检测到255,517个SNP位点,SNP密度为0.036(每28 bp一个SNP)。我们比较了短株和长株之间的定位位点数量、SNP密度和核苷酸多样性,没有发现显著差异(见表2)1).
种群结构和系统发育分析
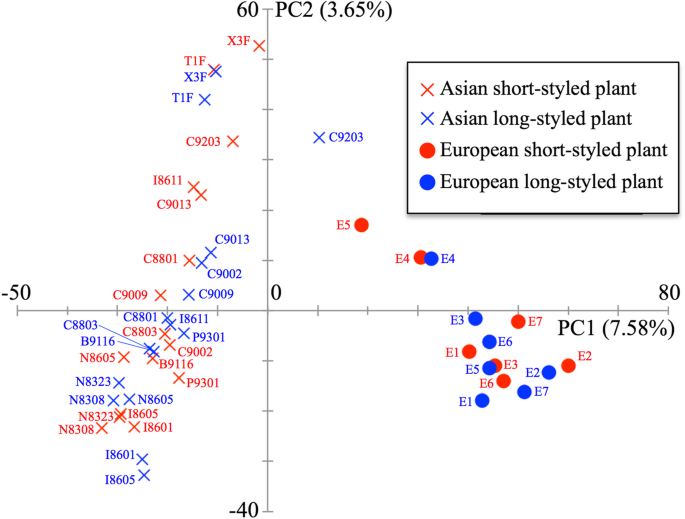
通过计算荞麦地方品种间的遗传距离,利用NJ方法构建系统发育树,共选取7154,454个位点,包含255,517个SNP位点。NJ株系显示,46个品种大致分为两组,亚洲(32个品种)和欧洲(14个品种)。1).栽培荞麦之间的系统发育关系与地理分布良好相关;来自一个国家的样本在NJ树(例如,对于尼泊尔的I8601和I8605,对于尼泊尔N8323和N8605,以及日本的T1F和X1F),来自NJ树(例如,I8601和I 8605,以及日本的T1F和X1F)中的样本。PCA图还表明,46种品种分别主要分为由亚洲和欧洲品种组成的两组(图。2).
为了明确荞麦种群结构,我们对46 GBS中分离的255,517个snp进行了混合分析。交叉验证误差在K.= 1,逐渐增大为K.增加(附加文件1:图S1)。在K.= 2时,交叉验证误差也较低,说明最优祖先种群数量为K.= 1或K.= 2.Although the lowest cross-validation error was actually observed atK. = 1, the NJ tree and PCA implied that there were two groups (one including only Asian landraces and the other only European landraces), indicating thatK.= 2适合将46个普通荞麦地方品种分组。以后我们把这两个集团称为亚洲集团和欧洲集团。
为了比较亚洲和欧洲群体之间的核苷酸多样性,我们鉴定了SNP,并计算了每组的基因组平均核苷酸多样性(表1).欧洲组的SNP密度远低于亚洲组(0.034)。欧洲(0.0055)的核苷酸多样性也比亚洲(0.065)组更低。然而,即使NJ树和PCA图表明有两组,亚洲和欧洲品种之间的FST值不是很高(FST = 0.068)。
S型脚手架的多样性
我们之前确认了S.- 由包含332个脚手架组成的区域,包括5,393,196磅,映射了短风格特定的网站[11.].为了分析该地区的核苷酸多样性,我们在23个短风景植物中鉴定了这332个支架上的SNP。从60,108个地点(332支架的总长度的0.11%),我们检测到1123个SNP。SNP密度(0.019)和遗传多样性(0.0039)S.-等位基因支架低于所有基因组支架1).
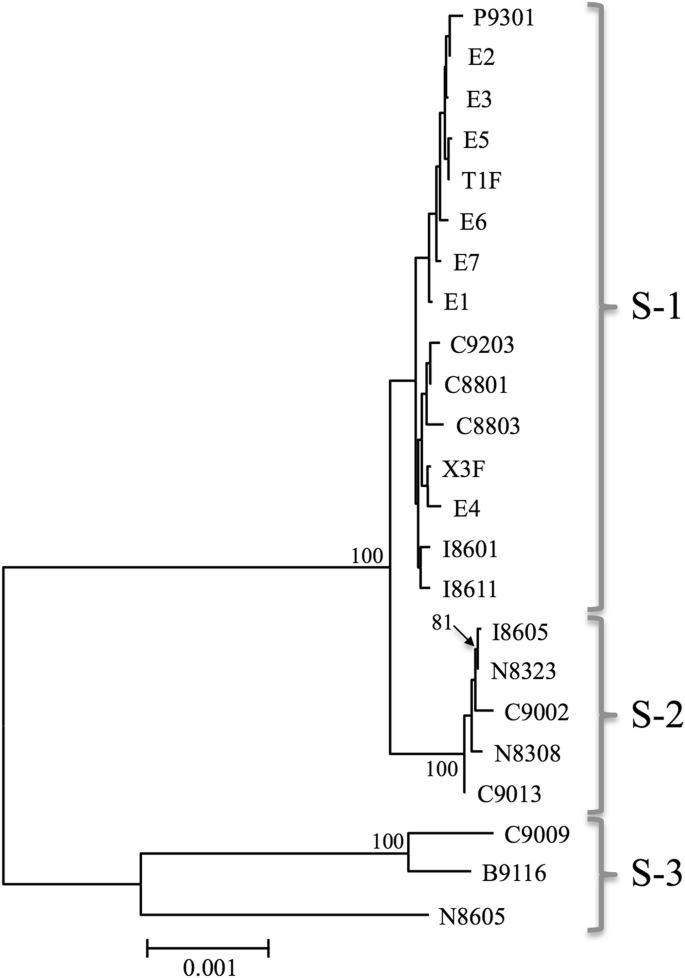
为了揭示物种间的系统发育关系S.从23株短花柱植物中获得-等位基因区,构建了NJ树S.共60108个位点,包括1123个SNP位点。NJ树基于S.- allalle地区显示普通荞麦植物的短风格表型(S / S基因型)大致分为三组,分别命名为S-1、S-2和S-3(图1)。3.).系统发育关系鉴定的基础上S.-链接支架的分析结果与S-ELF和SSG2[15.但不同于基于全基因组SNPs的NJ树(图。1).S-1、S-2和S-3系统发育组分别由15个、5个和3个品种组成。这些组也被PCA检测(附加文件2:图S2)。核苷酸的多样性S.-等位基因区(0.0039)低于所有支架1).系统发育基团之间的平均核苷酸距离和FST值S.-链接支架高(表2).特别是,S-3与S-1和S-2之间的关系远比它们之间的关系要远。
讨论
普通荞麦全基因组和s等位基因区特异性核苷酸多样性
在本研究中,我们利用GBS方法获得了255,517个全基因组SNPs,这比以往对该物种的研究获得了更多的全基因组SNPs。此外,公布的基因组序列[11.]使我们能够估计核苷酸多样性,表明常见荞麦(0.0065)的核苷酸多样性与其他侧交叉植物(如玉米)相当Zea Mays.,0.0064)[24.[向日葵(向日葵;0.0056) (25.],高于水稻等自交作物(奥雅萨苜蓿,0.0024)[26.]和大豆(大豆;0.0019) (27.].这种更高的核苷酸多样性可能是由于荞麦的异交交配系统和/或野生荞麦的基因流动,f . esculentumSSP。抱怨.
异型SI系统的遗传基础与植物和动物界性染色体的遗传基础相似。有许多关于性染色体核苷酸多样性的报告[28.那29.那30.那31.].例如,在人类中,X和Y染色体的核苷酸多样性与常染色体染色体的比例分别为0.62和0.20 [32.].性染色体中观察到的核苷酸多样性是较低的有效群体尺寸的预期结果:人X和Y染色体的有效种群尺寸分别对应于常染色体染色体的3/4和1/4。在这项研究中,我们证实了核苷酸的多样性S.-等位基因区域的核苷酸多样性比率同样低于整个基因组的核苷酸多样性比率S.- 整个基因组的区域为0.62。该数字远高于预期值(0.25)可能是由于功能基因的低密度S.- 区域[11.和/或最近的多种基因流动,如下节讨论。更高的遗传多样性S.- 相连的区域比预期也表明了S.- 相连的区域不是在净化选择下。
从s等位基因差异推断的基因流
系统发育关系从332S.等位基因支架(图3.)与基于全基因组SNPs的结果不一致(图。1):在图中所示的数据中,检测到三个差异很大的系统发育群,S-1、S-2和S-3。3.(从S.-等位基因区域),但在图中没有。1(来自整个基因组)。值得注意的是S.- 良好的区域分歧。特别是,S-3从其他两组强烈发散:S-3和其他两组之间的核苷酸多样性约为0.006(表2).使用每百万年0.01个同义核苷酸替换的粗略分子时钟速率(例如,为0.011Gossypium种,为0.016落花生物种) [33.那34.,这表明S-3类群在30万年前从其他类群中分离出来。在中国北方发现的最早的荞麦考古花粉标本可追溯到5000 - 6000 BP [35.].因此,检测到突变不太可能S.- 源自荞麦栽培的起源。考虑到肥沃杂交种导致交叉兼容性的可能性[36.]栖息地的重叠[19.]在栽培和野生荞麦之间,我们得出结论,分歧S.- 来自野生荞麦的序列术,f . esculentumSSP。抱怨,野生和栽培荞麦之间的血液均可归因于高基因组的多样性。
S-allele地区作为系统发育工具,用于阐明植物碎片碎片的起源和扩散
将混合物和系统发育分析和PCA分类为46种荞麦品种分为两组,亚洲和欧洲群,具有低FS值(FST = 0.068)。欧洲团体表现出低于亚洲集团的核苷酸多样性(表1).从花粉和大甲甲基的考古学研究(即烧焦的种子)记录,已经提出了荞麦在4000-2800 BP期间被引入欧洲,虽然它没有成为晚期中世纪期间的流行作物[37.].欧洲集团的核苷酸多样性和亚洲和欧洲群体之间的弱区分都可能是由于与从亚洲到欧洲的分散相关的遗传瓶颈,以及近期的培养和选择。通过同工学分析检测到的低遗传变异和失去等位基因也支持了这一假设[38.].
Y染色体保留着核苷酸多样性积累的顺序记录,已被用于检测祖先的单倍型和追踪人类迁移[39.].虽然基因组的SNP已经照亮了种群结构和种群分化的栽培荞麦,SNPS.- 连接的支架可以在物种的扩散历史的不同方面上脱光。在这项研究中,我们检测到三个专业S.-等位基因组,S-1, S-2和S-3(图1)。3.).由…所暗示的系统发育关系S.-链接支架表明来自野生亲缘的基因渗入,如上所述。这是由于抑制了重组S.- 像性染色体这样的阶级区域。因此,系统发育分析使用S.-等位基因区有望更好地了解荞麦种植的起源和全球扩散。
在这项研究中,我们没有发现S-1类型 S.-等位基因序列在尼泊尔和不丹的种群。基于我们对这三个分歧的识别S.-等位基因群,栽培荞麦有多个起源的可能性也应考虑。考虑到荞麦种子的遗骸在3000 BP出现在尼泊尔中西部[40],我们应考虑荞麦的可能性在喜马拉雅地区独立驯化,包括尼泊尔,不丹和中国西南部。为了阐明栽培荞麦的起源和全球扩散历史,我们需要扩展我们的样本集,以包括来自世界各地的更多栽培荞麦样本,以及野生荞麦的样品,f . esculentumSSP。抱怨,来自中国。
结论
基于使用GBS技术获得的基因组SNP,我们成功地估计了荞麦的核苷酸多样性(0.0065),这与其他侧交叉植物的核苷酸多样性(0.0065),例如玉米和向日葵。基于基因组SNP的系统发育分析还显示出培养的荞麦由两组,亚洲和欧洲组成。欧洲集团的低核苷酸多样性和亚洲和欧洲群体之间的低分化与与亚洲到欧洲的分散相关的遗传瓶颈,以及近期的欧洲荞麦的耕种和选择。基于基因组SNP的这些结果与基于同工酶变异的先前研究的结果一致。使用SNP估计的核苷酸多样性(0.0039)S.-等位基因区域比全基因组SNPs估计的低,反映了较小的群体规模S.-等位基因区域与整个基因组相比。这些数据还表明了野生荞麦基因向栽培荞麦流动的可能性,以及栽培荞麦有多种来源的可能性。综上所述,系统发育分析采用S.-等位基因区可以更好地了解荞麦栽培的起源和全球栽培扩散历史。
方法
基因型测序数据和SNP检测
我们使用发布的GBS读取EcoR我和均方误差I限制性内切酶(DRA accession number DRA004489,ftp://ftp.ddbj.nig.ac.jp/ddbj_database/dra/fastq/dra004/dra004489.)[11.].GBS数据集代表了23个短风格和23个长风格荞麦地方品种,它们来自欧亚大陆范围广泛的地区(附加文件3.:表S1)。低质量的读取和适配器使用Trimmomatic-0.36 [41使用选项SLIDINGWINDOW:5:25和MINLEN:40。使用的接头序列为CACGACGCTCTTCCGATCT和ACCGCTCTTCCGATCTGTAA。使用BWA 0.7.15 (Li and Durbin 2009)将修剪后的对端(PE) reads与荞麦参考序列(FES_r1.0)进行映射[42]与-l 10和-b 10选项。使用SAMTOOLS 1.3.1从BAM文件过滤单端读取。[43].变量由samtools的mpileup函数和在samtools 1.3.1中实现的bcftools调用。深度小于4和超过40的站点使用VCFtools 0.1.13转换为缺失数据[44].然后,使用VCFtools 0.1.13对缺失数据比例大于0.2的站点进行过滤。
系统发育树的构建及结构分析
掺合料v1.22 [45对46个荞麦地方品种的种群结构进行了研究。对于每个值K.,用不同的随机种子进行10个混合物分析运行。根据日志似然的最高值选择了最佳运行。邻居加入(nj)树[46]使用SEQBOOT构造,具有100个重复,然后是DNAdist(带有Kimura双参数方法),邻居和来自Phylip包3.6的定期节目[47].NJ树使用中点生根并使用FigTree 1.4.2进行可视化[48].采用Tassel 5.2.37进行基于协方差的主成分分析(PCA) [49].
核苷酸多样性和f统计量
缺失数据比例大于0.2的站点使用VCFtools 0.1.13进行过滤。然后,利用VCFtools 0.1.13计算种内和各分类/组内的核苷酸多样性。威尔和科克汉姆的重量F统计数据(置)50]采用VCFtools 0.1.13进行计算。
缩写
- 妊娠:
-
扩增的片段长度多态性
- GBS:
-
Genotyping-by-sequencing
- gbs:
-
颗粒结合的淀粉合成酶
- PCA:
-
主成分分析
- S.轨迹:
-
Self-incompatibility轨迹
- S-ELF3:
-
s位点早花3
- 如果:
-
Self-incompatibility
- 单核苷酸多态性:
-
简单的核苷酸多态性
- SSG2:
-
Short-style-specific基因2
- SSRs:
-
简单序列重复(SSRs)
参考文献
- 1。
Konishi T,Yasui Y,Ohnishi O.培养普通荞麦的原始出生地从培养种群和野生常见荞麦面的遗传关系推断出AFLP分析。基因类型系统。2005; 80:113-9。
- 2。
ohnishi O.基于普通荞麦栽培和野生种群的植物分析培养常见荞麦的起源。Fagopyrum。2009; 26:3-9。
- 3.
BONAFACCIA G,MAROCCHINI M,KREFT I.面粉和麸皮的组成和技术特性来自普通和鞑靼荞麦。食品化学。2003; 80:9-15。
- 4.
Ahmed A, Khalid N, Ahmed A, Abbasi NA, Latif MSZ, Randhawa MA。(2014)荞麦的植物化学成分及生物功能特性研究进展。农业科学。2014;152:349-69
- 5.
Giménezbastida JA, Zieliński H.(2015)荞麦作为功能食品及其对健康的影响。农业食品化学。2015;63:7896-913。
- 6.
Ohnishi O,Ohta T.普通荞麦中的联系地图的建设,Fagopyrum esculentumMoench。日本J Genet 1987; 62:397-414。
- 7.
小西,岩田,Yashiro K, Tsumura Y, osawa R, Yasui Y, Ohnishi O.普通荞麦微卫星标记的开发与鉴定。Sci繁殖。2006;56:277 - 85。
- 8.
Yasui Y, Wang Y, Ohnishi O, Campbell CG。普通荞麦扩增片段长度多态性连锁分析(Fagopyrum esculentum)和它的野生自花近亲Fagopyrum homotropicum.基因组。2004;47:345-51。
- 9.
王志强,王志强,王志强,等。利用DNA微阵列快速分型进行普通荞麦高密度连锁定位和QTL定位(Fagopyrum esculentumMoench)。Sci繁殖。2014;64:291-9。
- 10.
王志强,王志强,王志强,等。同殖元作物大规模选择育种中基因组选择的潜力:增加普通荞麦产量的实证研究。前植物SCI。2018; 9:276。
- 11.
安井,平川,上野,等。荞麦基因组草图的组装及其在鉴定农艺有用基因中的应用。DNA研究》2016。2016; 23:215-24。
- 12.
Matsui K,Hisano T,Yasui Y,Mori M,Walker Ar,Morishita T,Katsu K.编码亮绿素蛋白还原酶的基因的分离与表征(Felar.)和花青素还原酶(FeANR)在荞麦(Fagopyrum esculentum).植物生理学杂志。2016;205:41-7。
- 13。
王志强,王志强,王志强,等。一种新的荞麦二氢黄酮醇4-还原酶(DFR)具有独特的底物结合结构,改变了底物的特异性。BMC医学杂志。2017.2017; 17:239。
- 14。
Matsui K,Oshima Y,Mitsuda N,Sakamoto S,Nishiba Y,Walker Ar,Ohme-Takagi M,Robinson SP,Yasui Y,Mori M,Takami H.荞麦R2R3 MyB转录因子FeMYBF1调节黄酮酚生物合成。植物SCI。2018; 21(9):276。
- 15.
Yasui Y, Mori M, Aii J, Abe T, Matsumoto D, Sato S, Hayashi Y, Ohnishi O, Ota T。s位点早花3专门存在于表现出异常自我不相容性的短风格荞麦植物的基因组。Plos一个。2012; 7:E31264。
- 16.
2 .异型自交不亲和的遗传分析(S.)荞麦的基因座。在:周M,编辑。荞麦的分子育种和营养方面:学术出版社;2016. p。411-22。
- 17.
加尔伯r,Quisenberry Ks。荞麦中的自我施肥。JAGRIC RES。1927年; 34:185-90。
- 18.
Ohnishi O.搜查荞麦III的野生祖先。栽培普通荞麦的野生祖先,鞑靼荞麦。econ bot。1998年; 52:123-33。
- 19.
在三江地区普通荞麦栽培群体和自然群体之间的密切遗传关系不是由于他们之间最近的基因流动——利用微卫星标记的分析。基因工程学报2007;82:53-64。
- 20.
Nei M.分子进化遗传学。纽约:哥伦比亚大学。按;1987年。
- 21.
Elshire RJ, Glaubitz JC, Sun Q, Poland JA, Kawamoto K, Buckler ES, Mitchell SE。一个强大的,简单的基因分型测序(GBS)方法的高多样性物种。《公共科学图书馆•综合》。2011;6:e19379。
- 22.
波兰贾,普里夫。促植物育种和遗传学的基因分型测序。植物基因组。2012; 5:92-102。
- 23.
Poznik Gd,Henn Bm,Yee Mc,Sliwerska E,Euskirchen Gm,林AA,斯奈德M,Quintana-Murci L,Kidd JM,UnderHill Pa,Bustamante CD。测序Y染色体可以及时解决差异,以便与女性的共同祖先。科学。2013; 341:562-5。
- 24。
Wright Si,Bi IV,Schroeder SG,Yamasaki M,Doebuly JF,McMullen MD,Gaut BS。人工选择对玉米基因组的影响。科学。2005; 308:1310-4。
- 25。
刘a,柏克。野生和栽培向日葵的核苷酸多样性模式。遗传学。2006;173:321-30。
- 26。
黄X,kurata n,wei x,王zx,王a,zhao q等。水稻基因组变异的地图揭示了栽培水稻的起源。自然。2012; 490:497-501。
- 27.
林慧敏,徐旭,刘旭,陈伟,杨光,王福林,等。通过对31个野生和栽培大豆基因组的重测序,确定了遗传多样性和选择模式。Nat麝猫。2010;42:1053-9。
- 28.
张晓东,张晓东,张晓东,等。一株y -连锁植物基因的低变异及其对y染色体进化的影响。大自然。2000;404:388 - 90。
- 29.
引用本文:张志刚,张志刚,张志刚。植物雌雄异株y染色体退化的DNA序列多样性的间接证据SlY4 / SlX4和DD44-X / DD44-Y基因对。生物学杂志。2005;18:337-47。
- 30。
Begun Dj,Holloway Ak,Stevens K,Hillier LW,Poh YP,Hahn MW,Nista PM,Jones CD,肯尼斯州,杜威CN,Pachter L,Myers E,Langley Ch。人口基因组学:多态性和分歧的全基因组分析果蝇模拟人.公共科学图书馆杂志。2007;5:e310。
- 31.
沃尔彭·r,zeng f,chen c,zhang z,wai cm,han j等。木瓜YH染色体的起源和驯化。Genome Res。2015; 25:524-33。
- 32.
Sachidanandam R,Weissman D,Schmidt Sc,Kakol Jm,Stein Ld,Marth G等。含有142万单核苷酸多态性的人类基因组序列变化的地图。自然。2001; 409:928-33。
- 33.
Abdurakhmonov IY, Buriev ZT, Logan-Young CJ, Abdukarimov A, Pepper AE。棉花光敏色素感光基因家族的复制、分化和持久性(Gossypiumspp)。BMC Plant Biol. 2010;10:119。
- 34.
宋华,高华,刘军,田鹏,南志。密码子使用偏倚与基因表达及替代率的相关性分析arachis duranensis.和arachisipaënsis.直接同源。Sci众议员2017;7:14853。
- 35.
荞麦:一种来自中国主要驯化中心以外的作物?古植物学、孢粉学和遗传学证据综述。Veg Hist Archaeobot. 2018; 27:493-506。
- 36。
OHNISHI O.非破碎的习惯基因(sht),叶绿素缺陷和其他有害基因隐藏在普通荞麦的野生祖先的自然群体中。Fagopyrum。1999; 16:23-8。
- 37。
de Klerk P,Couwenberg J,Joosten H.在中世纪晚期欧洲亚洲西部驻弗平莫里姆的Parenten占Parenten:一个洲际谜团。Palaeogeogr Palaeoclimatol帕拉西奥。2015; 440:1-21。
- 38。
栽培荞麦的群体遗传学研究,Fagopyrum esculentumMoench。VIII。欧洲和丝绸之路的局部区别。JPN J Genet。1993; 68:303-16。
- 39.
利用Y染色体和线粒体DNA群体结构追踪人类迁移。Annu Rev Genet 2007; 41:539-64。
- 40。
Knörzerkh。3000年在高喜马拉雅山的山谷中农业。植被神父古群。2007; 9:219-22。
- 41。
Bolger Am,Lohse M,Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014; 30:2114-20。
- 42。
李H,Durbin R.快速准确的短读对齐与挖洞轮转变换。生物信息学。2009; 25:1754-60。
- 43.
李H,手机B,Wysoker A,Fennell T,Ruan J,Homer N,Marth G,Abecasis G,Durbin R. 1000基因组项目数据处理子组。序列对齐/地图格式和samtools。生物信息学。2009; 25:2078-9。
- 44.
Danecek P, Auton A, Abecasis G, Albers CA, Banks E, DePristo MA, Handsaker RE, Lunter G, Marth GT, Sherry ST, McVean G, Durbin R. 1000基因组项目分析组。不同的调用格式和VCFtools。生物信息学。2011;27:2156-8。
- 45.
基于快速模型的非亲缘关系个体祖先估计。基因组研究》2009;19:1655 - 64。
- 46.
Saitou N,Nei M.邻居加入方法:一种重建系统发育树的新方法。mol Biol Evol。1987; 4:406-25。
- 47.
Phylip包3.6。http://www.evolution.genetics.washington.edu/phylip.htlm
- 48。
Figtree 1.4.2。http://tree.bio.ed.ac.uk/software/figtree/
- 49。
Bradbury PJ,张Z,Kroon de,Casstevens TM,Ramdoss Y,Buckler ES。TASSEL:多样化样本中复杂性状的关联映射软件。生物信息学。2007; 23:2633-5。
- 50.
王志强。种群结构分析的f -统计量估计。进化。1984;38:1358 - 70。
确认
我们感谢T. Ota和H.寻找有价值的建议,而K.L.Farquharson用于稿件的语言编辑支持。本研究得到了JSPS Kakenhi(授予号码18H02177和18KK0172)的支持。
资金
本研究得到了JSPS Kakenhi(授予号码18H02177和18KK0172)的支持。
资助机构没有参与这项研究的设计;收集、分析或解释数据;在手稿的撰写和发表结果的决定中。
数据和材料的可用性
在GBS分析中使用的Illumina读取可在DDBJ序列读取档案(DRA)中获得(DRA登录号是DRA004489,ftp://ftp.ddbj.nig.ac.jp/ddbj_database/dra/fastq/dra004/dra004489.).
作者信息
从属关系
贡献
NM分析并解释了GBS数据,并是编写手稿的主要贡献者。yy构思了这项研究,参与了它的设计,进行了分析,并写了稿件。这位作者都读到并批准了最终手稿。
相应的作者
伦理宣言
作者的信息
作物展位,农业研究生院,京都岛,日本京都京都,京都,京都,京都,京都,京都,京都,京都岛,京都,京都,京都岛,浅谈探矿实验室。
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
额外的文件1:
图S1。人口结构46次荞麦。a)祖先人口分配的交叉验证误差通过混合物的不同数量的群集(k = 1-10)。使用标准偏差显示10个掺合符号的平均交叉验证错误。b)46个常见荞麦地体的人口结构推断出来(k = 2)。使用255,517个SNP来估计个体的祖先比例。棒的彩色代码(青色和洋红色)表示推断亚群的典型基因型。红色和蓝色的牧草分别是短而长的植物。(PNG 620 KB)
附加文件2:
图S2。基于GBS数据的23个短风格植物的主分量分析(PCA)映射在332 S-等位基因脚手架上。从PCA示出了前两个轴(用于PC1和PC2的y轴的X轴)的曲线图。由每个分量解释的方差的比例在括号中沿每个轴给出。(PNG 457 KB)
附加文件3:
表S1.本研究中使用的荞麦地兰列表。(PDF 51 KB)
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
关键词:基因流动S.- 栽培荞麦地区。BMC植物杂志19,125(2019)。https://doi.org/10.1186/s12870-019-1730-1.
收到了:
公认:
发表:
关键字
- 荞麦
- 作物演变
- GBS
- 异形的self-incompatibility