摘要
背景
微小反重复转座子和长末端重复反转座子在植物基因组中普遍存在,对植物基因组的进化和多样性具有重要意义。然而,它们的插入/放大机制和作用柑橘类基因组的进化/多样性仍然明白很差。
结果
为了解决这一知识缺口,我们开发了不同的计算管道,以分析,注释和分类螨虫,并在六种不同测序中分析螨虫和LTR回收路标柑橘类物种。共鉴定了110个科的62010只全长螨类。我们观察到螨倾向于插入基因相关区域,并在启动子中富集。我们发现DTM63可能是一个活跃分子突变在可追溯的过去的鸟类家庭,可能仍然有效柑橘类.螨的插入导致了大量的多态性,并发挥了重要的作用柑橘类基因组多样性和基因结构变异。此外,鉴定出6630个完整的LTR反转录转座子和13371个单独的LTR。其中12个LTR谱系在单子叶和双子叶植物分化前就已分离。我们观察到LTR反转录转座子的插入和缺失是在动态平衡的情况下完成的,它们的半衰期在柑橘类是〜180万年。
结论
这些发现为了解螨和LTR反转座子及其在不同物种基因组多样性中的作用提供了依据柑橘类基因组。
背景
微小反重复转座子是一种缺乏自身转座酶的非自主DNA转座子[1].它们广泛存在于真核生物中,特别是植物基因组中。螨虫有以下特点:(1)一样自主DNA转座子,螨具有重复两侧小直接反向重复(目标站点重复TSD)和较短的长度(通常是< 800个基点),(2)一些螨可能转录并形成双链rna,这可能进一步推导为小分子rna (srna) [2],(3)与宿主基因组中的其他转座子相比,螨可以获得非常高的拷贝数[3.那4.].
Jiang和他的同事发现了一种活跃的螨虫m在米饭中,后来他们发现了两个自主DNA转座子p和发出难闻的气味利用的守恒端点进行同源搜索m.他们还观察到发出难闻的气味可以激活换位m并命名这个现象“跨动员”[5.].后来,Yang和同事们通过实验证实了交叉动员假说,并报告说,螨虫也可以被不同家族的自主DNA转座子转座[6.].此外,一些研究观察到,螨倾向于更接近遗传区域[1那7.]并且可以调节基因表达[8.].一些螨的转录本可以形成发夹结构,进一步被dicer-like (DCL)识别并产生小rna (sRNAs)。这种现象在拟南芥,米和人类[9.那10.].螨源srna约占水稻基因组所有srna的1/4 [2,表明螨在转录过程中起着重要的调控作用。螨的插入和消除可导致宿主基因组中存在和缺失多态性,这被认为是宿主基因组进化的一个重要方面[11.].
通常,长末端重复(LTR)逆转录转座子以5 ' -TG-3 '开始,以5 ' -CA-3 '结束。整合后,在受体位点两侧有4~6 bp的TSD。一般来说,自主LTR逆转录转座子的内部区域包含两个基因:呕吐和波尔.呕吐编码病毒样衣壳的结构蛋白波尔编码包括天冬氨酸蛋白酶(AP),逆转录酶(RT), RNaseH (RH)和整合酶(INT)的多蛋白。根据RT/INT的顺序和包膜蛋白(ENV)的发生情况,可以将LTR反转录转座子进一步划分Copia那吉普赛那ERV那Bel-Pao.和逆转录酶病毒超家族[12.].
以前的研究表明,在宿主基因组中存在LTR回析链烷酮的相对平衡和缺失。LTR回收转换的删除主要是由不平等的同源重组引起的[13.那14.]及非法重组[15.].然而,元件内不相等的同源重组导致形成单独的LTR,其结构与完整的LTR逆转录转座子的5 ' LTR或3 ' LTR末端相同[14.在它们的末端有TSD侧翼。非法重组会删除LTR反转录转座子的内部序列,并在缺失位点形成一个较短的转发重复序列。以往的研究表明,非法重组所删除的序列比不平等同源重组所删除的序列高5倍拟南芥[15.].LTR反转座子的扩增被认为是导致基因组显著大小扩张的主要驱动因素之一。研究表明,即使在密切相关的物种中,某些LTR逆转录转座子家族的扩增也会导致基因组大小的显著变化[16.].霍金斯等人。发现具体放大GORGE3.LTR回收器族家族导致了基因组大小的显着差异Gossypium.[17.].此外,LTR回收转移倾向于插入下游基因的增强剂,阻遏物或启动子,随后调节下游基因的表达[18.那19.].血橙的形成是基于LTR回收的基于基因表达调节的一个很好的例子[20.].LTR反转座子也可诱发染色体重排和基因易位[21.].在一项系统发育研究中,Du等人发现5吉普赛血统和6Copia在双子叶植物和单子叶植物分化之前,谱系就已经分离了[22.].这可以用作以下研究中的注释参考和分类标记,以及用于在注释基因组中改进LTR回收衰退的指南。在Du的研究中,他们发现Solo-Ltr形成和LTR尺寸之间存在正相关[22.].后来,El-Baidouri和Panaud发现Solo-Ltr形成与LTR /内部的比例之间存在更强的正相关性[23.].他们还发现,LTR反转座子的水平转移在基因组进化中发挥了重要作用[23.].
柑橘类是人体健康的重要维生素来源,在所有水果中名列前茅。先前的研究已经从已发表的文献中确定了转座因子(TEs)柑橘类基因组(20.那24.那25.那26.那27.,但是TE注释管道中的差异使得这些结果无法比较。螨和LTR反转录转座子积累和扩增的机制及其在柑橘类对基因组进化和多样性的了解仍然很贫乏。不同的出版物柑橘类基因组为比较te和研究它们在基因组多样性方面的进化特征打开了大门。在本研究中,我们研究了6柑橘类物种:c . sinensis(甜橙,基因组大小〜367 MB,Contig N50 49.89 KB),Atalantia buxifolia(中国盒子橙,基因组大小〜370 MB,CONTIG N50 23.89 KB),c .茅(柚,基因组大小~ 380 Mb, contig N50 10.62 Mb),c . ichangensis(changpapeda,基因组大小~ 391 Mb, contig N50 76.56 kb),C. Medica.(香橼,基因组大小406 Mb, contig N50 46.50 kb)c·克莱门蒂娜(柑橘,基因组大小〜370 MB,CONTIG N50 115.90 KB)[26.那27.那28.那29.那30.].
我们开发了两个综合管道,以注释和分析上面的螨虫相关和相关的序列柑橘类物种,然后研究了一些的放大模型柑橘类螨虫族,并比较甜橙与其他5之间的螨虫存在和缺失多态性比率柑橘类物种。我们研究了螨在不同基因组区域的相对丰度,并分析了螨在基因结构变异中的作用。研究了螨源小rna及其相对位置。采用相对保守的方法,我们注释了完整的LTR反转座子和单独的LTR,并研究了不同谱系和家族的激活情况。我们还研究了不同LTR反转录转座子家族的相对单独-LTR丰度。此外,我们还研究了甜橙中ltr相关序列的分布,并探讨了单独ltr形成的可能因素。
结果
使用基于螨虫猎人的管道对螨虫进行标注和分类
我们使用附加文件中所示的管道对螨虫进行了注释和分类1:图S1。在手工管理螨种子序列(附加文件8.:数据集S1),我们发现许多螨虫的终端区域不存在(终端缺席/存在= 47:5),这表明由螨猎物计算的螨种子的边界校正的必要性[31.].所有与螨虫相关的序列被分类为突变总科,PIF-Harbinger总科,帽子超家族,又进一步划分为110个家族。每个家族命名为DT(A/M/H)1-n (帽子对应于DTA,突变对应于DTM,PIF-Harbinger对应DTH,数字1-n对应特定族号)。突变超级携带82个家庭,帽子超级家庭有20个家庭PIF-Harbinger超级家族含有8个家庭。总共有61,980个全长螨虫注释柑橘类各基因组中与螨虫相关的序列平均长度约占总基因组长度的3% (8~11 Mb,表)1).我们发现99.4%的全长螨型短于800磅,平均长度为约302磅。平均全长突变总科,PIF-harbinger总科和帽子Superfamily分别为256,431和563 bp。全长螨虫帽子超级家族明显比其他两个超级家族长(威尔科克斯检验,P.-value < 0.001),以及突变Superfamily是最短的。我们还观察到前10%的螨类全基因组拷贝数占全部全基因组拷贝数的50%以上。
一轮扩音爆占主导地位柑橘类螨虫
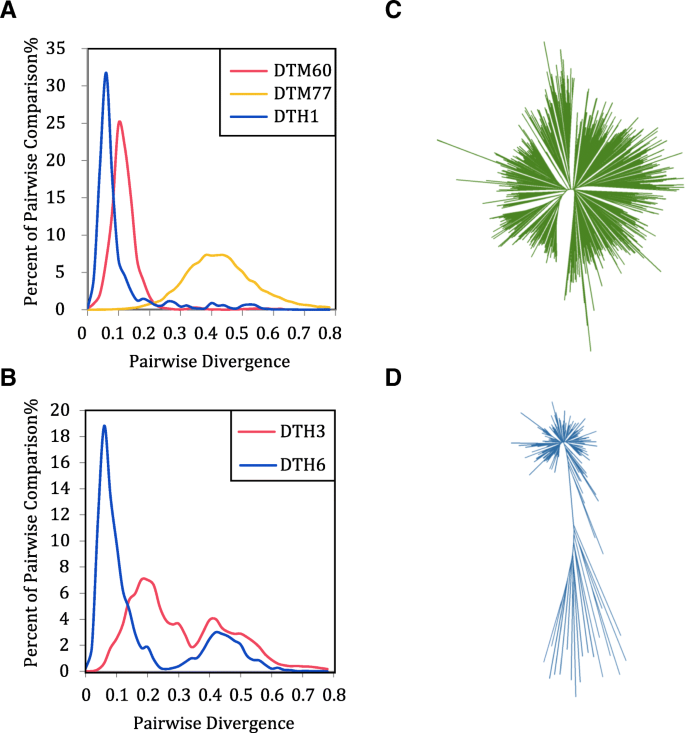
据报道,水稻中螨虫家族主要经历了一轮或多轮扩增[2].在我们的研究中使用类似的方法,我们比较了在柑橘类.我们发现螨虫家族的单峰分布占主导地位柑橘类,如DTM60、DTM77、DTH1等。1a),而几个螨虫族(包括DTH3和DTH6)显示了双峰或多模峰分布(图。1b)。我们观察到具有单峰分布的螨虫族的系统发育树显示出明星形状(图。1c),而双峰或多峰分布则显示出两个或多个枝。1d).综上所述,单峰分布的螨科主要是同一螨的一个副本在一定时间内发生一轮扩增或同一科的一个紧密副本发生显著扩增。而双峰型或多峰型分布的螨主要经历两次或多次扩增,源于同一螨科不同成员在不同时间点的扩增事件。
古老的活动螨科和可追踪的可能的活动螨科
DTM53在六个中有超过1000个全长副本柑橘类物种的两两散度分布非常相似,均为单峰曲线,平均两两散度约为0.26(图2)。2a).它响应的是约2300万年的辐散时间,在辐散之前柑橘类和Atalantia属.同样,DTM58(平均两两发散0.45)和DTM77(平均两两发散0.41)在发散之前也经历了放大爆发柑橘类和Atalantia属。但是,全长DTM63的副本数量答:buxifolia那c . sinensis那c·克莱门蒂娜那C. ichangensis, C. medica和c .茅分别为48、302、201、323、174和496,DTM63数差异较大,说明DTM63家族近期扩增。有一些非常相似或相同的副本c . sinensis那c·克莱门蒂娜那c . ichangensis和检测结果。DTM63的成对分歧显示原点周围的峰(图。2b)表示DTM63仍然是这四种物种中的活性螨家族,并且需要进一步的研究来确认。发现的活跃螨主要来自TC1 / MARINER.和论坛/预兆而DTM63可能是第一个发现的螨类活性家族突变总科。由于DTM63缺乏转座酶,其转座可能依赖于自主的DNA转座子。为了找出激活DTM63转位的自主DNA转座子,我们对候选的DNA转座子进行了注释,获得了572和585个候选区域c . sinensis和c·克莱门汀分别。随后,我们用保守的7个BP终端和9〜10 BP TSD扫描了候选地区,但我们没有找到任何突变- 麦克酸自主DNA转座子,其保持与DTM63相同的结束序列。
P/A多态性之间的比率不同柑橘类基因组反映遗传距离
螨的插入和缺失不可避免地导致宿主基因组存在或缺失(P/A)多态性[2].以甜橙为参考基因组,计算甜橙与其他5种基因组的P/A多态性比柑橘类物种(表2).我们发现甜橙与原代之间的P/A多态性比柑橘类种类答:buxifolia是其他四种的1.3~2.2倍。因此,MITE P/A多态性与系统发育关系有较强的相关性,最接近甜橙的系统发育关系为:c·克莱门蒂娜(多态性比率38.38%),c .茅(46.70%), ichangensis(59.89%),C. Medica.(65.79%)和答:buxifolia(84.49%)分别.上述结果表明,P / A多态性螨虫比例反映了不同的遗传关系柑橘类物种。
螨优先插入基因侧翼区域,在基因组多样性中起重要作用
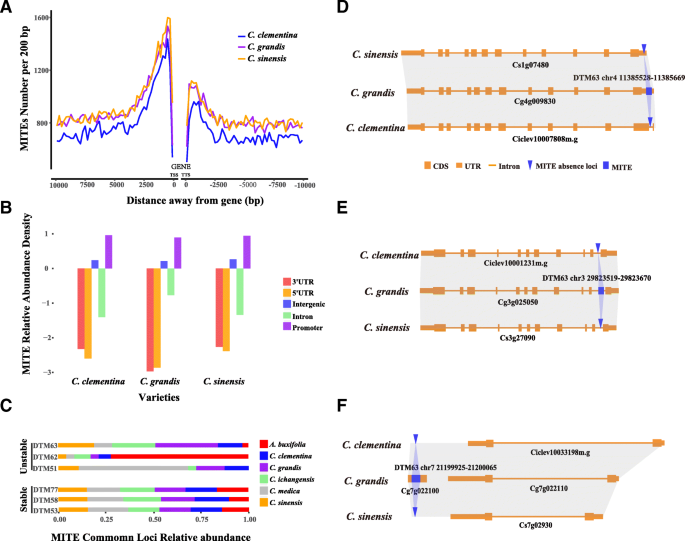
为了研究螨虫是否有利于插入基因相关区域,我们计算了甜橙、柚子和小柑橘基因区域上游和下游基因区域(转录起始位点(TSS)到转录终止位点(TTS))中螨虫插入的分布。我们观察到三个地区的螨类插入分布格局非常相似柑橘类在上游基因区和下游基因区1kb内分别出现不同的峰值(图2)。3.a),表明在基因侧翼区域中优先插入螨虫。然后,我们分析了不同基因组区域的螨虫的分布,包括5'和3'未翻译的区域(UTRS),内含子,启动子(定义为TSS的1kb)和基因组区域。考虑到大多数基因组区域是代族区域,我们计算了相对丰富的螨虫,并且观察到螨虫的相对密度是启动子区中最丰富的密度,基因区域(5'和3'UTR中最少丰富,内含子。3.b).因此,螨优先插入基因侧翼区域,特别是启动子区域,说明aCIS.- 螨虫对其下游基因的作用[32.那33.].
通过比较不同螨科的拷贝数,探讨其分布规律及其在螨病中的作用柑橘类基因组进化,不同螨科丰度在6柑橘类基因组见附加文件3.:图S3。有两种类型的螨虫分布模式。稳定的基因组分布图案指示的螨虫家族与不同的拷贝数相似柑橘类如DTM53、DTM58和DTM77。这些螨科在物种形成前经历了扩增爆发性柑橘类和Atalantia最近很少有螨虫的插入或缺失。然而,螨科的基因组分布不稳定,在不同物种间存在明显的分布偏倚。例如,DTM63在c .茅那c . ichangensis和c . sinensis比C. Medica.和答:buxifolia(无花果。3.C),由于最近的换位。DTM62有更多副本答:buxifolia比其他5柑橘类DTM51有更多的拷贝C.媒体比其他人。与稳定的螨虫家庭相比,不稳定的螨虫家庭促成了更多的分歧柑橘类并可能在多样性中扮演重要角色柑橘类基因组.
我们定义了所有物种中存在的螨虫,作为保守的螨虫,另一种是非保守的。插入促进剂(18.13%)和5'/ 3'UTR区域(1.72%)的保守螨的比例低于非保守螨(促进剂25.76%,在UTR中4.31%),这表明了强烈的选择效果螨虫插入这些区域(附加文件5.:表S1)。有趣的是,新插入的螨虫倾向于促进区域(26.08%)c·克莱门蒂娜27.17%c . sinensis和26.63%c .茅)与古代插入的螨虫(23.97%)相比c·克莱门蒂娜25.21%c . sinensis和23.69%c .茅)(额外的文件6.:表S2)。数字3.D显示一只螨虫,唯一插入5'UTRc .茅, 图。3.e显示螨插入内含子的例子c .茅在内含子中插入螨没有改变基因结构,图。3.f显示插入螨虫c .茅插入区注释了一个基因,共线区没有注释的基因c·克莱门蒂娜和c . sinensis基因组分别。以上例子表明,螨虫在人类基因组结构变化中起着重要的作用柑橘类基因组。
螨虫SRNA主要来自螨虫终端
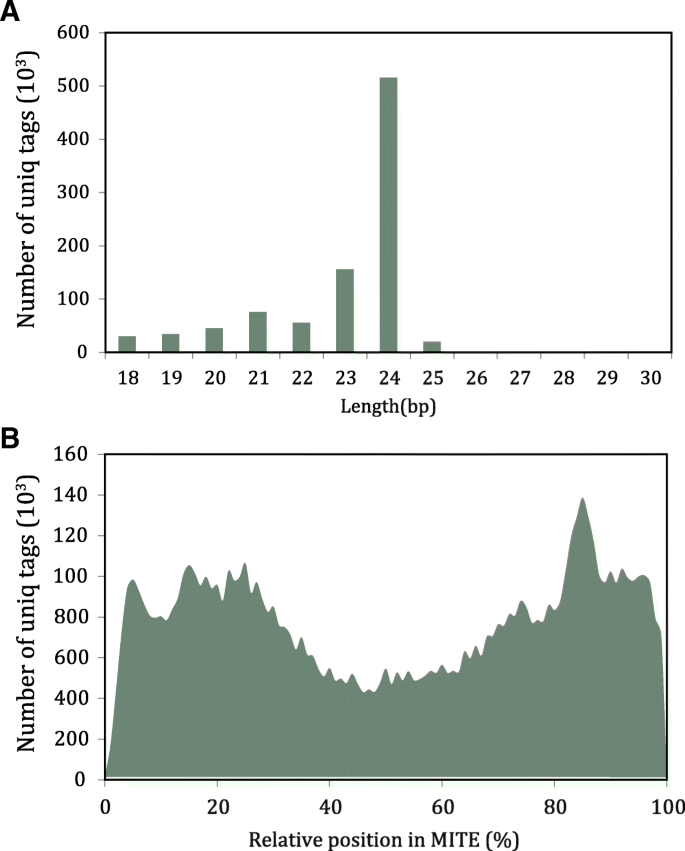
从先前的研究中,我们收集了总共14,664,233个独特的SRNA标签,其中7,258,262标签可以与甜橙参考基因组对齐,并且935,213标签可以与螨类序列对齐。在过滤掉未映射的标签时,螨虫SRNA占总金额的12.9%。通过调查螨螨衍生的SRNA的长度分布,我们观察到它们主要是24bp(图。4.a)和从不同位置的螨(图。4.b).与srna来源的相对位置相比,我们发现它们在甜橙中的分布与水稻不同[2].两端只有两个峰,中间有一个谷,表明甜橙中螨的中部比其他区域衍生的sRNAs少,而水稻中则有一个峰[2].
LTR反转座子的注释、分类和表征
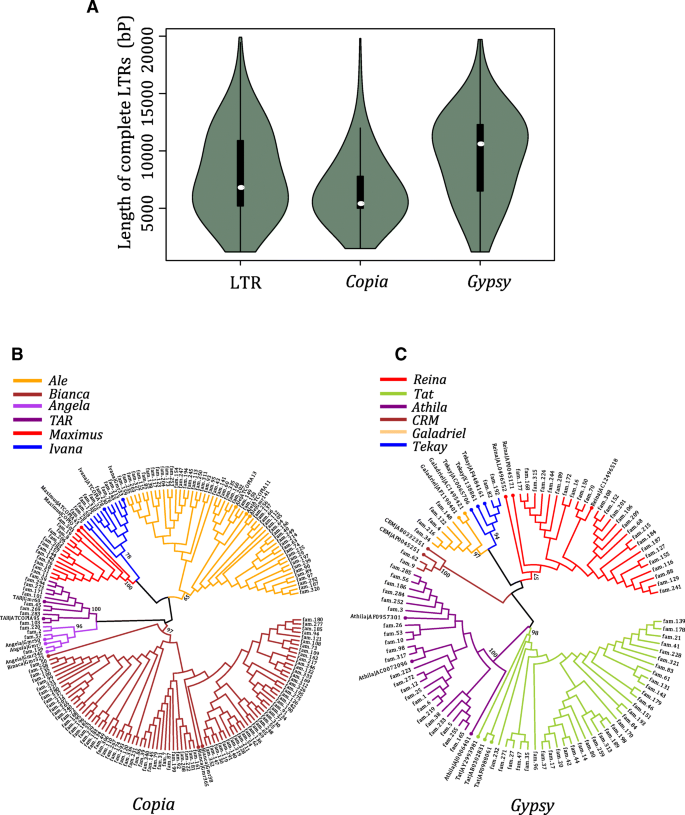
尽管Wicker和他的同事们提出了“80-80-80”规则来进行TEs族分类[34],在不同的LTR研究中仍然存在争议。在最近的一项研究中,建议使用另一种截止(60%的身份和70%覆盖率)更适合UWUM家族Zea Mays.和rlc_gmr6 / 18家庭大豆[23.].考虑到完整的LTR反转座子和单独的LTR都具有完整的LTR,我们选择75%的截断值将所有LTR反转座子分为不同的柑橘类家庭。我们总共从340个家族中获得13371个单独LTR和6670个完整的LTR反转录转座子(见表)3.;附加文件9.:数据集S2)。除了6种之外,Solo-LTR的数量大致相当于Atalantia buxifolia和c . sinensis而完整的LTR反转录转座子从392到1904不等。考虑到c .茅其LTR全转座子区域的组装质量也明显优于其他5个基因组。此外,我们使用的策略可能会遗漏一些低拷贝的LTR逆转录转座子家族答:buxifolia.通过这种方式,c .茅将是进一步调查的更合适的选择。完整的LTR回收器的长度分布中有两个不同的峰值(图。5.A),一个峰在~ 5.4 kb左右,另一个峰在~ 12.4 kb左右,这两个峰对应于Copia和吉普赛分别super-families。全LTR反转录转座子的平均长度Copia全长约6.5 kb,完整LTR逆转录转座子的平均长度吉普赛Superfamily为~ 9.3 kb。与之前的研究一致,吉普赛LTR逆转录转座子明显长于CopiaLTR回收转换(Wilcox测试P.-value < 0.001) [22.那23.那26.那35那36].的比例吉普赛例如元素Copia类元素为1.5英寸c .茅,这略高于大豆(1.4)[22.]但远低于大米(4.9)[35]及高粱(3.7)[36].此外,SOLO-LTR完成LTR回收的比率为1.4,其高于玉米,大豆和稻米[23.].
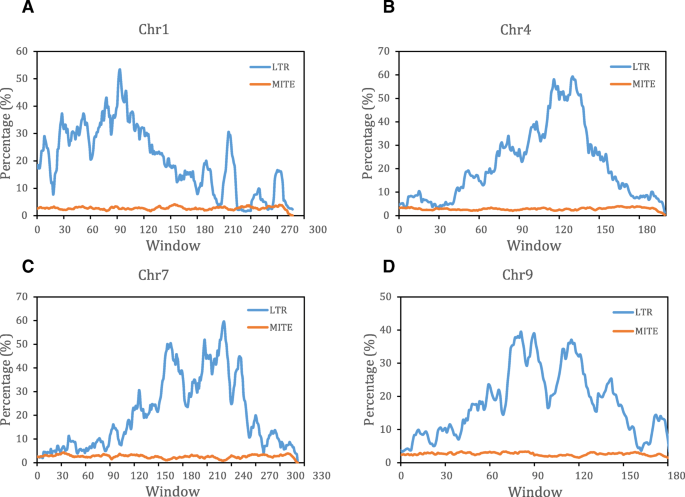
通过计算甜橙基因组中LTR转回转体的分布,我们发现与螨虫的相对均匀的基因组分布不同,LTR回收弯囊的分布非常异质。沿着不同染色体的焦点近端区域中存在显着的峰,这与对其他物种的先前研究一致[37].对这一现象的一种可能解释是,着丝粒近端区域受到重组抑制,导致不相等同源重组和不合理重组被抑制,从而积累了着丝粒近端区域的LTR逆转录转座子。
12个保守的LTR逆转录转座子谱系存在于柑橘类
在以前的研究中,Copia类超家族被分为6个谱系,安吉拉那啤酒那Bianca.那伊凡娜那马克西姆斯和焦油[12.那38那39),而吉普赛- 将超家族进一步分为另外6个谱系,Tekay.那凯兰崔尔女王那CRM.那雷纳那萨利拉和答[40].在这些当中,Tekay,Galadriel,CRM和雷纳是Chromovirus演化支.这12个谱系在双子叶和单子叶植物分化之前就被分离了。我们从公共数据中检索了RT域的序列和本研究中鉴定的LTR逆转录转座子,包括91个吉普赛- 就像RT和174Copia- 麦克雷特序列。然后我们构建了RT结构域的系统发育树(图。5.b, c).从系统发育树中观察到,所有12个谱系都被鉴定出柑橘类.此外,根据系统发育树的拓扑结构对相应的家族的分支进行分类。
为了调查这12个谱系的激活和状态,我们统计了每个谱系的家族和元素数(见表)4.).在吉普赛超级家族,答有最多的家庭(33),和萨利拉包含最大完整的LTR RORTRANSPOSONS(1754)。但是,在Copia超级家族,Bianca.有最多的家族(86个)和完整的LTR反转座子(1071个),而安吉拉有最少的家庭(5个)和焦油包含最不完整的LTR RORTORANSPOSONS(96)。
我们进一步计算了每个谱系的完整LTR回收器和独奏LTR的平均长度。完整的LTR回收输送不同Copia和吉普赛谱系表现出显着的长度差(Kruskal-Wallis等级和测试,P.-value < 0.001), solo-LTRs长度差异显著(Kruskal-Wallis秩和检验,P.值< 0.001)。的马克西姆斯谱系的平均长度为〜10 kB,这是完整的最长CopiaLTR回收转换。与以前的研究相比,平均长度Copia血统的柑橘类虽然Bianca.有更多的家庭和完整的LTR RODRORANSPOSON成员柑橘类比米饭,拟南芥和麦芽汁[12.].
几个LTR回收吊装家庭在可追认的过去中处于活跃,并在进口角色柑橘类基因组多样性
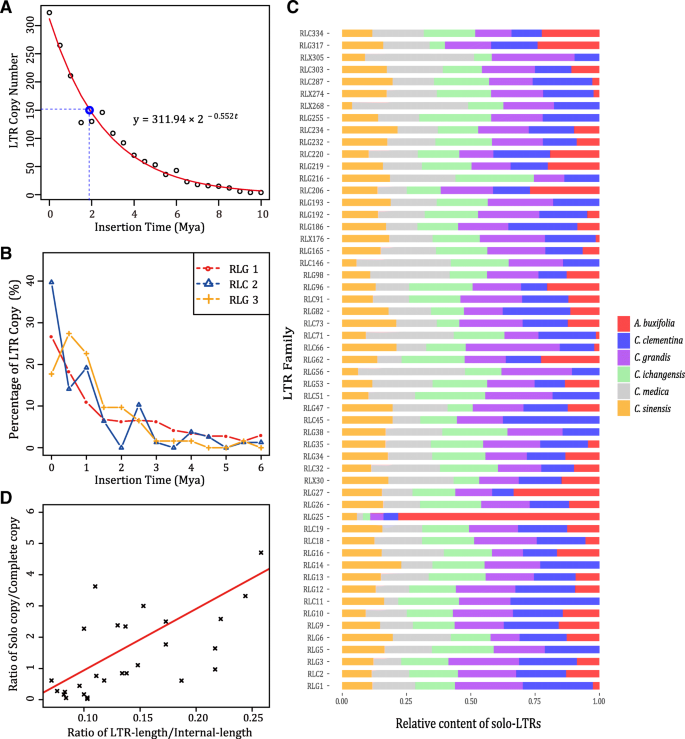
一般来说,LTR反转录转座子在一个长期的循环中不断插入和消除,并保持宿主基因组大小的动态平衡。通过计算LTR反转录转座子的插入时间,我们得到LTR反转录转座子插入时间曲线(图。6.a),发现LTR反转座子插入时间呈指数分布,半衰期在柑橘类是〜180万年。同时,我们注意到,只有几个LTR回收器家庭在可跟踪的过去中活跃,这与前一项研究一致[23.].在c .茅那only eight families, i.e., RLG1, RLG2, RLG3, RLG4, RLG5, RLC7, RLG9 and RLG12 contained more than 30 complete LTR retrotransposons, and the member of RLG1 (476) were larger than the total members (322) of the other seven families. In addition, LTR retrotransposon families with the most copy numbers of complete LTR retrotransposons (such as RLG1, RLG2, RLG3) were usually active recently (Fig.6.b)表示,通过不平等的同源重组和非法重组,从基因组快速地除去古代LTR转回转储。虽然完整的LTR回收分子拷贝的副本高度依赖于基因组完整性,但是Solo-LTR的副本表现出较少的依赖(表4.).因此,我们比较了不同的LTR回收吊装家庭的副本来调查独奏LTR是否扮演了任何角色柑橘类基因组多样性(图。6.c).在6个家族中,有些家族(如RLC2、RLG5和RLG16)的拷贝数相似柑橘类基因组,表明这些LTR回收转换在不同的过程中不太活跃和发散柑橘类我们称它们为稳定的LTR家族。而在6个家族中,一些不稳定的LTR家族组成明显不同柑橘类物种。例如,发现136个单独的ltr属于RLG25答:buxifolia,但在其他5中的每一个柑橘类基因组少于10个拷贝。以上发现表明RLG25可能在答:buxifolia通过比其他5的不等重组累积了独奏LTR的更多副本柑橘类物种。
插入时间可能有助于单独长链的形成
为了避免Miss Innoto-LTR的注释,主要由完整的LTR回收器和截短的LTR回收器复合的内部不平衡同源重组和截断的LTR回收分子,只有在有两个4〜6bp精确的TSD侧翼时才将区域视为SOLO-LTR。与以前的研究不同,仅采用完整的LTR回收路标,因为没有TSD的LTR回收分子可能是装配误差,边界注释误差和元素间不等重组的结果,这显示出罕见拟南芥[15.].我们取S/C值(单独-LTR/完全LTR)来评估单独-LTR形成与一些相关因素的关系(图。6.d)。我们的结果揭示了S / C和LTR回收ansposons插入时间之间的显着相关性(Spearman的等级相关性R. = 0.455,P.-value < 0.01),与大豆的结果不一致[22.].S/C与LTR大小之间存在相似的显著相关(Spearman秩相关)R. = 0.627,P.值< 0.01)。我们证实了S/C与LTR/INTERNAL比值之间存在很强的相关性(Spearman秩相关)R. = 0.691,P.-value < 0.01),与El-Baidouri的研究结果一致[23.].综上所述,我们发现solo-LTRs形成与几个因素有关,和更长的LTR将有利于更稳定的一双5’和3’LTR如果他们不太遥远,并且插入LTR反转位子活动时间越长,不平等的重组的可能性就越高。
讨论
来自多个基因组的螨虫和LTR回收的注释和比较提供了新的洞察力柑橘类进化和基因组多样性
虽然少数研究揭示了螨和LTR反转录转座子在肿瘤中的重要作用柑橘类[20.那24.那27.的关键作用柑橘类进化和多样性长期被忽视。这是第一项研究,重点关注螨虫和LTR回收转运展示和比较柑橘类调查它们的作用,尤其是在基因组多样性和进化中。螨虫和LTR Retrotransposons为有兴趣的研究人员提供了有用的资源柑橘类螨虫和LTR回收器。
螨的插入柑橘类基因组导致巨大的多态性,其中螨插入多态性(MIP)的基因间多态性比远高于内部内部,这反映了不同物种之间的遗传关系。螨虫和LTR回收的比较揭示了一些螨虫和LTR回收器在相对稳定的螨虫中柑橘类基因组的组成差异不大柑橘类例如DTM53和RLC2。相比之下,一些螨和LTR反转录转座子非常不稳定,如DTM63和RLG25,它们极大地重塑了柑橘类基因组和一些螨甚至在基因结构变异中发挥作用。其他植物是否在不同物种间表现出相似的MIP,是否具有相同的螨虫和LTR反转座子稳定和不稳定的组成模式,尚有待观察。
螨和LTR反转座子:异同
螨虫和LTR回收器是最重要的转座子类型,在植物基因组中具有高度特征。螨虫占2.51〜2.90%,而LTR回收转换占6种不同的20.66%至29.31%柑橘类物种。一般情况下,LTR反转座子的平均长度远长于螨,占基因组的绝大部分。螨虫和LTR逆转录转座子都由几个家族的全长(完整)拷贝所主导。然而,这两种转座子的分布差异很大,LTR反转座子相关序列在着丝粒近端区域富集,而mite相关序列相对均匀分布在染色体上(图)。7.广告)。螨虫和LTR回收转回都可以插入启动子区域以调节下游基因表达,如前一项研究所揭示的[20.那27.].
DTM63活跃于可跟踪的过去,并在基因结构变化中发挥作用
6号DTM63拷贝数的巨大差异柑橘类以及Ke等人在柑橘芽突变体中发现的DTM63新插入[41]表示DTM63可能是活动的突变类螨家族在可追踪的过去和可能仍然活跃柑橘类.DTM63的平均长度为约150bp。我们发现了一些在启动子区域中的DTM63插入的病例,表明基因结构变异中的DTM63潜在作用,可以调节下游基因表达(附加文件4.:图S4;在附加文件中列出的相应区域7.:表S3)。在我们之前的研究中,我们报道了螨插入参与了核多胚的出现柑橘类[27.,这也突出了螨在启动子区域的关键调控作用。在进一步的研究中,DTM63插入是否会改变下游基因的表达,甚至引起表型突变柑橘类将被调查。
完整的LTR反转座子的数量高度依赖于基因组的完整性
我们注意到完整的LTR回收器数量高度取决于基因组完整性。例如,c .茅Pacbio长reads (contig N50, 10.62 Mb)和c·克莱门蒂娜采用Sanger测序法(contig N50, 115.9 kb)进行测序,其余4个基因组采用下一代测序法(NGS)进行测序和组装(所有的contig N50 < 80 kb)。因此,在c .茅是最高的,其次是c·克莱门蒂娜,而其他4个基因组的完整LTR反转座子较少。5 'ltr和3 '当你完整的LTR反转位子活动,特别是新插入的LTR反转位子活动应该是相同的,但在这种情况下,挥动组装软件倾向于破坏5 'ltr和3 'ltr成不同的叠连群,虽然长的读取横跨5 ' / 3 'ltr与旁侧序列通常克服这个biasness。螨的长度相对较短(通常< 800 bp),因此全长螨的数量基本不受基因组完整性的影响。
全基因组比较揭示了LTR转回转移在基因组多样性中的作用
我们对整个基因组LTR的注释和比较,使我们深入了解了不同LTR逆转录转座子家族在基因组多样性中的作用。近年来,一些LTR反转录转座子的激活程度较低,很少有新的单独LTR形成,导致不同LTR之间的差异较小柑橘类基因组,而有些家族在不同的家族之间是高度分化的柑橘类并可能在其中起作用柑橘类基因组的多样性。正如我们上面所强调的,完整的LTR逆转录转座子的数量高度依赖于基因组的完整性,进一步的研究更完整柑橘类需要基因组来验证完整的LTR反转录转座子是否显示出单独LTR的相同模式。虽然插入了CopiaMYB转录激活子Ruby上游的类逆转录转座子调控Ruby的表达[20.,进一步揭示了LTR插入对下游基因的调控作用。其他ltr插入是否表现出类似的效果,甚至影响表型还有待观察柑橘类与否。
结论
在这项研究中,我们专注于6次螨虫和LTR回收吊装柑橘类各基因组中与螨虫相关的序列约占3%,而LTR反转座子约占21.2% ~ 33.1%。高拷贝数螨科以单轮扩增为主。其中DTM63可能是最早发现的活性物质突变类螨科柑橘类.甜橙中12.9%的小rna来源于螨虫相关序列,表明螨虫具有重要的调控作用。螨的插入柑橘类属间MIP比例远高于属内,反映了遗传关系。此外,通过比较染色体上的LTR反转座子含量,我们发现LTR主要富集在着丝粒的近端区域,并且单独的LTR的形成与LTR插入时间、LTR大小和LTR/INTERNAL比值呈正相关。LTR-RTs的插入和消除是通过动态平衡来完成的柑橘类而LTR-RTs的半衰期较长拟南芥、大米和Medicago Truncatula..这些发现为了解螨和LTR反转座子及其在不同物种基因组多样性中的作用提供了依据柑橘类物种的基因组。
方法
基因组序列
黄柏、黄柏那c .茅那C. ichangensis和C. medica由我们进行排序[27.],可于c . sinensis注释项目数据库(http://citrus.hzau.edu.cn/Orange/).而且,基因组c·克莱门汀是下载柑橘类基因组数据库(https://www.citrusgenomedb.org/).
MITE注释、手工保存和分类
MITE-Hunter [31.](默认参数)用于从选择的基因组中搜索所有的螨候选基因组,然后进行UCLUST, identity 80%用于对候选基因组进行聚类。我们从每个聚类中选取至少1个具有代表性的样本,对TSD和末端倒置重复序列(TIR)进行人工校正。我们去掉了不具有TSD和TIR特征的假阳性序列,然后去掉长度大于800 bp的序列,使f41与之前的研究保持一致。由于一些序列发生了变化,我们使用相同的参数执行第二次聚类(族)。螨超科分类参考了以前的研究[42].最后,我们从每个家族中手工选取一个具有完整TIR和TSD的螨作为代表进行构建柑橘类螨数据库。重复掩码4.0.2(参数" -pa 6 -s -nolow -xsmall - exn ") [43]用于注释所有6个选定基因组的所有与螨虫相关的序列,并编写一个内部Perl脚本来检索所有与螨虫相关的序列(附加文件1:图S1)。
螨放大方式和时间
螨相关序列覆盖了90%以上的代表,以全长螨为研究对象。肌肉(44]对同一科的全长螨类进行比对,然后使用MEGA6 [45]用邻居连接法构建系统发生树。根据Jukes-Cantor方法[46,我们编写了一个Perl脚本来计算每个MITE族的成对散度。采用猕猴桃平均替代率为2.81 × 10−9编码区每年每同义地点的替换量[47],非编码区速率的2倍(R.= 5.62 × 10−9)和公式t = k / 2r [13.,我们估计了几个高拷贝数的螨科的扩增时间。
推定突变-iker dde地区c . sinensis和c·克莱门蒂娜用TARGeT (E.值< = 0.01)管道48].检索到推定区域的上游和下游的侧翼序列。写入Perl脚本以查找5'-ggacttg-3',5'-caggtcc-3'(允许在终端中的1个不匹配)的保守终端序列,以及所有候选者的9〜10bp tsd。
螨类的P/A多态性
从所有六个基因组中检索到全长螨虫的上游和下游的侧翼序列。首先,使用BRASK对与其他五个基因组的侧翼曲线与其他五个基因组对齐(E.价值<10.−50).然后将甜橙的侧翼序列与其他5个基因组进行比对。如果这对侧翼序列是最佳的,锚定在同一支架/染色体、同一条链上,且两个锚定位点之间的距离小于1 kb,则认为该螨位点是等位基因。如果在上述目标等位基因中存在同一螨家族的相关序列,则认为在两个基因组中均存在螨插入,否则认为在查询基因组中存在螨插入,而在目标基因组中不存在。根据螨的配对关系,建立了螨的基因座指数c . sinensis其他五个柑橘类物种,分析其六个六个地区的分销和作用柑橘类基因组。我们将基因转录起始位点的前1 kb序列区域定义为启动子区域。Bedtools [49v2-2.26.0用于鉴定不同基因组区域(5'UTR, 3'UTR,内含子,基因间,启动子)中螨虫的插入/存在。MEME套件的DREME工具(http://meme-suite.org/tools/dreme)进行MOTIF分析。编写了几个内部PERL脚本来测量中螨的相对丰度密度柑橘类基因组。
螨虫小RNA
为了防止由不完全螨引起的相对位置统计的偏差,从每个家庭中选择差异的所有螨虫的序列的长度差异的差异。Bowtie2 [50允许碱基错配的方法被用来比较先前研究中得到的小rna与所选的螨序列。小rna被认为来自于螨,如果它们显著与螨对齐。
LTR反转座子的注释和分类
tRNAScan-SE [51],用默认参数预测6个物种的tRNAs,然后过滤伪基因和冗余序列,将所有tRNAs合并,制成一个柑橘类TRNA数据库。使用TRNA数据来预测LTR_FINDER的LTR回收器的PBS [52],利用开发的tRNA数据库。LTR_FINDER参数“- w2”用于预测LTR反转录转座子。类似地,在之前的研究中[53],进行聚类和过滤以减少假阳性,进一步分析只考虑5’tg、5’ca、PBS、PPT、3’ca、3’tg的候选人。附加文件中逐步显示了LTR的注释工作流程2:图S2。
LTR反转录转座子系统发育树
采用内部序列三帧平移,hmmsearch (e值≤10)- 6)用于搜索的侧面吉普赛-象RT (PF00078)和Copia例如RT (PF07727)。RT域氨基酸序列长度大于100个aa。我们从每个家族中选择一个典型的RT序列,并从序列中去除过早终止密码子。结合我们之前的研究中收集的代表,使用Muscle对RT序列进行比对。MEGA6 [45](默认参数),采用邻居连接法构建系统发生树。
LTR回析碳膜对染色体的分布
利用以上LTR注释结果,以甜橙为例,将染色体拆分为1 Mb的窗口,在进一步分析中不考虑每条染色体的窗口< 1 Mb。计算LTR在染色体上的分布。
当你插入时间
与转座过程一样,新生成的LTR逆转录转座子只会以其中一个LTR作为模板。这样,新生成的LTR逆转录转座子的5'LTR和3'LTR将是相同的。在进化过程中,5'LTR和3'LTR可能会自发积累突变。LTR插入时间(T)公式T = k/2r,采用替代率为R.= 5.62 × 10−9每年计算的替换计算。使用肌肉用于对准每个完整的LTR回收的5'-LTR和3'-LTR,并通过JUKES-CANTOR方法校正LTR之间的距离[46].
此外,我们还推导出LTR在指数分布中的插入时间。采用R语言拟合非线性最小二乘(NLS)函数(拟合方程y = a∗ 2B.∗X),初始值a = 270, b =−0.5和指数函数,我们解释了LTR插入的次数和时间。此外,对于具有较高拷贝数的LTR家族柑橘类物种,分别计算每个构件的插入时间,然后映射家庭成员放大时间。
Solo-LTR形成
c .茅是用PacBio测序技术测序的,所以我们选择了c .茅作为Solo-LTR形成分析的代表。本研究中未包含少于10个完整的LTR回收的家庭,因为某些家庭成员的错误注释和遗漏是不可避免的,并且具有更完整的LTR回收滚装的家庭将更加强大。由于Solo-LTR只有一个LTR,用于完整的LTR回收器的方法对于独立的LTRS研究不可行。因此,我们使用了每个系列的完整LTR回收器的平均插入时间来表示Solo-LTR的插入时间。还计算平均内序长度。使用R软件完成S / C与LTR插入时间,LTR大小和LTR /内部之间的关系的计算。
缩写
- LTR:
-
长终端重复
- 螨虫:
-
微型倒置重复转换元素
- 再保险:
-
逆转录酶
- srna:
-
小rna
- TSD中:
-
目标站点重复
- TSS:
-
转录开始网站
- TTS:
-
转录终止位点
参考
- 1。
局TE,罗纳德PC,Wessler SR。基于计算机的系统调查揭示了野生型水稻基因中的小倒置元素的优势。Proc Natl Acad Sci U S A. 1996; 93:8524-9。
- 2。
微反重复转座元件(Miniature inverted-repeat transposable element, MITEs)是通过扩增的方式积累起来的,在植物基因表达和物种多样性中起着重要的作用栽培稻.mol Biol Evol。2012; 29:1005-17。
- 3.
王志强,王志强,王志强,等。植物转座因子与基因组学的关系。Nat Rev Genet, 2002; 3:329-41。
- 4.
Smit A, Riggs AD。人类基因组中的跳跳虎和DNA转座子化石。美国国家科学学院学报1996;93:1443-8。
- 5.
姜n,宝z,张x,hirochika h,eddy sr,mccouch sr,wessler sr。米中的活跃的DNA转座子家庭。自然。2003; 421:163-7。
- 6.
杨国华,杨国华,杨国华,杨国华。基因转位的调控:隐匿性螨异常活动的分子决定因素。科学。2009;325:1391-4。
- 7.
Oki N,Yano K,Okumoto Y,Tsukiyama T,Teraishi M,Tanisaka T.一种基因组视图的微型倒置重复转移元素(螨虫),栽培稻ssp。japonica..基因与遗传。2008;83:321-9。
- 8.
Zerjal T,Joets J,Alix K,Grandbastien Ma,Tenaillon Mi。玉米基因组三个旅游螨系列中的对比进化模式和靶特异性。植物mol biol。2009; 71:99-114。
- 9.
Piriyapongsa J, Mariño-Ramírez L,约旦IK。从转座因子研究人类microrna的起源与进化。遗传学。2007;176:1323-37。
- 10。
Piriyapongsa J,约旦IK。植物转座子对sirna和miRNAs的双重编码。RNA。2008; 14:814-21。
- 11.
Sampath P,Murukarthick J,Izzah NK等人。20微型倒置重复转换元素家族的基因组比较分析Brassica Rapa.和b . oleracea.《公共科学图书馆•综合》。2014;9:e94499。
- 12.
在小麦科、水稻和拟南芥中,copia反转录转座子的全基因组比较分析揭示了保守的古代进化谱系和单个copia家族的独特动态。基因组研究》2007;17:1072 - 81。
- 13。
MA J,Bennetzen JL。近期水稻核基因组的快速增长和分歧。Proc Natl Acad Sci U S A. 2004; 101:12404-10。
- 14.
陈志刚,陈志刚。水稻LTR反转录转座子的克隆与表达分析。中国水稻科学(英文版);2003;
- 15.
德沃斯KM,布朗JK,贝内特岑JL。在拟南芥中,通过非法重组减少基因组大小可以抵消基因组扩展。基因组研究》2002;12:1075-9。
- 16.
Bennetzen JL。开花植物基因组膨胀与收缩的机制和率。genetica。2002; 115:29-36。
- 17.
Hawkins JS, Kim H, Nason JD, Wing RA, Wendel JF。转座元件的差异谱系特异性扩增是基因组大小变化的原因Gossypium..基因组研究》2006;16:1252 - 61。
- 18.
Kashkush K, Feldman M, Levy AA。反转录转座子的转录激活改变了小麦中相邻基因的表达。Nat遗传学。2003;33:102-6。
- 19.
发夹rna和逆转录转座子LTRs影响RNAi和染色质基因沉默。科学。2003;301:1069 - 74。
- 20。
张永强,张永强,张永强,等。反转录转座子控制血橙中特定的、冷依赖的花青素积累。植物细胞。2012;24:1242-55。
- 21。
萧H,江南,Schaffner E,Stockinger EJ,Van der Knaap E.一种转回转录介导的基因复制隐蔽了番茄果实的形态变异。科学.2008; 319:1527-30。
- 22.
杜军,田志,韩春生,等。开花植物ltr反转录转座子的进化保护、多样性和特异性:来自全基因组分析和多特异性比较的见解植物j . 2010; 63:584 - 98。
- 23.
El Baidouri M,Panaud O.植物王国的比较基因组古生物学揭示了TE驱动基因组进化的动态。基因组Biol Evol。2013; 5:954-65。
- 24.
Bernet GP,AsínsMJ。吉普赛等吉普赛人的鉴定和基因组分布柑橘类和Poncirus..Al Appl Genet。2003; 108:121-30。
- 25.
徐强,郭伟,邓旭东。利用bac末端序列挖掘、表征和利用新的SSR标记柑橘类.Mol Biol rep 2012; 39:5373-86。
- 26.
徐Q,Chen Ll,Ruan X等人。甜橙的基因组草案(柑橘sinensis.).NAT Genet。2013; 45:59-66。
- 27.
王X,徐y,张某,等。原始,野生和栽培的基因组分析柑橘类提供对无性繁殖的见解。NAT Genet。2017; 49:765-72。
- 28.
吴国强,王志强,王志强,等。不同的柑橘,柚子和橙子基因组的测序揭示了复杂的历史期间的混合柑橘类驯化。生物科技Nat》。2014;32:656 - 62。
- 29.
关键词:水稻,基因组资源,遗传多样性,遗传多样性柑橘克莱门蒂娜: 3个深度覆盖BAC文库的特征和46000个BAC末端序列的分析。BMC基因组学。2008;9:423。
- 30.
Wu Ga,Terol J,Ibanez V等人。原产地和演化的基因组学柑橘类.大自然。2018;554:311-6。
- 31。
《螨虫猎人:从基因组序列中发现微小反重复转座元件的程序》。核酸学报2010;38:e199。
- 32。
杨克,李毅,江y,施X,凯特邦特S,Hall Tc。可转换元素的两端角色Kiddo.在米疹2启动子。植物细胞。2005; 17:1559-68。
- 33。
张峰,张海涛,张海涛,等。突变和大量转座子扩增对水稻基因表达的意外后果。大自然。2009;461:1130-4。
- 34。
柳条t,sabot f,hua-van a等。真核转换元素的统一分类系统。NAT。Rev. Genet。2007; 8:973-82。
- 35。
田志强,李志强,杜建军,等。基因重组和基因密度是否决定了水稻长端重复反转录转座子的DNA消除模式?基因组研究》2009;19:2221-30。
- 36。
等。高粱双色基因组与禾本科植物的多样性。大自然。2009;457:551-6。
- 37。
杜建军,田志强,王志强,马建军。转基因大豆中转录转座子相互作用的研究进展。植物细胞。2010;22:48 - 61。
- 38.
王鹤,刘杰斯。LTR RODRANSPOSON景观Medicago Truncatula.:清除速度比大米快。BMC基因组学。2008;9:382。
- 39.
ltr -逆转录转座子多样性及其对该属进化的影响直升机(l)。BMC基因组学。2017;18:634。
- 40。
多明戈,克鲁兹GM,梅特卡夫CJ,等。植物ltr反转录转座子在精细的家族水平上的分析揭示了个体的分子模式。BMC基因组学。2012;十三1。
- 41。
柯林,余辉,徐辉,谢志刚,邓旭,徐强柑橘类利用转座子显示技术获得芽型突变体。园艺学报,2017;44:1207-16。
- 42。
韩永强,秦胜,魏斯勒。谷草2类转座元件超家族分辨率的比较研究。BMC基因组学。2013;14:1。
- 43。
Zhi D,Raphael Bj,Price Al,Tang H,PEVZNER PA。在大型基因组中识别重复域。基因组Biol。2006; 7:R7。
- 44。
Edgar Rc。肌肉:高精度和高吞吐量的多个序列对齐。核酸RES。2004; 32:1792-7。
- 45。
Tamura K, Stecher G, Peterson D, Filipski A, Kumar S. MEGA6:分子进化遗传学分析版本6.0。生物学杂志。2013;30:2725-9。
- 46。
jukes th,cantor cr。蛋白质分子的进化。哺乳动物蛋白质代谢。1969; 3:132。
- 47.
史涛,黄海涛,黄海涛,等。猕猴桃及其亲缘植物进化过程中基因组的古老复制。安植物学。2010;106:497 - 504。
- 48.
目标:一个基于网络的管道,用于从基因组序列中检索和表征基因和转座元件家族。核酸学报2009;37:e78。
- 49.
昆兰AR, Hall IM。BEDTools:用于比较基因组特征的一套灵活的工具。生物信息学。2010;26:841-2。
- 50.
Langmead B,Salzberg SL。与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9:357-9。
- 51.
tRNAscan-SE:用于改进基因组序列中转移RNA基因检测的程序。核酸Res. 1997; 25:955-64。
- 52.
Xu Z, Wang H. LTR_FINDER:一个预测全长LTR反转录转座子的有效工具。核酸学报2007;35:W265-8。
- 53.
Steinbiss S, Willhoeft U, Gremme G, Kurtz S. de novo预测LTR逆转录转座子的精细注释和分类。核酸Res. 2009; 37:7002-13。
确认
提交人士要感谢高级编辑和宣传编辑,以及慷慨地审查我们的工作的匿名审查员,并给予我们对改进的有用建议。
资金
中国国家自然科学基金(31871269,31571351)和中央大学的基本研究资金(2662017点)支持这项工作得到了中国重点研究和发展方案(2018YFD10001)的支持(2018年,2018年)(31871269,31571351)。作者非常感谢这些组织。
数据和材料的可用性
在这项研究中产生或分析的所有数据都包含在这篇发表的文章中[及其附加文件]。
作者信息
隶属关系
贡献
YL和MTQ为分析的构思和设计、数据的获取、数据的分析、实验的进行以及手稿初稿的撰写做出了贡献。JWF、YD和SW贡献了LTR反转录转座子和螨的生物信息学分析。GW、LK和QX对基因组数据进行分析,并对手稿进行编辑/审阅。LLC获得资助,参与构思和设计的分析和数据获取,监督和分析数据,完善稿件。所有作者阅读并批准了最终的手稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用
同意出版
不适用
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
附加文件1:
图S1。螨分类标注流水线。多克斯(151 kb)
额外的文件2:
图S2。LTR反转座子的分类与注释。多克斯(475 kb)
额外的文件3:
图S3。螨科相对丰度柑橘类物种。(docx 1605 kB)
额外的文件4:
图S4。手动确认DTM63插入位置。(一)人工确诊病例检测结果。(b)。手动确认的情况制成。(c)手动确认的情况c·克莱门蒂娜.(D)人工确诊病例c . ichngensis.蓝色表示TSD和“N”中的“N”代表了复制全长DTM63。相应的同源区域列于附加文件中7.:表S3。(DOCX 584 KB)
额外的文件5:
表S1。不同基因组区域间保守和非保守螨插入信息(docx12kb)
额外的文件6:
表S2。新插入螨在三个不同基因组区域的信息柑橘类物种。(多克斯13 kb)
额外的文件7:
表S3。DTM63插入位点信息。(多克斯14 kb)
额外的文件8:
数据集S1。螨虫的种子文件。(FA 43 kb)
额外的文件9:
数据集S2。LTR反转录转座子的种子文件。(FA 2229 kb)
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
刘,Y.,塔希尔Qamar,M.,Feng,JW。等等。微型倒置重复转换元素(螨虫)和长终端重复(LTR)回复六的比较分析柑橘类物种。BMC植物杂志19,140(2019)。https://doi.org/10.1186/s12870-019-1757-3
已收到:
公认:
发表:
关键词
- 活跃的转座子的家庭
- 柑橘类
- 基因组多样性
- 长终端重复
- Retrotransposons.
- 微型反重复转座元件(螨)