抽象的
背景
与白肉甘薯(WFSP)相比,紫肉甘薯(PFSP)块根中富含花青素,是一种理想的功能性食品开发资源。有研究表明,miRNA介导的表达调控在植物花青素生物合成中发挥重要作用。然而,目前对甘薯块根中与花青素合成相关的mirna及其功能了解甚少。
结果
在该研究中,分别构建了来自WFSP(XUSHU-18)和PFSP(Xuzishu-3)的小区根部的小RNA(SRNA)和降低组文库。通过SRNA测序鉴定了191个已知的和33个新的miRNA,通过降低测量测序鉴定了180个已知的IB-miRNA和5个新型IB-miRNA的180个靶基因。其中,121个miRNA在Xushu-18和Xuzishu-3之间表达了不同的表达。SRNA的综合分析,降低测量,GO,KEGG和QRT-PCR显示,26个差异表达的miRNA和36个相应的靶标可能参与花青素生物合成。其中,IB-MIR156表达与其目标之间的反比相关性IBSPL.通过qRT-PCR和sRNA测序检测WFSP和PFSP的表达。随后,ib-miR156在拟南芥.有趣的是,过表达ib-miR156的植物表现出抑制SPL.和紫色表现型。同时,在转基因植株中检测到四种花青素途径基因的表达上调拟南芥植物。最后,提出了甜菜蛋白中涉及花青素生物合成的推定的IB-miRNA-靶模型。
结论
该结果为研究甘薯中与花青素积累相关的miRNA的表达谱提供了重要线索,为了解块茎作物中miRNA介导的花青素生物合成调控网络提供了重要线索。
背景
地瓜(番薯甘薯L.),六倍倍增(2n = 6x = 90)二坐子植物旋花科植物家庭,是世界各地的重要作物,因为其高产,适应性宽,富裕营养丰富[1].茎根的肉有多种颜色,如白色,黄色,橙色和紫色[2].紫色肉质甘薯(PFSP)不仅是白皮甘薯(WFSP)的营养,而且还富集了高含量的花青素。花青素是属于类黄酮基团的水溶性颜料。许多研究表明,花青素具有强烈的抗氧化活性,因此对肥胖症和癌症等多种疾病具有治疗作用[3.].更重要的是,PFSP的花青素苷具有较高的热稳定性和光稳定性,酰基化程度高于草莓、覆盆子和苹果[4.那5.那6.].因此,PFSP最近被提出作为一种潜在的药物作物,用于开发药物,如抗肿瘤、抗炎和抗氧化剂[6.那7.].
花青素生物合成途径在植物中得到了广泛的研究[8.那9.].参与花青素生物合成的基因可以分为结构基因和调节基因。结构基因编码一系列酶,包括苯丙氨酸氨基丙氨酸(朋友)、查尔酮合酶(CHS)、查尔酮异构酶(气)、类黄酮3 ' -羟化酶(F3'H),黄酮醇-3'5'-羟化酶(F3'5'H.),黄烷酮3-羟基化酶(F3H)、二氢黄酮醇还原酶(DFR.)、udp -葡萄糖类黄酮3-o-糖基转移酶(UFGT)和花青素合成酶(答)[10].这些酶基因可被MYB-bHLH- wdr (MBW)复合物转录调控,该复合物由MYB、bHLH和WD40重复[11].此外,其他调控基因也被报道调控花青素生物合成,如组成型光形态建成1 (COP1)[12],斋月Zim-域(JAZ)[13],Squamosa启动子结合蛋白样(SPL.)[14),而南汽[15].除了结构基因和调节基因外,最近的证据表明,MicroRNAS(miRNA)还可以在培养植物中的花青素生物合成中发挥重要作用[16那17那18].
MiRNA广泛存在于真核生物中,是一种内源性非编码小RNA,长度约为20-24 nt。大量证据表明,mirna主要通过切割靶mrna或在转录后水平上阻止基因翻译参与调控基因表达[19那20.].在植物中,miRNA参与多种生物过程,包括生长和发展[12,应激反应[21那22],植物素信令[23那24]以及次生新陈代谢。例如,已识别MiR156,MiR165 / 166,MiR828和MiR858参与其中的花青素生物合成拟南芥[14那16那17那25那26),茄属植物lycopersicum[18].通过高通量测序,越来越多的miRNA已被识别和特征在小麦中[27那28],大豆[29)、大米(30.],甜橙[31],芦笋[32],中国萝卜[33],玉米[34], 茶 [35)等。到目前为止,在最新的miRBase数据库中共记录了来自271种不同植物的38589个mirna (http://www.mirbase.org/).一些甘薯mirna和它们的靶标也已被确定[36那37].然而,在甘薯中参与调控花青素生物合成的mirna尚未有系统的报道。
在该研究中,使用SRNA和降低测量测序鉴定MiRNA及其相应的靶标,这些靶标可能参与甘薯的花青素生物合成。构建并测序来自WFSP的小核(品种Xushu-18,XS-18)和PFSP(品种Xuzishu-3,XZS-3)的小RNA和降低组文库。SRNA的综合分析,降低测量,GO,KEGG和QRT-PCR透露了IB-MiRNA群体的全面叙述,相应的目标,表达丰度以及可能参与花青素代谢的MIRNA。通过QRT-PCR验证了八种不同表达的miRNA的表达水平及其靶标,这与测序数据一致。此外,我们证明了IB-MIR156的过表达拟南芥强烈抑制丰富的丰富IBSPL.并导致紫色表型。最后,说明了与甘薯结节根系中与花青素生物合成相关的可能的IB-miRNA靶标调节模型。我们的调查结果提供了IB-MiRNA的综合表达分析,并提出MiRNA涉及在甘薯中调节花青素生物合成。
结果
甘薯块根中的小RNA群体
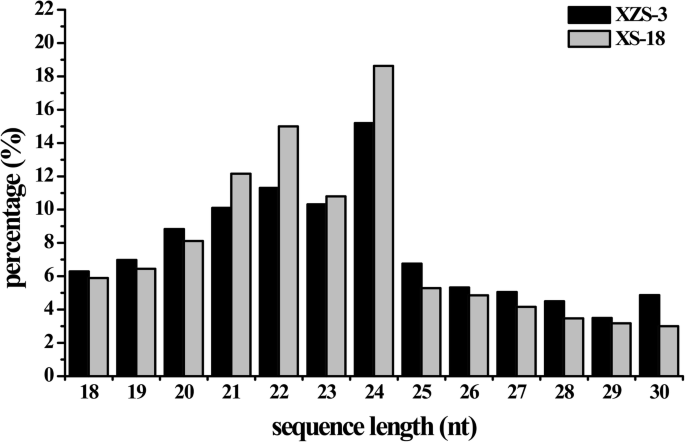
为了鉴定参与花青素生物合成过程的miRNA,WFSP(XS-18)和PFSP(XZS-3)的结节根分别用于构建SRNA文库并分别对Illumina Hiseq2000平台进行测序。产生了27,705,914(XS-18)和28,947,914(XZS-3)原始读数的总和。除去低质量读取和污染的适配器序列,分别从XS-18和XZS-3获得26,825,634(96.82%)和27,331,707(94.42%)清洁读数。总共15,764,489(XZS-3,91.61%)和13,904,486(XS-18,73.32%)映射到甘薯的相应组装的未植物(附加文件1).将映射的reads在Pfam数据库中进一步注释,并将其分为rrna、tRNAs、snrna、snoRNAs、ta-siRNA (TAS)等。内源性srna被鉴定为已知mirna和新的mirna(附加文件)2).SRNA的长度分布模式在两个库中类似。它们的范围为18至30nt,其中24 nt SRNA分别是XS-18和XZS-3库中最丰富的尺寸,18.63和15.19%(图。1).
甘薯中已知和新的ib- mirna的鉴定
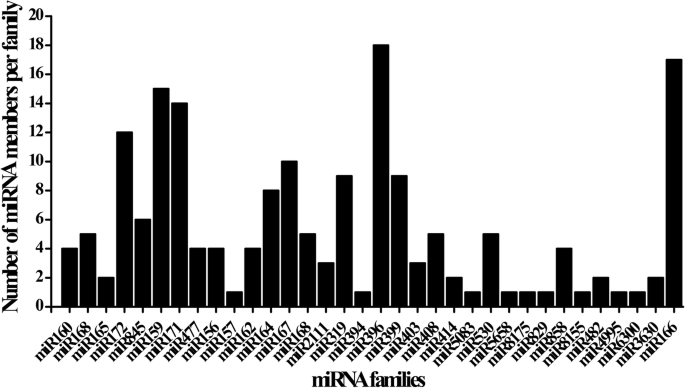
发现已知的miRNA在植物生长,发展和许多其他生物过程中是重要的[38].为了鉴定甘薯中已知的miRNA,将sRNA序列与储存在miRBase 22.1中的成熟miRNA序列进行比对。两个文库共鉴定出43个miRNA家族的191个已知miRNA,其中XS-18和XZS-3分别鉴定出185和145个已知miRNA(附加文件)3.).在鉴定的43个miRNA家族中,每个miRNA家族的成员数量差异显著。其中ib-miR396家族成员最多,为18人,其次为ib-miR166和ib-miR159家族,分别为17人和15人。相比之下,9个家族只有一个成员,如ib-miR157、ib-miR394、ib-miR5083和ib-miR5658(图2)。2).
miRNA的丰度估计为每百万份转录本(TPM),在43个已知的miRNA家族中差异很大(附加文件)4.).一些mirna在超过1000个TPM的情况下高表达,如ib-miR159a-1和ib-miR319a-1,而一些mirna在低于2个TPM的情况下低表达,如ib-miR160-1、ib-miR1030a-j和ib-miR1128。此外,同一miRNA家族中不同成员的表达水平也不同。例如,在miR159家族中,ib-miR159a-1在XS-18和XZS-3文库中分别以5978和1106 TPM丰度最高。而ib-miR159f在XS-18和XZS-3库中分别只出现了4个和1个TPM(附加文件4.).
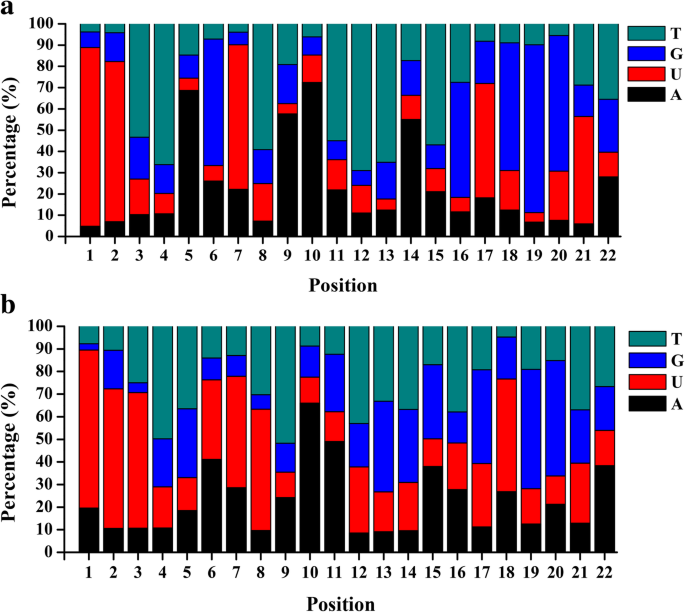
已知mirna的长度从18 nt到24 nt不等,其中21 nt的mirna数量最多(附加文件)3.).要了解甘薯麦芽育麦芽肉的基础偏好,我们分析了已知miRNA的每个位置的基本分布。在两个文库中,核苷酸序列分析显示尿苷(U)是5'末端最常见的核苷酸。靶基因的切割位点通常与miRNA的第10或第11核苷酸匹配。已知的IB-miRNA的大多数核苷酸在第10次和第11℃的第10次和胸腺嘧啶(T)或胸腺嘧啶(图11)处的腺嘌呤(A)(图。3.一种)。
将剩余的SRNA序列映射到甘薯转录组数据库,并且使用发夹结构来鉴定甘薯的新型miRNA。基于已发布的标准预测了新的miRNA [39].完全,在XS-18和XZS-3库中预先预测33和33个新颖的miRNA(附加文件5.).新mirna的长度分布在20 ~ 25 nt之间。与已知mirna相似,核苷酸偏倚分析显示U主要出现在新mirna的5 '末端(图5)。3.b).新的miRNA前体的长度从57 nt到293 nt不等,平均长度为140 nt(附加文件5.),这与植物中常见的miRNA前体长度一致[40].新miRNA前体的预测发夹结构和成熟miRNA在前体中的位置显示在附加文件中6..发夹结构的负折叠自由能量范围为-116.21至-22.20 kcal mol−1平均为−57.62千卡mol−1.最小折叠自由能量指数(MFEI)的范围为0.51至2.44,平均为1.31,大多数MFEIS> 0.8(附加文件5.).通常,互补miRNA星(miR *)股的存在对新的miRNA的真实性提出了强烈的支持[39].与已知的mirna相似,在甘薯中发现的新型ib- mirna的丰度差异很大。MiR*链被发现存在于33种新的ib- mirna中的17种,且表达水平低于相应的MiR链(附加文件)5.),其同意MIR * Strands大多降级的报告[40].
通过降解组测序验证ib-miRNAs的靶标
探索甘薯麦克纳斯,计算程序PSRNATarget的功能(http://plantgrn.noble.org/v1_psrnatarget/进行)进行以预测其靶基因。预计总共3278名转录物是224 MiRNA的目标(附加文件7.).然后进行高通量降解组测序,验证miRNA靶点。共获得1451万条原始reads, 1886192条unique reads,其中766328条unique reads与甘薯转录组数据库中汇集的参考序列相匹配(附加文件)8.).通过CleaveLand4分析,共鉴定出115个已知ib- mirna和5个新ib- mirna切割的180个靶基因(http://sites.psu.edu/axtell/software/cleaveland4/)(额外的文件9.).
根据目标站点的标签的相对丰度,将切割的目标转录物分为五类,名为0,1,2,3和4类别。在附加文件中显示了五类中的miRNA和相应的目标9..在180个目标中,0、1、2、3、4类目标分别为55个、8个、34个、1个和23个。
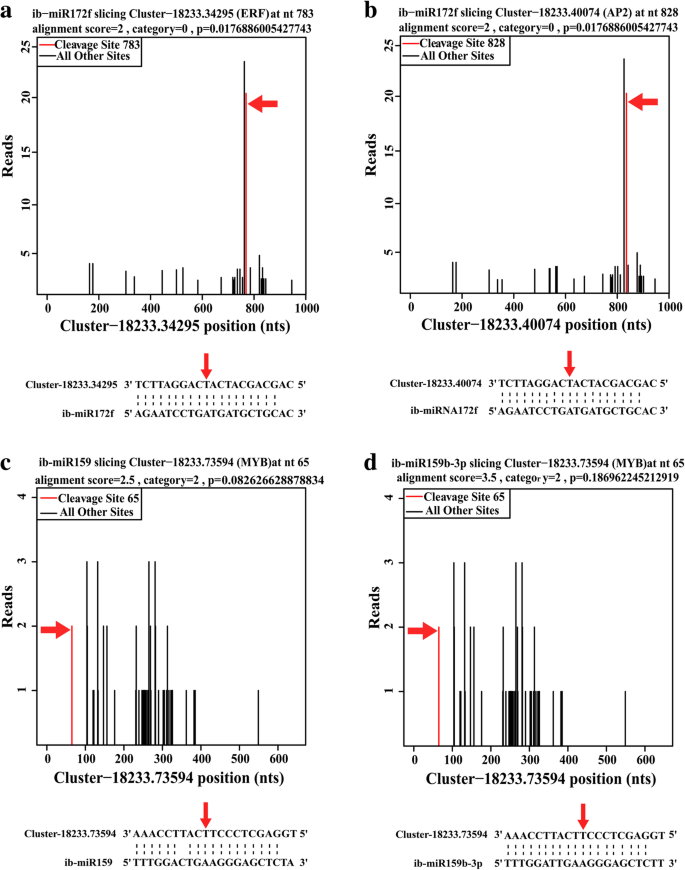
靶基因分析显示,ib- mirna的许多剪切靶基因转录本是TF基因,包括my那怀疑那南汽那SPL.那东盟地区论坛年代,小块土地那WDR.那HD-ZIPIII等(附加文件9.).许多IB-miRNA有多个成绩单作为靶基因。例如,IB-MIR172可以瞄准小块土地(集群- 18233 .34295集群- 18233 .74214),APETALA 2(集群- 18233 .40074集群- 18233 .74210) (图。4.a, b).此外,同一转录本被多个ib-miRNA靶向。例如,ib-miRNA159家族成员,ib-miR159、ib-miR159a、ib-miR159b-3p和ib-miR159c具有相同的靶标IBMYB.(集群- 18233 .73594) (图。4.C,D)。my基因(集群- 18233 .73594)也被ib-miR858、ib-miR319、ib-miR159和ib-miR156靶向(附加文件9.).
WFSP和PFSP之间ib-miRNAs的差异表达
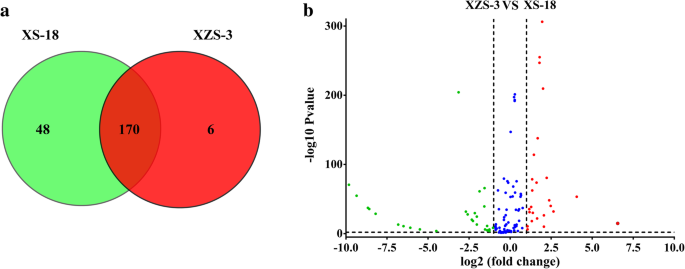
比较miRNA的正常化表达水平,比较XS-18和XZS-3文库,以鉴定差异表达的miRNA。遵循差异表达分析标准的已知和新的miRNA(QValue <0.01和log2(折叠变化)| > 1). The Veen diagram analysis showed that 170 miRNAs co-expressed between the two libraries. Forty eight miRNAs (41 known and 7 novel miRNAs) presented specifically in XS-18 while 6 miRNAs (ib-miR162b, ib-miR164c, ib-miR166m, ib-miR166j, ib-miR414 and ib-miR845d) particularly expressed in XZS-3 (Fig.5.一种;额外的文件10).火山图显示,与XS-18相比,XZS-3中有47个mirna表达上调,74个mirna表达下调。5.B;额外的文件10).
QRT-PCR验证IB-MIRNA及其靶基因
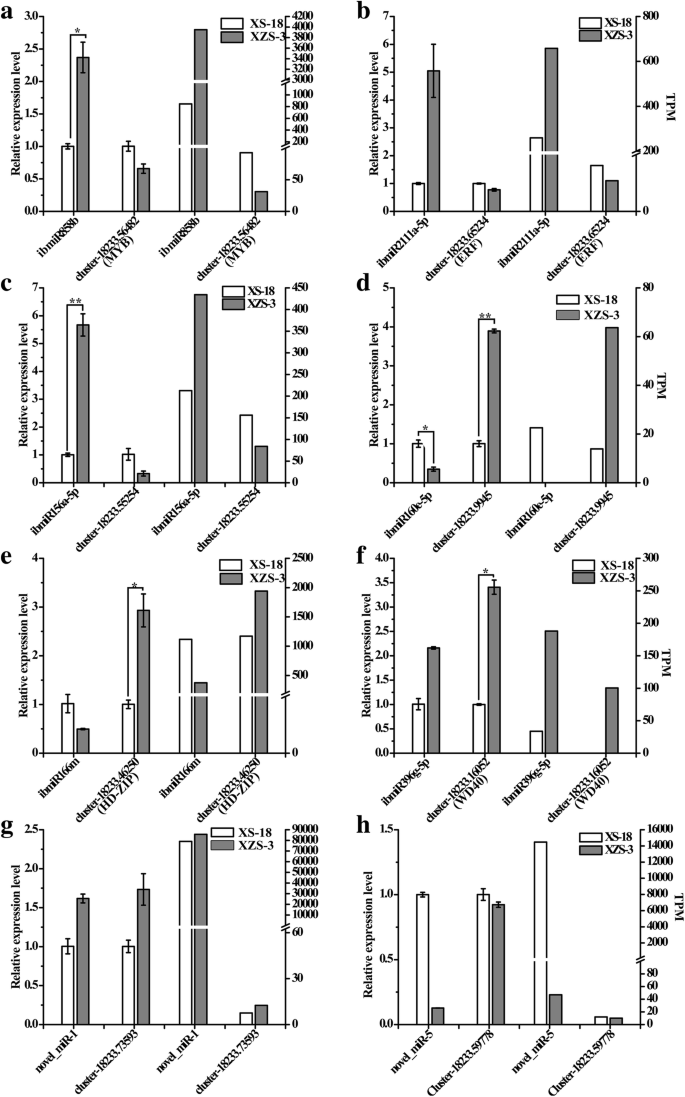
随机选取XS-18和XZS-3之间差异表达的8个mirna及其对应的靶标进行qRT-PCR,验证sRNA和降解组测序结果。总的来说,qRT-PCR所选取的8个mirna的表达模式与高通量测序结果一致,说明sRNA测序数据是可靠的(图)。6.).与XS-18相比,在XZS-3中检测到上调IB-MiR858B,IB-MIR2111A-5P和IB-MIR156A-5P的上调表达水平,而它们相应的靶基因的丰富my那小块土地和SPL.分别被调节(图。6.A,B,C)。IB-miR160e-5p和Ib-miR166m的表达量减少,而其相应的靶基因东盟地区论坛和HD-zip.与XS-18相比,XZS-3中的表达增加了(图。6.d,e)。miRNA与其对应靶之间的反向相关的表达模式确认了降低测量测序数据。然而,IB-MiR396G-5P,Novel-Mir-1和Novel-5的表达显示了与其相应的转录物相同的趋势,集群- 18233 .16052(WD40),集群- 18233 .73593(my),集群- 18233 .59778(东盟地区论坛),提示转录本可能不是这些mirna的剪切靶点(图。6.f,g,h)。表中列出了另外五个miRNA的表达1qRT-PCR也进行了验证(附加文件11).
通过差异表达IB-MiRNA调节的目标的Go和Kegg分析
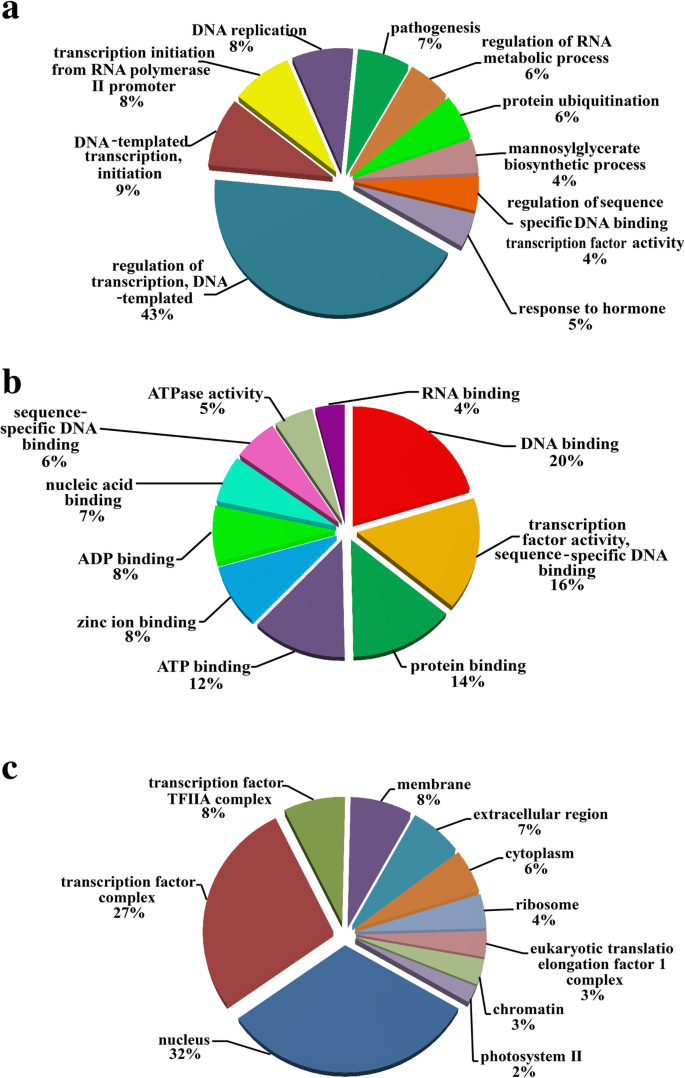
通过计算程序psRNATarget预测,共有3278个转录本成为224个mirna的靶标。然后,对通过降解组测序鉴定的180个靶标进行基因本体(GO)分析,在GO数据库中发现了124个靶标,分别涉及生物学过程(79个)、分子功能(83个)和细胞成分(71个)12).在生物过程类中,超过40%的靶基因参与了转录调控、DNA模板注释(GO:0006355),其次是DNA模板转录、起始(GO:0006352)和DNA复制(GO:0006260)。7.一种)。在分子函数类别中,三种最占优势术语是DNA结合(GO:0003677),序列特异性DNA结合转录因子活性(GO:0003700)和蛋白质结合(GO:0005515)(图。7.b).在细胞成分类别中,两种主要富集物是细胞核(GO:0005634)和转录因子复合物(GO:0005667)(图。7.c)。
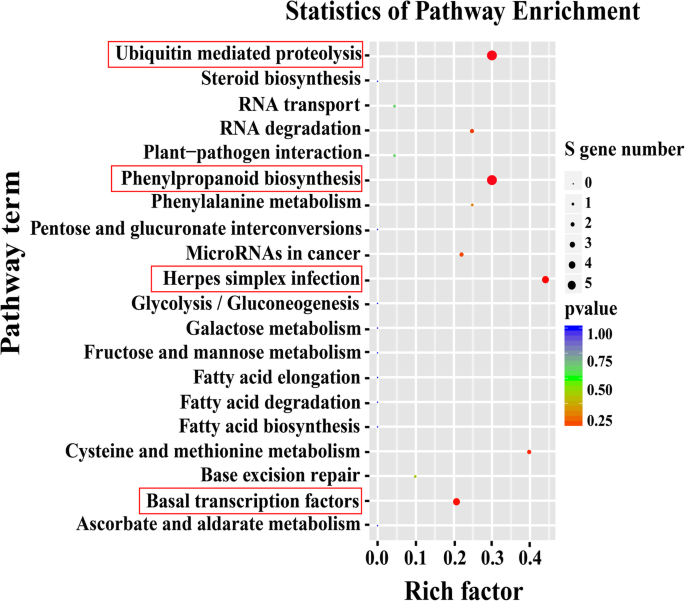
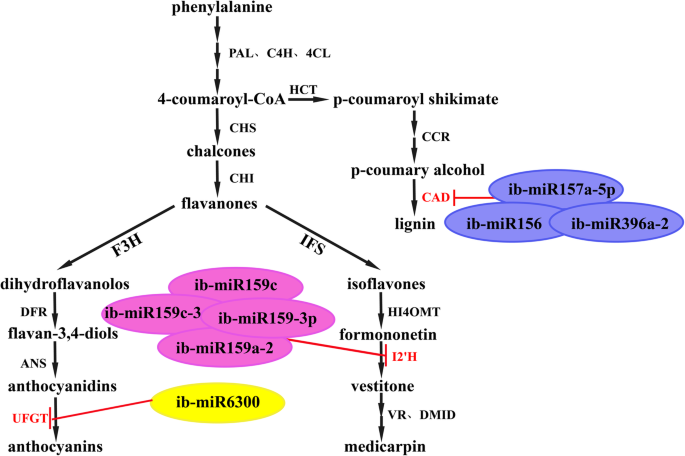
通过KEGG富集进行途径分析。12个不同mirna调控的22个靶点被分配到11个KEGG通路(图)。8.;额外的文件13).泛素介导的蛋白水解(ko04120, 5个靶点)和苯丙类生物合成(ko00940, 5个靶点)是两个最多的富集途径,其次是单纯疱疹感染(ko05168;4个靶标)和基础转录因子(ko03022;4目标)(无花果。8.;额外的文件13).花青素的生物合成来源于一般的苯基丙素代谢途径[41].在大多数物种中,与苯丙烷丙烷途径相关的基因和miRNA可以参与花青素生物合成。基于苯丙醇丙烷代谢中靶基因的功能,在甘薯在甘薯中提出了一种苯丙烷化调控的途径面板(图。9.).特别是8个ib- mirna (ib-miR157a-5p, ib-miR156, ib-miR396a-2, ib-miR159c;ib-miR159-3p、ib-miR159c-3、ib-miR6300和ib-miR159a-2参与甘薯中苯基丙素通路(图2)。9.),可能在调控甘薯花色苷的生物合成方面发挥重要作用。
鉴定IB-MiRNA及其与甘薯的花青素生物合成相关的目标
鉴定WFSP和PFSP在块根中差异表达的mirna有助于更好地理解参与花青素生物合成的mirna的生物学功能。结合sRNA、降解体测序、GO和KEGG分析,在121个差异表达mirna中鉴定出26个可能参与甘薯花青素生物合成(见表)1额外的文件14).
正如预期的那样,在本研究中也差异表达了与花青素生物合成相关的先前报告的miRNA(表1,无花果。6.).例如,IB-MIR156A-5P和IB-MIR858B的表达水平高于XS-3的XS-18。除了上面提到的miRNA之外,首先识别出9个家庭的其他24个miRNA,旨在涉及花青素生物合成(表1额外的文件14).例如,ib-miR172c、ib-miR172e-3p的表达。2那ib-miR172i, ib-miR172b, miR319, miR396 and miR2111 increased significantly in XZS-3. In particular, ib-miR164c was only expressed in XZS-3. However, family members of ib-miR159, ib-miR165, and ib-miR166 showed reduced or even no expression in XZS-3.
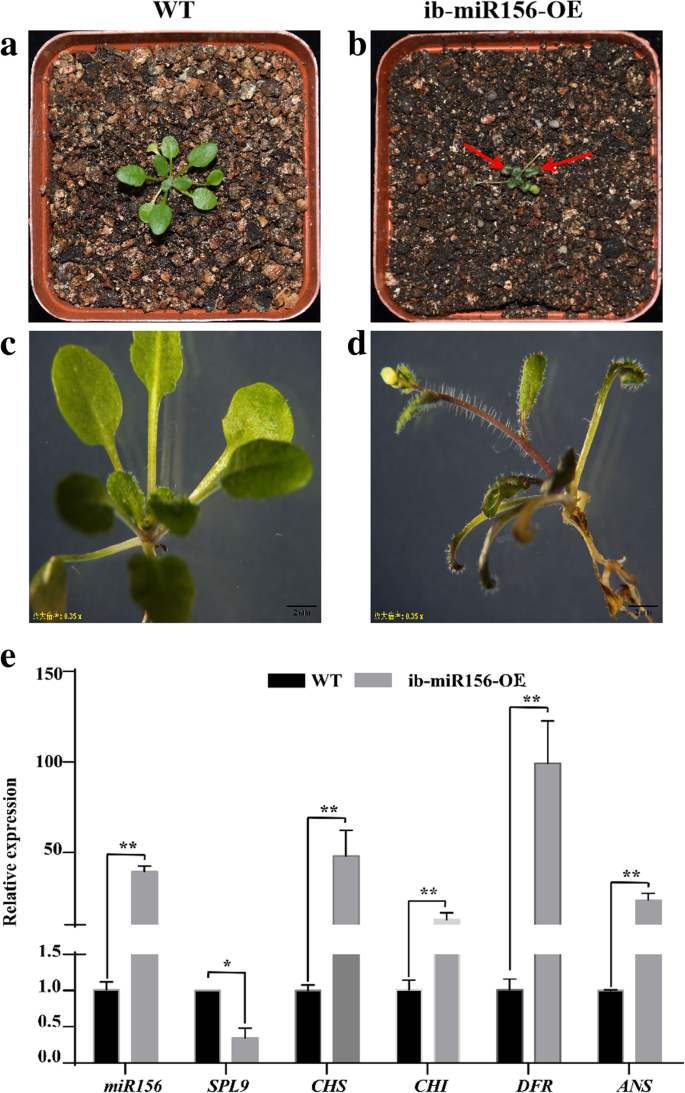
过表达ib-pri-MIR156诱导花青素积累拟南芥
miR156的直字基因拟南芥已被证明可以调节花青素的生物合成[14].在本研究中,ib-miR156的积累与IBSPL.在WFSP和PFSP中,ib-miR156的靶点提示ib-miR156可能也参与了花青素的生物合成。为了验证这一假设,我们生成了重组载体pc2300-pOT2-ib-pri-MIR156,并将其转化为拟南芥.有趣的是,在IB-PRI-MIR156的幼苗上观察到紫色表型在IB-PRI-MIR156过度表达中拟南芥在正常生长条件下(图10).通过qRT-PCR分析,miR156的丰度大大增加,而表达SPL.转基因中显着减少拟南芥(无花果。10e).此外,花青素生物合成相关的4个结构基因表达上调,包括CHS那气那DFR.和答在转基因中拟南芥(无花果。10e)。结果表明,IB-PRI-MIR156通过抑制表达式来积极调节花青素积累SPL..
讨论
通过高通量测序识别IB-MIRNA
MiRNA作为关键的转录后调节因子,参与植物的各种生物过程。最近,增加证据表明,植物miRNA也参与次生新陈代谢[14那38那42].花青素是重要的二级代谢产物之一。据报道,许多MIRNA在调节植物中调节花青素生物合成的重要作用,例如MIR156 [14], miR393 [43], miR828 [18那25那26], miR163 [44], miR165/166 [17]和miR858 [45].然而,在甘薯块根中鉴定与花青素生物合成相关的mirna尚未见报道。
在本研究中,通过sRNA测序数据分析,从WFSP和PFSP文库中共鉴定出191个已知ib- mirna。这一数字远高于此前有关甘薯的报道。在第一份关于甘薯miRNA的报告中,仅计算确定了8个潜在的miRNA候选[46].然后,在Ningzishu 1的索尔氏菌测序中鉴定了24名已知的miRNA [36].在这两项研究中,由于使用EST序列作为参考,鉴定出的miRNA数量相当有限。该研究已知的miRNAs数量与最近关于甘薯对低温胁迫反应的miRNAs的报告相当,在该报告中发现了190个已知的miRNAs [47].该研究是基于Kazusa SweetPotato Garden Database,该数据库是SweetPotato的野生二倍体祖先的基因组序列,Ipomoea Trifida.,虽然组装序列的总长度只有712 Mb,大小远低于估计的基因组序列[48].
谢等人检测到的大多数已知的miRNA家庭也存在于我们的数据集中,除MiR1508,MiR5253,MiR5298和MiR2911之外还存在[47].另一方面,我们鉴定出了此前在甘薯中未见报道的miR6478、miR6300、miR6173和miR5508。由于mirna的表达水平存在时间和空间特异性,这一结果并不令人惊讶[49].基于Pre-MiRNA的发夹结构(附加文件6.),共鉴定出33个新的候选mirna。新miRNAs的数量明显多于以EST序列为参考的研究,但远少于对甘薯低温胁迫反应的miRNAs的报道。虽然台中6号甘薯品种的基因组序列已于去年公布,但由于装配不完整和不准确,限制了其作为六倍体甘薯优良参考基因组的应用[50那51].为了消除遗传背景、物种和发育阶段造成的差异,本研究以sRNA深度测序得到的相同甘薯品种的转录组序列作为参考,预测新的mirna。降解组测序已经成功应用于许多植物物种的miRNA靶标鉴定[52那53].在该研究中,通过降级测序方法获得了115个IB-miRNA的总量为180个靶标。降级支持的目标数量远远低于使用计算PSRNATarget预测的目标。正如预期的那样,大多数靶基因是植物物种的转录因子,一些IB-miRNA有多个目标。如,IB-MIR156A-5P和IB-MIR5658都有10个目标,IB-MIR159A有6个目标。此外,属于同一家族的一些IB-MiRNA共享相同的成绩单。根据靶标的注释,发现这些靶基因参与宽的生物过程。
ib - mirna参与甘薯花青素生物合成
生长素是植物发育所必需的。高水平的生长素可以抑制MBW复合物的表达,从而调节花青素的生物合成[54那55].ARF,WRKY.和HD-zip.基因在生长素介导的信号转导中发挥重要作用,通过Aux/IAA-ARF途径调控苹果花青素的生物合成[56].在植物中,东盟地区论坛miR160和miR390调控的基因[24那57].WRKY主要参与生长素介导的信号转导,对类黄酮和花青素的生物合成有相当大的影响[58那59].HD-ZIP TF在植物茎尖分生组织和器官极性中起关键作用[23].本课题组前期研究发现,miR165/166的阻断导致HD-ZIP TFs上调,IAA含量增加,花青素含量增加拟南芥[17].在这项研究中,SRNA分析和降低测量测序证明了这一点怀疑和东盟地区论坛分别被ib-miR172e-3p和ib-miR160e-5p靶向;而HD-ZIPs被ib-miR164c和ib-miR166m靶向(表1).这些结果表明,IB-MIR172E-3P,IB-MIR160E-5P,IB-MIR164C和IB-MIR166M可以通过助长信号传导调节花青素生物合成。
CAD.是木质素生物合成的关键酶基因之一[60,发现ib-miR156、ib-miR157和ib-miR396对其有靶向作用(图。9.).MiR156和miR159已被报道为植物次生壁生物合成的潜在调控因子金合欢曼尼姆[61].在这项研究中,我们发现ib-miR159家族的4个成员有一个共同的新靶点I2'H(无花果。9.),可以促进Medicarpin的积累[62].MiR6300,仅在少数豆科植物中检测到Catharanthus roseus也叫和山茶花Sinensis.,被发现是目标UFGT[63那64].发现八个miRNA调节本研究中甘薯苷途径参与苯丙砜途径的酶基因。基于MiRNAS和酶基因在苯丙醇丙烷生物合成中的相互作用,我们提出了甘薯苯基丙醇调节的途径面板(图。9.).
MYB TFs已被确定为花青素生物合成途径的主要调控因子之一[65那66那67].例如,ATPAP1.(ATMYB75.)及其同系物已被证明能有效地诱导多种植物产生花青素[68那69那70].表达的MYB1在甘薯和苹果中诱导花青素积累[65那71].my已被证明由许多miRNA成员调节。例如,MiR828通过调节,参与了花青素生产,培养的毛细血管和棉纤维开发my在植物(16那18那72那73].发现miR858在通过切割的裂解中发挥积极作用。mybl2.在拟南芥[9.].MiR858也被确定为66的靶标my苹果会员[74那75].同样,我们最近的研究表明,Sl-miR858调节了两个SlMYB转录物和在番茄中的花青素积累作用[45].在这项研究中,my此外,ib-miR858、ib-miR319、ib-miR159和ib-miR156也能靶向这些基因。与WFSP相比,ib-miR858b表达明显上调,而其相应的靶基因表达明显上调IBMYB.PFSP下调(表1;无花果。6.).据报道,WD40蛋白在甘薯花青素生物合成MYB-bHLH复合物中是必不可少的和不可替代的[76].的成绩单ibWD40虽然它是psRNATarget预测的ib-miR396g-5p的一个潜在靶点,但在降解组测序中没有被ib-miR396g-5p切割(Additional file7.).Ib-miR396g-5p和ibWD40qRT-PCR显示,PFSP中的表达均高于WFSP。6.).一个可能的原因是,mirna可能对它们的目标施加翻译调控,而降解组测序无法检测到这一点。转录因子bHLH对R2R3-MYB伴侣的活性至关重要,可以直接结合的启动子DFR.和UFGT激活或抑制花青素的生物合成[77那78].然而,没有miRNA被鉴定为靶向Bhlh.在这项研究中。酶基因LcUFGT在花青素积累中发挥重要作用[79].在SweetPotato,我们发现IB-MiR6300直接针对性IBUFGT.,提示b-miR6300可能通过调控参与花青素的生物合成IBUFGT.(附加文件9.).但其调控机制尚需进一步验证。
SPLS是植物中广泛存在的转录因子,其在植物生长和发育,初级和次生新陈代谢以及其他生物过程中起重要作用。在拟南芥那SPL.被证明是由miR156调控的,并通过破坏拟南芥[14].在本研究中,ib-miR156a-5p被鉴定为可剪切IBSPL.基因(附加文件9.).SRNA测序和QRT-PCR的结果显示,MiR156A-5P显着上调,而其目标IBSPL.与WFSP相比,在PFSP中表达下调(图。6.).通过过度表达IB-MIR156拟南芥,紫色表型,花青素在转基因主茎中积累量高拟南芥被观察到。qRT-PCR结果显示,ib-miR156在大肠杆菌中过表达拟南芥强烈抑制丰富的丰富SPL.(无花果。10),这与Gou等人的结果一致[14].另外,在IB-MiR156过度表达转基因植物中检测到四种结构基因的上调转录物,在IB-MiR156中检测到(图。10),表明IB-MIR156可以通过调节苯基丙烷途径中的结构基因调节花青素生物合成。这些结果表明,IB-MIR156的调节功能类似于AT-MIR156。
推定的IB-miRNA-target模型涉及甘薯的花青素生物合成
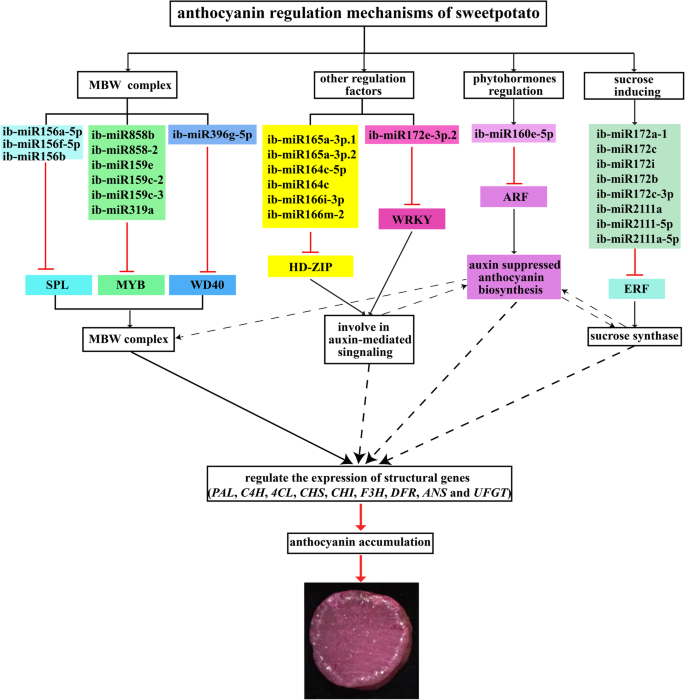
花青素的生物合成是由调控基因调控的,包括my那WDR.那Bhlh.那SPL.那东盟地区论坛和疯了.此外,蔗糖合酶,ABC转运蛋白和糖/肌醇转运蛋白也可能参与花青素生物合成。在这项研究中,根据靶的注释,我们发现26个差异表达的miRNA和36个相应的调节基因更有可能参与甘薯的花青素生物合成(表1额外的文件14).基于目前的数据和以往的报道,提出了一个可能与花青素生物合成相关的ib- mirna靶标模型。如图所示。11, ib-miR156、ib-miR159、ib-miR396和ib-miR858的miRNA家族可能被靶向SPL.那my和WDR.塑造一个有监管的MBW综合设施;ib-miR164、ib-miR165/166和ib-miR172e-3p的miRNA家族。2可能是有针对性的怀疑和HD-zip.涉及养肝信号传导;IB-MIR160,IB-MIR172和IB-MIR2111的MiRNA系列可能会规范东盟地区论坛和小块土地激活生长素介导的信号传导和蔗糖信号传导。所有这些生物学过程都会影响结构基因的表达,进而形成复杂的调控网络,调控花青素的生物合成。
结论
综上所述,这是对甘薯块根中mirna及其靶点调控花青素合成的系统鉴定、表达分析及潜在作用的首次报道。通过生物信息学分析和表达验证,26个差异表达mirna和36个相应靶点更有可能与花青素的生物合成有关。通过农杆菌属介导基因转化,ib-miR156过表达转基因拟南芥呈紫色表型。在转基因品系中,ib-miR156的表达量上调,而其靶标上调SPL.显示下调表达。四种特异性三种花青素酶基因,CHS那气那DFR.和答,在转基因中表达显着高得多拟南芥表明ib-miR156可以通过调控相关结构基因正向介导花青素合成。在此基础上,提出了一个与甘薯花色苷生物合成相关的ib- mirna靶标模型。我们的发现为研究花青素特异性mirna及其作用靶点提供了全面的信息,也为研究甘薯花青素生物合成mirna的机制提供了一个起点。
材料和方法
植物材料
地瓜(番薯甘薯L.)Whing的白皮Xushu-18(XS-18)和紫色肉体Xuzishu-3(XZS-3)的栽培品种用于SRNA和降低组测序。该品种由中国江苏省甘普洛特研究所提供,并在正常情况下在山西农业大学(Taigu)的实验领域成长。在90天阶段的根部形成后,选择具有相似尺寸和形状的白色和紫色枝条。在轻轻洗涤后,通过液氮立即冷冻并储存在-80°C冷冻机。我们将三个茎根样本混合在一起,作为一种生物学重复,以消除个体遗传方差的影响。总共为每个样品收集三种独立的生物重复。
小RNA库的构建和测序
根据生产说明书,使用Trizol试剂(Invitrogen公司,美国)从甘薯根中提取总rna。从每个基因型中分离出3个生物重复的总RNA,并用纳米光度计(IMPLEN, CA, USA)测定RNA的质量和数量。然后,根据制造商的建议,使用NEBNext®Multiplex Small RNA library Prep Set for Illumina®(NEB, USA.)构建了两个测序库(XS-18和XZS-3),并在中国北京诺金生物信息学研究所Illumina Hiseq 2000平台上进行测序。
使用深度测序的甘薯MiRNA的鉴定及表达分析
在先前的报告方法后进行甘薯结节根部的已知和新麦芽糖的方法[39].首先,从原始数据中去除包含ploy- n,带有5 '适配器污染物的更大的读,没有3 '适配器或插入标签,包含ploy A或T或G或C和低质量读的读,从而获得干净的读。小于18个核苷酸(nt)和大于30个核苷酸的序列也被去除。然后由Bowtie将sRNA标签映射到参考序列[80不允许错配。利用相同的甘薯品种(XS-18和XZS-3)进行转录组测序,并进行sRNA深度测序(未发表)。将转录组数据库中的组装序列作为预测miRNA前体的参考序列。对映射的序列进行blast搜索,使用Repeat Masker (http://www.repeatmasker.org/)和RFAM数据库(http://rfam.xfam.org/)除去源自蛋白质编码基因,重复序列,RRNA,TRNA,SNRNA和SnRNA和SnRNA的标签,或来自指定物种本身的这些类型的数据。通常,我们遵循以下优先级规则:已知miRNA> rRNA> TRNA> SnRNA> SnRNA> SnRNA>重复>基因> NAT-siRNA>基因>新型miRNA> TA-siRNA。剩余的读数与沉积在miRBase 22.1数据库中的miRNA序列对齐(http://www.mirbase.org/)[81].没有错配的匹配序列被认为是已知的miRNA。然后通过探索二次结构,Dicer切割位点和最小自由能量来预定读取的读取软件Mirevo和Mirdeep2以预测新的MiRNA候选者[82].使用RNAFOLD软件还预先预测了前miRNA的茎环发夹结构。通过以下归一化公式的TPM(每百万百万)估计miRNA表达水平:标准化表达式=映射读数/总读数* 1000000 [83].
为了揭示与XS-18和XZS-3之间的花青素生物合成相关的差异表达的miRNA,使用DEGSEQ R包分别分析MiRNA表达。P.-value使用qvalue调整[84].Qvalue < 0.01,默认设置显著差异表达阈值|log2 (fold change)| > 1。
降级图书馆构建,测序和数据分析
为鉴定甘薯mirna的潜在靶mrna,以XS-18和XZS-3混合根构建degramdome文库。首先,利用Oligotex试剂盒(Qiagen, Germany),提取总RNA 200 μg,将poly (A) RNA连接到一个3 '端具有EcoP15 I识别位点的5 '适配器上。连接后,生成第一链cDNA,用PCR进行扩增。PCR产物分别用PAGE-gel和EcoP15 I酶切。然后将EcoP15 I剪切片段连接到一个3 '双链DNA适配器上,然后进行PAGE-gel纯化,得到连接的产物。PCR扩增后,第三次使用PAGE-gel纯化最终产物。最后将纯化的cDNA文库用于Illumina HiSeq2000测序系统(LC-BIO Sciences, China)的深度测序。
测序后,从原始reads中过滤出适配器序列、低质量reads和含n的片段。如前所述,剩余的x -18和XZS-3甘薯转录组数据库中的序列被用于识别CleaveLand4管道可能切割的靶标[85].sRNA介导的降解体事件根据其在靶转录本上的cleaved tag reads的丰度分为0、1、2、3和4 [86].类别0:丰度等于目标转录本丰度的最大值,裂解标签读值只有一个最大值。第1类:丰度等于目标转录本丰度的最大值,而cleaved tag reads有一个以上的最大值。第2类表示cleaved tag的丰度小于最大值,但高于转录本丰度的平均值。第3类:裂解标签丰度小于或等于转录本丰度的平均值。第4类表示位置的深度等于1。根据miRNA在转录本上的分布和丰度,构建t图分析miRNA靶点和RNA降解模式。通过PAREsnip软件分析mirna的潜在靶点,p值< 0.05 [87].
去丰富和Kegg途径分析
利用在线GO分析工具,根据miRNAs靶点的生物学作用、分子功能和细胞组分注释GO词条(http://www.geneontology.org/page/go- enrichment-analysis.)[88].KEGG途径候选靶基因的统计富集是由KOBAS软件设置的[89那90.].
IB-PRI-MIR156的施工过度表达载体和遗传转化拟南芥
从PFSP中分离IB-PRI-MIR156,并插入35S2启动子和35s终止子之间的POT2载体中。然后,将POT2转化为修饰的PCAMBIA2300二元载体,以构建过度表达的载体PC2300-POT2-IB-PRI-MIR156。向量被引入农杆菌属GV3101,然后是农杆菌属介导的转化获得转基因拟南芥[91.].用于质粒结构的所有引物都列于附加文件15.
qRT-PCR分析ib- mirna、相应靶标及花青素特异酶基因的表达
通过QRT-PCR评估miRNA的表达及其相应的靶标。通过使用Trizol(Invitrogen,USA)从XS-18和XZS-3中提取总RNA。对于miRNA表达分析,根据具有一些修改的报告的方法设计了特定的杆环Rt,正向和通用反向引物[92.].1 μg经DNase处理的总RNA按照PrimeScript™1st Strand cDNA synthesis kit (TaKaRa,中国)的程序进行cDNA合成。所有qRT-PCR分析均使用SYBR进行预混料Taq交货™(Tli RNaseH Plus) (TaKaRa,中国),并在Bio-Rad CFX96 Real-Time PCR系统(Bio-Rad,美国)上进行。地瓜,IB5S.和ibActin基因作为内参,分别在每个反应中对miRNA和靶基因进行精确归一化[93.].对转基因拟南芥过度表达miR156,atU6和attint.被使用。用2——ΔΔCt方法(94.].来自三种生物重复的每个样品一式三份进行。结果呈现为平均值±SD。Dunnett两尾T检验的方法用于进行RT-PCR结果的统计分析,统计学显着差异显示在P.≤0.05(带*号)和P.≤0.01(标**)。所有引物的序列列于附加文件中15.
缩写
- 答:
-
花青素合成酶
- 东盟地区论坛:
-
助线响应因子
- CAD:
-
肉桂醇脱氢酶
- Chi:
-
查耳酮异构酶
- CHS:
-
查耳酮合酶
- DFR:
-
Dihydroflavonol 4-reductase
- 小块土地:
-
乙烯响应转录因子
- 美东时间:
-
表达序列标签
- F3'5'H:
-
类黄酮3 ',5 ' -羟化酶
- F3'H:
-
黄烷酮3-羟化酶
- F3H:
-
黄烷酮3-羟化酶
- 去:
-
基因本体论
- HD-ZIP:
-
Homeodomain-leucine拉链蛋白质
- I2'H:
-
异黄酮2》羟化酶
- 国际宇航科学院:
-
Indole-3-acetic酸
- Kegg:
-
京都基因和基因组百科全书
- 朋友:
-
苯丙氨酸氨裂解酶
- SPL:
-
Squamosa启动子结合蛋白样
- SRNA:
-
小核糖核酸
- TF:
-
转录因子
- UFGT:
-
UDP-甘糖黄酮糖基转移酶
- 世界发展报告》:
-
WD repeat-containing蛋白质
参考
- 1.
Bovell-Benjamin AC。甘薯:对其过去,现在和未来的人类营养作用的审查。adv food nutr res。2007; 52(06):1-59。
- 2.
苏田等。紫肉红薯花色苷的生理功能及其在食品中的利用。日本农业研究2003;37:167-73。
- 3.
Matsui T,EBUCHI S,Kobayashi M,Fukui K,Sugita K,Terahara N,Matsumoto K.通过α-葡萄糖苷酶抑制作用来实现衍生自Imomoea Batatas品种Ayamurasaki的二酰胺化的花青素的抗高血糖作用。JAgric Food Chem。2002; 50(25):7244-8。
- 4.
山川O.新加工在日本新加工厂的发展。九州国家农业实验站。日本。普满农业部门。1998; 75(2):284-7。
- 5.
安第斯紫玉米和红肉甘薯花青素水提取物的稳定性与合成和天然色素的比较。食品化学。2004;86(1):69 - 77。
- 6.
Johnson M,Pace Rd。甘薯叶:促进健康和预防疾病的性质和协同相互作用。Nutr Rev.2010; 68(10):604-15。
- 7.
关键词:紫甘薯,突变体,转录组,De novo测序番薯甘薯l .)。基因。2015;575(3):641 - 9。
- 8.
Park Ki,Ishikawa N,Morita Y,Choi JD,Hoshino A,Iida S. aBhlh.牵牛花的调节基因,番薯紫竹,控制种子中的花,原花青素和植物细胞素色素沉着的花青素生物合成,以及种子培养体形成。工厂J. 2007; 49(4):641-54。
- 9。
王玉玲,王玉强,宋智,张华mybl2.通过microRNA858a和HY5的激活导致花青素生物合成途径拟南芥.莫尔植物。2016; 9(10):1395-405。
- 10.
Castellarin SD,Pfeiffer AP,Degan M,Peterlunger E,Gaspero GDI。季节性水缺损下葡萄树成熟果实中花青素生物合成的转录调控。植物细胞环境。2007; 30(11):1381-99。
- 11.
徐W,Dubos C,Lepiniec L. myB-BHLH-WDR复合物对类黄酮生物合成的转录控制。趋势植物SCI。2015; 20(3):176-85。
- 12.
迈尔A,Schrader A,Kokkelink L,Falke C,焊机B,Inisto E等人。光和E3泛素连接酶COP1 / SPA控制MYB转录因子PAP1和PAP2参与的蛋白质稳定性,参与了花青素积累拟南芥.植物j . 2013; 74(4): 638 - 51。
- 13.
齐涛涛,宋少森,任QC,吴德伟,黄辉,陈勇,等。茉莉酸- zim结构域蛋白与WD-repeat/bHLH/MYB复合物相互作用,调控茉莉酸介导的花青素积累和毛状体起始拟南芥.植物细胞。2011;23(5):1795 - 814。
- 14.
苟杰,Felippes FF,刘CJ,Weigel D,Wang JW。花青素生物合成的负调节拟南芥通过MIR156靶向的SPL转录因子。植物细胞。2011; 23(4):1512-22。
- 15.
Zhou H, lin Wang K, Wang H, Gu C, Dare AP, Espley RV, He H, Allan AC, Han Y. Molecular genetics: NAC转录因子激活花青素生物合成。植物j . 2015; 82(1): 105 - 21所示。
- 16.
杨福克,蔡继,杨y,刘泽。MicroRNA828的过度表达减少了花青素的积累拟南芥.植物细胞组织器官邪教。2013; 115(2):159-67。
- 17.
贾XY,丁n,风扇wx,yan j,gu yy,唐xq,li rz,唐gl。MiR165 / 166在植物开发中的功能可塑性,促进小串联靶模模仿。植物SCI。2015; 233:11-21。
- 18.
贾晓燕,刘洪,沈杰,李飞,丁宁,孙勇,高春艳,李仁忠。microRNA828对番茄缺磷条件下花青素合成的负调控。中国农业科学,2015;48(15):2911-24。
- 19.
植物的RNA沉默。自然。2004;431(7006):356 - 63。
- 20。
吴林,周洪,张强,张军,倪飞,刘超,等。由microRNA途径介导的DNA甲基化。38摩尔细胞。2010;(3):465 - 75。
- 21。
丁德华,张丽丽,王海涛,刘志华,张志华,郑永强。玉米根系mirna表达差异对盐胁迫响应的影响。安机器人。2009;103(1):第29 ~。
- 22.
Sunkar R,Chinnusamy v,朱家,朱杰克。小RNA作为植物非生物应激反应和营养剥夺的大玩家。趋势植物SCI。2007; 12(7):301-9。
- 23.
Williams L,Grigg Sp,谢米,克莱斯滕斯,弗莱彻·杰克。规范拟南芥microRNA miR166g及其thd - zip靶基因对茎尖分生组织和侧器官形成的影响。发展。2005;132(16):3657 - 68。
- 24.
夏锐,徐建军。miR390-的出现、演变和多样化TAS3-东盟地区论坛陆地植物的途径。植物细胞。2016; 29(6):1232-47。
- 25.
罗QJ,米特A,贾F,摇滚CD。涉及PAP1和TAS4的自动调节反馈回路响应于拟南芥中的糖。植物mol biol。2012; 80(1):117-29。
- 26.
贾晓燕,于志强,梁建平,唐国良,金丽华,张丽,何丽华,李瑞珍。克隆拟南芥at-pri-miR828基因及其在番茄中的遗传转化。园艺学报。2013;40(12):2419-28。
- 27.
李涛,马林,耿玉坤,郝春英,陈晓辉,张学勇。小RNA和降解组测序揭示了mirna的复杂作用及其在发育小麦粒中的靶点。《公共科学图书馆•综合》。2015;10 (10):e0139658。
- 28.
kwon y,哦,je,no,hong sw,bhoo sh,lee h.乙烯信号通路对蔗糖诱导的花青素积累产生负面影响拟南芥.acta botanica yunnanica(云南植物研究)2011;
- 29.
宋庆新,刘艳芳,胡晓燕,张文坤,马斌,陈世英,张建生。大豆种子发育中mirna及其靶基因的深度测序鉴定植物学报。2011;11(1):5-20。
- 30.
李云飞,郑勇,张磊,张磊。水稻microRNA靶点的转录组范围鉴定。植物j . 2010; 62(5): 742 - 59。
- 31.
吴九,郑S,冯·叶肝,麦克纳斯与其野生型麦克兰类及其野生型对比分析及其野生型。前植物SCI。2016; 7:1416。
- 32.
陈建军,郑勇,秦磊,王勇,陈磊,何勇,等。通过高通量测序和降解组分析在男性和女性中鉴定mirna及其靶点芦笋officinalis.植物学报。2016;16(1):80-99。
- 33.
孙y,秋y,段m,王j,张x,王h等。鉴定南萝卜鲜明植物生物合成相关微大稻草(Raphanus Sativus通过高通量测序。中国生物医学工程学报,2016;
- 34.
刘虎,秦c,陈z,zuo t,杨x,周h等。基于小RNA和降解测序鉴定MIRNA及其在玉米耳中发展玉米耳的靶基因。BMC基因组学。2014; 15(1):25。
- 35.
jeyaraj a,张x,hou y,shangguan m,gajjeraman p,li y,wei c.全基因组识别一个芽和两个嫩茶植物的嫩叶和两个嫩叶(山茶花Sinensis.),小RNA测序,微阵列杂交和基因组调查支架序列。植物学报。2017;17(1):212-28。
- 36.
BIAN X,E Z,MA P,JIA Z,GUO X,Xie Y.通过SOLEX的测序鉴定MIRKAT MAYNAS。Russ J植物理体。2016; 63(2):283-92。
- 37.
孙瑞荣,郭涛涛,Cobb J,王清良,张波林。小分子rna在甘薯花和贮藏根发育中的作用。植物学报2015;33(6):1731-9。
- 38.
Jonesrhoades MW,Bartel DP,Bartel B. Micrornas及其在植物中的监管角色。Annu Rev植物Biol。2006; 57(1):19-53。
- 39.
Meyers Bc,Axtell MJ,Bonnie B,Bartel DP,David B,Bowman JL等。植物微稻草注释标准。植物细胞。2008; 20(12):3186-90。
- 40。
Thakur V,Wanchana S,Xu M,Bruskiewich R,Quick WP,Mosig A等人。植物微稻田预测统计特征的特征。BMC基因组学。2011; 12(1):108。
- 41。
卢斌,程光新,张震,孙金堂,阿里梅,贾庆林,等。新转录因子CaMYC调控花青素生物合成(Capsicum Annuum.l .)。植物生长规律。2018:1-12。https://doi.org/10.1007/s00344-018-9871-2.
- 42。
刘锐,赖斌,胡斌,秦勇,胡刚,赵建军。与花青素积累相关的microrna及其靶基因的鉴定荔枝中华人员通过高通量测序和降低分析。前植物SCI。2017; 7:2059。
- 43。
王志强,王志强,王志强,等。microRNA miR393将次生代谢产物的生物合成从卡马毒素转向硫代葡萄糖苷。植物j . 2011; 67(2): 218 - 31所示。
- 44。
吴德伟,张超,王志强,等。独联体- - -trans- miR163和靶基因的调控在两个方面产生了次级代谢物的自然变异拟南芥物种及其全多利多片。植物细胞。2011; 23(5):1729-40。
- 45。
贾晓燕,沈静,刘辉,李飞,丁宁,高春艳,等。小串联目标模拟介导的microRNA858阻断诱导番茄花青素积累。足底。2015;242(1):283 - 93。
- 46.
王志强,王志强,王志强,等。甘薯中保守mirna及其靶基因的硅质鉴定与鉴定(番薯甘薯表达序列标签(est)。植物学报。2013;8(12):e26543。
- 47.
谢Z,王A,李h,yu j,江j,唐z等。高通量深度测序揭示了MicroRNA在甘薯储存过程中的重要作用在冷却温度下。SCI批准。2017; 7(1):16578。
- 48.
Hirakawa H, Okada Y, Tabuchi H, Shirasawa K, Watanabe A, Tsuruoka H, et al.;野生甘薯基因组序列调查,Ipomoea Trifida.(h . b . k .)g .堂。DNA研究》2015;22(2):171 - 9。
- 49.
张B. microRNA。一种改善植物耐受性应激的新靶点。J Exp Bot。2015; 66(7):1749-61。
- 50.
杨建军,莫因扎德H H .,胡晓东,肖鹏,等。单倍型分解甘薯基因组可追溯其六倍化历史。Nat植物。2017;3(9):696 - 703。
- 51.
吴胜,刘国强,曹强,Hamilton JP,孙辉,周超,Eserman L, Gemenet DC, Olukolu BA, Wang H,等。栽培甘薯的两个二倍体野生近缘种的基因组序列揭示了遗传改良的目标。Nat Commun。2018;9(1):4580。
- 52.
Ku Ys,Wong JW,Mui Z,Liu X,Hui JH,Chan TF,Lam HM。植物对非生物胁迫的小RNA。监管作用和学习方法。int j mol sci。2015; 16(10):24532-54。
- 53.
方yn,郑伯,王l,杨,吴思,徐q,郭ww。高通量测序和降级分析揭示了MiRNA的改变表达及其在雄性无菌纤维糖Pummelo中的靶标的表达(柑橘祖母).BMC基因组学。2016;17(1):591。
- 54.
刘铮,施明忠,谢大勇。植物花青素生物合成的调控拟南芥红pap1-D细胞由生长素代谢程序。足底。2014;239(4):765 - 81。
- 55.
吉Xh,王yt,张r,wu sj,mm,li m等。蟾蜍素,细胞素和氮在红肉苹果愈伤组织培养中的血清素生物合成效果(海棠).植物细胞。2015;120(1):325 - 37。
- 56.
王永春,王宁,徐海峰,姜树华,方慧琴,苏美美,等。生长素通过aux/IAA-ARF信号通路调控苹果花青素的生物合成。Hortic杂志2018;5:59。
- 57.
王吉,王立,毛耶,蔡维,薛鹤,陈XY。通过MicroRNA靶向养蛋白响应因子控制根帽形成拟南芥.植物细胞。2005;17(8):2204 - 16。
- 58.
江益,梁梁,杨某,雅典族人Wrky57用作茉莉酸 - 诱导的叶片衰老中茉莉酸和生长素介导的信号传导的收敛节点。植物细胞。2014; 26(1):230-45。
- 59.
王,刘某,张t,姜S,徐H,王Y,张Z,王c,陈x。红肉苹果的转录组分析揭示了Mdwrky11在黄酮类化合物和花青素生物合成中的新作用。JAgric Food Chem。2018; 66(27):7076-86。
- 60.
Boerjan W,Ralph J,Baucher M. Lignin生物合成。Annu Rev植物Biol。2003; 54(1):519-46。
- 61.
关键词:微rna,生物合成,微rna金合欢曼尼姆.7。2012;(11):e49662。
- 62。
细胞色素p450在类黄酮代谢中的作用。Phytochem启2006;5(2 - 3):271 - 82。
- 63。
Lelandais-Briere C,Naya L,Sallet E,Calenge F,Frugier F,Hartmann C,Gouzy J,Crespi M. Genome-Wide Medicago Truncatula Small RNA分析显示了新的MicroRNA和同种型在根和结节中差异调节的小植物和同种型。植物细胞。2009; 21(9):2780-96。
- 64。
Shen Em,Singh Sk,Ghosh Js,Patra B,Paul P,Ling Y,Pattanaik S. miraneomeCatharanthus roseus也叫.微rna在萜类吲哚生物碱生物合成中的鉴定、表达分析及潜在作用。Sci众议员2017;7(1):43027。
- 65。
Mano H,Ogasawara F,Sato K,Higo H,Minobe Y,Hironori M,Fumiaki O,Kazuhito S,Hiromi H,Yuzo M.紫红色甜薯茎秆枯萎病中的调节基因分离。植物理性。2007; 143(3):1252-68。
- 66。
赖B,李XJ,胡b,秦耶,黄奇,王英,胡锦涛。lcmyb1.是基因型,组织,发育阶段和ABA和荔枝中的光刺激之间的差异化花青素积累的关键决定因素。Plos一个。2014; 9(1):E86293。
- 67。
沈欣,赵凯,刘丽,张凯,袁华,廖晓东,等。PacMYBA在aba调控红樱桃花色苷生物合成中的作用。香港邓(李属鸟结核l .)。植物细胞生理。2014; 55(5):862。
- 68。
Zuluaga DL,Gonzali S,Loreti E,Pucciariello C,Degl'Inocenti E,Guidi L等。拟南芥MYB75 / PAP1转录因子诱导转基因番茄植物中的花青素产生。Funct植物Biol。2008; 35(7):606-18。
- 69。
罗文DD,Cao M,Lin-Wang K,Cooney JM,Jensen DJ,Austin Pt等。红色35s中的叶子颜色的环境调节.PAP1拟南芥.新植醇。2010;182(1):102 - 15所示。
- 70.
Cutanda-Perez MC,useorges A,Gomez C,Vialet S,Terrier N,Romieu C,Torregrosa L.VlmyBA1在葡萄中的异位表达激活了涉及花青素合成和运输的狭窄基因。植物mol biol。2009; 69(6):633。
- 71.
周LJ,李伊,张立雷,张克,谢XB,赵开,郝yj。sumo e3连接酶MDSIZ1.通过磺酰化促进花青素积累mdmyb1.在苹果低温条件下。植物学报。2017;40(10):2068-80。
- 72.
谢勇,孙勇,李丹。MicroRNA828负调控蔗糖诱导的花青素生物合成拟南芥.植物生理学杂志。2013;49(2):188-94。
- 73.
关X,庞米,不,谢X,叶W,碑架DM,陈ZJ。mir828和mir858调节同源性MYB2基因功能拟南芥毛状体和棉纤维的开发。Nat Commun。2014;5(1):3050 - 63。
- 74.
Moxon S,Jing R,Szittya G,Schwach F,Rusholme Pilcher RL,Moulton v,Dalmay T.番茄短RNA的深度测序识别涉及果实成熟的MicroRNA靶向基因。Genome Res。2008; 18(10):1602-9。
- 75.
夏锐,朱华,安永强,Beers EP,刘志强。新型调控网络的苹果miRNAs和tasiRNAs。基因组医学杂志。2012;13(6):47 - 64。
- 76.
Dong W,NIU LL,GU J,GU JT,GAO F. D40重复基因的分离调节紫肉甘薯贮藏根贮藏根储存根。acta physiol植物。2014; 36(5):1123-32。
- 77.
Feller A, Machemer K, Braun EL, Grotewold E.进化与比较分析my和Bhlh.植物转录因子。植物j . 2011; 66(1): 94 - 116。
- 78.
谢晓波,李松,张瑞芬,赵军,陈永春,赵强。bHLH转录因子MdbHLH3促进低温胁迫下苹果花色苷积累和果实着色。植物学报。2012;35(11):1884-97。
- 79.
Wei YZ,Hu Fc,Hu GB,李XJ,黄XM,王HC等。花青素生物合成基因在果皮中与花青素积累相关的差异表达荔枝比如说.Plos一个。2011; 6(4):E19455。
- 80。
Langmead B,Salzberg SL。与Bowtie 2. NAT方法的快速喷射读取对齐。2012; 9(4):357-9。
- 81。
Kozomara A, Griffiths-Jones S. miRBase:利用深度测序数据注释高可信度microrna。核酸学报2014;42(1):68-73。
- 82.
张BH,PAN XP,COX SB,COBB GP,Anderson TA。证据表明miRNA与其他RNA不同。细胞mol寿命。2006; 63(2):246-54。
- 83.
周磊,陈建军,李志强,胡昕,黄勇,等。microRNAs和mRNAs的整合分析。位于Xq27.3的microrna与透明细胞肾细胞癌相关。《公共科学图书馆•综合》。2010;5 (12):e15224。
- 84.
层JD。正错误发现率为贝叶斯解释和q价值.安Stat。2003;31:2013-35。
- 85.
addo-quaye c,miller w,axtell mj。克利夫兰。用于使用降级数据寻找切割的小型RNA目标的管道。生物信息学。2009; 25(1):130-1。
- 86。
Charles AQ, Eshoo TW, Bartel DP, Axtell MJ。内源性siRNA和miRNA靶标通过测序确定拟南芥降级。Curr Biol。2008; 18(10):758-62。
- 87。
Leighton F,Simon M,Woolfenden HC,Szittya G,Dalmay MV。Paresnip。通过降低测量测序证明了一种用于快速基因组/靶相互作用的快速基因组发现的工具。核酸RES。2012; 40(13):E103。
- 88。
Young Md,Wakey MJ,Smyth GK,Oshlack A. RNA-SEQ的Geneontology分析:选择偏差。基因组Biol。2010; 11:R14。
- 89。
毛XZ,Cai T,Olyarchuk JG,Wei LP。使用Kegg orthology(Ko)作为受控词汇的自动基因组注释和路径识别。生物信息学。2005; 21(19):3787-93。
- 90。
Kanehisa M, Araki M, Goto S, Hattori M, Hirakawa M, Itoh M,等。KEGG将基因组与生命和环境连接起来。核酸学报2008;36:480-4。
- 91。
Smith CJS, Watson CF, Bird CR, Ray J, Schuch W, Grierson D.在转基因植物中表达截短的番茄多半乳糖醛酸酶基因抑制内源基因的表达。Mol Gen Genet, 1990; 24:47 - 81。
- 92。
Chen CF,Ridzon Da,扫帚AJ,周Zh,Lee Dh,Nguyen JT等。茎环RT-PCR的实时定量微小RNA。核酸RES。2005; 33(20):E179。
- 93。
Lin JS, Lin CC, Lin HH, Chen YC, jst . MicroR828调控木质素和H2O.2积累在甘薯上伤人。新植醇。2012;196(2):427 - 40。
- 94.
Livak KJ, Schmittgen TD。利用实时荧光定量PCR技术分析相关基因表达数据(-Delta delta c(t))方法。方法。2001;25(4):402 - 8。
致谢
作者感谢曹清河(中国江苏省甘薯研究所)提供的实验材料(徐书18号和徐子书3号)。
资金
基因克隆、载体构建及遗传转化拟南芥国家重点研发计划项目(2018YFD1000705, 2018YFD1000700)和山西省重点研发计划项目(201803D221008-6)资助miRNAs及基因表达检测项目由山西农业大学科技创新基金项目(2018yz001)和山西省国家自然科学基金项目(201801D121238)资助。工厂处理和样品采集由山西省煤基科技重点项目(FT-2014-01)资助。sRNA测序和降解组测序数据分别由国家自然科学基金资助项目(31101555,31571611)和国家山西农业流域建设科研专项(SXNGJSKYZX201701-03)资助。
数据和材料的可用性
在当前研究期间生成和/或分析的数据集包含在额外的表中。
作者信息
隶属关系
贡献
XYJ,RZL,LHH,QHC和WBW构思了原始的研究计划;LHH,RMT和XWS进行了实验并分析了数据。QHC提供了一些实验材料。LHH,YS,TW,XYL和HMZ收集了这些材料。LHH,RMT,XWS和XYJ起草了稿件。RZL批判性地修改了手稿。所有作者均阅读并批准了手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
额外的文件1:
甘薯文库SRNA序列及转录组统计学的统计。(DOC 32 kb)
额外的文件2:
独特sRNA序列在甘薯不同分类中的分布。(DOC 32 kb)
额外的文件3:
在甘薯中鉴定的已知miRNAs的详细信息。(XLS 69 kb)
额外的文件4:
已知miRNA的正常读数。(XLS 37 KB)
额外的文件5:
在甘薯中发现的新miRNAs的详细信息。(XLS 36 kb)
额外的文件6:
新型miRNA前体的二次结构。(TIF 269 KB)
额外的文件7:
通过psRNATarget预测甘薯mirna的靶基因。(XLS 935 kb)
额外的文件8:
甘薯degedrom测序reads的统计。(医生22 kb)
额外的文件9:
利用降解组测序鉴定甘薯mirna的靶基因。(XLS 126 kb)
额外的文件10:
xzs -18与XZS-3之间差异表达的mirna列表(XLS 33 kb)
额外的文件11:
比较qRT-PCR和深度测序检测的mirna表达水平。(TIF 901 kb)
额外的文件12:
GO分析甘薯降解组测序鉴定的差异miRNAs靶点。(XLS 63 kb)
额外的文件13:
通过降解组测序鉴定甘薯差异mirna靶点的KEGG分析。(XLS 24 kb)
额外的文件14:
甘薯中可能与花青素合成相关的mirna及其靶基因。(XLS 24 kb)
额外的文件15:
本研究使用的引物列表。(XLS 26 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
何磊,唐瑞,石旭东。et al。通过小RNA和降解组测序发现甘薯块根中花青素生物合成相关的microrna及其靶基因。BMC植物杂志19,232(2019)。https://doi.org/10.1186/s12870-019-1790-2
收到了:
公认:
发表:
关键字
- 花青素生物合成
- 降低分析
- MiR156
- 小核糖核酸测序
- 地瓜