- 研究文章
- 开放获取
- 发表:
拟南芥蒂利亚纳细胞器模拟T7噬菌体DNA replisome与闪烁蛋白和DNA聚合酶Pol1A和Pol1B之间的特定相互作用
BMC植物生物学体积19文章编号:241(2019)
摘要
背景
植物叶绿体和线粒体利用核编码蛋白来复制其DNA。这些蛋白质在细胞器环境中被用作复制,并且与参与核基因组的复制中的那些不同。这些细胞器局部蛋白质具有细菌和噬菌体基因中的祖传根,支持其起源的内肌病理论。我们检查了三种蛋白质之间的相互作用拟南芥蒂利亚纳:一种类似于噬菌体T7 GP4蛋白和动物线粒体闪烁的DNA螺旋酶 - 原序,以及两个DNA聚合酶,POL1a和POL1b。我们利用三管齐下的方法来分析相互作用,包括酵母 - 二杂化分析,直接偶联分析(DCA)和耐热化。
结果
酵母双杂交分析显示,Twinkle的120-295残基仍然可以与Pol1A或Pol1B相互作用。该区域是蛋白质引物酶结构域的一部分,与位于其中的锌指和RNA聚合酶亚结构域略有重叠。此外,我们观察到拟南芥与Pol1A相比,Twinkle与Pol1A的交互要强烈得多。热电泳还证实了Twinkle的引物结构域比蛋白质的任何其他区域具有更高的结合亲和力。直接偶联分析确定了Twinkle中的特定残基和DNA聚合酶,这对两个蛋白之间的正相互作用至关重要。
结论
Twinkle与Pol1A或Pol1B的相互作用模拟了T7噬菌体和哺乳动物线粒体中存在的最小DNA replisomes。然而,虽然T7和哺乳动物绝对需要其同源的Twinkle DNA解旋酶引物酶,拟南芥闪烁突变体似乎不受此损失的影响。这意味着拟南芥线粒体模仿来自T7和哺乳动物线粒体的最小replisomes,有一个额外水平的冗余特定于闪烁功能的丧失。
背景
线粒体和叶绿体具有各自独特的基因组。内共生理论为真核细胞中线粒体和叶绿体的存在提供了主要解释。在这个理论中,现代线粒体和叶绿体是古老的自由生活的α-变形菌和蓝藻细菌与宿主细胞共生的结果[1].随着时间的推移,内共生的α-变形菌和蓝藻细菌失去了越来越多的DNA编码能力,直到它们失去了自由生活的能力。然而,时间并没有从这些细胞器中移除所有的DNA,留下了我们今天熟悉的线粒体和叶绿体基因组。这种DNA不是人造的,它与基因一起完全发挥功能,这些基因被复制、转录和翻译,以产生细胞器功能所需的基本蛋白质[2那3.].维持这种DNA的机制类似于噬菌体系统,比真核细胞核DNA复制和细菌染色体复制的机制简单得多[4.那5.].
许多植物细胞器DNA复制蛋白已经在不同的物种中被鉴定和描述[4.那6.].在本报告中,我们专注于拟南芥细胞器DNA复制蛋白闪烁和DNA聚合酶1a和1b之间的相互作用。我们提出在植物叶绿体和线粒体中的最小功能性DNA覆盖物由DNA预级螺旋酶蛋白闪烁,与DNA聚合酶1a或1b一起组成。我们还建议SSB1参与这一最小的额。该系统已被描述为哺乳动物中最小的线粒体DNA克利物[7.,并模仿T7噬菌体的复制机制[8.].
拟南芥闪烁蛋白得名于人类同源物[9.,类似于噬菌体T7 gp4 DNA引物酶/解旋酶蛋白。在人类中,由于引物酶结构域的氨基酸序列差异,这种蛋白质缺乏DNA引物酶活性[10].在植物中,这种蛋白有两个DNA螺旋和引物酶的活动[11那12].T7 GP4蛋白既用作DNA解旋酶和引发酶,而且是中心其基因组的复制机制[8.].从结构上看,拟南芥闪烁和T7 GP4非常相似;然而,拟南芥与噬菌体gp4相比,Twinkle在引物酶和解旋酶结构域之间有轻微的延伸,并且有较长的n端区域。
锌指通常与DNA结合域有关,在转录因子中发现了许多例子。锌指还可能参与蛋白质折叠和组装、RNA包装和脂质结合[13].拟南芥闪烁蛋白质,类似于T7 GP4蛋白质,具有功能性锌指结构域。功能性Primase活性需要T7 GP4的锌手指[14].在人类和哺乳动物中,氨基酸的变化,包括祖先锌指基序中半胱氨酸残基的丢失,导致非功能的引物酶结构域[15].
植物细胞石DNA聚合酶的祖先起源是络合。最初,拟南芥蛋白质聚合酶1A和1B分别指定分别聚合酶伽马2和1,在参照人类线粒体DNAPOLγ[16].然而,进一步的研究对该表征有争议,发现植物蛋白与细菌DNA聚合酶I具有更多的共同之处;因此,pol1a和pol1b的名称[17].此外,其他的研究经常提到这些蛋白作为植物细胞器DNA聚合酶,或持久性有机污染物[18].因此,同一种蛋白质有三个不同的名字。在本文中,我们提到的蛋白质是DNA聚合酶1A和1B。这些是已知的唯一在植物叶绿体和线粒体中起作用的DNA聚合酶。Pol1A和Pol1B的噬菌体T7同源物是gp5蛋白。虽然Pol1A和Pol1B彼此之间大约有70%的氨基酸相同,但研究已经确定了各自的几个不同特征。Parent等人报道Pol1B在DNA修复中比Pol1A发挥更大的作用[17,而Pol1A的复制精度更高,能够比Pol1B更有效地取代DNA [19那20.].这些以前的作品表明,POL1A和POL1B不仅仅是冗余的DNA聚合酶,而且每个都具有特定的功能。
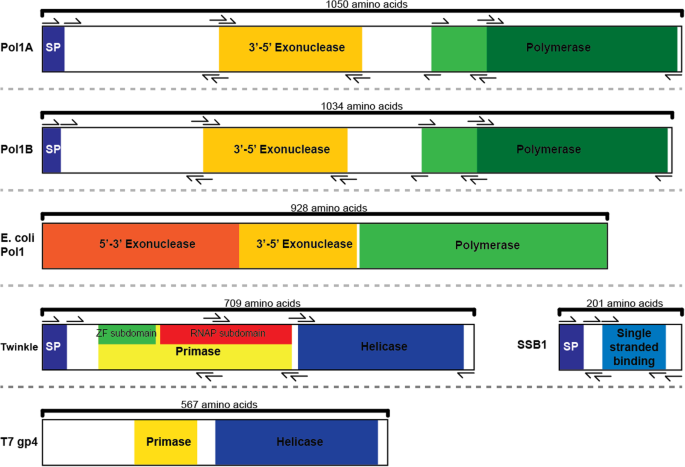
两者之间有一些结构上的区别大肠杆菌DNA聚合酶I和拟南芥Pol1A Pol1B。最明显的是Pol1A和Pol1B缺乏中发现的5 ' -3 '外切酶域大肠杆菌DNA聚合酶i的位置是一长串没有功能分配的氨基酸。在大肠杆菌,这个5 ' -3 '外切酶活性涉及冈崎片段的去除。植物DNA聚合酶中缺乏这一结构域表明,引物的去除已被另一种机制或其他蛋白质所取代。因此,在植物DNA聚合酶的进化过程中,5 ' -3 '外切酶区域可能随着突变的积累而失去了功能。植物和细菌DNA聚合酶的第二个区别是插入3 ' -5 '外切酶结构域和聚合酶结构域之间的氨基酸的延伸(图)。1).在结构预测这需要在一个大的循环结构。
拟南芥Pol1a,pol1b,twinkle,ssb1,大肠杆菌DNA聚合酶I和T7 gp4。“SP”代表SignalP服务器预测的“信号肽”。功能域基于NCBI保守域数据库Uniprot的结果进行了说明[57]由伯恩斯坦数据库和发表作品[58], 灰色的 [15]和理查森[59].利用SignalP预测信号肽区域[60)服务器。除SSB1外,这些蛋白质异常大,特别是与在细胞核中起作用的同源植物和动物蛋白质的长度相比[61].Pol1A和Pol1B中的浅绿色区域是类质体DNA pol a结构域的边界,与代表更一般的DNA pol a结构域的深绿色区域重叠。基因以cDNA和氨基酸长度的比例显示。黑色箭头表示用于创建每个蛋白质截尾的PCR引物。值得注意的是,在我们的酵母双杂交实验中,所有可能的截尾都是通过在选择培养基上交配和生长的单倍体酵母中创建截尾库来同时进行测试的
SSB1与在许多其它生物的同源物的单链DNA结合蛋白。当进行比较,本文将参考T7噬菌体SSB1同源,gp2.5。较早的研究已经表明,在人类中,可以在体外形成,只需颦POLγ[最小线粒体DNA复制体7.].添加mtSSB1蛋白可增强该replisome。这些人类蛋白之间的相互作用模仿在T7噬菌体中发现的简单系统,该系统由gp4 DNA引物解旋酶、gp5 (DNA聚合酶)和gp2.5 (SSB)蛋白组成[8.].
植物利用相似的replisome来维持其细胞器DNA是合乎情理的。我们认为,植物细胞器中的最小DNA replisome由在人类中发现的类似成分组成,并被T7噬菌体利用,即Twinkle、Pol1A或Pol1B和SSB1蛋白。在这篇论文中,我们将展示这些蛋白质的相互作用,重点是Twinkle和DNA聚合酶的特性。我们已经确定了一个预测的无序结构的特定区域,并表明该区域是闪烁与Pol1A和Pol1B相互作用的关键。
结果
我们假设Pol1A, Pol1B和Twinkle相互作用。这是基于之前的研究,详细的DNA复制机制在T7噬菌体以及对人类同源物Twinkle, Po1A, Pol1B和SSB1的体外研究[7.那8.].为了验证我们的假设,我们使用了直接耦合分析(DCA)、酵母双杂交和热电泳。
直接耦合分析闪光,POL1A和POL1B
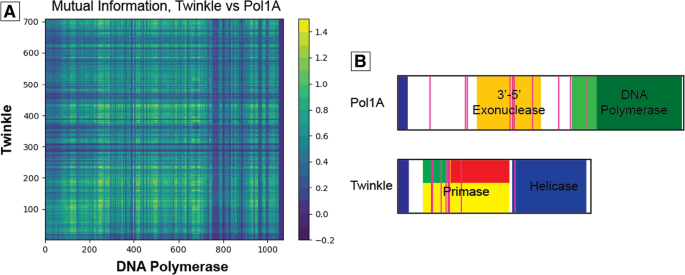
直接耦合分析(Direct coupling analysis, DCA)是一种生物信息学技术,可量化序列中两个位置之间的相互作用,并可通过检查同源基因保存来评估进化约束[21].在我们的案例中,我们计算了从90种植物中提取的DNA聚合酶与Twinkle的直接耦合。虽然DCA通常是在数百个蛋白质序列上进行的,因为我们的模型旨在评估直接耦合拟南芥蒂利亚纳,我们限制了多序列比对,只包括源自植物基因组的蛋白质序列。此外,当蛋白质序列之间的序列相似性小于80%时,DCA会系统地提高[21].为了确保序列数量与仅对少数蛋白质序列非常相似的物种进行抽样所产生的潜在偏差之间的平衡,我们从不同物种的90个可用基因组中取样,只从每个物种中选取了一个蛋白质序列。根据这些数据,我们生成了一张热图,并从每个蛋白质中选择了相互作用可能性最高的10个残基。在Twinkle中识别的10个残基中,有8个在Twinkle的引物域内彼此非常接近(图)。2).对于DNA聚合酶来说,前10个残基在整个蛋白质中分散得更远。每个候选残差的确切位置报告在表中1.
酵母双杂交分析
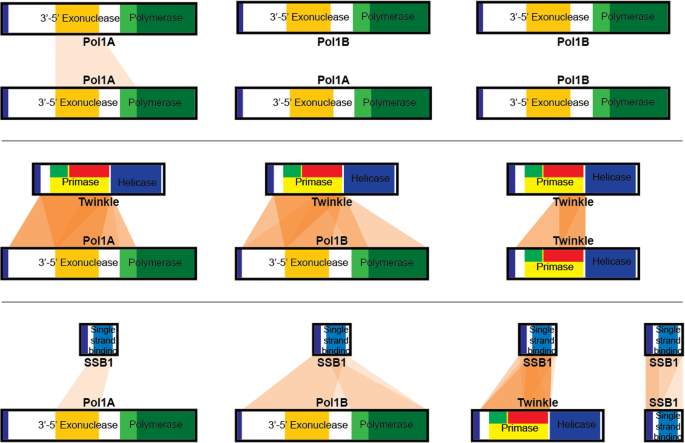
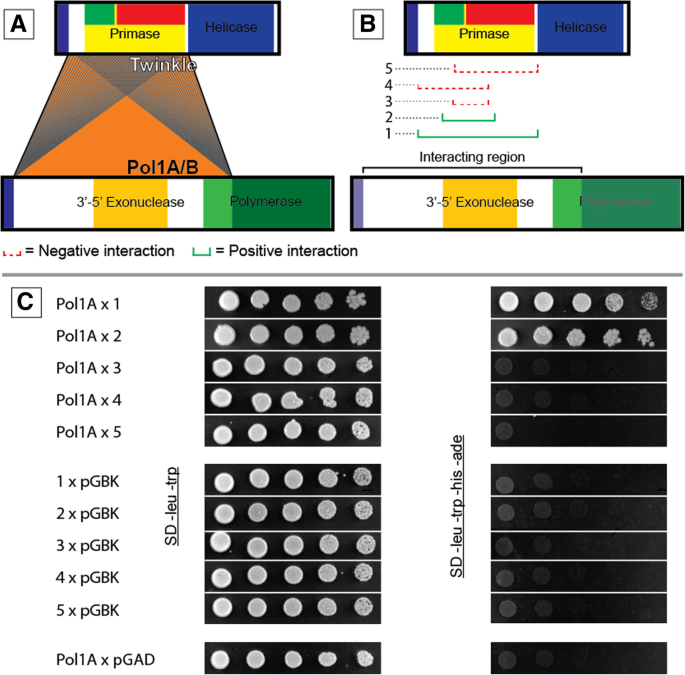
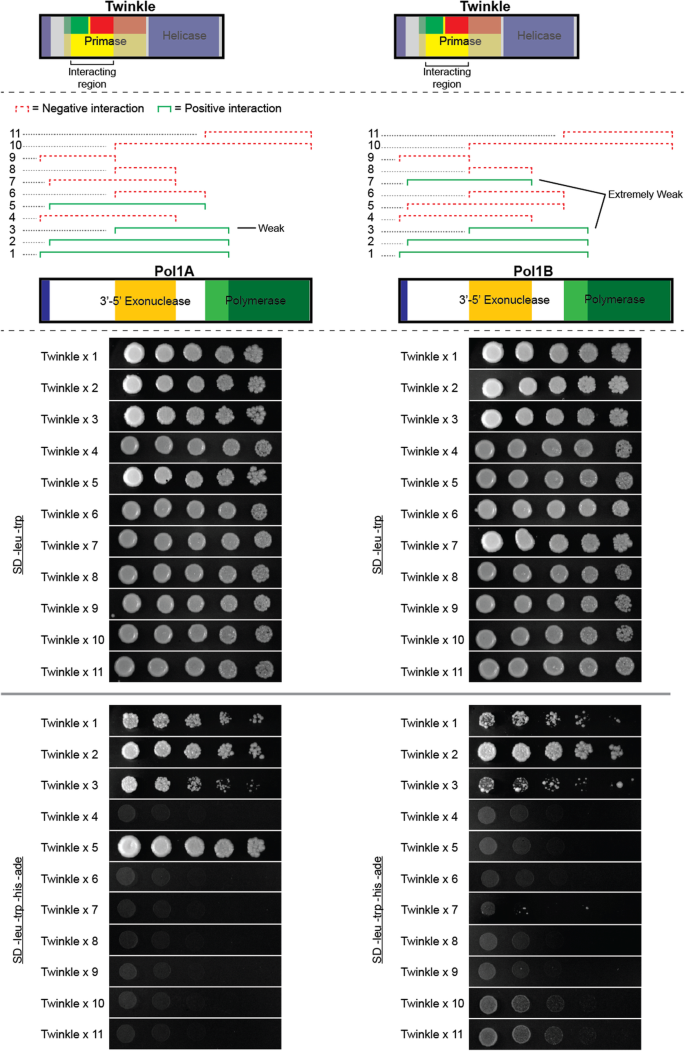
为了确保我们没有错过任何相互作用,我们创造了一系列的蛋白质截断。这些截断的设计如图所示。1.将所有可能的截断片段编译到单倍体酵母细胞中,建立Pol1A、Pol1B、Twinkle和SSB1的截断文库。将单倍体酵母截体文库进行配对,然后在培养基上选择性地进行正互作。这使得我们可以用一种蛋白质的每一个可能的截断来测试另一种蛋白质的每一个可能的截断。图中总结了Pol1A、Pol1B、Twinkle和SSB1截断后的蛋白结构之间的相互作用。3..
我们的图书馆屏幕上显示一闪的特定区域和其他三个蛋白质之间的相互作用一致;Pol1A,Pol1B和SSB1。特别地,闪烁的从预测信号肽和解螺旋酶域之前下游的区域在许多相互作用多次出现。类似的模式与DNA聚合酶发生;从预测的信号肽的下游,并略微聚合酶结构域中内侧的区域出现在许多相互作用。相互作用区域被突出显示图4..这些结果支持我们的假设,即闪烁的N末端区域负责与DNA聚合酶相互作用。然而,这并不一定表明只有聚合酶的N末端区域与闪光相互作用。观察到的聚合酶的相互作用区域非常大并且蛋白质的几个结构域,而不仅仅是n末端区域。因为我们在两种蛋白质中鉴定的区域非常大,所以我们决定通过进一步截断这些区域进一步研究闪光和聚合酶之间的相互作用。
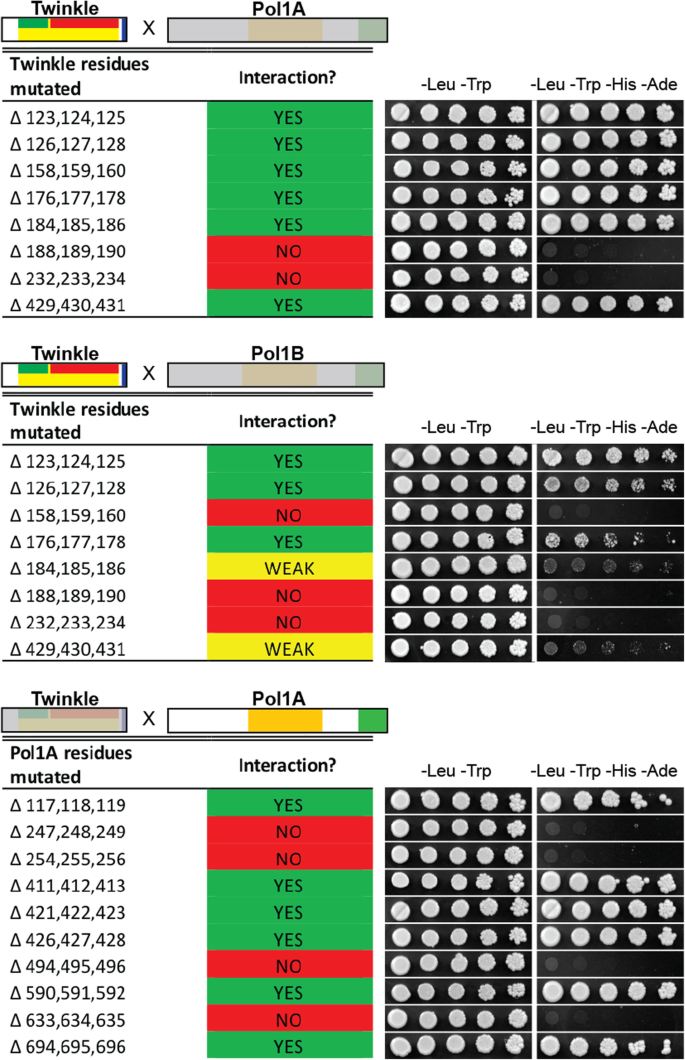
我们还对DCA分析鉴定的10个残基中的每个残基及其在Twinkle和Pol1A中的邻近残基进行突变,以确定它们是否会破坏相互作用(图)。5.).我们选择了Pol1A突变,因为我们之前的工作显示了与Twinkle更积极的关联,因此互动的缺失会更加明显。所有突变都是通过用丙氨酸取代原始残基及其邻基而产生的三丙氨酸突变体。由于DCA分析基于许多物种的基因比对,我们对候选残基及其近邻进行了突变,以纠正Twinkle或Pol1A中关键相互作用残基的不完美定位。Twinkle中的一些候选残基非常接近,我们可以用一个克隆使它们都发生突变。与之前的分析类似,我们观察到Twinkle和Pol1A与Pol1B之间的相互作用要强得多(图1)。5.).我们还检测到Pol1B和Twinkle之间由于点突变而产生的更多相互作用中断。基于DCA结果和我们最初的酵母双杂交分析,我们假设在一个相对较小的区域,Twinkle与Pol1A和Pol1B相互作用。
删除闪烁
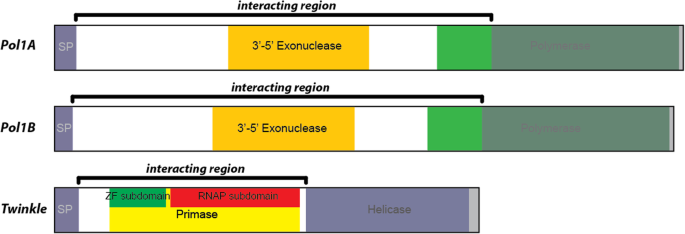
制作闪烁的截短我们设计了从相互作用区域的N-末端或C末端侧缩短10氨基酸增量的蛋白质的引物。我们扩增并测试了不同的截短,直到互动丢失。在找到保留阳性交互的N终端和C终端区域的最大截断后,我们创建了一个组合这些边界的最终截断。闪烁的残留物120-295构成了与Pol1a或pol1b相互作用的最小截断,如图4所示。6.Pol1B结果显示在附加文件中4.).这一区域不存在于任何预测的功能域,进一步加强了我们的假设,即Twinkle的n端区域与DNA聚合酶相互作用。
截断pol1a和pol1b
我们接下来截断DNA聚合酶基因,因为我们用闪烁闪烁,以磨合在特定的相互作用区域上。为此,我们使用从闪烁截头标识的最小区域作为绑定伙伴。在为10个氨基酸缺失设计引物之前,我们使用最初设计在我们的图书馆方法中的引物产生截短(图。1).目的是确定DNA聚合酶中一个明显更小的区域,该区域仍将与Twinkle相互作用,以减少产生数十个氨基酸截断所需的工作量。然而,我们发现,我们不能减少的相互作用区域的聚合酶远比我们最初的筛选显示(图。7.),附加的文件5..我们还注意到Pol1A和Pol1B的不同区域能够与我们已经确定的Twinkle的小区域相互作用。每次相互作用的斑点稀释暗示了Pol1A和Twinkle与Pol1B和Twinkle之间更强的关联。
Twinkle/DNA聚合酶相互作用的热电泳分析
热泳是生化测定,通过使用一个温度梯度的分子之间的相互作用的措施。分子,在这种情况下的蛋白质悬浮在其由激光加热的液体。激光聚焦加热液体的特定点,从而在周围区域的温度梯度。通过该温度梯度的分子运动指示与更小的分子更快地移动比大的分子的相对大小。如果两个分子或蛋白质彼此相互作用,所述复合物延缓的较大尺寸通过液体的运动。此运动可被用于计算两个相互作用的分子的解离常数。使用热泳,我们能够确定颦Pol1B的不同区域之间的结合强度。
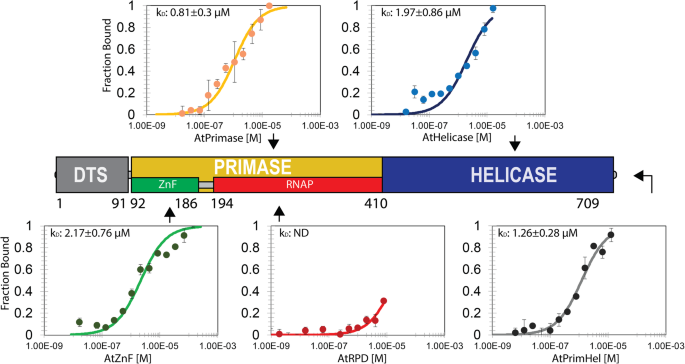
虽然酵母-双杂交实验表明Pol1A和Twinkle之间有更强的相互作用,Pol1B用于确定与Twinkle及其不同结构模块的结合常数,因为这种重组蛋白在蛋白纯化过程中有更好的产量,而且Pol1A和Pol1B之间存在70%的氨基酸同源性。在这个实验中,一个多组氨酸标记的Pol1B和配体(全长Twinkle, Zn++使用手指结构域、RNAP结构域、引物酶模块和解旋酶模块)。他标记的Pol1B用荧光染料标记,在结合实验中,荧光染料的数量保持在10 nM不变。随着配体浓度的增加孵育后,样品被装入MST标准毛细血管。Pol1B与全长Twinkle、引物和解旋酶结构域以及锌指亚结构域均有特异性结合。RNAP区域与Pol1B没有明显的相互作用(数据未显示;区域如图所示。8.).热电泳拟合得到配体的解离常数Pol1B-Twinkle为1.26 μM, Pol1B-Primase为0.81 μM, pol1B-Helicase为1.97 μM, pol1B-zf为2.17 μM(图)。8.).这些值与T7 DNA聚合酶和双官能T7丙酶 - 螺旋酶之间的结合亲和力相当[22那23那24].在DNA存在下,这种相互作用的解离常数约为0.44 μM [25].在细菌中,DNA引物酶和解旋酶活性被编码为两个独立的多肽。在枯草芽孢杆菌dnaG引物酶蛋白与DNA聚合酶dnaE解旋酶相互作用,解离常数为0.75 μM [26].最后,含锌指蛋白被描述为与DNA聚合酶相互作用的伙伴,在这种情况下,锌指蛋白对两种蛋白质之间的物理相互作用至关重要[27那28].对Twinkle和Pol1B相互作用的证实进一步支持了这些酶在细胞复制中的作用拟南芥蒂利亚纳细胞细胞基因组。
DNA Pol1B与一闪域热泳结合。The DNA polymerase 1B was labeled as the target protein at 20 nM concentration and assayed in microscale thermophoresis experiments with Twinkle, the primase and helicase domains, and the zinc finger subdomain. The thermophoretic data was fitted to the Kd equation described in materials and methods. The zinc finger subdomain was able to bind to the PolB and a Kd was measured at 2.17 μM. The RNAP subdomain (red) did not show binding (data not shown). The primase and helicase domain were able interact with the polymerase with a Kd of 0.81 and 1.97 μM respectively. The full-length protein interacts with a Kd of 1.26 μM. All proteins were titrated in 16 serial dilutions from different concentrations. Graphs were plotted at x axis with enzyme concentration and at the y axis with normalized fluorescence. Error bars represent the standard error for three measurements
闪烁T-DNA突变分析
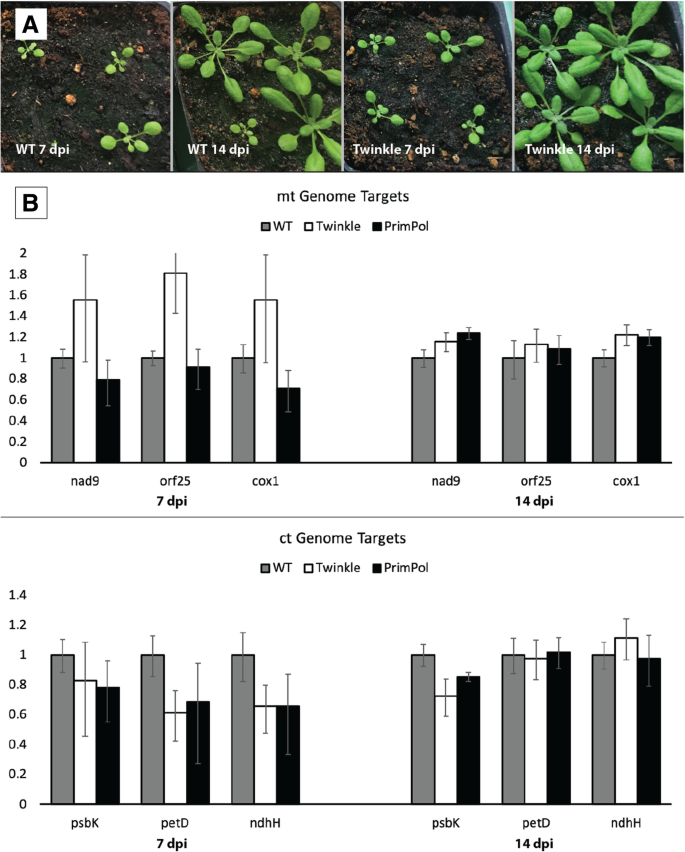
在确定了Twinkle与DNA聚合酶之间的正相互作用后,我们获得了T-DNA突变体拟南芥Twinkle (T-DNA突变体SALK_152246)和另一种潜在引物蛋白PrimPol (At5g52800;T-DNA插入系Salk_090163).令人惊讶的是,Twinkle突变体似乎不受这种蛋白质丢失的影响(图。9.一个);PrimPol突变体也没有明显的表型(未显示)。随后对基因组拷贝数进行相对qPCR分析,发现线粒体或叶绿体DNA水平相对于核基因组没有显著变化,这是由于Twinkle或PrimPol插入突变(图)。9.b).考虑到Twinkle同源物对于T7噬菌体和动物线粒体基因组的维持都是必不可少的,我们惊讶地发现,与野生型植物相比,突变型植物没有明显的表型或基因组拷贝数变化。
讨论
我们已经确定了一个区域拟南芥闪烁DNA引物酶/解旋酶是与两种细胞器DNA聚合酶相互作用的关键。在这个区域内,几个关键残基的突变导致Pol1A或Pol1B完全解离,这是由酵母双杂交分析确定的。我们还发现,Pol1A关键残基的突变破坏了与Twinkle的相互作用。虽然Twinkle中的关键残基聚类相当紧密,但这些DNA聚合酶中的关键残基间距要大得多。我们怀疑这是我们不能产生更小的Pol1A或Pol1B截断的主要原因,维持与Twinkle的相互作用,而Twinkle的相互作用区域定位于一个非常狭窄的蛋白质区域。这支持了我们的假设,即Twinkle的n端区域与Pol1A或Pol1B相互作用。然而,它并没有证实DNA聚合酶的n端区域是与Twinkle正相互作用的关键。我们之前的研究结果也显示,Twinkle的这一区域与SSB1有关,支持了Twinkle协调最小DNA replisome组装的观点。由于大多数维管植物具有相同的细胞器复制蛋白,这一模式很可能在其他物种的高等植物叶绿体和线粒体中重复。
虽然我们已提供的证据显示,星星和DNA聚合酶可能形成一个最小的DNA复制体,我们不知道这是否是在植物细胞器中使用的唯一的DNA复制体。此外,其他的蛋白质可提供辅助功能,例如在大肠杆菌[29那30.].例如,几乎没有关于底漆去除的研究。2个5 ' -3 '外切酶已在拟南芥细胞器[31那32一种定位于线粒体,另一种定位于叶绿体,但对它们的作用机制还没有任何研究。核糖核酸酶已被报道在植物线粒体和叶绿体中,但其特征功能是在mRNA和tRNA的加工过程中[33].我们也展示了强烈的相互作用拟南芥闪烁与SSB1,它可能形成一个关键部分的DNA replisome。
Pol1A和Pol1B已被证明具有足够的过程性,可以复制整个细胞器基因组[17那34].这比加工得多大肠杆菌DNA聚合酶I,主要参与冈崎片段加工和DNA修复,不能复制超过几十个碱基[35].T7 DNA聚合酶gp5本身也缺乏加工能力,但当与gp5结合时大肠杆菌患有毒素变得高度加工。有趣的是,重组版本的大肠杆菌能够结合硫氧还蛋白的DNA聚合酶I在过程中显示出戏剧性的增加[36].植物细胞器DNA聚合酶也可能结合硫氧还蛋白或其他加工因子以实现更大的加工能力,但这从未被证实。如果这种情况发生,这将有助于解释为什么这些酶具有比大肠杆菌DNA聚合酶I,尽管它们在序列和功能上非常相似。
到目前为止,在植物叶绿体和线粒体中除了Pol1A和Pol1B之外,没有发现其他细胞器DNA聚合酶。除了DNA聚合酶(s),确定什么帮助解开和启动DNA复制对我们理解植物细胞器中的DNA复制至关重要。我们已经证明了DNA聚合酶和Twinkle之间的关联,表明这两种蛋白质是这些细胞器中DNA replisome的一部分。然而,纯合子的闪烁T-DNA插入突变体拟南芥显示在植物的生长没有显着的缺陷并且在细胞器基因组拷贝数没有显着的降低相比于野生型植物(图9.).这种植物的生长方式与WT相似。这令人费解,因为T7 gp4解旋酶-引物酶之间的类似相互作用对于噬菌体DNA的过程复制至关重要[24].此外,有条件的Twinkle敲除小鼠未能存活,并显示出心脏和骨骼肌组织中线粒体DNA的快速损耗[37].如果“闪”基因敲除的植物生长良好,这强烈表明另一种蛋白质可能提供DNA聚合酶功能所需的引物酶活性。然而,之前的工作表明,Twinkle可以有效地启动Pol1A和Pol1B的DNA合成[12].同样的研究表明拟南芥闪烁不能启动T7或大肠杆菌DNA聚合酶I.我们和其他(Brieba和Morley,未发表的观察)表明,如果它们具有至少一种功能性细胞细胞素DNA聚合酶,植物可以在几乎没有可见的生长缺陷中存活。尽管缺乏表型,但突变POL1A或POL1B导致细胞器基因组拷贝数减少近似30%[38].在这些减少的细胞器基因组拷贝数上的生长似乎不受弱光条件或干旱的影响(数据未显示)。
对“闪”基因敲除植物正常生长的一个简单解释是,有另一种DNA复制蛋白1)在复制叉处有主要的解旋酶/引物酶活性,或2)另一种蛋白补偿了“闪”基因活性的丧失。在这两种情况下,拟南芥的Twinkle功能存在明显的冗余。其他候选蛋白包括Twinky (At1g30660)和PrimPol (At5g52800),一种独特的引物/聚合酶蛋白。然而,这两种蛋白质中的任何一种的T-DNA插入突变也不会导致可识别的生长缺陷(未发表的观察结果)。无论如何,Pol1A和Pol1B必须参与其中,因为它们是在细胞器中发现的唯一的DNA聚合酶,而我们和其他人已经无法在两个基因中制造双纯合突变体。
在之前的研究中,我们报道了具有Pol1B突变的植物相对于具有Pol1A突变的植物,细胞器基因组拷贝数下降更大,特别是在线粒体中[38那39].我们还观察到这些植物的生长有轻微的延迟。这表明,Pol1B在细胞器DNA复制中可能比Pol1A更重要。如果最小的replisome是由Twinkle和DNA聚合酶组成的,根据我们之前的研究结果,我们预计Twinkle会与Pol1B有很强的相关性。然而,我们的研究表明Pol1A有更强的关联性。这可以用不同的情况来解释:与Twinkle的强相互作用可能不利于DNA复制,或者Pol1A和Pol1B的作用可能没有那么多冗余,彼此之间更加不同。支持这一观点的是,一项研究表明,Pol1B主要参与DNA修复[17].其他研究表明,这两种DNA聚合酶都可以执行翻译修复,但Pol1B在链置换中比Pol1A更有效[19那40].这表明,在线粒体和叶绿体中,Pol1A可能更多地参与DNA复制,而Pol1B更多地参与DNA修复。这与玉米的研究结果相反,在玉米中,两种细胞器DNA聚合酶中只有一种被证明是负责玉米质体基因组复制的,因为这一单一基因的突变本质上消除了叶绿体DNA复制[41].这个突变体的玉米有大约在线粒体DNA的两倍的降低,这意味着该第二DNA聚合酶可以在线粒体中起作用。
此外,重组依赖复制(recombination dependent replication, RDR)可以解释Twinkle突变体缺乏表型。RDR在植物细胞器中的广泛应用已被证明是维护和修复线粒体和质体DNA的一种手段[42].This is especially true in mitochondria where direct and inverted sequence repeats of ≥50 bp are present throughout the genome. Mutations in recombination proteins lead to genome instability and are often lethal. One prominent example is RecA, a homolog of the bacterial replication protein RecA. Arabidopsis possess three RecA homologs that localize to the organelles (RecA1 [At1g79050], RecA2 [At2g19490], and RecA3 [At3g10140]). Mutations to these homologs lead to delayed phenotypes, increased recombination, and in the case of RecA2 plant death beyond the seedling stage [43那44那45].其他定位于细胞器的重组蛋白包括单链DNA结合蛋白Whirly [46那47]和osb [48],以及名为Msh1的MutS同系物[49].这些蛋白质的任何突变都会导致非法重组的增加,并对植物的发育产生不利影响。
结论
我们用三种独立的方法证实了Twinkle与细胞器DNA聚合酶Pol1A和Pol1B之间的正相互作用。我们使用了经典的分子生物学技术(酵母双杂交)、生物信息学(DCA)和生物化学(热电泳)来定义这些相互作用的关键区域和残基。这三种方法提供了相互作用的确认,并可应用于其他蛋白质-蛋白质相互作用的研究。
对植物细胞器的完整DNA replisome的进一步研究将确定参与线粒体DNA复制的其他蛋白质拟南芥,包括“闪”突变的时候。研究对象包括Twinky, PrimPol, RecA, Whirly和MutS。我们用同样的方法来证明Twinkle和DNA聚合酶之间的相互作用,可能会揭示这些候选蛋白中对DNA replisome组装和功能至关重要的特定区域。
方法
直接耦合分析
我们通过直接偶联分析来自90种植物物种的直接偶联分析,计算DNA聚合酶(POLIA,AT1G50840和POLIB,AT3G20540)和闪烁(AT1G30680)基因之间的直接和间接相互作用。首先,我们手动从国家生物技术信息中心下载每个DNA聚合酶和闪烁基因(NCBI)[50,确保每种植物都包含两种基因的完整基因注释。将下载的基因分离成两个FASTA文件,每个FASTA文件对应一个基因,我们分别对每个FASTA文件进行多序列比对。我们使用以下CLUSTAL OMEGA [51,其中${INPUT}是包含未对齐基因的FASTA文件,${OUTPUT}是包含对齐基因的FASTA文件:
clustalo -i ${INPUT} > ${OUTPUT}
不同的多个序列对准器通过派斯鲁伊奎拉[A比较52]表明,CLUSTAL OMEGA执行相对较好为全长基因序列,类似序列在我们的分析中使用。
在对每一组序列进行单独比对之后,我们通过在聚合酶II基因的末端添加20个天冬酰胺残基,然后连接每个物种的Twinkle序列,将每个物种的比对序列连接到一个单独的FASTA文件中。这种人工缓冲确保了两个基因之间的相互作用不受组合基因的影响(例如,残基邻近性),并促进了我们快速区分两个基因的能力。我们使用MATLAB实现DCA [53]为了在两种基因之间识别每双对残留物比较中的直接信息和相互信息。输出文件包含四列:第一氨基酸残基的位置,比较氨基酸残基的位置,两个残基之间的直接信息的量,以及两个残留物之间的相互信息的量。我们使用以下命令执行直接耦合分析,其中$ {输入}是组合的FASTA文件,$ {output}包含每个成对比较的直接信息和相互信息:
MATLAB -nodisplay -nojvm -nosplash -r “DCA $ {INPUT} $ {} OUTPUT”
自氨基酸残基在DCA分析报告来自结合多重序列比对(即第一残留的位置闪烁开始后20天冬酰胺残留,最后残留的聚合酶II),我们决定这残留残留在原始的对齐拟南芥蒂利亚纳序列并仅从DCA输出文件中提取那些成对比较。然后我们重新编号一致的氨基酸残基序列与原对齐(即第一个聚合酶II的残渣标记输出文件的位置在第一列和第一残渣闪烁的标签位置输出文件的第二列)。最后,使用这些标签,我们使用Python 2.7版的matplotlib和scipy.interpolate.griddata库创建了计算出的互信息的热图。我们使用热图直观地识别两个基因之间较高互信息的区域。
酵母双杂交分析
使用来自Clontech的材料和协议进行酵母 - 二杂化分析。这包括使用Matchmaker金菌株的酵母酵母,用于小型乙酸锂方案后的小规模酵母转化。通过将所有可能的截短进入PGADT7(捕食)和PGBK(诱饵)质粒(额外文件所示的引物,通过克隆所有可能的截断来创建POL1A,POL1B,闪光和SSB1(AT4G11060)的截断库(AT4G11060)1:表S1和附加文件2:表S2).这些截断部分首先被克隆到大肠杆菌以产生大量的质粒DNA。将获得的质粒截断文库克隆到MATa和MATa α单倍体酵母中。随后将合适的单倍体酵母文库进行配对,并在培养基上选择性地进行正互作。从酵母菌落中提取DNA, PCR扩增质粒DNA,并对PCR产物进行测序,确定截断边界。所有在阳性相互作用中鉴定的构建物都在诱饵和猎物质粒中进行测试,并通过将目标蛋白转化为空诱饵或猎物质粒来进行自激活,以消除假阳性相互作用的可能性。在交配实验中,Matchmaker gold酵母与从克隆技术公司购买的规范化cdna拟南芥文库一起使用。
建立了三丙氨酸取代突变体,以测试DCA分析突出的残基的突变效果。这些突变体是利用“环角PCR”创造的。在该方法中,我们使用正确插入了Twinkle或Pol1A的pad或pGBK质粒作为模板DNA进行PCR。利用正向引物和反向引物,分别与质粒的碱基对为5 ',利用高保真DNA聚合酶延长质粒的长度。该引物具有一个约15 bp的三丙氨酸错配区,两侧有Twinkle或Pol1A的同源序列。得到的PCR产物是一个质粒大小的钝端DNA分子,具有我们设计的三丙氨酸取代。这个分子的钝端被连接在一起并转化成大肠杆菌。采集菌落,培养和收获质粒DNA,通过Sanger测序检查三丙氨酸替代的正确插入。一旦证实,质粒转化为酵母,并使用选择性培养基测量相互作用。
热泳
克隆和表达
闪烁缺乏前91个密码子用作用于构建DNA引用和旋光酶结构域的模板,以及RNAP和锌指亚域。将预ase结构域(残基92-410),RNAP和锌指亚域(分别将残基194-410和92-186分别)克隆到PET-19b中并如所述纯化[12那19].解旋酶结构域(401-709)克隆到pcr -1b载体中[54如前所述,将Tris-HCl转换为pH7.0的缓冲液组成的磷酸钾。使用PreScission蛋白酶去除pET结构的标签,使用TEV蛋白酶去除pcr -1b的结构。
微尺度热泳
POL1B用纳米罐的整体标签标记套件红润-NTA标记为100nm浓度。荧光标记的蛋白质以恒定浓度的20nM使用。在16.8μm的16.8μm中滴定标记的POL1b,对于闪光酶结构域的12.8μm,对于螺旋酶结构域,70μm,对于锌指子域的30μm,对于锌指子域,30μm。将反应在PBS缓冲液中孵育+ 0.05%吐温。使用纳米液体单片NT.115P仪器进行测量,分析以10%的LED功率和50%MST功率进行标准毛细管进行。根据等式装配来自硫定量变化的数据:
其中U为未结合态的响应值,B为结合态的响应值,CT为荧光分子的最终浓度。
qPCR
从野生型、Twinkle和Primpol突变体植株中收获7和14 dpi的叶片组织。采用米纳斯等人采用的十六烷基三甲基溴化铵(CTAB)方案从组织样本中分离出适合qPCR的高质量DNA。55].采用相对qPCR分析,通过测量3个线粒体和3个叶绿体DNA靶点来测量细胞器基因组拷贝数(附加文件)3.:表S3)。包含的nad9线粒体靶(NADH脱氢酶铁硫蛋白3),orf25(开放阅读框,编码基于结构相似性线粒体ATP合酶的植物B亚基),和COX1(细胞色素C氧化酶亚基1)。叶绿体目标包括psbK(光系统II反应中心:蛋白酶K的前体),petD(细胞色素B6-F复合体亚基4)和ndhH(NAD(P)H醌氧化还原酶亚基H)。这些目标是独一无二的线粒体和叶绿体DNA,以避免重叠与核基因组。AtRpoTp(噬菌体样RNA聚合酶,核编码的,质体定位的)被用作标准来衡量拷贝水平。使用Applied Biosystems的StepOne加定量PCR机并在加电SYBR Green试剂相对基因拷贝数进行了分析。技术和生物重复进行了汇编,并使用ΔΔCT方法[进行分析56].
数据和材料的可用性
在本研究中使用和/或分析的数据集可从通讯作者在合理要求。对齐文件存放在Treebase, ID 24443;http://purl.org/phylo/treebase/phylows/study/TB2:S24443
GenBank登录号码为拟南芥蒂利亚纳本研究使用的基因:
DNA PolIA, At1g50840, GenBank登录NM_103965。
DNA PolIB, At3g20540, GenBank登录NM_001203013。
闪烁,At1g30680, GenBank登录NM_179404。
SSB1, At4g11060, GenBank登录NM_117176。
PrimPol, At5g52800, GenBank登录NM_001161303。
Twinky, At1g30660, GenBank登录NM_102803。
缩写
- DCA:
-
直接耦合分析
- mt:
-
线粒体
- PolIA:
-
DNA聚合酶IA
- PolIB:
-
DNA聚合酶IB
- 单边带:
-
单链DNA结合蛋白
- zf:
-
锌指
参考文献
- 1.
阿奇博尔德JM。内共生与真核细胞进化。咕咕叫医学杂志。2015;25 (19):R911-R21。
- 2.
莫利SA,Nielsen BL。植物线粒体DNA。前面Biosci-landmrk。2017; 22:1023-32。
- 3.
黄志强,王志强,王志强,等。植物叶绿体的还原进化。咕咕叫麝猫。2018;64(2):365 - 87。
- 4.
植物线粒体的DNA复制。线粒体。2014;19:231-7。
- 5.
奥德斯堡DJ,Bendich AJ。植物血浆中的DNA维持和线粒体。前植物SCI。2015; 6。
- 6.
植物线粒体基因组:动态与维护。Biochimie。2014;100:107-20。
- 7.
Korhonen Ja,Pham Xh,Pellegrini M,Falkenberg M.在体外重建最小的MTDNA重新替换。Embo J. 2004; 23(12):2423-9。
- 8。
李SJ,理查德森CC。噬菌体T7 DNA复制的编排。CURR OPIN化学生物学。2011; 15(5):580-6。
- 9。
李凤英,李凤英,李凤英,等。人类线粒体DNA缺失与编码Twinkle的基因突变有关,Twinkle是一种定位于线粒体的噬菌体T7基因4样蛋白(vol 28, pg 223, 2001)。Nat麝猫。2001;29(1):100。
- 10。
TWINKLE具有5 '-> 3 ' DNA解旋酶活性,可被线粒体单链DNA结合蛋白特异性刺激。中国生物医学工程学报。2003;27(4):491 - 497。
- 11.
拟南芥At1g30680基因编码与噬菌体T7 gp4蛋白同源物,具有DNA引物酶和DNA解旋酶活性。13.生物谷bioon . 2013;
- 12.
Peralta-Castro A, Baruch-Torres N, Brieba LG。植物细胞器DNA引物解旋酶利用独特的识别序列为细胞器DNA聚合酶合成RNA引物。核酸学报2017;45(18):10764-74。
- 13.
Laity JH, Lee BM, Wright PE。锌指蛋白:结构和功能多样性的新见解。中国生物医学工程学报。2001;11(1):39-46。
- 14.
噬菌体T7引物复合物的分子相互作用。美国国立科学院。109(24): 9408 - 2012; 13。
- 15.
关闭TE,灰色MW。闪烁是一种线粒体复制DNA解旋酶,广泛存在于真核生物的辐射中,在大多数真核生物中也可能是线粒体DNA引物酶。中国科学(d辑:地球科学)2006;62(5):588-99。
- 16.
Roy S, Choudhury SR, Singh SK, Das KP。AtPol lambda是拟南芥中哺乳动物DNA聚合酶lambda的同源物,通过暗修复途径参与UV-B诱导的DNA损伤修复。植物生理学杂志。2011;52(2):448-67。
- 17.
亲本JS, Lepage E, Brisson N.拟南芥的两个polielle细胞器DNA聚合酶的不同作用。植物杂志。2011;156(1):254 - 62。
- 18.
森山T,寺泽K,持久性有机污染物的佐藤N.保护,所述植物细胞器DNA聚合酶,在真核细胞中。单细胞生物。2011; 162(1):177-87。
- 19。
Baruch-Torres N, Brieba LG。植物细胞器DNA聚合酶是复制型和转译型DNA合成聚合酶。核酸学报2017;45(18):10751-63。
- 20。
阿亚拉-加西亚VM, Baruch-Torres N, Garcia-Medel PL, Brieba LG。植物细胞器DNA聚合酶具有不同的核苷酸掺入保真度。2月j . 2018; 285(21): 4005 - 18。
- 21。
Morcos F, Pagnani A, Lunt B, Bertolino A, Marks DS, Sander C, et al.;残基协同进化的直接耦合分析捕获了许多蛋白质家族的天然联系。美国国立科学院。2011; 108(49):E1293-E301。
- 22。
Hamdan Sm,Johnson de,Tanner Na,Lee JB,Qimron U,Tabor S等人。动态DNA直升机-DNA聚合酶相互作用确保加工复制叉运动。Mol细胞。2007; 27(4):539-49。
- 23.
张高清,李SJ,朱镕基,TRAN NQ,塔博尔S,Richardson CC。直升机-DNA聚合酶相互作用对于引发前导股DNA合成至关重要。美国国立科学院。2011; 108(23):9372-7。
- 24.
Kulczyk AW,Akabayov B,李SJ,Bostina男,贝尔科维奇SA,理查德森CC。DNA聚合酶解旋酶和之间的相互作用是噬菌体T7复制体的高持续合成能力是必不可少的。J Biol Chem。2012; 287(46):39050-60。
- 25.
关键词:噬菌体T7, DNA解旋酶,引物酶,聚合酶,复制中间体生物化学学报。2016;291(3):1472-80。
- 26.
Rannou O, Le Chatelier E, Larson MA, Nouri H, Dalmais B, Laughton C, et al.枯草芽孢杆菌中DnaE聚合酶、DnaG引物酶和DnaC解旋酶在三元复合物中的功能相互作用及滞后链DNA复制过程中引物酶与聚合酶的传递。核酸学报2013;41(10):5303-20。
- 27.
蛋白-蛋白相互作用在复制蛋白a (RPA)功能中的作用:RPA通过多种机制调节DNA聚合酶a的活性。Biochemistry-Us。1997, 36(28): 8443 - 54。
- 28.
DNA连接酶III和DNA聚合酶gamma之间的新相互作用在线粒体DNA稳定性中起着重要作用。j . 2007; 402:175 - 86。
- 29.
replisomes介导的DNA复制。生物化学学报2001;70:181-208。
- 30.
姚N,细菌O 'DM,机器ER。JSM生物化学和分子生物学。杂志2016;3(1):1013。
- 31。
Theologis A, Ecker JR, Palm CJ, Federspiel NA, Kaul S, White O, et al.;拟南芥1号染色体的序列分析。大自然。2000;408(6814):816 - 20。
- 32。
Salanoubat M, Lemcke K, Rieger M, Ansorge W, Unseld M, Fartmann B,等。拟南芥3号染色体的序列分析。大自然。2000;408(6814):820 - 2。
- 33。
绿色PJ。高等植物的核糖核酸酶。植物学报1994;45:421-45。
- 34。
森山T,佐藤N.酶参与光合细胞器真核生物DNA复制。前植物SCI。2014; 5。
- 35.
恩氯。6 - DNA聚合酶。殷H-M,编辑。重组DNA技术的酶学引物。圣地亚哥:学术出版社;1996.p . 345 - 489。
- 36.
噬菌体T7 DNA聚合酶的硫氧还蛋白结合域对大肠杆菌DNA聚合酶具有加工性。1997, 94(2): 479 - 84。
- 37.
动物线粒体复制DNA解旋酶的结构、功能及进化。生物化学学报2016;51(1):53-64。
- 38.
莫利SA,Nielsen BL。细胞器DNA聚合酶突变体的植物发育过程中叶绿体DNA拷贝数变化。前植物SCI。2016; 7。
- 39.
Cupp JD, Nielsen BL.拟南芥(Arabidopsis thaliana)细胞器DNA聚合酶IB突变体表现出mtDNA水平随线粒体面积密度的降低而降低。杂志杆菌。2013;149(1):91 - 103。
- 40.
Trasvina-Arenas CH, Baruch-Torres N, Cordoba-Andrade FJ, Ayala-Garcia VM, Garcia-Medel PL, Diaz-Quezada C, et al.;植物细胞器DNA聚合酶中负责5 '-dRP裂解酶和链置换活性的独特插入的鉴定:对碱基切除修复的影响DNA修复。2018;65:1-10。
- 41.
黄志强,王志强,王志强,等。玉米叶绿体基因拷贝数降低对叶绿体基因表达的影响。植物杂志。2012;160(3):1420 - 31所示。
- 42.
马雷夏尔A,布里松N.重组和植物细胞器基因组稳定性的维护。新植物。2010; 186(2):299-317。
- 43。
Shedge V, Arrieta-Montiel M, Christensen AC, Mackenzie SA。植物线粒体重组监测需要不同寻常的RecA和MutS同源物。植物细胞。2007;19(4):1251 - 64。
- 44。
罗文Ba,奥德斯堡DJ,林教岛AJ。RECA保持拟南芥中叶绿体DNA分子的完整性。J Exp Bot。2010; 61(10):2575-88。
- 45。
Miller-Messmer M, Kuhn K, Bichara M, Le Ret M, Imbault P, Gualberto JM。依赖reca的DNA修复导致拟南芥线粒体基因组的异质性增加。植物杂志。2012;159(1):211 - 26所示。
- 46。
拟南芥涡旋家族的dna结合蛋白以细胞器为靶点。2月。2005;579(17):3707 - 12所示。
- 47.
在拟南芥中,Whirly蛋白维持质体基因组的稳定性。美国国立科学院。2009; 106(34): 14693 - 8。
- 48.
张志强,张志强,张志强,等。植物特异性ssDNA结合蛋白OSB1参与拟南芥线粒体DNA的化学计量传递。植物细胞。2006;18(12):3548 - 63。
- 49.
Abdelnoor RV, Yule R, Elo A, Christensen AC, Meyer-Gauen G, Mackenzie SA。植物线粒体基因组的亚化学计量转移受到MutS同源基因的影响。美国国立科学院。2003, 100(10): 5968 - 73。
- 50.
国家生物技术信息中心数据库资源协调员。核酸Res. 2018;46(D1): D8-D13。
- 51.
西弗斯F,希金斯DG。Clustal Omega用于对许多蛋白质序列进行精确比对。蛋白质科学。2018;27(1):135 - 45。
- 52.
派斯FS,鲁伊奎拉PC,Oliveira的G,科英布拉RS。评估多个序列比对程序的效率。算法分子生物学。2014; 9(1):4。
- 53.
Morcos F, Pagnani A, Lunt B, Bertolino A, Marks DS, Sander C, et al.;残基协同进化的直接耦合分析捕获了许多蛋白质家族的天然联系。中国科学(d辑:地球科学)2011;108(49):E1293-301。
- 54.
Goulas T, Cuppari A, Garcia-Castellanos R, Snipas S, Glockshuber R, Arolas JL, et al.;pcr系统:用于重组蛋白表达和纯化的载体收集。《公共科学图书馆•综合》。2014;9 (11):e112643。
- 55。
Minas K,Mcewan NR,Newbold CJ,Scott KP。从瘤胃流体,植物和细菌纯培养物提取QPCR级DNA的高通量CTAB方案的优化。有限元微生物吧。2011; 325(2):162-9。
- 56。
Schmittgen TD, Livak KJ。采用比较C-T法分析实时PCR数据。Nat Protoc。2008;3(6):1101 - 8。
- 57。
罗伊克A,Consortium U. Uniprot:通用蛋白质知识库(第45 Vol 45,PG D158,2017)。核酸RES。2018; 46(5):2699。
- 58。
噬菌体T7基因4蛋白的7-kDa区域对引物酶是必需的,但对解旋酶活性不是必需的。中国科学院院刊1988;85(2):396-400。
- 59.
日下T,理查德森CC。锌基序的序列识别通过DNA primases的作用。J Biol Chem。1996; 271(32):19563-70。
- 60.
利用SignalP预测分泌蛋白。出自:Kihara D,编辑。蛋白质功能预测:方法与方案。纽约,NY:施普林格纽约;2017.59 - 73页。
- 61.
因为它们是由比动物蛋白的外显子编码的较少拉米雷斯桑切斯O,佩雷斯-Rodriguez的P,Delaye L,迪森A.植物蛋白是小。GENOM蛋白质组Bioinf。2016; 14(6):357-70。
确认
我们感谢来自博士的有益的意见和建议。乔尔Griffitts和朱丽安·格罗斯,微生物学和分子生物学的杨百翰大学部。
资金
这项研究在支持植物线粒体DNA replicaion和重组的尼尔森实验室由BYU指导环境格兰特的支持;杨百翰大学研究生研究奖学金到SM到的植物线粒体DNA基因组的维护,并从微生物学和分子生物学系的本科生和研究生的学生支持支持分析。在LGB实验室热泳研究是由国家科学技术委员会,西恩西亚BASICA支持授予#253757。
作者信息
隶属关系
贡献
BLN和SM设计了整体研究并写了稿件。SM执行了大部分酵母 - 二杂化实验和分析。APC和LGB进行了热透镜实验。JM和Pr执行了直接耦合-AnayLSIS。KLO,AO和SA辅助酵母二杂交实验。所有作者均阅读并批准了手稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
莫雷,南卡罗来纳州,佩拉尔塔-卡斯特罗,布里巴,L.G.et al。拟南芥蒂利亚纳细胞器模拟T7噬菌体DNA replisome与闪烁蛋白和DNA聚合酶Pol1A和Pol1B之间的特定相互作用。BMC植物杂志19,241(2019)。https://doi.org/10.1186/s12870-019-1854-3
已收到:
公认:
发表:
关键字
- 拟南芥
- Organellar DNA复制
- Yeast-two-hybrid
- 直接耦合分析
- 热泳