抽象的
背景
具有巨大基因组和密切相关的种类的种类的强大文献可以建立用于调查这些基因组胶像的起源和演变的进化框架。巴黎粳稻(黑莓科)拥有迄今已在真核生物中被证实的最大基因组;然而,其系统发育地位仍未确定。因此,基因组的进化史巨幅展开p .粳稻仍然知之甚少。
结果
我们使用下一代测序技术来生成完整的质体p .粳稻那P. Verticillata.那Trillium govanianum.那ypsilandra thibetica和Y. Yunnanensis..与发表的塑料一起,印度血管生成的塑料,在香料群岛和丙肾上腺发育中巴黎,并计算其发散时间。结果表明,已存在的祖先基因组扩增巴黎和trillium.发病率约为59.16至38.21米。姐妹关系p .粳稻和部分Euthyra它们在渐新世/中新世(20米亚)的过渡时期发生了分化,当时日本群岛从亚洲大陆分离出来。
结论
基因组大小的扩大在最近的共同祖先巴黎和trillium.很可能是一个持续了大约2000万年的渐进过程。的散度p .粳稻(部分Kinugasa)其他具有厚实的根茎的分类群可能被亚洲大陆的日本岛屿孤立所引发的。这种长期分离,因为寡烯/中间烯边界,在基因组胶像的形成和演变中起着重要作用粳稻。此外,我们的结果支持分类处理巴黎作为一个属而不是把它分成三个属,但不支持承认T. Govanianum.作为单独的属特罗里尼亚.
背景
Agiosperms在基因组大小上表现出极端的多样性,定义为单倍体核DNA量,在最小和最大的基因组之间变化约2400倍[1那2那3.那4.].尽管在心血管中的基因组大小的分布朝向小基因组偏斜(平均值为1c = 5.7 gb,并且模态值为1c = 0.6 gb)[4.],到目前为止,已有5个基因组大小为1C > 100gb的物种被记录在案。这些植物属于单子叶植物黑莓科(一种巴黎,两种物种trillium.),百合科(一种贝母)和eudicot家族粘度酵e(一种物种槲寄生)[5.那6.那7.那8.那9.,表明基因组巨人症可能仅在少数世系中独立起源和进化[1那10].
由于对非常大或非常小的基因组进行测序的技术挑战,对驱动基因组巨人症形成的机制的了解仍然有限[9.].具有巨基因组的物种及其密切相关类群之间高分辨率和良好支持的系统发育关系可以构建进化框架,以阐明这些基因组巨基因组的进化史[9.那10那11那12].不幸的是,为属的强大的系统发作巴黎那trillium.和槲寄生,其中包括基因组巨量,仍然难以捉摸[13那14那15这阻碍了我们对巨型基因组形成和进化机制的理解。
虽然基因组的大小Polychaos dubia,一种单细胞真核生物,估计有超过670 Gb [16],这种测量被认为是不可靠和不准确的[4.].迄今为止,已观察到真核生物中确诊的最大基因组巴黎粳稻(法语。等干腊肠)。法兰治(姓氏)(也称为Kinugasa japonica(Franch。等等)Tatew.etSutô。),1C值为148.88 GB [1那17].这种植物是一种多年生草本植物,属于单子叶科黑莓科部落黑莓科[18那19],在日本中央和北部北部的中央和北部20.那21].细胞学研究表明p .粳稻是染色体数目为2n = 8x = 40的八倍体[1那22那23].由于其独特的特征,如艳丽的白色萼片和八倍体染色体计数,p .粳稻已经历来陷入了属于属巴黎(部分Kinugasa)[21那23]或被视为单调型属Kinugasa[20.那24].此外,进化关系p .粳稻在最近的基于单个或多基因座DNA序列的近期分析中,有相关的分类群仍然存在争议。使用塑体的分析:区域显示p .粳稻是属的姐妹吗trillium.[25].塑体的组合分析:和matK核透露了它的DNA地区p .粳稻与属有密切关系吗Daiswa(=巴黎部分Euthyra)[13那26].相比之下,两项基于质体的独立研究psba-trn.那trnL-F及核ITS序列资料[27]和五个塑性区域的组合(atpB那:那matK那NDHF.和trnL-F)[28),解决p .粳稻作为姐妹小组的部分巴黎.这些冲突表明关系之间的关系p .粳稻相关类群有待进一步研究。
使用过少的DNA序列的系统发育分析可能导致不同序列区域之间的冲突[29那30.];在这种情况下,不可能重建一个健壮和可靠的系统发育,特别是在低分类学水平[31].完整的质体DNA序列可以为植物复杂进化关系的分析提供有价值的信息[32那33那34].随着下一代DNA测序技术的出现,质体近年来被广泛应用于重建一些系统发育困难的植物类群的健壮系统发育[31那35那36那37];这些病例表明,整个质体测序可能为阐明两者之间的关系提供新的证据p .粳稻和盟军的分类群。尽管潜在的遗产DNA基因座的分析可能无法证明物种的完整历史,但基于完全的基于塑性的系统发育可以给我们一些有价值的信息来阐明基因组胶质组织的母体起源和演变粳稻。
在目前的研究中,我们使用了低覆盖基因组霰弹枪测序[38产生的质体p .粳稻那P. Verticillata.那Trillium govanianum.那ypsilandra thibetica和Y. Yunnanensis.通过与其他已发表的质体结构和基因含量的比较,推测其分子进化过程。然后,我们重建了该家族内部的进化关系,以研究该家族的系统发育位置p .粳稻.最后,我们确定了分歧的日期p .粳稻为了深入了解最大的真核生物基因组持有者的进化史。
结果
plastid基因组特征
质体系的p .粳稻那P. Verticillata.那T. Govanianum.那Y. Thibetica.和Y. Yunnanensis.完全组装。每个塑料的测序覆盖范围从283×到1086×(附加文件2:表S2)。基因内容(附加文件3.:表S3,附加文件4.:表S4,附加文件5.:表S5,附加文件6.:S6,附加文件7.:S7)和安排(附加文件8.:图S1,附加文件9.:图S2,附加文件10:图S3,附加文件11:图S4,附加文件12:图S5)在五个塑料上几乎相同。这些新产生的塑料的尺寸范围为155,957至158,806bp,其典型的四胞胎结构具有由LSC(83,635-85,301bp)和SSC(18,337-19,586bp)区分离的IRS(26,805-27,602bp)。(桌子1).除了TRND-GUC已从塑料中删除y thibitica,其他质体编码114个独特基因,包括80个蛋白编码基因、30个tRNA基因和4个rRNA基因(见表)2).
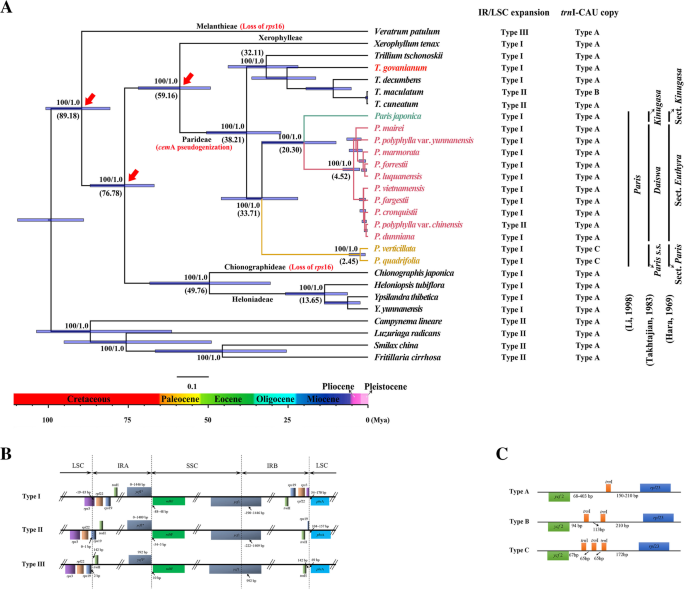
虽然基因的含量和排列几乎相同,但假殖化和基因丢失也时有发生。由于在编码区存在几个内部终止密码子,cemA被确定为伪基因巴黎和trillium.质体系(无花果。1一种)。此外,失去了第一个外显子的石头剪刀16个基因被发现在质体中Veratrum patulum和Chionographis粳稻(无花果。1a).将IR区域扩展为ycf1基因在黑莓科的所有细胞体中都存在相同的IR/SSC边界,但它们的IR/LSC连接存在显著差异。在黑莓科和外群类群中观察到三种类型的IR/LSC边界(图。1b). IR膨胀成TRNH-RPS19基因间间隔(III型)仅见于V. Patulum.,而IR的扩展为RPS19(II型)发生在延龄草cuneatum那天然珠,和巴黎polyphylla.var。中国人以及除了小组的分类群中。相比之下,其特征在于IR / LSC边界落入rps3,在其余类群中观察到I型(图。1一种)。
之间的代际区域的长度rpl23岁,ycf2在黑莓科的质体间表现出很大的差异,其中的单副本、重复和三副本的环境观察I-CAU(图)。1C)。三倍的环境观察到I-CAU(C型)P. Quadrifolia和P. Verticillata.(部分巴黎),而重复的环境I-CAU(B型)被发现T. Maculatum..单份的的环境I-CAU (A型)在其他细胞体中也有发现(图)。1一种)。
系统核发科学分析和分歧估计
从ML和BI分析的树拓扑是相同的。各质体之间的系统发育关系如图所示。1一种。由Zomlefer认识到的五个支点(Bs = 100%,pp = 1),对应于五个部落(Melanthieae,ChionographiDeae,Heloniadeae,Xerophyleae和Parideae)对应18],被恢复了。Tribe Melanthieae是姐姐到剩下的香菇(Bs = 100%,pp = 1)。ChionographIdeae和Heloniadeae之间以及Xerophylleae和Paridea之间的姐妹关系得到了完全支持(BS = 100%,PP = 1)。从我们的系统核算科学分析中的部落内关系与基于核核糖体的先前研究的关系一致的环境L -的环境F地区[18];plastid DNA序列的组合[28那39];和塑性基因组测序[15].
在部落中,姐姐之间的关系trillium.和巴黎已回收(Bs = 100%,pp = 1)。这巴黎种进一步分为三个完全支持谱系(BS = 100%, PP =1),分别对应三个狭义定义属(巴黎轮上。那Kinugas.和Daiswa,分别)Takhtajan [24或三个部分(部分巴黎, 部分Kinugasa和部分Euthyra,分别)受Hara限制[23].其中,p .粳稻(部分Kinugasa)姐姐是该科的Euthyra(bs = 100%,pp = 1)和该部分巴黎妹妹是由节组成的枝吗Kinugasa和部分Euthyra.这里获得的交叉关系与前一项研究的交叉关系一致[40].
黑蕨科的三个校准点(图。1A)先前研究的建议[41]用来限制基于质体的系统发生树。结果表明,Parideae部落最近的共同祖先(MRCA)约为59.16 Mya (95% HPD: 73.01-49.11 Mya)及其属巴黎和trillium.(95% HPD: 52.17-26.84米亚)。在属巴黎,该段的MRCA巴黎约为33.71 mya(95%HPD:47.47-22.03 mya),以及单调部分之间的分歧Kinugasa(p .粳稻)和部分Euthyra约20.30 Mya (95% HPD: 34.64-9.96 Mya)。
讨论
具有巨大基因组的物种和相关类群的健壮系统发育可以建立进化框架,以阐明这些基因组巨体的起源和进化[9.那10那11那12].先前的研究[13那25那26那27揭示了很难重建高分辨率和有良好支持的系统发育关系p .粳稻美国是最大的真核生物基因组持有者,而它的同族类群基于太少的DNA序列区域。在这项研究中,我们对整个质体进行了测序p .粳稻, 也P. Verticillata.那Trillium govanianum.那ypsilandra thibetica和Y. Yunnanensis..再加上印度可用的塑料,我们对整个塑料进行了比较和系统发育分析,以阐明进化关系p .粳稻与其密切相关的类群。这项研究为我们提供了一些关于基因组巨兽起源和进化的新信息粳稻。
质体系比较
失去了第一个外显子RPS16在灵感独特的部落调味料和ChionographIdeae中观察到。此外,失去了的环境D-GUC只在y thibitica。这些结果支持扣除某些塑性基因的丧失可能独立地发生在Anuiaperms的进化史上[32那42].因此,某些塑性基因的丧失可能不提供相关的进化信息。然而,在任何调味料血糖中没有观察到基因损失和基因迁移,这意味着家庭中的基因含量和塑性结构是高度保守的。
以往的研究表明,蛋白质编码基因cemA在几种非光合寄生厂丢失了[43那44那45].据我们所知,这种基因在光合肌肤营养上的假期化才能在密切相关的属中检测到巴黎和trillium.(无花果。1).虽然其功能尚不清楚[46],这种突变可能提供一个分子突触形态来识别Parideae部落[47].此外,正如先前一项研究所建议的那样[15,谱系特异性的三倍性TRNI-CAU.在P. Quadrifolia和P. Verticillata.可以作为分子突触结构来界定这部分吗巴黎(无花果。1一种)。
单子叶塑料的IR / LSC边界通常扩展到trnH-RPS19基因簇和红外展开重复trnH基因,它不同于那些非单孢被子植物[47].在这项研究中,我们确定了调味料内的三种类型的IR / LSC扩展;其中,II型和III展示了典型的单码IR / LSC结,而I类型的IR / LSC结合在一起rps3.尽管IR/LSC扩展到RPS19-rpl22代际垫片或者rpl22已经在一些单子目中被观察到,如天冬酰胺目、天门冬酰胺目、天门冬酰胺目和天门冬酰胺目[48那49那50, IR/LSC逐步扩展为rps3迄今为止只在黑莓科中发现。三种类型的IR/LSC边界在树状拓扑中的系统发育分布表明,III型可以作为黑莓科的祖先状态,与IR区域扩展为rps3发生在派生部落,如chionographidae, Heloniadeae, Xerophylleae和Parideae(图。1一种)。此外,观察II型II / LSC结t . cuneatum那天然珠,和巴黎polyphylla.var。中国人可能是由于IR区域从rps3来RPS19.
系统发育推论
我们的系统染色组织分析在调味膜内回收了五种良好的支持良好的谱系(Bs = 100%,pp = 1),其对应于由Zomlefer认识的五个部落[18].这项研究中恢复的进化关系与之前的研究一致[18那28那40那51]但具有更高的分支支持(BS = 100%,PP = 1)。结果进一步证明整个塑性基因组测序可以改善某种谱系的系统发育分辨率[33那34].
我们扩大了对Parideae的胞浆体的采样,为重建该群体中强大的属内系统发育提供了机会。蝶亚科的基本分化大约发生在38.21 Mya,形成两个完全支持的谱系(巴黎和trillium.)在树拓扑中(BS = 100%,PP = 1)。两种属股同股同步,包括在阀杆顶点,茎顶进呈孤零花的茎尖和染色体基数的单螺纹叶片N = 5 [16].在进化枝巴黎,三节(节巴黎, 部分Kinugasa,部分Euthyra)由Hara概述[23]以及三个狭义定义的属巴黎轮上。那大和和Kinugasatakhtajan [24在ML和BI分析中均为支持强的单系分支(BS = 100%, PP = 1)。考虑到那个物种巴黎Clade分享了鲜花和叶子的形态突录,与颗粒的条件相比,4-至15个优秀trillium.[27,我们相应地倾向于接受的分类处理巴黎作为单一属[21那23]而不是三个分开的属于[24].
因为之前的研究没有包括p .粳稻在系统发育分析中,它与其他物种的进化关系巴黎物种仍未得到解决[15].ML和BI分析均表明p .粳稻(部分Kinugasa)是该组的姊妹Euthyra,这与塑料的分析一致:那matK和TRNL-TRNF.地区[13那26那40].但是,我们数据恢复的关系在很大程度上不同于塑植的组合分析结果公安局一种的环境H和的环境L.-F和核ITS序列[27]和塑料atpB那:那matK那NDHF.和trnL-F地区[28].值得注意的是,支持良好的姐妹关系p .粳稻和部分Euthyra(BS = 100%,PP = 1)在本研究中恢复,可以通过它们共享的形态兴趣形态学,例如厚的根茎和角卵巢,与长而细长的根茎和圆形的卵巢物种相比部分巴黎(无花果。2).此外,该种不同寻常的形态特征(即华丽的白色萼片和八倍体染色体数目)证明了其分类处理的合理性p .粳稻作为属的独特部分巴黎哈拉[23].
我们的数据不仅恢复了巴黎同时也为澄清关于物种系统发育地位的争论提供了证据T. Govanianum.产于喜马拉雅山脉。虽然T. Govanianum.有一个颗粒花,像那些一样叶子trillium.物种,它分享形态特征,如狭窄的萼片和丝状花瓣,与巴黎物种(图。3.).因此,T. Govanianum.被认为是单独的属特罗里尼亚[13那52].但是,ML也不是双树拓扑分开T. Govanianum.从trillium.物种,但将它们分组成一个受到良好的疏水板(BS = 100%,PP = 1)。值得注意的是,基于五种塑性DNA区域,在系统发育分析中显示了类似的发现,其具有更广泛的香料群体的分类素[28].综上所述,研究结果表明T. Govanianum.应该留在属中trillium.不承认它的属特罗里尼亚.
深入了解基因组巨人症的起源和进化巴黎粳稻
本研究重建的健壮系统发育为深入了解中国基因组巨人症的起源和进化提供了理论依据p .粳稻.Musthiseae的大多数物种具有小或非常小的基因组,而大型或巨大的基因组在两个属中被专门发现:巴黎和trillium.[40].性状重建表明,旱叶科和旱叶科分化后基因组大小可能增加4倍以上,而旱叶科和旱叶科分化前基因组大小可能增加4倍以上巴黎和trillium.[40].分子约定表明,PariCeae的茎年龄和皇冠年龄分别为约59.16 mya和38.21 mya,表明大规模的基因组扩张将持续长期约有2000万年。在此期间,祖先的祖先基因组巴黎和trillium.可能会逐渐扩大,这意味着像之前的一项研究提出的那样,蝶虱科的基因组大小可能是在数千万年的缓慢积累[40].
系统核糖分析表明该部分巴黎包括姐妹枝吗p .粳稻(部分Kinugasa)和部分Euthyra.这些关系表明,一个巨大基因组的形成p .粳稻最有可能发生在板块分化之后Euthyra和p .粳稻.除了p .粳稻,两个物种(T。×hagae和T.菱形)在Genome尺寸的情况下,在属中发现了100 GBtrillium.[5.那6.那9.].由于我们未获得这两种植物的样本,因此它们内部的系统发育位置trillium.尚不清楚。然而,进化关系p .粳稻与相关类群的研究发现,巨型基因组的形成p .粳稻和trillium.物种可能是独立的事件。
的质体的结合p .粳稻和部分Euthyra围绕寡烯/中茂的过渡(20.30 Mya,95%HPD:34.64-9.96 Mya),当时日本海的开放将日本岛屿从亚洲大陆分开[53].虽然p .粳稻和部分Euthyra是密切相关的,它们占据着不同的分布:p .粳稻是日本特有的,而物种来自该地区Euthyra主要分布于中国亚热带和喜马拉雅山脉[23].因此,散度p .粳稻和部分Euthyra可能被日本岛屿从亚洲大陆的孤立所引发的。
值得注意的是,基因组大小p .粳稻是否比那些属于剖面的物种大约大2-3倍Euthyra[40].一系列证据证明,植物基因组大小的变化是在选择性约束下的,并不是通过纯粹的漂移过程进化的[54那55那56].因此,基因组大小可能与物种的环境和生态严格相关[57].一般来说,基因组较大的植物具有一些共同的形态特征,如大的身体和气孔大小[58].由于气孔大的植物易受干旱影响,只有那些发生在潮湿生境的物种才能维持更大的基因组[56那58].与季伏和喜马拉雅州的明显降水季节性为特征的季风气候相比[59那60]、日本列岛的海洋性气候[61]会产生相对更潮湿的栖息地,促进了进化p .粳稻对基因组巨大。
结论
最大的真核生物基因组持有者,p .粳稻,通过其完全塑性DNA序列的比较和系统发育分析来研究其密切相关的分类群。塑料塑料中的比较分析显示,它们的结构和基因含量高度保守,并为Parideae的一些谱系提供了分子突录。系统染色的分析和分子约会回收了进化骨干巴黎从而阐明了的系统发育地位p .粳稻.树拓扑和分子约会表明,现存的祖先基因组的扩张巴黎和trillium.可能是持续约2000万年的渐进过程;分歧p .粳稻和部分Euthyra可能已被日本海的开放引发,这些海上围绕寡烯/中新世(20.30 Mya)的过渡,将日本岛屿与亚洲大陆分开。这种长期分离将在基因组胶像中的形成和演变中发挥着重要作用p .粳稻.的系统发育地位p .粳稻意味着巨大的基因组巴黎和trillium.可能是独立形成和进化的,即使这两个属是密切相关的。此外,我们的系统基因组分析有力地支持了植物的分类处理巴黎作为一个属,而不是将它划分为三个属,但不支持识别T. Govanianum.作为单独的属特罗里尼亚.
方法
植物材料和猎枪测序
叶组织p .粳稻那P. Verticillata.那T. Govanianum.那Y. Thibetica.和Y. Yunnanensis.在该领域收集,用硅胶干燥(每种物种一个单独).经纪云恒博士鉴定,保存于中国科学院昆明植物研究所植物标本馆;凭证信息见表3..提取基因组DNA〜20毫克的叶子组织,使用改良的CTAB法[62].约5 μg纯化的基因组DNA经超声剪切。使用TruSeq DNA样品准备试剂盒(Illumina, Inc., USA),根据制造商的协议制备平均插入尺寸为350 bp的配对端文库。Shotgun测序是在Illumina HiSeq 2000平台上进行的。
质体组装、注释、比较
原始Illumina读取经过NGS QC工具包过滤[63]删除适配器和低质量读数。Jin等人开发的管道。[64]用于De Novo Plastome组装。干净的阅读巴黎物种,T. Govaniamum.和Ypsilandra种被标记到参考质体上P. Quadrifolia(GenBank加入:KX784051),T. Tschonoskii.(Genbank入库:KR780076)和Heloniopsis Tubiflora(Genbank登录:KM078036)使用Bowtie v2.2.6软件[65]的默认参数和预设选项。SPAdes v3.10.1将所有类质体reads组装成contigs [66[K.-mer定义为75,85,95和105.自定义Python脚本[64]可以使用爆炸和内置库来搜索塑体样谱,用于将已验证的体积连接到SHADE V3.10.1中的塑料中[66],使用默认参数。重新组装的结果用绷带v.8.0进行可视化编辑[67].
所得的质体由Dual Organellar Genome Annotator数据库注释[68].使用佐埃v10.3手动打印注释[69].人工检查蛋白质编码基因的起始和终止密码子。所有鉴定的tRNA均经tRNAscan-SE v1.21验证[70],预设参数。通过参考在线数据库cpbase来确定塑性基因的功能分类(http://rocaplab.ocean.washington.edu/old_website/tools/cpbase).利用Organellar Genome DRAW程序构建了质体图谱[71].
质体的一般特征,如结构重排、基因丢失/假殖化、基因复制和IR区域的扩张/收缩等,在以前的研究中提供了进化信息[15那32那72].因此,我们在调味料素塑料中进行了这些特征的比较。基因含量和布置被可视化并与Mummer 3.0程序进行比较[73].使用geneiv10.2.3比较每个质体中LSC、IR和SSC区域的边界[69].
系统核发科学分析
检查系统发育位置p .粳稻,在系统孕组学分析中包括24种代表族聚糖中的宽系统发生多样性的塑料(附加档案1:表S1)。塑料Campynema线性那贝母cirrhosa那Luzuriaga Radicans.和菝葜中国都是用来给树生根的。其中5个细胞体是在本研究中新生成的(见表)3.),并且从NCBI数据库下载REST塑料(附加文件1:表格。
使用MAFFT对完整的质体DNA序列进行比对[74集成在geneiv .10.2.3 [69],如果需要,手动调整。用标准的最大似然(ml)和贝叶斯推理(BI)方法进行系统染料组织分析。使用RAXML-HPC Blackbox V8.1.24进行M1分析[75在GTR-GAMMA模型下进行1000次快速自举(BS)。搜索最佳评分ML树和快速引导在一次运行中执行。BI分析的最佳核苷酸序列替代模型的选择使用Modeltest v3.7确定[51]与赤池资讯准则[76].BI采用MRBAYES v.3.1.2进行[77]使用所选模型(TVM + I + G)。两个独立的并行马尔可夫链蒙特卡罗(MCMC)用树采样每100多代采样,丢弃25%,被抛弃为烧伤。当分流频率的平均标准偏差<0.01时,考虑了达到的稳定性。后验概率值(PP)由剩余的0.75亿树木确定。
分子的约会
迄今为止,没有针对Family Melanthiaceae和其近亲鉴定化石。以前的一项研究,在单焦点和高级植物植物的主要碎片上校准了17个化石[41]的冠龄约为84.8米,而Parideae- xerophyllidae和chionographidae - heloniadeae分支分化约为74米,Parideae和xerophyllidae分支分化约为52.3米。我们利用这些事件校准系统发生树(图。1一种)。
使用集成在PAML程序包中的MCMCTree V4.9C程序进行分子约会[78].采用ML树拓扑来估计节点的发散时间。以独立率分子钟为时钟模型,以HKY85为取代模型。根龄设为小于100 Mya。用最小年龄为84.8米亚的方法校正了黑莓科的分化度。拟合菌科-旱叶菌科和雪蛾科的分枝最小年龄为74 Mya,拟合菌科和旱叶菌科的分枝最小年龄为52.3 Mya。其他参数被定义为默认值。MCMC链运行了10,100,000次迭代。前10万次迭代被丢弃为老化,每10次迭代对树进行采样,直到收集到100万次样本。
数据和材料的可用性
完整的cp基因组序列p .粳稻那P. Verticillata.那Trillium govanianum.那ypsilandra thibetica和Y. Yunnanensis.在加入号码MF796668-MF796672下可在Genbank提供。分析中使用的数据包括在文章和附加文件中。
缩写
- 双:
-
贝叶斯推理
- 英国石油公司:
-
基对
- BS:
-
引导
- CTAB:
-
溴化十六烷基trimethylammonium
- HPD:
-
后验密度最高
- 红外:
-
反向重复
- 其:
-
核核糖瘤DNA的内部转录间隔物
- 密度:
-
马尔可夫链蒙特卡洛
- ML:
-
最大似然
- 米娅:
-
百万年前
- NCBI:
-
国家生物技术信息中心
- 核糖体rna:
-
核糖体RNA
- SSC:
-
小单拷贝
- tRNA:
-
转移核糖核酸
- LSC:
-
大单副本
参考
- 1.
Pellicer J, Fay MF, Leitch IJ。最大的真核生物基因组?acta photonica sinica, 2010; 40(4): 481 - 486。
- 2.
Pellicer J,Kelly LJ,Magdalena C,Leitch IJ。洞察早期分歧性患者血米腹部腹膜血管(睡莲)的基因组大小和染色体演变的洞察。基因组。2013; 56:437-49。
- 3.
转座元件、表观遗传学和基因组进化。科学。2012;338:758 - 66。
- 4.
Dodsworth S,Leitch Ar,Leitch IJ。Angiospers的基因组大小分集及其对基因空间的影响。CurrOpin Genet Dev。2015; 35:73-8。https://doi.org/10.1016/j.gde.2015.10.006.
- 5.
DNA c值的系统发育分析提供了开花植物祖先基因组较小的证据。Ann Bot. 1998;82(增刊A): 85-94。
- 6.
Leitch IJ,Beaulieu JM,Chase MW,Leitch Ar,Fay MF。单焦度的基因组大小动态和演化。j bot。2010; 2010年:862516。https://doi.org/10.1155/2010/862516.
- 7.
Zonneveld BJM,Leitch IJ,Bennett MD。第一个核DNA含量超过300个贪眼。Ann Bot。2005; 96:229-44。https://doi.org/10.1093/aob/mci170.
- 8.
陆生植物基因组大小的多样性与进化。在:Leitch IJ, Greilhuber J, Dolezel J, Wendel JE,编辑。植物基因组多样性2。奥地利维也纳:斯普林格出版社;2013.p . 307 - 322。
- 9.
Hidalgo O,Pellicer J,Christenhusz M,Schneider H,Leitch Ar,Leitch IJ。是否存在基因组大小的上限?趋势植物SCI。2017; 22:567-72。https://doi.org/10.1016/j.tplants.2017.04.005.
- 10.
马建新,王志强,王志强,等。植物基因组大小变化的研究进展。安机器人。2005;95:127-32。https://doi.org/10.1093/aob/mci008..
- 11.
混合交配群体的转座元件数。麝猫杂志2001;77:261 - 75。https://doi.org/10.1017/S0016672301005067.
- 12.
温德尔JF, Cronn RC, Johnston JS, Price HJ。植物基因组中的盛宴与饥荒。遗传。2002;115:37-47。https://doi.org/10.1023/a:1016020030189..
- 13.
农民SB, Schilling EE。基于形态学和分子数据的Trilliaceae系统发育分析。系统机器人。2002;27:674 - 92。https://doi.org/10.1043/0363-6445-27.4.674.
- 14.
默里房车,尼克伦特区。槲寄生属的分子系统发育槲寄生.Trans Illin State academy Sci. 2004; 97:44-5。
- 15.
黄Y,李X,杨Z,杨C,阳j,ji y。完全叶绿体基因组序列的分析改善了系统发育分辨率巴黎(Melanthiaceae)。植物学报2016;7:1797。https://doi.org/10.3389/fpls.2016.01797.
- 16.
Gregory Tr,Hebert Pd。DNA含量的调节:近似的原因和最终后果。Genome Res。1999; 9:317-24。
- 17.
Hidalgo O,Pellicer J,Christenhusz MJM,Schneider H,Leitch IJ。扫须粉蕨家族(Psilotaceae)的基因组胶像:tmesipteris obliqua挑战纪录保持者巴黎粳稻.acta photonica sinica, 2017b;https://doi.org/10.1093/botlinnean/box003.
- 18.
Zomlefer WB, Williams NH, Whitten WM, Judd WS。属的界限和关系的部落黑百合科(百合目,黑百合科),重点Zigadenus.:来自它的证据trnL-F序列数据。我是J机器人。2001; 88:1657-69。https://doi.org/10.2307/3558411.
- 19.
被子植物系统学。开花植物目和科被子植物系统发育分类的最新进展:APG IV. Bot J Linn Soc. 2016; 181:1-20。
- 20.
竹胁木,Sutô T.论金龟新属。Sapporo Not Hist Soc, 1935; 14:34-6。
- 21.
李H.属的系统发育巴黎L.在:李H,编辑。巴黎北京属的属:科学出版社;1998. p。8-65。
- 22.
哈加氏体Kinugasa japonica特别提到它的起源和行为。Cytologia。1938;8:137-41。https://doi.org/10.1508/cytologia.8.137.
- 23.
哈拉h变奏曲巴黎polyphylla.以其他亚洲种为参照。东京科学大学第三节。1969; 10:141 - 80。
- 24.
《中华民国史记》的修订Daiswa(蛛丝)。布里塔尼亚。1983; 35:255-70。https://doi.org/10.2307/2806025.
- 25.
陈志强,陈志强,陈志强,等。三叶藓科植物的分子系统学研究理智lato从中推断出来RBC.L序列数据。中国科学院地理科学与资源研究所。https://doi.org/10.1006/mpev.1995.1018.
- 26.
奥斯罗斯SK,川南S. Trilliaceae II的分子系统。系统发育分析trillium.它的盟友使用一系列RBC.土地垫cpDNA的K基因和18S-26S nrDNA的转录间隔。植物物种学报1999;14:75-94。https://doi.org/10.1046/j.1442-1984.1999.00009.x.
- 27.
吉y,fritcech pw,李h,肖t,周z.系统发育和分类巴黎(调味料)从DNA序列数据推断。Ann Bot。2006; 98:245-56。https://doi.org/10.1093/aob/mcl095..
- 28.
李志刚,李志刚,李志刚。百合科植物叶绿体DNA序列的分子系统学研究进展。Bot J Linn Soc. 2016; 181:567-84。https://doi.org/10.1111/boj.12405..
- 29.
更多的基因还是更多的类群?基因数和分类单元数对系统发育准确性的相对贡献。Mol Biol Evol. 2005; 22:1337-44。https://doi.org/10.1093/molbev/msi121.
- 30.
作者简介:Philippe H, Brinkmann H, Lavrov DV, Littlewood DTJ, Manuel M, Wörheide G, et al.;解决困难的系统发育问题:为什么更多的序列是不够的。公共科学图书馆杂志。2011;9:e1000602。https://doi.org/10.1371/journal.pbio.1000602.
- 31。
利用叶绿体基因组的大规模平行测序技术提高系统发育分辨率。BMC医学杂志。2009;7:84。https://doi.org/10.1186/1741-7007-7-84.
- 32.
Jansen RK, Cai Z, Raubeson LA, Daniell H, Leebens-Mack J, Müller KF, guisinger - belllian M,等。通过对64个质体基因组中的81个基因的分析,揭示了被子植物的亲缘关系,并确定了基因组规模的进化模式。美国国家科学研究院2007;104:19369-74。https://doi.org/10.1073/pnas.0709121104.
- 33.
利用质体基因组规模数据分析基部被子植物之间的神秘关系。中国科学院院刊2007;104:19363-8。https://doi.org/10.1073/pnas.0708072104.
- 34.
Moore MJ, Soltis PS, Bell CD, Burleigh JG, Soltis DE. 83个质体基因的系统发育分析进一步解决了eudicots的早期分化。美国国家科学研究院2010;107:4623-8。https://doi.org/10.1073/pnas.0907801107.
- 35.
Barrett CF,Specht CD,Leebens-Mack J,Stevenson DW,Zomlefer WB,Davis Ji。解决古代辐射:可以完全塑性基因套装阐明热带刺刀(Zingiberales)之间的深层关系吗?Ann Bot。2014; 113:119-33。https://doi.org/10.1093/aob/mct264.
- 36.
Stull GW,Dunod SR,Soltis De,Soltis PS。用塑料级数据集分辨钴基氏素的基础鳞片系统和纤维酸的周边。我是J机器人。2015; 102:1794-813。https://doi.org/10.3732/ajb.1500298.
- 37.
Attigala L, Wysocki WP, Duvall MR, Clark LG。基于胞浆体系统发育分析的青竹科系统发育及形态进化研究。Mol Phylogen Evol. 2016;101: 111 - 121。doi: org/https://doi.org/10.1016/j.ympev.2016.05.008.
- 38.
Straub SC, Parks M, Weitemier K, Fishbein M, Cronn RC, Liston A.导航基因组冰山的尖端:植物系统学的下一代测序。Am J Bot. 2012; 99:349-64。https://doi.org/10.3732/ajb.1100335.
- 39.
Kim JS,Hong JK,Chase MW,Fay MF,Kim JH。基于四种塑体基因座的分子系统肝脏分子发育分析的单子宫秩序智慧的家族关系:matK那:那atpB和atpF-H.Bot J Linn Soc. 2013; 172:5-21。https://doi.org/10.1111/boj.12039.
- 40。
Pellicer J, Kelly LJ, Leitch IJ, Zomlefer WB, Fay MF。侏儒和巨人的宇宙:单花木科黑莓科的基因组大小和染色体进化。新植醇。2014;201:1484 - 97。
- 41。
马奎斯I,林VKY,戈麦斯MS,伊尔斯WJD等。单子叶百合目的系统发育学和历史生物地理学:从澳大利亚到南极洲。支序分类学。2016;32:581 - 605。https://doi.org/10.1111/cla.12153.
- 42.
等。在被子植物进化过程中,叶绿体DNA中有许多平行的infA损失,并有多个独立的转移到细胞核。植物细胞。2001;13:645-58。https://doi.org/10.2307/3871412.
- 43.
Wolfe KH, modern CW, Palmer JD。非光合寄生植物最小质体基因组的功能和进化。美国国家科学学院1992;89:10648-52。https://doi.org/10.1073/pnas.89.22.10648.
- 44.
Logacheva MD,Schelkunov M,Penin AA。肌营养植物兰花奈西奈斯 - AVIS中塑性基因组的测序与分析。基因组Biol Evol。2011; 3:1296-303。https://doi.org/10.1093/gbe/evr102.
- 45.
王志强,王志强,王志强,等。植物非光合作用过程中进化速率变化的机制模型。美国国立科学院学报2016;113:9045-50。https://doi.org/10.1073/pnas.1607576113.
- 46.
Willey DL,Gray Jc。编码atuptationhaem结合多肽的开放阅读框是用豌豆叶绿体基因用于呼吸中心f的。植物mol biol。1990; 15:57-356。
- 47.
做HDK, Kim JS, Kim JH。全叶绿体基因组中trnI_CAU三倍增殖事件巴黎verticillataM. Bieb。(调味料,莱利斯)。基因组Biol Evol。2014; 6:1699-706。https://doi.org/10.1093/gbe/evu138.
- 48.
王RJ,Cheng Cl,Chanc Cc,Wu Cl,Su TM,Chaw Sm。单焦炭叶绿体基因组中倒置重复的单拷贝交配的动态与演化。BMC EVOL BIOL。2008; 8:36。https://doi.org/10.1186/1471-2148-8-36.
- 49.
朱安,郭伟,顾磊杰,范伟,Mower JP。质体反向重复的进化动力学:扩张、收缩和损失对替代率的影响。新植醇。2016;209:1747-56。https://doi.org/10.1111/nph.13743.
- 50.
Lopes广告,Pacheco Tg,Nimz T,Vieira Ldn,Guerra MP,Nodari Ro,等。金刚鹦鹉棕榈[acrocomia Aculeata,(JACQ。)LODD的完整塑料。EX MART。]并广泛分析羊皮酸盐塑性基因的演变。Planta。2018; 247:1011-30。https://doi.org/10.1007/S00425-018-2841-X..
- 51.
波萨达,克兰德尔MODELTEST: DNA替代模型测试。生物信息学。1998;14:817-8。https://doi.org/10.1093/bioinformatics/14.9.817.
- 52。
哈拉,斯登WT,威廉姆斯LHJ。尼泊尔开花植物的一种枚举。分类单元。1978;第三:384。
- 53。
超大陆的历史及其与日本岛屿起源的关系。J Geodyn。2011;120:100-14。https://doi.org/10.5026/jgeography.120.100.
- 54。
康敏,陶杰,王军,任超,齐青,向清云,等。中国喀斯特特有植物区系的适应和非适应基因组大小演变。新植醇。2014;202:1371 - 81。https://doi.org/10.1111/nph.12726.
- 55。
飞行的代谢“引擎”驱动鸟类基因组的缩小。中国生物医学工程学报。2014;281:20132780。https://doi.org/10.1098/rspb.2013.2780.
- 56.
李志刚,李志刚。百合科植物的大基因组限制假说:植物性状、生境和气候季节性。新植醇。2016;210:709-16。https://doi.org/10.1111/3ph.13769.
- 57.
Knight Ca,Molinari N,Petrov D.大基因组约束假设:进化,生态和表型。Ann Bot。2005; 95:177-90。https://doi.org/10.1093/aob/mci011.
- 58.
骑士CA,Beaulieu JM。基因组大小通过表型空间缩放。Ann Bot。2008; 101:759-66。https://doi.org/10.1093/aob/mcm321.
- 59.
万姆,李交流,克利夫特PD,斯图特JBW。东亚季风的发展:南海北部20万自南海的矿物学和沉积学纪录。Palaeogeogr Palaeoclimatol帕拉西奥。2007; 254:561-82。https://doi.org/10.1016/j.palaeo.2007.07.009.
- 60。
Jacques FM,Guo Sx,Su T,xing Yw,Huang Yj,Liu Ys等。西南部季风季风气候定量重构 - 以云南林阳植物为例。Palaeogeogr Palaeoclimatol帕拉西奥。2011; 304:318-27。
- 61。
Fukutome S,Frei C,Schärc。SST对日本夏季降水际变率的影响。J Meteorol Soc Jap。2003; 81:1435-56。
- 62。
Doyle JJ,Doyle JL。少量新鲜叶组织的快速DNA隔离程序。Phytochem Buart。1987; 19:11-5。
- 63。
Jain M. NGS QC工具包:用于下一代测序数据质量控制的工具包。《公共科学图书馆•综合》。2012;7:e30619。https://doi.org/10.1371/journal.pone.0030619.
- 64。
金俊杰,于文斌,杨建斌,宋勇,易天生,李德志。GetOrganelle:一个简单和快速的管道从头组装一个完整的环状叶绿体基因组使用基因组略读数据。bioRxiv。2018;256479.doi:https://doi.org/10.1101/256479
- 65。
Bankevich A,Nurk S,Antipov D,Gurevich AA,Dvorkin M,Kulikov As等。黑桃:一种新的基因组装配算法及其在单细胞排序的应用。J Comp Biol。2012; 19:455-77。https://doi.org/10.1089/cmb.2012.0021.
- 66.
Langmead B, Salzberg SL. Bowtie2快速间隙读对准。Nat方法。2012;9:357-9。https://doi.org/10.1038/nmeth.1923.
- 67.
Wick RR,Schultz MB,Zobel J,Holt Ke。绷带:De Novo基因组组件的交互式可视化。生物信息学。2015; 31:3350-2。https://doi.org/10.1093/bioinformatics/btv383.
- 68.
Wyman SK, Jansen RK, Boore JL。用DOGMA自动注释细胞器基因组。生物信息学。2004;20:3252-5。https://doi.org/10.1093/bioinformatics/bth352.
- 69.
Kearse M,Moir R,Wilson A,Stoneshavas S,Cheung M,Sturrock S等人。佐芙基本:用于组织的集成和可扩展的桌面软件平台和序列数据分析。生物信息学。2012; 28:1647-9。https://doi.org/10.1093/bioinformatics/bts199.
- 70.
Schatter P,Brooks An,Lowe TM。用于检测TRNA和SNORNAS的TRNASCAN-SE,SNOSCAN和SNOGPS Web服务器。Nucl酸res。2005; 33:W686-9。
- 71.
Lohse M, Drechsel O, Bock R. OrganellarGenomeDRAW (OGDRAW):一种易于生成高质量定制质体和线粒体基因组图形图的工具。咕咕叫麝猫。2007;52:267 - 74。https://doi.org/10.1007/s00294-007-0161-y.
- 72.
Raubeson La,Peery R,Chumley Tw,Dziubek C,Fourcade HM,Boore JL等。对比叶绿体基因组学:分析包括来自Angiospers的新序列Nupha Radvena.和毛茛属植物macranthus.BMC基因组学。2007;8:174。https://doi.org/10.1186/1471-2164-8-174.
- 73.
Kurtz S,Phillippy A,Delcher Al,Smoot M,Shumway M,Antonescu C,等。用于比较大型基因组的多功能和开放软件。基因组Biol。2004; 5:R12。https://doi.org/10.1186/gb-200-5-2-R12.
- 74.
kaoh K, Standley DM. MAFFT多序列比对软件版本7:性能和可用性的改进。生物学杂志。2013;30:772-80。https://doi.org/10.1093/molbev/mst010.
- 75.
基于最大可能性的系统发育分析与数千种分类群和混合模型。生物信息学。2006;22:2688e2690。https://doi.org/10.1093/bioinformatics/btl446.
- 76。
Posada D,Buckley Tr。系统血症中的模型选择和模型平均值:Akaike信息标准和贝叶斯曲目过度偏移比率的优点。系统中的生物学。2004; 53:793-808。https://doi.org/10.1080/10635150490522304.
- 77。
ronquist f,huelsenbeck jp。MRBAYES 3:混合模型下的贝叶斯系统发育推理。生物信息学。2003; 19:1572-4。https://doi.org/10.1093/bioinformatics/btg180.
- 78。
Yang Z.PAML:通过最大可能性进行系统发育分析的程序包。小屋。1997年; 13:555-6。https://doi.org/10.1093/bioinformatics/13.5.555.
确认
我们感谢三个匿名审查员在改善手稿方面的批判性评论。我们也感谢梁鑫王教授,以及J.Marut博士为本研究提供了一些样品。
资金
作者要感谢国家自然科学基金的主要计划(31590823),中国天然科学基金(31872673),云南省NSFC-联合基金会(U1802287),大 -中国科学院的规模科学设施(2017年 - LSF-GBOWS-02),以及跨学科研究(CAS)昆明研究所(CAS)的指导计划(CAS)的设计,以及收集,分析和解释数据,以及写作稿件。
作者信息
从属关系
贡献
YJ和JY2构思和设计了研究。LY,ZY,CL,ZZ,ZZ和JY1收集并分析了数据。yj,ly和zy准备了手稿。JY2和HL讨论了结果并修订了稿件。所有作者都阅读并批准了稿件。
相应的作者
道德声明
伦理批准和同意参与
所有样品的收集完全符合国家和地方法律的许可。这项研究没有涉及任何濒危或受保护的物种,研究中使用的植物样本也不是从国家公园或自然保护区采集的。根据国家和地方立法,收集这些植物不需要特别的许可。
同意出版
不适用。
利益争夺
作者宣称,这项研究是在没有任何商业或财务关系的情况下进行的,这些关系可以被解释为潜在的利益冲突。
附加信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
额外的文件1:
表S1。包含Genbank加入的系统发育分析中包含的塑料。(docx 19 kb)
附加文件2:
表S2。Illumina的测序结果总结巴黎粳稻那P. Verticillata.那Trillium govanianum.那ypsilandra thibetica和Y. Yunnanensis..(DocX 14 KB)
附加文件3:
表S3。在日本Paris japonica的质体中鉴定的基因列表。(多克斯16 kb)
附加文件4:
表S4。在质体中鉴定的基因列表巴黎verticillata.(多克斯16 kb)
附加文件5:
表S5。在质体中鉴定的基因列表Trillium govanianum..(多克斯16 kb)
额外的文件6:
表S6。在质体中鉴定的基因列表Ypsilandra thibetica。(多克斯16 kb)
额外的文件7:
表S7。在质体中鉴定的基因列表ypsilandra yunnanensis..(多克斯16 kb)
额外的文件8:
图S1。的地图巴黎粳稻质体系。圆圈外的基因是顺时针转录的,圆圈内的基因是逆时针转录的。内圆的深灰色区域表示质体的CG含量。(JPG 5223 KB)
额外的文件9:
图S2。的地图巴黎verticillata质体系。圆圈外的基因是顺时针转录的,而圆圈内的基因是逆时针转录的。内圆的深灰色区域表示质体的CG含量。(JPG 5226 kb)
额外的文件10:
图S3。的地图Trillium govanianum.质体系。圆圈外的基因是顺时针转录的,圆圈内的基因是逆时针转录的。内圆的深灰色区域表示质体的CG含量。(JPG 5223 KB)
额外的文件11:
图S4。的地图ypsilandra thibetica质体系。圆圈外的基因是顺时针转录的,圆圈内的基因是逆时针转录的。内圆的深灰色区域表示质体的CG含量。(JPG 5244 KB)
额外的文件12:
图S5。的地图ypsilandra yunnanensis.质体系。圆圈外的基因是顺时针转录的,圆圈内的基因是逆时针转录的。内圆的深灰色区域表示质体的CG含量。(JPG 5287 kb)
权利和权限
开放获取本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
杨磊,杨志强,刘超。等等。叶绿体系统核拍摄体分析为最大的真核基因组持有者的演变提供了见解,巴黎粳稻(Melanthiaceae)。BMC植物杂志19,293(2019)。https://doi.org/10.1186/s12870-019-1879-7
已收到:
公认:
发表:
关键词
- 质体系
- Phylogenomics
- 巨大的基因组
- 进化
- 巴黎粳稻
- 巴黎
- Trillium govanianum.