摘要
背景
光学直流。是菊科中体型最大、形态异质性最强的属之一。在的关系光学很可能是由于早期快速辐射造成的。研究质体的进化并解决其中的骨干关系光学,我们对代表所有4个亚属的17个物种的完整质体进行了测序。
结果
全部光学质体与大多数菊科质体具有相同的基因含量和结构。分子进化分析表明,大多数质体蛋白编码基因已经过纯化选择。系统基因组分析20光学另一种选择是包含所有蛋白质编码基因的核苷酸或氨基酸序列,而不是整个质体的核苷酸序列,支持单系光学并在其中发现了三个分支。传统的4个亚属中有3个被恢复为副生。七个质体区域被鉴定为含有最高的核苷酸变异性。
结论
我们的分析揭示了质体结构的保守性和在同属类群中解决关系的能力。数据集之间拓扑结构的差异很可能主要是由于简约信息字符数量的差异造成的。我们的研究表明,目前的分类光学可能至少部分基于收敛的形态特征状态。更大的分类单元取样将是必要的,以探索特征进化和生物地理学的属。我们的研究结果有助于深入了解哪些位点将提供最多的系统发育信号光学和cardueae。
背景
光学直流。是菊科中最大的属之一[1,2].它由大约300种组成,分布在亚洲、欧洲和北美,在喜马拉雅山脉和中亚的多样性最高[3.,4].光学表现出极端的形态多样性,存在于5000米以上的草原、潮湿森林和寒冷干燥的高山草甸[3.,5].
已进行了一些系统发育研究光学但该属的界限和属内关系仍有争议[5,6,7,8,9,10,11,12].李普希茨(13共鉴定出390种,隶属于6个亚属。光学直流。jurinocera.(扑砸。)嘴唇。,埃利奥尼(直流)。钩。f。amphilaena.(Stschegl)。Lipsch。Theodorea(Cass)。Lipsch。和Frolovia(直流)。Lipsch。然而,分子证据[7,10,14]已经表明了该subg。jurinocera.,subg。Frolovia和教派。Elataesubg。光学应该被排除在外光学作为独立的属来对待:Lipschitziella随机变数Kamelin,Frolovia(直流)。Lipsch。和Himalaiellaraab-Straube分别。使用分子和形态证据,shi和raab-straube [3.]建议光学教派。Aucklandia应该被视为新属,AucklandiaFalc。基于五个基因座的序列(rbcL ndhF matK trnL-F及其)和形态,Wang等人。[11]建立了属尚花王玉杰,Raab-Straube, Susanna,刘建强Jacea,剩下四个亚属(光学、Eriocoryne Amphilaena和Theodorea)作为构成光学尽管有了这些进展,但由于潜在的快速辐射,这四个亚属之间和内部的关系一直没有很好地解决,在更深的节点上留下了不充分的系统发育信号[5].虽然最近使用核基因的靶向核基因的富集来解决Cardueae的关系,但最近的近期的研究尚未评估这些关系。光学代表两个亚属[15].
质体已经被证明是探索植物生命之树深层关系的有力工具[16,17,18,19].他们帮助解决了特别顽固的谱系的模糊关系,例如经历过快速进化辐射的速度(例如[20.,21,22,23])。完整的塑料序列还提供了与基因重排,重复和损失相关的分子进化模式的洞察(例如[24,25,26]),并且在某些情况下,这些结构变化是本身和本身的系统源性信息性状,例如,这两个大反转(〜20kb和〜3 kB逆),其表征了大多数奥尔西西的大单拷贝(LSC)区域塑料[27,28,29].
质体的不同区域有不同的选择约束,这可能产生对系统发育的不同估计,例如,非编码区与编码区[30.,31].选择性力也可能在驱动质体结构中发挥作用[32],包括重新排列[33]和基因丢失[34,35].然而,在菊科中,选择性力量在质体进化中的作用尚不清楚。
到目前为止,只有三个光学有报道称:美国involucrata[36],美国chabyoungsanica[37),美国polylepis[38].在这里,我们对17个物种进行了测序,它们代表了所有4个亚属光学为了(1)阐明质体进化,包括结构变异和分子选择信号,(2)估计不同质体数据集在解决这一辐射谱系中的关系方面的有效性,(3)研究其中的主干关系光学.
结果
的特点光学塑料
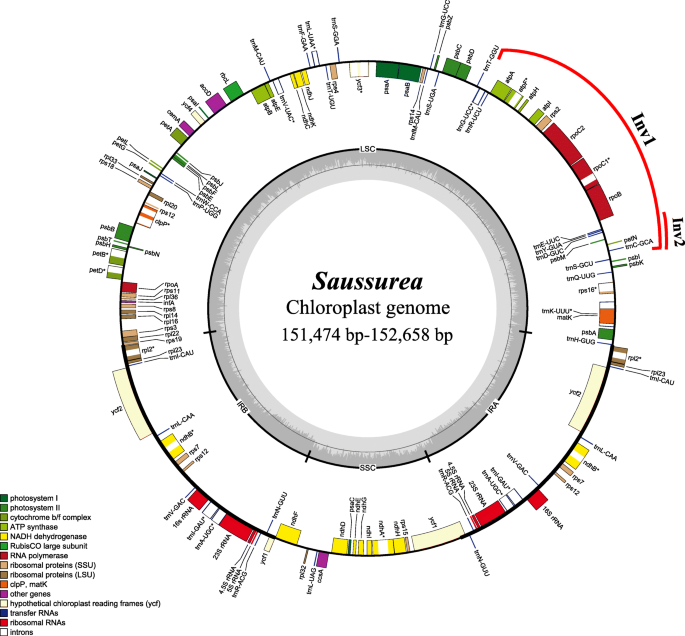
经过重新组装和参考引导组装,我们获得了每个质体的单个支架。表中提供了排序和装配信息1和额外的文件1:表S2。17的大小光学胞浆大小相近,范围为151474 bp美国tridactyla到152,658 bp美国przewalskii.17个质体均具有典型的被子植物四体结构,包含113个独特基因,其中蛋白编码基因79个,转移RNA (tRNA)基因30个,核糖体RNA (rRNA)基因4个。共有18个基因(包括11个蛋白编码基因和7个tRNA基因)含有内含子,其中15个基因含有1个内含子,3个基因含有2个内含子。IR区域也高度一致,均包含17个基因(6个蛋白编码基因,7个tRNA基因,4个rRNA基因)。在所有的质体中rps12基因被发现是反式剪接的,它的一个外显子位于LSC区域,另一个在IR区域复制(图。1).
一般的质体图光学。表中提出了每个物种的塑料的具体尺寸1.发票:反演
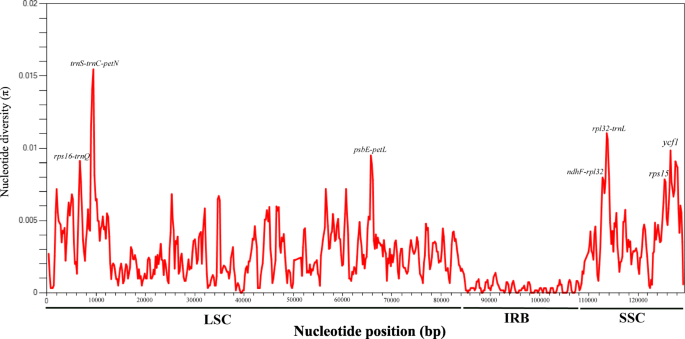
大部分菊科植物的~ 20kb和~ 3kb倒置(Inv1和Inv2)均被检测到光学质体系(无花果。1).inv1位于TRNG-UCC.和trnS-GCU基因;Inv2(位于trnS-GCU和trnE-UUC基因)嵌套在大反转内,与Inv1共享一个端点(图。1).滑动窗口分析显示单拷贝区域中的可变部位比在IR区中的更高比例。七个相对高度的可变地区(rps16-trnQ,trnS-trnC-petN, psbE-petL, ndhF-rpl32, rpl32-trnL, rps15和YCF1.),从质体序列中鉴定(图。3.).
选择分析
多数蛋白编码基因的dN/dS比值较低(ω;额外的文件2:图S1),表明它们已经过提纯筛选。只有三个基因(psbL, psbZ和YCF2.)有ω > 1,但分支模型结果显示前景和背景分支之间没有显著差异(表2).
系统发育分析
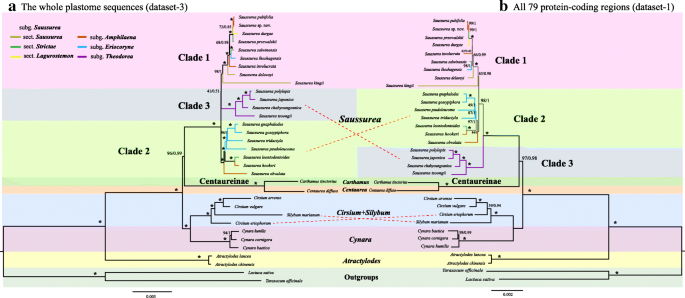
我们的系统基因组分析大大提高了分辨率,并提供了可靠的主干关系光学(无花果。4额外的文件3.:图S2)。三个连接数据集的特征如表所示3..DataSet-3具有最高数量的定义信息(PI)字符,然后是DataSet-1和DataSet-2。Centaureinae被解除为姐妹光学在数据集-1和-3中有强大的支持,但在数据集-2中没有。所有三个数据集也都有力地支持了单系光学(BS = 100);埃里科诺珊,amphilaena,和光学)四种传统的亚因子被解析为琼脂糖素。三个主要的光学被确定。枝1包括亚群的3种。amphilaena.(S . publifolia年代。sp。11月。,美国involucrata),子之一。埃利奥尼(美国lhozhagensis)和五个subg。光学(S. Durgae,S.przewalskii,S. Salwinensis,S. Delavayi,S. Kingii).枝2包括亚群2种。amphilaena.(S. hookeri, S. obvalata)和4个subg。埃利奥尼(S.Gnaphalodes,S.Gossypiphora,S. Psedolecoma,和美国tridactyla).Clade 3包括两个Subg。Theodorea(S. japonica和美国tsoongii)及两种韩国品种(沙布youngsanica, polylepis).DataSets-1和-2已解决的Subg。Theodorea作为剩下的姐妹光学,而数据集3将分支2解析为remaining的姐妹光学,虽然具有低支持(图。4额外的文件3.:图S2)。基于聚并的结果得到了与基于连接的系统发育(数据集-1)几乎相同的拓扑结构,除了位置S. Kingii.,它被解析为进化枝1 +进化枝2的姐妹(附加文件)4:图S3)。
讨论
质体基因组进化
20.光学在我们的分析中,质体表明该属的质体进化是保守的。全部光学质体具有大多数菊科的典型质体结构,包括几乎存在于所有菊科的LSC倒位摘要以[27],艾[29], Lasthenia[28],塔克斯卡姆[39),Mikania[40].IR区域的扩张和收缩已经被证明是某些细胞体长度变化的重要来源,例如早期分化的双二体[41,42]和apiales [43].然而,在本研究中,没有检测到显着的IR长度变化光学质体系(无花果。2).
在我们的分子进化分析中,大多数蛋白编码基因被发现处于纯化选择(附加文件2:图S1)。这种模式也已经在其他芽服血浆中证明,例如Mikania cordata[40),直升机[44],反映了绿色植物中塑性基因的典型保守演变。实际上,放松纯化选择的最佳证据是在损失光合作用的植物中,由于功能丧失,直接在光合作用中涉及的基因更快,通常导致伪析化和最终的基因损失[32,34,35].然而,考虑到大多数质体蛋白编码于细胞核,基于全基因组和转录组的分析对于充分研究质体蛋白编码位点选择的重要性是必要的。
系统发育信息信息网站
为了解决近缘物种间的关系,必须确定快速进化的位点。以前的系统发育研究光学主要偏向于三个质体位点(trnL-F,psbA-trnH,和matK)但这些已经未能解决整个属的关系(例如[5,6,7,8])。我们的分析显示,这三个区域的核苷酸多样性相对较低(图。3.),解释先前分析的低分辨率,并强调探索更多的质体以获取更多信息的地点和区域的重要性。我们发现了七个相对可变的区域:rps16-trnQ,trnS-trnC-petN, psbE-petL, ndhF-rpl32, rpl32-trnL, rps15和ycf1。这些,rps16-trnQ,trnC-petN,Psbe-petl,rpl32-trnl,rps15和YCF1.已被报道为分化热点,并被广泛用于重建植物分类系统发育[40,45,46,47,48,49,50].在这里确定的谱系特异性、快速进化的区域将有助于进一步确定大而多样的系统发育光学.
系统发育关系光学
主干关系光学在以往的分子系统发育研究中(例如,[5,6,7,8,10,11,12])。我们的分析在普遍的强大支持下大大提高了分辨率(图。4额外的文件3.:图S2)。除了subg。Theodora.(唯一的单系亚属),这里恢复的关系与用来定义节和亚属的形态特征之间的一致性相对较低[3.,4,13].事实上,这些形态特征已经被证明有适应价值,例如稠密的毛状体和彩色苞片,用来界定亚格。埃利奥尼和subg。amphilaena.分别。这两种特征状态在高山物种中普遍存在,被认为可以保护植物免受高海拔地区寒冷和UV-B辐射的影响[5,51,52,53].因此,系统发育和形态的不一致可能反映了在光学.同样重要的是要注意,我们对系统发育的估计仅基于快速辐射组中的塑性。鉴于不完全的谱系分类(ILS)或杂交最有可能在近亲中掩盖物种系统发育,可以增加核动作性数据的添加可能导致不同的关系估计光学.因此,有必要扩大分类单元和位点的取样光学为了更好地理解性状状态演化和生物地理学的模式。
由subg。Theodorea和教派。Laguranthera.(美国durgae)在系统发育研究中被确定为早期分化组光学基于ITS和trnL-trnF[7].在我们的连接数据集-1和-2和基于聚合的方法中,Subg的早期发散位置。Theodorea也得到了支持,尽管它与宗派在谱系上是相对遥远的。Laguranthera..跨所有连接数据集,S. Kingii.有迄今为止最长的树枝(图。4额外的文件3.:图S2),也检测到王和刘的系统发育研究中检测到[12].正如上面提到的,这可能是由于它的两年生习性造成的,因为众所周知,代期较短的物种的替代率较高[54].此外,系统的位置S. Kingii.在连接和基于聚合的方法之间不稳定,可能需要进一步调查。
我们的三种基于连接的分析所导致的树之间更深层次的不一致可能与数据集之间的简约信息(PI)字符数量的差异有关,其中在dataset-3(表)中PI字符的数量最高3.).这些差异可能解释了对脊梁更好的整体支持光学在基于数据集-3的树(图。4A)与其他树相比。鉴于我们的研究相对较低的分类水平(在一个属内),包括核苷酸序列,特别是非编码区域,将最大限度地发挥解决关系的力量是有道理的。因此,我们推荐在这些情况下完整的质体数据集。考虑到这些分支是多么的短,在一些主干节点上的不一致并不令人惊讶;很可能这些分支中很少存在PI特征,因此这些节点对系统发育分析的条件非常敏感[23,55].
结论
我们的分析揭示了质体结构的保守性和在同属类群中解决关系的能力。通过检查蛋白质编码基因座的选择信号,我们能够消除由于选择性偏差作为拓扑不一致的来源而产生的系统误差。因此,数据集中拓扑的差异很可能主要是由于差异信息性字符数的差异。我们的研究进一步证明目前接受的亚基团体光学可能至少部分地基于收敛性的字符状态,因此需要修改。此外,需要更大的分类种类来解除仅在这里暗示的性格演化和生物地理的模式。我们的结果在这里提供有用的洞察力,其中洛卡将提供最多的PI网站光学和Cardueae,但他们还表明,完全的塑料测序将是解决这种困难的属中的关系的宝贵技术。
方法
分类单元取样,叶绿体DNA分离,高通量测序
我们对17个新的质体进行了测序,代表了16个目前被描述的物种和一个未被描述的物种光学;收集和凭证信息在附加文件中提供1S1:表。这些被添加到GenBank中之前报道的三个质体中(附加文件)1:表S1)。的界限和亚基化处理光学跟着中国植物志和植物的Pan-Himalaya[3.,4].所有物种的总DNA都是用改良的CTAB(十六烷基三甲基溴化铵)方法从新鲜或硅胶干燥的叶子中提取的[56].按照Sun等人介绍的方法构建测序库并进行量化[41].以2.5 ~ 5.0 ng超声DNA为输入,构建TruSeq Illumina (Illumina Inc., San Diego, CA, USA) 500bp DNA序列库。使用安捷伦2100生物分析仪(安捷伦技术公司,Santa Clara, CA, USA)和实时定量PCR对文库进行定量。然后,利用Illumina HiSeq 2000平台在中国云南昆明Novogene进行2 × 125 bp测序。
质体组装、注释和比较分析
原始序列序列随后使用Trimmomatic v.0.36进行过滤[57[以下参数:滑动窗口= 4:20,Minlen = 50,前导= 3,尾随= 3,头部作物= 12,Avgqual = 20.剩余的高质量读数将De Novo组装到Contig中,最小使用CLC Genomics Workbench 11.0的长度为1000 bp(https://www.qiagenbioinformatics.com/)默认参数。然后由此产生的de novo contigs参考塑料组装美国chabyoungsanica.使用教条注释成品塑料[58]和GeSeq [59].在genei9.0.5版本中,手动调整起始/终止密码子和内含子/外显子边界[60,利用已发表的质体光学作为引用。tRNA基因通过tRNAscan-SE鉴定[61].圆形塑料的物理贴图用OGDRAW进行了可视化[62].
我们进行了质体比较光学polylepis及其他六属山茱萸科(金缕梅、红花、山茱萸、白花矢车菊、水飞蓟、白术).所有7个完整的质体都与progressiveemauve对齐[63,假设基因组共线进行全面比对。为了评估序列差异并确定高度系统发育信息位点,在DnaSP版本6.11.01中采用滑动窗口分析计算核苷酸变化率(π) [64]所有对齐的塑料序列光学.为了对齐,必要时在Geneious中手动反转SSC区域。步长设置为200 bp,窗长为600 bp。
系统发育分析
三十一个分类赛(附加档案1:表S1)光学+ 11个山茱萸科外群属和2个菊苣科外群分类群(莴苣,蒲公英)被包含在系统发育分析中。进行级联和基于聚合的分析。对于基于串联的方法,分析了三个数据集:数据集-1包括所有79个蛋白质编码序列(CD)的核苷酸序列;DataSet-2包括这些79 CD的氨基酸序列;并且数据集-3包括完全塑性核苷酸序列,包括仅IR区域的一个副本。通过使用Phylosuite版本1.1.15连接对齐来创建数据集-1和-2 [65].所有三个数据集的特征均使用MEGA X进行计算[66].对于所有连接数据集,ModelTest版本3.7 [67用于估计Akaike信息标准(AIC)下的最佳模型。使用RAXML版本8.2.10进行最大似然(ML)分析[68]在一般时间可逆的核苷酸替代模型下,采用速率异质性的gamma模型(数据集-1和daset-3为GTRGAMMA;PROTGAMMAAUTO dataset-2)。使用RAxML的“快速Bootstrap”算法估计了Bootstrap (BS)支持有1000个Bootstrap重复。贝叶斯推理(BI)使用MrBayes版本3.2.3进行[69].两次试验采用四种马尔可夫链(一种冷链,三种热链)并行进行,每次试验从随机的起始树中抽取50万代样本,每5000代进行一次抽样。通过检测分裂频率的平均标准差(ASDF)来评估收敛性。当ASDF < 0.01时,前25%的树被丢弃为烧成树,剩余的树被用来构建多数规则共识树。
对于基于聚结的分析,在GTRGAMMA模型下,使用RAxML分别估算了79个CDS序列的ML无根树,共500个bootstrap重复。ASTRAL III版本5.6.2算法[70]用于从79个基因树中估计物种树,节点支持作为局部后验概率计算。
选择签名分析
为了检验质体蛋白编码基因选择的证据,我们使用CodeML估算了所有79个蛋白编码基因的非同义(dN)和同义(dS)替换(ω)的比率[71设置如下:model = 0, seqtype =1, NSsites = 0。显示较高ω的基因与分支模型一致[72,73来确定系特异性的质体选择光学.按照CodeML中的建议,使用基于连接的方法,RAxML用数据集-1确定的最佳ML树作为输入拓扑,由光学设置为前景分支。似然比和P值用于测试前景分支上的正面选择的模型(“模型= 2”)是在空模型(“型号= 0”)上的显着改进,其中在前台分支没有正选择。
数据和材料的可用性
本研究中使用的所有序列可从国家生物技术信息(NCBI)中获得(见附加文件1:表S1)。
缩写
- 另类投资会议:
-
akaike信息标准
- asdf:
-
分裂频率的平均标准偏差
- BI:
-
贝叶斯推断
- cd:
-
蛋白质编码序列
- CTAB:
-
溴化十六烷基trimethylammonium
- DnaSP:
-
DNA序列多态性
- 信条:
-
双细胞器基因组注释器
- GTR:
-
一般时间可逆
- 红外:
-
倒置重复
- 它的:
-
核糖体DNA的内部转录间隔
- LSC:
-
大单副本
- ML:
-
最大似然
- NCBI:
-
国家生物技术信息中心
- PI:
-
Parsimony-informative.
- 核糖体rna:
-
核糖体核糖核酸
- SSC:
-
小单副本
- tRNA:
-
转移核糖核酸
参考文献
- 1。
Bremer K.Asteraceae:信守分类。波特兰:木材出版社;1994年。
- 2.
杜鹃科(杜鹃科)。在:Funk VA, Susanna A, Stuessy TF, Bayer RJ,编辑。菊科的分类学、进化和生物地理学。维也纳:IAPT;2009.p . 293 - 313。
- 3.
Shi Z,Raab-Straube EV:Cardueae。在:中国植物群。由吴泽,乌鸦,P. H.&Hong,D. Y.,Vol。20-21。北京和圣路易斯:科学出版社和密苏里州植物园新闻出版社2011:42-194。
- 4.
陈燕。菊科II光学,卷。48(2)。北京:科学出版社;2015年。
- 5.
王永杰,刘建强。冷云的辐射光学(Asteraceae:Cardueae)由青藏高原隆起引发。Biol J Linn Soc。2009; 97(4):893-903https://doi.org/10.1111/j.1095-8312.2009.01225.x.
- 6.
Häffner E.山梨亚族(山梨科,菊科)的系统发育研究。Englera。2000;21:3 - 208https://doi.org/10.2307/3776757.
- 7.
Raab-Straube EV。系统发育关系光学(菊科,山茱萸科)感叶,由形态学、ITS和trn L-trn F序列数据,带有摘要Himalaiella11月,将军Lipschitziella和Frolovia.Willdenowia。2003;33 (2):379 - 402https://doi.org/10.3372/wi.33.33214.
- 8.
Susanna A, Garcia-Jacas N, Hidalgo O, Vilatersana R, Garnatje T. The Cardueae (Compositae) revisited: insights from ITS,trnL-trnF,和matK核和叶绿体DNA分析。Ann Mo Bot Gard, 2006:150-71https://doi.org/10.3417/00266493 (2006) 93 (150: TCCRIF) 2.0.CO; 2.
- 9.
傅ZX,焦佛,聂湾,张GJ,高Tg。向日葵家族的综合仿制性文学系统:对中国煤层科的系统学的影响。J Syst Evol。2016; 54(4):416-37https://doi.org/10.1111/jse.12216.
- 10。
李志刚,李志刚,李志刚,等。龙葵属植物的分子系统发育分析及系统学研究光学及相关属(菊科、山茱萸科)。分类单元。2004;53 (3):679 - 90https://doi.org/10.2307/4135443.
- 11.
王永杰,冯瑞博,刘建强。尚花(Compositae),来自青藏高原和喜马拉雅山脉的新属。分类。2013; 62(5):984-96https://doi.org/10.12705/625.19..
- 12.
王永军,刘建强。系统发育分析光学教派。Pseudoeriocoryne(菊科:山茱萸科)基于叶绿体DNATRNL-F.序列。生物化学学报。2004;32(11):1009-23https://doi.org/10.1016/j.bse.2004.04.005.
- 13.
Lipschitz S:Rod Saussurea DC。(菊科)。Leningrad:“Nauka”,Leningradskoje otdelenie;1979年。
- 14.
王永军,刘建强,米和。喜马拉雅特有植物的系统发育起源Dolomiaea,Diplazoptilon和Xanthopappus(Asteraceae:Cardueae)基于三个DNA区域。Ann Bot。2007; 99(2):311-22https://doi.org/10.1093/aob/mcl259.
- 15.
Herrando-Moraira S, Calleja JA, Carnicero P, Fujikawa K, Galbany-Casals M, Garcia-Jacas N, Im H-T, Kim S-C, Liu J- q, López-Alvarado J, López-Pujol J, Mandel JR, Massó S, Mehregan I, Montes-Moreno N, Pyak E, Roquet C, Sáez L, Sennikov A, Susanna A,基于NGS靶区富集的数据处理策略。分子系统学进展。2018;128:69-87https://doi.org/10.1016/j.ympev.2018.07.012.
- 16.
Jansen RK, Cai Z, Raubeson LA, Daniell H, Leebens-Mack J, Müller KF, Guisinger-Bellian M, Haberle RC, Hansen AK, Chumley TW。通过对64个质体基因组中的81个基因的分析,揭示了被子植物的亲缘关系,并确定了基因组规模的进化模式。中国科学(d辑:地球科学)2007;104(49):19369-74https://doi.org/10.1073/pnas.0709121104.
- 17.
利用叶绿体基因组的大规模平行测序技术提高系统发育分辨率。BMC医学杂志。2009;7 (1):84https://doi.org/10.1186/1741-7007-7-84.
- 18.
利用质体基因组规模数据分析基部被子植物之间的神秘关系。中国科学院院刊。2007;104(49):19363-8https://doi.org/10.1073/pnas.0708072104.
- 19.
Gitzendanner MA,Soltis PS,Wong GKS,Ruhfel Br,Soltis de。绿色植物的体积素描学分析:十亿年的进化史。我是J机器人。2018; 105(3):291-301https://doi.org/10.1002/AJB2.1048.
- 20。
吴志强,葛松。草中BEP分支的系统发育:来自叶绿体全基因组序列的证据。acta physica sinica, 2012;62(1): 573-8https://doi.org/10.1016/j.ympev.2011.10.019.
- 21。
张n,文j,zimmer ea。另一种观察葡萄秩序生命的系统发育位置:叶绿体系统核科学学具有展开的关键谱系的膨胀抽样。mol phylocyet evol。2016; 101:216-23https://doi.org/10.1016/j.ympev.2016.04.034.
- 22。
质体基因组揭示了棕榈树和其他单子叶品系之间的深度系统发育关系和广泛的比率变异。新植醇。2016;209 (2):855 - 70https://doi.org/10.1111/nph.13617.
- 23.
MA PF,张YX,ZENG CX,GUO ZH,LI DZ。叶绿体系统核糖组分析解决了顽固的竹部落arundinarieae(Poaceae)的深层关系。系统中的生物学。2014; 63(6):933-50https://doi.org/10.1093/sysbio/syu054.
- 24.
Sun Y, Moore MJ, Lin N, Adelalu KF,孟A, Jian S, Yang L, Li J, Wang hNDH.基因家族。BMC基因组学。2017;18 (1):592https://doi.org/10.1186/s12864-017-3956-3.
- 25.
闫米,弗里奇PW,摩尔MJ,冯T,孟安,杨杰,邓T,赵开,姚X,孙H.Plastid系统源神学学决定了突发术的依赖突发术的侵犯关系,并在肉体骨干关系上脱落。mol phylocyet evol。2018; 121:198-211https://doi.org/10.1016/j.ympev.2018.01.004.
- 26.
植物叶绿体基因组系统发育学研究进展[j]。植物科学进展。2005;10(5):203-9https://doi.org/10.1016/j.tplants.2005.03.007.
- 27.
Kim K-J, Choi K-S, Jansen RK。在向日葵科(菊科)的早期进化过程中,两个叶绿体DNA倒置同时发生。中国生物医学工程学报。2005;22(9):1783-92https://doi.org/10.1093/molbev/msi174.
- 28.
沃克JF,詹尼斯MJ,埃默里NC。叶绿体全基因组序列与倒转变异的比较分析Lasthenia burkei(Madieae菊科)。中国科学(d辑:地球科学)2014https://doi.org/10.3732/AJB.1400049..
- 29.
刘啊,霍恩,董立,王,张某,年轻的哈,冯x,顾yq。完全蒙古医学叶绿体基因组序列艾frigida与其他植物的系统发育关系。Plos一个。2013; 8(2):E57533https://doi.org/10.1371/journal.pone.0057533.
- 30。
Lam Vky,Darby H,Merckx Vsft,Lim G,Yukawa T,Neubig Km,Abbott Jr,Beatty Ge,Provan J,Soto Gomez M,Graham SW。极端的系统染料或肌肉发育方法:用肌肌孢子塑料进行案例研究。我是J机器人。2018; 105(3):480-94https://doi.org/10.1002/ajb2.1070.
- 31。
Walker JF, Stull GW, Walker- hale N, Vargas OM, Larson DA。在质体推断的系统发育中描述基因树冲突。bioRxiv。2019:512079https://doi.org/10.1101/512079.
- 32。
Bock DG,Andrew Rl,Rieseberg LH。论植物细胞质基因组的自适应价值。Mol Ecol。2014; 23(20):4899-911https://doi.org/10.1111/mec.12920.
- 33。
Weng ML,Blazier JC,Govindu M,Jansen RK。Geraniaceae中祖先塑性基因组的重建揭示了基因组重排,重复和核苷酸取代率之间的相关性。mol Biol Evol。2014; 31(3):645-59https://doi.org/10.1093/molbev/mst257.
- 34。
苏H-J, Barkman TJ,郝W, Jones SS, Naumann J, Skippington E, Wafula EK, Hu J- m。全寄生植物质体基因组的新编码和at丰富度记录Balanophora.中国科学院院刊。2019;116(3):934-43https://doi.org/10.1073/pnas.1816822116.
- 35。
王志强,王志强,王志强。植物非光合作用过程中进化速率变化的机制模型。中国科学院院刊。2016;113(32):9045-50https://doi.org/10.1073/pnas.1607576113.
- 36.
谢强,沈南南,郝旭,南PN, Hieu BTN,陈超,朱超,林永昌,肖cd .天山雪莲叶绿体全基因组研究(英文)光学involucrata),是中国著名的菊科药用植物。线粒体DNA Part A. 2015;28(2): 294-5https://doi.org/10.3109/19401736.2015.1118086.
- 37.
Cheon KS, Kim HJ, Han JS, Kim KA, Yoo KO。完整的叶绿体基因组序列光学chabyoungsanica(菊科),为韩国特有植物。conservation Genet resource . 2016:1-3https://doi.org/10.1007/s12686-016-0617-9.
- 38.
关键词:水稻,叶绿体,全基因组序列光学polylepis(菊科),韩国特有的易危种。线粒体DNA Part B. 2017;2(2): 650-1https://doi.org/10.1080/23802359.2017.1375881.
- 39.
来自无融合生殖的完整叶绿体基因组蒲公英(菊科):三个小种的身份和变异。《公共科学图书馆•综合》。2017;12 (2):e0168008https://doi.org/10.1371/journal.pone.0168008.
- 40。
苏勇,黄磊,王震,王涛。入侵杂草叶绿体基因组学的比较Mikania Micrantha.和它的本土同类Mikania cordata:结构变异、高度分化区域识别、分化时间估计和系统发育分析。分子系统学进展。2018;S1055790317307212https://doi.org/10.1016/j.ympev.2018.04.015.
- 41。
Sun Y, Moore MJ, Zhang S, Soltis PS, Soltis DE, Zhao T,孟A, Li X, Li J, Wang H. early- derived eudiicots几乎所有科18个完整质体的系统基因组学和结构分析,包括被子植物IR基因含量的进化分析。分子系统学进展。2016;96:93-101https://doi.org/10.1016/j.ympev.2015.12.006.
- 42。
Sun Y-X, Moore MJ,孟A-P, Soltis PS, Soltis DE, Li J-Q, Wang H-C。对齿齿科植物的质体基因组进行了测序,结果表明,齿齿科植物和齿齿科植物在古近纪存在差异。《公共科学图书馆•综合》。2013;8 (4):e60429https://doi.org/10.1371/journal.pone.0060429.
- 43。
唐妮斯,Jansen RK。来自蚜虫的全体积基因组的对比分析:倒置重复的膨胀和收缩,线粒体对DNA的塑性转移,以及高度发散的非分量区的鉴定。SYST BOT。2015; 40(1):336-51https://doi.org/10.1600/036364415X686620.
- 44。
Lee-Yaw JA, Grassa CJ, Joly S, Andrew RL, Rieseberg LH。对一年生向日葵中广泛的细胞核不一致的其他解释的评价(直升机).新植物。2019; 221(1):515-26。https://doi.org/10.1111/3/15386.
- 45。
Nolan K,Saemundur S,Hannes D,Yong Yj,Dapeng Z,Engels JMM,Quentin C.甲豆(Cacao)超级条形码(Theobromaspp。利用全叶绿体基因组和核糖体DNA。中国科学(d辑:地球科学)2012https://doi.org/10.3732/ajb.1100570.
- 46。
东路,徐C,李成,太阳j,zuo y,shi s,cheng t,guo J,Zhou S.YCF1.是陆生植物质体DNA条形码研究中最有前途的一种。Sci众议员2015;5:8348https://doi.org/10.1038/srep08348.
- 47.
王子LM。缓解植物植物和辐射地理物的塑性引物。苹果。植物SCI。2015; 3(6):1400085https://doi.org/10.3732/apps.1400085.
- 48.
于晓青,Drew BT,杨建斌,高利明,李德志。11个比较叶绿体基因组Schima(山茶科)种:DNA条形码和系统发育研究。《公共科学图书馆•综合》。2017;12 (6):e0178026https://doi.org/10.1371/journal.pone.0178026.
- 49.
高X,张X,萌H,李继,张达,刘C.对比叶绿体基因组巴黎马尔莫拉塔教派:对重复区域和进化意义的洞察。BMC基因组学。2018;19 (10):878https://doi.org/10.1186/s12864-018-5281-x.
- 50.
nebig KM, Whitten WM, Carlsward BS, Blanco MA, Endara L, Williams NH, Moore M.系统发育效用YCF1.兰花中的一种质体基因matK.植物学报。2009;277(1):75-84https://doi.org/10.1007/s00606-008-0105-0.
- 51.
Omori Y,Takayama H,OHBA H. Himalayan Giant Glasshouse植物中半透明苞片的选择性透光率感冒高贵的Hook.f.&thomson(Polygonaceae)。Bot J Linn Soc。2000; 132(1):19-27https://doi.org/10.1111/j.1095-8339.2000.tb01852.x..
- 52.
Tsukaya H,Tsuge T.春季低温发展的植物中花序的形态调整:“柔软植物”的收敛演变。植物BIOL。2010; 3(5):536-43https://doi.org/10.1055/s-2001-17727.
- 53。
杨y,körnerc,太阳h。青春期的生态意义光学美杜莎这是一种高海拔的喜马拉雅“毛茸茸的植物”。高原气象。2008;40(1):250-5https://doi.org/10.1657/1523 - 0430(07 - 009)(杨)2.0.CO; 2.
- 54。
Yang Y,Moore MJ,Brockington SF,Soltis de,Wong GK-S,Carpenter EJ,Zhang Y,Chen L,Yan Z,谢Y,Sage RF,Covshoff S,Hibberd JM,Nelson Mn,Smith SA。用转录组测序对高度多样化的植物湿度胰蛋白酶体解剖分子演变。mol Biol Evol。2015; 32(8):2001-14https://doi.org/10.1093/molbev/msv081.
- 55。
Valderrama E, Richardson JE, Kidner CA, Madriñán S, Stone GN。最近辐射的热带植物属中系统发育标记的转录组挖掘(RenealmiaL.f姜科)。分子系统学进展。2018;119:13-24https://doi.org/10.1016/j.ympev.2017.10.001.
- 56。
杨建斌,李德志,李海涛。九种新的通用引物对对被子植物叶绿体基因组进行高效测序。生态学报。2014;14(5):1024-31https://doi.org/10.1111/1755-0998.12251..
- 57。
Trimmomatic:用于Illumina序列数据的灵活微调器。生物信息学。2014;30:2114-20https://doi.org/10.1093/bioinformatics/btu170.
- 58。
Wyman SK, Jansen RK, Boore JL。用DOGMA自动注释细胞器基因组。生物信息学,2004;20 (17):3252 - 5https://doi.org/10.1093/bioinformatics/bth352.
- 59。
蒂利希M, Lehwark P, Pellizzer T, Ulbricht-Jones ES, Fischer A, Bock R, Greiner S. geseq -细胞器基因组的通用和准确注释。核酸Res. 2017;45(W1): W6-W11https://doi.org/10.1093/nar/gkx391.
- 60.
Geneious basic:一个集成的、可扩展的用于序列数据组织和分析的桌面软件平台。生物信息学。2012;28 (12):1647 - 9https://doi.org/10.1093/bioinformatics/bts199.
- 61.
Lowe TM,EDDY SR。Trnascan-SE:一种改进基因组序列转移RNA基因检测的程序。核酸RES。1997年; 25(5):955-64https://www.ncbi.nlm.nih.gov/pubmed/9023104.
- 62.
Lohse M,Drechsel O,Kahlau S,Bock R. Uniterellargenomedraw-一套用于生成塑体和线粒体基因组的物理图和可视化表达数据集的工具。核酸RES。2013; 41(W1):W575-81https://doi.org/10.1093/nar/gkt289.
- 63.
Darling Ae,Mau B.Perna NT:进展:与基因增益的多种基因组对齐。损失和重排普罗斯。2010; 5(6):E11147https://doi.org/10.1371/journal.pone.0011147.
- 64。
Rozas J, Ferrer-Mata A. SÃ nchez-DelBarrio JC, Guirao-Rico S, Librado P, Ramos-Onsins SE, Sã n-GA: DnaSP 6:大数据集DNA序列多态性分析。生物医学进展。2017;34(12):3299-302https://doi.org/10.1093/molbev/msx248.
- 65。
张D,高F,Li Wx,Jakovlići,Zou H,张J,王GT:文学:一种集成和可伸缩的桌面平台,用于简化的分子序数据管理和进化系统源研究。Biorxiv 2018:489088。https://doi.org/10.1101/489088.
- 66。
王志强,王志强,王志强,等。基于遗传算法的分子进化遗传分析。生物医学进展。2018;35(6):1547-9https://doi.org/10.1093/molbev/msy096.
- 67。
Posada D,Crandall Ka。ModelTest:测试DNA替换模型。生物信息学(牛津,英格兰)。1998年; 14(9):817-8https://doi.org/10.1093/bioinformatics/14.9.817.
- 68。
Stamatakis a . RAxML版本8:系统发育分析和大系统发育后期分析的工具。生物信息学。2014;30 (9):1312 - 3https://doi.org/10.1093/bioinformatics/btu033.
- 69。
Ronquist F, Huelsenbeck JP。MRBAYES:系统发育树的贝叶斯推理。生物信息学。2001;17 (8):754 - 5https://doi.org/10.1093/bioinformatics/17.8.754.
- 70。
张超,张志强。基于部分解析基因树的多项式时间物种树重建。BMC生物信息学。2018;19 (S6): 153https://doi.org/10.1186/s12859-018-2129-y.
- 71。
杨志。PAML 4:最大似然法的系统发育分析。中国生物医学工程学报。2007;24(8):1586-91https://doi.org/10.1093/molbev/msm088.
- 72.
张建军,杨志强。一种改进的分枝位点似然法在分子水平上检测阳性选择。中国生物医学工程学报。2005;22(12):2472-9https://doi.org/10.1093/molbev/msi237.
- 73.
杨志。阳性选择的似然比检验及其在灵长类溶菌酶进化中的应用。中国生物医学工程学报。1998;15(5):568-73https://doi.org/10.1093/oxfordjournals.molbev.a025957.
确认
不适用。
资金
中国科学院战略性先导科研计划项目(no . XDA20050203);国家重点研发计划项目(no . 2017YFC0505200);国家自然科学基金重大项目资助项目(no . 31590823)。关键词:边坡,边坡稳定性,边坡稳定性
作者信息
隶属关系
贡献
HW, YS和HS构思和设计了这项研究。XZ进行了从头组装、基因组注释、系统发育等分析。由XZ、TD、MJM、YS、YJ、HW、HS起草。XZ、TD和NL收集叶片材料。XZ、NL、HZ和AM进行实验。所有的作者讨论结果,并帮助形成研究,分析和最终的手稿。所有作者阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
附加文件1:
表S1。分类群包括在本研究中。为新排序提供了收集地点和凭证信息表S2。新测序的质体的测序和装配信息。Phred质量分数大于30的碱基在总碱基中所占的百分比。(多克斯27 kb)
附加文件2:
图S1。在PAML中,通过CodeML计算每个蛋白编码基因内的非同义替换和同义替换的比率(ω, dN/dS)。ω > 1的基因用红色表示。(PDF 1045 kb)
附加文件3:
图S2。推断来自ml(最大似然)和Bi(贝叶斯推理)的分子系统和Bi(贝叶斯推理)分析(79个CD;数据集2)的氨基酸序列分析。最大似然引导值(BS)和后验概率(PP)在节点处示出。分支机构*具有100%的引导支持和1.0后概率。(PDF 272 KB)
额外的文件4:
图S3。利用基于合并的方法估算了79个物种树。局部后验概率标记在节点处。带有*的分支的后验概率为1.0。的进化枝S. Kingii.是用红色标出的。(PDF 548 kb)
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
张,X.,邓,T.,摩尔,M.J.et al。塑料系统的血糖光学(菊科:Cardueae)。BMC植物杂志19,290(2019)。https://doi.org/10.1186/s12870-019-1896-6
已收到:
接受:
发表:
关键字
- 光学
- 快速辐射
- 质体系
- 基因组
- 系统核发科学分析
- 净化选择