摘要
背景
DNA甲基化是一种重要的表观遗传修饰,涉及基因表达调控、胚胎发育、细胞分化和基因组印迹等诸多生物学过程。它还涉及真核生物中的许多关键调控基因。通过追溯甲基化相关基因的进化史,我们可以了解这些基因的起源和扩展时间,这有助于了解植物的进化史,也可以了解不同物种的DNA甲基化模式的变化。然而,大多数关于甲基化相关基因进化的研究并没有针对整个DNA甲基化途径进行。
结果
在这项研究中,我们在77种中综合鉴定了33种甲基化相关基因,并在整个植物王国中研究了基因起源和进化。我们发现,原因,对甲基化维持和去甲基化的原因早期演变,而大多数新甲基化相关基因出现晚期。通过全基因组重复和串联复制扩增甲基化相关基因,但也伴随着不同物种中大量基因缺失事件。基因长度和内部长度在不同的物种中变化很大,但外显子结构和功能域相对节省。追踪与甲基化相关基因的系统发育关系揭示不同物种中DNA甲基化的演化病史。在物种的演变过程中,甲基化相关基因的表达模式已经改变,并且可以将这些基因的这些基因的表达模式聚集成四类。
结论
该研究描述了植物王国中DNA甲基化相关基因的全局表征。这些基因的原始时间,基因结构和系统发育关系的相似性和差异导致我们了解植物中DNA甲基化的进化节约和动态。
背景
经典DNA甲基化方法是通过不同的DNA甲基转移酶转移S-腺苷甲基乙硫氨酸的甲基,形成C5-甲基胞嘧啶(5-MC)[1].在植物中,在三个序列环境下的胞嘧啶(C)处发现DNA甲基化,包括CG、CHG和CHH (H = A、T或C),这与动物中在CG序列环境中占优势的DNA甲基化存在差异[2,3.,4].DNA甲基化在植物生长发育中起重要作用,并在基因组印记、春化、对生物和非生物胁迫的响应以及杂种优势中发挥作用[1,5,6,7,8,9,10,11].
在拟南芥,所有序列环境中的DNA甲基化(CG、CHG和CHH)都是通过rna定向DNA甲基化(RdDM)从头建立的[5,12,13,14,15,16].CG和CHG位点的甲基化维持主要依赖于DNA甲基转移酶1 (MET1) [5]和Chormomethylase3(CMT3)[17].CHH位点的甲基化不能维持,必须由CHROMOMETHYLASE2 (CMT2)和RdDM重新建立[18].在异染色质中,从头DNA甲基化和维持需要DNA甲基化1 (DDM1)的作用[18].DDM1是一种核心重新耦合器,可以改变核小体的组成和位置,这使得DNA甲基转移酶更易获得染色质[19].JUMONJI 14 (JMJ14)去除组蛋白上的一些活性标记[20.,组蛋白去乙酰化酶6 (hda6) [21.,泛素特异性蛋白酶26 (ubp26) [22.],而被抑制的标记H3K9me2由SUVH4维持[23.],可以维持DNA甲基化或由DNA甲基化引起的沉默状态。
RdDM途径依赖于一种专门的转录机制,该转录机制以RNA聚合酶II (Pol II)的两个植物特异性同源物为中心:Pol IV和Pol V [24.,25.,26.,27.].简而言之,典型的RdDM过程包括以下步骤:首先,在Pol IV依赖的小干扰RNA (sirna)生物发生过程中,单链RNA被Pol IV转录,然后在RNA依赖的RNA聚合酶(RDR2)的作用下转化为双链RNA [28.]在染色质Remodeler Classy 1(Clsy1)的帮助下,[29.];然后,双链RNA在DICER-LIKE 3 (DCL3)的作用下被切割成24 nt sinas [30.];24 nt siRNA的3 '端被HUA ENHANCER 1 (HEN1)甲基化[31.];最后,一条甲基化的24-nt siRNA被加载到ARGONAUTE 4 (AGO4)上[32.].其次,在POL V介导的DE Novo甲基化的过程中,在RNA定向的DNA甲基化1(DRD1)中染色染色质重塑剂将打开DNA双链体,并在Meristem沉默3(DMS3)和RNA中形成DDR络合物。定向DNA甲基化1(RDM1)稳定松散的染色质状态[33.,34.], MICRORCHIDIA 6 (MORC6)将有助于稳定松散状态[35.,36.];Pol V在SU(VAR) 3-9 HOMOLOG 2/9 (SUVH2/9)的协助下被招募到目标位点[37.];装载sirna的AGO4蛋白在Pol V的羧基末端结构域(CTD)和含KOW结构域的转录因子1 (KTF1)的作用下也被募集到靶位点,然后sirna与Pol V转录的支架RNA配对[38.];RDM1可以通过重新排列甲基转移酶1/2(DRM1 / 2)来连接蛋白,以激活DE Novo建立DNA甲基化。第三,在染色质变化的过程中,涉及de novo 2(Idn2) - Idn2副寄生酮(Idp)复合物与swi / snf复合物相互作用以改变核小体定位,从而使DNA更容易甲基化[39.].
虽然DNA甲基化是一种相对稳定的标记,但是细胞分裂后的子细胞可以继承,仍然是可逆的并且动态调节。在真核生物中,DNA甲基化水平不仅与建立和维持过程有关,还与去甲基化的方法有关。DNA去甲基化分为被动去甲基化和活性去甲基化。被动去甲基化是由于DNA复制过程中甲基化维持的损失,DNA甲基化的水平降低。活性去甲基化是通过DNA糖基酶/裂化酶和其他酶用非甲基化胞嘧啶代替5-甲基胞嘧啶碱基的方法[40.].在拟南芥,主DNA糖基酶包括沉默1(ROS1),遗迹(DME),DME样2(DML2)和DME样3(DML3)的阻遏物[5].这四种去甲基酶可以从所有序列中切除5-mC。之前的研究表明ROS1.可以在所有组织中进行活性DNA去甲基化[41.),而DME.优先在中间细胞中表达,并且雌性配子体的合并效应[40.,42.].此外,转录(ET)因子的效应表征在去甲基化中具有潜在作用[43.].
近年来,许多研究对植物甲基化相关基因的进化进行了探索。在六倍体小麦中,全部MET1基因分为三类,这些基因源于草家庭和基因重复内的全基因组重复,特别是在麦芽汁部落(44.].在不同植物和真菌中发现DNA甲基转移酶的发现表明MET1高度保守[11].RNA聚合酶亚基的进化分析表明,编码Pol IV和Pol V的基因相对于Pol II进化迅速。对15种植物RdDM通路基因的分析表明,RdDM出现在早期陆生植物中[45.].最近,利用443种陆地植物的数据推断了CMT科的起源[46.].
在这里,我们探讨了与藻类到开花植物相当长的进化尺度的甲基化相关基因的系统发育关系。我们在77种中鉴定了33种甲基化相关基因,并探讨了Novo甲基化,维持甲基化和去甲基化方法中基因的起源和演化。通过外显子性结构分析研究了基因结构变化,通过域分析研究了功能变化。此外,系统发育分析表明,这些甲基化相关基因分为三类,即早期植物,单子叶植物和双旋蛋植物。
结果
甲基化相关基因起源的不一致性
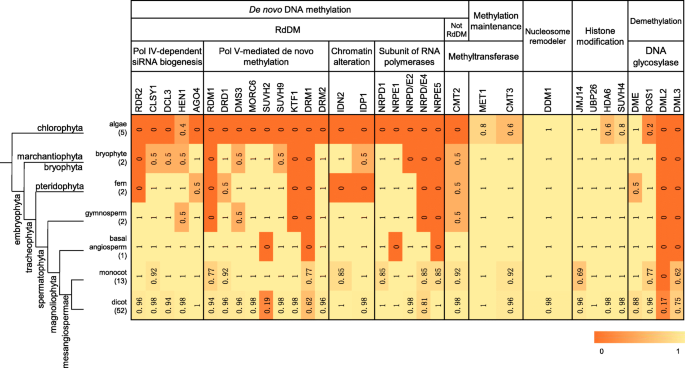
甲基化相关基因已在A. Thaliana..在这项研究中,所有这些基因被分为五个类别,包括八个组。这五类基因包括主要在DNA初始甲基化、甲基化维持、核小体重塑、组蛋白修饰和去甲基化中发挥作用的基因(图5)。1).典型的DNA甲基化RdDM过程包括三个步骤:Pol iv依赖的siRNA生物发生,Pol v介导的DNA甲基化和染色质改变。我们在涉及藻类和植物的77种物种中鉴定了所有这些类别的甲基化相关基因(附加文件)4:表S1)。这些藻类包括五种,植物包括苔藓植物、蕨类植物、裸子植物、基生被子植物、单子叶植物和双子叶植物。我们总结了各物种甲基化相关基因的含量(附加文件)5:表S2)。
在五个类别中的每一个中,我们计算了含甲基化相关基因的物种数量与物种总数的比率,并探讨了这些基因的藻类对较高开花植物的进化(图。1).在RDDM过程中,我们研究了Pol IV依赖性siRNA生物发生所需的5个基因。他们只有HEN1在藻类中发现,杂散,蕨类植物,单焦炭和双斑黄中的一些基因缺失。Pol V型介导的DE Novo甲基化有9个基因。SUVH2在基础上缺乏缺席,也缺乏71%的Dicots,而它在所有单像中发现。有2个基因参与染色质变化过程,它们在藻类中不存在。对于RNA聚合酶,我们探索了核RNA聚合酶D1(NRPD1),nrpe1.,NRPD2 / E2,NRPD4 / E4,nrpe5..有趣的是,藻类中没有发现所有五个亚基。发现CHH位点的甲基化通过在每个细胞周期中的DE Novo DNA甲基化过程重新建立,而De Novo DNA甲基化的CHH位点包括RDDM和另一种独立于RDDM的方式[12].后者需要的参与CMT2这在藻类中是不存在的。综上所述,藻类中只有少数基因参与DNA甲基化,但这些基因在苔藓植物和高等植物中广泛分布。因此,在绿藻和胚胎植物分化后,可能会出现DNA的重新甲基化过程。
在维持DNA甲基化的过程中,不同的酶以不同的方式起作用。CG的甲基化需要MET1基本上存在于所有这些物种中,包括五种下藻类中的四种。CHG的甲基化需要参与CMT3这也存在于这些被研究的物种中。因此,CG和CHG甲基化的维持可能来源于绿藻和胚胎植物的共同祖先。核小体的改造DDM1这对于介导异染色质区域的甲基化至关重要,基本上存在于所有被研究的物种中。其发生时间与DNA甲基转移酶的发生时间一致(MET1和CMT3).有4个基因(JMJ14,UBP26,HDA6和SUVH4)参与组蛋白修饰过程,它们都在藻类中发现。这些基因是组蛋白调控的重要组成部分,因此它们影响DNA甲基化,并影响植物的开花时间和耐旱性等表型性状[47.,48.,49.,50.].
对于DNA去甲基化过程,活性去甲基化是由四种DNA糖基化酶介导的(DME.,ROS1.,DML2.和DML3.).先前的研究表明DME.被发现只在dicots [42.,但我们的研究结果表明DME.和ROS1.从藻类到高等开花植物都存在。DML2.仅在双子叶中可见,比例相对较小(17%)。分布的分析DML2.在不同种类的植物中显示,它只存在于十字花科植物中(附加文件5:表S2)。DML3.存在于双子叶和单子叶。这些结果表明,DNA的去甲基化可能起源于绿藻门和胚胎门的共同祖先DML2.在十字花科植物和DML3.在Dicots和单焦点中有限。
甲基化相关基因的丰富和重复
为了探讨与甲基化相关的基因的发展方式,我们研究了不同物种中甲基化相关基因的丰富和复制模式。首先,我们总结了不同物种中甲基化相关基因的丰富(附加文件5:表S2)。观察到大量的基因扩增事件。在28个二倍体物种中,例如Setaria italica,Brachypodium distachyon,Fraxinus精益求精的,马吕斯有明显,Lotus对虾,大豆,杨树trichocarpa和素类,包含多个拷贝的基因数量显著高于其他43个二倍体物种(方差分析,p < 0.01) (Additional file5:表S2)。在6个多倍体物种中,包括5个四倍体(Panicum Virgatum.,烟草,gossypium hirsutum,海岛棉,芸苔栗鸟)及一个六倍体(小麦),甲基化相关基因的数量在二倍体水平上进行标准化。结果表明,含多个拷贝的基因数量相对较多P.Virgatum.,t . aestivum和B. Napus.(方差分析,p < 0.016:表S3)。
对于一些甲基化相关基因,也观察到基因缺失。在藻类中,存在有关甲基化相关基因的显着不存在(附加文件5:表S2)。所有五种藻类物种中都不存在23种甲基化相关基因。只发现4种甲基化相关基因,JMJ14,UBP26,DDM1和DME.,均存在于5种藻类中。在高等植物中,甲基化相关基因的缺失率要高得多。在单子叶植物中,有1到10个基因缺失。在双子叶植物中,有48个物种发生了基因缺失事件。在这三个拟南芥物种中,甲基化相关基因A. Thaliana.和A. Lyrata.是保守的,而在A. Halleri., 4个基因(RDM1,SUVH9,HDA6和NRPD / E2)缺席。在Solanum Tuberosum.在美国,基因缺失非常明显,33个基因中有10个缺失。
甲基化相关基因的扩增可能与全基因组复制(WGD)和串联复制有关。例如,基因组复制事件g·马克斯大约13-59百万年前,有助于形成近75%的基因拷贝[51.].大约5000万年前,有一个WGD活动M. Domestica.,显著增加了基因数量[52.].二idn2.基因在Gossypium raimondii就,Gorai.010G201400和Gorai.010G201300,从串联复制中演变。在这里,我们分析了与甲基化相关基因的进化模式奥雅萨苜蓿,茄属植物lycopersicum,vitis Vinifera,Amborella trichopoda.,Cucumis巨大成功和菜豆(附加文件7:表S4)。通过对这5个物种的WGD和串联复制信息的分析[53.,54.,我们发现这两种复制模式都有助于这些物种的基因扩增。一式两份MET1基因o.苜蓿是由WGD演变而来的。一式三份AGO4基因p .寻常的,Phvul.008G206600和Phvul.006G021200由WGD演变而来,Phvul.008G206600和Phvul.008G206500是由串联复制进化而来的。类似的现象也可以在美国lycopersicum,诉酿酒用葡萄,答:trichopoda和C. Sativus..
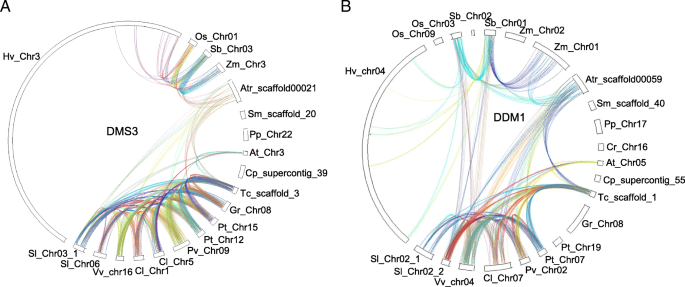
我们使用了77种中的17种,用于基因共同分析,包括四种早期植物,四种单粒细胞和九个Dicots。在每个甲基化过程中,我们对一些基因进行了共线分析(图。2额外的文件1:图S1)。共分析了10个甲基化相关基因。含有甲基化相关基因的染色体区域共线性表明这些基因是WGD或片段复制的产物。为AGO4,DMS3而其他基因、某些染色体片段在同一物种中具有较好的共线性。我们还比较了不同物种间的共线性。大部分基因与单子叶或双子叶之间不存在共线性关系,大部分基因与单子叶和双子叶之间也不存在共线性关系。在不同的早期植物中,染色体没有共线性。在单子叶植物中,的共线性玉米,高粱二色的,奥雅萨苜蓿找到了大多数基因。在Dicots中,观察到大多数基因的共同性,然而,一些基因A. Thaliana.,Citrullus lanatus,美国lycopersicum和卡里卡番木瓜共线性较差。这些数据表明,基因组进化对不同物种中甲基化相关基因的染色体共线性有破坏作用。
包含20个甲基化相关基因上下游相邻基因的染色体片段共线分析。一个不同物种DMS3基因的共线性。bDDM1基因在不同物种中的共线性。物种的名称是衣藻reinhardtii(Cr),Physcomitrella金属盘(PP),卷柏moellendorffii(Sm),Amborella trichopoda.(Atr),玉米(ZM),高粱二色的(SB),奥雅萨苜蓿(操作系统),Hordeum Vulgare.(高压)茄属植物lycopersicum(SL),vitis Vinifera(vv),Citrullus lanatus(CL),菜豆(PV),杨树trichocarpa(Pt),Gossypium raimondii就(gr),Theobroma Cacao.(TC),卡里卡番木瓜(CP),拟南芥(在)
甲基化相关基因基因结构的保护与变化
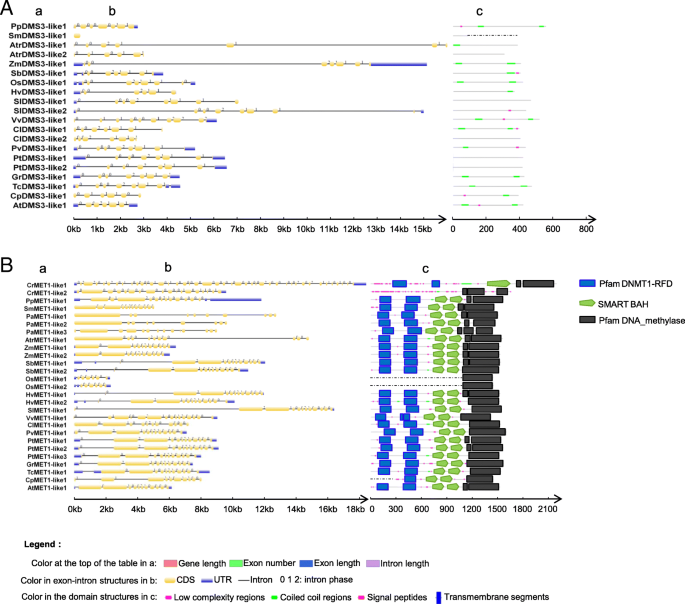
为了探讨进化期间基因结构如何发生变化,我们使用了18种代表性物种进行外部内含子结构分析。通过在物种级别检查这些甲基化相关基因的外显子内结构来开始该分析(附加文件8:表S5)。一般来说,基因长度和内含子长度差异很大(附加文件8表S5A-B, S5G-H),但大多数基因的外显子数和外显子长度是保守的(附加文件8:表S5C-F)。相关分析表明,基因长度的变化主要是由于内含子长度的变化(r = 0.79,p < 0.01). For example, in答:trichopoda,z梅斯,A. Thaliana.和诉酿酒用葡萄,大多数甲基化相关基因的基因长度和内含子长度变化了很多,但外显子长度变化很小。有趣的是,在c . reinhardtii,P. Abies.和C. Papaya.,大部分甲基化相关基因的基因长度和内含子长度变化较大,但部分甲基化相关基因的外显子长度和外显子数目也有一定变化。
然后,在蛋白质序列功能域水平研究外显子-内含子结构(图)。3.和额外的文件2:图S2)。首先,研究了参与DNA甲基化的5个基因(附加文件)2:图S2A-E)Rdr2, clsy1, dcl3, hen1和AGO4在Pol IV依赖性siRNA生物发生的过程中。外显子内部结构AGO4被保守,其他四种基因的外显子内结构的变化大,主要是因为内含子变化。这些基因的域结构基本保守。在Pol V型介导的de Novo甲基化(图。3.一个,附加文件2图S2F-M),这些基因的外显子-内含子结构变化显著KTF1.领域结构RDM1,DRD1,Morc6.和KTF1是高度保守的早期植物。人们发现DMS3只包含低复杂度区域和盘绕线圈区域。SUVH2/9在种子植物中均存在SRA、PreSET和SET结构域,但在早期植物中均仅存在这三个结构域中的一个。在染色质改变过程中(附加文件2:图S2 N-O),基因和内含子长度idn2.和IDP1改变了很多,但他们的蛋白质域很高度保守。在RNA聚合酶的亚基中(附加文件2图S2 P-T),外显子-内含子结构的变化NRPD1,NRPD / E4和nrpe5.主要是因为内含子的改变。它们的功能域是保守的,关键域存在于最古老的物种中。我们还发现在早期植物中(P. Abies.和答:trichopoda),一些关键基因的结构域发生了很大的变化。在单子叶植物中也观察到类似的趋势o.苜蓿和H.Vulgare.和双子叶植物C. Papaya..在从头CHH甲基化过程中(附加文件2的外显子-内含子结构CMT2改变了很多。的CMT2在C. Papaya.是短的,只包含部分域,和CMT2在冷杉和俗棘它们很短,不包含其他物种存在的域。
外显子-内含子结构和结构域。(A) DMS3的外显子-内含子结构和结构域。a: DMS3基因长度、外显子数、外显子长度、内含子长度综述;b:通过在线工具GSDS显示基因结构。c:域结构由在线工具SMART显示。(B) DMS3的外显子-内含子结构和结构域。a: DMS3基因长度、外显子数、外显子长度、内含子长度综述;b:通过在线工具GSDS显示基因结构。c:域结构由在线工具SMART显示。表中的颜色表示:黄色表示与平均趋势有较大的正差异; Green represents a large negative difference from the average trend. The species names are衣藻reinhardtii(Cr),Physcomitrella金属盘(PP),卷柏moellendorffii(Sm),Amborella trichopoda.(Atr),玉米(ZM),高粱二色的(SB),奥雅萨苜蓿(操作系统),Hordeum Vulgare.(高压)茄属植物lycopersicum(SL),vitis Vinifera(vv),Citrullus lanatus(CL),菜豆(PV),杨树trichocarpa(Pt),Gossypium raimondii就(gr),Theobroma Cacao.(TC),卡里卡番木瓜(CP),拟南芥(在)
其次,我们研究了负责维持DNA甲基化的基因(图。3.b,附加文件2:图S2V)。两个基因的外显子-内含子结构域(MET1和CMT3)一般是保守的。但是,对于两个重复MET1在水稻基因中,每个拷贝中只发现了DNA_methylase结构域,且蛋白质序列较短。在c . reinhardtii,CMT3含有一些低复杂性的肽段,但缺乏BAH和Chromo结构域。在P. Abies.,这些领域CMT3没有。域名CMT3在水稻上也不同于在拟南芥.至于核细胞体重塑因子DDM1(附加文件2:图S2W),外显子内和域结构在高等植物中高度保守。在组蛋白修改过程中(附加文件2图S2X-Z, S2AA),基因的外显子-内含子结构发生了很大变化,但其蛋白质结构域在早期植物中高度保守。
最后,我们研究了负责DNA去甲基化的基因(附加档案2:图S2BB-DD)。外显子,内含子和结构域DME.,ROS1.和DML3.被保守了。只有一些例外。外显子长度DME.在c . reinhardtii很长。对于域结构,DME.在c . reinhardtii除了部分领域之外,还包含大量的低复杂性区域答:芥。蛋白质序列DME.和ROS1.在P. Abies.都很短,只包含部分域。ROS1.在C. Papaya.很短,不包含任何域。
总之,外显子系统结构和功能域在早期植物中变化很大,但它们在单焦和双丁中的大多数基因被保守。注意是观察单极管中一些基因的少数域H.Vulgare.和o.苜蓿差异很大,而双子叶植物的种类C. Papaya.在大多数基因的结构域有相当大的变化。结构域改变的基因外显子发生了显著的变化,这与物种水平上外显子-内含子结构的整体变化是一致的(Additional file)8:表S5)。
甲基化相关基因的系统发育关系及纯化选择
在超过77个物种中对基因的识别已经导致了对甲基化途径起源的研究。通过外显子-内含子和结构域分析,我们知道虽然基因长度差异很大,但功能结构相对保守。接下来,我们试图探索甲基化相关基因的系统发育关系(图。4和额外的文件3.:图S3)。
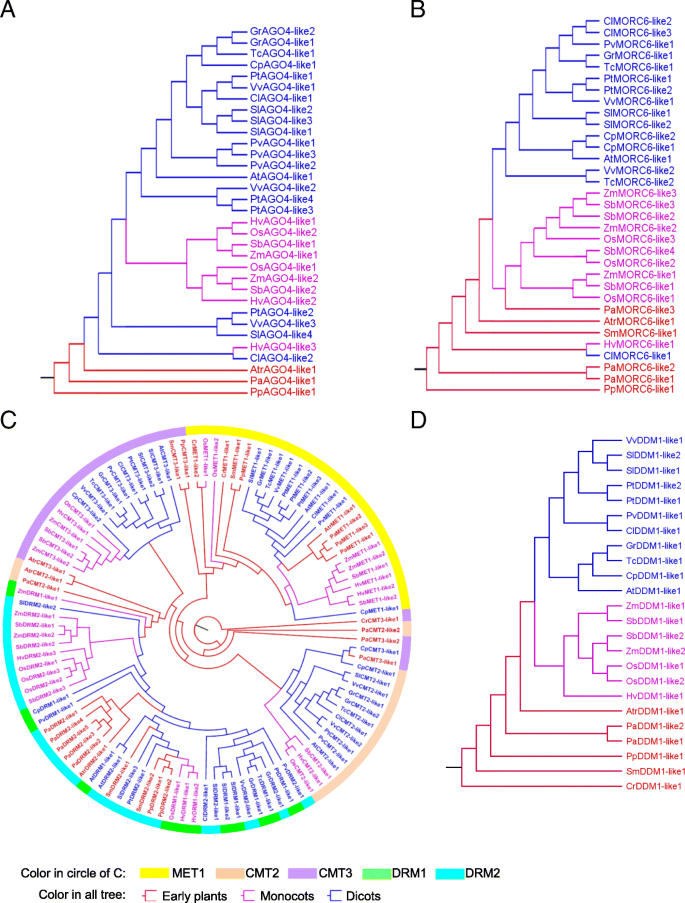
首先,我们研究了DNA甲基化相关基因(图。4a - b,附加的文件3.:图S3A-O)。DNA甲基化过程涉及许多基因,系统发育分析将它们分为三组,包括早期植物、双子叶植物和单子叶植物。这些数据的详细分析表明,在单子叶中,大多数具有多个同源性的甲基化相关基因倾向于聚集在不同的分支中,而在双子叶中,这些基因大多倾向于聚集在一个分支中。我们假设,单子叶和双子叶分化之前,单子叶甲基化相关基因可能已经复制,而双子叶中的大多数基因可能在分化之后进行复制。的Morc6.单子叶植物的基因分成三个分支Morc6.基因在双子叶植物c . lanatus,p . trichocarpa,C. Papaya.和美国lycopersicum被聚集在一个分支中。获得了类似的观察AGO4,DCL3.,DRD1和IDP1.因为两个NRPD1和nrpe1.来自NRPB1和序列相似性NRPD1和nrpe1.我们很高,我们合并了NRPD1和nrpe1.亚单位一起探索它们的进化关系。在早期的植物,NRPD1和nrpe1.被聚集在一起,但它们被聚集在一个高昂的Angiosperms中,这表明了这一点NRPD1和nrpe1.可能是从单焦点和仙人床的祖先分歧。
然后,我们研究了那些负责维持DNA甲基化的基因(图。4c).为了探索这些甲基转移酶的完整系统发育关系,我们将它们与那些负责从头甲基化的酶结合在一起,构建了一个进化树。可以看出MET1,CMT2和CMT3都很好地聚集在一起,而DRM1和DRM2在单子叶植物中簇生为两个分枝,但在双子叶和早期植物中簇生在一起。对于核小体重塑因子DDM1,我们发现DDM1将不同物种的基因聚集成三个分支(图。4d).在组蛋白修饰过程中(附加文件3.:图S3P-S),除了一些例外,JMJ14,UBP26,HDA6和SUVH4基本上分成三个分支。
最后,我们分析了去甲基化相关基因。在单子叶和双子叶中,大多数DNA糖基酶分别聚集成簇DME.分支机构ROS1.分支机构或DML3.分枝,但在早期的植物中它们是聚集在一起的(附加文件3.:图S3T)。这一结果表明,祖先的去甲基化相关基因可能在单子叶和双子叶分化后分化为不同的去甲基化相关基因。
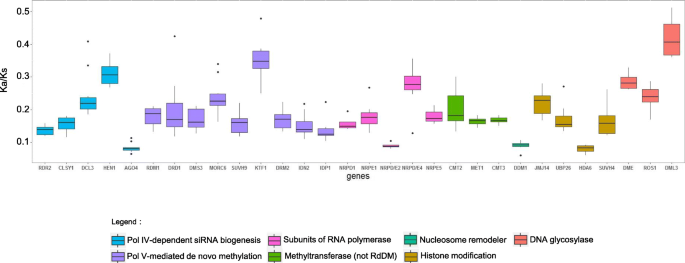
此外,我们通过计算9个双子叶中每个同源基因对的非同义位点上的非同义替换率(Ka)和同义位点上的同义替换率(Ks) (omega),研究了甲基化相关基因的选择压力A. Thaliana..我们发现所有的基因都处于负选择(图。5).这说明这些基因在双子叶植物的进化过程中是高度保守的,这也反映了DNA甲基化对双子叶植物的重要性。
不同物种中甲基化相关基因的发散表达模式
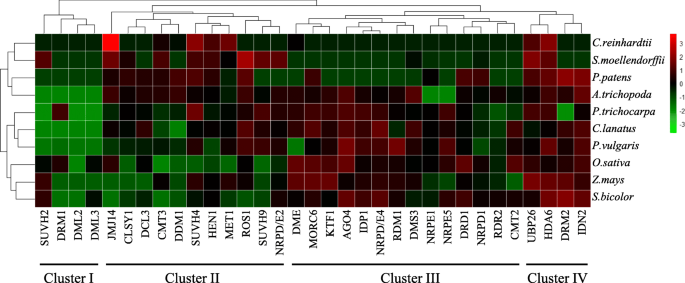
在追溯甲基化相关基因从早期植物到高等植物的进化历史的基础上,我们下一步的目标是探究甲基化相关基因在不同物种表达水平上是如何进化的。选择藻类、苔藓植物、蕨类、基部被子植物、双子叶植物和单子叶植物等10种植物进行表达模式分析。在三种早期植物中-c . reinhardtii,P. Paten.和美国moellendorffii,使用整个工厂的样品;在基底被子植物A.trichopoda分别为3个双子叶和3个单子叶。基因的归一化表达水平(TPM)以热图的形式表示(图2)。6).
首先,我们将甲基化相关基因的表达模式与物种水平进行了比较。我们可以看到早期的植物,单码和双点分别聚集成一个分支。这表明甲基化相关基因的表达模式可能在早期植物到更高植物的进化期间发生变化,以及在单子叶氏醇和双点的分化期间。在这些基因的演变中,不仅存在重复模式和基因结构,而且还存在于基因表达模式,这为我们提供了研究其他基因的起源和演化的想法。从基因水平来看,我们可以看到这些基因的表达模式可以聚集成四类。在这里,我们主要分析了Angiospers的表达模式。在簇III和IV中,表达水平很高,这可能反映了物种演化中基因表达的守恒,并解释了这些基因在植物生长和发育中的重要性。UBP26和HDA6是组蛋白调制中涉及的重要组成部分,从而影响DNA甲基化并影响表型特征[47.,48.,49.,50.].DRM2是RDDM途径的关键基因[5,12,13,14,15,16].在聚类I和聚类II中,这些基因的表达水平较低。
讨论
DNA甲基化在整个真核生物物种中很常见,对植物的生长发育至关重要,可导致基因和转座子沉默和基因组印记,并调节其他生物过程[1,5,6,7,8,9,10].已系统地研究了甲基化过程A. Thaliana..在本研究中,我们将重点研究甲基化相关基因在整个植物界的起源和进化。
甲基化相关基因的起源
DE Novo DNA甲基化过程可以从叶绿藻和胚胎症状后的繁体症和气管粥样气管粥样祖氏的祖先建立(图。1).CG和CHG甲基化的维持可以来自叶绿素和胚芽的共同祖先。假设,可以首先建立DNA甲基化,然后在甲基化的基础上,维持甲基化的维持,即,DE Novo甲基化可能比维护早期出现。但是,在我们的研究中,与维持CG和CHG甲基化相关的基因早于De Novo甲基化的大多数基因。DNA去甲基化可能源自叶绿藻和胚胎的共同祖先,其类似于CG和CHG甲基化的维持。但DML2.在十字花果植物中鉴定出来,这可能来自于DME.在十字架分化后,和DML3.在Mesangiospermae中有限,可能来自于此DME.在Mesangiospermae和Basal Angiosperm的分歧后。换句话说,Mesangiospermae和十字架植物也可能已经进化了新的去甲基化过程,这取决于DML3.和DML2..需要进一步调查来了解这两种酶的存在意味着去甲基化过程出现在甲基昔甲醚优于其他物种。对称甲基化(CG和CHG)可以通过先前出现的MET1和CMT2维持,并且大多数De Novo甲基化相关基因出现晚期。并且与其他与甲基化相关基因的对称甲基化保守没有系统的比较。在下面的研究中,我们可以探索是否可以保持对称甲基化更容易,因此进化地保守。
组蛋白改性与植物中的DNA甲基化相互作用。H3K9ME2是一个沉默的标记,70%的RDDM靶由H3K9ME2进行修改[55.].SUVH4维持H3K9me2,因此有助于DNA甲基化。H2B和H3K4me的乙酰化和泛素化是活性标记。为了维持DNA甲基化和由DNA甲基化引起的沉默状态,需要去除RdDM靶点的活性标记[20.,21.,22.,23.].这四种组蛋白修饰比从头甲基化基因进化得早,说明在从头甲基化之前,组蛋白修饰可能以其他方式影响植物的生长发育。
没有甲基化相关的基因
植物中的基因缺席(附加文件5:表S2)。在77种,73种有基因缺席。据报道,CMT3基因已经丢失了两种 -Eutrema salsugineum和链条Planisiliqua)[56].在藻类中,基因的普遍缺失可能是由于它们较晚的进化;在高等植物中,如单子叶植物和双子叶植物,这些常见的缺失事件指出了被子植物个体谱系中表观遗传基因可能丢失或获得的可能性。这些基因缺失对植物表观修饰的影响还有待进一步研究。
物种的外显子-内含子结构和甲基化水平
在本研究中,我们发现大多数物种的甲基化相关基因表现出保守的外显子长度和数量。同时,与基因功能密切相关的区域序列也高度保守。在C. Papaya.时,外显子-内含子结构和结构域发生了很大变化。其中一个原因可能是基因组组装的质量相对较差,另一个原因是这些基因在进化过程中确实发生了巨大的进化。许多基因的外显子-内含子结构和结构域发生了很大的变化o.苜蓿,如MET1(无花果。3.).水稻的MET1只有DNA甲基化酶结构域,没有BAH和DNMT1-RFD结构域,而其他物种的MET1包含所有这三种结构域。根据全基因组甲基化数据o.苜蓿和其他物种,我们可以看到CG甲基化的含量o.苜蓿高于A. Thaliana.,t .可可和诉酿酒用葡萄[57],说明MET1的改变并没有导致中CG甲基化水平的整体下降o.苜蓿.而MET1缺失关键域,CG甲基化含量居高不下的原因还有待进一步探索。
结论
DNA甲基化是植物最常见的修饰之一,涉及从头甲基化、维持甲基化和去甲基化的动态平衡。不同甲基化相关基因的起源时间并不一致,这反映了植物DNA甲基化的进化复杂性。基因序列结构域的保守性和基因的负选择性反映了甲基化过程的保守性,说明了DNA甲基化对植物的功能重要性。
方法
甲基化相关基因的鉴定
甲基化相关基因A. Thaliana.从TAIR (www.arabidopsis.org/)数据库。其他物种的蛋白序列和编码序列(CDS),以及一般特征格式3 (GFF3)和注释文件从附加文件所示的网站下载9:表S6。甲基化相关基因的蛋白质序列A. Thaliana.用作使用BLASTP程序(序列覆盖> 30%,身份> 30%和电子值截止1E-3,以识别其他76种的同源基因以鉴定同源基因。同时,将每个候选同源基因与每个物种的注释文件组合手动检查。在序列比对后手动排除未完成基因组组件的部分基因。物种分类树根据NCBI的分类数据库中的信息进行修改(https://www.ncbi.nlm.nih.gov/taxonomy/).
联合性分析
根据GFF3文件获得每个基因组中甲基化相关基因的染色体位置。当同源甲基化相关基因位于不超过一个中间基因的相同染色体中时,这些基因被定义为串联复制基因。我们使用了Maher等人所述的方法。识别大规模复制事件[58].如果两个基因位于同一个复制区段,则其侧翼的蛋白质序列在氨基酸水平上高度相似。因此,我们将甲基化相关基因定位到基因组上,并将此位置作为初始锚定位点,分别获得每个位点上下游的20个蛋白编码基因[[58].该染色体区域包含41个蛋白编码基因,使用MCScanX共线性分析(blastp E-value cutoff 1e-10) [59].
外显子内和域结构分析
使用内部Perl脚本从GFF3文件中提取每个物种中甲基化相关基因的外显子内部结构信息。基因结构分析和系统发育分析中使用的基因名称在附加文件中显示10:表S7。当其他物种中的多种同源基因对应于一种与甲基化相关基因相对应A. Thaliana.,用最长的基因计算基因长度、外显子数、外显子长度和内含子长度。外显子-内含子结构图使用在线工具GSDS (http://gsds.cbi.pku.edu.cn/).利用在线工具SMART (http://smart.embl-heidelberg.de/).采用SMART和Pfam数据库进行域预测。在外显子-内含子结构中,去除两个极值后取基因长度值的平均值。当基因长度超过平均值的1.5倍时,则判定为长。当基因长度小于平均值的一半时,则认为长度较短。这两个值称为极值。当长度在平均值的0.5 ~ 1.5倍之间时,判断为中等长度。外显子数、外显子长度和内含子长度的处理方法相同。
系统发育分析
使用Clustal X (version 2.0)比对所有甲基化相关基因的蛋白序列[60].系统发育分析采用PHYLIP软件,采用neighbor-joining (NJ)方法(bootstrap值1000)[61].使用Figtree (version 1.4.3)软件显示系统发生树。
甲基化相关基因选择压力的估计
采用Clustal X (v2.0)对甲基化相关基因的蛋白序列进行多序列比对,以PAL2NAL软件为指导,以nogap参数[62].用PAML包中的yn00程序计算每个同源基因对的每个非同义位点的非同义替换率(Ka)和每个同义位点的同义替换率(Ks) [63].根据Ka/Ks的定义,小于1的值代表消极选择或净化选择,大于1的值代表积极选择。用Ks > 2.5去除基因对,排除了饱和效应。
甲基化相关基因的表达分析
不同物种的RNA-Seq数据来源于NCBI序列读取档案(SRA)数据库(附加文件)11:表S8)。在三种早期植物中-c . reinhardtii,P. Paten.和美国moellendorffii,使用整个植物;在基底被子植物A.trichopoda, 3个双子叶和3个单子叶,叶组织。首先,使用trimmomatic-0.32软件对这些原始读取进行修剪[64].然后,使用HISAT2软件(版本2.1.0)将干净的数据映射到基因组中[65].最后,使用StringTie软件(1.3.4版)计算每个归一化为Transcripts Per Million (TPM)的基因的表达水平[66].当在物种中不存在甲基化相关的基因时,不能转录基因,我们记录了基因的表达为0;当物种中有多个同源拷贝时,将同源副本的表达和记录为物种中的表达水平。使用R包Pheatmap以Heatmap的形式显示基因(TPM)的归一化表达水平。
数据和材料的可用性
本研究中分析的所有数据都包含在本发布的文章及其补充信息文件中。
缩写
- CD:
-
编码序列
- GFF3:
-
通用特性格式3
- 卡:
-
每个非同义站点的非同义替换
- Ks:
-
每个同义地点的同义替换
- RDDM:
-
RNA-directed DNA甲基化
- sirnas:
-
小干扰rna
- WGD:
-
全基因组复制
参考
- 1.
DNA甲基化与表观遗传学。植物学报2004;55:41-68。
- 2。
Goll MG, best TH。真核生物的胞嘧啶甲基转移酶。植物学报2005;74:481-514。
- 3.
Feng S, Cokus SJ, Zhang X, Chen PY, Bostick M, Goll MG, Hetzel J, Jain J, Strauss SH, Halpern ME, et al.;动植物甲基化模式的保存与分化。美国国家科学研究院2010;107:8689-94。
- 4.
关键词:甲基化,全基因组,进化分析,真核生物科学。2010;328:916-9。
- 5。
Matzke Ma,Mosher Ra。RNA定向的DNA甲基化:一种增加复杂性的表观遗传途径。NAT Rev Genet。2014; 15:394-408。
- 6.
植物有性生殖过程中的DNA甲基化重编程?趋势麝猫。2010;26:394-9。
- 7.
Bauer MJ, Fischer RL。胚乳中的基因组去甲基化和印记。植物生态学报。2011;14:162-7。
- 8.
陈志强,陈志强。DNA甲基化模式的研究进展。生物化学学报2012;81:97-117。
- 9.
Diez CM,Roessler K,Gaut BS。表观生物学和植物基因组进化。CurrOp植物BIOL。2014; 18:1-8。
- 10.
听到E,Martienssen Ra。转基因表观遗传遗传:神话和机制。细胞。2014; 157:95-109。
- 11.
钟旭。比较表观基因组学:理解DNA甲基化进化的有力工具。新植醇。2016;210:76 - 80。
- 12.
曹X, Aufsatz W, Zilberman D, Mette MF, Huang MS, Matzke M, Jacobsen SE。DRM和CMT3甲基转移酶在rna导向DNA甲基化中的作用。咕咕叫医学杂志。2003;13:2212-7。
- 13。
Henderson IR,Deleris A,Wong W,Zhong X,Chin Hg,Horwitz Ga,Kelly Ka,Pradhan S,Jacobsen SE。DE Novo胞嘧啶甲基转移酶DRM2在拟南芥中的RNA定向的DNA甲基化期间需要完整的UBA结构域和催化突变的寄生虫DRM3。Plos Genet。2010; 6:E1001182。
- 14。
Stroud H, Greenberg MV, Feng S, Bernatavichute YV, Jacobsen SE。综合分析沉默突变体揭示了拟南芥甲基组的复杂调控。细胞。2013;152:352 - 64。
- 15.
Law JA, Jacobsen SE。建立,维持和修改动植物DNA甲基化模式。Nat Rev Genet. 2010; 11:204-20。
- 16.
MA L,Hatlen A,Kelly LJ,Becher H,Wang W,Kovarik A,Leitch IJ,Leitch Ar。在RNA定向的DNA甲基化(RDDM)途径中,在植物谱系中是陆地植物谱系中的独特性。基因组Biol Evol。2015; 7:2648-62。
- 17.
曹旭,杨志强。DRM和CMT3甲基转移酶基因对不对称和CpNpG甲基化的位点特异性控制。美国国立科学院科学技术研究所2002;99(增刊4):16491-8。
- 18.
拟南芥核小体重组酶DDM1允许DNA甲基转移酶访问含有h1的异染色质。细胞。2013;153:193 - 205。
- 19.
snf2家族蛋白:任何场合的染色质重塑者。2011; 15:649-56。
- 20。
拟南芥H3K4me2/3去甲基化酶JMJ14突变通过减少转基因转录抑制转录后基因沉默。植物细胞。2012;24:3603-12。
- 21.
Liu X,Luo M,Wu K.HDA6介导的组蛋白修饰和DNA甲基化的表观遗传相互作用。植物信号表现。2012; 7:633-5。
- 22.
Sridhar VV,Kapoor A,Zhang K,Zhu J,周T,Hasegawa PM,Bressan Ra,Zhu JK。通过组蛋白H2B脱氮控制DNA甲基化和异度沉默。自然。2007; 447:735-8。
- 23.
Johnson LM, Bostick M, Zhang X, Kraft E, Henderson I, Callis J, Jacobsen SE。SRA甲基胞嘧啶结合区域连接DNA和组蛋白甲基化。咕咕叫医学杂志。2007;17:379 - 84。
- 24.
Herr AJ,Jensen M,Dalmay T,BaulCombe DC。RNA聚合酶IV引导沉默内源性DNA。科学。2005; 308:118-20。
- 25.
Onodera Y, Haag JR, Ream T, Costa Nunes P, Pontes O, Pikaard CS。植物核RNA聚合酶IV介导siRNA和DNA甲基化依赖的异染色质形成。细胞。2005;120:613-22。
- 26.
Haag Jr,Pikaard CS。多管RNA聚合酶IV和V:用于植物基因沉默的非编码RNA的输送器。NAT Rev Mol Cell Biol。2011; 12:483-92。
- 27.
Tucker SL, Reece J, Ream TS, Pikaard CS。植物多亚基RNA聚合酶IV和V的进化史:通过全基因组和节段基因复制、逆转录转位和谱系特异性亚功能化的亚基起源。冷泉Harb Symp Quant Biol. 2010; 75:285-97
- 28.
Haag JR, Ream TS, Marasco M, Nicora CD, Norbeck AD, Pasa-Tolic L, Pikaard CS。pol IV、pol V和RDR2的体外转录活性表明,pol IV和RDR2偶联在植物RNA沉默中合成dsRNA。摩尔细胞。2012;48:811-8。
- 29.
Smith LM, Pontes O, Searle I, Yelina N, Yousafzai FK, Herr AJ, Pikaard CS, Baulcombe DC。拟南芥中与核RNA沉默和沉默信号在细胞间传播相关的SNF2蛋白。植物细胞。2007;19:1507-21。
- 30.
Xie Z, Johansen LK, Gustafson AM, Kasschau KD, Lellis AD, Zilberman D, Jacobsen SE, Carrington JC。植物小RNA通路的遗传和功能多样化。公共科学图书馆杂志。2004;2:e104。
- 31.
关键词:小RNA,甲基化,稳定性细胞研究》2012;22:624-36。
- 32.
Havecker Er,Wallbridge Lm,Hardcastle TJ,布什MS,Kelly Ka,Dunn RM,Schwach F,Doonan JH,BaulCombe DC。拟南芥RNA定向的DNA甲基化Argonona基于它们的表达和与靶基因座的相互作用来功能偏离。植物细胞。2010; 22:321-34。
- 33.
董玲,刘敏,方洋洋,赵建华,何晓峰,应晓波,张洋洋,谢强,蔡宁华,郭海生。DRD1-pol v依赖于外源消音器的自沉默限制了内源性靶基因的非细胞自主沉默。植物j . 2011; 68:633-45。
- 34.
Zhong X, Hale CJ, Law JA, Johnson LM, Feng S, Tu A, Jacobsen SE。DDR复合物促进RNA聚合酶V与启动子和进化年轻转座子的全球关联。《自然结构与生物》2012;19:870-5。
- 35.
Moissiard G, Cokus SJ, Cary J, Feng S, Billi AC, Stroud H, Husmann D, Zhan Y, Lajoie BR, McCord RP, et al.;异染色质凝聚和基因沉默所需的MORC家族atp酶。科学。2012;336:1448-51。
- 36.
寄生原生生物和其他真核生物中转录因子和染色质蛋白的比较基因组学。国际寄生虫学杂志。2008;38:1-31。
- 37。
刘志伟,邵春荣,张建军,周建新,张文文,李丽,陈胜,黄宏文,蔡涛,何新军。SET结构域蛋白SUVH2和SUVH9是pol V占据rna导向DNA甲基化位点所必需的。公共科学图书馆麝猫。2014;10:e1003948。
- 38。
Bies-Etheve N, Pontier D, Lahmy S, Picart C, Vega D, Cooke R, Lagrange T. rna导向的DNA甲基化需要一个与ago4相互作用的SPT5延伸因子家族成员。EMBO众议员2009;10:649-54。
- 39。
张cj,宁yq,张sw,陈q,邵克,郭yw,周jx,李l,陈某,他xj。IDN2及其副病剂形成RNA定向的DNA甲基化所需的复合物。Plos Genet。2012; 8:E1002693。
- 40。
朱镕基JK。DNA糖基化酶介导的活性DNA去甲基化。Annu Rev Genet, 2009; 43:143-66。
- 41。
Gong ZZ, Morales-Ruiz T, Ariza RR, roldan - arjoa T, David L, Zhu JK。ROS1是拟南芥转录基因沉默的抑制因子,编码DNA糖基化酶/裂解酶。细胞。2002;111:803-14。
- 42。
Choi Y,Gehring M,Johnson L,Hannon M,Harada JJ,Goldberg RB,Jacobsen SE,Fischer RL。Demeter是DNA糖基酶结构域蛋白质,是拟南芥中的胚乳基因印迹和种子活力所必需的。细胞。2002; 110:33-42。
- 43。
Tedeschi F, Rizzo P, Huong BTM, Czihal A, Rutten T, Altschmied L, Scharfenberg S, Grosse I, Becker C, Weigel D,等。转录因子的效应器是与拟南芥基因组DNA甲基化相关的新型植物特异性调节因子。新植醇。2019;221:261 - 78。
- 44。
王志强,王志强,王志强,等。小麦甲基转移酶1基因的克隆及表达分析。BMC基因组学。2014;15:922。
- 45.
Huang Y, Kendall T, Forsythe ES, Dorantes-Acosta A, Li S, Caballero-Perez J, Chen X, Arteaga-Vazquez M, Beilstein MA, Mosher RA。RNA聚合酶IV和v的古代起源和最近的创新。
- 46.
Bewick AJ,Niederhuth CE,Ji L,Rohr Na,Griffin Pt,Leebens-Mack J,Schmitz RJ。植物中色谱酶和基因体DNA甲基化的演变。基因组Biol。2017; 18:65。
- 47.
Lu F,Cui x,张S,刘C,Cao X.JMJ14是拟南芥中的H3K4去甲基化酶调节开花时间。细胞res。2010; 20:387-90。
- 48.
Schmitz RJ, Tamada Y, Doyle MR, Zhang X, Amasino RM。在拟南芥中,组蛋白H2B去泛素化是转录激活开花位点C和调控开花的必要条件。植物杂志。2009;149:1196 - 204。
- 49.
Tanaka M,Kikuchi A,Kamada H.拟南芥组蛋白脱乙酰酶HDA6和HDA19有助于萌发后的胚胎性质。植物理性。2008; 146:149-61。
- 50.
组蛋白甲基化控制着端粒酶独立的端粒延长。Dev杂志。2007;306:838-46。
- 51.
宋庆林,陈建军,程建军,等。古多倍体大豆的基因组序列。大自然。2010;463:178 - 83。
- 52.
Velasco R,Zharkikh A,Comuourtit J,Dephra A,Cestaro A,Kalyanaraman A,Fontana P,Bhatnagar Sk,Troggio M,Pruss D等人。驯化苹果的基因组(Malus x Domestica Borkh)。NAT Genet。2010; 42:833-9。
- 53.
唐华,王旭,王志强。被子植物基因组的比较揭示了单子叶植物谱系中的早期多倍体。美国国家科学研究院2010;107:472-7。
- 54.
贾荣杰,贾荣杰,贾荣杰。通过染色体复制事件的系统发育分析揭示被子植物基因组进化。大自然。2003;422:433-8。
- 55.
张旭,张志强,张志强,张志强。组蛋白H3赖氨酸9甲基化与CHG DNA甲基化的全基因组关联拟南芥.Plos一个。2008; 3:E3156。
- 56.
Bewick Aj,Ji L,Niederhuth CE,Willing Em,Hofmeister Bt,Shi X,Wang L,Lu Z,Rohr Na,Hartwig B等人。基因体DNA甲基化的起源和进化后果。Proc Natl Acad Sci U S A. 2016; 113:9111-6。
- 57.
Niederhuth CE,Bewick Aj,Ji L,Alabady Ms,Kim Kd,Li Q,Rohr Na,Rambani A,Burke Jm,Udall Ja等。在高血管培养物内广泛的DNA甲基化的自然变化。基因组Biol。2016; 17:194。
- 58.
王志强,王志强,王志强,等。拟南芥microRNA家族的基因克隆与表达分析。基因组研究》2006;16:510-9。
- 59.
Wang Y, Tang H, Debarry JD, Tan X, Li J, Wang X, Lee TH, Jin H, Marler B, Guo H, et al. .MCScanX:基因共线性和共线性的检测和进化分析工具包。核酸学报2012;40:e49。
- 60。
Larkin Ma,Blackshields G,Brown NP,Chenna R,McGettigan Pa,McWilliam H,Valentin F,Wallace Im,Wilm A,Lopez R等人。Clustal W和Clustal X 2.0版。生物信息学。2007; 23:2947-8。
- 61。
Felsenstein J. Phylip-Phylogy I推理包(版本3.2)。克莱斯。1989; 5:164-6。
- 62。
Suyama M,Torrent D,Bork P. Pal2nal:蛋白质序列对准的鲁棒转化为相应的密码子对齐。核酸RES。2006; 34:W609-12。
- 63。
杨Z.PAML 4:通过最大可能性进行系统发育分析。mol Biol Evol。2007; 24:1586-91。
- 64。
Bolger Am,Lohse M,Usadel B. Trimmomatic:用于Illumina序列数据的灵活修剪器。生物信息学。2014; 30:2114-20。
- 65。
HISAT:低内存要求的快速拼接对准器。Nat方法。2015;12:357-60。
- 66。
Pertea M, Pertea GM, Antonescu CM, Chang TC, Mendell JT, Salzberg SL. StringTie能够从RNA-seq读取改进的转录组重建。生物科技Nat》。2015;33:290-5。
确认
不适用
资金
国家自然科学基金(31801405)和中国科协青年英才资助项目资助。资助机构在研究的设计、数据的收集、分析和解释以及手稿的撰写中没有作用。
作者信息
隶属关系
贡献
MW和XZ构思了这个实验。LP、LZ、JL、CS、PQ进行生物信息学分析,并对结果进行分析。LP撰写稿件草稿,MW和LT进行修改。所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
附加文件
额外的文件1:
图S1。包含20个甲基化相关基因上下游相邻基因的染色体片段共线分析。(A)共线性RDR2.不同物种的基因。(B)共线性AGO4不同物种的基因。(c)联合性Morc6.不同物种的基因。(D)共线性DRM2不同物种的基因。(e)联合性UBP26不同物种的基因。(F)共线性NRPD1不同物种的基因。(G)共线性nrpe5.不同物种的基因。(H)的共线性MET1不同物种的基因。物种名称是衣藻reinhardtii(Cr),Physcomitrella金属盘(PP),卷柏moellendorffii(Sm),Amborella trichopoda.(Atr),玉米(ZM),高粱二色的(SB),奥雅萨苜蓿(操作系统),Hordeum Vulgare.(高压)茄属植物lycopersicum(SL),vitis Vinifera(vv),Citrullus lanatus(CL),菜豆(PV),杨树trichocarpa(Pt),Gossypium raimondii就(gr),Theobroma Cacao.(TC),卡里卡番木瓜(CP),拟南芥(在)。(PDF 584 kb)
额外的文件2:
图S2。外显子-内含子结构和结构域。(A-DD)的外显子-内含子结构和结构域Rdr2, clsy1, dcl3, hen1, ago4, rdm1, drd1, morc6, suvh2, suvh9, ktf1, drm1, drm2, jmj14, ubp26, hda6, suvh4, idn2, idp1, nrpd1, nrpe1, nrpd / e2, nrpd / e4, nrpe5, cmt2, cmt3, ddm,DME,ROS1和DML3..a:基因长度、外显子数、外显子长度和内含子长度综述。表格中的绿色和黄色表示具有极端值的基因。b:通过在线工具GSDS显示基因结构。c:域结构由在线工具SMART显示。物种名称是衣藻reinhardtii(Cr),Physcomitrella金属盘(PP),卷柏moellendorffii(Sm),Amborella trichopoda.(Atr),玉米(ZM),高粱二色的(SB),奥雅萨苜蓿(操作系统),Hordeum Vulgare.(高压)茄属植物lycopersicum(SL),vitis Vinifera(vv),Citrullus lanatus(CL),菜豆(PV),杨树trichocarpa(Pt),Gossypium raimondii就(gr),Theobroma Cacao.(TC),卡里卡番木瓜(CP),拟南芥(在)。(PDF 4727 KB)
额外的文件3:
图S3。用邻居连接法构建18个物种甲基化相关基因的系统发育树。(A-S) RDR2、DCL3、CLSY1、HEN1、RDM1、DRD1、DMS3、SUVH2/9、KTF1、JMJ14、UBP26、HDA6、SUVH4、IDN2、IDP1、NRPD1_NRPE1、NRPD/E2、NRPD/E4、NRPE5、DME_ROS1_DML3基因的进化树。(PDF 824 kb)
额外的文件4:
表S1。77个物种甲基化相关基因的基因ID。(XLSX 13399 kb)
额外的文件5:
表S2。在77种,甲基化相关基因的数量,单拷贝基因的数量,多拷贝基因的数量,缺失基因的数量,以及测序物种的染色体倍增。(XLSX 58 KB)
额外的文件6:
表S3。6种多倍体植物中甲基化相关基因的归一化数、单拷贝基因的归一化数、多拷贝基因的归一化数、缺失基因的归一化数以及被测序物种的染色体倍性。(XLSX 15 KB)
额外的文件7:
表S4。本文综述了5个物种中甲基化相关基因的复制模式。(XLSX 44 kb)
额外的文件8:
表S5。18个物种甲基化相关基因的基因长度、外显子数、外显子长度、内含子长度。答:18个物种甲基化相关基因的基因长度。B:基因长度极端值的基因数。C: 18个物种中甲基化相关基因的外显子数。D:外显子数目极端的基因数。E: 18个物种甲基化相关基因的基因长度。F:外显子长度极端值的基因数量。G: 18个物种甲基化相关基因内含子长度。H:内含子长度极端值的基因数。(XLSX 70 kb)
额外的文件9:
表S6。概述用于鉴定甲基化相关基因的77个测序基因组。(XLSX 15 KB)
额外的文件10:
表S7。用于基因结构分析和系统发育分析的基因名称。(XLSX 72 kb)
额外的文件11:
表S8。本研究中使用的RNA-Seq数据的SRA登录数。(XLSX 31 kb)
权利和权限
开放访问本文根据创意公约署署署的条款分发了4.0国际许可证(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Pei,L.,张,L.,Li,J.等等。追溯植物甲基化相关基因的起源和进化史。BMC植物BIOL.19,307(2019)。https://doi.org/10.1186/s12870-019-1923-7
收到了:
接受:
发表:
关键词
- DNA甲基化
- 基因的起源
- 重复
- 基因结构
- 系统发育分析