摘要
背景
APETALA2- 样基因编码特定的植物的转录因子,其中一些具有一个microRNA172(miR172)结合位点。miR172及其目标euap2.基因参与了许多植物的相变和花器官发育过程。然而,miR172的作用及其目标AP2.基因在很大程度上是未知的芸苔属植物显著(显著).
结果
在这项研究中,19euap2.和4个miR172基因鉴定显著基因组。序列分析表明17euap2.Bna-miR172在3 '编码区以基因为靶点。euap2.S被分为5大类甘蓝型.这种分类与外显子-内含子结构和基序组织一致。对非同义替代率和同义替代率的分析表明euap2.基因经历了净化选择。全基因组复制(WGD)或节段复制事件在扩增中起主要作用euap2.基因家庭。CIS-COMMENTOVATION元素(CRE)分析表明euap2.S参与了对光,激素,应力和发育过程的反应,包括昼夜宿主和熟练表达。MiR172目标的表达分析euAP2s在9种不同的组织中表现出不同的时空表达模式。大多数euap2.基因在花器官中高度表达,表明它们在花发育过程中具有特殊的功能。BnaAP2-1,BnaAP2-5和BNATOE1-2基于RNA-SEQ和QRT-PCR的早期开花材料具有更高的开花材料表达水平,表明它们可以充当花抑制剂。
结论
综上所述,分析了其进化、结构、组织特异性和表达euap2.基因在甘蓝型.基于RNA-SEQ和实验数据,euap2.可能与花的发育有关。三个euap2.基因(BnaAP2-1,BnaAP2-5和BNATOE1-2)可能被认为是花的抑制因子。本研究的结果为miR172 /的进一步功能表征提供了见解euap2.模块甘蓝型.
背景
MicroRNA是一类内源性,小的非编码RNA(20-24个核苷酸),具有在基因表达后调节中的关键作用[1].据报道,植物miRNAs参与了许多生物过程,如生长素信号转导[2、花期调节[3.,叶片发育[4和应激反应[5,6].最近的研究也表明,miRNAs可以通过其相应的靶基因调控多种植物的发育时间和花的诱导[7,8,9,10,11,12,13].特别是,两种高度保守的microrna,即miR156和miR172,已经被确定为营养阶段变化的主要调控因子[14,15,16,17,18,19].
首先是通过拟南芥中的小RNA测序获得miR172,并且由于其高保性而在许多植物中发现了[20.].miR172家族是拟南芥中MIR172a-e基因编码的年龄通路的主要组成部分。该家族在拟南芥中的靶基因有6个APETALA-2 (AP2)类型的基因:APETALA2(AP2.),饮食目标1(TOE1,TOE2, 和脚趾3),SCHLAFMUTZE (SMZ)和SCHNARCHZAPFEN (SNZ).AP2.参与花器官的形成,其他5个ap2样基因主要是花抑制因子[3.,21,22].AP2蛋白有一个或多个AP2结构域,分别由60-70个高度保守的氨基酸(aa)和AP2结构域的N-端YRG和c -端RAYDmotifs组成[23,24,25].
miR172及其目标基因在开花时间和花器官分化中发挥关键作用。在拟南芥中,过表达所有miR172靶基因以外TOE3导致花期延迟[18,22,24].相比之下,Toe1Toe2.双突变体,toe1toe2smzsnz四倍的突变和toe1toe2toe3smzsnzap2.六组突变均表现出开花早,且随着突变基因数量的增加开花时间提前[26,27].一方面,这一结果显示了功能冗余AP2.基因;另一方面,它证实了AP2.基因负调控拟南芥开花时间。在玉米突变体中glossy15(gl15),由于以mir172为靶点,幼叶表现出早熟的成叶特征GL15在青少年向成年人的转变过程中发挥了作用[28].在拟南芥中,过度表达miR172不仅加速了开花,而且导致了异常的花发育,包括花瓣的丢失和萼片向心皮的同源转化[3.,22,26],其表型类似于ap2突变体。当在miR172结合位点引入6碱基突变时AP2.为了产生一个miR172抗性基因,需要产生大量的花瓣和退化的雄蕊[22].水稻中过表达miR172b延缓了小穗分生组织向花分生组织的转变,导致花器官的同源性转化[29].烟草中MiR172的过度表达也导致萼片转化为花瓣,而且35S :: AP2M3植物表现出花的图案缺陷,包括大量的花瓣、雄蕊和心皮的增殖[30.].在大麦,Cleistogamy1(Cly1)取代了miR172靶位点的同义核苷酸,导致开花失败和封闭受精表型[31].
芸苔属植物显著是世界上第二大油料作物。该物种是一个异源多倍体(AACC, 2n = 4x = 38),由天然的种间杂交进化而来芸苔属植物拉伯(基因组AA,2n = 20)和Brassica oleracea(基因组CC, 2n = 18) [32].开花代表从营养期向生殖期的过渡。适当的开花是保证繁殖成功的关键,决定了生育期、产量和种子质量显著.尽管扮演着AP2.在拟南芥中,miR172的作用及其靶基因已经被很好地描述AP2.确定开花和花卉发展的基因仍不清楚显著.虽然MiRNA172C-Apetala2-1节点已被鉴定为共同豆类中氮固定的关键调节器[33,之间的相互作用AP2.和MiR172甘蓝型还没明白。因此,对miR172/进行全基因组功能分析将是有趣的euap2.在甘蓝型.本研究系统分析了miR172成员的染色体位置、进化关系、复制事件、共线性、基因结构、保守基序和上游顺式元件euap2.靶基因。还进行了RNA测序(RNA-SEQ)和定量实时PCR(QRT-PCR),以分析表达模式euAP2s在不同的组织和发育阶段。
结果
的识别euap2.和miR172基因显著
以拟南芥中8条经鉴定的AP2蛋白序列作为查询序列,对拟南芥中的AP2蛋白进行了鉴定euap2.基因家庭显著基因组使用BLASTP搜索,e值≤10−10和19个推定euap2.鉴定了家庭基因。然后,使用SMART,PFAM,CDD和HMMER数据库来验证AP2域,并且所有19个预测蛋白质都有一个或两个AP2域。如表所示1,我们给显著euAP2根据他们最近的拟南芥同源物。
分析了染色体位置、蛋白质序列长度、分子量、等电点和亚细胞定位。euAP2蛋白的大小从103 (BnaTOE1-5)到454 (BnaTOE2-3)不等。预测分子量为11.00 ~ 49.63 kDa, PI值为6.02 ~ 9.42。预测的亚细胞定位结果显示,9个euAP2蛋白位于核区域,4个蛋白位于细胞质区域,另外6个蛋白位于核和细胞质区域。这些结果表明,不同的euAP2家族蛋白可能具有不同的功能。
4个假定的miR172家族成员(Bna-miR172a, Bna-miR172b, Bna-miR172c和Bna-miR172d)显著在miRbase中预测,它们的前体和成熟序列显示在附加文件1:表S1。miR172前体和成熟序列的多序列对准在附加文件中显示8:图S1a, b.四个成熟的miR172序列除了第一个碱基外都高度保守,而前体序列存在分歧。Mfold预测了Bna-MIRl72基因转录本的二级结构8:图S1c),四个前体序列形成了不同的茎环结构,这可能导致成员之间的功能差异。
miRNAs的目标预测是基于植物中miRNAs与其目标基因的高度同源性[34].两个目标预测服务器,即Tap r和psrnatarget用于识别MiR172的潜在目标显著.共有17个携带miR172的miRNA响应元件(MRE)的靶点被预测,miRNA结合位点的位置显示在表中1.比较Bna-miR172成熟序列与euap2.序列显示,17个euap2.s包含与Bna-miR172成熟序列互补的序列,有1到3个碱基错配(图。1a).所鉴定的Bna-miR172靶位点均位于编码区euap2.基因。
染色体的分布euap2.基因和Bna-miR172前体如图所示。1b。19euAP2s4个Bna-miR172s位于13条已知染色体上,4个位于支架上。这些基因和miRNA前体在细胞中分布不均匀B .napus亚基因组,有6和9euap2.分别位于A和C基因组的基因。其他四个euap2.基因定位到a01 -随机,a09 -随机,ann -随机和cnn -随机。Bna-miR172基因分布于A03、A09、C02、C03和Ann_random。但是,没有euap2.基因和BNA-miR172前体位于A01,A04,A05,A06,A08和CO1染色体上。在染色体上没有基因和对亚基因组的不均匀分布euap2.基因在进化过程中澄清了它们的易位。
系统发育与选择压力分析euap2.和bna-mir172基因
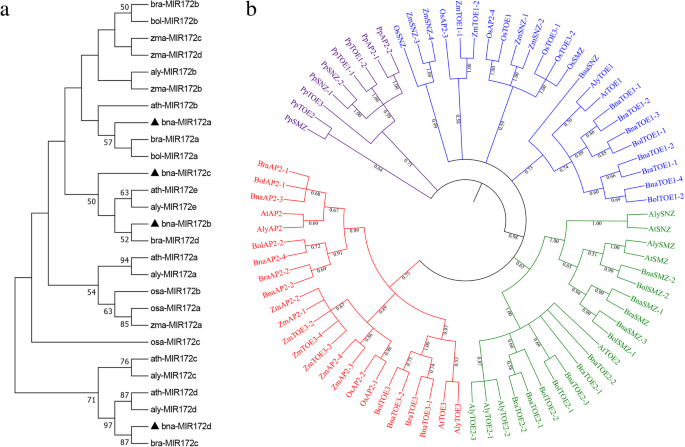
来评估动物的进化关系AP2.和miR172基因成员,利用miR172前体序列和miR172的氨基酸序列建立了一个未根邻连接系统发育树AP2.从拟南芥基因,显著,b·拉伯,b . oleracea,水稻,玉米,苔藓和拟南芥lyrata.然而,在苔藓中未发现miR172基因。根据系统发育分析,其他7种植物的MIR172基因可分为4类。每组至少有一个来自拟南芥的MIR172基因。2一个)。AP2.基因from the eight species were assigned to four groups according to the phylogenetic topology (Fig.2b)。BnaAP2基因首先聚集在一起AP2.S来自于显著,其次是拟南芥和答:lyrata,形成了一个特殊的十字花科分支。来自水稻的EuAP2蛋白与玉米的亲缘关系更密切,并形成了单子叶分支。双子叶和单子叶植物中的EuAP2共同形成较大的枝支。苔藓簇生成一个独立的枝。结果表明,euAP2蛋白可能在单子叶植物和双子叶植物中高度保守,在苔藓植物和维管植物分裂后出现分化。
我们还预测了AP2蛋白在拟南芥中的副骨和正交关系,显著,b·拉伯和B. oleracea。七对副蛋白酶被识别出来显著,在物种间鉴定出11对推测的同源蛋白。的非同义/同义替代比(Ka/Ks)为基因重复后基因分化的驱动力euap2.计算基因对(表2).除了一对基因(BnaTOE1-1 / BraTOE1-2),其中Ka/Ks为> 1,占大多数euap2.基因对的Ka/Ks < 1,表明AP2.基因家族在进化期间经历了强烈的净化选择性压力。
我们使用MCScanX程序检测了五种类型的基因重复,即单leton、分散、近端、串联和WGD或片段重复。没有串联重复的事件euap2.发现基因家族,鉴定了五个分散和11个分段重复事件(表3.).这些结果表明euap2.基因可能是由基因重复产生的,而WGD或片段重复事件是基因重复的主要驱动力euap2.进化(35].
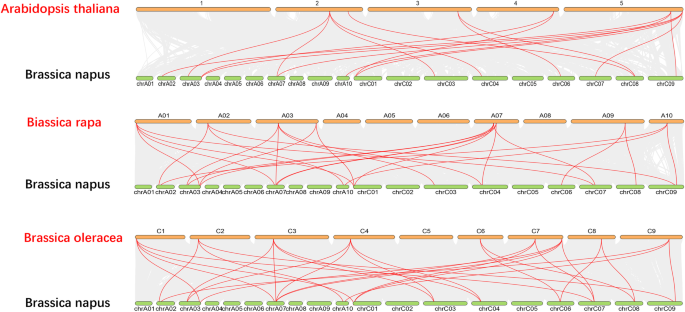
进一步推断出的系统发育关系BnaAP2家族,我们构建了三个比较共系图显著与拟南芥有关, b·拉伯和b . oleracea(无花果。3.).共13BnAP2基因表现出同系关系拟南芥,紧随其后的是b·拉伯(13)和b . oleracea(13)。有趣的是,共线对(13BnAP2之间的基因显著和其他三个种,表明这些同源对可能在分化前就已经存在(附加文件2:表S2)。
结构组织与保守域分析euap2.基因
为了进一步了解euap2.我们将它们的编码序列与基因组序列对齐(附加文件9:图S2)。的euap2.基因被分为五个亚组。在19个基因中评估外显子数(4至10),内含子长度和未转过来的区域(UTR)的差异,表明AP2.型基因虽然在同一亚家族中,但可能具有不同的功能。然而,位于同一分支末端的基因总是表现出相似的结构。各类群的外显子-内含子结构与系统发育关系一致且保守,但部分同源基因存在差异。
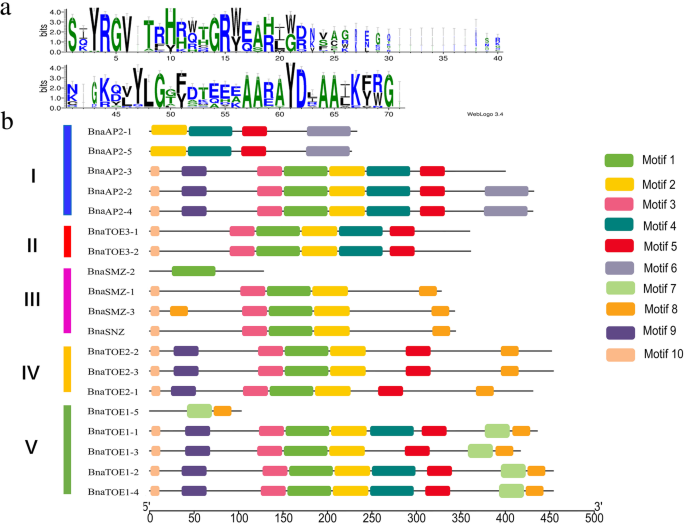
模因被用来识别假定的保守母题euap2.基因(图。4).认识到10个最保守的基序,图案的长度范围为11至50个氨基酸。图案注释列于其他文件中3.:表S3。图案1和4作为AP2-ERF域注释,其他主题具有未知的功能。保守的AP2结构域是由WebLogo创建的,并且所有Euap2蛋白质包含一个或两个图案1和4,这进一步证实了表中提出的识别结果1.同一类群中的大多数euAP2s含有相似的基序组成。同一亚群中ap2型蛋白的基序数目和排列相似,表明ap2型蛋白的功能保守在一个特定亚家族内。但也有一些例外:BnaAP2-1和BnaAP2-5有4个母序(母序2、4、5、6);BnaAP2-3有7个基序;BnaAP2-2 / BnaAP2-4包含8个基序;BnaSNZ只有一个AP2-ERF基序;BnaTOE1-5只有2个基序,远少于同亚家族其他蛋白的9个基序。基序6和基序7分别在支系I和支系V中具有独特性,可能与BnaAP2和BnaTOE蛋白的功能有关。这些特定的母题和母题构成可能导致了语篇的功能分化euap2.基因。
组织假定的主题euap2.基因。一个中共识AP2域序列序列的序列标识视图显著。每个位置上字母的高度(氨基酸)代表保守程度。b由MEME鉴定的EUAP2蛋白的推定基序。编号,彩色盒子代表不同的推定图案。i to v从附加文件中指出系统发育子组9S2:图
总的来说,同类群中euAP2成员相似的基因结构和保守的基序组成,以及系统发育分析的结果,进一步证实了进化分类的可靠性。
Bna-miR172的顺式调节元件(cre)分析BnaAP2基因
时间和空间基因表达是通过在启动子中存在不同的顺式调节元素的调节[36].的转录起始位点(TSS)上游1000 bp区域,了解其表达差异euap2.提取Bna-miR172前体序列上游1500 bp,利用PlantCARE数据库分析cre(附加文件4:表S4)。将已鉴定的CREs按其功能分为5大类:光响应元件、激素响应元件、应激响应元件、生长发育响应元件等。所有启动子序列均含有TATA-box和CAAT-box。多数基因中含有G-Box、GAG-motif、Box 4、AAAC-motif和Box 1等光响应元件。CRE如ERE、ABRE TGA element、GARE-motif、P-box、CGTCA和TGACG motif对乙烯、脱落酸、生长素、赤霉素和MeJA等多种植物激素均有响应。ARE、MBS、TC-rich repeats、LTR、Box-W1和HSE等应激响应元件已被报道用于响应各种非生物胁迫。此外,其他生长发育响应元件,如CCAAT-box, 5 ' UTR pyy -rich stretch和CCGTCC-box被发现与胚乳和分生组织的表达和昼夜节律控制相关。一般而言,Bna-miR172的表达及其靶点euap2.基因可受不同环境和内部发育因素的调控。
不同的表达谱euap2.基因
不同组织的基因表达模式
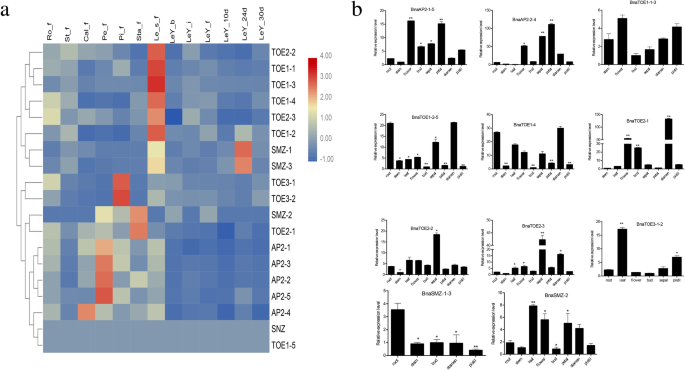
调查推定的作用euap2.基因,所有的表达模式euAP2s基于RNA-seq数据在不同组织中进行分析(图。5一个)。euAP2s用层次聚类法将其分为7类。五个AP2.第一组的基因,即,BnaTOE2-2,BNATOE1-1,BNATOE1-3,BNATOE1-4和BnaTOE2-3,在幼苗阶段的幼叶中的高水平表达。第二组有三个基因(BNATOE1-2,BnaSMZ-1, 和BnaSMZ-3),从苗期开始到开花后24 d,在幼叶中均有高表达。的转录euap2.第三~第六组基因在雌蕊、雄蕊、萼片和花萼中富集,表明它们在花器官发育中具有特殊的功能。BnaSNZ和BNATOE1-5第七组在这些组织中几乎没有表达。
表达谱的欧盟AP2.ZS11不同植物和生殖组织中的基因.一个欧盟表达谱的层次聚类AP2.基于ZS11 RNA-Seq数据对13个样本进行基因分析。显示log2转换后的FPKM值的热图.用于表达谱分析的组织显示在每一栏的顶部。每个组织都有三次复制。基因在表达条的右边。Ro-f、St-f、Cal-f、Pe-f、Pi-f和Sta-f分别代表从盛花期采集的根、茎、萼、花瓣、雌蕊和雄蕊组织;Le_s_f、LeY-b、LeY-i、LeY-f、LeY-10d、LeY-24d、LeY-30d分别代表苗期、芽期、早花期、全花期和开花后10d、24d、30d的幼叶。bqRT-qPCR分析欧盟AP2.九个组织中的基因。根作为控制。数据被归一化为肌动蛋白基因和误差杆表示平均值的标准误差(SE)。学生们t-测试: *P< 0.05, * *P< 0.01
qRT-PCR用于确认miR172靶基因的表达水平euap2.ZS11基因在根、茎、叶、花、芽、萼片、花瓣、雄蕊和雌蕊等9个组织中的表达。5b)。大部分的euap2.S表现出不同的组织特异性表达模式,与根系相比达到显着差异。BnaTOE1-2-5和BNATOE1-4根和雄蕊的转录本丰度最高;BnaTOE3-1-2和BnaSMZ-1-3分别在叶和根中表达量最高;BnaTOE2-2和BnaTOE2-3在萼片中特异性高表达;另外五个euap2.基因(BnaAP2-1-5,BnaAP2-2-4,BNATOE1-1-3,BnaTOE2-1, 和BnaSMZ-2)在花器官(花、芽、萼片、花瓣、雄蕊和雌蕊)中表达量增加,表明这些基因可能参与了花期和花器官发育的调控。
早花系和晚花系的基因表达模式
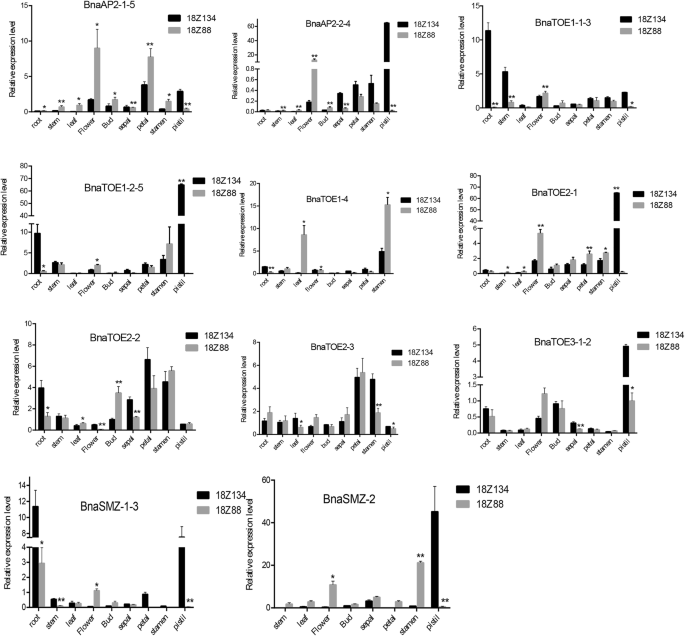
调查假定的AP2.提取了花期调控相关基因的RNA-seq数据,分别来自早花期块材料和晚花期块材料。首先,分析了营养期早花和晚花散装材料的茎部组织(S)和叶片(L)的表达水平10:图S3a)。的euap2.两种细胞系的两种组织之间的表达模式分为3类。第一组在四个样本中均无或低表达BnaSMZ-1,BnaSMZ-2,BnaSMZ-3,BNATOE1-5和BnaTOE2-1;第二组表现为中等水平的表达,四个样本和纳入样本之间无明显差异BNATOE1-1,BNATOE1-4,BNATOE2-2,BnaTOE2-3, BnaAP2-2和BnaAP2-3;第三组在四个样本中表现出相对较高的表达,BnaAP2-1、BnaAP2-5 BnaTOE1-2和BNATOE1-3在开花后期的茎部组织(S)和叶片(L)中表达量均高于开花前期的材料,表明它们在开花过程中起着负调控作用BnaAP2-4,BnaTOE3-1和BnaTOE3-2没有显示上述规则。其次,我们进一步研究了早花材料(18Z134)和晚花材料(18Z88)营养期和生殖期幼叶的RNA-seq数据,得到了类似的结果(附加文件)10:图S3b)。BnaSMZ-1,BnaSMZ-2,BnaSMZ-3,BnaSNZ,BNATOE1-1,BNATOE1-5和BnaTOE2-1属于第一组,没有或较低的表达;BNATOE1-3,BnaTOE2-2,BnaTOE2-3,BnaTOE3-1和BnaTOE3-2属于第二组,四个样本之间均保持高表达水平,但无明显差异;BnaAP2(AP2-1,AP2-2,AP2-3,AP2-4, 和AP2-5),BNATOE1-2和BNATOE1-4属于第三组,并在营养阶段叶片中表现出升高的表达和晚开花材料中的更高的转录物丰度,而不是早期开花的材料,进一步证实了它们的负调节作用。
采用qRT-PCR检测AP2.s中用于RNA-SEQ的相同材料(18Z88和18Z134)(图。6).大多数euap2.18Z88的花器官基因表达量高于18Z134,且差异显著。组织特异性表达分析显示BNATOE1-1-3,BnaTOE1-2-5,BnaTOE2-2和BnaSMZ-1-3在18Z134根中大量表达。所有euap2.除了基因BNATOE1-4和BnaTOE2-3在18z134的雌蕊中具有比18z88的雌蕊更高。表达AP2.S在其他组织中的表达均较低。
讨论
MiR172和它的目标AP2.芸薹属的基因及其进化
转录因子(Transcription factors, TFs)在植物生长发育中起着重要作用。AP2/ERF (APETALA2/Ethylene Responsive Factor, AP2/ERF)蛋白是一个大的转录因子家族,具有响应生物和非生物胁迫以及发育阶段的关键功能,已在包括拟南芥(Arabidopsis)在内的多种植物中被发现[37)、大米(38],b·拉伯[39],显著[40,41,大麦和小麦[42],杨树[43]和大豆[44].在这篇手稿中,我们主要集中在AP2.基因属于euap2.组。的euap2.拟南芥中有6种AP2.基因(AP2.,TOE1,TOE2,TOE3,SMZ, 和SNZ).每一个拟南芥euap2.基因中有1到5个同源基因显著.在本研究中,19euap2.基因在显著,这大约超过其祖细胞中鉴定的两倍b·拉伯(8)和b . oleracea(9)、表明有些euap2.基因在显著,b·拉伯和b . oleracea由于WGD和多倍体化,拷贝数增加。
在系统发育分析的基础上euap2.年代显著可分为五组(附加文件9:图S2)。大多数euap2.同一亚组中的基因具有类似的外显子系统结构,尽管外显子数和内部长度在不同亚家族的成员之间有显着变化(附加档案9:图S2)。属于同一类群的euAP2蛋白大部分含有相似的基序分布,而euAP2蛋白在亚群间具有不同的基序构成和组织(图1)。4b),宋也报道了这一发现[40].两者在基因结构和基序组成上的分歧euap2.基因在进化过程中提示功能分化。基序识别和基因结构分析也高度证实了我们的系统发育分类。在植物中发现的大多数miRNA靶基因都是转录因子[45].MiR172被认为是调节euap2.组AP2-像TFS通过成绩易转裂和拟南芥的翻译镇压[3.,21,22].在这项研究中,4个miR172家族成员在显著被识别,目标预测显示19个euap2.基因中含有miR172的MRE(图。1一个;表格1).另外,在3'编码区中的miRNA172结合位点也被鉴定在其中AP2.大麦基因和一些具有代表性的euAP2蛋白[38,42],表明miR172的互补位点在euap2.基因在不同的植物物种中都是保守的。
重复类型和共时性分析AP2.基因在显著和其他物种
七对平行推理显著通过系统发育分析鉴定物种之间的11对原蛋白质(图。2b).正选择是基因复制后euAP2蛋白功能分化的主要驱动力。数量的比较euap2.基因在显著与拟南芥和祖先亲本基因组显示显著有更多的基因(图。3.).进化过程中发生的基因复制事件有助于基因家族的扩展[35].根据重复分析,WGD或分段重复事件在此期间有重大影响euap2.进化。因此,WGD或分段重复可能是数量增加的一个原因euap2.基因。
的表达模式分析euap2.年代显著
的时空表达模式euap2.已经在许多物种中进行了分析。Jofuku表明,与其他花卉归属基因不同,AP2.在拟南芥的非花组织(茎和叶)和花组织(萼片、花瓣、雄蕊、心皮、发育中的胚珠和花序分生组织)中均有表达[46].javier研究了表情AP2.在小麦和大麦不同发育阶段的根、茎、幼叶和穗中发现了类似的基因AP2.类基因在所有组织中均有表达,在穗发育早期达到高峰,随着穗成熟逐渐减少[42].的euap2.水稻的基因转录在幼叶、成熟叶、根尖、花序和穗的各个阶段[38].表达AP2 /小块土地在显著之前也检查过:庄和朱的调查显著EST数据库AP2 / ERF.基因在茎中高度表达,其次是花和叶[47];哈贾尔透露,大部分BnaAP2亚家族在花蕾,根,叶子中显示出高表达,并通过转录组测序茎[41];Song使用了Chahoub报道的RNA-seq数据[32,并得出结论AP2.基因在根中的表达量高于叶片[40].
有报道称AP2.基因在花上生的关键功能,包括花卉公司的建立[27,48,49,50],花器官特性[3.,51,52]和花的同源性基因表达[44].在这项研究中BnaAP2S在不同组织间表达模式不同。超过一半的人euap2.基因主要在花、花蕾、萼片、花瓣、雄蕊和雌蕊等生殖器官中表达euap2.基因在花发育中也起着重要作用显著[53].
来进一步验证是否euap2.我们分析了影响开花时间的基因在早花期和晚花期不同器官中的表达。某些euap2.基因,如BnaAP2-1,BnaAP2-5和BNATOE1-2,在晚花材料(18Z88)花器官中表达量高于早花材料(18Z134),表明可能是花抑制基因。这一观察结果与之前在拟南芥上的研究结果一致[21,22,27].
结论
在这项研究中,19euap2.基因和4个miR172成员被鉴定显著基因组和19euap2.S被分为五个亚组。这17个euAP2基因被miR172预测为靶向,具有组织特异性的表达模式。大部分的euap2.AP2/miR172在花器官(萼片、花瓣、雄蕊和雌蕊)中表达量较高,表明AP2/miR172模块在花器官发育中起重要作用。此外,还有三个基因,BnaAP2-1,BnaAP2-5和BNATOE1-2,在花期晚的品系中表达量高于花期早的品系,这揭示他们可能充当花卉抑制器。携带在一起,MiR172 / Euap2基因的鉴定和表达为进一步研究MiR172 / Euap2相互作用和花卉开发提供了见解显著.
材料和方法
Bna-miR172及其靶标的鉴定euap2.基因
的核苷酸和蛋白质序列AP2.家族基因答:芥,显著,b·拉伯, 和b . oleracea从拟南芥信息资源下载(Tair,http://www.arabidopsis.org)及BRAD (http://brassicadb.org/brad/index.php).序列从答:lyrata、水稻、玉米和苔藓的来源是EnsemblePlants (http://plants.ensembl.org/index.html).使用拟南芥AP2蛋白序列对具有默认参数的BRAD和EnsemblePlants数据库进行BLASTP搜索。e值≤10的序列−10被认为是候选蛋白质。进一步确认候选人euap2.家族基因,使用智能评估蛋白质中AP2结构域的存在(http://smart.embl-heidelberg.de/)[54]和pfam(http://pfam.xfam.org/)[55)数据库。为了进一步验证所鉴定的基因,将所鉴定基因的氨基酸序列也提交到NCBI的保守域数据库(CDD,https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)及HMMER (https://www.ebi.ac.uk/Tools/hmmer/search/hmmscan).
物理化学参数,包括理论等电点(pI)和分子量(Mw),使用ExPasy pI/Mw工具(http://web.expasy.org/protparam/)[56].AP2家族蛋白的亚细胞定位通过Cell-PLoc网站(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc/).
植物miRNA172家族的前体和成熟序列来自miRbase22数据库(http://www.mirbase.org/)[57].miR172基因被命名为miRbase (Bna-miR172),并有新的序号(如a-e)。Bna-miR172前体的二级结构由Mfold web服务器(http://mfold.rna.albany.edu/?Q = Mfold.)[58].miR172的假定靶基因是通过网络工具TAPIR (http://bioinformatics.psb.ugent.be/webtools/tapir/)[59]和psRNATarget (http://plantgrn.noble.org/psRNATarget/)[60.通过同时上传miR172成熟序列和euap2.带有默认参数的基因序列。最后,筛选出具有1个或2个AP2保守域的两个服务器预测的潜在靶点作为候选靶点基因。
染色体位置信息euap2.和miR172基因显著数据库(http://www.genoscope.cns.fr/brassicanapus/).MapChart版本2.2用于映射euap2.和Bna-miR172基因[61.].
系统发育和KA / KS分析
多序列比对的蛋白质序列euap2.家族基因和前体序列miR172由ClustalW使用默认参数执行[62.].然后使用对齐的序列来构建Mega V6.0的邻居连接(NJ)树[63.]和1000个引导复制和成对删除。iTOL网站(https://itol.embl.de/)来更好地显示系统发生树[64.].的同源关系和谬误关系AP2.从系统发育树中推断基因[65.,66.].采用dnaspv6软件计算非同义替代率(Ka)和同义替代率(Ks)。最后,根据Ka/Ks比值评估基因对所经历的选择压力[67.].
重复类型和共时性分析euap2.年代显著和其他物种
采用多重共线性扫描工具(MCScanX)分析基因重复事件,默认参数为[68.].的共时关系AP2.基因的获得显著和其他三个物种,共系分析图使用Dual Synteny Plotter软件(https://github.com/CJ-Chen/TBtools)[69.].
外显子内部结构与图案分析euap2.基因
芸苔的外显子内含子结构euap2.通过编码序列(CDSs)与相应的基因组序列比对,获得家族基因。外显子-内含子结构图是使用在线的基因结构显示服务器(GSDS 2.0:http://gsds.cbi.pku.edu.cn)[70].
euAP2域的序列标识由WebLogo (http://weblogo.berkeley.edu/logo.cgi.).通过MEME程序对确定的euAP2家族进行保守基序分析(http://meme-suite.org/),具有以下参数:最佳宽度,6-300个氨基酸;一个主题重复的次数,任何;最多10个主题[71.].基序注释用InterPro (https://www.ebi.ac.uk/Interpro/beta//)[72.].
中顺式作用元素分析显著mir172和euap2.基因启动子
启动子区域选择Bna-miR172前体序列预测转录起始位点上游1.5 kb和euAP2转录起始位点上游1 kb。利用PlantCare (http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)[73.].
植物材料
RNA-seq数据来自测序的13种不同组织显著中国农业科学院油料作物研究所株中双11 (ZS11)(根、茎、花萼、花瓣、雄蕊在花期采集,苗期、芽期、开花期、全花期和开花后10、24、30 d采集)(生物工程ID, PRJNA358784)。从本实验室构建的重组自交系(RIL)中筛选极早开花和极晚开花材料[74.].其开花时间列于附加文件中5:表S5。收集早期开花散装材料(18Z43,18Z71和18Z134)的芽组织和叶子(L)的表达数据,并收集在营养期的晚在开花的散装材料(18Z44,18Z88和18Z163),以及加入数字是SRP108958。此外,还分析了来自早期开花材料(18Z134)的幼叶(18Z134)和后开花材料(18Z88)的RNA-SEQ数据,并在营养和生殖阶段(18Z88),加入号为PrJNA540020。每个样品分析两个独立的生物重复。记录的丰富度euap2.基因计算采用每千碱基外显子模型每百万测序片段(FPKM)。采用log2变换的FPKM值进行研究euap2.基因表达(附加文件6:表S6)。
组织表达分析
表达分析采用ZS11、18Z134、18Z88。在同一发育时期采集5个营养组织(根、茎、叶、花和芽)和4个花器官(萼片、花瓣、雄蕊和雌蕊)进行qRT-PCR。所有的植物材料都种植在中国重庆西南大学的试验田。
根据制造商的说明,通过用RNAEysy萃取试剂盒(Invitrogen,Carlsbad,CA)的所有样品中提取总RNA。使用Agilent 2100 Bioanalyzer(Agilent,Böblingen,德国)评估RNA的质量,并通过电泳通过电泳进行1.5%琼脂糖凝胶。对于QRT-PCR,使用Superscript™第一链合成系统III(Invitrogen)将1μgRNA逆转录成第一链cDNA。在实时PCR机器(Bio-rad,Hercules,USA)的Sybr Green PCR Supermix上进行定量实时PCR。使用2计算相对基因表达水平-ΔΔct方法,BnaACTIN7用作内部控制[75.].qPCR引物列于附加文件7: S7表。至少使用2个生物重复,每个样本进行3个技术重复。
数据和材料的可用性
所有支持数据都可以在稿件和其附加支持文件中找到。
缩写
- AP2.:
-
APETALA2
- CRE:
-
顺式元件
- FPKM:
-
每千个碱基的片段每百万个片段的外显子
- K a:
-
每个非同义位点的非同义替换数之比
- KA / KS:
-
非同义/同义替代比
- Ks:
-
每个同义位点的同义替换数之比
- 绝笔:
-
microrna的响应元件
- 存在:
-
定量实时聚合酶链反应
- SMZ:
-
SCHLAFMUTZE
- SNZ:
-
SCHNARCHZAPFEN
- 脚趾:
-
饮食目标1
- WGD:
-
全基因组复制
参考文献
- 1.
MicroRNAs:基因组学,生物发生,机制和功能。细胞。2007;116:281 - 97。
- 2.
Curaba J,Singh MB,Bhalla PL。MiRNA在狂欢狂暴信号通路之间的串扰中。J Exp Bot。2014; 65(6):1425。
- 3.
Aukerman MJ,Sakai H. MicroRNA的开花时间和花器官身份的调节及其Apetala2样靶基因。植物细胞。2003; 15(11):2730-41。
- 4.
关键词:microrna,调控,叶片形态发生,Palatnik大自然。2003;425(6955):257。
- 5.
Fujii H, Chiou TJ, Lin SI, Aung K, Zhu JK。在拟南芥中参与磷酸盐饥饿反应的miRNA。咕咕叫医学杂志2005;15(22):2038 - 43。
- 6。
高盐诱导miR-169家族成员,并短暂抑制NF-YA转录因子。中国生物医学工程学报,2009;
- 7。
黄建平,李建平。植物生长发育阶段的调控。发展。2011;138(19):4117 - 29。
- 8。
小rna与植物发育时间。2009;19(4): 374-8。
- 9。
Fornara F, Coupland G. Plant phase transitions make a SPLash。细胞。2009;138(4):625 - 7。
- 10。
Chuck G,Hake S.发育过渡的调节。CurrOp植物BIOL。2005; 8(1):67-70。
- 11.
周成明,王建文。microrna调控开花时间。遗传学报。2013;40(5):211-5。
- 12.
Spanudakis E,杰克逊S. MicroRNA在开花时间控制中的作用。J Exp Bot。2014; 65(2):365-80。
- 13.
srikanth a,施密米的开花时间的调节:所有道路都通往罗马。细胞mol寿命。2011; 68(12):2013-37。
- 14.
Wang JW,捷克B,Weigel D.Mir156调节的SPL转录因子定义内源性开花途径拟南芥蒂利亚纳.细胞。2009;138(4):738 - 49。
- 15.
吴克,公园我,康威,王家,伟莱D,Poithig卢比。miR156和miR172的顺序作用调节拟南芥的发育时间。细胞。2009; 138(4):750-9。
- 16.
Yamaguchi A,Wu Mf,Yang L,Wu G,Poethig Rs,Wagner D. MicroRNA调节的SBP箱转录因子SPL3是直接上游活化剂多叶的,FRUITFULL, 和APETALA1.Dev细胞。2009;17(2):268 - 78。
- 17。
吴刚,蒲秀珍。植物茎部发育的时间调控。植物学报,2003,54 (5):574 - 578拟南芥蒂利亚纳miR156和它的目标SPL3.发展。2006; 133(18):3539-47。
- 18。
关键词:miR172, miR172,开花抑制SMZ.Plos Biol。2009; 7(7):E1000148。
- 19。
朱QH,Helliwell Ca.MiR172的开花时间和花卉图案的调节。J Exp Bot。2011; 62(2):487-95。
- 20.
CARPEL FACTORY, a dicer homolog, and HEN1 (a new protein, a new protein, a new protein, a new protein, a new protein, a new protein, a new protein, a new protein, a new protein, a new protein, a new protein, a new protein拟南芥蒂利亚纳.咕咕叫医学杂志。2002;12(17):1484 - 95。
- 21.
施密德(Schmid),胡伦豪(Uhlenhaut),戈达尔(Godard),德玛尔(Demar),布列桑(Bressan),韦格尔(Weigel),罗曼(Lohmann)。花诱导途径的解剖使用全局表达分析。发展。2003;130(24):6001 - 12所示。
- 22.
Chen X.一个MicroRNA作为翻译压缩机APETALA2在拟南芥的花发育中。科学。2004;303(5666):2022 - 5。
- 23。
Okamuro JK,Caster B,Villarroel R,Montagu MV,Jofuku KD。Apetala2的AP2结构域定义了拟南芥中的大型DNA结合蛋白质。Proc Natl Acad Sci U S A. 1997; 94(13):7076-81。
- 24。
jung jh,seo yh,seo pj,reyes jl,ju y,chua nh,park cm。的Gigantea.-调控的microRNA172介导的光周期开花独立于君士坦斯在拟南芥。植物细胞。2007;19(9):2736 - 48。
- 25。
关键词:水稻,miR172,嫁接,马铃薯,马铃薯块茎发展。2009;136(17):2873 - 81。
- 26。
Jung Jh,Seo PJ,康SK,公园厘米。MiR172信号纳入MIR156信号通路中SPL3/4/5拟南芥发育过渡的基因。中国生物医学工程学报,2011;
- 27。
关键词:拟南芥,APETALA2,花过渡,花发育,双功能转录因子植物细胞。2010;22(7):2156 - 70。
- 28.
Lauter N,Kampani A,Carlson S,Goebel M,Moose SP,Freeling M. MicroRNA172下调glossy15促进玉米植物相变。Proc Natl Acad SCI U S A. 2005; 102(26):9412-7。
- 29.
miR172的过度表达导致水稻小穗确定性丧失和花器官异常(栽培稻).中国生物医学工程学报,2009;
- 30.
关键词:拟南芥,花模式缺陷,拟南芥APETALA2和microRNA172表达烟草benthamiana.中国生物医学工程学报,2006;
- 31。
Nair SK, Wang N, Turuspekov Y, pourkheirdish M, Sinsuwongwat S, Chen GX, Sameri M, Tagiri A, Honda I, Watanabe Y.通过抑制microrna引导的HvAP2 mRNA的切割来抑制大麦闭锁花。国家自然科学基金资助项目:国家自然科学基金资助项目。
- 32。
Liu S, Liu S, Parkin IAP, Tang H, Wang X, Chiquet J, Belcram H, Tong C, Samans B.早期异源多倍体进化芸苔属植物显著油料基因组。科学。2014;345(6199):950 - 3。
- 33。
Nova-Franco B, Íñiguez LP, Valdés-López O, alvarados - affantranger X, Leija A, Fuentes SI, Ramírez M, Paul S, Reyes JL, Girard L.等。植物杂志。2015;168(1):273 - 91。
- 34。
Fahlgren N, Carrington JC。MiRNA在植物中的靶标预测。方法《Mol Biol》2010;592:51-7。
- 35.
关键词:基因重复,基因片段,串联复制,遗传进化拟南芥蒂利亚纳.BMC植物BIOL。2004; 4(1):10。
- 36.
埃尔南德斯-加西亚首席技术官,Finer JJ。启动子和顺式调控元件的鉴定和验证。植物科学。2014;217 - 218(1):109 - 19所示。
- 37.
关键词:基因组,全基因组分析,生物信息学小块土地拟南芥和水稻的基因家族。植物杂志。2006;140(2):411。
- 38.
唐米,李G,陈的发展史和表达模式APETALA2水稻中的基因。j遗传学基因组学。2007; 34(10):930-8。
- 39.
歌曲X,李啊,侯X.全面分析AP2 / ERF转录因子超家族在大白菜中(芸苔属植物拉伯SSP。pekinensis)。BMC基因组学。2013; 14(1):573。
- 40.
[16]宋晓,王军,马晓,李勇,雷涛,王磊,葛伟,郭丹,王智,李超AP2 / ERF.总科在芸苔属植物显著.植物保护学报。2016;30(6):729 - 736。
- 41.
AP2/ERF转录因子的鉴定和功能预测芸苔属植物显著通过基因组的分析。计算Biol Chem。2017; 71:32。
- 42.
关键词:基因工程,基因工程,基因组分析,表达APETALA2来自大麦、小麦和大麦-小麦双倍体的类似基因。中国生物医学工程学报,2009;
- 43.
庄军,蔡斌,彭瑞荣,朱斌,金晓峰,薛勇,高峰,付晓燕,田永生,赵伟AP2 / ERF.基因家族在杨树trichocarpa.2015;5(1): 132-7。
- 44.
大豆ERF基因家族的系统发育、基因结构及表达模式大豆L.)。J Exp Bot。2008; 59(15):4095-107。
- 45.
Rhoades MW, Reinhart BJ, Lim LP, Burge CB, Bartel B, Bartel DP。植物microRNA靶标的预测。细胞。2002;110(4):513 - 20。
- 46.
Jofuku KD, Boer BGD, Montagu MV, Okamuro JK。同种异体基因对拟南芥花和种子发育的控制APETALA2.植物细胞。1994年; 6(9):1211-25。
- 47。
庄杰,朱波芸苔属植物显著ESTs: AP2/ erf家族转录因子的基因发现及表达模式。Mol Biol rep 2014;41(1): 45-56。
- 48。
爱尔兰VF,苏塞克斯IM。的函数apetala-1拟南芥花卉发育期间的基因。植物细胞。1990; 2(8):741-53。
- 49。
Bowman JL,Alvarez J,Weigel D,Meyerowitz Em,Smyth Dr。拟南芥拟南芥的花卉发展控制APETALA1和相互作用的基因。发展。1993;119(3):721 - 43。
- 50.
舒尔茨EA,霍恩GW。拟南芥花起始过程(FLIP)的遗传分析。发展。1993;119(3):745 - 65。
- 51.
Kunst L,Klenz Je,Martinez-Zapater J,Haughn GW。AP2.基因决定花的花被器官的身份拟南芥蒂利亚纳.植物细胞。1989;1(12):1195 - 208。
- 52.
拟南芥花发育的基因调控。植物细胞。1989;1(1):37-52。
- 53.
Lohmann Ju,Weigel D.拟南芥归属基因的负调节无性生殖的由APETALA2产品。细胞。1991;65(6):991 - 1002。
- 54。
Letunic I, Doerks T, Bork P. SMART 7:蛋白质结构域注释资源的最新更新。核酸研究2012;40(数据库版):302-5。
- 55。
Finn Rd,Bateman A,Clements J,Coggill P,Eberhardt Ry,Eddy SR,Heger A,Hethington K,Holm L,Mistry J. PFAM:蛋白质家庭数据库。核酸RES。2014; 42(数据库问题):222-30。
- 56。
Gasteiger E,Hoogland C,Gattiker A,Duvaud Se,Wilkins Mr,Appel Rd,Bairoch A.蛋白质识别和分析工具在扩展服务器上:Humana Press;2005年。
- 57。
Kozomara A,Griffithsjones S. Mirbase:使用深度测序数据注释高置信微润荷。核酸RES。2014; 42(数据库问题):D68。
- 58。
Mfold网络服务器用于核酸折叠和杂交预测。核酸杂志2003;31(13):3406-15。
- 59.
Bonnet E,He Y,Billiau K,Van YDP。TapIr,用于预测工厂MicroRNA目标的Web服务器,包括目标模拟。生物信息学。2010; 26(12):1566-8。
- 60.
戴旭,赵px。PsRNATarget:植物小RNA靶标分析服务器。核酸杂志2011;39:W155。
- 61.
vooorres重新。MapChart:用于链接地图和QTL的图形演示软件。j hered。2002; 93(1):77-8。
- 62.
Larkin MA, Blackshields G, Brown NP, Chenna R, Mcgettigan PA, Mcwilliam H, Valentin F, Wallace IM, Wilm A, Lopez R. Clustal W和Clustal X 2.0版本。生物信息学。2007;23(21):2947 - 8。
- 63.
Tamura K, Peterson D, Peterson N, Stecher G, Nei M, Kumar S. MEGA5:基于最大似然、进化距离和最大简化方法的分子进化遗传学分析。中国生物医学工程学报。2011;28(10):2731。
- 64.
Leatunic I,Bork P.互动生活树(ITOL)V3:用于显示和诠释系统发育和其他树木的在线工具。核酸RES。2016; 44:W242-5。
- 65.
宋A,GAO T,Li P,Chen S,Guan Z,Wu D,Xin J,风扇Q,Zhao K,陈F.转录组型识别和DOF转录因子基因家族的表达探讨菊花.植物保护学报。2016;40(5):559 - 564。
- 66.
关键词:拟南芥,SBP-box,基因家族,水稻基因。2008;407(1):1 - 11。
- 67.
Rozas J, Ferrer-Mata A, Sánchez-DelBarrio JC, Guirao-Rico S, Librado P, Ramos-Onsins SE, Sánchez-Gracia A. dnasp6:大数据集DNA序列多态性分析。生物学报。2017;34(12):3299-302。
- 68.
王Y,唐H, DeBarry JD,谭X,李J,王X,李的张茵,金H,马勒B,郭H, et al。MCScanX:一个用于基因共线性和共线性检测和进化分析的工具包。2012;40(7):e49。
- 69.
[10]陶晓,陈超,李超,刘杰,刘超,何勇WRKY菠萝基因家族:在发育和胁迫过程中的进化和表达谱。BMC基因组学。2018;19(1):490。
- 70.
[11]胡斌,金军,郭爱娥,张宏,罗军,高光。GSDS 2.0:基因特征可视化升级服务器。生物信息学,2014,31(8):1296。
- 71.
Bailey TL,Johnson J,Grant Ce,Noble Ws。MEME套件。核酸RES。2015; 43:W39-49。
- 72.
Finn RD, Attwood TK, Babbitt PC, Bateman A, Bork P, Bridge AJ, Chang HY, Dosztányi Z, Elgebali S, Fraser M. InterPro 2017-beyond protein family and domain annotation。核酸研究2017;45(数据库版):D190-9。
- 73.
Lescot M, Déhais P, Thijs G, Marchal K, Moreau Y, Peer YVD, Rouz P, Rombauts S. PlantCARE:植物顺式调控元件数据库和启动子序列分析工具的入口。中国生物医学工程学报,2002;30(1):125 - 125。
- 74.
刘L,曲c,wittkop b,yi b,小y,他,snowdon rj,li J.一种高密度SNP地图,用于精确映射种子纤维QTL芸苔属植物显著L. Plos一个。2013; 8(12):E83052。
- 75.
Livak KJ,Schmittgen TD。使用实时定量PCR和2分析相对基因表达数据——ΔΔCt方法。方法。2001; 25(4):402-8。
致谢
感谢西南大学的陆坤给我们提供的表达数据。
资金
国家重点研发计划(no . 2016YFD0100202);中央高校基本科研业务费专项资金(no . XDJK2017A009);重庆市科委(no . cstc2016shmszx80083);资助机构参与了这项研究的设计,提供了实验材料,并资助了转录组分析。
作者信息
从属关系
贡献
WTY和LLZ构思设计了实验;WTY进行数据分析并撰写稿件;LJN、XXF、LK参与了部分稿件的起草和论文的修改;JHJ和WJ进行系统发育分析;PXK、CYR、GYM和TYC进行RNA提取和qRT-PCR。所有作者阅读并批准最终稿件。
通讯作者
道德声明
伦理批准并同意参与
不适用.
同意出版
不适用。
相互竞争的利益
提交人声明他们没有竞争利益。
额外的信息
出版商的注意
《自然》杂志对已出版的地图和附属机构的管辖权主张保持中立。
附加文件
附加文件1
:表S1miR172前体和成熟序列在甘蓝型基因组。(XLSX 9 kb)
附加文件2
:表S2共线的euap2.基因之间的双显著,拟南芥和它的祖先(B.Rapa.和B. Oleracea).(XLSX 12 kb)
额外的文件3
:表S3MEME鉴定的euAP2蛋白推测基序注释(XLSX 9 kb)
附加文件4.
:表S4Bna-miR172启动子区预测的顺式调控元件euap2.基因(XLSX 11 KB)
额外的文件5
:表S5重庆5年极端材料开花时间(XLSX 9 kb)
额外的文件6
:表S6RNA-seq数据euap2.ZS11各组织及早、晚花期株系叶片和茎部组织的基因(XLSX 15 kb)
附加文件7.
:表S7引物用于qRT-PCReuap2.基因在B.napus(XLSX 10 KB)
附加文件8.
:图S1对bna-miR172成熟序列和前体序列及其二级结构进行多序列比对。一个BNA-miR172成熟序列的多序列比对;bbna-miR172前体序列多序列比对;c中前期mir172序列二级结构显著.(TIF 4456 kb)
附加文件9.
:图S2系统发育树和基因结构euap2.基因在显著.子树分支线是着色的,以指示不同的片状。蓝色框表明未经翻译的5'和3'地区;黄色框表示外显子;黑线表示内含子。CD,编码序列;UTR,未经翻译的地区。(TIF 1769 KB)
附加文件10.
:图S3eu表达谱AP2.基于RNA-Seq数据分析早期和晚期开花系的基因。在热图的左侧显示分层聚类结果,在热图右侧的彩色比尺条表示用log2转换的FPKM值。每个样本都有两次重复。一个eu表达谱AP2.叶片和茎部组织中的基因。EL,早花材料叶片;LL,晚开花材料的叶子;ES,早花材料的茎尖区域;LS为晚开花材料的茎尖区。beu表达谱AP2.营养和生殖阶段叶片中的基因。EV,营养期早花物质的叶片;LV,营养期开花后期的叶片;ER,生殖期早花物质的叶片;LR为生殖期花期后期的叶片。(TIF 2402 kb)
权利和权限
开放获取本文在知识共享归属4.0国际许可条款下发布(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介中不受限制地使用、分发和复制,只要您给予原作者和来源适当的信任,提供知识共享许可的链接,并说明是否有更改。“创作共用公共领域”豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
王涛,平晓,曹勇。et al。miR172的全基因组探索和表征euap2.基因在芸苔属植物显著L.可能在花器官发育中起作用。BMC植物杂志19,336(2019)。https://doi.org/10.1186/s12870-019-1936.2.
已收到:
接受:
发表:
关键字
- 芸苔属植物显著
- miR172
- euap2.
- 进化
- 表达分析