摘要
背景
适应非生物胁迫是多年生植物在自然环境中生存的关键。然而,人们对其潜在机制知之甚少。在此,我们采用液体培养系统来研究植物对重复盐胁迫的适应杨树树木。
结果
我们首先评估了表型反应,发现植物在盐胁迫预处理后表现出更好的抗逆性。然后进行时间过程RNA测序(RNA-seq)来分析盐处理12小时内基因表达的变化。差异表达基因(DEGs)的分析表明,发生了显著的转录重编程和对重复盐处理的适应。聚类分析确定了两个共同表达基因模块,这两个模块对重复盐胁迫适应具有潜在的关键作用,以及一个关键模块对盐胁迫反应的一般作用。基因本体(GO)富集分析在这些基因模块中发现了激素信号、细胞壁生物合成和修饰、生长负调控和表观遗传调控等途径。
结论
本研究说明了水稻的表型和转录适应杨树揭示了新的基因模块对响应和适应盐胁迫具有潜在的关键作用。
背景
对各种非生物应激的适应对于术治疗术的生存和生物质积累至关重要,并且由于其相对较长的循环而对多年生树种尤其如此。杨树,是一种模式树种,因为有一套近乎完整的实验资源,如易于繁殖、转化方法和丰富的遗传和基因组材料[1那2[提供理想的系统,以发现常年树如何适应非生物应激。
最近的研究在拟南芥其他模式植物表明,经历亚致死非生物胁迫的植物可能会在生理和转录水平上记住这种胁迫,以在再次遇到相同胁迫时提高表现[3.那4.那5.那6.那7.那8.].例如,与没有脱水经历的植物相比,经历了一种或多种脱水应力和浇水回收循环的拟南芥和玉米植物表现出更高的能够保持叶相对含水量(RWC)的能力[9.那10.那11.].在转录水平,压力反应的基因可分为两种类型根据连续压力响应:“记忆基因”显示显著不同级别的,或下调在随后的应力比前一个,和“非基因”,显示每个压力(类似的反应12.].拟南芥,玉米和切换之间的比较研究发现,存在保守的脱水记忆基因,但脱水记忆基因的总数和同源物也显着差异[10.那13.,表明存在进化保守和物种特异性的机制来调节植物对重复非生物胁迫的反应。
转录调控是动态的,不同类型的基因通常以不同的动力学和模式对干扰作出反应。因此,用多个时间点的基因表达数据而不是单个时间点来描绘植物对非生物胁迫的转录反应将会有更丰富的信息[14.那15.那16.].高通量测序能力的提高使得用时间序列RNA-seq实验来表征全基因组转录动态变得可行。结合计算分析,利用这种时间序列表达数据,可以识别关键基因模块、枢纽基因或推断调控网络的层次结构[16.那17.那18.那19.那20.那21.那22.那23.,并提供了一个更好的基本生物过程是如何调控的概述。
高盐度通常由土壤中NaCl的积累引起渗透和离子胁迫,是限制植物生长和分布的最广泛的非生物胁迫之一[24.那25.].因此,研究植物适应盐胁迫的机制实际上是很重要的。在这项研究中,我们建立了一种精确控制的基于液体培养的实验程序,以研究植物适应盐胁迫杨树。我们首先评估对盐胁迫的表型反应,然后进行时间疗程RNA-SEQ,以在盐处理过程中表征转录组动力学。我们的结果表明杨树植物对盐胁迫表现出快速的表型和转录适应。通过共表达分析鉴定关键基因模块,并分析这些基因模块的生物学相关性。
结果
盐胁迫的表型适应杨树
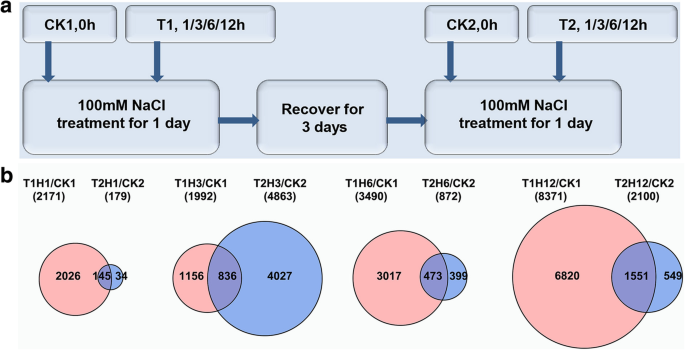
为了研究植物适应盐胁迫,我们采用了液体培养系统杨树允许精确控制NaCl处理。我们设置了两组实验:第一组采用200 mM NaCl直接进行高盐处理;第二组采用100 mM NaCl低盐处理1 d,然后恢复培养3 d,最后采用200 mM NaCl高盐处理。第一组植株迅速表现出明显的严重胁迫表型,茎尖弯曲强,叶片脱落。相比之下,在第二组中,低盐处理的植株表现与对照植株相似,在恢复3天后表型与对照植株无明显差异,在200 mM NaCl处理后表现出轻微的反应(图2)。1),时间进程分析进一步支持改善植物耐氯化钠治疗第二组:植物对200毫米氯化钠治疗后0.5小时和6 h后开始复苏而植物直接接受200毫米氯化钠0.5 h后表现出更强的表型,12 h(额外的文件后开始复苏1:图S1)。对叶片表型的详细检查表明,结果与整个植株的反应一致(图。1b) 总的来说,这些结果表明杨树经过预处理后,植物在生理上能迅速适应盐胁迫。
RNA-seq揭示了重复盐胁迫下大量的转录重编程
接下来,我们进行了RNA-SEQ,以简要重复盐处理期间基因组转录物丰度的动态变化(方法)。我们首先将植物用100mm NaCl处理1天,然后进行3天的回收培养,然后再次重复100mM NaCl处理。Samples were collected after 0 h (control samples, designated as CK1 and CK2, respectively), 1 h, 3 h, 6 h, and 12 h (designated as T1H1, T1H3, T1H6, T1H12, T2H1, T2H3, T2H6, and T2H12, respectively) of each salt treatment (Fig.2a) 。值得注意的是,对于RNA-seq样本制备,我们使用了100 为避免不同NaCl浓度对转录的影响,两种盐处理的mM-NaCl,每个处理的所有时间点均在2小时内收集 h尽量减少昼夜节律或其他环境因素的影响。分离出高质量的总RNA,并提交RNA-seq文库构建和测序。至少4500-7600万1.5亿 为每个库(附加文件)获得bp成对末端清洁读数7.:表S1)。
为了识别每个盐处理期间的差异表达基因(DEGs),我们在个体时间点和相应的对照样品之间进行了成对edgeR分析。随着每次处理的时间进程,deg的数量发生了显著变化(附加文件8.:表S2;附加文件9.:表S3)。第一次处理(T1)与第二次处理(T2)同一时间点的DEGs差异显著。例如,在T1H1/CK1组中有2171个deg,而在T2H1/CK2组中只有179个。重叠研究发现,每个时间点共有145、836、473、1551个共同基因(图1)。2b)分别占81,41,54和73%的较小数据集。总体而言,DEG的总数和低重叠率的差异表明发生重复盐处理期间的大规模转录重编程。值得注意的是,CK1和CK2之间存在1051只,具有649个上调和402个下调的次数。GO功能注释显示,1051次中的43个响应盐胁迫参与其中。在那些43次下,19种被上调,包括膜转运蛋白和MYB转录因子;24被抑制,包括调色剂内在蛋白质Tip1,干旱诱导的蛋白质DI19,嗜酸甘油蛋白调节蛋白质GA114和钙信号蛋白CDPK和GAP1(附加文件9.:表S3)。
共表达分析响应于重复盐胁迫识别关键基因模块
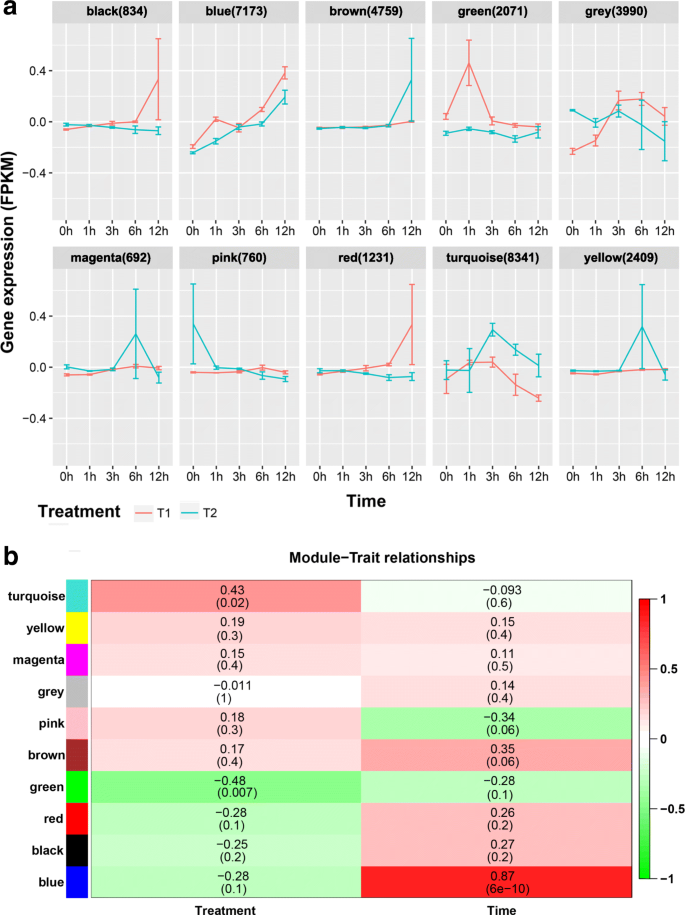
为了更好地了解时间过程治疗之间基因表达的相关性,进行加权基因相关网络分析(WGCNA)。鉴定了总,鉴定了10种Co表达的基因模块,其显示在NaCl处理期间的不同表达曲线和峰(图。3.一个;额外的文件2:图S2;附加文件10.:表S4)。模块尺寸范围为692至8341个基因,绿色模块含有适度的量(2071个基因),而蓝色(7173基因)和绿松石(8341个基因)模块包含最多的基因数量(这三个模块与含有显着的相关性稍后讨论的盐应激反应或适应(表1).
基因表达谱用Eigengene值(转录物轮廓的第一个主要成分)对每个模块进行了可视化,并且在模块上显示了不同的共表达模式(图。3.一个):蓝色模块增加沿时间进程和显示相似的变化之间的第一个和第二个治疗,绿色模块显示明显的峰值在1 h(第一个治疗相比,第二,和绿松石模块显示第二个治疗的一个重要达到3 h第一治疗相比,其他7个模块在整个时间过程中没有显著变化。模块-性状关系一致显示,绿色和绿松石模块与“处理”(T1和T2)高度相关,而蓝色模块与“时间”(1 h、3 h、6 h和12 h)显著相关(图2)。3.b)。其他七个模块与任何变量相对较低。这些结果表明,绿色和绿松石模块可能对适应重复的盐胁迫至关重要,而蓝色模块似乎对每个盐应激反应至关重要。因此,我们专注于分析绿色,绿松石和蓝色模块。
盐过敏感(SOS)途径对植物耐盐性至关重要[26.].SOS1,两个SOS2和SOS3中有三种同源物杨树基因组。我们检查了我们的共表达分析,发现SOS1的两个同源物分为绿松石模块,SOS2中的两个被分为蓝色模块,其中一个SOS3是绿色模块,一个是在绿松石模块中(附加文件)10.:表S4)。
转录因子(TFs)是植物发育和应激反应的重要调控因子[27.那28.那29.那30.那31.那32.那33.].因此,我们研究了这三种基因模块中TF基因的分布。与整个基因组相比,绿色和蓝色模块含有显着更高的TF百分比,而绿松石模块含有显着较低的TF百分比(表1;额外的文件11.:表S5)。额外的文件12.:表S6显示了绿色、绿松石色和蓝色模块中TF族个体的总结。统计分析发现,绿色模块在TFs的MYB/MYB相关、ERF、bHLH和HD-ZIP家族中显著富集,而蓝色模块在TFs的NAC、bZIP、WRKY、C3H、B3和HSF家族中显著富集。这些结果为确定植物对盐胁迫反应的关键调控因子提供了有价值的信息,但更重要的是,对盐胁迫的适应。
关键基因模块的基因本体富集分析
为了进一步探讨关键基因模块的功能意义,我们进行了基因本体(GO)富集分析,确定了每个基因模块显著富集的生物通路(BP)、分子功能(MF)和细胞组分(CC) GO项。在蓝色、绿色和蓝绿色模块中,分别鉴定出了399、154和681个富集BP GO项(附加文件13.:表S7)。
植物激素是植物非生物胁迫反应的重要调节因子。在本研究中,我们在选定的基因模块中发现了所有经典植物激素途径的富集氧化石墨烯术语(表1)2).有趣的是,在绿松石模块中主要富集在悬雍垂极地传输和信号传导中的基因,而在脱落酸(ABA)和水杨酸(SA)信号传导途径中起作用的基因仅富集在蓝色模块中。茉莉酸(JA)信号传导途径在蓝色模块中高度富集。在对植物激素嗜酸盐(GA),芸苔类固醇(BR)和乙烯(ET)的反应中的基因也富集在绿色模块中。这些结果表明,植物激素,尤其是植物素对于在ABA,SA和JA而对植物反应至关重要的同时,植物激素对于植物适应来说是至关重要的,但对于每种盐胁迫来说,植物适应性不足以足够。
详细分析确认每个模块具有特定的丰富术语。例如,绿色模块在增长的负调节中显示富集(附加文件3.:图S3;额外的文件13.:表S7);绿松石模块以参与RNA运输和分解代谢,蛋白质修改和运输以及公司发育的富集(附加档案)富集4.:图S4;额外的文件13.:表S7);蓝色模块富集在有丝分裂细胞周期,蛋白质去磷酸化,编程的细胞死亡和对非生物应激的反应的调节中(附加档案5.:图S5;额外的文件13.:表S7)。值得注意的是,与细胞壁相关的GO术语在绿色模块中高度富集,如植物型细胞壁生物发生、植物型细胞壁修饰、细胞壁疏松和木质素/果胶代谢或分解代谢过程(表S7)2);而绿松石模块显著富集于表观遗传修饰途径,如组蛋白甲基化、DNA甲基化和染色质组织(附加文件)4.:图S4;额外的文件13.:表S7)。
验证绿色,绿松石和蓝色模块的基因表达谱
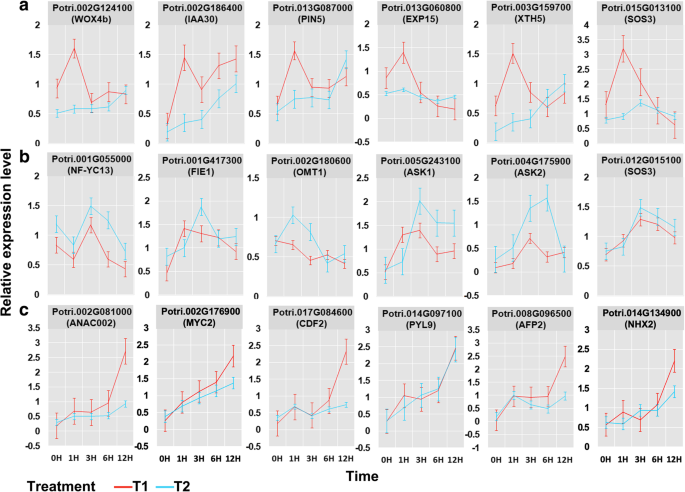
为了验证从RNA-SEQ分析衍生的基因表达谱,我们随机选择了具有相对较高的模块隶属值(MM值)的绿色,绿松石和蓝色模块的六个基因,以及用于定量PCR(QPCR)测试的表达水平。发现所有选定的基因的类似表达模式(图。4.;额外的文件6.图S6),表明我们的共表达分析结果与RNA-seq数据是可靠的,用于分析响应重复盐胁迫的基因表达杨树。
共同表达网络探索
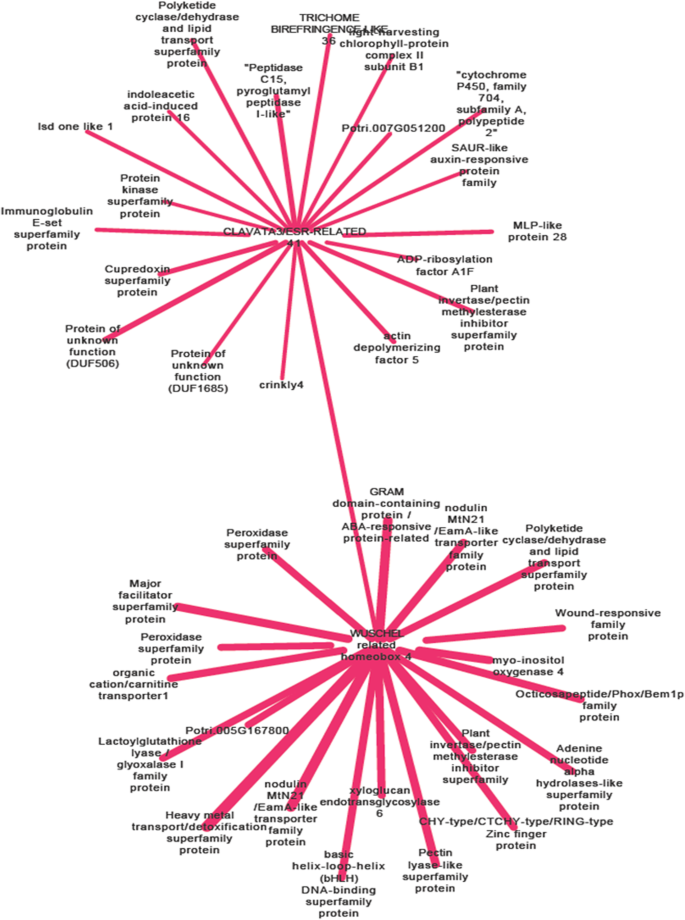
共表达网络分析显示了基于表达模式的基因相关性,显示了在特定实验条件下共表达基因表达变化的可能因果关系。WOX4-CLE41信号通路已经显示为控制二次生长速率的挂钩细胞命运的中央调节器杨树[34.那35.]因此,我们使用WOX4b和CLE41a以绿色模块中的基因为例,进一步探索本研究衍生的共表达网络。每个基因显示前20个共同表达的基因(图)。5.;额外的文件14.:表S8)。在WOX4b中的基因,miox4.那Bhlh.TF,两种过氧化物酶,几种转运蛋白基因,以及细胞壁生物发生和血糖相关基因,如果胶裂解酶样超家族蛋白,植物转化酶/果胶甲基酯酶抑制剂超家族,以及木糖葡聚糖内毒葡聚糖转移酶XTH6被发现。在CLE41a共表达基因,肌动蛋白解聚因子ADF5.那LOL1那MLP28,编码肽酶C15和蛋白激酶的基因,WOX4b而且,,CLE41a有一个saur样生长素应答基因和IAA16.之间CLE41a前20个共表达基因。这些共表达基因可能对盐胁迫下植物生长的调节很重要。
讨论
转录调节已被证明在植物对非生物胁迫的反应和适应中发挥重要作用[5.那36.那37.].因此,鉴定参与对非生物胁迫的植物反应调节的关键转录调节基因和信号通路对于植物存活率和生物质产生的遗传或生物技术改善至关重要。最近,有几项关于植物适应对非生物应激的综合研究。然而,我们知道多年生树木如何应对反复的非生物压力仍然有限。在这项研究中,我们使用杂种杨树克隆84k(Populus Alba.X杨树glandulosa)作为调查植物反应反复盐胁迫的模型系统,并证明了常年树对非生物胁迫的适应。
杨树植物可以在生理和转录水平迅速适应盐胁迫
我们开发了一种液体培养系统,使我们能够精确地控制NaCl剂量和治疗时间,调查如何杨树植物的反应反复盐胁迫。观察到整个植物反应和叶片表型对重复的盐胁迫表明,经历了1天的低NaCl预处理的植物在高NaCl处理中的恢复比直接用高NaCl处理的植物更好地表现得更好(图。1).这表明杨树可以在生理水平迅速适应盐胁迫。
我们还进行了时间过程RNA-seq来分析NaCl处理对转录动态的响应,这提供了比单时间点RNA-seq数据更好的分辨率。DEGs在时间进程中的总数和DEGs成对重叠研究表明,在连续的盐处理中存在动态和大规模的转录重编程(图)。2;额外的文件8.:表S2)。值得注意的是,除了3小时之外的所有时间点,T2的T2中的缺点较少,其中T2H3 / CK2具有比T1H3 / CK1更多的可切换。此外,来自我们的共表达分析的绿松石模块,其是最大的模块并含有8341个基因,在T2H3中具有比T1H3在T2H3中的表达峰值相对较高。绿松石模块富含GO类别,如营养素的极地传输和信号,RNA运输和分解代谢,蛋白质改性和运输,术语改性途径,对盐适应可能很重要。因此,我们认为这可能是对植物反应对重复盐胁迫的反应。来自DEG分析的另一个有趣点是,仍有超过1000个基因,3天回收培养后表现出显着差异(附加档案8.:表S2)即使植物表型恢复到预处理状态,表明转录调节比生理调节更敏感,3天回收培养物不足以完全逆转转录变化。这种现象与其他人报告的结果一致,植物保留了非生物胁迫的转录记忆,最多5天[8.那9.].研究它们的转录水平在恢复期间是如何保持的将是很有趣的。
共表达分析鉴定盐胁迫适应至关重要的推定基因模块
利用该时间序列RNA-SEQ数据,使用WGCNA封装进行基因共表达分析,并鉴定10个模块。每个模块沿着NaCl治疗具有不同的基因表达谱。基于对模块特征相关性的分析,我们决定专注于绿色,绿松石和蓝色模块,其显示出明显高的相关性与重复“治疗”或“时间”(图。3.).总体而言,绿色模块中的基因在第一次处理1 h达到了表达峰值,而第二次处理没有,而绿松石模块中的基因在第二次处理3 h出现了表达峰值,而第一次处理没有。这表明来自这两个模块的基因可以区分第一次和重复的盐胁迫,并可能导致盐胁迫适应。另一方面,蓝色模块中基因的表达量随着时间的推移而增加,并在第一次和第二次盐处理之间表现出相似的模式,表明该模块中的基因在植物对盐胁迫的响应中具有重要作用。
全基因组TF分布研究发现,绿色和蓝色模块包含的TF基因明显更多,而绿松石模块包含的TF基因明显更少,这表明绿色和蓝色模块可能在转录调控的更高层次上起作用,启动转录信号级联来调控下游基因的表达。绿色模块在TFs的MYB/MYB相关、ERF、bHLH和HD-ZIP家族中显著富集,蓝色模块在TFs的NAC、bZIP、WRKY、C3H、B3和HSF家族中显著富集。所有这些TF家族都被证明是非生物应激反应的重要调节者[27.那28.那30.那31.那32.那33.那38.那39.].但是,他们是否参与压力适应的调节仍需要进一步调查。该分析为植物转录反应对非生物压力的遗传研究遗传研究提供了推定的候选基因。
维持离子稳态对于植物耐受性和适应盐胁迫至关重要。盐过敏(SOS)途径,包括SOS1,SOS2和SOS3,已显示为盐胁迫下的钠离子流出中的中央球员[40那41.那42.],并在不同的植物物种中保存,如拟南芥[26.)、大米(43.], 番茄 [44.和白杨树[45.]在我们的共表达分析中,SOS1的两个同源物被划分为蓝绿色模块,SOS2的两个同源物被划分为蓝色模块,SOS3的一个同源物被划分为绿色模块,一个同源物被划分为蓝绿色模块(图。4.;额外的文件10.:表S4)。H.+跨膜的电化学梯度可以驱动钠离子+分区化为液压或排除细胞,由H产生+泵,如H+- 磷酸盐VP1 [46.].二VP1同源基因属于绿松石模块在我们的共表达分析(附加文件10.表S4),其中一种已在杨树中进行了功能表征,能促进植物在盐胁迫下的生长[47.].总之,这些结果表明杨树树可以通过改变离子传感和运输途径来适应重复的盐胁迫。
基因模块的功能分析表明,植物激素和表观遗传修饰是影响基因表达的关键因素杨树植物对盐胁迫的适应
植物激素是对非生物胁迫的重要调节因素,并且复杂串扰发生在不同激素的信号传导途径中,用于协调植物生长[29.那41.那48.那49.那50.那51.那52.].一般来说,ABA、SA、JA和ET被认为是“应激激素”,负责植物对生物或非生物胁迫的快速反应,而生长素、GA、BR和细胞分裂素被认为是“生长促进激素”,在长期内改变植物的发育过程[49.那53.].有趣的是,我们的富集分析与共同表达基因模块建议植物激素在其中发挥重要作用杨树反应和适应重复盐胁迫。此外,上述两种类型的植物激素可在植物反应的不同方面和对非生物应激的适应性。例如,我们发现ABA信号通路仅富集在蓝色模块中,表明ABA信令是植物响应所必需的,但是植物适应对重复的盐胁迫,这与先前的报道一致[9.那54.].JA和SA信号传导途径也专门富集在蓝色模块中。另一方面,在绿色或绿松石模块中高度富集的制造宾,GA和BR信号传导或传输途径,这表明这些植物激素在植物适应植物的适应中可能在长时间调节植物生长模式来发挥关键作用-学期。还有高度富集的GO术语,参考绿色模块中生长和细胞壁生物发生/修饰的负调节。分析这些植物激素的改变和鉴定植物反应和适应非生物应激的串扰信号通路是很重要的,从而揭示关键信号基因以改善非生物应激期间的植物生长。
表观遗传调控被认为是转录应激记忆的潜在机制之一[37.那55.那56.那57.那58.].例如,组蛋白三甲基化标记物H3K4ME3和H3K27ME3与脱水应力中的靶基因的转录记忆相关[9.那59.那60.].同时,DNA甲基化在特定区域发生变化拟南芥基因组负责盐处理引起的高渗透应激记忆[61.].在我们的研究结果中,我们发现,在与“治疗”相关的绿松石模块中,组蛋白甲基化、组蛋白泛素化、DNA甲基化和染色质组织等表观遗传途径高度富集,并在T2H3中表现出峰值表达水平(图)。3.).为了进一步揭示植物对非生物胁迫的适应机制,研究这些表观遗传修饰何时建立,这些表观遗传修饰对目的基因表达的影响,以及它们在恢复期的动态变化具有重要意义。
结论
在这项研究中,我们报道了杨树植物在生理和转录水平上都能快速适应盐胁迫。我们还做了时间序列RNA-seq来描述重复盐处理期间的转录组动态,并表明在转录水平上存在显著的重编程。我们利用这些时间序列RNA-seq数据进行共表达分析,确定了10个共表达基因模块,其中两个模块与重复盐胁迫高度相关,一个模块与一般盐胁迫响应高度相关。基因本体论(GO)分析表明,植物激素调控植物生长,特别是生长素信号通路可能在植物对反复盐胁迫的适应中起着关键作用,而ABA信号通路在植物对盐胁迫的普遍响应中起着关键作用。细胞壁生物合成/修饰和表观遗传调控也是植物适应盐胁迫的重要调控途径。综上所述,我们的研究结果为深入分析植物盐胁迫适应的信号通路和识别关键调控基因提供了框架杨树。
方法
植物栽培和样品收集
杂交杨树(Populus Alba.X杨树glandulosa)克隆84k,在16小时光/ 8小时深度光周期条件下生长,25℃,用于所有实验。所有植物都在山东农业大学(山东省)繁殖。在受控生长室的洋红色盒中转移了84k幼苗,然后1个月后转移到土壤中,最终转移到全部的Hoagland营养溶液再在NaCl治疗前进行3周生长。收集来自顶点至第四个间的茎,并立即在液氮中冷冻以进行RNA提取。每个样品收集三种生物重复,并将三种植物组合用于每种生物重复。
RNA提取和qPCR
从茎尖至第4节间采集的样品在液氮中研磨成细粉。采用CTAB法提取总RNA,用DNase (TaKaRa, 2270)处理后,从TaKaRa MiniBEST植物RNA提取试剂盒(TaKaRa, 9769)中柱纯化。用NanoDrop 2000和Agilent 2100 Bioanalyzer检测RNA的纯度和完整性,用HiScript II Q Select RT SuperMix进行qPCR (+gDNA wiper) (Vazyme, R233-01),用0.5 μg总RNA合成cDNA。采用Vazyme- chamq SYBY Color qPCR Master Mix (Vazyme, Q411-02)进行qPCR。基因特异性引物列于附加文件15.:表S9。以肌动蛋白为内参,计算基因相对表达量。为每个时间点制备三种生物重复。
RNA序列数据分析
提交高质量RNA用于mRNA测序文库的制备和150 在Hiseq X 10平台(Illumina)上进行bp配对末端测序。将干净的测序读数映射到p . trichocarpa使用hisat2的V3.0基因组组装[62.那63.],使用默认参数。使用htseq-count统计每个样本的原始映射读取[64.].edgeR包[65.,以Fisher’s exact test false discovery rate (FDR) < 0.05为统计显著性阈值。
基因共表达网络分析
计算基因表达丰度,用于加权基因共表达网络分析(WGCNA) [66.].基因间共表达关系邻接矩阵的软阈值幂为12。分层聚类执行时,最小模块大小为300,切割高度为0.994。不同的模块被分配到不同的颜色。加权相关网络分析(WGCNA)包括检验共表达模块与样本性状之间的相关性的方法。通过主成分分析(PCA)计算模块的基因表达矩阵,确定代表模块的PC1,模块特征基因(ME)。然后,利用所有模块MEs和性状数据计算模块-性状相关性。P.值<0.05用作统计显着性阈值。使用cytoscape分析了共表达网络[67.].转录因子(TF)数据库来源于植物TFDB(http://planttfdb.cbi.pku.edu.cn/index.php?sp = ptr.)[68.].
基因本体(GO)富集分析
P.trichocarpa使用V3基因本体(GO)注释,使用GoStats和GSEABase包进行富集进行富集[69.)与P.-Value <0.01。revigo [70使用默认参数用于可视化丰富的GO术语。
数据和材料的可用性
本文报道的原始序列数据已保存在BIG data Center的Genome sequence Archive [71.那72.[中国科学院基因组学(大)北京基因组学研究所,可公开访问的下载号码CRA001067http://bigd.big.ac.cn/gsa。
缩写
- 阿坝:
-
脱落酸
- 英国石油公司:
-
生物通路
- BR:
-
芸苔类固醇
- 答:
-
蜂窝组件
- 对照:
-
第一次盐处理的对照样品
- CK2:
-
第二次盐处理的对照样品
- 度:
-
差异表达基因
- ET:
-
乙烯
- GA:
-
赤霉素
- 走:
-
基因本体论
- H1 / H3 / H6 / H12:
-
1/3/6/12 H盐处理
- 是:
-
茉莉酸
- MF:
-
分子功能
- RNA-SEQ:
-
RNA测序
- RWC:
-
相对含水量
- 山:
-
水杨酸
- T1:
-
第一次盐处理
- T2:
-
第二次盐处理
- TF:
-
转录因子
- WGCNA:
-
加权基因相关网络分析
参考文献
- 1.
Jansson S, Douglas CJ。杨树:植物生物学的模式系统。植物学报2007;58:435-58。
- 2。
托斯康········乔西奥,詹森斯,波利曼·j,格里戈里艾尔,我,地狱,普通n,拉尔夫斯,rombauts s,萨拉玛夫A等。黑色棉布的基因组,Populus trichocarpa(&灰色)。科学。2006; 313(5793):1596-604。
- 3.
杜强,张超,刘宁,Fromm M, Avramova Z, Russo SE。脱水应激记忆:与玉米重复胁迫下生理反应相关的基因网络。植物科学学报2018;
- 4.
Hilker M,Schwachtje J,Baier M,Balazadeh S,Baurle I,Geiselhardt S,Hincha DK,Kunze R,Mueller Roeber B,Rillig MC,等。缺乏神经系统的生物体应激反应的启动和记忆。Biol Rev Camb Philos Soc.2016;91(4):1118-33。
- 5。
Avramova Z.应力的转录'记忆':瞬时染色质和记忆(表观遗传)标记应激反应基因。植物J. 2015; 83(1):149-59。
- 6。
王志强,王志强。细胞重编程。全球基因组调控的角色?植物杂志。2004;136(1):2579 - 86。
- 7。
Molinier J,Ries G,Zipfel C,Hohn B.在植物中应力的转换记忆。自然。2006; 442(7106):1046-9。
- 8。
胡涛,金莹,李洪波,傅建军。胁迫记忆诱导多年生黑麦草(Lolium perenne)转录和代谢的变化。杂志。2016;156(1):54 - 69。
- 9。
关键词:拟南芥,干旱,转录反应Nat Commun。2012;3:740。
- 10.
丁Y,Virlouvet L,Liu N,Riethoven JJ,Fromm M,Avramova Z.Zea Mays的脱水应力记忆基因;与拟南芥的比较。BMC植物BIOL。2014; 14:141。
- 11.
Virlouvet L,Fromm M.重复脱水胁迫期间保卫细胞的生理和转录记忆。新植物志。2015;205(2):596-607。
- 12.
丁艳艳,刘宁,刘丽娟,李丽娟,刘宁。拟南芥脱水胁迫记忆基因的研究进展。BMC Plant Biol. 2013;13:229。
- 13。
张超,彭晓霞,郭晓霞,唐国刚,孙飞,刘树喜。柳枝草脱水记忆行为的转录和生理特性研究。Biotechnol生物燃料。2018;11:91。
- 14。
Bar-Joseph Z,Gitter A,Simon I.使用时间序列基因表达数据学习和建模动态生物过程。NAT Rev Genet。2012; 13(8):552-64。
- 15.
杨志刚,李志刚,李志刚,等。杨树茎秆对重力刺激的转录和时间响应。植物学报,2018;60(7):578-90。
- 16。
林YC,李波,孙耶,熊,魏鹤,李琦,屯门奥基特S,Sederoff Rr,Chiang VL。SND1转录因子定向在杨树幼虫Paplume中木材形成中的定量功能分层遗传监管网络。植物细胞。2013; 25(11):4324-41。
- 17。
Gerttula S,Zinkgraf M,Muday GK,Lewis Dr,Ibatullin FM,Brumer H,Hart F,Mansfield SD,Filkov V,Gruver A.木质茎在杨树茎的转录和激素调节。植物细胞。2015; 27(10):2800-13。
- 18。
Klepikova Av,Logacheva MD,Dmitriev Se,Penin AA。APICTEMERISTEM时间序列的RNA-SEQ分析揭示了拟南芥花火的关键点。BMC基因组学。2015; 16:466。
- 19。
王志强,王志强,王志强,等。短肢植物共表达网络构建及应力响应分析。方法:生物医学。2018;1667:203-21。
- 20
Bassel Gw,Lan H,Glaab E,Gibbs DJ,Gerjets T,Krasnogor N,Bonner AJ,Holdsworth MJ,Pronte NJ。基因组网络模型捕获种子萌发显示植物细胞相变的协调调节。Proc Natl Acad Sci U S A. 2011; 108(23):9709-14。
- 21.
水稻干旱响应模块和基因的功能特征:基于网络的方法。麝猫。2015;6:256。
- 22.
Zinkgraf M,Liu L,Gruover A,Filkov V.在不同环境条件下鉴定基因共抑制网络基础杨树成型组织的动态调节。新植物。2017; 214(4):1464-78。
- 23.
Liu L ., Ramsay T ., Zinkgraf M ., Sundell D ., Street NR ., Filkov V ., Groover A.一种描述杨树次生生长和木材形成过程中转录因子的全基因组结合和潜在靶基因的资源。工厂j . 2015, 82(5): 887 - 98。
- 24。
陈静,张军,胡军,熊伟,杜超,陆敏。综合调控网络揭示胡杨早期耐盐机制。Sci众议员2017;7(1):6769。
- 25。
于磊,马杰,牛Z,白X,雷W,邵X,陈N,周F,万D。组织特异性转录组分析揭示胡杨幼苗对盐胁迫的多重反应。基因(巴塞尔)。2017;8(12):372。
- 26。
朱杰克。植物中的盐和干旱胁迫信号转导。Annu Rev植物Biol。2002; 53(1):247-73。
- 27。
朱Q,张俊,高X,童俊,肖L,李W,张H.拟南芥AP2 / ERF转录因子RAP2.6参与ABA,盐和渗透胁迫应答。基因。2010; 457(1-2):1-12。
- 28。
杨奥,波普鹿,萨特夫u,leking i,dietz kj,golldack d。拟南芥碱性亮氨酸拉链转录因子Atbzip24调节涉及非生物胁迫性的复杂转录网络。基因。2009; 436(1-2):45-55。
- 29。
程麦,廖PM,Kuo WW,林TP。拟南芥乙烯响应因子1通过与不同的CIS作用元件结合响应于不同的应力信号来调节非生物应激响应基因表达。植物理性。2013; 162(3):1566-82。
- 30
AtMyb41调节拟南芥对渗透胁迫的转录和代谢反应。植物杂志。2009;149(4):1761 - 72。
- 31
关键词:水稻,mir164, NAC基因,抗旱性[J] .机器人。2014;65(8):2119-35 .]
- 32
姜毅,Deyholos MK.拟南芥NaCl诱导的WRKY25和WRKY33转录因子在非生物胁迫中的功能表征.植物分子生物学.2009;69(1-2):91-105。
- 33.
江y,杨b,deyholos mk。拟南芥BHLH92转录因子在非生物应激中的功能表征。Mol Gen Genomics。2009; 282(5):503-16。
- 34.
PXY-CLE41受体配体对定义了一个控制血管细胞分裂的速率和方向的多功能途径。发展。2010;137(5):767 - 74。
- 35.
Kucukoglu M,Nilsson J,Zheng B,Chaabouni S,Nilsson O. Wuschel相关的Homeobox4(WOX4) - 麦克基基因调节山脉分裂活动和杨树中的二次生长。新植物。2017; 215(2):642-57。
- 36。
杨洋。植物抗旱性的研究进展[j]。植物学报2011;30(8):1383-91。
- 37。
Kim JM,Sasaki T,Ueda M,Sako K,Seki M.植物中染色质对干旱、盐分、热和冷胁迫的响应。前沿植物科学。2015;6:114。
- 38。
卢X,张X,段H,莲C,刘C,Yin W,XIA X.三种应激响应性NAC转基因转基因植物中的盐和耐旱性耐旱性。physiol植物。2018; 162(1):73-97。
- 39。
关键词:番茄,bZIP转录因子,SlAREB,水分亏缺,盐胁迫足底。2010;231(6):1459 - 73。
- 40。
刘杰,石谷明,Halfter U,金世忠,朱家杰。拟南芥SOS2基因编码一种耐盐所需的蛋白激酶。中国科学院院刊。2000;97(7):3730-4。
- 41。
熊磊,朱建军。寒冷、干旱和盐胁迫下的细胞信号。植物细胞。2002;14(增刊):s165 - 83。
- 42
姜旭,金文耀,阿里Z, Fujii H, Mendoza I, Yun DJ, Zhu JK,等。通过自抑制c端结构域的磷酸化激活质膜Na/H逆向转运体盐敏感1 (SOS1)。中国科学院院刊2011;108(6):2611-6。
- 43
Martinez Atienza J,Jiang X,Garciadeblas B,Mendoza I,Zhu JK,Pardo JM,Quintero FJ.水稻盐敏感途径的保护.植物生理学.2007;143(2):1001-12。
- 44
Olias R,Eljakaoui Z,Li J,De Morales PA,Marin Manzano MC,Pardo JM,Belver A.质膜Na+/H+逆向转运蛋白SOS1对番茄的耐盐性至关重要,并影响植物器官之间Na+的分配.植物细胞环境.2009;32(7):904-16。
- 45.
唐荣杰,刘洪,鲍勇,吕启东,杨玲,张海霞。木本植物杨树在响应盐胁迫中具有一个功能保守的盐过敏感途径。acta botanica sinica(云南植物学报),2010;40(4):457 - 461。
- 46.
Gaxiola RA, Li J, Undurraga S, Dang LM, Allen GJ, Alper SL, Fink GR. AVP1 H+泵的过表达导致植物耐干旱和耐盐。中国科学院院刊2001;98(20):11444-9。
- 47.
杨y,唐rj,李b,王hh,金yl,江厘米,宝y,su hy,zhao n,ma xj等。过表达杨树Trichocarpa H + -pyrophosphatase Gene PTVP1.1对转基因杨树的耐盐性赋予耐盐性。树生理。2015; 35(6):663-77。
- 48。
脱落酸与非生物胁迫耐受性的不同调控层。植物生理学报。2014;171(7):486-96。
- 49。
植物激素介导的胁迫反应调控。BMC Plant Biol. 2016;16:86。
- 50.
Unterholzner SJ,Rozhon W,Papacek M,Ciomas J,Lange T,Kugler Kg,Mayer Kf,Sieberer T,Poppenberger B. Brassinesteroids是拟南芥中吉布林素生物合成的母稳态因素。植物细胞。2015; 27(8):2261-72。
- 51。
任国华,李丽,黄玉华,王玉华,张伟,郑瑞,钟超,王旭东。WIP锌指蛋白GhWIP2通过介导赤霉素、脱落酸和生长素之间的相互作用抑制非洲菊细胞增殖。新植醇。2018;219(2):728 - 42。
- 52。
盐胁迫通过积累脱落酸和乙烯产生的相关变化抑制柱花草种子的萌发。植物生理生化学报。2018;30:397 - 398。
- 53。
赤霉素信号通路在植物对非生物胁迫响应中的作用。临床生物学杂志。2014;217(p1): 67-75。
- 54
关键词:拟南芥,干旱胁迫,RD29B, ABA,转录记忆植物j . 2014; 79(1): 150 - 61。
- 55
d'urso a,icknner jh。表观遗传转录记忆。Curr Genet。2017; 63(3):435-9。
- 56
Chinnusamy v,Zhu JK。植物应激反应的表观遗传调节。CurrOp植物BIOL。2009; 12(2):133-9。
- 57.
acta botanica yunnanica(云南植物研究中心);acta botanica yunnanica(云南植物研究中心);足底。2009;231(1):27-34。
- 58.
高渗透启动拟南芥幼苗建立了长期的体细胞记忆,并伴随表观基因组的特定变化。基因组医学杂志。2013;14 (6):R59。
- 59.
Super诱导脱水应力记忆基因的Super-Oraphicopsis Zaliana的Super脱水应力记忆基因,ZH3K27ME3和H3K4ME3 H3K4ME3和H3K4ME3染色质环境。莫尔植物。2014; 7(3):502-13。
- 60.
Kim JM,TK,Ishida J,Matsui A,Kimura H,Seki M.染色质地在拟南芥中的干旱胁迫下恢复过程中的转变。植物细胞生理。2012; 53(5):847-56。
- 61.
Wibowo A,Becker C,Marconi G,Durr J,Price J,Hagmann J,PapareDy R,Putra H,Kageyama J,Becker J等人。拟南芥中的Hypermotmist Regress Memory由基因组中的不同外观不稳定位点介导,并且通过DNA糖基酶活性受到雄性种系。Elife。2016; 5。
- 62.
Pertea M, Kim D, Pertea GM, Leek JT, Salzberg SL. HISAT、StringTie和Ballgown RNA-seq实验的转录水平表达分析。Nat Protoc。2016;11(9):1650 - 67。
- 63。
Kim D,Langmead B,Salzberg SL.HISAT:一种低内存要求的快速拼接对准器.Nat方法.2015;12(4):357-60。
- 64。
Anders S,Pyl Pt,Huber W. htseq - 使用高吞吐量排序数据的Python框架。生物信息学。2015; 31(2):166-9。
- 65。
Wibowo A,Becker C,Marconi G,Durr J,Price J,Hagmann J,PapareDy R,Putra H,Kageyama J,Becker J等人。拟南芥中的Hypermotmist Regress Memory由基因组中的不同外观不稳定位点介导,并且通过DNA糖基酶活性受到雄性种系。Elife 2016; 5:E13546。
- 66
加权相关网络分析的R包。BMC Bioinf。2008;9:559。
- 67
Shannon P, Markiel A, Ozier O, Baliga NS, Wang JT, Ramage D, Amin N, Schwikowski B, Ideker T. Cytoscape:一种生物分子相互作用网络集成模型的软件环境。基因组研究》2003;13(11):2498 - 504。
- 68
金娟,田芳,杨大成,杨志强,孔林,罗娟,高光。植物ttfdb 4.0:转录因子的中心枢纽及其调控相互作用。核酸Res. 2017;45(D1): D1040-5。
- 69.
Falcon S,Gentler R.使用GOstats测试GO术语关联的基因列表。生物信息学。2007;23(2):257-8。
- 70.
Supek F,Bosnjak M,Skunca N,Smuc T. Revigo总结并可视化了基因本体论术语的长名单。Plos一个。2011; 6(7):E21800。
- 71.
王Y,歌,朱茹,张某,杨y,陈t,唐b,dong l,ding n,zhang q等。GSA:基因组序列存档。基因组学蛋白质组学生物信息学。2017; 15(1):14-8。
- 72.
大数据中心成员:2018年大数据中心数据库资源。核酸的研究。2018; 46 (D1): D14-D20。
致谢
我们感谢Jiu-Hai Zhao教授,为计算服务器和数据分析管道,以及Qing-Hua Shi教授进行了建议对液体培养的建议杨树.我们感谢Li liu教授的宝贵讨论和意见。
资金
中国天然科学基金会支持李军刘国家自然科学基金的支持(批准31700583),并从广东省南方农业大学森林植物种质创新开发和利用的开放资金。资金机构在研究中没有作用,在收集,分析和解释中没有作用或写作稿件。
作者信息
隶属关系
贡献
LL,AW和XH构思和设计了实验。JL,TY和WC进行了实验和原始数据生成。XH分析了数据。LL,JL和XH写了稿件,CF和BW修订了手稿。所有作者阅读并认可的终稿。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
提交人声明他们没有竞争利益。
附加信息
出版商说明
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
附加文件
附加文件1:
图S1。全植物沿着NaCl治疗时间的反应。A,没有盐处理的植物;B,用100mm NaCl处理的植物;C,用100mM NaCl处理的植物,然后用3天回收,然后用200mM NaCl处理;D,植物直接用200mM NaCl处理。在左侧指示的时间点拍摄照片。(JPG 17970 KB)
附加文件2:
图S2。基因簇树木图基于时间课程RNA-SEQ数据。(TIF 4128 KB)
附加文件3:
图S3。在绿色模块中富集的生物途径(BP)。(PNG 206 KB)
附加文件4:
图S4。绿松石模块中富集的生物途径(BP)。(PNG 255 KB)
附加文件5:
图S5。蓝色模块中富集了生物通路(BP)。(PNG 301 kb)
额外的文件6:
图S6。具有RNA-SEQ数据的绿色(A),绿松石(B)和蓝色(C)模块的代表性基因的表达谱。(PDF 291 KB)
额外的文件7:
表S1。测序数据摘要。(XLSX 12 KB)
额外的文件8:
表S2。个体时间点与相应的控制样本之间的差异(DEGs)的总结(edgeR分析识别)。(XLSX 9 kb)
额外的文件9:
表S3。edgeR分析确定的单个时间点和相应对照样品之间的DEG。(XLSX 18432 kb)
附加文件10:
表S4。WGCNA分析衍生的共表达模块综述。(XLSX 9753 kb)
额外的文件11:
表S5。所有WGCNA模块中的TF基因。(XLSX 770 KB)
附加文件12:
表S6。绿色,绿松石和蓝色模块中的TF系列摘要。(XLSX 11 kb)
附加文件13:
表S7。所有模块的丰富术语。(XLSX 2113 KB)
附加文件14:
表S8。共同表达的前20个基因WOX4b和CLE41a从我们的RNA-seq数据。(XLSX 11 kb)
附加文件15:
表S9。用于QPCR的引物。(XLSX 11 kb)
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条中提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
刘,詹。那Han, X., Yang, T.et al。全基因组对盐胁迫的转录适应杨树。BMC植物BIOL.19,367(2019)。https://doi.org/10.1186/s12870-019-1952-2
收到:
公认:
发表:
关键字
- 非生物压力
- 适应
- 多年生植物
- 杨树
- 基因模块