摘要
背景
累积的证据表明,SPL.s是植物抗非生物胁迫的关键调控因子和高度保守的模块miR156/SPL.似乎平衡植物生长和压力反应。哈菲特Tamarix chinensis.对盐树具有高度的抗性。SPL.年代的t对(TcSPLs)他们的盐压力反应中的角色仍然难以捉摸。
结果
在本研究中,我们进行了系统的分析TcSPLs基因家庭,包括属于7组的12名成员。物理化学性质和保守的基序在每组中的群体和相似性之间表现出分歧。MicroRNA响应元素(MRES)在位置和序列中保存,除了第一个MRE之外TcSPL5. MIR156目标SPL.s通过MRE-miR156相互作用的双荧光素酶报告基因检测鉴定。数字表达基因簇显示mir156靶向的潜在不同功能SPL.年代vs一道SPL.S对盐胁迫的反应。mir156靶向的表达模式分析SPL.s与TcmiR156呈反向表达趋势,提示1 h(盐胁迫时间)可能是盐胁迫反应转录后调控的关键时刻。
结论
我们的工作证明的后转录调控miR156靶向TcSPLs和转录监管非目标TcSPLs在盐压力响应中,令人有帮助阐述miR156 /SPL.介导的分子机制t对盐胁迫宽容。
背景
转录因子(TFS)是与靶基因的CIS作用元素结合的蛋白质,以激活或抑制其转录[1].数十种植物TF基因家族精确地协调了与非生物应激相关的下游基因的空间和时间表达,例如AP2(Apetala2),NAC(NAM,ATAF1 / 2和CUC2),生长素响应因子(ARFS)和Squamosa启动子- 粘合(SBP)蛋白质样蛋白(SPL)[2,3.,4,5].SBP-box是由两个锌指结构组成的具有高度保守DNA结合域的植物特异性tf [6].彻底建立了植物发育中SPLs在植物发育中的基本作用,包括芽和叶形态发生,花器官发展,开花和果实成熟[7,8].
最近的研究表明SPL.在许多植物中,基因是抗非生物胁迫的关键调节因子[9,10.,11.].基因组型材研究SPL.植物物种的家庭已经找到了许多SPL.响应盐,干旱,冷和热应力[12.,13.,14.].其中一些SPL.通过调节信号转导、活性氧(ROS)清除、POD/SOD积累、脯氨酸合成和花青素代谢等相关基因的丰度来参与盐/干旱胁迫响应。例如,vpsbp16.的葡萄属pseudoreticulata增强过表达的盐和干旱胁迫耐受性拟南芥通过调节盐过度敏感(SOS)和ROS信号传导[10.].BpSPL9具有耐盐耐旱能力Betula Platyphylla.通过改善活性氧的清除[15.].
此外,一个子集SPL.s的miRNA的响应元件(MRE)互补miR156通过miR156进行调节介导的mRNA切割和/或翻译抑制在转录后水平[16.,17.].例如,SPL13miRNA156引起的沉默改善了苜蓿的干旱胁迫耐受性[18.].mir156 /SPL.越来越多的研究表明,miR156/SPL.模块作为平衡植物非生物胁迫反应和发育的关键桥梁出现[9,11.].在拟南芥而在水稻中,miR156过表达增加了对盐胁迫的耐受性和延迟开花。miR156系列和SPL9过表达系通过花青素代谢调控表现出相反的表型(对盐胁迫敏感和早花)。在miR156/中也发现了类似的结果SPL3开花时序及热力记忆研究拟南芥.累积证据表明MIR156 /SPL.模块在陆生植物中高度保守,似乎是植物生长(生物量/产量/流动时间)和抗逆性改善的有用分子工具[19.,20.].作为一个典型的案例,OsSPL14和OsSPL16在水稻提高粮食品质和产量以及SPL.玉米同源办[21.,22.].此外,下调SPL8改善了转基因苜蓿的生物质产量和盐/耐旱性,表明miR156靶向SPL8在豆类育种上有相当大的潜力[23.].
Tamarix chinensis.(t对)作为对盐度具有高抗性的卤素物,是用于研究植物盐胁迫耐受性的合适材料[24.,25.].虽然已经确定了与盐或干旱胁迫相关的许多TF基因Tamarix SPP..[26.,27.,28.,29.,30.,31.], 这SPL.年代的t对(TcSPLs)它们仍有待阐明盐应激反应的作用。在这项研究中,系统地识别TcSPL家族分类、结构分析和表达谱分析提供了序列表征TcSPLs以及盐胁迫下的表达模式。此外,我们还对mir156靶向的表达模式进行了初步研究SPL.通过实验验证,确定其在盐胁迫反应中的作用。
结果
的识别TcSPL基因和miR156目标
进一步探讨其重要作用SPL.基因在t对(TcSPLs)的cDNA序列TcSPL从RNA-SEQ数据预测家庭[32.].全部14TcSPLs, 除了TcSPL13和TcSPL14,包含保守的sbp -box(表1).的六名成员TcSPL含有家族的miRNA反应元件(MRE)是TcmiR156的潜在目标(表2).值得注意的是,两个女朋友预测TCSPL5,包括mRNA切割位点和翻译抑制位点。
推导出的蛋白质长度、等电点(pI)和分子量TcSPL蛋白是一个大的变化范围内(表2).TcSPL蛋白质从191(TcSPL4)至1098(TcSPL9)氨基酸的预测分子量为21.7 (TcSPL4)至120.7(TcSPL9)KDA,PI值范围从6.28(TcSPL5)至9.84(TcSPL2).这些结果表明了SPL蛋白家族的多样性t对.
系统发育分析SPL.基因
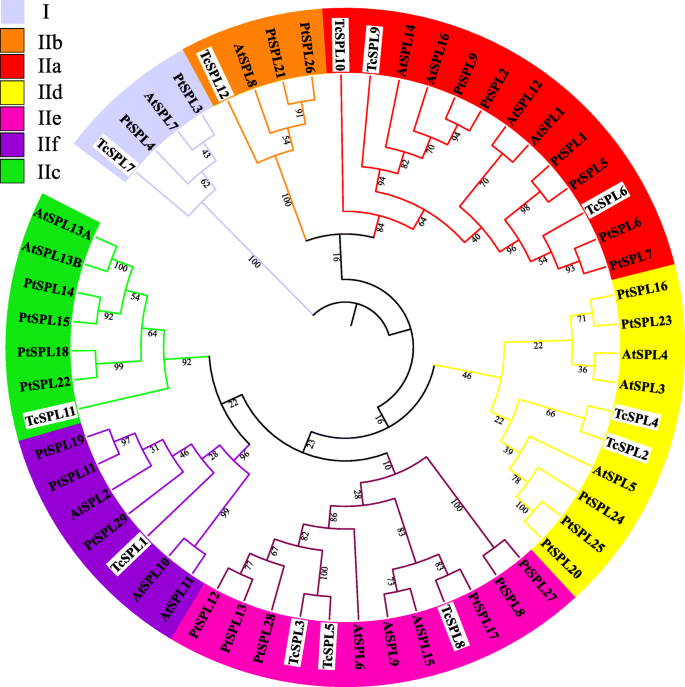
的SPL.基因功能已在国内外得到充分阐明拟南芥和人口众多因为它在植物发育调节中的重要性。进一步研究SPL.之间的进化关系t对,拟南芥(http://planttfdb.cbi.pku.edu.cn/family.php?sp=ath&fam=sbp.), 和人口trichocarpa(http://planttfdb.cbi.pku.edu.cn/family.php?sp = ptr&fam=sbp.),SBP盒序列为12TcSPLs,17atspls.从拟南芥30.PtSPL年代从P. Trichocarpa.用于构造最大似然法(m1)树(图。1和表格2).所有59SPL.基因被分成7组,至少包含一个SPL.从每个物种(图1).ML-tree的拓扑结构及其对应SPL.成员与多个一致SPL.根据代表性的研究的研究,研究和群体被命名为I和IIA-F组。
IID包括最小的SPL.IIa的成员编码的氨基酸最多,而IIa的成员编码的氨基酸最多SPL.蛋白质,约1000个氨基酸。类似的蛋白质特征SPL.在每个组中,S表明SBP-Box是一个相对节约的域SPL.不同物种的基因,其保存程度与物种的相同SPL.蛋白质。此外,除了IIC-F的所有成员之外,保守的MRE是识别的TcSPL11和PtSPL11.
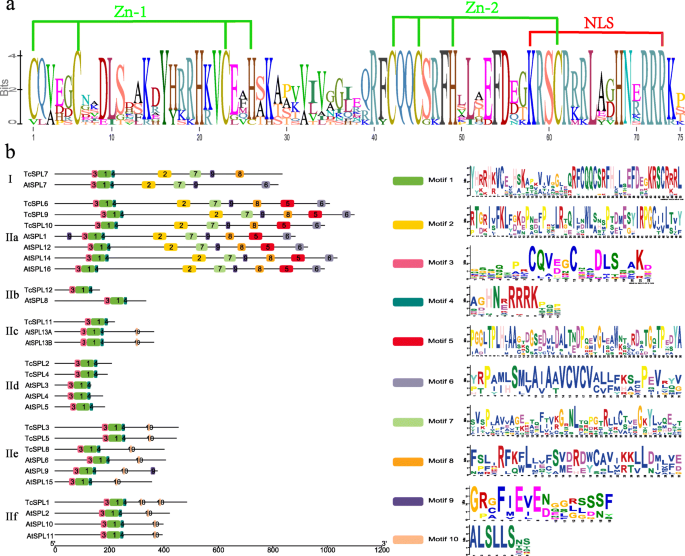
在保守基序鉴定TcSPLs
所有全部分享的SBP盒TcSPLsNCBI CD-Search (conservationdomain Search Service)预测。序列比对(图。2a)显示SBP盒TcSPLs是包含两个锌指基序(Zn-1/2)和一个核定位信号(NLS)的保守结构。Zn-1基序为CCCH (C3H)型锌指结构TcSPLs,除了第一个CYS残留物TcSPL8代Val残基,CCCC (C4)型在TcSPL7. Zn-2基序(即,C2HC型锌指结构)全部保守TcSPLs,这与重叠的NLS与四个残基。
在MEME中预测的前8个最保守的基序和2个相对保守基序被指定为基序10(图。2b)。大多数域之间相对节省了t .摘要和拟南芥[12.].同一组的成员具有类似的主题组合物。图案3,1和4代表Zn-1/2和全部识别的SBP盒的NLSTcSPLs.TcSPLs的IIb和IId只包含SBP-box的图案。一些主题特别出现在一两个组中。Motif 5为IIa所特有,而Motif 2、6、7和8为I和IIa所特有(图5)。2b)。对于2个相对基序,在四分之一中发现了包括含有ankylin重复域的基序9TcSPLs(TcSPL6,− 7, − 9, − 10), indicating that the biological function of theseTcSPLs可能与蛋白质相互作用有关。此外,motif 10由miR156的MRE编码的保守motif组成。
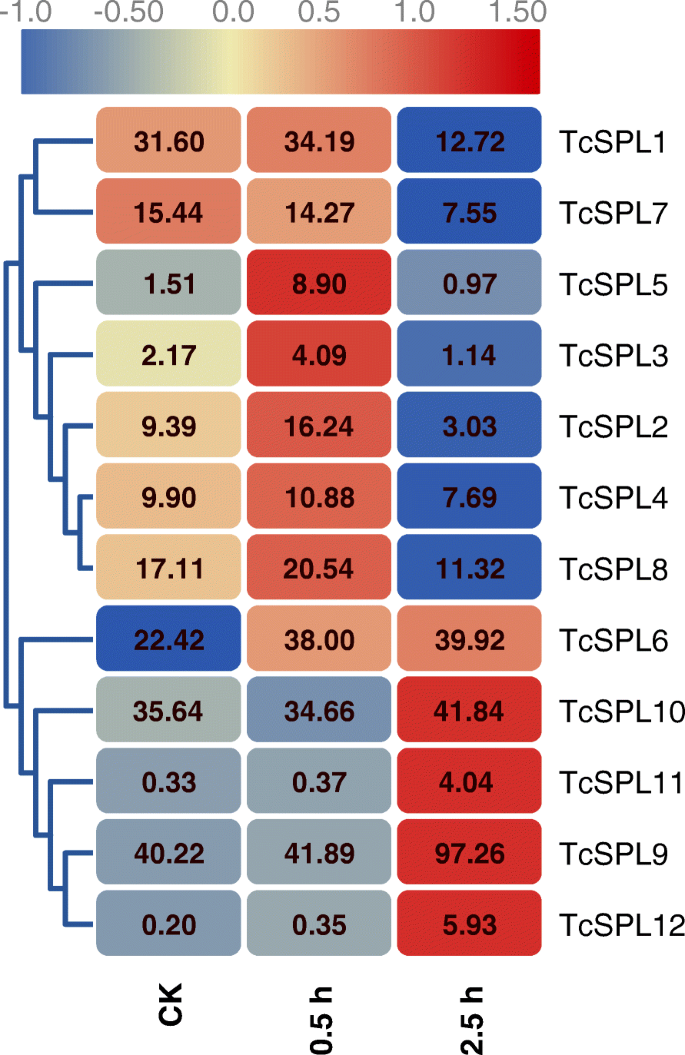
数字基因表达概况TcSPLs盐胁迫下
系统发育和蛋白质基序分析显示了保护和多样性TcSPLs.数字基因表达谱(DGE)的TcSPLs在t对构建了根,以探讨其在盐胁迫中的潜在作用。基于0、0.5、2.5 h NaCl处理时间序列的聚类分析,12TcSPLs被分为两个集群,即一个下调表达式模式和另一个上调表达式模式集群(图。3.).所有TcSPLs用的MRE聚为下调的簇与其他的相反的表达模式TcSPLs(除了TcSPL7),这意味着TcmiR156针对性TcSPLs在可以扮演不同的角色t对与非靶向相比的盐应激反应TcSPLs.TcSPL3和TcSPL5的the downregulated cluster showed a significant upregulation in 0.5 h, indicating that the expression patterns of the twoTcSPLs很复杂。五TcSPLsshowed relatively high abundance (FPKM > 20), and three of them (TcSPL6,−9,−10)属于同一组(IIa)。
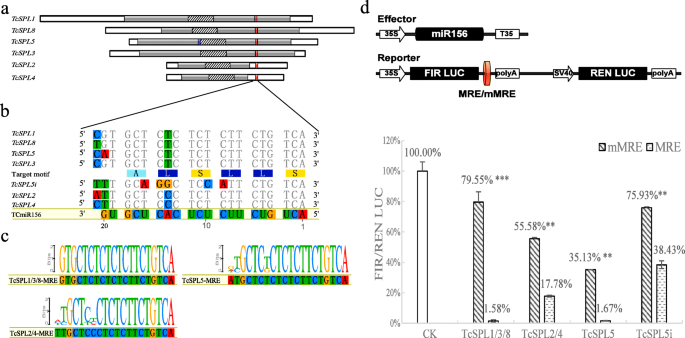
验证TCMIR156目标TcSPLs
六个TcSPLs靶向的TcmiR156含有7个MREsSPL.的mRNA:6倍mRNA切割的MRE(SPL.-MRE)和一个翻译抑制MRE (tcspl5i.绝笔)。所有SPL.-mres位于保守的SBP-Box的下游,是最后一个外显子的编码序列(CDS)的一部分(附加文件3.:图S1)与外TcSPL2绝笔,TcSPL4- 位于3'未转换区域(UTR)中。不像SPLc研究硕士,tcspl5i.- MRE位于第一个外显子并与SBP箱的4个上游核苷酸重叠(图。4一种)。TCMIR156的对准反向补充SPL.研究硕士(无花果。4b)显示完美的互补性TcSPL-MREs与TcmiR156的5 '和TcmiR156的3 '有两次不匹配(第14个和最后一个核苷酸)。除了第14和最后一个核苷酸外,TcmiR156还有4个错配(第9、10、15和16个核苷酸)表明TcSPL5可以由MiR156禁止翻译tcspl5i.-mre。MiR156相关的MRE除外tcspl5i.-MRE是保守的,假设对齐SPL.orthologues拟南芥和人口众多[12.,33.].TcSPL1/3/8-MRE与mre在最后一个外显子中完全保守SPL.s(atspls.和PtSPL年代),TcSPL2/4-MRE在3'UTR中高度保守SPL.S(图4c)。TcSPL1/3/8绝笔,TcSPL5- 在相同的阅读框架中,参与编码嵌入在非保守蛋白质区中的保守基序(图)(图。4c)。特别是tcspl5i.-MRE在5'中保存不足,并用不同的帧编码非保守的主题LQAPFC。
mir156靶向的鉴定与验证SPL.年代的Tamarix chinensis.(TcSPLs).一个所有cdna的图形表示TcSPLs携带着预测的MRE(红框和蓝框)。编码序列采用灰色阴影,保守的SBP-box采用对角线阴影。b预测的MRE序列与MiR156逆向互补序列(TCMIR156)相比对准。与TCMIR156相比的核苷酸分歧是用颜色突出的。通过三重密码子表示阅读框架,目标基质由氨基酸残基表示。cMRE的序列标志TcSPLs和假定的SPL.orthologues拟南芥蒂利亚纳和杨树trichocarpa(atspls.和PtSPL年代)。TcSPL1/3 / 8-MRE与,TcSPL2/4 mre对齐,TcSPL1/3 / 8-MRE与,和tcspl5i.-MRE有针对性的主题与。d双荧光素酶报告(DLR)测定的示意图。记者和效应被转移到杨树过夜转化后的原生质体。Firefly Luciferase至Renilla Luciferase的四个MRE的比例代表了MIR156-的活性TcSPL交互。MMRE在TCMIR156的10-11核苷酸中突变。数据由SES的百分比表示(n = 4). Significant differences were determined by Student’s t-test (**P< 0.01, * * *P < 0.001)
四种类型的mir156相关的MRE,具体TcSPL1/3/8 -绝笔,TcSPL2/4-MRE,TcSPL5-MRE和tcspl5i.-MRE,通过双荧光素酶报告基因(DLR)检测验证为TcmiR156的靶位点(图)。4d)。对照组的荧光强度(即,DLR载体和miRNA载体共转化)设定为100%。Compared with the control group, fluorescence decreased in four MRE groups (i.e., MRE vectors and miRNA vector co-transformation) and recovered in four mutated MRE (mMRE) groups mismatched to 10th and 11th nucleotides of TcmiR156 (i.e., mMRE vectors and miRNA vector co-transformation) to different intensities. The results demonstrated thatTcSPL1/3/8 -mre和TcSPL5-MRE被TcmiR156显著抑制(< 2%的荧光)TcSPL2/4 -mre和tcspl5i.-MRE被TcmiR156抑制到一定程度(荧光< 40%)。高荧光(> 50%)在mMRE组除TcSPL5-mmre和显着的复苏(P< 0.01)表明第10和第11核苷酸是miR156-的关键位点TcSPL交互。DLR分析表明TcSPL1 / 2/3/4/5/8由TCMIR156以MRE相互作用的方式定位。
miR156目标的盐应激响应TcSPLs
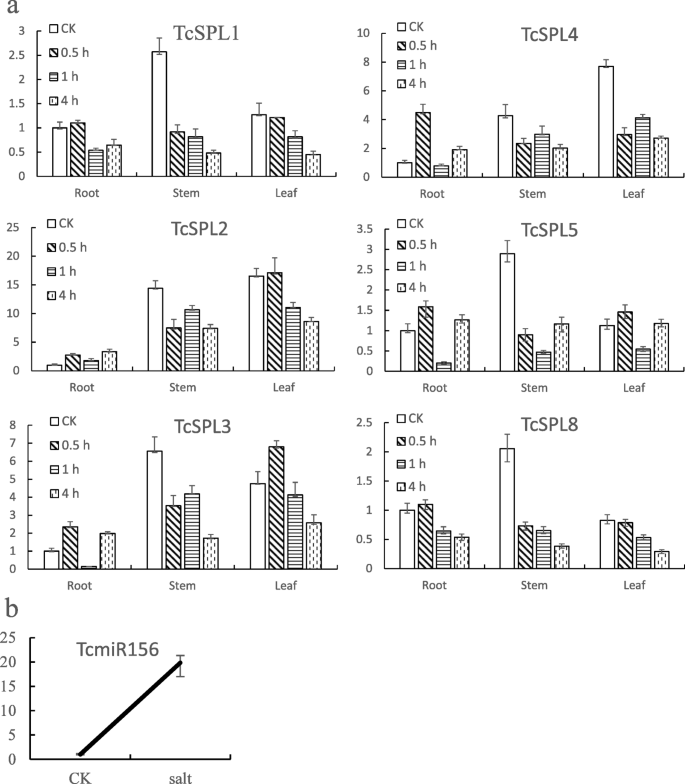
为了探索盐应激响应模式,在2%NaCl处理期间,在根,茎和叶中检查六种miR156靶基因的表达水平(图。5一个)。TcSPLs在三种组织中表达的表达没有组织特异性表达模式。平均表达水平表明,三个组织的下降mRNA丰富如下:茎,叶和根。特别是丰富的SPL.S IN茎是2-14大于在根没有盐胁迫下次,并SPL.在根和茎之间,2丰度显着不同。
盐应激反应模式SPL.S在同一组织中表现为两种时间表达模式,即地上组织响应模式和地下组织响应模式。对于射击反应模式,SPL.S先下调(0.5 h),然后稳定(1 h和4 h)。其中茎(1.9 ~ 6.2倍)的下调显著高于叶(1.8 ~ 2.8倍)SPL.S在茎的盐应激反应中发挥重要作用。对于根响应模式,TcSPLs受盐胁迫时间增加的“上下向上”调节。特别是,TcSPL3 TcSPL4,和TcSPL5在1小时盐处理的根中被大致压抑。有趣的是,TcSPLs在NaCl处理1 h时,所有组织的表达均下调,提示1 h是时间表达模式的关键时间点。因此,我们进一步检测了2% nacl处理后TcmiR156的表达水平t对(无花果。5b).实验结果表明,盐胁迫反应TcSPLs与TcmiR156表达水平呈负相关。所有结果表明TcSPLs在TCMIR156的阴性控制下,在盐应激反应中起重要作用。
讨论
保守和分歧TcSPLs
SPL.家庭已经在绿藻,苔藓,lycophyte和种子植物已经确定,他们的DNA结合结构域SBP-箱在整个植物界中高度保守[8].我们的研究确定了14个TcSPL基因与保守的SBP箱(两个不完整的SBP盒)。7组系统发育树的分类和名称密切关注群体假设SPL.进化研究[8],除了组CR绿藻SPL.年代。SPL.源于陆地植物,我最古老的群体SPL.使用c4型锌指,包括TcSPL7,而其他TcSPLs使用C3H型锌指分为较年轻的IIA-F.另外,位于Zn-1结构的N末端的保守DLS基序的第三残基分歧TcSPL7和TcSPL12,这表明TcSPL12似乎相对较早起源。独特的图案,包括图案5(IIA独特的IIA)和图案2,6,7和8(I和IIa的独特),表明I和IIA可能与SBP系列的功能多样性相关联。
根据数量SPL.家庭成员,SPL.不同植物的科大致可分为两种类型:(1)典型SPL.家庭,如SPL.家庭Physcomitrella patens.[34.,番茄35.],拟南芥, 白饭 [36.]与的13,15,16,19名成员,分别与(2)膨胀SPL.家庭由于基因重复,如SPL.杨树的家庭[12.]和玉米[14.]分别为28和31名成员。的TcSPL有14名成员的家庭应该属于规范家庭。与一件事不同PtSPL副骨头或更多符合一个AtSPL基因,只有2个副骨头(TCSPL2 / 4.和TCSPL3 / 5.)的预测TcSPLs,数量TcSPLs基本上和那样的AtSPL在每组。这些结果表明SPL.基因重复甘蒙柽柳和拟南芥仍然线性依赖,似乎发生在谱系发散之前。规范的情况类似SPL.家庭如米,却在相反的扩张中SPL.科,如杨树在谱系分化后有节段复制。
内的绝笔TcSPLs
MiRNA靶标网站内SPL.S可能存在于苔藓和维管植物分化之前[8].在这项研究中,几乎所有SPL.IIC-IIF中的S有MiRNA靶标在MiR156的靶网站,MRE的位置和序列都被保守TcSPLs.这与结果一致拟南芥和杨树SPL.S [12.,33.],建议SPL.转录后调控是植物类似。六倍规范的MRE和非规范tcspl5i.- 是预测的TcSPL1-5和- 8.TcSPL1/3/5 /位于其最后的外显子8的MRE具有较高的保护比TcSPL2/4-的MRE位于其3'-UTR。据更高的保护和保守的靶基序ALSLLS的证据,我们认为,位于最后一个外显子的MRE可能遭受比UTR更多的选择压力。为了tcspl5i.- 它的表征是所有推定的非规范SPL.直系同源物,如互补性较差,miR156,低级配对吉布斯自由能,第一外显子的位置和靶基序。我们推测这一点tcspl5i.- 似乎是一个特定的翻译抑制位点SPL.家庭相比atspl3.[33.].DLR实验证实了miR156与MREs的相互作用,且相互作用强度由高到低依次递减SPL.1/3/8-MRE > SPL5 > SPL2 / 4 > SPL5i,这与它们序列的互补性正相关。此外,DLR分析结果显示,10-11个核苷酸是所有典型MREs的关键位点,以及tcspl5i.- 似乎证明了翻译抑制的唯一性。
miR156目标的响应模式TcSPLs
DGE分析表明,TcSPLs可能在应对盐胁迫中发挥不同的作用。的一道TcSPLs除了TcSPL7是诱导的,同时是靶向的TcSPLs被盐抑制。在玉米方面,57% (11/19)SPL.S被盐或/和干旱抑制,而且大多数是非靶向的SPL.s的诱导[14.],也表明目标之间的表达式模式SPL.s和非目标SPL.小号有所不同。此外,高度丰富的非目标TcSPLs(TcSPL6,−9,−10)可能与IIa中确定的许多独特图案有关。
此外,我们调查了靶向的时间表达模式TcSPLs在不同的组织中确定其盐应激应激特征。目标TcSPLs在快速下调后,在叶片/茎保持稳定,显示出简单的抑制响应模式与盐胁迫。抑制作用在茎中更占优势。在根,有针对性的TcSPLs表现出一种复杂的“自上而下”调节的反应模式。盐胁迫0.5 h时表达上调与DGE分析一致。在盐胁迫1 h时,在2.5 h的DEG谱中,DEG的下调是一致的,这表明与茎/叶相比存在延迟抑制。在盐胁迫4 h后,随着丰度的恢复,目标物TcSPLs在盐胁迫下呈谷型(U曲线)表达模式,1h是一个关键的反转时间点。TcmiR156在1 h盐胁迫下与茎环qPCR呈显著负相关,证实了这一可能性。负表达趋势是mirna靶谱调控的指标[37.,38.].的TcSPLs在TcmiR156的负调控下,在盐胁迫响应中发挥重要作用。我们认为,TcmiR156可能通过负调控在盐胁迫响应中发挥重要作用TcSPLs.许多研究表明,两者之间存在功能冗余TcSPLs在植物开发法规中[39.,40].类似的盐应激响应模式表明6 miR156目标TcSPLs在转录后调节中可能是多余的。因此,MIR156目标的功能研究TcSPLs应该采用实验设计,避免由于冗余导致的潜在障碍。
结论
在本研究中,我们进行了系统的分析SPL.基因家庭t对,检查12TcSPL基因分成7组。所示的保守基序和MREs显示出组间差异和组内相似性。DEG提示mir156靶向可能有不同的功能SPL.年代vs一道SPL.S在盐胁迫反应中。MREs在位置和序列上都是保守的,除了tcspl5i.-mre。的miR156-targetedSPL.通过MRE-MIR156相互作用的DLR测定证明了S.不同组织的表达模式表明,1小时(盐应力时间)可能是miR156目标的临界时间点SPL.盐应激反应。我们的工作为进一步转录后调节研究提供了基础SPL.s,并有助于确定mir156 /SPL.介导的分子机制t对盐胁迫宽容。
方法
植物材料
所有材料均采自同一品种1个月的幼苗t .摘要奥托特在南京林业大学(江苏南京)校园里种植。将播种的根部浸入340mM NaCl溶液中,0小时,0.5小时,1小时和4小时。在收集后清洁根,茎和叶子,并立即在液氮中冷冻并储存在-80℃。
预测与鉴定TcSPL和其他来源:
从RNA-SEQ数据的组装转录物中获得基因和蛋白质序列[32.].通过隐藏的Markov模型(HMM)预测来自PFAM的SBP域配置文件(PFAM03110,http://pfam.sanger.ac.uk/),TcSPL成绩单是预测。所有的候选人TcSPL通过BLASTP搜索,在植物转录因子数据库中进一步鉴定出含有sbp -box的基因。全cDNA序列TcSPL1-5根据SMARTer RACE 5 ' /3 ' Kit (Clotech, CA, USA)手册,采用cDNA末端快速扩增(Rapid Amplification of cDNA End, RACE)鉴定,略有改进[41.,42.,43.].预测的完全CDSTcSPL6-10通过序列扩增鉴定。TcSPL11-14缺乏完整的CD,它们的部分CD或蛋白质序列用于以下分析。TcSPL13,−14个完整SBP-box的缺失在系统发育分析中被拒绝。所有序列都是列出的附加文件1:表S1。
另外,基因和蛋白质序列AtSPL沙PtSPLs从中下载拟南芥信息资源数据库(http://www.arabidopsis.org/)及Phytozome数据库(https:////phytozome.jgi.doe.gov/),分别(附加文件1:表S1)。
序列比对和系统发育分析
的sbp盒的多重序列比对TcSPLs,AtSPL沙PtSPLS通过对准方法肌肉获得。使用Mega 7.0进行系统发育分析[44.]与邻接法和1000个引导重复。
主题分析和MRE分析SPL.基因
使用PROTPARAM程序计算诱剂蛋白质的氨基酸序列长度,分子量和等电点(http://web.expasy.org/protparam/).使用模因程序(http://meme-suite.org/tools/meme.),使用默认参数。的miR156-targetedSPL.psrnatarget预测基因和女士(http:// plantgrn.noble.org/psRNATarget/)。内部MRE的比较TcSPLs用mir156和养护mreTcSPLs,AtSPL沙PtSPL通过与软件群集X的多个序列对齐进行分析S [45.].
表达分析TcSPL由TCMIR156为目标
来自不同组织的总RNA提取t对根据制造商的说明,使用RNAPREP纯植物套件(DP441,Tiangen,北京,中国)和Primescript™RT主混音RT Master(RR036Q,大连,中国),根据制造商的说明,实现了cDNA合成。QRT-PCR分析在VIIA 7实时PCR系统(ABI,加利福尼亚,美国)进行了饱和染料Evagreen(Biotium,CA,USA),然后推荐QPCR主混合物反应混合物和扩增程序。在附加文件中列出的基因特异性引物2:表S2设计采用ExpressionsUite软件(ABI,CA,USA)。使用2计算相对表达水平-ΔΔct基于参考基因TCTIF的方法(附加文件2:表S2)。
TCMIR156的表达分析
每1μg混合组织的总RNAt对在无胁迫和1 h NaCl胁迫条件下,均进行逆转录primescript.RT试剂盒(Takara,大连,中国),具有额外的茎环底漆[46.] TCMIR156(附加文件2:表S2)。如上面提到的定量RT-PCR和分析方法是相同的。
MRE的双荧光素酶报告基因(DLR)测定
所述双报告是指在单个系统中同时表达和两个单独的报告酶的测定。在DLR测定中,萤火虫的活动(Photinus pyralis)及Renilla (Renilla eniformis.)萤光素酶被顺序地从一个单一的样品进行测定。与TcmiR156插入P2GW7载体用作效应器20-NT的瞬时过表达成熟miR156。35SGLO载体通过在萤火虫萤光素酶的3'UTR插入MRE设置作为报告。效应和记者共转化成后杨树原生质体,从萤火虫萤光素酶和海肾荧光素生成的荧光强度被自动在Glomax-96微孔板发光与每个DLR测定的四个重复检测。制备荧光素底采用双荧光素酶报告基因检测试剂盒(Promega,WI,USA)。
数据和材料的可用性
所有TcSPLCDNA序列和引物可在附加文件中获得。
缩写
- CD:
-
编码序列
- DGE:
-
数字基因表达谱
- 经销商:
-
Dual-luciferase记者
- 唔:
-
隐藏的马尔可夫模型
- ml:
-
最大似然方法
- mMRE:
-
突变MRE序列
- 绝笔:
-
miRNA响应元素
- NLS:
-
核定位信号
- PI:
-
等电点
- 种族:
-
cDNA末端的快速扩增
- ROS:
-
反应性氧气
- SBP:
-
Squamosa启动子结合
- SOS:
-
盐过度敏感
- SPL:
-
SBP蛋白样蛋白质
- SPL-MRE:
-
mRNA在SPL中裂解
- TCSPLI-MRE:
-
SPL内翻译抑制MRE
- UTR:
-
未经翻译的地区
参考文献
- 1。
金杰,田F,杨德,孟y,孔L,罗杰,等。Planttfdb 4.0:朝向植物中的转录因子和监管相互作用的中心集线器。核酸RES。2017; 45:D1040-5。
- 2。
Riechmann JL,听到J,Martin G,Reuber L,Jiang C,Keddie J等人。拟南芥转录因子:真核生物中的基因组对比分析。科学。2000; 290:2105-10。
- 3.
李爽,谢震,胡超,张军。植物生长素响应因子研究进展。植物学报。2016;7:47。
- 4.
王平,程T,露玛,刘G,李毅男,石J,等。在种子植物AP2基因组的膨胀和功能分化通过分子进化和突变拟南芥分析确定。前植物SCI。2016; 7:1383。
- 5。
NAC转录因子在植物非生物胁迫反应中的作用。生物物理学报。2012;19:97 - 103。
- 6。
Moreno Ma,Harper LC,Krueger Rw,Dellaporta SL,Freeling M.Ligulless1编码含有核局部化蛋白质所需的核局部化蛋白,玉米叶片器官发生在玉米叶片器官期间。基因开发。1997; 11:616。
- 7。
陈晓霞,张志强,刘东,张凯,李安,毛磊。Squamosa启动子结合蛋白样转录因子在植物生长发育中的作用。acta botanica yunnanica(云南植物研究),2010;52:946-51。
- 8.
郭安生,朱强,顾晓霞,葛树华,杨静,罗建军。植物特异性sbp-box转录因子家族的全基因组鉴定及进化分析。基因。2008;418:1-8。
- 9.
STIEV A,Altmann S,Hoffmann K,Pant BD,Scheible WR,Baurle I. Arabidopsis MiR156通过SPL转录因子调节对重复环境压力的耐受性。植物细胞。2014; 26:1792-807。
- 10。
Hou H,Jia H,YAN Q,王X.来自拟南芥中野生血管病毒的SBP-BOX基因(VPSBP16)的过度表达提高了盐度和干旱胁迫耐受性。int j mol sci。2018; 19:940。
- 11.
Cui L,Shan J,Shi M,Gao J,Lin H.MIR156-SPL9-DFR途径坐标坐标植物中发育和非生物胁迫耐受性之间的关系。工厂J. 2014; 80:1108-17。
- 12.
Li C,Lu S. Populus Trichocarpa中SPL基因家族的分子表征。BMC植物BIOL。2014; 14:131。
- 13。
王P,陈德,郑y,金S,杨j,ye n。sbp-box基因的鉴定及表达分析揭示了茶叶(Camellia sinensis)中非生物胁迫和激素应激的参与。int j mol sci。2018; 19:3404。
- 14。
毛华,于林,李志,严艳,韩锐,刘洪,等。玉米spl家族转录因子的全基因组分析及其对非生物胁迫的响应。植物基因。2016;6:1-12。
- 15.
宁凯,陈胜,黄洪,姜军,袁华,李红梅。转bpspl9转基因白桦spl基因家族的分子特征及表达分析。植物细胞组织器官邪教。2017;130:469-81。
- 16。
Rhoades MW, Reinhart BJ, Lim LP, Burge CB, Bartel B, Bartel DP。植物小rna靶标的预测。细胞。2002;110:513-20。
- 17。
yu y,jia t,陈x.'如何'和'在植物microRnas的何处。新植物。2017; 216:1002-17。
- 18.
Arshad M, Feyissa BA, Amyot L, Aung B, Hannoufa A. Microrna156通过沉默提高紫花苜蓿(medicago sativa)的抗旱性SPL13.植物SCI。2017; 258:122-36。
- 19.
AUNG B,Gruber My,Hannoufa A. MicroRNA156系统:植物生物技术的工具。生物发作的生物科技。2015; 4:432-42。
- 20。
王H,王H. MIR156 / SPL模块,一个监管中心和多功能工具箱,齿轮造成农作物,以增强农艺性状。莫尔植物。2015; 8:677-88。
- 21。
王胜,李胜,刘强,吴科,张军,王胜,等。osspl16-gw7调控模块决定了籽粒形状,同时提高了水稻产量和品质。Nat麝猫。2015;47:949。
- 22。
三浦K,池田M,松原A,宋晓杰,伊东M,浅野K,等。Osspl14促进水稻穗分枝和高产。Nat麝猫。2010;42:545-9。
- 23。
郭建军,孙立军,唐勇,姜强,等。从型号到裁剪:SPL8 in的功能特性m . truncatula导致苜蓿生物量产量和非生物胁迫耐受性的遗传改进。植物Biotechnol J. 2018; 16:951-62。
- 24。
张立,李克,东·吴,王米,迪D,克朗萨克HJ等。硝酸盐势态的特征及比较Tamarix Ramosissima.和模拟干旱条件下的棉花根系。树杂志。2019;39:628-40。
- 25。
丁芳,杨军,袁芳,王斌。再生盐细胞排盐机制的研究进展。医学杂志。2010;5:164 - 70。
- 26。
ji x,nie x,liu y,zheng l,zhao h,zhang b等。一种Bhlh.基因刚毛柽柳通过提高渗透势和降低反应性氧物种积累来提高非生物胁迫耐受性。树生理。2016; 36:193-207。
- 27。
郑湖,刘克,萌x,刘y,ji x,li y等。一种怀疑基因刚毛柽柳,ThWrky4通过调节反应性氧物种和应激响应基因的表达来介导非生物应激反应。植物mol biol。2013; 82:303-20。
- 28。
王丽丽,李志强,陆敏,王玉英。柽柳nac转录因子Thnac13对转基因拟南芥耐盐和渗透胁迫能力的影响。植物学报,2017;
- 29。
季欣,王颖,刘刚。柽柳myc基因在不同非生物胁迫下的表达分析。中华医学会分子生物学分会。
- 30.
王丽丽,秦丽丽,刘伟,张德华,王玉英。拟南芥乙烯响应因子therf1的克隆及表达分析。杂志。2014;152:84 - 97。
- 31。
臧东,王超,吉鑫,王玉英。柽柳锌指蛋白thzfp1通过提高脯氨酸含量、sod和pod活性参与盐胁迫和渗透胁迫。植物科学。2015;235:111-21。
- 32。
王j,徐m,顾y,徐L.略带表达的Tamarix Chinensis的基因分析为NaCl-应激反应提供了见解。树木。2017; 31:645-58。
- 33。
Gandikota M, Birkenbihl RP, Höhmann S, Cardon GH, Saedler H, Huijser P. miRNA156/157识别元件在3 ' UTR拟南芥SBP盒基因SPL3通过幼苗中的平移抑制来预防早期开花。工厂J. 2007; 49:683-93。
- 34。
在SBP-box基因中Physcomitrella patens.;2006年。
- 35。
Silva GFFE, Silva EM, Azevedo MDS, Guivin MAC, Ramiro DA, Figueiredo CR等。针对microrna156的spl/sbp box转录因子调控番茄子房和果实发育。植物j . 2014; 78:604-18。
- 36。
杨Z,王X,顾S,Hu Z,徐H,徐C.拟南芥SBP-Box基因家族的比较研究。基因。2008; 407:1-11。
- 37。
陈光,李军,刘艳,张强,高艳,方锴,等。ga介导的spl基因家族和mir156在板栗花发育中的作用国际分子生物学杂志。2019;20:1577。
- 38。
张伟,韩军,郑涛,等。葡萄孤雌结实过程中Vvmir160s/vvarfs的相互作用及其时空表达/切割产物植物学报。2019;19(1):111。
- 39。
Cardon GH,Höhmanns,Nettesheim K,Saedler H,Huijser P.拟南芥SBP-Box基因SPL3的功能分析:涉及花卉过渡的新型基因。工厂J.2010; 12:367-77。
- 40。
王JW,捷克B,威格尔D. Mir156监管的SPL转录因子定义拟南芥的内源性开花途径。细胞。2009; 138:738-49。
- 41。
王辉,张伟,王敏,程强。杨树PtVIP1基因的克隆与鉴定;2018.https://doi.org/10.1007/S11676-018-0745-Z..
- 42。
王H,王米,程Q.使用3'-血和高通量测序的组合捕获杨树中14个NAC基因的替代裂解和多苯基化位点。分子。2018; 23:608。
- 43。
程T,Shi J,Dong Y,Ma Y,Peng Y,Hu X等。硫化氢通过增加S-亚硝基葡萄醇还原酶(GSNOR)活性并降低反应性氧/氮气损伤,增强了高温应激的杨树耐受性。植物生长调节。2017; 84:1-13。
- 44。
Kumar S,SteCher G,Tamura K. Mega7:用于更大数据集的分子进化遗传学分析版本7.0。mol Biol Evol。2016; 33:1870。
- 45。
Jeanmougin F,Thompson JD,Gouy M,Higgins DG,Gibson TJ。与群集X的多个序列对齐。趋势Biochem SCI。1998年; 23:403-5。
- 46。
通过茎环RT-PCR的microRNA陈C.实时定量。核酸RES。2005; 33:E179。
致谢
不适用。
资金
本研究由江苏省科技支撑计划项目(no . BE2011321)和江苏省高校学术发展重点项目(no . PAPD)资助。
作者信息
隶属关系
贡献
LX和MX构思并设计了这个项目。YY和JW负责分子生物学实验。JW和YY参与了数据分析。JW和LF起草了手稿。LX和JW对手稿进行了修改。所有作者都已阅读并批准该手稿出版。
通讯作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版
不适用。
利益争夺
提交人声明他们没有竞争利益。
附加信息
出版商的注意事项
施普林格《自然》杂志对已出版的地图和机构附属机构的管辖权要求保持中立。
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
王建军,叶彦,徐敏。et al。的角色SPL.柽柳盐胁迫反应中的基因家族与miR156 (Tamarix chinensis.).BMC植物BIOL.19,370(2019)。https://doi.org/10.1186/s12870-019-1977-6
已收到:
接受:
发表:
关键字
- SPL.基因家族
- miR156
- 盐胁迫
- Tamarix chinensis.