摘要
背景
通过育种,突变和转基因方法进行大豆进行遗传操作,以产生增值的品质性状。在那些遗传方法中,通过快速中子辐射诱变是有趣的,因为它产生多种突变,包括单/多基因缺失和/或重复。表征快速中子突变体的种子组成及其与基因突变的关系可用于了解大豆中的油和蛋白质性状。
结果
从大量的快速中子诱变植物中,我们选择了十个突变体,基于近红外线光谱法筛选总油和蛋白质含量的筛选。将这些十个突变体重新生长,通过GC-MS,通过SDS-PAGE和基因映射通过比较基因组杂交来分析种子。与野生型相比,突变体2R29C14C11233CMN15(本研究中的绰号为L10)显示出更高的蛋白质和较低的油含量,其次是三种其他线(本研究中的绰号为L03,L05和L06)。我们以反式酯化油的脂肪酸甲酯谱进行了特征,并在突变体中的不同比例下发现了五种主要脂肪酸(棕榈酸,硬脂酸,油炸,亚油酸和亚麻酸)的存在。使用10个突变体的SDS-PAGE的蛋白质谱确实在储存(甘氨酸和β-甘氨酸)和抗营养因子(胰蛋白酶抑制剂)蛋白之间具有可辨别的变化。此外,我们使用比较基因组杂交物理地映射了基因缺失或重复性的重复性。
结论
大豆快中子突变体中油脂和蛋白质的特性将有助于科学家和育种家开发具有改进蛋白质和油脂品质性状的新型增值大豆。
背景
大豆的遗传操作(大豆(L。)稳定)是自2010年参考基因组序列开始以来重新引起人们兴趣的一个领域[1].Because of the higher protein (~ 40%) and oil (~ 20%) content of soybean, it has been targeted by the food industry to produce a variety of nutritionally enhanced food products [2那3.那4.].有几种方法被用来操纵大豆的基因组组成,以产生理想的质量性状。这包括但不限于转座子标签[5.]化学处理[6.,辐射诱变[7.],遗传转化和基因编辑[4.那8.].辐射诱变,特别是快中子(FN)辐射,通过缺失、复制、易位和逆转录引起广泛的变异,可诱发强烈的突变表型[7.那9.].随着全基因组测序能力的提高,这一领域的兴趣进一步增强[7.那9.].辐射诱变删除相邻基因簇,包括串联重复序列,不需要插入任何外源基因。Bolon出版社。[7.使用FN轰炸制作了超过27,000个独特的大豆突变体(https://www.soybase.org/mutants/about.php).使用阵列的比较基因组杂交(CGHs)与下一代测序沿(NGS)技术,研究者亮相FN的一个子集的全基因组结构变化突变大豆[9.].此外,也有报道称,在一个与大豆三色发育迟缓相关的NAP1基因模型上,不同的fn诱导的序列重排[10.].此外,FN诱导的倒数染色体易位发现背后一个突变表型表现出种子高蔗糖,低油[11.].Stacey等人[12.]使用Fn诱变来阐明功能的功能网络GmHGO1与同质化异化分解代谢相关的基因导致大豆中棕色种子表型。
目前,有删除/复制配置文件数百FN突变系,这是信息的基因组分析[社区资源9.].然而,为了充分利用Fn突变体,与质量属性有关的突变体的综合生化分析至关重要。在这项研究中,我们筛选了大量的FN突变体,以鉴定具有较大的油和蛋白质分析的副本。10突变体的一个子集进行了详细的种子组成分析中,包括酯交换油和蛋白质分布的脂肪酸酸甲酯(FAME)的组合物。此外,我们物理地映射了由诱变引起的基因缺失/重复措施。该信息将帮助育种者/生物技术学家纳入所需的高油和蛋白质特征,并开发新的增值大豆。
结果
种子组成性状改变的突变系鉴定
遗传背景M92-220大豆FN群体的突变体[11.]在田间(明尼苏达大学,圣保罗,MN)生长数年,允许连续轮自花授粉和稳定表型。在突变群体中观察到广泛的表型[7.幸存的突变体每一代都能产生健康的种子。通过近红外光谱(NIR)分析,初步筛选2015年大田收获的数千个FN品系(明尼苏达大学,圣保罗,MN)的种子,估计种子成分的相对比例(表)1;额外的文件3.:图S1)。本研究选取了10个种子蛋白质和/或油脂水平异常的品系进行进一步分析。田间收获的10个品系种子均未表现出明显的种子形态表型和单株种子质量差异。本研究中使用的这些行的全名见表1.然而,为了简单起见,为了本手稿的目的,这些突变体被昵称为L01-L10。
对于十种选定的突变线,总种子蛋白质含量在30.5至58.0%之间,总种子油含量在10.9〜25.7%之间变化。10个突变豆种子中的总种子蛋白和油(P + O)含量在55.9至69.4%之间变化。野生型大豆种子中的总蛋白质,油和P + O含量分别为41.8,20.5和62.3%。与野生型相比,六条线(L03,L05,L06,L08,L09和L10)显示出更高的蛋白质含量(表1)然而,与野生型相比,四个突变系L01(30.5%)、L02(33.1%)、L04(41.0%)和L07(40.7%)的蛋白质含量较低。在这些突变系中,L10的蛋白质含量最高(58.04%),L01的含油量较高(25.7%),L05的蛋白质含量最高(P + O(69.4%)含量。
FN突变体脂肪酸甲酯(FAMEs)分析
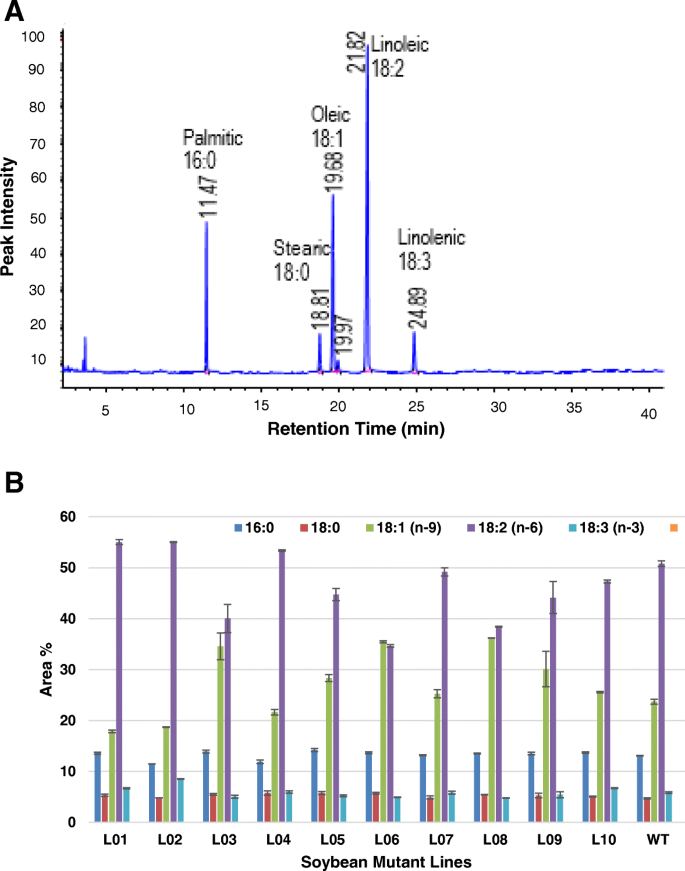
2016年,10个蛋白质和含油量不同的亲本突变体在田间(明尼苏达大学,圣保罗,MN)重新生长,以收集更多的种子进行分析(附加文件)3.:图S1)。采用气相色谱-火焰离子化检测器(GC-FID)测定种子中脂肪酸甲酯的含量。GC-FID对酯交换后的油进行分析,发现存在五种主要的脂肪酸甲酯(棕榈酸、硬脂酸、油酸、亚油酸和亚麻酸)。图中显示了典型的酯交换大豆油样品的GC-FID谱图。1一种。如从图可见一斑。1a,从10个突变体提取的酯交换油中检测到5个主要的FAMEs,占总FAMEs的95%以上。这些被鉴定为棕榈酸甲酯(16:0),硬脂酸甲酯(18:0),油酸甲酯(18:1),亚油酸甲酯(18:2)和亚麻酸甲酯(18:3)。10个突变体的饱和脂肪酸(棕榈酸和硬脂酸)平均含量在16.3 ~ 20.0%之间,不饱和脂肪酸(油酸、亚油酸和亚麻酸)平均含量在75.0 ~ 82.3%之间。在野生型大豆亲本M92-220中,饱和脂肪酸与不饱和脂肪酸的比例相似(17.8比80.3%)。1b). 10个突变株系间的FAMEs谱存在显著差异。这些变化主要表现在油酸和亚油酸的含量上。油酸的变化范围为17.9 ~ 36.2%,亚油酸的变化范围为34.6 ~ 55.0%。我们观察到油酸和亚油酸含量之间存在负相关(R2 =−0.96)。
差异表达蛋白图谱
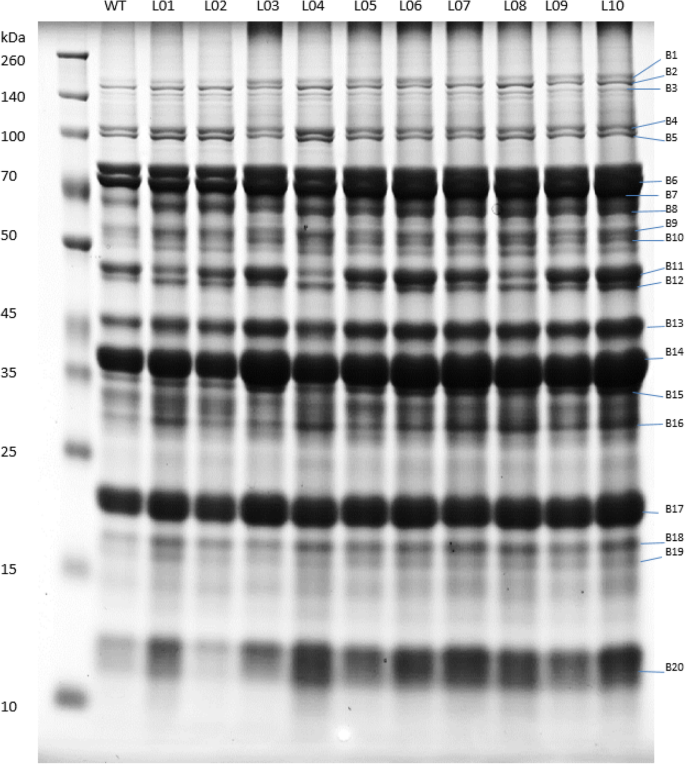
2016年大田收获的种子也用于蛋白质表达分析(附加文件3.:图S1)。通过SDS-聚丙烯酰胺凝胶电泳分析十个突变体的总种子蛋白质图谱(图。2).在贮藏蛋白和抗营养蛋白中观察到10个突变体的蛋白谱变化。大约有20条蛋白带被显著观察到,其中一些似乎是根据相对丰度差异表达。将差异表达的蛋白条带人工切除,用胰蛋白酶消化。用MALDI/TOF/TOF- ms对所得多肽进行鉴定,结果见表2和额外的文件1:表S1。The mutants L03, L06, and L10 showed higher abundance of several bands such as, B6 (β-conglycinin, alpha’ chain precursor), B13 (β-conglycinin, β chain), B14 (glycinin G2 precursor) and B17 (trypsin inhibitor subtype A precursor) as compared to the wild type soybean M92–220. Band B11, identified as β-conglycinin β chain precursor, exhibited higher abundances in mutants L03, L05, L06, L09 and L10. Similarly, B13, identified as β-conglycinin (β chain) showed higher abundances in all lines except L01, L02 and L04. The list of the peptides used to identify the proteins are shown in Additional file1:表S1与除了独特肽谱计数蛋白质登录号。基于所述分子量和贮藏蛋白(β伴大豆球蛋白和大豆球蛋白)和胰蛋白酶抑制剂的条带模式,我们细分他们分为不同的组。在10突变体的贮藏蛋白可以被细分为三组(第1组 - L01,L04,L08;第2组 - L02,L03,L05,L07,L09;以及第3组 - L06和L10)。大豆球蛋白A1b2B2-784前体(B20)在L02被显著低于其他突变株系。基于所述的胰蛋白酶抑制剂的带型(B16和B17),十突变体分为3组,除L03突变体(组1- L07,L08,L10;组2- L02,L05;组3- L01,L04,L06,L09)。带19(胰蛋白酶抑制剂)显示出低蛋白强度,因此不能进行分组。
基因缺失/新生力量突变的复制
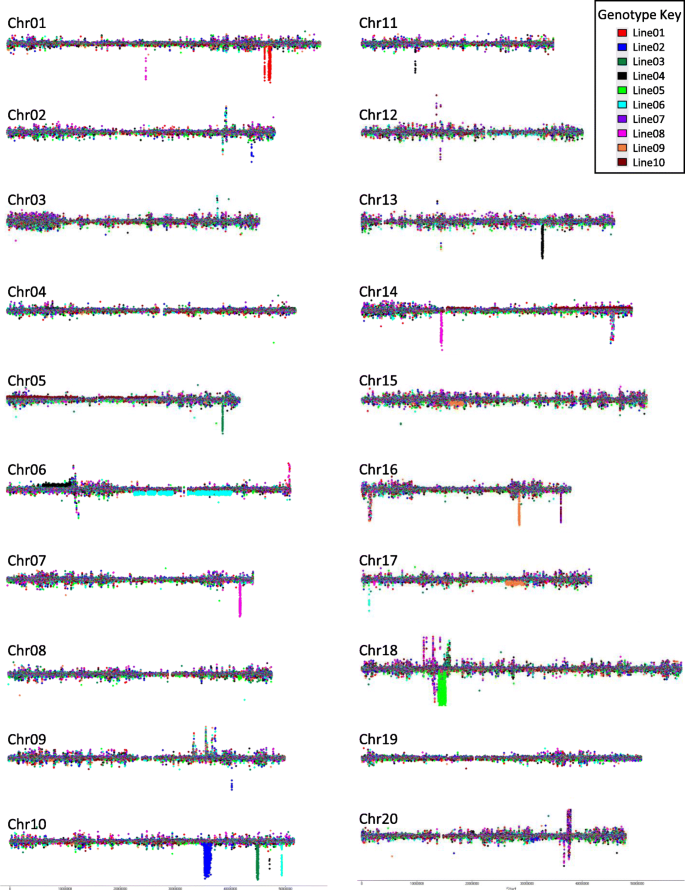
我们利用CGH分析来识别和估算与野生型父母直系M92-220相比,在10个突变体中识别和估计基因缺失和重复的位置。2017年在温室中生长的单一植物用于代表每个突变线(附加文件3.:图S1)。在图20染色体上的十个突变体的CGH分布如图2所示。3.,原始数据可通过GEO加入号GSE118594获得。在这些分析中,水平线表示对数2FN系和野生型系之间每个染色体的零(无差异)比率。垂直方向上的任何信号表示突变体之间的变异(可能重复或缺失)。如图所示。3.,在10个突变系中观察到基因重复/缺失的显著变化。FN突变以及缺失和重复的位置如表所示3.。表中还显示了参与删除和复制的基因数量3.,基因模型名称见附加文件2:表S2。在一些线中,由于FN辐射,删除了几百个基因(纯合或杂合)/复制。在一些线路中观察到一些大的杂合缺失。例如,L06显示出位于杂合缺失(246基因)内的最多基因,其次是L09(103基因)。然而,CGH信号的幅度表明这些是杂合缺失,几乎肯定会使生物体在纯合的缺失偏差中不可行。另一方面,在L10中观察到大量的重复(包括总共1743个基因)。在基因组中其他地方的邻近区内的区域中发生的大缺失可能具有增加的纯合缺失状态的存活概率。然而,这种情况预计在大豆中将罕见,因为最近复制的事件发生了数百万年前,大多数古老的职业地区不再高度保守。另一方面,大量的重复可能没有严重的表型后果,并且实际上在这项研究中观察到这一点。对于L10尤其如此,其中重复包括总共1743个基因。 The details of the genes that were deleted or duplicated due to the FN radiation are provided in the Additional file2:表S2。
为了确认基因缺失,我们进行了PCR分析。我们从缺失和复制的每个区域中选择至少一个基因用于纯合植物。但在L07、L08、L09等突变体中,由于引物的非特异性或扩增效果差/失败,无法进行分析。基于特异性,对8个基因进行了PCR分析,凝胶图像见附加文件4.:图S2。如图所示,除了将所选基因重复的L10之外,所有突变体都没有表现出基因产物或扩增。
讨论
通过FN诱变改善种子组成的前景
我们进行了几次分析,以表征大豆快上中子突变体,包括CGH以定位基因缺失/重复,NIR估计蛋白质和油水平,SDS页面用于种子蛋白质分析,以及针对着名组合的GC-FID。在该稿件中提出的结果表明,使用快的中子辐射可以提高5%至15%(干重基)在大豆种子中蛋白质含量增加。在测试(L01-L10)的十个突变体中,与野生型亲本M92-220(41.8%)相比,L10显示出58%的总种子蛋白质含量。大豆在其他豆类中具有最高的蛋白质含量,平均约40%至42%(干重)。然而,随着较高的蛋白质含量增加大豆的价值,优选大豆种子中的蛋白质含量的增加。由中心咨询集团LLC进行的一项研究得出结论认为,当产量和油水平保持不变时,蛋白质含量增加1%增加了每英亩的作物价值(https://www.agprofessional.com).根据美国大豆委员会(USB) (https://unitedsoybean.org.),家禽和牲畜农民更喜欢大豆含量高蛋白质含量。
提高大豆种子蛋白质含量的方法有好几种。在这项努力中,通过改进育种实践的遗传获得策略正在被采用,与蛋白质相关的基因的详细分子图谱已经被记录下来[13.].据报道,染色体20(连锁群I (LG-一世))和15(LG-E.)包含大豆蛋白质变异的主要数量性状位点[13.].然而,通过改良育种获得基因的策略涉及到几个挑战。例如,驯化的大豆是古多倍体,大约75%的基因有多个拷贝[1].因此,缺失或添加靶基因并不总是提供预期的结果。此外,还采用转基因方法来增加大豆蛋白质含量[14.那15.].在这些研究中,引入了一个外源基因来提高大豆种子中的蛋白质水平。然而,这种方法需要昂贵和漫长的监管批准,才能在市场上释放这些特性。基于FN的突变不涉及外源基因的引入,因此不需要通过调控过程的批准。
虽然FN诱变显然能够大幅增加种子蛋白总量,但在商业化之前,需要对这些品系的其他性状进行广泛评估。这包括农艺性状,特别是产量。在一条线内观察到表现出根本改变的种子组成性状的有害性状是不足为奇的。此外,可能需要识别种子蛋白增加背后的特定缺失和/或重复,因为这些可以回交成精英品种。如果FN种子组成位点也造成有害性状,它本身可能对育种没有用处。然而,它可能仍然有助于识别控制这些性状的基因,从而导致有针对性的育种策略,利用这些基因的自然或其他形式的诱导变异。
种子蛋白,油和脂肪酸组成之间的关系
我们观察到负相关(r2=−0.8302)1).这一结果表明,fn改变的基因组变化,导致更多的蛋白质含量通常也导致低总含油量,反之亦然。其他大规模调查亦有类似结果[16.那17.],人们普遍认为大豆籽粒蛋白质和油脂在自然变异中呈负相关,这可能与碳分布有关。尽管这种负相关的确切原因还不清楚,Wilson [18.]提出了一种通过估计组分值来克服蛋白质和油含量之间障碍的模型。如前所述,根据美国农业部收集的大豆种质的平均蛋白质(42%)和油(19%)含量,可以为蛋白质含量为44%至45%,油含量不低于18%的品种设定一个实用的目标。在我们的研究中,FN诱导在蛋白质(40-58%)和油(10-25%)方面产生了广泛的变化。目前的研究表明,一些FN品系具有改善大豆品质性状的潜力[18.].但是,在考虑这些数据时,必须考虑一些其他因素。例如,本研究中的突变体未进行可收获的产量或其他农艺性能特征。如上所述,可以改变种子组合物性状或这些植物中的其他突变可能影响其他对种植者重要的特征。
与油含量一样,还研究了从十种突变大豆中提取的反式酯化油的脂肪酸含量的变化。虽然L10表现出低总油含量,但所有十个突变体和野生型大豆的脂肪酸组成显示出五种突出的脂肪酸,即棕榈,硬脂,油酸,亚油酸和亚麻酸的存在。类似的脂肪酸组合物(13%的棕榈酸(16:0),4%硬脂酸(18:0),20%油酸(18:1),55%亚油酸(18:2)和8%的亚麻酸(18:3))之前已在文献中报告[19.那20.].其中,棕榈酸和硬脂酸是饱和脂肪酸,并且其余的是不饱和脂肪酸。
所述不饱和脂肪酸谱显示出10突变体样品之间显著变化。范围为油酸17.9之间变化以36.2%,与L08表现出最高量和L01示出最低量。据报道,由Bellaloui等文献。[21.油酸组成的变化可能是由于农业实践,包括种植日期、播种率和生长条件。作者认为,在种子灌浆期,温度影响了参与脂肪酸生物合成的酶。在FN突变体中,亚油酸含量的变化范围为34.6 ~ 55.0%,其中L02含量最高,L06含量最低。突变株L06的油酸含量增加(34.6%),亚麻酸含量降低(4.9%)。该突变株可能会对有兴趣开发新的高附加值大豆的育种家产生重大的兴趣,这些大豆的营养价值有显著的提高。减少油中多不饱和脂肪酸(18:3)的浓度是可取的,因为它会由于氧化而减少保质期,而氧化会产生难闻的气味[22.].
蛋白质和基因缺失/复制谱的变异
在本研究中观察到10个FN突变体的种子蛋白质谱的清晰变化,特别是对于储存蛋白和胰蛋白酶抑制剂(图。2).有趣的是,如B6 (β-聚甘氨酸,α '链前体),B11 (β-聚甘氨酸,β链前体),B14(甘氨酸G2前体)和B17(胰蛋白酶抑制剂亚型A前体)等较高丰度的条带与前面讨论的高蛋白FN突变体相对应。因此,我们推测β-聚甘氨酸可能是种子中蛋白质含量较高的原因。Krishnan和Nelson(2011)对9种大豆品种的蛋白质含量进行了研究,得出总的较高的蛋白质含量主要由球蛋白贡献[23.包括β-甘氨酸和甘氨酸。然而,在我们的研究中尚不清楚FN照射是如何在某些大豆品系中诱导更高的蛋白质含量的。我们还检查了缺失/重复区域,以评估该区域相应的基因。一些突变体没有显示储存蛋白基因的复制。而L10则表现出双功能抑制剂/脂转移蛋白/种子贮藏2S白蛋白超家族蛋白基因的重复。2S白蛋白根据沉降系数定义,是一组含有含硫氨基酸的贮藏蛋白[24.那25.].2S白蛋白还包括酶蛋白如蛋白酶抑制剂包括Bowman Birk和Kunitz胰蛋白酶抑制剂[26.].因此,我们预期缺失/重复可能具有其他掺杂效应,其有助于一些FN突变体中的蛋白质含量较高。
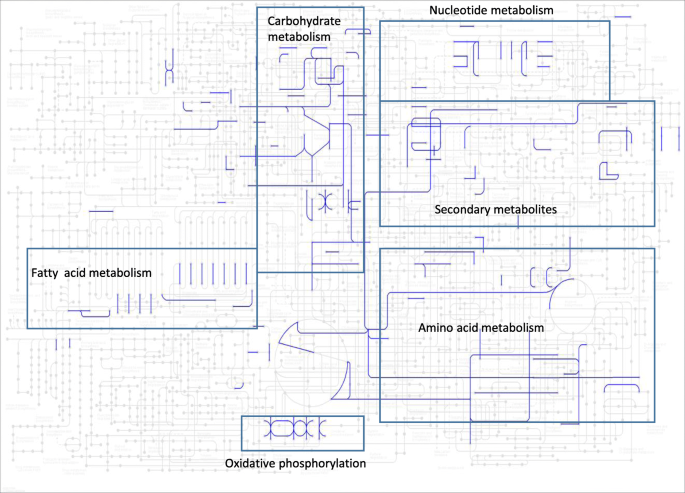
为了了解导致种子蛋白和油改变的潜在机制,我们在全球代谢途径上映射了L10的重复基因。如从图可见一斑。4.,糖酵解/糖异生、脂肪酸降解、嘌呤代谢、氨基酸生物合成、核糖体、内质网蛋白质加工、氧化磷酸化等多种途径得到富集。L10蛋白含量较高可能是由于核糖体、内质网蛋白质加工的富集途径。另一方面,L10的低含油量可能与从脂肪酸到蛋白质合成过程的碳分布有关。虽然对重复基因的全局途径分析提供了途径的变化,但不知道蛋白质增加或油减少的作用是一种还是多种因素的组合为了了解基因效应,正在进行与目标基因的分离分析。
总的来说,CGH分析显示突变系中存在广泛的缺失和重复。我们用PCR方法确认了被删除或重复的基因的CGH结果。结果与CGH分析一致(附加文件)4.:图S2)。确定导致种子成分变化的原因缺失/重复需要进一步的实验,特别是回交或异交群体的共分离分析[11.].此外,使用Fn和CGH存在重要的局限性,以识别这些特征的原因改变或基因。如果致病缺失/重复性大,则它们可能包含许多基因并使致病基因难以识别。此外,CGH通常不会解析小删除/重复,也不会检测到反转/易位,除非它们改变DNA段拷贝数。此外,CGH并不完全解决大型删除/重复的精确断点。因此,表中提供的数据3.和额外的文件2:表S2代表主要基于大缺失和重复基因/基因组的FN破坏。最后,考虑到这些材料的后期产生,推测大多数缺失和重复性在谱系内固定为纯合。然而,CGH表明一些缺失是杂合的,因此可能仍然在群体内进行分离。然而,CGH不容易区分纯合杂合象,因此目前尚不清楚这些事件中的哪一个仍然可以在这些群体中进行分离。
结论
本研究利用FN辐射诱变技术对大豆基因型M92-220的基因组组成进行调控。通过该方法获得的突变体在M5-M8代表型稳定。选取10个FN突变体进行详细分析。综合分析了种子的油脂和蛋白质含量等组成属性。突变体的蛋白质和含油量存在很大的变异。此外,通过全基因组CGH分析估算了FN突变过程中缺失/重复基因的位置和数量。这些信息和突变体对于科学家和育种家改变种子组成性状以生产增值大豆是有用的。
方法
突变体材料及初始种子成分筛选
在加利福尼亚州大学麦克莱伦核辐射中心完成了FN突变群和FN辐射剂量,并在以前的出版物中描述了[7.那9.].所有突变体都是在大豆品系‘M92-220’中发育的,该品系来源于品种‘MN1302’的种子库[27.].从2015年从2015年进行大量筛选的田间收获的种子,用NIR光谱进行种子组成分析[7.].鉴定出一组蛋白质和油脂水平变化较大的突变体。这10个突变体于2016年在大田条件下(圣保罗,MN)生长,并对收获的种子进行成分分析(见下文方法)。表中列出了本研究中使用的FN突变体1.FN突变体的产生范围从M5到M9。
脂肪酸甲酯分析
从磨碎的大豆籽粉(100 mg)中提取油,用己烷(5 mL)在超声波浴(功率600瓦)中提取两次,时间为15分钟。提取液在5000 rpm下离心10分钟,上清液收集在一个单独的小瓶中。用5ml新鲜己烷萃取残渣。池上清液在缓慢流动的氮气下蒸发干燥。将浓缩的大豆油用2ml己烷重新悬浮。将其中一部分mL蒸发至干燥,并使用5 mL酸化甲醇(10 mL乙酰氯至90 mL冷甲醇)进行酯交换至FAMEs。将混合物在室温下惰性氮气气氛下搅拌过夜。在上述混合物中加入3 mL水,用2 mL己烷提取脂肪酸甲酯。己烷层分离,气相色谱分析[28.].对脂肪酸甲酯的滞留时间与真实的FAMEs标准进行了比较。所有分析都是三次重复进行,并计算标准误差。
蛋白质提取
使用苯酚提取方案[提取蛋白质29.].简单地说,从每个突变系中提取200毫克的地面种子,首先用己烷脱脂[30.].Approximately 1 mL of the extraction buffer containing sucrose (0.7 M), tris(0.5 M), EDTA (50 mM), KC l(0.1 M), DTT (25 mM) and PMSF (2 mM) was added and the mixture was incubated for 30 min at ambient room temperature with shaking. The supernatant was collected after centrifugation (8000 g) for 30 min. An equal amount of water saturated phenol was added to the supernatant and the sample was mixed well for 10 min and centrifuged (30 min at 4 °C). The proteins from the phenol phase was precipitated by 0.1 M ammonium acetate in methanol and incubated at -20 °C overnight. Protein pellets were collected after centrifugation at 15000 g for 30 min followed by washing with cold acetone for three times. The protein pellets were re-suspended in 6 M urea, 100 mM Tris-HCl and the concentration was estimated by bicinchoninic acid assay (Pierce, Rockford, IL). All analyses and extractions were performed in three replicates.
通过SDS页面分离蛋白质
对分离蛋白质进行聚丙烯酰胺凝胶电泳。简而言之,每孔10μg蛋白质加载15%(w / v)聚丙烯酰胺 - 凝胶,使用Tris /甘氨酸/十二烷基硫酸钠缓冲液(Mini-Protean Tetra System,Bio-rad)以100V达到45分钟的分离。,赫拉克勒斯,加利福尼亚州)。使用Fermentas Spectra多色低范围蛋白梯(Thermo Fisher Scientific,Waltham,MA)估计蛋白质的分子量。使用生物安全Coomassie G-250(Bio-rad)染色凝胶。
蛋白质的消化和鉴定
蛋白质条带从考马斯染色的切下凝胶和消化用猪胰蛋白酶(Promega)。将得到的肽与AB SCIEX TOF / TOF 5800 MALDI-MS,使用蒸馏器的Mascot版本系统进行分析。2.3.0.0(www.matrixscience.com.).使用Mascot搜索引擎进行蛋白质鉴定(http://www.matrixscience.com),它使用基于概率的评分系统。使用NCBInr数据库进行肽查询。数据库搜索的参数与MS / MS谱如下:片段公差:0.60 Da(单一同位素的),父母宽容:50 PPM(单一同位素的),固定的修改:+ 57 C (Carbamidomethyl),变量修改:−18 n (Glu - > pyro-Glu),−17 n (Gln - > pyro-Glu), + 16 M(氧化)、马克斯错过分裂:1。
CGH分析
2017年,温室里种植了大豆植株3.:图S1)。杨叶组织收获并使用Qiagen的DNeasy方法用于下游分析CGH DNA提取。CGH如先前所述进行[11.].CGH微阵列(Agilent Technologies, Santa Clara, CA, USA)包括超过94万个探针,可在国家生物技术信息基因表达综合中心(GEO)获得登录号为GPL22907的登录号(GPL22907)。http://www.ncbi.nlm.nih.gov/geo.).该微阵列平台基本上铺贴了大豆基因组,在遗传区域内具有更大的探针密度。CGH杂交条件,扫描和数据分析所有遵循先前描述的方法[11.].通过jbrowse分析了这些删除或重复的段(https://phytozome.jgi.doe.gov/jbrowse/index.html)大豆Wm82 a2v1检索相应基因。
PCR分析
基因从纯合子缺失区选择,除了L10,其中一个基因从复制区选择作为对照。引物采用NCBI引物爆破设计https://www.ncbi.nlm.nih.gov/tools/primer-blast/反对这大豆全基因组序列。然后利用e-PCR程序检测引物序列在大豆基因组中的特异性,只使用缺失区序列特异性的引物进行缺失验证。引物序列由IDT (https://www.idt.com/).采用以下引物进行PCR分析。L01,5 ' -GCATATGCTGATTGGTGGCAA-3 '(正向)和5 ' -TTCCATGAGAAAGGGGTGCC-3 '(反向)(Glyma.01G139500);L02, 5 ' - ACCAATGCTCCTCCGCATTT-3 '(正向)和5 ' - tctgtggcagtcaacagagt -3 '(反向)(Glyma.09G180800);L03, 5 ' -ACCAATACTGACTTTTGATTCCCT-3 '(正向)和5 ' -TGAAAGGGATGGCTCGGATG-3 '(反向)(Glyma.05G208700);L04, 5 ' - TGTCCACTGTCCAGTTGTGAT-3 '(正向)和5 ' - ccttgggcttgcctgaagtt -3 '(反向)(Glyma.13G213700);L04, 5 ' -TGCATTGCACTGTCATTACCC-3 '(正向)和5 ' - gcatggcaagccgaaacta -3 '(反向)(Glyma.13G215400);L05, 5 ' - GTGGCAACAGTGTGCTTAGG-3 '(正向)和5 ' - aatccagtctgcccctctct -3 '(反向)(Glyma.18G117500);L06, 5 ' -TATGGTACCTCAGGCGGACA-3 '(正向)和5 ' -TGTTGTGTGTCAAGTAGGGTT-3 '(反向)(Glyma.10G271200);l10,5 ' -GGTGGCAGCTATACAGCACT-3 '(正向)和5 ' -ACCTTAATTCAGACACTCTCAAGGA-3 '(反向)(Glyma.05G002600)。
PCR混合物包含150 ng DNA,正反引物4 μM,每个核苷酸250 μM, 1X PCR Buffer,和Taq将总体积为15 μL的DNA聚合酶在94℃下加热3 min, PCR循环。PCR循环包括94℃变性40 s, 52℃退火30 s, 72℃延伸40 s 35个循环,72℃延伸10 min。PCR产物在2.0%琼脂糖凝胶上进行分析,溴化乙啶染色(0.5 μg/ml)并进行凝胶成像。
数据和材料的可用性
在这项研究中产生的或分析所有的数据都包括在此发表的文章和其他文件英寸
缩写
- CGHs:
-
比较基因组杂交
- 名声:
-
脂肪酸甲酯
- FN:
-
快中子
- GC-FID:
-
气相色谱-火焰电离检测器
- 谱:
-
Matrix-assisted激光解吸/电离
- 女士:
-
质谱
- 门店:
-
下一代测序
- 聚合酶链反应:
-
聚合酶链反应
- QTL:
-
数量性状位点
- SDS-PAGE:
-
十二烷基硫酸钠聚丙烯酰胺凝胶电泳
- 到F:
-
飞行时间质谱
参考
- 1。
宋庆林,陈建军,程建军,等。古多倍体大豆的基因组序列。自然。2010;463(7278):178 - 83。
- 2。
张志军。通过siRNA沉默GmFAD3基因导致大豆fad3突变表型的α -亚麻酸(18:3)降低[Glycine max (Merr.)]。转基因研究》2008;17(5):839 - 50。
- 3.
Chennareddy S,Cicak T,Clark L,Russell S,Skokut M,Beringer J,Yang X,Jia Y,Gupta M.一种新的甘蓝型油菜双向启动子在大豆中的表达。转基因决议,2017年;26(6):727–38.
- 4.
Demorest ZL,Coffman A,Baltes NJ,Stoddard TJ,Clasen BM,Luo S,Retterath A,Yabandith A,Gamo Me,Bissen J等人。直接堆叠序列特异性核酸酶诱导的突变,以产生高油酸和低亚麻酸大豆油。BMC植物BIOL。2016; 16(1):225。
- 5。
Wang S, Wang S, Eckert H, Luth D, Paz M, Donovan C, Zhang Z, et al., et al. .大豆(Glycine max Merr.)的建立。L)转座子诱变库。足底。2009;229(2):279 - 89。
- 6。
李志强,姜磊,马毅,魏志强,洪浩,刘志强,雷军,刘毅,关若,郭毅,等.一种新的高突变密度化学诱导大豆文库的开发与利用.整合植物生物学杂志.2017;59(1):60-74。
- 7。
Bolon YT, Haun WJ, Xu WW, Grant D, Stacey MG, Nelson RT, Gerhardt DJ, Jeddeloh JA, Stacey G, Muehlbauer GJ,等。大豆快中子突变种群资源的表型和基因组分析。植物杂志。2011;156(1):240 - 53年。
- 8.
Haun W, Coffman A, Clasen BM, Demorest ZL, Lowy A, Ray E, Retterath A, Stoddard T, Juillerat A, Cedrone F,等。脂肪酸去饱和酶2基因家族的定向诱变改良大豆油品质。生物技术学报;2014;12(7):934-40。
- 9.
Bolon YT, Stec AO, Michno JM, Roessler J, Bhaskar PB, Ries L, Dobbels AA, Campbell BW, Young NP, Anderson JE等。大豆快中子辐照后基因组恢复力和节段复制的流行。遗传学。2014;198(3):967 - 81。
- 10.
Campbell BW,Hofstad An,Sreekanta S,Fu F,Kono TJ,O'Orourke Ja,Vance Cp,Muehlbauer GJ,Stupar RM。在大豆NAP1基因座的快速中子诱导的结构重排导致粗糙的粒状。Al Appl Genet。2016; 129(9):1725-38。
- 11.
Dobbels AA,Michno JM,坎贝尔BW,VIRDI KS,丝太克AO,MUEHLBAUER GJ,Naeve SL,Stupar RM。在大豆中破坏的感应染色体易位一个KASI直向同源物,并且与高蔗糖,低油的种子表型相关联。G3(Bethesda)。2017; 7(4):1215至1223年。
- 12.
崔永强,张志强,张志强,等。油籽中维生素E生物强化的均质双加氧酶的鉴定。植物杂志。2016;172(3):1506 - 18。
- 13。
Patil G,Mian R,Vuong T,Pantalone V,Song Q,Chen P,Shannon GJ,Carter TC,Nguyen HT。大豆种子蛋白的分子映射和基因组学:未来的审查与观点。Al Appl Genet。2017; 130(10):1975-91。
- 14。
李磊,伍尔泰等。拟南芥的QQS孤儿基因调控大豆的碳氮分配。植物生物技术杂志。2015;13(2):177-87。
- 15.
李磊,郑伟,朱永强,叶海涛,唐斌,Arendsee ZW, Jones D,李锐,Ortiz D,赵旭,等。QQS孤儿基因通过NF-YC互作调控物种间碳氮分配。中国科学(d辑:地球科学)2015;
- 16。
Phansak P,Soonsun W,Hyten DL,Song Q,Cregan PB,Graef GL,Specht JE.鉴定大豆[大豆(L.)Merr.]种子蛋白质和油QTL的多群体选择性基因分型.G3(Bethesda).2016;6(6):1635-48。
- 17。
Vaughn JN,Nelson RL,Song Q,Cregan PB,Li Z.大豆种子成分的遗传结构通过跨多个群体的全基因组关联扫描得到细化。G3(Bethesda)。2014;4(11):2283-94。
- 18.
理查德WF。种子组成。在“大豆:改进,生产和使用”。农学美国社会,美国农作物科学学会和美国土壤学会;2004年第621-77。
- 19.
Goettel W,夏E,升起r,王ml,陈p,yq。油组合物和含量不同豆系转录多态性的鉴定与表征。BMC基因组学。2014; 15:299。
- 20.
杰·多·李KDB,香JG。遗传学和育种修饰的脂肪酸分布。Ĵ作物科学生物技术。2007; 10:201。
- 21.
Bellaloui N,Bruns Ha,Abbas HK,Mengistu A,Fisher DK,Reddy Kn。农业实践改变了仲南美国的大豆种子蛋白,油,脂肪酸,糖和矿物质。前植物SCI。2015; 6:31。
- 22.
杨树属3个部分6种杨树的无性系、品种和变种的微卫星DNA指纹、分化和亲缘关系。基因组。2002;45(6):1083 - 94。
- 23。
Krishnan HB, Nelson RL。高蛋白大豆(Glycine max)的蛋白质组学分析表明新型甘氨酸亚基的贡献。农业食品化学。2011;59(6):2432-9。
- 24。
伊斯兰教N,乌帕德亚雅NM,坎贝尔PM,Akhurst R,哈根N,希金斯TJ。在米粒组成型表达葵花籽白蛋白基因降低谷类型的积累。植物化学。2005; 66(21):2534-9。
- 25。
2S白蛋白储存蛋白:是什么使它们成为食物过敏原?Open Biochem J. 2008; 2:16-28。
- 26。
Lin J,Shewry Pr,Archer DB,Beyer K,Niggemann B,Haas H,Wilson P,Alcocer MJ。来自大豆(甘氨酸Max)的两种2S次蛋白的潜在过敏性:蛋白质微阵列方法。int arch verlergy免疫素。2006; 141(2):91-102。
- 27。
奥尔夫·杰赫,丹尼·雷尔。“MN1302”大豆的注册。作物科学。2004;44(2):693。
- 28。
花生四烯酸和6,9,12-十八碳三烯酸的过氧化物酶体氧化过程中积累的酰基辅酶as的分析。生物化学学报。1998;349(2):371-5。
- 29。
赫克曼WJ,Tanaka CK。二维凝胶电泳的溶解植物膜蛋白分析。植物理性。1986; 81(3):802-6。
- 30
Wang X, Shan G, Schafer B, Herman RA, Tobias R, Shippar J, Malayappan B, Sheng L, et al. .开发、验证和实验室间评价一种定量多路复用方法来评估大豆种子中10种内源过敏原的水平,以及它在跨越三个生长季节的大田试验中的应用。中国农业科学。2017;65(27):5531-44。
致谢
部分结果作为海报在ASPB会议植物生物学2018 (https://www.eventsce.com/2018/psbb/postertitles.asp?postersortorder = num.),植物生物学2019 (http://www.cvent.com/events/plant-biology-2019-posters/agenda-4a7213fd4e4045398ed575400f5ed3e5.aspx),中大西洋植物分子生物学学会,2019 (MAPMBS)http://wp.towson.edu/mapmbs/
资金
这项工作由美国农业部农业研究服务局CRIS项目8042–21220-234-00D资助。资助机构为研究项目、研究设计、数据收集、分析和手稿准备提供资金支持。
作者信息
从属关系
贡献
NI进行实验室实验,分析数据,起草手稿;RMS设计了CGH实验;QS PCR分析;DLL进行脂肪酸分析;WG通过MS鉴定蛋白质;AOS进行了实地研究;JR进行了CGH分析;SSN构思、设计和监督该项目,并对手稿的草稿版本进行了工作。所有作者阅读并批准了最终的手稿。
相应的作者
伦理宣言
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
所有作者声明没有利益冲突,除了Robert Stupar博士,他曾担任本杂志的副编辑。
补充资料
出版商说明
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放访问本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条中提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
伊斯兰,N.;斯图塔尔,R.M.;et al。快中子诱导大豆突变体中子集中种子蛋白和油含量的基因组变化和生化改变。BMC植物BIOL.19,420(2019)。https://doi.org/10.1186/s12870-019-1981-x.
收到了:
公认:
发表:
关键字
- 大豆
- 蛋白质
- 基因删除
- 脂肪酸
- 石油
- 突变体
- 比较基因组杂交
- 蛋白质