摘要
背景
棉花是最重要的商业作物之一,是天然纤维、油和饲料的来源。为了保护棉花免受有害害虫的侵害,已经开发了许多新的转基因品系。为了在成功的农业中快速检测表达,qPCR(定量聚合酶链反应)选择合适的参考基因对实验结果起着至关重要的作用,因为该方法量化了目标基因与参考基因的表达。传统上最常用的参考基因是“看家基因”,参与基本的细胞过程。然而,这些基因的表达水平往往随着实验条件的变化而变化,这迫使研究人员验证每个实验平台的参考基因。这项研究提出了一种数据科学驱动的无偏全基因组搜索,通过评估变异来选择参考基因n共> 一个公开的棉花品种RNA-seq数据集中的50000个基因gossypium hirsutum.
结果
五个基因(TMN5,TBL6,UTR5B,AT1G65240和CYP76B6),以及文献中发现的两种常用的参考基因(PP2A1和UBQ14),对转基因和非转基因植物的不同组织(叶、方、茎、根)、叶片不同发育阶段(幼、成熟)和方发育阶段(小、中、大)的33份实验样品进行qPCR检测。采用geNorm、BestKeeper、NormFinder和RefFinder四种算法评价基因的表达稳定性。
结论
根据结果,我们建议使用TMN5.和TBL6作为正常和转基因棉花组织qPCR实验的最佳候选参考基因。AT1g65240和PP2A1如果表达研究包括正方形,也可以使用。本研究首次成功地显示了数据科学驱动的基因组视图方法,然后是实验验证作为选择基于单独的功能的选择选择稳定参考基因的方法。
背景
实时定量PCR(qPCR)是研究差异基因表达的最可靠、准确和经济有效的技术[1].该技术在基因表达方面比Northern blot杂交和RT-PCR(逆转录聚合酶链反应)等常用方法具有优势,具有较高的敏感性、特异性、准确性和广泛的动态范围[2那3.].然而,影响QPCR数据的主要缺陷是多种因素的可变性,例如RNA样本的质量和完整性,CDNA合成效率和实验的PCR效率[4.].像的良好参考基因的选择合适的归一化的策略是至关重要的,在从实验的qPCR生物学意义的基因表达的结果[到达5.].
一个好的内参基因应该在各种实验参数中具有恒定的表达水平[6.那7.或在不同组织的细胞之间[8.]传统上用于qPCR研究的参考基因是半定量方法的携带物[9.].通常发现许多这些基因的表达水平非常高,导致低表达靶基因在高度敏感的QPCR实验中的低表达靶基因的归一化[10].有人警告停止他们的qPCR的使用情况,许多研究开始表明,这些基因的转录水平可以不同的生理条件下有很大的不同[11].
通过微阵列等高通量实验获得的全基因组表达数据有助于识别稳定基因,可用于qPCR实验的标准化[1].在初步完成的工作中[12],主要重点是评估一组已知的内参候选基因的表达稳定性,使用几种标准化算法- geNorm [13],NormFinder [14]和最好的人[15].然而,一些研究人员也尝试使用生物信息学方法评估基因的稳定性[16],统计测量像的变化(CV)的系数17]以及驱动内参基因和组织特异性基因表达的启动子的DNA熵的差异[18].假设真实内参基因在样本间的分布符合正态分布,尝试利用CV和CV在大豆中发现内参基因进行表达研究P.-正态测试的值[19].在另一种尝试中,一个名为findrg的自动工作流程[20.],以CV作为主要检测手段,在不同植物物种和人类癌症中寻找内参基因。然而,使用高通量转录组测序(RNA-seq)数据来识别变异最小的基因的方法最近变得相当流行,从人类等多种生物的许多研究中可以明显看出[21),苹果(22],molusc[23),C. Savignyi.,一种海洋生物[10].
棉花是经济上最重要的栽培作物之一。它是纺织工业天然纤维的主要来源,因此是基因改造的重要目标。转基因技术已应用于棉花改良农艺性状、抗虫性、抗除草剂性和纤维品质[24那25].在本研究中,我们使用了Bollgard II(孟山都)转基因棉花,其中含有两种Bacillus thuringiensis.基因Cry1Ac和cry2ab.证明有抗鳞翅目幼虫良好的杀虫效力(棉铃虫:Helicoverpa armigera).许多研究重点是在生物和非生物胁迫下鉴定不同棉质种类的最合适的参考基因[11那24那26那27那28那29那30.那31那32那33那34那35那36].然而,对转基因表达效果的研究变得非常重要,因为各种研究表明,由于转基因在两种植物中的表达,内源性基因的表达发生了改变[37那38[动物[39]因此,需要彻底评估转基因对内源基因表达水平的影响,尤其是那些用作参考基因的基因[39在开始一项研究之前。
在这项研究中,我们已经从公共可用的基因表达数据集中使用了一个无偏见的基因组搜索,用于棉花,以识别潜在的参考基因候选者,以最终选择棉花中最稳定的基因表达分析基因。我们已经解决了在非转基因和转基因中使用的潜在候选参考基因的表达稳定性g .分子在不同组织(叶、茎、方)、年龄类别(1 - 3月龄植株)、叶片发育阶段(幼叶和成熟叶)和正方形(小、中、大正方形)的不同实验条件下进行。本研究采用的数据驱动分析方法与实验验证相辅相成,可推广到其他具有大量数据的科学模型系统。
结果
候选基因的选择
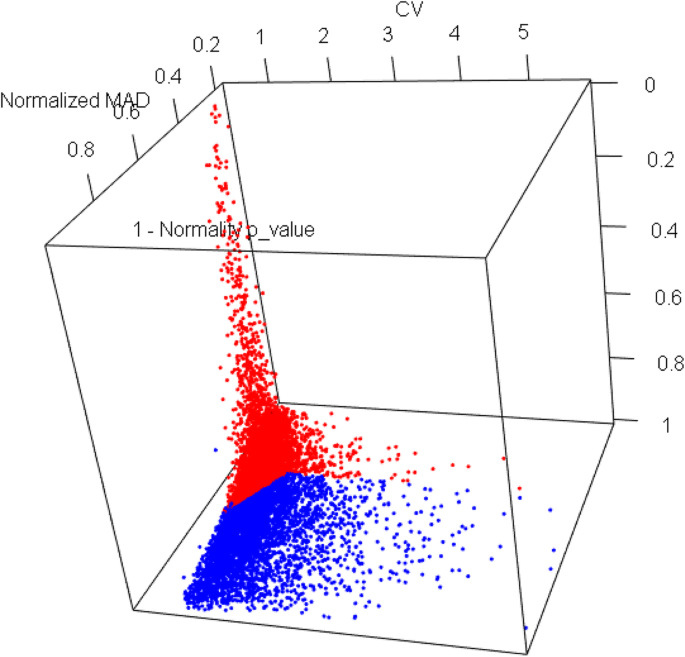
从公开的棉花FGD数据集(www.cottonfgd.org.)含有66,577个基因的RNA-SEQ FPKM值。在此设置中,只有51,272个基因可以从JGI注释中映射到基因名称作为相同数据集的一部分。从该注释的组中,消除11,137个基因作为低表达基因(中值FPKM≤0),并且使用剩余的40,135个基因进行分析。剪影分析表明,分析只有两个群集最佳(附加文件)3.)(CV,MAD,1-p)超空间中两个星团的表示如图所示。1在表格中给出的细节1.
从簇2中选择来自簇1的基因,并根据计算的欧几里德距离以升序排序。根据其亚细胞位置和分子函数进一步滤除前100个基因。本地化到核的基因产品更可能参与转录,并且可以在导致差分mRNA表达的不同实验条件下进行变化。因此,不考虑进一步分析的非细胞质表达基因或参与翻译中的基因。整个工作流程总结在图4中。2和考虑进一步分析表中的前十个基因2.在生物应激下棉花研究中使用的两个最普遍的参考基因,UBQ14,一个多泛素和PP2A1,蛋白质磷酸酶[11,纳入实验验证,对比见表2.
选择底漆
以棉花植株各部位汇集的cDNA为目标,对筛选出的前10个基因进行熔化曲线分析,筛选出5个基因TMN5,TBL6,UTR5B,AT1G65240和CYP76B6即符合条件者良好的引物。The use of these primers resulted in a single amplification product of expected size with the templates and no amplification (more than 35 Cq) for non-template controls (Additional file4.).使用cDNA的5倍稀释液为五个参考基因的引物引物效率的计算,得到R.2 > 0.97和效率(E)值为1.9–2.0(附加文件2和表3.).其余引物的引物效率都很低。
qPCR分析候选内参基因的表达
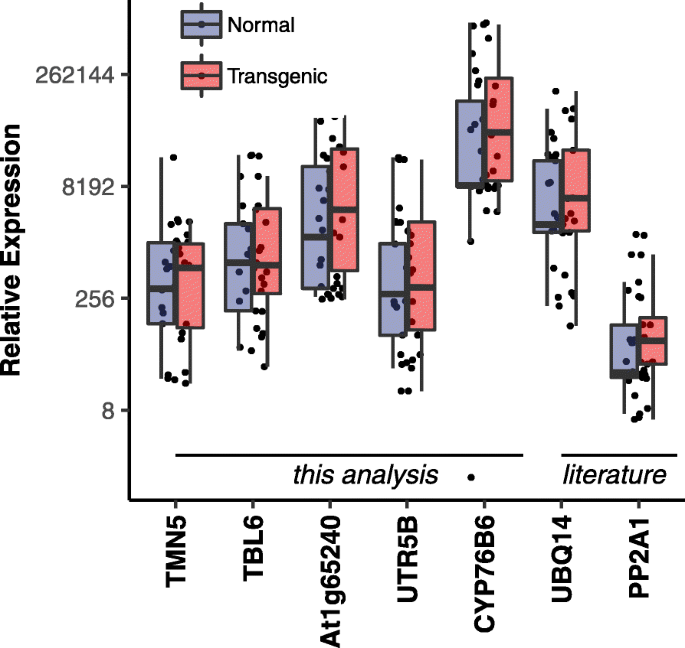
在转基因和非转基因品系之间的所有组织样本(叶、方和茎)中,分析选择的候选内参基因的表达(图)。3.).由于鳞翅目不侵染根,因此只在非转基因品系中纳入鳞翅目。RNA质量已在附加文件中显示1.在无花果。3.那TBL6和UTR5B在两个类别之间的差异最小,其次是TMN5.和AT1g65240.常用的基因PP2A1尽管在研究组中表现出较低的表达中表达,但在转基因和非转基因系之间表现出更大的变化。与另一种流行的基因可以观察到相同的趋势UBQ14.
不同年龄类别中内参基因在不同植物器官中的表达稳定性
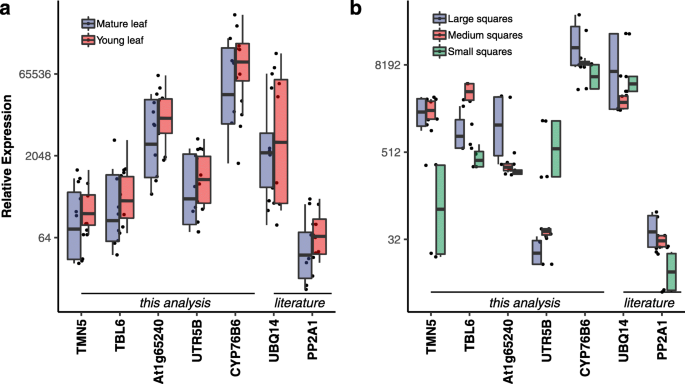
我们进一步分析了内参基因在不同植物组织(叶、方和茎)和不同年龄类别(1和3月龄)中的行为,并在每个器官和年龄类别中选取了4个生物复制的组织样本。从图中可以看出。4.A和B,那TBL6和TMN5.在不同的组织和年龄类别中表现出一致的表达,表明在分析外源基因或转基因基因的表达水平时,这些基因的组合是最优的。这一结果也与geNorm分析结果一致,geNorm将这两个基因排在第1位,稳定性参数值最小(附加文件)5.).
跨叶和方形组织的差动发育阶段的参考基因表达稳定性
分析了内参基因在叶片不同发育阶段的表达情况TMN5.(无花果。5.a)至具有在成熟底部叶片和年轻终端叶之间中值表达的至少变性。然而,在正方形的组织的不同发育阶段的候选参考基因的表达谱表现出不同的图案。CYP76B6和At1g65240(无花果。5.B)在方形组织的不同发育阶段表达更为一致TMN5, TBL6和UTR5B表现出更大的可变性。
候选内参基因的综合稳定性分析
候选基因的稳定性由BestKeeper和RefFinder从原始CP.值和通过geNorm和NormFinder从相对表达式(附加文件5.).在每种算法中分配的稳定性等级的几何平均值用于产生所有候选参考基因的综合稳定性排序(图。6.).分析表明,TMN5.和TBL6在所有组织类型中最稳定。然而,包括来自文献的常用参考基因的稳定性分析表明PP2A1一种T.最高等级后面TMN5.和TBL6(附加文件5.).
讨论
qPCR结果的可靠性在很大程度上依赖于目的基因表达水平归一化的内参基因。因此,内参基因的选择是比较基因表达研究中最关键的参数。大多数可靠的内参基因不应跨样本类型进行调控,如从植物不同部位分离的组织,跨不同年龄和发育阶段,不同的非生物和生物胁迫条件以及不同的基因型。然而,很难确定一个单一的内参基因在所有实验条件下都是最优的。Joseph等人在一篇综述论文中汇编[11]和两篇最近的文献[24那36],20多个参考基因已用于棉花植株的表达分析。福斯托等人[47甚至用miRNA作为参考基因,以表达棉花mRNA。然而,所有这些基因都是基于其在其他系统中的功能或传统使用的基础。目前的研究是第一次尝试无偏见的基因组宽搜索,以发现各种组织和转基因应力的表达分析最稳定的基因。
在引物效率检验的基础上,我们提出了5个新基因以供进一步实验。通过qPCR检测这些基因在不同组织、年龄类别、发育阶段和不同基因型的实验集中的表达稳定性。两个基因,TMN5和TBL6作为最整体稳定基因出现(图。6.).虽然两者之间没有太大的区别AT1g65240和UTR5B总体稳定,AT1g65240在Cry基因表达防御生物胁迫的目标植物器官中,正方形的变异较小。因此,总体稳定TMN5,TBL6和AT1G65240出现清除赢家.在测试的五个基因CYP76B6具有最高的表达和最不稳定性,在与其他生物的研究中观察到的模式[10].
对这些基因功能的研究揭示了这一点TMN5.编码跨膜9超家族成员5蛋白,该蛋白在细胞膜的蛋白质定位中起作用拟南芥蒂利亚纳[48].有趣的是,最近的基因组广泛寻找参考基因海鞘,一种软体动物,也已确定TMN.是一个稳定的基因[10].TBL6编码毛状体双折射样6,参与乙酰基转移酶活性拟南芥[49].编码的蛋白质AT1g65240已经显示出在种子荚中表达的天冬氨酸蛋白酶样蛋白,并提出在种子中储存蛋白的加工和降解中发挥作用[50[因此,解释了方形组织中的高表达水平。相似地,CYP76B6,香叶醇羟化酶,属于细胞色素P450酶CYP76家族,催化所有线性单萜醇的单或双氧化,其衍生物在花、果和幼叶中特别常见[51].由蛋白质编码的蛋白质UTR5B参与碳水化合物的运输[52].
两个基因来自文献PP2A1和UBQ14,与本研究中发现的5个基因在同一实验平台上进行比较,发现其表达水平较低PP2A1是类似于TMN5.和TBL6。由蛋白质编码的蛋白质PP2A1是蛋白磷酸酶2A的催化亚基[26三聚体全酶,广泛表达丝氨酸/苏氨酸蛋白磷酸酶,在真核生物中保守。虽然全酶在许多功能中参与调节病原体的反应[53,催化亚基在非转基因和转基因棉花植株之间表现出稳定性。
如本研究所述,这种分析发现的最稳定的基因具有非常多样化的功能。因此,我们基于功能选择参考基因的传统观念并不需要良好。
这是第一次结合RNA-seq数据分析和对非转基因和转基因棉花植株的实验验证,对qPCR中使用的内参基因进行验证。这项研究变得很重要,因为转基因棉花占印度棉花种植总面积的95% [54而转基因表达水平的周期性估计是确定这些转基因作物功能的关键检查点。但本研究仅涵盖了内参基因在正常野外条件下的表达稳定性,不包括生物和非生物胁迫条件对候选内参基因的影响。
结论
本研究采用数据驱动的方法对内参基因进行鉴定和实验验证,用于转基因和非转基因品系的qPCR研究gossypium hirsutum.在五个新的候选内参基因中TMN5.那TBL6那At1g65240那CYP76B6和UTR5B经过分析,前三个基因在我们实验中测试的所有条件下都表现出稳定的表达水平,因此是棉花品种中潜在的参考基因g .分子。
方法
本研究的工作流程(图。2)由两大部分组成:(i)对公开可用的RNA-seq数据进行统计分析,以确定表达变化最小的候选内参基因;(ii)选定内参基因的实验验证。
基因表达数据采集
RNA-SEQ每百万百万读(FPKM)值的RNA-SEQ片段g .分子从棉花功能基因组学数据库(CottonFGD)下载[40],据我们所知,这是唯一公开可用的RNA-seq表达数据集g .分子基因。FPKM值固有地包含两个标准化因子—(i)基因间转录长度的差异和(ii)由于在本次分析中,我们的目的是鉴定样本间变异最小的稳定基因,因此没有进行进一步的标准化以消除此类变异。最近发表的关于其他生物体类似转录组分析的文献[10那21那22那23还没有进一步规范化的FPKM或TPM值。
棉花fgd包含种子萌发(时间序列和器官比较)、组织器官、胚珠发育(时间序列)和胁迫实验(热、冷、热、冷、热、低温)等不同实验组55个不同实验条件下66,577个基因的基因表达数据。盐和聚乙二醇与对照组一起处理样品)。我们下载了联合基因组研究所(JGI)汇编[41]的注释g .分子从同一数据库中,只有映射基因名称和染色体位置只能签订基因。
在任何基因表达的实验中,低表达的基因经常创建用于下游分析的问题这样的基因的表达值往往从采样噪声区分。To eliminate such genes from the analysis, we used a filtering criterion of median FPKM value > 0 (or in other words non-zero FPKM value in at least 28 out of 55 samples). Genes those passed the above criteria was selected for the statistical analysis (Fig.2).
统计分析
为了评估基因的稳定性,采用了两种措施 - (i)CV\(= \划线{X} / {\西格玛} _x \)在哪里\(\眉题{x} \)和σX平均值和标准差是一个变量吗X分别和(ii)常态性P.-由夏皮罗-威尔克斯试验测定的值(P.-value < 0.05表示偏离正态分布)[18].CV,尽管最常用的,是由异常的影响,并且因此不会成为一个强大的措施。为了解决这个问题,第三个参数 - 平均绝对偏差(\(\ mathrm {mad} = \ kern0.5em中位数\ kern0.75em \ lef | x- \ hat {x} \右| \)在哪里\ ({x} \ \帽子)是X) [42中位数归一化后使用。MAD是一种分布扩散的度量,基于中值,因此不太容易受到异常值的影响。
一组理想的内参基因应该在样本之间具有较低或相似的统计变异(CV和MAD值较低),并且表现得像正态分布(正态p值较高或1 - p值较低)。因此,我们使用Kaufman和Rousseeuw最初提出的PAM (Partitioning around Medoids)算法,根据CV、MAD(归一化为各自的z-scores)和1 - p-value三个统计参数的值对基因进行k-medoids聚类[43].选择基于麦细基的聚类方法,更常用的K均值方法选择,以减少异常基因在聚类测定中的效果。使用ROSSEEUW的轮廓图形方法计算所需的最佳簇数[44].选取三个参数中medoid值最低的聚类,利用欧氏距离对聚类中的基因进行排序\(d=\sqrt{CV^2+{MAD}^2+{\left(1-p\ right)}^2}\)在这三个参数的超空间中,选择欧氏距离最小的前100个基因,并使用基因本体注释分析其亚细胞位置和功能。
从文献中选择常用的内参基因
最常用的参考基因是从文献中介绍了他们在2016年从棉花上发表的科学文学中的使用频率。研究人员没有使用的唯一关键词来报告表达研究中的参考基因。因此,采用了一种非常广泛的方法,其中搜索了术语中的任何术语“参考基因”或“对照基因”或“家务基因”的所有文章以及该术语的共同发生gossypium hirsutum在标题和摘要的任何地方。所获得的摘要进行人工监管,发现特别是在棉花植株的上下文中描述的参考基因研究的相关文章。
植物材料
采用1、3月龄转基因(Bollgard II,孟山都15985,抗虫品系)和非转基因品系进行试验gossypium hirsutum植物在正常场条件下生长,温度范围为25°C±4°C和自然光周期。在研究过程中,没有生物或非生物应激施加到植物。使用的器官是茎,末端叶,底部成熟的叶子和不同发育阶段的平方(小:销和匹配头正方形,中等:方形生长中点和大:蜡烛正方形)。根仅来自非转基因植物。根据Artico等人确定重复。[26].简而言之,从三种不同的棉植物中收获材料以获得一个游泳池。用三种不同的植物重复该过程,以获得第二池,生物重复。制备来自转基因和非转基因系的33类实验类别,包括叶,正方形,根和干组织。对于以各种实验类别进行的每个统计分析,分析了每种组织的四种重复。从所有组织中刚提取总RNA并储存在-80℃。
总RNA分离和cDNA合成
如前所述从新组织中提取总RNA [45略作修改。简单地说,新鲜组织在预冷的研钵和杵中均质成细膏状,每克植物组织使用10ml提取缓冲液(400 μl β -巯基乙醇和4%聚乙烯吡咯烷酮)。以25:24:1的比例加入等量的水饱和酚、氯仿和异戊醇,充分混合并离心。用氯仿-异戊醇萃取分离的水相,然后加入最终浓度为3 M的氯化锂(LiCl)。在−20°C孵育过夜后,RNA沉淀在2 M LiCl中重新悬浮,离心,用70%的冰冷乙醇洗涤。颗粒风干后溶解于500 μl无菌水中。使用NanoDrop™2000/2000c分光光度计(Thermo Scientific)测定RNA的质量和纯度,并用1%琼脂糖凝胶电泳和溴化乙酯染色检查RNA的完整性(附加文件)1).
通过将50μm的寡核苷酸(DT)引物,10mm的DNTPS(MBT079,HEMEDIA),AMV RT(NEB,#M0277S),1X第一链缓冲液,RNase抑制剂(Applied Biosystems,N8080119)加入1μg总RNA。将混合物在制造商的指示下在42℃下孵育1小时。通过在80℃下将混合物温育5分钟来灭活逆转录酶,并将CDNA溶液在-20℃下储存。
实时定量聚合酶链反应
底漆设计
从筛选出的前10个基因中设计引物(表)2)使用Primer Quest工具(https://eu.idtdna.com/pages/tools/oligoanalyzer.,2019年5月24日,DNA Technologies,Inc。)用于从集成的DNA技术实时PCR使用标准,扩增产物的范围为90至110bp,TM为62±1°C。对前10个选定的候选基因进行熔体曲线分析,并选择显示单个扩增子的引物进行进一步的实验。通过使用公式,效率(%)=(10,通过使用汇集的cDNA的连续稀释液的标准曲线法测定每个引物对的引物效率。(%)=(10-1 /斜坡−1)× 100(表13.和额外的文件2).
qPCR在罗氏LightCycler 480 II仪器上进行,使用KAPA SyBr green通用试剂盒(Sigma, #KK4600),使用96孔或384孔光学板(罗氏LightCycler®480 Multiwell plates 96或384,clear, C0687653 384或C1468659)。反应混合物中含有稀释后的cDNA (1:1) 1 μl,每个引物的浓度为0.2 μM, KAPA SyBr Green混合物的浓度为2.5 μl,水的浓度为1.3 μl,总体积为5 μl。反应混合物在95°C孵育10 s,然后在95°C扩增45个10 s循环;60°C时15秒,72°C时15秒。对每个样品进行3个技术重复的qPCR。每个引物对还包括3个阴性对照,其中cDNA被核酸酶游离水取代。
基因表达稳定性分析
qPCR实验中每个基因的定量周期(Cq)值减去非模板对照(NTC) Cq值的平均值,得到ΔC问:= C问:(样品) - 平均数Ç问:(NTC)和相对表达为e——ΔCq对于每次复制,其中E是从标准曲线获得的每个基因的引物效率。如结果所示,在不同样品上绘制复制的表达值的几何平均值。
为了分析表达的稳定性,我们使用了三个不同的算法族造产[13],NormFinder [14]和最好的人[15),以及基于网络的RefFinder [19这个工具集成了所有这三种算法和deltaact方法。使用SLqPCR R包运行geNorm算法[46],而作者提供的R封装或Excel工作表分别用于诺考文献和Bestkeeper。所有33个实验的每个基因的平均CQ值被用作BestKeeper和Reffinder的输入,而对于Genorm和NARMFINDER使用相对表达值。由于常常规方法使用基于模型的方法来量化和局部内变化,因此正常和转基因样本用作两组用于奈斯法官分析。
最好的算法的结果,独立使用或作为reffinder的一部分使用的相当。然而,排名不同,因为Bestkeeper使用与Bestkeeper Index的相关系数作为排序参数,而其Reffinder实现使用标准偏差来对基因进行排名。在使用不同的输入时,尚未将改进工具和Reminfinder分析的结果和Reanfinder分析的结果进行比较。每个基因的综合稳定性等级计算为每种方法给出的稳定性等级的几何平均值。
数据和材料的可用性
目前的研究中分析的数据集可从合理要求通讯作者。
在这项研究中产生的或分析所有的数据都包括在此发表的文章[及其附加文件]英寸
缩写
- AT1G65240:
-
天冬氨酸蛋白酶样蛋白2
- AZG1:
-
腺嘌呤和鸟嘌呤通透酶AZG1
- cDNA:
-
互补脱氧核糖核酸
- COV1:
-
蛋白连续血管环1
- CQ:
-
量化周期
- 简历:
-
变异系数
- CYP76B6:
-
香叶醇8-hydroxylase
- 脱氧核糖核酸:
-
脱氧核糖核酸
- EMB8:
-
Embryogenesis-associated蛋白质EMB8
- FPKM:
-
每百万百万映射的成绩单每千碱基映射的碎片
- 走:
-
基因本体论
- 变得更:
-
联合基因组研究所
- 疯狂的:
-
中位绝对偏离
- NCBI:
-
国家生物技术信息中心
- 全国过渡委员会:
-
Non-template控制
- 帕姆:
-
围绕中心点分区
- PEG:
-
聚乙二醇
- PP2A1:
-
蛋白质磷酸酶2a.
- QPCR:
-
定量实时聚合酶链
- RNA:
-
核糖核酸
- RPK2:
-
类受体丝氨酸/苏氨酸蛋白激酶RPK2
- rt - pcr:
-
逆转录聚合酶链式反应
- TBL6:
-
蛋白毛状体双折射样
- TMK3:
-
受体激酶TMK3
- TMN5:
-
跨膜9超家族成员5
- UBQ14:
-
聚泛素
- UTR5B:
-
UDP-半乳糖/ UDP-葡萄糖转运蛋白5B
参考文献
- 1.
Kozera B,Rapacz M.实时PCR中的参考基因。J Appl Genet。2013; 54(4):391-406。
- 2.
孙R,何Q,张B,王Q.雷蒙地棉中可靠参考基因的选择与验证.生物科技.Lett.2015;37(7):1483-93。
- 3.
巴斯廷SA。实时的,基于荧光定量RT-PCR:目前的程序和喜好的快照。专家Rev Mol Diagn。2005; 5(4):493-8。
- 4.
Lü J,杨超,张勇,潘华。昆虫RT-qPCR数据规范化的内参基因选择:系统综述。杂志。2018;9:1560。
- 5.
朱旭,李昕,陈伟,陈建军,卢伟,陈磊,等。在不同实验条件下评价木瓜中新的内参基因的准确转录标准。《公共科学图书馆•综合》。2012;7 (8):e44405。
- 6.
Schmittgen TD, Zakrajsek BA。实验处理对管家基因表达的影响:实时定量RT-PCR验证。生物化学学报。2000;46(1-2):69-81。
- 7.
Thellin O, Zorzi W, Lakaye B, De Borman B, Coumans B, Hennen G,等。内务管理基因作为内部标准:使用和限制。生物科技J》。1999;75(2 - 3):291 - 5。
- 8.
定量实时逆转录聚合酶链反应的缺陷。生物化学学报。2004;15(3):155。
- 9.
Real-time RT-PCR归一化;策略和注意事项。基因Immun。2005;6(4):279。
- 10。
Zhan A,Huang X,Li S.基因组 - 在Ciona Savignyi中温度和盐度应力下基因表达分析的新参考基因的评价。前群体。2019; 10:71。
- 11.
Joseph JT, Poolakkalody NJ, Shah JM。植物发育和胁迫反应研究的内参基因。J Biosci。2018;43(1):173 - 87。
- 12.
Almeida TA, quispes - ricalde A, de Oca FM, Foronda P, Hernández MM.高通量开放阵列qPCR基因板鉴定适合肌层和平滑肌瘤表达分析的管家基因。Gynecol杂志。2014;134(1):138 - 43。
- 13.
VandesompeleĴ,德译码K,PATTYN楼波佩B,范罗伊N,德Paepe A,等人。通过的多个内部对照基因几何平均实时定量RT-PCR数据的精确正常化。基因组Biol。2002; 3(7):research0034。
- 14.
安德森Cl,Jensen JL,ØrntoftTF。实时定量逆转录PCR数据的归一化:基于模型的方差估计的方式来确定适合于标准化基因,施加到膀胱癌和结肠癌的数据集。癌症res。2004; 64(15):5245-50。
- 15.
Pfaffl MW,Tichopad A,PrGomet C,Neuvians TP。使用配对相关性测定稳定监管靶基因,差异调节靶基因和样品完整性:基于Beykeeper-Excel的工具。Biotechnol Lett。2004; 26(6):509-15。
- 16.
Beer L,Mlitz V,Gschwandtner M,Berger T,Narzt MS,Gruber F等。研究人类角质形成细胞基因表达时选择正确参考基因的生物信息学方法。Exp Detrical.2015;24(10):742-7。
- 17.
王志强,王志强,王志强,等。应用微阵列技术筛选中国仓鼠卵巢细胞的管家基因。Biotechnol Bioeng。2009;104(5):1041 - 6。
- 18.
Thomas D, Finan C, Newport MJ, Jones S. DNA熵揭示了家务和组织特异性基因启动子之间的复杂性的显著差异。compput Biol Chem. 2015; 58:19-24。
- 19.
谢飞,肖鹏,陈东,徐磊,张斌。miRDeepFinder:一种用于植物小rna深度测序的miRNA分析工具。植物学报。2012;80(1):75-84。
- 20。
卡莫纳R,阿罗约男,门尼斯-Quesada的MJ,Seoane的P,扎弗拉A,Larrosa R,等人。自动识别的基于RNA-SEQ数据参考基因。BioMed Eng在线。2017; 16(1):65。
- 21。
MacRae T, Sargeant T, Lemieux S, Hébert J, Deneault É, Sauvageau G. RNA-Seq揭示了剪接体和蛋白酶体基因是人类癌细胞中最一致的转录本。《公共科学图书馆•综合》。2013;8 (9):e72884。
- 22。
周志刚,丛萍,田颖,朱艳。利用RNA-seq数据筛选苹果根基因表达的内参基因。《公共科学图书馆•综合》。2017;12 (9):e0185288。
- 23。
李啊,张立,李河,张米,李伊,王H,等。系统鉴定和验证来自60 RNA-SEQ文库中的参考基因在扇形mizuhopecten yessoensis中。BMC基因组学。2019; 20(1):288。
- 24.
Yi C,HONG Y.通过实时定量PCR估算转化棉中转基因的拷贝数。转基因棉。在:张B.(EDS)转基因棉。分子生物学的方法。纽约:人类新闻。2019; 1902:137-157。
- 25.
Hamid R,Tomar Rs,Marashi H,Shafaroudi Sm,Golakiya Ba,Mohsenpour M.转录组分析和编目舒适的棉花花芽和无菌线的花芽(Gossypium hirsutum L.)。基因。2018; 660:80-91。
- 26.
陆地棉(Gossypium hirsutum)新内参基因的鉴定和评价及其实时定量RT-PCR数据的精确归一化。植物学报。2010;10(1):49。
- 27.
王M,王Q,张B.棉花中非生物胁迫下基因表达可靠参考基因的评价与选择(Gossymium hirsutum L.)。基因。2013; 530(1):44-50。
- 28.
朱永宁,史德强,阮明波,张丽丽,孟Z-H,刘杰,等。转录组分析揭示了棉花对多种非生物胁迫响应基因的串扰(gossypium hirsutuml .)。8。2013;(11):e80218。
- 29.
梁C,孟Z,孟Z,Malik W,Yan R,Lwin Km,等。ghabf2,bzip转录因子,棉花的促进和盐度耐受性(gossypium hirsutuml .)。SCI批准。2016; 6:35040。
- 30.
林敏,庞超,范森,宋敏,魏华,于森gossypium hirsutumL.转录组在RNA-SEQ叶片衰老期间。BMC植物BIOL。2015; 15(1):43。
- 31。
郭勇,魏华,宋敏,等。新型棉花GhNAC18NAC基因,参与叶片衰老和各种胁迫反应。生物技术学报。2016;15(24):1233-45。
- 32。
李东东,张明,洪丽丽,陈晓波,葛杨,葛兴奇,等。新型棉花GhMAPKKK49 (gossypium hirsutuml .)MAPKKK基因,参与多种应激反应。植物生理学报,2016;38(1):13。
- 33。
萌C,CAI C,张T,郭W.六种新型NAC基因的表征及其对Gossymium Hirsutum L.植物SCI中非生物胁迫的反应。2009; 176(3):352-9。
- 34。
黄晓智,陈建勇,肖慧娟,肖云涛,吴杰,吴建新,等。陆地棉对棉铃虫响应的动态转录组分析和挥发性分析Sci众议员2015;5:11867。
- 35。
关键词:ZmSUT1,棉花,衰老诱导,叶片衰老,籽皮特异表达植物学报2019;38:1-10。
- 36.
曹A,邵d,崔B,童X,郑Y,太阳J,等。在具有不同的SE功能4个陆地棉品种SE初始去分化过程中筛选通过RT-qPCR定量基因表达的参考基因。基因。2019; 10(7):497。
- 37.
Stam M, Mol JN, Kooter JM。转基因植物中基因的沉默。安机器人。1997;79(1):3 - 12。
- 38.
Antosiewicz DM,Barabasz A,Siemianowski O.表型和金属 - 稳态基因过表达的分子的后果。前植物SCI。2014; 5:80。
- 39.
关键词:转基因,基因插入位点,内源基因,基因互作神经科学。2018;11:385。
- 40。
朱T,梁C,孟Z,孙G,孟Z,郭S等人。CottonFGD:综合功能基因组数据库的棉花。BMC植物BIOL。2017; 17(1):101。
- 41。
帕特森啊,温德尔JF,Gundlach H,Guo H,Jenkins J,Jin D等。重复多倍化的Gossypium Genomes和可旋转棉纤维的演化。自然。2012; 492(7429):423。
- 42。
PHAM-GIA T,洪T.均值和中位数绝对偏差。数学计算模型。2001; 34(7-8):921-36。
- 43。
Kaufman L,Rousseeuw PJ.在数据中发现群体:聚类分析导论:Wiley;2009。https://doi.org/10.1002/9780470316801.
- 44。
Rousseeuw PJ。轮廓:一个图形辅助解释和验证聚类分析。计算机应用数学。1987;20:53-65。
- 45。
Sajeevan RS,Shivanna MB,KN塔罗闍。一种有效的协议,用于从桑树的健康,并强调组织中总RNA分离(桑属)等种类。am j植物sci。2014; 5(13):2057。
- 46。
Kohl M. SLqPCR:用于SIRS-Lab GmbH实时定量PCR数据分析的功能。Jena: R Package, SIRS-Lab GmbH;2007.
- 47。
Fausto Aks,Da Franca St,Romanel E,Vaslin MF。Micrornas作为棉花中定量PCR的参考基因。Plos一个。2017; 12(4):E0174722。
- 48.
UniProt KB。https://www.uniprot.org/uniprot/f4hw17.Accessed Aug 15 2019。
- 49.
UniProt KB。https://www.uniprot.org/uniprot/q9lzq1..Accessed Aug 15 2019。
- 50。
拟南芥中3个典型的天冬氨酸蛋白酶基因的差异表达。生物化学学报。2002;269(18):4675-84。
- 51。
ILC T,坐标C,贝梭B,Navrot N,Werck-Reichhart D. monoterpenol氧化代谢:在植物适应和潜在应用中的作用。前植物SCI。2016; 7:509。
- 52。
UniProt KB。https://www.uniprot.org/uniprot/A0A1U8PXU0.Accessed Aug 15 2019。
- 53。
非典型蛋白磷酸酶2A基因家族不通过古多倍化扩增。植物杂志。2017;173(2):1283 - 300。
- 54。
Priyadarshini SE,Chakraborty TE.《生物技术:可持续社会经济转型的代理人》,印度政府科技部生物技术司,30年出版。古尔冈:NPG India;2016年。
确认
作者感谢Nehanjali Dwivedi,MSMF,班加罗尔对稿件的严格审查,欢乐库里,Haresh Dagale和Chandramani辛格,电子系统工程,IISC,班加罗尔部好心提供计算基础设施和数据分析过程的严格审查。
资金
本研究由超越抗体LLP资助。
作者信息
隶属关系
贡献
SPK设计并完成了实验,并起草了手稿。AKM和CB有助于植物生长、样本收集和RNA提取。ASK和VK帮助进行了qPCR。SKD构思了这项研究,进行了统计和数据分析,并起草了手稿。CKN指导了这项研究,MD协调了这项研究,并对文献和手稿进行了严格的审查。所有作者阅读并批准了最终文本。
相应的作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
附加信息
出版商的注意事项
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
权利和权限
开放获取本文根据知识共享署名4.0国际许可的条款分发(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上无限制地使用、分发和复制,前提是你给予原作者和来源适当的荣誉,提供一个到知识共享许可协议的链接,并指出是否作出了更改。创作共用及公共领域专用豁免书(http://creativecommons.org/publicdomain/zero/1.0/)除非另有说明,否则适用于本文中提供的数据。
关于这篇文章
引用这篇文章
Smitha,P.K.,Vishnupriyan,K.,Kar,A.S.等基因组广泛搜索,以确定参考基因候选基因的基因表达分析gossypium hirsutum.BMC植物杂志19,405(2019)。https://doi.org/10.1186/s12870-019-1988-3
已收到:
公认:
发表:
关键词
- 参考基因
- qPCR
- gossypium hirsutum
- 转基因
- 数据科学
- 棉花