摘要
背景
黄麻(Corchorus就种植面积和产量而言,棉花是仅次于棉花的最重要的天然纤维作物。盐胁迫严重制约植物的生长发育。高密度遗传连锁图谱是定量性状位点(qtl)定位的基础。通过下一代测序,许多作物品种已经开发出了与耐盐性相关的高密度遗传图谱和qtl图谱。然而,对黄麻的研究很少。迄今为止,黄麻只构建了几个低密度的遗传图谱,没有发现与耐盐相关的QTL。
结果
以F2黄麻群体为研究对象,建立了7个连锁群(lgg)的高密度遗传图谱,共4839个单核苷酸多态性标记,长度1375.41 cM,相邻标记间平均距离0.28 cM, lgg2(299个标记,长度113.66 cM)和LG7(1542个标记,长度350.18 cM)。相邻标记间间隔小于5 cM的占99.57%。在4个遗传位点上鉴定出3个显著qtl和13个次要qtl,解释了0.58 ~ 19.61%的表型方差。QTL作图的区间长度为1.3 ~ 20.2 cM。在2种盐胁迫条件下检测到主要QTL qJST-1,分别解释了11.81和19.61%的表型变异,在LG4上的峰值为19.3 cM。
结论
我们使用基因分型测序方法绘制了迄今为止第一个高密度和最完整的黄麻遗传图谱。在黄麻中进行了首次耐盐QTL定位。这些结果为黄麻萌发期耐盐基因的标记辅助选择和转基因育种提供了有用的资源。
背景
黄麻是一种二倍体(2n = 14)锦葵科草本植物,包括50-60种[1].无论是种植面积还是产量,它都是仅次于棉花的世界上最重要的天然纤维作物[2].Corchorus capsularis而且Corchorus olitorius是仅有的两个栽培品种,基因组大小分别为338 Mb和445 Mb [3.].黄麻可以在营养贫乏的土壤中快速而容易地生长,同时仍能产生大量的生物量。黄麻纤维柔软,有光泽,易干燥,具有良好的吸湿性和抗菌性,可降解,可回收,环保[4].近年来,黄麻的应用不断扩大,目前常用于造纸和纺织工业,并作为草药、叶菜和可再生生物燃料来源[5,6,7,8].因此,全球对黄麻的需求正在增加[9].然而,很少有新的黄麻育种方法被开发出来,在过去几十年里,与其他韧皮纤维作物相比,育种和遗传改良进展缓慢[10].
盐胁迫是限制农业作物生产力和威胁粮食安全的主要环境因素[11].大约20%的灌溉土地受到盐度的影响[12],而在全球气候变化和环境污染的影响下,这些问题预计会变得更加严重和广泛[13].盐胁迫对植物有主要和次要影响。主要作用包括对细胞的渗透作用和离子毒性作用,而次要作用则很复杂,包括氧化应激、对细胞成分的损伤和代谢功能障碍[11].因此,了解植物耐盐性的遗传机制,定位与耐盐性相关的数量性状位点(qtl),对于提高具有重要经济价值的植物的耐盐性至关重要。
高密度遗传连锁图谱是qtl定位的基础。在许多作物品种中,使用下一代测序技术已经开发了一些与耐盐性相关的高密度遗传图谱和qtl图谱[14,15,16].然而,对黄麻的研究很少。尽管如此,在该植物中已经报道了一些与遗传图谱构建和qtl鉴定相关的研究。例如,Tao等人。10]利用100个F8株系构建了913个多态标记的遗传图谱,检测出11个株高相关qtl。c . capsularisL.),这是迄今为止所报道的最高密度的黄麻遗传连锁图谱。Kerdu等人[2]随后绘制了包含503个限制性内切位点相关DNA标记的遗传图谱,跨度358.5 cM,平均标记间隔0.72 cM,覆盖87.0%c . olitoriusl .基因组。检测到9个与组织纤维含量相关的qtl。比斯瓦斯等人。17]开发了黄麻的第三个密度图,包括48个简单序列重复(SSRs)和410个单核苷酸多态性(SNPs)c . olitoriusL.此外,利用基于传统聚合酶链反应(PCR)技术的分子标记构建了一些低密度遗传连锁图谱,如简单序列重复序列(SSRs)、扩增片段长度多态性等,基于遗传连锁揭示了一些与纤维产量和品质性状相关的qtl [18,19,20.,21,22,23].然而,黄麻耐盐性相关的QTL尚未被发现。
在这项研究中,我们利用来自一种野生种质杂交的F2群体,开发了迄今为止报道的最高密度的遗传图谱c . olitoriusL. (J009)和广丰长果(GFG)品种。利用遗传图谱鉴定了水稻耐盐性的主要qtl和次要qtl,并确定了表型方差。这些结果为黄麻萌发期耐盐基因的标记辅助选择和转基因育种提供了有用的资源。
结果
基因分型(GBS)
为了提高检测到尽可能多的SNP标记的机会,对两个亲本进行了高水平测序。GFG和J009两个亲本每次读取150 bp,共产生16.68 Gb清洁碱基(平均Q20 = 95.30%),分别为8,282,642,100 bp和8,399,779,200 bp(见附加文件)1:表S1))。对于150 F2个体,共获得113.47 Gb清洁碱基,平均Q20值为95.56%,范围447.55 Mb ~ 1087.85 Mb,平均长度756.57 Mb /人(见附录文件)1:表S1)。原始数据存入NCBI,登录号为PRJNA544984。将高质量的清洁reads与黄麻参考基因组进行比对后,平均93.12%的清洁数据被映射,覆盖97.24%的参考基因组(至少覆盖一个碱基),在两个亲本中平均覆盖深度为20.97×(见附加文件)3.:图S1)。平均94.92%的清洁数据在子代中对齐,覆盖黄麻参考基因组的21.36%,平均覆盖深度为9.71×(见附加文件)3.:图S1)。只有在参考基因组上排列到唯一位置的reads被用于SNP调用和基因分型。
SNP发现和基因分型
在两个亲本中共检测到1959,620个SNPs,根据JoinMap 4.0的CP模型将其分为8种分离类型(见附加文件)4:图S2)。分离型hk × hk、nn × np和aa × bb分别占检测到SNP标记的51.93、27.51和11.09%。F2群体遗传图谱只能使用aa × bb标记。根据标记型(aa × bb)在基因组中的位置,在150株子代植株中选择相应的基因型。共使用8150个有效标记进行遗传定位,分离畸变不显著,完整性良好(> 75%),无异常碱基。
高分辨率基因图谱构建
最终,有效标记的4839个snp被成功分配到独特的遗传位置(见附加文件)2:表S2)。因此,一个高密度的遗传图谱被开发出来,包括7个标记为ll1 - 7的连锁群上的4839个标记。1).遗传图谱总长度为1375.41 cM,相邻标记间平均距离为0.28 cM1),其中99.57%为小于5厘米。在7个LG7位点中,LG7位点的连锁群最大,共有1542个标记,长度350.18 cM,相邻标记间平均距离0.23 cM。第二大连锁群为LG6,共有991个标记,长度为232.68 cM,平均距离为0.23 cM。LG2最短,为113.66 cM,共有299个标记,标记间平均距离为0.38 cM。LG5的标记数最少(278个),相邻标记间平均距离最大(0.50 cM)。
耐盐相关qtl的鉴定
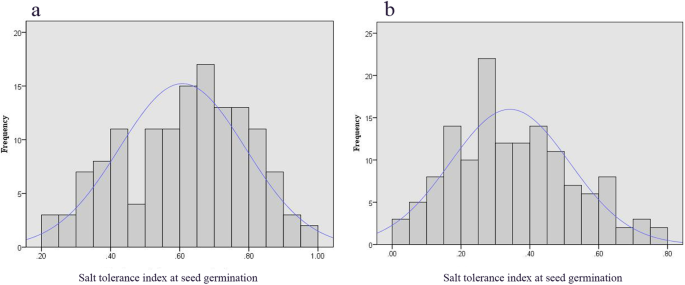
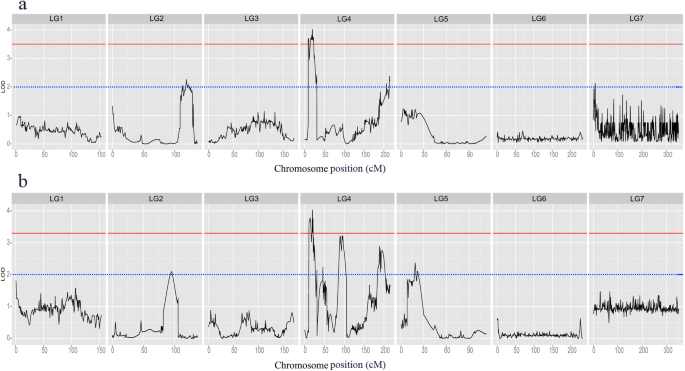
我们从150 F开始记录种子萌发时的耐盐指数(STIG)2:3品系和两个亲本在两种盐胁迫条件下试验6天。第4天的STIG数据呈正态分布(图2)。2),作为qtl的表型数据。根据1000排列检验,确定两种盐胁迫条件下LOD评分阈值约为3.5。在两种盐胁迫条件下,我们在LG4上鉴定了3个LOD值大于3.5的明显QTL,并在两种条件下同时检测到一个主要QTL,命名为qJST-1(表jst -1)2;无花果。3.).在140 mM盐胁迫条件下,qJST-1定位于LG4标记mk5633和mk6723之间的11.4 ~ 23.7 cM,在160 mM盐胁迫条件下,qJST-1定位于LG4标记mk6160和mk6484之间的16.9 ~ 21.6 cM。在140 mM和160 mM盐胁迫条件下,qJST-1在LG4上的LOD峰值均位于19.31 cM,分别占总表型变异的11.81和19.61%。在140 mM盐胁迫条件下,QTL qJST-2定位于标记mk7047和mk5638之间(LG4的9 ~ 11.4 cM位置),LOD峰值定位于LG4的10.01 cM,解释了3.74%的表型变异。在160 mM盐胁迫条件下检测到qJST-3,位于LG4位点mk6393和mk6391标记之间(10.4 ~ 16.9 cM),与qJST-2部分重叠。qJST-3的LOD峰值定位在LG4的13.41 cM,解释了8.84%的表型变异。所有qtl的加性效应均为负,表明耐盐性值的增加是由J009等位基因决定的。此外,虽然在一些位置LOD值低于3.5,但在这些位置发现了明显的峰值(LOD > 2.0),这些位置被认为是微有效(即次要)qtl所涉及的区域。在140 mM下共鉴定出13个次要qtl (n= 5)及160毫米(n= 8)盐胁迫条件(表2,无花果。3.),可映射到LG2、LG4、LG5和LG7,解释表型方差的0.58 ~ 8.12%。QTL作图的区间长度为1.3 ~ 20.2 cM。
讨论
构建迄今为止最高密度的黄麻遗传图谱
虽然已经构建了黄麻的几个遗传图谱,并鉴定出具有重要经济性状的qtl,但大部分工作涉及使用传统的DNA标记,如扩增片段长度多态性、随机扩增多态性DNA和简单间序列重复[18,19,20.,21,22,23]]。这种标记的生成是劳动密集型的、昂贵的、低通量的,因此不适合构建高分辨率遗传图谱和精细qtl作图[10].新一代测序技术的发展为SNP标记的识别提供了一种自动化、低成本、高通量的方法,也有利于构建高密度遗传图谱和精细QTL定位。然而,只有少数研究[2,10,17]已将高通量技术开发的标记用于构建黄麻遗传和QTL图谱。据我们所知,迄今为止构建的最密集的黄麻遗传图谱是使用特定位点扩增片段测序开发的,仅包括11个LGs中的913个标记,覆盖1621.4 cM,每个位点的平均密度为1.61 cM [10].在本研究中,我们使用GBS方法绘制了黄麻的第一个高密度和最完整的遗传图谱。遗传图谱包含7个LGs上的4839个标记,与黄麻的染色体数量一致(n= 7),长度为1375.41 cM,平均每个位点距离为0.28 cM,低于以往报道。因此,该遗传图谱将极大地加强黄麻QTL/基因定位和标记辅助育种。
黄麻耐盐表型研究
耐盐性是许多作物的一种由多基因控制的数量遗传特性[15].精确的表型是qtl定位的重要组成部分[24,25].因此,我们测定了在两种盐胁迫条件下耐盐品系和对照品系之间6天的相对种子发芽率,并进行了3次生物重复。所有F2:3在盐胁迫条件下,前两天的产量非常低。耐盐表型在前2 d呈偏态分布,第4 d基本符合正态分布。据此,我们选择第4天耐盐表型数据进行QTL定位。
黄麻耐盐性QTL定位
盐胁迫是一种主要的环境胁迫源,它限制植物的生长发育和作物的产量[26].作物耐盐性可以通过精确育种来提高,这需要鉴定耐盐相关基因或分子标记。然而,以往对黄麻耐盐性的研究大多只集中在形态、生理和蛋白质组学成分上[26].据我们所知,只有两项研究[9,27]通过转录组测序对黄麻耐盐性相关基因进行了研究,揭示了大量耐盐性的差异表达基因。然而,大量的差异表达基因使得很难确定与耐盐性相关的最佳候选基因进行实际应用。
因此,我们进行了qtl定位,在基因组的有限区间内定位候选基因。我们成功定位了16个耐盐相关qtl。有趣的是,在LG4中发现了3个明显的QTL,包括一个主要QTL和6个次要QTL。这些结果表明,该LG可能在黄麻抗逆性中起着至关重要的作用。3个显著qtl的加性效应均为负,表明性状值的增加是由J009等位基因决定的。
总的来说,这些结果强调了在野生黄麻植物中存在优秀的抗性基因。利用耐盐野生种质资源,有利于加快培育耐盐黄麻品种。在两种盐胁迫条件下均检测到主要QTL qJST-1,其中160 mM盐胁迫条件下的定位区间(16.9 ~ 21.6 cM)也包含在140 mM盐胁迫条件下的定位区间内。因此,这个位置可能是黄麻耐盐基因的温床。因此,我们计划在未来的工作中重点研究这一区域的基因,进一步鉴定耐盐候选基因。克隆这些基因在标记辅助选择和作物耐盐转基因育种中具有潜在的应用价值。
结论
我们使用GBS方法绘制了迄今为止最完整的黄麻高密度遗传图谱。此外,还首次在黄麻中进行了耐盐QTL定位。在4个碱基上鉴定出3个显著qtl和13个次要qtl。在2种盐胁迫条件下检测到主要QTL qJST-1,分别解释了11.81%和19.61%的表型变异。这些结果为黄麻萌发期耐盐基因的标记辅助选择和转基因育种提供了有用的资源。然而,由于黄麻参考基因组只是一个没有固定在染色体上的支架版本,这些qtl无法定位到染色体上的区间。因此,这些QTL区间所包含的基因无法被识别。此外,F2:3用于QTL作图。这比精细制图更适合QLT初步制图。未来,黄麻的重组自交系和高质量的参考基因组将被开发和构建,这对克隆这些耐盐基因并将其应用于黄麻耐盐育种具有重要价值。
方法
绘制种群和DNA提取
本研究中使用的两种母本植物都是野生的c . olitoriusL.种质资源J009和c . olitoriusL品种广凤长果(GFG)。它们分别来自尼泊尔和中国江西省广丰市。J009比GFG更耐盐。均保存在中国国家韧皮纤维种质中期资源库(位于湖南省长沙市)。以150株F2群体及其F2:3子代为研究对象,构建了盐胁迫下的高分辨率遗传图谱,并进行了qtl定位。收集150个F2个体和2个亲本的幼叶组织进行基因组DNA提取。使用DNeasy Plant Mini Kit (TIANGEN, Beijing, China)提取每个个体的总基因组DNA,并用于基因分型测序(GBS)文库制备以发现SNP。在1%琼脂糖凝胶上监测DNA降解和污染情况。使用nanoophotometer®分光光度计(IMPLEN, CA, USA)检查DNA纯度,使用Qubit®DNA Assay Kit和Qubit®2.0荧光计(Life Technologies, CA, USA)测量DNA浓度。
萌发期耐盐性评价
耐盐实验在光照培养箱中进行,盐浓度分别为140 mM和160 mM,光周期均为12 h - 12 h,温度为28±0.5℃/25±0.5℃,相对湿度为75%。定位群体的表面不育种子(150 F2:3用两张均匀的无菌滤纸将株系播种到干净的无菌培养皿中。每一行在两个培养皿上播种,每盘30粒种子。一盘作为盐胁迫条件,另一盘作为对照。盐胁迫诱导时,第一天分别在盐胁迫盘和对照盘上加入3ml NaCl溶液和3ml水,然后分别在相应的盘子上加入2ml NaCl溶液和2ml水,连续6天。每种盐胁迫条件均采用3个生物重复的随机完全设计。在为期6天的试验中,每天观察并记录种子萌发情况。当芽长大于3 mm时定义为发芽种子。根据以下公式计算STIG:
GBS库的构建和排序
GBS库根据标准GBS协议[28,29].用MseI和HaeIII酶切制备F2群体和亲本个体DNA,然后用条形码接头结扎进行个体标记。使用agcourt AMPure XP (Beckman)对不同条形码序列的限制性连接样品进行纯化。将纯化后的样品用Phusion Master Mix (NEB)万能引物和索引引物进行PCR扩增i5和i7完整序列。PCR产物用Agencourt AMPure XP (Beckman)纯化,聚合,然后在2%琼脂糖凝胶上运行。375-400 bp片段(包括指标和适配器)使用Qiagen凝胶提取试剂盒分离。然后使用Agencourt AMPure XP (Beckman)对这些片段产物进行纯化,并使用Illumina HiSeq™2500平台进行对端测序。
SNP呼叫和基因分型
每个样品的序列根据单独的条形码进行排序,原始数据按照Nie等人描述的标准经过一系列基于内部C脚本的质量控制程序处理。[30.]。布伦斯-惠勒校准仪[31]用于将每个样本的清洁reads与参考基因组进行比对(设置:mem -t 4 -k 32 -M -R)。使用SAMtools软件将对齐文件转换为BAM文件[32(设置:-bS -t)。如果多个读对显示相同的外部坐标,则只保留映射质量最高的读对。使用GATK对所有样本进行SNP调用[33软件,并用perl脚本进行过滤。
基因图谱构建
亲本多态性标记分为8种分离模式(ab × cd、ef × eg、hk × hk、lm × ll、nn × np、aa × bb、ab × cc、cc × ab)。显示明显偏析的标记(P< 0.001),完整性(< 75%),或含有异常碱基使用JoinMap 4.0进行过滤[34].采用aa × bb分离模式构建F2群体遗传图谱。采用JoinMap 4.0软件对各连锁组中的标记进行排序。利用Kosambi映射函数进行标记距离计算。log of odds (LOD)值设置为2.0-30。使用Perl脚本SVG绘制链接映射并构造用于评估的热图。
耐盐QTL定位
将7个连锁图谱上snp的基因分型数据与单株耐盐表型数据进行整合,进行QTL定位。QTL分析使用QTL Cartographer v2.5进行[35]基于复合区间映射法(CIM)。对于每个性状,LOD评分阈值由所有标记的1000-排列测试确定,显著性水平为PMapQTL < 0.05 [36].根据每个QTL的LOD峰值位置及超过评分阈值的周边区域,确定其映射区间。
数据和材料的可用性
在本研究过程中产生或分析的所有数据都包含在手稿和附加文件中。本研究使用的所有原始数据均已存入NCBI,登录号为PRJNA544984。
缩写
- CIM:
-
复合区间映射法
- GBS:
-
Genotyping-by测序
- LGs:
-
连杆组
- 聚合酶链反应:
-
聚合酶链反应
- QTL:
-
数量性状位点
- SNP:
-
单核苷酸多态性
- SSRs:
-
简单序列重复
- 斯蒂格:
-
种子萌发耐盐指数
参考文献
杨震,陆瑞,戴志,闫安,陈杰,白志,谢东,唐强,程超,徐艳。世界范围内植物遗传多样性与群体结构分析黄胸草。利用微卫星标记的种质资源。biotechnology Biotec Eq 2018:1-7。
Kundu A, Chakraborty A, Mandal NA, Das D, Karmakar PG, Singh NK, Sarkar D.黄麻组织纤维含量与粗皮纤维产量及其主要成分的QTL鉴定(黄栌花,锦葵花。).分子育种,2015;35(1):19。
Islam MS, Saito JA, Emdad EM, Ahmed B, Islam MM, Halim A, Hossen QMM, Hossain MZ, Ahmed R, Hossain MS.两种黄麻的比较基因组学和纤维生物成因的研究。《自然植物》2017;3:16223。
Islam MK, Alam I, Khanam MS, Siyoung L, Waghmode TR, Mooryong H. 9个黄麻品种铬的积累及耐铬特性(Corchorus spp。而且木槿。).植物组学。2014;7(5):392-402。
Dansi A, Adjatin A, Adoukonousagbadja H, Faladé V, Yedomonhan H, Odou D, Dossou B.贝宁共和国的传统叶菜及其使用。遗传资源与作物进化。2008;55(8):1239-56。
Zeghichi S, Kallithraka S, Simopoulos AP.墨鱼营养成分(Corchorus olitorius)和stamnagathi (Cichorium棘).世界营养学杂志,2003;91(91):1-21。
瓦兹尼MW, Islam AS, Taliaferro JM, Anwar N, Sathasivan K.黄麻的新型est (黄胸草。) cDNA文库。植物学报。2009;17(2):173-82。
黄麻的生物化学、药用和食用价值(囊状Corchorus L。而且C. olitorius;叶子:一篇评论。清华大学学报(自然科学版). 2013;2(11):35-44。
杨震,吕荣,戴志,闫安,唐强,程超,徐勇,杨伟,苏娟。耐盐和敏感Corchorus属植物盐胁迫应答机制的新转录组测序。基因型与基因,2017;8(9):226。
陶安,吴刚,齐娟,徐娟,方鹏,林玲,张玲,林萍。白黄麻株高高密度遗传图谱构建及qtl鉴定(囊状Corchorus L。)使用特异性位点扩增片段(SLAF)测序。Bmc基因组学,2017;18(1)):355。
朱镕基JK。植物的非生物胁迫信号及其响应。细胞。2016;167(2):313 - 24。
Al-Tamimi N, Brien C, Oakey H, Berger B, Saade S, Ho YS, Schmöckel SM, Tester M, Negrão S.水稻耐盐基因座的高通量无创表型分析。Nat Commun. 2016;7:13342。
Fedoroff NV, Battisti DS, Beachy RN, Cooper PJ, Fischhoff DA, Hodges CN, Knauf VC, Lobell D, Mazur BJ, Molden D.从根本上重新思考21世纪的农业。科学。2010;327(5967):833 - 4。
Diouf L,潘志,何sp,龚wf,贾玉华,Magwanga RO, Romy K, Or HR, Kirungu JN,杜霞。陆地棉苗期耐盐qtl的GBS基因分型构建与定位。中华分子化学杂志,2017;18(12):249。
罗敏,赵颖,张锐,邢娟,段敏,李娟,王楠,王伟,张松,陈忠。基于SNP标记的玉米成熟田间耐盐QTL定位。中国生物医学工程学报,2017;17(1):140。
佩蒂尔G, Do T, Vuong TD, Valliyodan B, Lee JD, Chaudhary J, Shannon JG, Nguyen HT。大豆耐盐基因辅助单倍型分析及高通量SNP标记的开发。科学报告。2016;6(199)。
黄麻RIL群体中大规模SNP标记的发现及连锁图谱的构建(Corchorus capsularis).Mol Breed. 2015;35(5): 1-10。
陈艳,张磊,齐娟,陈宏,陶安,徐娟,林玲,樊平。白黄麻遗传连锁图谱的构建(囊状Corchorus L。)使用SRAP、ISSR和RAPD标记。植物营养学报。2015;33(6):344 - 344。
Topdar N, Kundu A, Sinha MK, Sarkar D, Das M, Banerjee S, Kar CS, Satya P, Balyan HS, Mahapatra BS。黄麻皮纤维品质性状、产量及产量组成的完整遗传连锁图谱及QTL分析(黄胸草。).烟草学报。2013;47(3):129-37。
Das M, Banerjee S, Topdar N, Kundu A, Mir RR, Sarkar D, Sinha MK, Balyan HS, Gupta PK.黄麻纤维产量和纤维品质性状分子育种的QTL鉴定。Euphytica。2012;187(2):175 - 89。
张玲,刘霞,张玲,万霞,陶安,方萍,林萍,齐娟。黄麻株高QTL SSR标记构建遗传图谱(Corchorus capsularis).2016年作物j。
Sultana N, Khan H, Ashraf N, Sharkar MTK。黄麻种内连锁图谱的构建。中国植物科学,2006;5(5):758-62。
Haque S, Ashraf N, Begum S, Sarkar RH, Khan H.黄麻遗传图谱的构建(黄胸草。)基于RAPD标记。植物组织科学。2009;18(2):165-72。
石勇,高磊,吴震,张旭,王敏,张超,张峰,周勇,李震。水稻种子萌发期耐盐性的全基因组关联研究。中国生物医学工程学报,2017;17(1):92。
杨震,黄东,唐伟,郑勇,梁凯,Cutler AJ,吴伟。水稻耐寒性相关数量性状位点的高通量测序。公共科学学报,2013;8(7):e68433。
马宏,杨锐,宋丽,杨勇,王强,王志,任晨。黄麻盐胁迫响应的差异蛋白质组学分析(Corchorus capsularis&olitorius L。)幼苗根。中国机械工程学报。2015;47(2):385-96。
杨志,闫安,陆瑞,戴志,唐强,程超,徐勇,苏娟。盐度胁迫下两种栽培黄麻的转录组序列分析。公共科学学报,2017;12(10):e0185863。
Elshire RJ, Glaubitz JC, Sun Q, Poland JA, Kawamoto K, Buckler ES, Mitchell SE。一种用于高多样性物种的强大、简单的基因测序(GBS)方法。公共科学学报,2011;6(5):e19379。
Sonah H, Bastien M, Iquira E, Tardivel A, Légaré G, Boyle B, Normandeau É, Laroche J, Larose S, Jean M.一种改进的基因分型测序(GBS)方法,提高了SNP发现和基因分型的多功能性和效率。公共科学学报,2013;8(1):e54603。
聂华,闫霞,霍震,姜丽,陈鹏,刘辉,丁俊,杨峰。马尼拉蛤高密度遗传图谱的构建及数量性状位点的定位。科学通报2017;7(1):229。
Li H, Durbin R.快速准确的短读对齐与挖洞轮变换:牛津大学出版社;2009.
李华,Handsaker B, Wysoker A, Fennell T, ruj, Homer N, Marth G, Abecasis G, Durbin R.序列比对/映射格式与SAMtools。生物信息学。2009;25(16):2078 - 9。
Auwera GAVD, Carneiro MO, Hartl C, Poplin R, Angel GD, Levymoonshine A, Jordan T, Shakir K, Roazen D, Thibault J.从FastQ数据到高置信度变量调用:基因组分析工具包最佳实践流水线。《生物信息学》,2013;43(1110):11.10.11。
用一种新的计算机程序:连接图构建综合遗传连锁图。植物学报,2010;3(5):739-44。
王山。Windows QTL制图师2.5。2007.
Ooijen JWV, Maliepaard C. MapQTL (tm) 3.0版:用于计算遗传图谱上QTL位置的软件。序——序集理论及其应用。2000;501(12):2412-21。
致谢
我们衷心感谢Editage (www.editage.cn)进行英文编辑。
资金
国家自然科学基金(no . 31601351)、湖南省自然科学基金(no . 2017JJ3350)、中国农业科学院科技创新计划(no . caas - astp -2017- ibfc01和CAAS-XTCX2016016-2)、国家大麻产业技术体系(no . CARS-16-E01)和种质资源保护计划(no . 2016NWB044)资助。资助机构没有参与研究的设计、数据的收集、分析和解释,也没有参与手稿的撰写。
作者信息
从属关系
贡献
SJG和ZMY设计了这项研究。ZMY完成了大部分实验。ZGD, JGS, ZMY和JQC为植物材料的获取做出了贡献。ZMY, YXY, DWX, QT和CHD对数据进行分析。ZMY、CHC、YX、CL进行表型数据收集。ZMY和JGS撰写了手稿。所有作者都阅读并批准了最终的手稿。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格自然对出版的地图和机构从属关系中的管辖权主张保持中立。
权利和权限
开放获取本文根据创作共用属性4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),允许在任何媒介上不受限制地使用、分发和复制,前提是您对原作者和来源给予适当的赞扬,提供到创作共用许可证的链接,并注明是否进行了更改。创作共用公共领域奉献弃权书(http://creativecommons.org/publicdomain/zero/1.0/)除另有说明外,适用于本条所提供的资料。
关于本文
引用本文
杨,Z.,杨,Y.,戴,Z。et al。黄麻耐盐高分辨率遗传图谱的构建及数量性状位点的鉴定(Corchousspp)。BMC植物生物学19日,391(2019)。https://doi.org/10.1186/s12870-019-2004-7
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-019-2004-7
关键字
- 黄麻
- 连杆组
- 数量性状位点定位
- 盐耐受性
- 基因分型-测序(GBS)