摘要
背景
增加小麦(小麦为了养活不断增长的人口,粮食生产是必需的。为了完成这项任务,深入了解栽培小麦的遗传结构和检测与重要农艺性状调控显著相关的基因组区域是必要的步骤。为了更好地了解适应性和产量相关性状的遗传基础和关系,我们使用了102个阿根廷六倍体小麦品种,在3个不同的地方生长2 - 6年,用35k snp阵列进行基因分型。基于单核苷酸多态性(SNPs)和基因相关分子标记,我们对该种质进行了单倍型区组鉴定和全基因组关联研究(GWAS)。
结果
遗传结构显示了4个亚居群,反映了阿根廷主要育种项目所使用的种质的起源。单倍型分析显示,基因组中分布着1268个大小不同的区段,其中包括高度保守的1BS染色体臂,即1BL/1RS小麦/黑麦易位的位置。基于GWAS,我们鉴定出97个与抽穗期、株高、千粒重、穗粒数和收获结实效率相关的染色体区域。特别是FEh是提高阿根廷小麦产量潜力的一个有希望的性状;我们检测到15个单倍型/标记与增加的FEh值相关,其中11个在所有三个评估位点显示显著影响。在适应的情况下Ppd-D1基因被巩固为阿根廷小麦品种生命周期的主要决定因素。
结论
利用一系列与Ref Seq v1.0同源的分子标记,揭示了阿根廷六倍体小麦种质资源的遗传结构。此外,GWAS检测与重要产量和适应组分相关的染色体区域(单倍型),这将允许通过标记辅助选择改进这些性状。
背景
世界人口预计到2050年将增长到近100亿,到2100年将超过110亿。在全球范围内,农业扩张已经放缓,产量增长主要是通过农业集约化实现的[1].满足日益增长的饲料和粮食需求将主要来自提高产量:未来几十年,小麦(和其他主粮作物)产量将至少提高50% [2],这将取决于在其他因素中提高产量潜力[3.].如果我们考虑到全球平均气温每升高1摄氏度,就会平均减少全球小麦产量,特别是阿根廷小麦产量6.0%,这种情况就会变得更加复杂[4,5].此外,未来的作物管理需要在环境上更加可持续[1].在这种限制性背景下,到2050年,产量遗传增益需要每年增加1.16-1.31%,以满足粮食、饲料和生物燃料对谷物的预期需求[6].不幸的是,在过去的几十年里,不同国家报告的小麦基因收益的增长似乎低于所需[7- - - - - -9].
在阿根廷,2012-2016年小麦平均种植面积为4.46公顷,是仅次于大豆和玉米的第三大重要作物[10],涵盖了广泛的环境。自1999年以来,地方品种的遗传增益表现出停滞的迹象,遗传增益值仅为0.18% /年,主要原因是单位面积粒数趋于稳定,而粒重没有变化。现代品种在单位面积穗数不变的情况下增加了每穗粒数;结果效率(又称穗肥力指数)是能更好地解释单位面积粒数变化的性状,它的改善可能是促进粒数增加而不影响粒重的一种方法[11- - - - - -13].
数量性状位点(QTL)定位是目前了解复杂性状遗传基础的关键[14],包括产量组成和适应性。目前,用于基因研究的具有成本效益的高通量基因分型工具,如单核苷酸多态性(SNP)阵列,可用于所有主要作物物种,包括小麦[15],从而能够表征(i)全基因组群体多样性和结构;(ii)选择性扫瞄和方向选择;和(iii)全基因组关联研究(GWAS)中的标记-性状关系[16,17].然而,在后一种情况下,我们发现了一些局限性:(i) snp只提供双等位基因信息[18];(ii)关联可能是由于连锁失衡(LD)位点与基因或控制序列,而不是潜在的因果原因[19].为了克服双等位问题,提高候选区域的分辨率,可以采用单倍型分析,即组合来自某一染色体区域内多态位点的共同遗传标记。以这种方式探索单倍型可以充分利用古老的重组事件,以相对高的分辨率识别潜在性状的遗传位点[20.].
最近发布的小麦参考基因组汇编(IWGSC Ref. Seq. Seq.)v1.0),由国际小麦基因组测序联盟(IWGSC)开发[21,可以更准确地比较来自独立研究的与农艺性状相关的染色体区域,并有助于加快克隆用于小麦育种的进一步重要基因。
本研究利用35k Axiom小麦育种者基因分型阵列对阿根廷102个小麦品种进行了基因分型[22].多态snp被物理锚定在小麦参考基因组装配[21],确定标本的遗传结构。在多环境试验数据的基础上,采用单倍型GWAS方法识别影响作物适应性和产量建设的染色体区域。适应性性状包括抽穗期(这是一个相关性状,因为它将决定作物在生殖阶段所处的环境)和株高(这是阿根廷品种中与倒伏联系最密切的性状)[23].与产量相关的性状包括千粒重和每穗粒数,目前称为“产量的数值成分”,是[24]来分析品种间产量的差异。此外,(25的研究中,将粒数(GN)作为两个变量的乘积:开花期穗干重(SDWA)和结实效率(FE,即穗育性指数,即GNS与SDWA的商),这两个变量目前被称为“生理生态成分”。后(26]提出用无粒(或谷壳)采收时穗干重来替代SDWA。然后,以每单位谷壳产生的粒数(FEh)计算出结果效率。在阿根廷,FEh已显示出与GN的高度关联[13,27- - - - - -29]和外国的[30.遗传力中等高的品种[12,28,29].GWAS为更好地了解面包小麦适应性的主要遗传成分和产量成分提供了有价值的信息,对旨在提高新品种产量潜力的育种计划具有重要意义,从而为粮食安全作出贡献。
结果
阿根廷小麦采集的单核苷酸多态性遗传结构
对102个阿根廷六倍体小麦品种进行基因分型,共获得7972个多态性snp位点,MAF≥10%。基于之前描述的5个双亲本群体的图谱信息,一组6486个多态snp被遗传锚定在特定的小麦染色体上[22].第二组909个额外的snp使用三个本地双亲本群体锚定在特定的染色体上,另外577个snp使用上述关联方法锚定(附加文件)1:表S1)。在所有的锚定snp中,A亚基因组上有3034个,B亚基因组上有4010个,D亚基因组上最少,有928个。为了揭示阿根廷102个六倍体小麦品种间的遗传关系,我们采用基于模型的方法,利用R STRUCTURE软件[31].基于模型的方法是一种不使用先验信息评估基因型之间遗传相似性的聚类分析。为了进行分析,我们使用了之前描述的固定在小麦基因组上的7972个snp。我们在收集的材料中发现了4个亚居群,这反映了阿根廷主要育种项目所使用的种质资源的起源2:表S2)。包括STRUCTURE Q矩阵的图形表示(附加文件3.:图S1)。程序1(表1)只包括法国起源的引进品种(100%),Nidera、Syngenta和Sursem育种公司在这一组中独家代表。亚居2包括大部分老品种(1930 - 1990年发布的12个品种中有9个),也称为传统种质[32,33以及2010年之前Don Mario育种公司推出的所有品种。亚群体3包括Klein育种公司发布的品种(占46.15%),ACA、Buck、INTA和Relmó育种公司发布的品种出现频率较低,以CIMMYT等品种为家系的品种居多。第4亚群体包括INTA(33.33%)、Buck、Klein、ACA和Relmó发布的品种,其中大部分也有包括CIMMYT种质在内的家系。
基于SNP和基因标记的阿根廷小麦品种单倍型块的构建与鉴定
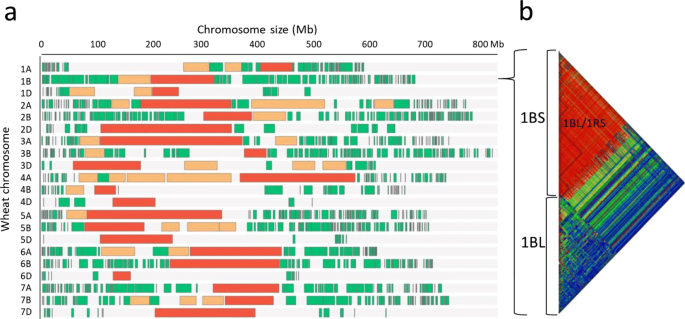
关于阿根廷小麦收藏单倍型地图构造的详细信息见表2.共构建了1268个单倍型块,其中A亚基因组518个,B亚基因组641个,D亚基因组109个。94%的SNP和基因标记与这些HB相关,只有495个SNP/基因相关标记不包含在任何HB中。D亚基因组染色体的平均块大小明显大于A或B亚基因组,观察到的平均块大小分别为13.5、6.7和4.8 Mb2).一般而言,在每条染色体中,最大的HBs (> 30 Mb)位于重组率较低的中心区域,而最小的HBs位于重组率和基因密度较高的端粒区域(图3)。1a). D亚基因组HBs之间的平均距离是B和a亚基因组的8倍,分别为16.1 Mb、2.7 Mb和2.4 Mb。D亚基因组片段之间的最大间隔也显著高于B和A,平均值分别为106.6、57.1和44.2 Mb (Table2).一个有趣的例子可以描述为染色体1b,我们在短臂上观察到几个大的HB,从hb9到hb20(该区域的HB平均大小为24 Mb)。一种可能的解释是存在的1BL/1RS小麦/黑麦转运,在阿根廷品种中保持高频率。易位中缺乏重组事件,产生了一个高度保守的区块,几乎涉及1B染色体的整个短臂(图1)。1b)(表2).
性状间的广义遗传力和相关性
所有评价性状均表现出较高的遗传力,作物适应性性状HD和PH广义遗传力值均高于0.95,产量相关性状FEh、TGW和GNS均高于0.80(见表3.).
在不同位置性状之间的相关性方面,FEh和GNS的模式最一致,在南部位置、Azul和Balcarce的大多数年份,以及在北部位置Marcos Juárez的所有年份都呈正相关4).除Azul 2014外,FEh与TGW在所有测试环境中均无显著相关性。
PH与TGW在Azul(1年)和Marcos Juárez各年份均呈显著正相关,且显著值较高。南方大部分年份PH值与GNS呈负相关,而Marcos Juárez在大部分年份PH值与GNS无显著相关。PH与FEh也可以描述类似的模式,南部地区的大多数年份PH与FEh呈负相关,而Marcos Juárez在所有年份均不存在显著相关4).
南部地区HD与GNS(2年)呈正相关,而Marcos Juárez(3年)呈负相关。HD与TGW在Azul(1年)和Marcos Juárez(2年)均呈负显著相关。HD与FEh在Azul(2年)呈显著正相关,与Marcos Juárez不同,大多数年份均无显著相关。在马科斯Juárez, HD与PH在南部地区呈显著相关,而在大多数年份均不显著相关。TGW与GNS的相关性在Marcos Juárez(3年)显示为正值,在大多数南方环境中没有显著相关性(表4).
对比观察到的南部和北部地区PH与GNS、PH与FEh、HD与GNS、HD与FEh的相关性,以及在较小程度上PH与TGW的相关性,可能表明构建特定地点产量成分表现涉及不同的策略:适应北纬地区的基因型易受高株提高TGW的影响,但受晚开花对TGW和GNS的影响,而适应南纬地区的基因型易受晚开花提高GNS和FEh的影响,但受高株降低FEh的影响。有趣的是,独立于环境,具有高FEh的基因型将有利于GNS,这是最重要的产量组成部分之一。
Haplotype-based gWAS分析
采收结实效率(FEh):
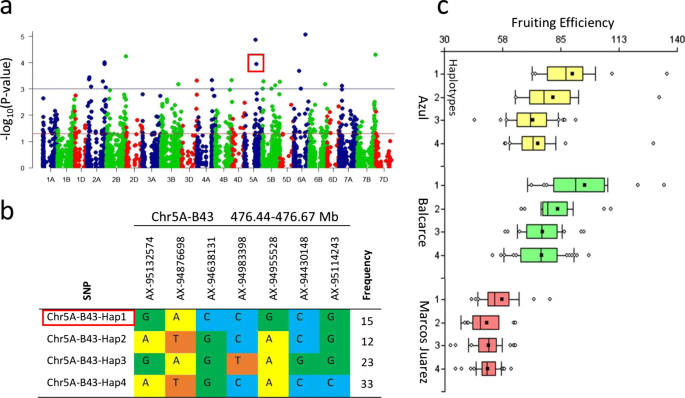
我们鉴定了17个与FEh相关的单倍型/标记,其中1个属于1A染色体,7个属于2A染色体,1个属于3B染色体,2个属于4A染色体,2个属于5A染色体,3个属于6A染色体,1个属于7A染色体(表)5;额外的文件6:表S5)。11个与FEh相关的单倍型在至少一年的时间内在三个地点的每一个都显著,即南部(Azul和Balcarce)和北部(Marcos Juárez)。5个单倍型仅在南部位点显著,只有位于2A染色体的SNP AX-94874921 (707.1Mb)仅在Marcos Juárez中显著相关。
一个在南部和北部与FEh显著相关的单倍型的例子是位于染色体5A (476.4-476.7 Mb)上的Chr5A-B43-Hap1。2a). HB Chr5A-B43由7个snp和显著相关的单倍型Hap1组成。102个评价品种中有16个检测到Chr5A-B43-Hap1(图;2b).在三个测试地点,Chr5A-B43-Hap1的存在显著增加了FEh,尽管强度不同,但在南部地点的影响更强(图1)。2c)。
一个Balcarce 2013中21条小麦染色体的收获结实效率(FEh)曼哈顿图。红线表示的GWAS阈值P< 0.05 =−日志10(P-value) = 1.3,蓝色线表示的是的GWAS阈值P< 0.001 =−日志10(P值)= 3。红色方框突出了在十个测试环境中的五个中与FEh相关的Chr5A-B43-Hap1。b基于位于5A染色体476.44 ~ 476.67Mb的7个SNP标记的单倍型区块命名为Chr5A-B43。四种不同的单倍型变异(Hap1-Hap4)在分析的人群中以不同的频率出现。红色矩形突出了与FEh相关的Chr5A-B43-Hap1。c箱线图显示了三个评估位置对应的四个不同单倍型组的表型值。Hap1在所有位点均与高FEh显著相关
与FEh有显著关联的3个单倍型与其他性状均有同源性:2A上的Chr2A-B49-Hap2 (704.8 ~ 705.8 Mb)与GNS同源,5A上的chr5a - b33 - ha4 (445.2 ~ 445.2 Mb)与PH同源,6A上的Chr6A-B24-Hap2 (205.1 ~ 233.3 Mb)与TGW同源。前两种单倍型的相关性仅在南部位点(Azul和Balcarce)显著,而第三种单倍型在所有位点均显著。Chr2A-B49-Hap2与GNS呈正相关,如表所示5单倍型高FEh的品种GNS也较高。以Chr5A-B33-Hap4为例,其与PH呈负相关5),单倍型FEh值较高的品种ph值较低。最后,Chr6A-B24-Hap2也呈负相关5):单倍型高FEh的品种TGW值较低。
千粒重(TGW):
我们鉴定了11个与TGW相关的单倍型/标记,其中1个属于染色体2D, 2个属于3A, 1个属于3B, 4个属于6A, 3个属于7B (Table5).位于3A染色体(686.8 Mb)的SNP AX-95257035在5个测试年份中的4个年份中仅在Marcos Juárez中与TGW显著相关。其余10个单倍型/标记与TGW在南部和北部的显著相关,一般来说,北部位点的显著性较高(表5).
检测到2个与TGW显著相关的单倍型,并与其他性状定位。3B染色体上的Chr3B-B111-246型Hap4 (817.4-817.8 Mb)与PH位点同源,6A染色体上的Chr6A-B24-247型Hap2 (205.1-233.3Mb)与FEh位点同源。以Chr3B-B111-Hap4为例,其与PH呈正相关,高TGW单倍型的品种也表达较高的PH(表)5).
每穗粒数:
我们鉴定出了两个与GNS相关的单倍型,一个属于染色体2A,另一个属于染色体4A (Table5).Chr2A-B49-Hap2在所有位点均显著相关,Chr4A-B19-Hap5在Azul和Marcos Juárez中显著相关,但在Balcarce中没有显著影响。与FEh定位的单倍型Chr2A-B49-Hap2及其效应先前已被描述(表5).
标题日期(HD):
16个单倍型/标记与HD呈显著相关。区域分布如下:1B上3个,2B上2个,2D上1个,3A上4个,3D上1个,5B上1个,6A上2个,6B上1个,7A上1个。对于这一特征,强调功能标记的一致效果对于Ppd-D12号染色体上的基因,在所有评估年份中,在三个测试地点显示了显著的相关性。Marcos Juárez与纬度5的相关性最高∘低于南部地区(Azul和Balcarce)。品种携带Ppd-D1敏感等位基因(102个供试品种中的42个)在所有环境下都延迟开花。值得一提的是,所有影响HD的单倍型在两年或两年以上的时间里都在北方位置显示出显著的相关性,与此相反,南方位置Azul显示出3个单倍型对HD没有显著影响,Balcarce显示出9个单倍型在相同的情况下。此外,我们发现了一个与PH定位的单倍型Chr6B-B26-Hap3 (157.6-157.8Mb),该单倍型增加了HD值,并对PH产生负面影响(表5).
株高(PH):
在单倍型GWAS中,有51个染色体区域与该性状相关。其中1个位于1A染色体,3个位于1B染色体,3个位于1D染色体,2个位于2A染色体,8个位于2B染色体,3个位于3A染色体,5个位于3B染色体,1个位于4A染色体,2个位于4B染色体,4个位于5A染色体,3个位于5B染色体,6个位于6A染色体,7个位于6B染色体,1个位于7A染色体,2个位于7B染色体。46个单倍型/标记在所有位点均显示显著相关,包括单倍型Chr1B-B17-Hap4和6B染色体上的SNP x -94943227,在所有检测位点和年份均显示一致的显著影响。3个单倍型与PH显著相关,并与FEh、TGW和HD性状定位,其贡献先前描述(表5).
讨论
遗传结构和单倍型鉴定
在这项工作中,基于模型的聚类分析的结果区分了四个亚种群,而以前[32],利用相同的102个品种和少量与农艺感兴趣基因相关的标记和中性标记(SSRs和isbp),检测到3个亚群体,尽管如此,正如预期的那样,这与本研究中确定的4个重叠。总的来说,本研究的亚居群1和亚居群2与前人研究的亚居群1和亚居群3相匹配,分别包括欧洲起源和传统种质的引种。我们研究中的第3和第4亚居与之前的研究中的第2亚居相匹配,在大多数情况下都包括了来自CIMMYT家系的种质。第3和第4亚居之间的差异可能与存在1BL/1RS小麦-黑麦易位有关,第3亚居中有24/26个品种携带这种易位,而第4亚居中没有品种具有类似的情况(数据未显示)。这种易位已被广泛用于育种,以获得对几种病原体和昆虫的抗性,扩大适应性和增加产量[34].阿根廷也不例外,携带1BL/1RS的现代品种已经表明:Klein Gladiador, Klein Nutria, Klein Yarará, LE 2333, LE 2341和ACA 906,在2009 - 2010年发布,证实了易位对生物和非生物抗逆性的持续贡献。尽管有这些优点,但由于1BL/1RS对面筋强度和面包制作质量的不利影响,应特别注意其使用,尽管它们因遗传背景而异[35].
小麦自驯化以来,一直处于激烈的人工和自然选择中,优良种质中观察到大量HBs [18,36].这一问题可以在我们的工作中观察到,在我们刚刚提到的1BL/1RS小麦-黑麦易位的存在(图。1b).无论如何,我们还必须考虑HB在染色体中的位置,如图所示。1a,大的单倍型(> 30 Mb)形成于每条染色体的着丝点和着丝点周围区域。如以前在[37],当在该区域引入感兴趣的等位变异并减少HBs的大小时,这些区域可能是一个挑战。
GWAS分析
产量潜在相关性状:
结果效率被认为是提高小麦产量潜力的一个有希望的性状[12,13,28,38,39].我们检测到15个显著的单倍型/标记对FEh有正影响,只有2个阴性(表5).FEh最强的遗传关联在南部的Azul和Balcarce位点被检测到,而在Marcos Juárez位点检测到较低的关联。这些结果表明,在产量潜力高的地区,如我们研究中的南部地区,FEh可以提供一种提高产量的途径。
在之前的研究中,FEh与粒数呈显著正相关[13,39].在这里,我们在2A染色体上检测到一个Chr2A-B49-Hap2 (704.8-705.8Mb)单倍型,该单倍型同时正向修饰FEh和GNS性状(表5).在进一步研究中,[40]检测到一个与FE和每小穗粒数相关的SNP (RFL_Contig3780_64),位于2A染色体676.24 Mb。他们提出CONSTANS 4 (CO4)和TaVrs1基因作为解释检测到的变异的候选基因。然而,基于IWGSC的Ref Seq。V1.0, CO4位于594.58 Mb (RFL_Contig3780_64近端81.66 Mb)。我们的单倍型Chr2A-B49-Hap2位于RFL_Contig3780_64远端28.56 Mb。Chr2A-B49-Hap2与CO4之间的物理距离估计为110.22 Mb,考虑到连锁不平衡(LD)衰减的临界值为0.1,估计为小麦基因组的平均50 Mb [41],将弃用CO4作为在Chr2A-B49-Hap2位点检测到的FEh QTL的候选。另一方面,基因TaVrs1,也叫GNI-A1,最近被克隆并以[42];不幸的是,GNI-A1该基因未组装在IWGSC Ref Seq上。V1.0染色体2A及其位置不能与Chr2A-B49-Hap2单倍型进行比较。
相反,FEh与TGW呈负相关[28,39].在目前的工作中,我们检测到单倍型Chr6A-B24-Hap2 (205.1-233.3Mb),增加FEh值,但惩罚TGW。
我们在5A染色体上检测到一个单倍型Chr5A-B33-Hap4 (445.2 Mb),该单倍型与FEh和ph均相关,但方向相反。
决定产量潜力的另一个相关性状是粒重。我们检测到6个显著的正效应单倍型/标记,5个显著的负效应单倍型/标记5).该列表包括TGW相关的6A染色体上的4个单倍型,其中一个为Chr6A-B24-Hap2 (205.1-233.3Mb)。该单倍型与TGW呈负相关,与FEh呈正相关,位于该基因近端4.5Mb处TaGW2-A1,位于6A染色体237.75Mb,已被证明与粒重有关[43].这种基因的非功能性突变会增加小麦的重量和大小[44].共享功能和物理位置将转向TaGW2-A1转化为Chr6A-B24-Hap2的候选基因。另一方面,基因TaGS5-3A位于3A染色体176.55Mb的基因也与较大的粒大小和较高的千粒重有关[45].我们在3A染色体上发现了一个与TGW相关的单倍型和一个SNP (Table5),但它们的位置太远了TaGS5-3A(分别为123和510Mb)被认为是候选基因。与FEh相反的是,在北部位置Marcos Juárez发现了最强的TGW遗传关联,促进TGW作为一个有趣的性状在这些纬度地区增加产量。
畿尼,46]利用GWAS检测到2A染色体691.22Mb的一个区域,该区域显著影响穗粒数。我们检测到位于704.8-705.8 Mb的Chr2A-B49-Hap2单倍型,它正向影响GNS,距离[46)地区。
作物适应相关性状:
对于HD,我们发现Ppd-D1基因标记(47,尤其是在我们研究中纬度最低的Marcos Juárez地区。这些结果与[33),Ppd-D1发现基因是阿根廷小麦品种生命周期的主要决定因素。
在1B染色体上,我们检测到3个与HD相关的单倍型。[48提出了基因TaFT-B3(581.4Mb)作为该染色体上短天的HD修饰符。我们最近的有效单倍型Chr1B-B76-Hap2 (Table5),位于61.7 Mb远端TaFT-B3,比估计为50 Mb的LD衰减的临界值0.1略远[41].
另一方面,[49显示功能丧失突变体PHYTOCLOCK 1 (WPCL1)基因位于3A染色体740.1Mb,与极早开花时间有关。我们在HD相关的3A染色体上检测到4个单倍型。我们最近的有效单倍型Chr3A-B-Hap4 (Table5),位于邻近39.5MbWPCL1将WPCL1定位为Chr3A-B-Hap4 HD联盟的候选。
最后,没有检测到HD与基因的显著关联Vrn-A1(5) [50),Vrn-B1(5 b),Vrn-D1(5 d) [51),Ppd-B1(2 b) [52],表明这组基因在我们的品种采集中似乎不影响抽穗时间。最可能的解释是,可能来自于采集的田间条件,满足春化要求,或春化基因之间潜在的高互作,导致难以检测对开花时间的微小影响。
在PH的情况下,我们没有发现该性状和“绿色革命”侏儒症基因的分子标记之间的显著关联Rht-B1而且Rht-D1,由[53].尽管采集到的小麦样品PH值变化较大,但需要指出的是,本研究所采用的小麦样品主要由半矮秆优良种质组成Rht-B1而且Rht-D1基因是平衡的。
在检测到的51个显著关联中,1B染色体单倍型Chr1B-B17-Hap4和6B染色体SNP AX-94943227 (234.8 Mb)尤其值得注意,因为它们在所有分析年份的三个位置都具有显著性。据我们所知,在这些染色体上没有描述与植物高度相关的基因,这表明这些一致的PH-HB/SNP关联是未来基因克隆项目有希望的目标。
我们在5A染色体上检测到4个与ph相关的单倍型。54]定位的ga响应Rht基因Rht9而且Rht12在5AL号染色体臂上Rht9与SSR barc151 (558.34 Mb)和Rht12定位于SSR Xgwm291 (698.19 Mb) 5.4cM处。我们的单倍型Chr5A-B54-Hap3映射在516.1-527.9 Mb可能与Rht9SSR barc151在558.34Mb上连接。然而,这样的联系Rht12在我们的研究中必须丢弃,因为额外的PH单倍型被放置在与该PH基因相距较远的遗传区域。
在6A染色体上,我们检测到6个与PH相关的单倍型;特别是,在三个地点和所有评价年份(Balcarce 2015年除外),单倍型Chr6A-B54-Hap5 (610.0 Mb)与PH显著相关。然而,相关的单倍型均未位于靠近6A上任何已知PH基因的物理位置,即Rht25(144.0-148.3 Mb)被[55),Rht18 / Rht14 / Rht24(413.73 Mb) [56].
需要强调的是,小型关联面板已被证明会增加1型和2型错误率,无法检测出真正的关联,同时也会产生更高的假阳性关联[57].在这项研究中,我们使用了一个小的关联面板(102个品种),但我们在GWAS中使用了一个保守的方法,以减少假关联。然而,我们可能无法检测到表型值解释率低或在面板中发现频率低的基因组区域。建议在应用于小麦育种计划之前,对独立群体中报告的相关性进行验证。
结论
利用SNP标记确定阿根廷六倍体小麦种质资源的遗传结构,发现获得的亚群体与阿根廷主要育种项目使用的种质来源之间存在较强的相关性。基于SNP和物理锚定在IWGSC Ref Seq v1.0上的基因相关标记,构建了单倍型图,可以检测到高度保守和选择的区域,如1BL/1RS易位。GWAS检测到97个与产量组成和适应性相关的染色体区域。在产量组成方面,我们突出了1A、2A、3B、4A、5A、6A和7A染色体上与FEh相关的区域,特别是在产量潜力较高的位置。在适应方面,HD最相关的效果是Ppd-D1该基因是阿根廷小麦生命周期变异的主要决定因素。使用IWGSC Ref Seq v1.0使我们能够精确地比较所有检测到的与先前研究中报道的基因和qtl相关的区域。
方法
植物材料
先前描述的[32一组102个面包小麦品种,包括老的(如cv。选用1927年发布的Barletta 77)和最近(截至2010年)的阿根廷主要小麦育种公司的商品品种,进行单倍型区段构建和GWAS分析。种子库由国家研究所Tecnología Agropecuaria (INTA)马科斯实验站Juárez、小麦种质资源库(马科斯Juárez,阿根廷)提供。
基因型的数据
该群体在阿根廷拉普拉塔的Genética兽医研究所(IGEVET)基因分型实验室使用35k Axiom小麦育种者基因分型阵列进行基因分型[22].关于35k snp Axiom阵列的信息在CerealsDB中公开脚注1.等位基因频率(MAF) <0.10和/或缺失数据超过10%的snp被丢弃。根据之前描述的地图信息,这些多态snp被基因锚定在特定的小麦染色体上[22].第二步,利用来自三个双亲本定位群体的连锁信息将新标记固定到小麦染色体上。此外,通过将SNP核苷酸信息转化为数字格式,并将该信息作为关联研究的表型值,几个SNP被锚定。与关联的snpP≤0.0001的染色体分配给该染色体(附加文件1:表S1)。将每个SNP标记的侧翼序列与IWGSC Ref Seq v1.0基因组组合进行blast比对,确定其在每个小麦染色体中的物理位置[21].先前描述的与农艺性状相关的标记分子数据[32]也包括在内。在适应性状的情况下,对基因进行标记Rht-B1, Rht-D1[53),Vrn-A1[50),Vrn-B1, Vrn-D1[51),Elf3-D1[58),Ppd-B1[52),Ppd-D1[47].病害方面,叶锈病抗性基因标记Lr10[59),Lr34[60),Lr24[61)合并。此外,工业质量相关标志,Ppo-A1, Ppo-D1[62),R-A1、R-B1 R-D1[63),Glu-B1al[64),Wx-A1, Wx-B1[65),Vp1-B3[66),Glu-A3[67),PinA-D1而且PinB-D1[68)被包括在内。最后,高分子量谷蛋白亚基(HMW-GS)基因座Glu-A1,Glu-B1而且Glu-D1的特征是根据[69].
SNP-based单体型结构
利用Haploview 4.2软件包对小麦每条染色体的单倍型结构进行评估[70].该软件包定义了单倍型块(HB),并提供了每个块的单倍型数量及其物理长度(bp),以及基于连锁不平衡的实体脊柱(LD)(如果D’> 0.8则为extended spine)标记的snp数量。这意味着一个区块中的第一个和最后一个标记是强LD,中间标记之间不一定是相互LD [18,70].Haploview中的HBs通过使用GitHub上的内部python脚本转换为一个表脚注2.该脚本将表格文件结果转换为“基因组关联与预测集成工具”(GAPIT)软件的合适输入[71].关于构造的单倍型地图的详细信息在附加文件中给出4:表S3。
田间试验与表型性状鉴定
在马科斯Juárez, 2011年至2016年(6年)在INTA马科斯试验站Juárez (32∘42”62年代,∘07”114 W, m.a.s.l)。在Azul, 2013年至2014年期间,在布宜诺斯艾利斯市国立中央省大学农学院的试验田进行了实地实验(36∘48”年代;59∘51”W, 137 m a.s.l)。在Balcarce, 2013年、2014年和2015年在INTA Balcarce试验站(37∘45”年代;55∘18”W;130 a.s.l)。除Marcos Juárez 2011和Balcarce 2013外,所有实验均采用两个重复的随机完全块设计(RCBD)。表中描述了GWAS进行的更详细的现场试验信息6.利用抽穗期(HD)和株高(PH)两个作物适应性性状和3个产量组成相关性状:收获结实效率(即穗肥力指数;每穗粒数(GNS)和千粒重(TGW)。HD(以天为单位)是测量从羽化到穗状穗从旗叶中脱出50%的时间[72].成熟期PH(单位cm)取随机选取的10 ~ 20株(试验),除芒外,从地面至穗顶测量每株主分蘖的平均值。成熟期随机取样5 ~ 15个刺突(根据试验)。它们在最小的小穗层被割下,称重,脱粒。以脱粒前穗部总干重与籽粒总重之差为单位计算穗部谷壳干重(g)。收穗时FEh(以籽粒g-1为单位)按[26].GNS测量为所选穗粒总数的平均值。重1000粒测定TGW (g)。MsJz采用phenbook软件收集表型数据[73].所有用于GWAS的表型数据在附加文件中给出5:表S4。
统计分析
获得了各环境下性状的最佳线性无偏预测因子(除Marcos Juárez 2011和Balcarce 2013,未进行实验重复),并用于Pearson相关分析和广义遗传力估计。广义遗传力(H2),利用该公式进行估算
在哪里\ \(σ_ {G} ^ {2} \)为基因型方差,\ \(σ_ {E} ^ {2} \)残差方差是,和r是环境的数量。误差方差采用环境方差对应的基因型左(\ \(\σ_ {e} ^{2} = \σ_ {\ mathit {GxE}} ^ {2} \) \).
Haplotype-based GWAS
使用4516个单倍型(来自1268个HB)和495个固定在小麦基因组上的信息性SNP/基因标记以及SUPER方法[74在R包GAPIT中实现[71].为了减少伪关联,种群水平的遗传结构(Q值)使用R structure软件进行评估[31].用于GWAS的R脚本可以在GitHub上找到脚注3.从每个性状的GWAS中,我们选择了显著的单倍型/标记(P<0.05,标记方面),至少有一个环境具有高度显著差异(P< 0.001)。表中列出了所有满足这些标准的单倍型/标记5.根据[所述公式]75]时,4个GWAS的单倍型/标记偶然同时显著的概率估计小于1.25E-07(0.05x0.05x0.05x0.001)。根据这个数字,估计5011个单倍型/ snp中至少有一个错误的概率为1 - (1 - 1.25E-07)5011= 0.00063(每个性状)。
数据和材料的可用性
支持本文结论的数据集包含在本文中。
缩写
- \ \(σ_ {E} ^ {2} \):
-
误差方差
- \ \(σ_ {E} ^ {2} \):
-
剩余方差
- \ \(σ_ {G} ^ {2} \):
-
基因型差异
- \ \(σ_ {GxE} ^ {2} \):
-
环境变异的基因型
- 英国石油公司:
-
碱基对
- BLUP:
-
最好的线性无偏预测器
- 国际玉米和小麦改良中心:
-
墨西哥梅亚门托国际中心
- 切:
-
收获时的结果效率
- 畿尼:
-
每穗粒数
- GWAS:
-
全基因组关联研究
- H2:
-
广义遗传
- HB:
-
单体型块
- 高清:
-
标题日期
- 国际商标:
-
国家农业技术研究所
- ISBP:
-
插入实地多态性
- IWGSC:
-
国际小麦基因组测序联盟
- LD:
-
连锁不平衡
- 加:
-
轻微的等位基因频率
- m:
-
兆基
- PH值:
-
株高
- 接待员:
-
的环境中
- RCBD:
-
随机完全块设计
- SNP:
-
单核苷酸多态性
- 苏维埃社会主义共和国:
-
简单序列重复
- TGW:
-
千粒重
参考文献
- 1
Ramankutty N, Mehrabi Z, Waha K, Jarvis L, Kremen C, Herrero M, Rieseberg LH。全球农业土地利用趋势:对环境健康和粮食安全的影响。植物学报。2018;69:789 - 815。
- 2
Reynolds M, Foulkes MJ, Slafer GA, Berry P, Parry MA, Snape JW, Angus WJ。提高小麦产量潜力。J Exp Bot. 2009;60(7): 1899 - 918。
- 3.
Hall AJ, Richards RA。主要粮食作物产量潜力和限水产量遗传改良预测。大田作物研究,2013;143:18-33。
- 4
赵晨,刘波,朴松,王旭,罗贝尔,黄勇,黄梅,姚勇,巴苏,P Ciais,等。据四项独立估计,气温上升会导致全球主要作物减产。中国科学院学报。2017;114(35): 9326 - 31所示。
- 5
García GA, Miralles DJ, Serrago RA, Alzueta I, Huth N, Dreccer MF。阿根廷潘帕斯草原的温暖夜晚:模拟其对小麦和大麦的影响显示产量下降。阿格利司系统。2018;162:259 - 68。
- 6
育种的挑战:提高产量潜力。见:作物生理学(第二版)。爱思唯尔:2015。p . 397 - 421。https://doi.org/10.1016/b978-0-12-417104-6.00016-9.
- 7
刘志强,刘志强,刘志强,等。新州小麦品种1901 - 2014年最佳同步开花期的遗传收益分析。欧元J艾格龙,2018;98:1-13。
- 8
Peltonen-Sainio P, Jauhiainen L, Laurila IP。北欧条件下谷物产量趋势:产量潜力的变化及其实现。大田作物研究,2009;110(1): 85 - 90。
- 9
罗培思,刘志强,刘志强,刘志强,刘志强,刘志强,刘志强。春面包小麦三十年来的遗传增收及其相关性状的变化。作物科学。2012;52(3): 1123 - 31所示。
- 10
François O.与r. r导师Popul Genet U Grenoble-Alpes进行类结构群体遗传分析。2016:1-9。http://membrestimc.imag.fr/Olivier.Francois/tutoRstructure.pdf.2019年9月13日访问。
- 11
Lo Valvo PJ, Miralles DJ, Serrago RA。1918 - 2011年阿根廷面包小麦品种的遗传进展:生理和数值产量组成的变化《大田作物》2018;221:314-21。
- 12
王志强,张志强,张志强,等。高穗肥力指标的选择促进了小麦产量和稳定性的遗传进展。Euphytica。2018;214(7): 112。
- 13
刘志强,刘志强,刘志强,等。阿根廷小麦新品种的增产效应。作物科学。1998;38(5): 1203 - 9。
- 14
植物复杂性状的遗传结构。植物学报。2007;10(2): 156 - 61。https://doi.org/10.1016/j.pbi.2007.01.003.
- 15
黄欣,韩斌。作物植物的自然变异与全基因组关联研究。植物学报,2014;65:531-51。
- 16
王志强,刘志强,刘志强,Röder MS.植物Snp基因的鉴定。植物学报。2009;12(2): 211 - 7。
- 17
田飞,白普理PJ,布朗PJ,洪H,孙Q, Flint-Garcia S, Rocheford TR, McMullen MD, Holland JB, Buckler ES。玉米巢式关联定位群体叶片构型的全基因组关联研究。Nat麝猫。2011;43(2): 159 - 62。https://doi.org/10.1038/ng.746.
- 18
郝超,王勇,晁生,李涛,刘洪,王磊,张欣。iselect 9k snp分析揭示了多倍体诱导的革命性变化和强烈的人类选择导致的强单倍型块体。Sci众议员2017;7:41247。
- 19
刘志强,刘志强。基于gwas的特征分析方法的研究进展。工厂方法。2013;9(1): 29。
- 20.
L钱,Hickey LT, Stahl A, Werner CR, Hayes B,斯诺登RJ, Voss-Fels KP。探索和利用单倍型多样性提高作物产量稳定性。植物学报。2017;8:1534。
- 21
黄晓明,黄晓明,黄晓明,黄晓明,黄晓明,黄晓明等。利用全注释参考基因组改变小麦研究和育种的限制。科学。2018;361(6403): 7191。
- 22
Allen AM, Winfield MO, Burridge AJ, Downie RC, Benbow HR, Barker GL, Wilkinson PA, Coghill J, Waterfall C, Davassi A,等。适用于六倍体面包小麦(triticum aestivum)全球资源高通量snp基因分型的小麦育种家阵列的特征植物生物技术学报。2017;15(3): 390 - 401。
- 23
张志强,张志强,张志强,等。小麦倒伏性状的研究进展。草业科学。2019;70(2): 95 - 106。
- 24
刘志强,刘志强。植物产量性状研究。农业学报,1923;13:390 - 439。
- 25
费舍尔RA。小麦。不同环境下大田作物的生产力潜力:国际水稻研究所,菲律宾洛斯Baños;1984年,129 - 54页。
- 26
杨志强,李志强,李志强,等。小麦穗肥力的筛选方法研究。农业科学学报,2013;151(3): 322 - 30。
- 27
González F, Terrile II, Falcón M.穗育性和茎长持续时间是提高潜在粒数(和产量)的有希望性状:阿根廷现代小麦的变异。作物科学。2011;51(4): 1693 - 702。
- 28
黄志强,李志强,李志强,等。小麦品种和早期育种材料穗部育性的遗传变异。农业科学学报2016;154(1): 13-22。
- 29
李志强,李志强,李志强,李志强,等。小麦穗肥力:早期世代的遗传及其与穗产量组成的关系。植物育种。2015;134(3): 264 - 70。
- 30.
Fischer R. Wheat生理学:最新进展综述。作物草地科学。2011;62(2): 95 - 114。
- 31
François O.与r. r导师Popul Genet U Grenoble-Alpes进行类结构群体遗传分析。2016: 1 - 9。
- 32
王志强,王志强,王志强,等。阿根廷六倍体小麦种质资源的遗传结构研究。Genet Mol Biol, 2013;36(3): 391 - 9。
- 33
Gomez D, Vanzetti L, Helguera M, Lombardo L, Fraschina J, Miralles DJ。vrn-1、ppd-1基因及早熟性对阿根廷面包小麦品种抽穗时间的影响《大田作物研究》2014;158:73 - 81。
- 34
李志强,李志强,李志强,李志强,李志强等。小麦-黑麦“Amigo”和“Kavkaz”染色体1RS. 1al易位重组1RS基因与白粉病抗性基因的连锁图谱分析。基因组。2004;47(2): 292 - 8。https://doi.org/10.1139/g03-101.
- 35
Graybosch RA。不稳定结合:黑麦染色质转移到小麦的质量影响。中国粮食科学;2001;33(1): 3-16。https://doi.org/10.1006/jcrs.2000.0336.
- 36
张志刚,张志刚,张志刚,等。小麦种质资源库遗传多样性研究进展与展望。中国农业大学学报(自然科学版)。Sci众议员2018;8(1): 12527。
- 37
王晓明,刘文华,刘文华,刘文华,刘文华。基因组技术在作物改良中的应用。大自然。2017;543(7645): 346。
- 38
Slafer GA, Elia M, Savin R, García GA, Terrile II, Ferrante A, Miralles DJ, González FG。结果效率:进一步提高小麦产量的一个替代性状。粮食能源安全,2015;4(2): 92 - 109。
- 39
Terrile II, Miralles DJ, González FG。小麦(triticum aestivum l)结实效率:各性状对不同生长条件的响应及其与开花时穗干重和收获时粒重的关系。《大田作物》2017;201:86 - 96。
- 40
郭志刚,陈丹,陈晓燕,陈晓燕,陈晓燕,陈晓燕,陈晓燕,陈晓燕,Röder MS, gannal MW, Schnurbusch T. 54个性状的全基因组关联分析鉴定出了决定小麦小花育性的多个位点。新植醇。2017;214(1): 257 - 70。
- 41
Juliana RP, Singh P, Singh PK, Poland JA, Bergstrom GC, Huerta-Espino J, Bhavani S, Crossa J, Sorrells ME。小麦抗叶锈病、条锈病和褐斑病的全基因组关联图谱揭示了潜在的候选基因。Theor app Genet. 2018;131:1-18。
- 42
郭志刚,张志刚,张志勇,张志勇,张志勇,张志勇,等。通过同源框基因突变在小麦中释放小花育性。中国科学院学报,2019;116(11): 5182 - 7。
- 43
苏震,郝超,王磊,董勇,张旭。面包小麦中与粒重相关的tagw2功能标记的鉴定与构建Theor app Genet. 2011;122(1): 211 - 23所示。
- 44
Simmonds J, Scott P, Brinton J, Mestre TC, Bush M, Del Blanco A, Dubcovsky J, Uauy C. tagw2-a1剪接受体位点突变增加了四倍体和六倍体小麦千粒重。Theor app Genet. 2016;129(6): 1099 - 112。
- 45
马玲,李涛,郝超,王勇,陈鑫,张晓霞。Ta gs 5-3a基因在小麦大粒高产改良中的应用。植物生物技术学报。2016;14(5): 1269 - 80。
- 46
刘杰,徐智,范昕,周强,曹军,王峰,纪刚,杨玲,冯斌,王涛。中国小麦穗部相关性状的全基因组关联研究。植物学报,2018;9:1584。
- 47
比尔斯·J、特纳·A、格里菲思·S、斯内普·J·w、劳丽·达·迪。在光周期不敏感的小麦ppd-d1a突变体中,一个伪响应调节器被错误表达。Theor Appl Genet. 2007;115(5): 721 - 33所示。
- 48
刘志强,刘志强,刘志强,等。小麦小麦开花位点t3-b1 (taft3-b1)和eat1靶基因tatoe1-b1 (tatoe1-b1)对小麦短日光周期响应的影响。植物细胞环境。2017;40(11): 2678 - 90。
- 49
普通小麦中Mizuno N、Kinoshita M、Kinoshita S、Nishida H、Fujita M、Kato K、Murai K、Nasuda S三个同源植物时钟1基因的功能缺失突变与超早开花表型相关。PLoS ONE。2016;11(10): 0165618。
- 50
张志强,张志强,张志强,等。多倍体小麦vrn-1启动子区的等位变异。Theor app Genet. 2004;109(8): 1677 - 86。
- 51
Fu D, Szűcs P, Yan L, Helguera M, Skinner JS, Von Zitzewitz J, Hayes PM, Dubcovsky J.大麦和小麦vrn-1第一个内含子的大缺失与春季生长习性有关。Mol Genet Genomics. 2005;273(1): 54 - 65。
- 52
Díaz A,齐khali M, Turner AS, Isaac P, Laurie DA。光周期-b1和春化-a1基因的拷贝数变异与小麦开花时间的改变有关。PLoS ONE。2012;7(3): 33234。
- 53
Ellis M, Spielmeyer W, Gale K, Rebetzke G, Richards R. rht-b1b和rht-d1b矮化基因的“完美”标记。Theor Appl Genet. 2002;105(6): 1038 - 42。
- 54
王志强,李志强,李志强,等。小麦赤霉素矮化基因的分子定位研究。Theor Appl Genet. 2005;111(3): 423 - 30。
- 55
刘志强,刘志强,刘志强,刘志强,刘志强,刘志强,刘志强,刘志强,刘志强,刘志强,刘志强,等。小麦染色体臂6as上一个影响株高、抽穗时间和穗发育的基因座rht25的鉴定与鉴定。Theor app Genet. 2018;131(10): 2021 - 35。
- 56
Ford BA, Foo E, Sharwood R, Karafiatova M, Vrána J, MacMillan C, Nichols DS, Steuernagel B, Uauy C, Doležel J,等。小麦rht18半矮化现象与ga 2-oxidasea9表达增加和ga含量降低有关。植物杂志。2018;177(1): 168 - 80。
- 57
王辉,李志强,李志强,李志强,李志强。群体大小和不平衡数据集对大麦育种种质qtl全基因组关联定位的影响Theor app Genet, 2012;124(1): 111 - 24。
- 58
刘志强,刘文文,刘志强,等。小麦早熟性d1 (ep -d1)开花基因与亚端粒染色体缺失的关系。J Exp Bot. 2015;67(1): 287 - 99。
- 59
王晓燕,王晓燕,王晓燕,等。不同遗传背景下小麦叶锈病抗性基因lr10的分子标记检测方法。摩尔品种。1997;3(1): 65 - 74。
- 60
刘志强,刘志强,刘志强,等。小麦lr34/yr18耐慢锈基因区段的分子遗传分析。Theor Appl Genet. 2006;114(1): 21 - 30。
- 61
王志强,王志强,王志强,等。小麦叶锈病抗性基因lr24的分子标记鉴定。Theor Appl Genet. 1995;90(7 - 8): 982 - 90。
- 62
何欣,何珍,张玲,孙东,毛瑞霞,傅尔斯特,夏霞。小麦多酚氧化酶(ppo)基因2a和2d等位变异及其功能标记的构建。Theor Appl Genet. 2007;115(1): 47-58。
- 63
王志刚,张志强,张志强,等。小麦红粒颜色基因tamyb10的pcr标记研究。Theor app Genet. 2011;122(8): 1561 - 76。
- 64
B, Gale K, Ikea J, Juhasz A, Bedö Z, Tamas L, Gianibelli M.利用新型pcr和反相高效液相色谱技术揭示小麦中高表达bx7谷蛋白亚基(glutenin -b1al等位基因)的传播。Theor app Genet. 2004;109(7): 1525 - 35。
- 65
王志强,李志强,李志强,等。阿根廷面包小麦种质资源中蜡质基因的遗传变异。生物技术学报。2009;12(1): 4 - 5。
- 66
杨艳,赵霞,夏玲,陈霞,夏霞,于智,何铮,Röder M.小麦收获前耐发芽胎生-1 sts标记的开发与验证。Theor Appl Genet. 2007;115(7): 971 - 80。
- 67
张文华,张志刚,张志刚,等。小麦低分子量谷蛋白- 3基因的克隆与表达分析。l). Theor Appl Genet. 2004;108(7): 1409 - 19所示。
- 68
黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟,黄丽娟。植物化学学报1994;25(1): 43-57。
- 69
Pflüger L, D 'Ovidio R, Margiotta B, Pena R, Mujeeb-Kazi A, Lafiandra D.人工合成六倍体小麦D基因组相关高分子量和低分子量谷蛋白亚基的特征。Theor Appl Genet. 2001;103(8): 1293 - 301。
- 70
Barrett JC, Fry B, Maller J, Daly MJ。Haploview:分析和可视化的ld和单倍型地图。生物信息学。2004;21(2): 263 - 5。
- 71
张志强,王琪,李敏,张志强,张志强,张志强,张志强,张志强。基因组关联与预测的研究进展。生物信息学。2012;28(18): 2397 - 9。
- 72
张特勤,刘志刚,刘志刚。谷物生长阶段的十进制编码。杂草研究》1974年;14(6): 415 - 21所示。
- 73
Crescente JM, Guidobaldi F, Demichelis M, Formica MB, Helguera M, Vanzetti LS。表现型数据收集的开源软件。GigaScience。2017;6(4): 1 - 5。
- 74
王强,田芳,潘勇,Buckler ES,张震。一种超强大的全基因组关联研究方法。PLoS ONE。2014;9(9): 107684。
- 75
张军,张志强,张志强,张志强,等。不同水分处理对冬小麦产量和植株水分性状qtl的影响。Theor app Genet. 2018;131:1-19。
确认
作者希望感谢国际小麦基因组测序联盟(IWGSC)在发表前获得IWGSC RefSeq v1.0的资料。SMLB和JMC是阿根廷国家科学和技术理事会(CONICET)的研究员。IAR得到了孟山都公司比彻尔-博洛格奖学金项目的资助。作者感谢PICT-2012-0660项目负责人Viviana Echenique和Gabriela Tranquilli为基因分型提供的资金支持。作者感谢罗米娜·奥蕾莉亚、玛丽亚·多纳托、洛雷纳·科尔蒂佐、莫罗·马丁内斯和霍拉西奥·达拉·瓦莱的技术援助。
资金
这项工作得到了以下项目的支持:阿根廷国家农业技术研究所(INTA) PNBIO1131042;FONCYT皮克特人- 2012 - 0660;CONICET PIP 11420090100291;UNICEN 03 / A192;UNICEN 03 / A215;和CIC-PBAN∘673/06。IAR得到了孟山都的比彻尔-博洛格奖学金514项目的资助。
作者信息
从属关系
贡献
本研究由SMLB、IAR、PA、MH、WJR、AP、LV设计。SMLB、IAR、JC、LV和WJR进行了实验。SMLB、IAR、MD和LV收集表型数据。JC开发了计算机程序。SMLB, LV, MBC和JC进行数据分析。SMLB、LV、JC起草稿件。WJR, PA, MH, AP参与了手稿的改进。所有作者都认真阅读并通过了稿件。
相应的作者
道德声明
伦理认可和同意参与
不适用。
同意出版
不适用。
相互竞争的利益
IAR得到了孟山都公司比彻尔-博洛格奖学金项目的资助。作者声明他们之间没有利益冲突。
额外的信息
出版商的注意
施普林格《自然》对出版的地图和机构附属关系中的管辖权要求保持中立。
补充信息
额外的文件1
表S1.与IWGSC Ref Seq v1.0基因固定的多态snp和基因相关分子标记。基于102个阿根廷六倍体小麦
额外的文件2
表S2.阿根廷102个六倍体小麦品种的Q结构矩阵。
额外的文件3
图S1.阿根廷102个六倍体小麦品种Q结构矩阵的图解。
额外的文件4
表S3.阿根廷六倍体小麦采集中用于HB表征和GWAS的单倍型地图。
额外的文件5
表S4.Balcarce(2013-2015)、Azul(2013-2014)和Marcos Juárez(2011-2016)对GWAS中FEh、GNS、TGW、HD和PH的表型数据进行了分析。
额外的文件6
表S5.利用GWAS技术对102份阿根廷六倍体小麦样品进行FEh、TGW、GNS、PH和HD的相关单倍型(snp /marker ID)检测。
权利和权限
开放获取本文根据知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许不受限制地在任何媒体上使用、分发和复制,前提是您要适当地注明原作者和来源,提供创作共用许可的链接,并说明是否进行了更改。知识共享公共领域转让豁免书(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
Luján Basile, s.m., Ramírez, i.a., Crescente, J.M.et al。阿根廷六倍体小麦采集和GWAS产量组成及适应性的单倍型区块分析。BMC植物杂志19日,553(2019)。https://doi.org/10.1186/s12870-019-2015-4
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12870-019-2015-4
关键字
- 小麦
- 单体型
- GWAS
- 标题日期
- 株高
- 千粒重
- 每穗粒数
- 收获时的结果效率