摘要
背景
NAC转录因子包含5个高度保守的子结构域,这是蛋白质二聚化和DNA结合所必需的。在这些亚结构域中,很少有残基被确定为蛋白质功能所必需的,而且更少的残基被证明具有生物学相关性在足底。我们在小麦中使用了一种正向衰老调节剂,NAM-A1,以测试在特定的、高度保守的NAC结构域残基上错义突变对蛋白质功能的影响。
结果
我们发现了在NAC结构域的5个高度保守残基的错义突变NAM-A1在四倍体TILLING种群中。含有这些突变的TILLING系,与同义和非保守突变对照一起,在温室条件下生长,并对衰老进行评分。5个突变中有4个突变对花梗衰老表现出显著且一致的延迟,但对旗叶衰老没有一致的影响。在酵母双杂交实验中,所有四个具有延迟衰老表型的突变等位基因也失去了与同源基因NAM-B1相互作用的能力。这些残基中的两个先前被证明参与拟南芥NAC结构域功能,表明残基功能在物种之间具有守恒性。与野生型相比,这四个等位基因中的三个导致细胞死亡反应减弱NAM-A1当短暂的过度表达在烟草benthamiana。其中一个突变在田间条件下进行了进一步的测试,在该突变中,花梗和叶片的衰老都有显著和一致的延迟。
结论
我们将一系列突变等位基因的田间和温室研究与生化分析相结合,确定了NAC结构域所需的四个残基NAM-A1功能和蛋白质相互作用。我们表明,这些残基的突变导致表型梯度,提高了对农艺重要性状产生等位基因系列突变的可能性。我们也展示了突变NAM-A1与更普遍研究的旗叶衰老相比,更严重地影响花梗衰老,突出表明这是一个值得进一步研究的领域。这种综合方法的结果为NAC转录因子功能域内的保守残基具有生物学意义提供了强有力的证据在足底。
背景
NAC转录因子是植物特异性转录因子(TFs)的一个大家族。NACs调节广泛的生物过程,包括许多基本的发育过程,如侧根形成、茎尖分生组织发育和叶片衰老[1,2,3.,4,5]。它们还调节广泛的非生物和生物应激反应[6,7,8]。虽然许多早期的研究都是在模式物种中进行的,比如拟南芥近年来,越来越多的作物研究发现,NAC TFs是多种农艺相关性状的重要调控因子。例如,在小麦中,NAC TFs参与调节非生物和生物胁迫反应[9,10,11,12,13,14,15,16,17,18],以及与决定谷物品质有关的性状[5,19]。
NAC转录因子的特点是在蛋白质的n端有一个高度保守的NAC结构域,随后在c端有一个非保守的、内在无序的区域[3.,20.]。NAC结构域由5个子结构域(子结构域i至子结构域v)组成,它们本身在植物界高度保守,并具有关键的功能作用[20.,21,22]。对拟南芥的早期研究表明,NAC TFs以同二聚体或异二聚体的形式结合DNA,并且这种二聚体是稳定DNA结合所必需的[23,24]。以同型二聚体形式形成的NAC结构域的结晶将蛋白质二聚界面定位于子结构域i和ii [20.]。随后,结合DNA的NAC二聚体的晶体结构鉴定了位于DNA-蛋白质相互作用界面的亚结构域iii、iv和v中的残基[21]。
尽管它们很重要,但目前尚不清楚NAC结构域内的残基在不同的NAC转录因子中,甚至在不同的物种中,在多大程度上具有共同的功能。早期的研究表明,拟南芥ANAC019 NAC结构域iii亚结构域的特定残基突变导致体外DNA结合缺失(图2)。1) (24]。进一步的研究发现了ANAC019蛋白二聚化所必需的一对残基(图2)。1A,子域i) [24]。最近,ANAC019 NAC结构域内的各种残基被预测在NAC结构域(子结构域i,以绿色突出显示)的ph依赖性稳定中起作用[25]。然而,据我们所知,没有实验表明NAC结构域的其他残基是蛋白质二聚化所必需的,也没有表明上述残基具有重要的生物学作用在足底。
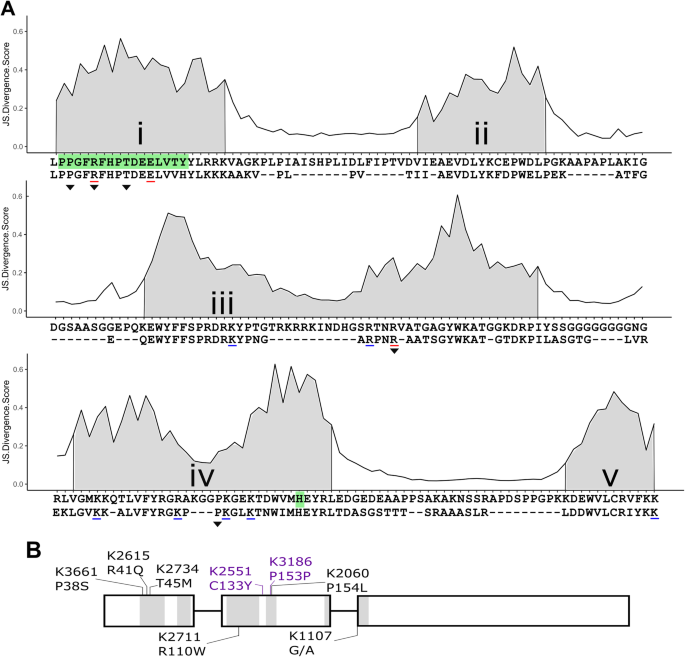
Kronos TILLING栽培群体中NAM-A1高度保守残基的鉴定。来自7种植物物种的含有NAC结构域的蛋白质的一致性排列的Jensen-Shannon Divergence评分(见方法)突出了5个NAC亚结构域的保守性,用灰色表示(一个电流-电压)。在NAC转录因子集合中少于10%的序列中存在的残基被排除在比对之外。先前的工作测试了特定残基在蛋白质结合(子结构域i)和DNA结合(子结构域iii至v)中的作用;相应工艺所需的残留物以红色下划线表示,不需要的残留物以蓝色下划线表示[24]。子结构域iv中的组氨酸残基通过与部分蛋白质二聚化结构域(子结构域i;均以绿色标示)[25]。从这种比对中获得的一致NAC结构域序列显示在守恒分数(第一行)下面。的排列nac结构域NAM-A1如下所示的是共识序列。在Kronos TILLING群体中发现的带有错义突变的残基用三角形(黑色)突出显示。注意,K2551中非保守残基(C133Y)的错义突变不存在于一致性比对中,因为它在NAC蛋白中不保守。错义突变,以及非保守和同义控制(紫色)和剪接受体变异K1107,显示在该基因的示意图上NAM-A1结构(b)
在小麦中,一个NAC转录因子(由NAM-B1)曾被证明对衰老起积极的调节作用[5]。在现代的六倍体(面包)和四倍体(面食)小麦中,这个基因要么没有功能,要么被删除了。5]。随后对六倍体小麦的研究表明,a基因组同源基因的截断突变体,NAM-A1,本身就足以显著延缓叶片衰老[26]。在某些测试环境中,NAM-A1的缺失也导致籽粒蛋白含量(GPC)降低,尽管这比D基因组截断或双A和D基因组突变的情况要轻。的截断变体NAM-A1也能显著延缓四倍体小麦的叶片衰老,这也对应于GPC的显著降低[27]。然而,目前尚不清楚这种影响会达到何种程度NAM-A1错义突变中的等位基因变异可能影响这些表型。此外,尽管这些研究报告花梗通常保持绿色康南1号变种人,没有人专门研究过NAM-A1(或其同源物)调节花梗衰老。
最近,一项基因组靶向诱导局部病变(Targeting Induced Local lesion in Genome)资源为四倍体和六倍体小麦的点突变变异提供了一个无与伦比的来源[28]。由于小麦的多倍体特性,在TILLING群体的生产中,可以使用比通常用于二倍体植株更高剂量的诱变化学物质甲烷磺酸乙酯。这导致了一组突变密集的细胞系,这些细胞系在外显子组捕获后被测序,现在可以很容易地通过EnsemblPlants进行查询[29]。当与最新的基因组参考进行比对时[30.], 99%被测序的小麦基因至少有一个错义突变,平均每个基因有30个错义等位基因。这提供了一个有用的资源在足底特定残基在蛋白质功能中的作用研究。
在这里,我们利用四倍体的硅TILLING资源来研究特定错义突变对基因的影响NAM-A1。在四倍体小麦中,大多数小麦基因以功能拷贝的形式存在于两个基因组上NAM-A1是功能性的。因此,我们可以研究特定突变对NAM-A1在二倍体环境中。我们在NAC结构域中发现了一组五种错义突变,预计它们是高度有害的[20.,24,25]。基于这些突变,我们描述了它们对旗叶和花梗衰老和细胞死亡的影响在足底。我们还评估了它们在体外阻止蛋白质结合的能力。我们确定了导致植物衰老延迟和蛋白质结合能力丧失的四个残基。我们认为这些残基的功能可能在物种之间是保守的,并且这些残基是必需的NAM-A1函数。
结果
TILLING种群在基因的高度保守残基中含有错义突变NAM-A1南京汽车领域
为了可视化NAC结构域在植物界的保守性,我们从植物界7个物种的1404个含有NAC结构域的蛋白质中获得了NAC结构域序列(小麦,普通小麦,水稻var。粳稻、小立霉、毛杨,玉米,拟南芥;额外的文件1)。我们使用了Jensen-Shannon散度(JSD)方法,正如Capra和Singh [31],以表征对齐的NAC结构域中每个残基的估计保存水平(图2)。1A).在整个对齐的NAC域中,我们获得了0.21的平均JSD值。然后,我们将我们的共识NAC结构域序列与前面描述的子结构域相关联[20.]。将NAC域分解为子域,我们发现五个子域中每个子域的平均JSD值都高于整个域的JSD值,从异质性较强的子域iii的0.24到高度保守的子域i的0.39不等(图2)。1A)。在一致性NAC结构域序列的比较中,子结构域的较高保守性也很明显(上序列,图2)。1A)与小麦NAC转录因子的NAC结构域序列对齐NAM-A1(底部序列,图2)1一个)。
基于上述高度保守的残基,我们选择了Kronos TILLING品种中存在的5个错义突变,这些突变预计会对蛋白质功能产生有害影响(图2)。1B,表1、附加文件2:表1)。Kronos TILLING系K3661、K2615和K2734中的三个突变位于子结构域i的预测蛋白二聚化界面。其余两个突变(系K2711和K2060)位于子结构域iii和iv的预测DNA结合界面。另外选择两个系作为对照。其中一个编码高度保守残基(K3186)的同源突变,另一个编码子结构域iii和子结构域iv之间的非保守残基(K2551)的错义突变。最后,K1107被鉴定为剪接受体突变,该突变导致包含子结构域v大部分的预测的11个残基缺失(附加文件)2:图S1)。这些突变中的两个先前在拟南芥中被证明在蛋白质二聚化(K2615, R41Q)和DNA结合(K2711, R110W)中发挥作用(表2)1) [24]。在本文的其余部分中,TILLING系将被称为TILLING系和相应的突变(例如K2711)R110W)。Line K1107包含一个剪接受体变体(SAV),将被称为K1107干腊肠。
TILLING突变导致延缓衰老
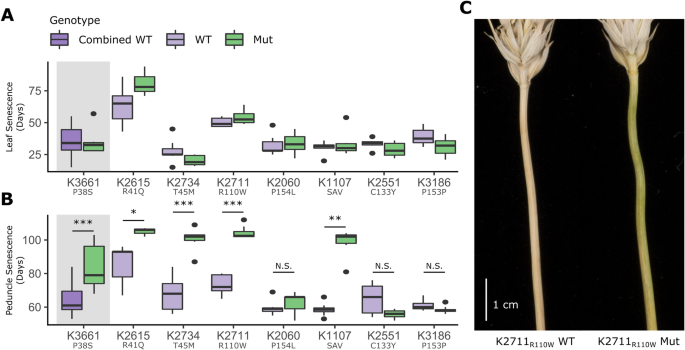
米5对所选TILLING系的代植株进行生长,并在温室条件下对旗叶和花梗的视觉衰老进行评分(图2)。2、附加文件2:图S2)。在两个独立的实验中,没有一个突变系在旗叶衰老方面表现出与野生型兄弟姐妹一致的延迟。实验1中有4个品系旗叶衰老发生明显延迟(双样本Wilcoxon检验);p< 0.05,附加文件2:图S2),然而,当在实验2中重复时,这种差异并没有重现(图S2)。2A)。试验2中,野生型和突变系的相对旗叶叶绿素含量在衰老过程中也无显著差异(附加文件2:图S3)。
然而,旗叶衰老并不一致地延迟NAM-A1突变体,这不是花梗衰老的情况。四行,包括三个错义,K2615R41Q, K2734T45M, K2711R110W剪接突变为K1107干腊肠结果显示,与野生型植株相比,突变植株的花梗衰老明显延迟(图2)。2B和C、附加文件2:图S2)。在实验2中,K2711R110W突变株抽穗后花梗衰老的平均时间为104天,而野生株为73天,显著差异为41天。同样,K2615的花梗衰老明显延迟了大约23、33和40天R41Q, K2734T45M和K1107干腊肠变种人,分别p< 0.05)。K2060P154L在花梗衰老方面表现出更微妙的延迟,尽管这仅在实验1中具有显著性(p < 0.05, 4天)。第四线,K3661p38的基因中含有一个纯合突变5因此,在相同的突变背景下,不能直接与野生型系进行比较。然而,当与所有野生型的数据相比时NAM-A1不同的等位基因,K3661p38花序梗衰老显著延迟,平均发生在抽穗后83天(p< 0.001)。K3186的同义突变P153P和K2551的非保守错义突变C133Y在两个实验中,与野生型兄弟姐妹相比,没有显著延迟花梗衰老。
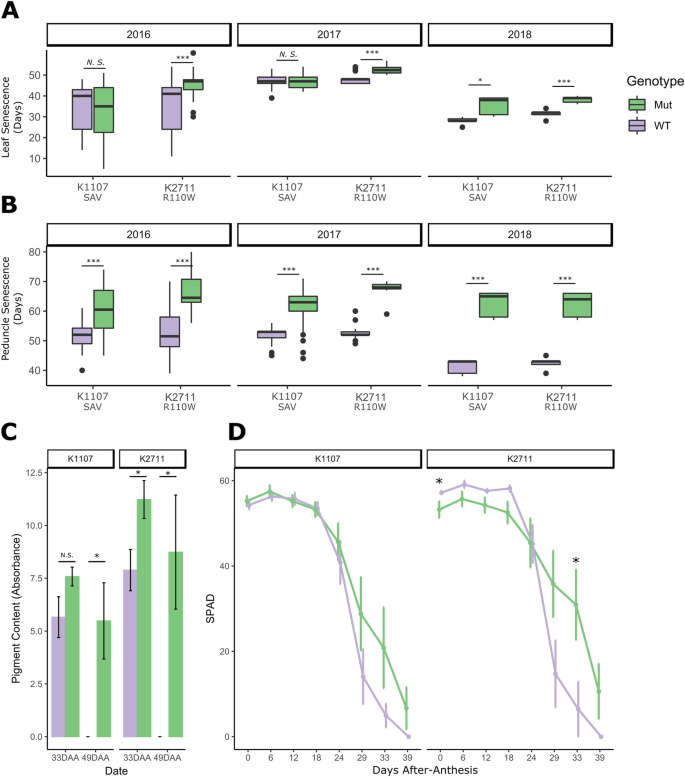
根据温室试验,我们选择了品系K2711R110W和K1107干腊肠用于现场条件下的测试。这些系与野生型亲本Kronos回交一次,以减少ems诱导的背景突变的数量。三年(2016年,2018年在英国,2017年在美国加利福尼亚州),K2711R110W一致显示旗叶和花梗衰老的显著延迟(图2)。3.A和B,学生t检验,p< 0.001)。这种效应比K1107强干腊肠(p < 0.001),但旗叶衰老仅在2018年(p< 0.05)。在2018年的田间试验中,在花后33天和49天(DAA)对花梗叶绿素含量进行了量化(图2)。3.C)。在33 DAA时,K2711的叶绿素水平显著升高R110W突变株比野生型多(学生t检验;p < 0.05)。到49 DAA时,两种野生型系的叶绿素A含量均降至零,而两种野生型系的叶绿素含量均显著升高NAM-A1突变系(K2711)R110W和K1107干腊肠,p< 0.05)。
在2018年的田间试验中,也使用SPAD测量对旗叶衰老进程进行了量化。从开花期到大约24 DAA,突变型和野生型植株之间没有显著差异;尽管K2711R110W野生型系在开花期含有更多的SPAD单位,这种差异在接下来的时间点没有保持(图2)。3.D)。在29 DAA时,两个突变系的突变体和野生型植株之间可以看到分离。然而,只有K2711R110W在33 DAA时,突变体旗叶SPAD单位与野生型差异显著(Kruskal-Wallis秩和检验,p < 0.05)。这与在K2711中观察到的更强的旗叶衰老表型一致R110W与K1107相比干腊肠。
TILLING突变导致蛋白质含量降低,但晶粒尺寸参数没有变化
颗粒样品取自K1107干腊肠和K2711R110W2016年和2018年(英国)种植的品系,并测量了晶粒尺寸参数。两种K2711在田间均未观察到对晶粒尺寸参数的一致影响R110W或K1107干腊肠(附加文件2:图S4)。这种缺乏晶粒尺寸效应与先前在六倍体小麦中观察到的NAM-A1截断等位基因一致[26]。
对K2711大田试验的同一组籽粒样品进行了籽粒蛋白质近红外(NIR)测定R110W和K1107干腊肠。2016年,K2711的突变植株R110W种群的籽粒蛋白质含量(GPC)显著低于野生型(p< 0.05,图。4)。的K1107干腊肠然而,突变植株的GPC没有持续降低。2018年,突变系中GPC的降低在K2711中是一致的R110W和K1107干腊肠种群GPC分别降低14.7%和11.5%,p < 0.05。4)。这些结果与先前在六倍体小麦上的研究一致,其中截断突变体在NAM-A1和NAM-D1导致油田中GPC含量的减少,尽管这在不同年份有所不同[26]。
蛋白质结合区域的突变阻止了异源二聚化
为了确定错义突变是如何导致衰老延迟的,我们描述了它们对蛋白质功能的影响。此前,对小麦NAM-B1蛋白进行了酵母双杂交(Y2H)筛选,确定了NAM-A1同源物作为推定的结合伙伴(未发表结果)。我们通过酵母双杂交和共免疫沉淀证实了这种相互作用,证明NAM-B1和NAM-A1可以在体内相互作用(图2)。5、附加文件2:图5)。然后,我们使用这种已知的相互作用来测试NAM-A1的保守残基中选择的突变是否(表1)1)导致蛋白质失去与NAM-B1的相互作用。
克隆了NAM-A1突变等位基因,在Kronos TILLING系中发现了相应的突变,详见表1。同样的残基也突变为丙氨酸残基,以测试任何表型是由于TILLING系的特定突变,还是由于保守残基本身的功能特性[32]。在Y2H中,只有NAM-A1等位基因的NAC结构域被用来阻止酵母GAL4启动子被c端转录激活结构域自动激活(附加文件)3.)。在Y2H实验中,NAC转录因子c端结构域引起的自激活已经被观察到[33],表明NAC转录因子具有转录激活因子的功能。去除c端结构域预计不会显著改变蛋白质的二聚化性质[25]。
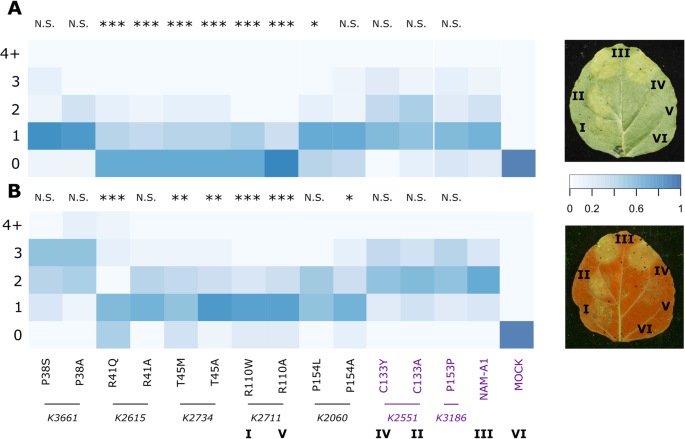
在选择性培养基上筛选相互作用发现,保守残基的5个错义突变中,有4个导致与TILLING和丙氨酸突变完全失去相互作用(图2)。5、附加文件2:图S6)。这包括预测蛋白结合域的三个突变等位基因(P38S, R41Q, t45m)和预测dna结合域的两个突变等位基因中的一个(R110W)。一个突变(P154L)仅部分降低了结合NAM-B1的能力,因为与对照相比,TILLING突变体的相互作用水平降低。然而,这种相互作用在丙氨酸突变中完全恢复。在选择条件下,所有的对照都保留了相互作用,包括K2551中非保守氨基酸C133Y的错义突变。
错义突变减少细胞死亡诱导
在Y2H屏幕之后,我们短暂地过度表达NAM-A1等位基因的烟草benthamiana以确定它们对细胞死亡反应的影响(见方法)。在白光下,细胞死于黄化,而在紫外光下,酚类化合物的积累导致了自身荧光。34]。野生型过度表达NAM-A1导致细胞死亡表型类似于在弱超敏反应期间观察到的,有证据表明黄化和自身荧光明显高于模拟(p< 0.001,图。6)。这与的作用是一致的NAM-A1在小麦中作为衰老的积极调节剂[5,26,27]。
的突变等位基因NAM-A1细胞死亡反应减少。突变体的异源表达NAM-A1等位基因的n benthamiana显示细胞死亡反应在两种情况下均显著减少(一个)及细胞分解(b)与野生型等位基因相比。根据Maqbool等人的评分标准,对细胞死于黄化和坏死进行评分。[34],0表示没有褪绿或坏死,6表示高度的褪绿或坏死。热图描述了被分配特定分数的复制的比例,其中深蓝色表示复制的密度较高。请注意,所有大于或等于4分的分数都被分配到同一组(“4+”),因为分数大于4分的发生率很低;N在20到35之间变化。所有对照等位基因(紫色)与野生型没有显著差异。TILLING/丙氨酸突变对对应的TILLING线如下所示。典型的斑块浸染叶片的褪绿评分(顶部,正面)和紫外线评分(底部,背面);数字标签与热图下面的数字标签相对应;M是模拟治疗。UV图像(底部)已被水平翻转,以便与白光图像进行比较。 Statistical comparisons shown are between the mutant allele and wild-typeNAM-A1基于符合泊松分布的线性模型。*,p< 0.05;**p< 0.01;***,p< 0.001
然后我们筛选了NAM-A1突变等位基因,包括TILLING和丙氨酸突变,用于减少细胞死亡反应。总的来说,我们发现两种细胞死亡评分方法(黄化和自身荧光)与在Y2H筛选中观察到的蛋白质相互作用的丧失密切相关。在酵母中显示蛋白质相互作用完全丧失的四个突变等位基因中(图2)。5),与野生型NAM-A1相比,其中3种也表现出明显减弱的黄化和细胞死亡反应(图2)。6,p< 0.01;R41q, t45m, r110w)。只有缺乏蛋白相互作用的P38S对细胞死亡反应没有影响。P154L的替换部分降低了蛋白质相互作用,但也具有临界显著性(p= 0.03)。两种对照等位基因均未显示萎黄或细胞死亡的减少,包括非保守错义突变(C133Y)。使用Western blot验证所有构建体的蛋白表达(附加文件)2:图S7)。
讨论
NAC转录因子已在几种植物中被鉴定[22,23,35并被发现在调节各种压力和发育反应(包括衰老)方面起着核心作用。在这项研究中,我们已经鉴定和表征了小麦NAC转录因子NAM-A1与其同源物NAM-B1之间蛋白质相互作用所需的一组残基。
NAC结构域的高度保守残基是跨物种蛋白质功能所必需的
虽然NAC结构域在物种之间是保守的,但尚不确定物种之间的蛋白质功能是否需要相同的残基,以及同一物种内的NAC TFs是否需要相同的残基。在这里,我们描述了保守残基错义突变对蛋白质功能的影响。Olsen等人(2005)先前研究表明拟南芥NAC转录因子ANAC092 (R19)中高度保守的盐桥形成精氨酸残基的突变足以破坏蛋白质二聚化[24]。在这里,我们已经独立地证实了这个残基(NAM-A1中的R41)在蛋白-蛋白相互作用中的重要性(R41Q;无花果。5),并表明该残基的突变导致小麦衰老的延迟(K2615;无花果。2)和减少细胞死亡反应n benthamiana(无花果。6)。同样,亚结构域iii中第二个精氨酸的R110W突变位于与ANAC019 (R88)中鉴定的DNA结合所必需的相同残基上[24]。在这里,我们发现这个残基对蛋白质-蛋白质相互作用也很重要,这是一个有点出乎意料的结果,因为残基位于蛋白质的假定dna结合区域。某些蛋白质结构域,如Dof和HMG盒子结构域,已知在蛋白质相互作用和DNA结合中都有作用[36,37R110周围的区域可能参与了这两个过程。另一种可能是,特定的残基对建立蛋白质结构至关重要,从而在突变时防止二聚化。此外,我们还描述了这种残留物的影响在足底结果表明,R110W在K2711中的替换导致田间小麦旗叶和花梗衰老的显著延迟,以及细胞死亡反应的减少n benthamiana。
在本研究过程中,Kang等人研究了拟南芥蛋白ANAC019中特定残基在不同pH水平下稳定蛋白结构的作用[25]。这些作者报道了ANAC019的H135组氨酸残基的质子化状态(图2)。1一个;子结构域iv中绿色部分)定义了蛋白质的二聚体形式及其与DNA形成复合物的能力。基于蛋白质结构模拟,作者提出,当H135残基没有质子化时,他们所谓的“完美”二聚体就形成了,这阻止了与亚结构域i中的D24残基形成盐桥。在这种状态下,二聚蛋白残基之间发生各种化学相互作用,有助于稳定“完美”二聚体(图1)。1A).如果H135残基被质子化,预计它会与残基D24形成盐桥,从而破坏大部分这些相互作用。鉴于这些结果,在“完美”二聚体状态下形成二聚体内键的残基可能是蛋白质二聚化所必需的,这似乎是合理的。
我们在这里研究的三个突变位于Kang等人突出显示的二聚化结构域内。1一个;子域i的绿色高亮部分[25]。两个残基P38S (K3661)和R41Q (K2615)直接对应于预测在“完美”二聚体状态下形成稳定键的残基(分别为ANAC019中的P16和R19)。第三个突变T45 M (K2734)位于这个二聚化区域,位于NAC一致序列中关键盐桥形成残基(D24)附近(图2)。1A)。在本研究中,我们发现NAM-A1的这三个残基突变导致蛋白质相互作用能力完全丧失,并延迟了花梗衰老。这些结果与Kang等人预测的这些残基在稳定NAC二聚体形成中的作用一致,并为其生物学相关性提供了强有力的证据在足底(25]。我们还发现R110残基位于DNA结合域中,是蛋白质二聚化所必需的,这表明H135可能不是DNA结合域中唯一参与蛋白质-蛋白质相互作用的残基。我们在小麦中总结和支持这些发现的能力也强调了NAC转录因子的功能域和必要残基可能在物种间高度保守的事实。
Kronos TILLING群体为小麦新等位基因变异的研究提供了便利
在本研究中,我们还表明,小麦TILLING群体,特别是四倍体Kronos群体,为研究小麦大等位基因系列突变提供了宝贵的资源。使用NAM-A1由于该基因在四倍体小麦中作为单拷贝基因发挥作用(考虑到其同源性),为本研究提供了便利NAM-B1含有导致Kronos中无功能蛋白质的帧移位)[5,27]。因此,我们能够描述M5突变系直接没有混杂效应,由于同源冗余。在克罗诺斯人群中,大约60%具有至少一种突变的基因包含至少一种过早截断突变[28]。这表明,对于很大一部分基因,应该有可能通过一个同源基因中期望的错义突变和另一个同源基因中常见的过早截断突变之间的单次杂交来研究相似的等位基因序列。我们还强调了使用多个独立TILLING突变来验证您感兴趣的候选基因的影响的重要性。我们在多个独立的TILLING系中观察到类似的延迟衰老效应NAM-A1基因,表明这种影响很可能是由于NAM-A1突变本身,而不是由于另一种密切相关的突变。这也提高了对野生型亲本(在本例中是Kronos)进行回交的重要性,以减少背景突变负荷[38]。
对小麦和其他作物的等位基因系列进行研究,可以帮助识别导致表型梯度的特定等位基因。我们发现,突变对温室中花梗衰老的影响从延迟几天到超过20天不等。同样,在K2711和K2711之间观察到表型的强度R110W和K1107干腊肠在田间也各不相同,特别是对旗叶的衰老。我们目前正在开发种质,以在田间试验中测试额外的错义突变对衰老的影响。众所周知,种群内的等位基因变异可以解释不同环境下的不同成功[39,40,41]。事实上,之前对自然变异的研究NAM-A1鉴定了一个错义突变(单倍型NAM-A1c),该突变与野生型等位基因和第三个单倍型之间的中间谷物蛋白含量表型相关,该单倍型包含错义突变和c端结构域的帧移位突变[42]。这个错义突变位于亚结构域iii末端的一个相对不保守的残基中。随着更多农学上重要的基因被鉴定出来,挖掘TILLING种群的新变异或碱基编辑等位基因将变得越来越可能[43],以智能地引入基于保守域的预测结果的变化。
NAM-A1突变体对花梗衰老的影响比对旗叶衰老的影响更严重
我们的等位基因系列研究NAM-A1突变体也强调了当突变在NAM-A1可导致旗叶衰老的延迟,特别是在田间,在温室和田间试验中对花梗衰老也有强烈而显著的影响。值得注意的是,旗叶和花序梗的视觉衰老性状得分确实不同。当25%的叶片变黄时,旗叶衰老被评分-衰老开始的量度。相反,当花梗顶部一英寸完全变黄时,花梗衰老就被评分了——这是衰老终止的一种衡量标准。这种差异是由于确定花梗何时开始衰老所固有的困难。大多数情况下,旗叶的衰老是以一种定向的方式发生的,从叶尖向基部移动。相比之下,花梗衰老可以同时发生在茎干周围,因此很难确定衰老的明确发生位置和时间。然而,通过SPAD测量旗叶衰老进程,也可以评估旗叶的衰老终止。这使得与花梗衰老的视觉测量进行更有意义的比较。在花梗衰老的显著延迟的温室试验中发现NAM-A1突变体(无花果。2B)。然而,使用SPAD测量没有观察到任何突变体旗叶衰老进程的显著延迟(附加文件)2:图S3)。相似的结果也被观察到NAM-A1野外突变体(图2)3.B和D)。这一数据证实了在比较视觉衰老评分时观察到的结果;也就是NAM-A1在温室和田间试验中,突变体对花梗衰老的影响比对旗叶衰老的影响更为严重和实质性。
以往的研究不结盟运动基因主要集中在旗叶衰老表型,尽管已注意到延迟花梗衰老表型[5,26,27,44]。众所周知,不结盟运动基因调节着营养物质进入穗的再活化,而穗在到达籽粒之前必须经过花梗。收获时花梗含氮量较高康南1号而NAM RNAi敲除谱则显示了谷物中微量营养素水平的改变[26,45]。早期的工作未能识别出任何表达不结盟运动基因在14 DAA的花梗组织中,初步表明NAM基因可能间接作用于花梗的营养物质再动员和衰老[46]。然而,最近来自全面发育时间过程的RNA-Seq数据已经确定了这种基因的表达不结盟运动灌浆过程中花梗上的基因(附加文件2:表S2) [47,48]。在可用于花梗组织的最晚时间点(乳粒期,约15 DAA),所有的水平不结盟运动花梗中的基因比旗叶中的基因要高,这支持了花梗的直接作用不结盟运动花梗中的基因。这证实了最近的表达分析,发现更高的表达水平不结盟运动与7 DAA的旗叶叶片相比,花序梗中基因的差异更大[49]。很容易让人猜测变异的NAM-A1等位基因延缓花梗衰老,从而将营养物质隔离在花梗中,阻止它们进入籽粒。对这一假设的支持来自于RNAi敲除线的研究不结盟运动该家族表明,突变系在衰老过程中比野生型系在花梗中保留了更多的铁、锌、氮和果聚糖含量[26,45,50]。
研究旗叶与其他组织(如花梗)的衰老情况比较,是可以理解的,因为大部分糖分和营养物质都是由旗叶提供的。[51,52]。此外,旗叶衰老的进程是相对容易测量使用非破坏性的SPAD计。然而,除了黄化的视觉评分外,花梗衰老的定量评分依赖于色素含量的破坏性测量。然而,我们的研究结果表明,了解花梗衰老的时间和调控机制对于了解旗叶衰老如何影响籽粒品质和产量也是必要的。
结论
在这里,我们结合了在NAC结构域的关键残基与生化和在足底研究它们在功能中的作用NAM-A1。利用Kronos TILLING群体,我们能够快速识别和筛选错义突变NAM-A1在温室和田地里。在确定了4个导致延迟花梗衰老的突变体后,我们使用酵母2-杂交系统来证明突变导致蛋白质相互作用的丧失,并表明这是突变体破坏蛋白质功能的机制。这些突变的特征证实了先前在拟南芥中进行的NAC TFs研究,并确定了蛋白质功能所需的先前未表征的残基。总之,这些结果突出了我们利用小麦新遗传资源(如TILLING群体)来探究特定突变对基因功能和表型的影响的能力。展望未来,这提出了一种利用小麦深入表征其他保守蛋白结构域的方法,然后可以将其与其他物种产生的知识相结合。
方法
植物生长和表型
温室实验
Kronos TILLING突变系[53含有基因突变的NAM-A1基因的选择如前所述[28]。简单地说,含有预测或已知影响蛋白质功能的错义突变的系被鉴定出来,与具有同义、非保守错义或剪接变异体突变的对照系一起被鉴定出来1) [20.,24]。米5将选择的TILLING系种子在4℃湿滤纸上预发芽48 h。然后将种子播种到P96托盘中,其中含有85%的细泥炭和15%的园艺砂砾。在2-3叶期,将单株移栽到彼得菲尔德谷物混合(彼得菲尔德,莱斯特,英国)的1升圆盆中。植物在标准的温室条件下生长,以16:8 h的光照:黑暗周期,并进行基因分型,以鉴定纯合突变体和野生型系(见下面的基因分型方法)。初生穗在抽穗处被标记(75%的穗从旗叶鞘脱出;Zadoks生长阶段57)[54]。当25%的主旗叶失去叶绿素并呈现视觉黄色时,旗叶开始衰老。当主花梗顶端1英寸完全变黄时,说明花梗衰老。实验在相同条件下重复2次(文中称为实验1和2)。
本菌烟生长
来自John Innes中心的野生型幼苗被播种在F2土壤(泥炭,2.5 kg/m)的半种盘中3.白云石,1.3 kg/m3.底肥2.7 kg/m3.3-4个月,0.25 kg/m3.润湿剂,0.3 kg/m3.Exemptor)。播种11天后移栽到FP8花盆中。在400瓦的钠灯下,植物在16:8的明暗环境下生长,白天温度为25°C,晚上温度为22°C。
JIC田间试验
选择2个品系(K2711和K1107)进行田间进一步评价。在每种情况下,都是纯合子NAM-A1突变植物与野生型的原始亲本克洛诺斯杂交。F1植物自花授粉产生F2突变型或野生型的群体分离NAM-A1等位基因。K1107获得2个独立居群(分别为K1107 - 4和K1107 - 6), K2711获得1个独立居群。F2种群于2016年3月在Bawburgh的Church Farm(52°38′n 1°10′e)播种。在2016年的生长季,施氮量为119公斤/公顷。
得分2表型和基因型的植株,个体F2手工播种,播种面积6 × 61 m236株植物组成的网格,每株植物之间间隔约17厘米(K2711;n= 158 f2植物;K1107-4,n= 218;K1107-6,n= 212)。个体F2对植株进行抽穗日期(两个分蘖,穗状花序出现75%)、叶片衰老(两个分蘖,旗叶从顶端衰老25%)和花梗衰老(两个花梗,顶部英寸衰老)评分。衰老评分与温室试验结果一致。通过对纯合突变体和野生型的鉴定2我们为每个品系和基因型选择了13 - 16株单株,以便在2017年进行进一步的表型分析(Davis)。2018年,这些选择的纯合子系的一个子集也被用于表型分析(JIC)。2016年的数据分析仅包括基因型为纯合子野生型或突变型的个体。
2018年4月,从独立的F22016年大田试验的植株是在教堂农场播种的K2711 (WT)NAM-A1,n= 5;突变体NAM-A1, n = 5)和K1107 (WTNAM-A1, n = 5;突变体NAM-A1, n = 5)。每个品系都被认为是突变型或野生型基因型的独立生物复制,以考虑来自原始TILLING突变的分离背景突变的潜在混淆影响。每个生物重复以双行1 m播种,以单空行隔开,按完全随机设计排列。采用与前面类似的方法对整行进行衰老评分,但在这里,当75%的主分蘖表现出叶或花梗衰老表型时,我们将双行评分为“衰老”。在2018年的田间试验中,施氮量为124.5 kg/ha。
戴维斯田间试验
纯合子系来源于个体F2播种K2711 (WT)植株NAM-A1 n= 13,变种人NAM-A1 n= 15)和K1107 (WTNAM-A1 n= 14,变种人NAM-A1 n= 16),于2016年11月在加州戴维斯(38°31 ' N, 121°46 ' W)的加州大学野外研究站(University of California Field Station, Davis, California)获得。如上所述,每个系都被认为是突变型或野生型基因型的独立生物复制。K2711和K1107作为两个独立的完全随机设计试验。与JIC 2018试验相同,每个生物重复以两行1米的方式播种,中间间隔一行。衰老的评分方式与2018年JIC试验相同,详见上文。总共200磅。每英亩施用硫酸铵,一半在播种前施用,另一半在拔节开始时施用(Zadoks 30, 3月312,017)。戴维斯和JIC的试验都喷洒了适当的商业杀菌剂,以避免疾病的发生。
叶绿素含量
采用柯尼卡美能达SPAD-502型叶绿素仪对叶片的相对叶绿素含量进行无损检测。在温室试验中,沿着旗帜叶片进行8次读数,并取平均值以获得每片叶片的总体读数(n每个品系和基因型= 5)。在2017年和2018年的田间测量中,每个生物重复至少测量2个叶片(每个品系和基因型n = 5),并在每个时间点进行平均测量。
在JIC 2018田间试验中,还从取样的花梗组织中化学测量了叶绿素含量。花后33天和49天分别取花梗顶端1英寸,切成小段(约0.5 cm长)。在叶绿素提取前,每个生物重复取样两个花梗。每个纯合基因型(K2711 WT、K2711突变体、K1107 WT和K1107突变体)取样5个生物重复。将植物组织在3 mL N,N-二甲基甲酰胺(分析级,Sigma Aldrich,英国)中4°C浸泡4 - 5天,直到所有色素从组织中浸出。叶绿素和类胡萝卜素含量按先前描述的方法定量测定[55]。
晶粒尺寸
使用MARVIN种子分析仪(GTA Sensorik GmbH)分析单株(2016年温室和田间)和汇总样本(2017年和2018年田间)的谷物样本。获得了籽粒形态计量参数,包括面积、长度和宽度,以及近似千粒重(TGW)。
谷物蛋白质含量
采用Perten DA7250近红外分析仪测定籽粒蛋白质含量。对20 ~ 700粒的籽粒样品进行蛋白质含量测定(蛋白质干基%)。小于300粒的样品使用静态杯进行分析;超过300粒的样品用旋转杯进行了分析。在基因型之间没有观察到水分含量的显著变化,因此在进一步的分析中没有考虑到。
基因分型
验证的存在NAM-A1TILLING突变,我们使用KASP基因分型(LGC Genomics),如前所述[56]。引物的基因分型设计使用Polymarker,在可能的情况下,否则设计基于对齐序列NAM-A1同源物以确保同源物特异性(附加文件)2:表S3) [57]。
NAC域对准
含有NAC结构域的蛋白质拟南芥,玉米,杨树trichocarpa,Physcomitrella金属盘使用Interpro ID IPR003441从EnsemblPlants中提取。来自大麦芽和栽培稻从Borrill et al. 2017的策划列表中获得[22]。含有nac结构域的蛋白质来自小麦从RefSeqv1.1的基因注释中获得[30.]。
序列使用Clustal Omega v1.2.0进行对齐,并在Jalview v2.10.5中进行手动整理[58]。比对后,在NAC转录因子集合中占用率低于10%的残基被排除在进一步分析之外,如Borrill等人2017年所述[22]。非保守的c端结构域也被排除在进一步的分析之外。在此基础上,每个残基上的Jensen-Shannon散度评分按照Capra和Singh(2007)的方法计算,使用“蛋白质残基守恒预测”web服务器[31]。
NAM-A1和NAM-B1等位基因克隆
TILLING突变NAM-A1基因(TraesCS6A02G108300)在TILLING数据库中得到鉴定www.wheat-tilling.com(28]。根据NAC结构域残基的保守性选择特定等位基因(PSSM viewer, pfam02365)并预测SIFT分数[59]。为简单起见,克罗诺斯突变系被标记为“K”,后面跟着它们的四位标识符(例如,克罗诺斯2711被称为K2711)。
的NAM-A1序列从t . aestivum(Transcript traescs6a02g1083002)合成pUC57载体(Genewiz),使用TOPO克隆试剂盒(ThermoFisher Scientific)使用附加文件中的引物克隆到pCR8载体上2表S4。利用附加文件中的引物进行定点诱变2:表S5,获取的等位基因序列NAM-A1变体。所有克隆产物均使用Sanger测序(Eurofins Genomics)进行确认。的NAM-B1序列由肿脓杆菌dicoccoides(GenBank登录号DQ869673.1),并按上述方法合成(附加文件)3.)。
Co-immunoprecipitation
的野生型等位基因NAM-A1在pGWB21载体(n端Myc标签)和NAM-B1在pGWB12矢量(n端FLAG标签)中[60被共同渗透n benthamianaP19的总外径为0.6,单个外径为0.2。在浸润后3天收获叶片并速冻。在标准缓冲液中提取蛋白质(100 mM Tris-HCl, pH 7.5;150mm NaCl;0.1% (v/v) Triton x-100;1% (w/v) PVPP;5毫米EDTA;10%甘油;2 mM DTT;1%蛋白酶抑制剂(Sigma))。 1 mL of protein extraction was applied to M5 anti-FLAG magnetic beads (Sigma). The beads were then boiled and the resulting fraction was run on a 12% SDS-PAGE gel, alongside the input and unbound fractions. The proteins were transferred to a nitrocellulose membrane and probed with both an anti-FLAG HRP conjugated antibody and an anti-Myc HRP conjugated antibody (Abcam). Total input was visualised by staining the membrane with Ponceau following antibody probing. The experiment was repeated three times with consistent results.
Yeast-two-hybrid
使用ProQuest Two-Hybrid系统进行轻微修改,以验证蛋白质之间是否存在相互作用NAM-B1和NAM-A1等位基因。简单地说,编码NAM-A1和NAM-B1 NAC结构域的序列分别为残基1-217和1-215;额外的文件3.)扩增并克隆到pCR8中,使用附加文件中的引物2表S4。等位基因的NAM-A1(1-217)和野生型NAM-B1将(1-215)序列克隆到pDEST22和pDEST32载体上。这些等位基因包括TILLING系中存在的突变和相关的丙氨酸突变,如上所述(附加文件)2表5)。仅使用NAC结构域来避免由NAC转录因子c端结构域的转录激活结构域引起的Y2H系统的自激活[23]。
的NAM-A1(1-217)两个载体中的等位基因共转化NAM-B1(1-215)在替代载体中转化为具有化学活性的MaV203细胞。同时,单个向量有和没有NAM-A1将(1-217)等位基因转化为MaV203作为自动激活对照。转化在SC-Leu-Trp介质上恢复。
最初,转化后的单个菌落在200 μL的dH中重悬20.将2 μL菌落悬液分别镀于SC-Leu-Trp(对照)和SC-Leu-Trp- his + 10 μM 3AT(选择)上。这些滴落试验对选择培养基上的生长进行评分,作为蛋白质相互作用的指示。每次转化测试6个个体菌落。实验重复了三次,结果一致。
还进行了稀释试验,以突出蛋白质相互作用的相对强度。在这里,单个菌落在液体SC-Leu-Trp培养基中培养过夜,OD为0.1。由此,在dH中进行连续1:10稀释20.分别对pDEST32中NAM-B1和pDEST22中NAM-A1等位基因的组合进行了3个独立菌落的检测。实验重复了两次,结果一致。
细胞死亡评分n benthamiana
全身的NAM-A1等位基因克隆到Gateway二进制载体pGWB12中,带有n端FLAG标签[60]。这些转化为农杆菌(菌株LBA4404)并共浸润到3周龄的小鼠体内n benthamiana与沉默抑制剂P19一起生长的叶片,总OD值为0.4,每个构造的个体OD值为0.2。入渗后3 d收获叶片,在液态氮中速冻2。叶片组织在液氮中研磨2,按照上述方法提取蛋白质,并在12% SDS-PAGE凝胶上运行。将该蛋白转移到硝化纤维素膜上,并用抗flag酶标抗体(Abcam)进行探针检测。用Ponceau染色观察总蛋白输入。
的NAM-A1等位基因也被斑块浸润n benthamiana叶子在上面详细说明的OD。浸润后5天对浸润斑块进行细胞死亡评分,详见Maqbool et al. 2015 [34]。简单地说,利用紫外线和白光分别对叶片的背面和正面进行成像。然后根据荧光或褪绿的强度对浸润斑块进行从0到6的离散评分。每个等位基因至少在20个独立叶片上以叶片周围随机模式重复渗透。
数据分析和可视化
所有数据分析均使用R (v3.5.1)进行。使用ggplot2 [61]。使用基础R包进行统计。使用的统计测试在可能的情况下在文本中注明。当温室和田间试验数据的分析不能满足正态性假设时,使用非参数统计检验(双样本Wilcoxon, Kruskal-Wallis秩和)。当满足正态性假设时,使用学生t检验。中数据的非正态和离散性质n benthamiana细胞死亡实验,统计比较突变等位基因对野生型的细胞死亡反应NAM-A1采用泊松分布拟合线性模型。
数据和材料的可用性
本研究过程中产生或分析的所有数据都包含在这篇发表的文章及其补充信息文件中。试验中使用的原始耕作系可从SeedStor (https://www.seedstor.ac.uk/)。所有的质粒都含有野生型NAM-A1和NAM-B1本文中使用的层序沉积于Addgene (https://www.addgene.org/Cristobal_Uauy/)。
缩写
- DAA:
-
开花后几天
- JSD:
-
Jensen-Shannon散度
- TF:
-
转录因子
- 耕作:
-
靶向诱导的基因组局部病变
- Y2H:
-
酵母混动
参考文献
- 1.
李建军,李建军,李建军,李建军。无顶端分生组织基因牵牛花胚和花的模式形成所必需的,并在分生组织和原基边界表达。细胞。1996;85(2):159 - 70。
- 2.
Aida M, Ishida T, Fukaki H, Fujisawa H, Tasaka M.拟南芥杯形子叶突变体的器官分离相关基因分析。植物学报。1997;9(6):841。
- 3.
谢强,刘国强,蔡永华。拟南芥母转导生长素信号下游TIR1促进侧根发育。基因工程学报,2000;14(23):3024-36。
- 4.
郭勇,甘思。AtNAPNAC家族转录因子在叶片衰老过程中起重要作用。植物学报,2006;46(4):691 - 691。
- 5.
王晓明,王晓明,王晓明,等。NAC基因对小麦籽粒蛋白质、锌和铁含量的影响。科学。2006;314(5803):1298 - 301。
- 6.
colinge M, Boller T.马铃薯两个基因的差异诱导;Stprx2和StNAC,以应对感染5种还有伤害。植物化学学报,2001;46(5):521 - 521。
- 7.
陈少林,Nakashima K, Sakuma Y, Simpson SD, Fujita Y, Maruyama K,等。拟南芥干旱胁迫诱导NAC转录因子的分离与功能分析独联体-元素对脱水应激反应早启动子。植物学报。2004;16(9):2481-98。
- 8.
Jensen MK, Hagedorn PH, de Torres-Zabala M, Grant MR, Rung JH, Collinge DB,等。NAC (nam - ataf1,2 - cuc2)转录因子的转录调控可减弱ABA信号,从而实现有效的基础防御Blumeria茎f . sp。hordei在拟南芥。植物学报,2008;36(6):867 - 868。
- 9.
薛国平,倪鲍尔,McIntyre CL, Riding GA, Kazan K, Shorter R。TaNAC69来自NAC超家族的转录因子在小麦非生物胁迫下上调,并识别两种一致的dna结合序列。植物学报,2006;33(1):43-57。
- 10.
夏宁,张刚,刘学祥,邓磊,蔡国良,张勇,等。小麦抗条锈病病原菌侵染和非生物胁迫的新NAC转录因子基因的鉴定中国生物医学工程学报,2010;37(8):391 - 391。
- 11.
夏宁,张刚,孙云峰,朱丽,徐丽生,陈晓明,等。TaNAC8NAC转录因子是小麦对条锈病病原菌侵染和非生物胁迫的应答基因。植物生理学报,2010;34(5):394-402。
- 12.
唐勇,刘敏,高松,张志,赵旭,赵超,等。新型分子表征TaNAC小麦基因与过表达TaNAC2a赋予烟草耐旱性。植物物理学报,2012;44(3):210-24。
- 13.
毛翔,张宏,钱翔,李安,赵刚,景睿。TaNAC2一种nac型小麦转录因子,增强了小麦的多种非生物胁迫耐受性拟南芥。实验学报,2012;63(8):2933-46。
- 14.
冯宏,段鑫,张强,李鑫,王斌,黄磊,等。来自NAM亚家族的新型NAC转录因子tae-miR164的靶基因负调控小麦对条锈病的抗性。植物病理学杂志,2014;15(3):284-96。
- 15.
毛翔,陈生,李安,翟超,景睿。新型NAC转录因子TaNAC67赋予拟南芥增强的多非生物胁迫耐受性。科学通报,2014;9(1):84359。
- 16.
黄强,王勇,李斌,常军,陈敏,李凯,等。TaNAC29小麦NAC转录因子提高转基因拟南芥的耐盐性和耐旱性。植物学报,2015;15(3):268。
- 17.
王峰,林荣,冯军,陈伟,邱东,徐生。TaNAC1作为小麦抗条锈病的负调节因子,提高对条锈病的敏感性两,并促进转基因植株侧根发育拟南芥。植物科学,2015;6:108。
- 18.
陈东,柴松,McIntyre CL .,薛国平。小麦根系中NAC转录因子的过度表达可提高根系长度、生物量和抗旱性。植物学报,2018;37(2):225-37。
- 19.
赵东,刘立东,赵德平,赵德平,刘志强,何志强。NAC转录因子过表达可延缓小麦叶片衰老,提高籽粒氮浓度。植物学报,2015,17(4):904-13。
- 20.
Ernst HA, Olsen AN, Larsen S, Lo Leggio L.转录因子家族成员ANAC的保守结构域结构。中华医学杂志2004;5(3):297-303。
- 21.
Welner DH, Lindemose S, Grossmann JG, Mollegaard NE, Olsen AN, Helgstrand C,等。植物特异性NAC转录因子在晶体和溶液中的DNA结合:与WRKY和GCM转录因子的紧密联系。中国生物医学工程学报;2009;31(3):394 - 394。
- 22.
李春华,李春华,李春华,等。小麦NAC转录因子家族的全基因组序列和表达分析。G3(马里兰州贝塞斯达)。2017; 7(9): 3019 - 29。
- 23.
陈建军,陈建军,李建军,等。NAC转录因子的研究进展。植物科学进展,2005;10(2):79-87。
- 24.
王晓明,李建军,李建军,等。NAC转录因子的分子功能及其与dna结合的特异性。植物科学与技术,2005;32(4):785 - 797。
- 25.
姜敏,金生,金海军,Shrestha P, Yun JH, phebk,等。NAC转录因子ANAC019的c结构域是通过n结构域的组氨酸开关进行ph调谐DNA结合所必需的。中国生物医学工程学报,2018;22(5):1141-50。
- 26.
李建军,赵荣,李建军,李建军,李建军,李建军等。功能表征GPC-1六倍体小麦的基因。足底。2014;239(2):313 - 24。
- 27.
Pearce S, Tabbita F, Cantu D, Buffalo V, Avni R, Vazquez-Gross H,等。锌和铁转运体的调控GPC1小麦单果早衰过程中的基因研究。植物学报,2014;14(1):368。
- 28.
Krasileva KV, Vasquez-Gross HA, Howell T, Bailey P, Paraiso F, Clissold L,等。揭示多倍体小麦的隐性变异。自然科学进展,2017;39(6):913。
- 29.
Vullo A, Allot A, Zadissia A, Yates A, Luciani A, Moore B等。集成基因组2018:非脊椎动物物种的集成组学基础设施。核酸学报,2017;46(D1): D802-D8。
- 30.
李建军,李建军,李建军,等。利用全注释参考基因组改变小麦研究和育种的限制。科学。2018;361 (6403):eaar7191。
- 31.
李建军,李建军。基于序列守恒的功能重要残基预测。生物信息学。2007;23(15):1875 - 82。
- 32.
坎宁安BC,威尔斯JA。利用丙氨酸扫描诱变技术对hgh受体相互作用进行高分辨率表位定位。科学。1989;244(4908):1081 - 5。
- 33.
郑家祥,朴玉涛,郑海,朴世华,金建康。水稻NAC蛋白具有同型二聚体和异源二聚体的作用。植物学报,2009;3(2):127-34。
- 34.
Maqbool A, Saitoh, Franceschetti M, Stevenson CEM, Uemura A, Kanzaki H,等。植物NLR免疫受体中整合HMA结构域识别病原体的结构基础。eLife。2015; 4: e08709。
- 35.
Ooka H, Satoh K, Doi K, Nagata T, Otomo Y, Murakami K,等。NAC家族基因的综合分析栽培稻和拟南芥。中国生物医学工程学报,2003;10(6):239-47。
- 36.
Štros M, Launholt D, Grasser KD。HMG-box:一种多用途的蛋白质结构域,存在于多种dna结合蛋白中。细胞与生命科学,2007;64(19):2590。
- 37.
王晓明,王晓明,王晓明,等。植物dna结合锌指转录因子家族的研究进展。植物科学,2013;29(1):32 - 45。
- 38.
Uauy C, Wulff BBH, Dubcovsky J.传统诱变与新型高通量测序和基因组编辑相结合揭示多倍体小麦隐藏变异。热疗学报,2017,51(1):435-54。
- 39.
Worland AJ。开花时间基因对欧洲小麦环境适应性的影响。Euphytica。1996;89(1):49-57。
- 40.
刘建军,李建军,李建军,等。美国大平原地区冬小麦发育基因等位变异及其对抽穗期的影响。科学通报,2016;11(4):e0152852。
- 41.
杨晓明,王晓明,王晓明,等。北美高纬度冬小麦春化和光周期响应基因的等位基因变异。PloS one。2018; 13 (8): e0203068-e。
- 42.
李建军,李建军,李建军,等。检测NAM-A1面包小麦的自然变异揭示了世界范围内核心种质与欧洲精英种质之间单倍型分布的差异。农学。2015;5(2)。
- 43.
宗毅,王毅,李超,张锐,陈坤,冉毅,等。利用cas9 -胞苷脱氨酶融合对水稻、小麦和玉米进行精确碱基编辑。生物工程学报。2017;35:438。
- 44.
李建军,李建军,李建军,等。小麦衰老调控因子的基因网络模型研究。bioRxiv。2018:456749。
- 45.
张建军,张建军,张建军,等。小麦(小麦) NAM蛋白调节铁、锌和氮化合物从营养组织向籽粒的转运。[J] .中国生物医学工程学报,2009;30(5):563 - 564。
- 46.
李建军,李建军,李建军。营养分配与籽粒产量TaNAM非生物胁迫下的rnai小麦。植物学报,2013,31(1):573-91。
- 47.
Borrill P, Ramirez-Gonzalez R, uway C. expVIP:可定制的RNA-seq数据分析和可视化平台。植物生理学报,2016;17(4):2172。
- 48.
Ramírez-González RH, Borrill P, Lang D, Harrington SA, Brinton J, Venturini L,等。多倍体小麦的转录景观。科学。2018;361 (6403):eaar6089。
- 49.
Borrill PGM。的NAM-B1转录因子与小麦籽粒组成的调控。英国:东安格利亚大学;2014.//www.cinefiend.com/articles/10.1186/s12870-016-0889-y。
- 50.
Borrill P, Fahy B, Smith AM, uway C.小麦籽粒灌浆受到籽粒灌浆能力的限制,而不是旗叶光合作用持续时间的限制不结盟运动RNAi植物。科学通报,2015;10(8):e0134947。
- 51.
李建平,李建平。小麦中铁和铜的分布与再活化。中国生物医学工程学报,2009;35(5):817 - 826。
- 52.
Kichey T, Hirel B, Heumez E, Dubois F, Le Gouis j。小麦l),开花后氮素的吸收和再动员与农艺性状和氮素生理标志物相关。作物学报,2009,32(1):1 - 4。
- 53.
uway C, Paraiso F, colasonno P, Tran RK, Tsai H, Berardi S,等。一种改良TILLING方法检测四倍体和六倍体小麦诱导突变。植物学报,2009;9(1):115。
- 54.
赵建军,张涛,孔泽克,等。谷物生长阶段的十进制编码。植物科学。1974;14(6):415-21。
- 55.
Wellburn AR。叶绿素a和b以及总类胡萝卜素的光谱测定,使用不同的溶剂和不同分辨率的分光光度计。植物生理学报,2004;19(3):387 - 398。
- 56.
刘建军,刘建军,李建军,等。RNA-Seq散装分离分析为六倍体小麦育种提供了高分辨率的遗传标记。植物学报,2015;13(5):613-24。
- 57.
Ramirez-Gonzalez RH, uway C, PolyMarker CM。快速多倍体引物设计流水线。生物信息学,2015,31(12):2038 - 9。
- 58.
Sievers F, Wilm A, Dineen D, Gibson TJ, Karplus K, Li W,等。使用Clustal omega快速,可扩展地生成高质量的蛋白质多序列比对。生物化学学报,2011;7:539。
- 59.
Ng PC, Henikoff SSIFT。预测影响蛋白质功能的氨基酸变化。核酸学报,2003;31(13):3812-4。
- 60.
Nakagawa T, Kurose T, Hino T, Tanaka K, Kawamukai M, Niwa Y,等。为实现植物转化融合基因的高效构建,开发了一系列门户二值载体pgwb。中国生物医学工程学报,2007;34(1):444 - 444。
- 61.
Wickham H. ggplot2:数据分析的优雅图形。纽约:斯普林格出版社;2016.
致谢
作者要感谢J. Dubcovsky(加州大学戴维斯分校)在加利福尼亚州戴维斯的田间试验期间的支持,以及约翰英纳斯中心和加州大学戴维斯分校的温室和实地小组。我们也感谢约翰英纳斯中心的种质资源组进行近红外分析。他们还想感谢M. Banfield和A. Smith (JIC)在本手稿开发过程中提供的有用意见。
资金
这项工作由英国生物技术和生物科学研究委员会(BBSRC)资助,授予BB/P013511/1, BB/P016855/1和周年未来领袖奖学金(BB/M014045/1)。SAH由John Innes基金会资助。LEO由BBSRC诺维奇研究园区生物科学博士培训合作伙伴研究经验安置计划(BB/M011216/1)支持。NC由Fulbright和CONICYT Becas-Chile 72111195资助。资助者没有参与研究的设计、数据的收集、分析或解释,也没有参与论文的撰写。
作者信息
从属关系
贡献
SAH, PB和CU构思了这项研究。SAH和LEO克隆了NAM-A1等位基因,并进行了酵母2杂交筛选。SAH、PB和NC进行了现场试验。SAH进行了温室试验n benthamiana筛选并分析NAC结构域的保守性。SAH和CU撰写了这篇论文,所有作者都对手稿发表了评论。所有作者已阅读并同意稿件。
相应的作者
道德声明
伦理批准并同意参与
不适用。
发表同意书
不适用。
相互竞争的利益
作者宣称他们没有竞争利益。
额外的信息
出版商的注意
施普林格·自然对已出版的地图和机构关系中的管辖权要求保持中立。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/),它允许在任何媒体上不受限制地使用、分发和复制,前提是您要适当地注明原作者和来源,提供到知识共享许可协议的链接,并注明是否进行了修改。创作共用公共领域免责声明(http://creativecommons.org/publicdomain/zero/1.0/)适用于本文中提供的数据,除非另有说明。
关于本文
引用本文
哈林顿,南卡罗莱纳州,奥弗恩,路易斯安那州,科博,北卡罗来纳州。et al。小麦中的保守残留物(小麦NAC结构域是蛋白质结合所必需的,当发生突变时,会导致花梗延迟和旗叶衰老。BMC Plant Biol19日,407(2019)。https://doi.org/10.1186/s12870-019-2022-5
收到了:
接受:
发表:
关键字
- 小麦
- 衰老
- 南京汽车领域
- 突变体
- 耕作
- NAM-A1
- 酵母双杂交