摘要
背景
转座因子(TEs)占小麦基因组的80%以上,通常对特定的超级家族和家族具有独特的特征。然而,TEs在小麦进化和重塑小麦基因组中的作用仍然很大程度上不清楚。
结果
在这项研究中,我们在野生emmer小麦的两种过程中发现了一种在基因的外显子6中的微型(307bp长度)Te样序列,其用于5-甲酰基四氢脱溶胶(t . turgidumSSP。dicoccoides),并通过一个终止密码子产生一个更短的阅读框来干扰基因翻译。这个序列被命名了玛丽安,并没有显示出任何结构上的相似性。它不具有末端倒置重复序列(TIR),这将允许我们将该元件分配到TIR DNA超级家族之一,而且它不具有SINE的特征,如Pol-III启动子或poly-A尾部。在硅片里对五份公开的基因组草图的分析小麦属植物和山羊草属物种透露,玛利亚姆元素出现在二倍体小麦物种中的非常低的拷贝数(1-3插入)中,在四倍体和六倍倍性小麦物种中〜12插入。此外,玛利亚姆发现元素是小麦独有的,因为它在其他植物基因组中未发现。动态性质玛利亚姆通过特异性PCR分析评估小麦基因组,并揭示其以人群特异性方式在野生emmer群体中保留活性。
结论
这项研究为TEs在小麦中的进化影响提供了新的见解。
背景
转移元素(TES)是具有在其宿主内增殖的能力的DNA段,因此它们可以构成大部分真核基因组[1].小麦基因组含有数千个已知TE家族,占基因组的80%左右,其中LTR逆转录转座子最为丰富[2那3.那4.那5.].TEs分为Class I元件(反转录转座子)和Class II元件(DNA转座子),后者又分为super- family和family [6.].
te可被各种生物或非生物胁迫刺激,如热休克、创伤或细菌感染[7.那8.那9.,以及基因组的压力,如杂交和多倍体化[10.].基因组中te的活性可以改变其基因和表观遗传结构[11.那12那13那14].TES进入编码区(外显子)可能导致函数的破坏和突变表型的产生[15],而插入内含子可能会干扰转录本编辑或导致内含子保留[16那17那18].在基因附近插入te会影响它们的表达,例如通过干扰启动子的活性[15那16].TES扩增的另一个已知后果是宿主基因组大小的膨胀[14那19].所有这些效应在植物的等位基因和表型多样性中起着关键作用[14].
小麦 (Triticum-Aegilops.类群)起源于大约400万年前,由三个二倍体物种从一个共同的祖先分化而来。四倍体的t . turgidumSSP。dicoccoides(野生二粒体,AB基因组)由异源多倍体化事件产生,包括杂交小麦属植物urartu(A基因组供体)和未知山羊草属物种(截面sitopsis., B基因组的供体)。野生二粒小麦的初步驯化及其带壳驯化二粒小麦的进化(t . turgidumSSP。dicoccun.)诱导选择自由脱粒的杜兰姆(t . turgidumSSP。杜伦姆AB基因)。六倍体的小麦(面包小麦,ABD基因组)是在大约1万年前由第二个主要的多倍体化事件产生的,该事件涉及到驯化的二粒小麦(t . turgidumSSP。dicoccun.),山羊草属tauschii(D基因组供体)[4.那20.那21].野生二粒是作为野生物种在自然界中被发现的,自从它在1913年被阿哈隆·阿哈隆森重新发现以来[22],这一种已被广泛研究,作为一种潜在的供体,为驯化小麦提供有益性状[22那23那24那25那26].野生二粒是一种一年生的、主要自花传粉的物种,在中东地区不同的环境条件下以斑块的方式分布,这些环境条件在年平均温度、海拔、土壤类型和其他条件下都不同。在以色列,在北部的黑门山和南部的亚玛撒山(犹太沙漠)之间有20多个种群(孤立或半孤立)的野生二聚体[27那28].最近,我们发现转座子可以在野生二粒小麦中以群体特异性的方式增殖,从而产生等位变异[29].
在这项研究中,我们发现了一个小麦独特的微型te样序列玛利亚姆在两个野生二粒小麦的边缘群体。5个最近更新的小麦基因组草图的可用性;t . turgidumSSP。dicoccoides那t . turgidumSSP。杜伦姆那T. Aestivum.那Ae。Tauschii和t . urartu,促进了计算机辅助分析玛利亚姆在这些小麦品种的含量和动态。此外,小说的影响玛利亚姆在评估用于编码5-甲酰基四氢脱氢的基因中,以及动态玛利亚姆在野生二粒小麦种群中。为此,进化的影响玛利亚姆讨论了插入。
结果
在黑门山野生二粒小麦群体5-甲酰基四氢叶酸基因的编码序列中发现了一个新的te样DNA片段
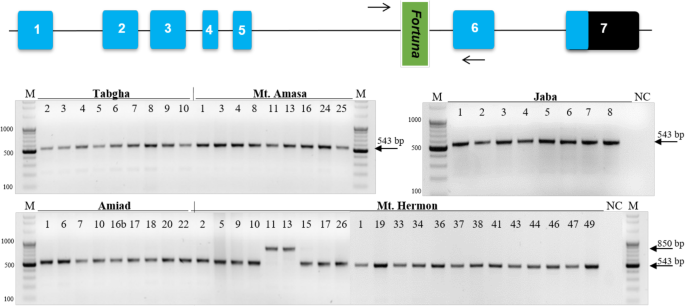
作为研究的一部分[29我们在5个野生二粒小麦群体中发现了一个长度为307 bp的DNA短插入序列,该序列编码5-甲酰基四氢叶酸(TRIDC2AG023940,EnsemblPlants)在山的两种录取中。Hermon人口狂野emmer群体(图。1最佳)。基因组特异性引物由内部5上游到螨虫插入设计命运,从基因的外显子6(图。1额外的文件1:表S1)。完整网站的预期大小为543 bp。特定于网站的PCR实验表明命运元素在黑门山、亚米德山、Tabgha山、雅巴山和亚玛撒山5个居群中均存在,但在黑门山居群的2个居群中扩增了850 bp的高频段,而不是预期的全频段或空频段(图2)。1底部)。
上图:TRIDC2AG023940基因的结构示意图。基因序列由7个外显子(框)和1个外显子组成命运外显子5和外显子6.注意到基因的编码序列(CDS)在外显子7中末端。SSPCR的正向和反向引物由箭头表示。底部:特异性PCR分析展示命运在所有群体中插入TRIDC2AG023940基因的内含子5。在黑门山居群(左下)的两份材料中,插入的下限(543 bp)和上限(850 bp)分别表示为插入点。M为大小标记(左边数字为bp), NC为阴性对照(PCR以水为模板)。本ssPCR所使用的引物(见顶方案)为:Forward = TCTTTGTGTATTCTCTAGCTCTGT, Reverse = ACTCCACCCTTTCTCTTTAGCA
307 bp插入序列分析(图。2)显示该序列未与包括基因组及转录组数据库的任何已知序列相匹配(https://plants.ensembl.org/Triticum_aestivum/Info/Annotation/[30.],http://plants.ensembl.org/Triticum_dicoccoides/Info/Annotation/[4.]),以及ITMI等重复数据库[31),Triticeae重复数据库(http://botserv2.uzh.ch/kelldata/trep-db/index.html)和Giri数据库[32)(http://www.girinst.org/censor/index.php),没有产生与注释的转座元素的匹配。有趣的是,307 bp序列的两侧有一个9 bp的序列(CCAAGAACT),类似于靶位点复制(TSD),可由转座元件插入产生(图)。2).缺失9 bp位点的基因仅在一个拷贝中发现,表明307 bp的序列与te类似,形成了TSD。此外,307 bp插入序列不具有末端倒置重复序列(TIR),这将允许我们将该序列分配给TIR DNA超级家族之一。此外,它不具有典型的sine特征,如Pol-III启动子或poly-A尾部。
测试类te插入的其他副本是否出现在Triticum-Aegilops.利用MAK软件从最近的基因组草图中检索到te样插入序列。我们找到了一个插入t . urartu(AA基因组),3个插入Ae。Tauschii.(DD Genome),12插入(8个亚基A和4中的8个)t . turgidumSSP。dicoccoides(野生Emmer,AABB Genome)那6插入(4在亚组A和2中的4个)t . turgidumSSP。杜伦姆(杜兰姆,AABB基因组)和11插入(3在亚基A,4中的3个,亚基组中的4个)T. Aestivum.(面包小麦,AABBDD基因组)。可以在附加文件中找到所有检索的插入的序列及其分子charactration,包括亚基因组和染色体位置1S1:表。注意,这个插入被发现是唯一的Triticum-Aegilops.在其他植物基因组中未发现。
从使用WebLogo软件从5个基因组草稿中检索的33全长307bp插入的侧翼序列创建序列徽标[33)(http://weblogo.threeplusone.com/额外的文件4.:图S1)。33个插入中有16个显示明显的TSD,其余17个插入不显示明显的TSD(附加文件1:表S1)。序列徽标在第4和9处的特定核苷酸中显示了某种序列偏好,但是9-BP重复序列不受高度保守。缺乏可检测的TSDS对于17个插入可以通过围绕每个插入的前方相同的序列破坏的插入围绕插入的突变/重排来解释。请注意,缺少TSD的插入超过50%(17个)位于基因组BB中,已知是非常动态的[20.那21].为此,在大约50%的插入中检测到清晰的保守长度的tsd,这一事实支持了类似转座子行为的假设,因此我们将其命名为无tir的微小元素。”Mariam“.注意,多个序列对准分析显示33个全长的高序列守恒(超过90%)玛利亚姆从五个基因组草稿中恢复过来的元素。高序列保护玛利亚姆二倍体和多倍体物种中可能强烈表示该元素在小麦中的最近促进剂。
插入多态性的玛利亚姆在Triticum-Aegilops.群内野生二粒种群
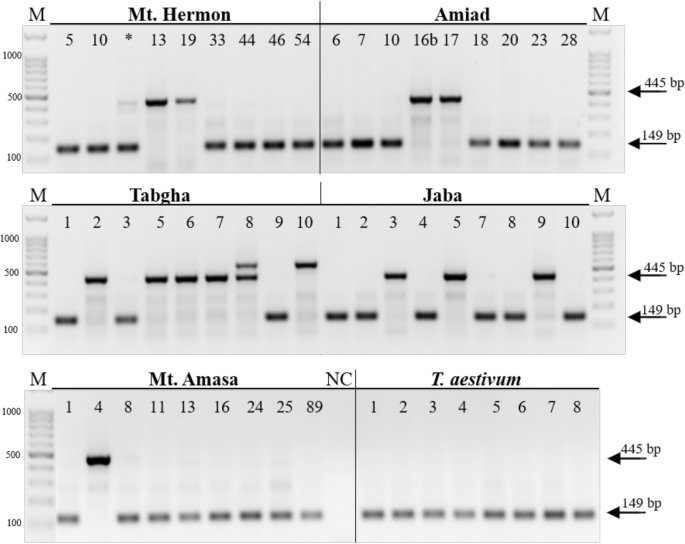
如上所述,存在玛利亚姆元素t . urartu和Ae。Tauschii.,在面包小麦的三个亚基因组中表明该元素可能存在于野生二粒小麦和面包小麦的所有二倍体祖先中,并在多倍体物种中扩增。的7玛利亚姆在A和B子基因组中发现的插入T. Aestivum.,6是常见的emmer常见的(单数插入)。此外,所有3玛利亚姆中插入Ae。Tauschii.是D亚基因组中常见的T. Aestivum..最后,插入中找到t . urartu是野生二粒和T. Aestivum..的动力玛利亚姆使用特异性PCR分析评估野外emmer小麦和面包小麦涂抹中的元素,从45种野生emmer摘录中分离出来(从5种不同的独立孤立的人群中收集; Mt. Hermon,Amiad,Tabgha,Jaba和Mt.Amasa - 9从每个人口的access)和8个面包小麦加入(见附加文件2:表S2)。总体而言,引物从10个基因的侧翼序列中设计玛利亚姆野生二粒小麦和/或面包小麦的A和B亚基因组(附加文件)3.:表S3)。
PCR结果表明,该菌株具有较高的插入多态性水平玛利亚姆基于野生二粒小麦的存在(全位点)与缺失(空位点)。只检查了一个插入,A5-6(表1),存在于野生emmer和面包小麦的所有含义中(单数插入),而6插入(A4-4(图。3.)、A6-2、A7-5、B3-4、B7-4、B7-6(附加文件4.(图S2)在野生二粒小麦中以群体特异性的方式具有多态性,而在面包小麦中没有检测到(表S2)1).一个插入(B1-4,表1)缺乏所有野生emmer小麦的牧草,但在所有面包小麦acce中存在。在另外两个案例中(a4-1(附加文件4.:图S2f), B1-4(附加文件4.(图S2g)),野生二粒小麦和面包小麦的插入均具有多态性。8个面包小麦品种中只有1个品种存在a1 - 4插入,7个品种存在B1-4插入。
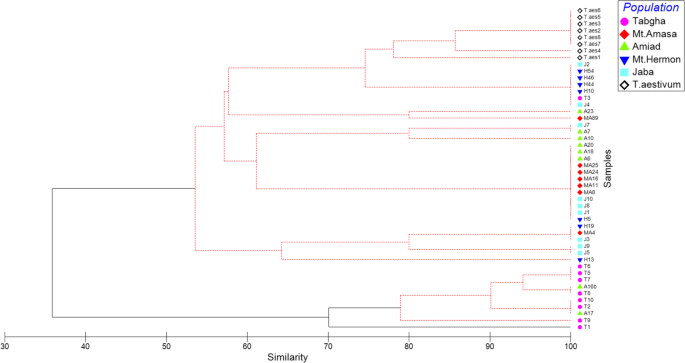
的插入模式玛利亚姆(根据ssPCR分析结果)构建层次凝聚聚类生成的系统发生树(见图)。4.).系统发育树显示显著(P.-value< 0.05),表明Tabgha种群(除T3加入外)与其他种群的分离程度与我们之前根据迈诺斯(一个螨科)插入模式评估转座子显示分析[29].所有T. Aestivum.加入将集聚在一起,以及Mt.除了他们的两种进入之外,Amasa和Amiad通常被他们的人口聚集在一起。JABA和Hermon种群在人口中的甲型中显示出高度的变化。这些发现可能表明人口特定的动态玛利亚姆在野生二粒小麦和面包小麦品种。
玛利亚姆插入与小麦基因有关
序列的注释玛利亚姆插入网站显示33中的11个玛利亚姆插入位于其他TE元素附近,包括class I和class II元素(附加文件1表S1), 12个插入位点位于或邻近基因(下游或上游高达500 bp),如甲酰基四氢叶酸环连接酶、蔗糖-磷酸酶3、soleucyl-tRNA合成酶、丝氨酸/苏氨酸蛋白激酶、木葡聚糖酶等。这是在前面描述的之外玛利亚姆TridC2AG023940基因的插入(A2-MH)在编码预测的线粒体5-甲酰基四羟氢脱落酶的基因中。
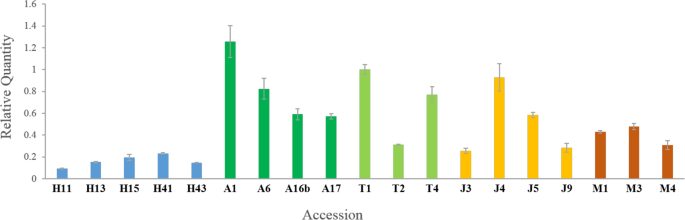
测试是否玛利亚姆本研究设计引物对TRIDC2AG023940基因和/或其B基因组同源物进行RT-qPCR分析。将每个加入的相对表达水平归一化到任意选择的加入的相对表达水平(T1),以便在样本之间进行比较。TRIDC2AG023940和/或其同源物来自B亚基因组的相对表达量在野生二粒小麦群体内和群体间存在差异(图2)。5.).
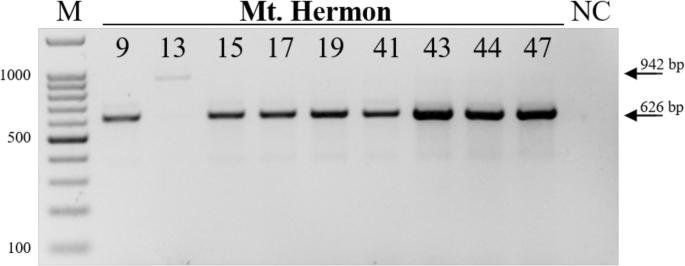
Amiad、Tabgha和Jaba群体间该基因表达水平差异超过2倍,而Amasa山群体内的差异不明显。黑门山群体的表达水平显著低于阿米德群体(单因素方差分析,F= 4.3655,P.-value = 0.0169,图5.).黑门山居群不同种质间差异约为2倍玛利亚姆在TridC2AG023940基因内插入显示表达式最低水平。例如,加入H11显示了在Mt中的TridC2AG023940基因的表达水平的降低。赫蒙人口。这些结果可能表明了对该基因表达的复杂性,并且不允许验证或裁定出来的可能效果玛利亚姆插入文字数量。然而,在这种情况下,插入存在于外显子中,因此可能会干扰剪接或保留在成熟RNA中。为了测试这些可能性,引物与预测的cDNA序列(港玛利亚姆设计用于RT-PCR分析。使用来自Mt的9种附加的cDNA进行表达分析。Hermon人口作为模板。在所有检测到外显子6中没有插入的载体中(参见图11),可以看到对应于预期的626bp尺寸的拼接产品的单个清晰条带(图。6.).在H13登录中,外显子6的插入在DNA水平上存在(图。1),有一个更高的频带,对应于在外显子6中插入的拼接产品的预期942bp尺寸(图。6.).为了验证,我们从琼脂糖凝胶中提取了H13接入的这条较高的条带,并进行了纯化和测序,发现该转录本确实含有全长玛利亚姆插入。该实验表明,外显子6中的插入改变了以基因组特异性方式从基因产生的成熟mRNA序列。
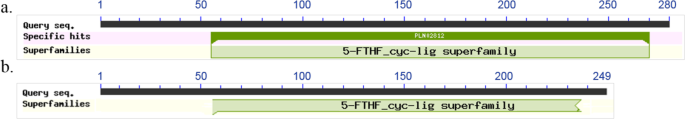
根据该基因的编码序列(CDS)预测EnsemblPlants在数据库中,CDS开始于外显子1,结束于外显子7。因此,如果转录本被翻译,外显子6中的新插入将干扰翻译。通过对新插入的cDNA的ORF分析,在新插入的307个核苷酸中,有一个位于41位的终止密码子,因此插入第6外显子的等位基因产生的蛋白产物具有缩短和改变的c端。用Blastp搜索TRIDC2AG023940基因的翻译后的cDNA,没有插入(图)。7.a)和插入外显子6(图。7.b)在NCBI数据库中显示插入干扰环连接酶结构域c端。
讨论
在这项研究中,我们介绍了一种在野生emmer小麦中首先发现的新型微型TE样序列。这个序列最初发现在Mt的两次换乘中。Hermon狂野埃默米麦的人口被称为玛利亚姆,是低拷贝数重复序列。该片段在不同的小麦物种中以相对较少的数量发现,特别是单个分子物种(如)t . urartu在狂野emmer和面包小麦的12个插入左右。第一次插入玛利亚姆发现,当测序时,类似于TE插入,它产生了TSD。但在重复序列和te数据库中均未发现相似序列。此外,其体积小(307 bp)玛利亚姆表示它是非自主的,并且可能被分类为微型反转重复转换元素(螨虫)。
的顺序玛利亚姆没有与已知的TES显示任何功能相似性。它没有允许我们将该元素分配给TIR DNA元素超级家庭的TIR。此外,它不具有诸如POL-III促进剂或多尾的任何特征特征。唯一的特征玛利亚姆元素与已知的TE超家族共同,是序列重复的长度在〜50%的情况下的完整插入。目标网站重复(TSD)长度为9bp是特征腐败师TIR DNA元素。已经提出了这一点腐败师元素可能会引起螨虫衍生物,产生9个BP-long tsds以及一些问题腐败师元素可能非常短,甚至缺失[6.].不过,所有腐败师-衍生的螨类,最近在草类中注释和特征,具有tir(见例子,[34那35])。因此,当玛利亚姆可能是一个腐败师-衍生的螨,单凭其TSD大小很难对其进行分类。
为了进一步研究玛利亚姆为了研究它在不同小麦品种中的插入模式,玛利亚姆从5个公开的小麦物种基因组草图中检索到插入,发现33个插入,其中一些是常见的小麦物种(附加文件1:表S1)。生物信息学分析表明玛利亚姆是小麦组独有的,在二倍体中以低拷贝数存在t . urartu(一个基因组)和Ae。Tauschii.(D基因组)物种,因此很可能存在于多倍体小麦的B祖祖中。检索到玛利亚姆插入t . turgidumSSP。dicoccoides在5个不同群体和8份面包小麦材料中进行了验证,发现不同群体材料之间存在较高的多态性水平,某些情况下存在群体特异性(表1).
一些玛利亚姆在大多数情况下,在遗传区域内发现插入玛利亚姆位于UTR或内含子内部。A2-MH插入(第一个玛利亚姆发现在emmer中发现的插入)被发现位于用于5-甲酰基四氢溶胶环连接酶线粒体酶的基因的外显子6内。特异性PCR分析显示这种插入仅在Mt的两种含义中发现。Hermon百姓野生小麦,而Q-Realtime PCR分析表明,所有MT中该基因的表达降低。与其他野生艾梅麦群体相比,赫蒙特的探查了。
结论
一个新的移动元件的发现,一个不具有任何已知TEs的内部特征的序列,表明小麦基因组可能具有其他难以检测的移动序列,具有更高的拷贝数和可能显著的功能影响。这项研究强调了最新的基因组组合对小麦研究的巨大贡献,同时揭示了野生二粒小麦群体以及不同小麦多倍体中潜在的有趣的遗传变异。此外,尽管迄今收集和分析的数据不允许对新发现的移动元素进行分类,但它为小麦基因组中移动DNA的多样性提供了额外的见解。
方法
基因组和转录组数据
我们使用了五份基因组草图小麦属植物和山羊草属物种:(1)t . urartu,基因组的供体(https://www.ncbi.nlm.nih.gov/assembly/GCA_003073215.1)[36].(2)Ae。Tauschii.,D基因组的供体(https://www.ncbi.nlm.nih.gov/assembly/GCA_000347335.2)[37].(3)t . turgidumSSP。Diccococides,野生二粒小麦,基因组AB (WEWseq:http://wewseq.wix.com/consortium.)[4.].(4)t . aestivum面包小麦,基因组ABD。(https://plants.ensembl.org/Triticum_aestivum/Info/Index)[5.那30.].(5)t . turgidumSSP。杜伦姆,硬粒小麦,基因组AB [38].
RNA-SEQ数据库
更新的公开可用的RNA-seq数据库T. Aestivum.和t . turgidumSSP。dicoccoides中发现的Ensemblplants用于本研究[5.那30.].该文库包括cDNA、CDS和ncRNA序列(https://plants.ensembl.org/Triticum_aestivum/Info/Annotation/那http://plants.ensembl.org/Triticum_dicoccoides/Info/Annotation/).
检索玛利亚姆插入
我们已经使用了第一个玛利亚姆序列识别(A2-MH)作为查询在螨分析试剂盒(MAK)软件(http://labs.csb.utorto.ca/yang/mak/),(39那40].MAK是一个基于同源性的软件,这意味着它使用共识序列作为查询,使用全局对齐的BLASTN算法。我们用的e值是1e−3以及20个核苷酸的末端错配耐受性。此外,检索两侧序列(每端1000 bp),以确定插入位点。
系统发育分析
Primer6软件版本6.1.6 [41[根据插入多态性,用于构建系统发育树聚类野生emmer和面包小麦涂抹玛利亚姆基于ss-PCR。Primer6软件采用Bray-Curtis相似性对各矩阵进行分层聚类分析,并对各节点进行SIMPROF相似性检验,评估系统发育树的统计显著性。SIMPROF通过随机化每个变量的值并重新计算概要文件来计算平均概要文件。pi统计量被计算为相似矩阵的实际相似轮廓与平均轮廓的偏差。这与进一步随机生成的剖面的偏差进行比较,以检验显著性。
统计分析
采用Statistica statsoft进行单因素方差分析。以相对数量(q-real - time- pcr对TRIDC2AG023940基因的相对表达量)为因变量,以种群数量为分类变量。采用Post hoc和Tukey检验分析群体间的具体差异。
植物材料和DNA和RNA提取
野生艾梅麦的一系列(t . turgidumSSP。dicoccoides)在这项研究中使用了以色列五个地理上孤立的地点的种群;黑门山,亚米得山,他迦山,雅巴山,亚玛撒山。以前的刊物使用了同一套资料[42].此外,还有8种T. Aestivum.被使用(附加文件2:表S2)。种子由Sergei Volis博士(Hermon山、Amiad山和Amasa山种群)和海法大学的Eviatar Nevo教授(Tabgha和Jaba种群)好心提供。面包小麦由美国农业部(USDA)提供。所有野生二粒种子均由以色列种子中心提供。海法大学进化研究所的野生谷物基因库(WCGB)中有Tabgha和Jaba以色列种群的种子。以色列黑门山、阿米德山和阿马萨山种群的种子可在特拉维夫大学谷物改良研究所的利伯曼种质库中找到。每个种群在普通花园条件下的温室中种植10-20份种子。大约在萌发后4周收集叶材料,使用DNeasy植物迷你试剂盒(Qiagen)提取DNA,使用TriReagent (Sigma)提取总RNA。
特定站点聚合酶链反应
使用Primer3软件设计特异性PCR引物(http://bioinfo.ut.ee/primer3-0.4.0/primer3/)测试特定的插入站点。用13.2μl超纯水(蜂胶),2μl109AQDNA聚合酶缓冲液C(EURX),0.8μl,25mM MgCl,制备PCR扩增2Taq DNA聚合酶(5 U/μl, EURx) 0.2 μl,每个位点特异性引物各1 μl (50 ng/μl),基因组DNA各1 μl (50 ng/μl)。PCR反应条件为:94℃孵育3min,循环(94℃1 min, 57℃1 min, 72℃1 min) 30次,72℃3 min。PCR产物在1.5%琼脂糖凝胶上等量10 μl,用溴化乙锭(Amresco)观察。预期产品大小由DNA大小标准(100 bp阶梯,SMOBIO)确定。
长串互补脱氧核糖核酸的合成
采用5xAll-In-One RT MasterMix (abm), 20 μl反应合成用于基因表达分析的cDNA。每个反应包含4 μl MasterMix和多达2 μg RNA溶解在16 μl超纯水(HyLabs)。42℃孵育15 min。每个cDNA样品的纯度通过PCR检测,使用位点特异性引物互补的两个外显子肌动蛋白基因,产生不同的cDNA和基因组DNA扩增产物。未检测到基因组DNA污染。
实时定量RT-PCR分析基因表达
采用实时荧光定量PCR检测TRIDC2AG023940的表达水平。利用Primer Express v2.0软件设计了针对该基因cDNA序列的引物。其中一个引物与外显子-外显子连接互补,以降低反应对RNA样本可能的DNA污染的敏感性。正向引物被设计成跨越外显子1和2之间的连接,以确保反应对DNA污染不敏感。需要注意的是,没有一对基因组特异性引物可以用于RT-qPCR。对5个群体的19份dna样本进行实时RT-qPCR分析。qPCR实验使用7500 Fast Real-Time PCR系统进行,使用7500 version 2.0.5软件(Applied Biosystems)进行分析。每个反应包含7.5 μl KAPA SYBR FAST qPCR Master Mix, 0.3 μl ROX Low 509 (KAPA Biosystems), 1 μl正向引物和1 μl反向引物(10 μM), 0.2 μl ddH2o和5μl模板cDNA。
通过使用模板cDNA混合物的串联稀释液与每个引物对的实时QPCR测试引物效率,以产生标准曲线。效率通过[(10-1 / y) - 1]×100%计算,其中Y是标准曲线斜率。基于标准曲线,在基于实时QPCR扩增的基因表达分析中,使用50倍的cDNA稀释CDNA。为了验证产品特异性,产生熔化曲线并显示每个引物对的单个特定产品。如上所述使用比较2进行实时QPCR分析——ΔΔCT方法,有肌动蛋白用作内源控制的表达水平和作为参考样品的一个随机选择的加入。使用比较2评估靶基因的相对表达水平——ΔΔCT方法,如前所述。
数据和材料的可用性
本研究中生成的所有数据都包含在纸张中以及支持信息文件中。
缩写
- 螨:
-
微型反重复转座元件
- TE:
-
转座的元素
- TIR:
-
终端反向重复
- TSD中:
-
目标站点重复
参考文献
- 1。
kidwell mg。可转座元素和真核生物中基因组大小的演变。genetica。2002; 115(1):49-63。
- 2.
Charles M, Belcram H, Just J, Huneau C, Viollet A, Couloux A, Segurens B, Carter M, Huteau V, Coriton O.转座因子在小麦B和A基因组进化中的动态和差异增殖。遗传学。2008;180(2):1071 - 86。
- 3.
Clavijo BJ,Venturini L,Schudoma C,Accinelli GG,Kaithakottil G,Wright J,Borrill P,Kettleborough G,Hogens D,Chapman H.一种改进的组装和注释对Allohexaploid小麦基因组的改进和注释鉴定了农艺基因的完整系列,并提供了基因组基因的完整系列用于染色体易位。Genome Res。2017; 27(5):885-96。
- 4.
Avni R, Nave M, Barad O, Baruch K, Twardziok SO, Gundlach H, Hale I, Mascher M, Spannagl M, Wiebe K.野生二粒基因组结构和多样性阐明了小麦的进化和驯化。科学。2017;357(6346):93 - 7。
- 5.
基于染色体的面包小麦(Triticum aestivum)基因组草图序列。科学。2014;345(6194):1251788。
- 6.
柳条T,Sabot F,Hua-Van A,Bennetzen JL,Capy P,Chalhoub B,Flavell A,Leroy P,Morgante M,Panaud O.一种统一的真核转换元素的分类系统。NAT Rev Genet。2007; 8(12):973-82。
- 7.
LISCH D.植物演进的转座子有多重要?NAT Rev Genet。2012; 14(1):49-61。
- 8.
库马尔,贝内特岑JL。植物反转位子活动。acta photonica sinica . 1999;33(1): 479-532。
- 9.
压力和转座因子:共同进化还是有用的寄生虫?遗传。2000;85(2):101 - 6。
- 10。
多倍体:轮回形成与基因组进化。生态学报。1999;14(9):348-52。
- 11.
植物ltr反转座子与螨:转座的控制及其对植物基因和基因组进化的影响。基因。2003;311:1-11。
- 12.
Mansour A.基因组回析吊子的表观遗传激活。J细胞mol Biol。2007; 6(2):99-107。
- 13.
转座元件与基因组的表观遗传调控。(1)中国科学(d辑:地球科学)2007;
- 14.
赵丹丹,陈建平,蒋宁。植物基因组的组成:转座元件与基因之间的消失线。生物物理学报。2016;1859(2):366-80。
- 15.
Rebollo R,罗马语MT, Mager DL。转座元件:宿主基因调控序列的丰富和自然来源。Annu Rev Genet. 2012; 46:21-42。
- 16.
Wessler SR。真核转换元素:教授旧基因组新技巧。隐含基因组。2006.第138-65页。
- 17.
Schmitz J,Brosius J.转置元素的出口:进化的挑战和机遇。生物chimie。2011; 93(11):1928-34。
- 18.
关键词:小麦,反转录转座子,Au - sin,基因组,亚基因组,染色体植物学报2018;37(2):193-208。
- 19.
Hawkins JS, Proulx SR, Rapp RA, Wendel JF。植物中通过反转录转座子增殖来平衡基因组扩张的快速DNA损失。中国科学(d辑:地球科学)2009;106(42):17811-6。
- 20.
Feldman M,Levy A. AllopolyPloidy-A在小麦基因组的演变中的成型力。CytoOgenet基因组Res。2005; 109(1-3):250-8。
- 21。
陈志强,陈志强,陈志强,等。小麦A、B、D基因组起源的研究进展[j]。分子系统学进展。2006;39(1):70-82。
- 22。
Aaronsohn A.发现野麦小麦。城市俱乐部公牛芝加哥。1913; 6:167-73。
- 23。
小麦双球菌对小麦赤霉病的抗性研究。Euphytica。1984;33(1):41-7。
- 24.
陈志强,王志强,王志强,等。小麦(Triticum dicoccoides)和大麦(Hordeum spontaneum)野生祖先耐盐性的遗传资源。植物品种。1993;110(4):338 - 41。
- 25.
Krugman T,Korol A,Nevo E,Snape JW,Levy O,Rubin B.氯脲抗性基因(SU1)在栽培小麦(Triticum Aestivum)和野外(Triticum dicoccoides)中的比较RFLP测绘。Al Appl Genet。1997; 94(1):46-51。
- 26.
Krugman T,ChaguéV,Peleg Z,Balzergue S,Just J,Korol AB,Nevo E,Saranga Y,Chalhoub B,Fahima T.多级调节和信号传导过程,与适应野生EMMER小麦的终端干旱相关。Funct综合基因组学。2010; 10(2):167-86。
- 27.
王志强,王志强,王志强,等。小麦遗传多样性及其在小麦育种中的应用。中国科学(d辑:地球科学)1989;77(3):421-55。
- 28.
宋敏,张玉华,张玉华。二粒小麦精细尺度空间遗传结构及其居群范围定位的作用。另一个星球杂志。2014;41(1):166 - 73。
- 29.
关键词:野生二粒小麦,转座因子,插入模式,等位基因变异小麦属植物turgidum ssp。dicoccoides).植物学报。2017;17(1):175。
- 30.
Appels R, Eversole K, Feuillet C, Keller B, Rogers J, Stein N, Pozniak CJ, Choulet F, Distelfeld A, Poland J.使用完全注释的参考基因组改变小麦研究和育种的限制。科学。2018;361 (6403):eaar7191。
- 31.
Wicker T, Matthews DE, Keller B. TREP:麦类重复元素数据库。植物科学进展,2002;7(12):561-2。
- 32.
宝W,Kojima KK,Kohany O. Repbase更新,真核生物基因组中重复元素的数据库。暴毒DNA。2015; 6(1):11。
- 33。
Crooks Ge,Hon G,Chandonia J-M,Brenner SE。WebLogo:序列徽标生成器。Genome Res。2004; 14(6):1188-90。
- 34。
杨堂,哈特TC。MDM-1和MDM-2:水稻中的两个致毒剂衍生的螨虫族。J Mol Evol。2003; 56(3):255-64。
- 35。
Mo Y- j, Kim K-Y, Shin W-C, Lee G-M, Ko J-C, Nam J-K, Kim B-K, Ko J-K, Yu Y, Yang T-J。水稻基因组中突变体样螨家族Imcrop的特征分析。基因组的基因。2012;34(2):189 - 98。
- 36.
ling h-q,ma b,shi x,liu h,dong l,sun h,cay,gao q,zhengs,li y。小麦祖细胞祖细胞祖细胞祖的基因组序列。自然。2018; 557(7705):424。
- 37.
Luo M-C, Gu yicq, Puiu D, Wang H, Twardziok SO, Deal KR, Huo N, Zhu T, Wang L, Wang Y.小麦D基因组Aegilops tauschii祖先的基因组序列。大自然。2017;551:498 - 502。
- 38.
Maccaferri M, Harris NS, Twardziok SO, Pasam RK, Gundlach H, Spannagl M, Ormanbekova D, Lux T, Prade VM, Milner SG。硬粒小麦基因组突出了过去的驯化特征和未来的改良目标。Nat麝猫。2019;51:885 - 95。
- 39.
王志强,王志强,王志强,等。真核生物转座元件的生物信息学和基因组学分析。Chromosom杂志2011;19(6):787 - 808。
- 40.
杨堂,哈特TC。MAK,一种用于自动螨分析的计算工具套件。核酸RES。2003; 31(13):3659-65。
- 41.
Clarke Kr。非参数多变量分析社区结构的变化。AUST J ECOL。1993年; 18(1):117-43。
- 42.
关键词:野生二粒小麦,DNA甲基化,表观遗传变异,遗传多样性dicoccoides)的人口。BMC Plant Biol. 2015;15:200。
确认
感谢多伦多大学杨国君博士提供MAK的单机版。我们也感谢美国农业部提供了面包小麦。
资金
由以色列科学基金会资助(批准# 322/15)https://www.isf.org.il/#/.
资金用于购买实验中使用的材料。资金机构在实验设计和数据分析中没有任何作用。
作者信息
隶属关系
贡献
KD进行了硅片分析和湿凳实验,参加了准备原始稿件草案。DKF经过验证的生物信息学数据和PCR分析,参加了准备原稿草稿。KK进行了研究设计,编写,修订,并提交了稿件的最终版本。所有作者都已经阅读并赞成最终的手稿。
通讯作者
道德声明
伦理批准和同意参与
不适用。
同意出版物
不适用。
利益争夺
两位作者宣称他们没有相互竞争的利益。
额外的信息
出版商的注意
Springer Nature在发表地图和机构附属机构中的司法管辖权索赔方面仍然是中立的。
补充信息
附加文件1:表S1。
从五种小麦中检索到完整的玛丽亚姆插入。
附加文件2:表S2。
T. Aestivum.本研究使用的材料及其地理来源。
附加文件3:表S3。
位点特异性引物侧翼插入玛利亚姆.
附加文件4:图S1。
序列标志代表目标站点的偏好玛利亚姆.图S2。特异性PCR分析玛利亚姆插入。
权利和权限
开放获取本文遵循知识共享署名4.0国际许可协议(http://creativecommons.org/licenses/by/4.0/)如果您向原始作者和源给出适当的信用,则允许在任何介质中进行不受限制的使用,分发和再现,提供指向Creative Commons许可证的链接,并指示是否进行了更改。Creative Commons公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于本条提供的数据,除非另有说明。
关于这篇文章
引用这篇文章
在小麦中编码5-甲酰基四氢叶酸的基因编码序列中发现了一种新的微型转座子样元素。BMC植物杂志19,461(2019)。https://doi.org/10.1186/s12870-019-2034-1
收到了:
公认:
发表:
关键字
- 微型转座的元素
- 小麦属植物
- 山羊草属
- 5-formyltetrahydrofolate
- 基因组进化
- 多倍性
- 小麦